转谷氨酰胺酶(EC 2.3.2.13 TGase)催化蛋白质肽链中谷氨酰胺残基和不同酰基受体之间形成ε-(γ-谷氨酰胺)异肽,导致蛋白质交联或被小分子修饰[1]。微生物TGase最初从茂原链霉菌(Streptomyces mobaraensis)中分离得到。与动植物来源的酶相比,S.mobaraensis TGase具有不依赖Ca2+和生产成本低的优点[1],是商品化TGase的主要来源。迄今为止,S.mobaraensis TGase被广泛用于蛋白质类食品的改良(如肉类,乳制品和大豆制品的质地改善)[2]。此外,该酶在抗体偶联药物[3],组织工程[4]和蛋白质修饰[5]等方面也具有良好的应用前景。然而,S.mobaraensis发酵产生的野生型TGase在50 ℃和60 ℃下的半衰期分别仅为41.8 min和2 min,不能满足众多高温条件下的应用。例如,牛肉凝胶通常在60 ℃下生产[6],而豆腐加工的交联反应需要在50 ℃下进行[7-8]。因此,高效生产耐热TGase对其工业应用具有重要意义。
目前,研究者已经使用随机突变或半理性策略来改善S.mobaraensis TGase的热稳定性。其中,随机突变[9]和组合突变[10]可得到TGase耐热突变体TGm1(S2P-S23V-Y24N-S199A-K294L),其60 ℃半衰期达到24.3 min,是野生型TGase的12.2倍[11]。本课题组此前通过脯氨酸扫描和蛋白质表面静电荷设计构建了热稳定突变体TGm2(S2P-S23V-Y24N-E28T-S199A-A265P-A287P-K294L)和TGm3(S2P-S23V-Y24N-E28T-N96E-S144E-N163D-R183E-S199A-R208E-A265P-A287P-K294L-K325E)[12]。结果显示,TGm2和TGm3在60 ℃下的半衰期分别达到66.87 min和122.91 min[12]。然而,S.mobaraensis TGase热稳定突变体仅在大肠杆菌中表达,其安全性和发酵水平仍有待提高。
除大肠杆菌外,S.mobaraensis TGase已在不同宿主中表达,如变铅青链霉菌(Streptomyces lividans)[13]、乳酸乳球菌(Lactococcus lactis)[14]、谷氨酸棒杆菌(Corynebacterium glutamicum)[15-16]、枯草芽孢杆菌(Bacillus subtilis)[17]、毕赤酵母(Pichia pastoris)[18]和解脂耶氏酵母(Yarrowia lipolytica)[19]。在S.mobaraensis中,TGase以前体形式(pro-TGase)合成,被胞外蛋白酶切除其N-端酶原区后转化为活性酶[20]。研究显示,N-端酶原区调控TGase的折叠[21]并通过抑制其交联活性来降低对宿主的毒性[22]。因此,通常通过共表达pro-TGase及其活化蛋白酶才能实现异源宿主中活性S.mobaraensis TGase的合成[15]。尽管不断有新的活性表策略提出,但活性TGase在异源宿主中产量较低[23]或不能分泌[24]。前期研究中,本课题组通过突变育种得到能高泌TGase的S.mobaraensis突变株smY2019(20 U/mL),并通过增加基因拷贝数使S.mobaraensis的TGase产量提高至40 U/mL[25]。上述结果表明,S.mobaraensis仍然是最具竞争力的TGase产生菌株之一。
本研究中,通过双交换同源重组将TGase耐热突变体TGm3基因整合至smY2019-Δtg(缺失TGase基因的smY2019衍生菌株)[25]的TGase基因位点,构建了可分泌表达活性TGase的重组S.mobaraensis,并对其分泌TGm3进行了纯化及酶学性质分析。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
本研究所使用的菌株和质粒均来自于实验室保藏。smY2019为S.mobaraensis DSM40587突变株,smY2019-Δtg为缺失TGase基因的smY2019衍生菌株[25]。E.coli JM109用于质粒构建。E.coli ET12567/pUZ8002用于链霉菌接合转移。pUC-TGm3为pUC19衍生质质粒,编码TGm3基因表达框。质粒pJTU1278用于链霉菌双交换同源重组。
1.1.2 试剂
高保真DNA聚合酶(Phanta Max Master Mix)、一步克隆连接试剂盒One Step Cloning Kit(ClonExpressTM Ⅱ),南京诺唯赞生物科技股份有限公司;蛋白预制胶、蛋白质Marker、MES蛋白电泳缓冲液,赛默飞世尔科技(中国)有限公司;硫链丝菌素、安普霉素、氨苄青霉素、质粒抽提试剂盒,生工生物工程(上海)股份有限公司;酵母粉、胰蛋白胨等,OXIOD(英国);Bradford蛋白检测试剂盒,碧云天生物公司;CBZ-Gln-Gly、L-谷氨酸-γ-单羟胺酸、盐酸羟胺,默克生命科学(上海)有限公司;其他常规试剂及药品为国产或进口分装。
1.1.3 培养基
GYM琼脂培养基(g/L):葡萄糖10,酵母粉4,麦芽粉3,琼脂粉20。
MS琼脂培养基(g/L):甘露醇20,麦芽提取物2.5,大豆粉20,琼脂20;50 mmol/L MgCl。
种子培养基(g/L):甘油20, 酵母粉5, 胰蛋白胨20, K2HPO4 4, MgSO4 2, pH 7.2。
发酵培养基(g/L):甘油20, 鱼粉蛋白胨25,酵母粉5,玉米浆粉5.5,(NH4)2SO4 5.5, K2HPO4 2, MgSO4 2, pH 7.2~7.4。
1.2 实验方法
1.2.1 双交换同源重组质粒的构建
使用引物TGm3-FF/TGm3-FR和TGm3-RF/TGm3-RR分别从smY2019-Δtg基因组扩增得到TGase基因上(1 000 bp)和下(1 000 bp)游同源臂基因序列。以引物对TGm3-F/TGm3-R从pUC-TGm3 扩增得到TGm3表达框。基于一步克隆试剂盒,将上述3个基因片段连接至pJTU1278载体的EcoR Ⅰ和BamH Ⅰ位点之间并转化E.coli JM109,构建得到双交换同源重组质粒pJTU1278-TGm3。引物序列如表1所示。
表1 本研究引物序列
Table 1 Primer sequences

名称核苷酸序列(5′-3′)TGm3-FFGATAAGCTTGATATCGAATTGCGTCGTCGACCGTGACGACGAACTCTGm3-FRCCAGTTAACTCCCCGGCAACACCACTGm3-RFGAGCATCCGGCTCCCCTCCCCACCTGm3-RRCGCTCTAGAACTAGTGGATCGACAAACGAACGCATGCTTCCGAACGCTGm3-FGTGGTGTTGCCGGGGAGTTAACTGGTGm3-RGGTGGGGAGGGGAGCCGGATGCTCVer-FGGGGTTACGAGGACCTGGCGGACVer-RTTCATCACCGCCAGCCAGCAGGCG
1.2.2 S.mobaraensis转化及双交换同源重组菌株筛选
将pJTU1278-TGm3转化至非甲基化的E.coli ET12567/pUZ8002,并在LB液体培养基(包含25 μg/mL氯霉素,50 μg/mL卡那霉素和100 μg/mL氨苄西林)中37 ℃培养2 h。收集E.coli细胞并和S.mobaraensis孢子悬液混合,于50 ℃水浴预热10 min。将预热后的悬液涂布在MS琼脂培养基上,于30 ℃培养16 h。用终质量浓度为10 μg/mL的硫链丝菌素溶液覆盖琼脂培养基,30 ℃培养48 h后长出转化子。为促进双交换同源重组,将转化子转移至不含硫链丝菌素抗性的GYM平板上进行松弛培养。松弛培养后的菌落分别转移至含有硫链丝菌素和不含硫链丝菌素抗性GYM平板培养,利用引物对Ver-F/Ver-R(表1)对无硫链丝菌素抗性的菌落进行PCR及测序分析,筛选得到双交换同源重组整合TGm3表达框的S.mobaraensis菌株smY2019-TGm3。
1.2.3 重组S.mobaraensis的发酵培养
S.mobaraensis在GYM琼脂平板30 ℃培养4~5 d,取孢子和菌丝接种至含有25 mL种子培养基的摇瓶中,于30 ℃和220 r/min摇床培养24 h。取2 mL的种子培养物转移到含有25 mL发酵培养基的摇瓶中,于30 ℃和220 r/min条件下发酵96 h。
1.2.4 野生型TGase和TGm3的纯化
利用PrePack SP Purose 6 Fast Flow阳离子交换柱[26] 分别从smY2019和smY2019-TGm3发酵上清液中纯化野生型TGase和TGm3。向发酵上清液中加入-20 ℃预冷的乙醇至浓度达到70%(体积分数),于4 ℃静置30 min后,离心收集蛋白质沉淀。用乙酸-乙酸钠缓(50 mmol/L, pH 5.5)溶解蛋白质沉淀,过滤后制得纯化前的粗酶液。将粗酶液加入到经乙酸-乙酸钠缓冲液(50 mmol/L, pH 5.5)平衡的阳离子交换柱中,用含150 mmol/L NaCl的乙酸-乙酸钠缓冲液洗脱。收集具有TGase活性的洗脱液,用Tris-HCl缓冲液(50 mmol/L, pH 8.0)透析得到纯化后的酶液。
1.2.5 TGase酶学性质测定
60 ℃半衰期:用Tris-HCl缓冲液(50 mmol/L,pH 8.0)将纯化后的酶稀释至0.4~0.6 mg/mL,于60 ℃下孵育120 min。在特定的时间间隔采集样品测定TGase活力,如公式(1)所示:
各样品残余活力![]()
(1)
基于上述残余酶活力,使用Origin 2019的ExpDec1函数拟合得热失活曲线方程,并计算得到酶的60 ℃半衰期。
最适反应温度:用Tris-HCl缓冲液(50 mmol/L,pH 8.0)将纯化后的酶稀释至0.5 mg/mL,在37~70 ℃下测定比酶活力,确定最适反应温度。
最适反应pH及pH稳定性:发酵上清液经乙醇沉淀和冷冻干燥制备酶粉,并溶于Tris-HCl缓冲液(pH 3.5~8,50 mmol/L)。将不同pH的酶溶液与对应pH的底物混合于37 ℃进行酶反应10 min,确定酶的最适反应pH。将不同pH的酶溶液于37 ℃孵育1 h后,再于37 ℃和pH 6.0测定其催化活性,确定酶的pH稳定性。
1.2.6 S.mobaraensis生物量测定
采用干重法测定S.mobaraensis生物量。每隔一定时间,收集S.mobaraensis发酵液1~1.5 mL EP管中,12 000 r/min、10 min进行离心,弃去上清液,留下菌体。EP管开盖放至65 ℃烘箱中干燥直到恒重,取出称量。计算菌体在不同时间点的质量,绘制细胞干重曲线。实验采用3个样品重复测定。
1.2.7 DNA及蛋白质分析
取0.3 g琼脂糖溶于30 mL 1×TAE溶液溶解制备DNA电泳胶,于120 V条件下电泳15 min。使用Bradford Protein Assay Kit试剂盒测定蛋白浓度。用10%凝胶SDS-PAGE凝胶分离蛋白,用考马斯亮蓝R250染色。SDS-PAGE分析在120 V下进行60 min。用PageRuler Prestained Protein Ladder (10~180 kDa)作为蛋白质分子质量标准。
1.2.8 TGase酶活力测定
以N-CBZ-Gln-Gly和羟胺为底物,采用比色法测定TGase活力[27]。将60 μL TGase溶液与150 μL底物溶液(30 mmol/L N-CBZ-Gln-Gly,100 mmol/L羟胺,10 mmol/L谷胱甘肽,200 mmol/L Tris-HCl,pH 6.0)混合于37 ℃孵育10 min后,加入60 μL终止剂(2% FeCl3·6H2O、3 mol/L HCl,4%三氯乙酸)终止酶反应。根据反应液的吸光度(A525)计算形成的反应产物L-谷氨酸γ-单羟肟酸酯的浓度。1个TGase活力单位定义为在37 ℃条件下每分钟催化CBZ-Glu-Gly生成1 μmol的羟肟酸所需酶的量。
1.2.9 生物信息学分析
采用ColabFold[28]构建野生型S.mobaraensis TGase及TGm3的结构模型,并使用Rosetta_Relax[29]进行侧链构象优化。使用Discovery Studio-2019(Dassault, Omaha, 美国)的Non-bond Interactions Monitor模块统计酶分子的非共价相互作用。使用GROMACS-2020[30]进行酶分子动力学分析。主要分析条件如下:力场为FF14 sb;将酶的模拟结构置于填充SPC/E水模型的立方盒子内,酶与盒子边界距离为15 Å;使用Na+和Cl- 平衡体系电荷,并采用最陡下坡法对系统进行能量最小化;等容等温系综和等温等体积系综各进行200 ps后解除限制。在330 K下进行分子动力学模拟40 ns,计算模拟过程中均方根偏差(root mean square deviation,RMSD)和均方根波动(root mean square fluctuation,RMSF),并选择最终平衡构象。
2 结果与分析
2.1 耐热TGase生产菌的构建
前期研究中,课题组通过理性设计对S.mobaraensis TGase耐热突变体TGm1(S2P-S23Y-S24 N-H289Y-K294L)进行了系统的改造,得到突变体TGm3(S2P-S23V-Y24 N-E28T-N96E-S144E-N163D-R183E-S199A-R208E-A265P-A287P-K294L-K325E)[12]。通过E.coli制备的上述突变体样品中,TGm3的60 ℃半衰期(122.91 min)分别较TGm1和野生型TGase提高2.2倍和67.2倍。为实现TGm3在S.mobaraensis中的高效表达,合成了TGm3的表达框(1 745 bp),包含了S.mobaraensis TGase天然启动子、酶原区、终止子及TGm3编码序列(基于链霉菌密码子偏好性优化)(图1)。

图1 TGm3的表达框
Fig.1 The expression cassette of TGm3
注:左边数字:苷酸序列;波浪线:启动子区及转录终止区;方框:核糖体结合位点;双下划线:信号肽;虚线:酶原区;单下划线:TGm3。
基于一步克隆策略将TGm3表达框与S.mobaraensis基因组TGase上(1 000 bp)、下(1 000 bp)游序列融合并连接至载体pJTU1278的EcoR Ⅰ和BamH Ⅰ位点之间,经PCR验证获得重组质粒pJTU1278-TGm3(结果未显示)。smY2019是S.mobaraensis DSM40587高产诱变菌[25],而smY2019-Δtg则是后者缺失TGase基因的衍生菌株。为实现TGm3在S.mobaraensis中的高效表达,将该重组质粒转化smY2019-Δtg [25],筛选在TGase基因位点发生双交换同源重组的转化子(图2-a)。经验证引物对Ver-F/Ver-R(图2-a)菌落PCR扩增,smY2019-TGm3得到了约4 000 bp的DNA条带,而对照菌smY2019-Δtg仅为2 000 bp(图2-b)。PCR产物测序进一步确认了smY2019-TGm3 基因组通过双交换成功整合了TGm3表达框。
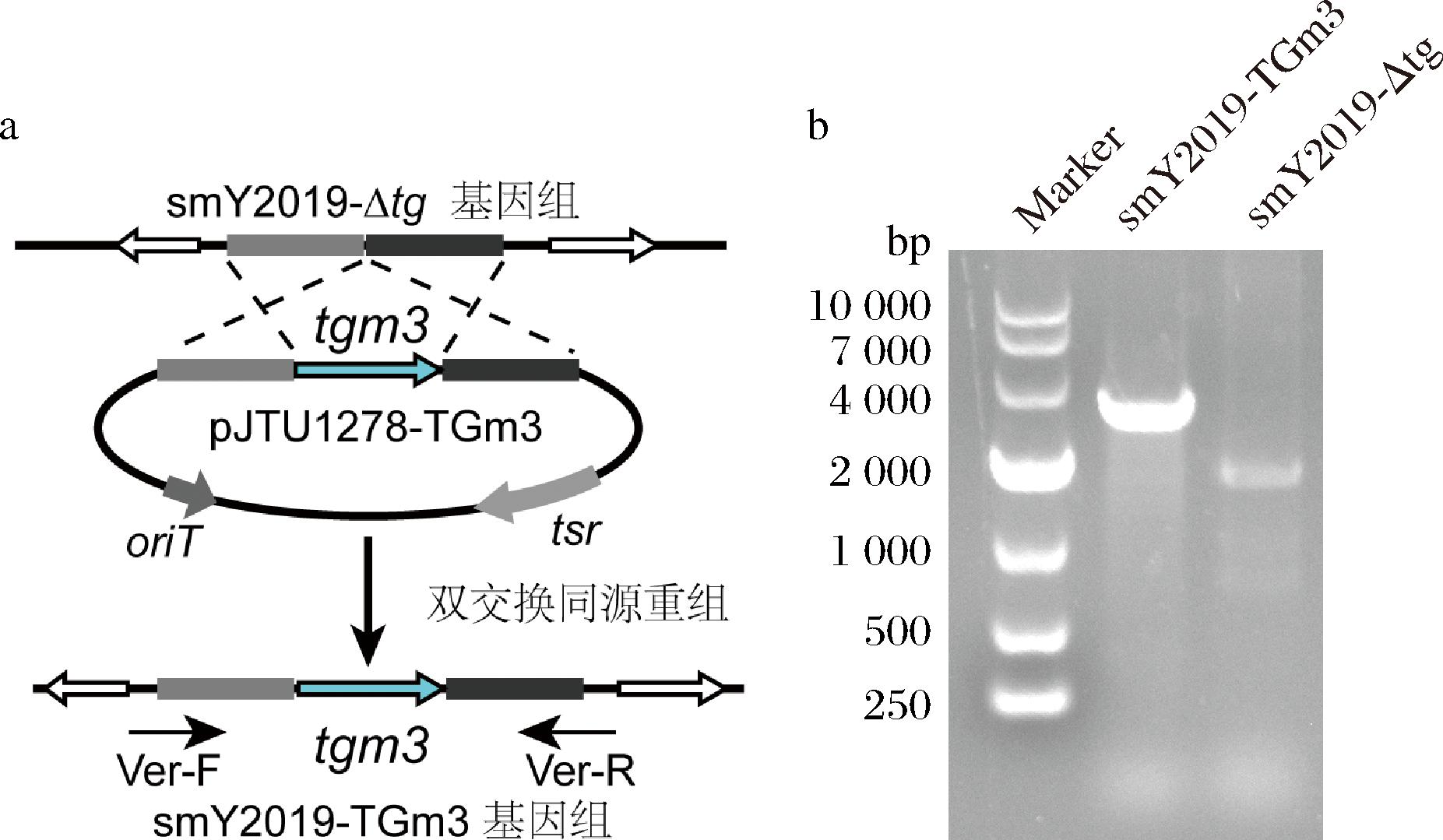
a-单交换同源重组流程图;b-PCR验证双交换同源重组菌基因组(引物对为Ver-F/Ver-R)
图2 重组smY2019-TGm3的构建
Fig.2 Construction of smY2019-TGm3
2.2 smY2019-TGm3发酵过程分析
基于摇瓶发酵,比较了smY2019-TGm3和smY2019的TGase合成能力及菌株生长的差异。发酵前24 h,TGase增长较慢,尽管菌体量已接近最大值;发酵24~60 h,菌体量缓慢增长至最大值(36 h)后开始下降,而smY2019-TGm3和smY2019胞外TGase酶活力增长至最大值,分别达到19.5 U/mL和18.9 U/mL;发酵60 h后,smY2019-TGm3和smY2019胞外TGase均开始下降,但前者保持了更高的酶活力(图3-a)。基因组分析显示,除TGase活化蛋白酶之外,S.mobaraensis DSM40587 基因组(GenBank No.CP083590)编码了40余种胞外蛋白酶,可能是其后期酶活力下降的重要原因。SDS-PAGE分析显示,smY2019-TGm3与smY2019均在24 h合成了大量的pro-TGase(43 kDa),但直至发酵36 h才被大量转化为成熟TGase(38 kDa)(图3-b)。值得注意是,smY2019产生的pro-TGase在发酵60 h基本转化为成熟TGase,而smY2019-TGm3的pro-TGase则直至96 h才被完全活化(图3-b)。
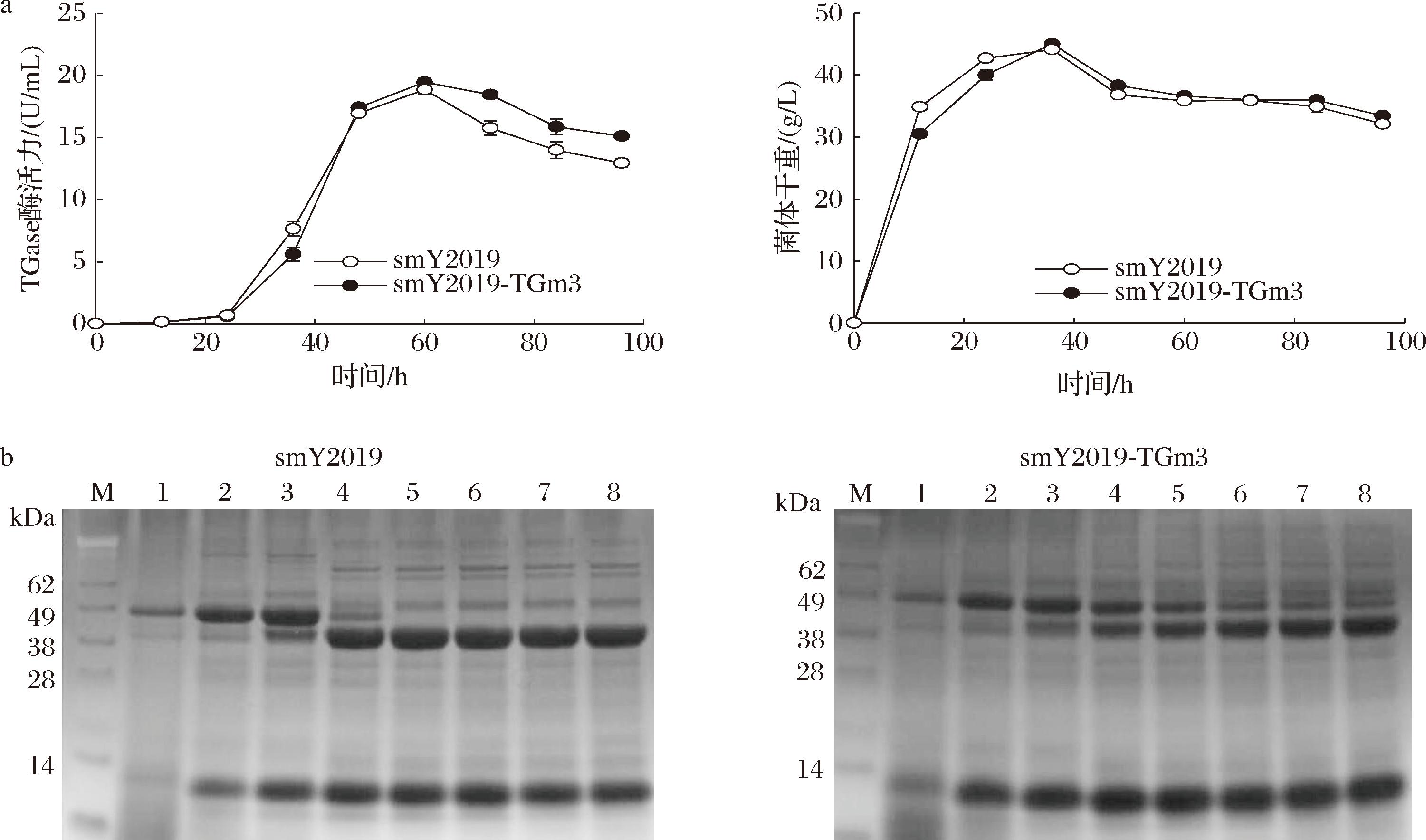
M-蛋白质标准分子质量;1~8-12、24、36、48、60、72、84、96 h a-发酵过程曲线;b-发酵上清液SDS-PAGE分析
图3 smY2019和smY2019-TGm3发酵过程比较
Fig.3 The fermentation process comparison between smY2019 and smY2019-TGm3
上述结果表明,smY2019-TGm3与smY2019的产酶和菌株生长能力基本一致,便后者的酶原活化效率更低。前期研究显示,E.coli表达的TGm3比酶活力是野生型TGase的3.4倍[12]。因此,TGm3较高的比酶活可能弥补了smY2019-TGm3中pro-TGase活化效率的不足,从而在60 h与smY2019有相近的胞外TGase酶活力。在发酵60 h后,smY2019-TGm3持续的活化也可能是其维持高于smY2019胞外酶活力的重要原因之一。
2.3 smY2019-TGm3酶学性质及酶反应动力学分析
为进行酶学性质分析,采用离子交换层析从smY2019和smY2019-TGm3发酵上清液中分别纯化得到单一电泳条带的野生型TGase和TGm3(图4-a)。以N-CBZ-Gln-Gly和羟胺为底物,比较了野生型TGase和TGm3的酶学性质及反应动力学。
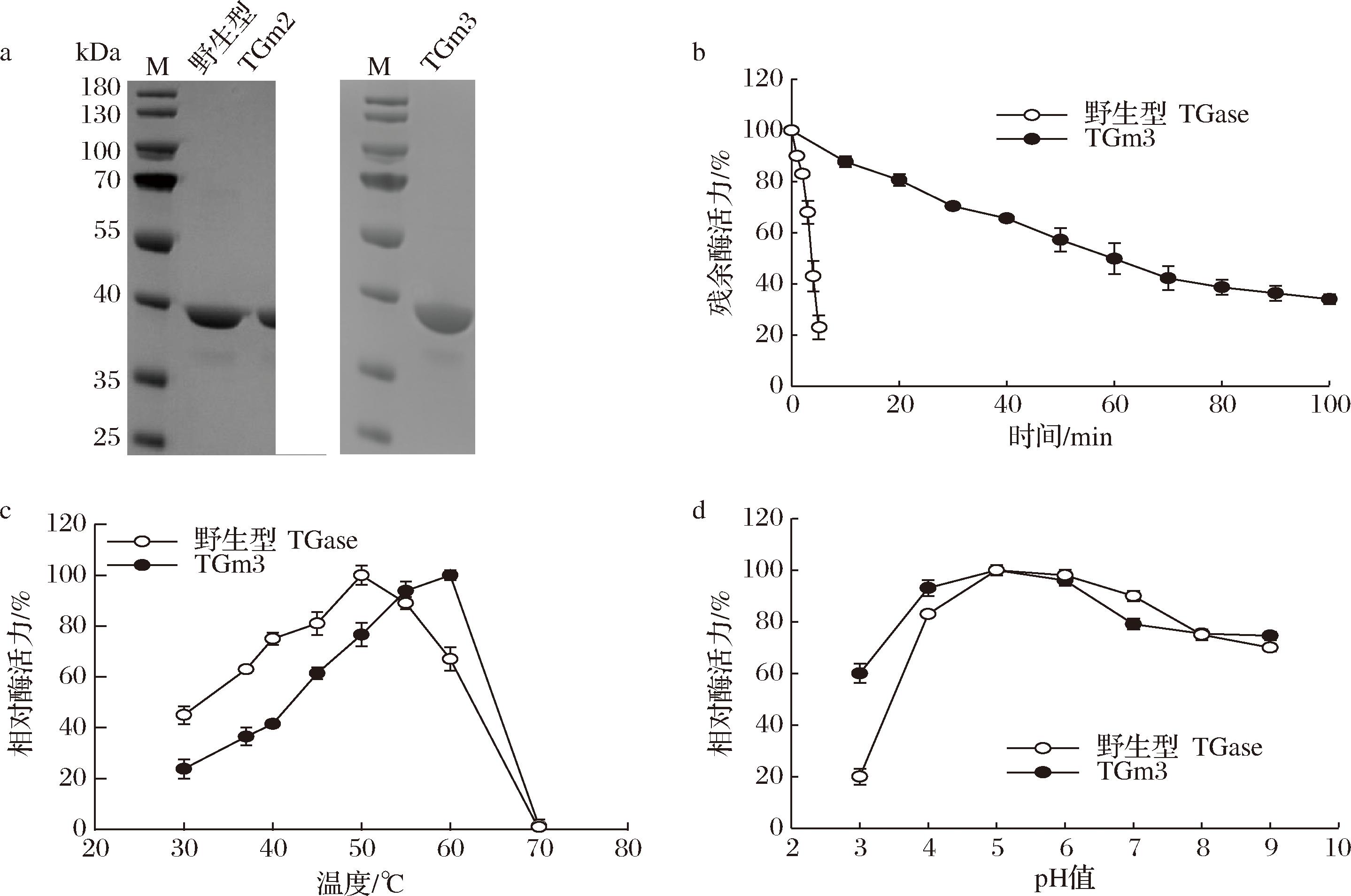
a-纯化酶SDS-PAGE分析;b-酶热稳定性分析;c-温度对酶活力的影响;d-pH对酶活力的影响
图4 野生型TGase和TGm3的酶学性质比较
Fig.4 The catalytic property comparison between wild-type TGase and TGm3
如表2所示,smY2019-TGm3表达的TGm3比酶活力及kcat/Km分别较野生型TGase提高70%和54%,表明TGm3具有更高的催化效率。将上述纯酶于60 ℃和pH 8.0条件下热处理,测定了不同时间的残余酶,并拟合计算得到60 ℃半衰期。如图4-b所示,TGm3在热处理100 min后仍保持34%的初始酶活力,而野生型TGase残余酶活力则在2 min下降至50%以下。结果,TGm3的60 ℃半衰期较野生型TGase提高32.6倍(表2)。同时,前者的最适反应温度也较后者提高了10 ℃(图4-c),但两者在pH 5和pH 6均维持较高的催化反应活性(图4-d)。因此,smY2019-TGm3发酵生产的TGase较野生型TGase更适于高温条件下的应用。
表2 酶学性质及酶反应动力学
Table 2 The catalytic properties and reaction kinetics
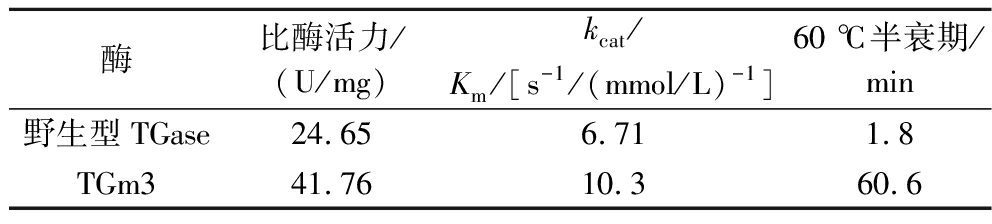
酶比酶活力/(U/mg)kcat/Km/[s-1/(mmol/L)-1]60 ℃半衰期/min野生型TGase24.656.711.8TGm341.7610.360.6
然而,与E.coli系统制备的TGm3相比,本研究表达的S.mobaraensis表达的TGm3显示出更低的热稳定性及催化活性。尽管与本研究的TGm3氨基酸序列一致,E.coli TGm3的60 ℃半衰期和比酶活力分别达到122.9 min和83.7 U/mg[12]。如前所述,TGm3相较于野生型TGase增加了14个氨基酸突变,而且多数突变位于酶分子表面[12]。这些突变可能引入了可被S.mobaraensis大量内源蛋白酶识别的切割位点,从而导致TGm3肽链被部分切割,最终引起酶学性质的改变。
2.4 TGm3稳定化机制的解析
如前所述,与野生型TGase 相比,TGm3包含了14个氨基酸突变[12]。为了揭示酶稳定化机制,在330 K下对野生型TGase和TGm3进行了40 ns的分子动力学模拟。结果如图5所示。

a-分子动力学分析;b-TGm3柔性降低的区域(Cys64为催化活性中心)
图5 野生型TGase和TGm3的分子动力学及结构分析
Fig.5 The molecular dynamics and structural analysis between wild-type TGase and TGm3
TGm3的平均RMSD值(0.164 nm)和最终RMSD值(0.132 nm)均低于野生型TGase(平均值0.169 nm,最终值0.191)(图5-a)。因此,整体结构刚性的增加可能是TGm3高热稳定性的重要原因。基于RMSF值可知,TGm3较野生型刚性增加的区域主要有4个;其中,氨基酸残基246~250和301接近酶催化活性残基Cys64(图5-b)。研究表明,酶催化活性中心刚性的提高有助于酶热稳定性[31]。因此,残基246~250和301刚性的增加可能对于TGm3的热稳定性至关重要。尽管TGm3中静电相互作用较野生型减少2个,但前者的氢键和疏水相互作用分别较后者增加26个和8个(表3)。这表明氢键和疏水相互作用可能是TGm3结构刚性增加的关键因素。此外,TGm3包含了可增加酶分子间静电斥力的6个突变(N96E、S144E、N163D、R183E、R208E和K325E),可能有助于抑制酶分子在高温条件下的聚集,从而使其较野生型具有更的高热稳定性[12]。
表3 酶分子内非共价相互作用数量
Table 3 The amount of non-covalent interactions within the enzyme molecules
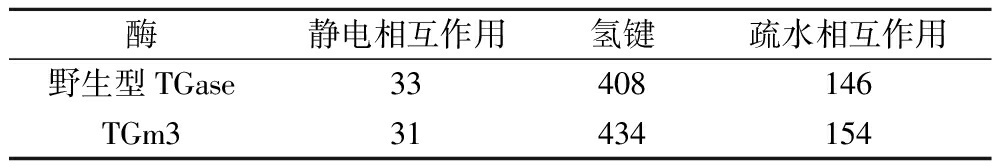
酶静电相互作用氢键疏水相互作用野生型TGase33408146TGm331434154
3 结论
长期以来,S.mobaraensis是我国食品添加剂国家标准GB 2760规定的唯一可用于食品级TGase。上述菌株生产TGase亦通过了美国食品药品监督管理局认证为GRAS认证,也是世界范围广泛接收的食品级TGase生产菌株。然而,S.mobaraensis 生产的野生型TGase的热稳定性较低,不适于诸多工业化应用场景。因此,构建能高效合成耐热型TGase的S.mobaraensis菌株,对于进一步拓宽其工业化应用十分重要。本研究通过双交换同源重组将TGase耐热突变体TGm3基因整合至高产TGase的突变株S.mobaraensis,构建了可分泌表达活性TGase的重组菌smY2019-TGm3,所产TGase突变体催化活性和热稳定性均明显高于目前野生型S.mobaraensis TGase。因此,smY2019-TGm3具工业化应用的潜力。为进一步提高TGm3在S.mobaraensis中的合成水平,敲除宿主内源蛋白酶基因及发酵工艺优化将是下一步工作的研究重点。
[1] SANTHI D, KALAIKANNAN A, MALAIRAJ P, et al.Application of microbial transglutaminase in meat foods:A review [J].Critical Reviews in Food Science and Nutrition, 2017, 57(10):2071-2076.
[2] DUARTE L, MATTE C R, BIZARRO C V, et al.Review transglutaminases:Part Ⅱ-industrial applications in food, biotechnology, textiles and leather products [J].World Journal of Microbiology &Biotechnology, 2019, 36(1):11.
[3] DENNLER P, CHIOTELLIS A, FISCHER E, et al.Transglutaminase-based chemo-enzymatic conjugation approach yields homogeneous antibody-drug conjugates [J].Bioconjugate Chemistry, 2014, 25(3):569-578.
[4] MILCZEK E M.Commercial applications for enzyme-mediated protein conjugation:New developments in enzymatic processes to deliver functionalized proteins on the commercial scale [J].Chemical Reviews, 2018, 118(1):119-141.
[5] DA SILVA FREITAS D, MERO A, PASUT G.Chemical and enzymatic site specific PEGylation of hGH [J].Bioconjugate Chemistry, 2013, 24(3):456-463.
[6] DONDERO M, FIGUEROA V, MORALES X, et al.Transglutaminase effects on gelation capacity of thermally induced beef protein gels [J].Food Chemistry, 2006, 99(3):546-554.
[7] CHANG Y H, SHIAU S Y, CHEN F B, et al.Effect of microbial transglutaminase on the rheological and textural characteristics of black soybean packed tofu coagulating with Agar [J].LWT-Food Science and Technology, 2011, 44(4):1107-1112.
[8] WANG L J, LI D, TATSUMI E, et al.Application of two-stage ohmic heating to tofu processing [J].Chemical Engineering and Processing:Process Intensification, 2007, 46(5):486-490.
[9] MARX C K, HERTEL T C, PIETZSCH M.Random mutagenesis of a recombinant microbial transglutaminase for the generation of thermostable and heat-sensitive variants [J].Journal of Biotechnology, 2008, 136(3-4):156-162.
[10] MU D D, LU J J, SHU C, et al.Improvement of the activity and thermostability of microbial transglutaminase by multiple-site mutagenesis [J].Bioscience Biotechnology and Biochemistry, 2018, 82(1):106-109.
[11] BUETTNER K, HERTEL T C, PIETZSCH M.Increased thermostability of microbial transglutaminase by combination of several hot spots evolved by random and saturation mutagenesis [J].Amino Acids, 2012, 42(2-3):987-996.
[12] WANG X L, DU J H, ZHAO B C, et al.Significantly Improving the thermostability and catalytic efficiency of Streptomyces mobaraenesis transglutaminase through combined rational design [J].Journal of Agricultural and Food Chemistry, 2021, 69(50):15268-15278.
[13] NODA S, MIYAZAKI T, TANAKA T, et al.High-level production of mature active-form Streptomyces mobaraensis transglutaminase via pro-transglutaminase processing using Streptomyces lividans as a host [J].Biochemical Engineering Journal, 2013, 74:76-80.
[14] MA T G, LU J J, ZHU J, et al.The secretion of Streptomyces monbaraensis transglutaminase from Lactococcus lactis and immobilization on porous magnetic nanoparticles [J].Frontiers in Microbiology, 2019, 10:1675.
[15] KIKUCHI Y, DATE M, YOKOYAMA K I, et al.Secretion of active-form Streptoverticillium mobaraense transglutaminase by Corynebacterium glutamicum:Processing of the pro-transglutaminase by a cosecreted subtilisin-Like protease from Streptomyces albogriseolus [J].Applied and Environmental Microbiology, 2003, 69(1):358-366.
[16] DATE M, YOKOYAMA K I, UMEZAWA Y, et al.Production of native-type Streptoverticillium mobaraense transglutaminase in Corynebactetium glutamicum [J].Applied and Environmental Microbiology, 2003, 69(5):3011-3014.
[17] MU D D, LU J J, QIAO M Q, et al.Heterologous signal peptides-directing secretion of Streptomyces mobaraensis transglutaminase by Bacillus subtilis [J].Applied Microbiology and Biotechnology, 2018, 102(13):5533-5543.
[18] YANG X Q, ZHANG Y M.Expression of recombinant transglutaminase gene in Pichia pastoris and its uses in restructured meat products [J].Food Chemistry, 2019, 291:245-252.
[19] LIU S, WAN D, WANG M, et al.Overproduction of pro-transglutaminase from Streptomyces hygroscopicus in Yarrowia lipolytica and its biochemical characterization [J].BMC Biotechnology, 2015, 15:75.
[20] ZOTZEL J, PASTERNACK R, PELZER C, et al.Activated transglutaminase from Streptomyces mobaraensis is processed by a tripeptidyl aminopeptidase in the final step [J].European Journal of Biochemistry, 2003, 270(20):4149-4155.
[21] LIU S, ZHANG D X, WANG M, et al.The pro-region of Streptomyces hygroscopicus transglutaminase affects its secretion by Escherichia coli [J].FEMS Microbiology Letters, 2011, 324(2):98-105.
[22] JUETTNER N E, SCHMELZ S, ANDERL A, et al.The N-terminal peptide of the transglutaminase-activating metalloprotease inhibitor from Streptomyces mobaraensis accommodates both inhibition and glutamine cross-linking sites [J].The FEBS Journal, 2020, 287(4):708-720.
[23] WANG L M, YU B, WANG R X, et al.Biotechnological routes for transglutaminase production:Recent achievements, perspectives and limits [J].Trends in Food Science &Technology, 2018, 81:116-120.
[24] ZHANG N, ZHANG S, HE Y Z, et al.Intein-mediated intracellular production of active microbial transglutaminase in Corynebacterium glutamicum [J].Enzyme and Microbial Technology, 2020, 142:109680.
[25] YIN X Q, LI Y Y, ZHOU J W, et al.Enhanced production of transglutaminase in Streptomyces mobaraensis through random mutagenesis and site-directed genetic modification [J].Journal of Agricultural and Food Chemistry, 2021, 69(10):3144-3153.
[26] PASTERNACK R, DORSCH S, OTTERBACH J T, et al.Bacterial pro-transglutaminase from Streptoverticillium mobaraense[J].European Journal of Biochemistry, 1998, 257(3):570-576.
[27] LIU S, WANG M, DU G C, et al.Improving the active expression of transglutaminase in Streptomyces lividans by promoter engineering and codon optimization [J].BMC Biotechnology, 2016, 16(1):75.
[28] MIRDITA M, SCHÜTZE K, MORIWAKI Y, et al.ColabFold:Making protein folding accessible to all [J].Nature Methods, 2022, 19(6):679-682.
[29] CONWAY P, TYKA M D, DIMAIO F, et al.Relaxation of backbone bond geometry improves protein energy landscape modeling [J].Protein Science, 2014, 23(1):47-55.
[30] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers [J].SoftwareX, 2015, 1:19-25.
[31] XIE Y, AN J, YANG G Y, et al.Enhanced enzyme kinetic stability by increasing rigidity within the active site [J].Journal of Biological Chemistry, 2014, 289(11):7994-8006.