副干酪乳酪杆菌(Lacticaseibacillus paracasei)是乳杆菌属、革兰氏阳性异型发酵乳酸菌(lactic acid bacteria,LAB),常见于乳制品中[1-2]。副干酪乳酪杆菌具有较多的益生功能,如增强机体免疫、调节肠道菌群稳态、降低血糖水平等,对人体健康具有积极影响[3]。基于副干酪乳酪杆菌的益生功能,现多将副干酪乳酪杆菌添加至发酵制品中生产功能性产品,如奶酪、酸奶[4]。
乳制品(如牛奶)的主要成分是酪蛋白,其约占总蛋白质含量的80%[5]。乳制品中含有重要的益生菌——LAB。LAB的生长需要外源氨基酸或肽,而LAB中的蛋白水解系统可以水解乳制品中的酪蛋白为其提供氨基酸或肽[6-7]。LAB的蛋白水解能力较弱,但其具有完整的蛋白酶/肽酶系统,主要有细胞包膜相关蛋白酶PrtP、细胞内寡肽内肽酶(PepO、PapF)、氨肽酶(PepN、PepC、PepG)、谷氨酰氨肽酶PepA、亮氨酰氨肽酶PepL、脯氨酰二肽氨肽酶PepX以及转氨酶BcaT;这些蛋白酶基因和氨基酸转化酶编码基因的存在是LAB在奶酪中生长以及奶酪风味化合物形成的先决条件[8]。
全基因组测序技术不仅揭示了菌株基因组的异同,也使得细菌的表型特征同发挥作用的基因型紧密联系起来,便于深入分析某一表观现象的具体作用机制。比较基因组学通过对不同菌株间基因组的比较,从而更加深入探究菌株的基因组及功能基因[9]。刘慧敏[10]通过全基因组测序手段测定6株LAB全基因草图并注释,系统分析了这6株LAB基因组的结构组成和特征基因,并通过验证特征基因,明确了种属内新菌株。LI等[11]应用宏基因组学和基因组学技术手段从传统发酵食品中快速鉴定出新抗菌LAB菌株,并挖掘出防止李斯特菌等致病菌污染、改善奶酪的质地和感官品质的菌株,可作为奶酪理想的附属发酵剂。HOSSAIN[12]通过比较基因组学方法发现,发酵黏液乳杆菌LAB-1含有编码营养保健品、发酵食品等具有益生功能、编码压力诱导蛋白及编码防御系统的优良基因,这些基因使其具有提高食品质量、在肠道和工业条件下生存、抵御噬菌体感染的益生能力。吕瑞瑞等[13]通过三代测序技术对副干酪乳酪杆菌PC-01进行全基因组测序,比较基因组学方法分析39株副干酪乳酪杆菌菌株间的基因组差异和遗传背景。基因组学技术的应用使得更多新的基因被发现,进一步挖掘出更多功能性更好的菌株,并将其应用于食品中,增强食品的益生作用,从而利于人体健康。
本研究基于蛋白酶活力对5株副干酪乳酪杆菌BDⅡ、J4、J8、J15、J17进行筛选,得到3株蛋白酶活力相对较高的菌株,并对其进行全基因组分析,最终得到1株蛋白酶活力最高的菌株BDⅡ。本研究通过表型分析深入挖掘蛋白水解基因,了解副干酪乳酪杆菌蛋白水解机制,为益生菌类乳制品功能物质产生机制的研究提供依据。
1 材料与仪器
1.1 实验菌株
菌株:副干酪乳酪杆菌BDⅡ、J4、J8、J15、J17,鼠李糖乳酪杆菌B6,光明乳业研究院菌种库-80 ℃保存。
1.2 试剂与仪器
MRS肉汤培养基、酪蛋白,德国MERCK公司;琼脂、福林酚试剂、无水Na2CO3、NaOH、盐酸,国药集团化学试剂有限公司;细菌基因组DNA抽提试剂盒(B518255),生工生物有限公司;三氯乙酸、L-酪氨酸,上海阿拉丁生化科技股份有限公司;1×磷酸盐缓冲液,Gibco公司。
Spectramax M5酶标仪,美国Molecular Devices公司。
2 实验方法
2.1 菌株培养
将冻存于-80 ℃冰箱的5株副干酪乳酪杆菌在MRS液体培养基中活化3代,37 ℃厌氧培养24 h。将菌液以6 000 r/min离心3 min,收集沉淀和上清液备用;用0.9%(质量分数)无菌生理盐水清洗菌沉淀3次后提取基因组DNA[14]。
2.2 菌株产蛋白酶能力及活力检测
取上述离心得到的上清液10 μL于脱脂乳平板上,37 ℃放置24 h,观察透明圈的情况。取无菌生理盐水清洗的菌沉淀以体积分数3%接种量接入脱脂乳中培养24 h[15]。按照国标GB/T 23527—2009《蛋白酶制剂》中福林法测定蛋白酶活力。标准曲线方程:Y=0.061 5X-0.056 7(R2=0.998 6),X表示酪氨酸质量浓度(μg/mL),Y表示680 nm处吸光度。
2.3 基因组分析
2.3.1 基因组DNA提取、测序
根据生工生物公司的说明书提取细菌基因组DNA,所提DNA(OD260/280=1.8~2.0,DNA总量≥10 μg,质量浓度≥50 ng/μL)用于建库测序[16]。
2.3.2 Illumina全基因组草图测序、组装及质控分析
采用二代Illumina HiSeq PE150基因组de novo测序方法对副干酪乳酪杆菌的基因组进行测序。文库构建具体参考肖宇等[17]的方法。桥式PCR部分具体参考尹明华等[18]的方法。该部分委托上海美吉生物医药科技有限公司完成。
2.3.3 基因组核苷酸平均一致性(average nucleotide identity,ANI)分析
为了进一步评估副干酪乳酪杆菌的进化距离关系和所属种属,自NCBI(https://www.ncbi.nlm.nih.gov/)数据库检索副干酪乳酪杆菌的基因组数据,使用美吉生物云平台数据库对菌株全基因组序列进行ANI分析[19]。
2.3.4 基因组功能分析
在美吉生物云平台对3株副干酪乳酪杆菌的全基因组进行直系同源聚类群(clusters of orthologous groups,COG)、基因本体论(gene ontology,GO)、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)、碳水化合物活性酶(carbohydrate-active enzymes,CAZys)功能分析,所有数据库中序列比对阈值设置E-value≤10-5[20-21]。
2.3.5 GT51和肽酶分析
在NCBI数据库获得阴性对照B6的全基因组信息,RefSeq序列编号为GCF_016599675.1,B6存在pepX(WP_005711613.1)和GT51(WP_020751873.1),通过Boxshade(https://directory.fsf.org/wiki/Boxshade)比对BDⅡ和B6基因pepX和GT51的氨基酸序列。通过Swiss Model网站(https://swissmodel.expasy.org)在线对副干酪乳酪杆菌BDⅡ特有肽酶的三维结构进行同源建模[22]。在PyMol软件进行可视化[23]。
2.4 数据处理
CAZys数据图使用Excel 2019软件完成。
3 结果与分析
3.1 菌株蛋白分解特性分析
副干酪乳酪杆菌可水解酪蛋白,因此在脱脂乳平板上会产生透明圈。如图1所示,5株菌均可水解酪蛋白,产生透明圈。张传丽等[24]将芽胞杆菌接种于蛋白酶检测培养基上,发现其可水解蛋白产生透明圈。徐美余[25]将副干酪乳酪杆菌接种于蛋白酶培养基上,发现该菌也可产生水解透明圈,与本实验结果一致。

a-BDⅡ;b-J4;c-J17;d-J8;e-J15
图1 五株菌的蛋白水解透明圈图
Fig.1 Transparent circle diagram of protein hydrolysis of five strains of bacteria
3.2 蛋白酶活力分析结果
蛋白酶活力高的菌株可以催化蛋白分解产生更多的有益活性物质,赋予食品益生功能,对人体健康有益。由图2可知,6株菌的蛋白酶活力从大到小依次为BDⅡ、J4、J17、J8、J15、B6,其中BDⅡ、J4、J17蛋白酶活力相对较高,6株菌蛋白酶活力具有显著差异(P<0.05),BDⅡ的蛋白酶活力最高,为22.017 U/mL。吴涵清等[15]发现LAB的酶活力低于10 U/mL,与本实验研究结果一致。许倩[26]检测副干酪乳酪杆菌H9的蛋白酶活力为(1.068±0.155) U/mL,本实验6株菌蛋白水解能力均优于H9。MA等[27]通过筛选蛋白酶活力>15 U/mL的益生菌LAB用于发酵生产饲料,LAB的蛋白酶活力最高为27.4 U/mL,本实验副干酪乳酪杆菌的蛋白酶活力处于其间,可能是菌株间的蛋白酶基因表达水平、代谢等的差异使得蛋白酶活力不同。
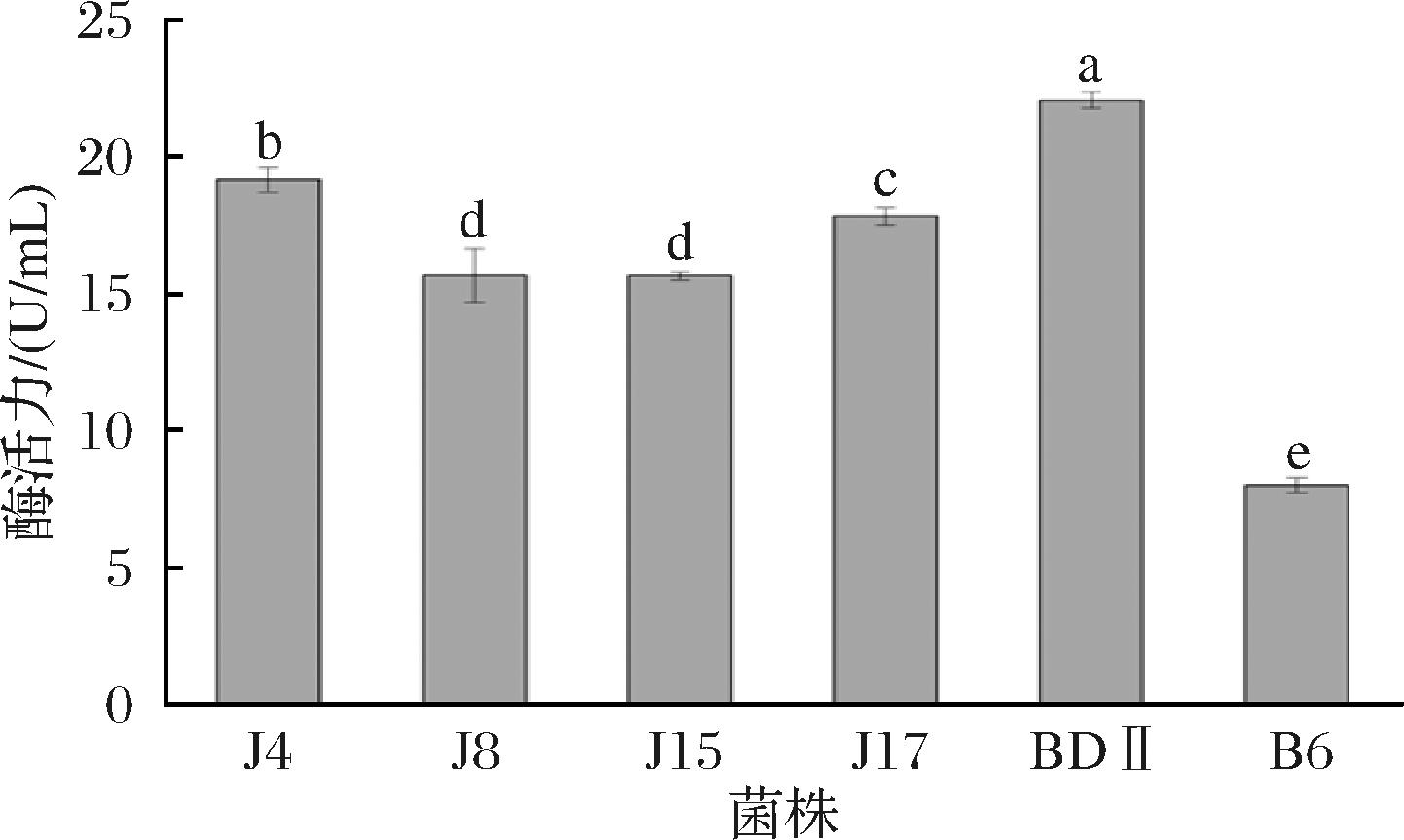
图2 六株菌的蛋白酶活性检测结果
Fig.2 Results of protease activity assay of six strains of bacteria
注:不同小写字母代表差异显著(P<0.05)。
3.3 副干酪乳酪杆菌基因组信息
BDⅡ、J4、J17蛋白酶活力较高,对其进行数据评估和序列组装,得到基因组组装结果和基因组基本特征、GC含量、基因组大小、蛋白质编码区域(coding sequence,CDS)数量、tRNA数量等。由表1和表2可知,J4经组装后有108个scaffolds,46个质粒,基因组大小为2 980 874 bp,GC含量为46.25%,有55个tRNA,5个rRNA,含有2 921个CDS。J17经组装后有95个scaffolds,35个质粒,基因组大小为2 975 575 bp,GC含量为46.27%,有55个tRNA,4个rRNA,含有2 903个CDS。BDⅡ经组装后有82个scaffolds,36个质粒,基因组大小为3 033 819 bp,GC含量为46.24%,有52个tRNA,2个rRNA,含有3 008个CDS。不同的菌具有不同的基因组信息,其中BDⅡ的CDS区明显多于J4,J4多于J17。吕瑞瑞等[13]对39株副干酪乳酪杆菌进行全基因组分析,发现副干酪乳酪杆菌的GC含量为46.14%~46.64%,基因组大小为2 383 240~3 246 143 bp,与本研究结果基本一致;GHOSH等[28]研究发现副干酪乳酪杆菌基因组大小为2.3~3.3 Mb,与本研究结果一致。
表1 副干酪乳酪杆菌J4、J17、BDⅡ的基因组组装结果统计
Table 1 Statistics of genome assembly results of Lacticaseibacillus paracasei J4, J17, and BDⅡ
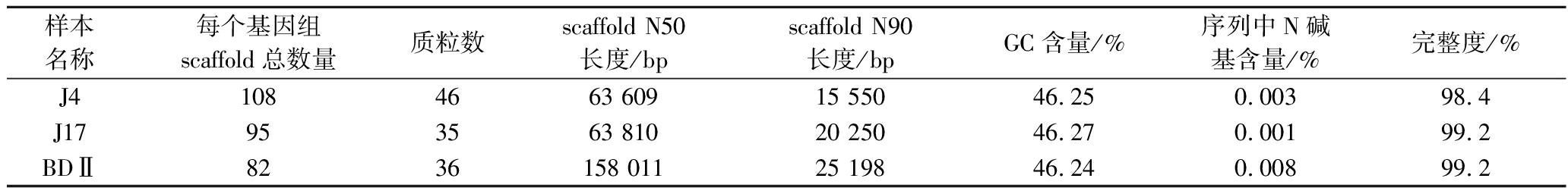
样本名称每个基因组scaffold总数量质粒数scaffold N50长度/bpscaffold N90长度/bpGC含量/%序列中N碱基含量/%完整度/%J41084663 60915 55046.250.00398.4J17953563 81020 25046.270.00199.2BDⅡ8236158 01125 19846.240.00899.2
表2 副干酪乳酪杆菌 J4、J17、BDⅡ基因组基本特征
Table 2 Basic characterization of the genomes of L. paracasei J8, J17, and BDⅡ
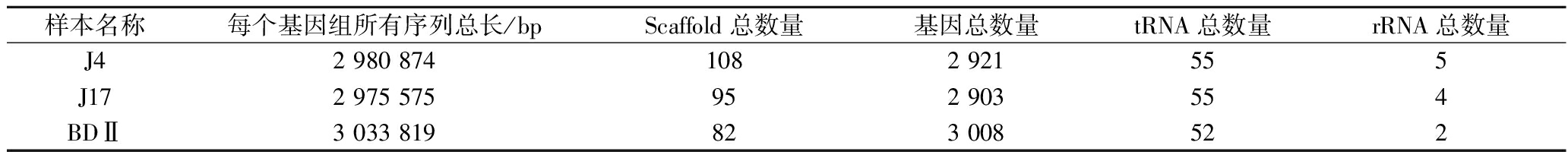
样本名称每个基因组所有序列总长/bpScaffold总数量基因总数量tRNA总数量rRNA总数量J42 980 8741082 921555J172 975 575952 903554BDⅡ3 033 819823 008522
注:每个基因组所有序列总长:如果存在染色体、质粒,取其总和。
3.4 基因组圈图
基于组装的基因组序列和蛋白质编码基因的预测结果绘制基因组图谱,提供基因组特征的可视化。使用CGView软件绘制的副干酪乳酪杆菌的基因组圈图可以更加全面的、直观的显示基因组特征[18]。由图3-a可知,BDⅡ的圆形基因组图谱的最外圈长度为3 033 819 bp。第2圈和第3圈分别是正链和负链编码区,不同的颜色表示CDS不同的COG的功能分类,共鉴定出3 008个蛋白质编码基因,其中2 315(76.96%)个基因被COG注释。由图3-b可知,J4的圆形基因组图谱的最外圈长度为2 980 874 bp。共鉴定出2 921个蛋白质编码基因,其中2 221(76.04%)个基因被COG注释。由图3-c可知,J17的圆形基因组图谱的最外圈长度为2 975 575 bp。共鉴定出2 903个蛋白质编码基因,其中2 213(76.23%)个基因被COG注释。3株菌均有4种主要功能类型被注释,分别为代谢、信息存储和处理、细胞功能和信号传递及非典型特征;共注释23种类型,包括碳水化合物转运和代谢、转录、一般功能预测、氨基酸转运和代谢、未知功能和其他几种功能。第4圈为rRNA、tRNA、IS、噬菌体等,其中黑色和红色代表tRNA和rRNA。第5圈为GC含量,其中橙色和蓝色表示GC含量分别高于或低于全基因组的平均值。第6圈为GC偏移值,图中黄绿色(朝向外侧)和橘黄色(朝向内侧)分别表示前导链和滞后链。3株副干酪乳酪杆菌的基因组图谱直接显示了菌株的基因组分布情况。
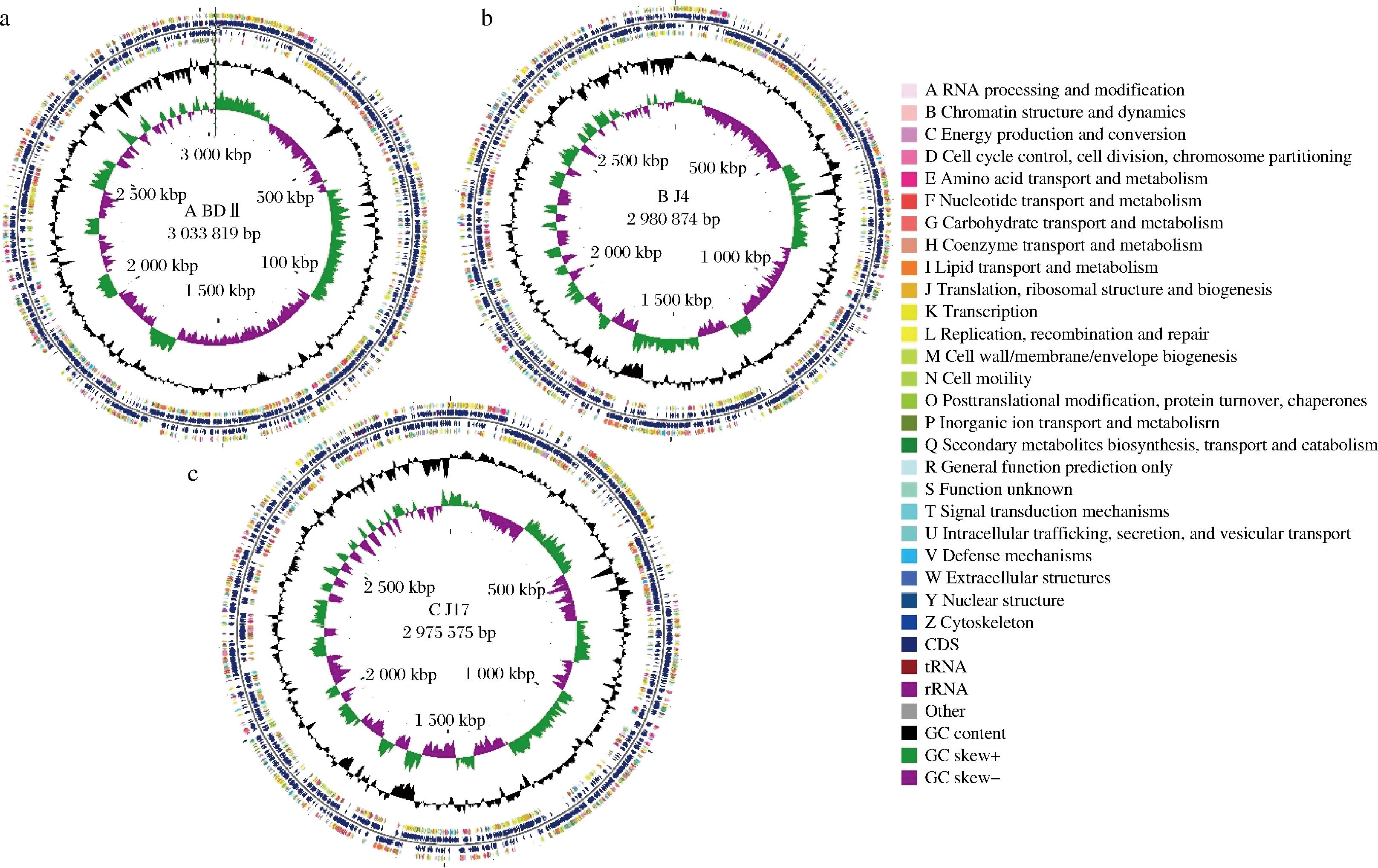
a-BDⅡ;b-J4;c-J17
图3 三株副干酪乳酪杆菌的基因组圈图
Fig.3 Genomic circle map of three strains of L. paracasei
注:圈图从外到内第1圈和第4圈为正链、负链上的CDS,不同的颜色表示不同的COG功能分类;第2圈和第3圈分别为正链、负链上的CDS、tRNA、rRNA;第5圈为GC含量;第6圈为GC-Skew值;最内1圈为基因组大小标识。A RNA processing and modification RNA 加工和修饰,B Chromatin structure and dynamics染色质结构和动力学,C Energy production and conversion能量产生和转换,D Cell cycle control, cell division, chromosome partitioning细胞周期控制、细胞分裂、染色体分配,E Amino acid transport and metabolism氨基酸转运和代谢,F Nucleotide transport and metabolism核苷酸转运和代谢,G Carbohydrate transport and metabolism碳水化合物转运和代谢,H Coenzyme transport and metabolism辅酶转运和代谢,I Lipid transport and metabolism脂质转运和代谢,J Translation, ribosomal structure and biogenesis翻译、核糖体结构和生物合成,K Transcription转录,L Replication, recombination and repair复制、重组和修复,M Cell wall/membrane/envelope biogenesis细胞壁/膜/被膜的生物合成,N Cell motility细胞运动,O Posttranslational modification, protein turnover, chaperones 翻译后修饰、蛋白质折叠和伴侣蛋白,P Inorganic ion transport and metabolism无机离子转运代谢,Q Secondary metabolites biosynthesis, transport and catabolism次级代谢物生物合成、转运和代谢,R General function prediction only主要功能预测,S Function unknown未知功能,T Signal transduction mechanisms信号转导机制,U Intracellular trafficking, secretion, and vesicular transport胞内转运、分泌和小泡运输,V Defense mechanisms抵御机制,W Extracellular structures胞外结构,Y Nuclear structures 核结构,Z Cytoskeleton细胞骨架。
3.5 ANI分析
ANI是用来辨认相同物种或进化关系密切的物种的1种方法,根据范围常分为:亚种间相似(90%~96%)、种间相似(70%~90%)和属间相似(50%~70%),同一物种多以95%为阈值[29]。为评估副干酪乳酪杆菌进化关系,对副干酪乳酪杆菌基因组及其ANI值计算并绘制热图。
如电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.039758)所示,为了比较LAB种属间的异同,从NCBI数据库下载已完成全基因组测序未受污染的15株代表性LAB进行ANI分析(包括同源相似性较高的13株副干酪乳酪杆菌,同源相似性相对较低的2株约氏乳杆菌)。由图4可知,对18株菌进行ANI值分析,16株副干酪乳酪杆菌ANI值>98%,表明本研究3株乳杆菌与种间其他菌株同源相似性较高,均为副干酪乳酪杆菌,为后续对副干酪乳酪杆菌的功能研究及其在乳制品中的应用提供依据。吕瑞瑞等[13]对39株副干酪乳酪杆菌进行ANI分析,发现39株菌株的ANI值均大于98.09%,与本研究结果一致。

图4 菌株基因组ANI热图
Fig.4 Heat map of strains genome ANI
3.6 COG注释分析结果
为了进一步探究3株菌核心基因功能,利用COG注释数据库对3株乳杆菌已鉴定的蛋白质编码基因进行功能注释分类。
如图5-a所示,BDⅡ注释了23类2 315个蛋白质编码基因,其中345个(15%)蛋白质编码基因在碳水化合物转运和代谢类别中富集,212个(9.2%)蛋白质编码基因在翻译、核糖体结构和生物发生类别中富集,205个蛋白质编码基因在转录类别中富集。由图5-b所示,J4注释了23类2 221个蛋白质编码基因,其中314个(14%)蛋白质编码基因在碳水化合物转运和代谢类别中富集,210个(9.4%)蛋白质编码基因在翻译、核糖体结构和生物发生类别中富集,207个(9.3%)蛋白质编码基因在转录中富集,201(9%)个蛋白质编码基因仅在一般功能预测、氨基酸转运和代谢中富集;由图5-c所示,J17注释了23类2 213个蛋白质编码基因,其中314个(14%)蛋白质编码基因在碳水化合物转运和代谢中富集,209个(9.4%)蛋白质编码基因在翻译、核糖体结构和生物发生类别中富集,206个(9.3%)蛋白质编码基因在转录中富集。因此这3株菌均为碳水化合物转运和代谢类别蛋白基因富集较多,预测3株副干酪乳酪杆菌碳水化合物利用能力较强。值得注意的是,BDⅡ被注释的蛋白编码基因最多。

a-BDⅡ;b-J4;c-J17
图5 三株副干酪乳酪杆菌COG功能注释
Fig.5 Functional annotation of three strains of L. paracasei COG
注:C Energy production and conversion能量生产与转换,D Cell cycle control, cell division, chromosome partitioning细胞周期控制、细胞分裂、染色体分配,E Amino acid transport and metabolism氨基酸转运和代谢,F Nucleotide transport and metabolism核苷酸转运和代谢,G Carbohydrate transport and metabolism碳水化合物转运和代谢,H Coenzyme transport and metabolism辅酶转运和代谢,I Lipid transport and metabolism脂质转运和代谢,J Translation, ribosomal structure and biogenesis翻译、核糖体结构和生物合成,K Transcription转录,L Replication, recombination and repair复制、重组和修复,M Cell wall/membrane/envelope biogenesis细胞壁/膜/被膜的生物合成,N Cell motility细胞运动,O Posttranslational modification, protein turnover, chaperones 翻译后修饰、蛋白质折叠和伴侣蛋白,P Inorganic ion transport and metabolism无机离子转运代谢,Q Secondary metabolites biosynthesis, transport and catabolism次级代谢物生物合成、转运和代谢,R General function prediction only主要功能预测,S Function unknown未知功能,T Signal transduction mechanisms信号转导机制,U Intracellular trafficking, secretion, and vesicular transport胞内转运、分泌和小泡运输,V Defense mechanisms抵御机制,W Extracellular structures胞外结构,X Mobilome: prophages, transposons动员组 噬菌体原、转座子,Z Cytoskeleton细胞骨架。
3.7 GO注释分析结果
通过GO注释可以获得基因标准化的功能描述信息。如图6所示,BDⅡ在GO数据库中共注释了2 309个基因;J4在GO数据库中共注释了2 138个基因;J17在GO数据库共注释了2 129个基因。GO数据库主要对菌株3部分进行注释,分别为生物过程、细胞组分和分子功能,生物过程主要有14个分类,细胞组分有14个分类,分子功能有14个分类。

a-BDⅡ;b-J4;c-J17
图6 三株副干酪乳酪杆菌GO分类功能统计
Fig.6 Functional statistics of GO classification of three strains of L. paracasei
注:图例部分:Biological Process生物过程,Cellular Component细胞组分,Molecular Function分子功能。横坐标部分:生物过程:phosphorylation磷酸化反应,phosphoenolpyruvate-dependent sugar phosphotransferase system依赖磷酸烯醇丙酮酸的糖磷酸转移酶系统,proteolysis蛋白酶解,translation翻译,carbohydrate metabolic process碳水化合物代谢过程,regulation of DNA-templated transcription DNA模板转录的调控,transmembrane transport跨膜转运,cell division细胞分裂,DNA repair DNA修复,methylation甲基化作用,regulation of cell shape细胞形态调控,DNA replication DNA复制,DNA recombination DNA重组,carbohydrate transport碳水化合物转运;细胞组分:membrane细胞膜,cytoplasm细胞质,plasma membrane质膜,ribosome核糖体,ribonucleoprotein complex核糖体蛋白复合体,ATP-binding cassette (ABC) transporter complex ABC转运复合体,DNA-directed RNA polymerase complex 以DNA导向的RNA聚合酶复合体,extracellular region胞外区域,periplasmic space周质空间,large ribosomal subunit核糖体大亚基,chromosome染色质,small ribosomal subunit核糖体小亚基,proton-transporting ATP synthase complex, catalytic core F(1) 质子转运 ATP 合酶复合物,催化核 F (1),envelope包膜;分子功能:ATP binding ATP结合,DNA binding DNA结合,hydrolase activity水解酶活性,metal ion binding金属离子结合,transmembrane transporter activity跨膜转运体活性,ATP hydrolysis activity ATP水解活性,transferase activity转移酶活性,DNA-binding transcription factor activity DNA 结合转录因子的活性,kinase activity激酶活性,oxidoreductase activity氧化还原酶活性,D-glucosamine PTS permease activity D-葡糖胺 PTS 渗透酶活性,zinc ion binding锌离子结合,structural constituent of ribosome核糖体结构成分,protein-N(PI)-phosphohistidine- sugar phosphotransferase activity蛋白-N(PI)-磷组氨酸-糖磷酸转移酶活性。
由图6-a所示,BDⅡ生物过程中注释前3位的是氧化磷酸化(118个)、磷酸烯醇式丙酮酸依赖性糖磷酸转移酶系统(113个)、蛋白质水解作用(70个)相关成分的基因。细胞组分中注释靠前的是细胞膜(539个)、细胞质(270个)、细胞质膜(261个)、核糖体(53个)相关成分基因。分子功能中注释靠前的是ATP结合区(325个)、DNA结合区(243个)、水解酶活性类(150个)、金属离子结合区(117个)、跨膜转运蛋白活性(113个)相关成分基因。由图6-b所示,J4生物过程中注释前3位的是氧化磷酸化(109个)、磷酸烯醇式丙酮酸依赖性糖磷酸转移酶系统(103个)、蛋白质水解作用(63个)相关成分的基因。细胞组分中注释靠前的是细胞膜(474个)、细胞质(260个)、细胞质膜(247个)、核糖体(51个)相关成分基因。分子功能中注释靠前的是ATP结合区(306个)、DNA结合区(221个)、水解酶活性类(135个)、转移酶活性类(110个)、跨膜转运蛋白活性(109个)相关成分基因。由图6-c所示,J17生物过程中注释前3位的是氧化磷酸化(108个)、磷酸烯醇式丙酮酸依赖性糖磷酸转移酶系统(101个)、蛋白质水解作用(62个)相关成分的基因。细胞组分中注释靠前的是细胞膜(468个)、细胞质(261个)、细胞质膜(248个)、核糖体(51个)相关成分基因。分子功能中注释靠前的是ATP结合区(306个)、DNA结合区(221个)、水解酶活性类(134个)、跨膜转运蛋白活性(110个)、转移酶活性类(110个)相关成分基因。值得注意的是,BDⅡ在GO通路中注释的基因是3株菌中最多的,J4和J17注释的功能基因数目相近,功能也相似。在分子功能注释中3株菌具有明显差异,BDⅡ注释靠前的是金属离子结合区,而J4和J17则是转移活性酶类。
3.8 KEGG注释分析结果
副干酪乳酪杆菌编码基因的KEGG注释如图7所示。BDⅡ共有2 099个基因富集于40条代谢途径,J4共有2 018个基因富集于40条代谢途径,J17共有2 017个基因富集于40条代谢途径,3株菌均有6个主要代谢功能组成,分别为细胞过程、代谢、遗传信息处理、人类疾病、生物系统和环境信息处理。

a-BDⅡ;b-J4;c-J17
图7 三株副干酪乳酪杆菌的KEGG代谢通路分类图
Fig.7 Classification map of KEGG metabolic pathways in three strains of L. paracasei
注:图例部分:Cellular Processes细胞过程,Environmental Infromation Processing环境信息处理,Genetic Information Processing基因信息处理,Human Diseases人类疾病,Metabolism代谢,Organismal Systems生物系统;图纵坐标部分:Cell growth and death细胞的生长和死亡,Cell motility细胞运动,Cellular community-prokaryotes细胞群落-原核生物,Transport and catabolism运输和分解代谢,Membrane transport膜运输,Signal transduction信号传导,Signaling molecules and interaction信号分子与相互作用,Folding, sorting and degradation折叠、分类和降解,Replication and repair复制和修复,Transcription转录,Translation翻译,Cancer: overview癌症:概述,Cancer: specific types癌症:特定类型;Cardiovascular disease心血管疾病,Drug resistance: antimicrobial耐药性:抗菌,Drug resistance: antineoplastic耐药性:抗肿瘤,Endocrine and metabolic disease内分泌代谢疾病,Immune disease免疫疾病,Infectious disease: bacterial传染病:细菌性,Infectious disease: parasitic传染病:寄生虫病,Infectious disease: viral传染病:病毒性,Neurodegenerative disease神经退行性疾病,Amino acid metabolism氨基酸代谢,Biosynthesis of other secondary metabolites其它次级代谢产物的生物合成,Carbohydrate metabolism碳水化合物代谢,Energy metabolism能量代谢,Global and overview maps全局和概览图,Glycan biosynthesis and metabolism甘聚糖的生物合成与代谢,Lipid metabolism脂质代谢,Metabolism of cofactors and vitamins辅因子和维生素的代谢,Metabolism of other amino acids其他氨基酸的代谢,Metabolism of terpenoids and polyketides萜类化合物和聚酮类化合物的代谢,Nucleotide metabolism核苷酸代谢,Xenobiotics biodegradation and metabolism异生物降解与代谢,Aging老化,Digestive system消化系统,Endocrine system内分泌系统,Environmental adaptation环境适应,Immune system免疫系统,Nervous system神经系统。
在代谢过程中,共注释了12条代谢途径。由图7-a所示,BDⅡ碳水化合物代谢和氨基酸代谢途径分别富集331、139个基因。在遗传信息处理类别中,有4条途径被注释,其中翻译途径的注释最多,共有84个基因被富集。在人类疾病类别中,共125个基因被富集。耐药性途径注释最多,共42个基因被富集,其次是细菌性传染病和癌症途径注释较多,分别有23和19个基因被富集。由此可知,BDⅡ与人类健康关系密切,特别是抗菌性,主要抵御致病菌和有害菌。在生物系统类别中,内分泌系统和老化注释最多,分别有22和8个基因被富集。环境信息处理过程不仅与益生菌本身密切相关,也与宿主密切相关。在环境信息处理类别中,信号转导和膜转运系统注释最多,分别82和246个基因被富集。由图7-b所示,J4碳水化合物代谢和氨基酸代谢途径分别富集307、138个基因。在遗传信息处理类别中,有4条途径被注释,其中翻译途径的注释最多,共有83个基因被富集。在人类疾病类别中,共113个基因被富集。耐药性途径注释最多,共39个基因被富集,其次是细菌性传染病和癌症途径注释较多,分别有16和20个基因被富集。在生物系统类别中,内分泌系统和老化注释最多,分别有23和6个基因被富集。在环境信息处理类别中,信号转导和膜转运系统注释最多,分别有80和233个基因被富集。由图7-c所示,J17在细胞过程中细胞群落与细胞生长和死亡类别注释较多,分别为73、14个基因被富集。碳水化合物代谢和氨基酸代谢途径分别富集307、138个基因。在遗传信息处理类别中,有4条途径被注释,其中翻译途径的注释最多,共有82个基因被富集,其次是复制和修复类别中有59个基因被富集。在人类疾病类别中耐药性途径注释最多,共39个基因被富集,其次是细菌性传染病和癌症途径注释较多,分别有16和20个基因被富集。在生物系统类别中,内分泌系统和老化注释最多,分别有22和6个基因被富集。在环境信息处理类别中,信号转导和膜转运系统注释最多,分别有80和233个基因被富集。值得注意的是,BDⅡ被注释的基因是3株菌中最多的,特别是碳水化合物代谢和氨基酸代谢途径富集基因最多;J4和J17注释的功能基因数目相近,功能相似。
3.9 CAZys分析
CAZys是根据蛋白质结构域中氨基酸序列的相似性分类的专业数据库,将CAZys分为糖苷水解酶(glycoside hydrolases,GHs)、糖基转移酶(glycosyltransferases,GTs)、碳水化合物酯酶(carbohydrate esterases,CEs)以及辅助氧化还原酶(auxiliary activities,AAs)等[30]。碳水化合物酶数据库注释结果如图8和表3,在图8-a中,J4共有33个GHs,分为16种GHs家族;共有28个GTs,分为9种GTs家族;共有16个CEs,分为7种CEs家族;共有10个AAs,分为5种AAs家族。由图8-b所示,J17共有33个GHs,分为16种GHs家族;共有29个GTs,分为9种GTs家族;共有16个CEs,分为7种CEs家族;共有9个AAs,分为5种AAs家族。由图8-b所示,BDⅡ共有36个GHs,分为16种GHs家族;共有31个GTs,分为10种GTs家族;共有16个CEs,分为7种CEs家族;共有9个AAs,分为5种AAs家族。其中,CEs家族主要有CE1、CE2、CE4、CE7、CE9、CE10、CE12;AAs家族主要有AA1、AA3、AA4、AA6、AA7;GTs家族主要有GT2、GT4、GT5、GT8、GT51、GT28、GT32、GT35、GT41、GT83;GHs家族主要有GH1、GH4、GH13、GH20、GH25、GH29、GH31、GH32、GH35、GH36、GH65、GH73、GH88、GH109、GH125、GH136;这些是3株菌共有的蛋白家族,GT51则是BDⅡ菌株特有的蛋白家族,注释GT51属于胞壁质聚合酶,与细菌细胞壁的合成密切相关。值得注意的是,在BDⅡ中GHs和GTs数量均高于另外2株副干酪乳酪杆菌,而仅BDⅡ在GTs家族中具有特有蛋白家族GT51。
表3 三株菌的碳水化合物酶家族分类情况表 单位:种
Table 3 Classification of carbohydrase families of the three strains of bacteria

菌株CEs家族GTs家族AAs家族GHs家族J479516J1779516BDⅡ710516

a-J4;b-J17;c-BDⅡ
图8 三株副干酪乳酪杆菌的CAZys注释统计图
Fig.8 CAZys annotation statistics for three strains of L. paracasei
3.10 蛋白酶基因比对结果与分析
LAB虽蛋白水解能力较弱,但蛋白水解体系完善,可通过降解外源蛋白质或肽得到氨基酸等营养物质供给自身生长。大多数LAB主要应用于发酵乳制品,在乳制品中蛋白水解方面研究较多,特别是酪蛋白[31]。LAB对酪蛋白的水解主要分为3部分,如胞外酶、转运系统和肽酶。胞外酶相关基因主要有PrtP、PrtB、PrtS、PrtH等。转运系统主要是DtpP和DtpT等转运二肽和三肽、Opp转运寡肽。肽酶主要是PepT等相关的酶基因[31]。
副干酪乳酪杆菌常作为附属发酵剂用于发酵食品的生产制作[32]。如电子版增强出版附表2所示,查找副干酪乳酪杆菌中主要报道的特征蛋白酶基因并进行序列比对以及通过3株菌的全基因组数据注释结果发现,基因数据中筛选到20个BDⅡ蛋白酶基因。20个BDⅡ蛋白酶基因包括19个比对值100%的基因和一个比对值99.87%的特有肽酶基因pepX,这些基因的表达对副干酪乳酪杆菌发挥蛋白水解作用具有十分重要的影响。
4 GT51和肽酶分析
通过对BDⅡ的蛋白酶基因进行总结,最终发现BDⅡ具有特有的肽酶基因pepX。因此对该特有肽酶进行同源建模,以详细观察肽酶的三维结构。如图9所示,通过Swiss Model网站对菌株BDⅡ特有肽酶的三维结构进行同源建模,形成相似度较高的三维模型。在图9-a中,以数据库中的B3 WEZ5.1.A模型为模板,预测模型为单体,模板和目的序列匹配度为100%,Coverage为100%,GMQE为0.97(可信度为0~1),因此该三维模型匹配度较好。在图9-b中,以数据库中的6nff.1.A模型为模板,预测模型为同源二聚体,模板和目的序列匹配度为39.36%(>30%),Coverage为98%,GMQE为0.77,QMEAN为0.76(区间-4~0,值越接近0,匹配度越好),判断该三维模型匹配度较好。

a-1号模型为单体;b-2号模型为同源二聚体
图9 肽酶pepX的三维蛋白结构
Fig.9 Three-dimensional protein structure of the peptidase pepX
通过Swiss Model网站对NCBI数据库的标准肽酶pepX(编号:EPC27423.1)的三维结构进行检索。将BDⅡ特有肽酶三维结构与数据库标准肽酶的三维结构进行比对,发现两者相似度极高。
如电子版增强出版附图1所示,在Boxshade网站上对阴性对照B6和BDⅡ的pepX对应氨基酸序列进行详细比对分析,发现有82%的相似性;根据背景色块分为3部分,黑色、灰色和无色,黑色背景为高度保守的氨基酸位点,灰色为较保守的氨基酸位点,特别关注灰色和无色背景,发现2株菌pepX对应氨基酸序列具有一定的差异。如电子版增强出版附图2所示,在Boxshade网站上对B6和BDⅡ的GT51氨基酸序列进行详细比对分析,发现有38%的相似性,发现2株菌GT51对应氨基酸序列差异较大。基于此,推测pepX和GT51对应氨基酸序列的差异可能导致其蛋白酶活力差异。
5 讨论
已有的关于副干酪乳酪杆菌的研究多关注于益生特点、安全性评价及系统发育分析,对其携带的蛋白水解基因研究较少。同时,不同菌株的基因组信息具有差异,因此需要将已知菌株与未知菌株的基因组信息进行比对,通过保守基因的比较推测菌株的基本功能,通过特有基因的比较推测菌株独特的功能特点。本研究对3株副干酪乳酪杆菌进行全基因组及比较基因组分析,发现J4和J17的功能基因差异性较小,BDⅡ的CDS和经注释的功能基因数量均最多,显著区别于其他2株菌,确是3株菌中蛋白酶活力最高的,表型分析与基因型推测结果相符。不同菌株的基因组数量、基因组大小、GC含量、CDS数量存在差异,但种间差异较小,与吕瑞瑞等[13]的研究结果一致。
3株菌中,蛋白酶活力实验分析表明,BDⅡ的蛋白酶活力最高。同时在功能基因分析中,COG注释结果表明,BDⅡ被注释的蛋白编码基因最多,特别是碳水化合物转运和代谢类别中蛋白基因富集最多;在KEGG通路中被注释的基因BDⅡ最多,特别是碳水化合物代谢和氨基酸代谢途径富集基因,因此BDⅡ的蛋白酶活力最高,蛋白水解能力最强,与XIANG等[33]的研究结果一致。此外,在KEGG代谢通路分析中,发现3株菌耐药性途径注释较多,与ZHAO等[34]发现的结果一致,因此推测BDⅡ具有一定的耐药性;还发现3株菌具有抗菌特性,与ISEPPI等[35]的发现一致。在CAZys分析中,发现BDⅡ与其他3株菌相比,具有一个特别的蛋白家族糖基转移酶GT51,该酶可调控蛋白质糖基化水平,间接参与蛋白质水解的调控过程;其次在细菌细胞壁生物合成中起关键作用并与抗生素的开发关系密切,长期以来研究者一致认为该酶是最有希望开发抗生素的[36],因此本课题组后期可针对该特点对BDⅡ进行进一步深入研究。
LAB的蛋白水解酶基因的表达是酪蛋白水解的主要原因。STRESSLER等[37]发现pepX和pepN可协同作用水解酪蛋白,并且对食品蛋白水解具有十分重要的意义。对3株菌的蛋白酶基因进行比对分析,发现BDⅡ拥有独特的肽酶pepX及糖基转移酶GT51。此外对BDⅡ和B6的pepX对应的氨基酸序列进行比对分析,发现两者间具有82%的相似性,具有一定差异;对BDⅡ和B6的GT51对应的氨基酸序列进行比对分析,发现两者间具有38%的相似性,具有较大差异;氨基酸序列的差异可能会导致功能的差异,推测pepX和GT51的表达是BDⅡ酪蛋白水解能力较强的原因[38]。本研究通过表型分析深入挖掘蛋白水解基因,为益生菌类乳制品功能物质产生机制的研究提供依据,因此具有十分重要的意义。但是该课题仍存在不足,没有对基因进行深入分析,并不了解具体发挥作用的基因及其是否真的能起到应有的作用,因此后期还需要进一步深入研究。
6 结论
随着科学技术的发展,高通量测序技术逐渐普遍,多物种已完成全基因组测序,从而可更好的将表型与基因型结合起来,了解菌株表现出某种特征的原因。本研究基于福林法、基因组学技术对副干酪乳酪杆菌的蛋白酶活力、蛋白酶基因进行分析,发现5株副干酪乳酪杆菌均可水解酪蛋白,其中有3株菌蛋白酶活力相对较高,BDⅡ蛋白酶活力最高,可能是其具有较多可编码蛋白水解酶的基因。此外,还发现同属不同种的菌株基因组大小不同,具有不同的功能。本研究通过表型分析深入挖掘蛋白水解基因,为益生菌类乳制品功能物质产生机制的研究提供依据。
[1] 张俊, 赵保堂, 杨富民.副干酪乳杆菌的益生特性及其应用研究进展[J].包装与食品机械, 2019, 37(4):47-52.ZHANG J, ZHAO B T, YANG F M.Review on the probiotic characteristics and application of Lactobacillus paracasei[J].Packaging and Food Machinery, 2019, 37(4):47-52.
[2] 刘凯龙, 康小红, 张哲, 等.副干酪乳杆菌PC-01益生特性和安全性研究[J].中国食品学报, 2021, 21(11):47-52.LIU K L, KANG X H, ZHANG Z, et al.Studies on probiotic properties and safety of Lactobacillus paracasei PC-01[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(11):47-52.
[3] 杨郑州, 侯保朝, 谢晓娜, 等.基于基因组学对副干酪乳杆菌BS 08黏附作用的分析[J].中国乳品工业, 2020, 48(8):10-14.YANG Z Z, HOU B C, XIE X N, et al.Adhesion analysis of Lactobacillus paracasei BS 08 based on genomics[J].China Dairy Industry, 2020, 48(8):10-14.
[4] 唐嘉琪, 唐霞, 张锋, 等.外源添加副干酪乳杆菌LZ9077对酸乳品质的影响[J].乳业科学与技术, 2023, 46(6):1-6.TANG J Q, TANG X, ZHANG F, et al.Effect of exogenous addition of Lactobacillus paracasei LZ9077 on properties of set-type yogurt[J].Journal of Dairy Science and Technology, 2023, 46(6):1-6.
[5] 陈森怡. 优良干酪用乳酸菌的筛选及新鲜干酪抗氧化活性的研究[D].上海:上海海洋大学, 2021.CHEN S Y.Screening of advantageous cheese-making lactic acid bacteria and study on antioxidant activity of fresh cheese[D].Shanghai:Shanghai Ocean University, 2021.
[6] LAW B A, KOLSTAD J.Proteolytic systems in lactic acid bacteria[J].Antonie Van Leeuwenhoek, 1983, 49(3):225-245.
[7] 李柏良, 丁秀云, 靳妲, 等.基于基因组学分析嗜热链球菌KLDS SM的蛋白质水解系统和氨基酸合成途径[J].食品科学, 2018, 39(18):120-126.LI B L, DING X Y, JIN D, et al.Genomic studies of proteolysis system and amino acid biosynthesis pathway in Streptococcus thermophilus KLDS SM[J].Food Science, 2018, 39(18):120-126.
[8] CAPLOVA Z, PANGALLO D, KRAKOVA L, et al.Detection of genes prtP, pepN, pepX and bcaT involved in formation of aroma-active compounds in lactic acid bacteria from ewes’ cheese[J].Journal of Food and Nutrition Research, 2018, 57(2):195-200.
[9] 杨淑娟, 周金萍, 李海燕, 等.基于比较基因组学解析植物乳植杆菌ST的功能基因组[J].食品科学, 2023, 44(14):86-93.YANG S J, ZHOU J P, LI H Y, et al.Comparative genomic analysis of functional genomics of Lactiplantibacillus plantarum ST[J].Food Science, 2023, 44(14):86-93.
[10] 刘慧敏. 乳酸菌嘌呤降解菌株的筛选、基因组特征及其应用研究[D].扬州:扬州大学, 2021.LIU H M.Screening, genomic characteristics and application of purine degrading lactic acid bacteria[D].Yangzhou:Yangzhou University, 2021.
[11] LI L, ZHANG L X, ZHANG T T, et al.(Meta)genomics-assisted screening of novel antibacterial lactic acid bacteria strains from traditional fermented milk from Western China and their bioprotective effects on cheese[J].LWT, 2023, 175:114507.
[12] HOSSAIN T J.Functional genomics of the lactic acid bacterium Limosilactobacillus fermentum LAB-1:Metabolic, probiotic and biotechnological perspectives[J].Heliyon, 2022, 8(11):e11412.
[13] 吕瑞瑞, 李伟程, 康小红, 等.副干酪乳杆菌PC-01全基因组测序及不同副干酪乳杆菌菌株比较基因组学分析[J].微生物学通报, 2021, 48(9):3025-3038.LYU R R, LI W C, KANG X H, et al.Whole genome sequencing of Lactobacillus paracasei PC-01 and comparative genomics analysis about Lactobacillus paracasei strains[J].Microbiology China, 2021, 48(9):3025-3038.
[14] 武朝霞. 嗜酸乳杆菌与地衣芽孢杆菌融合子制备及性能分析[D].太谷:山西农业大学, 2013.WU C X.Fusion and analysis of the fusant of Lactobacillus acidophilus and Lactobacillus bulgaricus [D].Taigu:Shanxi Agricultural University, 2013.
[15] 吴涵清, 洪青, 刘振民, 等.抗氧化乳酸菌的筛选及其对夸克奶酪品质的影响[J].食品与发酵工业, 2024,50(22):286-293.WU H Q, HONG Q, LIU Z M, et al.Screening of lactic acid bacteria with antioxidant capacity and its effect on the quality of quark cheese[J].Food and Fermentation Industries, 2024,50(22):286-293.
[16] 张婧, 吴影, 古绍彬, 等.凝结芽孢杆菌CGMCC 9951新型抗菌肽的挖掘、表达及活性测定[J].食品与发酵工业, 2022, 48(17):71-78.ZHANG J, WU Y, GU S B, et al.Genome mining, expression and activity determination of a novel antimicrobial peptide from Bacillus coagulans CGMCC 9951[J].Food and Fermentation Industries, 2022, 48(17):71-78.
[17] 肖宇, 刘洋, 刘建军, 等.1株高产几丁质脱乙酰酶红球菌的基因组测序及其应用潜力分析[J].食品科学, 2021, 42(18):157-163.XIAO Y, LIU Y, LIU J J, et al.Genomic analysis of a Rhodococcus strain with high chitin deacetylase activity and its application potential[J].Food Science, 2021, 42(18):157-163.
[18] 尹明华, 张艳红, 李淑娟, 等.广丰千金薯烟草花叶病毒lncRNA测序鉴定、原核蛋白表达及其序列分析[J].中草药, 2023, 54(11):3666-3675.YIN M H, ZHANG Y H, LI S J, et al.LncRNA sequencing identification, protein expression and sequence analysis of tobacco mosaic virus of Dioscorea polystachya Guangfeng Qianjin[J].Chinese Traditional and Herbal Drugs, 2023, 54(11):3666-3675.
[19] 杨瑞思. 五种不同乳杆菌基因组及益生效果的比较研究[D].昆明:昆明理工大学, 2024.YANG R S.Comparative study on genome and probiotic effects of five different Lactobacillus species[D].Kunming:Kunming University of Science and Technology, 2024.
[20] 李洪飞, 孙大庆, 曹龙奎.莱鲍迪甙C高效转化细菌Paenarthrobacter ilicis CR5301全基因组测序及关键糖苷酶分析[J].食品科学, 2022, 43(18):166-175.LI H F, SUN D Q, CAO L K.Whole genome sequencing and key glycosidase analysis of Paenarthrobacter ilicis CR5301, an efficient rebaudioside C-converting bacterium[J].Food Science, 2022, 43(18):166-175.
[21] 褚琪, 张艳芳, 周浩然, 等.L.plantarum SGJ-24全基因组测序及活性相关基因的挖掘和分析[J].食品工业科技, 2024, 45(14):131-138.CHU Q, ZHANG Y F, ZHOU H R, et al.Whole genome sequencing of L.plantarum SGJ-24 and mining and in-depth analysis of activity related genes[J].Science and Technology of Food Industry, 2024, 45(14):131-138.
[22] 王晓璇. 拮抗细菌Burkholderia seminalis R456的铁代谢及其调控机制研究[D].杭州:浙江大学, 2020.WANG X X.Study on iron metabolism of antagonistic bacteria Burkholderia seminalis R456 and its regulatory mechanism [D].Hangzhou:Zhejiang University, 2020.
[23] YUAN S G, STEPHEN CHAN H C, HU Z Q.Using PyMOL as a platform for computational drug design[J].Wiley Interdisciplinary Reviews:Computational Molecular Science, 2017, 7(2):e1298.
[24] 张传丽, 李同祥, 杨凡, 等.产淀粉酶和蛋白酶芽孢杆菌的筛选鉴定及其酶学性质分析[J].中国酿造, 2023, 42(12):110-116.ZHANG C L, LI T X, YANG F, et al.Screening and identification of amylase-and protease-producing Bacillus and their enzymatic property analysis[J].China Brewing, 2023, 42(12):110-116.
[25] 徐美余. 发酵中草药的制备及其饲喂对南丹瑶鸡的影响[D].昆明:昆明理工大学, 2023.XU M Y.Preparation of fermented Chinese herbs and its feeding on Nandan-Yao chickens [D].Kunming:Kunming University of Science and Technology, 2023.
[26] 许倩. 不同加工处理对牛乳蛋白抗原性及过敏原性的影响[D].北京:中国农业大学, 2017.XU Q.Effects of different processing ways on the antigenicity and allergenicity of main bovine milk proteins [D].Beijing:China Agricultural University, 2017.
[27] MA H, WANG L, YU H J, et al.Protease-producing lactic acid bacteria with antibacterial properties and their potential use in soybean meal fermentation[J].Chemical and Biological Technologies in Agriculture, 2022, 9(1):40.
[28] GHOSH S, SARANGI A N, MUKHERJEE M, et al.Reanalysis of Lactobacillus paracasei Lbs2 strain and large-scale comparative genomics places many strains into their correct taxonomic position[J].Microorganisms, 2019, 7(11):487.
[29] 田浩杰. 高温放线菌比较基因组学及其代谢初步研究[D].贵阳:贵州大学, 2023.TIAN H J.Comparative genomics of high-temperature actinomycetes and preliminary study of their metabolism [D].Guiyang:Guizhou University, 2023.
[30] 邹垚. 甘肃鼢鼠与罗氏鼢鼠肠道微生物功能特征及与土壤微生物关系研究[D].杨凌:西北农林科技大学, 2022.ZOU Y.Functional characteristics of intestinal microbes of Eospalax cansus and Eospalax rothschildi and their relationships with soil microbes[D].Yangling:Northwest A &F University, 2022.
[31] 杜越欧, 侯俊财.乳酸菌蛋白水解体系及相关基因表达的研究进展[J].食品工业科技, 2013, 34(3):383-386;391.DU Y O, HOU J C.Research progress in proteolysis system of lactic acid bacteria and related gene expression[J].Science and Technology of Food Industry, 2013, 34(3):383-386;391.
[32] YANG Y J, XIA Y J, LI C Y, et al.Metabolites, flavor profiles and ripening characteristics of Monascus-ripened cheese enhanced by Ligilactobacillus salivarius AR809 as adjunct culture[J].Food Chemistry, 2024, 436:137759.
[33] XIANG Q J, ZHAI J F, ZHANG M, et al.Detection of soluble expression and in vivo interactions of the inner membrane protein OppC using green fluorescent protein[J].Genetics and Molecular Research, 2015, 14(4):17834-17846.
[34] ZHAO X, HU R, LIU Y, et al.Genomics analysis of Lactobacillus paracasei SLP16[J].Letters in Applied Microbiology, 2022, 75(4):881-887.
[35] ISEPPI R, MESSI P, CAMELLINI S, et al.Bacteriocin activity of Lactobacillus brevis and Lactobacillus paracasei ssp.paracasei[J].Journal of Medical Microbiology, 2019, 68(9):1359-1366.
[36] YANG M, ZHOU L, ZUO Z, et al.Structure-based virtual screening for glycosyltransferase51[J].Molecular Simulation, 2008, 34(9):849-856.
[37] STRESSLER T, EISELE T, SCHLAYER M, et al.Characterization of the recombinant exopeptidases PepX and PepN from Lactobacillus helveticus ATCC 12046 important for food protein hydrolysis[J].PLoS One, 2013, 8(7):e70055.
[38] 凌莉, 任展宏, 王歆.不同物种肌动蛋白聚合功能及其差异分析[J].中国生物化学与分子生物学报, 2024, 40(9):1240-1249.LING L, REN Z H, WANG X.Functional analysis of actin polymerization in different species[J].Chinese Journal of Biochemistry and Molecular Biology, 2024, 40(9):1240-1249.