HMG-CoA 还原酶是生物合成甲戊二酸过程中最重要的限速酶,同时 HMG-CoA 还原酶是生物医学领域作为降低胆固醇药物的重要靶点,通过抑制 HMG-CoA 还原酶能够有效抑制人体内胆固醇的生成从而降低人体胆固醇的含量[1-3],达到预防以及治疗高胆固醇血症的目的。HERMANN等[4]和 GOLOMB等[5]发现他汀类药物目前虽然是治疗高胆固醇血症的主要途径,但长期服用会有严重副作用如横纹肌溶解和肌痛。他汀类药物对 HMG-CoA 还原酶竞争性抑制仍是降低人体胆固醇合成的主要途径,目前国内外研究人员已经从天然食物蛋白质的水提物中获取同样能够抑制 HMG-CoA 还原酶活性的生物活性肽,从而抑制胆固醇的生物合成[6]。
生物活性肽存在于蛋白质序列中,经酶解和微生物发酵等方式得以释放[7]。肽的功能活性与氨基酸的组成具有一定关系,同时,干酪蛋白质的降解会产生大量苦味肽,研究表明肽的苦味源自于 Leu、Gln、Ile 和 Pro 等疏水性氨基酸,这些疏水性氨基酸又与肽的生物活性密切相关[8]。牦牛乳干酪是高原地区居民传统食物,成熟时间需6个月至2年,易产生大量肽序列。本课题组前期研究牦牛乳硬质干酪分离鉴定出的14种苦味肽中具有抗菌活性[9-10]、抗糖活性[11]、抗氧化活性[12]和血管紧张素转换酶(angiotension converting enzyme, ACE)抑制活性[13],然而牦牛乳干酪中多肽对HMG-CoA 还原酶抑制活性大小及抑制作用机理尚处于空白。
结合 PeptideDB、ChemDraw和Biopep-UWM 数据库等生物信息学平台及工具可以高效准确地预测肽的生物活性。通过分子对接和分子动力学软件技术分析肽与受体蛋白之间的相互作用,阐明生物活性抑制机制。通过 Biopep-UWM 数据库中已知HMG-CoA 还原酶抑制肽进行比对,为继续完善 Biopep 数据库中HMG-CoA还原酶抑制肽数据奠定基础。
本试验利用课题组前期鉴定的牦牛乳干酪苦味肽RK7和KQ7,通过生物信息学平台及工具研究理化性质和序列特征,结合分子对接和分子动力学表述HMG-CoA 还原酶抑制肽的作用机制,并进行体外活性试验,为HMG-CoA 还原酶抑制肽的作用机制提供理论基础。
1 材料与方法
1.1 材料与试剂
RK7、KQ7,生工生物工程(上海)股份有限公司;猪乙酰辅酶A还原酶、ELISA试剂盒,江苏晶美生物科技有限公司;阿托伐他汀,齐鲁制药(海南)有限公司。
1.2 仪器与设备
Discovery Studio Client v16.1.0(DS), Dassault Systèmes公司;ChemBioDraw Ultra 11.0(CD),PerkinElmer公司;Versa Max酶标仪,Molecular Devices公司;VM-500S涡旋混合器,群安试验仪器有限公司;PHS-3C pH 计,上海仪电科学仪器股份有限公司;FR224CN电子分析天平,奥豪斯仪器有限公司;HWS26恒温水浴锅,上海一恒科技有限公司。
1.3 试验方法
1.3.1 苦味肽RK7和KQ7理化性质
运用 Peptide Property Calculator 平台测定 RK7和KQ7的等电点(isoelectric point,PI),净电荷及水溶性;ExPASy ProtParam平台测定2种肽的不稳定指数;PEPTIDE 2.0CE 测定2种肽的疏水性[14]。
1.3.2 配体和受体的结构优化及处理
运用ChemDraw Professional 20.0 软件绘制RK7、KQ7、阿托伐他汀、辛伐他汀、瑞舒伐他汀和普伐他汀的分子结构式,请见电子版增强出版附图1(https://doi.org/10.13995/j.cnki.11-1802/ts.040007)。在DS中的 Small molecules 模式下进行 Prepare Ligands 处理产生多构体,随后在 CHARMM 力场下进行能量最小化处理,以便后期与受体进行分子对接。
HMG-CoA 还原酶三维结构图请见电子版增强出版附图2。HMG-CoA 还原酶在 PDB 数据库下载(PDB ID:1HWI,分辨率为2.3 Å)蛋白质晶体X射线衍射三维结构[15]。使用 Discovery Studio 软件将下载蛋白文件打开,去除3D结构中的水分子和无关配体,并用 Hydorgens 做加氢处理,使用 Clean Protein 去除蛋白质多余配体构象,补充完整的氨基酸结构。
1.3.3 LibDock 分子对接及Biopep 数据库比对
在 DS 软件中打开 Dock Ligands(LibDock)模式,阿托伐他汀、辛伐他汀、瑞舒伐他汀和普伐他汀为阳性对照;将 RK7、KQ7 与 HMG-CoA 还原酶(PDB:1HWI)进行 Dock Ligands(LibDock)分子对接,对接成功后会生成对接分数、结合能以及对接位点,从而确定肽和对照药品活性位点的最佳姿态;在 DS 软件对接成功生成的2D 图中也可以直观地看到肽结合在配体上的氨基酸残基以及分子间的相互作用力如范德华力、氢键和疏水相互作用等。
通过对 Biopep 数据库比对与分子对接结果相结合,将苦味肽 RK7 和 KQ7与 Biopep 数据库中 HMG-CoA 还原酶抑制肽做比对[16],通过对数据库中已有的抑制肽序列与RK7和KQ7具有相同抑制作用的肽序列比对后预测RK7和KQ7抑制还原酶活性。
1.3.4 GROMACS分子动力学模拟
本次分子动力学试验采用Gromacs 2022.3 版本软件进行[17-18]。将RK7和KQ7及HMG-CoA还原酶结构文件导入Gromaceb并生成拓扑文件和模拟盒子,随后进行能量最小化处理,最后运行自由分子动力学模拟。该过程共 5 000 000 步,步长为2 fs,共计时长100 ns。在完成模拟后,采用软件分析轨迹,计算各氨基酸运动轨迹的均方根方差(root mean square deviation,RMSD)、均方根波动值(root mean square fluctuation,RMSF)和氢键(hydrogen bond,HBond)。
1.3.5 肽RK7、KQ7的合成及HMG-CoA还原酶抑制活性测定
试验采用固相合成法对肽RK7、KQ7合成,LC-MS分析肽的纯度和分子质量。此试验过程由生工生物公司(上海)股份有限公司完成。
HMG-CoA还原酶抑制活性使用分光光度法测定,并根据公式(1)计算HMG-CoA还原酶抑制率:
HMG-CoA还原酶抑制率![]()
(1)
式中:ODs,样品组吸光值;ODsb,样品空白组吸光值;ODc,对照组吸光值;ODb,空白组吸光值。
1.3.6 数据处理
试验样品每组重复3次,使用SPSS软件进行数据处理,使用Origin 2018及Graphpad软件作图。
2 结果与分析
2.1 RK7和KQ7理化性质预测
通过课题组前期试验可知,苦味肽 RPKHPIK(RK7)的分子质量为 875.07 Da,疏水性氨基酸比例为 42.86%;苦味肽 KVLPVPQ(KQ7)的分子质量为 779.98 Da,疏水性氨基酸比例为71.42%[10-11]。
RK7和KQ7 2种多肽对HMG-CoA还原酶的抑制活性受其理化性质的影响。RK7和KQ7分别含有42.86%和71.42%的疏水性氨基酸,这些疏水性氨基酸在与HMG-CoA还原酶结合的过程中起到了关键作用。研究表明,疏水性氨基酸如Ala、Gly、Ile、Leu和Pro在与HMG-CoA还原酶结合时,通过疏水作用和氢键稳定了复合物的构象,使得酶的活性受到抑制[19]。
2.2 分子对接分析
2.2.1 分子对接研究肽 RK7、KQ7对 HMG-CoA还原酶抑制机制
运用程序LibDock进行分子对接并生成RK7和HMG-CoA还原酶复合物3D及2D相互作用图见电子版增强出版附图3。RK7和HMG-CoA还原酶氨基酸残基Leu521(4.59 Å)、Ser520(5.12 Å)、Met523(5.36、5.47 Å)、Asn658(4.88 Å)、Ala525(3.14 Å)、Gly560(4.14 Å)、Gly807(3.48 Å)、Met655(3.24 Å)、Gln766(5.86 Å)形成氢键,其中Met523与HMG-CoA还原酶形成了2个氢键,键长为5.47 Å和5.36 Å,和Gly807及Met655键长最短,结合紧密;RK7与氨基酸残基Met659(4.67 Å)和Cys561(4.38 Å)形成疏水作用力的Pi-Alkyl 键。与Val522、Lys662、Ala654、Met657、Ile660、Thr804、Gly656、Thr809、Ile802、Val806、Gly765、Gly808、Asn810、Ala768、Thr557、Glu528形成范德华力;Glu559(3.89、4.45、4.64 Å)和Asp767(3.52 Å)形成静电相互作用。RK7与 氨基酸残基Gly524(4.68 Å)、Thr558(3.53 Å)、Gly803(2.95 Å)和 Gly806(4.85 Å)形成碳氢键。RK7通过范德华力、氢键等强相互作用力与 HMG-CoA还原酶紧密结合在一起。TOPPO等[20]研究发现,配体与受体蛋白之间的相互作用中氢键起到重要作用,能更加稳定复合物的构象,同时疏水相互作用、静电相互作用和范德华力在配体与受体蛋白相互作用中同样能够有效促进配体受体复合物的结合,从而达到降低酶活性的目的。
KQ7与 HMG-CoA还原酶对接构象图见电子版增强出版附图4。KQ7与HMG-CoA还原酶的Asn658(2.81、3.85 Å)、Ser626(4.24 Å)和Ala556(4.68、5.24 Å)形成氢键,其中Asn658和Ala556各自分别形成2个氢键,Asn658键长为2.81 Å和3.85 Å,Ala556键长为4.68 Å和5.24 Å,在KQ7和HMG-CoA还原酶的结合中起重要作用;KQ7与HMG-CoA还原酶氨基酸残基Lys662(3.76 Å)、Glu559(2.43 Å)和Asp767(4.03、6.87 Å)形成静电相互作用;与氨基酸残基Gly806(3.69 Å)、Gly807(3.38 Å)及Asp767(3.45、3.99 Å)形成碳氢键;与Met655(3.53、5.59 Å)形成疏水相互作用的Pi-Alkyl 键;Asn810、Gly656、Ala654、Gly803、Gly765、Thr809、Ile802、Val805、Gly808、Gln766、Thr558、Ala769、Asn755、Ala759、Ala768、Thr557、Thr758、Leu562、Ile762、Lys691、Gly560、Met659和Met657形成范德华力。MEDINA-FRANCO等[21]通过对α-花生四烯酸酯降胆固醇的研究中,Lys691和Glu559是抑制 HMG-CoA 还原酶重要的氨基酸。有研究表明,HMG-CoA 还原酶催化部分由 Cys688、Thr689、Asp690 和 Lys691 组成,Glu559和 Asp767 位于活性位点的前部,在研究中起到重要抑制作用[22]。
2.2.2 分子对接研究阳性对照药物对 HMG-CoA还原酶抑制机制
阿托伐他汀对 HMG-CoA 还原酶分子对接构象3D/2D图见电子版增强出版附图5。阿托伐他汀与 HMG-CoA 还原酶的氨基酸残基 Lys692(5.73 Å)、Lys735(5.54 Å)和 Arg590(6.35 Å)形成氢键;阿托伐他汀与氨基酸残基 Glu665(7.82 Å)、Leu862(6.04 Å)、Arg590(5.43 Å)、Lys692(6.51 Å)和 Lys735(7.11 Å)形成静电相互作用;与氨基酸残基 His861、Gly560、Leu562、Ala751、Asn686、Ser684、Cys688、Leu587、Lys691、Asn755、Asn658、Ser661 和Lys662 形成范德华力;与氨基酸残基 Ser565(3.80 Å)、His752(5.70 Å)、Asp690(4.50 Å)和 Glu559(5.48 Å)形成碳氢键;与氨基酸残基 Ala856(6.80 Å)、Leu853(5.33 Å)和 Met657(5.15 Å)形成疏水相互作用。阿托伐他汀通过与 HMG-CoA 还原酶氨基酸残基相互作用达到抑制作用。
辛伐他汀对 HMG-CoA还原酶分子对接构象3D/2D图见电子版增强出版附图6。辛伐他汀与HMG-CoA还原酶的氨基酸残基Arg590(5.99 Å)、Lys692(5.35 Å)和 Lsy735(5.91 Å)形成氢键;与 Ser684(3.95 Å)形成碳氢键;与 Leu862(4.39 Å)、Leu857(5.62 Å)、Val683(3.78、5.56 Å)和 Leu853(4.39 Å)形成疏水作用力中的 Pi-Alkyl 键;与氨基酸残基 Glu665、Ser661、Asn658、Met657、Gly560、Glu559、Cys561、Leu562、Ser565、Asn755、His752、Lys691、Ala751、Asp690 和 Asn686 形成范德华力。辛伐他汀通过与 HMG-CoA 还原酶分子对接作用机制可知与 HMG-CoA 还原酶氨基酸残基作用从而达到抑制作用。
瑞舒伐他汀对 HMG-CoA 还原酶分子对接构象3D/2D图见电子版增强出版附图7。瑞舒伐他汀与 HMG-CoA 还原酶氨基酸残基 Gly803(3.86 Å)、Gly765(4.21 Å)、Gln766(3.36 Å)、Asn658(4.48、4.87 Å)、Met655(3.76 Å)、Val805(4.68 Å)和 Asp767(3.35 Å)形成氢键,其中 Asn658 与瑞舒伐他汀形成2个氢键;与氨基酸残基 Ile802(4.42 Å)、Gln766(4.46 Å)和Asn658(4.03 Å)形成碳氢键;与 Ala654(6.91 Å)形成疏水作用力中的 Pi-Alkyl 键;与氨基酸残基 Glu801、Thr809、Gly656、Met657、Met659、Ala525、Cys526、Gly807、Gly808、Lys691、Gly806 和 Gln770 形成范德华力。
普伐他汀对HMG-CoA 还原酶分子对接构象3D/2D图见电子版增强出版附图8。普伐他汀与 HMG-CoA 还原酶的氨基酸残基 Ala769(3.98 Å)、Ala768(3.64 Å)、Gln766(5.82 Å)、Thr558(4.14 Å)和 Asn658(4.37 Å)形成5个不同距离的氢键,其中与氨基酸 Ala768形成的氢键距离最短,相互作用的最紧密;与氨基酸残基 Gly807(3.66、3.88 Å)和 Thr558(4.05 Å)形成碳氢键;普伐他汀与氨基酸残基 Met655(3.86、4.57、4.13 Å)和 Ala654(4.22 Å)形成疏水作用中的 Pi-Alkyl 键;普伐他汀与氨基酸残基 Gln770、Gly765、Ile802、Thr809、Gly803、Thr804、Val805、Met659、Gly806、Met657、Gly560、Ala525、Cys526、Thr557、Thr758和 Gly808形成范德华力。本实验通过对照药物的分子对接结果显示在氢键、碳氢键、疏水相互作用及范德华力的相互作用下,有效的抑制了HMG-CoA 还原酶的活性。
抑制 HMG-CoA 还原酶活性的强弱与肽链中部分特殊氨基酸相关,例如氨基酸残基中包含Glu528、Glu665、Asn686、Lys692、Lys735、Ile531、Met534和 Tyr644这些氨基酸残基会位于配体分子与蛋白复合物中的特殊位点,其大小、形状和距离会对蛋白质活性位点的结合产生影响[23]。本实验中牦牛乳硬质干酪肽 RK7和 KQ7中 RK7与 HMG-CoA 还原酶最佳对接构象的结果显示出较高的 LibDock 对接分数为188.726,KQ7最佳对接构象的 LibDock 分数为183.437。多数配体都能在结构域和疏水域与受体蛋白结合,氢键、碳氢键等疏水相互作用会其使锚定在活性位点上,所以 KQ7 与 HMG-CoA 还原酶有更强的结合力。Met、Leu和Cys 是疏水性氨基酸,它们可以与肽和受体蛋白复合物之间形成相互作用,在一定范围内,疏水性氨基酸越多,抑制活性也越强[24]。
2.2.3 分子KQ7、RK7及他汀类药物抑制 HMG-CoA 还原酶的氨基酸及相互作用力分析
分子对接分析显示,RK7和KQ7与HMG-CoA还原酶之间形成了多种相互作用,包括氢键、疏水作用、静电相互作用和范德华力。这些相互作用共同决定了肽的结合力和稳定性。例如,RK7通过氢键与HMG-CoA还原酶的残基形成紧密结合,而KQ7则由于其更高的疏水性,在疏水相互作用中表现出更强的结合力。氢键是抑制 HMG-CoA 还原酶活性强弱的重要因素之一,氢键的数量以及键长对维持肽-酶复合物活性结构至关重要[25]。如表1所示,RK7、KQ7、阿托伐他汀、辛伐他汀、瑞舒伐他汀及普伐他汀分别与 HMG-CoA 还原酶形成13、6、7、4、10、7个氢键,其中 KQ7与 Ala556、Asn658和Asp767各形成2个作用力较强的氢键,距离分别为4.68、5.24、2.81、3.85、3.45、3.99 Å,同时 KQ7 与氨基酸残基 Asp767 即形成2个碳氢键同时形成2个静电相互作用,与 Glu559形成1个静电相互作用。本实验中KQ7同样与Glu559和 Asp767相互作用并且 Asp767与 KQ7相互作用力更强。RK7 与 HMG-CoA 还原酶的氨基酸残基 Met523形成2个氢键,与 Glu559和 Asp767形成静电相互作用,其中 RK7 与氨基酸残基 Glu559形成3个静电相互作用,距离为3.89、4.45、4.64 Å,同时与 Cys561形成疏水相互作用,Met657和 Glu528形成范德华力,使 RK7 与 HMG-CoA 还原酶结合更加紧密,RK7和KQ7与羽扇豆肽[26](LILPKHSDAD和LTFPGSAED)对HMG-CoA 还原酶的效果非常相似,具有很好的抑制效果。
表1 HMG-CoA还原酶与RK7、KQ7氨基酸结合对照表
Table 1 HMG-CoA reductase and RK7, KQ7 amino acid binding control

编号分子间相互作用力RK7KQ71氢键Leu521(1),Ser520(1),Met523(2),Asn658(1),Ala525(1),Gly560(1),Gly807(1),Met655(1),Gln766(1),Gly524(1),Thr558(1),Gly803(1),Gly806(1)Ala556(2),Asn658(2),Ser626(1),Gly806(1),Gly807(1),Asp767(2)2疏水相互作用Met659(1)Cys561(1)Met655(2)
续表1
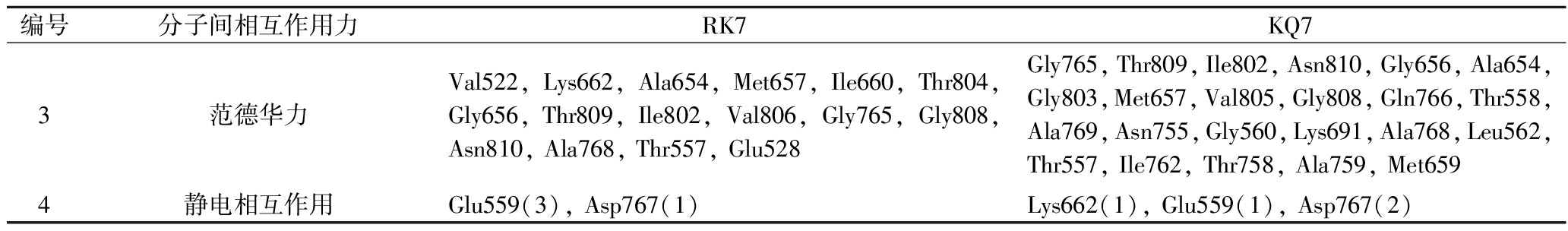
编号分子间相互作用力RK7KQ73范德华力Val522,Lys662,Ala654,Met657,Ile660,Thr804,Gly656,Thr809,Ile802,Val806,Gly765,Gly808,Asn810, Ala768, Thr557, Glu528Gly765,Thr809,Ile802,Asn810,Gly656,Ala654,Gly803,Met657,Val805,Gly808,Gln766,Thr558,Ala769,Asn755,Gly560,Lys691,Ala768,Leu562,Thr557, Ile762, Thr758, Ala759, Met6594静电相互作用Glu559(3), Asp767(1)Lys662(1), Glu559(1), Asp767(2)
注:Ala-丙氨酸、Cys-半胱氨酸、Asp-天冬氨酸、Glu-谷氨酸、Phe-苯丙氨酸、Gly-甘氨酸、His-组氨酸、Ile-异亮氨酸、Lys-赖氨酸、Leu-亮氨酸、Met-甲硫氨酸、Asn-天冬酰胺、Pro-脯氨酸、Gln-谷氨酰胺、Arg-精氨酸、Ser-丝氨酸、Thr-苏氨酸、Val-缬氨酸、Trp-色氨酸、Tyr-酪氨酸。
他汀类药物主要是降高胆固醇的药物,它们通常用于治疗高胆固醇血症、血脂异常和冠心病等心血管疾病。所有他汀类药物都具有3-羟基-3-甲基戊二酰(3-hydroxy-3-methylglutaryl,HMG)样部分,该部分与 HMG-CoA 还原酶具有高亲和力,因此空间阻断该酶,从而抑制甲羟戊酸的产生,这是胆固醇生物合成的第一步。如表2所示,他汀类药物中阿托伐他汀与 HMG-CoA 还原酶氨基酸残基形成7个氢键,同时 Lys692、Lys735、Arg590、Ser565、Asp690和 Glu559对 HMG-CoA 还原酶的抑制起到重要作用,阿托伐他汀与氨基酸残基 Met657形成疏水相互作用,与 Glu665、Arg590、Lys692和 Lys735重要氨基酸形成静电相互作用使得阿托伐他汀与HMG-CoA 还原酶结合的更加紧密。同时 KQ7 与阿托伐他汀具有相同的Asp767、Met657、Lys691和 Glu559关键氨基酸,因此KQ7相较RK7具有更强的抑制 HMG-CoA 还原酶的活性,但二者均低于阿托伐他汀对 HMG-CoA 还原酶抑制活性。辛伐他汀与重要氨基酸残基Arg590、Lys692和 Ser684形成氢键,与 Leu857、Met657、Cys561、Glu559、Ser565、Lys691、Asp690和Asn686形成疏水作用力和范德华力,对辛伐他汀抑制 HMG-CoA 还原酶活性至关重要,通过分子对接结果可知辛伐他汀与 HMG-CoA 还原酶氨基酸残基相互作用力之间没有静电相互作用。瑞舒伐他汀与 HMG-CoA 还原酶重要氨基酸残基 Asp767 形成氢键,与 Met657和 Lys691形成范德华力,参与抑制 HMG-CoA 还原酶活性的重要氨基酸明显少于阿托伐他汀与辛伐他汀,并且在分子对接结果中同样没有静电相互作用。普伐他汀与HMG-CoA 还原酶重要氨基酸残基 Met657形成范德华力,并且在对接结果中同样没有静电相互作用。疏水相互作用是肽-复合物中结合能的主要贡献者,因此是抑制 HMG-CoA 还原酶活性的主要驱动力[27],同时在相互作用中含有疏水性氨基酸 Ala、Gly、Ile、Leu和 Pro 会更加有利与肽对 HMG-CoA 还原酶活性的抑制[28]。通过分子对接实验得知阿托伐他汀在所有阳性对照样品中对 HMG-CoA 还原酶的抑制是最有效的,与 KARTHIK等[29]实验中他汀类药物对 HMG-CoA 还原酶作用结果相一致。
表2 HMG-CoA还原酶与阿托伐他汀、辛伐他汀、瑞舒伐他汀和普伐他汀氨基酸结合对照表
Table 2 Amino acid binding control of HMG-CoA reductase with atorvastatin, simvastatin, resuvastatin, and pravastatin

编号分子间相互作用阿托伐他汀辛伐他汀瑞舒伐他汀普伐他汀1氢键Lys692(1),Lys735(1),Arg590(1),Ser565(1),His752(1),Asp690(1),Glu559(1)Arg590(1),Lys692(1),Lsy735(1), Ser684(1)Gly803(1),Gly765(1),Gln766(1),Asn658(1),Met655(1),Val805(1),Asp767(1),Ile802(1),Gln766(1), Asn658(2)Ala769(1),Ala768(1),Gln766(1),Thr558(1),Asn658(1),Gly807(2),Thr558(1)2疏水相互作用Ala856(1),Leu853(1),Met657(1)Leu862(1),Leu857(1),Val683(2), Leu853(1)Ala654(1)Met655(3),Ala654(1)3范德华力His861,Gly560,Leu562,Ala751,Asn686,Ser684,Cys688,Leu587,Lys691,Asn755,Asn658,Ser661,Lys662Glu665,Ser661,Asn658,Met657,Gly560,Glu559,Cys561,Leu562,Ser565,Asn755,His752,Lys691,Ala751, Asp690, Asn686Glu801,Thr809,Gly656,Met657,Met659,Ala525,Cys526,Gly807,Gly808,Lys691, Gly806Gln770,Gly765,Ile802,Thr809,Gly803,Thr804,Val805,Met659,Gly806,Met657,Gly560,Ala525,Cys526,Thr557,Thr758,Gly8084静电相互作用Glu665(1),Leu862(1),Arg590(1),Lys692(1),Lys735(1)---
注:-表示无氨基酸残基具有该相互作用力。
2.2.4 肽KQ7与 RK7在 BIOPEP 数据库中的比对
BIOPEP数据库是目前最新颖全面的生物活性肽数据库[30],截至2022年11月,BIOPEP数据库已经记载了4 518种已知功能活性的肽段,其中抑制HMG-CoA还原酶活性的肽段数据库中共有28种,将KQ7和RK7与BIOPEP数据库中28种抑制HMG-CoA还原酶活性的肽段进行比对后发现,KQ7与已知的肽段LPYP(ID:9605)最高相似度达75%,RK7中均不含BIOPEP数据库中已知抑制HMG-CoA还原酶活性的肽片段,但通过前期分子对接结果表明,苦味肽RK7可以与HMG-CoA还原酶相结合从而达到抑制作用,所以预测RK7为新型抑制HMG-CoA还原酶活性肽片段。
2.3 分子动力学分析肽RK7和KQ7与HMG-CoA 还原酶复合物的稳定性
2.3.1 肽RK7和KQ7与HMG-CoA 还原酶复合物RMSD和RMSF模拟轨迹分析
RMSD是评估蛋白质结构变化的重要指标。本实验通过在 100 ns 的分子动力学模拟过程中分析HMG-CoA还原酶与肽RK7、KQ7复合物分子动力学轨迹,研究肽RK7、KQ7与HMG-CoA还原酶复合物的稳定性及动力学性质。计算了每个系统中所有主链原子相对于初始结构的RMSD随着时间的变化情况。详见电子版增强出版附图9、10,在整个模拟过程中,每个蛋白质的主链原子 RMSD 值均保持在较低的水平,RK7与HMG-CoA还原酶复合物在80 ns左右趋于稳定,RMSD值在0.2~0.25 nm波动。KQ7与HMG-CoA还原酶复合物在20 ns左右达到稳定,RMSD值在0.21~0.25 nm波动,波动浮动极小,说明体系处于平衡状态。
RMSF为原子位置变化对于时间的平均,可以表征蛋白质氨基酸在整个模拟过程中的柔性和运动剧烈程度。蛋白的 RMSF 被认为是确定每个残基与参考位置的偏差。这个参数决定了配体-蛋白质相互作用在模拟时间的适用性。远离活性位点的氨基酸会表现出较高的RMSF值。同时,RMSF 和蛋白柔性也有关,越是柔性大的区域,RMSF 值越大。HMG-CoA还原酶是一个同源四聚体,共有4个相同的亚基。见电子版增强出版附图11,在整个RMSF模拟过程中,波动值基本稳定在0.2 nm左右,这说明RK7与HMG-CoA还原酶在结合过程中基本保持稳定状态且柔性小保持稳定。KQ7与HMG-CoA还原酶RMSF见电子版增强出版附图12,虽然两边表现出的柔性大,这应该与底物活性位点位置的改变有关,但是整体复合物的RMSF值保持稳定,平均波动范围<0.2 nm,表明KQ7与HMG-CoA还原酶在结合过程中基本保持结构的稳定。
分子动力学模拟RMSD与RMSF进一步证明了这2种肽与HMG-CoA还原酶复合物的稳定性。在模拟过程中,RK7复合物在80 ns左右趋于稳定,而KQ7复合物在20 ns左右就达到了稳定状态,显示出更快的结合速度和稳定性。这可能是由于KQ7中较高的疏水性氨基酸比例,使其能够更迅速地形成稳定的结合。
2.3.2 肽RK7和KQ7与HMG-CoA 还原酶复合物氢键轨迹分析
氢键(hydrogen bond, HBond)是评估蛋白质与小分子之间氢键的指标,此外氨基酸之间的氢键可以改变HMG-CoA还原酶内部的几何结构,由此计算了HMG-CoA还原酶和RK7、KQ7形成复合物的氢键数目。在本次模拟中,试验计算了HMG-CoA还原酶与肽RK7和KQ7之间的HBond,并绘制了HBond 图表(见电子版增强出版附图13、14)。结果显示,在模拟的过程中,HMG-CoA还原酶与肽RK7之间形成了许多氢键,其中以HMG-COA还原酶中的一些关键残基和小分子中的一些重要基团之间的氢键为主,且随模拟时间进行,RK7和KQ7在80~100 ns,90~100 ns达到数量最多,这与分子对接数据相吻合,使RK7和KQ7与HMG-COA还原酶在整个对接过程中有较好的结构稳定性。
2.4 肽RK7和KQ7的合成与HMG-CoA 还原酶活性验证
2.4.1 肽RK7和KQ7的合成
肽RK7和KQ7的HPLC-LC/MS图见电子版增强出版附图15。RK7理论相对分子质量875.06 Da,实际测得相对分子质量为874.90 Da;KQ7理论相对分子质量为779.96 Da,实际测得相对分子质量为779.50 Da,两者分子质量均与理论值接近,RK7和KQ7纯度分别为98.07%和99.76%,合成结果达到预期标准。将合成的牦牛乳干酪苦味肽RK7、KQ7用于HMG-CoA 还原酶活性验证。
2.4.2 肽RK7和KQ7体外活性验证
通过紫外分光光度法对苦味肽RK7和KQ7进行HMG-CoA还原酶抑制活性的测定,实验结果见电子版增强出版附图16。RK7和KQ7与HMG-CoA还原酶抑制率随浓度的增加而增加。阿托伐他汀的HMG-CoA还原酶抑制率曲线为y=3.302 7x2+40.075x+15.088,R2=0.996 9,质量浓度在0.2~1.0 mg/mL范围变化时,阿托伐他汀的抑制率为22.72%~58.77%。肽RK7的HMG-CoA还原酶抑制率曲线为y=9.283 7x2+36.271x+6.798 1, R2=0.988 1, 肽质量浓度在0.2~1.0 mg/mL时,RK7的HMG-CoA还原酶抑制率为14.82%~51.37%,KQ7的HMG-CoA还原酶抑制率曲线为y=0.246 5x2+44.75x+3.285,R2=0.996 7,肽质量浓度在0.2~1.0 mg/mL时,KQ7的HMG-CoA还原酶抑制率为11.71%~48.58%。
RK7和KQ7在所有浓度下的HMG-CoA还原酶抑制率均显著高于对照组(P<0.05)。根据抑制率曲线得知,RK7与KQ7的IC50分别为1.045和1.228 mg/mL,阿托伐他汀为0.79 mg/mL。RK7活性强于KQ7,这与分子对接结果一致,但两者均低于阿托伐他汀的HMG-CoA还原酶抑制活性。体外活性验证试验进一步证明RK7和KQ7对HMG-CoA还原酶均具有良好的抑制作用,且HMG-CoA还原酶抑制效果随浓度的增加而增强。不同的氨基酸组成和肽序列影响肽的生物活性,疏水性氨基酸对HMG-CoA还原酶抑制活性有较大影响[31]。由于RK7与HMG-CoA还原酶的重要氨基酸结合数相对较多,因此,RK7相较于KQ7具有更高的抑制活性。
相比现有的他汀类药物,RK7和KQ7在分子对接和动力学模拟中的表现同样突出。阿托伐他汀等他汀类药物通过多种相互作用抑制HMG-CoA还原酶,显示了较高的抑制活性。与之相比,KQ7由于其高疏水性和快速稳定的结合状态,显示出强于RK7的潜在抑制活性。然而,体外实验结果显示,RK7的IC50值略优于KQ7,这可能与其在结合过程中形成的更稳定的氢键网络有关。RK7和KQ7通过与HMG-CoA还原酶的紧密结合,干扰了酶的正常功能,阻止了胆固醇的合成。这种抑制作用主要依赖于肽与酶之间的疏水作用和氢键形成。此外,静电相互作用和范德华力也在稳定复合物中起到了重要作用。这些发现为未来开发基于肽的HMG-CoA还原酶抑制剂提供了新的思路。
3 结论
本实验通过生物信息学及体外验证实验探讨了牦牛乳硬质干酪苦味肽 RK7 和KQ7 对 HMG-CoA 还原酶抑制活性作用机制。RK7 和KQ7所含的疏水性氨基酸 Ala、Gly、Ile、Leu和 Pro有利于抑制 HMG-CoA 还原酶活性。分子对接的结果显示出2种肽对 HMG-CoA 还原酶抑制活性:KQ7>RK7;分子动力学通过对HMG-CoA 还原酶与KQ7和RK7模拟中的RMSD,RMSF和氢键分析进一步判断HMG-CoA 还原酶与RK7 和KQ7之间的结合是否稳定。综合上述结果,认为HMG-CoA 还原酶与RK7 和KQ7之间的相互作用稳定,并且RK7 和KQ7与HMG-CoA 还原酶结合的更加紧凑,HMG-CoA 还原酶与多肽之间形成了许多氢键,这使得结合过程中复合物的结构更加稳定;目前4种市售降低胆固醇的药物与HMG-CoA 还原酶进行分子对接并分析得出阿托伐他汀抑制 HMG-CoA 还原酶中所含重要分子间的相互作用力及氨基酸数量最多且复合物构像稳定,对HMG-CoA 还原酶抑制活性:阿托伐他汀>辛伐他汀>普伐他汀>瑞舒伐他汀;体外实验证明RK7 和KQ7 HMG-CoA 还原酶的IC50 分别为1.045和1.228 mg/mL,HMG-CoA 还原酶抑制活性:RK7>KQ7。本实验通过生物信息学高效快速预测牦牛乳干酪中抑制 HMG-CoA 还原酶苦味肽生物活性及揭示干酪中苦味肽 KQ7,RK7抑制HMG-CoA 还原酶作用机理,为后续研究牦牛乳源HMG-CoA 还原酶抑制肽提供重要参考。
[1] MIHALJEVI -JURI
-JURI P, SOUSA S F.A QM/MM evaluation of the missing step in the reduction mechanism of HMG-CoA by human HMG-CoA reductase[J].Processes, 2021, 9(7):1085.
P, SOUSA S F.A QM/MM evaluation of the missing step in the reduction mechanism of HMG-CoA by human HMG-CoA reductase[J].Processes, 2021, 9(7):1085.
[2] SINGH N, TAMARIZ J, CHAMORRO G, et al.Inhibitors of HMG-CoA reductase:Current and future prospects[J].Mini Reviews in Medicinal Chemistry, 2009, 9(11):1272-1283.
[3] TOBERT J A.Lovastatin and beyond:The history of the HMG-CoA reductase inhibitors[J].Nature Reviews.Drug Discovery, 2003, 2(7):517-526.
[4] HERMANN M, BOGSRUD M P, MOLDEN E, et al.Exposure of atorvastatin is unchanged but lactone and acid metabolites are increased several-fold in patients with atorvastatin-induced myopathy[J].Clinical Pharmacology and Therapeutics, 2006, 79(6):532-539.
[5] GOLOMB B A, EVANS M A.Statin adverse effects:A review of the literature and evidence for a mitochondrial mechanism[J].American Journal of Cardiovascular Drugs, 2008, 8(6):373-418.
[6] KASHIMA Y, KANEMATSU S, ASAI S R, et al.Identification of a novel hypocholesterolemic protein, major royal jelly protein 1, derived from royal jelly[J].PLoS One, 2014, 9(8):e105073.
[7] DALIRI E, OH D, LEE B.Bioactive peptides[J].Foods, 2017, 6(5):32.
[8] HE S D, ZHANG Y, SUN H J, et al.Antioxidative peptides from proteolytic hydrolysates of false abalone (Volutharpa ampullacea perryi): Characterization, identification, and molecular docking[J].Marine Drugs, 2019, 17(2):116.
[9] 杨保军, 梁琪.干酪源生物活性肽的构效关系研究进展[J].食品与发酵工业, 2021, 47(16):288-293.YANG B J, LIANG Q.Advances on the structure-activity relationship of bioactive peptides in cheese[J].Food and Fermentation Industries, 2021, 47(16):288-293.
[10] 杨保军, 梁琪, 宋雪梅.基于计算机虚拟技术研究牦牛乳硬质干酪苦味肽的抑菌活性差异[J].食品与生物技术学报, 2021, 40(12):75-87.YANG B J, LIANG Q, SONG X M.Study on difference of antibacterial activity of bitter peptide from yak milk hard cheese based on computer virtual technology[J].Journal of Food Science and Biotechnology, 2021, 40(12):75-87.
[11] 李梦瑶, 梁琪, 宋雪梅.结合分子对接技术研究牦牛乳干酪苦味肽RK7和KQ7的α-淀粉酶抑制活性[J].食品科学, 2023, 44(2):132-138.LI M Y, LIANG Q, SONG X M.Using molecular docking to investigate the alpha-amylase inhibitory activity of bitter peptides RK7 and KQ7 derived from yak cheese[J].Food Science, 2023, 44(2):132-138.
[12] 杨保军, 梁琪, 宋雪梅.牦牛乳硬质干酪苦味肽RK7的抗氧化活性及其机制[J].中国食品学报, 2022, 22(8):40-50.YANG B J, LIANG Q, SONG X M.Antioxidant activity and mechanism of yak milk hard cheese bitter peptide RK7[J].Journal of Chinese Institute of Food Science and Technology, 2022, 22(8):40-50.
[13] 杨保军, 梁琪, 宋雪梅.牦牛乳干酪苦味肽ACE抑制活性表征的分子机制[J].中国食品学报, 2022, 22(5):8-17.YANG B J, LIANG Q, SONG X M.Molecular mechanism of ACE inhibitory activity characterization of yak milk cheese bitter peptide[J].Journal of Chinese Institute of Food Science and Technology, 2022, 22(5):8-17.
[14] SOLEYMANZADEH N, MIRDAMADI S, MIRZAEI M, et al.Novel β-casein derived antioxidant and ACE-inhibitory active peptide from camel milk fermented by Leuconostoc lactis PTCC1899:Identification and molecular docking[J].International Dairy Journal, 2019, 97:201-208.
[15] ISTVAN E S, DEISENHOFER J.Structural mechanism for statin inhibition of HMG-CoA reductase[J].Science, 2001, 292(5519):1160-1164.
[16] MINKIEWICZ P, DZIUBA J, IWANIAK A, et al.BIOPEP database and other programs for processing bioactive peptide sequences[J].Journal of AOAC International, 2008, 91(4):965-980.
[17] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX, 2015, 1:19-25.
[18] VAN DER SPOEL D, LINDAHL E, HESS B, et al.GROMACS:Fast, flexible, and free[J].Journal of Computational Chemistry, 2005, 26(16):1701-1718.
[19] GESTO D S, PEREIRA C M S, CERQUEIRA N M F S, et al.An atomic-level perspective of HMG-CoA-reductase:The target enzyme to treat hypercholesterolemia[J].Molecules, 2020, 25(17):3891.
[20] TOPPO A L, YADAV M, DHAGAT S, et al.Molecular docking and ADMET analysis of synthetic statins for HMG-CoA reductase inhibition activity[J].NISCAIR-CSIR, 2021, 58(2):127-134.
[21] MEDINA-FRANCO J L, L PEZ-VALLEJO F, RODR
PEZ-VALLEJO F, RODR GUEZ-MORALES S, et al.Molecular docking of the highly hypolipidemic agent α-asarone with the catalytic portion of HMG-CoA reductase[J].Bioorganic &Medicinal Chemistry Letters, 2005, 15(4):989-994.
GUEZ-MORALES S, et al.Molecular docking of the highly hypolipidemic agent α-asarone with the catalytic portion of HMG-CoA reductase[J].Bioorganic &Medicinal Chemistry Letters, 2005, 15(4):989-994.
[22] CHARAN J, RIYAD P, RAM H, et al.Ameliorations in dyslipidemia and atherosclerotic plaque by the inhibition of HMG-CoA reductase and antioxidant potential of phytoconstituents of an aqueous seed extract of Acacia senegal (L.) Willd in rabbits[J].PLoS One, 2022, 17(3):e0264646.
[23] GESTO D S, CERQUEIRA N M F S A, RAMOS M J, et al.Discovery of new druggable sites in the anti-cholesterol target HMG-CoA reductase by computational alanine scanning mutagenesis[J].Journal of Molecular Modeling, 2014, 20(4):2178.
[24] ISLAM B, SHARMA C, ADEM A, et al.Insight into the mechanism of polyphenols on the activity of HMGR by molecular docking[J].Drug Design, Development and Therapy, 2015, 9:4943-4951.
[25] WANG Z, CHENG L P, KAI Z P, et al.Molecular modeling studies of atorvastatin analogues as HMGR inhibitors using 3D-QSAR, molecular docking and molecular dynamics simulations[J].Bioorganic &Medicinal Chemistry Letters, 2014, 24(16):3869-3876.
[26] GALVEZ ALFREDO F.Identification of lunasin as the active component in soy protein responsible for reducing LDL cholesterol and risk of cardiovascular disease[J].Circulation, 2012, 126(21):A10693.
[27] ZHONG F, ZHANG X M, MA J G, et al.Fractionation and identification of a novel hypocholesterolemic peptide derived from soy protein Alcalase hydrolysates[J].Food Research International, 2007, 40(6):756-762.
[28] LAMMI C, AIELLO G, BOSCHIN G, et al.Multifunctional peptides for the prevention of cardiovascular disease:A new concept in the area of bioactive food-derived peptides[J].Journal of Functional Foods, 2019, 55:135-145.
[29] KARTHIK M V K, SATYA DEEPAK M V K N, SHUKLA P.Explication of interactions between HMGCR isoform 2 and various statins through in silico modeling and docking[J].Computers in Biology and Medicine, 2012, 42(2):156-163.
[30] MINKIEWICZ P, IWANIAK A, DAREWICZ M.BIOPEP-UWM virtual:A novel database of food-derived peptides with in silico-predicted biological activity[J].Applied Sciences, 2022, 12(14):7204.
[31] KUMAR J, SAHU A, DWIVEDI H, et al.Bioactivity assessment of peptide of mung bean on HMG-CoA reductase for anti-hypercholesterolemic activity[J].Journal of Medicinal Food, 2021, 24(8):819-828.