卤化物是由卤素分子取代化合物中的烷烃、芳烃或不饱和烃等位置而产生的一类次级代谢物质,具有强大的生物活性,被广泛用于制药、化学和农业领域[1],它存在于多种天然产物中,包括氯霉素、金霉素、万古霉素、吡咯烷酮、植物生长调节剂噻诺林和抗真菌剂吡咯硝酸酯等[2]。应用最为广泛的是色氨酸卤化物,其结构中的吲哚环是卤素(氟、氯、溴和碘)进行取代的位置。7-氯-色氨酸(7-Cl-Trp)是蝴蝶霉素等天然抗生素的前体[3],具有重要生物活性,因此近年来得到广泛关注[4]。
色氨酸卤化合成7-Cl-Trp的过程需要2种酶催化:黄素腺嘌呤二核苷酸递氢体(flavine adenine dinucleotide reduced, FADH2)依赖的卤化酶RebH和还原型辅酶Ⅰ(nicotinamide adenine dinucleotide,NADH)依赖的黄素还原酶RebF[5]。卤素酶RebH利用FADH2、分子氧(O2)和氯离子(Cl-)对底物色氨酸进行卤化处理,合成7-Cl-Trp。还原型辅因子FADH2的再循环需要NADH依赖的黄素还原酶RebF。有研究基于纯化酶和包括交联酶聚集物在内的醇脱氢酶,构建了一种克级卤化的生物催化过程[6]。有研究在色氨酸高产菌株Corynebacterium glutamicum中表达黄素腺嘌呤二核苷酸(flavin adenine dinucleotide,FAD)依赖的卤化酶RebH和NADH依赖的黄素还原酶RebF,通过代谢工程改造使7-Cl-Trp达到克级合成量[4]。有研究在Saccharomyces cerevisiae中构建了从头合成卤化色氨酸和色氨酸衍生物的途径[7]。但是目前7-Cl-Trp合成仍处于较低水平,迫切需要构建新的7-Cl-Trp合成方法。
全细胞催化具有无需进行酶的纯化、酶更加稳定、产物易于分离提纯、催化剂可重复使用等优点,因此得到越来越广泛的研究[8],利用全细胞催化合成7-Cl-Trp具有重要意义。前期研究中,在E.coli中同时表达FAD依赖型L-氨基酸脱氨酶(L-amino acid deaminase,L-AAD)和FADH2依赖型卤化酶,基于此FAD/FADH2再生体系,通过纯酶和全细胞催化合成7-Cl-Trp[9]。在全细胞催化中,通过L-AAD和卤化酶表达水平调节和增强FAD/FADH2供应,7-Cl-Trp合成提高了15%(从96 mg/L到110 mg/L)。最后,应用超声处理提高膜的通透性,调整活性比,使7-Cl-Trp产率提高1.6倍。但是该系统相对较低的7-Cl-Trp合成量归因于底物色氨酸降解副反应以及卤化酶对底物色氨酸催化效率低。本研究中,首先通过敲除色氨酸降解基因阻断副反应,然后通过结合定向进化和定点饱突变改造卤化酶,以提高其催化性能。最后,对全细胞催化条件进行优化,以进一步提高7-Cl-Trp合成量。
1 材料与方法
1.1 菌株、质粒和化学品
本研究中使用的菌株和质粒列于表1。前期研究中构建的同时表达L-AAD和卤化酶的重组E.coli 1作为基因敲除突变体的出发菌株[9]。E.coli 1敲除基因tnaA编码的色氨酸酶后命名为E.coli 1/ΔtnaA。
表1 菌株、质粒和引物
Table 1 Strains, plasmids, and primers used in this study

菌株、质粒、引物特征资源或参考文献菌株E.coli 1E.coli BL21(DE3)同时表达L-AAD和卤化酶[9]E.coli 1/ΔtnaAΔtnaA::FRT本研究质粒pKD13bla FRT-ahp-FRT[10]pKD46bla αβ exo (red recombinase), temperature conditional replicon[10]pCP20bla cat, temperature sensitive replicon, temperature inducible FLP recombinase[10]引物tnaA-Left-STAGCCTGGACGAGCCGGTTACtnaA-Left-AAGCTCCAGCCTACACGCGATGGTTCTCCAGGTTTACGtnaA-Right-STGGAGAACCATCGCGTGTAGGCTGGAGCTGCTTCGtnaA-Right-ACAGCCTGCTTTCCTGCACTGTCAAACATGAGAATTAATTCCGGtnaA-Middle-STTCTCATGTTTGACAGTGCAGGAAAGCAGGCTGGAGCtnaA-Middle-ACTTCGCTTTCTGGCGAGAAGGTF104 forwardGCGTTTATTTATGGGCAATATGGGTATTGATATF104 reverseGACCAGATACCCCCAGCGAGTACAACTTGT278 forwardTTATGCACTTAACCGGTGGCGGAGAGTTACT278 reverseGCCGCTTCTTTAGCTGTTTTGATCCACGN346 forwardTTATGCACTTAAACGGTGGCGGAGAGTTACN346 reverseTCACCTTTAATGATGCGAGTATTTAATGGTGTATC
注:粗体序列(对应为F104、T278和N346)替换为其他19种氨基酸:A(GCT)、R(CGT)、N(AAC)、D(GAC)、C(TGT)、E(GAA)、Q(CAG)、G(GGT)、H(CAC)、I(ATT)、L(TTA)、K(AAA)、M(ATG)、F(TTC)、P(CCG)、S(TCT)、T(ACC)、W(TGG)、Y(TAC)和 V(GTT)。
PrimeSTAR HS DNA聚合酶、2×Taq PCR混合液、基因组提取试剂盒、DNA纯化试剂盒、感受态细胞制备试剂盒,宝生物工程(大连)有限公司;GeneMorph Ⅱ随机突变试剂盒,美国安捷伦科技公司;MutanBEST kit,日本TaKaRa公司;所有化学试剂均购自上海阿拉丁生化科技股份有限公司。
1.2 仪器与设备
1200 HPLC系统,美国安捷伦科技公司;UV-2450紫外可见分光光度计,日本Shimadzu公司。
1.3 E.coli色氨酸降解途径改造
基因敲除的菌株E.coli/ΔtnaA和E.coli 1/ΔtnaA根据文献中报道的一步敲除法进行构建[10]。以E.coli BL21(DE3)基因组DNA为模板,以表1中tnaA-Left-S和tnaA-Left-A、tnaA-Right-S和tnaA-Right-A为引物分别扩增基因tnaA上游和下游区域(约1 000个碱基)。以质粒pKD13为模板,以含有上下游同源臂的tnaA-Middle-S和tnaA-Middle-A为引物,扩增基因tnaA的敲除框。通过融合PCR,将这3个片段融合到一起。纯化的PCR产物通过电转化导入E.coli和E.coli 1。在卡纳抗性平板上筛选阳性转化子。导入温敏型质粒pCP20,诱导表达FLP重组酶,消除基因组上的抗性片段。
在前期研究最优全细胞转化条件下:pH 7.4,4.2 g/L[细胞干重(dry cell weight,DCW)]细胞, 2.0 g/L色氨酸,总体积5 mL,35 ℃反应12 h,测定野生型E.coli及敲除菌株E.coli/ΔtnaA色氨酸降解量。
在前期研究最优全细胞转化条件下,在2、4、6、8、10、12 h,取样检测E.coli 1及敲除菌株E.coli 1/ΔtnaA催化过程中7-Cl-Trp合成量。
1.4 卤化酶的定向进化
利用GeneMorph Ⅱ随机突变试剂盒,通过易错PCR(ep-PCR)扩增卤化酶基因。50 μL反应混合物包含5 μL 10×Mutazyme Ⅱ反应缓冲,1 μL 40 mmol/L dNTP混合物(200 μmol/L),0.5 μL混合引物(每个引物250 ng/μL),1 μL Mutazyme Ⅱ DNA聚合酶(2.5 U/μL),1 μL模板,41.5 μL水。
为了在30个周期后产生较高的扩增产率及每个突变体的突变频率控制在3~16个核苷酸,以0.1~100 ng的目标DNA为模板。将突变体文库转化E.coli,37 ℃培养8 h,在含有100 μg/mL氨苄青霉素的Luria-Bertani(LB)固体培养基上筛选。突变体单菌落接种0.5 mL LB培养基(含有100 μg/mL氨苄青霉素),在96孔板上37 ℃过夜培养。然后LB培养物接种TB培养基(含有100 μg/mL氨苄青霉素),在96孔板上37 ℃培养2 h,加入异丙基-β-D-硫化半乳糖苷0.04 mmol/L,20 ℃诱导5 h。离心弃上清液,将细胞重悬,加入2.0 g/L色氨酸,在96孔板上35 ℃反应12 h,通过高效液相色谱检测7-Cl-Trp合成量。
1.5 卤化酶的定点突变
使用MutanBEST kit采用一步法PCR进行定点突变。反应使用质粒pRSFDuet-lsr-laad作为模板,引物见表1。纯化后,使用Blunting Kination Enzyme Mix对PCR产物处理,连接酶Ligation Solution I连接。突变质粒转化E.coli,菌落PCR及测序验证突变位点的正确性。然后在最优反应条件下,测试突变体全细胞催化7-Cl-Trp合成量。
1.6 检测
色氨酸和7-Cl-Trp含量采用RP-HPLC测定,采用惯性ODS-SP柱(250 mm×4.6 mm i.d,5 μm,日本)。采用甲醇(A)和体积分数0.05%磷酸(B)的混合溶液作为流动相进行梯度洗脱(0~3 min,2% A;3~22 min,2%~80% A,维持至25 min;25~26 min,80%~2% A,恢复到初始状态),流量为0.8 mL/min,在210 nm下检测样品的紫外吸收度[11]。细胞浓度通过UV-2450紫外可见分光光度计在波长600 nm处测定(OD600),DCW通过预先校准的关系(1 OD=0.492 g/L DCW)测定。
2 结果与分析
2.1 E.coli色氨酸降解途径改造
全细胞催化过程中经常发生底物降解副反应,生成其他副产物,导致底物转化率降低,并加大了下游分离纯化难度[12]。为了测试前期研究中构建的全细胞CombiAADHa系统是否会发生底物色氨酸降解副反应,首先将底物色氨酸加入洗涤后的野生型E.coli BL21(DE3)中,在与前期研究相同的全细胞催化条件下反应[9],测试反应过程中是否消耗色氨酸,生成其他副产物。结果表明,12 h内,色氨酸降解量为50 mg/(L·g DCW)细胞(图1-b)。
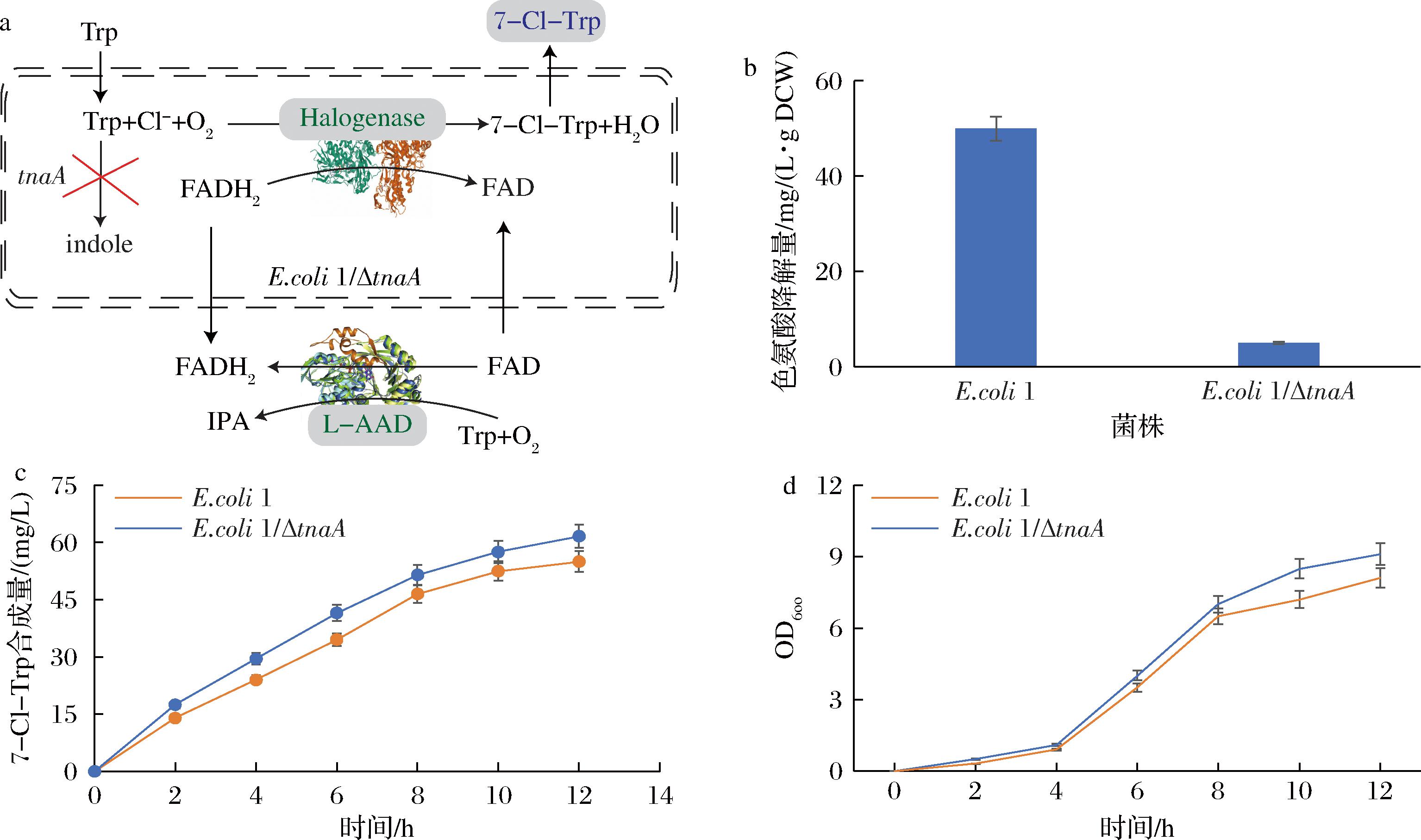
a- E.coli色氨酸降解途径改造;b-色氨酸降解量;c-7-Cl-Trp合成量;d-细胞生长量
图1 E.coli色氨酸降解途径改造及对色氨酸降解量、7-Cl-Trp合成量和细胞生长的影响
Fig.1 E.coli Trp degradation pathway modification and effects on Trp degradation, 7-Cl-Trp synthesis, and cell growth
前期报道中,tnaA编码的色氨酸酶能够将色氨酸催化为吲哚、酮酸和氨气,敲除tnaA基因能够极大提高色氨酸产量[13]。因此本研究构建了E.coli/ΔtnaA,将底物色氨酸加入洗涤后的野生型E.coli中,在与前期研究相同的全细胞催化条件下反应,结果表明,12 h内,色氨酸降解量降低为5 mg/(L·g DCW)(图1-b)。说明敲除tnaA能够有效消除底物降解副反应,有望提高全细胞催化7-Cl-Trp合成量。
前期研究中,在E.coli中同时表达L-AAD和卤化酶,构建了E.coli 1,全细胞催化色氨酸合成7-Cl-Trp。在此基础上,敲除tnaA基因,构建了E.coli 1/ΔtnaA,使进入全细胞催化剂中的色氨酸最大限度的用于催化合成7-Cl-Trp(图1-a)。然后考察了敲除tnaA基因对7-Cl-Trp合成和E.coli 1生长的影响。
由E.coli全细胞催化7-Cl-Trp合成时间曲线可知(图1-c),敲除tnaA基因后,敲除菌株E.coli 1/ΔtnaA整个催化过程中7-Cl-Trp合成量均高于原菌株。12 h时,相比E.coli 1,E.coli 1/ΔtnaA全细胞催化7-Cl-Trp合成量提高了15.8%(从53.2 mg/L提高到61.6 mg/L),说明敲除tnaA基因有利于提高色氨酸利用率,从而提高7-Cl-Trp合成量。
相比E.coli 1,E.coli 1/ΔtnaA最终细胞浓度降低了10%(图1-d),可能是由于色氨酸在生长大肠杆菌中积累对细胞产生不利影响,也可能是由于E.coli 1中多质粒表达造成细胞生长不稳定,细胞生长易于受到基因敲除的影响。但是最终细胞浓度的小幅度降低对全细胞催化合成7-Cl-Trp基本无影响。
2.2 卤化酶定向进化和定点突变
由于L-AAD的催化效率远高于卤化酶,全细胞CombiAADHa系统中L-AAD和卤化酶活性比率(L-AAD:卤化酶)为102∶1,前期研究中,重复表达卤化酶基因lsr后,卤化酶活性升高,两酶活性比率得到调整,全细胞催化7-Cl-Trp合成量提高[9],但是基因重复表达加重了菌体代谢负担,容易导致质粒不稳定,而且卤化酶活性仍然低于L-AAD活性。
由于Lechevalieria aerocolonigenes来源的卤化酶与其他卤化酶的相似性较低(19%),因此很难建立同源性模型来提高催化性能。因此,为了提高卤化酶的催化性能和底物色氨酸的转化率,利用易错PCR对卤化酶进行定向进化,构建了突变体文库。首先,通过改变模板初始用量来优化易错PCR条件。通过改变目标DNA的初始数量或扩增周期的数量来产生低、中或高突变率[14]。与低突变率相比,高突变率产生了更多的独特序列和有益突变[15]。因此本研究采用高突变率进行卤化酶的定向进化。
结果表明,在优化后的易错PCR条件下,每个突变体平均有2~5个氨基酸替换(3~16个核苷酸变化)。通过平板培养得到约5 000个菌落,在96孔板培养后,制备洗涤后的细胞,加入相同量色氨酸进行全细胞催化,通过高效液相色谱测试7-Cl-Trp合成量,筛选具有较高全细胞催化能力的突变体。如图2所示,一个突变体全细胞催化7-Cl-Trp合成量(86.3 mg/L),是突变前菌株E.coli 1/ΔtnaA(61.6 mg/L)全细胞催化7-Cl-Trp合成量的1.4倍。分析了野生型卤化酶和该突变体的催化性能,与野生型相比,该突变体的Kcat增加了20.0%,Km减少了5.0%,整体催化效率为8.2×10-4 s-1/(μmol/L),是卤化酶3-LSR的1.2倍。序列分析表明,该突变体有3个氨基酸突变(F104L、T278S、N346V),1个疏水氨基酸(苯丙氨酸)转变为不同的疏水氨基酸(亮氨酸),1个亲水氨基酸(苏氨酸)转变为不同的亲水氨基酸(丝氨酸)。然而,1个亲水氨基酸(天冬酰胺)已经变成了1个疏水氨基酸(缬氨酸)。这3个氨基酸突变位点与3-LSR的3个突变位点S130L/N166S/Q494R不同,是新的突变位点。这些结果表明,氨基酸F104、T278和N346对卤化酶的催化效率至关重要。因此,选择这3个位点作为进一步进化的目标,通过定点饱和突变和多点突变,以提高卤化酶催化效率,并对野生型卤化酶及其突变体的动力学参数进行比较。
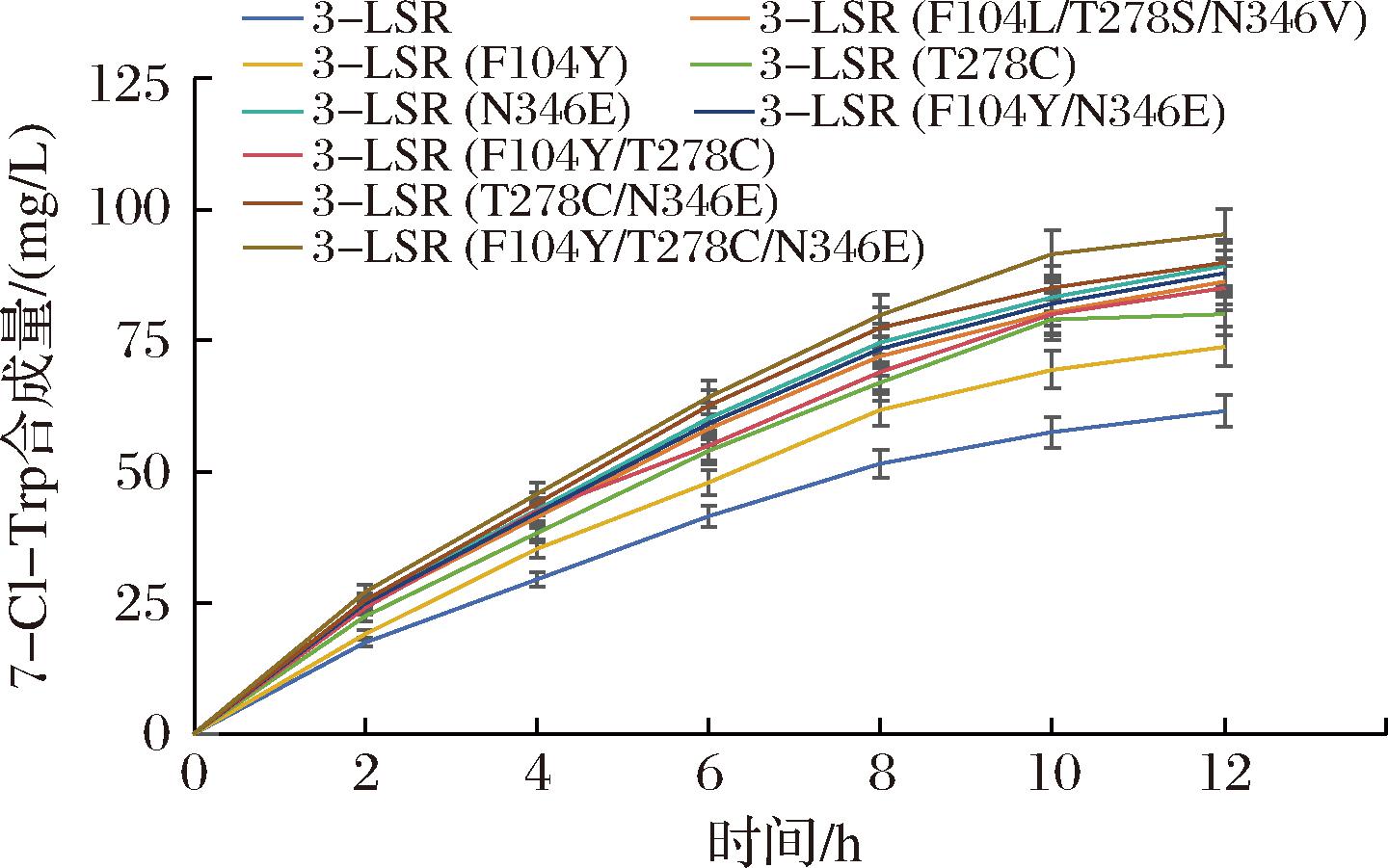
图2 卤化酶突变体全细胞催化7-Cl-Trp合成时间曲线
Fig.2 Time curve of 7-Cl-Trp synthesis by halogenase mutants
通过对3个位点分别进行定点饱和突变,每个突变体重组E.coli进行全细胞催化,得到7-Cl-Trp合成量最高的3个单点突变体F104Y、T278C、N346E,全细胞催化7-Cl-Trp合成量分别为73.9、80.1、89.3 mg/L(图2),比突变前菌株E.coli 1/ΔtnaA(61.6 mg/L)全细胞催化7-Cl-Trp合成量提高了20.0%、30.0%、44.9%。对卤化酶3-LSR及其突变体的动力学参数进行比较,3个单点突变体F104Y、T278C、N346E整体催化效率分别为7.3×10-4、7.8×10-4、8.8×10-4s-1/(μmol/L),是卤化酶3-LSR的1.1倍、1.2倍、1.3倍。
在单点饱和突变基础上,构建了两点和三点突变体重组E.coli,并测定了它们的全细胞催化能力。两点突变体重组E.coli F104Y/T278C、F104Y/N346E、T278C/N346E全细胞催化7-Cl-Trp合成量分别为85.1、87.9、89.9 mg/L(图2),比突变前菌株E.coli 1/ΔtnaA(61.6 mg/L)全细胞催化7-Cl-Trp合成量提高了38.1%、42.7%、45.9%。对卤化酶3-LSR及其突变体的动力学参数进行比较,3个两点突变体F104Y/T278C、F104Y/N346E、T278C/N346E整体催化效率分别为8.0×10-4、8.6×10-4、8.6×10-4 s-1/(μmol/L),是卤化酶3-LSR的1.2倍、1.3倍、1.3倍。
三点突变体重组E.coli F104Y/T278C/N346E全细胞催化7-Cl-Trp合成量最高为95.4 mg/L(图2),比突变前菌株E.coli 1/ΔtnaA(61.6 mg/L)全细胞催化7-Cl-Trp合成量提高了54.9%。对卤化酶3-LSR及其突变体的动力学参数进行比较(表2),三点突变体的Kcat增加了50.0%,Km减少了9.2%,整体催化效率提高了65.2%。所有卤化酶突变体均在E.coli 1/ΔtnaA中表达,并通过全细胞蛋白电泳分析,发现这些突变体重组E.coli中卤化酶具有相似表达水平(数据未展示),说明卤化酶分子改造并未影响其在E.coli中表达水平,全细胞催化效率的提高是因为卤化酶催化效率的提高。
表2 卤化酶和突变体动力学参数比较
Table 2 Comparison of kinetic parameters for halogenase and mutants
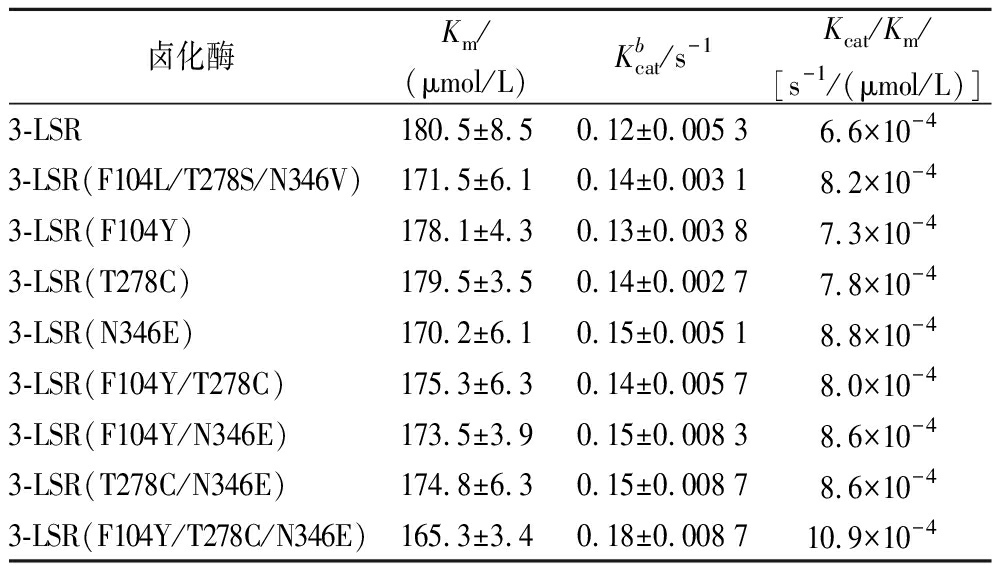
卤化酶Km/(μmol/L)Kbcat/s-1Kcat/Km/[s-1/(μmol/L)]3-LSR180.5±8.50.12±0.005 36.6×10-43-LSR(F104L/T278S/N346V)171.5±6.10.14±0.003 18.2×10-43-LSR(F104Y)178.1±4.30.13±0.003 87.3×10-43-LSR(T278C)179.5±3.50.14±0.002 77.8×10-43-LSR(N346E)170.2±6.10.15±0.005 18.8×10-43-LSR(F104Y/T278C)175.3±6.30.14±0.005 78.0×10-43-LSR(F104Y/N346E)173.5±3.90.15±0.008 38.6×10-43-LSR(T278C/N346E)174.8±6.30.15±0.008 78.6×10-43-LSR(F104Y/T278C/N346E)165.3±3.40.18±0.008 710.9×10-4
2.3 全细胞催化条件优化
前期研究中CombiAADHa全细胞催化条件为pH7.4,4.2 g/L(DCW),2.0 g/L色氨酸,总体积5 mL,35 ℃反应12 h[9]。基因敲除和卤化酶分子改造可能会导致E.coli全细胞催化剂对环境条件敏感,不稳定性提高,从而降低全细胞催化能力和7-Cl-Trp合成量。如图3-a所示,全细胞系统的最佳温度为45 ℃。然而,细胞在45 ℃下发生裂解,难以通过离心完全分离,不利于全细胞催化剂的循环使用。因此,验证了全细胞催化剂热稳定性(图3-a),孵育2 h后,将最佳温度定义为40 ℃。在40 ℃催化12 h,细胞始终保持完整,易于通过离心完全分离。全细胞催化剂最优pH与改造前相同,为pH 7.4(数据未展示)。
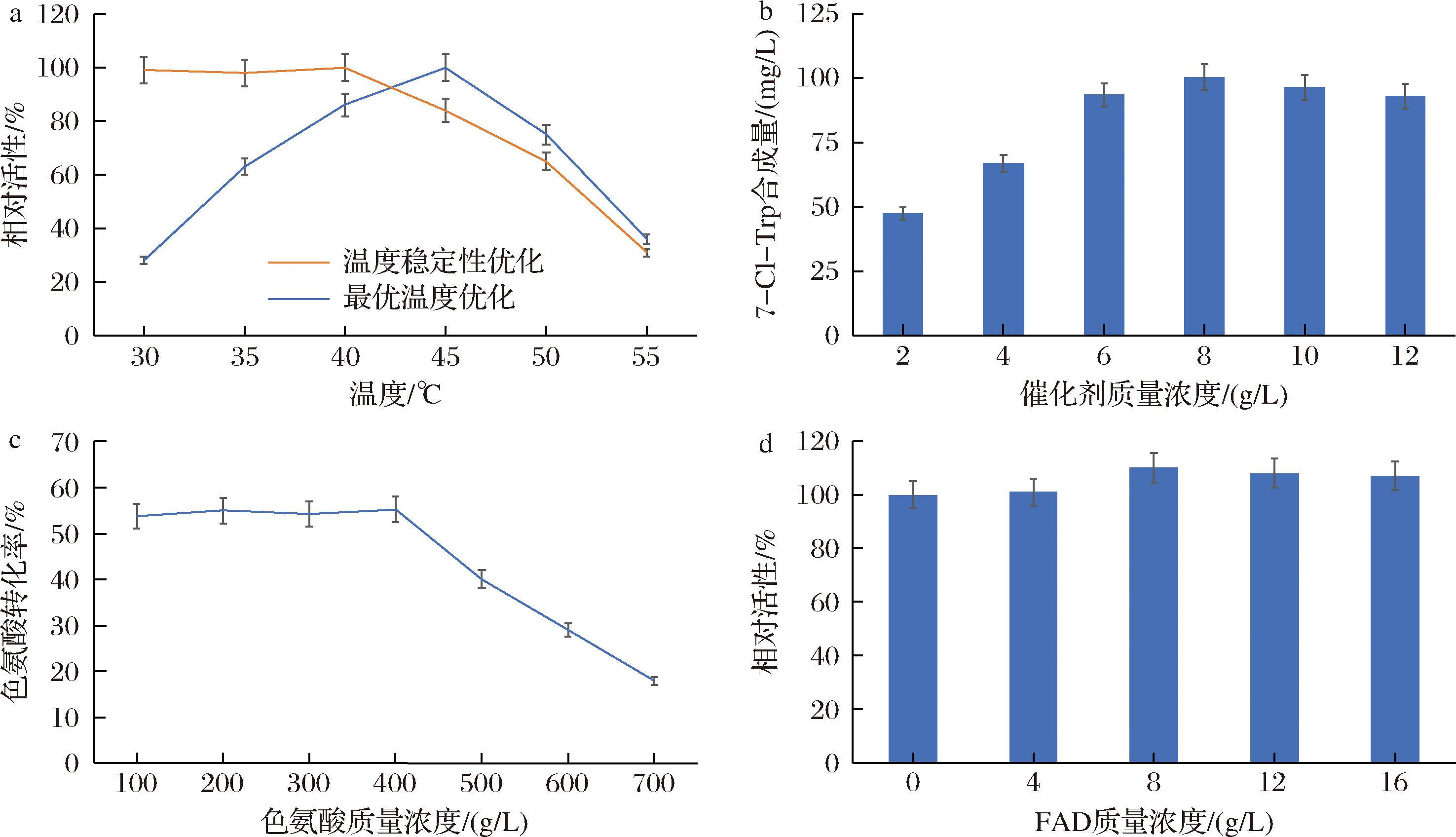
a-coli全细胞催化温度;b-催化剂质量浓度;c-色氨酸质量浓度;d-FAD质量浓度
图3 E.coli全细胞催化条件优化
Fig.3 Optimization of E. coli whole-cell catalytic conditions
如图3-b所示,随着E.coli全细胞催化剂质量浓度提高,7-Cl-Trp合成量提高,当催化剂为8 g/L(DCW)时,7-Cl-Trp合成量最高为101.4 mg/L。催化剂质量浓度的进一步增加导致7-Cl-Trp合成量略有下降,可能是由于较高的细胞密度会导致氧传递速率减慢。
如图3-c所示,在低质量浓度的色氨酸(100、200、300 mg/L)下,转化率超过50%。在400 mg/L色氨酸时,最大转化率为55.3%。随着色氨酸质量浓度的进一步提高,转化率降低,说明存在一定程度的底物抑制。
色氨酸和卤化酶都是FAD/FADH2依赖型酶,全细胞催化色氨酸合成7-Cl-Trp需要辅酶FAD/FADH2,因此研究外源添加不同量FAD对7-Cl-Trp合成量的影响。如图3-d所示,外源添加8 μmol/L FAD,7-Cl-Trp合成量提高10%。说明全细胞自身的辅酶FAD/FADH2再生系统不足以为氧化脱氨基和卤化反应提供足够辅酶,需要外源添加一定FAD/FADH2。继续提高FAD/FADH2添加量,7-Cl-Trp合成量不再提高。
在上述最优催化条件(40 ℃,pH 7.4,400 mg/L色氨酸,10 μmol/L FAD)下对色氨酸进行全细胞催化,12 h后,7-Cl-Trp合成量为105.7 mg/L。
3 结论与讨论
前期研究中,分别通过纯酶和全细胞催化合成7-Cl-Trp,虽然全细胞催化剂具有无需进行酶的纯化、细胞稳定性高、可回收再利用等优点,但是由于底物色氨酸降解副反应途径的存在及卤化酶催化活性较低,全细胞催化合成7-Cl-Trp产量较低。本研究通过结合色氨酸途径改造和卤化酶分子改造提高全细胞催化合成7-Cl-Trp的能力。
全细胞催化剂在非生长状态下,其酶仍能发挥催化作用[16],因此全细胞催化过程中,色氨酸酶仍能催化色氨酸导致副反应发生。敲除色氨酸酶后,E.coli色氨酸降解量降低,全细胞催化7-Cl-Trp合成量提高。与预期结果相同,阻断全细胞催化剂底物或中间体竞争途径可以阻止全细胞催化途径偏离所需的合成途径,通常会增加代谢流和产物合成[17]。类似现象发生在全细胞催化合成N-乙酰-D-神经氨酸(N-acetyl-D-neuraminic acid,Neu5Ac)中。敲除编码Neu5Ac醛缩酶和Neu5Ac转运体的nanA和nanT基因,可以消除唾液酸分解代谢,而敲除nanK(编码Neu5Ac转运体)和nanA基因可以阻止ManNAc和Neu5Ac从生物合成途径中转移。当敲除E.coli中nanTEK基因后,Neu5Ac的产量提高了3倍,得到173.8 mmol/L的Neu5Ac[18]。
黄素依赖型卤化酶(flavin-dependent halogenases,FDHs) 是一类依赖黄素参与,催化天然产物卤化生成卤代化合物的酶[19]。色氨酸卤化酶是将卤素原子特异性地卤化在色氨酸中的吲哚环上,产生相应卤化产物的酶。目前已知生产色氨酸卤化酶的菌大多是放线菌,因此对于该酶的研究多集中于基因克隆以及在E.coli中的异源表达[20]。目前已有研究对卤化酶进行结构上的突变改造以提高其催化效率或者扩大底物偏好性。有研究通过定向进化得到更耐高温的卤化酶突变体,但是催化效率并没有提高,说明热稳定性不一定与酶的催化效率相关[21]。对比色氨酸卤化酶结构,发现与底物色氨酸结合的关键位点,为卤化酶定点突变打下基础[22]。但是目前关于卤化酶定向进化和定点突变的研究仍然比较少。
卤化酶的定向进化和定点突变结果表明,与单点突变体相比,两点突变体具有更高的催化效率,且三点突变体的整体催化效率最高。这些结果表明,这3个突变位点对色氨酸的酶活性有协同作用。在对通过定向进化确定的突变位点进行微调后,发现通过饱和突变产生的新突变体全细胞催化效率更高。因此,定向进化和定点突变的结合使酶快速有效进化。类似策略也被应用于定向进化和定点突变结合,提高Paenibacillus campinasensis家族-11木聚糖酶的碱稳定性和热稳定性[23]。通过随机突变和定点突变,提高放线菌平面对一种棘球菌素B脱酰化酶的催化效率[24]。
总之,通过敲除底物降解基因,构建了代谢工程E.coli全细胞生物催化剂,以消除副反应,增加7-Cl-Trp合成量。卤化酶也通过易错PCR和定点突变进行了分子改造,最终三点突变体表现出了更高的催化效率。
[1] MARDIROSSIAN M, RUBINI M, ADAMO M F A, et al.Natural and synthetic halogenated amino acids-structural and bioactive features in antimicrobial peptides and peptidomimetics[J].Molecules, 2021, 26(23):7401.
[2] WAGNER C, EL OMARI M, KÖNIG G M.Biohalogenation:Nature’s way to synthesize halogenated metabolites[J].Journal of Natural Products, 2009, 72(3):540-553.
[3] VARALA R, ALAM M M, KURMARAYUNI C M, et al.Biosynthetic pathways for rebeccamycin family of natural products[J].Mini-Reviews in Organic Chemistry, 2023, 20(3):309-321.
[4] VELDMANN K H, MINGES H, SEWALD N, et al.Metabolic engineering of Corynebacterium glutamicum for the fermentative production of halogenated tryptophan[J].Journal of Biotechnology, 2019, 291:7-16.
[5] REED K B, BROOKS S M, WELLS J, et al.A modular and synthetic biosynthesis platform for de novo production of diverse halogenated tryptophan-derived molecules[J].Nature Communications, 2024, 15(1):3188.
[6] FRESE M, SEWALD P N.Enzymatic halogenation of tryptophan on a gram scale[J].Angewandte Chemie International Edition, 2015, 54(1):298-301.
[7] MILNE D N, S EZ-S
EZ-S EZ J, NIELSEN A M, et al.Engineering Saccharomyces cerevisiae for the de novo production of halogenated tryptophan and tryptamine derivatives[J].ChemistryOpen, 2023, 12(4):e202200266.
EZ J, NIELSEN A M, et al.Engineering Saccharomyces cerevisiae for the de novo production of halogenated tryptophan and tryptamine derivatives[J].ChemistryOpen, 2023, 12(4):e202200266.
[8] PRIEBE X, HOANG M D, RÜDIGER J, et al.Byproduct-free geraniol glycosylation by whole-cell biotransformation with recombinant Escherichia coli[J].Biotechnology Letters, 2021, 43(1):247-259.
[9] HOU Y, ZHAO W Y, DING X C, et al.Co-production of 7-chloro-tryptophan and indole pyruvic acid based on an efficient FAD/FADH2 regeneration system[J].Applied Microbiology and Biotechnology, 2023, 107(15):4873-4885.
[10] DATSENKO K A, WANNER B L.One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products[J].Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12):6640-6645.
[11] ZHAO Z J, ZOU C, ZHU Y X, et al.Development of L-tryptophan production strains by defined genetic modification in Escherichia coli[J].Journal of Industrial Microbiology &Biotechnology, 2011, 38(12):1921-1929.
[12] DE CARVALHO C C C R.Enzymatic and whole cell catalysis:Finding new strategies for old processes[J].Biotechnology Advances, 2011, 29(1):75-83.
[13] DING D Q, BAI D Y, LI J L, et al.Analyzing the genetic characteristics of a tryptophan-overproducing Escherichia coli[J].Bioprocess and Biosystems Engineering, 2021, 44(8):1685-1697.
[14] NEYLON C.Chemical and biochemical strategies for the randomization of protein encoding DNA sequences:Library construction methods for directed evolution[J].Nucleic Acids Research, 2004, 32(4):1448-1459.
[15] DRUMMOND D A, IVERSON B L, GEORGIOU G, et al.Why high-error-rate random mutagenesis libraries are enriched in functional and improved proteins[J].Journal of Molecular Biology, 2005, 350(4):806-816.
[16] NARANCIC T, RADIVOJEVIC J, JOVANOVIC P, et al.Highly efficient Michael-type addition of acetaldehyde to β-nitrostyrenes by whole resting cells of Escherichia coli expressing 4-oxalocrotonate tautomerase[J].Bioresource Technology, 2013, 142:462-468.
[17] THEODOSIOU E, TÜLLINGHOFF A, TOEPEL J, et al.Exploitation of hetero- and phototrophic metabolic modules for redox-intensive whole-cell biocatalysis[J].Frontiers in Bioengineering and Biotechnology, 2022, 10:855715.
[18] LIN B X, ZHANG Z J, LIU W F, et al.Enhanced production of N-acetyl-D-neuraminic acid by multi-approach whole-cell biocatalyst[J].Applied Microbiology and Biotechnology, 2013, 97(11):4775-4784.
[19] MENON B R K, RICHMOND D, MENON N.Halogenases for biosynthetic pathway engineering:Toward new routes to naturals and non-naturals[J].Catalysis Reviews, 2022, 64(3):533-591.
[20] VAN PÉE K H, MILBREDT D, PATALLO E P, et al.Chapter four application and modification of flavin-dependent halogenases[J].Methods in Enzymology, 2016, 575:65-92.
[21] POOR D C B, ANDORFER M C, LEWIS P J C.Improving the stability and catalyst lifetime of the halogenase RebH by directed evolution[J].ChemBioChem, 2014, 15(9):1286-1289.
[22] LANG D A, POLNICK S, NICKE T, et al.Changing the regioselectivity of the tryptophan 7-halogenase PrnA by site-directed mutagenesis[J].Angewandte Chemie International Edition, 2011, 50(13):2951-2953.
[23] ZHENG H C, LIU Y H, SUN M Z, et al.Improvement of alkali stability and thermostability of Paenibacillus campinasensis Family-11 xylanase by directed evolution and site-directed mutagenesis[J].Journal of Industrial Microbiology &Biotechnology, 2014, 41(1):153-162.
[24] CHENG Y N, QIU S, CHENG F, et al.Enhancing catalytic efficiency of an Actinoplanes utahensis echinocandin B deacylase through random mutagenesis and site-directed mutagenesis[J].Applied Biochemistry and Biotechnology, 2020, 190(4):1257-1270.