在代谢途径工程[1]、蛋白质复合物生产[2]或重组基因与辅因子的共同表达[3]等各种应用中,多种基因的有效共同表达非常重要。高效良好的多基因共表达是代谢工程和合成生物学中的一个共同挑战。这些蛋白质组分需要在累积表达、表达比率以及调节方面进行优化[4-6]。传统上,单向启动子(monodirectional promoters,MDPs)已经被广泛应用于这类场景,但在多基因共表达方面存在一定的局限性。因此,迫切需要一种在基因电路设计、代谢途径组装和优化方面具有更高灵活性的新工具,确保各个共表达基因达到最佳表达水平,从而最大限度地提高重组蛋白生产的整体性能。
发散性转录是所有生物体中普遍存在的一个保守特征。具体而言,发散性转录指的是在启动子+1转录起始位点两端7~25 bp内存在一个相反的转录位点。这种类型的启动子被称为双向启动子(bidirectional promoters, BDPs)[7]。发散性转录在原核生物与真核生物中广泛存在,包括大肠杆菌(Escherichia coli)[8-9]、酿酒酵母[10]、里氏木霉[11]、果蝇[12]、蓝氏贾第鞭毛虫[13]、植物[14-15]、人类[16-17]、和哺乳动物[18-19]。在真核生物中,BDPs是由2个RNA聚合酶(RNA polymerase, RNAP)同时汇集在无核小体的复制起始区的特性决定的,随后在2个方向上起动转录。相比之下,细菌中的BDPs序列更为简短且具有对称性,允许RNAP在双链DNA的同一区段上以任意方向结合,相当于一个RNAP进行一次转录事件[7]。利用BDPs,可以定量调节2个基因的累积表达,减弱使用多个单向启动子可能导致的负面影响[20],在一次克隆表达筛选实验中确定最佳的用于基因共表达的启动子。在真菌系统中,BDPs可通过组装和调节共同代谢途径中不同基因的转录来改善遗传电路。在酵母中,BDPs工程已被证明可用于优化多基因协同表达。例如,VOGL等[21]通过筛选天然和杂合的BDPs文库,实现了更高产量的β-胡萝卜素合成,这是通过多基因通路的组装和转录调控实现的。BDPs在原核生物中少有报道,主要集中在大肠杆菌表达系统。
瞬时表征蛋白传感器(sensor for transiently expressed proteins, STEP)是一种基因编码的荧光生物传感器,能够在几秒钟内检测到活细菌中的蛋白质表达[22]。将绿色荧光瞬时表征蛋白传感器蛋白1 (green fluorescent sensor for transiently expressed proteins 1, gSTEP1)与瞬时表征蛋白传感器蛋白质标签(sensor for transiently expressed proteins tag, STEPtag)基因序列分别插入到p19BDP载体两端,转化到大肠杆菌DH5α后2种蛋白迅速结合并发出绿色荧光。gSTEP1蛋白是一种完全成熟但暗淡的绿色荧光蛋白,在未结合STEPtag蛋白标签时荧光强度很低。
本文根据大肠杆菌与谷氨酸棒杆菌(Corynebacterium glutamicum)-10与-35区保守序列,设计了一系列BDPs突变文库,通过使用瞬时表征蛋白传感器与红绿荧光蛋白标记法,在大肠杆菌与谷氨酸棒杆菌中筛选出多个高活性BDPs。随后进行了发酵实验,将筛选出的BDPs应用到大肠杆菌甲羟戊酸(mevalonic Acid, MVA)生产中,在相同的发酵条件下,W6菌株48 h MVA产量达到了异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)诱导的对照菌产量的72.6%。本研究为原核生物中BDPs的筛选提供了一个有效的途径。
1 材料与方法
1.1 实验材料
1.1.1 主要试剂
2×PrimeSTAR Max DNA Polymerase,宝生物工程(大连)有限公司;2×Rapid Taq Master Mix,南京诺唯赞生物科技股份有限公司;2×MultiF Seamless Assembly Mix,武汉爱博泰克生物科技有限公司;2×Es Taq Master Mix(Dye)、质粒小量抽提试剂盒和胶回收试剂盒,泰州康为世纪有限公司;引物,苏州GENEWIZ生物科技有限公司。
1.1.2 菌株、质粒及引物
本实验中所用的菌株和构建的质粒列于表1。
本实验中所用的引物列于表2。
表1 本研究中所用的菌株与质粒
Table 1 Strains and plasmids used in this study

名称特性来源菌株DH5αF-Φ80 lacZΔM15 Δ(lacZYA-argF ) U169 recA1 endA1 hsdR17(rK-, mK+) phoA实验室保藏BL21(DE3)E. coli B dcm ompT hsdS(rB-mB-) gal实验室保藏ckBL21(DE3), p19-T7-mvaE-mvaS本研究S4BL21(DE3), p19-mvaE-S4-mvaS本研究W6BL21(DE3), p19-mvaE-W6-mvaS本研究Y19BL21(DE3), p19-mvaE-Y19-mvaS本研究SCB0-6BL21(DE3), p19-mvaE-SCB0-6-mvaS本研究SCB1-4BL21(DE3), p19-mvaE-SCB1-4-mvaS本研究SCB1-7BL21(DE3), p19-mvaE-SCB1-7-mvaS本研究SCB2-9BL21(DE3), p19-mvaE-SCB2-9-mvaS本研究SCB2-13BL21(DE3), p19-mvaE-SCB2-13-mvaS本研究SCB3-18BL21(DE3), p19-mvaE-SCB3-18-mvaS本研究SCB4-13BL21(DE3), p19-mvaE-SCB4-13-mvaS本研究SCB6-5BL21(DE3), p19-mvaE-SCB6-5-mvaS本研究质粒pACYC-ESKKDKp15A ori, KanR, pACYC-mvaEmvaS-mvK-pmK-mvaD实验室保藏p19-synCmR, p19-STEPtag-syn-cpGFP本研究p19-wzxBCmR, p19-STEPtag-wzxB-cpGFP本研究p19-aceACmR, p19-STEPtag-aceA-cpGFP本研究p19-ygaQCmR, p19-STEPtag-ygaQ-cpGFP本研究p19-RBGCmR, p19-mRFP1-BDP-eGFP本研究p19-ckCmR, p19-T7-mvaE-mvaS本研究p19-S4CmR, p19-mvaE-S4-mvaS本研究p19-W6CmR, p19-mvaE-W6-mvaS本研究p19-SCB2-9CmR, p19-mvaE-SCB2-9-mvaS本研究
表2 本研究中所用的引物
Table 2 Primers used in this study

引物名称引物序列(5′-3′)SCBsyn-FGTAAAACGACGGCCAGTCCCNNNNNNNNNNATNNTANNNNNNNNNNNNNNNTTGNNNCAANNNNNNNNNNNNNNNTANNATNNNNNNNNNNGG-GGTCATAGCTGTTTCCTGSTBwzxB-FGTAAAACGACGGCCAGTCCCNNNNNNNNNNTTGNNNNNNNNNNNNNNNNNNNNTATNATATATANNNNNNNNNNNNNNNNNNNACCGAANNNN-NNNNNNGGGGTCATAGCTGTTTCCTGSTBaceA-FGTAAAACGACGGCCAGTCCCNNNNNNNNNNTTGNNNNNNNNNNNNNNNNNNNANNAATNTNNNNNNNNNNNNNNNNNNCAANNNNNNNNNNG-GGGTCATAGCTGTTTCCTGSCBygaQ-FGTAAAACGACGGCCAGTCCCNNNNNNNNNNAAAATATNANNNNNNNNNNNNNATGAATTANNNNNNNNNNNTNNTAANTTNNNNNNNNNNGGG-GTCATAGCTGTTTCCTGBDP-backbone-RCATCGACATGGGAGGGTTACBDP-backbone-F2GTAACCCTCCCATGTCGATGBDP-backbone-R2GGGACTGGCCGTCGTTTTACBDP-PCR-FATCGCGGAACAGTTCGTTCABDP-PCR-RATCTTCGATGTTGTGGCGGALtsvJ-F1TGTTTCGTCCGCCGAAACGTACCTCTACAAATAATTTTGTTTAATACTAGAGAAAGAGGAGAAATACTAGATGGCGAGTAGCGAAGACGTLtsvJ-R1ACGTTTCGGCGGACGAAACAGCCTCTACAAATAATTTTGTTTAATACTAGAGAAAGAGGAGAAATACTAGATGGTGAGCAAGGGCGAGGALtsvJ-F2AGCTATGACCCCGGGACTGGCCGTCGTTTTACAGTACGTCTGAGCGTGATACCCGCTCACTGAAGATGGCCCGGTAGGGCCGAAACGTACCTCTACAALtsvJ-R2CCAGTCCCGGGGTCATAGCTGTTTCCTGAGCTGTCACCGGATGTGCTTTCCGGTCTGATGAGTCCGTGAGGACGAAACAGCCTCTACAASCB0-6GTAAAACGACGGCCAGTCCCCACTTTCTATGTCTCCTCCCTTGCTATATCAGAGTTTGAATATCTAGGCTGGGGTCATAGCTGTTTCCTGSCB1-4GTAAAACGACGGCCAGTCCCGGGACATCGATGAGCCACTCTCCTAATGCGCTTCGTGTAACATCATAAGGGGGTCATAGCTGTTTCCTGSCB1-7GTAAAACGACGGCCAGTCCCTAGCGGCCCCGACATCCTTGGGCTTAAATCACTGTTATGATCGTAGGGCGGGGTCATAGCTGTTTCCTGSCB2-9GTAAAACGACGGCCAGTCCCTTATTTATTTTCCAATACCATAATGCAAGACCAGGATGAATAGCTAGACTGGGGTCATAGCTGTTTCCTGSCB2-13GTAAAACGACGGCCAGTCCCTGTCTGGGAATCTGGATAGACCCTTAAGTTTAAAAGGAAGTGGTAGCTTGGGGGTCATAGCTGTTTCCTGSCB3-18GTAAAACGACGGCCAGTCCCCATGGTACCTGTGCTTAGATAAACATTGTGACCGCAACACCTACACTAGGGGGTCATAGCTGTTTCCTGSCB4-13GTAAAACGACGGCCAGTCCCAAATTCGCCCCAATATCCATCAACAGTTTACTCATTTGGAGGGTAGATTAGGGGTCATAGCTGTTTCCTGSCB6-5GTAAAACGACGGCCAGTCCCCTACAGTAGGACTCGGGCGTATGGATTTATGACCGTTAGTCAAACTCTACGGGGTCATAGCTGTTTCCTGmvaE-FAGGATCCTTATTGTTTTCTTAAATCATTTAAAATAGCCAAGGCTCmvaE-RGAAATACTAGATGAAAACAGTAGTTATTATTGATGCATTACGAACmvaS-FGAAATACTAGATGACAATTGGGATTGATAAAATTAGTTTTTTTGTGCmvaS-RAGCTGAATTCTTAGTTTCGATAAGAGCGAACGGTRiboJ-F1CTGTTTTCATCTAGTATTTCTCCTCTTTCTCTAGTATTAAACAAAATTATTTGTAGAGGTACGTTTCGGCCCTACCGGGCCATCRiboJ-R1AGCTATGACCCCGGGACTGGCCGTCGTTTTACAGTACGTCTGAGCGTGATACCCGCTCACTGAAGATGGCCCGGTAGGGCCGAARiboJ-F2CCAGTCCCGGGGTCATAGCTGTTTCCTGAGCTGTCACCGGATGTGCTTTCCGGTCTGATGAGTCCGTGAGGACGAAACAGCCTCRiboJ-R2CAATTGTCATCTAGTATTTCTCCTCTTTCTCTAGTATTAAACAAAATTATTTGTAGAGGCTGTTTCGTCCTCACGmva-backbone-F1TCGAAACTAAGAATTCAGCTTGGCTGTTTTGGmva-backbone-R1ACCCTCAAACTGCGAAGCACmva-backbone-F2GTGCTTCGCAGTTTGAGGGTmva-backbone-R2AAGAAAACAATAAGGATCCTCTAGAGTCGACCTGS4-FTAAGTATCATCATATCTGTCCCATCGTTTTTGGAAACAATCTGTATCACATTAATAACATATGATCGTCAGGGGTCATAGCTGTTTCCTGS4-RGACAGATATGATGATACTTAATGGGACTGGCCGTCGTTTTACW6-FTTTGGTTCCTCATATTAAGATGTTTATAATATATATATTTAAGAACCTTTTTTTACCGAATGTGCCATTTGGGGTCATAGCTGTTTCCTGW6-RTCTTAATATGAGGAACCAAAAATAGAATGGGGACTGGCCGTCGTTTTACY19-FCATAGATCCTAAAATATTAGCACTCTTAGTAGATGAATTACCAATCTCTTTTACTAAGTTATCGTACCATGGGGTCATAGCTGTTTCCTGY19-RCTAATATTTTAGGATCTATGGGGACTGGCCGTCGTTTTACSCB1-7-FGTAAAACGACGGCCAGTCCCTAGCGGCCCCGACATCCTTGGGCTTAAATCACTGTTATGATCGTAGGGCGGGGTCATAGCTGTTTCCTGSCB1-7-RGGGACTGGCCGTCGTTTTACSCB2-9-FGTAAAACGACGGCCAGTCCCTTATTTATTTTCCAATACCATAATGCAAGACCAGGATGAATAGCTAGACTGGGGTCATAGCTGTTTCCTGSCB2-9-RGGGACTGGCCGTCGTTTTAC
1.1.3 培养基
LB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10,用于大肠杆菌质粒构建以及种子发酵液的培养。LB固体培养基为LB液体培养基添加1.85 g/L琼脂粉制成。
M9培养基(g/L):Na2HPO4 6.6,KH2PO4 3, NaCl 0.5, NH4Cl 1,溶于980 mL ddH2O后,高温灭菌后加入20 mL 20% D-葡萄糖溶液, 2 mL 1 mol/L MgSO4溶液, 0.1 mL 1 mol/L CaCl2溶液,用于种子发酵液的培养。
LBB培养基(g/L):酵母提取物5,胰蛋白胨10, NaCl 10, 脑心浸出液10,用于谷氨酸棒杆菌电转过程恢复培养。
LBHis培养基(g/L):酵母膏2.5,胰蛋白胨5, NaCl 5,脑心浸出液18.5,山梨醇91,用于谷氨酸棒杆菌质粒构建。LBHis固体培养基为LBHis液体培养基添加1.85 g/L琼脂粉制成。
1.2 仪器与设备
759S紫外可见光分光光度计,上海棱光技术有限公司;LongGene A300 PCR仪,朗基科学仪器有限公司;Trace1310-ISQ LT气相色谱质谱联用仪,美国赛默飞世尔科技公司;BioTek多功能酶标仪,美国伯腾仪器有限公司。
1.3 实验方法
1.3.1 BDPs文库的构建与筛选
根据文献中报道的大肠杆菌核心区明确的BDPs为模板,保留-10与-35区的保守序列,设计引物交由苏州金唯智公司合成。实验室以p19-0为载体,将gSTEP1与STEPtag基因序列插入到载体上,在gSTEP1与STEPtag基因中间保留一段序列(M13) (gtaaaacgacggccagtcccggggtcatagctgtttcctg)作为后续插入BDPs所需的同源臂,构成p19-BDP质粒,当BDPs插入到p19-BDP载体中同时表达左右两端的gSTEP1与STEPtag基因,从而产生绿色荧光。
酶切载体与短片段同源重组法:从金唯智下单引物SCBsyn-F、STBwzxB-F、STBaceA-F与 SCBygaQ-F,得到4个120 bp左右的BDPs片段,前后分别为p19-BDP质粒Sma Ⅰ酶切位点(cccggg)20 bp同源序列,对质粒p19-BDP用Sma Ⅰ进行酶切后,使用 MultiF Seamless Assembly 重组酶50 ℃ 1 h进行同源重组后转化到DH5α 感受态细胞,涂布含氯霉素抗性的LB琼脂板,筛选荧光强度高的77个克隆划线平板,过夜培养后接种到96深孔板,37 ℃摇床24 h后测量荧光与OD值,计算单位荧光值后挑取强中弱各3个克隆接种LB+CM液体培养基,37 ℃培养12 h后提质粒送测金唯智公司,测量方法如图1所示。
使用引物SCBsyn-F(SCBygaQ-F、STBaceA-F、STBwzxB-F)与BDP-backbone-R, BDP-backbone-F2与BDP-backbone-R2环批质粒p19-BDP再进行Gibson 双片段组装转化大肠杆菌DH5α感受态,构建4种BDPs文库。
使用引物SCB0-F(SCB1-F、SCB2-F、SCB3-F、SCB4-F、SCB5-F、SCB6-F)与BDP-backbone-R, BDP-backbone-F2与BDP-backbone-R2环批质粒p19-BDP再进行Gibson 双片段组装转化大肠杆菌DH5α感受态,构建7种对称性BDPs文库。
1.3.2 大肠杆菌的转化与培养
转化:将大肠杆菌感受态取出,放在冰上化冻;将重组产物加入感受态细胞中,轻微混匀后冰浴20 min;42 ℃水浴90 s,冰浴6 min;EP管中加入700 μl LBB培养基,37 ℃、100 r/min摇床1 h;5 000 r/min离心1 min,弃掉保留适量上清液,均匀涂布于含有对应抗生素的LB琼脂板上,37 ℃过夜培养12 h。
培养:挑取目标单菌落划线于氯霉素抗性LB琼脂板上,37 ℃培养12 h后挑取目标菌落接种于96深孔板(LB液体培养基+30 ng/μL氯霉素),37 ℃、220 r/min摇床24 h后测量荧光与OD值,计算单位荧光值。挑取克隆接种LB液体培养基,37 ℃、12 h后提质粒送测金唯智公司。
1.3.3 谷氨酸棒状杆菌的转化与培养
转化:将制备的C.glutamicum CGMCC 1.15647感受态取出,放在冰上化冻;加入500 ng冰上预冷的目标质粒,轻微混匀后冰浴15 min;将感受态细胞转移至电转杯中,1.8 kV,5 ms点击2次;转移至含有915 μL LBHis培养基的EP管中;46 ℃水浴6 min,30 ℃、100 r/min摇床2 h;取适量菌液,均匀涂布于含有对应抗生素的LBHis琼脂板上,30 ℃过夜培养24~36 h。
培养:挑取目标单菌落划线于带氯霉素抗性的LBHis琼脂板上,30 ℃培养24 h后挑取目标菌落接种24深孔板(LBHis液体培养基+10 ng/μL 氯霉素),30 ℃、220 r/min摇床24 h后测量荧光与OD值,计算单位荧光值。使用引物BDP-PCR-F与BDP-PCR-R菌批目标菌落送测金唯智公司。
1.3.4 红绿荧光标记载体的构建
使用引物LtsvJ-F1与LtsvJ-R1、LtsvJ-F2与LtsvJ-R2,以RFP-Pmi-eGFP质粒为模板,通过overlap PCR法得到p19-RBG片段,进行Gibson组装得到质粒载体p19-RBG。使用引物SCB0-6(SCB1-4、SCB1-7、SCB2-9、SCB2-13、SCB3-18、SCB4-13、SCB6-5)与BDP-backbone-R、BDP-backbone-F2与BDP-backbone-R2,以p19-RBG质粒为模板,PCR扩增片段后进行Gibson组装,转化大肠杆菌DH5α感受态,涂布氯霉素抗性LB琼脂板,37 ℃过夜培养后进行菌落PCR,挑取正确克隆接种到24深孔板,37 ℃摇床24 h后测量荧光与OD值,计算单位荧光值。
挑取生长良好的单菌落接种于装有2 mL LBB液体培养基的10 mL接种管中,37 ℃、220 r/min培养12 h后提取质粒送测金唯智公司。将质粒电转转化到谷棒感受态中,涂布氯霉素抗性LBHis琼脂板,30 ℃培养36~48 h后挑取克隆接种24深孔板(LBHis液体培养基+10 ng/μL氯霉素),30 ℃摇床24 h后测量荧光与OD值,计算单位荧光值。
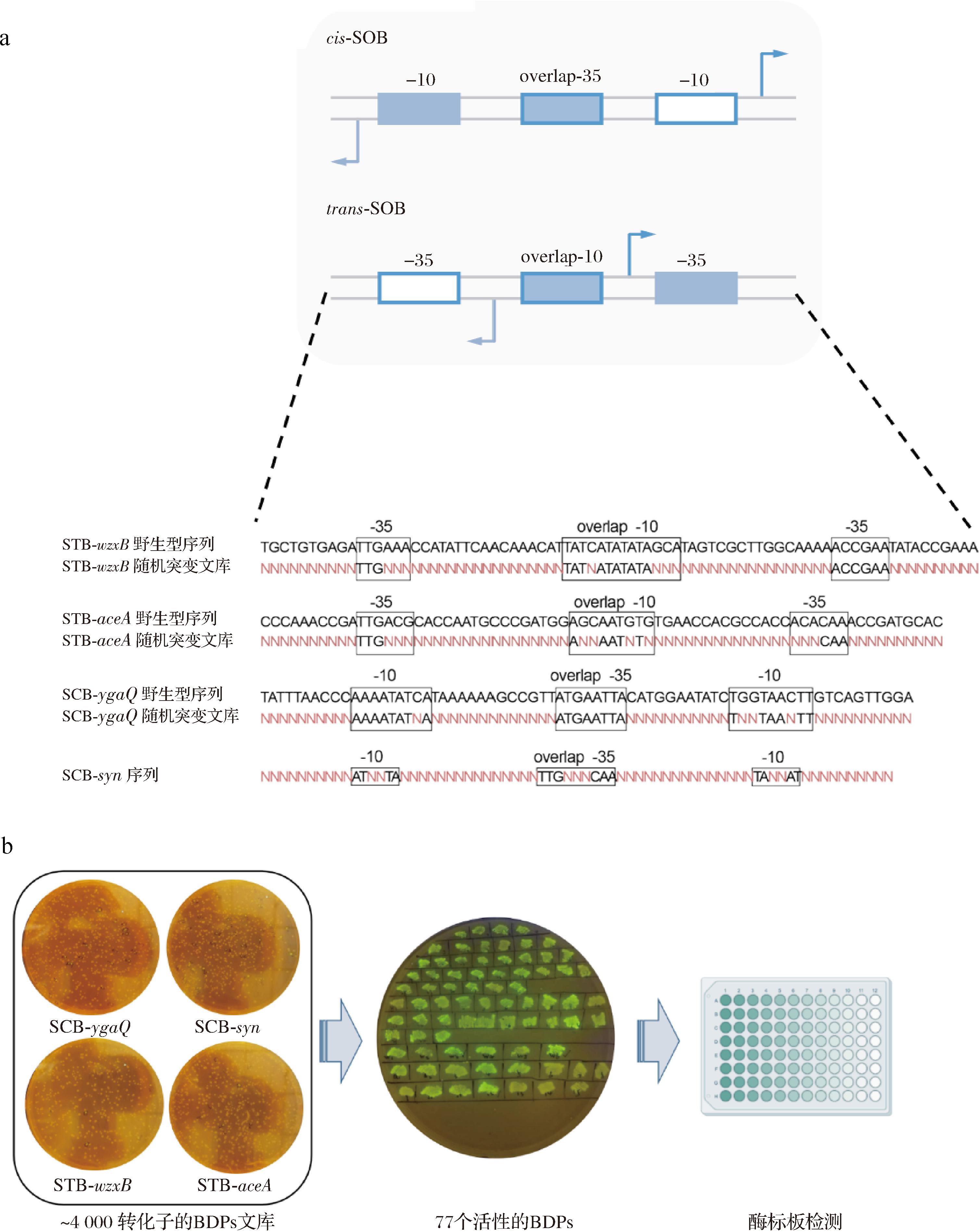
a-4种BDPs文库的构建;b-活性BDPs的筛选流程
图1 BDPs文库的构建与筛选
Fig.1 Construction and screening of BDPs libraries
注:图a中wzxB,ygaQ为文献中报道的大肠杆菌中核心区明确的基因,其中wzxB基因(编码O-抗原翻转酶)上下游序列-10区重合,称为单区重合反式BDPs(single-overlapping trans-bidirectional promoters,简称STB),ygaQ基因(编码蛋白未知)上下游序列-35区重合,称为单区重合顺式BDPs(single-overlapping cis-bidirectional promoters,简称SCB),aceA(编码异柠檬酸裂解酶)为文献中报道的谷氨酸棒杆菌中核心区明确的-10区重合基因,syn是基于谷氨酸棒杆菌中的特征保守序列设计出的-35区重合的对称序列;图b是活性BDPs的筛选流程,首先在LB氯霉素抗性板上构建了大约4 000转化子的库,选取了77个有活性的BDPs划线LB氯霉素平板,接种到96孔板中发酵24 h后测量单位荧光值。
1.3.5 荧光检测
培养后的菌液用培养基稀释到10倍,初始OD值为0.4~0.6,利用BioTek多功能酶标仪测定菌液的OD600值及荧光强度(STEP1:激发波长488 nm,发射波长520 nm,EGFP:激发波长485 nm,发射波长515 nm,RFP:激发波长584 nm,发射波长607 nm)。
1.3.6 MVA载体的构建
使用引物mvaE-F与mvaE-R、mvaS-F与mvaS-R,以pACYC-mvaEmvaS-mvK-pmK-mvaD质粒为模板,PCR扩增得到分别含有mvaE与mvaS的片段,使用引物RiboJ-F1与RiboJ-R1,RiboJ-F2与RiboJ-R2互为模板,PCR扩增得到mvaE与mvaS中间的BDPs插入片段,使用引物mva-backbone-F1与mva-backbone-R1,mva-backbone-F2与mva-backbone-R2,以p19-RBG质粒为模板,PCR扩增得到下游骨架片段,将6个片段Gibson组装后转化到大肠杆菌BL21(DE3),接种LB+CM液体培养基12 h后提取质粒得到大肠杆菌MVA载体p19-mvaE-BDP-mvaS。
使用引物S4-F(W6-F、Y19-F、SCB1-7-F、SCB2-9-F)与mva-backbone-R1、mva-backbone-F2与S4-R(W6-R、Y19-R、SCB1-7-R、SCB2-9-R),以p19-mvaE-BDP-mvaS为模板,PCR片段扩增后进行Gibson组装,转化大肠杆菌BL21(DE3)感受态,37 ℃ 过夜培养,挑取克隆接种于2 mL LB+CM的10 mL接种管,37 ℃、12 h后提取质粒送测金唯智公司。
1.3.7 发酵实验及MVA的检测
将p19-mvaE-S4(W6、Y19、SCB1-7、SCB2-9)-mvaS质粒转化大肠杆菌BL21(DE3)感受态,37 ℃ 过夜培养后接种到48 深孔板中,37 ℃摇床12 h后以初始OD值为0.1接种于30 mL LB+CM+2%(质量分数)葡萄糖+0.3%(体积分数)甘油与30 mL M9+CM+2%葡萄糖+0.3%甘油中,24 h与48 h分别检测MVA产量,间隔3 h测量1次OD600值。
取800 μL菌液与200 μL的300 mmol/L HCl常温放置5 min,然后加入1 mL乙酸乙酯,上下振荡5 min,9 000 r/min 离心5 min,取100 μL上清液用乙酸乙酯稀释10倍(产量低的话取400 μL上清液不稀释),加入0.5 g/mL无水硫酸钠除水,充分振荡3 min,4 ℃静置1 h后离心3 min,上清液过膜后取200 μL加入内插管。然后使用GC-MS分析溶剂层中的MVA。条件设定如下:进气口温度为250 ℃,氦载气流量为2 mL/min,离子源温度为250 ℃,扫描范围(m/z)为50~350。烘箱温度如下:50 ℃ 2 min,以20 ℃/min的速度升至230 ℃,保温3 min。与溶于乙酸乙酯的标品MVA相比,对产物进行定量。通过计算标准曲线,将峰面积转换为MVA浓度。
2 结果与分析
2.1 大肠杆菌中单区重合BDPs的筛选
为了筛选出大肠杆菌中双边活性启动子BDPs,选择了2个大肠杆菌来源的以及一个谷氨酸棒杆菌来源的核心区明确的BDPs序列,如图1所示,另外基于谷氨酸棒杆菌中-35区杂合对称的保守序列设计了一个全合成的BDPs文库(syn)。测量了4个文库的单位荧光值,结果如图2所示。4个BDPs文库均呈现强度的一个多样性(图2-a),最高值与最低值有大约6倍的差异(图2-b),测序结果证实了文库中的序列多样性(图2-c)。
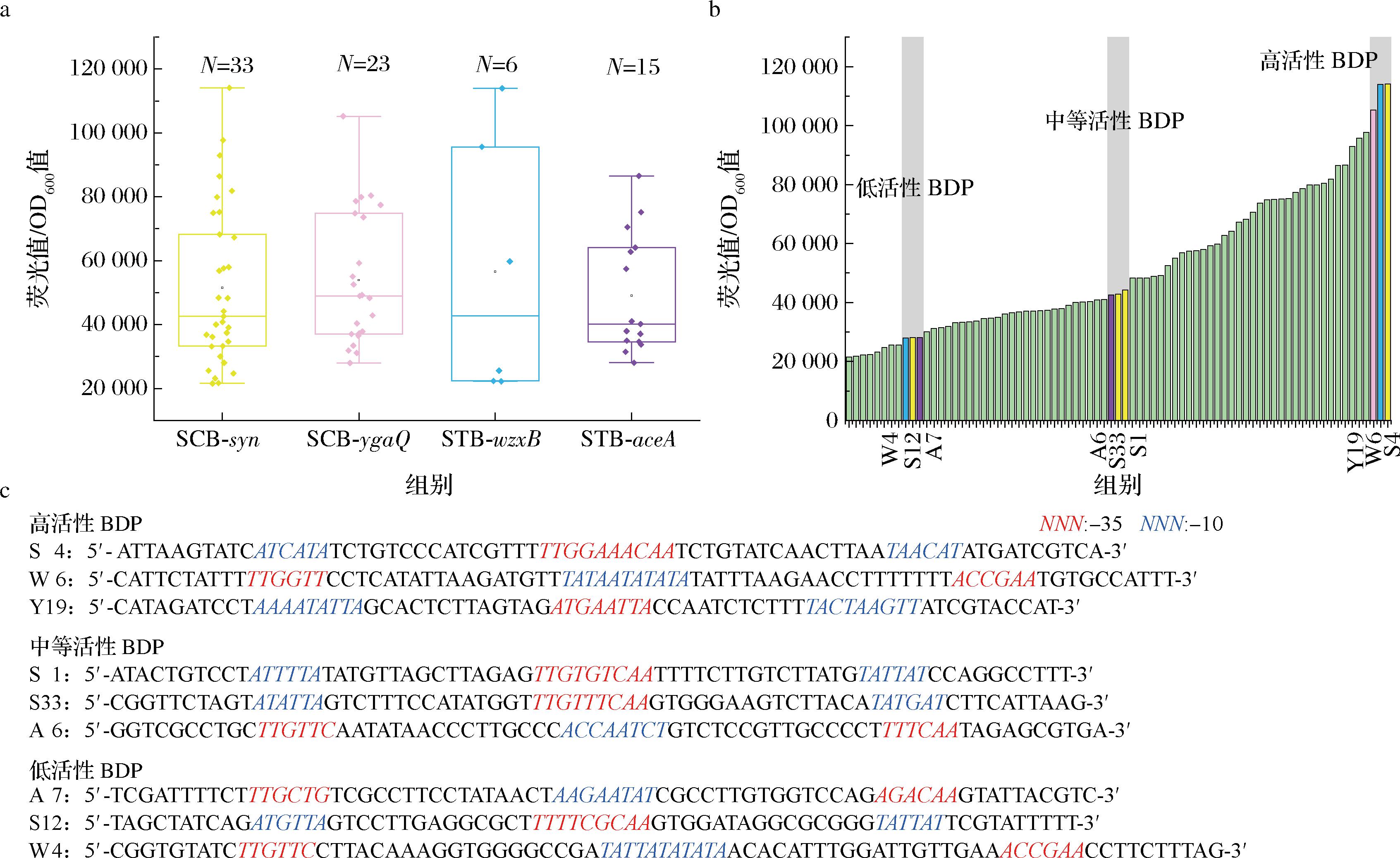
a-4种BDPs文库箱线图;b-77个BDPs单位荧光值;c-不同活性BDPs的测序结果
图2 大肠杆菌中77个BDPs单位荧光与序列
Fig.2 Fluorescence and sequence of 77 BDPs in E.coli
注:b图中,单位荧光值≥60 000的属于高荧光区,40 000≤单位荧光值≤60 000的属于中等荧光区,单位荧光值≤40 000的属于低荧光区;c图中,红色标记为序列-35区, 蓝色标记为序列-10区。
2.2 谷氨酸棒杆菌中对称性BDPs的筛选
与真核生物BDPs不同,原核BDPs仅靠7~23 bp的核心的对称保守序列就可实现双向的转录耦合。为了在谷氨酸棒杆菌中筛选出高活性BDPs,本文设计了7个对称性的文库(表3),SCB0-F(SCB1-F、SCB2-F、SCB3-F、SCB4-F、SCB5-F、SCB6-F)与BDP-backbone-R, BDP-backbone-F2与BDP-backbone-R2环批质粒p19-BDP,Gibson 双片段重组后转入E.coli DH5α,随后电转C.glutamicum CGMCC 1.15647,得到了8个高活性BDPs,单位荧光值与序列如图3所示。SCB2-9启动子两端gSTEP1蛋白与STEPtag蛋白结合活性最高。
表3 七个对称性文库的设计
Table 3 Design of seven symmetric libraries

片段名称片段长度/bp引物序列0UP3 634SCB0-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNNNNTANNNNNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC1UP3 634SCB1-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNNNTNANNNNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC2UP3 634SCB2-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNNNTNNANNNNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC3UP3 634SCB3-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNNaNNNtNNNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC4UP3 634SCB4-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNNANNNNTNNNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC5UP3 634SCB5-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNTNNNNNANNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTAC6UP3 634SCB6-FgtaaaacgacggccagtcccNNNNNNNNNNNNNNNNNNNNNTNNNNNNANNNNNNNNNNNNNNNNNNNNNggggtcatagctgtttcctgBDP-backbone-RCATCGACATGGGAGGGTTACDown3 686BDP-backbone-F2GTAACCCTCCCATGTCGATGBDP-backbone-R2GGGACTGGCCGTCGTTTTAC

a-7种对称性BDPs文库中活性BDPs的筛选;b-8个高活性BDPs单位荧光值;c-8个高活性BDPs测序结果
图3 谷氨酸棒杆菌中8个BDPs单位荧光与序列
Fig.3 Fluorescence and sequence of eight BDPs in C.glutamicum
2.3 红绿荧光检测对称性BDPs双边活性
STEP双分子荧光检测系统利于在培养基上迅速筛选出双边活性高的BDPs,为了分别得到BDPs两边的活性,本文构建了一个左边是红色荧光蛋白RFP,右边是绿色荧光蛋白eGFP的载体——p19-RBG,之后在大肠杆菌与谷氨酸棒杆菌中分别检测了BDPs左右两端的红绿荧光值,结果如图4所示。
在谷氨酸棒杆菌中筛到的BDPs在大肠杆菌中也具有双边活性,且活性更高(图4-a),这表明BDPs具有普遍性,在不同宿主菌中的活性也不同。通过比较图3-b与图4-b中的谷氨酸棒杆菌中不同菌株单位荧光值可以发现,BDPs双边的共同活性与单边活性不成正比关系。
2.4 BDPs在MVA生产过程中的应用
为了验证BDPs的适用性,本文将BDPs应用到了大肠杆菌MVA的生产过程中。基于实验室之前构建的质粒pACYC-ESKKDK,构建了载体p19-mvaE-BDP-mvaS,同时构建了T7启动子表达的mvaE、mvaS串联表达对照菌ck。mvaE与mvaS是MVA生产上游的2个基因,对MVA的生产起着至关重要的作用。
首先构建了菌株S4,菌株S4是将S4启动子插入到p19-mvaE-BDP-mvaS载体中构成的,将S4菌株接种到30 mL LB+CM培养基中培养12 h后,以初始OD600值为0.1接种到LB/M9+2%+0.3%甘油的培养基中发酵24 h与48 h后测量MVA产量(图5-a)。
24 h后M9培养基中MVA产量略高于T7启动子诱导的对照组产量为24 mg/L,LB培养基中MVA产量很低;48 h后对照组MVA产量高于S4菌株达到了43.5 mg/L,S4菌株在M9培养基中产量为29.8 g/L,为对照菌68.5%,LB培养基中MVA产量依旧很低。
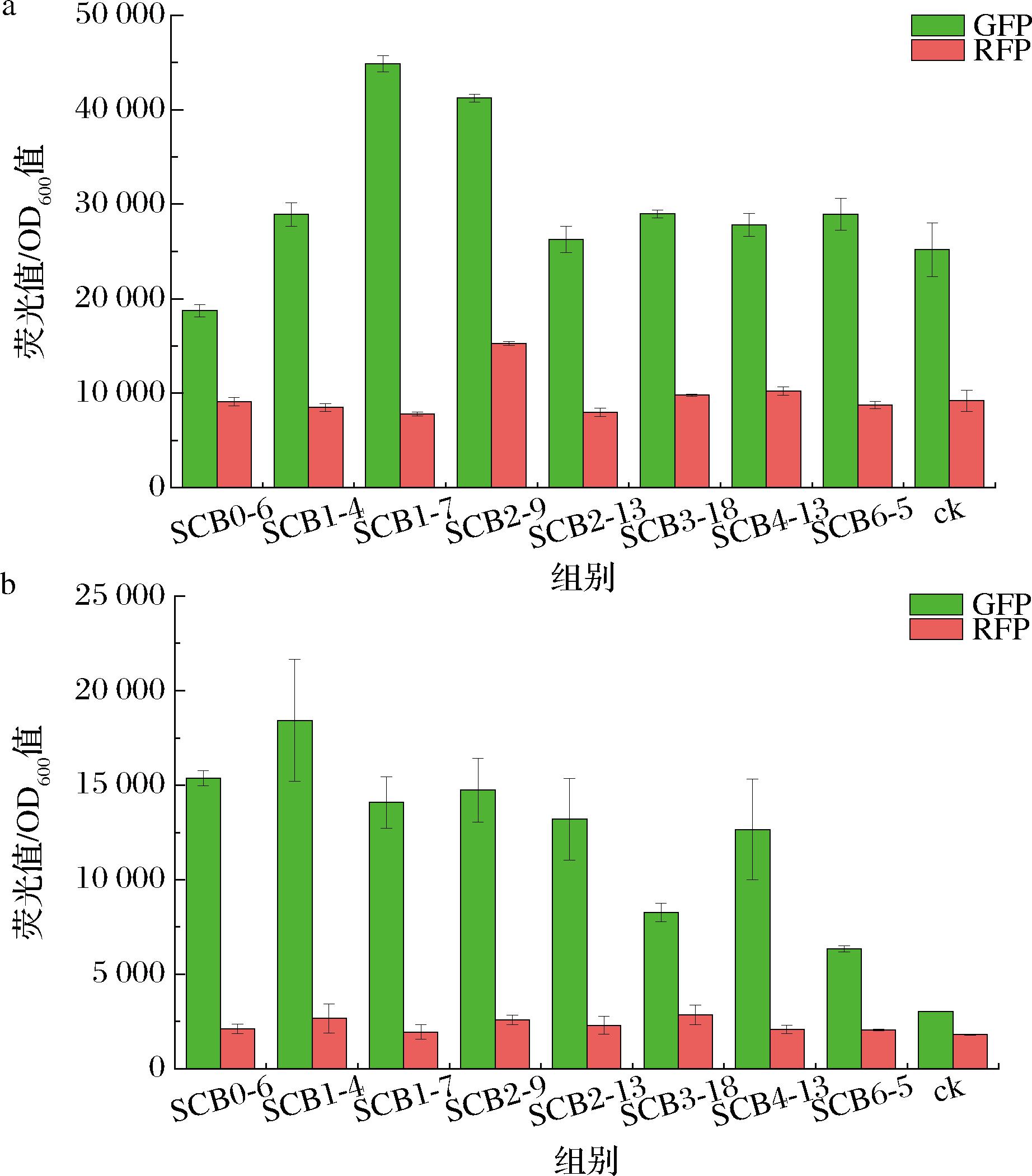
a-大肠杆菌单位荧光;b-谷氨酸棒杆菌单位荧光
图4 八个对称性BDPs在大肠杆菌与谷氨酸棒杆菌中的单位荧光
Fig.4 Unit fluorescence of eight symmetrical BDPs in E.coli and C.glutamicum
注:不同BDPs设置了3组生物学重复。
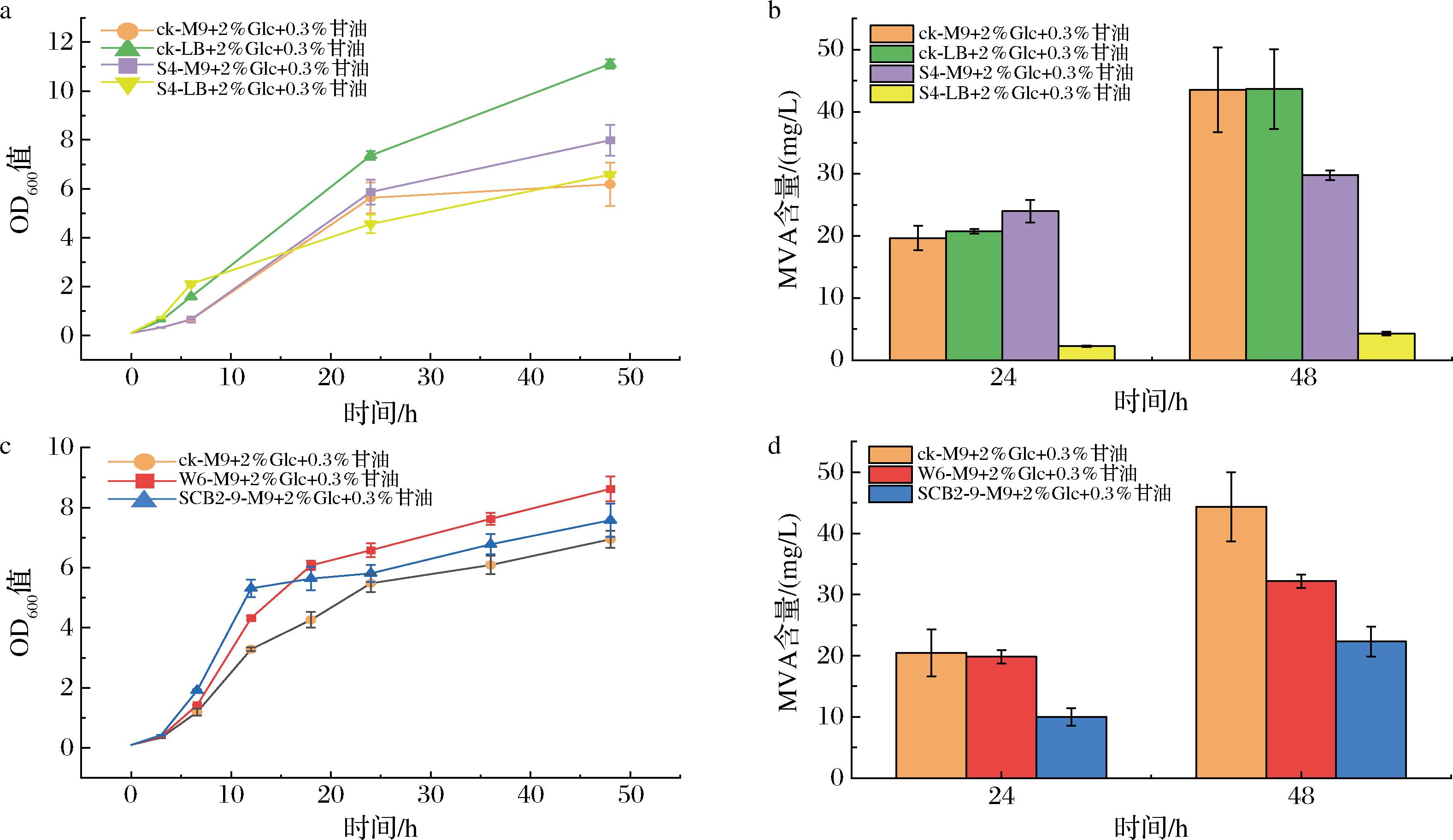
a-S4菌株与对照菌两种培养基48 h生长曲线与MVA产量;b-W6、SCB2-9菌株与对照菌M9培养基48 h生长曲线与MVA产量
图5 BDPs在MVA生产过程中的应用
Fig.5 Application of BDPs in MVA production process
随后将W6与SCB2-9启动子插入到p19-mvaE-BDP-mvaS载体构建了菌株W6与SCB2-9,接种到M9+2%葡萄糖+0.3%甘油的培养基中发酵24 h与48 h后测量MVA产量,间隔3 h测量OD值,结果如图5-b所示,W6菌株表现良好,24 h MVA产量为19.8 mg/L,与对照组接近,48 h MVA产量为32.2 mg/L,达到了对照组的72.6%,对照组MVA产量为44.35 mg/L,SCB2-9菌株MVA产量略低于W6菌株。
3 结论
双向启动子具有普遍性但其在生物技术领域中长期被忽略。实际上,相对于单向转录子,BDPs更多地参与基因的表达调控[7]。相对于单向启动子,BDPs更多的出现在2个相邻的基因间。BDPs曾用于酵母的多基因协同表达,通过协调两边基因的转录从而有效提高目标蛋白的产量[21]。
本实验前期根据自然界已知的BDPs序列以及谷氨酸棒杆菌中的固定保守序列设计了4种突变文库,利用STEP双分子荧光系统完成了在大肠杆菌中BDPs的快速筛选。随后设计了7种核心区对称的序列文库,完成了在谷氨酸棒杆菌中对BDPs的筛选。为了得到启动子两端的单独强度,设计了左红右绿的BDPs载体,得到了BDPs两端的转录活性,证明了BDPs的双边活性与单边活性不成正比。最后将筛选出的BDPs应用到了大肠杆菌MVA的生产过程中,证实了BDPs在协同表达不同基因的转录方面起到了很好的作用。然而,如何提高筛选出的BDPs活性是一个需要考虑的问题,本文中筛选出的BDPs调控的MVA产量最高只达到了IPTG诱导对照组的72.6%,扩大库容或是对筛选出的BDPs进行合理的改造或许能够筛选出活性更高的BDPs。同时,本研究仅对于BDPs两边的单个基因表达进行了研究,对于BDPs是否能有效应用于协调多个基因的表达还有待研究。BDPs的研究不仅可以应用于协调两边基因提高目标蛋白产量,同时还能用于遗传电路的设计与构建、遗传稳定性BDPs的筛选等实验中,具有很大的应用前景。
[1] AJIKUMAR P K, XIAO W H, TYO K E J, et al.Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli[J].Science, 2010, 330(6000):70-74.
[2] SMALL C L.Expression and assembly of active cyanobacterial ribulose 1, 5-biphosphate carboxylase/oxygenase in Escherichia coli[D].University of Texas at Austin, 1985.
[3] SONODA H, KUMADA Y, KATSUDA T, et al.Functional expression of single-chain Fv antibody in the cytoplasm of Escherichia coli by thioredoxin fusion and co-expression of molecular chaperones[J].Protein Expression and Purification, 2010, 70(2):248-253.
[4] PADDON C J, WESTFALL P J, PITERA D J, et al.High-level semi-synthetic production of the potent antimalarial artemisinin[J].Nature, 2013, 496(7446):528-532.
[5] GALANIE S, THODEY K, TRENCHARD I J, et al.Complete biosynthesis of opioids in yeast[J].Science, 2015, 349(6252):1095-1100.
[6] TAN S Z, PRATHER K L.Dynamic pathway regulation:Recent advances and methods of construction[J].Current Opinion in Chemical Biology, 2017, 41:28-35.
[7] WARMAN E A, FORREST D, GUEST T, et al.Widespread divergent transcription from bacterial and archaeal promoters is a consequence of DNA-sequence symmetry[J].Nature Microbiology, 2021, 6(6):746-756.
[8] YANG S, SLEIGHT S C, SAURO H M.Rationally designed bidirectional promoter improves the evolutionary stability of synthetic genetic circuits[J].Nucleic Acids Research, 2013, 41(1):e33-e33.
[9] JODLBAUER J, RIEDER L, GLIEDER A, et al.Bidirectional Promoter Libraries Enable the Balanced Co-expression of Two Target Genes in E.coli[M]//Inclusion Bodies:Methods and Protocols.New York, NY:Springer US, 2023:75-86.
[10] ÖZTÜRK S, ERGÜN B G, ÇAL K P.Double promoter expression systems for recombinant protein production by industrial microorganisms[J].Applied Microbiology and Biotechnology, 2017, 101(20):7459-7475.
K P.Double promoter expression systems for recombinant protein production by industrial microorganisms[J].Applied Microbiology and Biotechnology, 2017, 101(20):7459-7475.
[11] WU X X, LI F Z, YANG R F, et al.Identification of a bidirectional promoter from Trichoderma reesei and its application in dual gene expression[J].Journal of Fungi, 2022, 8(10):1059.
[12] YANG L, YU J.A comparative analysis of divergently-paired genes (DPGs) among Drosophila and vertebrate genomes[J].BMC Evolutionary Biology, 2009, 9:55.
[13] TEODOROVIC S, WALLS C D, ELMENDORF H G.Bidirectional transcription is an inherent feature of Giardia lamblia promoters and contributes to an abundance of sterile antisense transcripts throughout the genome[J].Nucleic Acids Research, 2007, 35(8):2544-2553.
[14] XIE M T, HE Y H, GAN S S.Bidirectionalization of polar promoters in plants[J].Nature Biotechnology, 2001, 19(7):677-679.
[15] DHADI S R, KROM N, RAMAKRISHNA W.Genome-wide comparative analysis of putative bidirectional promoters from rice, Arabidopsis and Populus[J].Gene, 2009, 429(1-2):65-73.
[16] ADACHI N, LIEBER M R.Bidirectional gene organization:A common architectural feature of the human genome[J].Cell, 2002, 109(7):807-809.
[17] TRINKLEIN N D, ALDRED S F, HARTMAN S J, et al.An abundance of bidirectional promoters in the human genome[J].Genome Research, 2004, 14(1):62-66.
[18] FUX C, FUSSENEGGER M.Bidirectional expression units enable streptogramin-adjustable gene expression in mammalian cells[J].Biotechnology and Bioengineering, 2003, 83(5):618-625.
[19] AMENDOLA M, VENNERI M A, BIFFI A, et al.Coordinate dual-gene transgenesis by lentiviral vectors carrying synthetic bidirectional promoters[J].Nature Biotechnology, 2005, 23(1):108-116.
[20] PEREMARTI A, TWYMAN R M, G MEZ-GALERA S, et al.Promoter diversity in multigene transformation[J].Plant Molecular Biology, 2010, 73(4):363-378.
MEZ-GALERA S, et al.Promoter diversity in multigene transformation[J].Plant Molecular Biology, 2010, 73(4):363-378.
[21] VOGL T, KICKENWEIZ T, PITZER J, et al.Engineered bidirectional promoters enable rapid multi-gene co-expression optimization[J].Nature Communications, 2018, 9(1):3589.
[22] EASON M G, PANDELIEVA A T, MAYER M M, et al.Genetically encoded fluorescent biosensor for rapid detection of protein expression[J].ACS Synthetic Biology, 2020, 9(11):2955-2963.