黄酮类化合物是植物中一种非常重要的次生代谢产物,在植物生长发育中起着重要作用[1]。其生物合成途径受到许多外在因素的影响,如温度,光照和植物激素等[2]。MYB、bHLH、WD40和DELLA蛋白等是主要参与调控黄酮生物合成的转录因子[3]。但DELLA蛋白和MYB蛋白的功能还在不断探索中,对于揭示植物的黄酮调控机制具有重要意义。
在拟南芥基因组中包含5个不同的DELLA蛋白编码基因(GAI、RGA、RGL1、RGL2、RGL3),GAI蛋白是DELLA家族的一个调控因子,能够参与植物激素的信号转导途径,对植物根的形成、茎的伸长和叶片衰老等生长发育过程起到重要作用[4]。MYB基因是一类编码转录因子的基因家族,它们在植物中参与调控多种生物过程,包括次生代谢物的合成[5]。GAI蛋白与MYB蛋白之间的结合和相互作用对于黄酮生物合成途径的调控至关重要[6]。
研究表明,GAI蛋白与MYB蛋白可以直接或间接地相互作用,从而共同调控黄酮生物合成途径中的关键酶的表达[7]。目前在拟南芥等模式植物中对DELLA蛋白和MYB蛋白相互作用的研究比较多。例如,在拟南芥中,DELLA蛋白与MYB转录因子TT8结合并协同作用,以促进花瓣中黄酮合成途径关键基因的表达[8];TAN等[9]发现拟南芥中的DELLA蛋白与SG7MYB(MYB12和MYB111)相互作用,减少黄酮醇生物合成来促进根部生长。
前期研究结果发现,胰蛋白酶可以作为一种新型生物保鲜剂显著诱导黄酮类化合物的合成,延长果蔬保质期,对果蔬有明显的保鲜作用[10]。黄酮类化合物具有很强的抗氧化能力,可以作为天然的抗氧化剂抑制果蔬中的氧化反应,对果蔬细胞表现出很好的保护作用[11]。诸多研究表明,DELLA蛋白可以参与黄酮类化合物的生物合成途径,影响黄酮类化合物的生成,但胰蛋白酶是否通过DELLA蛋白与MYB作用,影响黄酮类物质的生物合成,从而提高贮藏期间果实的品质,仍需进一步说明。
火龙果果皮薄嫩,果肉含水量高,容易腐烂变质[12],提高火龙果果实耐贮性是火龙果研究中的一项重要工作。本研究以火龙果果实为实验材料,通过基因沉默(virus-induced gene silencing, VIGS)技术及理化指标分析,验证了HuGAI1可以增强果实抗性。通过对转录组数据分析,筛选出HuGAI1基因沉默后受调控的关键MYB基因,并通过蛋白互作网络分析筛选出与黄酮相关基因,验证MYB基因和黄酮相关基因的相互作用,为胰蛋白酶通过影响HuGAI1基因表达而提高火龙果保鲜效果提供参考。
1 材料与方法
1.1 材料、试剂与仪器
采用周边市场购买的同一批次的新鲜火龙果果实,选择大小、形状相似,颜色相近,无机械损伤和虫害污染的果实进行试验。
胰蛋白酶(牛源,5×105 U/g),美国Amersco公司。
台式高速冷冻离心机,湘仪离心机仪器有限公司;722S型分光光度计,上海精密科学仪器有限公司;荧光定量PCR仪,伯乐生命医学产品上海有限公司。
1.2 实验方法
1.2.1 处理方法
将胰蛋白酶溶于蒸馏水中,终质量浓度为56.07 mg/L(2.41×10-6 mol/L)。用胰蛋白酶溶液浸泡火龙果15 s,对照组用蒸馏水同样处理。25 ℃放置,每隔24 h,观察、拍照、记录、测定失水率。贮藏1周后,用手术刀切下处理过的火龙果表皮,迅速放入液氮3~5 min,储存在-80 ℃下,为进行后续实验做准备[13]。
1.2.2 VIGS载体构建以及诱导火龙果HuGAI1基因沉默
采用RNAprep Pure植物总RNA提取试剂盒提取实验各组的火龙果果皮的总RNA[14],用HiFi-MMLV cDNA Kit合成cDNA,利用SnapGene软件设计引物,添加EcoR I和BamH I酶切位点。以获得的cDNA为模板,用表1中引物进行PCR扩增。将PCR产物与pTRV2载体同时进行双酶切,进行连接重组,重组产物转化入大肠杆菌Top10感受态细胞中。挑取阳性克隆,双酶切验证。将测序正确的阳性克隆培养提取质粒,转化农杆菌GV3101,于-80 ℃保存备用。分别将保藏的含有pTRV1、HuGAI1-pTRV2的菌液以1%的接种量接种到20 mL的LB液体培养基中,200 r/min、28 ℃培养至菌液混浊(24~36 h)后,吸取2 mL菌液至200 mL的LB培养基中,200 r/min、28 ℃培养至菌液OD600=0.6左右。5 000 r/min、离心10 min,弃上清液,收集菌体。用现配的侵染缓冲液(含有终浓度为200 μmol/L的乙酰丁香酮、10 mmol/L的2-(N-吗啉)乙磺酸和10 mmol/L的MgCl2)重悬菌体。将含有pTRV1的重悬液和含有HuGAI1-pTRV2的重悬液以1∶1等体积混匀,采用注射法侵染火龙果果皮,注射后的火龙果避光放置48 h。侵染后的火龙果用胰蛋白酶溶液浸泡15 s,正常光照贮藏。
表1 转录组数据RT-qPCR验证引物序列
Table 1 Primer sequences used in RT-qPCR validation of transcriptome data
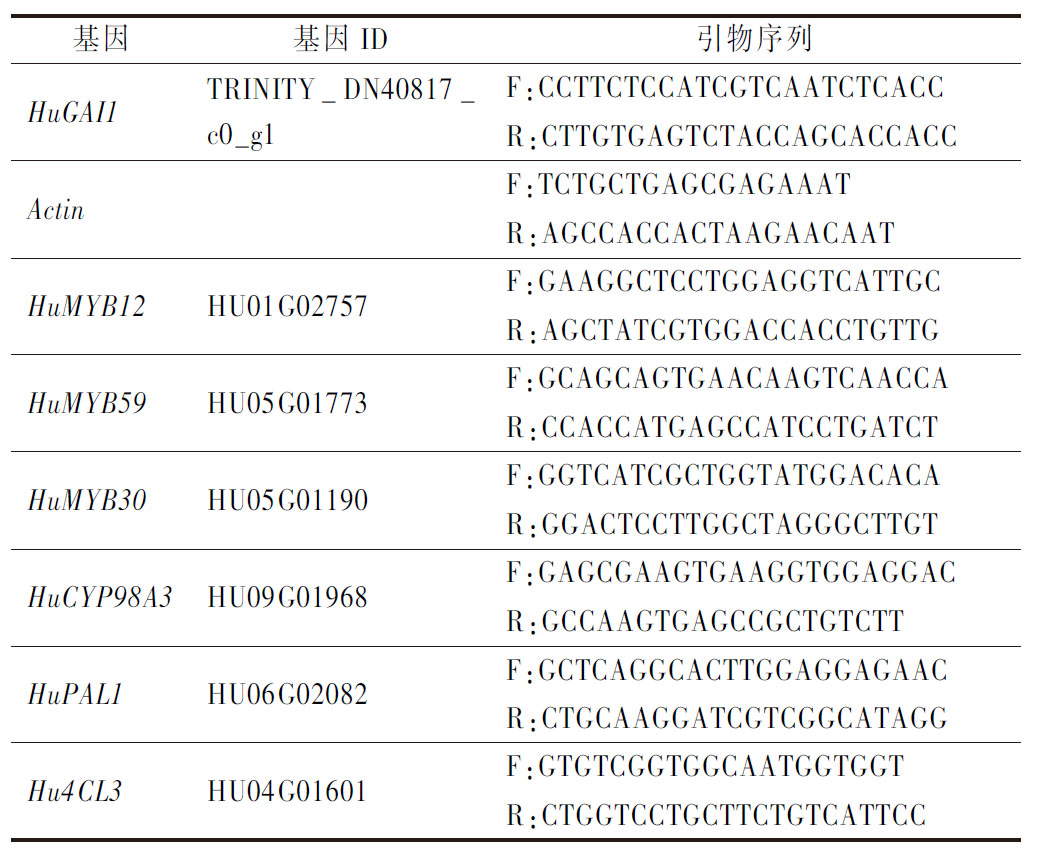
基因基因ID引物序列HuGAI1TRINITY_DN40817_c0_g1F:CCTTCTCCATCGTCAATCTCACCR:CTTGTGAGTCTACCAGCAC-CACCActinF:TCTGCTGAGCGAGAAATR:AGCCACCACTAAGAACAATHuMYB12HU01G02757F:GAAGGCTCCTGGAGGTCATTGCR:AGCTATCGTGGACCACCTGTTGHuMYB59HU05G01773F:GCAGCAGTGAACAAGTCAACCAR:CCACCATGAGCCATCCTGATCTHuMYB30HU05G01190F:GGTCATCGCTGGTATGGACACAR:GGACTCCTTGGCTAGGGCTTGTHuCYP98A3HU09G01968F:GAGCGAAGTGAAGGTGGAG-GACR:GCCAAGTGAGCCGCTGTCTTHuPAL1HU06G02082F:GCTCAGGCACTTGGAGGAGAACR:CTGCAAGGATCGTCGGCATAGGHu4CL3HU04G01601F:GTGTCGGTGGCAATGGTGGTR:CTGGTCCTGCTTCTGTCATTCC
1.2.3 转录组学分析
真核生物mRNAs测序技术主要是利用高通量测序技术,对某一特定时间点上某一特定组织和细胞中表达的mRNA进行序列分析。本研究利用Illumina TruSeqTM RNAsample prep kit的方法,进行了测序试验并构建文库[15]。从组织样本中提取总RNA,Nanodrop 2000检测提取RNA的含量及纯度,琼脂糖电泳检测其完整性,Agilent 2100检测其RIN值。Illumina HiSeq测序:PCR扩增15个循环对文库进行富集;在2%琼脂糖凝胶上回收目标条带;按照数据比例进行混合上机,TBS380定量(Picogreen);用cBot对其进行桥式PCR扩增,得到簇(cluster);Illumina HiSeq序列测定(PE文库,读长2×150 bp)。
1.2.4 RT-qPCR验证和理化指标测定
以火龙果Actin基因为内参基因[16],通过2-ΔΔCt计算差异基因表达水平的方法,实验重复3次[17]。所有引物均列于表1中。采用WANG等[13]的方法定期测定其失重率。
1.2.3 数据统计
统计分析采用SPSS统计软件包(11.0.1)进行。非参数检验(威尔科克森检验)用于分析整组样本之间的差异。使用配对样本T检验确定一个时间点样本间差异的显著性。显著或极显著差异用P<0.05或P<0.01评价[18]。
2 结果与分析
2.1 VIGS载体构建
HuGAI1的开放阅读框(open reading frame, ORF)全长1 755 bp,编码585个氨基酸的多肽,相对分子质量为64.59 kDa,理论等电点为5.202。通过DNAMAN软件将与HuGAI1同源性较高的不同物种氨基酸序列进行比对(图1-a),发现HuGAI1基因编码的蛋白质产物具有GRAS家族所具有的典型结构域,如在DELLA蛋白N端存在的DELLA与TVHYNP 2个保守区域,中间包含一个核定位信号域NLS;在后面有一个保守氨基酸结构域VHVID以及其两端的亮氨酸重复序列;在序列C端有保守的PFYRE、SAW结构域。表明HuGAI1属于DELLA家族的GAI转录因子。通过NCBI上Blast在线比对工具,将检索到的12条与HuGAI1同源性较高的蛋白进行多序列比对,使用MEGA 6.0软件构建进化树(图1-b),结果表明火龙果HuGAI1与DELLA家族的GAI具有同源性。利用HuGAI1的序列特征,在其上下游分别加入EcoR I和BamH I酶切位点设计特异性引物。以火龙果cDNA为模板扩增HuGAI1基因片段,将目的片段连接到pTRV2载体上(图1-c),转入大肠杆菌(Escherichia coli)Top10感受态细胞,提取成功连接的HuGAI1-pTRV2质粒,转入农杆菌GV3101保存备用。

a-HuGAI1蛋白序列比对图;b-HuGAI1序列比对发育树;c-基因沉默示意图
图1 载体构建图
Fig.1 Vector construction diagram
2.2 HuGAI1沉默对火龙果果实表型的影响
贮藏初期,各组果实外观红润,颜色鲜艳,硬度适中,贮藏6 d时,均颜色变深,硬度下降,色泽一般(图2-a)。其中TV-GAI1组(VIGS组)果实出现局部变软,表皮稍有萎蔫,鳞片略有干枯。贮藏时间增加到12 d,Try组(胰蛋白酶处理组)表现良好,果实表面开始出现少量斑点,色泽暗淡,CK组(空白处理组)鳞片干枯,TV-GAI1组果实完全萎蔫,出现大量病变斑点,不可食用。随着保藏时间的加长,果实都表现出了不同程度质量损失(图2-b),并且这种趋势是上升的,后期相对平缓,前3 d曲线斜率最大原因是火龙果最初贮藏时,蒸腾作用较明显,失重率较明显,随着时间的延长相对减弱,贮藏9 d后,质量损失率改变相对较小,后期组间差距也相对变大,贮藏12 d时,TV-GAI1组的果实失重率最大(13.59%),空白对照组次之(12.69%),胰蛋白酶处理组失重率最低(10.78%)。贮藏期间TV-GAI1组火龙果果皮黄酮含量整体较低,Try组黄酮含量整体较高,HuGAI1沉默影响了果皮中黄酮含量(图2-c)。说明胰蛋白酶处理可以延缓火龙果果实失水率,提高火龙果果皮黄酮含量。在火龙果果实外观也有一定的保鲜作用,延长了火龙果果实的保鲜时间。
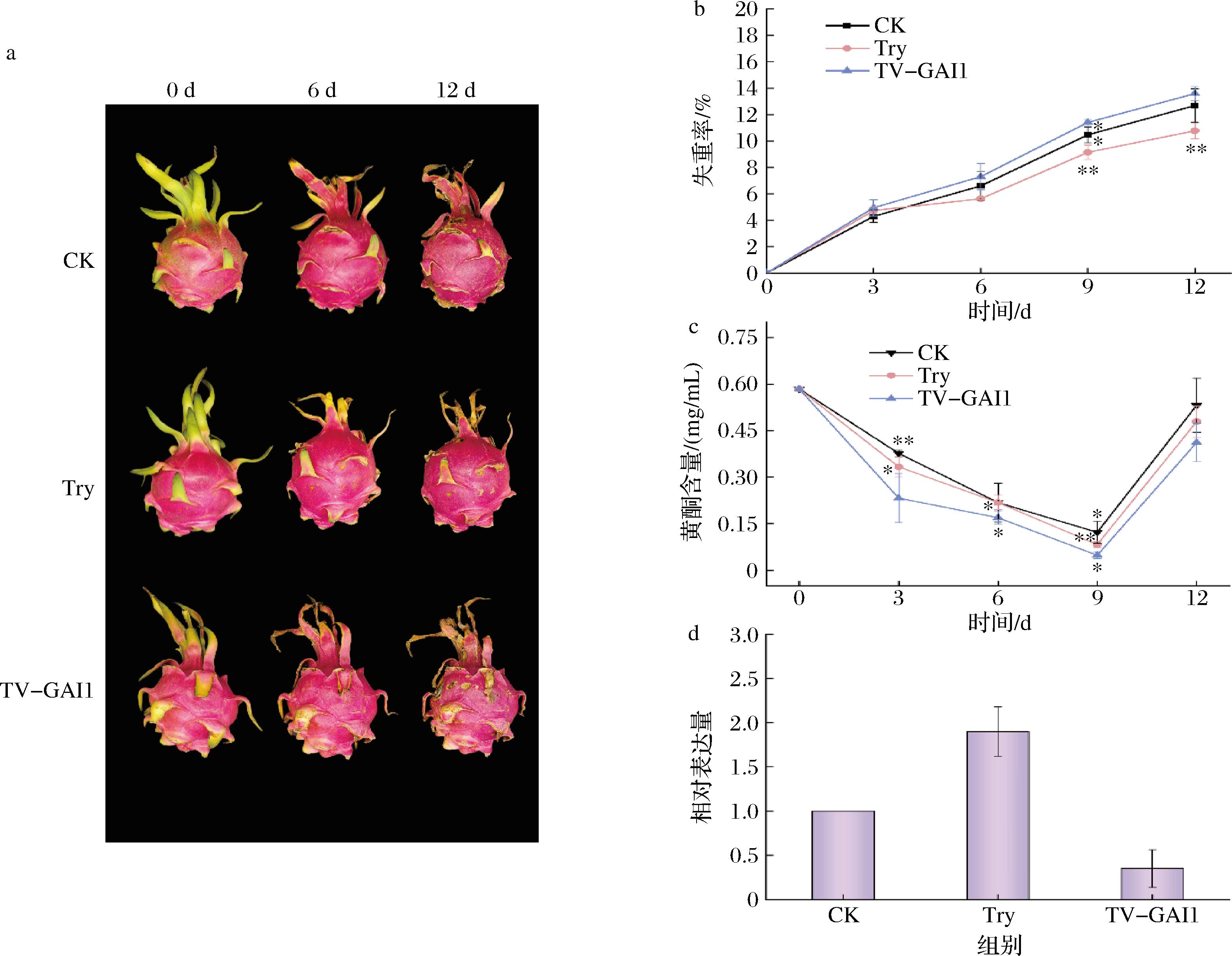
a-火龙果表型图;b-火龙果失重率;c-火龙果果皮黄酮含量;d-基因表达验证
图2 VIGS对火龙果的影响
Fig.2 Effect of VIGS on Hylocereus undatus
注:CK代表空白处理组,Try代表胰蛋白酶处理组,TV-GAI1代表VIGS组,*表示具有显著性差异(P<0.05),**表示具有显著性差异(P<0.01)。
通过荧光定量PCR验证发现(图2-d),与CK组相比,Try组的HuGAI1基因表达量明显升高,说明胰蛋白酶处理促进了HuGAI1基因的表达;而TV-GAI1组HuGAI1基因表达量降低,证明火龙果HuGAI1基因沉默是有效的。进一步证明胰蛋白酶可能通过促进HuGAI1的表达,起到了保鲜的作用。
2.3 HuGAI1和HuMYB12结合调控的次生代谢途径及关键的MYB基因
将Try组与HuGAI1沉默组的转录组数据进行比较,分析差异表达基因。主成分分析(principal component analysis, PCA)中(图3-a)主成分1(PC1)的贡献率为66.06%,主成分2(PC2)的贡献率为11.81%,Try组和沉默组同组别的3个样品聚集在一起,而不同组别的样本表现出明显的分离趋势,说明同一组别样本数据稳定性较好,而不同组别间具有一定的差异性。同时在两组别间共筛选出上调基因3 347个,下调基因4 021个(图3-b)。

a-样本间PCA图;b-样本间差异基因火山图;c-MYB基因聚类热图;d-MYB基因子聚类趋势图
图3 差异表达基因的转录组学分析
Fig.3 Transcriptomic analysis of differentially expressed genes
对火龙果沉默HuGAI1基因组样本进行转录因子分析,共筛选注释出19个MYB基因。在这19个MYB基因中,MYB基因热图(图3-c)显示差异倍数最高的为MYB12(HU01G02757),其中16个基因上调,3个基因下调(图3-d)。作为MYB家族的转录因子,猜测MYB12可能影响了相关物质的合成代谢,从而影响果实的抗病能力。
2.4 黄酮相关基因的互作网络
通过对沉默HuGAI1的转录组进行分析,筛选出受调控的98个黄酮相关基因,利用Cytoscape软件进行相关性分析。通过Cytoscape构建的共表达网络图由50个节点和175条边组成(图4-a)。利用软件内插件MCODE将基因分出一个簇,簇内各节点如C4H参与苯丙烷类物质的生物合成。基于cytoHubba对黄酮基因进行“BottleNeck”排序(图4-b),从基因之间的互作关系筛选出处于中心位置的3个黄酮基因,HuCYP98A3(HU09G01968)、HuPAL1(HU06G02082)和Hu4CL3(HU04G01601)。

a-黄酮相关基因共表达总网络图;b-黄酮差异基因Top10蛋白互作网络图
图4 表达相关性网络分析
Fig.4 Expression correlation network analysis
2.5 RT-qPCR验证转录组学数据
通过RT-qPCR验证分别检测了MYB12、MYB59和MYB30基因和黄酮基因的转录组数据的表达情况。基因的RT-qPCR与RNA-seq的结果一致,说明转录组学数据可靠(图5-a)。结果表明,这些基因在RNA-seq和RT-qPCR中的表达模式是相似的(图5-b),即可表明RNA-seq和RT-qPCR定量检测基因表达水平的结果是一致的。
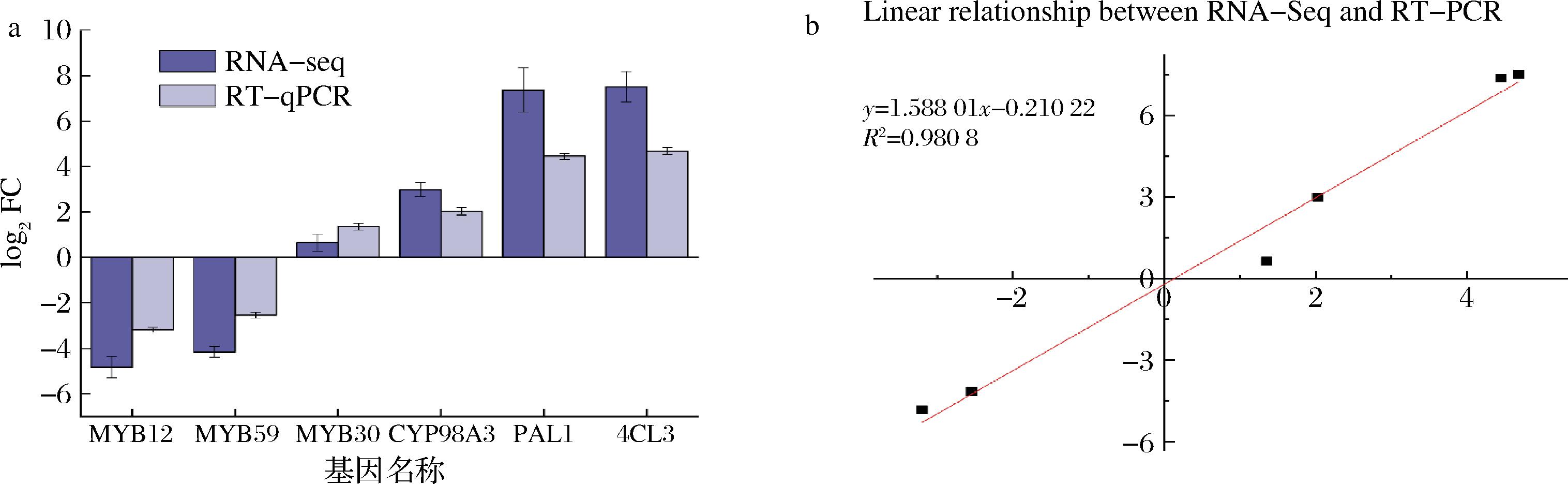
a-RT-qPCR验证不同处理组中基因的表达情况;b-基因表达量的线性关系
图5 RT-qPCR验证
Fig.5 RT-qPCR verify
3 结论
黄酮是一类在植物中广泛存在的天然化合物,具有多种生理活性和营养功能[19]。植物中的黄酮类化合物的生物合成途径受到多个基因的调控,包括苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL)、肉桂酸4-羟化酶(cinnamate 4-hydroxylase,C4H)、肉桂酸CoA连接酶(4-coumarate-CoA ligase, 4CL)等[20]。这些基因编码的酶参与了黄酮生物合成途径的各个关键步骤。GAI作为DELLA蛋白家族的转录因子参与植物的生长发育,但并不直接参与植物中的黄酮合成途径[21],GAI蛋白可以与MYB蛋白结合,激活或抑制黄酮生物合成途径中的关键酶的表达,从而影响植物中黄酮的合成和积累。
在本研究中,胰蛋白酶处理采后火龙果果实有效地减缓了果实衰老和腐败。研究采用VIGS技术沉默HuGAI1,实验结果表明,在贮藏12 d后,胰蛋白酶处理组火龙果果实表现良好,失重率最低,表面出现少量病斑,而沉默组果实果皮出现大量斑点,鳞片完全干枯。说明将HuGAI1基因沉默后,HuGAI1表达量下降,火龙果果实腐败加剧,胰蛋白酶能够上调HuGAI1表达,减缓火龙果果实失水率,提高果皮黄酮含量,延缓火龙果腐败。分析VIGS技术处理HuGAI1后的转录组学数据,结果表明,胰蛋白酶作为一种新型超氧化物清除剂,对类黄酮生物合成系统具有调节作用。此外,基于蛋白网络互作图筛选出的黄酮相关基因,核心位置排名前三的为HuCYP98A3、HuPAL1和Hu4CL3,最靠前的是HuCYP98A3,属于细胞色素P450酶家族,是一种参与植物次生代谢的酶,它主要参与花青素生物合成途径中的单体类黄酮的羟化反应。它能够催化单体类黄酮分子中的顺式-3′-羟基化反应,形成3′-羟基单体类黄酮,从而进入到下一步的花青素合成中[22]。PCL是一个植物转录因子家族,在植物的生长和发育中发挥重要作用[23]。4CL主要参与植物的苦草酸途径和苯丙烷类的合成途径,产生木质素等多种次生代谢产物,提高植物抗氧化及清除自由基的能力[24]。黄酮类物质在植物抗氧化方面发挥着积极影响,对果实减缓腐败起到一定作用。
总而言之,在胰蛋白酶作用下可以上调HuGAI1的表达,HuGAI1和HuMYB12基因相互作用参与到黄酮的生物合成,提高黄酮生物合成途径相关基因的表达,进而提高果实中黄酮类物质含量,增强果实细胞的抗氧化能力,实现了延缓火龙果果实衰老腐败的作用。本文研究对胰蛋白酶提高果蔬保鲜机理提供了新的理论支撑。
[1] MAL G, DONG C M, SONG C, et al.De novo genome assembly of the potent medicinal plant Rehmannia glutinosa using nanopore technology[J].Computational and Structural Biotechnology Journal, 2021, 19:3954-3963.
[2] TIAN S P, QIN G Z, LI B Q.Reactive oxygen species involved in regulating fruit senescence and fungal pathogenicity[J].Plant Molecular Biology, 2013, 82(6):593-602.
[3] XU Z S, YANG Q Q, FENG K, et al.DcMYB113, a root-specific R2R3-MYB, conditions anthocyanin biosynthesis and modification in carrot[J].Plant Biotechnology Journal, 2020, 18(7):1585-1597.
[4] KHAN Y, XIONG Z, ZHANG H, et al.Expression and roles of GRAS gene family in plant growth, signal transduction, biotic and abiotic stress resistance and symbiosis formation:A review[J].Plant Biology, 2022, 24(3):404-416.
[5] DELUC L, BARRIEU F, MARCHIVE C, et al.Characterization of a grapevine R2R3-MYB transcription factor that regulates the phenylpropanoid pathway[J].Plant Physiology, 2006, 140(2):499-511.
[6] 文慧. OsWRKY5与OsMYB1R调控Pik-H4介导稻瘟病抗性的研究[D].广州:华南农业大学, 2020.WEN H.Preliminary study of OsWRKY5 and OsMYB1R in regulating Pik-H4-mediated resistance to rice blast[D].Guangzhou:South China Agricultural University, 2020.
[7] MOROHASHI K, GROTEWOLD E.A systems approach reveals regulatory circuitry for Arabidopsis trichome initiation by the GL3 and GL1 selectors[J].PLoS Genetics, 2009, 5(2):e1000396.
[8] XIE Y, TAN H J, MA Z X, et al.DELLA proteins promote anthocyanin biosynthesis via sequestering MYBL2 and JAZ suppressors of the MYB/bHLH/WD40 complex in Arabidopsis thaliana[J].Molecular Plant, 2016, 9(5):711-721.
[9] TAN H J, MAN C, XIE Y, et al.A crucial role of GA-regulated flavonol biosynthesis in root growth of Arabidopsis[J].Molecular Plant, 2019, 12(4):521-537.
[10] LI X, LIU X R, YIN Y, et al.Transcriptomic analysis reveals key genes related to antioxidant mechanisms of Hylocereus undatus quality improvement by trypsin during storage[J].Food &Function, 2019, 10(12):8116-8128.
[11] XIE Y X, YANG W J, TANG F, et al.Antibacterial activities of flavonoids:Structure-activity relationship and mechanism[J].Current Medicinal Chemistry, 2015, 22(1):132-149.
[12] HUANG Y Y, BRENNAN M A, KASAPIS S, et al.Maturation process, nutritional profile, bioactivities and utilisation in food products of red pitaya fruits:A review[J].Foods, 2021, 10(11):2862.
[13] WANG J, TIAN P P, SUN J J, et al.CsMYC2 is involved in the regulation of phenylpropanoid biosynthesis induced by trypsin in cucumber (Cucumis sativus) during storage[J].Plant Physiology and Biochemistry, 2023, 196:65-74.
[14] ZHANG J Q, WU Z C, HU F C, et al.Aberrant seed development in Litchi chinensis is associated with the impaired expression of cell wall invertase genes[J].Horticulture Research, 2018, 5:39.
[15] ZHANG Y Y, LI B R, ZHANG M, et al.Transcriptome analyses and virus-induced gene silencing identify HuWRKY40 acting as a hub transcription factor in the preservation of Hylocereus undatus by trypsin[J].Journal of Food Biochemistry, 2022, 46(12):e14437.
[16] NIE Q, GAO G L, FAN Q J, et al. Isolation and characterization of a catalase gene “Hucat3” from pitaya (Hylocereus Undatus) and its expression under abiotic stress[J]. Gene, 2015, 563(1): 63-71.
[17] LI X, ZHANG Y Y, WU Y, et al.Lipid metabolism regulated by superoxide scavenger trypsin in Hylocereus undatus through multi-omics analyses[J].Journal of Food Biochemistry, 2022, 46(7):e14144.
[18] LIU J, GAO Y, GONG F F, et al.The transcriptome and metabolome reveal stress responses in sulfur-fumigated cucumber (Cucumis sativus L.)[J].Frontiers in Plant Science, 2021, 12:778956.
[19] FANG Y M, LI R, CHU Z, et al.Chemical and flavor profile changes of cocoa beans (Theobroma cacao L.) during primary fermentation[J].Food Science &Nutrition, 2020, 8(8):4121-4133.
[20] FISCHER T C, MIRBETH B, RENTSCH J, et al.Premature and ectopic anthocyanin formation by silencing of anthocyanidin reductase in strawberry (Fragaria×Ananassa)[J].New Phytologist, 2014, 201(2):440-451.
[21] ZHANG Z L, OGAWA M, FLEET C M, et al.Scarecrow-like 3 promotes gibberellin signaling by antagonizing master growth repressor DELLA in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(5):2160-2165.
[22] WANG Z H, WANG W P, WU W T, et al.Integrated analysis of transcriptome, metabolome, and histochemistry reveals the response mechanisms of different ages Panax notoginseng to root-knot nematode infection[J].Frontiers in Plant Science, 2023, 14:1258316.
[23] ZHU C, ZHANG S T, FU H F, et al.Transcriptome and phytochemical analyses provide new insights into long non-coding RNAs modulating characteristic secondary metabolites of oolong tea (Camellia sinensis) in solar-withering[J].Frontiers in Plant Science, 2019, 10:1638.
[24] ZHOU X, CAO J, LIU X M, et al.Cloning and functional analysis of Gb4CL1 and Gb4CL2 from Ginkgo biloba[J].The Plant Genome, 2024, 17(2):e20440.