农作物秸秆是地球上最丰富的生物质来源之一,主要由木质纤维素生物质(lignocellulosic biomass,LCB)组成[1]。目前,由于秸秆品种多、产量大、组成结构复杂、转化率低等问题,以及秸秆焚烧、掩埋等粗加工方法,造成秸秆资源大量浪费,环境污染严重[2]。因此,开发快速有效的秸秆处理技术至关重要,微生物降解是具有生态效益的生物处理方法。降解木质纤维素微生物(例如真菌、细菌等)通过分泌碳水化合物活性酶(carbohydrate active enzymes,CAZymes),将秸秆的木质纤维素降解为还原糖[3],同时有利于微生物生长,可以从土壤、腐烂枝叶、动物粪便等物质中分离获得[4]。
粘质沙雷氏菌广泛分布于多种环境中,具有降解木质纤维素等多种功能,此前有研究分离筛选粘质沙雷氏菌及其产酶情况。KWAK等[5]研究表明,粘质沙雷氏菌可以通过生化和分子生物学手段产生纤维素酶等细胞外酶;傅慧静等[6]发现,粘质沙雷氏菌是松褐天牛幼虫肠道细菌群落的优势细菌,可以分泌降解木质素的木质素过氧化物酶和锰过氧化物酶。NARENDRA KUMAR等[7]从含有腐烂树叶凋落物的土壤中分离出的粘质沙雷氏菌的内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶活力分别达1.1、1.1、1.2 U/mL,并对产酶条件进行了优化。但鲜见对粘质沙雷氏菌全基因组进行测序的研究,高通量测序技术是分析菌株全基因组并挖掘其相关功能基因的有效方法,是研究菌株特性、阐明其降解木质纤维素的酶活特性,进一步提高菌株在生物质转化中的应用价值的有效方法。
本研究采用刚果红染色法结合酶活性评价,从自然界特殊环境中筛选分离出一种具有木质纤维素降解能力的粘质沙雷氏菌EC-011。通过基因组测序和分析获取其生物信息特征,揭示了其降解木质纤维素的遗传基础,并对其产酶条件进行了优化,为更好地开发粘质沙雷氏菌EC-011降解木质纤维素提供了生物信息基础和理论依据。
1 材料与方法
1.1 实验材料
1.1.1 样品来源
样品采自黑龙江省、内蒙古自治区、山东省等地的林地腐木腐叶、青贮黄贮饲料、秸秆沼气发酵池、牛羊粪、腐熟秸秆堆等处,共109份样品。
1.1.2 试剂及培养基
微晶纤维素、木聚糖、水杨苷,源叶生物科技(上海)有限公司;羧甲基纤维素钠(sodium carboxymethylcellulose,CMC-Na)等其他试剂均为国产分析纯,福晨(天津)化学试剂有限公司。
纤维素培养基(g/L):MgSO4 0.5,(NH4)2SO4 2,K2HPO4 1,NaCl 0.5,CMC-Na 10,琼脂20。
木聚糖培养基(g/L):(NH4)2SO4 2,MgSO4 0.5,K2HPO41,NaCl 0.5,木聚糖2,琼脂20。
斜面培养基(g/L):牛肉膏3,蛋白胨10,NaCl 5,琼脂20。
种子培养基(g/L):蛋白胨5,牛肉膏10,葡萄糖5,酵母粉5,NaCl 5。
产酶培养基(g/L):(NH4)2 SO4 5,K2HPO41,MgSO4 0.5,酵母粉5,木聚糖5,CMC-Na 20。
1.1.3 实验仪器
ZHWY-2102C恒温培养振荡器,上海智城分析仪器制造有限公司;JA2003分析天平,上海良平仪器仪表有限公司;PHS-3C pH计,上海浦春计量仪器有限公司;DZKW-5-4电热恒温水浴锅,天津市天泰仪器有限公司;722S紫外分光光度计,上海菁华科技仪器公司;TGL-16 G台式离心机,上海安亭科学仪器厂。
1.2 筛选方法
初筛:100 mL锥形瓶中装入50 mL无菌水和5 g样品,30 ℃、180 r/min振荡30 min,吸取1.0 mL样品悬液梯度稀释至10-6;取不同稀释度的稀释液涂布于纤维素和木聚糖培养基中,30 ℃倒置培养48 h。之后用1%刚果红溶液浸染1 h,弃掉溶液,1 mol/L NaCl溶液进行脱色,选取有透明圈的菌株纯化保藏。
复筛:保存菌种活化后,按1%的接种量接种于产酶培养基中,30 ℃、180 r/min培养72 h。发酵液经8 000 r/min离心10 min后,将上清稀释数倍后采用DNS法测定酶活性,酶活性较高的菌株确定为研究菌株。
1.3 酶活性测定方法
1.3.1 纤维素酶活性测定
分别取1.5 mL不同浓度用柠檬酸-柠檬酸钠缓冲液配制的葡萄糖标准品溶液,加入3 mL DNS试剂煮沸5 min,迅速冷却至室温,定容至10 mL,540 nm处测吸光度值,绘制标准曲线。分别使用CMC-Na、微晶纤维素和水杨苷进行内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶测定,并使用滤纸酶活性(filter paper activity,FPA)测定总纤维素酶活性[8]。
1.3.2 FPA测定
取4支具塞试管,各放入一条Whatman滤纸(1 cm×2 cm),依次加入1.5 mL柠檬酸-柠檬酸钠缓冲液、0.5 mL酶液(对照组为灭活的酶液),涡旋混匀后37 ℃水浴反应1 h,加入3 mL DNS试剂后立即煮沸5 min,迅速冷却至室温,用蒸馏水定容至10 mL。在540 nm处测吸光度值A1(空白为A0)。
酶活力单位定义:1 mL酶液与底物反应30 min产生1 μmol葡萄糖为1个酶活力单位(U)。
1.4 菌种鉴定
生理生化鉴定:复筛菌株活化后在木聚糖平板上稀释涂布,37 ℃培养48 h后观察菌落形态并进行生理生化试验鉴定[9-10]。
根据方法进行细菌总DNA的提取。使用PCR引物(引物序列为27F:AGAGTTTGATCMTGGCTCAG,1492R:GGTTACCTTGTTACGACTT)对纯化菌种进行PCR扩增。
PCR反应体系[11]为:2×Taq PCR Master Mix 12.5 μL;引物F (10 μmol/L) 1 μL;引物R (10 μmol/L)1 μL;Template (DNA) 1 μL;9.5 μL ddH2O补足到25 μL。
PCR反应条件:95 ℃预变性5 min;94 ℃变性30 s、57 ℃退火30 s、72 ℃延伸90 s,共30个循环;72 ℃延伸10 min。
对PCR扩增产物进行1.5%琼脂糖凝胶电泳检测。符合预期要求后,将PCR产物送至生工生物工程(上海)股份有限公司进行16S RNA测序。
测序结果于2024年7月15日在NCBI(National Center of Biotechnology Information)网站(https://www.ncbi.nlm.nih.gov/genbank/)中进行blast序列比对,运用MEGA 11.0构建系统发育树。
1.5 全基因组测序
本次全基因测序委托上海美吉生物医药科技有限公司完成。采用二代测序和第三代高通量测序平台完成测序分析,使用fastp0.20.0等质量统计软件完成原始数据质控、去除低质量序列等工作。
1.6 基因组组装
细菌基因组扫描图利用短序列组装软件SOAPdenovo2.04(http://soap.genomics.org.cn/)对二代测序后的优化序列进行多个Kmer参数的拼接,得到最优的contigs组装结果,然后把reads比对到contig上,根据reads的paired-end和overlap关系,对组装结果进行局部组装和优化,形成scaffolds。
细菌基因组完成图利用组装软件unicycler v0.4.8进行三代序列组装,组装过程中会借助pilonjin软件进行序列校正,如果最终组装序列两端存在一定长度以上的overlap,则将序列成环并截去其中一端overlap序列,可得到完整的染色体及质粒序列,及最终组装结果[12]。
1.7 基因预测
利用Glimmer(http://ccb.jhu.edu/software/glimmer/index.shtml)、GeneMarkS、Prodigal软件对基因组中的编码序列(coding sequence,CDS)进行预测。扫描图组装结果默认使用Prodigal进行预测,完成图组装结果默认使用Prodigal预测染色体基因组,使用GeneMarkS 4.3预测质粒基因组。
利用tRNAscan-SE v2.0软件(http://trna.ucsc.edu/software/)对基因组中的tRNA进行预测,获得tRNA的核苷酸序列信息、反密码子信息及二级结构信息。利用Barrnap软件(https://github.com/tseemann/barrnap)对基因组中包含的rRNA进行预测,获得每个样本基因组中所有rRNA的种类、位置、序列信息[13]。
1.8 绘制基因组圈图
将测序得到的基因组序列、编码基因预测及非编码RNA等信息文件组装成GBK文件,通过CGView(http://wishart.biology.ualberta.ca/cgview/download.html)绘制基因组圈图,全方面展示测序基因组特征[14]。
1.9 基因功能分析
预测的编码蛋白序列分别与直系同源蛋白聚簇(cluster of orthologous groups of proteins,COG)、基因本体论(gene ontology,GO)、京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)、碳水化合物活性酶(carbohydrate-active enzymes database,CAZY)数据库中的蛋白序列进行blast比对和功能注释[15-16]。
1.10 产酶工艺优化
取活化好的菌液,接入液体产酶发酵培养基中,在不同发酵温度(25、30、35、40、45 ℃)、pH值(4、5、6、7、8)、培养时间(24、48、72、96、120 h)、接种量(2%、3%、4%、5%、6%)下测定FPA,进行单因素试验,每组各3个平行。
在单因素试验结果的初步分析基础上,进行菌株产酶条件的优化。以发酵温度、培养时间、接种量和pH作为4个因素,FPA作为响应值,采用Design expert 13.0进行Box-Behnken设计试验,建模得出响应面图,分析得到粘质沙雷氏菌EC-011的最佳产酶条件,并进行验证试验。
2 结果与分析
2.1 菌株酶活性初步测定
如图1-a和图1-b所示,经初筛、复筛得到一株菌株,在CMC-Na、木聚糖培养基中均出现水解圈,初步判断该菌株能降解纤维素和半纤维素,并测定其酶活性,经测定内切葡聚糖酶、外切葡聚糖酶、β-葡萄糖苷酶、滤纸酶活性、木聚糖酶酶活性分别为0.76、0.32、0.95、0.84、1.05 U/mL,将其命名为EC-011。
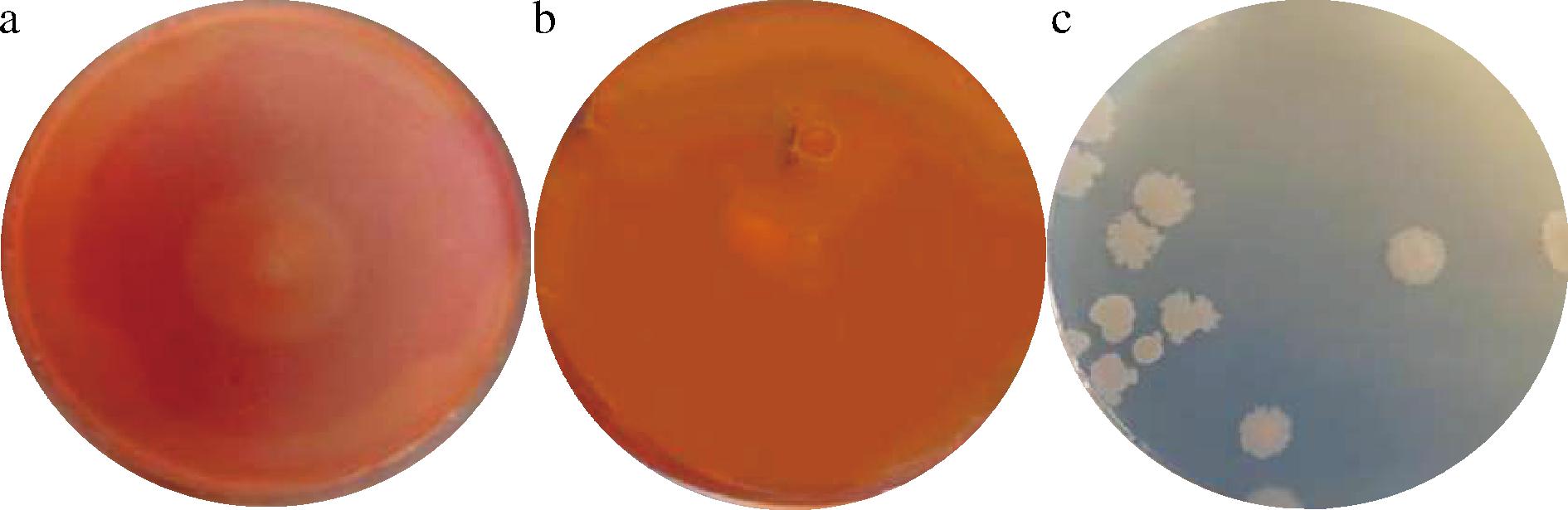
a-CMC-Na培养基;b-木聚糖培养基;c-单菌落形态
图1 粘质沙雷氏菌EC-011不同培养基生长情况
Fig.1 Growth of Serratia marcescens EC-011 on different culture media
2.2 菌株鉴定结果
如图1-c所示,在木聚糖平板上生长的菌落为白色,表面光滑,中心隆起,边缘整齐,易于拾取,生理生化特征测定结果如表1所示。如图2所示,成功扩增的菌落PCR产物全长约1 500 bp。
表1 粘质沙雷氏菌EC-011生理生化特征
Table 1 Physiological and Biochemical Characteristics of S.marcescensEC-011
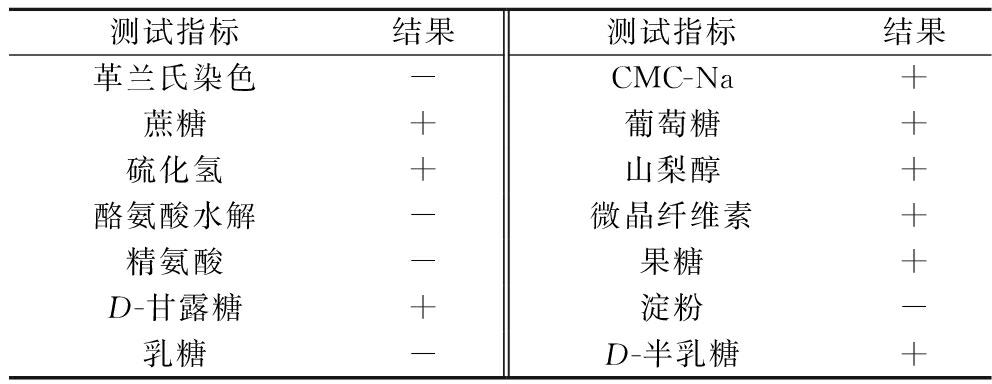
测试指标结果测试指标结果革兰氏染色-CMC-Na+蔗糖+葡萄糖+硫化氢+山梨醇+酪氨酸水解-微晶纤维素+精氨酸-果糖+D-甘露糖+淀粉-乳糖-D-半乳糖+
注:“+”表示阳性反应,“-”表示阴性反应。
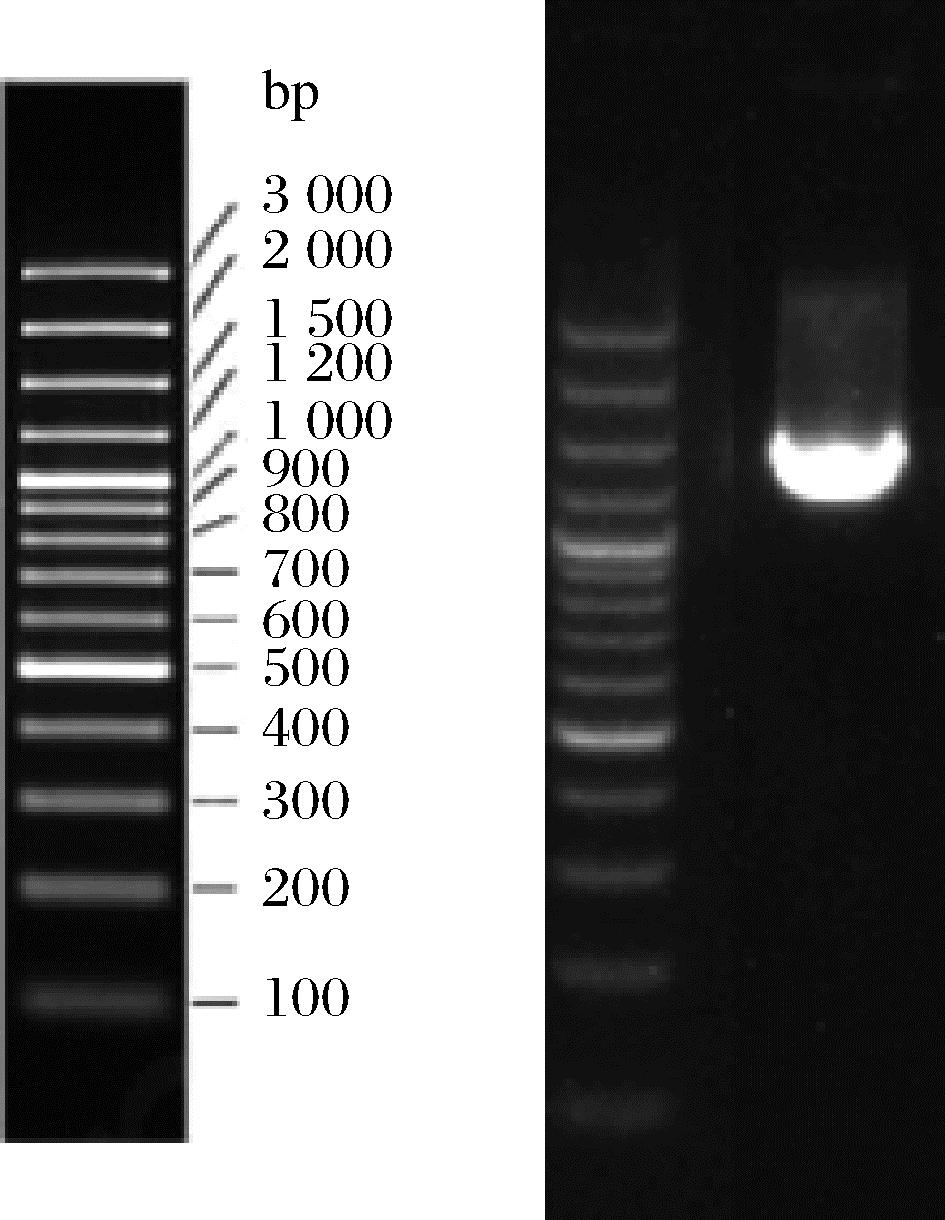
图2 PCR产物琼脂糖凝胶电泳结果图
Fig.2 PCR product agarose gel electrophoresis results
16S rDNA测序分析结果如图3所示,测序结果在NCBI网站上进行blast对比,运用MEGA11.0构建的系统发育树,与同一进化分支上的Serratia marcescens ATCC 13880的相似性达97%,确定并命名为粘质沙雷氏菌EC-011。并送至中国普通微生物菌种保藏管理中心保藏,保藏号为CGMCCC No.28580。
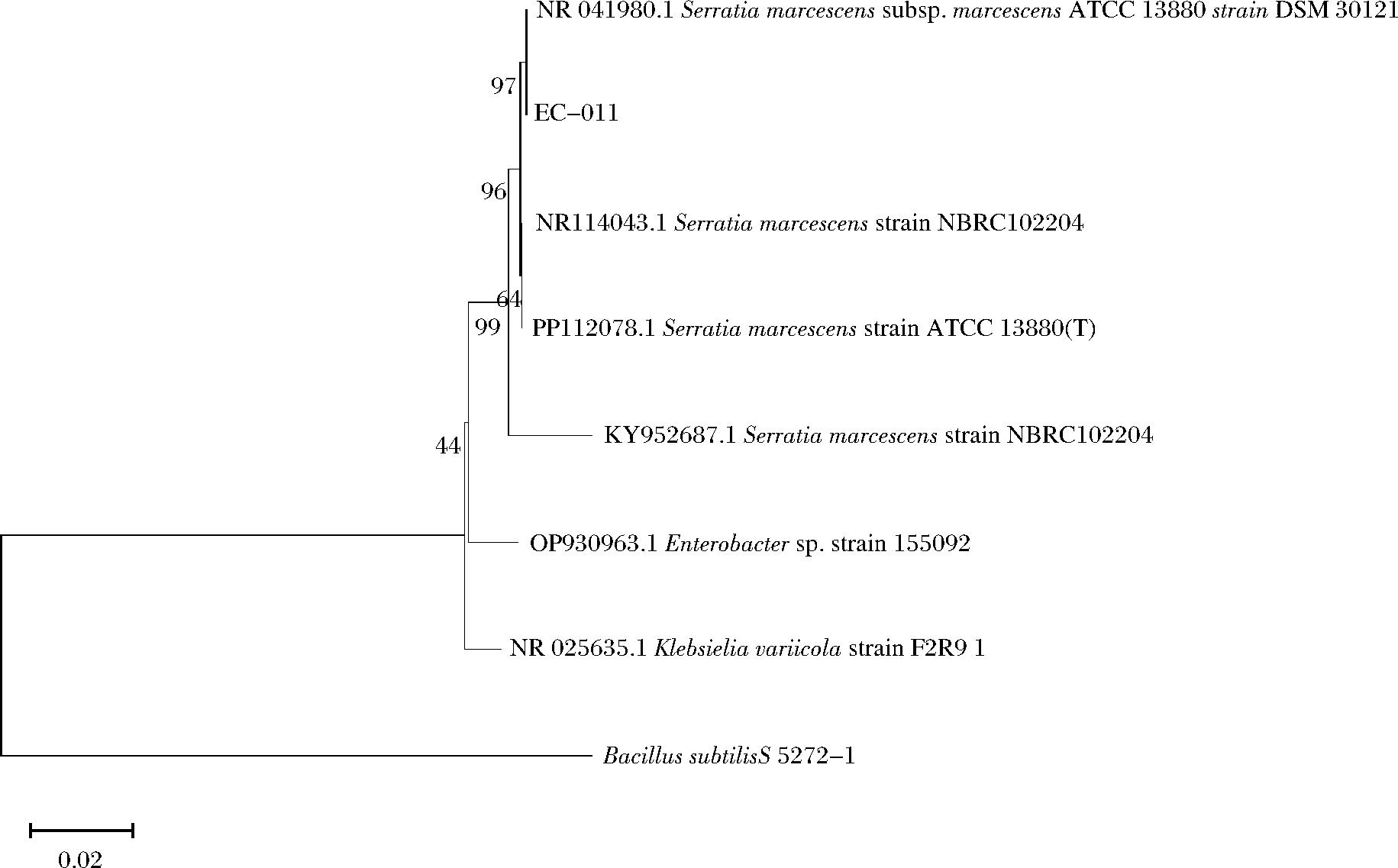
图3 粘质沙雷氏菌EC-011 16S r-DNA序列构建的系统发育树
Fig.3 Phylogenetic tree constructed from S. marcescens EC-011 16S r-DNA sequence
2.3 基因组组成与信息
粘质沙雷氏菌EC-011的基因组圈图、基因长度图分别如图4、图5所示。由图4可知,粘质沙雷氏菌EC-011的基因组为一条环状闭合DNA,基因组大小4 158 647 bp,共预测到蛋白质编码基因4 075个,tRNA、rRNA、sRNA基因分别为50个、9个、47个,GC含量占比61.87%。由图5可知,基因平均长度为912.21 bp,最长和最短基因长度为90、17 891 bp,长度500~1 000 bp的基因共有1 544个,长度大于1 000 bp的基因共有1 427个;串联重复序列为22个,重复比例为0.23%;散在重复序列为44个,重复比例为0.08%。
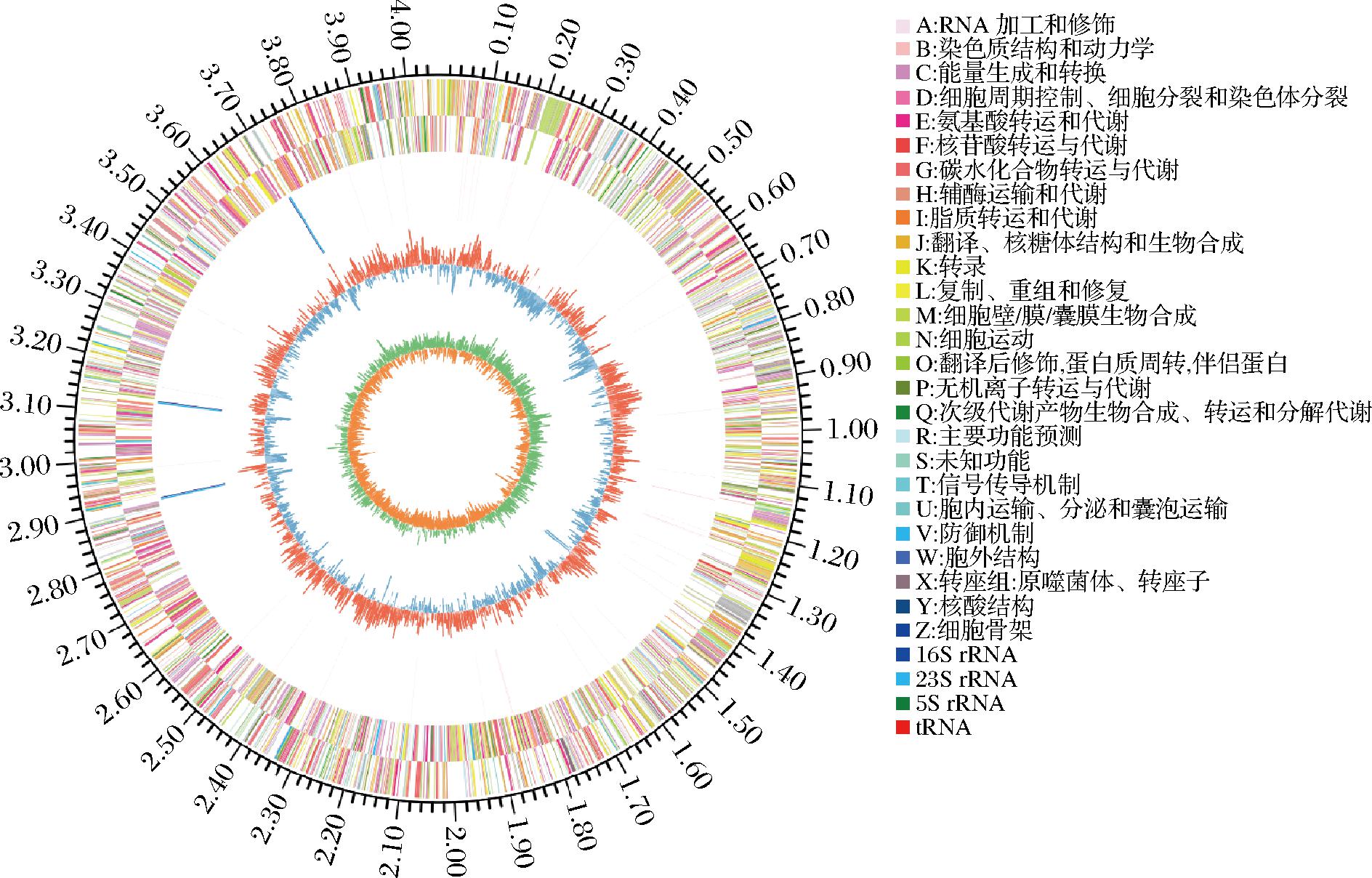
图4 粘质沙雷氏菌EC-011基因组圈图
Fig.4 Genome circle mapping of S. marcescens EC-011
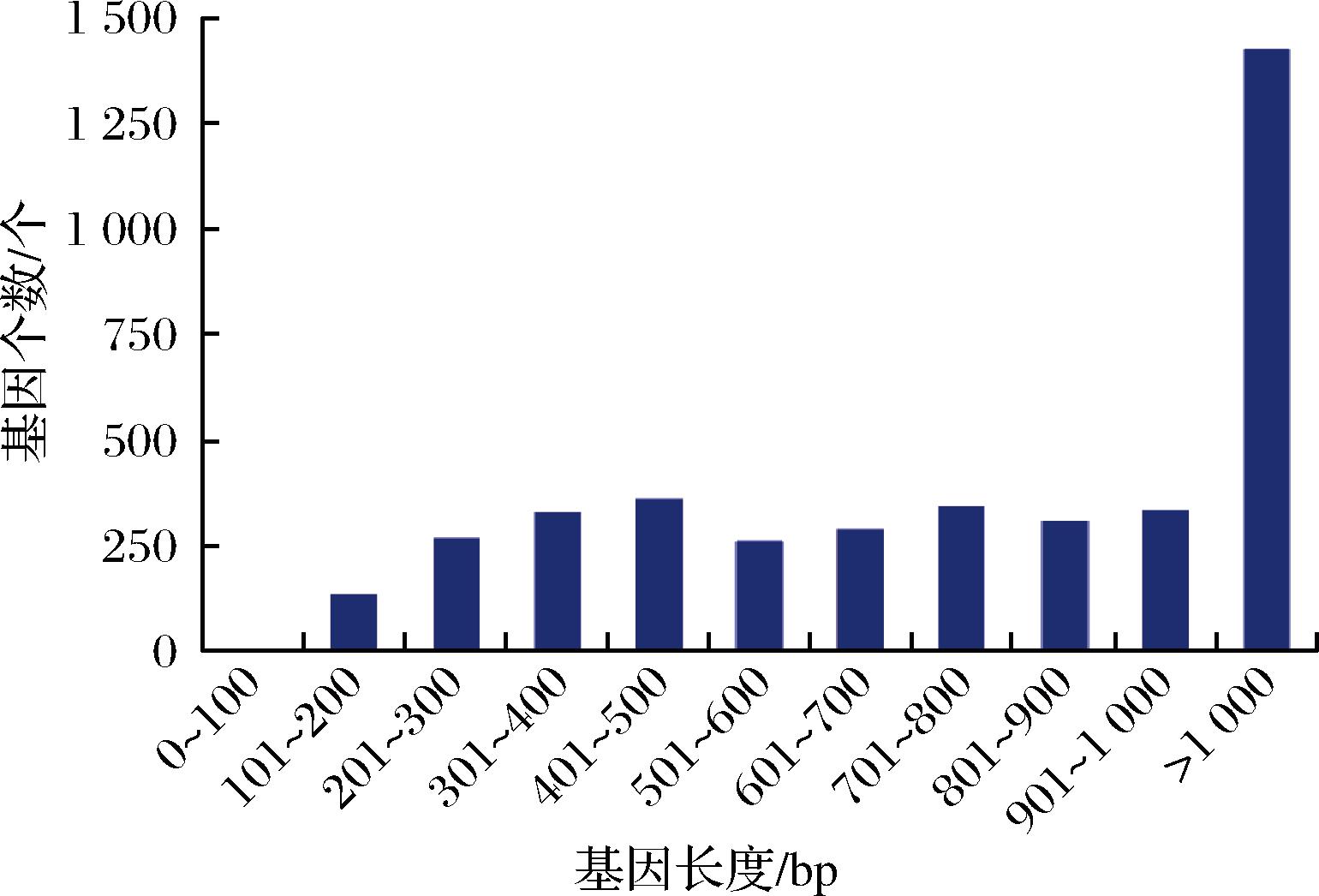
图5 粘质沙雷氏菌EC-011基因长度图
Fig.5 Length of S. marcescens EC-011 gene
2.4 基因功能注释
2.4.1 COG数据库注释
粘质沙雷氏菌EC-011共有3 398个蛋白质编码基因在COG数据库中得到注释,占总编码蛋白基因的83.39%,结果如图6所示。

图6 粘质沙雷氏菌EC-011 COG 注释分类
Fig.6 Classification of S. marcescens EC-011 COG annotations
由图6可知,注释结果共有22类,分别有224、43、392、98、311、185、187、215、258、132、224、51、193、227、72、302、211、226、41、85、16、45个基因注释分类到C-X。其中氨基酸转运与代谢(E:amino acid transport and metabolism)共392个(11.48%),占比最高。注释基因数量较多的功能分类为:转录(K:transcription)258个(7.59%)、无机离子转运与代谢(P:inorganic ion transport and metabolism)227个(6.68%)、信号转导机制(T:signal transduction mechanisms)226个(6.48%),能量生成和转化(C:energy production and conversion)和细胞壁/细胞膜/细胞被膜(M:cell wall/membrane/envelope biogenesis)均为224个(6.59%)。
其中碳水化合物运输与代谢(G:carbohydrate transport and metabolism)共注释311个(9.15%),其中鉴定出参与碳水化合物细胞转运的COG2814(主要促进子)20个基因,COG0726(肽聚糖/二聚糖/几丁质脱乙酰酶、PgdA/NodB/CDA1家族)3个基因、COG1349(糖代谢的DNA结合转录调节因子、DeoR/GlpR家族)2个基因和COG0697(药物/代谢物转运蛋白家族的渗透酶)17个基因[17]。
2.4.2 GO数据库注释
粘质沙雷氏菌EC-011共有2 573个蛋白质编码基因在GO数据库中得到注释,占总编码蛋白基因的63.14%,结果如图7所示。

图7 粘质沙雷氏菌EC-011 GO 注释分类
Fig.7 Classification of S. marcescens EC-011 GO annotations
由图7可知,有592个功能基因注释到生物过程(biological process),其中跨膜转运(transmembrane transport)89个(3.46%)和DNA模板化(regulation of transcription DNA-templated)78个(3.03%)占比最多;有1 600个功能基因注释到细胞组分(cellular component),其中膜整体组件(integral component of membrane)688个(26.74%)和等离子体膜(plasma membrane)372个(14.46%)占比最多;有1 471个功能基因注释到分子功能(molecular function),ATP结合(ATP binding)289个(11.23%)和金属离子结合(metal ion binding)214个(8.32%)占比最多。
2.4.3 KEGG数据库注释
粘质沙雷氏菌EC-011共有2 279个蛋白质编码基因在KEGG数据库中得到注释,占总编码蛋白基因的55.93%,结果如图8所示。
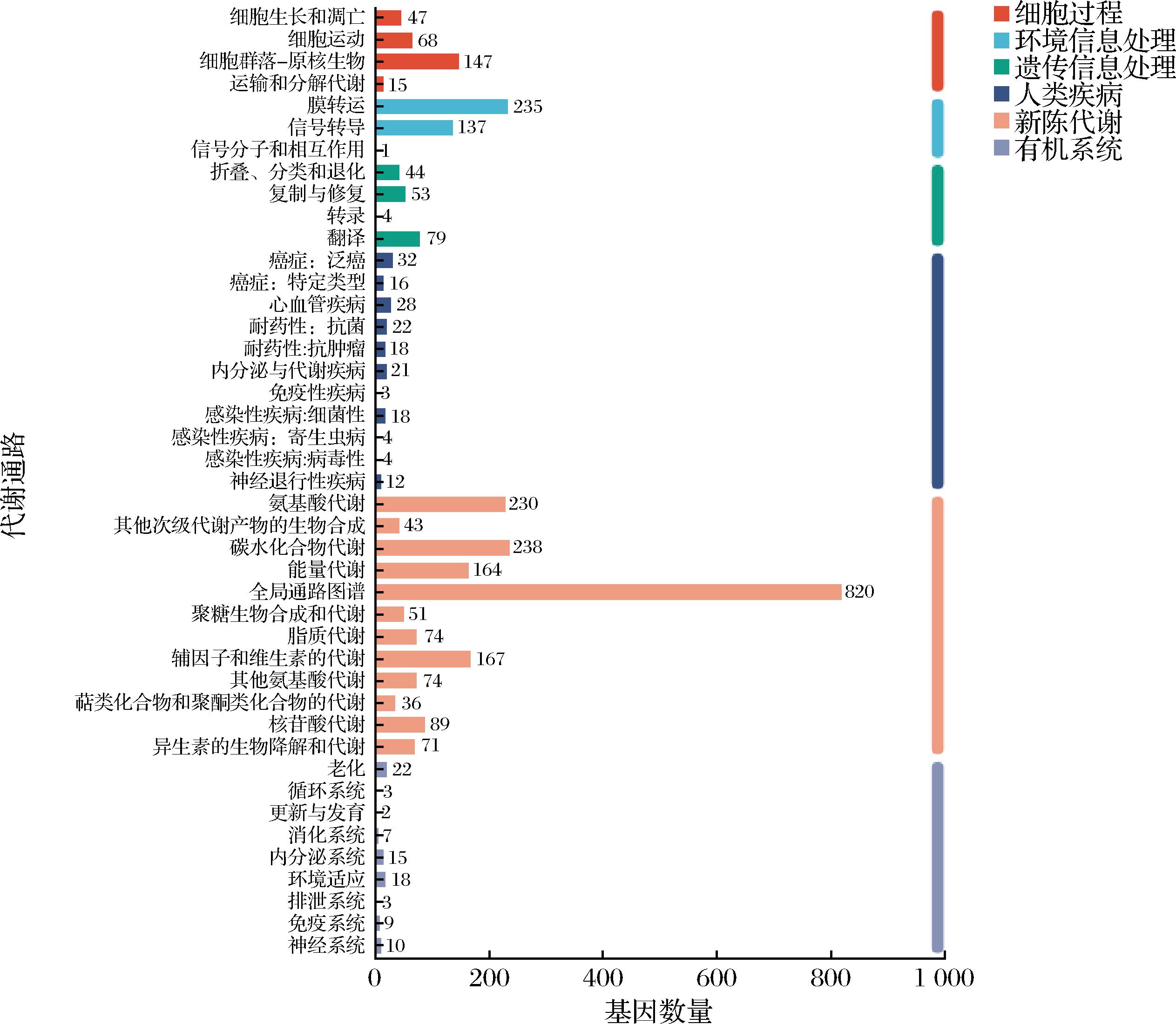
图8 粘质沙雷氏菌EC-011 KEGG 注释分类
Fig.8 Classification of S. marcescens EC-011 KEGG annotations
共有2 279个基因在KEGG Pathway中富集到六大功能中,其中细胞过程(cellular processes)275个,环境信息处理(environmental information processing)373个,遗传信息处理(genetic information processing)180个,人类疾病(human diseases)178个、新陈代谢(metabolism)2 057个,有机系统(organismal systems)89个,共计43条代谢通路。
其中参与碳水化合物代谢(carbohydrate metabolism)的相关基因共有238个,氨基糖和核苷酸糖代谢(ko00520,35个)、丙酮酸代谢(ko00620,45个)、糖酵解/糖异生(ko00010,32个)、淀粉和蔗糖代谢(ko00500,25个)和磷酸戊糖途径(ko00030,26个)是木质纤维素降解的主要能量代谢途径[18]。上述结果表明,粘质沙雷氏菌EC-011基因组包含许多代谢碳水化合物和其他营养物质所必需的基因,具有显著的碳水化合物降解能力。
2.4.4 CAZy数据库注释
碳水化合物活性酶(CAZymes)分为六大类,包括糖基转移酶(glycosyl transferase,GTs)、糖苷水解酶(glucoside hydrolase,GHs)、多糖裂合酶(polysaccharide lyase,PLs)、碳水化合物酯酶(carbohydrate esterase,CEs)、碳水化合物结合模块(carbohydrate binding module,CBMs)和辅助氧化还原酶(auxiliary activities,AAs),可以降解木质纤维素。其中,GHs、CEs、PLs和CBMs主要与纤维素和半纤维素降解有关,而AAs则与木质素降解有关。对粘质沙雷氏菌EC-011木质纤维素降解的碳水化合物酶基因进行注释,使用dbCAN2数据库鉴定CAZymes[18]。结果由图9所示,CAZyme家族共注释了117个基因,其中AAs19个、CEs 20个、GHs 44个、GTs 34个。
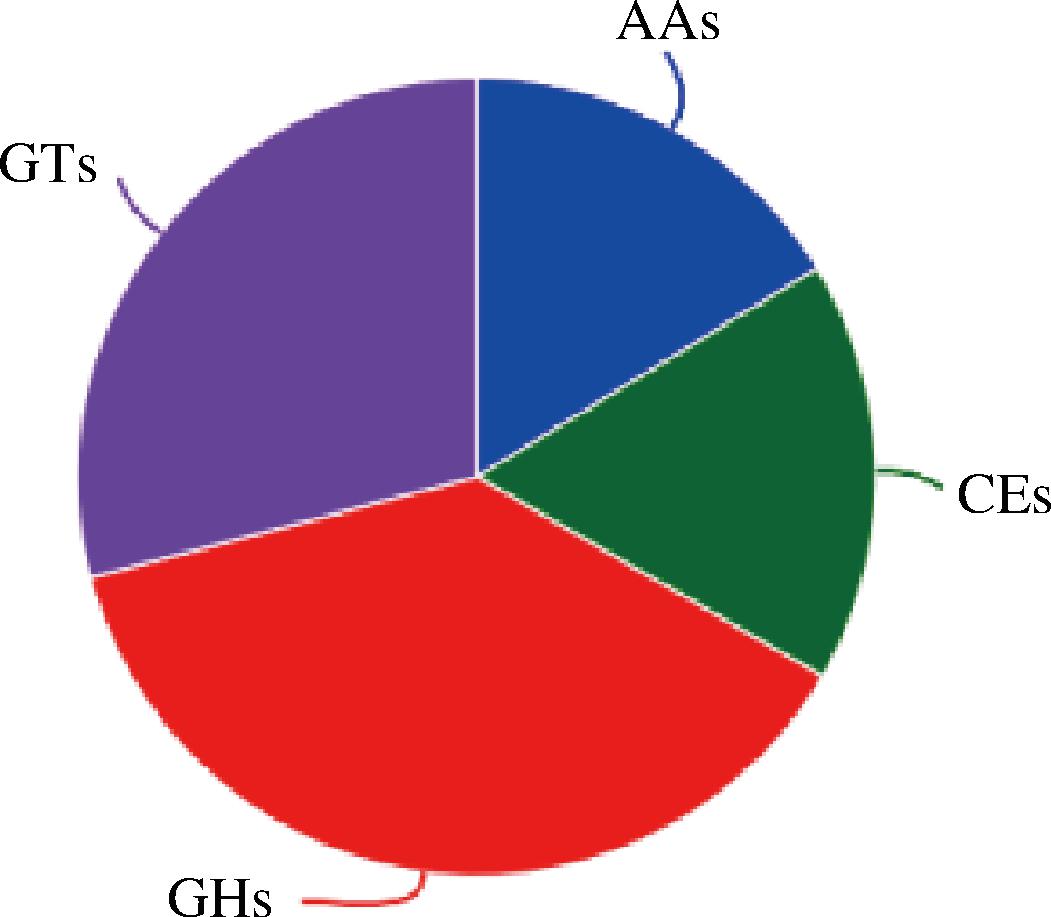
图9 粘质沙雷氏菌EC-011 CAZy注释分类
Fig.9 Classification of S. marcescens EC-011 CAZy annotations
由表2可知,编码假定纤维素酶的基因主要来自GH1、GH3、GH5、GH16、GH51和AA3家族,分别包括1个、1个、2个、1个、1个和5个基因。对于纤维素降解,注释到GHs的内切-1,4-β-葡聚糖酶和内切-β-1,3-1,4葡聚糖酶随机切割纤维素多糖链的无定形区域以产生纤维二糖,以及β-葡萄糖苷酶用于纤维二糖或纤维素低聚糖的水解[19]。AAs的纤维二糖脱氢酶在还原端的C-1氧化纤维二糖、纤维糊精或结构相关的寡糖如乳糖,纤维二糖被氧化为纤维蛋白内酯[20]。
表2 粘质沙雷氏菌EC-011 CAZymes注释基因
Table 2 Annotated genes encoding CAZymes of S.marcescensEC-011
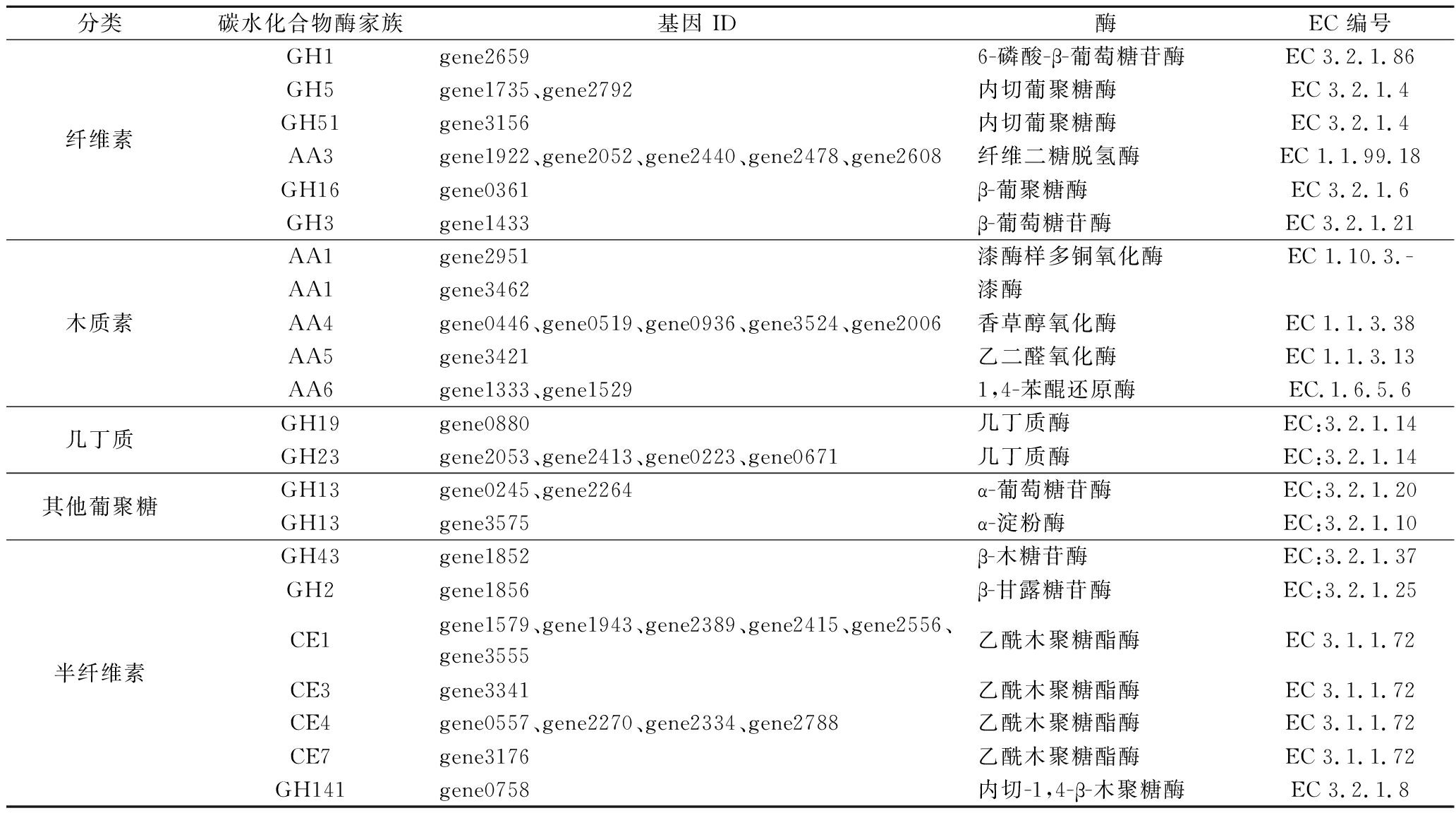
分类碳水化合物酶家族基因 ID酶EC 编号纤维素GH1gene26596-磷酸-β-葡萄糖苷酶EC 3.2.1.86GH5gene1735、gene2792内切葡聚糖酶EC 3.2.1.4GH51gene3156内切葡聚糖酶EC 3.2.1.4AA3gene1922、gene2052、gene2440、gene2478、gene2608纤维二糖脱氢酶EC 1.1.99.18GH16gene0361β-葡聚糖酶EC 3.2.1.6GH3gene1433β-葡萄糖苷酶EC 3.2.1.21木质素AA1gene2951漆酶样多铜氧化酶EC 1.10.3.-AA1gene3462漆酶AA4gene0446、gene0519、gene0936、gene3524、gene2006香草醇氧化酶EC 1.1.3.38AA5gene3421乙二醛氧化酶EC 1.1.3.13AA6gene1333、gene15291,4-苯醌还原酶EC.1.6.5.6几丁质GH19gene0880几丁质酶EC:3.2.1.14GH23gene2053、gene2413、gene0223、gene0671几丁质酶EC:3.2.1.14其他葡聚糖GH13gene0245、gene2264α-葡萄糖苷酶EC:3.2.1.20GH13gene3575α-淀粉酶EC:3.2.1.10半纤维素GH43gene1852β-木糖苷酶EC:3.2.1.37GH2gene1856β-甘露糖苷酶EC:3.2.1.25CE1gene1579、gene1943、gene2389、gene2415、gene2556、gene3555乙酰木聚糖酯酶EC 3.1.1.72CE3gene3341乙酰木聚糖酯酶EC 3.1.1.72CE4gene0557、gene2270、gene2334、gene2788乙酰木聚糖酯酶EC 3.1.1.72CE7gene3176乙酰木聚糖酯酶EC 3.1.1.72GH141gene0758内切-1,4-β-木聚糖酶EC 3.2.1.8
表3 粘质沙雷氏菌EC-011 antiSMASH注释结果
Table 3 Results of S.marcescensEC-011 antiSMASH annotation

分类N-乙酰谷氨酰胺酰谷氨酰胺萜类芳基多烯氧化还原辅因子核糖体合成肽类似物基因数目1344412415占比9.49%32.11%29.93%17.52%10.95%
由表2可知,编码假定半纤维素酶的基因主要来自GH43、GH2、CE3、CE1、CE4、CE7和GH141家族,分别包括1个、1个、1个、6个、1个、4个和1个基因。对于半纤维素降解,GHs的内切-1,4-β-木聚糖酶和1,4-β-木糖苷酶水解木聚糖的主链,在木聚糖裂解后产生短的低聚木糖[21]。β-木糖苷酶和β-甘露糖苷酶的功能是从木二糖和低聚木糖中释放木糖单元以及甘露聚糖多糖末端甘露糖水解。CEs具有从木聚糖去除乙酰或降解低聚木糖的功能,CE3可以增强木聚糖的溶解[22]。CE1、CE4和CE7含有高度特异性的乙酰基木聚糖酯酶,参与木聚糖降解[23],但不能降解乙酰半乳糖葡甘露聚糖或乙酰化甘露聚糖化合物[24]。
在木质素降解方面,编码假定木质素降解相关酶的基因主要来自AA1、AA4、AA5和AA6家族,分别包括2个、5个、1个和2个基因。香草醇氧化酶(vanillyl alcohol oxidases,VAO)属于AA4家族,可以转化侧链位于芳香环相反位置的各种酚类化合物[25]。AA5(乙二醛氧化酶/醇氧化酶)和AA6(1,4-苯醌还原酶)家族的酶也与源自木质素的酚类化合物的氧化或还原有关[26]。AAOs(芳醇氧化酶)通常催化一系列不同芳香族和脂肪族不饱和醇的伯醇氧化为相应的醛,其中许多不饱和醇由菌种分解木质纤维素时形成[20]。
进一步分析了参与其他类型葡聚糖代谢的CAZymes基因,包括几丁质和淀粉。鉴定出了一些几丁质酶家族(GH19和GH23分别1个和4个),鉴定出GH13家族(3个)的α-淀粉酶和α-葡萄糖苷酶参与淀粉水解[27]。综上所述,粘质沙雷氏菌EC-011基因组中的CAZymes编码基因参与各种类型的碳水化合物代谢,包括纤维素、半纤维素等木质纤维素降解、几丁质降解以及淀粉代谢。
2.4.5 抗生素和次级代谢产物(antibiotics &secondary metabolite analysis shell,antiSMASH)分析
antiSMASH分析则表明,具有抗氧化特性的分子基因41个,表现为在恶劣环境中的抗逆性[28]。核糖体合成肽类似物基因15个,可能在蛋白质合成中发挥作用,对木质纤维素降解酶的生成有重要作用。N-乙酰谷氨酰胺谷氨酰胺基因13个,可能与细胞壁合成有关。与萜类化合物基因44个,在植物与微生物间的相互作用中可能起到关键作用。氧化还原辅因子基因24个,在维持细胞内氧化还原稳态方面起着关键作用,对细胞代谢、生长性能和生物合成能力有重要影响。这些信息对于进一步探索粘质沙雷氏菌EC-011如何适应环境以及降解木质纤维素至关重要。
2.4.6 分泌系统分析
由图10可知,粘质沙雷氏菌EC-011具有多个分泌系统,包括Tat(双精氨酸易位)、Sec-SRP途径、Type Ⅰ、Type Ⅱ、Type Ⅲ、Type Ⅳ、Type Va、Vb、Vc和Type VI。Sec(一般分泌)途径是常见机制,关键组分有SecA、SecY、SecE、SecDF、YajC、YidC,粘质沙雷氏菌EC-011通过该机制可以将蛋白酶、水解酶等多种蛋白质分泌到细胞质外环境中[29]。SecA是一种膜结合蛋白,SecDF、YajC和YidC作为辅助蛋白在分泌过程中起到一定的调控作用[30]。SRP(信号识别颗粒)途径是介导内膜蛋白靶向质膜的一种广泛机制。Tat(双精氨酸转运酶)途径是一类同Sec途径平行运转的跨内膜途径,主要区别在于:Sec分泌途径主要分泌处于未折叠状态的蛋白都,而Tat途径分泌的是底物蛋白,具有折叠构象,主要由TatA、TatB和TatC三种内膜蛋白构成,因此通过Tat途径分泌的蛋白较少。Type Ⅰ能够直接将蛋白质从细胞质转移到外部环境,将蛋白质迅速释放到周围环境中,起到快速响应的作用。Type Ⅱ属于两步法,即底物蛋白先从胞质跨内膜分泌到周质空间再穿过外膜分泌到胞外,由该系统分泌的蛋白多为水解酶蛋白,如纤维素酶、果胶酶等。Type Ⅲ主要负责转运毒力因子到宿主细胞中,在某些情况下,也可能用于分泌降解酶。Type Ⅳ是一种多功能运输系统,类似于Type Ⅲ,可以用于分泌大型的多功能酶复合体。Type Va、Vb、Vc通过不同的通道和机制将蛋白质分泌到外部环境。Type Ⅵ虽然主要与抗微生物活性有关,主要负责将效应蛋白转移到靶细胞中,但它同样能将具有降解活性的大分子蛋白质运输到外部环境[31]。这些分泌系统的存在表明,粘质沙雷氏菌EC-011能够将内部产生的降解酶等蛋白质有效地分泌到胞外,能够应对不同的环境压力和生存需求,为菌体降解木质纤维素提供了不同途径。

图10 粘质沙雷氏菌EC-011分泌系统
Fig.10 S. marcescens EC-011 secretion system diagram
2.5 产酶条件优化
通过单因素试验可知(图11),滤纸酶活性的产生受时间的影响相对较小,随着温度、pH值和接种量的升高而增加,达到峰值后,酶活性随着温度、pH值和接种量的升高而减少。当温度维持在35 ℃时,滤纸酶活性达(0.83±0.01) U/mL,当接种量在3%时,滤纸酶活性达(0.84±0.01) U/mL,当pH值在6.0时,滤纸酶活性达(0.90±0.02) U/mL,当培养时间在72 h时,滤纸酶活达(0.92±0.03) U/mL。
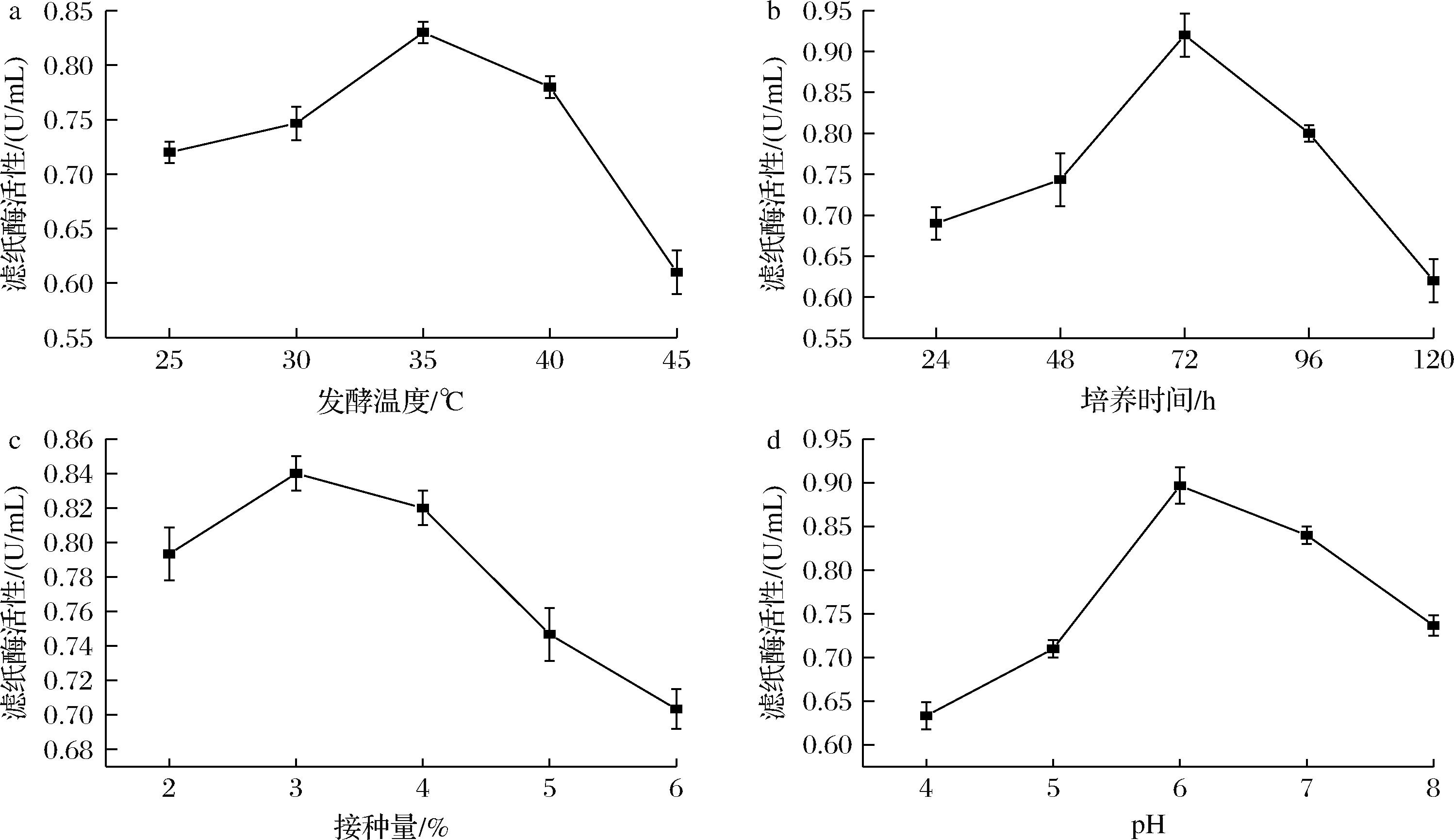
a-发酵温度;b-培养时间;c-接种量;d-pH
图11 各因素对滤纸酶活性的影响
Fig.11 The influence of various factors on the enzyme activity of filter paper
在单因素试验的结果上进行Box-Behnken试验,响应面试验设计及结果如表4所示。以Y(滤纸酶活性)为因变量,A(发酵温度)、B(培养时间)、C(pH)、D(接种量)为自变量进行回归拟合,得到方程:
Y=2.2+0.128 3A-0.020 0B-0.056 7C-0.036 7D-0.055 0AB+0.015 0AC-0.080 0AD+0.060 0BC+0.020 0BD+0.005 0CD-0.227 8A2-0.342 8B2-0.382 8C2-0.177 8D2
表4 响应面试验设计及结果
Table 4 Response surface test design and results
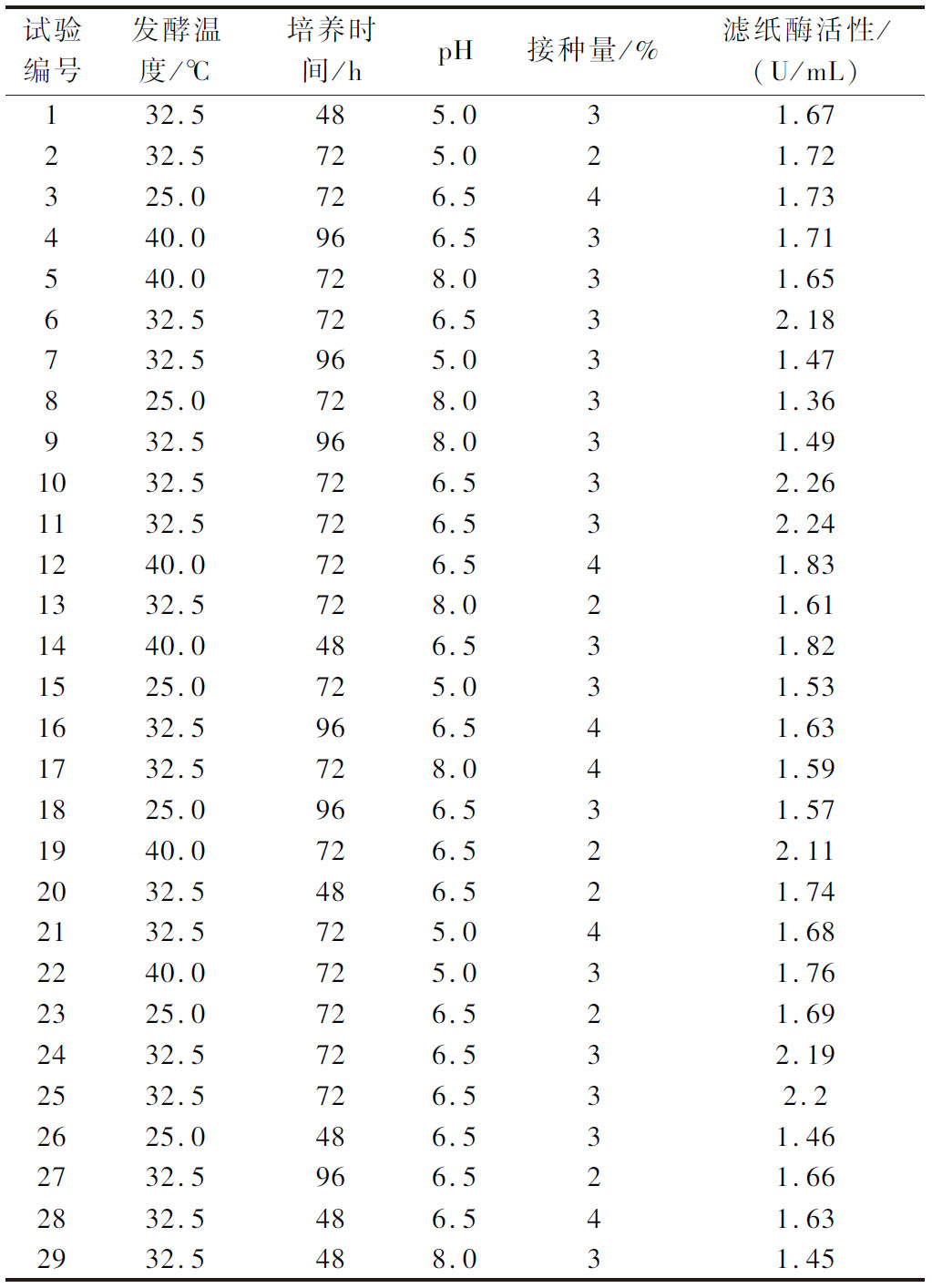
试验编号发酵温度/℃培养时间/hpH接种量/%滤纸酶活性/(U/mL)132.5485.031.67232.5725.021.72325.0726.541.73440.0966.531.71540.0728.031.65632.5726.532.18732.5965.031.47825.0728.031.36932.5968.031.491032.5726.532.261132.5726.532.241240.0726.541.831332.5728.021.611440.0486.531.821525.0725.031.531632.5966.541.631732.5728.041.591825.0966.531.571940.0726.522.112032.5486.521.742132.5725.041.682240.0725.031.762325.0726.521.692432.5726.532.192532.5726.532.22625.0486.531.462732.5966.521.662832.5486.541.632932.5488.031.45
由表5可知,该模型表现为极显著(P<0.000 1),失拟项不显著(P=0.272 9>0.05),R2=0.985 3,R2Adj=0.970 7,表明拟合度较好,可用于本试验预测。回归方程分析表明:一次项A、C、D,交互系数AB、AD、BC,二次项系数A2、B2、C2、D2影响显著或极显著。F值反映了各因素对于响应值的重要性,A>C>D>B,对滤纸酶活性大小的影响为:发酵温度>pH>接种量>培养时间。
表5 滤纸酶活性回归方差分析结果
Table 5 Results of variance analysis of filter paper activity regression
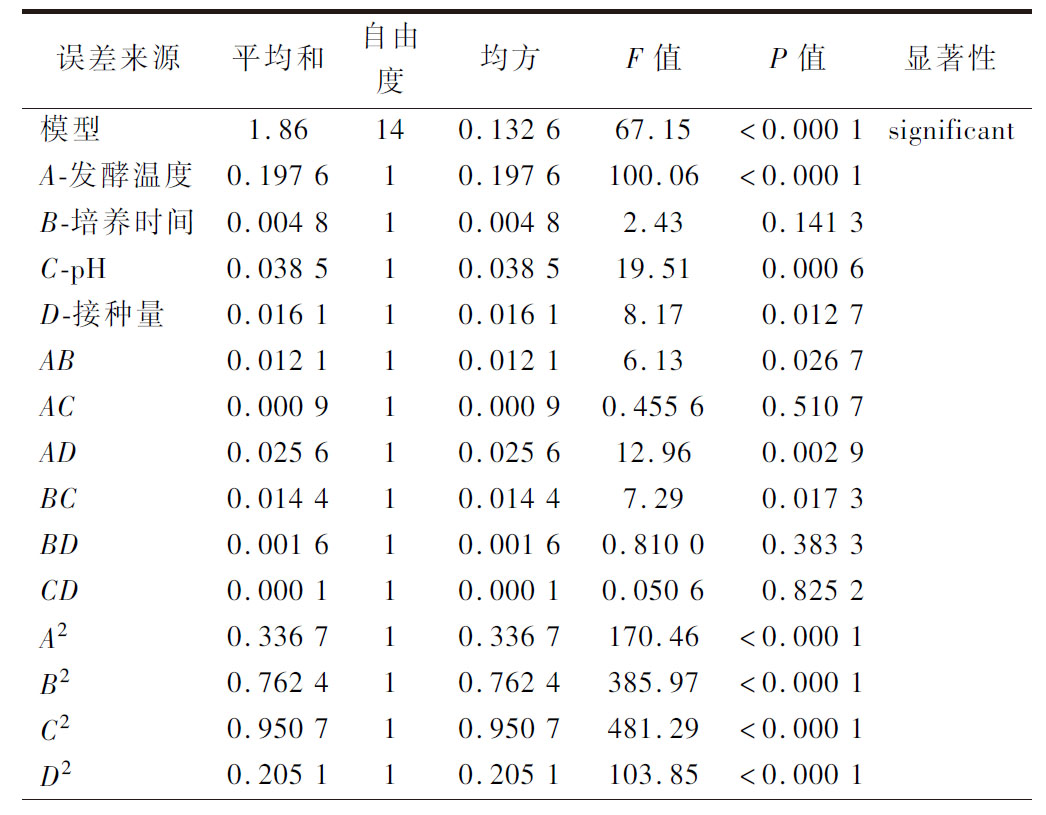
误差来源平均和自由度均方F值P值显著性模型1.86140.132 667.15<0.000 1significantA-发酵温度0.197 610.197 6100.06<0.000 1B-培养时间0.004 810.004 82.430.141 3C-pH0.038 510.038 519.510.000 6D-接种量0.016 110.016 18.170.012 7AB0.012 110.012 16.130.026 7AC0.000 910.000 90.455 60.510 7AD0.025 610.025 612.960.002 9BC0.014 410.014 47.290.017 3BD0.001 610.001 60.810 00.383 3CD0.000 110.000 10.050 60.825 2A20.336 710.336 7170.46<0.000 1B20.762 410.762 4385.97<0.000 1C20.950 710.950 7481.29<0.000 1D20.205 110.205 1103.85<0.000 1
续表5
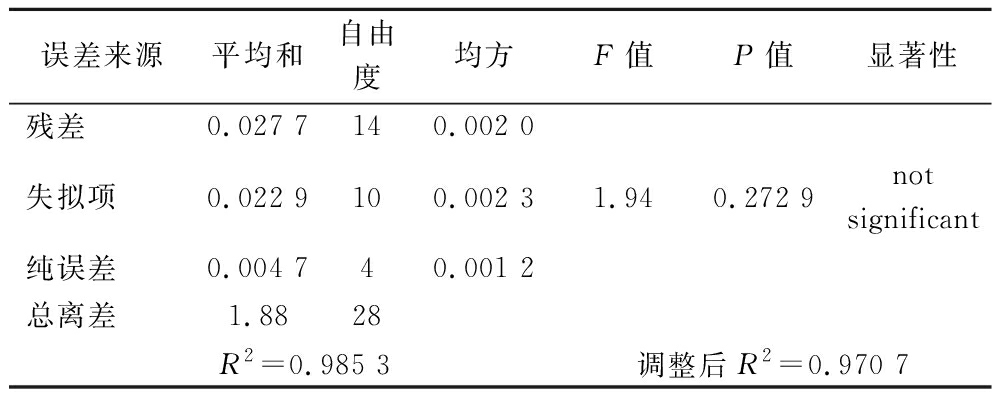
误差来源平均和自由度均方F值P值显著性残差0.027 7140.002 0失拟项0.022 9100.002 31.940.272 9not significant纯误差0.004 740.001 2总离差1.8828R2=0.985 3调整后R2=0.970 7
因素间交互效应的强弱可由等高线图的形状来分析。等高线图越近于椭圆,表明因素之间的交互作用越强烈。由图12-a、图12-d可知,发酵温度和培养时间的交互影响明显,随着发酵温度的增加,酶活性逐渐增大,发酵温度超过35 ℃,滤纸酶活性逐渐降低,培养时间也同单因素试验的结果相符。由图12-b、图12-e可知,发酵温度和接种量交互作用显著,滤纸酶活性在发酵温度34~37 ℃、接种量2%~3%范围内有最大值。由图12-c、图12-f可知,两者交互作用显著,随着培养时间、pH的增加,滤纸酶活性在培养时间72 h和pH 6.5呈现极大值点。

a、d-发酵温度和培养时间;b、e-发酵温度和接种量;c、f-培养时间和pH
图12 各因素交互作用对滤纸酶活性影响的等高线图(上)和响应面图(下)
Fig.12 Contour plot (top) and response surface plot (bottom) of the interaction effects of various factors on filter paper activity
根据响应面优化结果和回归方程,利用Design-expert13.0得到最佳参数为:发酵温度34.9 ℃,初始pH值为6.38,接种量2.24%,培养时间70.4 h,此条件下,优化后其滤纸酶活性达到2.24 U/mL。为了验证模型的有效性,在实际操作下结合方差分析结果,确定最佳培养条件为:培养时间70 h、发酵温度35 ℃,接种量2%,pH 6.5,进行3次验证试验后,此条件下测得2.19 U/mL,实际测量值接近预测值,证明了该条件的可行性,并确定为菌种的最佳产酶条件。
粘质沙雷氏菌EC-011基因组中含有大量的CAZymes编码基因,最佳产酶条件下能显著诱导木质纤维素降解相关基因表达,使得菌株能分泌纤维素酶、木聚糖酶、漆酶等降解酶,滤纸酶活在优化后能显著提高。但具体的基因表达情况需要后续的转录组学研究,对粘质沙雷氏菌EC-011的全基因分析,可以为后续的木质纤维素降解相关基因的克隆,构建工程菌株提高木质纤维素降解酶活性提供理论依据[32]。
3 结论
本研究筛选的粘质沙雷氏菌EC-011能分泌一些包括纤维素酶、木聚糖酶等酶类,具有高效的木质纤维素降解能力。全基因组测序分析表明,其含有大量碳水化合物活性酶的功能基因,antiSMASH和分泌系统分析表明该菌具有多样化的分泌机制,这些基因和机制对木质纤维素降解至关重要。经单因素及响应面优化得到最佳产酶条件为培养时间70 h,发酵温度35 ℃,接种量2%,pH 6.5,优化后滤纸酶活性达到2.19 U/mL。本研究将有利于木质纤维素降解微生物资源的挖掘和粘质沙雷氏菌EC-011的开发利用。
[1] YANG W, LI X H, ZHANG Y T.Research progress and the development trend of the utilization of crop straw biomass resources in China[J].Frontiers in Chemistry, 2022, 10:904660.
[2] MARRIOTT P E, G MEZ L D, MCQUEEN-MASON S J.Unlocking the potential of lignocellulosic biomass through plant science[J].New Phytologist, 2016, 209(4):1366-1381.
MEZ L D, MCQUEEN-MASON S J.Unlocking the potential of lignocellulosic biomass through plant science[J].New Phytologist, 2016, 209(4):1366-1381.
[3] SHARMA A, TEWARI R, RANA S S, et al.Cellulases:Classification, methods of determination and industrial applications[J].Applied Biochemistry and Biotechnology, 2016, 179(8):1346-1380.
[4] WANG X G, TIAN L, LI Y X, et al.Effects of exogenous cellulose-degrading bacteria on humus formation and bacterial community stability during composting[J].Bioresource Technology, 2022, 359:127458.
[5] KWAK J, LEE K, SHIN D H, et al.Biochemical and genetic characterization of arazyme, an extracellular metalloprotease produced from Serratia proteamaculans HY-3[J].Journal of Microbiology and Biotechnology, 2007, 17(5):761-768.
[6] 傅慧静, 胡霞, 吴松青, 等.松褐天牛幼虫肠道黏质沙雷氏菌培养条件与木质素降解功能[J].林业科学, 2020, 56(2):106-115.FU H J, HU X, WU S Q, et al.Culture conditions and lignin-degrading function of Serratia marcescens living in the larval gut of Monochamus alternatus[J].Scientia Silvae Sinicae, 2020, 56(2):106-115.
[7] NARENDRA KUMAR H K, CHANDRA MOHANA N, RAKSHITH D, et al.Multicomponent assessment and optimization of the cellulase activity by Serratia marcescens inhabiting decomposed leaf litter soil[J].Sustainable Chemistry and Pharmacy, 2023, 31:100951.
[8] WANG Z Y, WANG R X, ZHOU J S, et al.An assessment of the genomics, comparative genomics and cellulose degradation potential of Mucilaginibacter polytrichastri strain RG4-7[J].Bioresource Technology, 2020, 297:122389.
[9] 东秀珠, 蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社, 2001.DONG X Z, CAI M Y.Manual for Identification of Common Bacterial Systems[M].Beijing:Science Press, 2001.
[10] BUCHANAN R E.伯杰细菌鉴定手册[M].北京:科学出版社, 1984.BUCHANAN R E.Berger Bacterial Identification Manual[M].Beijing:Science Press, 1994.
[11] 孔晓双, 黄新新, 董应宏, 等.一株产β-葡萄糖苷酶甘草内生菌的筛选、全基因组分析及产酶优化[J].微生物学通报, 2024, 51(1):279-294.KONG X S, HUANG X X, DONG Y H, et al.Screening, whole genome analysis, and fermentation condition optimization of a β-glucosidase-producing endophyte from Glycyrrhiza uralensis Fisch[J].Microbiology China, 2024, 51(1):279-294.
[12] WICK R R, JUDD L M, GORRIE C L, et al.Unicycler:Resolving bacterial genome assemblies from short and long sequencing reads[J].PLoS Computational Biology, 2017, 13(6):e1005595.
[13] 纪帅奇, 乌日娜, 张淘崴, 等.枯草芽孢杆菌SNBS-3全基因组测序及其抑菌物质预测分析[J].食品科学, 2024, 45(2):57-63.JI S Q, WU R N, ZHANG T W, et al.Whole genome sequencing of Bacillus subtilis SNBS-3 and prediction of its antimicrobial substances[J].Food Science, 2024, 45(2):57-63.
[14] STOTHARD P, WISHART D S.Circular genome visualization and exploration using CGView[J].Bioinformatics, 2005, 21(4):537-539.
[15] The Gene Ontology Consortium.The gene ontology resource:20 years and still GOing strong[J].Nucleic Acids Research, 2019, 47(D1):D330-D338.
[16] LIU B, ZHENG D D, ZHOU S Y, et al.VFDB 2022:A general classification scheme for bacterial virulence factors[J].Nucleic Acids Research, 2022, 50(D1):D912-D917.
[17] ZHANG T J, WEI S L, LIU Y J, et al.Screening and genome-wide analysis of lignocellulose-degrading bacteria from humic soil[J].Frontiers in Microbiology, 2023, 14:1167293.
[18] TANG H, LI Y Q, ZHENG L, et al.Efficient saccharification of bamboo biomass by secretome protein of the cellulolytic bacterium Serratia marcescens LY1 based on whole-genome and secretome analysis[J].Renewable Energy, 2022, 193:32-40.
[19] WILSON D B.Microbial diversity of cellulose hydrolysis[J].Current Opinion in Microbiology, 2011, 14(3):259-263.
[20] SÜTZL L, LAURENT C V F P, ABRERA A T, et al.Multiplicity of enzymatic functions in the CAZy AA3 family[J].Applied Microbiology and Biotechnology, 2018, 102(6):2477-2492.
[21] MONICA P, KAPOOR M.Alkali-stable GH11 endo-β-1, 4 xylanase (XynB) from Bacillus subtilis strain CAM 21:Application in hydrolysis of agro-industrial wastes, fruit/vegetable peels and weeds[J].Preparative Biochemistry &Biotechnology, 2021, 51(5):475-487.
[22] MALGAS S, SUSAN VAN DYK J, PLETSCHKE B I.A review of the enzymatic hydrolysis of mannans and synergistic interactions between β-mannanase, β-mannosidase and α-galactosidase[J].World Journal of Microbiology &Biotechnology, 2015, 31(8):1167-1175.
[23] ZHANG J H, SIIKA-AHO M, TENKANEN M, et al.The role of acetyl xylan esterase in the solubilization of xylan and enzymatic hydrolysis of wheat straw and giant reed[J].Biotechnology for Biofuels, 2011, 4(1):60.
[24] BIELY P.Microbial carbohydrate esterases deacetylating plant polysaccharides[J].Biotechnology Advances, 2012, 30(6):1575-1588.
[25] XU C F, SU X, WANG J H, et al.Characteristics and functional bacteria in a microbial consortium for rice straw lignin-degrading[J].Bioresource Technology, 2021, 331:125066.
[26] BRENELLI L B, PERSINOTI G F, CAIRO J P L F, et al.Novel redox-active enzymes for ligninolytic applications revealed from multiomics analyses of Peniophora sp.CBMAI 1063, a laccase hyper-producer strain[J].Scientific Reports, 2019, 9(1):17564.
[27] GRAEBIN N G, DA N SCHÖFFER J, DE ANDRADES D, et al.Immobilization of glycoside hydrolase families GH1, GH13, and GH70:State of the art and perspectives[J].Molecules, 2016, 21(8):1074.
[28] 高帅. 塔里木河淤泥小单孢菌14个种次级代谢产物挖掘[D].阿拉尔:塔里木大学, 2023.GAO S.Mining of secondary metabolites from 14 species of Micromonospora from Tarim river sediment[D].Alaer:Tarim University, 2023.
[29] ZHANG W B, YAN H L, ZHU Z C, et al.Genome-wide identification of the Sec-dependent secretory protease genes in Erwinia amylovora and analysis of their expression during infection of immature pear fruit[J].Journal of Zhejiang University.Science.B, 2020, 21(9):716-726.
[30] JONES A M, VIRTANEN P, HAMMARLÖF D, et al.Genetic evidence for SecY translocon-mediated import of two contact-dependent growth inhibition (CDI) toxins[J].mBio, 2021, 12(1):e03367-20.
[31] 刘伟, 庞建, 刘占英, 等.革兰氏阴性细菌蛋白分泌系统研究进展[J].微生物学通报, 2022, 49(2):781-793.LIU W, PANG J, LIU Z Y, et al.Progress on the protein secretion system of Gram-negative bacteria[J].Microbiology China, 2022, 49(2):781-793.
[32] 盛文建, 白方敏, 戴璐, 等.重组粘质沙雷氏菌混合发酵木糖-葡萄糖生产2, 3-丁二醇[J].基因组学与应用生物学, 2019, 38(12):5475-5482.SHENG W J, BAI F M, DAI L, et al.Production of 2, 3-butanediol from xylose and glucose by recombinant Serratia marcescens MG1[J].Genomics and Applied Biology, 2019, 38(12):5475-5482.