胆汁酸又称胆酸,是动物胆汁的主要组成部分,是由肝脏实质细胞合成的亲水性、疏水性兼备的两极分子。据报道,胆汁酸不仅能够促进动物肠道中的脂类消化代谢,还具备通过受体以感知动物营养水平进而调节免疫功能和糖脂代谢、维持胆固醇与胆汁酸稳态[1]。动物体内常见胆汁酸分为两大类:结合型胆汁酸(conjugated bile acid,CBA)和初级胆汁酸。具有胆盐水解酶(bile salt hydrolase,BSH)活性的微生物可通过水解CBA释放胆盐和氨基酸(牛磺酸或甘氨酸)的方式形成胆汁耐受,从而帮助这些微生物在动物胃肠道进行定植,对宿主生理代谢也具有重要调节作用[2]。
近年来的研究发现,BSH通过水解胆盐和调节动物体内的胆固醇代谢效率[3-4],BSH可以显著提高动物的脂质代谢和抑制体重增加。此外,近期研究表明,BSH通过调节胆汁酸代谢还可能影响动物的炎症反应[5],BSH还可能通过调节肠道菌群组成间接影响宿主的代谢和免疫功能[6]。目前已知BSH存在于多个种属中,包括乳杆菌、双歧杆菌、肠球菌等,不同来源的BSH在种类和表达量上存在显著差异,而非保守的氨基酸序列和空间结构也导致了酶活性和底物选择性的差异[7-8]。植物乳植杆菌(Lactiplantibacillus plantarum)作为动物胃肠道主要的微生物群落之一,乳酸杆菌来源的BSH分布广泛、活性较高,可在动物胃肠道中降解结合胆盐的过程中发挥重要作用[9]。
当前,国内外关于植物乳植杆菌来源BSH异源表达的研究主要集中在酶的胆盐胁迫现象和独特的底物识别机制上,而对于酶的活性、诱导表达条件以及空间结构的系统性研究相对较少。尽管已有研究探讨了BSH的底物识别机制,但对其酶学性质、诱导表达条件及空间结构的研究仍然缺乏。本研究通过异源表达系统获得高活性的重组BSH,系统研究其酶学性质,并通过生物信息学方法探究其结构特征,为深入理解BSH的结构与功能关系及其在动物胆固醇代谢中的作用提供新的见解。这一研究不仅有助于阐明BSH的作用机制,还为开发高效BSH制剂提供了新的思路和科学依据。
1 材料与方法
1.1 菌株来源、培养及质粒
植物乳植杆菌分离株827(Lactiplantibacillus plantarum strain 827,Lp827)分离自猪消化道,由西南大学动物医学院实验室保藏。该菌株在37 ℃厌氧条件下于MRS培养基中培养。大肠杆菌感受态细胞Top10、BL21(DE3)购自生工生物工程(上海)股份有限公司,接种于LB培养基,37 ℃培养。
实验所用质粒为实验室改造的pET32a载体,通过插入麦芽糖结合蛋白(maltose binding protein,MBP)标签序列进行了优化,重命名为pET570a。
1.2 DNA提取、引物设计与扩增
将分离株Lp827接种到5 mL MRS肉汤培养基中,37 ℃厌氧培养至OD600值达0.8~1.0,然后通过细菌基因组抽提试剂盒进行DNA抽提。根据NCBI数据库中植物乳植杆菌BSH两端基因保守序列设计引物,下划线处为酶切位点:
bsh-F:5′-TCTGGATCCATGTGTACTGCCATAAC-3(BamH I);bsh-R:5′-GGCCTCGAGTTAACTGCATAGTATTG-3(Sac I),引物委托生工生物工程(上海)股份有限公司合成。以DNA为模板进行PCR扩增、测序,得到BSH全序列。扩增条件:95 ℃预变性3 min;95 ℃变性30 s;55 ℃退火30 s;72 ℃延伸2 min,34个循环,72 ℃终延伸10 min。
1.3 重组LpBSH的诱导表达及蛋白纯化
PCR产物经琼脂糖凝胶电泳后,使用凝胶回收试剂盒纯化(omega E.Z.N.A®GEL Extraction Kit)。纯化产物与pET570a经BamH I和Sac I双酶切后,T4 DNA连接酶(TAKARA)16 ℃连接16 h。连接产物转化大肠杆菌TOP10感受态细胞,涂布于含100 mg/L氨苄西林(ampicillin,Amp)的LB平板,37 ℃培养过夜。挑取单菌落接种于5 mL含Amp的LB肉汤培养基中,37 ℃、220 r/min振荡过夜,提取质粒并经过鉴定后将质粒命名为pET570a-bsh。
将上述质粒入BL21(DE3)感受态细胞,涂布于含Amp的LB平板,挑取单菌落接种到含Amp的LB液体培养基,37 ℃,220 r/min培养至OD600值约为0.6,加入终浓度为0.5 mmol/L的IPTG诱导表达[1]。离心收集菌体(6 000×g,4 ℃,10 min),悬浮于裂解缓冲液(50 mmol/L Tris-HCl,300 mmol/L NaCl,10 mmol/L咪唑,pH 8.0)中,超声波破碎(250 W,工作3 s,间隔7 s,总时间10 min),12 000×g离心10 min收集上清液和沉淀,上清液经Ni-NTA亲和层析柱(生工)纯化目的蛋白,采用120 g/L SDS-PAGE分析重组蛋白的表达和纯化效果。
1.4 LpBSH活性测定及定义
采用改良的茚三酮法测定LpBSH活性[2]。首先建立牛磺酸标准曲线:配制0~50 mmol/L以10 mmol/L为梯度的牛磺酸标准液(1 mL体系),与10 g/L茚三酮试剂,溶于0.5 mmol/L柠檬酸缓冲液,调节pH值为5.5,经100 ℃水浴13 min后测定OD570。以浓度为横坐标,OD570为纵坐标绘制牛磺酸标准曲线。
BSH活性测定反应体系(2 mL)包含:0.1 mL纯化BSH溶液,1.8 mL 50 mmol/L磷酸缓冲液(pH 6.0),0.1 mL 200 mmol/L 牛磺胆酸(taurocholate acid, TCA)。37 ℃反应30 min后,加入0.2 mL 150 g/L三氯乙酸终止反应。离心(10 000×g,4 ℃,10 min)后取0.2 mL上清液与0.8 mL茚三酮显色剂混合,100 ℃水浴13 min,冷却至室温后测定OD570。
一个单位(U)BSH活性定义为:在标准反应条件下(37 ℃,pH=6.0),每分钟催化底物生成1 μmol牛磺酸的酶量。根据标准曲线换算酶活力,计算如公式(1)所示:
BSH活力![]()
(1)
1.5 重组LpBSH酶学性质的测定
1.5.1 pH对LpBSH活性的影响
在pH 4.5~8.0,以0.5为间隔测定重组LpBSH的酶活性。采用50 mmol/L柠檬酸钠缓冲液(pH 3.5~6.0)和50 mmol/L磷酸钠缓冲液(pH 6.0~8.5)调节pH。在每个pH点设置3个平行样本。
1.5.2 温度对LpBSH稳定性及活性的影响
将纯化后的LpBSH溶液分别在15~75 ℃下处理1 h,每5 ℃为1个梯度,处理后按照1.4节中方法测定LpBSH活性,每组设置3平行样本。以未经温度处理的BSH活性为100%,计算各温度点BSH的相对残余活性,以确定LpBSH蛋白热稳定性。
为测定重组LpBSH的最适反应温度,在24~52 ℃范围内,以3 ℃为间隔进行酶活性测定。以最大酶活性为100%,计算每个反应温度下LpBSH的相对酶活性,每组设置3个平行样本。
1.5.3 金属离子对LpBSH的影响
在1.4节酶活性测定体系中分别加入Fe3+、Fe2+、Cu2+、Mn2+、Zn2+、Na+、K+、Ca2+,并设置金属离子浓度为0.5 mmol/L,在最适pH和温度下反应30 min。为了突显金属离子对LpBSH的影响,以不含金属离子的反应体系作为对照计算实验组BSH的催化活性。
1.5.4 LpBSH反应动力学模型测试
为了对比重组LpBSH和野生型LpBSH的催化曲线是否发生变化,设置若干梯度0~20 mmol/L的TCA划分为20组,分别作为反应底物。在最适温度和pH下分别使用野生型和重组LpBSH进行酶促反应。避光孵育30 min,根据BSH水解产物牛磺酸的标准曲线,计算BSH催化水解反应的速率。对每个底物浓度点,测定3个平行样本,实验重复2次。使用Origin 2022软件进行数据拟合,置信区间设为95%,根据Hill方程进行模型拟合,推算出BSH反应动力学参数。Hill计算方程如公式(2)所示:
(2)
式中:y,底物分解速率,单位时间内底物的水解量,μmol/(L·min);k,半最大速率常数,达到1/2Vmax时的底物浓度;n,希尔系数,表征底物结合的协同性;x,底物浓度,mmol/L;Vmax,最大反应速率,LpBSH在饱和底物浓度下的最高催化效率,μmol/(L·min)。
1.6 BSH的生物信息学分析
1.6.1 BSH氨基酸序列的同源比对及进化分析
将测序得到的核酸序列翻译成蛋白质序列,在NCBI蛋白质数据库中检索其他来源的BSH序列:双歧杆菌(Bifidobacterium longum)、加氏粘液乳杆菌(Limosilactobacillus gasseri)、唾液联合乳杆菌(Ligilactobacillus salivarius)、粪肠球菌(Enterococcus faecium)等的BSH序列。采用MEGA 11软件进行多序列比对和构建系统发育树。采用Neighbor-Joining法构建系统发育树,Bootstrap值设为1 000。
1.6.2 LpBSH空间结构和疏水性的预测与分子对接
使用SWISS-MODEL在线服务器预测BSH的三维结构,选取同源性最高的BSH晶体结构作为模板。采用PyMOL 2.3软件进行结构可视化分析。利用ExPASy ProtScale工具预测BSH的疏水性。通过PLIP在线服务预测BSH与底物TCA和甘氨胆酸(glycocholic acid, GCA)的相互作用,采用AutoDock Vina进行分子对接模拟。基于对接结果,确定底物结合口袋(距离<5 Å)并标注活性中心。结合氨基酸序列分析,寻找潜在影响水解胆盐的关键位点。
1.7 统计分析
所有实验均进行3次独立重复。数据以“平均值±标准差”表示。使用Graphpad Prism8软件进行统计分析,采用单因素方差分析(One-way ANOVA)进行组间比较,P<0.05认为差异具有统计学意义。
2 结果
2.1 BSH基因扩增结果
从L.plantarum strain 827中成功扩增出约1 000 bp的bsh基因片段(图1)。测序结果与NCBI数据库中已知L.plantarum BSH基因序列同源性达99%,确认为目的基因。将BSH基因成功克隆至pET570a载体中,构建重组表达质粒pET570a-bsh。
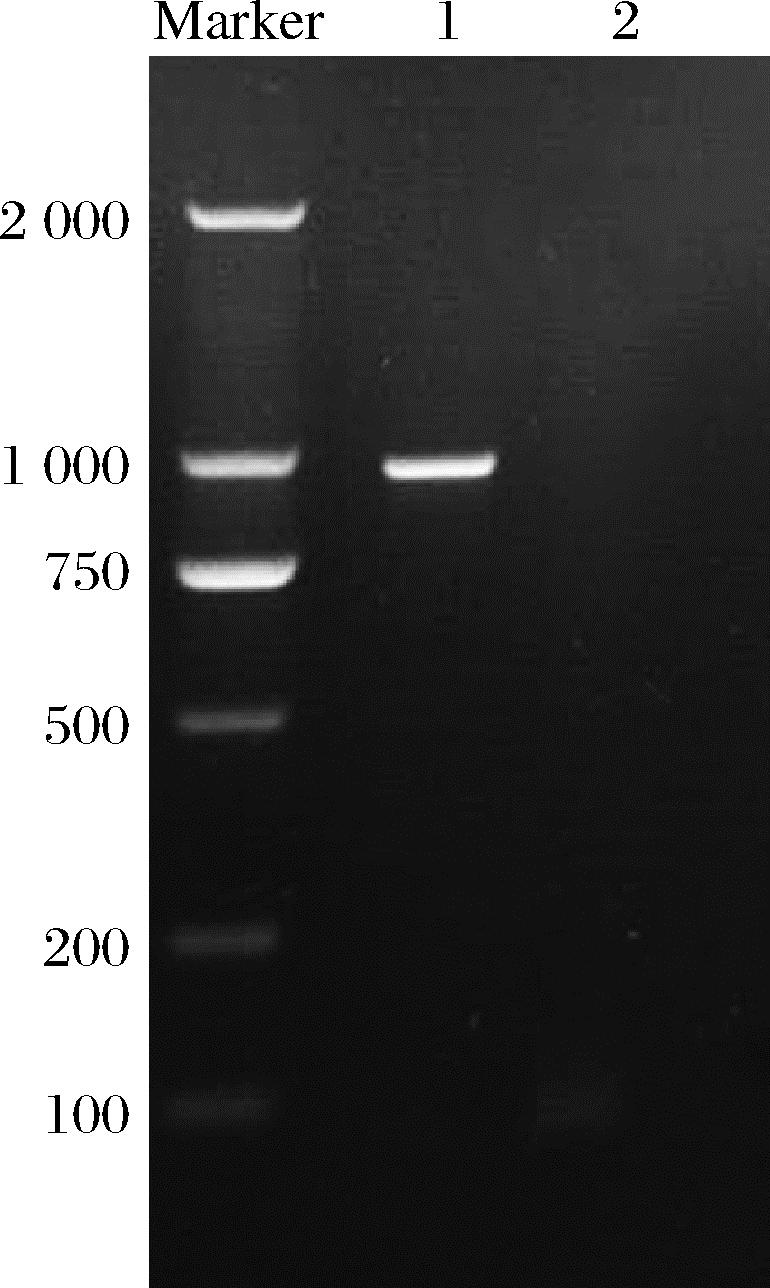
Marker-2 000 bp DNA Ladder;1-植物乳植杆菌bsh;2-阴性对照。
图1 Lp827中bsh的PCR结果
Fig.1 PCR amplification of bsh from Lp827
2.2 重组LpBSH的诱导表达与纯化
SDS-PAGE分析结果(图2)显示,与空载质粒组相比,0.5 mmol/L IPTG诱导后在约75 kDa处出现明显的蛋白条带,与预期的融合蛋白(BSH - MBP)分子质量相符。Ni-NTA亲和层析纯化后得到单一蛋白条带,说明bsh基因经pET570a载体在大肠杆菌BL21(DE3)中正常表达。
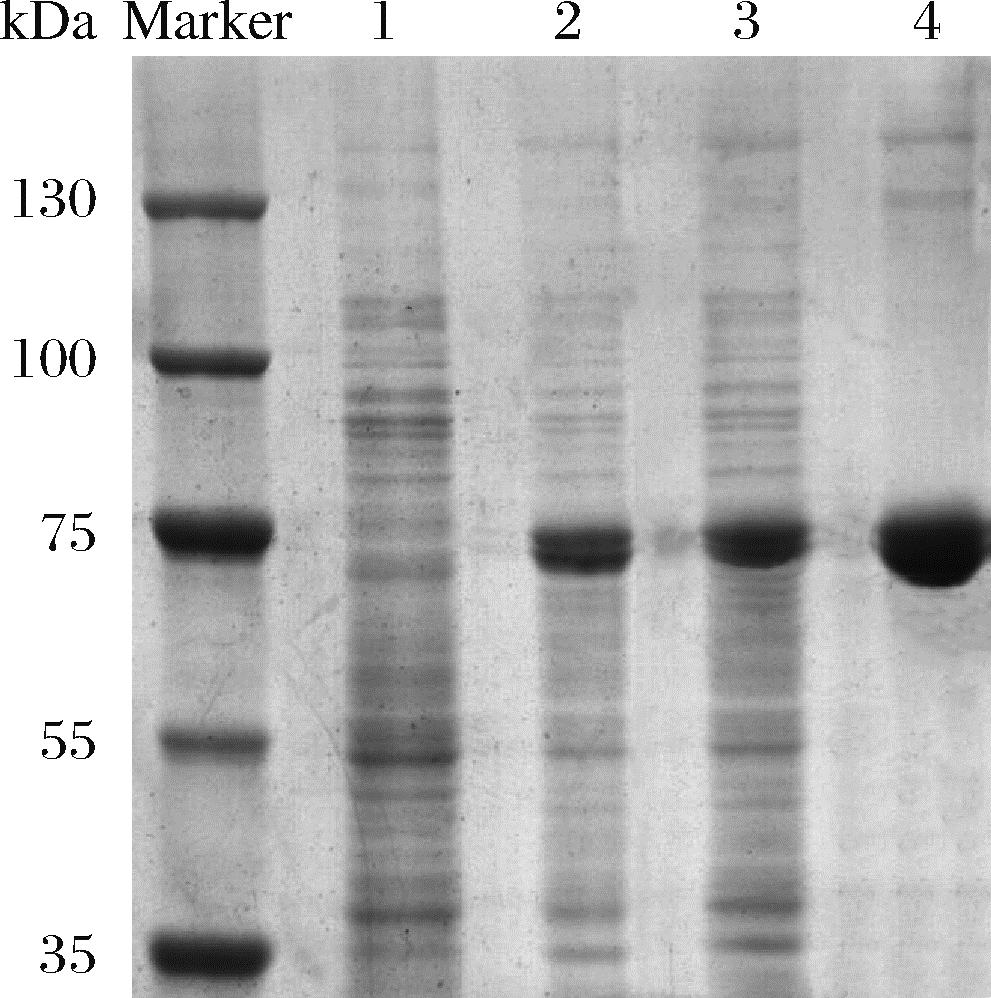
Marker-130 kDa彩色预染蛋白;1-BL21空载;2-破碎后上清液;3-破碎后沉淀;4-纯化后蛋白。
图2 LpBSH蛋白表达与纯化结果
Fig.2 Results of LpBSH protein expression and purification
2.3 LpBSH活性检测结果
建立的牛磺酸浓度标准曲线方程如公式(3)所示:
y=0.172x-0.409(R2>0.999)
(3)
式中:y,OD570处的吸光度;x,牛磺酸浓度,mmol/L。
在不同诱导时间分别采样,用破碎离心后的上清液进行酶活性测定以优化诱导条件。结果见图3,可以看出,0.5 mmol/L IPTG诱导下0~6 h BSH活性不断升高,6 h时达到最大酶活性,为(88.97±3.21) U/mL。值得注意的是,BSH活性在6 h后有缓慢下降的趋势,在诱导12 h后BSH活性仅有峰值的86.55%,这可能是由于IPTG具有一定的细胞毒性导致。
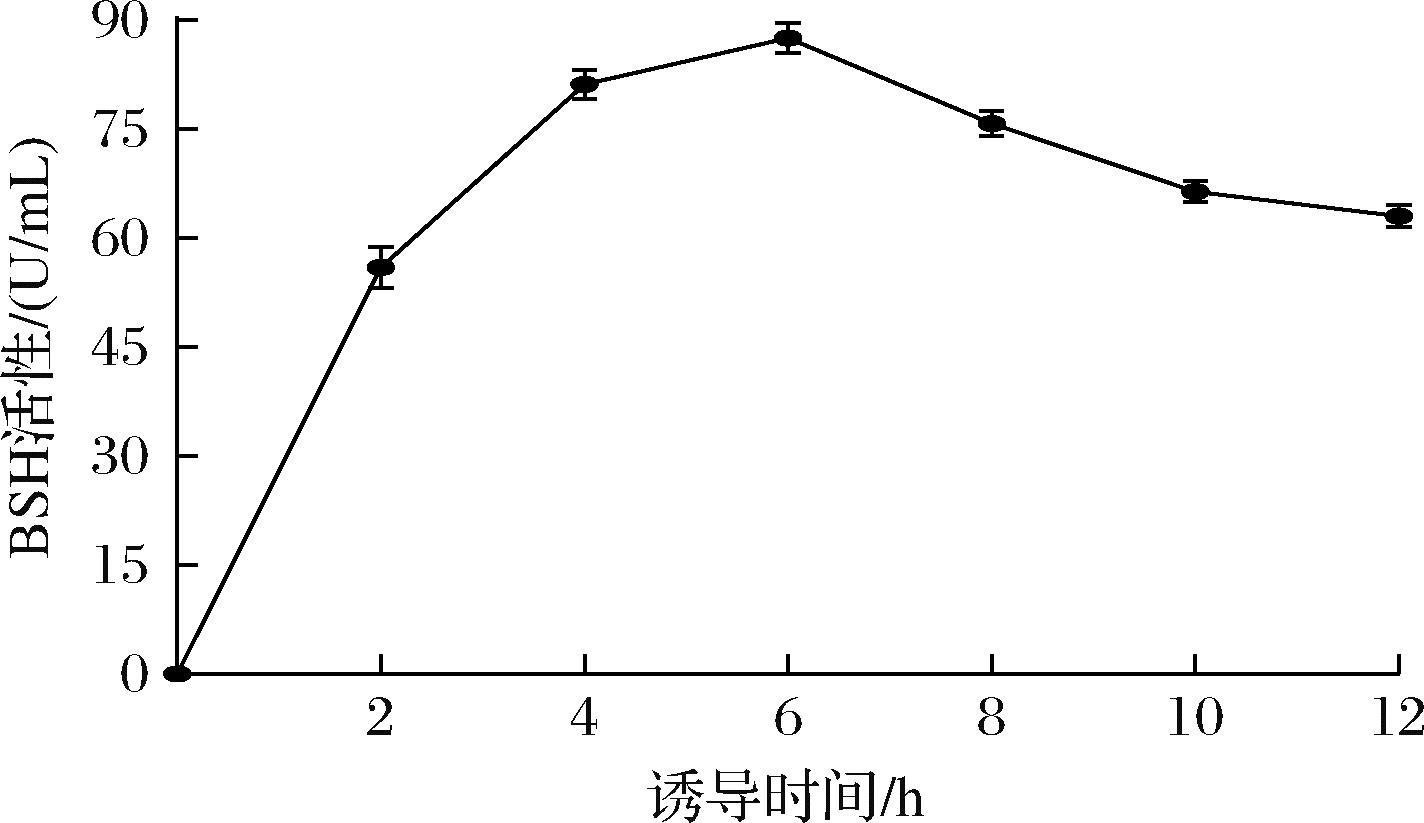
图3 不同诱导时间对表达LpBSH活性的影响
Fig.3 Effect of different induction time on the activity of LpBSH expression
2.4 重组LpBSH的酶学性质
2.4.1 PH对LpBSH活性的影响
pH活性曲线(图4)显示,对于LpBSH最适pH=5.5,当pH>7.0时LpBSH活性迅速下降,这也印证了植物乳植杆菌在动物胃肠道中存在的胆盐胁迫现象。相比之下,重组LpBSH在pH=6.5时其活性达到峰值,为(88.97±3.21) U/mL,在pH 4.5~8.0保持较高活性(>70%最大活性)。这一结果表明,重组BSH具有更宽的pH适应范围。
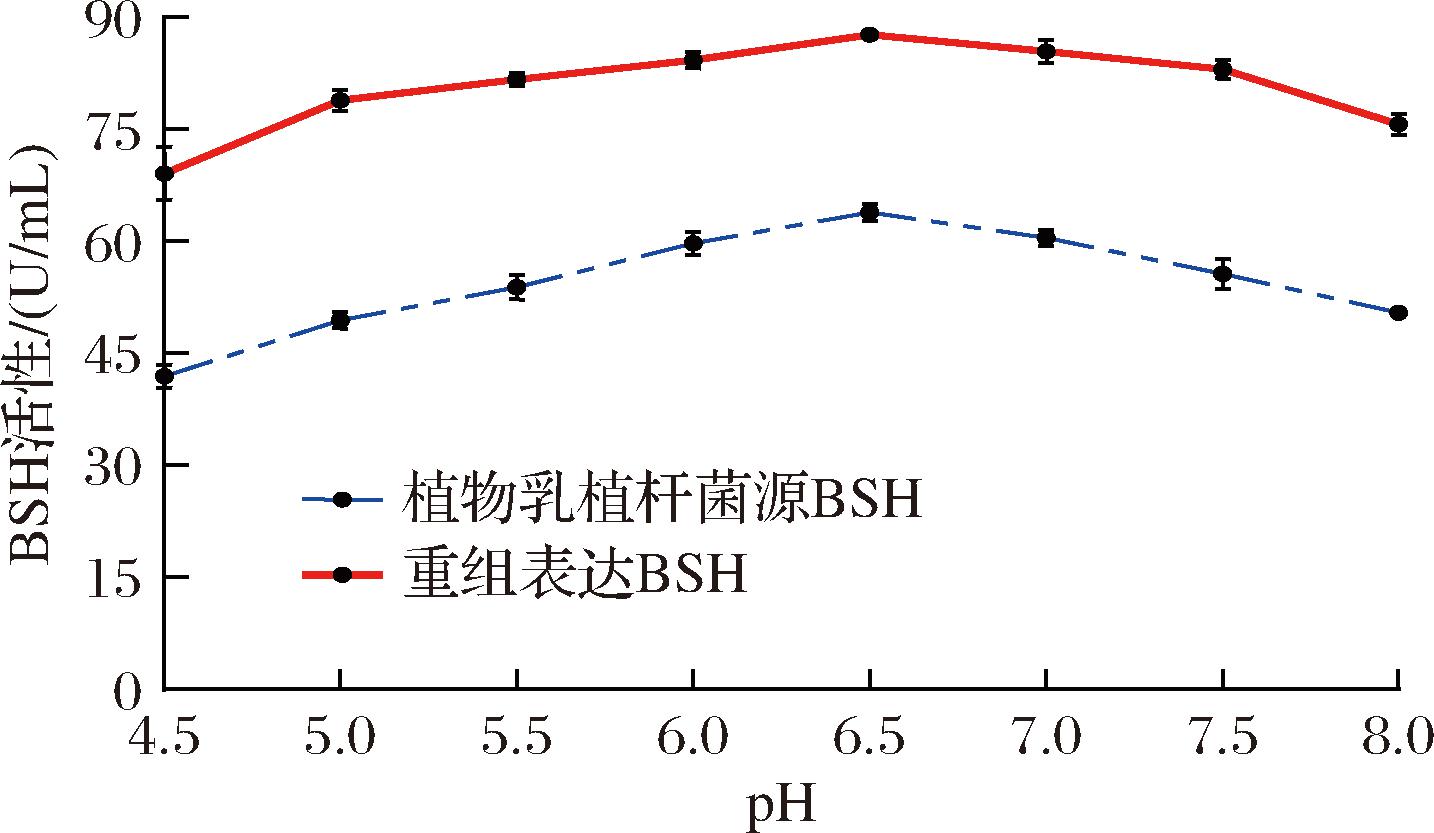
图4 pH对LpBSH活性的影响
Fig.4 Effect of pH on LpBSH activity
2.4.2 温度对LpBSH活性及稳定性的影响
温度活性曲线(图5-a)显示,重组LpBSH在37 ℃时达到最大催化活性,在31~40 ℃范围内保持较高活性(>90%最大活性)。温度稳定性实验(图5-b)表明,在15~35 ℃范围内处理1 h后,BSH仍保留(64.44±0.35)%以上的残余活性。65 ℃处理1 h后仅保留(2.9±0.17)%的残余活性,70 ℃及以上完全失活,表明BSH对高温敏感。
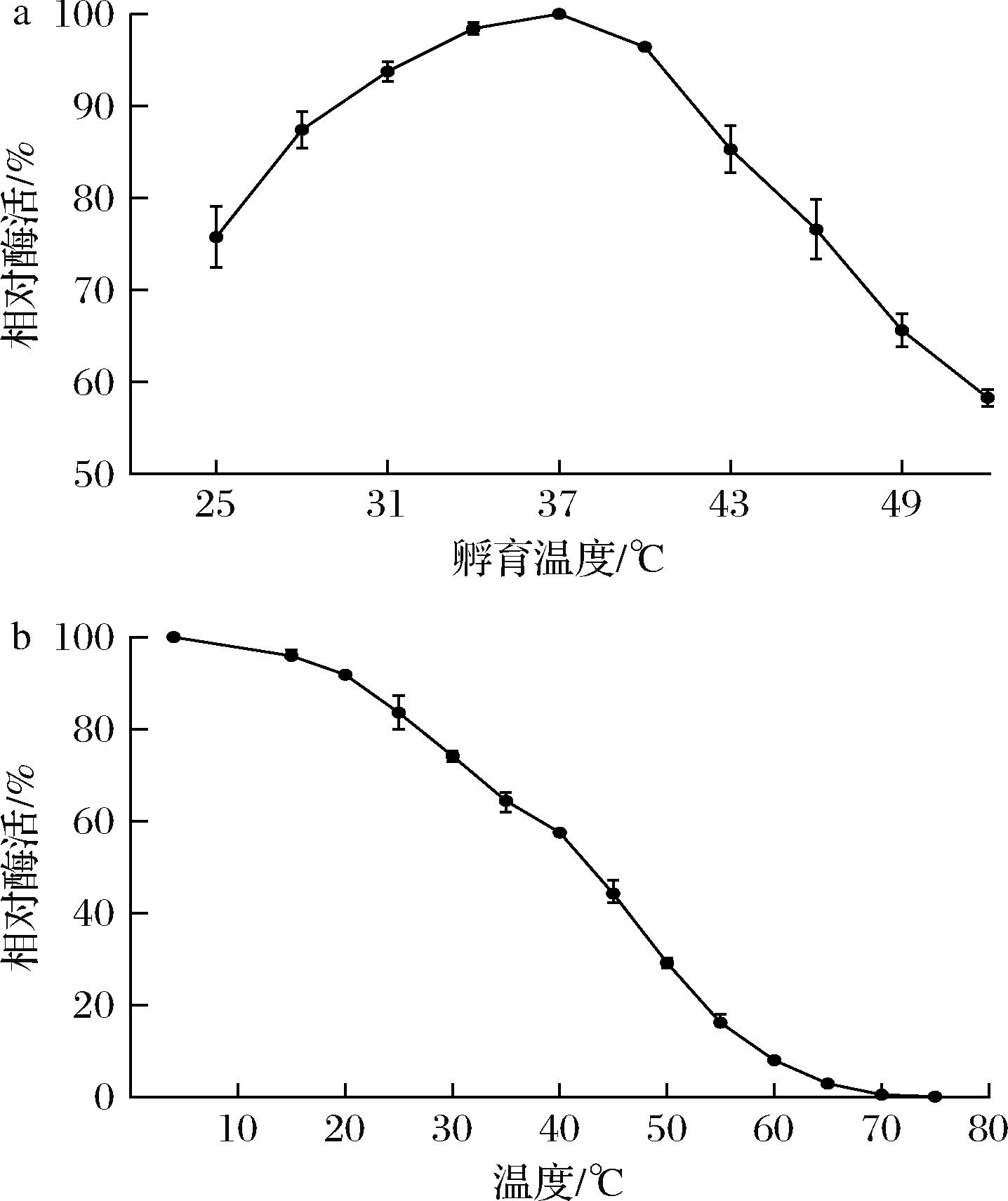
a-温度活性曲线;b-温度稳定性
图5 温度对LpBSH活性及稳定性的影响
Fig.5 Effect of temperature on the activity and stability of LpBSH
2.4.3 不同金属离子对LpBSH活性的影响
金属离子对LpBSH活性的影响如图6所示,各金属离子对LpBSH活性均有明显促进作用,仅K+有细微促进作用(P<0.05)。而Cu2+、Ca2+、Mn2+对BSH有显著的抑制作用(P<0.001),Fe3+和Fe2+的抑制作用最为显著。这可能是重金属离子与BSH 活性位点Cys2 的巯基形成了金属螯合物或络合物使部分催化残基失去催化功能导致的。野生型BSH表现出类似的金属离子敏感性模式,见图6蓝色部分。
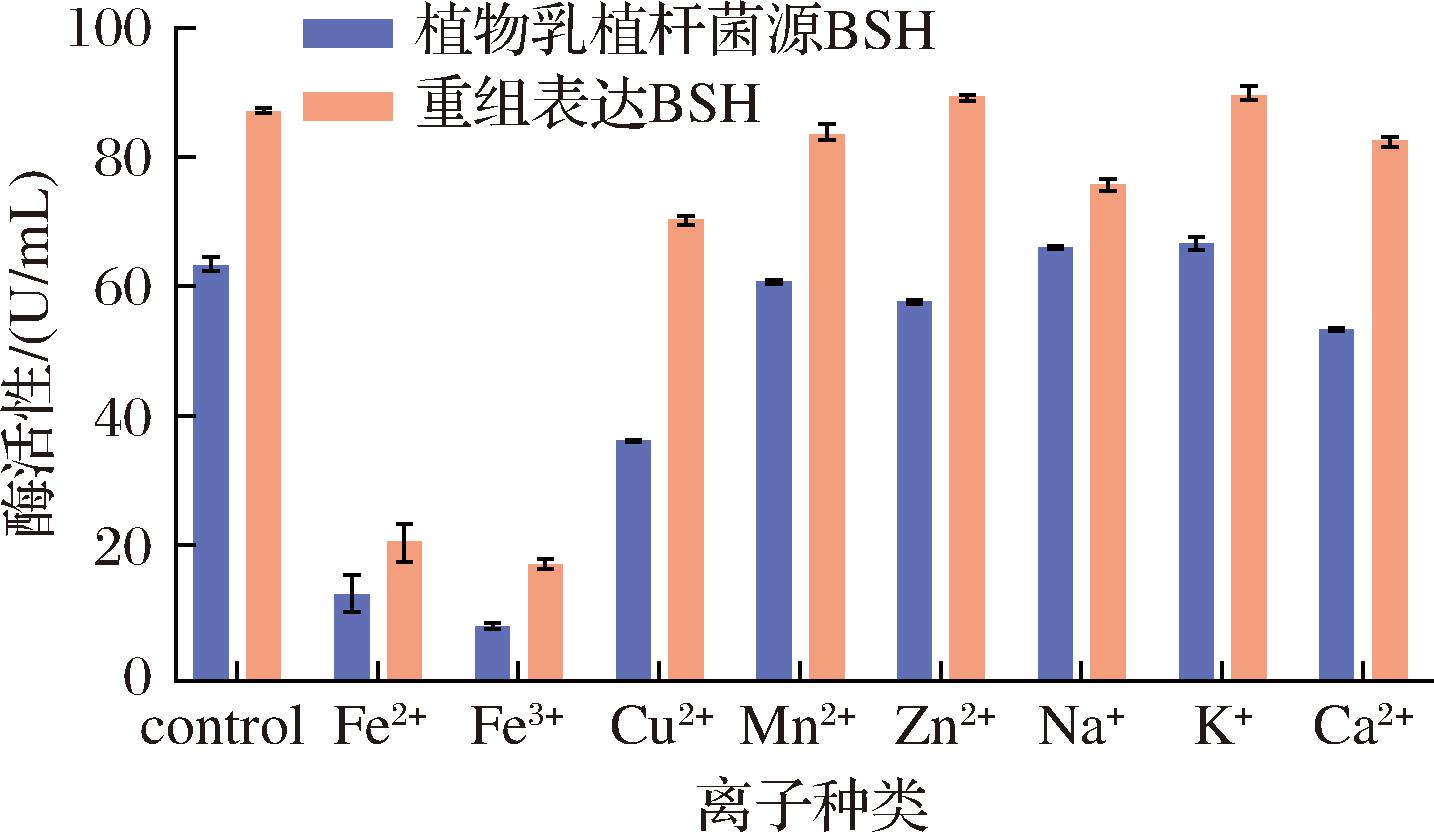
图6 金属离子对LpBSH催化活性的影响
Fig.6 Effect of metal ions on catalytic activity of LpBSH
2.5 LpBSH反应动力学模型
动力学分析结果(图7、表1)显示,重组LpBSH和野生型LpBSH的催化反应均符合Hill方程(P<0.05)。2种酶的Hill系数n均大于1,表明底物对BSH催化反应具有正协同作用。且图7中底物浓度与BSH催化速率曲线成“S”形而非双曲线形,说明BSH属于别构酶。此外,重组BSH表现出更高的最大反应速率(Vmax)和更低的半饱和底物浓度(K),说明其具有更高的催化效率和底物亲和力。
表1 LpBSH反应动力学参数
Table 1 Kinetic parameters of LpBSH

类别底物Hill系数(n)Vmax/[μmol/(L·min)]Kr2植物乳植杆菌BSHTCA1.159±0.102433.660±10.9691.049±0.0860.988重组BSHTCA1.701±0.193715.722±14.3961.046±0.0750.982
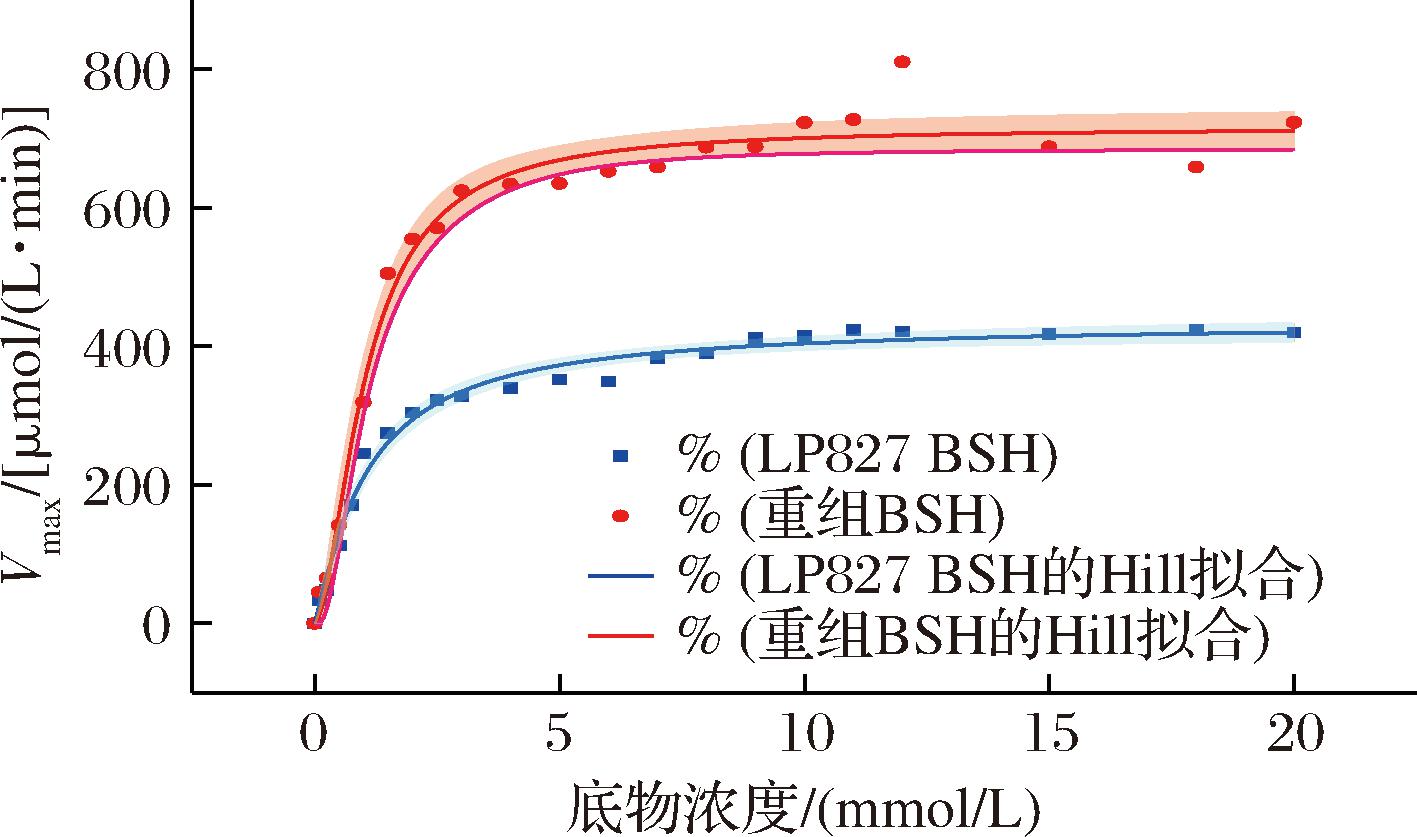
图7 LpBSH反应动力学模型
Fig.7 Kinetic model of LpBSH
2.6 LpBSH的生物信息学分析
2.6.1 BSH同源比对及系统发育树构建
将2.1节中PCR产物测序结果转换为氨基酸序列进行BSH同源比对并建树,结果见图8。可以看出实验室分离的L. plantarum strain 827和数据库中的植物乳植杆菌BSH亲缘关系非常近,且在同一分支中,而与林氏果实乳杆菌(Fructilactobacillus lindneri)和鼠李糖乳酪杆菌(Lacticaseibacillus rhamnosus)BSH亲缘性较远。
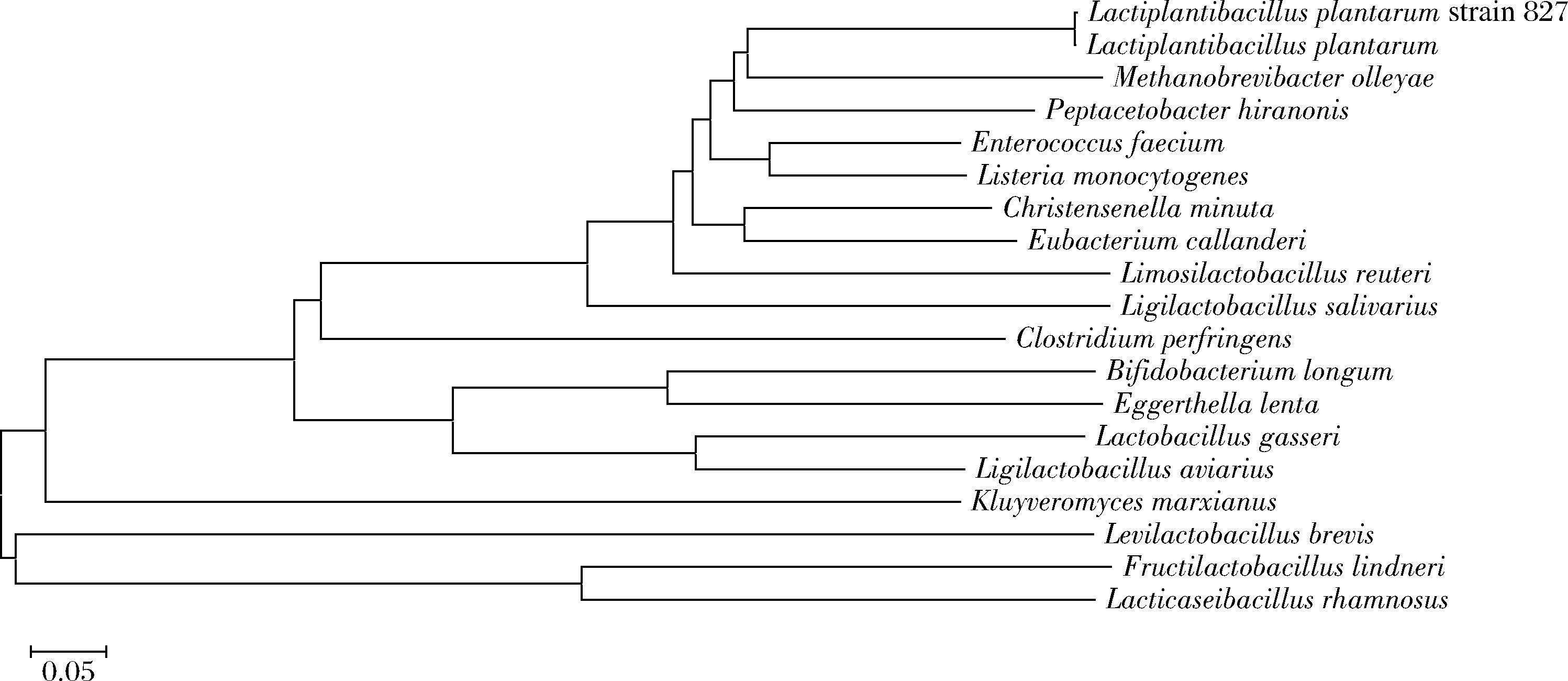
图8 基于N-J法构建的系统发育树
Fig.8 Neighbor-Joining phylogenetic tree based on BSH sequence
2.6.2 BSH疏水性分析
利用ProtScale工具对LpBSH进行疏水性预测,结果见图9。可以看出,LpBSH的“疏水性-亲水性”分布呈现周期性波动,这种模式与典型的胞内酶特征相符。值得注意的是,在预测的活性位点和底物结合口袋附近,存在明显的疏水性-亲水性交替区域。这种特殊的氨基酸分布可能对LpBSH与底物的结合和催化起关键作用。结合已报道的BSH底物结合口袋、活性位点[3],和SWISS MODLE预测出的空间结构来看,构成BSH的α螺旋和β折叠的氨基酸多呈现疏水性,这可能是由于疏水区域的蛋白质结构更稳定,可以通过疏水键与底物中的疏水性结构结合。具体而言,疏水区域可能通过疏水相互作用稳定LpBSH的空间结构,并与底物的疏水部分结合;而亲水区域则可能参与催化反应和产物释放。这也印证了与LpBSH底物结合口袋的特异性与氨基酸特性及蛋白质空间结构是分不开的[4-5]。
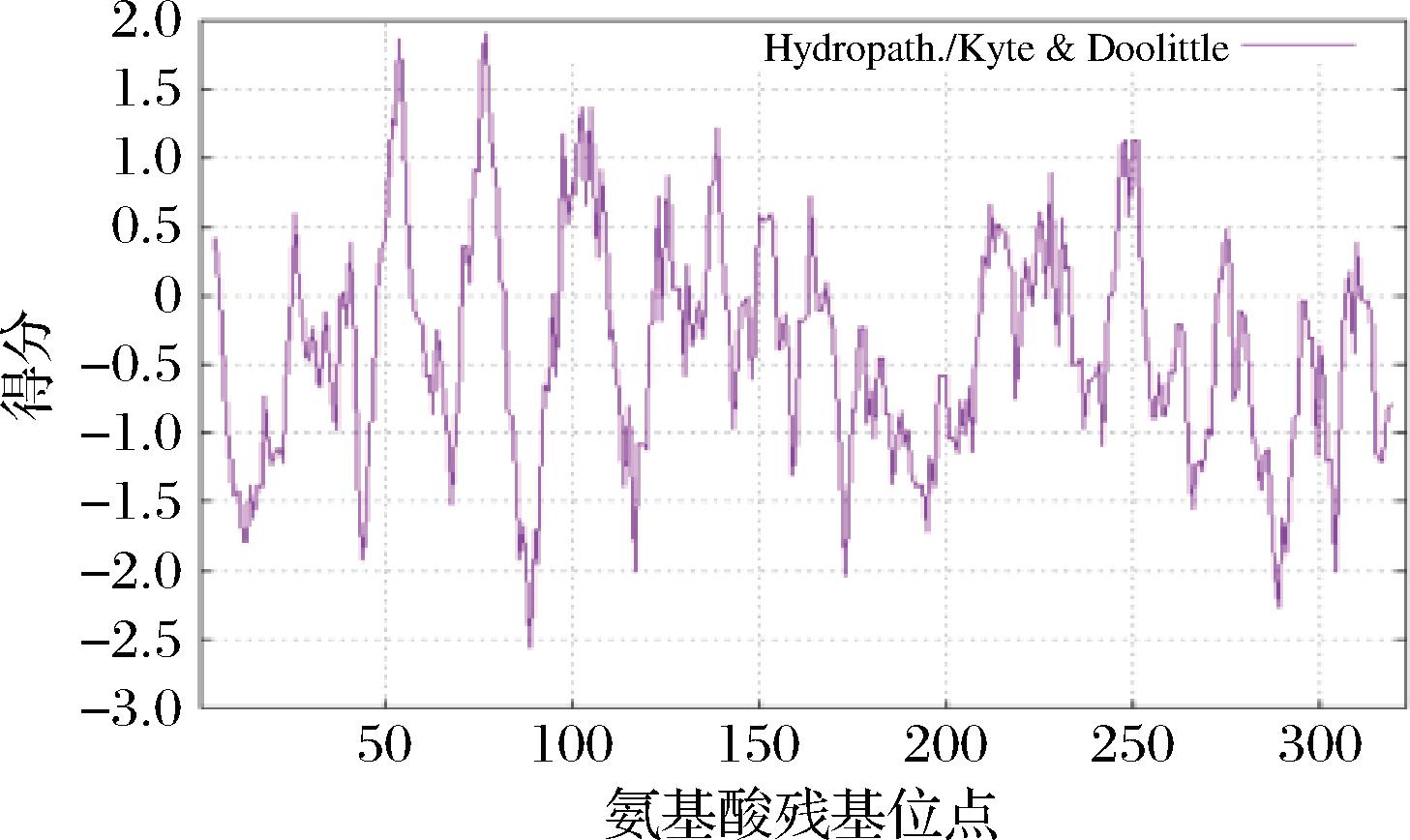
图9 LpBSH疏水性预测结果
Fig.9 Prediction results of LpBSH hydrophobicity
注:Hydropath./Kyte&Doolittle代表Kyte&Doolittle法预测的疏水性评分。
2.6.3 BSH的三维结构预测
通过SWISS MODEL的同源建模,预测了分离株Lp827 BSH的三维结构,所使用的模板(PDB ID:5 gs7.1)与目标序列的同源性为87%,QMEAN分数为0.98,表明预测模型具有较高的可信度。与已报道的其他种属细菌的BSH一样[6-8],植物乳植杆菌BSH同样是多聚体,具体来说是四聚体,见图10-a。为了便于寻找到活性位点和环区,将其拆分为单体,见图10-b。单体结构展示了典型(N-terminal nucleophile,NTN-)水解酶家族特征,包括α-β-β-α四层三级结构折叠[7,9]。4个单体通过静电作用、疏水相互作用和氢键形成稳定的四聚体,这种四聚体结构可能对维持BSH的催化活性和底物特异性至关重要。特别是,4个单体中的loop结构(第185~219位氨基酸)共同形成的氧阴离子洞(oxyanion hole)可能在平衡电荷和决定底物特异性方面发挥关键作用[3]。此外,ROSSOCHA等[7]则发现,BSH的底物结合口袋是由2个BSH单体的3个β折叠形成的,严格保守的的Cry2和Arg18是催化反应的关键残基,对LpBSH催化活性起关键作用。
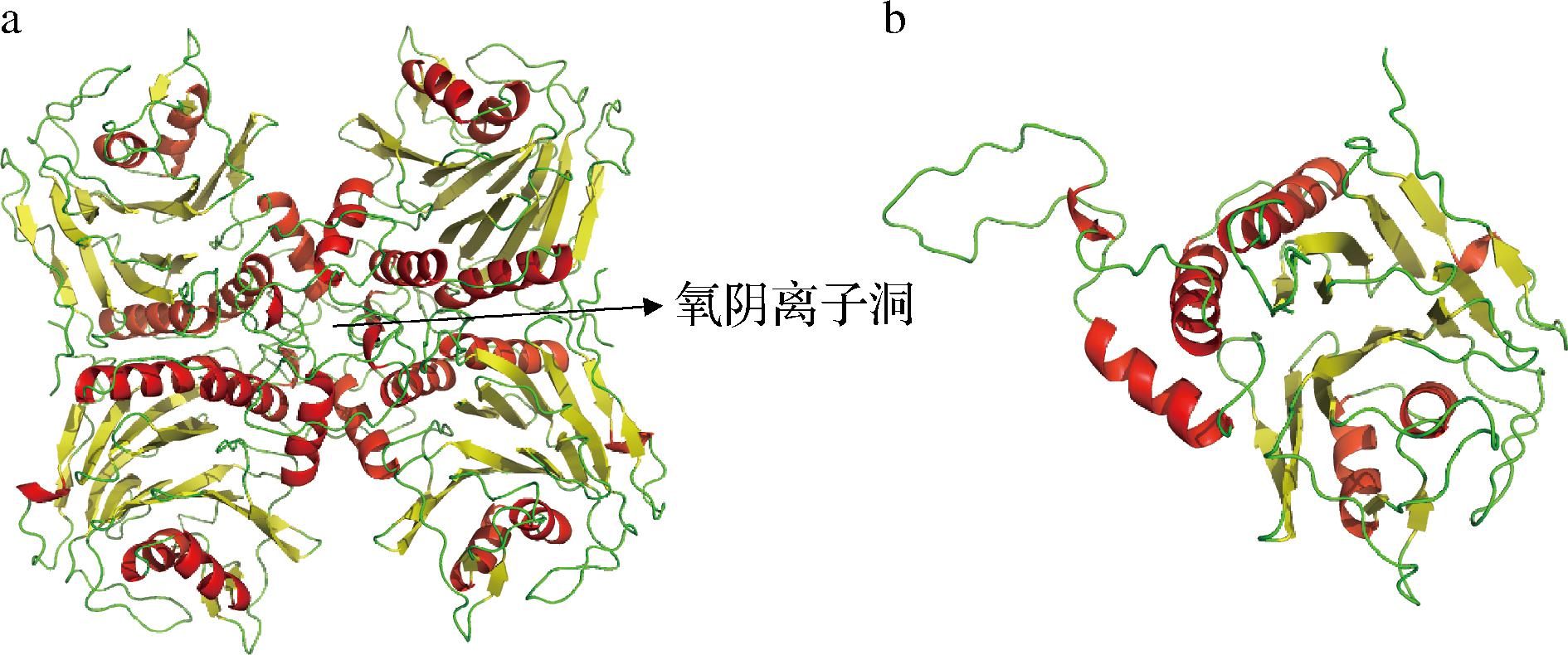
a-植物乳植杆菌BSH空间结构预测(四聚体);b-植物乳植杆菌BSH空间结构预测(单体)
图10 LpBSH空间结构预测
Fig.10 Spatial Structure Prediction of LpBSH
注:红色为α螺旋,黄色为β折叠。
2.6.4 氨基酸序列比对
对多个已报道具有底物特异性的BSH和L.plantarum strain 827的氨基酸序列进行多序列比对分析[10-12],见图11。
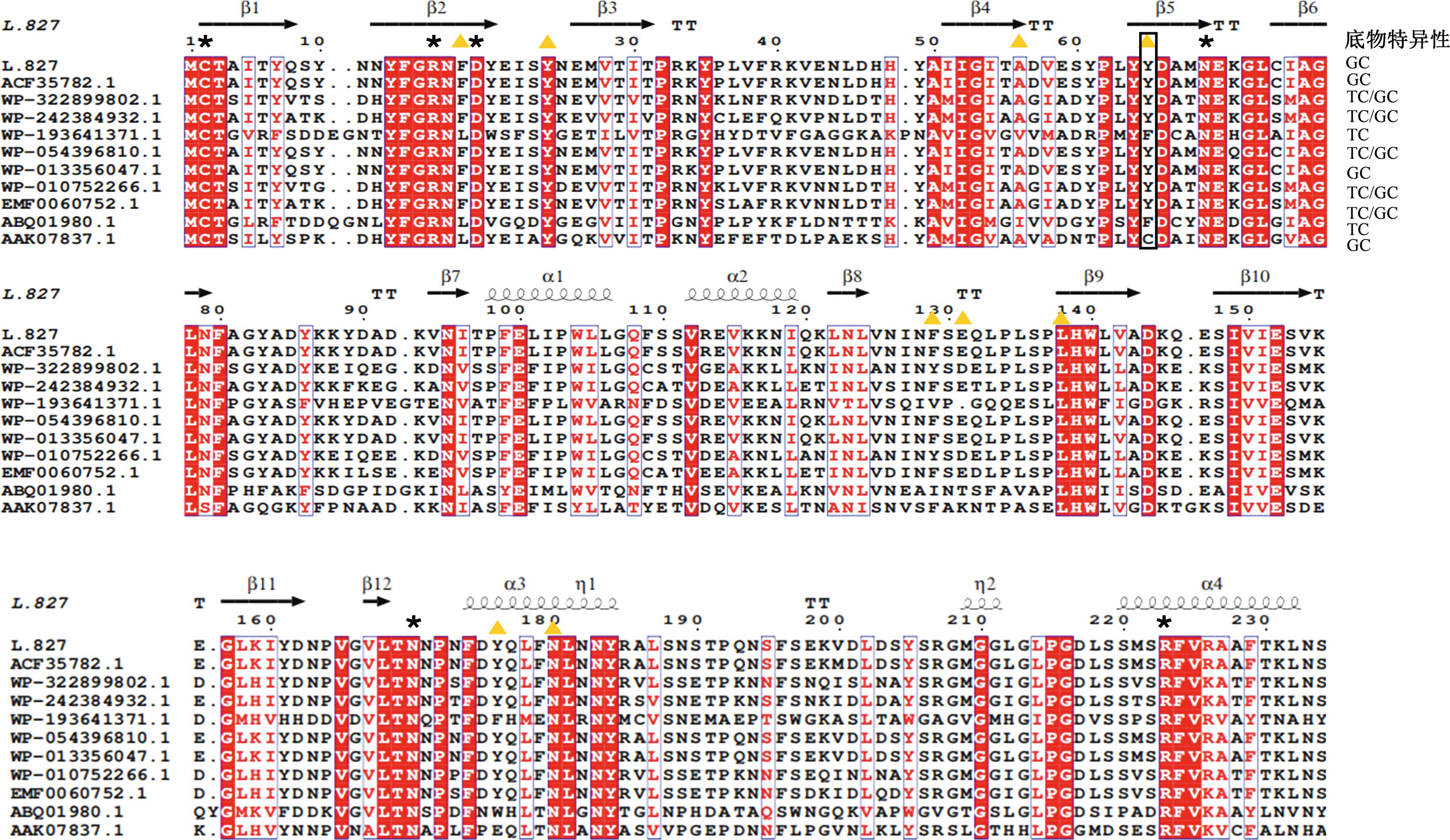
图11 不同来源BSH氨基酸序列比对
Fig.11 Amino acid sequence comparison of BSH from different sources
注:序列上方二级结构来源于Swiss modle 预测的L.plantarum strain 827;*代表BSH活性位点;黄色三角,代表底物结合口袋;黑色方框,代表L.plantarum strain 827 67-Try对应的BSH底物结合位点;红色阴影,代表同源性100%;蓝色方框,代表同源性大于80%。
结果显示这些序列的相似度范围为87.24%~99.9%。基于SWISS MODEL预测的晶体结构和现有文献[13-14],确定了BSH的活性位点、二级结构元件和底物结合口袋。分析发现,活性位点的氨基酸(Cys、Asp、Asn、Arg)高度保守(同源性为100%);相比之下,底物结合口袋的氨基酸多为非保守的疏水性氨基酸,这种差异性可能是不同来源BSH底物特异性差异的分子基础。特别值得注意的是,在以L.plantarum strain 827为参考的第67位氨基酸(Tyr67)对BSH底物特异性识别有显著影响。当该位置为非极性氨基酸(如Phe或Ala)时,BSH通常表现出对牛磺型结合胆盐的偏好性;当为极性氨基酸(如Cys或Tyr)时,则倾向于识别甘氨型结合胆盐,这一发现与CHAE等[11]的报道一致,为BSH底物特异性的调控提供了潜在的分子靶点。此外,即使在同一属的BSH中也观察到底物偏好性的差异,这可能与BSH的表达环境有关,提示了BSH功能适应性的复杂性。
2.6.5 BSH与底物的分子对接
基于已报道的BSH-TCA相互作用位点和保守序列分析[15-17],使用AutoDock Vina进行分子对接,预测Lp827 BSH与TCA的结合模式(对接过程中使用了柔性配体-刚性受体策略,搜索空间覆盖整个BSH单体,使用默认的评分函数评估结合亲和力)。对接结果(图12)显示,可以看出与TCA形成稳定分子作用力的10个氨基酸残基(Cys2、Arg18、Leu38、Ala56、Leu63、Asn79、Pro98、Phe129、Ser136、Leu138)主要集中在α-β-β-α结构的两个β层之间,其中Cys2、Arg18、Asn79在不同来源的BSH中都是高度保守的催化活性位点。TCA主要与BSH单体的一个疏水口袋相互作用,该口袋由多个保守残基组成。氢键和疏水相互作用是稳定BSH-TCA复合物的主要力量。基于分子对接,预测了BSH催化水解结合胆盐的过程(以TCA为例),当Asn79、Asn175识别到反应潜在底物时[8],BSH N-末端会对底物中酰胺键中的碳原子亲核进攻,随后Cys2会被激活、形成一个自由化的α-氨基基团并通过氢键与水分子结合,这种结合桥接到亲核的半胱氨酸硫原子。这样,半胱氨酸的亲核性得到了增强,有利于进一步的催化反应[18-19]。随后Arg18将稳定带有负电荷的硫氢基团,同时启动自催化的功能[7]。经过氨基酸的释放后,Cys2对底物中的硫酯键进行第2次亲核进攻。这一步骤释放了胆酸部分,并通过另一个四面体中间体使酶再生,从而恢复酶的催化能力。在BSH与底物的相互作用中,对接上的其他氨基酸的作用可能是使得底物能够更好地适配到催化位点或维持氧阴离子洞以维持反应正常进行,从而提高了反应的效率。
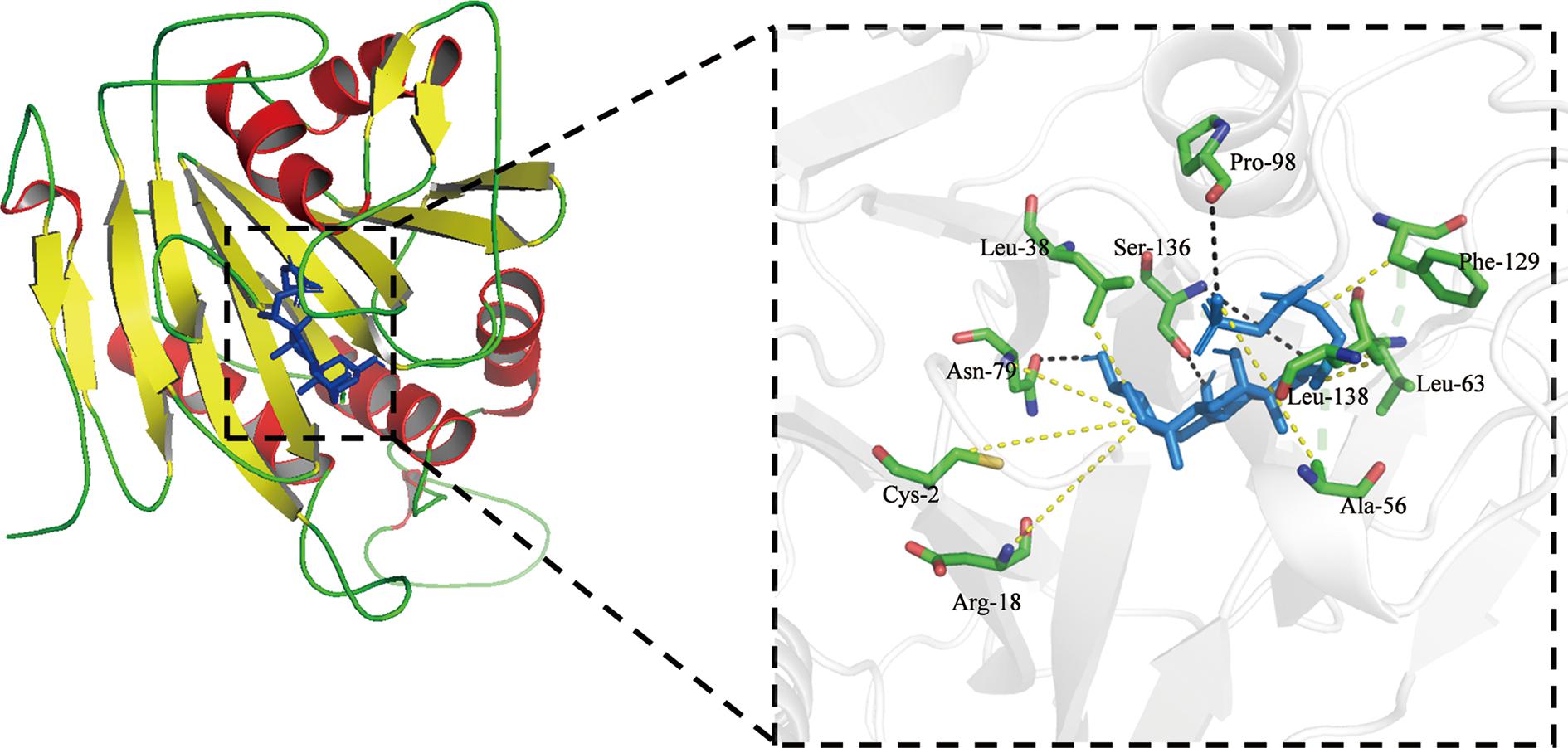
图12 LpBSH与TCA结合残基
Fig.12 LpBSH and TCA binding residues
注:虚线框中,淡蓝色部分为TCA,白色部分为LpBSH单体,其余部分为底物识别相关残基;黄色虚线代表疏水作用力、深蓝色虚线代表氢键。
此外,有研究报道,BSH的空间结构和酶分子的底物选择性及作用机理密切相关[4,18],已知BSH类有4个结合环(loop1~loop4)[20],它们构成底物结合口袋,其中loop1~3中存在催化反应的关键残基,而1oop4中未发现相关催化残基,这些环区的空间结构被认为会影响酶活力及底物亲和能力[5, 8]。通过Autodock分子对接,进一步分析BSH与甘氨酸结合GCA和TCA的结合模式(图13)发现,2种底物的结合口袋存在微妙差异。GCA主要与环区2(57~64位氨基酸)和部分环区3(125~137位氨基酸)相互作用,而TCA主要与环区3以及部分环区2和环区4(255~269位氨基酸)结合。环区2在空间上更接近BSH的关键活性位点,这可能也是Lp827 BSH对GCA的催化活性高于TCA的实验观察的原因之一。
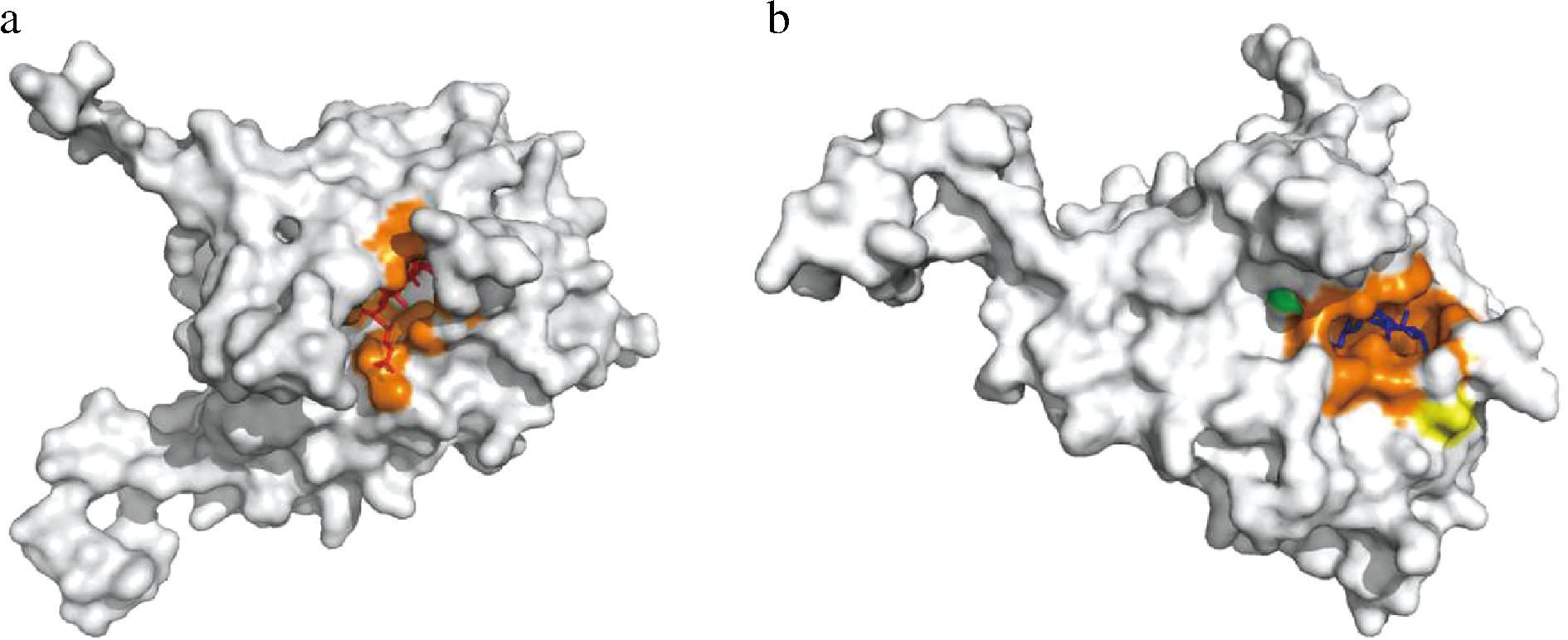
a-LpBSH与GCA的结合口袋;b-LpBSH与TCA的结合口袋
图13 LpBSH的底物结合口袋
Fig.13 The substrate-binding pockets of LpBSH
注:橙色为结合口袋;绿色、黄色为活性位点。
3 讨论
BSH能够水解动物体内结合态胆盐生产氨基酸和初级胆汁酸,初级胆汁酸与胆固醇结合形成沉淀并随粪便排出,是降低机体内胆固醇含量的重要途径之一。据目前的报道,BSH主要来源于乳杆菌,是乳杆菌自身代谢产物之一的胞内蛋白[5,21]。本研究成功实现了植物乳植杆菌来源BSH在大肠杆菌中的高效重组表达,并对其酶学性质和结构特征进行了深入探讨。通过添加MBP标签以减少包涵体的生成同时优化表达条件和纯化方法,获得了活性显著高于野生型的重组LpBSH,为进一步开发高效BSH制剂奠定了基础。
大量包涵体的产生是BSH原核表达存在的问题之一,其本质由于BSH在宿主细胞中被过度表达,超过了细胞的折叠能力,产生的蛋白错误折叠和聚集。在已有的报道中,通过尿素或苯甲基磺酰氟对BSH包涵体复性后会引起活性的降低[16,22]。本研究中通过在目的片段前添加MBP标签作为分子伴侣,不仅增加了目标蛋白的溶解度,还有助于维持其正确折叠。添加MBP标签后,重组LpBSH的活性显著高于野生型,这可能是由于MBP标签促进了BSH的正确折叠和稳定性。这一发现为优化其他难以表达的酶提供了新的思路。
通过对分离株Lp827酶学性质的研究,发现重组LpBSH在pH 4.5~8.0范围内保持较高活性,最适pH值为6.5,这比野生型LpBSH(最适pH 5.5)具有更宽的pH适应范围。这一特性可使重组LpBSH在模拟胃肠道复杂环境中表现出更好的适应性。温度稳定性实验表明,重组LpBSH在15~35 ℃保持良好的稳定性,这与其潜在的应用环境(动物体内温度)相符。然而,65 ℃以上处理会导致酶活性显著下降,这可能与预测的BSH四聚体结构有关,高温可能破坏四聚体间的相互作用,导致结构解离和活性丧失。这一发现为LpBSH在实际应用中的温度控制提供了重要参考。此外,本研究发现重金属离子尤其是Fe2+、Fe3+对BSH有明显的抑制作用,推测这些金属离子可能与BSH活性位点附近的特定氨基酸残基(如Cys2)相互作用,形成金属螯合物或络合物,从而影响了催化功能。在动力学试验中可以发现,重组LpBSH与野生型LpBSH均为“S”型催化曲线,同属别构酶,而重组LpBSH最大催化速率比野生型高出65.1%。基于上述野生型LpBSH和重组LpBSH的酶学性质和动力学的试验结果,可以得出相同培养时间、相同反应体系下重组LpBSH在pH稳定性和催化反应速率上均优于LpBSH,这可能是由于植物乳植杆菌和大肠杆菌对营养需求不同,导致单位体积培养基中的大肠杆菌密度高于植物乳植杆菌密度,此外由于LpBSH催化水解反应需要稳定的四聚体结构,不同表达系统可能会导致BSH分子间的相互作用发生变化,影响其四聚体结构的形成或稳定性,最终导致酶学性质差异和动力学差异,此外,MBP标签的插入一定程度增加了LpBSH的稳定性和溶解度,这也是导致二者之间酶学性质差异和动力学差异的原因之一。
通过与已报道的BSH空间结构或底物特异性对比[6-8, 23],结合生物信息学分析分子对接研究,探讨了BSH的结构特征与其底物特异性的关系。预测的Lp827 BSH四聚体结构具有与NTN-水解酶家族相似的结构特征,包括α-β-β-α四层三级结构折叠。此外,本研究发现底物结合口袋由多个非保守的疏水性氨基酸组成,其中第67位残基(以Lp827为参照)对BSH的底物偏好性有显著影响,该位点上的氨基酸是否有极性与BSH水解结合胆盐的偏好性有关,这可能是不同来源BSH底物偏好性的分子基础之一。
分子对接揭示了BSH与不同底物(GCA和TCA)结合模式的差异。本研究发现GCA的结合口袋多集中于环区2(第57~64位氨基酸)和部分环区3(第125~137位氨基酸),而TCA的结合口袋则主要集中在环区3,少部分在环区2和环区4(第255~269位氨基酸)。Cys2作为BSH公认的活性位点,在空间结构上与Lp827 BSH的环区2更为接近,这可能使得GCA更容易地被催化水解,这也可能是BSH底物偏好性的原因之一。
本研究成功实现了植物乳植杆菌来源BSH在大肠杆菌中的高效重组表达,在此基础上研究了其酶学性质和结构特征。通过系统的实验设计和生物信息学分析,不仅优化了BSH的表达和纯化方法,而且推测出其结构与功能关系的几个原因,为理解BSH催化机制及底物偏好性奠定了理论基础。
[1] 周晓玲, 张娟, 陈坚, 等.双歧杆菌胆盐水解酶基因的重组表达、纯化与酶学性质[J].食品与生物技术学报, 2016, 35(8):792-800.ZHOU X L, ZHANG J, CHEN J, et al.Expression, purification and enzymatic properties study of the bile salt hydrolase from Bifidobacterium in Escherichia coli[J].Journal of Food Science and Biotechnology, 2016, 35(8):792-800.
[2] SRIDEVI N, VISHWE P, PRABHUNE A.Hypocholesteremic effect of bile salt hydrolase from Lactobacillus buchneri ATCC 4005[J].Food Research International, 2009, 42(4):516-520.
[3] GOPAL A, SHAH N P, ROGINSKI H.Bile tolerance, taurocholate deconjugation and cholesterol removal by Lactobacillus acidophilus and Bifidobacterium spp.[J].Milchwissenschaft, 1996, 51(11):619-623.
[4] ÖZTÜRK M, HAC BEYO
BEYO LU K, ÖNAL C, et al.Construction of R16F and D19L mutations in the loop I of bile salt hydrolase (BSH) enzyme from Lactobacillus plantarum B14 and structural and functional analysis of the mutant BSHs[J].Food Biotechnology, 2019, 33(2):125-141.
LU K, ÖNAL C, et al.Construction of R16F and D19L mutations in the loop I of bile salt hydrolase (BSH) enzyme from Lactobacillus plantarum B14 and structural and functional analysis of the mutant BSHs[J].Food Biotechnology, 2019, 33(2):125-141.
[5] 卢方云, 杨彪, 马晶晶, 等.胆盐水解酶酶学性质与基因结构研究进展[J].食品工业科技, 2023, 44(8):469-475.LU F Y, YANG B, MA J J, et al.Progress on enzymatic properties and gene structure of bile salt hydrolase[J].Science and Technology of Food Industry, 2023, 44(8):469-475.
[6] KUMAR R S, BRANNIGAN J A, PRABHUNE A A, et al.Structural and functional analysis of a conjugated bile salt hydrolase from Bifidobacterium longum reveals an evolutionary relationship with penicillin V acylase[J].Journal of Biological Chemistry, 2006, 281(43):32516-32525.
[7] ROSSOCHA M, SCHULTZ-HEIENBROK R, VON MOELLER H, et al.Conjugated bile acid hydrolase is a tetrameric N-terminal thiol hydrolase with specific recognition of its cholyl but not of its tauryl product[J].Biochemistry, 2005, 44(15):5739-5748.
[8] CHAND D, PANIGRAHI P, VARSHNEY N, et al.Structure and function of a highly active Bile Salt Hydrolase (BSH) from Enterococcus faecalis and post-translational processing of BSH enzymes[J].Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2018, 1866(4):507-518.
[9] AVINASH V S, PANIGRAHI P, CHAND D, et al.Structural analysis of a penicillin V acylase from Pectobacterium atrosepticum confirms the importance of two Trp residues for activity and specificity[J].Journal of Structural Biology, 2016, 193(2):85-94.
[10] 姜金康. 乳杆菌属胆汁盐水解酶的活性多样性、底物特异性及相关基因研究[D].上海:上海交通大学, 2008.JIANG J K.Diversity of BSH activities in different Lactobacilli towards human bile salts[D].Shanghai:Shanghai Jiaotong University, 2008.
[11] CHAE J P, VALERIANO V D, KIM G B, et al.Molecular cloning, characterization and comparison of bile salt hydrolases from Lactobacillus johnsonii PF01[J].Journal of Applied Microbiology, 2013, 114(1):121-133.
[12] GARC A-RUIZ A, GONZ
A-RUIZ A, GONZ LEZ DE LLANO D, ESTEBAN-FERN
LEZ DE LLANO D, ESTEBAN-FERN NDEZ A, et al.Assessment of probiotic properties in lactic acid bacteria isolated from wine[J].Food Microbiology, 2014, 44:220-225.
NDEZ A, et al.Assessment of probiotic properties in lactic acid bacteria isolated from wine[J].Food Microbiology, 2014, 44:220-225.
[13] 陈仪婷, 张红星, 谢远红, 等.降胆固醇乳酸菌的筛选鉴定及其耐酸耐胆盐性能研究[J].食品与发酵工业, 2018, 44(5):29-33.CHEN Y T, ZHANG H X, XIE Y H, et al.Selection of cholesterol-lowering lactic acid bacteria in vitro and study on it’s tolerance of acid and bile salts[J].Food and Fermentation Industries, 2018, 44(5):29-33.
[14] 任全路. Ligilactobacillus sp.BD7642关键胆盐水解酶的发掘[J].食品与发酵工业, 2024, 50(6):72-77.REN Q L.Discovery of core bile salt hydrolase in Ligilactobacillus sp.BD7642[J].Food and Fermentation Industries, 2024, 50(6):72-77.
[15] XU F Z, GUO F F, HU X J, et al.Crystal structure of bile salt hydrolase from Lactobacillus salivarius[J].Acta Crystallographica Section F, 2016, 72(5):376-381.
[16] ROHAWI N S, RAMASAMY K, AGATONOVIC-KUSTRIN S, et al.A new high-performance thin-layer chromatographic method for determining bile salt hydrolase activity[J].Journal of Chromatography B, 2018, 1092:145-151.
[17] ÖZTÜRK M, ÖNAL C.Asparagine 79 is an important amino acid for catalytic activity and substrate specificity of bile salt hydrolase (BSH)[J].Molecular Biology Reports, 2019, 46(4):4361-4368.
[18] OINONEN C, ROUVINEN J.Structural comparison of ntn-hydrolases[J].Protein Science, 2000, 9(12):2329-2337.
[19] BRANNIGAN J A, DODSON G, DUGGLEBY H J, et al.A protein catalytic framework with an N-terminal nucleophile is capable of self-activation[J].Nature, 1995, 378(6555):416-419.
[20] DALY J W, KEELY S J, GAHAN C G M.Functional and phylogenetic diversity of BSH and PVA enzymes[J].Microorganisms, 2021, 9(4):732.
[21] 汤慧勤, 姚晓敏, 徐一涵, 等.产胆盐水解酶植物乳杆菌降胆固醇作用的研究[J].上海交通大学学报(农业科学版), 2018, 36(4):35-40.TANG H Q, YAO X M, XU Y H, et al.Study on cholesterol-lowering effect of bile salt hydrolase-active Lactobacillus plantarum[J].Journal of Shanghai Jiao Tong University (Agricultural Science), 2018, 36(4):35-40.
[22] HA C, CHO J, CHAI Y, et al.Purification and characterization of bile salt hydrolase from Lactobacillus plantarum CK 102[J].Journal of Microbiology and Biotechnology, 2006, 16(7):1047-1052.
[23] XU F Z, HU X J, SINGH W, et al.The complex structure of bile salt hydrolase from Lactobacillus salivarius reveals the structural basis of substrate specificity[J].Scientific Reports, 2019, 9(1):12438.