近年来,由于多重耐药病原体引发的传染病爆发频率不断上升,抗生素耐药性问题已成为全球公共卫生领域的重大关注点[1]。预计到2050年,抗生素耐药性问题将导致每年约1 000万人死亡,成为全球主要的死亡原因[2]。抗生素在临床环境中的不当使用是抗菌耐药危机加剧的主要因素之一,这进一步加剧了多重耐药性感染的传播[3]。对于这些多重耐药性病原体引起的感染,往往需要使用具有一定肾毒性和神经毒性的多粘菌素类药物进行治疗[4],增加此类抗生素的使用不仅会使机体承受沉重的负担,更会使耐药性危机加剧。目前亟需找到一种具有独特作用机制且抗耐药性较强的新型治疗方法来应对抗生素耐药威胁。
抗菌肽(antimicrobial peptides, AMPs)是一个有力的替抗候选者,它是天然防御系统的重要组成部分,通常由10~50个氨基酸组成,具有广谱的抗菌活性,广泛分布于细菌、植物和动物中[5-6]。它能够调节先天免疫系统中的关键免疫机制来达到保护宿主的作用,更能对病原体进行直接杀伤[7]。目前绝大部分的抗菌肽的抗菌机制是膜破坏机制,带正电荷的抗菌肽在静电力的作用下与带负电荷的微生物膜相结合,导致孔的形成或膜的不稳定化,致使细胞内容物泄露、细胞死亡[8-9]。这种非靶点式的作用机制是其不易产生耐药性的原因(图1)。
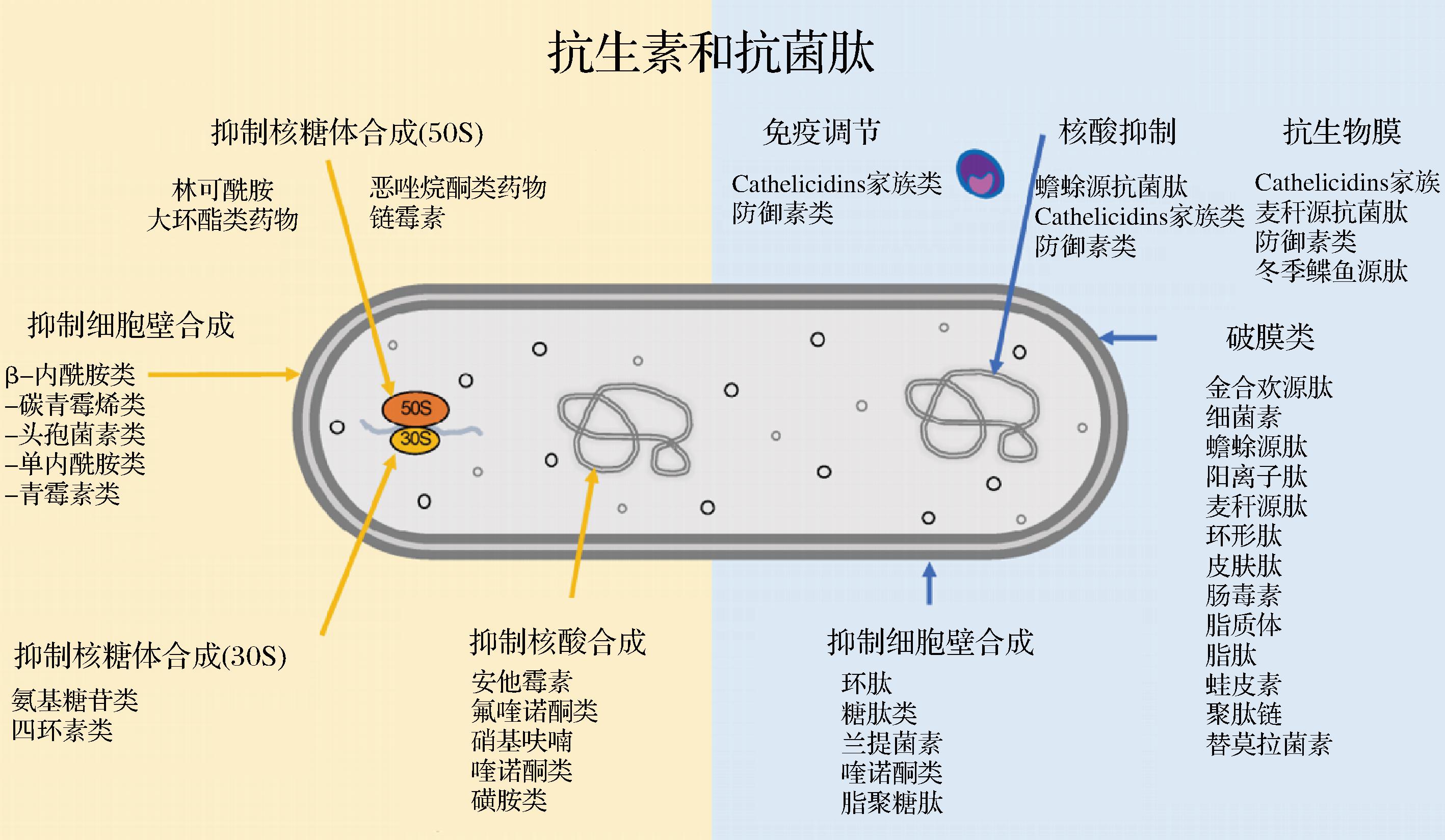
图1 各类抗生素和抗菌肽的作用机制[7]
Fig.1 Mechanisms of action of various antibiotics and antimicrobial peptides[7]
尽管目前已有部分抗菌肽进入临床或应用试验阶段,但数量仍然有限。在医学领域,目前已经有抗菌肽通过了临床试验,比如抗菌肽pexiganan,它是从非洲爪蟾中提取的,对革兰氏阴性菌、阳性菌以及真菌都具有生物活性,对几种多重耐药微生物也有足够的生物效力。在2项治疗糖尿病足溃疡轻度感染的Ⅲ期临床试验(NCT01590758、NCT01594762)中,pexiganan已经被评估为外用乳膏类药物[10-11]。在农业中,抗菌肽被视为一种环保的有害微生物控制方法,为化学农药提供了替代选择。可以帮助作物防御细菌和真菌病原体,并且不会伤害有益微生物[12]。此外,在食品行业中,抗菌肽被视为一种天然防腐剂,研究表明它们可抑制食品中腐败菌和病原体生长[13-14]。然而,由于大多数抗菌肽存在稳定性差、活性低及合成成本高等问题,其市场化仍面临严峻挑战。为了让抗菌肽能够规模应用,需要对其进行改造优化来弥补其存在的短板。
结构决定功能,分析利用抗菌肽的构效关系是抗菌肽改造的核心策略。抗菌肽普遍具有两亲性结构,使得它们能够与富含阴离子脂质的微生物膜相互作用并渗透。两亲性对于其区分宿主细胞与病原体至关重要[15]。疏水性及螺旋性的提高有助于增强抗菌肽的细胞渗透性,从而提高其活性[16]。抗菌肽的作用模式可能会因其结构、电荷以及靶标微生物的特性而有所不同[17]。本文系统梳理了当前抗菌肽的改造策略,针对其稳定性差、活性不足、合成成本高等关键短板,归纳了相应的优化途径。与此同时,针对人工设计改造抗菌肽存在的成本高、效率低问题,引入深度学习模型作为辅助工具,以提升抗菌肽的智能化设计效率与精度。通过归纳总结现有的抗菌肽改造策略对抗菌肽整体研发进展具有重要意义。
1 人工设计的抗菌肽改造策略
1.1 抗菌肽稳定性提升策略
抗菌肽具有作为新型治疗药物的巨大潜力,但其在临床应用中仍面临着许多挑战,其中一个主要问题是其稳定性差,极易被蛋白酶降解,这限制了其半衰期和治疗效果。另外,低pH、高离子强度以及血液环境也会对其生物活性造成负面影响。为解决这个问题,人们探索了许多方法来提高抗菌肽的稳定性,常见策略包括D型氨基酸或非天然氨基酸的替换、肽模拟物、N端乙酰化、C端乙酰化、环化、聚乙二醇化、氟化、脯氨酸的引入等等[18-25]。这些都是基于对抗菌肽序列的改造或是添加修饰基团来提高抗菌肽的稳定性。然而,部分策略可能会影响其原本的生物效能,例如KLUBTHAWEE等[26]设计了一条完全由D型氨基酸组成的抗菌肽,显著增强了抗酶解能力,但其抗菌活性明显下降。随后,他们采用壳聚糖与硫酸葡聚糖复合形成的纳米纤维网络对原肽进行包埋,成功在不影响抗菌活性的前提下保护抗菌肽免受胰蛋白酶降解。
1.2 抗菌肽靶向性提升策略
由于抗菌肽大多具有广谱的抗菌活性,其在体内发挥作用极大可能会误伤有益菌或正常组织细胞。为了解决这一问题,人们将抗菌肽与各种靶向域相结合,得到各种靶向肽。例如将物种特异性信息素cCF10与广谱抗菌肽C4相链接并修饰抗菌肽活性区域,得到了靶向粪肠球菌的抗菌肽cCF10-C4,其仅对粪肠球菌有高效的杀菌作用[27];利用噬菌体展示技术,在巨量的抗体文库中筛选出对金黄色葡萄球菌特异性最强的序列,合成出其对应的肽序列后,利用3个甘氨酸将该序列与抗菌肽库中的一条广谱肽相链接,并进行尾端修饰,提高电荷数从而加强整体的抗菌活性,得到了靶向金黄色葡萄球菌的抗菌肽[28]。上述靶向策略的核心均是导入一个能够与目标特异性结合的靶向域,将广谱抗菌肽改造成靶向某一类菌的窄谱抗菌肽,显著提高抗菌肽的选择性。
1.3 抗菌肽活性提升策略
大部分抗菌肽都是动植物体合成的天然产物,其生物活性远不如商业抗生素,为了提高其活性,YANG等[29]从猪髓细胞抗菌肽-36的α-螺旋区截断出模板肽KV,之后对KV进行一系列加长和残基取代,引入芳香疏水氨基酸(色氨酸和苯丙氨酸)增强疏水性,显著提高肽对白色念珠菌(包括氟康唑耐药菌株)的活性。SONG等[30]利用单一疏水残基进行末端修饰,并系统探讨该手段对抗菌肽W4生物活性的影响,证明此方法可以提高肽的活性与细胞选择性。LIU等[31]合成了一系列含有简单重复序列的肽(RW)n- NH2(其中n等于1、2、3、4或5),并测定它们的抗菌和溶血活性,证明选择合适的肽链长度有助于提高抗菌肽的抗菌活性。WANG等[28]在尾端添加了带有正电荷的氨基酸(如赖氨酸、精氨酸),通过提高电荷数来提高抗菌活性。提高抗菌肽活性的策略主要通过增强疏水性和正电荷密度,促进肽与细菌细胞膜的相互作用及破膜能力,从而提升其抗菌效能。
1.4 抗菌肽合成成本的降低策略
抗菌肽的规模化生产也是一个巨大挑战,大规模合成抗菌肽的成本和复杂性限制了其商业化进程。近年来,利用细菌或酵母等微生物的重组表达系统实现抗菌肽的高效生产,为规模化合成提供了新的技术途径[32];无细胞生物合成技术的出现也显著降低抗菌肽在筛选优化过程中的合成成本[33]。此外,通过截短抗菌肽长度,只保留其核心序列也能够降低抗菌肽合成成本与难度,例如arasin-1是从蜘蛛蟹中分离出来的富含37个氨基酸的抗菌肽,其核心序列位于肽的富含脯氨酸/精氨酸的NH2端,片段arasin-1(1-23)几乎与全长肽具有相同的活性[34]。
1.5 人工设计抗菌肽的弊端
还有许多不定向的改造策略,例如,使用丙氨酸扫描对抗菌肽核心序列进行多个位点的丙氨酸替换突变,再通过大批量肽的试验筛选,挖掘出抗菌肽的关键突变位点,最后,点对点地进行氨基酸替换,从而达到改良抗菌肽的目的[35]。上述方法均需要反复合成抗菌肽进行试验测试,这样通过人工设计改良抗菌肽,迭代慢、验证成本高,即使对抗菌肽的各项参数之间的关系有较为深入的理解,也很难一次性设计出符合期望的抗菌肽。深度学习的引入能够弥补这一弊端,深度学习模型能够快速预测肽序列的抗菌活性和选择性,并优化设计方案,从而显著提高研发效率,降低验证成本,为研发新型抗菌肽提供了全新的思路。
2 深度学习概述
2.1 深度学习的基本概念
深度学习是机器学习的一个子集,广泛应用于图像识别、语音处理和自然语言理解等领域,彻底改变了人工智能的发展。人工神经网络是构建深度学习模型的核心,这些神经网络由多个相互连接的节点组成,从而模仿人类大脑的架构。深度学习的主要构建模块包括前馈神经网络和卷积神经网络,其中卷积神经网络在处理图像数据时尤其有效[36]。深度学习主要依赖反向传播算法,通过梯度下降不断调整权重以最小化预测误差,从而优化模型性能[37]。由于训练深度模型通常需要处理庞大的数据量和复杂的参数计算,因此对计算资源的需求极高。图形处理单元的并行计算能力显著提升了模型训练的效率,极大推动了深度学习的快速发展[38]。
深度学习最具潜力的特点之一是其能够从数据中自动学习。传统的机器学习模型通常需要从原始数据中手动提取特征,而深度学习模型能够自动学习层次化的特征表示,从而减少对领域特定知识的需求。然而,深度学习也面临着过拟合等挑战。过拟合是指模型在训练过程中对训练数据的噪声和细节进行了过度学习,导致模型不仅捕捉到了数据中的潜在规律,还将数据中的随机波动或异常值作为规律进行学习。这种现象使得模型在训练数据上表现得非常好,但在面对新的、未见过的数据时,模型的泛化能力显著下降,从而影响其预测准确性。正则化技术,如丢弃法和提前停止法,都可以有效缓解过拟合问题[39]。此外,对于具有多层的深度学习模型,由于缺乏透明性,它们常被视为“黑箱”,很难理解它们如何得出具体的决策。这一现象推动了更多关于可解释人工智能模型的研究,其目的在于深入探索模型决策背后的逻辑和原理。
2.2 深度学习中的常见模型及其功能
深度学习有多种模型架构,每种架构都是为了特定类型的任务而设计的。最常用的模型之一是卷积神经网络(convolutional neural networks, CNN),它主要用于图像处理任务,如图像分类、目标检测和分割[40]。另一种广泛使用的模型是循环神经网络(recurrent neural network, RNN),它特别适用于处理序列数据,如时间序列预测、自然语言处理和语音识别[41]。对于涉及序列数据和长期依赖关系的复杂任务,长短时记忆网络(long short-term memory, LSTM)通常更为优选。LSTM是RNN的一种变种,专门设计用于解决梯度消失等问题。它通过引入记忆单元来存储信息,并能够在较长时间内有效地检索这些信息,因此,非常适合用于机器翻译和语音转文本等任务[42]。生成对抗网络(generative adversarial network, GAN)是另一种强大模型,尤其适用于图像和视频生成等任务。GAN主要由2部分组成——生成器和判别器,它们相互对抗训练,从而使生成器能够从随机噪声中创造出逼真的数据[43]。最近Transformer模型在自然语言处理中获得了广泛关注,如BERT(bidirectional encoder representations from transformers)和GPT(generative pre-trained transformer)等模型,这类模型使用自注意力机制来并行处理输入数据,而不是像传统模型那样按顺序处理,这使得它们在涉及大数据集的任务中(如文本生成、翻译和摘要)更加高效[44]。
3 深度学习在抗菌肽研究中的应用
3.1 抗菌肽功能的预测与优化
抗菌肽预测是指使用计算算法和深度学习技术,分析氨基酸序列的特征和结构,以预测肽段的功能性。预测抗菌肽的功能是一个重要且具有挑战性的任务,特别是在预测抗菌肽多重功能时,其复杂性会显著增加。抗菌肽的功能预测通常遵循以下流程(图2):首先,从各大数据库中收集已知的抗菌肽资源;其次,提取其结构特征,如疏水性、电荷数、分子质量等;最后,通过降维技术将这些特征转化为特征向量,并将其输入到不同的深度学习模型中进行训练。经过训练的模型能够对未知肽序列进行功能预测,从而实现对抗菌肽序列功能的预测与评估。
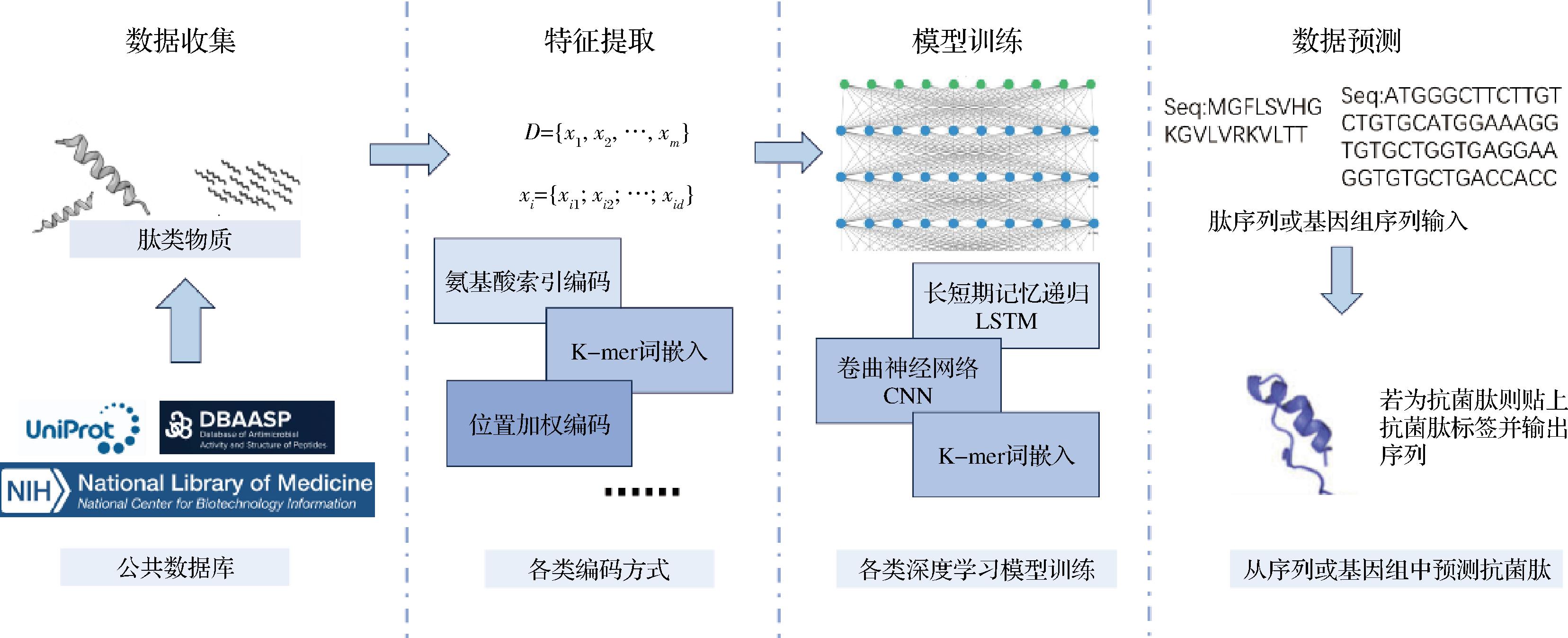
图2 深度学习预测抗菌肽的工作流程
Fig.2 Deep learning workflow for predicting antimicrobial peptides
ZHAO等[45]基于CNN-Attention_BiLSTM结构以及迁移学习方法,建立了一个名为discoverAMP(dsAMP)的抗菌肽预测模型,其在敏感性、特异性、马修相关系数、准确度、精密度、F1评分等多个指标中表现优异,展现出强大的预测准确性。GUO等[46]建立了基于Wassersreub AutoEncoder(WAE)构建了名为conotoxin prediction model(ConoPred)的模型,区别于ZHAO等[45]的方法以及训练数据集,GUO等[46]建立的是一个用来预测芋螺毒素的深度学习模型,可以专门预测与芋螺毒素相似度高的抗菌肽,在他们的研究中,这项模型主要是为他们的芋螺毒素生成模型所服务,用来减少抗菌肽筛选工作量。深度学习模型并不是只能利用肽序列进行预测,SANTOS-J NIOR等[47]运用了他们在2020年所构建的模型——Macrel,在2024年预测了全球微生物组中的抗菌肽,并利用来自环境和宿主相关的63 410个宏基因组和87 920个原核生物基因组的庞大数据集来预测抗菌肽,创建了名为“AMPSphere”的全新抗菌肽数据库,并合成了100条抗菌肽来验证其模型的准确性,共有79条肽具有生物活性,其中63条能够靶向病原体,展示出了Macrel模型的可靠性[48]。SHEN等[49]开发了一个深度学习模型,从反刍动物胃肠道的120个宏基因组和10 373个宏基因组组装基因组的数据集中识别可能的抗菌肽,之后通过分子动力学模拟及体内外试验对预测结果进行了验证,这种方法可以快速准确地鉴定反刍动物胃肠道来源的抗菌肽,从而有效地探索反刍动物细菌资源,促进下一代抗菌药物开发。SALEM等[50]结合迁移学习构建了AMPDeep管道,解决了训练数据不足的问题,成功利用深度学习模型对抗菌肽是否具有溶血活性进行了分类。由上可见,深度学习在抗菌肽预测方面的应用主要是基因组中抗菌肽数据的挖掘、减少抗菌肽筛选过程的成本以及对抗菌肽生成模型进行校验。表1为上述研究所涉及的模型信息汇总。
NIOR等[47]运用了他们在2020年所构建的模型——Macrel,在2024年预测了全球微生物组中的抗菌肽,并利用来自环境和宿主相关的63 410个宏基因组和87 920个原核生物基因组的庞大数据集来预测抗菌肽,创建了名为“AMPSphere”的全新抗菌肽数据库,并合成了100条抗菌肽来验证其模型的准确性,共有79条肽具有生物活性,其中63条能够靶向病原体,展示出了Macrel模型的可靠性[48]。SHEN等[49]开发了一个深度学习模型,从反刍动物胃肠道的120个宏基因组和10 373个宏基因组组装基因组的数据集中识别可能的抗菌肽,之后通过分子动力学模拟及体内外试验对预测结果进行了验证,这种方法可以快速准确地鉴定反刍动物胃肠道来源的抗菌肽,从而有效地探索反刍动物细菌资源,促进下一代抗菌药物开发。SALEM等[50]结合迁移学习构建了AMPDeep管道,解决了训练数据不足的问题,成功利用深度学习模型对抗菌肽是否具有溶血活性进行了分类。由上可见,深度学习在抗菌肽预测方面的应用主要是基因组中抗菌肽数据的挖掘、减少抗菌肽筛选过程的成本以及对抗菌肽生成模型进行校验。表1为上述研究所涉及的模型信息汇总。
表1 预测型抗菌肽深度学习模型
Table 1 Deep learning modeling of predictive antimicrobial peptides

名称主要使用模型文献成果年份dsAMP[45]CNN-Attention-BiLSTM在多个评估指标上均优于现有的分类器,准确性高2024ConoPred[46]WAE能够准确预测芋螺毒素2024Macrel[47]支持向量机(SVM)能从基因组序列中来预测抗菌肽,并标注其抗菌及溶血可能性2020Transformer and BERT[49]Hugging Face Transformers API和ProtBERT-BFDa有效挖掘了反刍动物胃肠道抗菌肽资源2025AMPDeep [50]Prot-BERT-BFD利用迁移学习克服小数据困难,预测了抗菌肽溶血活性2022
注:部分文章并未对其所构建的模型进行命名,未命名的模型名称则用其主要使用模型名称代替;Prot-BERT-BFD:protein bidirectional encoder representations from transformers trained on the big fantastic database。
3.2 新型抗菌肽的设计与生成
与预测抗菌肽不同,利用深度学习模型设计出新的抗菌肽是一个创造的过程,其基本原理是首先利用已有的抗菌肽库对模型进行预训练,随后可以通过迁移学习,利用特定的肽库对预训练模型进行微调,以便在特定的设计目标下生成具有所需特性的抗菌肽。这一方法不仅能够加速新抗菌肽的发现,还能够精确地引导生成过程,确保所设计的肽分子满足特定的功能需求。生成式学习模型主要依赖于变分自编码器(variational autoencoder,VAE)和GAN等深度学习架构。以VAE模型为例,其在抗菌肽生成方面的工作原理如图3所示,将用于训练的抗菌肽资源输入编码器,训练构成一个抗菌肽类潜在空间,之后在构成的潜在空间里随机取样,并通过解码器进行解码重构,得到新设计的抗菌肽。GAN的原理则是受到博弈论启发,假设一个两“人”(生成器和鉴别器)博弈的情况,即生成器不断优化生成数据,鉴别器也需要不断优化从而准确地区分生成器生成的数据与真实数据,两者形成对抗性博弈,最终达到平衡,鉴别器将无法区分真假数据,这也意味着生成器已经捕获了真实数据的分布,达到生成真实数据的目标(图4)。
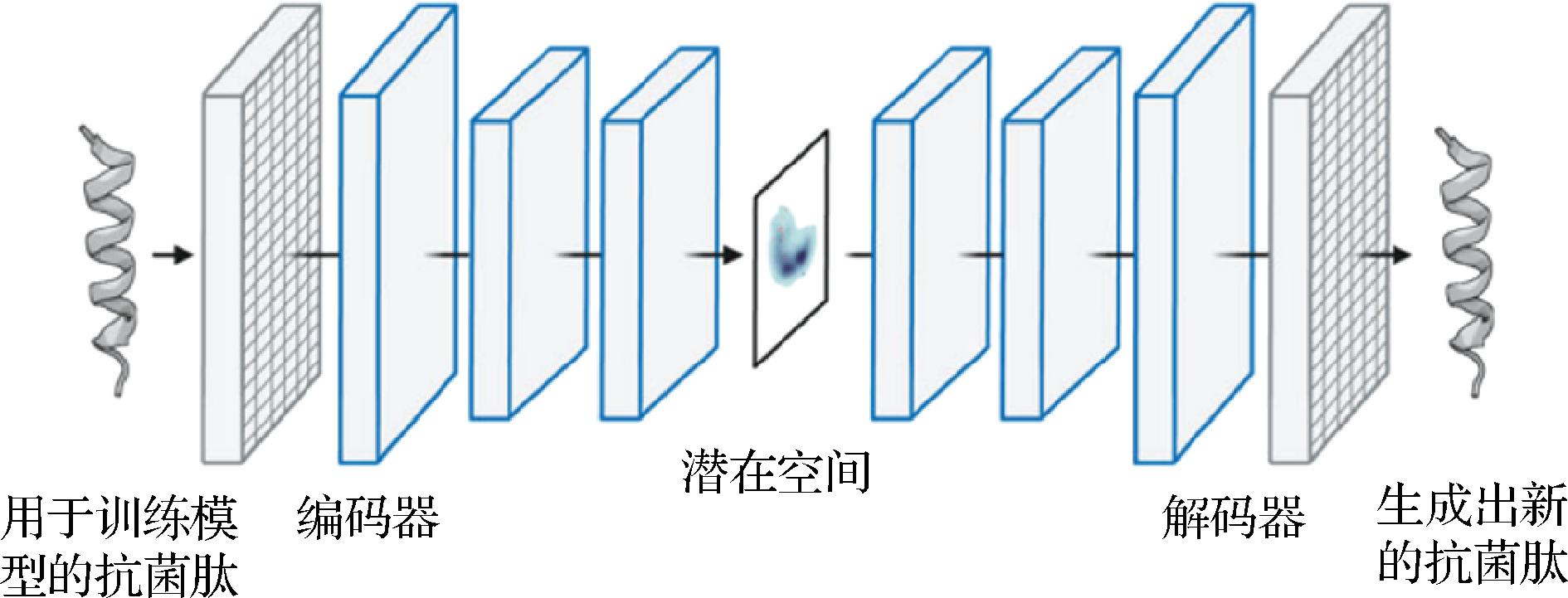
图3 VAE模型生成抗菌肽的工作流程图[33]
Fig.3 Workflow diagram for the generation of antimicrobial peptides from VAE models[33]
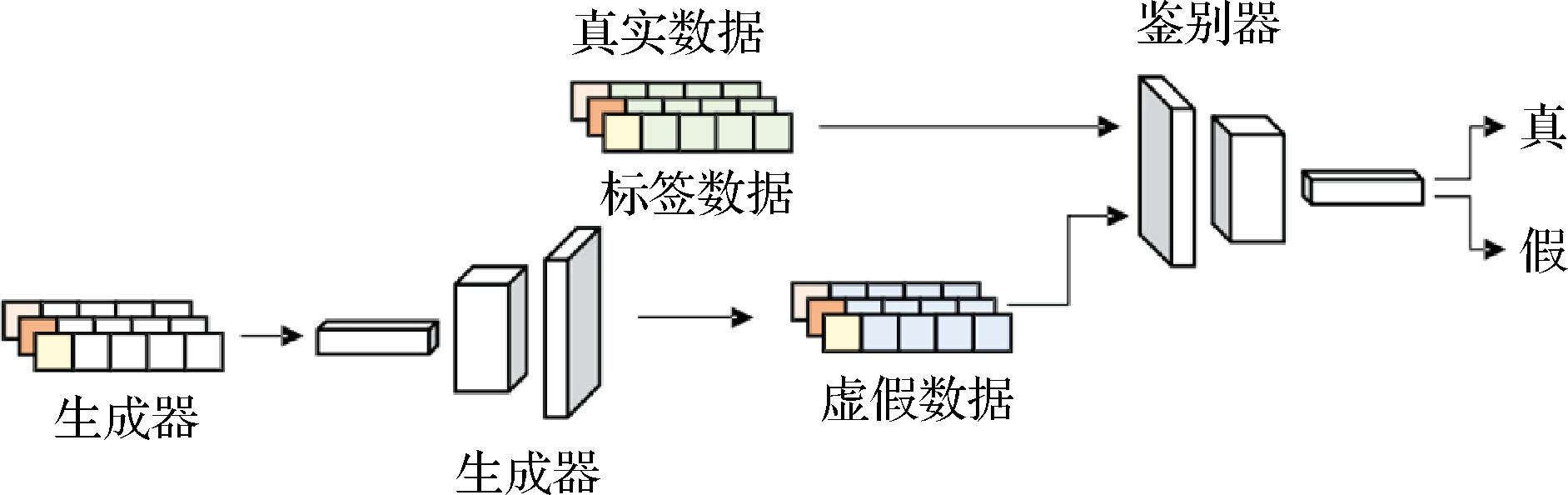
图4 GAN模型工作原理示意图[51]
Fig.4 Schematic diagram of the GAN model workflow[51]
PANDI等[33]使用了VAE生成式模型,并构建了相应的最小抑菌浓度(minimum inhibitory concentration,MIC)预测模型,将2个模型相结合,按照MIC顺序筛选出了500条新生成的优先候选肽,之后结合了无细胞蛋白质合成技术进行进一步的筛选,最终筛选出了6条可抵御多重耐药病原菌的广谱肽。GUO等[46]为了高效开发芋螺毒素建立了一个预测模型以验证其构建的芋螺毒素生成模型(conotoxin generation model,ConoGen),该生成模型的构造采用了迁移学习方法以及大语言模型,最后通过ConoPred去检验ConoGen产生的人工芋螺毒素,从而大幅加速了人工芋螺毒的研发进程。ZHAO等[45]则是基于GAN构建了名为dsAMPGAN的抗菌肽生成模型,配套预测模型形成一体化的AMP生成与功能预测框架。CAPECCHI等[52]使用DBAASP(database of antimicrobial activity and structure of peptides)数据库的数据训练递归神经网络来设计短的非溶血性抗菌肽,合成并测试了28个生成的肽,每个肽与训练数据至少有5个突变,最后鉴定出8个抗铜绿假单胞菌、鲍曼不动杆菌和耐甲氧西林金黄色葡萄球菌的全新非溶血性抗菌肽。YU等[51]利用二级结构、抗菌性能、毒性这三种类别的抗菌肽数据库训练了基于GAN构建的Multi-CGAN模型。该模型能通过学习多种单属性肽数据,生成多功能抗菌肽序列,并且通过后续的实验验证,证实新生肽的多功能化,且与训练数据同源的概率很低。DAS等[53]采用了WAE模型进行肽序列的训练。其生成的序列不仅具有较高的重建精度,还表现出较强的多样性。该研究还结合了通过高通量分子动力学模拟获得的物理化学特征来筛选生成的分子。在48 d内,成功鉴定并合成了20条候选肽,并通过体外实验和小鼠实验验证了其良好的生物活性。SZYMCZAK等[54]使用条件变分自编码器(conditional variational autoencoder,CVAE)建立了HydrAMP模型,并且通过试验验证了它的准确性。该模型具有两个功能:生成具有特定抗菌特性的现有肽的类似物和无约束式生成新型抗菌肽。综合以上案例可以看出,使用生成式深度学习模型用来构建抗菌肽是一条可行的道路,搭配预测类模型或者其他预测手段,将显著加速功能优化过程。表2为上述研究所涉及的模型信息汇总。
表2 生成式抗菌肽深度学习模型
Table 2 Deep learning modeling of generative antimicrobial peptides

名称主要使用模型文献成果年份ConoGen[46]Transformer生成了新型半胱氨酸支架的人工芋螺素2024VAE[33]VAE使用VAE生成模型结合MIC预测及无细胞蛋白质合成技术高通量筛选抗菌肽2023dsAMPGAN[45]GAN可以生成广泛新型抗菌肽2024ML[52]RNN设计并筛选出了8个新的抗3种致病菌的非溶血抗菌肽2021Multi-CGAN[51]GAN读取多个单功能肽库,来形成多功能新型肽2023WAE[53]WAE相较于VAE模型性能更加的优秀2021HydrAMP[54]CVAE无需额外手段预选,成功率极高2022
注:部分文章并未对其所构建的模型进行命名,未命名的模型名称则用其主要使用模型名称代替;ML:machine learning;Multi-CGAN:multi-conditional generative adversarial network。
4 结论与未来展望
本文总结了现阶段抗菌肽研究中所存在的问题和相对应的抗菌肽改造策略,指出可以用包被技术或各种序列修饰来提高抗菌肽的稳定性;构造或者寻找相对应的靶向域与抗菌肽相结合来提升抗菌肽的选择性;探究抗菌肽构效关系来建立高效抗菌肽模板;使用毕赤酵母表达系统、缩短肽链等方式来降低抗菌肽的合成成本。在抗菌肽改造领域引入深度学习技术可以大大缩短抗菌肽改造的迭代时间,能够显著减少实验验证成本,弥补了人工设计的弊端。尽管近年来深度学习在抗菌肽改造方面取得了显著进展,但仍存在着一些局限性和挑战,如数据不足、模型泛化能力有限等问题。因此,需要进一步引入更多先进的深度学习技术和模型。未来的研究应致力于跨学科融合,提升抗菌肽改造的效率,克服现有局限,推动抗菌肽在实践应用中的转化。
[1] WENP C, VANEGAS J M, REMPE S B, et al.Probing key elements of teixobactin-lipid Ⅱ interactions in membranes[J].Chemical Science, 2018, 9(34):6997-7008.
[2] DE KRAKER M E A, STEWARDSON A J, HARBARTH S.Will 10 million people die a year due to antimicrobial resistance by 2050?[J].PLoS Medicine, 2016, 13(11):e1002184.
[3] YEWALE V N.Antimicrobial resistance:A ticking bomb![J].Indian Pediatrics, 2014, 51(3):171-172.
[4] ABDELRAOUF K, BRAGGS K H, YIN T J, et al.Characterization of polymyxin B-induced nephrotoxicity:Implications for dosing regimen design[J].Antimicrobial Agents and Chemotherapy, 2012, 56(9):4625-4629.
[5] BROGDEN K A.Antimicrobial peptides:Pore formers or metabolic inhibitors in bacteria?[J].Nature Reviews.Microbiology, 2005, 3(3):238-250.
[6] BOULANGER N, BULET P, LOWENBERGER C.Antimicrobial peptides in the interactions between insects and flagellate parasites[J].Trends in Parasitology, 2006, 22(6):262-268.
[7] BROWNE K, CHAKRABORTY S, CHEN R X, et al.A new era of antibiotics:The clinical potential of antimicrobial peptides[J].International Journal of Molecular Sciences, 2020, 21(19):7047.
[8] LI Y M, XIANG Q, ZHANG Q H, et al.Overview on the recent study of antimicrobial peptides:Origins, functions, relative mechanisms and application[J].Peptides, 2012, 37(2):207-215.
[9] AHMAD A, AHMAD E, RABBANI G, et al.Identification and design of antimicrobial peptides for therapeutic applications[J].Current Protein &Peptide Science, 2012, 13(3):211-223.
[10] GREBERK E, DAWGUL M.Antimicrobial peptides under clinical trials[J].Current Topics in Medicinal Chemistry, 2017, 17(5):620-628.
[11] SIERRA J M, FUSTÉ E, RABANAL F, et al.An overview of antimicrobial peptides and the latest advances in their development[J].Expert Opinion on Biological Therapy, 2017, 17(6):663-676.
[12] TANG R, TAN H, DAI Y, et al.Application of antimicrobial peptides in plant protection:Making use of the overlooked merits[J].Frontiers in Plant Science, 2023, 14:1139539.
[13] 贺松, 龚芳红, 张德纯, 等.乳酸链球菌素对乳酸菌抑菌作用的研究[J].食品科学, 2009, 30(23):352-355.HE S, GONG F H, ZHANG D C, et al.Antimicrobial activity of nisin against lactic acid bacteria[J].Food Science, 2009, 30(23):352-355.
[14] YANG Z Y, WEI Y X, WU W P, et al.Characterization of simplified nonapeptides with broad-spectrum antimicrobial activities as potential food preservatives, and their antibacterial mechanism[J].Food &Function, 2023, 14(7):3139-3154.
[15] ZASLOFF M.Antimicrobial peptides of multicellular organisms[J].Nature, 2002, 415(6870):389-395.
[16] OBA M, NAKAJIMA S, MISAO K, et al.Effect of helicity and hydrophobicity on cell-penetrating ability of arginine-rich peptides[J].Bioorganic &Medicinal Chemistry, 2023, 91:117409.
[17] DASHPER S G, LIU S W, REYNOLDS E C.Antimicrobial peptides and their potential as oral therapeutic agents[J].International Journal of Peptide Research and Therapeutics, 2007, 13(4):505-516.
[18] LU J G, XU H J, XIA J H, et al.D- and unnatural amino acid substituted antimicrobial peptides with improved proteolytic resistance and their proteolytic degradation characteristics[J].Frontiers in Microbiology, 2020, 11:563030.
[19] WANG J J, SONG J, YANG Z Y, et al.Antimicrobial peptides with high proteolytic resistance for combating gram-negative bacteria[J].Journal of Medicinal Chemistry, 2019, 62(5):2286-2304.
[20] ZHU Y J, SHAO C X, LI G Y, et al.Rational avoidance of protease cleavage sites and symmetrical end-tagging significantly enhances the stability and therapeutic potential of antimicrobial peptides[J].Journal of Medicinal Chemistry, 2020, 63(17):9421-9435.
[21] YU W K, WANG J J, WANG Z H, et al.PEGylation of the antimicrobial peptide PG-1:A link between propensity for nanostructuring and capacity of the antitrypsin hydrolytic ability[J].Journal of Medicinal Chemistry, 2021, 64(14):10469-10481.
[22] WANG C S, SHAO C X, FANG Y X, et al.Binding loop of sunflower trypsin inhibitor 1 serves as a design motif for proteolysis-resistant antimicrobial peptides[J].Acta Biomaterialia, 2021, 124:254-269.
[23] LAI Z H, TAN P, ZHU Y J, et al.Highly stabilized α-helical coiled coils kill gram-negative bacteria by multicomplementary mechanisms under acidic condition[J].ACS Applied Materials &Interfaces, 2019, 11(25):22113-22128.
[24] LAI Z H, YUAN X J, CHEN H Y, et al.Strategies employed in the design of antimicrobial peptides with enhanced proteolytic stability[J].Biotechnology Advances, 2022, 59:107962.
[25] YU W K, SUN Y, LI W Y, et al.Self-assembly of antimicrobial peptide-based micelles breaks the limitation of trypsin[J].ACS Applied Materials &Interfaces, 2023, 15(1):494-510.
[26] KLUBTHAWEE N, BOVONE G, MARCO-DUFORT B, et al.Biopolymer nano-network for antimicrobial peptide protection and local delivery[J].Advanced Healthcare Materials, 2022, 11(7):2101426.
[27] XU L, SHAO C X, LI G Y, et al.Conversion of broad-spectrum antimicrobial peptides into species-specific antimicrobials capable of precisely targeting pathogenic bacteria[J].Scientific Reports, 2020, 10(1):944.
[28] WANG T, TAN P, TANG Q, et al.Phage-displayed heptapeptide sequence conjugation significantly improves the specific targeting ability of antimicrobial peptides against Staphylococcus aureus[J].mLife, 2024, 3(2):251-268.
[29] YANG Y, WANG C X, GAO N, et al.A novel dual-targeted α-helical peptide with potent antifungal activity against fluconazole-resistant Candida albicans clinical isolates[J].Frontiers in Microbiology, 2020, 11:548620.
[30] SONG J, WANG J J, ZHAN N, et al.Therapeutic potential of trp-rich engineered amphiphiles by single hydrophobic amino acid end-tagging[J].ACS Applied Materials &Interfaces, 2019, 11(47):43820-43834.
[31] LIU Z G, BRADY A, YOUNG A, et al.Length effects in antimicrobial peptides of the (RW)n series[J].Antimicrobial Agents and Chemotherapy, 2007, 51(2):597-603.
[32] 查曼, 闵勇, 刘晓艳, 等.抗菌肽在毕赤酵母中表达的研究进展[J/OL].食品与发酵工业, 2024:1-9.(2024-11-29).https://link.cnki.net/doi/10.13995/j.cnki.11-1802/ts.041234.ZHA M, MIN Y, LIU X Y, et al.Research progress on expression of antimicrobial peptides in Pichia pastoris[J/OL].Food and Fermentation Industries, 2024:1-9.(2024-11-29).https://link.cnki.net/doi/10.13995/j.cnki.11-1802/ts.041234.
[33] PANDI A, ADAM D, ZARE A, et al.Cell-free biosynthesis combined with deep learning accelerates de novo-development of antimicrobial peptides[J].Nature Communications, 2023, 14(1):7197.
[34] PAULSEN V S, BLENCKE H M, BENINCASA M, et al.Structure-activity relationships of the antimicrobial peptide arasin 1 - and mode of action studies of the N-terminal, proline-rich region[J].PLoS One, 2013, 8(1):e53326.
[35] WU W P, SONG J, LI T, et al.Unlocking antibacterial potential:Key-site-based regulation of antibacterial spectrum of peptides[J].Journal of Medicinal Chemistry, 2024, 67(5):4131-4149.
[36] LECUN Y, BENGIO Y, HINTON G.Deep learning[J].Nature, 2015, 521(7553):436-444.
[37] RUMELHART D E, HINTON G E, WILLIAMS R J.Learning representations by back-propagating errors[J].Nature, 1986, 323(6088):533-536.
[38] RAINA R, MADHAVAN A, NG A Y.Large-scale deep unsupervised learning using graphics processors[C].Proceedings of the 26th Annual International Conference on Machine Learning.ACM, 2009:873-880.
[39] SRIVASTAVA N, HINTON G, KRIZHEVSKY A, et al.Dropout:a simple way to prevent neural networks from overfitting[J].Journal of Machine Learning Research, 2014, 15:1929-1958.
[40] LECUN Y, BOTTOU L, BENGIO Y, et al.Gradient-based learning applied to document recognition[J].Proceedings of the IEEE, 1998, 86(11):2278-2324.
[41] ELMAN J.Finding structure in time[J].Cognitive Science, 1990, 14(2):179-211.
[42] HOCHREITER S, SCHMIDHUBER J.Long short-term memory[J].Neural Computation, 1997, 9(8):1735-1780.
[43] GOODFELLOW I J, POUGET-ABADIE J, MIRZA M, et al.Generative adversarial nets [C].Proceedings of the 28th International Conference on Neural Information Processing Systems - Volume 2.Montreal:MIT Press, 2014:2672-2680.
[44] VASWANI A, SHAZEER N, PARMAR N, et al.Attention is all you need [C].Proceedings of the 31 st International Conference on Neural Information Processing Systems.Long Beach:Curran Associates Inc, 2017:6000-6010.
[45] ZHAO M, ZHANG Y, WANG M L, et al.dsAMP and dsAMPGAN:Deep learning networks for antimicrobial peptides recognition and generation[J].Antibiotics, 2024, 13(10):948.
[46] GUO M H, LI Z P, DENG X J, et al.ConoDL:A deep learning framework for rapid generation and prediction of conotoxins[J].Journal of Computer-Aided Molecular Design, 2024, 39(1):4.
[47] SANTOS-J NIOR C D, PAN S J, ZHAO X M, et al.Macrel:Antimicrobial peptide screening in genomes and metagenomes[J].PeerJ, 2020, 8:e10555.
NIOR C D, PAN S J, ZHAO X M, et al.Macrel:Antimicrobial peptide screening in genomes and metagenomes[J].PeerJ, 2020, 8:e10555.
[48] SANTOS-J NIOR C D, TORRES M D T, DUAN Y Q, et al.Discovery of antimicrobial peptides in the global microbiome with machine learning[J].Cell, 2024, 187(14):3761-3778.
NIOR C D, TORRES M D T, DUAN Y Q, et al.Discovery of antimicrobial peptides in the global microbiome with machine learning[J].Cell, 2024, 187(14):3761-3778.
[49] SHEN H, LI Y R, PI Q J, et al.Unveiling novel antimicrobial peptides from the ruminant gastrointestinal microbiomes:A deep learning-driven approach yields an anti-MRSA candidate[J].Journal of Advanced Research, 2025.DOI:10.1016/j.jare.2025.01.005.
[50] SALEM M, KESHAVARZI ARSHADI A, YUAN J S.AMPDeep:Hemolytic activity prediction of antimicrobial peptides using transfer learning[J].BMC Bioinformatics, 2022, 23(1):389.
[51] YU H Q, WANG R H, QIAO J B, et al.Multi-CGAN:Deep generative model-based multiproperty antimicrobial peptide design[J].Journal of Chemical Information and Modeling, 2024, 64(1):316-326.
[52] CAPECCHI A, CAI X G, PERSONNE H, et al.Machine learning designs non-hemolytic antimicrobial peptides[J].Chemical Science, 2021, 12(26):9221-9232.
[53] DAS P, SERCU T, WADHAWAN K, et al.Accelerated antimicrobial discovery via deep generative models and molecular dynamics simulations[J].Nature Biomedical Engineering, 2021, 5(6):613-623.
[54] SZYMCZAK P, ![]() M, GRZEGORZEK T, et al.Discovering highly potent antimicrobial peptides with deep generative model HydrAMP[J].Nature Communications, 2023, 14(1):1453.
M, GRZEGORZEK T, et al.Discovering highly potent antimicrobial peptides with deep generative model HydrAMP[J].Nature Communications, 2023, 14(1):1453.