生长期特异性启动子对脂肪酶表达优化的研究
杜菲凡1,2,黎菁菁1,2,郑蓝昕1,2,张迪1,2,叶慧敏1,2,邓俊威1,2,秦煜南1,2,黄秋燕1,2,曹诗林1,2*,曾新安1,2*
1(佛山大学,广东省食品智能制造重点实验室,广东 佛山,528000)2(佛山大学 食品科学与工程学院,广东 佛山,528000)
摘 要 脂肪酶是一种生物催化剂,在多种生化反应中具有重要的作用。枯草芽孢杆菌脂肪酶A(Bacillus subtilis lipase A, Lip A)是目前分子质量最小的脂肪酶之一,由于其对底物的适应性强、耐碱性强、无盖子结构、不需要界面活化等优点,在食品、医药、造纸、洗涤剂等方面具有重要的应用前景。为提高LipA的发酵水平,该研究将Lip A表达于大肠杆菌(Escherichia coli)BL21(DE3)中,并构建生长期启动子ynhG的Lip A表达菌株,通过上调ynhG启动子在生长期的表达来增加Lip A的表达量。以增强型绿色荧光蛋白(enhanced green fluorescent protein, EGFP)表征,在ynhG启动子的-10区、-35区和RBS等区域进行饱和突变,以获得启动强度提高的增强型ynhG启动子的序列。根据所获得的增强型ynhG启动子序列,通过定点突变构建增强型的ynhG-Lip A-BL21(DE3)表达菌株后进行表达鉴定,结果表明,所获得的增强型ynhG启动子的酶活产量比野生型的ynhG启动子增加了31%~137%。研究成果将为提升枯草芽孢杆菌脂肪酶在工业领域的应用提供重要的基础数据。
关键词 枯草芽孢杆菌脂肪酶A;大肠杆菌;生长期启动子;载体构建;发酵优化
DOI:10.13995/j.cnki.11-1802/ts.039232
引用格式:杜菲凡,黎菁菁,郑蓝昕,等.生长期特异性启动子对脂肪酶表达优化的研究[J].食品与发酵工业,2025,51(14):106-112.
DU Feifan,LI Jingjing,ZHENG Lanxin, et al.Optimization of lipase expression by anagen-specific promoters[J].Food and Fermentation Industries,2025,51(14):106-112.
第一作者:硕士研究生(曹诗林讲师和曾新安教授为共同通信作者,E-mail:shilin.cao@fosu.edu.cn;xazeng@scut.edu.cn)
基金项目:国家重点研发计划项目(2023YFD2101003)
收稿日期:2024-03-15,改回日期:2024-09-29
脂肪酶(lipase,EC 3.1.1.3),是一类能特异性地在油水界面上水解三脂酰甘油酯键的酶[1-2],广泛存在于微生物、人体、动物以及植物中。枯草芽孢杆菌脂肪酶A(Bacillus subtilis lipase A, Lip A)的分子质量只有19.3 kDa,是分子质量最小的脂肪酶之一[3]。由于Lip A具有耐碱性强[4]、缺乏盖子结构、无界面激活作用[5]等特点,在食品、洗涤剂和造纸等方面有重要应用。由于微生物繁殖速度快、培养简单,其产生的脂肪酶种类多,具有较强的稳定性、底物特异性,水解作用的温度和pH范围更广[6],并且大多数的脂肪酶为胞外酶[7],使得更方便快捷地在工业生产中得到纯度较高的酶制剂,因此微生物发酵产脂肪酶已成为工业生产脂肪酶的主要途径,有关微生物脂肪酶生产的相关研究也越来越多。
大肠杆菌(Escherichia coli)表达系统由于具有生长速度快、培养条件简单、遗传背景清晰和经济效能高等特点,目前已被广泛应用于异源蛋白的工业生产[8-9]。其中启动子的启动强度会影响重组蛋白的表达,一般可以分为诱导型启动子、组成型启动子和生长期特异性启动子等[10]。诱导型启动子是需要添加诱导剂调控表达的启动子,一般使用异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)进行诱导,但是IPTG价格昂贵且对人体有潜在的毒性;组成型启动子是不需要添加诱导剂即能在细胞生长的整个时期持续性表达的启动子,但外源蛋白的过早表达,也会增加宿主细胞的负担,影响重组蛋白的表达,而且它容易发生有害突变、质粒易丢失,不适宜应用于工业生产中;生长期特异性启动子是不需要添加诱导剂就能够在某一个特定的生长时期自主表达的基因调控元件,不需要严格控制表达条件就能相对精准地在特定的时期表达,节约生产成本,具有较大的应用价值[11-13]。
本文从NCBI数据库中挖掘出一个在对数生长期(OD600nm≈0.6)时开始表达的生长期启动子ynhG,以增强型绿色荧光蛋白(enhanced green fluorescent protein, EGFP)为表征,对ynhG启动子进行表达鉴定,再构建出ynhG启动子引导的枯草芽孢杆菌脂肪酶表达菌株,进行表达鉴定后选择效果较优的ynhG启动子引导的枯草芽孢杆菌脂肪酶表达菌株进行条件优化。但是由于野生型ynhG启动子的启动强度较低,获得的Lip A也较低,为了使重组Lip A高表达,需要对ynhG启动子进行改造,使其定向进化。启动子转录起始位点上游的保守序列(-35区和-10区)改变会显著影响启动子的活性[10]。例如,BR ZIO等[14]构建了-35区突变的P(1)启动子,促进了IMP-5碳青霉烯酶的高水平表达;VINUÉ等[15]构建了-10区突变的P2启动子,使得P2启动子的强度提高了1倍;改变核糖体结合位点(ribosomal binding site, RBS)序列也会对启动子活性产生较大影响[10]。CHEN等[16]对大肠杆菌lacZ启动子的RBS进行突变,获得了启动强度提升0.17~8.6倍的RBS文库,使用启动强度提升的启动子组合调节dcuB和dcuC基因(能够表达控制琥珀酸排出的主要转运蛋白)的表达,导致琥珀酸滴度增加34%。因此,为了提高ynhG启动子的活性,本研究分别对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行了饱和突变,构建出ynhG启动子文库[17]。
ZIO等[14]构建了-35区突变的P(1)启动子,促进了IMP-5碳青霉烯酶的高水平表达;VINUÉ等[15]构建了-10区突变的P2启动子,使得P2启动子的强度提高了1倍;改变核糖体结合位点(ribosomal binding site, RBS)序列也会对启动子活性产生较大影响[10]。CHEN等[16]对大肠杆菌lacZ启动子的RBS进行突变,获得了启动强度提升0.17~8.6倍的RBS文库,使用启动强度提升的启动子组合调节dcuB和dcuC基因(能够表达控制琥珀酸排出的主要转运蛋白)的表达,导致琥珀酸滴度增加34%。因此,为了提高ynhG启动子的活性,本研究分别对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行了饱和突变,构建出ynhG启动子文库[17]。
本研究首先设计出ynhG启动子的-35区、-10区和RBS等几个区域的引物,对ynhG-EGFP-pETDuet-1表达质粒进行饱和突变构建出ynhG启动子文库,然后随机挑取-35区、-10区和RBS等几个区域的启动子文库的菌落进行培养,并以EGFP为表征测定相对荧光强度,选出启动强度提高的ynhG生长期启动子,再用于构建Lip A表达菌株,以提高Lip A的表达量。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
大肠杆菌DH5α、BL21(DE3)和LipA表达片段均保存于本实验室。ynhG-EGFP-pETDuet-1和ynhG-Lip A-EGFP为本实验室前期已构建的质粒。
1.1.2 试剂
质粒提取试剂盒、琼脂糖凝胶DNA回收试剂盒,上海捷瑞生物工程有限公司;胰蛋白胨、酵母提取物、技术琼脂粉,广州环凯微生物科技有限公司;甘油、异丙醇,天津大茂化学试剂有限公司;无水氯化钙,国药集团化学试剂有限公司;琼脂糖,上海阿拉丁生化科技股份公司;QuickCut DpnI、PrimeSTAR HS (Premix),宝日医生物技术(北京)有限公司。其他的常规试剂为国产或进口分装。
1.1.3 实验溶液的配制
LB液体培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,pH值中性。
LB固体培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,技术琼脂粉20,pH值中性。
氨苄青霉素溶液(100 mg/mL):氨苄青霉素0.5 g,无菌水5 mL,无菌滤头(0.22 μm)过滤除菌后,分装在无菌的1.5 mL离心管中,-20 ℃冰箱贮存。
PBS缓冲液(g/L):KH2PO4 23.1,K2HPO4 125.4,pH 7.4。
1.2 实验方法
1.2.1 构建ynhG启动子突变文库
以EGFP作为报告基因,使用ynhG-EGFP-pETDuet-1质粒作为模板,对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行饱和突变,ynhG启动子序列如图1所示,具体突变位点如表1所示,所用引物如表2所示,扩增程序为:98 ℃预变性2 min;98 ℃变性10 s;65 ℃退火10 s;72 ℃延伸6.5 min,循环25次;最后72 ℃延伸7 min。使用DpnI快切酶对突变了-35区、-10区、RBS前端和RBS后端序列的PCR产物酶切15 min去除ynhG-EGFP-pETDuet-1模板质粒,再通过琼脂糖凝胶电泳进行检测。分别将突变了4个区域序列的PCR产物通过热激法转化到大肠杆菌BL21(DE3)中,构建出ynhG启动子的-35区、-10区、RBS前端和RBS后端突变文库。
表1 ynhG启动子的突变位点
Table 1 Mutation sites of ynhG promoter

突变区域突变位点(5'-3')-35区gaaa-10区caacatRBS前端序列ggcaaatRBS后端序列gtttaca
表2 ynhG启动子的突变引物
Table 2 Primers of ynhG promoter mutation
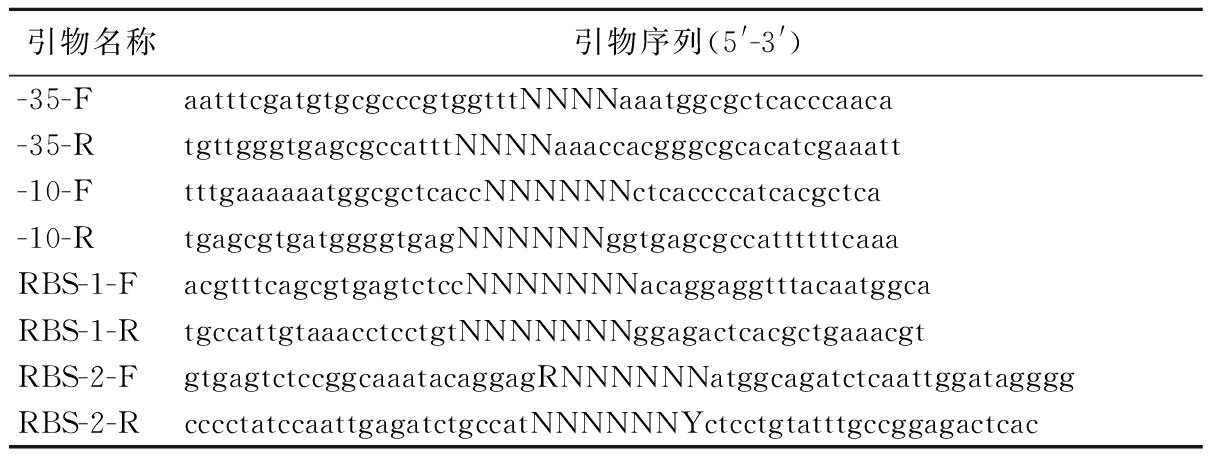
引物名称引物序列(5'-3')-35-FaatttcgatgtgcgcccgtggtttNNNNaaatggcgctcacccaaca-35-RtgttgggtgagcgccatttNNNNaaaccacgggcgcacatcgaaatt-10-FtttgaaaaaatggcgctcaccNNNNNNctcaccccatcacgctca-10-RtgagcgtgatggggtgagNNNNNNggtgagcgccattttttcaaaRBS-1-FacgtttcagcgtgagtctccNNNNNNNacaggaggtttacaatggcaRBS-1-RtgccattgtaaacctcctgtNNNNNNNggagactcacgctgaaacgtRBS-2-FgtgagtctccggcaaatacaggagRNNNNNNatggcagatctcaattggataggggRBS-2-RcccctatccaattgagatctgccatNNNNNNYctcctgtatttgccggagactcac
注:RBS-1:突变RBS前端序列,RBS-2:突变RBS后端序列。
1.2.2 测定EGFP荧光强度并筛选ynhG启动子
根据平板中菌落数目,在-35区、-10区和RBS前、后端突变后的平板上,分别随机挑取了66、120、117、90个菌落接种至含有氨苄青霉素抗性的LB培养基中37 ℃过夜培养。以1%的接种量接种于含有氨苄青霉素抗性的LB培养基中37 ℃,200 r/min条件下培养24 h。离心并收集菌体,用PBS缓冲液(pH=7.4)重悬洗涤2次,再用PBS缓冲液将菌体重悬,测定OD600nm,并在激发波长485 nm,发射波长528 nm 的条件下,测定荧光强度值。本文的荧光强度为在如上条件下每单位OD600nm的荧光强度值,空白对照为原始的ynhG启动子。计算出相应的荧光强度后,选取荧光强度较高的阳性克隆子寄去北京擎科生物科技有限公司进行测序。
1.2.3 ynhG-Lip A表达质粒的构建
取出一管ynhG-Lip A-EGFP-BL21(DE3)甘油菌,置于冰上溶解后,接种于含有氨苄青霉素抗性的LB液体培养基中,37 ℃过夜培养。使用质粒提取试剂盒(小量)提取ynhG-Lip A-EGFP质粒,将其作为PCR模板进行反向PCR,将ynhG-Lip A-EGFP质粒上的EGFP基因片段截短,构建ynhG-Lip A表达质粒,PCR扩增引物如表3所示,扩增程序为:98 ℃预变性2 min;98 ℃变性10 s;55 ℃退火10 s;72 ℃延伸6 min 20 s,循环25次;最后72 ℃延伸7 min。PCR产物使用1%琼脂糖凝胶电泳检测,之后将分子质量为6 172 bp的目的条带切割下来,并使用琼脂糖凝胶DNA回收试剂盒进行纯化回收,将经过纯化的片段转化进大肠杆菌DH5α感受态细胞,经质粒提取验证成功的阳性克隆子,扩种后使用50%甘油保菌,取一管菌液送北京擎科生物科技有限公司进行测序,并且将工程菌分别命名为ynhG-Lip A-DH5α。
表3 PCR扩增引物
Table 3 PCR amplification primers
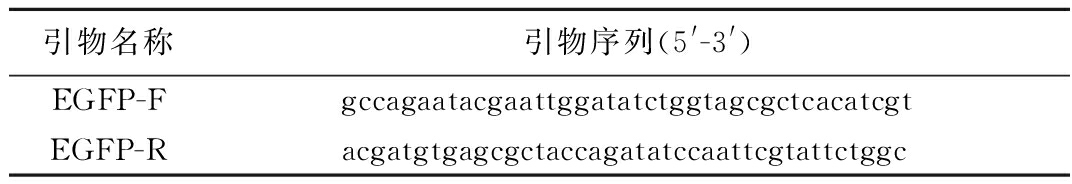
引物名称引物序列(5'-3')EGFP-FgccagaatacgaattggatatctggtagcgctcacatcgtEGFP-Racgatgtgagcgctaccagatatccaattcgtattctggc
1.2.4 构建增强型ynhG启动子的Lip A表达菌株
根据1.2.2节的结果,ynhG启动子的RBS后端突变后ynhG启动子活性明显提高,因此按照RBS后端突变后的表达菌株相对于原始的ynhG启动子的表达菌株的荧光强度提升值进行排序,选取了排行前8个的样品,根据启动子的测序结果进行点突变,构建出8个不同的增强型ynhG-Lip A表达菌株。
1.2.4.1 使用点突变进行增强型ynhG-Lip A表达质粒的构建
点突变构建增强型ynhG-Lip A表达质粒,PCR模板为:ynhG-Lip A表达质粒,PCR扩增引物如表4所示,扩增程序为:98 ℃预变性2 min;98 ℃变性10 s,58 ℃退火10 s,72 ℃延伸6 min 20 s,循环25次;最后72 ℃延伸7 min。PCR产物使用DpnI酶切去除模板质粒后,1%琼脂糖凝胶电泳检测。
表4 点突变引物
Table 4 Point mutation primers

引物名称引物序列(5'-3')RBS-2.1-FctccggcaaatacaggagACACGAaatgaaatacctgctgccgacRBS-2.1-RcggcagcaggtatttcattTCGTGTctcctgtatttgccggagacRBS-2.2-FctccggcaaatacaggaggAAAAACatgaaatacctgctgccgacRBS-2.2-RcggcagcaggtatttcatGTTTTTcctcctgtatttgccggagacRBS-2.3-FctccggcaaatacaggagAACAAAaatgaaatacctgctgccgacRBS-2.3-RcggcagcaggtatttcattTTTGTTctcctgtatttgccggagacRBS-2.4-FctccggcaaatacaggagAACAGcaatgaaatacctgctgccgacRBS-2.4-RcggcagcaggtatttcattgCTGTTctcctgtatttgccggagacRBS-2.5-FctccggcaaatacaggaggGTCCTaatgaaatacctgctgccgacRBS-2.5-RcggcagcaggtatttcattAGGACcctcctgtatttgccggagacRBS-2.6-FctccggcaaatacaggaggAACCTTatgaaatacctgctgccgacRBS-2.6-RcggcagcaggtatttcatAAGGTTcctcctgtatttgccggagacRBS-2.7-FctccggcaaatacaggagATTAAAaatgaaatacctgctgccgacRBS-2.7-RcggcagcaggtatttcattTTTAATctcctgtatttgccggagacRBS-2.8-FctccggcaaatacaggaggGGAAAGatgaaatacctgctgccgacRBS-2.8-RcggcagcaggtatttcatCTTTCCcctcctgtatttgccggagac
注:大写字母的碱基为突变位点。
1.2.4.2 转化、测序验证
将构建的8个增强型ynhG-Lip A表达质粒转化到大肠杆菌BL21(DE3)感受态细胞,经质粒提取验证成功的阳性克隆子,扩种后使用50%甘油保菌,各取一管菌液送北京擎科生物科技有限公司进行测序,并且将工程菌分别命名为ynhG-RBS-2.1-Lip A-BL21(DE3)、ynhG-RBS-2.2-LipA-BL21(DE3)、ynhG-RBS-2.3- LipA-BL21(DE3)、ynhG-RBS-2.4-LipA-BL21(DE3)、ynhG-RBS-2.5-LipA-BL21(DE3)、ynhG-RBS-2.6-LipA-BL21(DE3)、ynhG-RBS-2.7-Lip A-BL21(DE3)和ynhG-RBS-2.8-Lip A-BL21(DE3)。
1.2.5 表达鉴定
ynhG启动子能够在OD600nm≈0.6时开始自主表达,将测序验证成功的8个增强型ynhG-Lip A-BL21(DE3)和原始的ynhG-Lip A-BL21(DE3)分别接种于含有氨苄青霉素抗性的LB培养基中37 ℃培养12~16 h;按1%接种量接种于50 mL的LB液体培养基里,37 ℃、220 r/min,培养到OD值为0.6时,转移到30 ℃摇床,诱导16 h;在4 ℃、8 000 r/min(下同)条件下离心10 min,收集菌体,使用PBS缓冲液(pH=7.4)重悬洗涤,离心后去除上清液,重复操作2次;再加入PBS缓冲液10 mL进行重悬,然后超声破碎,具体条件为:功率比20%,超声工作0.8 s,间歇1 s,超声破碎时间为2 min/mL细胞悬浮液,8 000 r/min离心10 min,收集细胞裂解的上清液进行酶活力的测定。
1.2.6 酶活力的测定
采用时间过程曲线法测定脂肪酶酶活力,总体系为250 μL,将200 μL 50 mmol/L磷酸盐缓冲液与25 μL 酶液混合,加入25 μL底物溶液(20 mmol/L棕榈酸对硝基苯酯溶于异丙醇中)后,立即读取410 nm 波长下0~2 min的吸光值,并以该过程变化曲线的斜率来表征相对酶活力,重复实验3次,取平均值。本文的相对酶活力的计算为1 L发酵体系下的总酶活力与菌液OD600nm的比值,空白对照为原始的ynhG-Lip A-BL21(DE3)表达菌株的相对酶活力。
2 结果与分析
2.1 ynhG启动子突变结果
2.1.1 重组质粒的构建
以ynhG-EGFP-pETDuet-1质粒作为模板,对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行饱和突变,然后使用DpnI快切酶酶切后,通过琼脂糖凝胶电泳进行检测,如图2所示,成功跑出了分子质量大小为6 172 bp的-35区突变(泳道1)、-10区突变(泳道2)、RBS前端突变(泳道3)和RBS后端突变(泳道4)的ynhG-EGFP-pETDuet-1质粒。
2.1.2 ynhG启动子突变文库对EGFP的表达活性
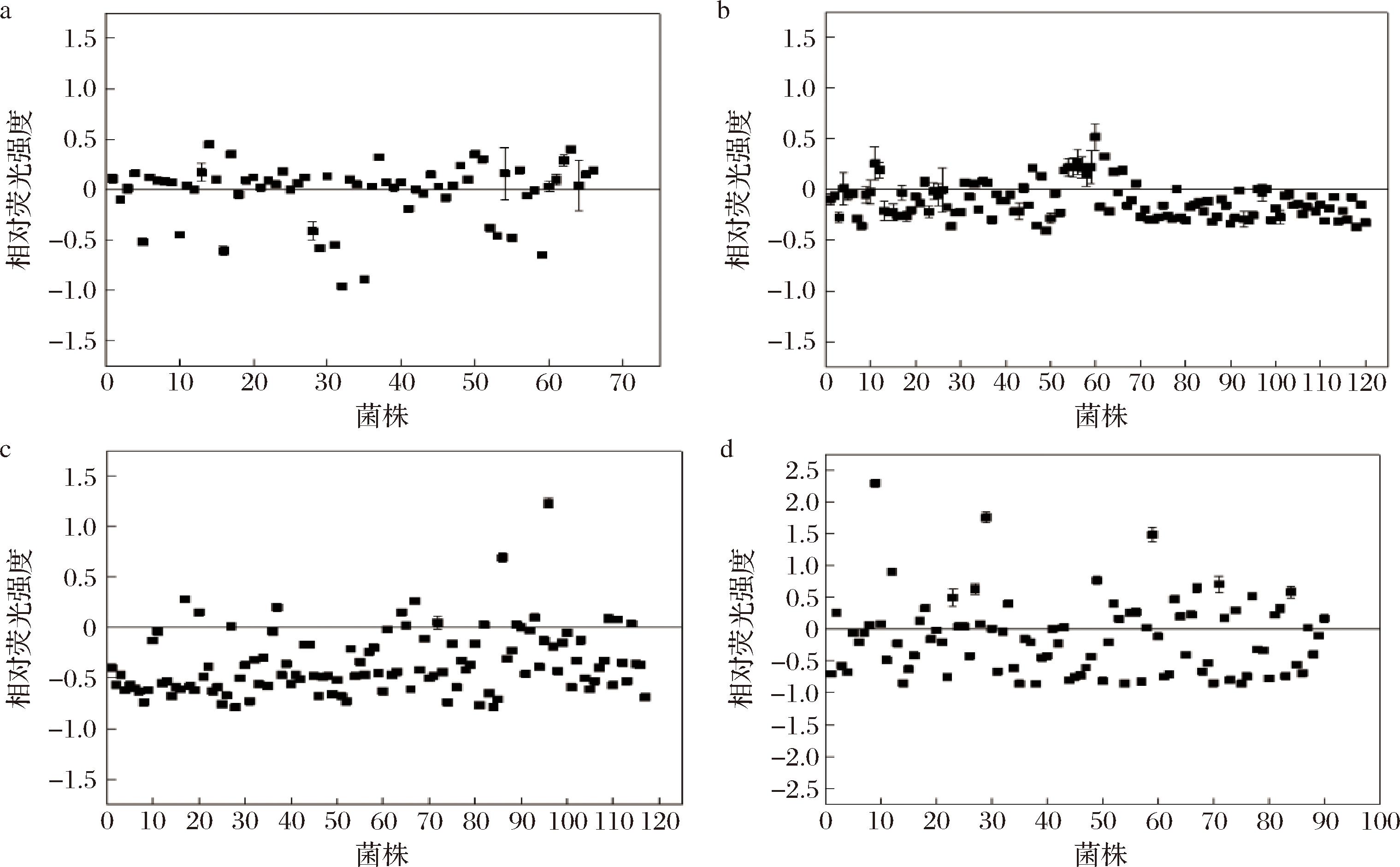
根据LB平板上的菌落数目,从4个不同的区域中随机挑选了多个突变菌株,其中-35区66个、-10区120个、RBS前端117个、RBS后端90个,并使用EGFP作为报告基因对这些突变菌株的荧光强度进行测定,确定不同区域突变对ynhG启动子活性的影响。如图3所示,横坐标为随机挑选的菌株的编号,纵坐标为相对荧光强度,它具体反映了菌株的荧光强度相对于空白对照的变化情况。在图3中,y=0作为一条辅助线,其上方的点代表启动子活性有所增强,而位于y=0下方的点则表明启动子活性出现降低。在随机挑选的菌株中ynhG启动子的-35区突变后活性提升的比例为69.7%,活性下降的比例为30.3%,活性变化在-0.96~0.45倍;-10区突变后活性提升的比例为21.67%,活性下降的比例为78.33%,活性变化范围在-0.41~0.52倍;RBS前端突变后活性提升的比例为14.53%,活性下降的比例为85.47%,活性变化范围在-0.79~1.23倍;RBS后端突变后活性提升的比例为41.11%,活性下降的比例为58.89%,活性提升的最高值比空白对照高2.3倍,活性变化范围更广,在-0.86~2.3倍,且活性提升值高于空白对照0.5倍的比例占12.22%,提升更明显,说明突变RBS后端会导致更大范围的强度变化,更易获得表达强度高的启动子,因此,后续将对RBS后端突变的ynhG启动子文库进行研究。
2.1.3 ynhG启动子的RBS后端突变文库的研究
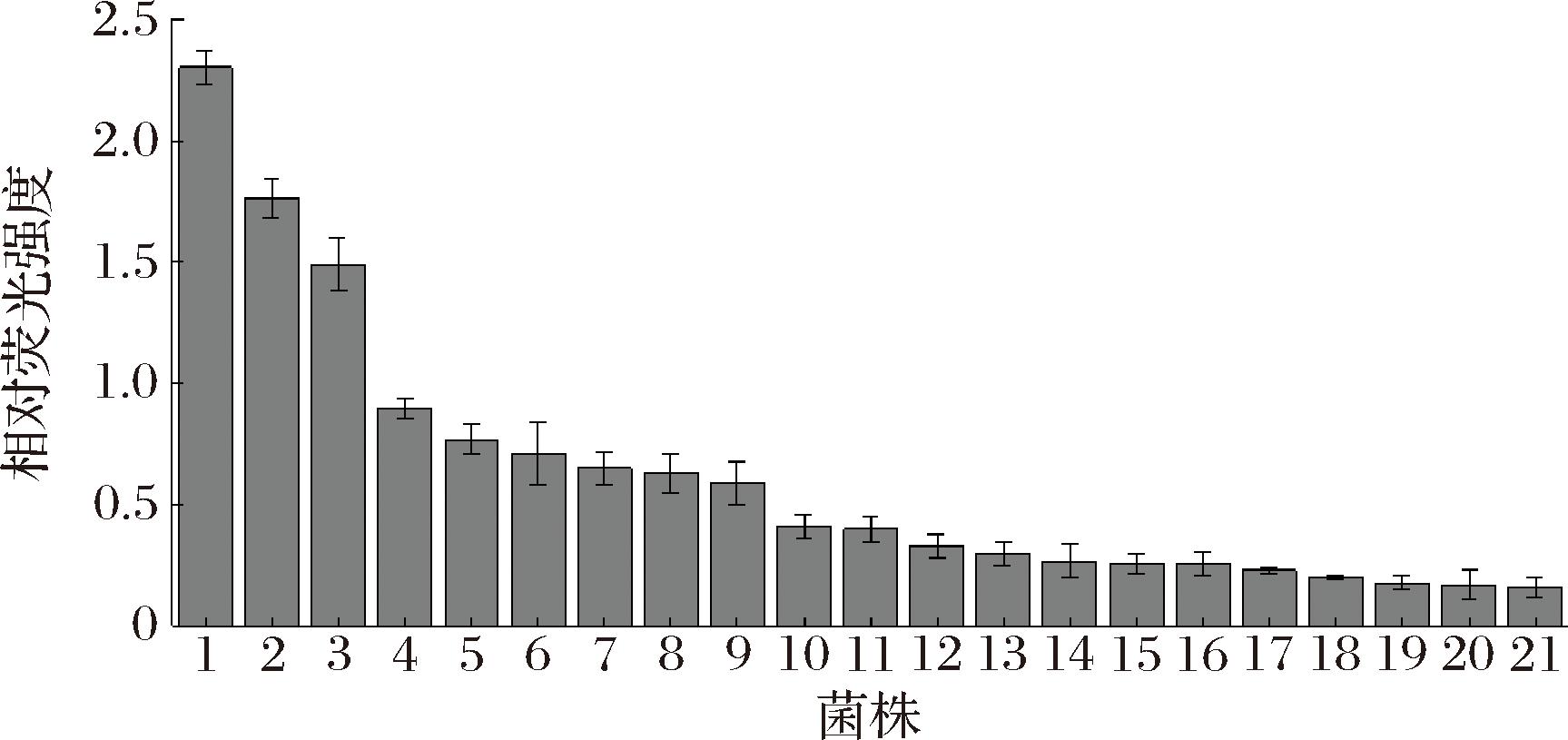
ynhG启动子的RBS后端突变文库展现出广泛的启动强度变化,并且获得了更多的活性提高的突变体。因此,从该文库中选取21个菌株,并依据活性大小进行重新排序和测序。如图4所示,所选的21个菌株对EGFP的表达强度相对于空白对照都有所提升,前8个菌株的表达强度均比空白对照提高了0.6倍以上,其余大都在提高0.15~0.5倍,各菌株对应的RBS序列如表5所示。
表5 RBS序列
Table 5 RBS sequence
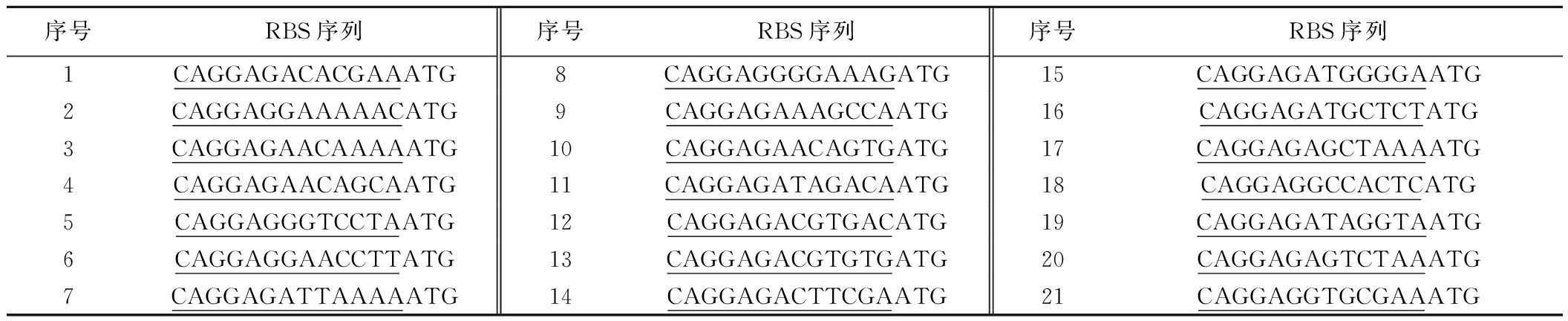
序号RBS序列序号RBS序列序号RBS序列1CAGGAGACACGAAATG8CAGGAGGGGAAAGATG15CAGGAGATGGGGAATG2CAGGAGGAAAAACATG9CAGGAGAAAGCCAATG16CAGGAGATGCTCTATG3CAGGAGAACAAAAATG10CAGGAGAACAGTGATG17CAGGAGAGCTAAAATG4CAGGAGAACAGCAATG11CAGGAGATAGACAATG18CAGGAGGCCACTCATG5CAGGAGGGTCCTAATG12CAGGAGACGTGACATG19CAGGAGATAGGTAATG6CAGGAGGAACCTTATG13CAGGAGACGTGTGATG20CAGGAGAGTCTAAATG7CAGGAGATTAAAAATG14CAGGAGACTTCGAATG21CAGGAGGTGCGAAATG
2.2 构建ynhG-Lip A表达质粒
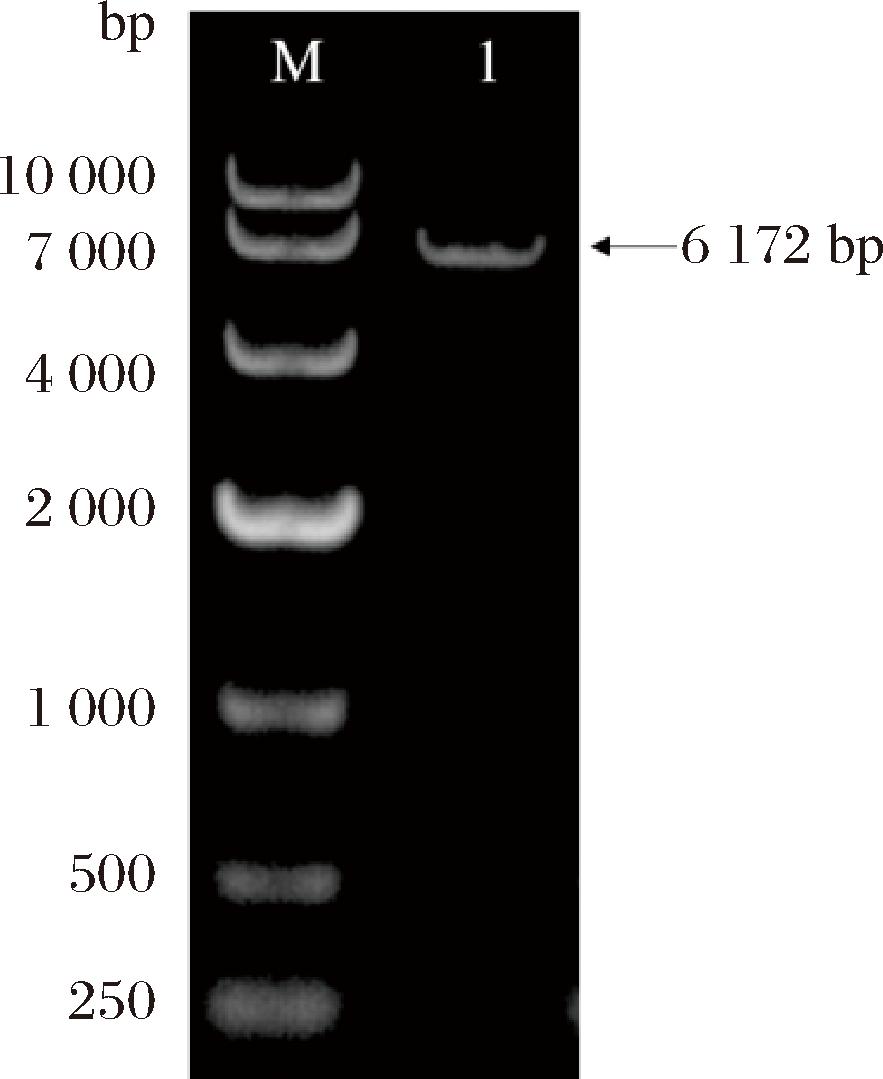
以ynhG-Lip A-EGFP表达质粒为模板,设计引物反向PCR截短EGFP片段,使用化学转化法将截短后的ynhG-Lip A表达质粒转化到大肠杆菌DH5α中,挑取阳性克隆子扩大培养,提取质粒进行琼脂糖凝胶电泳,如图5所示,成功得到分子质量大小为6 172 bp条带(泳道1)。保菌后送样,委托北京擎科生物科技有限公司进行测序,测序结果和预先设计的结果符合,表明ynhG-Lip A表达质粒构建成功。
2.3 增强型ynhG启动子的Lip A表达菌株的构建
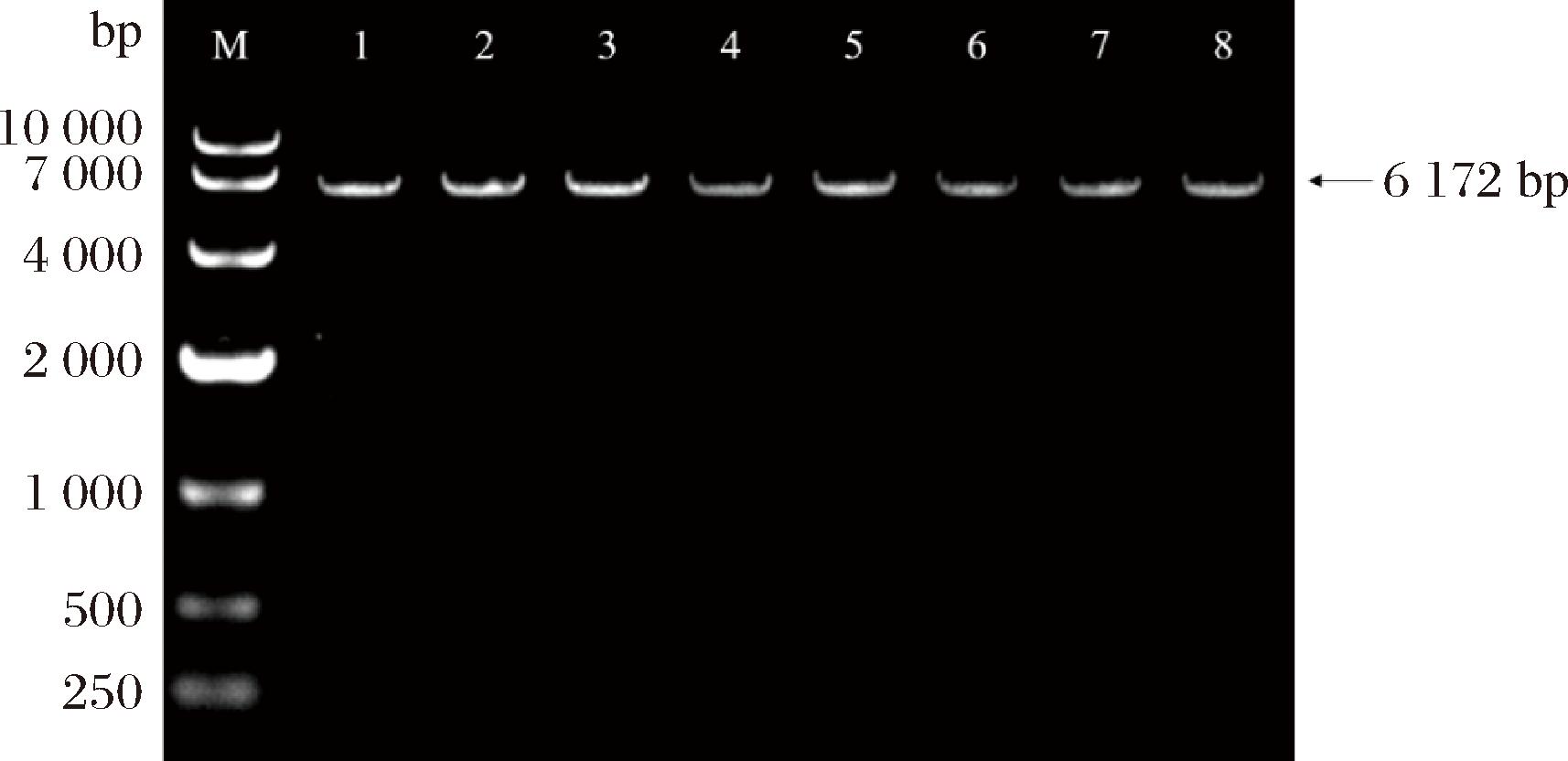
选取RBS后端突变文库中启动强度提升值排行前8位的菌株,根据表5的ynhG启动子的RBS序列的测序结果设计引物,以构建成功的ynhG-Lip A表达质粒作为模板进行点突变,转化到大肠杆菌BL21(DE3)中进行筛选,随机挑取阳性克隆子扩大培养,并提取质粒进行验证,PCR产物使用DpnI酶切去除模板质粒后,1%琼脂糖凝胶电泳检测。如图6所示,8个编号的表达质粒均成功跑出6 172 bp的条带,将其分别保菌测序后,成功得到突变成功的序列,表明成功获得了增强型的ynhG-RBS-2.1-Lip A-BL21(DE3)(泳道1)、ynhG-RBS-2.2-LipA-BL21(DE3)(泳道2)、ynhG-RBS-2.3-Lip A-BL21(DE3)(泳道3)、ynhG-RBS-2.4-Lip A-BL21(DE3)(泳道4)、ynhG-RBS-2.5-Lip A-BL21(DE3)(泳道5)、ynhG-RBS-2.6-Lip A-BL21(DE3)(泳道6)、ynhG-RBS-2.7-Lip A-BL21(DE3)(泳道7)和ynhG-RBS-2.8-Lip A-BL21(DE3)(泳道8)表达菌株。
2.4 表达鉴定结果
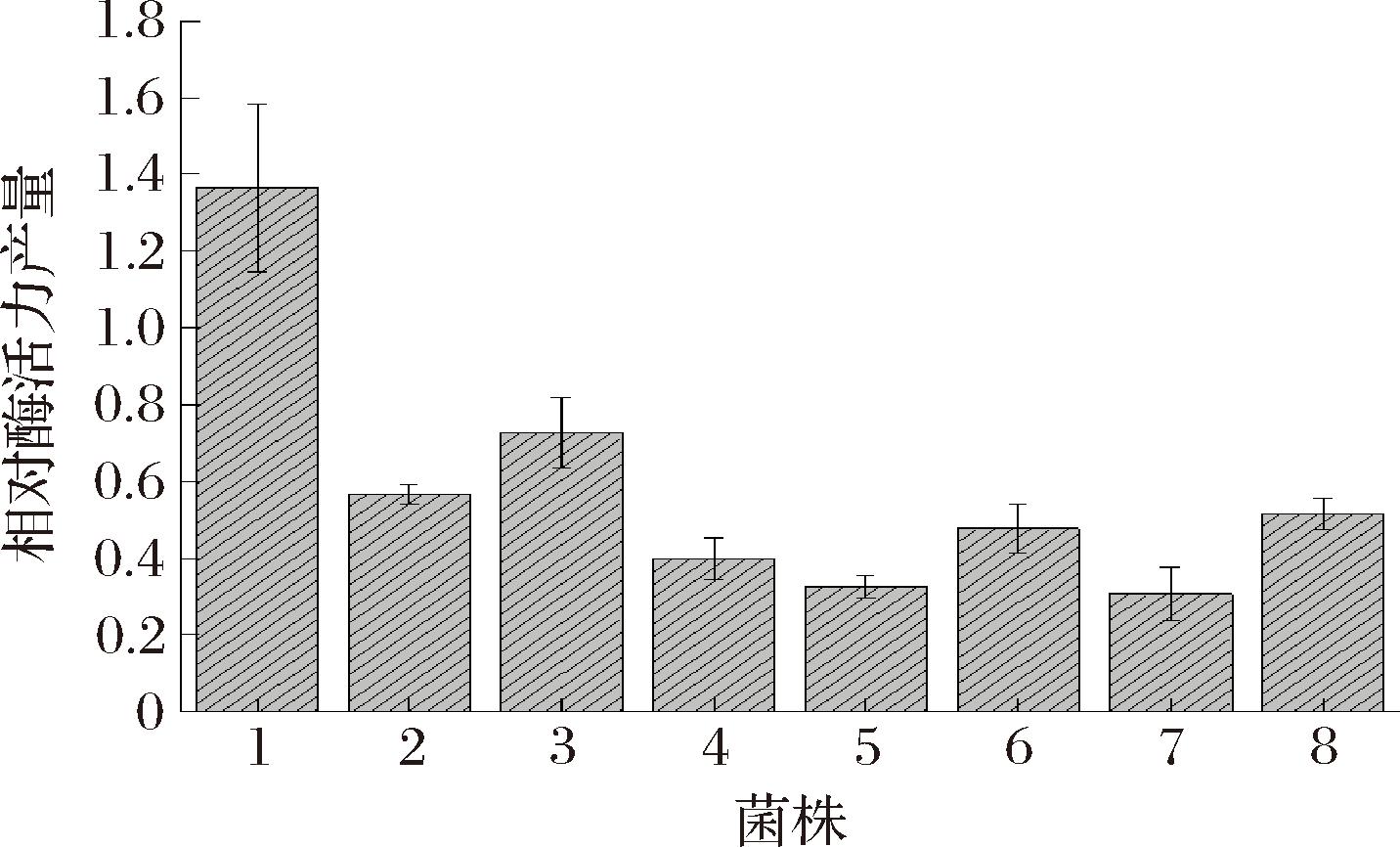
对8个增强型ynhG-Lip A-BL21(DE3)和原始的ynhG-Lip A-BL21(DE3)菌株进行发酵表达,如图7所示,发现构建的8个增强型的ynhG启动子表达枯草芽孢杆菌脂肪酶A的强度都有一定的提升,酶活力产量相对于空白对照提升了31%~137%,可能是由于发酵的条件不同,使得构建的8个增强型的ynhG启动子对Lip A的表达强度和对EGFP的表达强度(0.63~2.3倍)有一定的差异。但是8个增强型的ynhG启动子对Lip A的表达均有所提升,表明通过对ynhG启动子进行改造获得了表达强度更高的启动子,促进Lip A的高效表达,同时为后续对生长期特异性启动子的研究提供了一定的实验基础和合适的底盘。
3 结论与讨论
大肠杆菌(Escherichia coli)表达系统因其具有发酵周期短、操作简单、价格低廉等优点,目前已被广泛用于外源蛋白的工业化生产。本研究成功对ynhG启动子的-35区、-10区和RBS区域进行饱和突变,发现对RBS后端进行突变更易获得启动强度提高的启动子,并且通过测序成功获得启动强度提高的增强型ynhG启动子的序列。同时成功对ynhG-Lip A-EGFP的表达质粒进行截短,获得ynhG-Lip A表达质粒,并以ynhG-Lip A为模板对ynhG启动子进行点突变,成功获得增强型的ynhG-Lip A-BL21(DE3)表达菌株。最后,对8个增强型的ynhG-Lip A-BL21(DE3)表达菌株进行表达鉴定,发现所获得的增强型ynhG启动子的酶活产量比野生型的ynhG启动子增加了31%~137%。研究结果为进一步提高枯草芽孢杆菌脂肪酶的工业应用提供了重要的基础数据。
参考文献
[1] ALI S, KHAN S A, HAMAYUN M, et al.The recent advances in the utility of microbial lipases:A review[J].Microorganisms, 2023, 11(2):510.
[2] 黎菁菁, 赖昕珏, 李鑫尧, 等.枯草芽孢杆菌脂肪酶LipA的生物信息学分析[J].现代食品科技, 2022, 38(7):113-119;10.LI J J, LAI X J, LI X Y, et al.The bioinformatics analysis of the lipase LipA from Bacillus subtilis[J].Modern Food Science and Technology, 2022, 38(7):113-119;10.
[3] RATHI P C, FULTON A, JAEGER K E, et al.Application of rigidity theory to the thermostabilization of lipase A from Bacillus subtilis[J].PLoS Computational Biology, 2016, 12(3):e1004754.
[4] KÜBLER D, INGENBOSCH K N, BERGMANN A, et al.Fluorescence spectroscopic analysis of the structure and dynamics of Bacillus subtilis lipase A governing its activity profile under alkaline conditions[J].European Biophysics Journal, 2015, 44(8):655-665.
[5] VAN POUDEROYEN G, EGGERT T, JAEGER K E, et al.The crystal structure of Bacillus subtili lipase:A minimal α/β hydrolase fold enzyme[J].Journal of Molecular Biology, 2001, 309(1):215-226.
[6] 汪玲. 微生物脂肪酶的性质及应用[J].生物化工, 2020, 6(3):161-163.WANG L.Study on the properties and application of microbial fecal enzymes[J].Biological Chemical Engineering, 2020, 6(3):161-163.
[7] 胡兴翠, 刘建华.微生物脂肪酶特性及工业应用[J].基因组学与应用生物学, 2019, 38(8):3572-3579.HU X C, LIU J H.Characteristics and industrial applications of microbial lipase[J].Genomics and Applied Biology, 2019, 38(8):3572-3579.
[8] HULEANI S, ROBERTS M R, BEALES L, et al.Escherichia coli as an antibody expression host for the production of diagnostic proteins:Significance and expression[J].Critical Reviews in Biotechnology, 2022, 42(5):756-773.
[9] VINCENTELLI R, ROMIER C.Expression in Escherichia coli:Becoming faster and more complex[J].Current Opinion in Structural Biology, 2013, 23(3):326-334.
[10] 于慧敏, 郑煜堃, 杜岩, 等.合成生物学研究中的微生物启动子工程策略[J].合成生物学, 2021, 2(4):598-611.YU H M, ZHENG Y K, DU Y, et al.Microbial promoter engineering strategies in synthetic biology[J].Synthetic Biology Journal, 2021, 2(4):598-611.
[11] PAN X W, TANG M, YOU J J, et al.A novel method to screen strong constitutive promoters in Escherichia coli and Serratia marcescens for industrial applications[J].Biology, 2022, 12(1):71.
[12] ANILIONYTE O, LIANG H, MA X Q, et al.Short, auto-inducible promoters for well-controlled protein expression in Escherichia coli[J].Applied Microbiology and Biotechnology, 2018, 102(16):7007-7015.
[13] 刘博. 大肠杆菌稳定期特异性启动子的筛选及pSP表达载体的构建[D].大连:大连理工大学, 2007.LIU B.Stationary-phase promoters screening in Escherichia coli and construction of pSP expression vector[D].Dalian:Dalian University of Technology, 2007.
[14] BR ZIO A, CONCEIÇ
ZIO A, CONCEIÇ O T, PIMENTEL M, et al.High-level expression of IMP-5 carbapenemase owing to point mutation in the-35 promoter region of class 1 integron among Pseudomonas aeruginosa clinical isolates[J].International Journal of Antimicrobial Agents, 2006, 27(1):27-31.
O T, PIMENTEL M, et al.High-level expression of IMP-5 carbapenemase owing to point mutation in the-35 promoter region of class 1 integron among Pseudomonas aeruginosa clinical isolates[J].International Journal of Antimicrobial Agents, 2006, 27(1):27-31.
[15] VINUÉ L, JOVÉ T, TORRES C, et al.Diversity of class 1 integron gene cassette Pc promoter variants in clinical Escherichia coli strains and description of a new P2 promoter variant[J].International Journal of Antimicrobial Agents, 2011, 38(6):526-529.
[16] CHEN J, ZHU X N, TAN Z G, et al.Activating C4-dicarboxylate transporters DcuB and DcuC for improving succinate production[J].Applied Microbiology and Biotechnology, 2014, 98(5):2197-2205.
[17] 陈珏, 黄佳敏, 燕天鹤, 等.随机突变文库构建与筛选研究进展[J].生物工程学报, 2021, 37(1):163-177.CHEN J, HUANG J M, YAN T H, et al.Progress in the construction and screening of random mutation library[J].Chinese Journal of Biotechnology, 2021, 37(1):163-177.
Optimization of lipase expression by anagen-specific promoters
DU Feifan1,2, LI Jingjing1,2, ZHENG Lanxin1,2, ZHANG Di1,2, YE Huimin1,2, DENG Junwei1,2, QIN Yunan1,2, HUANG Qiuyan1,2, CAO Shilin1,2*, ZENG Xinan1,2*
1(Guangdong Provincial Key Laboratory of Intelligent Food Manufacturing, Foshan University, Foshan 528000, China)2(College of Food Science and Engineering, Foshan University, Foshan 528000, China)
ABSTRACT Lipase is a biocatalyst with an important role in several biochemical reactions.Bacillus subtilis lipase A (LipA) is one of the lipases with the smallest molecular weight.Due to its strong adaptability to substrate, strong alkaline resistance, no cover structure, and no need for interface activation, it has important application prospects in food, medicine, paper making, detergent, and other aspects.To improve the fermentation level of LipA, the study was expressed in Escherichia coli BL21 (DE3), and a LipA-expressing strain of the anagen promoter ynhG was constructed to increase the expression of LipA by up-regulating the expression of the ynhG promoter in the growth phase.Characterized with enhanced green fluorescent protein (EGFP), saturation mutation in the -10, -35, and RBS of the ynhG promoter to obtain the sequence of the enhanced ynhG promoter with increased initiation intensity.According to the enhanced ynhG promoter sequence, the enhanced ynhG-LipA-BL21 (DE3) expression strain showed that the enzyme yield of the enhanced ynhG promoter increased by 31% to 137% compared with the wild-type ynhG promoter.The research results will provide important basic data for improving the application of Bacillus subtilis lipase in the industrial field.
Key words Bacillus subtilis lipase A; Escherichia coli; growth stage promoter; vector construction; fermentation optimization
 ZIO等[14]构建了-35区突变的P(1)启动子,促进了IMP-5碳青霉烯酶的高水平表达;VINUÉ等[15]构建了-10区突变的P2启动子,使得P2启动子的强度提高了1倍;改变核糖体结合位点(ribosomal binding site, RBS)序列也会对启动子活性产生较大影响[10]。CHEN等[16]对大肠杆菌lacZ启动子的RBS进行突变,获得了启动强度提升0.17~8.6倍的RBS文库,使用启动强度提升的启动子组合调节dcuB和dcuC基因(能够表达控制琥珀酸排出的主要转运蛋白)的表达,导致琥珀酸滴度增加34%。因此,为了提高ynhG启动子的活性,本研究分别对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行了饱和突变,构建出ynhG启动子文库[17]。
ZIO等[14]构建了-35区突变的P(1)启动子,促进了IMP-5碳青霉烯酶的高水平表达;VINUÉ等[15]构建了-10区突变的P2启动子,使得P2启动子的强度提高了1倍;改变核糖体结合位点(ribosomal binding site, RBS)序列也会对启动子活性产生较大影响[10]。CHEN等[16]对大肠杆菌lacZ启动子的RBS进行突变,获得了启动强度提升0.17~8.6倍的RBS文库,使用启动强度提升的启动子组合调节dcuB和dcuC基因(能够表达控制琥珀酸排出的主要转运蛋白)的表达,导致琥珀酸滴度增加34%。因此,为了提高ynhG启动子的活性,本研究分别对ynhG启动子的-35区、-10区、RBS前端和RBS后端序列进行了饱和突变,构建出ynhG启动子文库[17]。





 ZIO A, CONCEIÇ
ZIO A, CONCEIÇ O T, PIMENTEL M, et al.High-level expression of IMP-5 carbapenemase owing to point mutation in the-35 promoter region of class 1 integron among
O T, PIMENTEL M, et al.High-level expression of IMP-5 carbapenemase owing to point mutation in the-35 promoter region of class 1 integron among