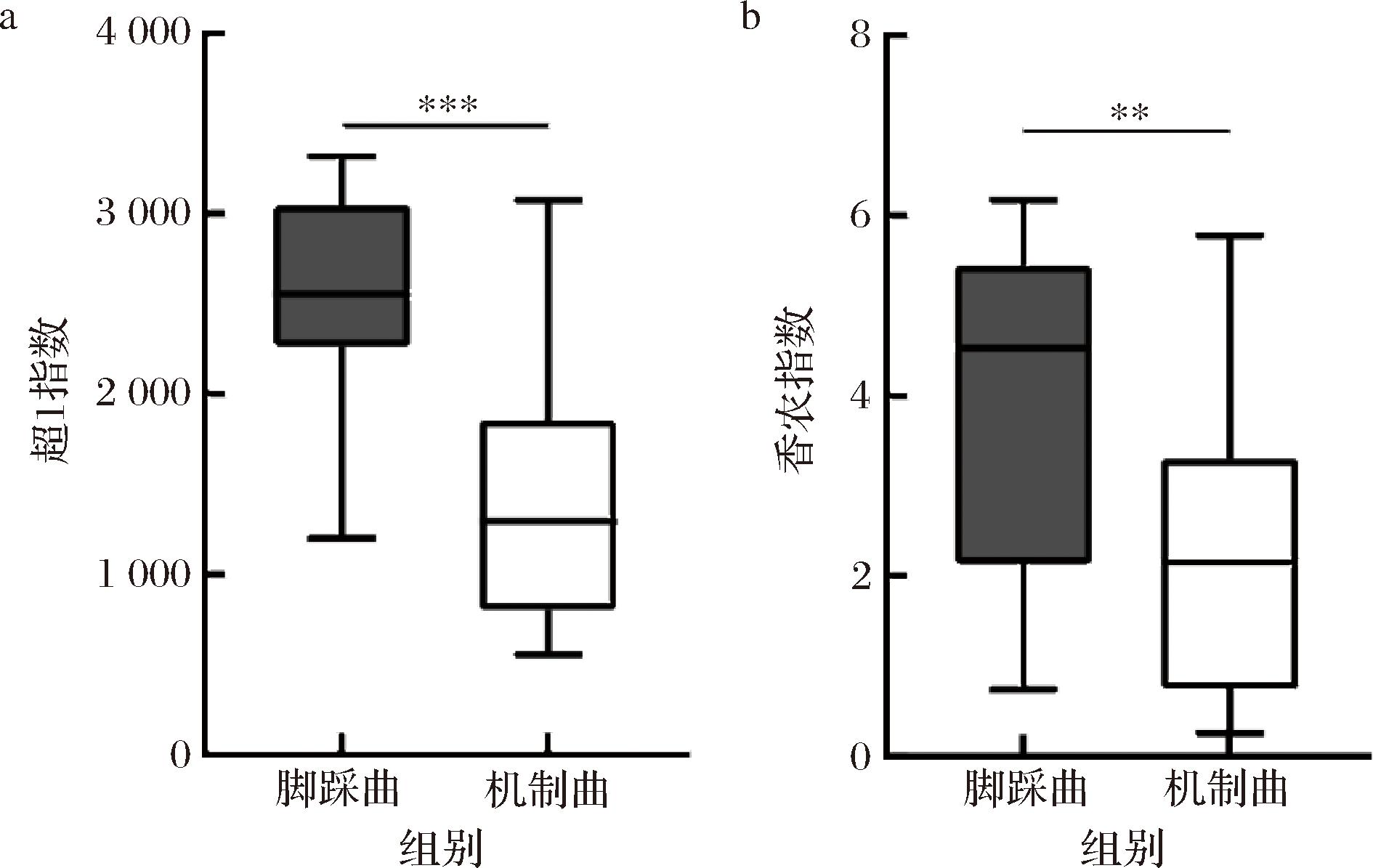
a-超1指数;b-香农指数
图1 机制曲和脚踩曲样品的α多样性分析
Fig.1 α diversity analysis of high-temperature Daqu samples prepared by mechanism and foot stepping
注:超1指数和香农指数均在测序量为28 010条序列时计算所得;***代表P<0.001,**代表P<0.01(下同)。
GE Dongying,CAI Wenchao,YU Peirong, et al.Comparative analysis of MiSeq high-throughput sequencing technology mechanical and artificial high-temperature Daqu fungal community[J].Food and Fermentation Industries,2025,51(14):313-319.
酱香型白酒以高温大曲为糖化发酵剂,经多菌株固态双边发酵而成,是中国传统白酒中复杂且典型的香型之一[1]。高温大曲以小麦为主要原料,经混合、成型、堆积、发酵以及熟化等工艺制成[2]。大曲较为开放的制作环境使得小麦和水等生产原料、空气和土地等生产环境以及生产工具和操作者等携带的微生物在大曲中繁殖,共同构成了高温大曲复杂的微生物系统[3]。当前,酱香型白酒酿造使用的高温大曲主要分为机制曲和脚踩曲两大类,从工艺上看,两类大曲的根本区别是压曲方式的不同,其中机制曲是用机械带动压缩机构,将曲料挤压成型,而脚踩曲是将曲料倒入模具中,由踩曲工人用脚来回踩踏压实[4]。而脚踩曲的制作耗时长且劳动力高,当前逐渐有向效率高的机制曲转变的趋势[5],但机械曲中微生物结构是否可以与传统脚踩曲相比还有待进一步研究。
相较于脚踩曲,机制曲缺少了人工脚踩的环节,WANG等[6]研究表明在脚踩过程中脚上附着的微生物可能会通过接触传入高温大曲中,从而对大曲中微生物的组成造成影响。HUANG等[7]采用高通量测序技术对采自四川宜宾的机械压制和人工脚踩制备的高温大曲中微生物类群进行了解析,结果发现在机械压制高温大曲中嗜热真菌属(Thermomyces)的含量偏高,而人工脚踩高温大曲中Apiotrichum、地霉属(Geotrichun)、丝孢酵母属(Trichosporon)、复膜孢子酵母属(Saccharomycopsis)和丝孢毕赤酵母属(Hyphopichia)的含量偏高。高温大曲中丰富的微生物可能主要来源于生产原料和生产环境两个方面,侯强川等[8]研究表明贵州和湖北地区生产高温大曲的菌群结构存在差异。此外,在真菌菌群中霉菌能够分泌产生各种酶,以提高大曲的糖化力,酵母菌能够产酒产香,对白酒风味品质的形成至关重要[9]。因此,比较分析不同地区机制与脚踩高温大曲的真菌菌群结构,对于探究两类高温大曲的差异性具有积极意义。
高通量测序技术可同时对数百万乃至数十亿个DNA分子进行测序,并且可通过深入全面的解析确定微生物的多样性和类别[10],该技术具有测序结果可靠且可重复等优点,目前在解析大曲中的真菌类群上具有较为广泛的应用[11]。故而,使用高通量测序技术可以达到对于机制曲和脚踩曲的真菌菌群进行解析与比较的目的。本研究采用Illumina MiSeq高通量测序对机制和脚踩的各29个高温大曲的真菌菌群进行了对比分析,通过该研究以期为认识机制曲和脚踩曲中真菌菌群结构的差异提供理论依据。
采集机制高温大曲(M)和脚踩高温大曲(F)各29块,所有样品均为同一批次生产并在曲库中贮存时间大于4个月,取样时从曲库的不同位置随机采集。具体而言,在曲库靠近门口、靠近窗户和曲库中间的不同高度随机采集,其中从脚踩曲曲房和机制曲曲房采集曲块的位置均相似。所有样品均以小麦为主要原料,原料粉碎后拌入母曲,于37 cm×18 cm×7 cm的模具中分别通过机械化压制和60 kg以下的踩曲工人脚踩成型,将成型后的曲块平置晾置后送往曲房侧立堆积发酵,在高温堆积发酵过程中进行2次翻曲,总发酵期大于40 d,待发酵完成后拆曲于曲库中贮存。两类高温大曲除成型方式的差异外,生产时间、制曲原料和工艺等均相同。大曲采集并常温带回实验室后,将各29个机制曲和脚踩曲的曲块分别打磨成粉末状,置于-40 ℃冰箱备用。
DNA基因组提取试剂盒,德国QIAGEN公司;FastPfu Fly DNA Polymerase(5 U/μL)、5×FastPfu Buffer、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)混合物和DNA聚合酶,宝生物工程(大连)有限公司;物ITS3F(5′-GCATCGATGAAGAACGCAGC-3′)/ITS4R(5′-TCCTCCGCTTATTGATATGC-3′),上海赛恒生物科技有限公司;Illumina MiSeq高通量测序平台配套试剂,美国Illumina公司。
JFSD-100Ⅱ粉碎机,上海隆拓仪器设备有限公司;ND-2000C微量紫外分光光度计,美国Thermo公司;Veriti FAST梯度聚合酶链式反应(polymerase chain reaction,PCR)仪,美国ABI公司;DYY-12型电泳仪,北京六一仪器厂;UVPCDS8000凝胶成像分析系统,美国ProteinSimple公司;MiSeq PE300高通量测序平台,美国Illumina公司;R930机架式服务器,美国DELL公司。
1.3.1 宏基因DNA提取、PCR扩增和Illumina MiSeq测序
参照杨少勇等[12]的方法,使用QIAGEN DNeasy maricon Food Kit试剂盒分别提取机制和脚踩的各29 个高温大曲样品中微生物的宏基因组DNA,参照KANG等[13]的扩增条件和体系对其真菌的ITS2序列进行PCR扩增,将通过琼脂糖凝胶电泳检测的扩增产物寄往上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序。
1.3.2 生物信息学分析
参照GUO等[14]的方法将双端序列进行拼接并对序列进行质控,通过QIIME(v1.9.1)分析平台[15]和PyNAST软件[16]对序列进行对齐,使用UCLUST方法划分操作分类单元(operational taxonomic units,OTU)[17],并通过与UNITE数据库比对注释各分类学地位[18]。以各样品中测序深度最小的序列数为基准,对两类高温大曲的α多样性指数进行了计算,并基于非加权和加权UniFrac距离使用主坐标(principal coordinates analysis,PCoA)分析对其β多样性进行了解析,使用线性判别分析效应量(linear discriminant analysis effect size,LEfSe)甄别了机制曲和脚踩曲中显著富集的真菌菌群[19]。
1.3.3 多元统计学分析
分别对机制和脚踩的各29个高温大曲样品的真菌菌群进行了测序后,本研究分为机制曲和脚踩曲两组进行了对比分析。使用两独立样本的秩和检验分析(Mann-Whitney)和置换多因素方差分析(permutational multivariate analysis of variance,PERMANOVA)对机制曲和脚踩曲的差异性进行了分析[20];使用R软件(v4.1.2)绘制PCoA图和upset图;使用STAMP软件(v2.1.3)绘制秩和检验条形图;使用gephi软件(v0.9.2)绘制真菌属的共现网络图。
本研究采用Illumina MiSeq高通量测序技术对两类高温大曲中的真菌菌群结构进行了解析,通过97%相似度划分并进行同源性比对明确了各分类学地位后,发现每个机制曲中真菌菌群平均可以划分为1 325个OTU,并被鉴定为3个门下的22个属,而脚踩曲划分为2 337个OTU,并被鉴定为3个门下的16个属。基于α多样性对两类高温大曲的真菌菌群进行了比较分析,结果如图1所示。

a-超1指数;b-香农指数
图1 机制曲和脚踩曲样品的α多样性分析
Fig.1 α diversity analysis of high-temperature Daqu samples prepared by mechanism and foot stepping
注:超1指数和香农指数均在测序量为28 010条序列时计算所得;***代表P<0.001,**代表P<0.01(下同)。
由图1可知,平均每个机制曲和脚踩曲样品中真菌菌群的超1指数分别为1 423和2 546,香农指数分别为2.21和3.67。通过Mann-Whitney检验发现,较之脚踩曲,机制曲真菌菌群超1指数极显著偏低(P<0.001),香农指数非常显著偏低(P<0.01)。这说明与脚踩曲样品相比,机制曲所蕴含真菌菌群的丰富度和多样性均显著偏低(P<0.01)。
本研究进一步基于非加权和加权UniFrac距离对两类高温大曲中真菌菌群结构的差异性进行了分析,其中非加权分析是定性分析,仅考虑了样品中是否存在该物种,而加权分析是基于物种相对含量进行计算的,其结果如图2所示。
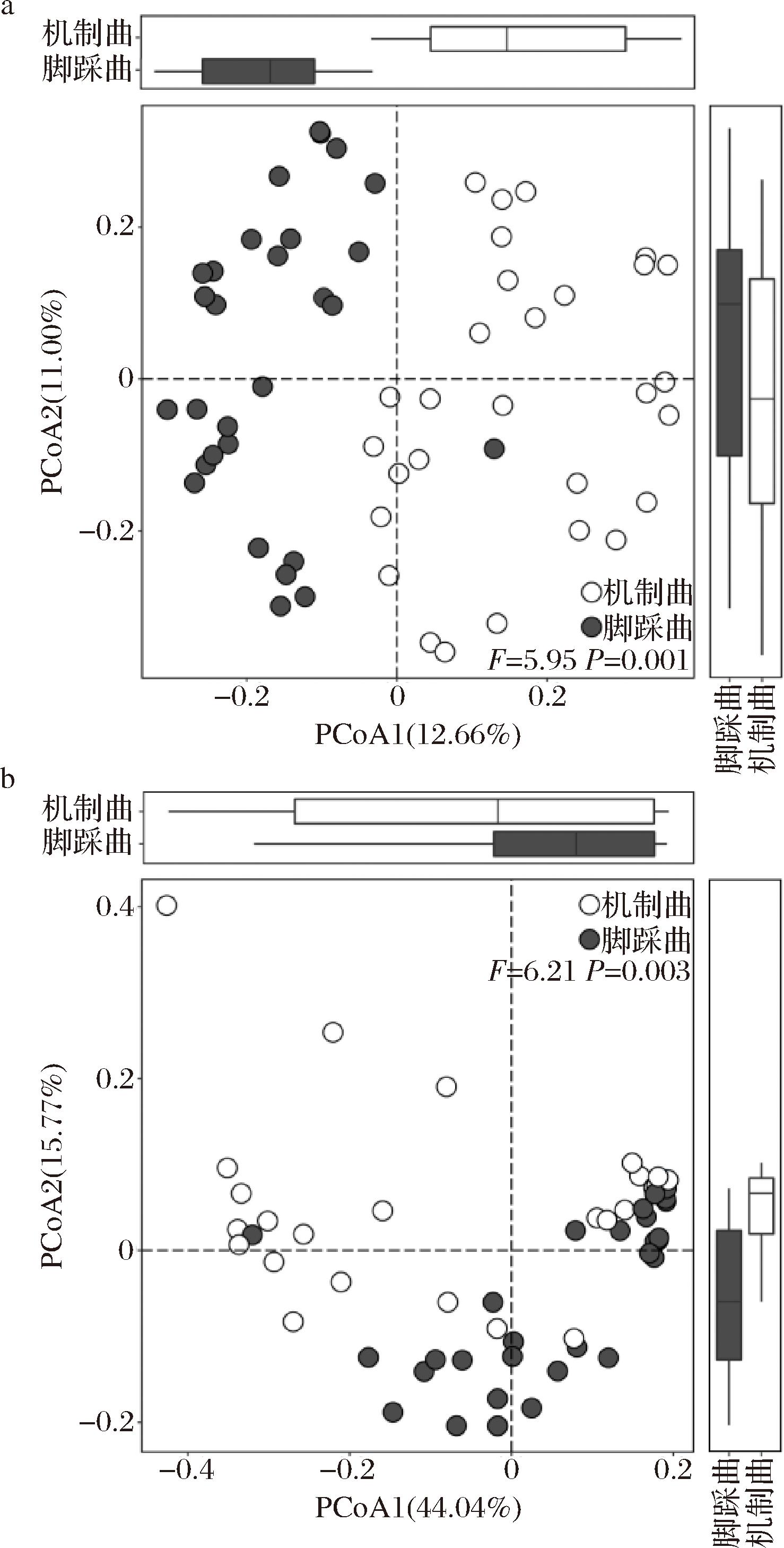
a-非加权;b-加权
图2 基于非加权和加权UniFrac距离的主坐标分析
Fig.2 Principal coordinate analysis based on unweighted and weighted UniFrac distance
由图2-a可知,仅考虑某一物种是否存在于样品中时,两类高温大曲样品呈现出明显的分离趋势。而由图2-b可知,进一步考虑物种的相对含量时发现,虽然两类高温大曲样品亦呈现出分离趋势,但该分离趋势较之图2-a较弱。此外,无论是否考虑物种的相对含量,PERMANOVA检验均发现两类高温大曲样品其真菌菌群的构成均存在非常显著差异(P<0.01)。由此可见,机制曲和脚踩曲真菌菌群存在显著差异,但这种差异主要体现在含量较低的物种上。
为甄别机制曲和脚踩曲中具有差异的真菌菌群,本研究对两类样品中平均相对含量大于1.0%的真菌门和属进行了对比分析,其秩和检验条形图如图3所示。
由图3-a可知,机制曲和脚踩曲中子囊菌门(Ascomycota)的平均相对含量分别为96.78%和97.24%,毛霉门(Mucoromycota)分别为3.34%和2.21%,经Mann-Whitney检验发现两个真菌门在两类样品中差异均不显著(P>0.05)。由图3-b可知,机制曲和脚踩曲中嗜热子囊菌属(Thermoascus)的平均相对含量分别为5.67%和27.82%,酵母共生菌属(Symbiotaphrina)分别为9.51%和0.02%,曲霉菌属(Aspergillus)分别为27.45%和14.25%,罗森氏菌属(Rasamsonia)分别为0.01%和1.47%,耐干霉菌属(Xeromyces)分别为3.52%和0.02%,嗜热毛霉属(Thermomucor)分别为2.89%和1.75%,嗜热真菌属(Thermomyces)分别为47.73%和50.63%。经Mann-Whitney检验发现,较之脚踩曲,机制曲样品中Symbiotaphrina和Xeromyces的含量均极显著偏高(P<0.001),而Thermoascus的含量极显著偏低(P<0.001),Rasamsonia的含量非常显著偏低(P<0.01)。
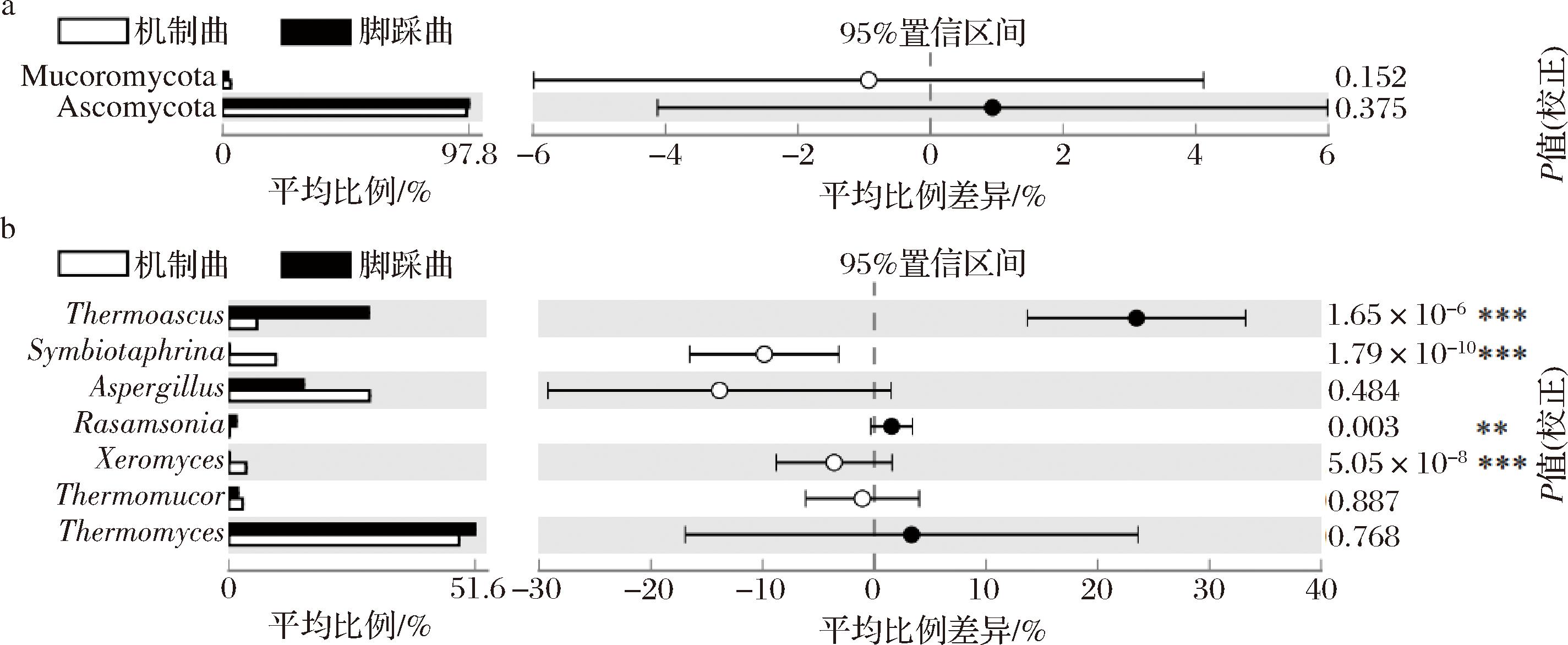
a-门水平;b-属水平
图3 机制曲和脚踩曲样品中平均相对含量大于1.0%的真菌门和属的秩和检验条形图
Fig.3 Rank-sum test bar chart of fungi and genera with average relative content of more than 1.0% in high-temperature Daqu samples were prepared by means of mechanism and foot stepping
虽然机制曲和脚踩曲样品的真菌菌群存在差异,但Thermomyces的含量均较高,DENG等[21]研究表明,高温大曲制作过程中长时间的高温阶段加剧了美拉德反应和焦糖化反应,导致高温大曲表面形成更多的黑色化合物,并且Thermomyces具有一定的耐热性,能够产生可降解碳水化合物的嗜热酶,提高高温大曲的酯化力和发酵力。作为纳入本研究的两类高温大曲样品中含量亦较为丰富的Aspergillus,ZENG等[22]研究表明Aspergillus可以产生蛋白酶、果胶分解酶和纤维素分解酶等,且其可能具有一定的耐热性,在高温固态发酵过程中可能有助于大曲中纤维素和蛋白质的水解。
本研究发现Thermoascus在脚踩高温大曲中含量显著偏高,GUAN等[23]得出类似的结论,其发现Thermoascus虽然在机械和传统大曲的整个贮藏期内均存在且平均相对含量均大于1.0%,但其在所有机械大曲样品中均以较低的丰度存在。本研究亦发现Rasamsonia在脚踩高温大曲中含量显著偏高,GUAN等[23]亦发现Rasamsonia仅在传统大曲中平均相对含量大于1.0%。此外,本研究发现酵母共生菌属(Symbiotaphrina)在机制曲和脚踩曲中平均相对含量分别为9.51%和0.02%,有研究指出其可为昆虫提供胆固醇、维生素和必需氨基酸,并且与有机酸、氨基酸和生物碱高度相关[24],然而对于其在大曲中存在的具体功能还有待进一步研究。罗方雯等[25]从白酒酿造环境和生产用高温大曲样品中均解析出Symbiotaphrina,且其在环境中的相对丰度高于大曲样本,由此可推测,大曲中Symbiotaphrina可能一部分是从环境中捕捉而来。
本研究进一步使用LEfSe分析对两类高温大曲的生物标志物进行了解析,结果如图4所示。

a-进化分支图;b-LDA分布直方图
图4 机制曲和脚踩曲样品中真菌类群的LEfSe分析
Fig.4 LEfSe analysis of fungi in high-temperature Daqu samples were prepared by means of mechanism and foot stepping
由图4-a可知,隶属于Xylonomycetes的Symbiotaphrinales是机制曲样品的生物标志物,隶属于散囊菌纲(Eurotiomycetes)的散囊菌目(Eurotiales)是脚踩曲样品的生物标志物。由图4-b可知,当LDA评分在3.5以上时,共检测到11个分类单元在机制曲和脚踩曲中具有显著性差异(P<0.05)。其中有3个真菌属在机制曲样品中显著富集(P<0.05),分别为Symbiotaphrina、Xeromyces和丝衣霉属(Byssochlamys),有2个真菌属在脚踩曲样品中显著富集(P<0.05),分别为Rasamsonia和Thermoascus。值得一提的是,作为机制曲中显著富集的Byssochlamys,其平均相对含量仅为0.90%,这进一步印证了机制曲和脚踩曲真菌菌群存在的差异主要体现在含量较低的物种上。Byssochlamys是大曲发酵过程中一个重要的属,拥有多种与风味相关的酶编码基因,并能降解原料中的淀粉或纤维素[26]。与本研究类似,ZHANG等[27]亦证明了Byssochlamys在机制大曲中的丰度显著高于脚踩大曲,并且由于Byssochlamys的生长Thermoascus的丰度呈降低的趋势。郭敏[28]采用LEfSe分析对贵州茅台的机制和传统高温大曲中显著富集的差异物种进行了解析,发现在真菌属水平上机械制曲中Saccharomycopsis为显著富集的差异物种,而传统制曲中无显著富集的差异物种,该研究与本研究不同的原因可能是由所采集样品的制曲环境和工艺等的差异所导致的。
在解析了机制曲和脚踩曲真菌菌群存在差异的基础上,本研究将在两类高温大曲的所有样品中均存在的OTU定义为核心OTU,并进一步基于OTU水平进行了比较分析,结果如图5所示。
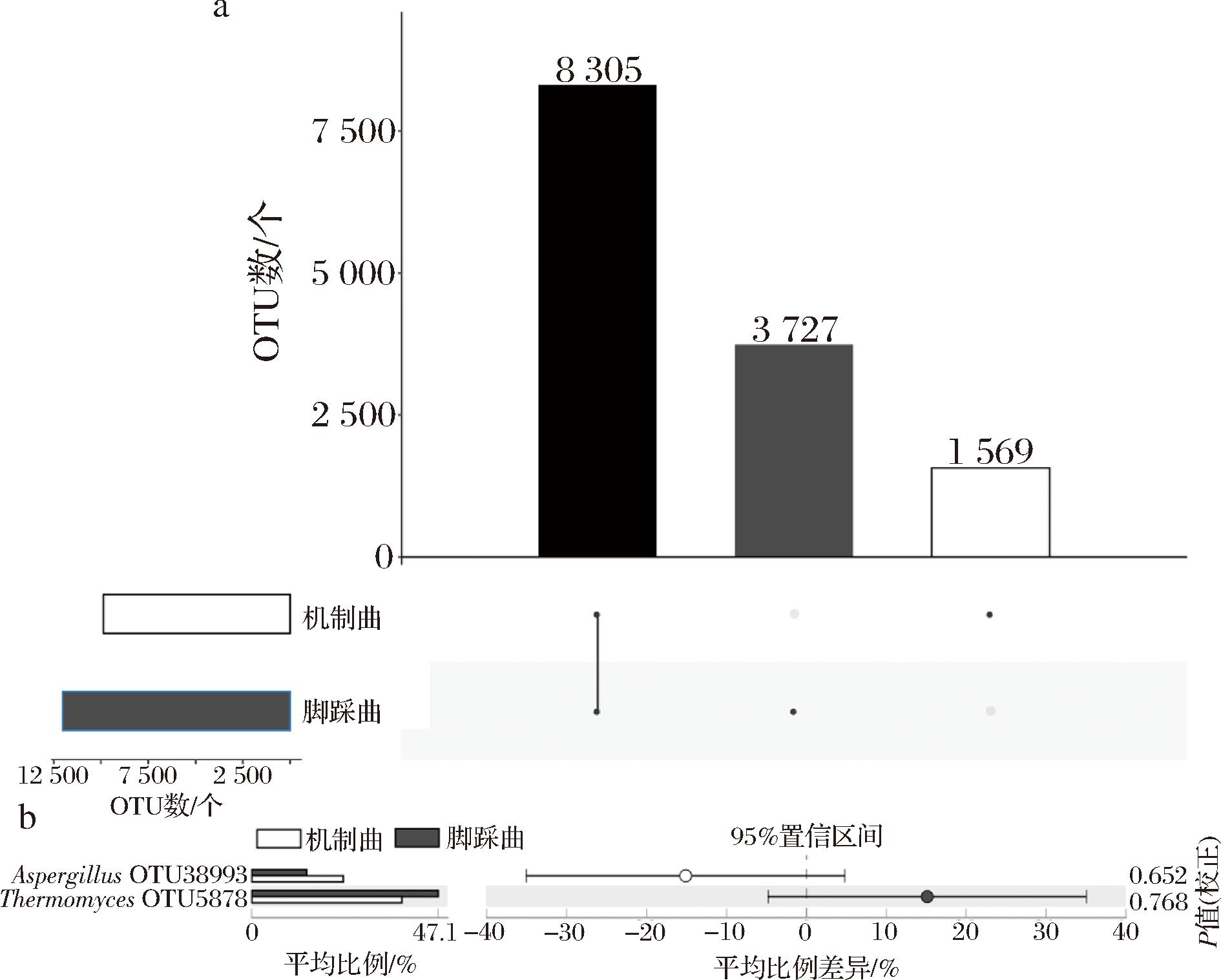
a-upset图;b-秩和检验条形图
图5 机制曲和脚踩曲真菌菌群中OTU的upset图及核心OTU的秩和检验条形图
Fig.5 Upset plot of OTUs and rank sum test bar chart of core OTUs in high-temperature Daqu fungal community prepared by means of mechanism and foot stepping
由图5-a可知,分别有1 569和3 727个OTU只在机制曲或脚踩曲真菌菌群中存在,但其包含的序列数较少,分别仅占总序列数的0.86%和1.58%。此外,有8 305个OTU同时存在于两类高温大曲的若干个样品中,占总序列数的97.56%。由图5-b可知,虽然仅有2个OTU在纳入本研究所有样品的真菌菌群中均存在,但它们所包含的序列数累计占比61.13%。本研究通过比对并对分类学地位进行了注释后发现,隶属于Aspergillus的OTU38993和隶属于Thermomyces的OTU5878,在机制曲中平均相对含量分别为20.80%和45.94%,在脚踩曲中平均相对含量分别为8.50%和47.04%,通过Mann-Whitney检验发现差异均不显著(P>0.05)。综上所述,虽然机制曲和脚踩曲中真菌菌群组成存在差异,但这种差异较小,有六成以上的真菌菌群共同存在于两类高温大曲的所有样品中,而仅有0.86%和1.58%的菌群分别单独存在于机制曲和脚踩曲中,这进一步印证了β多样性分析的结论。
生物共现网络可以用于解析物种之间的相互作用以及生态系统中微生物的稳定性[29],故而本研究在解析了机制曲和脚踩曲中真菌菌群结构的基础上,进一步通过共现网络图对两类高温大曲中真菌的相关性进行了分析,结果如图6所示。
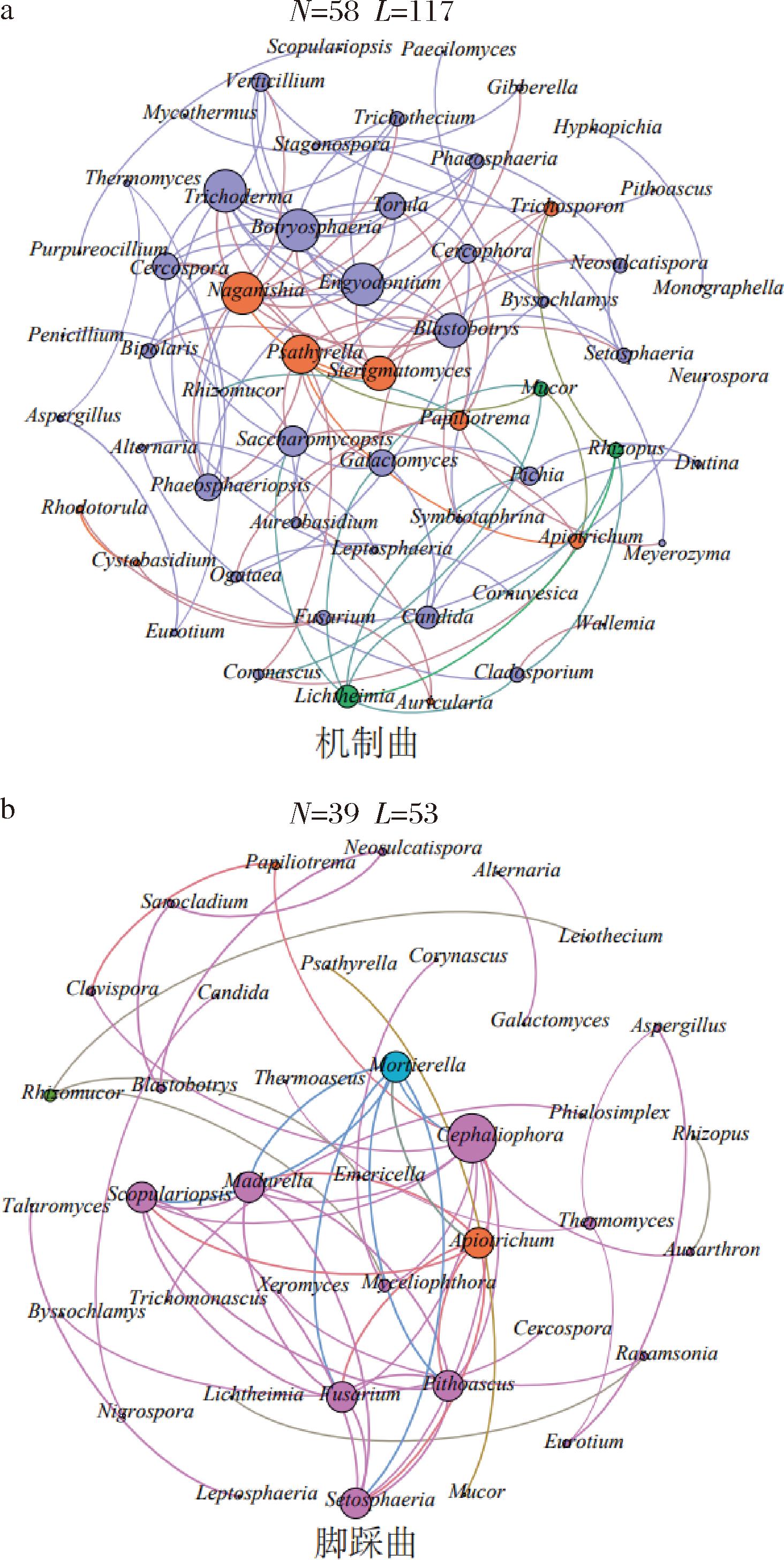
a-机制曲;b-脚踩曲
图6 机制曲和脚踩曲样品中真菌属的相关性分析
Fig.6 Correlation analysis of fungi in high-temperature Daqu samples were prepared by means of mechanism and foot stepping
由图6可知,机制曲中有58个属之间存在117个显著相关性(P<0.05),而脚踩曲中有39个属之间存在53个显著相关性(P<0.05)。为了评估共现网络的稳定性,本研究对网络中各节点的平均度进行了统计,结果发现机制曲和脚踩曲共现网络的平均度分别为4.034和2.718,这说明机制曲中构成网络的真菌类群之间关系更密切,并且网络的稳定性更高。由此可见,与脚踩曲相比,机制曲真菌属之间的相互作用更强且稳定性更高。
机制曲和脚踩曲的真菌菌群存在差异,但这种差异主要体现在低丰度的物种上。较之脚踩曲,机制曲中真菌菌群的丰富度和多样性均显著偏低,隶属于Thermoascus和Rasamsonia的菌株含量亦显著偏低,但隶属于Byssochlamys、Xeromyces和Symbiotaphrina的菌株更多。本研究亦发现机制曲中真菌属间的相互作用更强,稳定性更高。
[1] WANG Y R, GAI J S, HOU Q C, et al.Ultra-high-depth macrogenomic sequencing revealed differences in microbial composition and function between high temperature and medium-high temperature Daqu[J].World Journal of Microbiology &Biotechnology, 2023, 39(12):337.
[2] PANG Z M, LI W W, HAO J, et al.Correlational analysis of the physicochemical indexes, volatile flavor components, and microbial communities of high-temperature Daqu in the northern region of China[J].Foods, 2023, 12(2):326.
[3] DU H, WANG X S, ZHANG Y H, et al.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].International Journal of Food Microbiology, 2019, 297:32-40.
[4] 蒋思峡. 传统与机械化酱香大曲的菌群结构分析[D].贵阳:贵州大学, 2017.JIANG S X.The analysis of microfloras on traditional and mechanized Mao-tai flavor Daqu[D].Guiyang:Guizhou University, 2017.
[5] 左乾程, 黄永光, 郭敏, 等.酱香型白酒机械化制曲发酵细菌群落的演替[J].食品科学, 2021, 42(18):150-156.ZUO Q C, HUANG Y G, GUO M, et al.Succession of bacterial community during mechanized fermentation of Maotai-flavor Daqu[J].Food Science, 2021, 42(18):150-156.
[6] WANG Y R, CAI W C, WANG W P, et al.Analysis of microbial diversity and functional differences in different types of high-temperature Daqu[J].Food Science &Nutrition, 2020, 9(2):1003-1016.
[7] HUANG P, JIN Y, LIU M M, et al.Exploring the successions in microbial community and flavor of Daqu during fermentation produced by different pressing patterns[J].Foods, 2023, 12(13):2603.
[8] 侯强川, 王玉荣, 王文平, 等.茅台和尧治河高温大曲细菌群落结构差异及功能预测[J].食品与发酵工业, 2022, 48(1):36-44.HOU Q C, WANG Y R, WANG W P, et al.Difference of bacterial community structure and functional prediction in high-temperature Daqu of Maotai and Yaozhihe[J].Food and Fermentation Industries, 2022, 48(1):36-44.
[9] 郭世鑫, 姚孟琦, 马文瑞, 等.酱香型白酒的研究现状[J].中国酿造, 2021, 40(11):1-6.GUO S X, YAO M Q, MA W R, et al.Research status of sauce-flavor Baijiu[J].China Brewing, 2021, 40(11):1-6.
[10] REUTER J A, SPACEK D V, SNYDER M P.High-throughput sequencing technologies[J].Molecular Cell, 2015, 58(4):586-597.
[11] CAI W C, XUE Y A, WANG Y R, et al.The fungal communities and flavor profiles in different types of high-temperature Daqu as revealed by high-throughput sequencing and electronic senses[J].Frontiers in Microbiology, 2021, 12:784651.
[12] 杨少勇, 黎婷玉, 蔡文超, 等.襄阳地区高温大曲和中高温大曲真菌多样性解析[J].中国酿造, 2021, 40(5):76-80.YANG S Y, LI T Y, CAI W C, et al.Analysis of fungal diversity in high-temperature Daqu and medium-high-temperature Daqu from Xiangyang[J].China Brewing, 2021, 40(5):76-80.
[13] KANG J M, JIA L L, ZHANG Z X, et al.Comparison of physicochemical characteristics and microbiome profiles of low-temperature Daqu with and without adding Tartary buckwheat[J].Food Bioscience, 2022, 49:101931.
[14] GUO Z, WANG Y R, XIANG F S, et al.Bacterial diversity in pickled cowpea (Vigna unguiculata[linn.]walp) as determined by illumina MiSeq sequencing and culture-dependent methods[J].Current Microbiology, 2021, 78(4):1286-1297.
[15] ESTAKI M, JIANG L J, BOKULICH N A, et al.QIIME 2 enables comprehensive end-to-end analysis of diverse microbiome data and comparative studies with publicly available data[J].Current Protocols in Bioinformatics, 2020, 70(1):e100.
[16] GREGORY CAPORASO J, BITTINGER K, BUSHMAN F D, et al.PyNAST:A flexible tool for aligning sequences to a template alignment[J].Bioinformatics, 2010, 26(2):266-267.
[17] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics, 2010, 26(19):2460-2461.
[18] ABARENKOV K, HENRIK NILSSON R, LARSSON K H, et al.The UNITE database for molecular identification of fungi:Recent updates and future perspectives[J].New Phytologist, 2010, 186(2):281-285.
[19] LOZUPONE C, LLADSER M E, KNIGHTS D, et al.UniFrac:An effective distance metric for microbial community comparison[J].The ISME Journal, 2011, 5(2):169-172.
[20] TANG Z Z, CHEN G H, ALEKSEYENKO A V.PERMANOVA-S:Association test for microbial community composition that accommodates confounders and multiple distances[J].Bioinformatics, 2016, 32(17):2618-2625.
[21] DENG L, MAO X, LIU D, et al.Comparative analysis of physicochemical properties and microbial composition in high-temperature Daqu with different colors[J].Frontiers in Microbiology, 2020, 11:588117.
[22] ZENG Y, WANG Y, CHEN Q, et al.Dynamics of microbial community structure and enzyme activities during the solid-state fermentation of forgood Daqu:A starter of Chinese strong flavour Baijiu[J].Archives of Microbiology, 2022, 204(9):577.
[23] GUAN T W, YANG H, OU M Y, et al.Storage period affecting dynamic succession of microbiota and quality changes of strong-flavor Baijiu Daqu[J].LWT, 2021, 139:110544.
[24] LI X, ZHANG Y, YI Y L, et al.Revealing the effects of Moringa oleifera Lam.leaves addition on Fuzhuan Brick Tea by metabolomic and microbiota analysis[J].LWT, 2022, 156:113014.
[25] 罗方雯, 黄永光, 涂华彬, 等.基于高通量测序技术对茅台镇酱香白酒主酿区域酵母菌群结构多样性的解析[J].食品科学, 2020, 41(20):127-133.LUO F W, HUANG Y G, TU H B, et al.Analysis of the structure and diversity of yeast community in main Chinese Maotai-flavor liquor-producing areas of Maotai Town using high-throughput sequencing[J].Food Science, 2020, 41(20):127-133.
[26] YANG Y, WANG S T, LU Z M, et al.Metagenomics unveils microbial roles involved in metabolic network of flavor development in medium-temperature Daqu starter[J].Food Research International, 2021, 140:110037.
[27] ZHANG Y D, SHEN Y, CHENG W, et al.Understanding the shifts of microbial community and metabolite profile from wheat to mature Daqu[J].Frontiers in Microbiology, 2021, 12:714726.
[28] 郭敏. 基于高通量测序对酱香大曲制曲微生态多样性的研究[D].贵阳:贵州大学, 2018.GUO M.Study on micro-ecological diversity of Maotai-flavor Daqu based on high-throughput sequencing[D].Guiyang:Guizhou University, 2018.
[29] 万凌琳, 陈芷凡, 郭佳, 等.生物共现网络原理及其在淡水生态系统评估中的应用[J].湖泊科学, 2022, 34(6):1765-1789.WAN L L, CHEN Z F, GUO J, et al.Principle and application of co-occurrence networks for freshwater ecosystem assessment[J].Journal of Lake Sciences, 2022, 34(6):1765-1789.