植物乳植杆菌转录调控子YesN对蔗糖代谢及发酵香肠风味形成的影响
强艺1,张大革1,2,徐广谦1,2,徐文生1,2*
1(北京农学院 食品科学与工程学院,北京,102206)2(农产品有害微生物及农残安全检测与控制北京市重点实验室,北京,102206)
摘 要 该研究旨在揭示植物乳植杆菌转录调控子YesN调控蔗糖代谢的机理,并评估其在发酵香肠生产中对挥发性风味物质和生物胺生成的影响。以植物乳植杆菌yesN突变菌株(ΔyesN)为研究对象,采用转录组测序技术对比野生菌株在基础培养基中的差异表达基因;通过凝胶迁移实验(electrophoretic mobility shift assay,EMSA)和细菌单杂交技术,验证YesN对蔗糖代谢基因簇启动子PscrB的调控作用;此外,利用这2种菌株作为发酵剂制备香肠,分析基因yesN对发酵香肠中挥发性风味物和生物胺生成的影响。转录组分析发现ΔyesN菌株与野生菌株相比,有491个基因表达差异,其中109个基因表达下调,382个基因表达上调。KEGG富集分析显示,上调基因主要涉及核酸和氨基酸代谢,而下调基因主要涉及碳源代谢和酯类代谢。在以突变菌株和野生菌株为发酵剂制备的香肠中,检测到14种差异挥发性风味物质,包括7种酯类、5种醛类和2种醇类。结果表明转录调控子YesN在植物乳杆菌中对蔗糖代谢基因簇具有显著的调控作用,并且其在发酵香肠生产中对风味物质和生物胺的生成具有重要影响。
关键词 植物乳植杆菌;蔗糖代谢;转录调控子;转录组分析
DOI:10.13995/j.cnki.11-1802/ts.040655
引用格式:强艺,张大革,徐广谦,等.植物乳植杆菌转录调控子YesN对蔗糖代谢及发酵香肠风味形成的影响[J].食品与发酵工业,2025,51(14):320-326.
QIANG Yi,ZHANG Dage,XU Guangqian, et al.Research on impact of transcriptional regulator YesN in Lactiplantibacillus plantarum on sucrose metabolism and flavor formation in fermented sausages[J].Food and Fermentation Industries,2025,51(14):320-326.
第一作者:本科生(徐文生副教授为通信作者,E-mail:xuwinsh@bue.edu.cn)
基金项目:北京市教委面上项目(KM201610020016)
收稿日期:2024-08-05,改回日期:2024-10-30
植物乳植杆菌(Lactiplantibacillus plantarum)是一种常见于植物发酵原料的乳杆菌,以其环境适应性和碳源同化性强等特点而闻名。该菌不但在食品、饲料工业中广泛应用,也作为益生元添加于保健品中。此外,植物乳植杆菌天然存在于人的唾液和胃肠道中,可维持菌群平衡,提高免疫力,促进营养吸收,降低胆固醇,缓解乳糖不耐受等功能[1],具有较高的开发利用价值。
植物乳植杆菌具有广泛的生态分布和栖息能力,与其强大的碳水化合物利用能力密切相关。相对于其他乳酸菌,植物乳植杆菌具有非常高的遗传多样性和更强的碳水化合物利用能力,能够利用的碳源不仅包括蔗糖、果糖等单双糖,还包括纤维二糖、甘露糖、D-核糖、L-岩藻糖、低聚果糖和低聚半乳糖等多糖[2]。这与植物乳植杆菌基因组相对较大、基因数量远超其他类似菌株并存在大量和糖代谢相关基因有关[3]。
众所周知,细菌会通过细胞精确调控来减少代谢负担,其中乳酸菌碳源代谢全局调控机制主要由代谢控制蛋白A(catabolite control protein A,CcpA)、双组分系统(two-component systems,TCSs)等介导。双组分系统是细菌为适应复杂、多变的环境而发展、进化出的一种信号传导系统,促进细菌感应和应答环境刺激。典型的双组分系统由感应组氨酸激酶和转录调控子组成。对植物乳植杆菌基因组分析表明,该菌中双组分系统的含量高于其他乳酸菌。虽然有一些双组分系统如AgrC/AgrA、PlnB/PlnC和PleC/PleD等的功能已经被解析,但包括YesM/YesN在内的许多双组分系统的功能仍然有待进一步研究[4]。本课题组前期工作表明,缺失yesN会影响植物乳植杆菌蔗糖代谢相关基因簇的表达[5],但其机理仍然有待进一步研究。为了进一步阐明yesN对植物乳植杆菌蔗糖代谢调控机制,本文通过细菌单杂交系统和EMSA来验证YesN对蔗糖代谢的调控作用。同时通过转录组测序来研究yesN基因突变对植物乳植杆菌蔗糖代谢的影响。
1 材料与方法
1.1 材料与试剂
植物乳植杆菌野生菌株WT和突变菌株ΔyesN为实验室保存;MRS肉汤、MRS琼脂、LB肉汤、LB琼脂培养基,北京奥博星公司;基因组DNA提取试剂盒、总RNA提取试剂盒,美国Promega公司。
乙腈、甲酸、三氯乙酸、盐酸、氢氧化钠、色胺、精胺、苯乙胺、酪胺、组胺、亚精胺、腐胺、蛋白胨、蔗糖、氯霉素、链霉素、卡那霉素、正丁醇、三氯甲烷,国药集团化学试剂公司。
1.2 仪器与设备
梯度PCR仪,美国MJ公司;高速冷冻离心机,德国Eppendorf公司;核酸电泳仪PowerPac HC,美国伯乐公司;气质联用仪Agilent5957C-7980A、高效液相色谱1260、高效液相质谱联用仪Agilent 6410B,美国安捷伦公司。
1.3 实验方法
1.3.1 乳酸菌的培养与菌体的获得
植物乳植杆菌基因yesN缺失突变菌株的构建方法参见前文[5]。植物乳植杆菌野生菌株WT和突变菌株ΔyesN的常规培养均在MRS培养基中37 ℃静置培养。如进行转录组分析,则细胞培养在15%蛋白胨+蔗糖作为碳源的培养基中,蔗糖通过0.2 μm的滤膜过滤除菌后添加到灭菌的培养基中。
过夜培养的植物乳植杆菌按1%的菌液量接种到500 mL基础培养基补充过滤灭菌的蔗糖的培养基中,37 ℃静置培养9~10 h使菌体达到对数生长期,当OD600值达到0.3时,菌体通过4 ℃,8 000 r/min离心5 min收集菌体,收集的菌体置于液氮中快速冷却用于RNA提取,上清液通过0.22 μm的滤膜过滤,采用高效液相色谱检测乳酸。
1.3.2 生物胺检测
准确称取10.00 g香肠匀浆置于100 mL具塞锥形瓶中,加入20 mL 50 g/L三氯乙酸溶液,混匀,振荡提取60 min,转移至50 mL离心管中,8 000 r/min 4 ℃离心10 min,取上清液置50 mL容量瓶中,连续提取2次,合并上清液,用50 g/L三氯乙酸稀释至刻度,滤纸过滤。
移取10.00 mL滤液置于25 mL具塞试管中,加入10 mL正已烷涡旋振荡5 min,弃去上层有机相,重复进行2次。将上述除脂肪后溶液加入适量氯化钠使溶液饱和。准确移取上述饱和后的试样提取液5.00 mL,置于20 mL离心管中,用0.1 mol/L氢氧化钠溶液调节pH至12.0。加入5.0 mL的正丁醇-三氯甲烷(1∶1,体积比)混合溶液,涡旋振荡5 min,8 000 r/min离心10 min,吸取上层有机相,再重复萃取2次,最后一步萃取用分液漏斗分离,合并萃取液,混匀,取3.0 mL萃取液并加入0.2 mL 1 mol/L盐酸,混合后40 ℃水浴下氮气吹干,加入乙腈溶解。
柱温:35 ℃;流速:0.3 mL/min;流动相A:5 mmol/L乙酸铵-甲酸水,流动相B:乙腈,进样体积:5 μL,洗脱程序:0~7.50 min,95%A~70% A,7.50~10.00 min,70%A~10% A,10.00~12.00 min 10% A。质谱离子源:电喷雾离子源(electrospray ionization,ESI),正离子扫描,多反应离子监测(multiple reaction monitoring,MRM)。
1.3.3 电泳迁移率实验(electrophoretic mobility shift assay,EMSA)
采用5端标记cy5荧光的引物扩增蔗糖利用基因簇基因簇启动子(Pscr)区域,获得250~300 bp左右的cy5-DNA片段。在1~8号PCR管中分别加入20 ng cy5-DNA片段,200 ng poly(dI-dC),(0、0.25、0.5、0.75、1、1、1、1 μmol/L)磷酸化His6-YesN和竞争性DNA(0、0、0、0、0、100、500、1 000 ng),在25 ℃条件下温浴30 min后,冰浴电压100 V电泳2 h,注意全程避光,电泳结束后用Typhoon FLA9500扫描并拍照。
1.3.4 细菌单杂交
采用细菌单杂交系统在Escherichia coli中检测DNA-蛋白质间的相互作用。分别克隆yesN及其启动子片段pBXcmT和pTRG载体,质粒对pBXcmT-PscrB/pTRG-yesN作为阳性对照,采用pBXcmT-PscrBΔRBS I/Ⅱ作为阴性对照,并导入XL1Blue MRF感受态细胞构建共转化子,将共转化子在30 ℃ 200 r/min过夜培养后,将过夜培养的共转化子菌液浓度调整到OD600=1并10倍梯度稀释,分别取5个梯度稀释的菌液涂布在添加或不添加3-氨基-1,2,4-三氮唑(3-amino-1,2,4-triazole, 3-AT)的含有50 μg/mL卡那霉素、25 μg/mL氯霉素及12.5 μg/mL链霉素的LB培养基中30 ℃培养12 h后每隔8 h观察记录筛选平板的生长情况直至72 h。阳性相互作用的转化子接种到含有3-AT和12.5 μg/mL链霉素的平板中确认。
1.3.5 总RNA的提取
离心收集的菌体用无菌去离子水(ddH2O)洗2遍。操作依据厂家操作规程进行总RNA的提取,最终总RNA溶解到200 μL无RNase ddH2O中。用紫外吸收法检测RNA的浓度和纯度。质检合格的样品送上海生工科技有限公司进行转录组学测序,每个样品设2个平行。
使用Bowtie2通过默认参数将样本有效数据对比到参考基因组上,统计序列对比信息。使用featureCounts和已知基因模型评估基因的表达量并使用WGCNA进行基因共表达分析。使用DESeq2进行基因表达差异分析;使用topGO进行GO富集分析,使用clusterProfiler进行KEGG通路和COG分类富集分析并基于基因功能富集分析结果。
1.3.6 中式发酵香肠制备及其挥发性风味物的检测
将瘦肉、肥肉切丁后按3∶1的质量比混合,并加入适当的辅料充分搅拌混匀后分成2等份,分别以植物乳植杆菌野生菌株WT和突变菌株ΔyesN为发酵剂制备香肠发酵20 d。取30.0 g剪碎的发酵香肠装入100 mL样品瓶中80 ℃水浴30 min后采用顶空吸附固相微萃取联合气质联用检测挥发性风味物质[6]。
2 结果与分析
2.1 植物乳植杆菌野生菌株WT和突变菌株ΔyesN发酵特性
植物乳植杆菌野生菌株WT和突变菌株ΔyesN均生长在补充了蔗糖的基础培养基中,乳酸生产及其在发酵香肠中生物胺的生成结果见表1。
表1 植物乳杆菌WT和突变菌株ΔyesN乳酸、生物胺生成结果
Table 1 Results of lactic acid and biogenic amine production in Lactobacillus plantarum WT and mutant strain ΔyesN
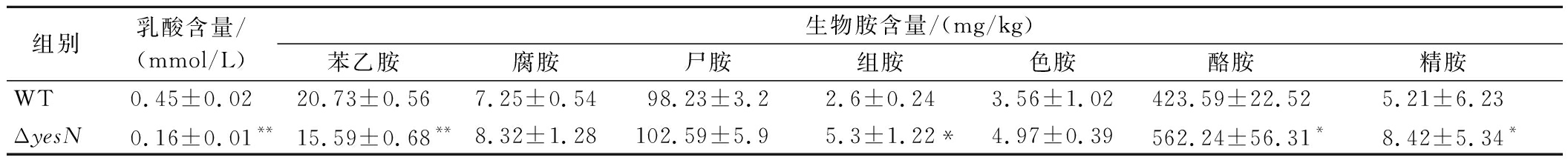
组别乳酸含量/(mmol/L)生物胺含量/(mg/kg)苯乙胺腐胺尸胺组胺色胺酪胺精胺WT0.45±0.0220.73±0.567.25±0.5498.23±3.22.6±0.243.56±1.02423.59±22.525.21±6.23ΔyesN0.16±0.01**15.59±0.68**8.32±1.28102.59±5.95.3±1.22*4.97±0.39562.24±56.31*8.42±5.34*
注:**表示数据差异极显著(P<0.01),*表示差异显著(P<0.05)。
乳酸生成方面,相对于菌株WT,ΔyesN菌株的乳酸生成能力较弱。而应用于生产发酵香肠结果显示,ΔyesN菌株生产的发酵香肠中除苯乙胺显著低于(P<0.01)WT菌株外,组胺、酪胺和精胺的生成量均显著高于WT菌株(P<0.05)。两者腐胺、尸胺、色胺的生成没有显著差异(P>0.05)。
2.2 细菌单杂交系统检测YesN调控scr基因簇启动子
细菌单杂交系统可以检测蛋白质与DNA间的相互作用,从而确定植物乳杆菌转录调控子YesN与蔗糖利用基因簇scr启动子间的相互作用。利用该系统将含有潜在scrB启动子的254 bp片段作为诱饵DNA片段,导入到含肠杆菌组氨酸合成酶基因his及链霉素抗性基因aadA的载体pBXcmT。同时,本研究将yesN基因的表达框与细菌双杂交系统的pTRG质粒上的RNA聚合酶(RNAP)α-亚基融合,这对重组载体共同转化到宿主菌XL1Blue大肠杆菌感受态细胞。如果宿主菌中表达的YesN能与靶DNA相互作用,那么RNA聚合酶α亚基将招募RNA聚合酶到启动子,使下游基因表达,合成组氨酸合成酶和链霉素抗性基因产物,并在含化合物3-AT和链霉素的筛选平板上正常生长。结果见图1,只有yesN/PscrB菌株和阳性菌株能在3-AT和Str的LB平皿上生长,证明YesN 能与蔗糖利用基因簇启动子DNA相互作用。
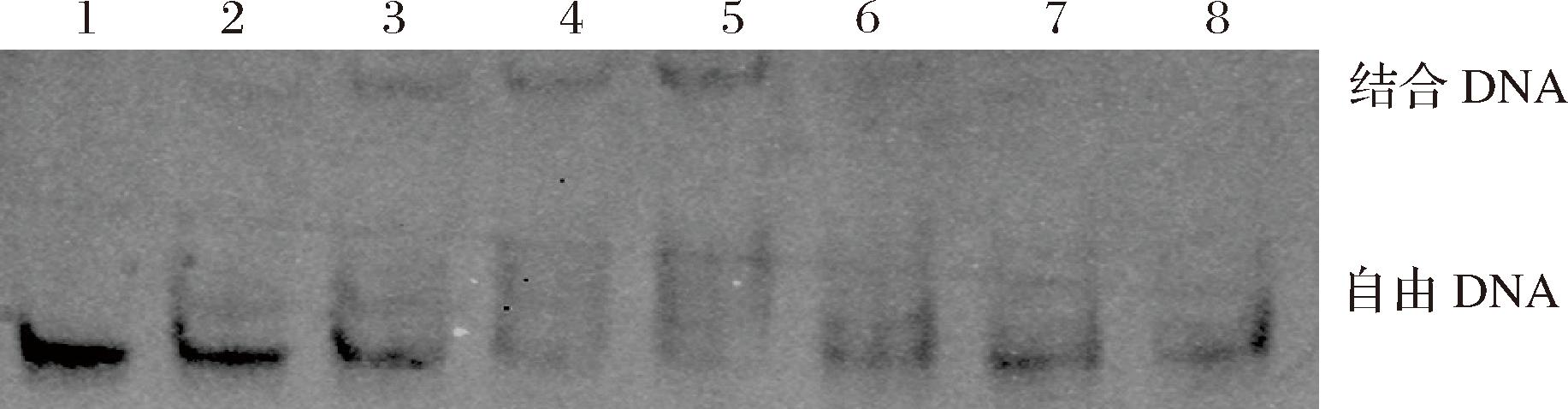
为了进一步验证转录调控子YesN与蔗糖代谢基因簇启动子PscrB的特异性结合作用,通过EMSA验证YesN和启动子PscrB的结合作用。结果见图2,在具有高浓度(10倍)非特异性竞争片段poly(dI:dC)存在的情况下,YesN能够特异性地结合到带有cy5荧光标记的scrB启动子片段上,随着YesN蛋白浓度的提高,启动子片段的阻滞作用越明显。由此证明YesN和能与启动子PscrB结合。
2.3 转录组基本特征
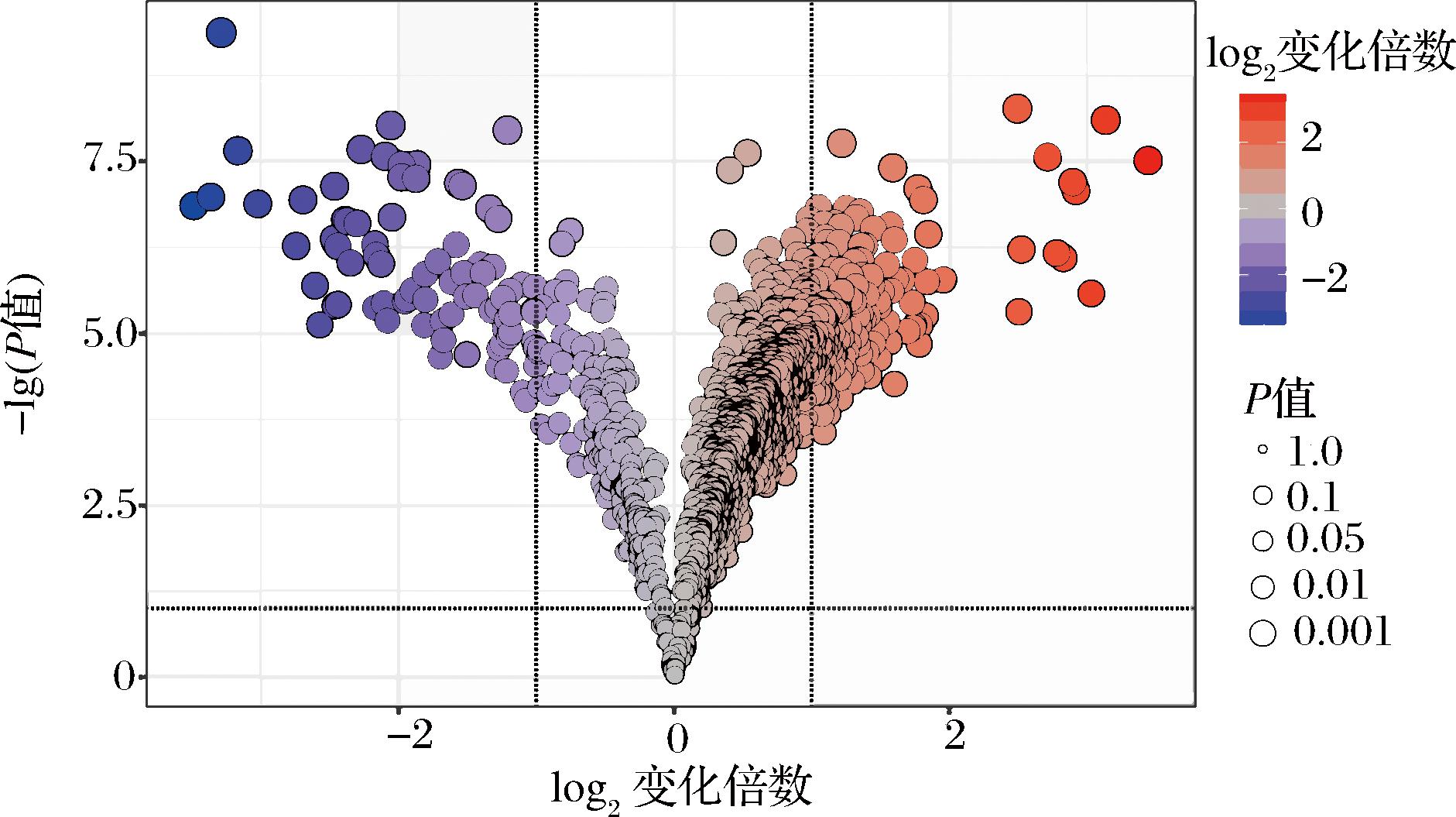
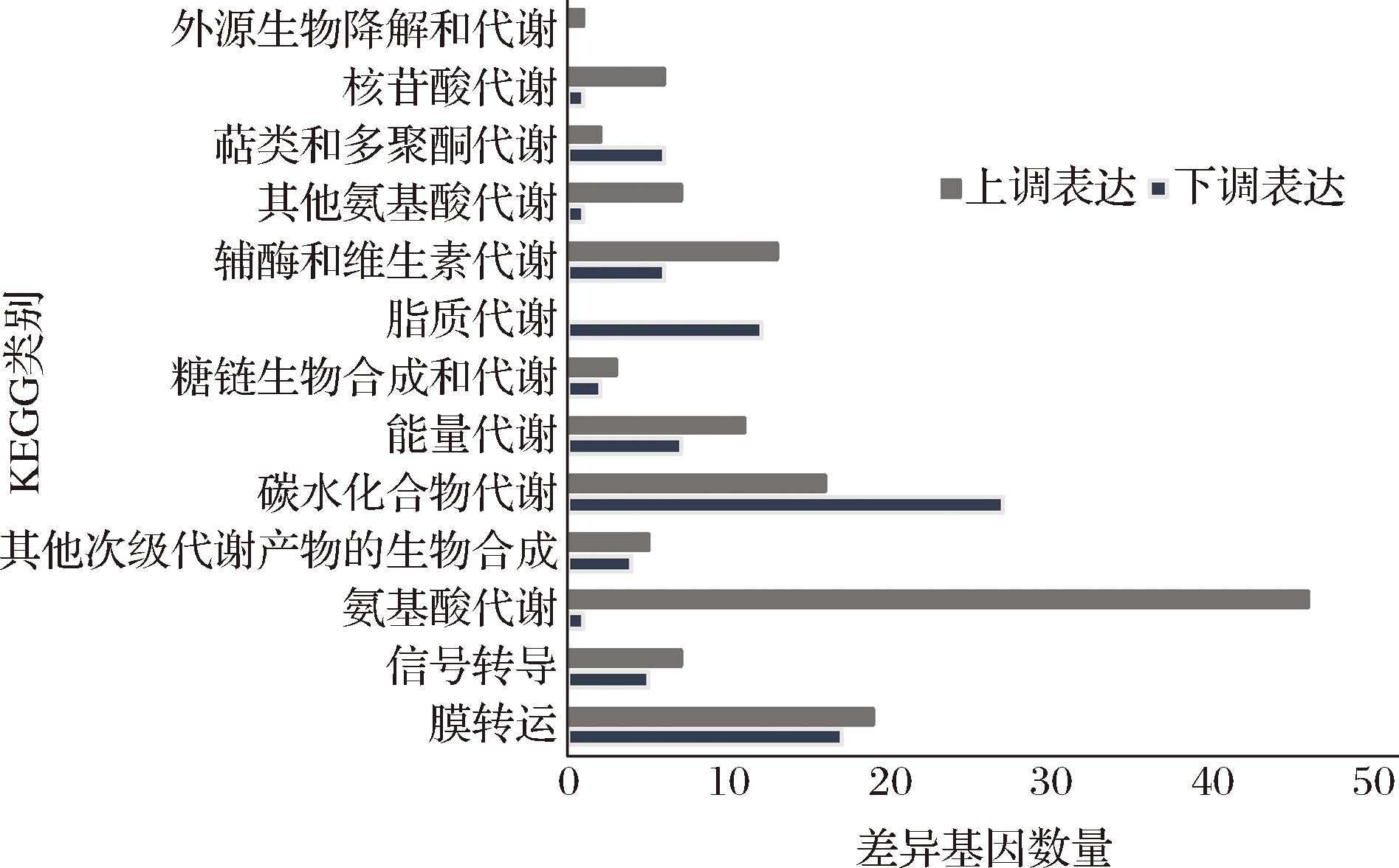
通过转录组测序比较植物乳植杆菌野生菌株和变异菌株基因表达情况,发现总共有491个基因差异表达基因。其中109个基因下调表达,382个基因上调表达(图3)。其中下调表达的基因主要涉及磷酸转移酶系统(phosphotransferase system,PTS),脂肪酸代谢,碳水化合物代谢等;而上调表达基因主要涉及氨基酸代谢、全局调控因子σ70,乳酸合成、己糖单磷酸途径(hexose monophosphate pathway,HMP)、涉及苹果酸和柠檬酸调控的双组分系统等(图4)。
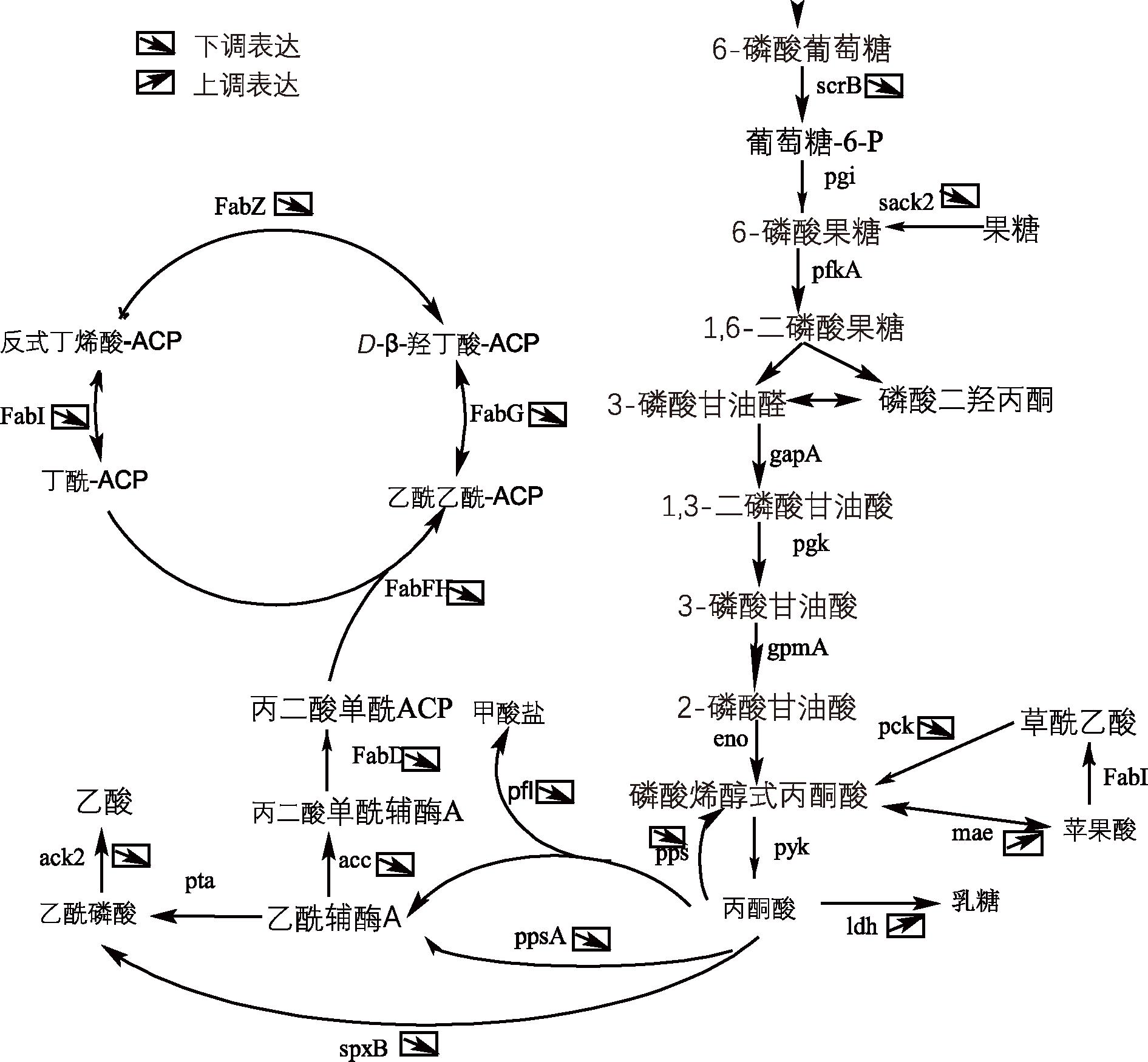
在突变菌株ΔyesN中,涉及全局调控因子σ70为上调表达,而与野生菌株相比碳源代谢相关的全局调控蛋白CcpA的表达则无差异。在中心碳源代谢途径中,丙酮酸代谢主要涉及的基因如ldh、pfl、pox、ack等,除ldh上调表达外,其余均下调表达(图5)。植物乳植杆菌可以通过HMP途径利用其他中间代谢产物替代产生细菌生长所需要的能量和NADH等还原力,突变菌株ΔyesN中HMP途径相关的2个关键的酶基因-转酮酶基因tkt和转醛酶基因tal均上调表达,而且与一碳代谢相关的酶编码也上调表达。植物乳植杆菌中与苹果酸利用相关的基因如苹果酸组氨酸激酶基因malK,转化苹果酸为丙酮酸的苹果酸酶基因mae等均上调表达。
糖类的转运系统主要有PTS转运蛋白和ABC(ATP-binding cassette,ABC)转运蛋白,PTS转运蛋白利用磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)作为磷酸基团的供体,通过催化磷酸基团转移来实现糖的磷酸化活化和转运。PTS主要参与葡萄糖、果糖、乳糖、甘露糖、半乳糖、山梨糖等单、双糖类的磷酸化转运;结果表明涉及单双糖转运PTS转运蛋白和多糖摄取的ABC转运蛋白均表达下调。
与糖类相关的转运系统受到抑制不同,氨基酸代谢相关基因表达显著上调(图4)。涉及氨基酸转运的ABC转运蛋白(如支链氨基酸摄取基因livA、livC、livD),涉及多肽摄取的转运蛋白(E2P15_15840-E2P15_15860,E2P15_00830-E2P15_00835);涉及氨基酸代谢的操纵子(如色氨酸合成相关的操纵子ser、精氨酸合成的操纵子arg、支链氨基酸合成的关键酶支链氨基酸转氨酶基因bcaT、蛋氨酸合成的关键酶基因metE、组氨酸合成操纵子his等)和与生物胺降解有关的多铜氧化酶基因mco等均上调表达。
2.4 发酵香肠挥发性风味分析
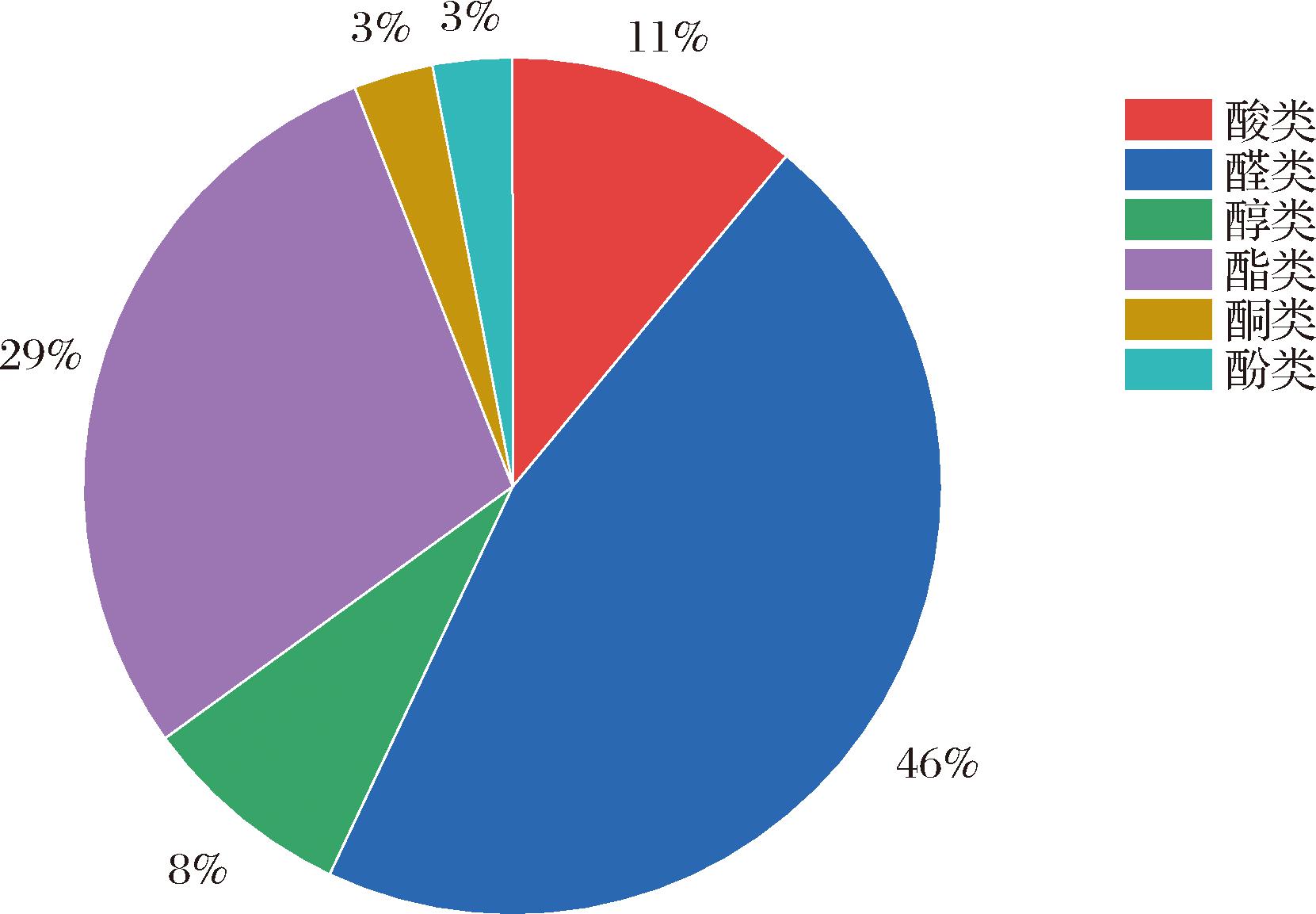
分别采用植物乳植杆菌野生菌株WT和突变菌株ΔyesN为发酵剂制备香肠,对2种样品的挥发性风味进行检测,共检测出35种挥发性风味。如图6所示,按照结构特征可以将其分为酸类(4种)、醛类(16种)、醇类(3种)、酯类(10种)、酮类(1种)、酚类(1种)等。
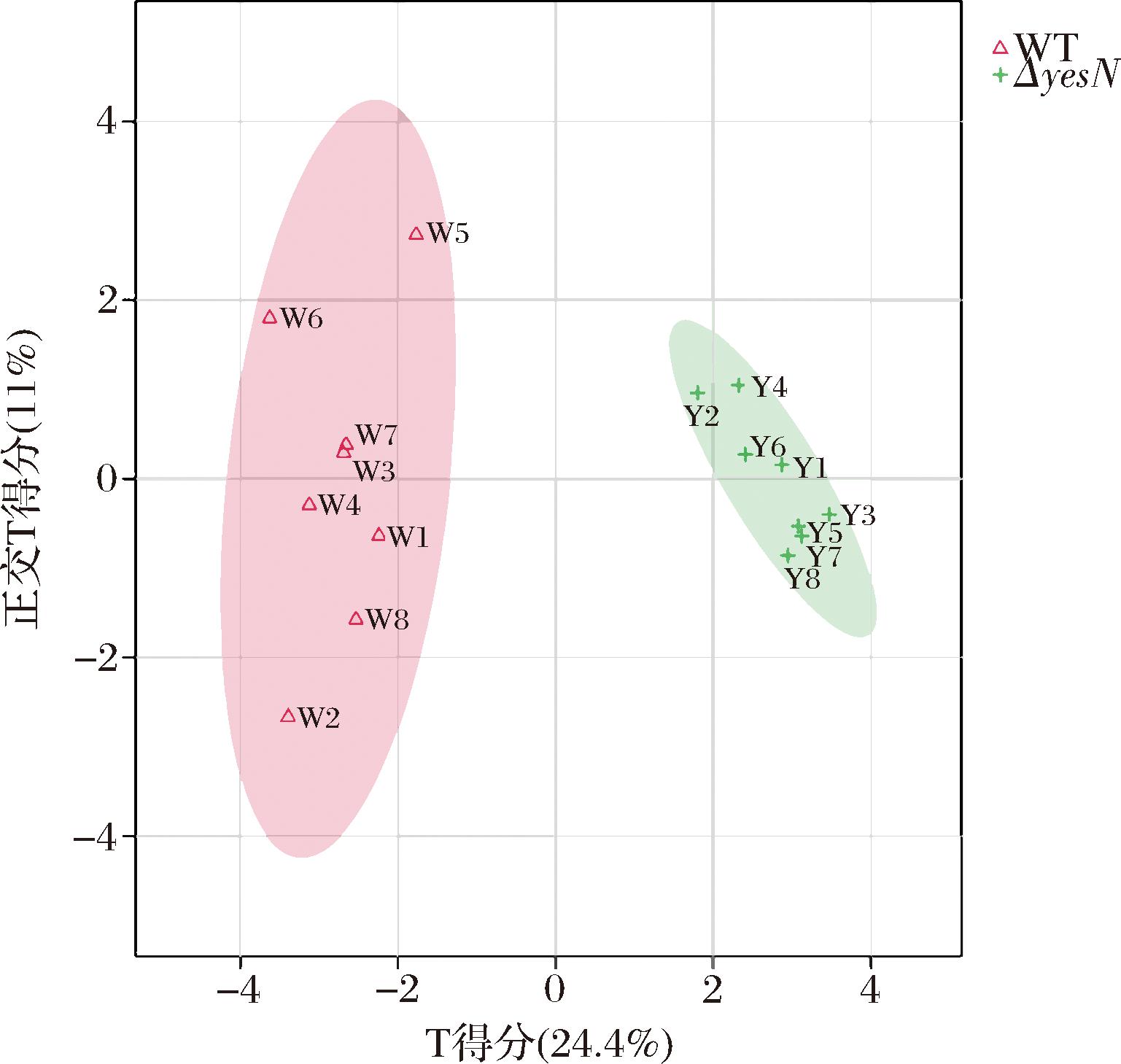
为了研究以植物乳植杆菌野生菌株WT和突变菌株ΔyesN为发酵剂发酵香肠挥发性物质表达量和样品类别间的差异程度,采用有监督的正交偏最小二乘判别分析(orthogonal partial least squares discriminant analysis,OPLS-DA)对2种发酵香肠的挥发性风味物进行分析。结果如图7所示,香肠挥发性风味物质具有很好的聚类效果,说明不同发酵剂对发酵香肠挥发性风味影响较大。对OPLS-DA模型进行置换检验,其R2Y=0.959,Q2=0.752,均高于0.5且差值小于0.3,说明该模型具有良好的准确性、预测和解释能力,并且不存在过拟合的现象。
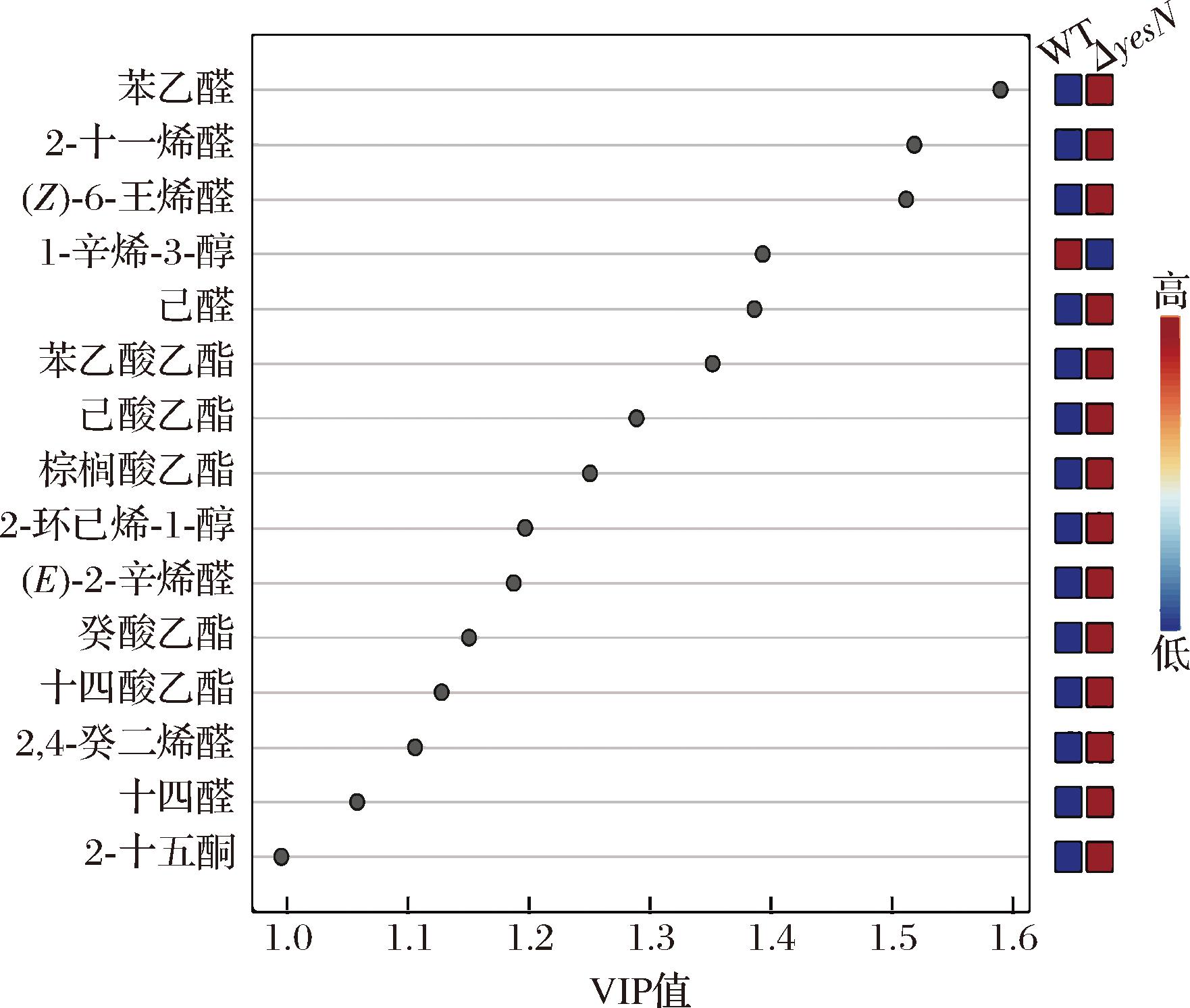
VIP值(variable importance in projection)可以反映不同风味物质对模型分类的贡献度。基于OPLS-DA模型,以VIP>1作为筛选差异风味物质的标准。结果见图8,共筛选出14种差异挥发性风味物质,其中包括7种酯类,5种醛类和2种醇类。
3 结论与讨论
葡萄糖、蔗糖等碳源是微生物偏好使用的碳源,当这些碳源的利用受到限制时,微生物的生长和代谢产物的形成可能会受到显著影响[5, 7]。在本研究中,观察到yesN突变菌株在乳酸生成方面显著下降(P<0.01)。同时对比了野生型(WT)菌株和ΔyesN突变菌株在香肠生产中的应用。结果显示,与WT菌株相比,ΔyesN突变菌株在发酵过程中除苯乙胺含量显著降低外(P<0.01),组胺、酪胺和精胺的含量均显著增加(P<0.05)。在发酵香肠的生产过程中,生物胺的积累与香肠中存在的乳杆菌属(Lactobacillus)和肠球菌属(Enterococcus)等微生物的活动密切相关[8]。而植物乳植杆菌中存在的多铜氧化酶McO能够降解酪胺、色胺等多种生物胺,从而有助于降低酒类和发酵肉制品中生物胺的总含量[9]。
调控糖代谢的双组分系统在细菌中普遍存在,如双组分系统SaeRS调控表皮葡萄球菌的葡萄糖代谢[10];产气荚膜梭菌中麦芽糖的代谢受到双组分系统MalNO的调控[11];在贝氏梭菌中D-木糖的感知和转运依赖双组分系统LytS/YesN[12]等。转录调控子YesN在细菌中广泛分布,一般认为其功能与碳源代谢调控相关[13],如在枯草芽孢杆菌中双组分系统yesM/N参与果胶和鼠李半乳糖醛酸的降解[14];在Paenibacillus sp.JDR-2中yesM/yesN双组分系统调控木聚糖的降解[15];在植物乳植杆菌中YesN对蔗糖代谢相关基因簇的表达有调控作用[5]。本研究通过细菌单杂交系统和EMSA验证转录调控子YesN与蔗糖代谢基因簇启动子PscrB直接作用证实了YesN具有调控植物乳植杆菌蔗糖代谢基因簇的功能。
环境中的细菌必须能够快速适应环境的变化。当环境中首选碳源缺乏时,细菌主要通过全局性调控因子调控细菌加强次生碳源的分解代谢存活。这些全局性调控因子主要包括CcpA、σ因子等。σ因子家族的的主要功能有指导基因转录、启动胁迫应激反应以及辅助代谢等,在碳源饥饿、高酸、高盐等胁迫条件下,σ70可调节相关基因的表达来响应外界环境的改变,指导细胞的代谢反应以增强微生物的生存能力[16]。CcpA是乳酸菌最重要的、高度保守的碳源代谢抑制蛋白,调控许多重要的细胞代谢,在有适合碳源存在的情况下,CcpA激活糖PTS转运蛋白摄入环境中的糖[7]。对链球菌的研究表明:CcpA能够介导ldh、pfl、ack等基因的表达,增加乳酸的产量,敲除ccpA的菌株会显著增加乙酸和甲酸的产量而显著减少乳酸的产量[17]。
细菌转运蛋白主要包括PTS转运蛋白和ABC转运蛋白2种,PTS转运蛋白对单双糖具有较高的选择性,而ABC转运蛋白则更多参与寡糖的转运[18]。目前在植物乳杆菌中已知的涉及蔗糖摄取的PTS主要有ScrA和PTS26BCA[19]。ScrA被认为是植物乳杆菌中一种受CcpA和YesN调控、参与蔗糖和/或果聚寡糖(fructooligosaccharides,FOS)代谢的胞内酶。当葡萄糖等碳水化合物高水平存在的情况下,糖代谢相关PTS转运蛋白上调表达,乳酸代谢相关基因如pdh, pfl, pox甚至ack基因的上调表达,以便细菌通过糖酵解途径消耗大量能量来产生乳酸、乙酸等终产物以维持菌体的生长[17]。
在碳水化合物饥饿或酸性条件时,磷酸转移酶(phosphotransferase,PTS)表达下调,细菌的碳水化合物摄入受到抑制,部分ABC转运蛋白基因的表达上调,细菌加强次级碳源的代谢[20]。在糖饥饿时,植物乳杆菌一方面上调表达HMP途径的2个关键酶(转酮醇酶和转醛醇酶)基因的表达,增加对一碳到七碳相关代谢产物的利用;另一方面在CcpA等因子调控下,pdh, pfl, pox和ack等基因下调而ldh基因的表达上调,细菌降低甲酸盐、乙酸等产物的形成,加强乳酸的合成[3,7]。如植物乳植杆菌在葡萄汁或葡萄酒中使用苹果酸作为首选能量源,增加苹果酸乳酸发酵途径基因的表达[21]。在乳制品中,碳水化合物饥饿和能量消耗会促使细菌将其代谢从糖酵解转变为氨基酸分解代谢,如细胞上调支链氨基酸代谢的关键酶BcaT的表达,促进支链氨基酸的摄入、合成和分解代谢[22]。支链氨基酸的分解代谢会将支链氨基酸分解形成挥发性支链脂肪酸,进而形成各种典型性风味成分,如3-甲基丁酮(图8)等,并为细胞生长代谢提供能量[23]。
分别采用野生菌株和ΔyesN突变菌株为发酵剂制备香肠,利用SPME-GC-MS技术检测挥发性风味物质,本研究共检测到35中挥发性风味物质,其中醛类16种,占总挥发性风味物质的46%,酯类11种,占29%等。由于醛类和酯类阈值较低,均对发酵香肠的风味形成贡献较大。其中醛类是脂肪降解的典型风味之一,主要来源于油酸等不饱和脂肪酸发降解;而酯类具有芳香味,具有典型的水果味或油脂味[24]。在多元统计方法方面,陈泓帆等[25]白酒对广式香肠质挥发性风味物质的影响,筛选出7种特征挥发性风味物质作为区分不同香型白酒广式香肠的主要标志物。本研究采用0PLS-DA模型,以VIP>1为标准,对比分析了2种香肠的风味特征筛选出14种关键差异风味物质,为进一步优化香肠发酵剂的筛选基础。
参考文献
[1] CUI Y H, WANG M H, ZHENG Y K, et al.The carbohydrate metabolism of Lactiplantibacillus plantarum[J].International Journal of Molecular Sciences, 2021, 22(24):13452.
[2] MENG X F, GANGOITI J, BAI Y X, et al.Structure-function relationships of family GH70 glucansucrase and 4,6-alpha-glucanotransferase enzymes, and their evolutionary relationships with family GH13 enzymes[J].Cellular and Molecular Life Sciences, 2016, 73(14):2681-2706.
[3] CHEN C, LU Y Q, WANG L L, et al.CcpA-dependent carbon catabolite repression cegulates fructooligosaccharides metabolism in Lactobacillus plantarum[J].Frontiers Microbiology, 2018, 9:1114.
[4] COPPINE J, KACZMARCZYK A, PETIT K, et al.Regulation of bacterial cell cycle progression by redundant phosphatases[J].Journal of Bacteriology, 2020, 202(17):e00345-20.
[5] XU W S, ZHANG Y Y, HUANG M Q, et al.The yesN gene encodes a carbohydrate utilization regulatory protein in Lactobacillus plantarum[J].Annals of Microbiology, 2015, 65(1):115-120.
[6] 张大革, 黄漫青, 高秀芝, 等.植物乳杆菌基因yesN对中式发酵香肠风味的影响[J].中国食品学报, 2019, 19(9):164-171.ZHANG D G, HUANG M Q, GAO X Z, et al.Effect of the gene yesN of Lactobacillus plantarum on the aroma compounds profiles in chinese fermented sausage[J].Journal of Chinese Institute of Food Science and Technology, 2019, 19(9):164-171.
[7] LU Y Q, SONG S C, TIAN H X, et al.Functional analysis of the role of CcpA in Lactobacillus plantarum grown on fructooligosaccharides or glucose:A transcriptomic perspective[J].Microbial Cell Factories, 2018, 17(1):201.
[8] 王德宝, 赵丽华, 田建军, 等.不同发酵剂对发酵香肠中风味物质释放及有害生物胺控制的影响[J].中国食品学报, 2019, 19(8):89-96.WANG D B, ZHAO L H, TIAN J J, et al.Effects of different starter cultures on the flavor release and control of the harmful biogenic amines in the fermented mutton sausages[J].Journal of Chinese Institute of Food Science and Technology, 2019, 19(8):89-96.
[9] 潘思弋, 徐佳敏, 张惠玲, 等.一株降解生物胺乳酸菌用于葡萄酒苹果酸乳酸发酵的特性研究[J].食品与发酵工业, 2023, 49(24):140-148.PAN S Y, XU J M, ZHANG H L, et al.Characteristics of wine malolactic fermentation by a biogenic amine-degrading lactic acid bacterium[J].Food and Fermentation Industries, 2023, 49(24):140-148.
[10] LOU Q, QI Y J, MA Y F, et al.Two-component signal transduction system SaeRS positively regulates Staphylococcus epidermidis glucose metabolism[J].The Scientific World Journal, 2014, 2014:908121.
[11] HISCOX T J, OHTANI K, SHIMIZU T, et al.Identification of a two-component signal transduction system that regulates maltose genes in Clostridium perfringens[J].Anaerobe, 2014, 30:199-204.
[12] SUN Z, CHEN Y X, YANG C, et al.A novel three-component system-based regulatory model for D-xylose sensing and transport in Clostridium beijerinckii[J].Molecular Microbiology, 2015, 95(4):576-589.
[13] PETTERSEN J S, NIELSEN F D, ANDREASSEN P R, et al.A comprehensive analysis of pneumococcal two-component system regulatory networks[J].NAR Genomics and Bioinformatics, 2024, 6(2):lqae039.
[14] PONCET S, SORET M, MERVELET P, et al.Transcriptional activator YesS is stimulated by histidine-phosphorylated HPr of the Bacillus subtilis phosphotransferase system[J].Journal of Biological Chemistry, 2009, 284(41):28188-28197.
[15] CHOW V, NONG G, STJOHN F J, et al.Bacterial xylan utilization regulons:Systems for coupling depolymerization of methylglucuronoxylans with assimilation and metabolism[J].Journal of Industrial Microbiology and Biotechnology, 2022, 49(2):kuab080.
[16] TODT T J, WELS M, BONGERS R S, et al.Genome-wide prediction and validation of sigma70 promoters in Lactobacillus plantarum WCFS1[J].PLoS One, 2012, 7(9):e45097.
[17] JIN Y Q, FAN Y T, SUN H, et al.Transcriptome analysis rreveals catabolite control protein a regulatory mechanisms underlying glucose-excess or-limited conditions in a ruminal bacterium, Streptococcus bovis[J].Frontiers in Microbiology, 2021, 12:767769.
[18] ANDERSEN J M, BARRANGOU R, HACHEM M A, et al.Transcriptional analysis of prebiotic uptake and catabolism by Lactobacillus acidophilus NCFM[J].PLoS One, 2012, 7(9):e44409.
[19] FRANCKE C, KERKHOVEN R, WELS M, et al.A generic approach to identify Transcription Factor-specific operator motifs;Inferences for LacI-family mediated regulation in Lactobacillus plantarum WCFS1[J].BMC Genomics, 2008, 9:145.
[20] HALSEY C R, LEI S L, WAX J K, et al.Amino acid catabolism in staphylococcus aureus and the function of carbon catabolite repression[J].mBio, 2017, 8(1):e01434-16.
[21] L PEZ DE FELIPE, DE LAS RIVAS B, MU
PEZ DE FELIPE, DE LAS RIVAS B, MU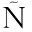 OZ R.Molecular responses of Lactobacilli to plant phenolic compounds:A comparative review of themechanisms involved[J].Antioxidants,2022,11(1):18.
OZ R.Molecular responses of Lactobacilli to plant phenolic compounds:A comparative review of themechanisms involved[J].Antioxidants,2022,11(1):18.
[22] 马勇, 魏从娇, 王雍雍, 等.发酵香肠中3-甲基丁醛产生途径的研究进展[J].食品与发酵工业, 2023,49(9):324-333.MA Y, WEI C J, WANG Y Y, et al.Research progress on the production pathway of 3-methylbutanal in fermented sausage[J].Food and Fermentation Industries, 2023, 49(9):324-333.
[23] CHEN L, LIU R, WU M G, et al.A review on aroma-active compounds derived from branched-chain amino acid in fermented meat products:Flavor contribution, formation pathways, and enhancement strategies[J].Trends in Food Science &Technology, 2024,145:104371.
[24] 吴双慧, 杨梓垚, 牛茵, 等.复配发酵剂对发酵香肠的品质及挥发性风味的影响[J].食品工业科技, 2024,45(5):126-133.WU S H, YANG Z Y, NIU Y, et al.Effects of mixed starter culture on quality and volatile flavor of fermented sausages[J].Science and Technology of Food Industry, 2024, 45(5):126-133.
[25] 陈泓帆, 聂鑫, 宋川, 等.浓香和酱香型白酒对广式香肠质构和挥发性风味物质的影响[J].中国调味品, 2023, 48(5):44-49.CHEN H F, NIE X, SONG C, et al.Effects of strong-flavor and sauce-flavor Baijiu on the texture and volatile flavor substances of Cantonese sausages[J].China Condiment, 2023, 48(5):44-49.
Research on impact of transcriptional regulator YesN in Lactiplantibacillus plantarum on sucrose metabolism and flavor formation in fermented sausages
QIANG Yi1, ZHANG Dage1,2, XU Guangqian1,2, XU Wensheng1,2*
1(Faculty of Food Science and Engineering, Beijing University of Agriculture, Beijing 102206, China)2(Key Laboratory of Agricultural Product Detection and Control of Spoilage Organisms and Pesticide Residue, Beijing 102206, China)
ABSTRACT The purpose of this study was to elucidate the mechanisms through which the transcriptional regulator YesN in Lactiplantibacillus plantarum modulates sucrose metabolism, as well as to assess its impact on the production of volatile flavor substances and biogenic amines in fermented sausage.The research subject, the L.plantarum yesN mutant strain (ΔyesN), was investigated using transcriptome sequencing technology to compare the differentially expressed genes to that of the wild-type strain in basal culture media.The regulatory function of YesN on the promoter PscrB of the sucrose metabolism gene cluster was ascertained through gel mobility shift assays (EMSA) and bacterial single hybridization techniques.Further, these two strains were employed as fermentation starters in sausage preparation, and the influence of the yesN gene on the creation of volatile flavors and biogenic amines in fermented sausages was analyzed.Transcriptomic results showed that about 491 genes were differentially expressed between the ΔyesN strain and the wild-type strain, with 109 genes downregulated and 382 genes upregulated.Kyoto Encyclopedia of Genes and Genomes (KEGG) enrichment analyses showed that upregulated genes were primarily involved in nucleic acid and amino acid metabolism, whereas downregulated genes were mainly associated with carbon source and ester metabolism.In sausages prepared with mutant and wild-type strains as fermenting agents, a total of 14 significantly different volatile flavor compounds were detected, including 7 esters, 5 aldehydes, and 2 alcohols.These results indicated that the transcriptional regulator YesN exerted a significant regulatory effect on the sucrose metabolism gene cluster in L.plantarum, and it had an important impact on the production of flavor compounds and biogenic amines in fermented sausage manufacturing.
Key words Lactiplantibacillus plantarum; sucrose metabolism; transcriptional regulator; transcriptomic analysis

 PEZ DE FELIPE, DE LAS RIVAS B, MU
PEZ DE FELIPE, DE LAS RIVAS B, MU OZ R.Molecular responses of
OZ R.Molecular responses of