监督式机器学习在食品中霉菌毒素检测的应用研究进展
张书鸣1,2,王欣1,郑玲春1,白亚敏3,聂旭元1,王锴1,2,许桐1,FRANCISCO PÉREZ NEVADO4,王强 1,2*
1(重庆第二师范学院 生物与化学工程学院,重庆,400067)2(重庆第二师范学院 油脂资源利用与创新重庆市工程研究中心,重庆,400067)3(重庆市食品药品检验检测研究院,重庆,401121)4(埃斯特雷马杜拉大学,巴达霍斯,06001,西班牙)
摘 要 霉菌毒素是霉菌生长过程中产生的有毒次生代谢产物,广泛存在于各类食品中,直接影响食品的质量和人体的健康。为了保障食品安全和人体健康,必须采取有效措施来防止霉菌毒素在食品中的产生和积累。检测霉菌毒素的传统方法存在检测效率较低、灵敏度较差、适用性低等缺点,机器学习作为一种辅助手段,在提升检测准确性和简化实验操作等方面具有促进作用。其中,监督式机器学习因其强大的数据处理能力、精准的预测和分类能力、挖掘潜在食品安全问题的能力以及与其他技术的结合应用能力,已被广泛应用到食品安全的检测中,为食品安全检测提供了有力的支持,有助于保障消费者的健康和权益。该文围绕监督学习在食品中霉菌毒素检测的应用进展,介绍了监督学习的工作流程和常用算法,根据不同算法类型阐述了监督学习在检测食品中霉菌毒素的应用,并对监督学习在食品安全检测领域的发展趋势进行了展望。
关键词 监督学习;霉菌毒素;食品安全;分析检测;食品分析化学
DOI:10.13995/j.cnki.11-1802/ts.042148
引用格式:张书鸣,王欣,郑玲春,等.监督式机器学习在食品中霉菌毒素检测的应用研究进展[J].食品与发酵工业,2025,51(14):422-432.
ZHANG Shuming,WANG Xin,ZHENG Lingchun, et al.Advances in supervised machine learning for mycotoxin detection in foods[J].Food and Fermentation Industries,2025,51(14):422-432.
第一作者:硕士,讲师(王强教授为通信作者,E-mail:52320980@qq.com)
基金项目:重庆市自然科学基金面上项目(cstc2021jcyj-msxmX0362);重庆市教委科技项目(KJQN202101616)
收稿日期:2025-01-15,改回日期:2025-03-12
食品安全始终是全社会高度关注的焦点问题,因为食品的安全性直接关系到人们的身体健康乃至生命安全[1]。在保障食品安全的众多工作中,对食品污染物进行准确识别和有效控制至关重要。食品污染物涵盖化学物质、微生物等多种类型,它们会严重影响食品的品质和安全性。其中,毒素作为一种特殊的食品污染物,其危害不容小觑[2]。毒素的存在不仅会降低食品的品质和营养价值,还可能损害人体的神经系统和生殖系统,引发各种急性和慢性疾病[3-5]。霉菌毒素便是其中一类严重威胁人类健康的毒素[6]。因此,为切实保障食品安全,必须采取一系列有效的检测措施,以降低霉菌毒素的摄入风险。
目前,液相色谱质谱法(liquid chromatography-mass spectrometry, LC-MS)[7]、高效液相色谱法(high-performance liquid chromatography, HPLC)[8]和酶联免疫吸附测定法(enzyme-linked immunosorbent assay, ELISA)[9]被认为是检测霉菌毒素的最普遍分析方法,表1中总结了以上3种方法在检测霉菌毒素时的优势和局限性。有研究表明可通过机器学习与其他无损检测方法(如电子鼻、光谱法、微波检测法等)结合可实现自动化智能化的快速无损检测,这种方式能够减少人为因素干扰,减少化学试剂依赖,在霉菌毒素检测和食品安全检测中越来越受欢迎,被广泛应用于食品图像分类[10]、食品识别[11]以及食品加工检测[12]等。机器学习方法根据学习方式的不同可分为3种类型:监督式学习、无监督式学习和强化机器学习。目前在食品检测行业应用中常见的机器学习方法是监督式机器学习,简称为监督学习,通过监督学习可以提取样品的特征值,筛选出更为关键的特征变量,保持对原始数据准确和完整的描述。监督学习对于霉菌毒素的检测,通过训练模型识别特定类型的霉菌毒素,不仅减少了误报和漏报的情况,还缩短了检测时间、减少了人为误差,使研究人员能够通过大量的食品图像和数据进行算法训练,实现食品安全检测的自动评估与监测。
表1 传统霉菌毒素检测方法优缺点对比
Table 1 Comparison of advantages and disadvantages of traditional mycotoxin detection methods
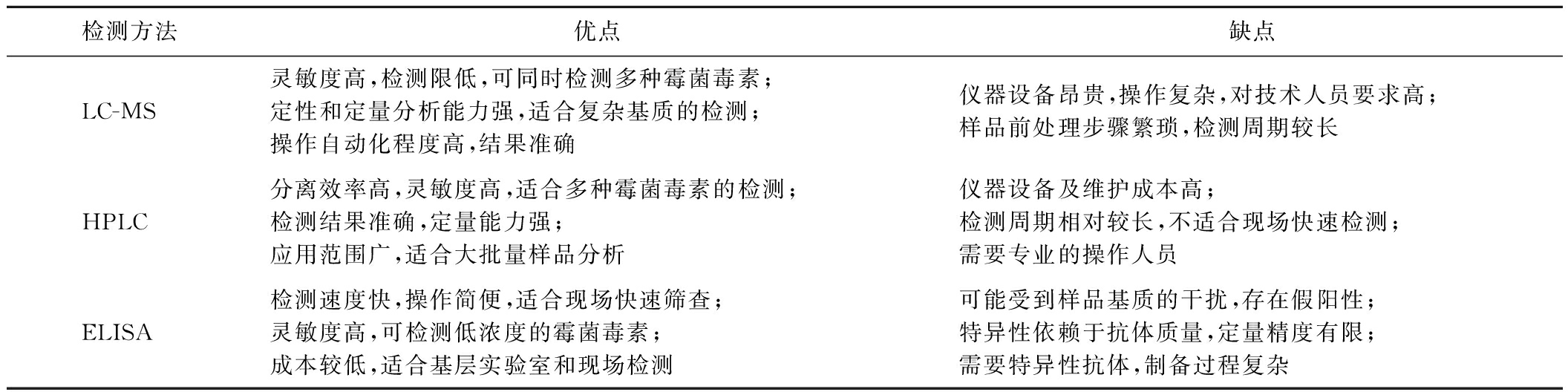
检测方法优点缺点LC-MS灵敏度高,检测限低,可同时检测多种霉菌毒素;定性和定量分析能力强,适合复杂基质的检测;操作自动化程度高,结果准确仪器设备昂贵,操作复杂,对技术人员要求高;样品前处理步骤繁琐,检测周期较长HPLC分离效率高,灵敏度高,适合多种霉菌毒素的检测;检测结果准确,定量能力强;应用范围广,适合大批量样品分析仪器设备及维护成本高;检测周期相对较长,不适合现场快速检测;需要专业的操作人员ELISA检测速度快,操作简便,适合现场快速筛查;灵敏度高,可检测低浓度的霉菌毒素;成本较低,适合基层实验室和现场检测可能受到样品基质的干扰,存在假阳性;特异性依赖于抗体质量,定量精度有限;需要特异性抗体,制备过程复杂
本文综述了在食品检测中广泛使用的监督学习研究进展,介绍了监督学习对于快速检测、预测食品中霉菌毒素的优势,讨论了监督学习在食品霉菌毒素检测的应用进展,评述了目前监督学习应用在食品霉菌毒素检测中存在的不足并展望了其应用前景。
1 监督学习概述
监督学习是机器学习中最常见的学习方式之一,其核心原理是通过已标注的训练数据来训练模型,使其能够预测或分类新的、未标注的数据。在监督学习中,每个训练样本都有一个对应的标签,模型通过学习这些“输入-输出”对之间的关系来进行预测,在食品检测中表现出卓越的优势。近年来,监督学习在食品质量评价[13]、医疗卫生[14]、数据分析[15]以及人工智能[16]等领域均有广泛的应用。随着各学科的交叉融合与渗透,监督学习算法目前已被广泛且深入地应用于霉菌毒素的检测与预测中[17],为食品安全检测提供了有力的工具。本文将从工作流程、常用的学习算法对监督学习进行介绍。
1.1 监督学习工作流程
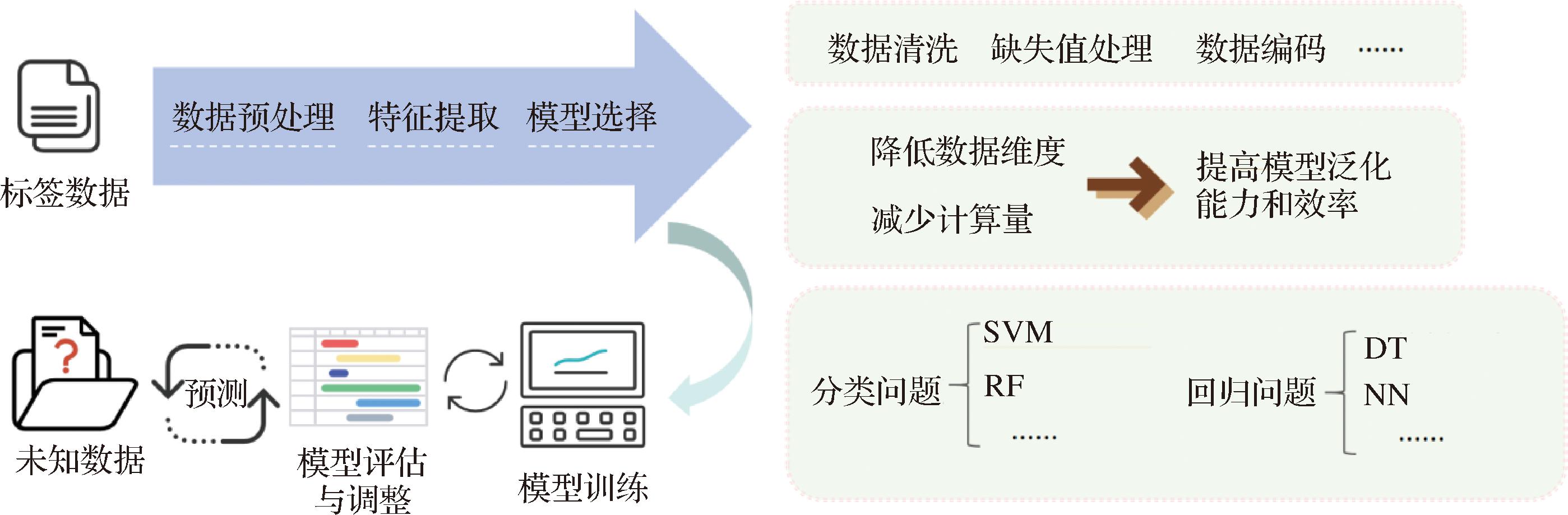
监督学习工作流程大致包括数据收集、数据预处理、特征提取、模型选择、模型训练、模型评估与调整等步骤(图1)。
在监督学习中,收集包含输入特征和输出标签的训练数据集,通过数据清洗、缺失值处理、特征缩放、特征选择或特征工程等步骤进行数据预处理与特征提取,根据问题的性质和数据的特性选择合适的监督学习模型,如支持向量机(support vector machire,SVM),随机森林(random forest,RF)等。
使用训练数据集训练模型,通常将训练数据集拆分为训练数据集、测试数据集和验证数据集,确定训练数据集的输入特征,确定适合模型的算法,如支持决策树(decision tree,DT)、神经网络(neural network,NN)等。然后在训练数据集上执行算法,最后通过提供测试集来评估模型的准确性。如果模型预测出正确的输出,即视为模型准确[18]。
1.2 常用的监督学习算法
监督学习算法通过在大量已知数据上训练模型挖掘数据本质规律,利用规律处理和分析未知数据,其模型效果常用正确率、精确率等指标评价。因数据隐含规律不同,需依目的建立不同监督学习模型,常用算法包括DT、NN、SVM和RF等。DT是能分类预测结局的技术,结果为自上而下树状图,可规避传统模型共线性缺点、体现影响因素交互作用,还能通过设置参数改变树层数,增加模型对变量的敏感性,如基于DT学习获得足够的细菌生长实验记录用于数据分析,该研究成功地预测了生长决策化学物质[19-20]。NN是模仿生物神经系统的信息处理模型,主要有卷积神经网络(convolutional neural network,CNN)和人工神经网络(artificial neural network,ANN)等类型。CNN通过卷积和池化操作提取特征,可与图像分割、图像增强和图像分类结合分析食品品质,现已应用于小样本的苹果外观质量分类,结果显著缩短了算法的运行时间,并更加高效地应用于了苹果分类市场[21]。ANN由输入、隐藏、输出层组成,每层含多个神经元,每个神经元接收一组输入信号,并通过激活函数对这些信号进行加权求和后产生输出,食品行业广泛用其解决食品品质[22]与成分含量[23]等问题。SVM可对已知分类样本集合二分式划分,判断新样本映射位置所属区域来分类,在食品领域通过数据指标映射关系训练识别,可通过一个指标预测对应指标,如预测在生产阶段估计面包片的大小和形状,以研究面包片根据一些环境和生产变量发生的形状变化[24-25]。RF是多种DT的集合形式,在建立回归模型过程中通过构建多个DT进行预测,每个DT输出一个连续的数值,然后将DT预测的结果,通过取平均值或加权平均值的方法整合得到最终结果[26],郭灿等[27]基于RF算法对我国花生黄曲霉毒素污染发生规律及其与地理、气候等因素的相关性进行研究,构建了适用于我国花生黄曲霉毒素污染的预警模型。
这些监督算法各具特色,在不同场景下均展现出了强大的应用潜力。表2所示为常用监督学习算法优缺点。
表2 常用监督学习算法优缺点
Table 2 Advantages and disadvantages of commonly used supervised learning algorithms

算法优点缺点DT训练后的模型具有良好的视觉可解释性,并且能够处理数字和分类数据在预测连续变量的结果时,预测效果较差,并且由于其高度灵活性,可能会导致过拟合NN学习能力较强,自动提取目标特征,发现样本集中特征规律,有较好的耐故障能力、信息融合能力、综合推理能力和较快的总体处理能力网络层数较多时,容易陷入局部最优解,也容易产生过拟合SVM能够处理高维数据、非线性问题、适用于小样本数据;具有较强的泛化能力、较好的鲁棒性和可解释性对参数的敏感性较高;计算复杂程度高;对数据的缩放、噪声数据敏感RF准确性高、能够处理大规模数据、决策规律合理、不易发生过拟合,具备良好的预测性能面对复杂的数据和大规模数据集时,需要对模型进行合适的参数调整来获得最佳的模型性能
为了克服单一算法的局限性,不少研究人员正探索多种算法相结合以取得更好的效果[28-29],如:a)减少标注成本:在许多实际应用中,获取大量有标签数据的成本高昂,而无标签数据通常比较丰富。通过结合监督学习和无监学习,可以显著减少对有标签数据的依赖,从而降低数据标注成本。b)提高模型性能:在有标签数据稀缺的情况下,通过引入无标签数据,可以提高模型的泛化能力和预测准确性。c)更好地利用数据:充分利用已有的无标签数据,避免数据浪费,提升模型的整体表现。
2 监督学习在食品中检测霉菌毒素的应用
常见的霉菌毒素有黄曲霉毒素、赭曲霉毒素、伏马菌素、玉米赤霉烯酮等,霉菌毒素在食品中的存在,严重威胁公众健康,迫切需要探索能够实现霉菌毒素快速、无损检测的技术。监督学习在检测食品中霉菌毒素的应用上展现出了显著的优势,极大地提高了食品安全检测的效率与准确性。传统上,霉菌毒素的检测依赖于繁琐的化学分析或生物学测试,这些方法不仅需要耗费大量时间,还可能受到人为操作误差的影响,限制了其在快速筛查和大规模监测中的应用。而监督学习技术,通过其强大的数据处理与模式识别能力,为这一挑战提供了创新解决方案。监督学习的应用,不仅为食品中霉菌毒素的检测注入了新的活力,也为保障食品安全、维护公众健康作出了积极贡献。
2.1 纯监督学习
监督学习是机器学习中的一种重要学习方式,其原理是利用一组已知类别的样本(即输入和对应的期望输出)来调整分类器的参数,使其达到所要求性能的过程。在这个过程中,每个样本都包含一个输入对象(通常为矢量)和一个期望的输出值(也称为监督信号)。监督学习的算法多样,包括但不限于DT、NN、SVM、RF等,主要应用于分类和回归问题。
2.1.1 DT
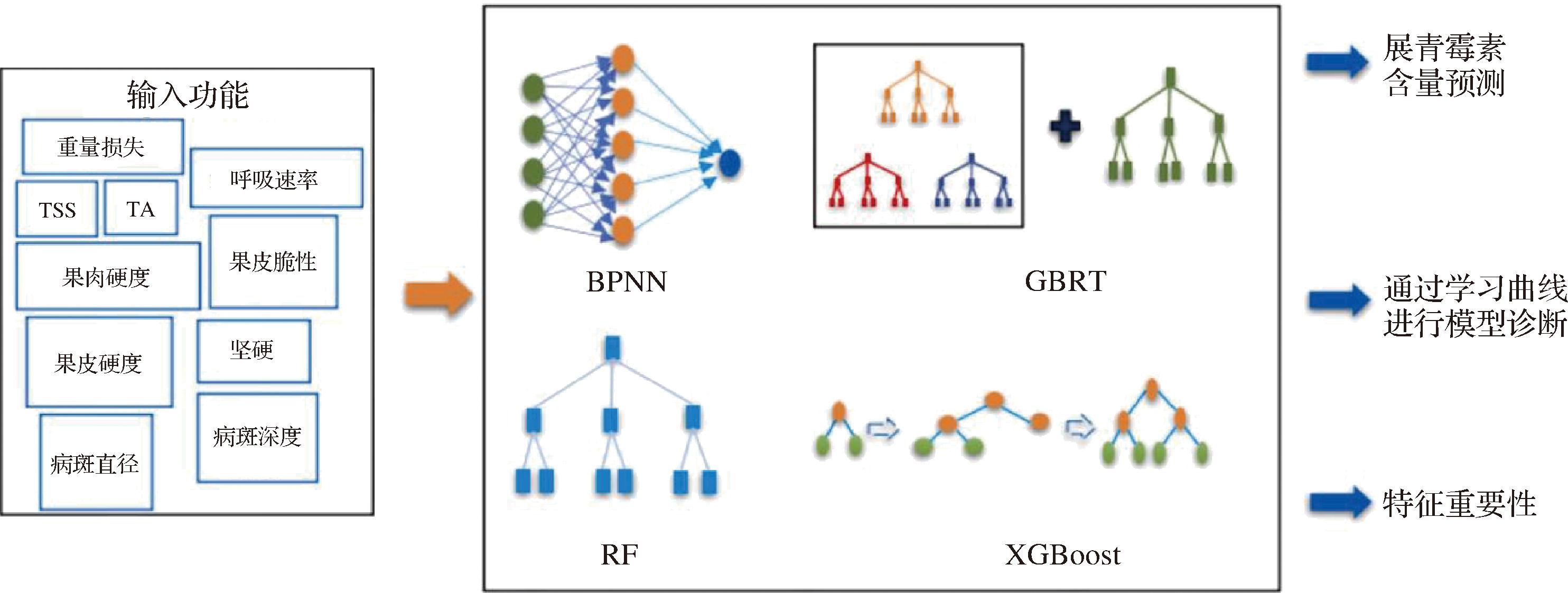
DT是在已知各种情况发生概率的基础上,通过构成DT来求取净现值的期望值≥0的概率,评价项目风险,判断其可行性的决策分析方法。RF是一种在机器学习中广泛应用的集成学习方法,它通过构建多个DT并将它们的预测结果结合起来,以提高模型的准确性和稳定性。与单一DT相比,RF可对复杂的数据集和预测变量进行随机选择,从而构建分类和回归树进行预测建模,提高预测准确性,避免发生过拟合现象。MA等[30]利用RF算法寻找识别商业带壳花生仁表面黄曲霉毒素污染的最佳特征波长,建立了简化的RF分类模型,与使用全光谱变量相比,该模型使用的光谱变量至少减少了94.82%,这有所提高。WANG等[31]应用RF算法来训练模型,该模型预测了在欧洲某个网格中种植的小麦被研究中的6种霉菌毒素中的至少一种污染的概率(低、中、高),结果表明该模型具有很高的预测性能,可以使用当前模型及其霉菌毒素预测功能来提升小麦供应链中的物流和基于风险的监测,从而提高小麦衍生产品的安全性,并改善粮食安全。梯度提升(gradient boosting, GB)算法也是一种集成学习算法,基本原理是将多个弱学习器组合成一个强学习器,通过每次迭代添加一个弱学习器来拟合前一个模型的预测误差。与RF不同之处在于,GB在学习过程中通过误差最小化原则依次构建每一棵DT,每棵树学习之前所有的预测值与目标值的差值更改权重大小,以此缩短原始训练样本与每次迭代构建的学习器间的误差,从而提高分类预测能力。通过不断学习和优化算法,逐渐提高无损检测的准确性,降低误报和漏报率,引入智能算法对提高食品安全检测技术具有积极的应用前景。近年来,不少研究人员将GB运用于农业中污染物的预测,CASTANO-DUQUE等[32]通过结合历史气象数据和相应的霉菌毒素污染数据实现关键特征的获取,使用梯度提升机(gradient boosting machine, GBM)和贝叶斯网络(bayesian network, BN)模型,预测伊利诺伊州玉米中黄曲霉毒素和伏马菌素的污染,总体准确率为94%,这些早期气象事件的检测可直接帮助农民和利益相关者做出明智决策,以防止伊利诺伊州种植的玉米受到霉菌毒素污染。WU等[33]利用碳点与机器学习算法适当地融合以实现对食品中黄曲霉毒素和赭曲霉毒素的超灵敏自动化检测,结果表明,extreme gradient boosting (XGBoost)是经过测试的8种算法中的优化模型。用小麦、大米、玉米、咖啡和牛奶的加标样品来评估测试模型,即使在10 pmol的霉菌毒素含量下,也达到了100%的示范准确度。该方法还能用于检测霉菌毒素这暗示了为智能检测食源性污染物而开发的方法的范围更广。CHENG等[34]使用GB算法根据病变和物理指标预测受感染苹果中的棒曲霉素水平,如图2所示,通过自动化设备和智能算法,减少人工干预,降低操作难度和误差,实现采后水果质量管理的自动化和智能化。
DT算法通过构建树形结构,将食品样本的特征与霉菌毒素的存在情况进行关联。其结构化的分类方式使得DT算法能够直观、清晰地展示霉菌毒素检测的逻辑关系和决策过程。与单一且传统的霉菌毒素检测方法相比,结合了DT算法的检测方法节约了时间和经济成本。DT与RF都是基于DT的集成学习方法,能够在短时间内处理大量的食品样本,快速识别出霉菌毒素的存在和浓度,为食品安全检测提供了实时的反馈,可见将多个DT模型作为基础模型组合在一起工作后,可表现出更优越的性能。
2.1.2 NN
NN的基本原理是模拟人脑神经系统的功能,通过多个节点(也叫神经元)的连接和计算,实现非线性模型的组合和输出。能够实现对输入数据的学习、模式识别和结果预测等功能。NN有多种类型,极为常见的是CNN,主要应用于图像处理领域,通过模拟生物视觉系统的机制,有效地识别和处理图像数据,是图像处理和识别领域最成功的(deep neural network,DNN)之一,在食品安全检测中发挥着至关重要的作用。早些年,研究人员就开始将NN应用在食品霉菌毒素检测中,并取得了一系列可观成就,例如,MATEO等[35]首次基于研究的因素使用NN预测小麦中脱氧雪腐镰刀菌烯醇的积累,结果表明径向基函数神经网络比多层感知器网络获得更低的错误和更好的泛化能力。后续评估了多层感知器人工神经网络(multilayer perceptron artificial neural network, MLP-ANN)和径向基函数网络(radial basis function network, RBFN)在不同条件下预测受镰刀菌污染的大麦种子中脱氧雪腐镰刀菌烯醇积累的能力,发现与 MLP-ANN 相比,RBFN 做到了更低的错误和更好的泛化性,但它们依然存在不足[36]。近年来,更多研究人员开发了更准确、更快速的新方法,如MARCO等[37]用ANN、逻辑回归和判别分析3种方法研究电子鼻是否能够分离黄曲霉毒素和伏马菌素的污染水平高于或低于法定限值的样品,结果表明,ANN的性能优于其他方法,黄曲霉毒素和伏马菌素的准确率分别为78%和77%。
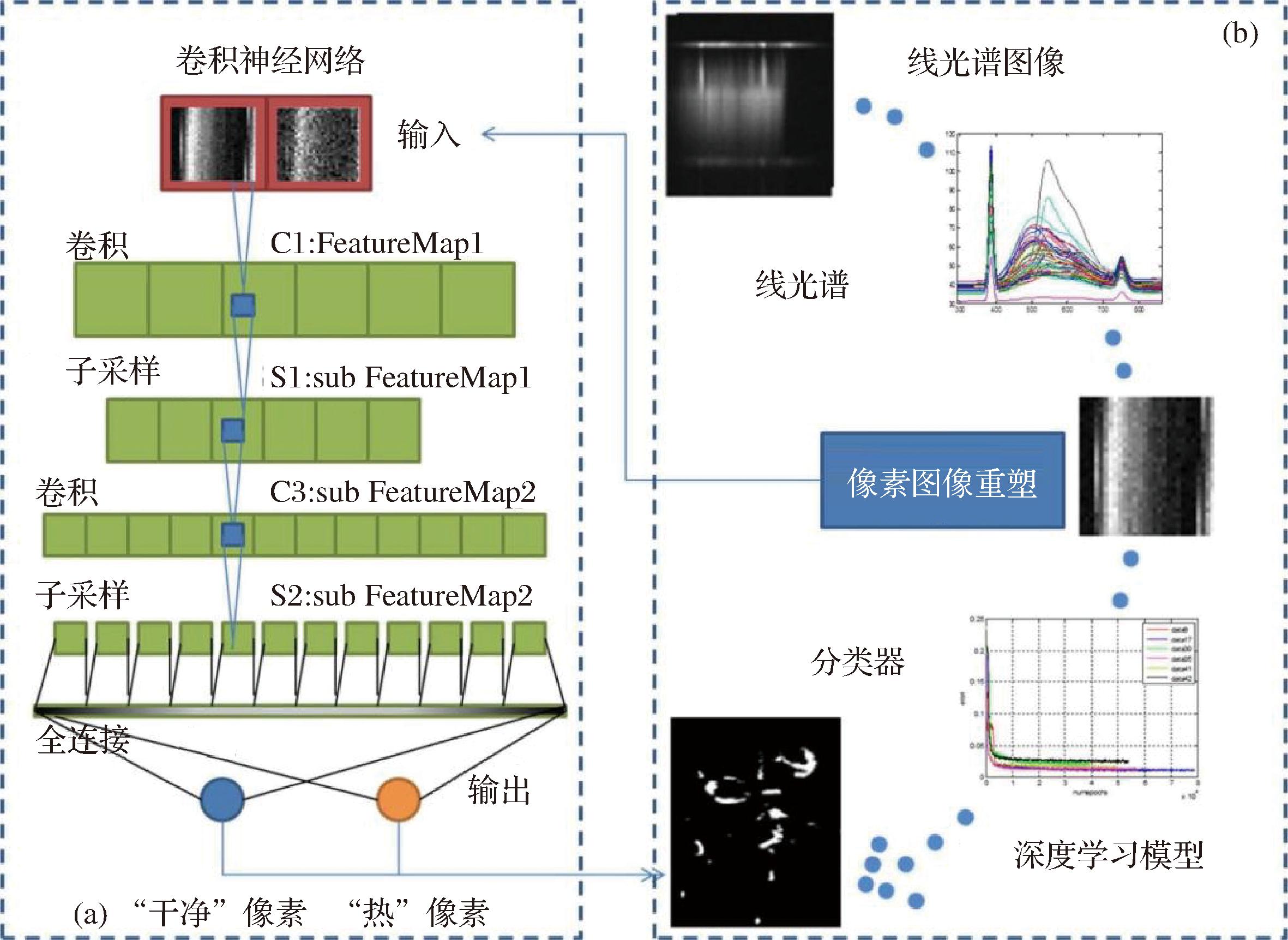
该团队进一步训练2个DNN模型来预测收获时哪些玉米田受到黄曲霉毒素和伏马菌素的污染超出法定限度,结果表明该模型具有稳健性[38]。HAN等[39]利用光栅模块、SCOMS 相机和电位移平台构建了高光谱成像系统(图3),并获取了黄曲霉毒素污染前后73个花生样品的146个高光谱图像立方体,提出了一种针对CNN方法的像素光谱重塑图像方法用于食品中黄曲霉毒素的检测。KIM等[40]基于紧凑卷积变压器 (compact convolutional transformer, CCT) 的模型对受脱氧雪腐镰刀菌烯醇和黄曲霉毒素污染的小麦进行分类,用CCT模型的功能来高效、无损地检测贮存小麦中的霉菌毒素水平。这种方法旨在最大限度地减少晶粒退化和损失,同时估计变质的程度,有助于在小麦存贮系统的运营管理中做出明智的决策。LIU等[41]采用多光谱成像技术结合不同的机器学习方法检测玉米中的玉米赤霉烯酮含量,结果表明通过反向传播神经网络遗传算法选择与玉米中玉米赤霉烯酮含量最相关的波长的方法是最佳方法,准确率为 93.33%。DENG等[42]提出了一种利用微波检测技术结合多任务学习策略对小麦霉变度和黄曲霉毒素进行定性定量分析的新方法,该方法是基于CNN结构的多任务学习模型,实现传输指标的自学习和模型标定,完成小麦霉变程度识别和黄曲霉毒素含量检测的分类和回归任务。SUN等[43]开发了一种双模式比色表面增强拉曼光谱侧向层析免疫测定法来检测脱氧雪腐镰刀菌烯醇,使用ANN和CNN分析比色读数和拉曼信号,以有效分类和精确预测脱氧雪腐镰刀菌烯醇污染水平,实现了98.8%的分类准确率。
近年来,NN在食品中霉菌毒素检测技术的应用被逐渐挖掘,其作为一种强大的监督学习工具,能够通过对大量数据的训练和学习,快速准确地识别出食品中的霉菌毒素。其具有高度的适应性和灵活性,通过调整网络结构和参数,可以应用于不同种类的霉菌毒素检测,实现对不同食品的无损检测,这种灵活性使得NN在应对复杂多变的食品安全问题时具有独特的优势。此外,NN与电子鼻、高光谱成像、多层感知器等技术相结合可以显著提高检测系统的效率和准确性,这种结合不仅提高了检测的自动化程度,还为食品质量检测提供了智能化的解决方案,能够在复杂的检测环境中快速、准确地识别和分类目标物质[44]。对以上文献数据的来源和特征、模型选择和应用场景进行简要概括,结果见表3。
表3 基于NN算法的食品中霉菌毒素检测应用
Table 3 Application of Neural Network Algorithms in Mycotoxin Detection in Foods
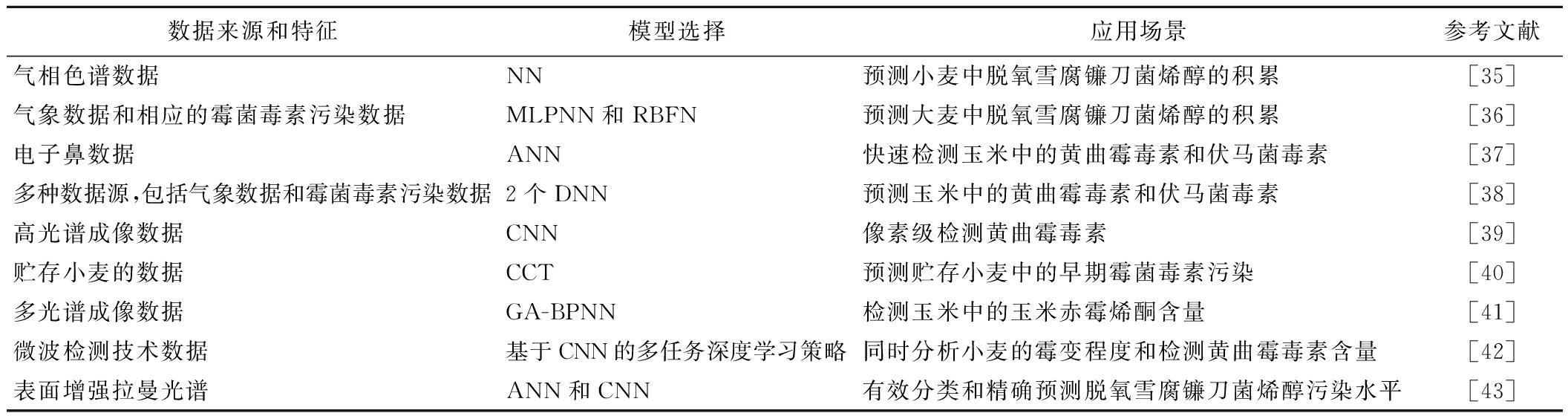
数据来源和特征模型选择应用场景参考文献气相色谱数据NN预测小麦中脱氧雪腐镰刀菌烯醇的积累[35]气象数据和相应的霉菌毒素污染数据MLPNN和RBFN预测大麦中脱氧雪腐镰刀菌烯醇的积累[36]电子鼻数据ANN快速检测玉米中的黄曲霉毒素和伏马菌毒素[37]多种数据源,包括气象数据和霉菌毒素污染数据2个DNN预测玉米中的黄曲霉毒素和伏马菌毒素[38]高光谱成像数据CNN像素级检测黄曲霉毒素[39]贮存小麦的数据CCT预测贮存小麦中的早期霉菌毒素污染[40]多光谱成像数据GA-BPNN检测玉米中的玉米赤霉烯酮含量[41]微波检测技术数据基于CNN的多任务深度学习策略同时分析小麦的霉变程度和检测黄曲霉毒素含量[42]表面增强拉曼光谱ANN和CNN有效分类和精确预测脱氧雪腐镰刀菌烯醇污染水平[43]
2.1.3 SVM
SVM主要用于解决模式识别领域中的数据分类问题,其基本原理是寻找一个最优的超平面,将不同类别的样本点有效地分开。这个最优超平面不仅要求能将两类样本正确分开,而且要求分类间隔最大,即两类样本点到该超平面的距离之和最大。这些最靠近超平面的样本点被称为支持向量,它们对于决策超平面的位置起到了关键作用。SVM的优点包括:高精度和强泛化能力;适用性强与灵活性高;有效处理不平衡数据集。近5年关于SVM在食品中霉菌毒素的检测研究数量显著增加,成为了学术界的一个热点。例如XU等[45]研制了一种基于自由空间测量的微型微波检测装置,用于获取不同霉变程度小麦样品在2.5~11.5 GHz频段的透射指数,构建了多种模型,其中自举软收缩-SVM 模型实现了最佳预测性能。ZHENG等[46]使用极端梯度提升算法与SVM相结合,建立了定量和二级分选方法,这种两级方法的定量模型预测值的均方根误差较小,排序方法的准确率高,可快速、准确检测,无需溶剂,无需样品预处理,可用于检测玉米中的黄曲霉毒素前体Ver A,便于实际使用。BERTANI等[47]使用基于SVM算法的二元分类模型进行频谱分析,并结合荧光光谱分析手段,检测杏仁浆中的黄曲霉毒素,通过这种方式可以达到94%的分类准确率,取得了良好的检测效果。此外,SVM分类模型结合不同的预处理方法,如光谱技术,来筛选样品中的污染物也得到了广泛应用。例如KIM等[48]提出并验证了一种基于高光谱荧光、近红外或短波红外成像结合SVM检测玉米样品中单一污染和混合污染的黄曲霉毒素和伏马菌毒素的快速检测方法,结果表明,带有SVM模型的短波红外显示出更高的准确性和泛化性能。ZHAO等[49]利用基于光谱仪的光谱检测系统采集不同霉变程度小麦面粉样品的吸光度,又将区间选择方法与SVM结合建立了基于特征区间选择的小麦玉米赤霉烯酮定量检测模型,用于定量检测小麦中的玉米赤霉烯酮,结果表明近红外光谱法结合适当的数据分析方法可作为定量检测小麦和其他谷物霉菌毒素的简单快速工具。CEBRI N等[50]通过SVM模型评估近红外光谱在检测干腌火腿琼脂上产生赭曲霉毒素的霉菌种类方面的潜力。DENG等[51]建立了基于近红外光谱技术的SVM模型,用于测试玉米中的黄曲霉毒素,如图4所示,结果表明,SVM 模型是有效的高精度模型。但是SVM在处理高维数据时,虽然具有强大的分类能力,但也面临过拟合和计算复杂度高的问题。通过引入松弛变量、减少特征维度、数据预处理等方法可以有效避免过拟合[52]。
N等[50]通过SVM模型评估近红外光谱在检测干腌火腿琼脂上产生赭曲霉毒素的霉菌种类方面的潜力。DENG等[51]建立了基于近红外光谱技术的SVM模型,用于测试玉米中的黄曲霉毒素,如图4所示,结果表明,SVM 模型是有效的高精度模型。但是SVM在处理高维数据时,虽然具有强大的分类能力,但也面临过拟合和计算复杂度高的问题。通过引入松弛变量、减少特征维度、数据预处理等方法可以有效避免过拟合[52]。
同时,通过优化算法、并行计算、分布式计算等技术可以显著提高SVM的计算效率,使其在大规模数据集上也能高效运行[53]。
SVM通常与其他技术相结合使用,尤其是光谱技术,以提高检测效率和准确性。如将SVM与近红外光谱技术相结合,利用近红外光谱仪采集食品样本的光谱信息,并通过SVM算法对光谱数据进行处理和分析,充分发挥两者优点,实现对霉菌毒素的快速无损检测。除近红外光谱外,高光谱成像、微波检测以及其他技术在与SVM相结合也表现出优异的性能,未来有望创新性结合更多技术,助力霉菌毒素在食品中的快速检测技术发展。
2.1.4 其他算法
除了DT、NN以及SVM外,线性回归(linear regression, LR)、线性判别分析(linear discriminant analysis,LDA)、批标准化(batch normalization,BN)等方法在食品霉菌毒素检测中也发挥着重要作用。LR是一种预测模型,主要用于建模输入变量(特征)和输出变量(目标)之间的线性关系,帮助了解模型的预测精度和拟合程度。LDA是一种分类模型,用于将数据投影到低维空间以便更好地分类。BN定理是分类统计模型,其核心思想在于利用已知的先验概率和条件概率来更新对某个事件发生的概率的估计。例如KIM等[54]通过LR开发了统计模型根据收获前的天气条件估计花生黄曲霉毒素污染的区域状况,在黄曲霉毒素风险方面表现良好。KIM等[55]使用LDA与紧凑的自动化拉曼系统结合进行快速、无损分析玉米中的黄曲霉毒素,用自然被黄曲霉毒素污染的玉米样品获得数据,并使用监督学习方法对数据进行分析的结果令人满意,有望在未来得到进一步的开发。ZHANG等[56]建立了基于全波段的BN定性判别模型用于探讨近红外光谱法检测花生中黄曲霉毒素污染的可行性,该模型判别精度高,验证集和预测集的综合总体准确率均超过91.00%。
不同的算法具有不同的优势,适用于不同的应用场景。LR可用于预测环境因素与霉菌毒素含量的关系[57],LDA可用于识别不同种类的霉菌毒素之间的特征差异[58],BN可用来整合不同来源的信息[59],3种技术各自具备一定的优势,但目前大多与检测技术联用应用于检测食品中的黄曲霉毒素,霉菌毒素类型较单一,随着技术的不断发展,相信在未来能够更广泛地应用于检测其他霉菌毒素并具有更好的效果。
2.2 融合监督学习与其他算法
单纯的监督学习是一种依赖于标注数据进行训练的机器学习方法,虽然在许多任务中表现出色,但也存在一些明显的不足,例如对标注数据依赖高、存在过拟合风险等。不少研究人员提出融合监督学习与其他算法即结合多种学习方法的策略,以提升模型性能和适应性,有效地解决问题。
2.2.1 监督学习算法与主成分分析(principal component analysis, PCA)融合
PCA是一种经典的降维技术,能够将高维数据映射到低维空间,同时保留数据的主要方差信息。通过PCA降维后的数据可以减少特征维度,降低计算复杂度,减少过拟合现象,同时去除噪声和冗余信息,提升监督学习模型的训练效率[60]。ATA 等[61]为了区分受黄曲霉毒素污染的辣椒和未受污染的辣椒,提出了一种基于高光谱成像和机器学习的紧凑型机器视觉系统,其利用PCA等技术选择显著特征,将LDA等技术用作分类器,使用所提出的特征和选择方法,可以用更少的光谱波段数量实现稳健和更高的分类性能,从而能够设计更简单的机器视觉系统。LI等[62]利用短波红外高光谱成像开发一种准确、快速检测黄曲霉污染的方法,且该方法能阐明玉米粒污染时间。应用 PCA可降低高光谱图像的维度,同时保留最重要的信息,后采用CNN从使用PCA降维后单个玉米粒的高光谱图像中提取特征。PCA流程在CNN特征提取阶段之前进行,以提高 CNN 的效率并提高其识别和分类关键特征的能力。该实验表明,将PCA与CNN相结合,可实现高效的特征提取,降低计算复杂性,并保留与分类任务最相关的信息。王蓓等[63]利用电子鼻技术,建立了花生中黑曲霉和黄曲霉毒素含量的快速检测方法,利用PCA检测分组模式,通过LDA和偏最小二乘判别分析对黄曲霉毒素含量超标与否进行定性分析。也有研究人员在PCA和LDA的基础上增加SVM算法,将这3种算法与激光诱导荧光光谱法结合,用于检测花生油、水稻中的黄曲霉毒素污染[64-65]。
等[61]为了区分受黄曲霉毒素污染的辣椒和未受污染的辣椒,提出了一种基于高光谱成像和机器学习的紧凑型机器视觉系统,其利用PCA等技术选择显著特征,将LDA等技术用作分类器,使用所提出的特征和选择方法,可以用更少的光谱波段数量实现稳健和更高的分类性能,从而能够设计更简单的机器视觉系统。LI等[62]利用短波红外高光谱成像开发一种准确、快速检测黄曲霉污染的方法,且该方法能阐明玉米粒污染时间。应用 PCA可降低高光谱图像的维度,同时保留最重要的信息,后采用CNN从使用PCA降维后单个玉米粒的高光谱图像中提取特征。PCA流程在CNN特征提取阶段之前进行,以提高 CNN 的效率并提高其识别和分类关键特征的能力。该实验表明,将PCA与CNN相结合,可实现高效的特征提取,降低计算复杂性,并保留与分类任务最相关的信息。王蓓等[63]利用电子鼻技术,建立了花生中黑曲霉和黄曲霉毒素含量的快速检测方法,利用PCA检测分组模式,通过LDA和偏最小二乘判别分析对黄曲霉毒素含量超标与否进行定性分析。也有研究人员在PCA和LDA的基础上增加SVM算法,将这3种算法与激光诱导荧光光谱法结合,用于检测花生油、水稻中的黄曲霉毒素污染[64-65]。
2.2.2 监督学习算法与k-means聚类融合
k-means聚类作为一种经典的特征提取方法,对数据进行预处理,不仅可以去除噪声和异常值,使监督学习模型能够在更干净的数据上进行训练,从而提高分类或回归的准确性,还可以将数据划分为多个子集,从而减少模型对整个数据集的过拟合[66]。RABBANI等[67]将ANN和SVM法用于预测其设计的传感器对霉菌毒素的响应,交叉验证的ANN和SVM模型能够将各种传感器按产生高或低荧光响应准确分类,后使用k-means聚类方法来检查其调查的每个阶段每个DNA序列的相似性和不相似性。PURCHASE等[68]使用k-means聚类来确定谷物成分与伏马菌素浓度之间的关系,初步得出浓度可能与因子值的相互作用非线性组合相关的结论,后使用DT回归器研究这种可能性并确定与高浓度最相关的因子值。ROCCHETTI等[69]将k-means聚类与正交偏最小二乘判别分析结合,显示了牛奶样品的2组判别性,从而揭示了整个饲喂系统以及其他变异因素对最终霉菌毒素污染概况的分层影响更高的结论。
将监督学习与其他学习算法结合可以形成强大的机器学习范式,这一结合不仅充分利用了监督机器学习在分类和预测方面的优势,还吸纳了其他学习算法在数据挖掘和模式识别上的长处。不仅提高了霉菌毒素检测的准确性和效率,还降低了对大量标记数据的依赖,节省了时间和成本。更重要的是,其提供了一个更为全面和深入的视角,有助于更好地理解和应对食品中的霉菌毒素污染问题,这对于食品安全监测有重要意义。
3 结语
监督学习在提高食品安全检测和消费者满意度方面具有巨大潜力,作为一种辅助手段与检测技术相结合,为精准预测提供了新思路。本文介绍了监督学习的过程以及方法,综述了监督学习在食品中霉菌毒素检测的应用进展。监督学习除了在食品中霉菌毒素检测以及食品各方面的应用外[70-71],在医学、风工程、海洋数据等领域也展现出广阔的应用前景[72-74]。尽管监督学习在食品中霉菌毒素的检测与预测中表现出巨大的潜力,但仍然可以从以下3方面做出努力:通过优化算法、优化硬件、模型简化等进一步缩短识别时间、提高实时性;通过物联网(如光谱传感器、电子鼻、电子舌)和边缘计算实现霉菌毒素检测的自动化和智能化;尝试荧光传感器阵列结合AI等方法实现多种毒素进行同时检测及高通量识别。
参考文献
[1] KING T, COLE M, FARBER J M, et al. Food safety for food security: Relationship between global megatrends and developments in food safety[J]. Trends in Food Science &Technology, 2017, 68:160-175.
[2] KUMAR P, MAHATO D K, KAMLE M, et al. Aflatoxins: A global concern for food safety, human health and their management[J]. Frontiers in Microbiology, 2017, 7:2170.
[3] 缪伊雯, 白菲, 童华荣. 茶叶中霉菌毒素危害与质量控制研究进展[J]. 食品科学, 2023, 44(17):352-362.MIAO Y W, BAI F, TONG H R. Research progress on the hazards and control of mycotoxins in tea[J]. Food Science, 2023, 44(17):352-362.
[4] XU H W, WANG L Z, SUN J D, et al. Microbial detoxification of mycotoxins in food and feed[J]. Critical Reviews in Food Science and Nutrition, 2022, 62(18):4951-4969.
[5] 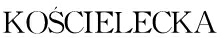 K,
K, 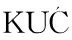 A, KUBIK-MACHURA D, et al. Endocrine effect of some mycotoxins on humans: A clinical review of the ways to mitigate the action of mycotoxins[J]. Toxins, 2023, 15(9):515.
A, KUBIK-MACHURA D, et al. Endocrine effect of some mycotoxins on humans: A clinical review of the ways to mitigate the action of mycotoxins[J]. Toxins, 2023, 15(9):515.
[6] ZAIN M E. Impact of mycotoxins on humans and animals[J]. Journal of Saudi Chemical Society, 2011, 15(2):129-144.
[7] PAVLENKO R, BERZINA Z, REINHOLDS I, et al. An occurrence study of mycotoxins in plant-based beverages using liquid chromatography-mass spectrometry[J]. Toxins, 2024, 16(1):53.
[8] TSAGKARIS A S, PRUSOVA N, DZUMAN Z, et al. Regulated and non-regulated mycotoxin detection in cereal matrices using an ultra-high-performance liquid chromatography high-resolution mass spectrometry (UHPLC-HRMS) method[J]. Toxins, 2021, 13(11):783.
[9] LIANG Y F, ZHOU X W, WANG F, et al. Development of a monoclonal antibody-based ELISA for the detection of Alternaria mycotoxin tenuazonic acid in food samples[J]. Food Analytical Methods, 2020, 13(8):1594-1602.
[10] MAGNUS I, VIRTE M, THIENPONT H, et al. Combining optical spectroscopy and machine learning to improve food classification[J]. Food Control, 2021, 130:108342.
[11] ELLIS D I, BROADHURST D, CLARKE S J, et al. Rapid identification of closely related muscle foods by vibrational spectroscopy and machine learning[J]. The Analyst, 2005, 130(12):1648-1654.
[12] MENICHETTI G, RAVANDI B, MOZAFFARIAN D, et al. Machine learning prediction of the degree of food processing[J]. Nature Communications, 2023, 14(1):2312.
[13] CHHETRI K B. Applications of artificial intelligence and machine learning in food quality control and safety assessment[J]. Food Engineering Reviews, 2024, 16(1):1-21.
[14] RASHIDI H H, TRAN N, ALBAHRA S, et al. Machine learning in health care and laboratory medicine: General overview of supervised learning and Auto-ML[J]. International Journal of Laboratory Hematology, 2021, 43(Suppl 1):15-22.
[15] ALSUBARI S N. Data analytics for the identification of fake reviews using supervised learning[J]. Computers, Materials &Continua, 2022, 70(2):3189-3204.
[16] SEBAG M. A tour of machine learning: An AI perspective[J]. AI Communications, 2014, 27(1):11-23.
[17] INGLIS A, PARNELL A C, SUBRAMANI N, et al. Machine learning applied to the detection of mycotoxin in food: A systematic review[J]. Toxins, 2024, 16(6):268.
[18] HENDRICKX K, PERINI L, VAN DER PLAS D, et al. Machine learning with a reject option: A survey[J]. Machine Learning, 2024, 113(5):3073-3110.
[19] GOODMAN K E, LESSLER J, COSGROVE S E, et al. A clinical decision tree to predict whether a bacteremic patient is infected with an extended-spectrum β-lactamase-producing organism[J]. Clinical Infectious Diseases, 2016, 63(7):896-903.
[20] ASHINO K, SUGANO K, AMAGASA T, et al. Predicting the decision making chemicals used for bacterial growth[J]. Scientific Reports, 2019, 9(1):7251.
[21] SUN L, LIANG K B, SONG Y X, et al. An improved CNN-based apple appearance quality classification method with small samples[J]. IEEE Access, 2021, 9:68054-68065.
[22] 章海亮, 周孝文, 刘雪梅, 等. 基于卷积神经网络和高光谱成像技术的多宝鱼新鲜度鉴别[J]. 光谱学与光谱分析, 2024, 44(2):367-371.ZHANG H L, ZHOU X W, LIU X M, et al. Freshness identification of turbot based on convolutional neural network and hyperspectral imaging technology[J]. Spectroscopy and Spectral Analysis, 2024, 44(2):367-371.
[23] 蔡健荣, 黄楚钧, 马立鑫, 等. 一维卷积神经网络的手持式可见/近红外柑橘可溶性固形物含量无损检测系统[J]. 光谱学与光谱分析, 2023, 43(9):2792-2798.CAI J R, HUANG C J, MA L X, et al. Hand-held visible/near infrared nondestructive detection system for soluble solid content in mandarin by 1D-CNN model[J]. Spectroscopy and Spectral Analysis, 2023, 43(9):2792-2798.
[24] 刘静, 杜广全, 管骁. 基于近红外光谱的果蔬脆片品质评价方法研究[J]. 分析科学学报, 2017, 33(1):71-75.LIU J, DU G Q, GUAN X. Study on quality evaluation of fruit and vegetable chips based on near infrared spectroscopy[J]. Journal of Analytical Science, 2017, 33(1):71-75.
[25] MANNARO K, BAIRE M, FANTI A, et al. A robust SVM color-based food segmentation algorithm for the production process of a traditional carasau bread[J]. IEEE Access, 2022, 10:15359-15377.
[26] ZOUNEMAT-KERMANI M, BATELAAN O, FADAEE M, et al. Ensemble machine learning paradigms in hydrology: A review[J]. Journal of Hydrology, 2021, 598:126266.
[27] 郭灿, 岳晓凤, 白艺珍, 等. 花生黄曲霉毒素平衡取样-随机森林风险预警模型的应用研究[J]. 中国农业科学, 2022, 55(17):3426-3449.GUO C, YUE X F, BAI Y Z, et al. Research on the application of a balanced sampling-random forest early warning model for aflatoxin risk in peanut[J]. Scientia Agricultura Sinica, 2022, 55(17):3426-3449.
[28] WU H, PRASAD S. Semi-supervised deep learning using pseudo labels for hyperspectral image classification[J]. IEEE Transactions on Image Processing, 2018, 27(3):1259-1270.
[29] VAN ENGELEN J E, HOOS H H. A survey on semi-supervised learning[J]. Machine Learning, 2020, 109(2):373-440.
[30] MA J N, GUAN Y, XING F G, et al. Accurate and non-destructive monitoring of mold contamination in foodstuffs based on whole-cell biosensor array coupling with machine-learning prediction models[J]. Journal of Hazardous Materials, 2023, 449:131030.
[31] WANG X X, LIU C, VAN DER FELS-KLERX H J. Regional prediction of multi-mycotoxin contamination of wheat in Europe using machine learning[J]. Food Research International, 2022, 159:111588.
[32] CASTANO-DUQUE L, VAUGHAN M, LINDSAY J, et al. Gradient boosting and Bayesian network machine learning models predict aflatoxin and fumonisin contamination of maize in Illinois-First USA case study[J]. Frontiers in Microbiology, 2022, 13:1039947.
[33] WU Q S, XU L J, ZOU Z Y, et al. Rapid nondestructive detection of peanut varieties and peanut mildew based on hyperspectral imaging and stacked machine learning models[J]. Frontiers in Plant Science, 2022, 13:1047479.
[34] CHENG X Y, LI R X, XIE P D, et al. Predictive modeling of patulin accumulation in apple lesions infected by Penicillium expansum using machine learning[J]. Postharvest Biology and Technology, 2024, 217:113115.
[35] MATEO F, GADEA R, MATEO R, et al. Neural network models for prediction of trichothecene content in wheat[J]. World Mycotoxin Journal, 2008, 1(3):349-356.
[36] MATEO F, GADEA R, MATEO E M, et al. Multilayer perceptron neural networks and radial-basis function networks as tools to forecast accumulation of deoxynivalenol in barley seeds contaminated with Fusarium culmorum[J]. Food Control, 2011, 22(1):88-95.
[37] CAMARDO LEGGIERI M, MAZZONI M, FODIL S, et al. An electronic nose supported by an artificial neural network for the rapid detection of aflatoxin B1 and fumonisins in maize[J]. Food Control, 2021, 123:107722.
[38] LEGGIERI M C, MAZZONI M, BATTILANI P. Machine learning for predicting mycotoxin occurrence in maize[J]. Frontiers in Microbiology, 2021, 12:661132.
[39] HAN Z Z, GAO J Y. Pixel-level aflatoxin detecting based on deep learning and hyperspectral imaging[J]. Computers and Electronics in Agriculture, 2019, 164:104888.
[40] KIM Y, KANG S, AJANI O S, et al. Predicting early mycotoxin contamination in stored wheat using machine learning[J]. Journal of Stored Products Research, 2024, 106:102294.
[41] LIU W, DENG H Y, SHI Y L, et al. Application of multispectral imaging combined with machine learning methods for rapid and non-destructive detection of zearalenone (ZEN) in maize[J]. Measurement, 2022, 203:111944.
[42] DENG J H, NI L H, BAI X, et al. Simultaneous analysis of mildew degree and aflatoxin B1 of wheat by a multi-task deep learning strategy based on microwave detection technology[J]. LWT, 2023, 184:115047.
[43] SUN B Y, WU H Y, FANG T R, et al. Dual-mode colorimetric/SERS lateral flow immunoassay with machine learning-driven optimization for ultrasensitive mycotoxin detection[J]. Analytical Chemistry, 2025, 97(9):4824-4831.
[44] MA P H, ZHANG Z K, JIA X X, et al. Neural network in food analytics[J]. Critical Reviews in Food Science and Nutrition, 2024, 64(13):4059-4077.
[45] Rezaee Z, Mohtasebi S S, Firouz S M. Monitoring pistachio health using data fusion of machine vision and electronic nose (E-nose)[J]. Journal of Food Measurement and Characterization, 2024, 19(3): 1-8.
[46] ZHENG S Y, WEI Z S, LI S, et al. Near-infrared reflectance spectroscopy-based fast versicolorin A detection in maize for early aflatoxin warning and safety sorting[J]. Food Chemistry, 2020, 332:127419.
[47] BERTANI F R, BUSINARO L, GAMBACORTA L, et al. Optical detection of aflatoxins B in grained almonds using fluorescence spectroscopy and machine learning algorithms[J]. Food Control, 2020, 112:107073.
[48] KIM Y K, BAEK I, LEE K M, et al. Rapid detection of single- and co-contaminant aflatoxins and fumonisins in ground maize using hyperspectral imaging techniques[J]. Toxins, 2023, 15(7):472.
[49] ZHAO Y Q, ZHU C Y, JIANG H. Quantitative detection of Zearalenone in wheat using intervals selection coupled to near-infrared spectroscopy[J]. Infrared Physics &Technology, 2024, 136:105004.
[50] CEBRI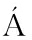 N E, N
N E, N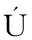
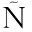 EZ F, RODR
EZ F, RODR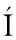 GUEZ M, et al. Potential of near infrared spectroscopy as a rapid method to discriminate OTA and non-OTA-producing mould species in a dry-cured ham model system[J]. Toxins, 2021, 13(9):620.
GUEZ M, et al. Potential of near infrared spectroscopy as a rapid method to discriminate OTA and non-OTA-producing mould species in a dry-cured ham model system[J]. Toxins, 2021, 13(9):620.
[51] DENG J H, JIANG H, CHEN Q S. Characteristic wavelengths optimization improved the predictive performance of near-infrared spectroscopy models for determination of aflatoxin B1 in maize[J]. Journal of Cereal Science, 2022, 105:103474.
[52] 杨承霖, 刘嘉祺, 郭芸成, 等. 结合太赫兹光谱与机器学习的小麦霉变程度判别[J]. 食品科学, 2023, 44(12):343-350.YANG C L, LIU J Q, GUO Y C, et al. Detection of mildew degree of wheat using terahertz spectroscopy and machine learning[J]. Food Science, 2023, 44(12):343-350.
[53] DIB A A, ASSAF J C, DEBS E, et al. A comparative review on methods of detection and quantification of mycotoxins in solid food and feed: a focus on cereals and nuts[J]. Mycotoxin Research, 39: 319-345.
[54] KIM D Y, GETACHEW F, TILLMAN B L, et al. Developing statistical models of aflatoxin risk in peanuts using historical weather data[J]. Agronomy Journal, 2024, 116(5):2346-2361.
[55] KIM Y K, QIN J W, BAEK I, et al. Detection of aflatoxins in ground maize using a compact and automated Raman spectroscopy system with machine learning[J]. Current Research in Food Science, 2023, 7:100647.
[56] ZHANG S, LI Z X, AN J, et al. Identification of aflatoxin B1 in peanut using near-infrared spectroscopy combined with naive Bayes classifier[J]. Spectroscopy Letters, 2021, 54(5):340-351.
[57] 赵雪, 靳欣迪, 刘斌, 等. 辣椒粉中黄曲霉菌生长及其产毒规律的预测模型构建[J]. 食品科学, 2021, 42(14):62-69.ZHAO X, JIN X D, LIU B, et al. Prediction model construction for Aspergillus flavus growth and toxin accumulation in chili powder[J]. Food Science, 2021, 42(14):62-69.
[58] RANBIR, SINGH G, KAUR N, et al. Machine learning driven metal oxide-based portable sensor array for on-site detection and discrimination of mycotoxins in corn sample[J]. Food Chemistry, 2025, 464(Pt 3):141869.
[59] WANG Q A, CHEN J, NI Y Q, et al. Application of Bayesian networks in reliability assessment: A systematic literature review[J]. Structures, 2025, 71:108098.
[60] RAHMAT F, ZULKAFLI Z, ISHAK A J, et al. Supervised feature selection using principal component analysis[J]. Knowledge and Information Systems, 2024, 66(3):1955-1995.
[61] ATA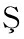 M, YARDIMCI Y, TEMIZEL A. A new approach to aflatoxin detection in chili pepper by machine vision[J]. Computers and Electronics in Agriculture, 2012, 87:129-141.
M, YARDIMCI Y, TEMIZEL A. A new approach to aflatoxin detection in chili pepper by machine vision[J]. Computers and Electronics in Agriculture, 2012, 87:129-141.
[62] LI S L, SHAO X J, GUO Z, et al. Novel detection method for Aspergillus flavus contamination in maize kernels based on spatial-spectral features using short-wave infrared hyperspectral imaging[J]. Journal of Food Composition and Analysis, 2025, 140:107219.
[63] 王蓓, 沈飞, 何学明, 等. 电子鼻同步检测花生霉菌及霉菌毒素[J]. 食品科学, 2022, 43(12): 310-316.WANG B, SHEN F, HE X M, et al.Simultaneous Detection of Harmful Fungi and Mycotoxin Contamination in Peanuts by Electronic Nose[J]. Food Science, 2022, 43(12): 310-316.
[64] CHEN M, HE X M, PANG Y Y, et al. Laser induced fluorescence spectroscopy for detection of Aflatoxin B1 contamination in peanut oil[J]. Journal of Food Measurement and Characterization, 2021, 15(3):2231-2239.
[65] WANG B, SHEN F, HE X M, et al. Simultaneous detection of Aspergillus moulds and aflatoxin B1 contamination in rice by laser induced fluorescence spectroscopy[J]. Food Control, 2023, 145:109485.
[66] SALEHI A, KHEDMATI M. Hybrid clustering strategies for effective oversampling and undersampling in multiclass classification[J]. Scientific Reports, 2025, 15(1):3460.
[67] RABBANI Y, BEHJATI S, LAMBERT B, et al. Prediction of mycotoxin response of DNA-wrapped nanotube sensor with machine learning[J]. ECS Meeting Abstracts, 2023, MA2023-01(10):1223.
[68] PURCHASE J, DONATO R, SACCO C, et al. The association of food ingredients in breakfast cereal products and fumonisins production: Risks identification and predictions[J]. Mycotoxin Research, 2023, 39(3):165-175.
[69] ROCCHETTI G, GHILARDELLI F, MASOERO F, et al. Screening of regulated and emerging mycotoxins in bulk milk samples by high-resolution mass spectrometry[J]. Foods, 2021, 10(9):2025.
[70] 陈靓, 阳佳红, 田星. 机器学习在食品风味领域的研究进展与未来趋势[J]. 食品科学, 2024, 45(10): 28-37.CHEN J, YANG J H, TIAN X. Research Progress and Future Trends of Machine Learning in the Field of Food Flavor[J]. Food Science, 2024, 45(10): 28-37.
[71] 郭香兰, 王立, 金学波, 等. 机器学习-基于GAN和DF结合的粮食加工过程污染物小样本数据扩充及预测[J]. 食品科学, 2024, 45(12): 22-30.GUO X L, YU L, JIN X B, et al. Machine Learning-Small Sample Data Expansion and Prediction of Pollutants in the Grain Processing Process Based on the Combination of GAN and DF[J]. Food Science, 2024, 45(12): 22-30.
[72] KOYAMA H. Machine learning application in otology[J]. Auris, Nasus, Larynx, 2024, 51(4):666-673.
[73] BANG J, YANG B. Application of machine learning to predict the engineering characteristics of construction material[J]. Multiscale Science and Engineering, 2023, 5(1):1-9.
[74] LOU R R, LV Z H, DANG S P, et al. Application of machine learning in ocean data[J]. Multimedia Systems, 2023, 29(3):1815-1824.
Advances in supervised machine learning for mycotoxin detection in foods
ZHANG Shuming1,2, WANG Xin1, ZHENG Lingchun1, BAI Yamin, NIE Xuyuan1, WANG Kai1,2, XU Tong1, FRANCISCO PÉREZ NEVADO4, WANG Qiang1,2*
1(School of Biological and Chemical Engineering, Chongqing University of Education, Chongqing 400067, China)2(Engineering Research Center of Lipid Resources Utilization and Innovation, Chongqing University of Education, Chongqing Education Commission of China, Chongqing 400067, China)3(Chongqing Institute for Food and Drug Control, Chongqing 401121, China)4(Universidad de Extremadura, Badajoz 06001, Spain)
ABSTRACT Mycotoxins are toxic secondary metabolites produced during the growth of molds and are widely present in various foods, directly affecting the quality of food and human health.To ensure food safety and human health, effective measures must be taken to prevent the production and accumulation of mycotoxins in food.Traditional methods for detecting mycotoxins have several drawbacks, such as low detection efficiency, poor sensitivity, and limited applicability.Machine learning, as an auxiliary tool, plays a promoting role in improving detection accuracy and simplifying experimental operations.Among these, supervised machine learning, with its powerful data processing capabilities, precise prediction and classification abilities, potential to uncover food safety issues, and the capacity to integrate with other technologies, has been widely applied in food safety detection, providing strong support for ensuring consumer health and rights.This article focuses on the application progress of supervised learning in the detection of mycotoxins in food, introduces the workflow and commonly used algorithms of supervised learning, discusses the application of supervised learning in detecting mycotoxins in food according to different algorithm types, and looks forward to the development trends of supervised learning in the field of food safety detection.
Key words supervised learning; mycotoxins; food safety; analytical testing; food analytical chemistry



 N等
N等
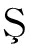 等
等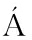 N E, N
N E, N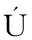
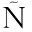 EZ F, RODR
EZ F, RODR GUEZ M, et al. Potential of near infrared spectroscopy as a rapid method to discriminate OTA and non-OTA-producing mould species in a dry-cured ham model system[J]. Toxins, 2021, 13(9):620.
GUEZ M, et al. Potential of near infrared spectroscopy as a rapid method to discriminate OTA and non-OTA-producing mould species in a dry-cured ham model system[J]. Toxins, 2021, 13(9):620. M, YARDIMCI Y, TEMIZEL A. A new approach to aflatoxin detection in chili pepper by machine vision[J]. Computers and Electronics in Agriculture, 2012, 87:129-141.
M, YARDIMCI Y, TEMIZEL A. A new approach to aflatoxin detection in chili pepper by machine vision[J]. Computers and Electronics in Agriculture, 2012, 87:129-141.