克雷伯杆菌烯还原酶基因的异源表达和性质研究
王婉莹1,李志建1*,李泽浩1,王子轩2,张佳月1,王果果1,王婧玥2,段至柔2,张璐璐1*
1(河南工业大学 粮油食品学院,河南 郑州,450001)2(河南工业大学 国际教育学院,河南 郑州,450001)
摘 要 烯还原酶能利用NAD(P)H还原α, β-不饱和烯烃的碳碳双键,将香芹酮还原生成天然香料二氢香芹酮。该研究从克雷伯杆菌O852(Klebsiella sp.O852)中获得烯还原酶基因(命名为KlebER2),在大肠杆菌中进行克隆和异源表达,对其表达条件进行优化,通过镍柱纯化后对其酶学性质和催化合成二氢香芹酮的能力进行研究。结果表明KlebER2蛋白由369个氨基酸组成,是亲水性蛋白,不含有跨膜结构;KlebER2的最佳表达条件为37 ℃培养菌体浓度OD600值至0.5时,用0.8 mmol/L的IPTG在20 ℃、100 r/min的条件下诱导22 h;酶学性质研究显示,KlebER2最适反应温度和pH分别为30 ℃和6.0,在30 ℃以下保持较好稳定性,Mn2+、Mg2+、Ba2+以及丙三醇对其表现出明显的促进作用,KlebER2对多种不饱和烯烃化合物有催化作用,能够催化香芹酮生成二氢香芹酮,转化率达50%以上。研究结果可为烯还原酶的异源表达及进一步工业应用提供指导。
关键词 烯还原酶;异源表达;酶学性质;蛋白纯化;二氢香芹酮
DOI:10.13995/j.cnki.11-1802/ts.040927
引用格式:王婉莹,李志建,李泽浩,等.克雷伯杆菌烯还原酶基因的异源表达和性质研究[J].食品与发酵工业,2025,51(14):65-73.
WANG Wanying,LI Zhijian,LI Zehao, et al.Heterologous expression and characterization of Klebsiella sp.ene reductase gene[J].Food and Fermentation Industries,2025,51(14):65-73.
第一作者:硕士研究生(李志建副教授和张璐璐讲师为共同通信作者,E-mail:zjli@haut.edu.cn;zhanglulu_haut@163.com)
基金项目:国家自然科学基金青年科学基金项目(32101868);河南工业大学校级大学生创新创业训练计划项目;郑州市科技局自然科学项目协同创新专项(22ZZRDZX26);河南工业大学青年骨干教师培养计划项目(21421235);河南工业大学粮油食品学院省部级科研平台开放课题(GO202420)
收稿日期:2024-09-02,改回日期:2024-10-19
含氧单萜二氢香芹酮具有薄荷香气,不仅可以作为风味添加剂用于食品工业中,赋予食品特殊的风味,还可以作为手性构建基用于合成不同的生物活性分子[1]。研究指出二氢香芹酮具有较好的抑菌活性、驱虫性能、抗肿瘤以及抗炎等生物活性[2],已经被广泛地应用于医药、食品、农业、化妆品、烟草以及化工等领域。
目前,二氢香芹酮的主要来源是从植物中直接提取或通过化学方法合成,这些方式不仅消耗大量的人力物力,而且成本高、工艺过程复杂、环境不友好[3]。与之相比,生物催化是利用酶或者生物有机体作为催化剂对底物进行催化转化,具有反应条件温和、副产物少、专一性强、催化效率高、产物易于分离、环境友好等优点,受到了广泛的关注。烯还原酶能够利用NAD(P)H还原α, β-不饱和烯烃的碳碳双键,将香芹酮还原生成二氢香芹酮,是生物催化合成的重要酶类之一。在过去的研究中,科研工作者已经在真菌、细菌、酵母和植物中鉴定出许多的烯还原酶,比如Syn7942ER(蓝藻Synechococcus sp.PCC 7942)[4]、Ppo-Er1(多粘芽孢杆菌Paenibacillus polymyxa)[5]和MgER(季也蒙念珠菌Meyerozyma guilliermondii)[6]。虽然这些酶具有巨大的研究价值,但是目前用于应用研究的烯还原酶类别较少,稳定性和耐受性较差,缺少真正能用于大规模生产应用的酶。
本实验室在前期研究中筛选得到一株可降解柠檬烯的克雷伯杆菌O852(Klebsiella sp.O852),能够氧化柠檬烯生成二氢香芹酮。通过基因组测序,筛选鉴定出一个催化二氢香芹酮合成的烯还原酶基因O852_GE001833,命名为KlebER2[7]。因此,本研究拟以KlebER2为研究对象,构建重组表达载体pET-28a-KlebER2,将其导入大肠杆菌中进行克隆和异源表达,并对重组蛋白进行纯化以及酶学性质表征,研究其催化香芹酮合成二氢香芹酮的性能。本研究不仅深化了对烯还原酶的认识,也为改造可还原α, β-不饱和化合物的烯还原酶提供基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
Klebsiella sp.O852为本课题组保存,表达载体pET-28a(+),以及Escherichia coli DH5α和E.coli BL21(DE3)分别用于基因的克隆和表达。
1.1.2 主要试剂
卡那霉素、5×蛋白上样缓冲液、IPTG、彩虹180广谱蛋白marker、BCA蛋白定量测定试剂盒、镍-琼脂糖凝胶6FF、层析柱均,北京索莱宝科技有限公司;香芹酮、二氢香芹酮,上海麦克林生化科技股份有限公司;还原型辅酶NADH和NADPH,上海源叶生物科技有限公司;其他相关试剂均为市售分析纯。
LB培养基(g/L):NaCl 10,蛋白胨10,酵母浸粉5,pH自然,121 ℃高压蒸汽灭菌30 min。
1.2 仪器与设备
Agilent 7890A型气相色谱仪,美国Agilent公司;YX280/15高压蒸汽灭菌锅,上海三申医疗器械有限公司;SW-CJ-1D型洁净工作台,上海力辰邦西仪器科技有限公司;固相微萃取纤维头(50/30 μmDVB/CAR/PDMS),美国Supeleo公司;BSD-TS270振荡培养箱,上海博迅医疗生物仪器股份有限公司厂;722 s分光光度计,上海精密科学仪器有限公司;JY88-IIN超声波细胞破碎仪,宁波新芝生物科技股份有限公司;DZCZ-24DN型电泳仪,北京市六一机械厂;TGL-16B高速冷冻离心机,上海安亭科学仪器厂。
1.3 实验方法
1.3.1 KlebER2生物信息学分析
生物信息学分析预测内容及网址见表1。
表1 生物信息学工具
Table 1 Bioinformatics tools

网址预测内容 https://web.expasy.org/protparam/理化性质分析https://services.healthtech.dtu.dk/services/SignalP-4.1/信号肽预测https://web.expasy.org/protscale/亲疏水性分析https://services.healthtech.dtu.dk/services/TMHMM-2.0/跨膜结构域预测https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa%20_sopma.html蛋白二级结构预测https://swissmodel.expasy.org/蛋白三级结构预测
1.3.2 重组质粒pET-28a-KlebER2构建
根据Primer 5软件设计引物,并委托上海生工生物有限公司合成,见表2。以Klebsiella sp.O852基因组为模板进行PCR扩增,将PCR产物进行1%琼脂糖凝胶电泳,试剂盒回收获得目的基因片段。使用限制性内切酶NcoI和XhoI对上述目的基因片段和载体pET-28a(+)进行双酶切,并通过T4-DNA连接酶将酶切后的产物和载体进行连接,构建重组质粒pET-28a-KlebER2,将重组质粒转化至E.coli DH5α感受态中,菌落PCR后筛选阳性克隆子,并进行测序验证。
表2 引物设计
Table 2 Primer design
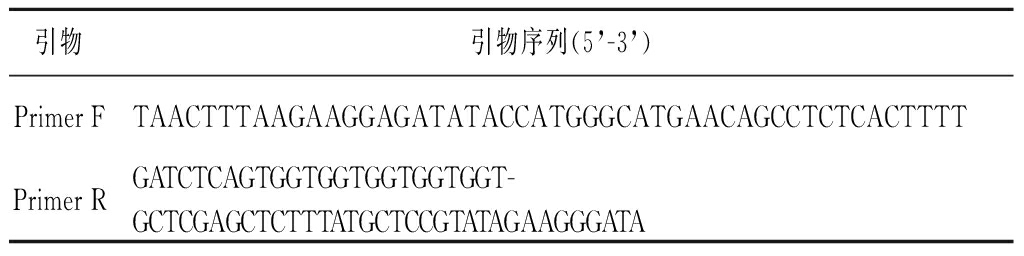
引物引物序列(5’-3’)Primer FTAACTTTAAGAAGGAGATATACCATGGGCATGAACAGCCTCTCACTTTTPrimer RGATCTCAGTGGTGGTGGTGGTGGT-GCTCGAGCTCTTTATGCTCCGTATAGAAGGGATA
1.3.3 重组蛋白诱导表达及优化
将上述验证成功的重组载体pET-28a-KlebER2导入E.coli BL21(DE3)中进行异源表达,获得重组菌株。将重组菌株活化,振荡培养后获得种子液。按1%的接种量,将种子液接种至含50 μg/mL卡那霉素的LB液体培养基中,培养至OD600=0.5~0.7,添加0.5 mmol/L IPTG溶液进行诱导,在24 ℃、120 r/min的条件下诱导培养10 h。离心收集菌体,向菌体内加入适量的Tris-HCl缓冲液重悬,超声破碎后再次离心收集破碎后的上清液和沉淀。分别取未诱导和诱导后的菌体、以及诱导后经过超声破碎的上清液和沉淀,进行SDS-PAGE,分析KlebER2蛋白的可溶性表达。
为了提高KlebER2蛋白的表达量,通过单因素实验对诱导温度(16、20、24、28、32、36 ℃)、诱导时间(6、10、14、18、22、26 h)、IPTG浓度(0.2、0.4、0.6、0.8、1 mmol/L)和转速(100、120、140、160、180 r/min)进行优化,通过SDS-PAGE电泳表征,分析蛋白表达情况。
1.3.4 重组蛋白纯化
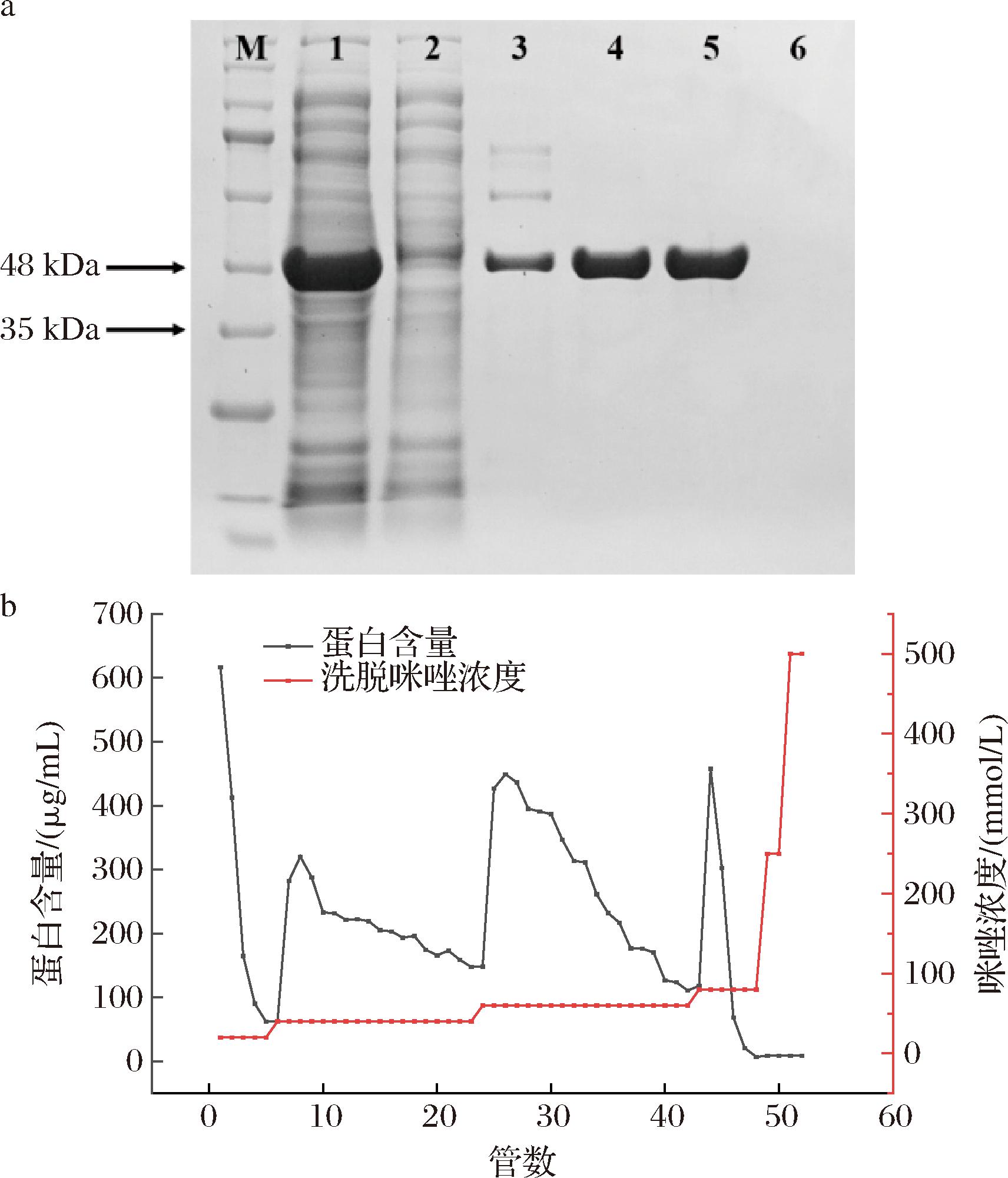
使用镍柱亲和层析对重组蛋白进行分离纯化。将超声破碎后离心得到的上清液用0.22 μm滤膜过滤后缓慢上样,分别用含20、40、60、80、250 mmol/L咪唑的Tris-HCl缓冲液进行阶段洗脱,分管收集各阶段的洗脱液,测定其蛋白浓度并进行SDS-PAGE电泳,检测蛋白的分子质量大小和纯度,选择电泳条带单一且符合相对分子质量的洗脱液进行透析浓缩,获得纯化后的酶。
1.3.5 酶活力的测定
酶活力的测定参照LI等[8]方法,反应总体系为1 mL,分别加入终浓度为25 μg/mL纯化后的酶、0.5 mmol/L NADH、6 mmol/L香芹酮,用所需pH的缓冲液补至1 mL。30 ℃下恒温振荡混匀10 h后,测定340 nm下吸光度的变化。一个酶活力单位(U)的定义:在上述条件下,每小时消耗1 mmol/L NADH所需的酶量。
1.3.6 KlebER2酶学性质研究
(1)最适温度和温度稳定性
将整个反应体系分别置于不同温度(20、30、40、50、60 ℃)下进行反应,确定KlebER2的最适反应温度。
将纯化后的酶分别置于不同温度下孵育3 h,随后在最适反应温度下进行反应,研究KlebER2的热稳定性,以未孵育的酶活力为100%进行计算。
(2)最适pH和pH稳定性
用不同pH(4.0、5.0、6.0、7.0、8.0、9.0、10.0)的缓冲液补至1 mL体系,在最适温度下反应,确定KlebER2的最适pH。
将纯化后的酶分别置于不同pH的缓冲液中孵育3 h,随后在最适反应温度和pH的条件下反应,研究KlebER2的pH稳定性,以未孵育的酶活力为100%进行计算。
(3)金属离子和有机溶剂
分别在反应体系中添加终浓度为1 mmol/L金属离子(K+、Fe2+、Ca2+、Mg2+、Cu2+、Mn2+、Ba2+)和20%有机溶剂(甲醇、丙三醇、正丁醇、乙酸乙酯、二氯甲烷、正己烷、二甲基亚砜),研究金属离子和有机溶剂对KlebER2酶活力的影响,以不添加金属离子或有机溶剂的酶活力为100%进行计算。
(4)底物特异性
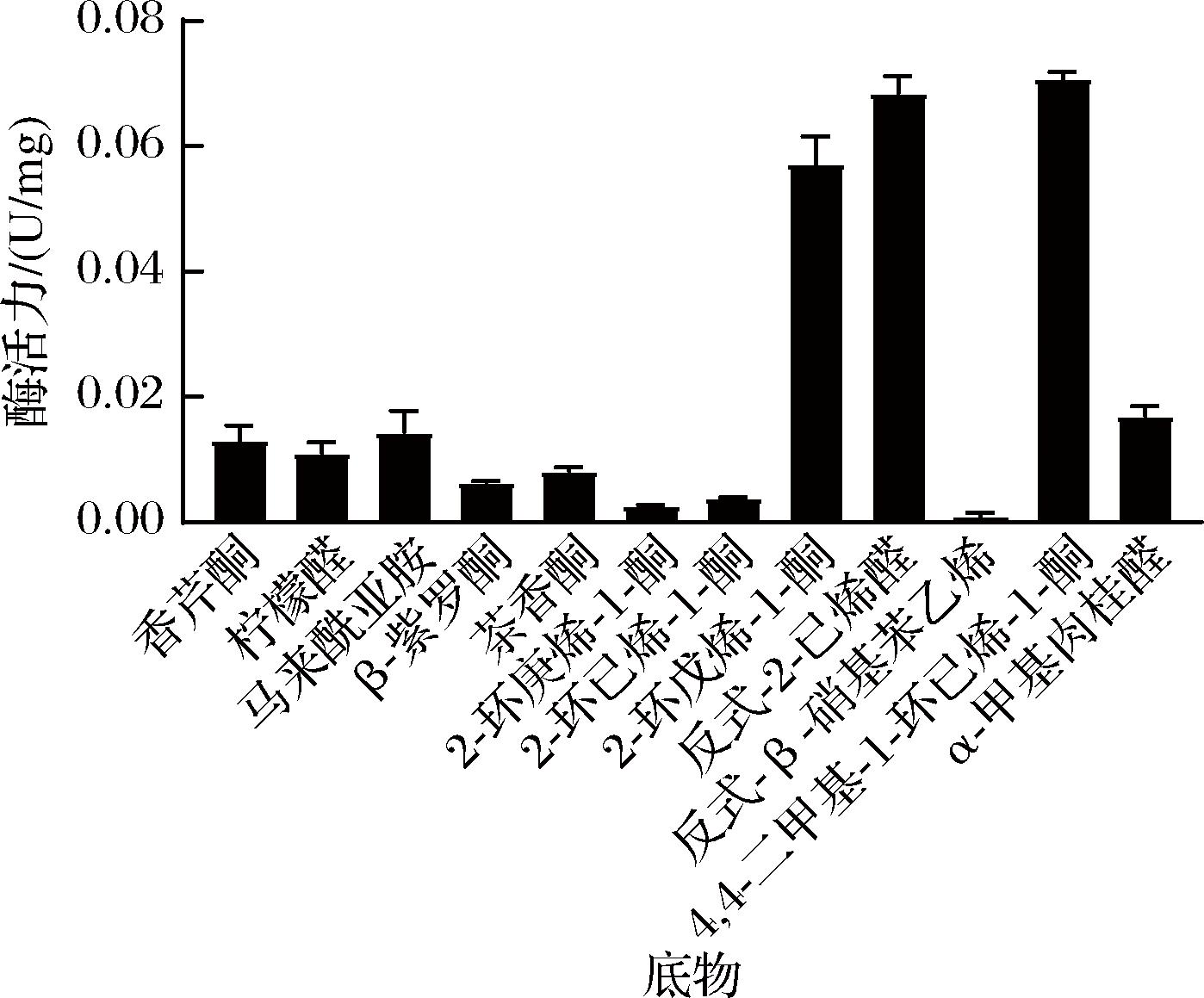
以香芹酮、柠檬醛、马来酰亚胺、β-紫罗酮、茶香酮、2-环庚烯-1-酮、2-环已烯-1-酮、2-环戊烯-1-酮、反式-2-已烯醛、反式-β-硝基苯乙烯、4,4-二甲基-1-环已烯-1-酮、α-甲基肉桂醛作为底物进行反应,研究KlebER2的底物特异性。
(5)辅酶偏好性
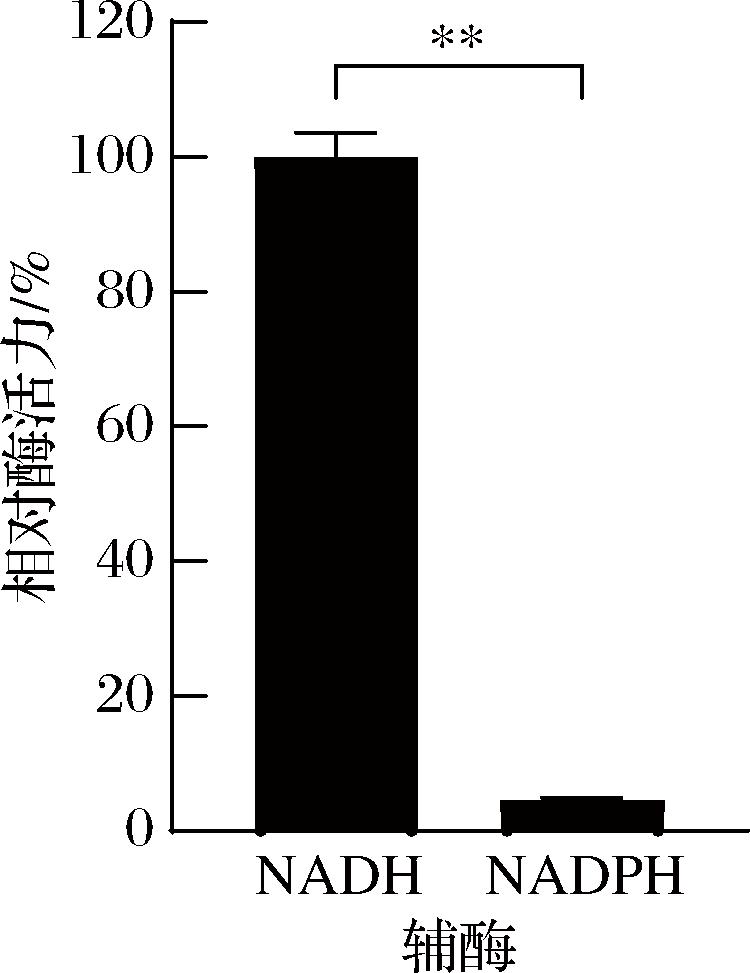
分别使用NADH和NADPH进行反应,研究KlebER2的辅酶偏好性。
1.3.7 KlebER2生物催化香芹酮合成二氢香芹酮
催化反应总体系与酶活测定体系相同,采用固相微萃取/气相色谱-质谱连用仪(solid-phase microextraction/gas chromatography- mass spectrometry,SPME/GC-MS)测定转化产物[8],实验重复3次。使用计算机谱和二氢香芹酮标准品进行定性分析,使用外标法进行二氢香芹酮定量分析。
1.4 数据处理
使用Image J对SDS-PAGE电泳条带的灰度值进行分析,使用SPSS 24.0对数据进行单因素方差分析,采用邓肯法进行显著性分析(P<0.05,P<0.01),使用GraphPad Prism 8.0和Origin 2024进行数据分析和绘图。所有数据均为3次实验重复所得。
2 结果与分析
2.1 KlebER2生物信息学分析
使用ProtParam在线工具分析KlebER2的理化性质,结果表明KlebER2由369个氨基酸组成,分子质量40.57 kDa,等电点6.21,脂肪族指数82.87;KlebER2不含跨膜结构,无信号肽。二级结构结果显示,该蛋白的二级结构主要以无规则卷曲为主,占比52.20%,其次为α螺旋、延伸链(图1-a)。以黄素还原酶V3PMM0.1.A的蛋白序列为基础进行三级结构建模预测(图1-b),并利用Ramachandran Plot评价该蛋白三维结构的合理性(图1-c)。结果表明,95.64%的氨基酸残基位于支持区域,说明该蛋白三维结构合理。
2.2 重组质粒pET-28a-KlebER2构建
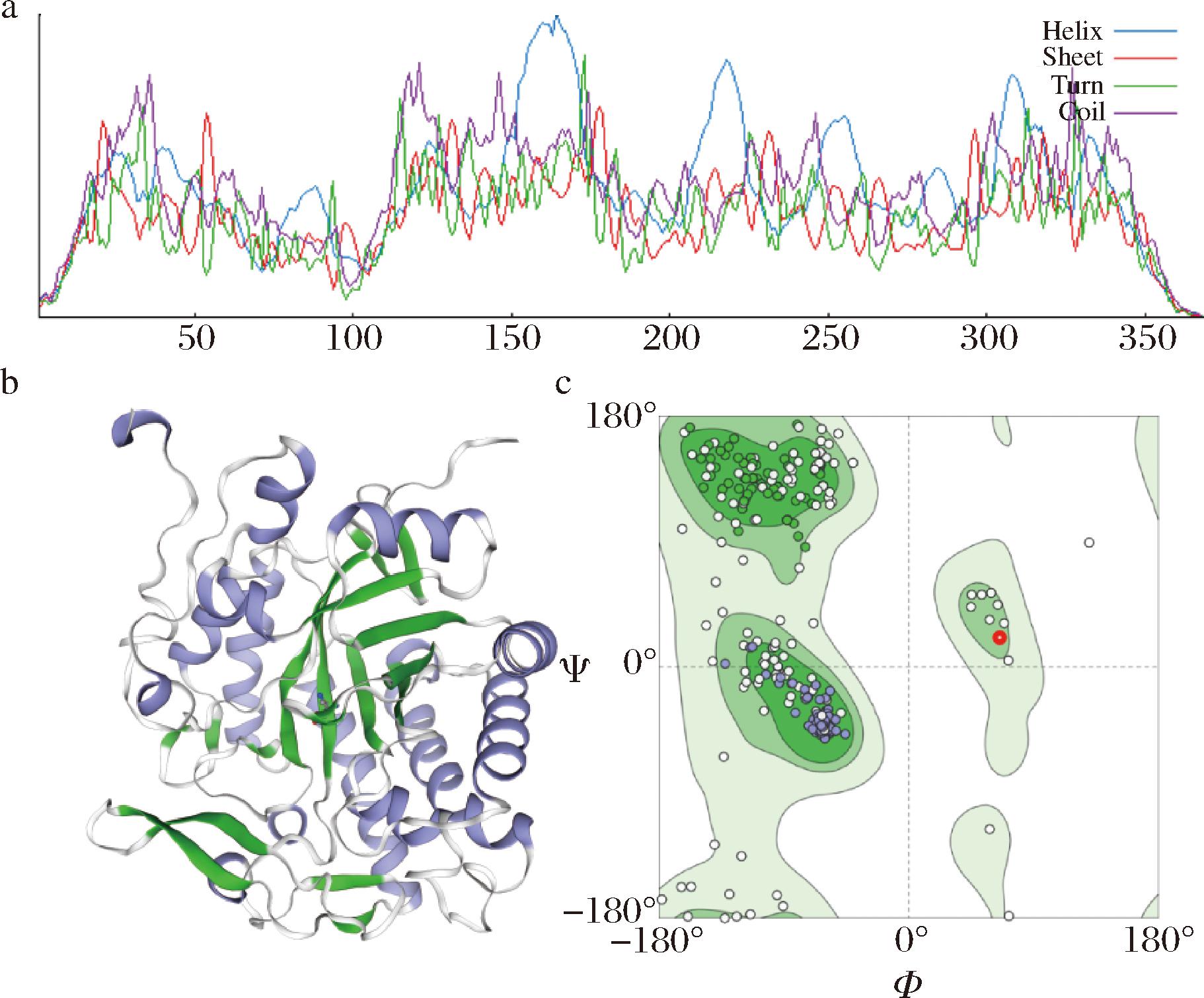
以Klebsiella sp.O852基因组为模板扩增目的基因KlebER2,琼脂糖凝胶电泳结果显示扩增条带大小与目的基因片段大小(1 110 bp)相符(图2-a)。随后,将克隆成功的目的基因片段与载体pET-28a(+)连接,构建重组质粒pET-28a-KlebER2,并转化至E.coli BL21(DE3) 感受态细胞,获得重组菌株,进行菌落PCR验证(图2-b)。
2.3 重组蛋白诱导表达及优化
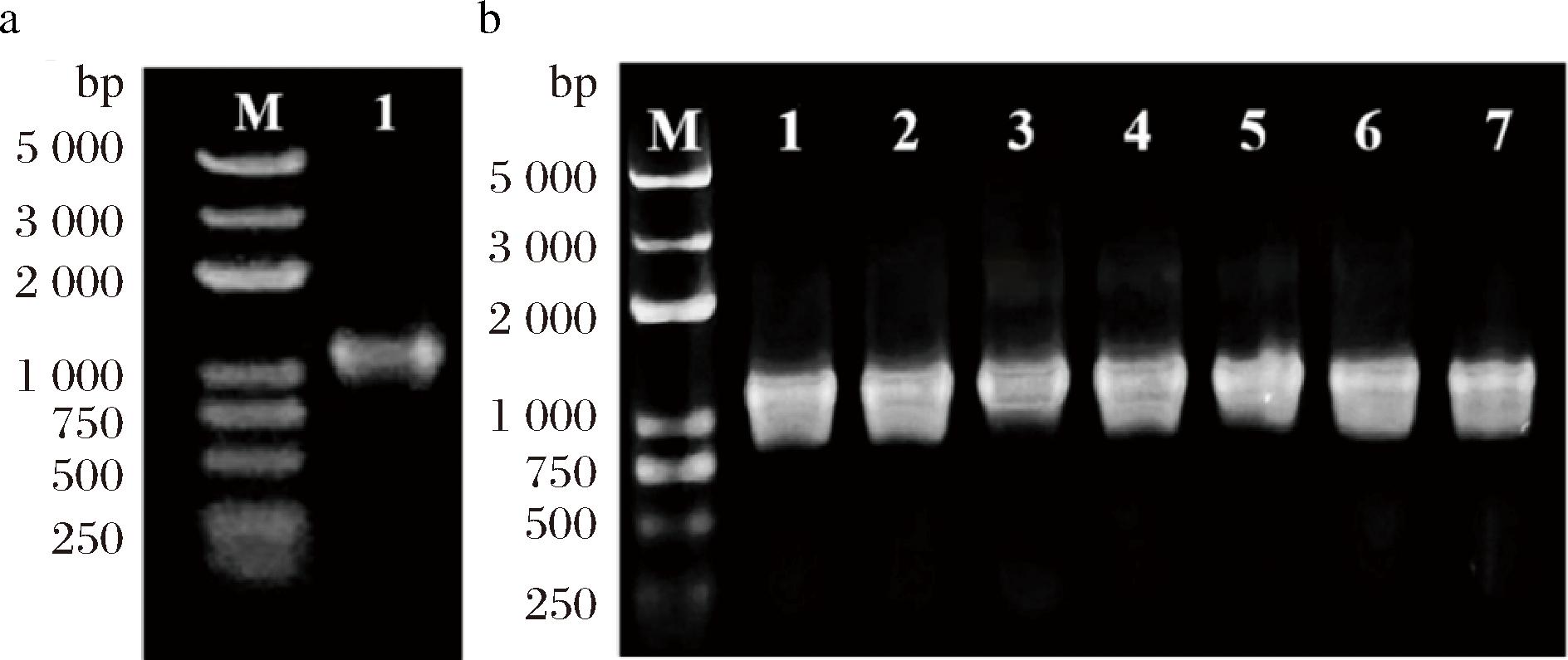
如图3所示,通过比对条带1和2可知,在分子质量为35~48 kDa处有一条明显的蛋白条带,在经过IPTG诱导之后该条带明显变粗,与KlebER2的分子质量(40.57 kDa)一致,说明KlebER2蛋白在大肠杆菌中成功表达。比对条带3和条带4可知,超声破碎之后的上清液中也存在KlebER2蛋白,但含量较低,大部分的蛋白都在沉淀当中,形成包涵体。因此,需要进一步进行优化实验以提高蛋白的可溶性表达。
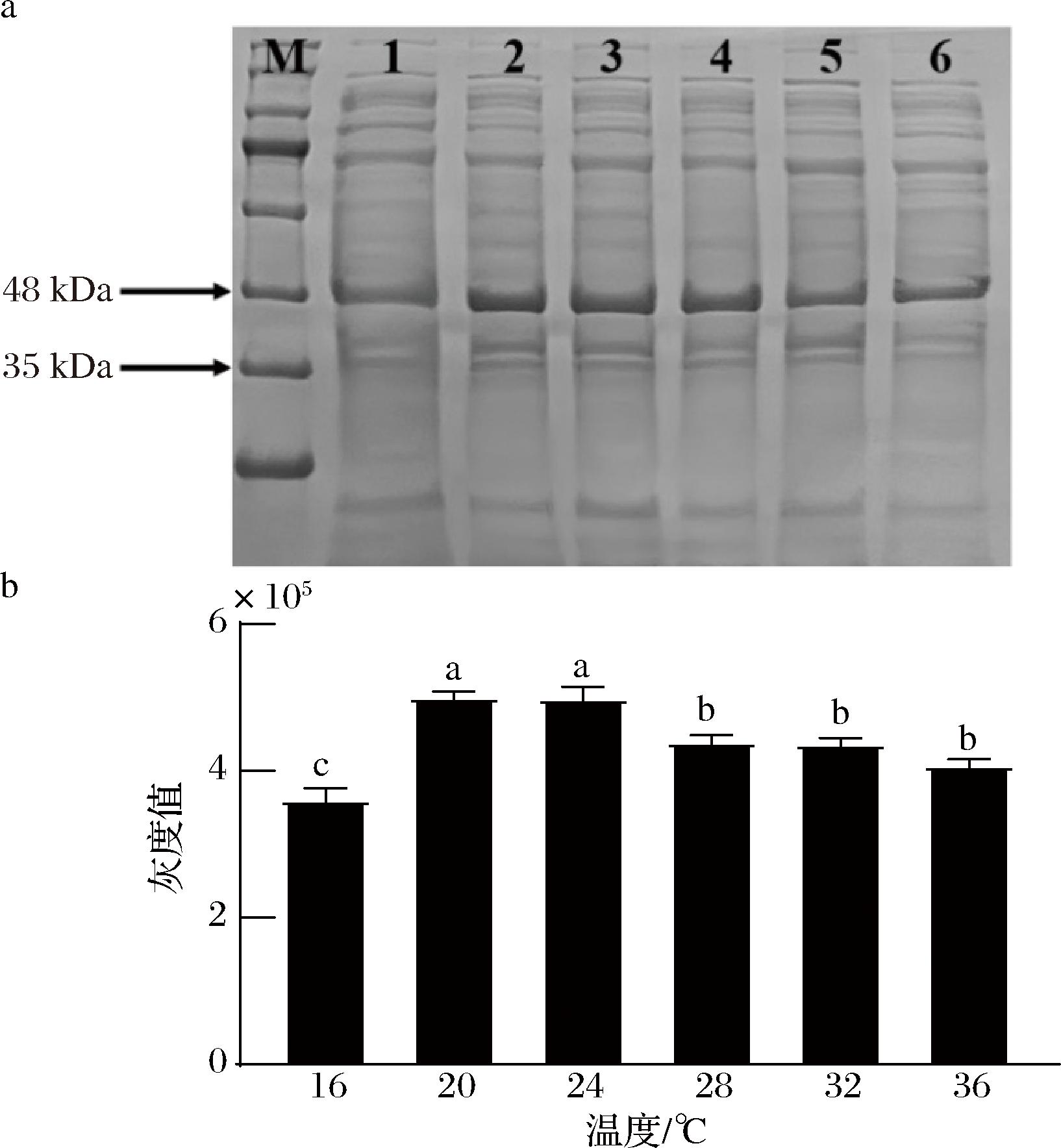
2.3.1 诱导温度优化
由图4可以明显看出,KlebER2蛋白的表达量随温度的升高呈现先升后降的趋势,当温度为20 ℃时,蛋白的表达量达到最大。诱导温度过高,一方面会影响质粒的稳定性,从而影响蛋白的表达,另一方面会导致蛋白不正确折叠、聚集,形成包涵体,并且高温可能会使蛋白失去活性[9];低温诱导能够减慢蛋白质合成的速率,影响多肽折叠的动力学,从而使得正确折叠的蛋白数量增加,减少包涵体的形成[10]。上述结果表明KlebER2的最适诱导温度为20 ℃。
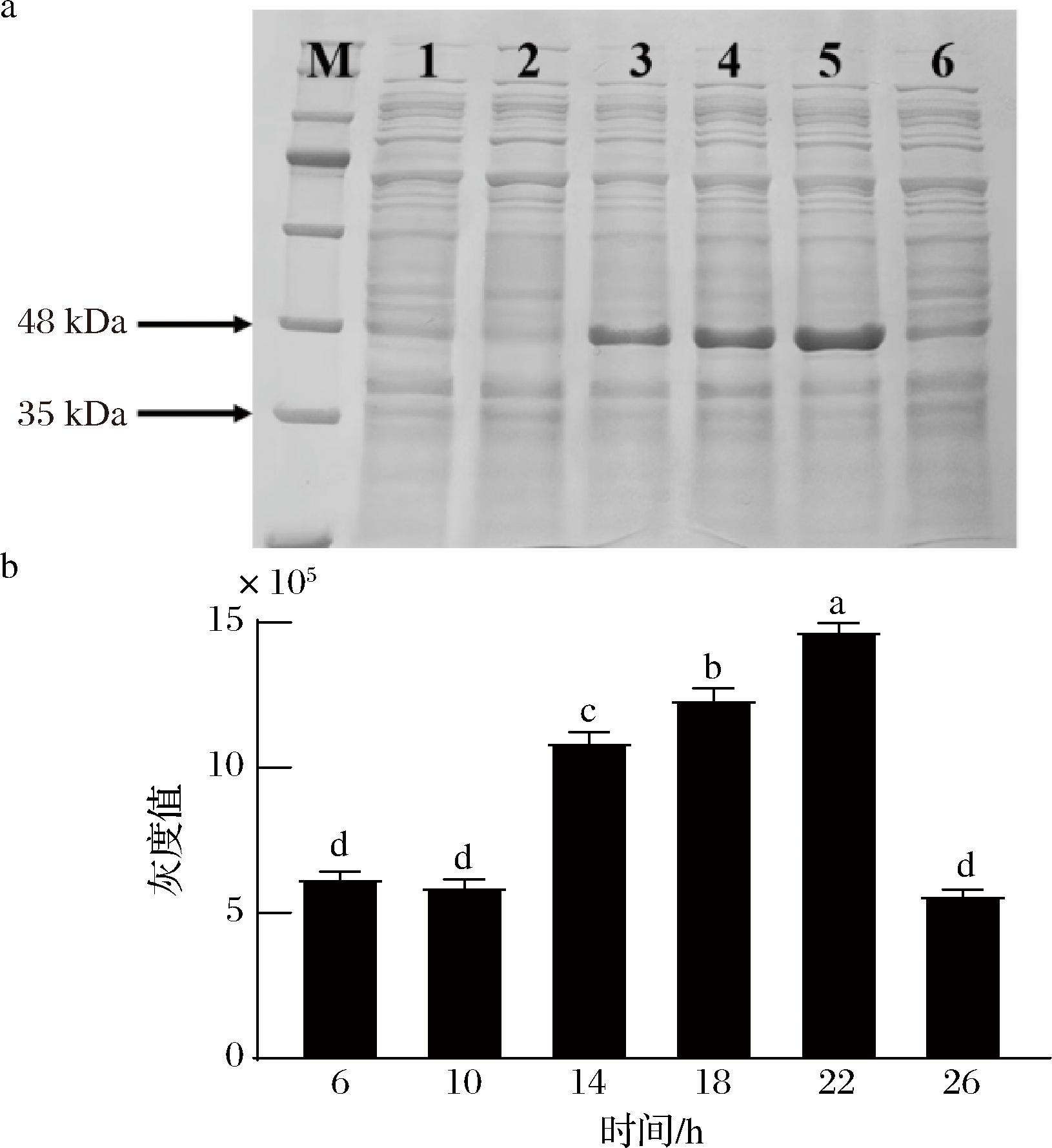
2.3.2 诱导时间优化
如图5所示,KlebER2蛋白的表达随着诱导时间的延长先升高后降低,诱导时间为22 h时,蛋白的表达量达到最大值,随着诱导时间的继续延长,KlebER2蛋白的表达量下降,这可能是因为诱导时间过短,重组蛋白积累量较少,表达量较低;当诱导时间过长时,培养基中营养物质减少,目的蛋白在表达的同时被降解,此外诱导时间过长,细菌会出现溶菌现象,也会使蛋白表达量降低[11]。因此,选择诱导时间为22 h进行后续实验。
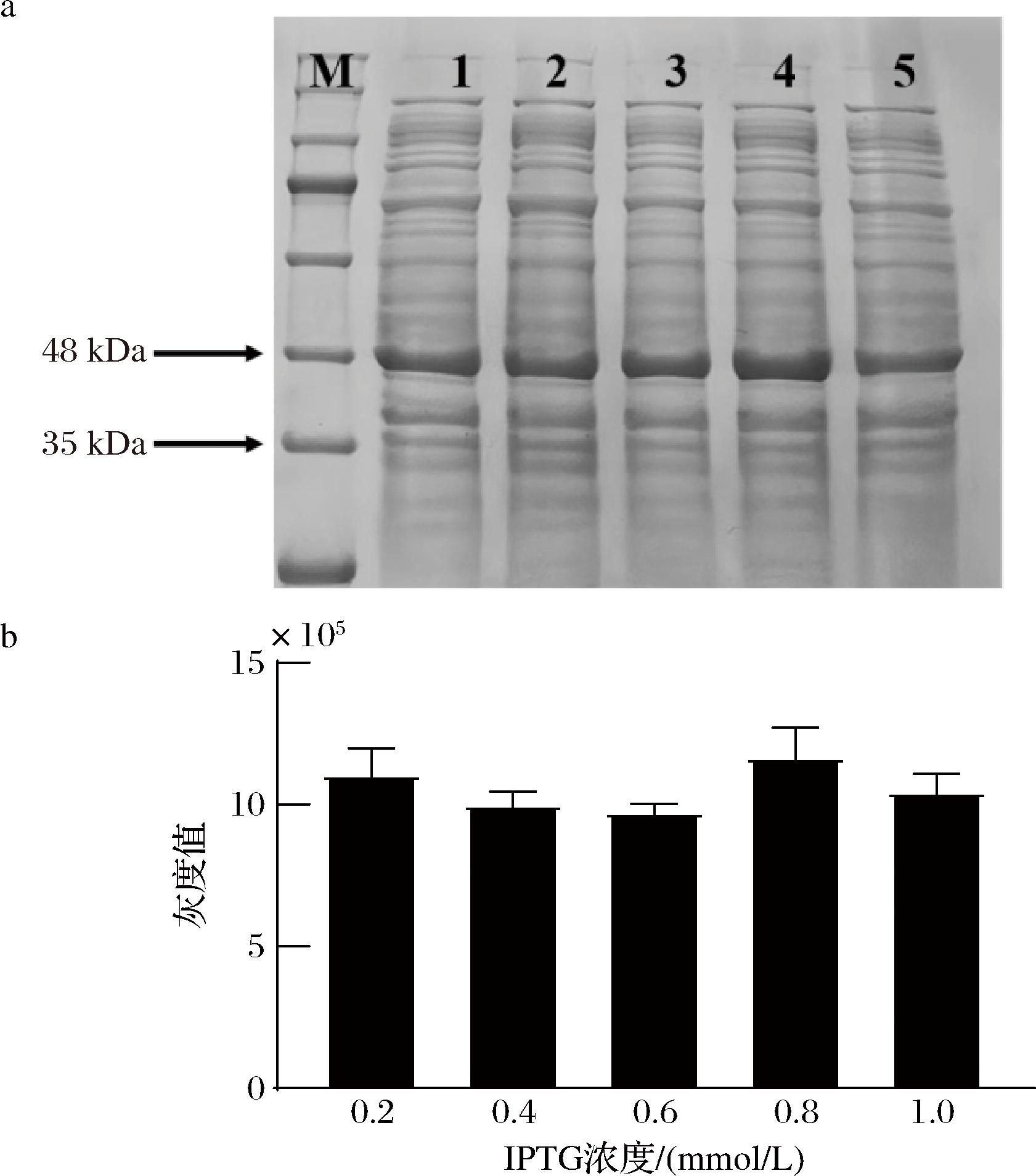
2.3.3 IPTG浓度优化
由图6可知,IPTG浓度对KlebER2蛋白的表达影响较小。当IPTG浓度为0.8 mmol/L时,KlebER2蛋白的表达量达到最大值,之后随着IPTG浓度的增加,KlebER2蛋白的表达量下降。IPTG浓度过低可能导致诱导剂的量不足以与阻遏蛋白结合,从而使蛋白表达量降低,浓度过高又可能会使蛋白表达过快,从而形成包涵体[12]。此外IPTG具有毒性,浓度过高也会影响菌体的生长,添加适量的IPTG既能降低菌体细胞的代谢负荷,也能提高蛋白的表达水平[13]。因此,IPTG的最佳浓度为0.8 mmol/L。
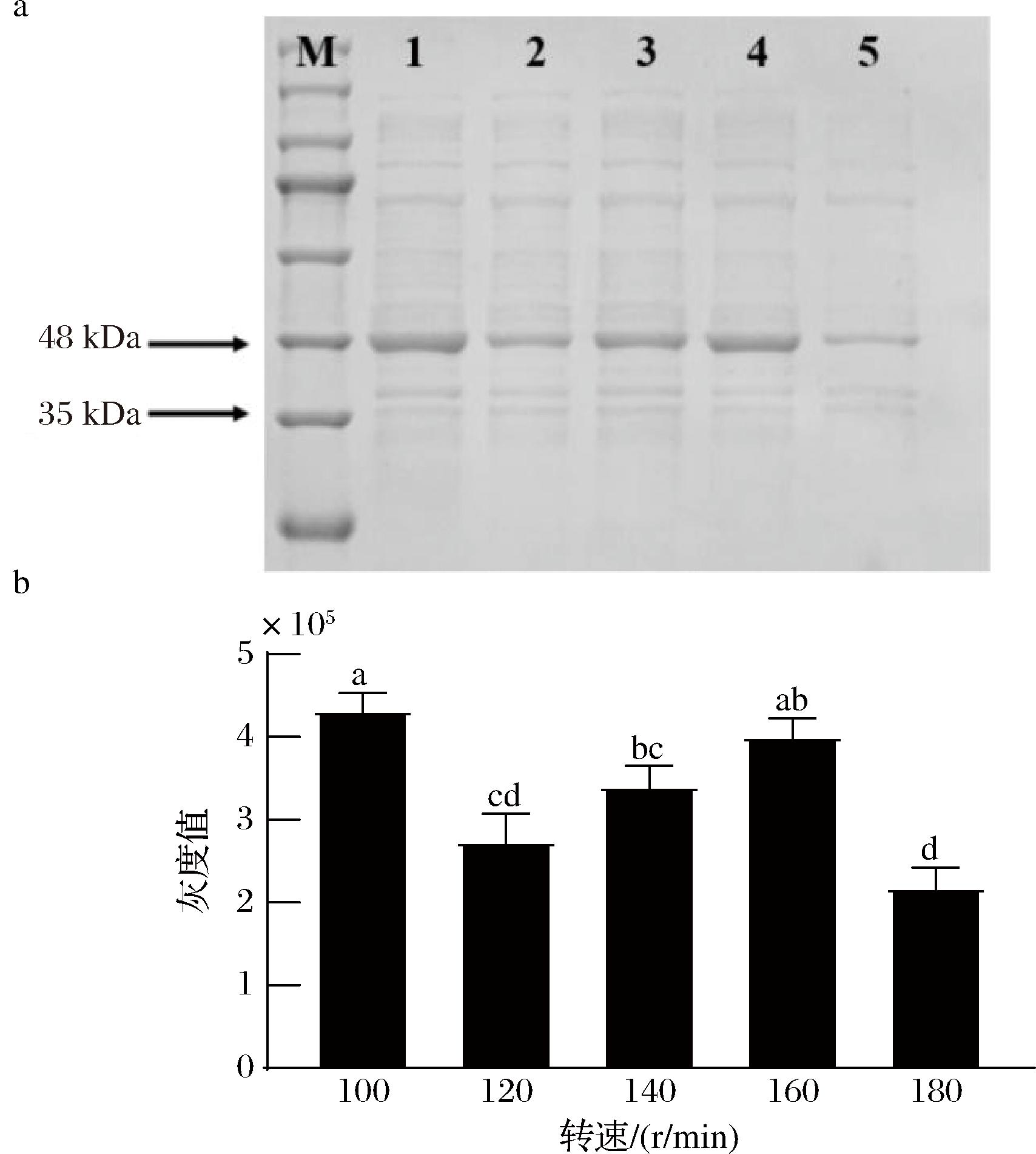
2.3.4 诱导转速优化
如图7所示,当诱导转速为100 r/min时,KlebER2蛋白的表达量达到最大值。随着转速的继续增加,蛋白的表达量下降,这可能是由于转速增加,对细胞的机械损伤以及蛋白所受到的剪切力增加,不利于蛋白的正确折叠,从而导致KlebER2表达量降低[14]。因此,KlebER2的最佳诱导转速为100 r/min。
2.4 重组蛋白纯化
由图8可知,在浓度为60、80 mmol/L咪唑缓冲液的作用下,均有一条明显且单一的蛋白条带被洗脱,分子质量为35~48 kDa,与预期一致(40.57 kDa),认为是该条带为KlebER2蛋白条带。相比于60 mmol/L咪唑缓冲液的纯化效果,80 mmol/L咪唑缓冲液纯化后的蛋白含量明显增加,表明80 mmol/L咪唑是KlebER2蛋白洗脱的最佳浓度。经计算,1 L发酵液收集的菌体经纯化后能获得23 mg纯酶,酶活力为0.056 U/mg。
2.5 酶学性质研究
2.5.1 最适温度和温度稳定性
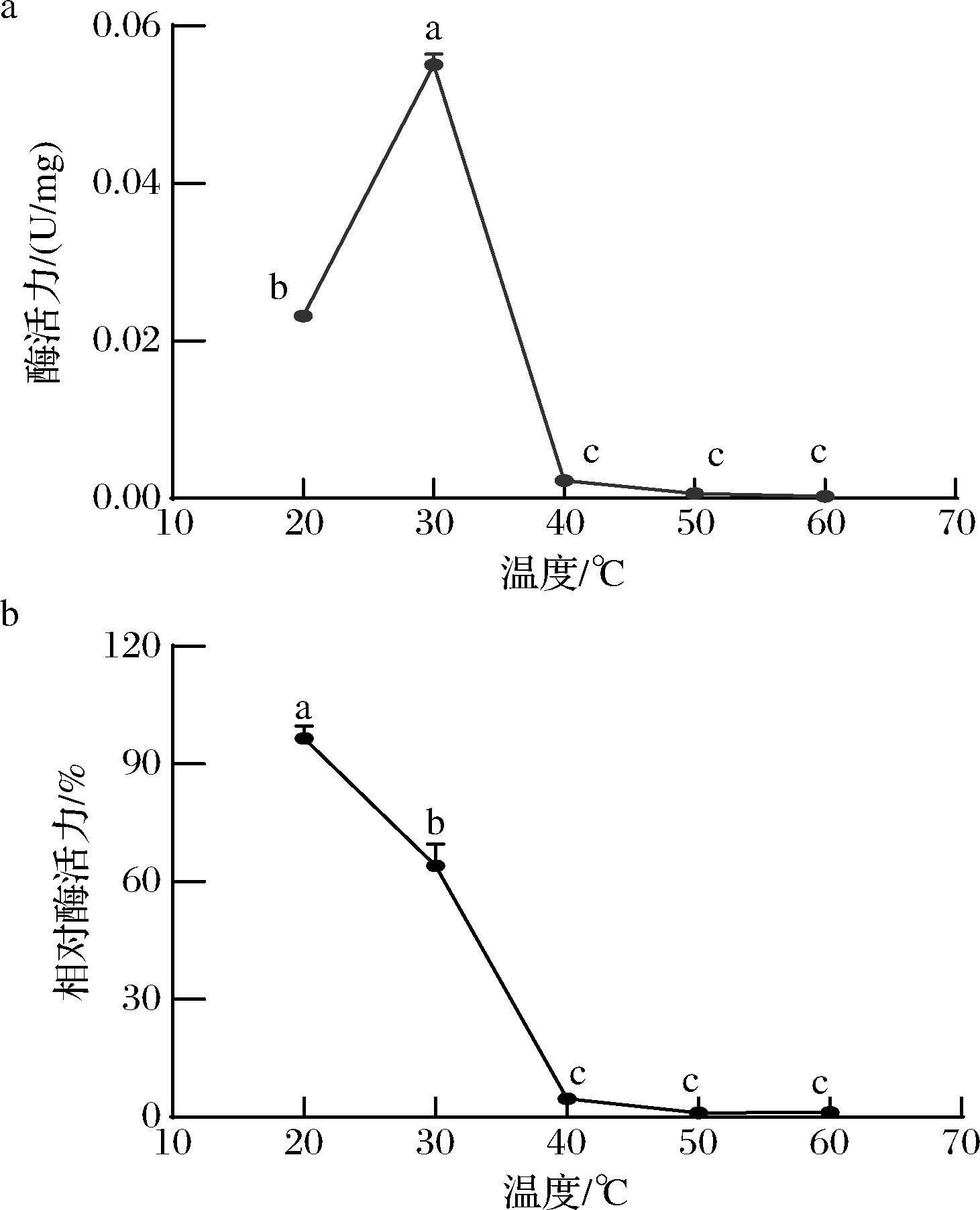
温度是影响酶活力的一个重要因素,温度过低会影响酶反应速率,酶活力较低,随着温度的升高,底物分子热运动加快,分子碰撞的机会增加,酶活力增加,但温度过高会破坏酶蛋白的结构,导致酶活力降低或失活。如图9-a所示,KlebER2的酶活力随着反应温度的增加呈先升后降的趋势,在30 ℃达到最高值。当反应温度达到40 ℃时,酶活力骤降,这可能是由于温度过高导致酶活力降低[15]。因此KlebER2最适反应温度为30 ℃,这和之前文献所报道的烯还原酶EaER2、PvER1和PvER2最适反应温度相同[16]。
温度稳定性结果如图9-b所示,KlebER2的酶活力随着温度的升高而降低,在孵育温度为20 ℃时,相对酶活力达90%以上,40 ℃孵育后相对酶活力仅剩10%左右,50 ℃孵育后酶基本失活。以上结果表明KlebER2对温度较为敏感,这在一定程度上限制其应用。
2.5.2 最适pH和pH稳定性
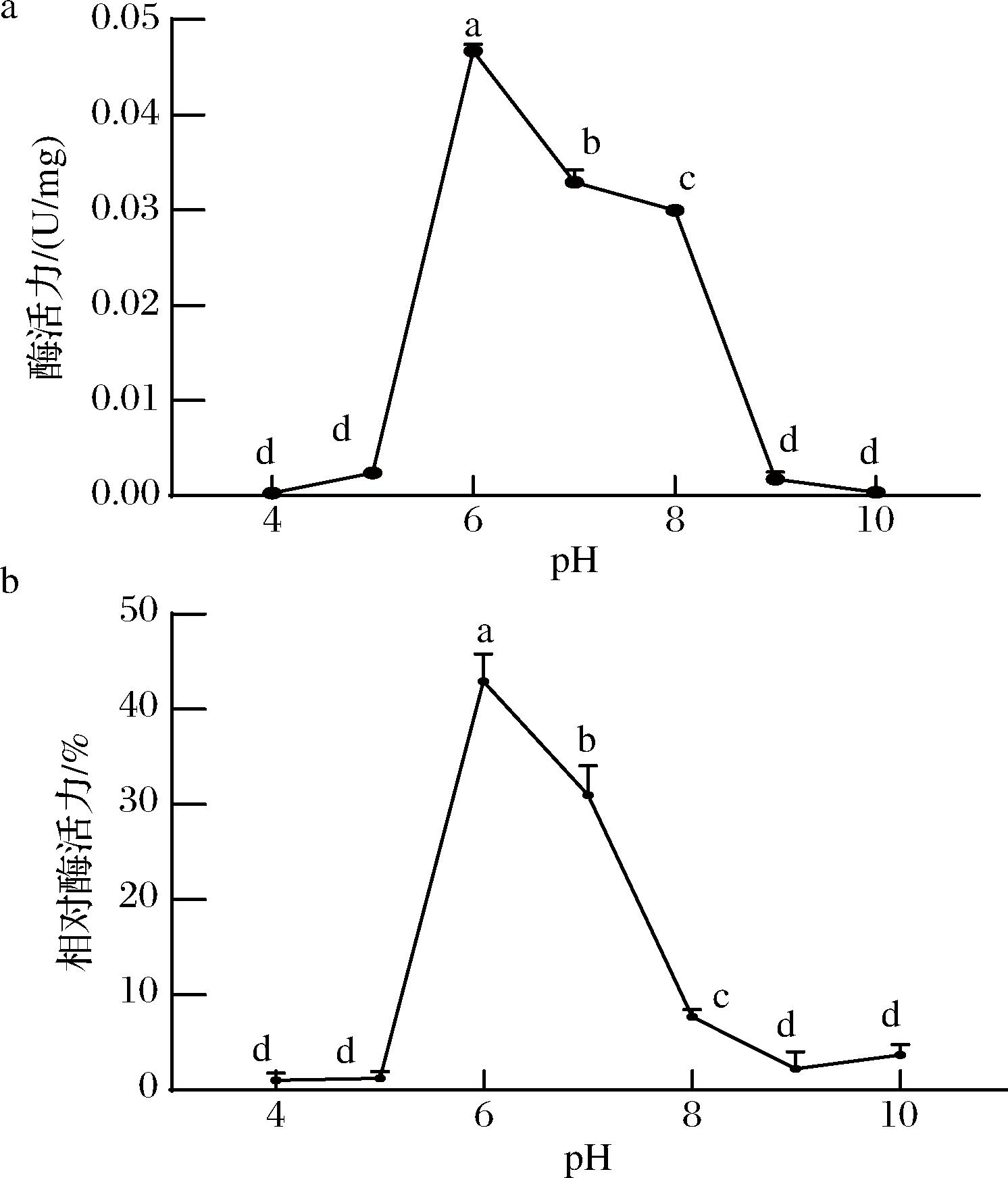
pH的改变会使酶的基团发生变化,空间结构发生改变,从而影响酶与底物之间的结合与解离,进而影响酶的催化功能,当酶处于过酸或过碱的环境时,都会造成酶的不可逆的失活[17]。如图10-a所示,KlebER2蛋白的最适反应pH为6.0。在pH 4.0~5.0时,酶活力较低;当pH>6.0时,KlebER2酶活力降低;当pH为10.0时,酶几乎完全失活,这表明KlebER2可能具有严格的pH依赖性,只在很窄的pH范围内起作用,在使用过程中必须严格控制反应系统的pH值[9]。
KlebER2的pH稳定结果如图10-b所示,KlebER2仅在pH值为6.0~7.0时能够保持较高的酶活力,在其余pH下丧失了大量酶活力,表明KlebER2对pH的耐受性较差。
2.5.3 金属离子和有机溶剂
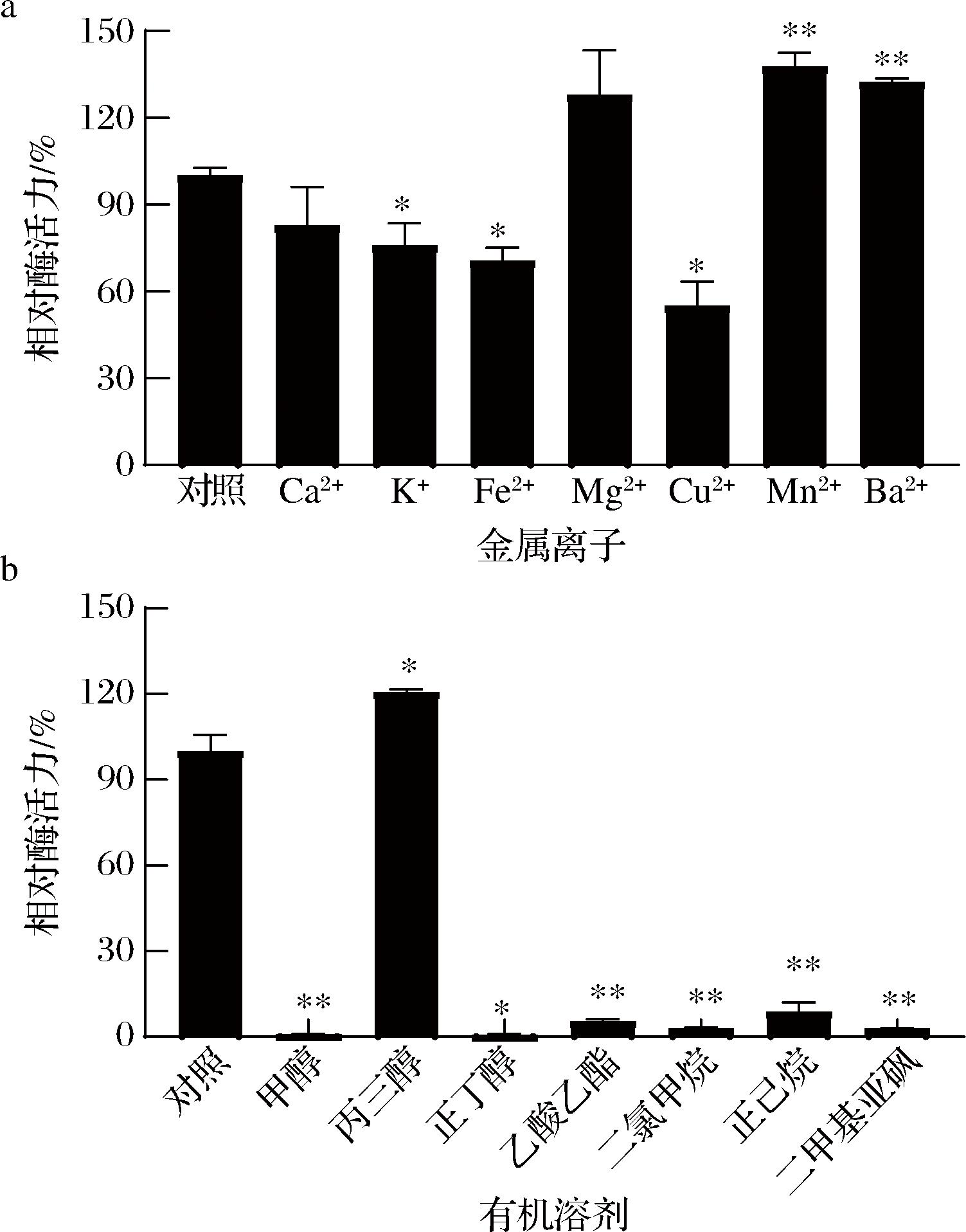
金属离子可以与酶结合改变酶的构象或者破坏酶的催化活性中心结构,从而影响酶的活力[17]。由图11-a可知,Mg2+、Mn2+以及Ba2+均对KlebER2起促进作用,其中Mn2+的促进作用最为明显,相对酶活力达137%。Ca2+、K+、Fe2+以及Cu2+对KlebER2均有不同程度的抑制,这可能是这些离子影响KlebER2的活性中心结构所致。
有机溶剂常用于生物催化反应,以增加底物的溶解度,对生物催化反应有重要影响[18]。然而,在某些溶液或试剂中,酶分子结构的化学键可能发生改变,导致活性基团被破坏,从而影响酶的活力[19]。由图11-b可知,KlebER2受有机溶剂影响较大,除丙三醇外,其余有机溶剂均对KlebER2有明显的抑制作用。
2.5.4 底物特异性
如图12所示,KlebER2对多种α-β不饱和双键化合物都具有催化作用[20],在2-环戊烯-1-酮、反式-2-己烯醛、4,4-二甲基-1-环己烯-1-酮为底物的反应体系中,KlebER2的酶活力较高,即对环烯类化合物有良好的催化能力,这与来源于Synechococcus sp.的烯还原酶Syn7942ER的底物特异性相似[4]。其次,KlebER2对香芹酮、柠檬醛、马来酰亚胺、β-紫罗酮、茶香酮以及α-甲基肉桂醛也具有一定的催化作用,对2-环庚烯-1-酮、反式-β-硝基苯乙烯则几乎没有催化能力。
2.5.5 辅酶偏好性
烯还原酶能够还原α, β-不饱和烯烃的碳碳双键,同时将NAD(P)H氧化为NAD(P)+,不同的烯还原酶可能具有不同的辅酶偏好性。在本实验中,以香芹酮为底物时,KlebER2更偏好NADH,对NADH的酶活力是NADPH的22倍(图13)。这与来源于无色杆菌(Achromobacter sp.)的烯还原酶Achr-OYE4的辅酶偏好性一致[21]。大多数烯还原酶是偏好NADPH[22],NADPH价格远高于NADH,因此,KlebER2偏好NADH的性质为其工业化应用奠定了良好基础。
2.6 KlebER2生物催化香芹酮合成二氢香芹酮
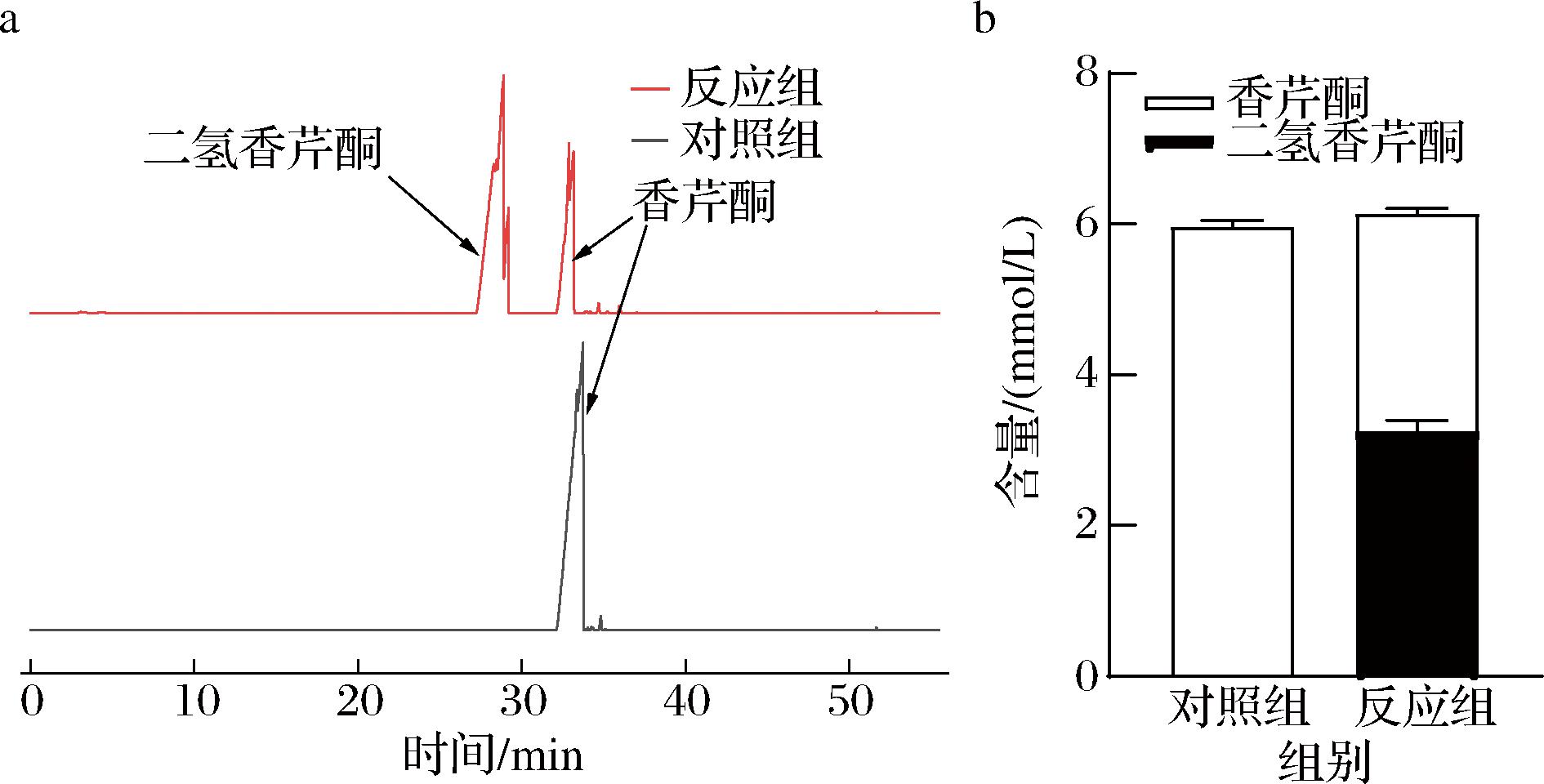
二氢香芹酮不仅是一种天然香料,还是一种重要的手性中间体,可用于合成多种活性化合物,常通过烯还原酶催化还原香芹酮的方法合成[20]。为了验证该酶是否具有还原香芹酮生成二氢香芹酮的能力,设置生物催化反应体系为1 mL,由50 mmol/L磷酸盐缓冲液(pH 6.0)、25 μg/mL纯化后的酶、0.5 mmol/L NADH和6 mmol/L香芹酮组成,在30 ℃反应10 h后,使用SPME/GC-MS对转化产物进行测定。结果如图14所示,二氢香芹酮保留时间在27 min左右,对照组在该处并没有明显的峰出现,而实验组在该处出现了明显的峰,说明KlebER2能够催化香芹酮合成二氢香芹酮,具有香芹酮还原酶活力,并且转化率达到50%以上。
3 结论
本研究对Klebsiella sp.O852来源的烯还原酶基因KlebER2进行克隆和异源表达,获得重组KlebER2蛋白,并对重组蛋白表达的条件进行优化,即37 ℃培养菌体浓度OD600值至0.5时,用0.8 mmol/L的IPTG在20 ℃、100 r/min的条件下诱导22 h,可实现蛋白的高表达;使用镍柱亲和层析对诱导后的蛋白进行分离纯化,以80 mmol/L咪唑缓冲液纯化蛋白能在保证蛋白纯度的情况下最大程度的收集KlebER2蛋白;KlebER2的酶学性质研究结果表明,其最适反应温度和pH分别为30 ℃和6.0,该酶对温度和pH较为敏感,在40 ℃孵育3 h后相对酶活力仅剩10%左右,在pH<6.0或pH>7.0的条件下,酶基本失活,这在一定程度上限制其应用,在以后研究中可通过蛋白质工程和固定化技术对KlebER2的稳定性进行改造,以满足工业化生产的需求;此外,Mn2+、Mg2+、Ba2+以及丙三醇对KlebER2表现出明显的促进作用,并且KlebER2对多种不饱和烯烃化合物都具有催化作用,使其应用更为广泛;偏好NADH的特性使其在今后的工业生产中具有明显的经济优势;最后对KlebER2催化香芹酮生成二氢香芹酮进行研究,转化率达50%以上。本研究明晰了烯还原酶KlebER2的性能,为二氢香芹酮的工业化生产奠定基础。
参考文献
[1] TISCHLER D, G DKE E, EGGERICHS D, et al.Applying a thermostable and organic solvent tolerant ene reductase for the asymmetric reduction of (R)-carvone[J].Chembiochem, 2020, 21(8):1217-1225.
DKE E, EGGERICHS D, et al.Applying a thermostable and organic solvent tolerant ene reductase for the asymmetric reduction of (R)-carvone[J].Chembiochem, 2020, 21(8):1217-1225.
[2] PORTO C, STÜKER C Z, MALLMANN A S, et al.(R)-(-)-carvone and (1R, 4R)-trans-(+)-dihydrocarvone from Poiretia latifolia vogel[J].Journal of the Brazilian Chemical Society, 2010, 21(5):782-786.
[3] 台亚楠, 董曼, 任婧楠, 等.柠檬烯微生物转化的研究进展[J].食品科学, 2014, 35(17):272-277.TAI Y N, DONG M, REN J N, et al.Recent advances in biotransformation of limonene[J].Food Science, 2014, 35(17):272-277.
[4] FU Y L, HOELSCH K, WEUSTER-BOTZ D.A novel ene-reductase from Synechococcus sp.PCC 7942 for the asymmetric reduction of alkenes[J].Process Biochemistry, 2012, 47(12):1988-1997.
[5] SOARES-CASTRO P, MONTENEGRO-SILVA P, HEIPIEPER H J, et al.Functional characterization of a 28-kilobase catabolic island from Pseudomonas sp.strain M1 involved in biotransformation of β-myrcene and related plant-derived volatiles[J].Applied and Environmental Microbiology, 2017, 83(9):e03 112-16.
[6] AMRAN A I, LIM S J, MUHD NOOR N D, et al.Enolase in Meyerozyma guilliermondii strain SO:Sequential and structural insights of MgEno4581 as a putative virulence factor and host-fungal interactions through comprehensive in silico approaches[J].Microbial Pathogenesis, 2023, 176:106025.
[7] ZHANG L L, FAN G, LI X, et al.Identification of functional genes associated with the biotransformation of limonene to trans-dihydrocarvone in Klebsiella sp.O852[J].Journal of the Science of Food and Agriculture, 2022, 102(8):3297-3307.
[8] LI Z H, WANG W Y, WANG G G, et al.Heterologous expression and characterization of the carveol dehydrogenase from Klebsiella sp.O852[J].Molecular Catalysis, 2024, 561:114158.
[9] SUN Y J, XU J Q, ZHOU H S, et al.Recombinant protein expression chassis library of Vibrio natriegens by fine-tuning the expression of T7 RNA polymerase[J].ACS Synthetic Biology, 2023, 12(2):555-564.
[10] BROWNING D F, GODFREY R E, RICHARDS K L, et al.Exploitation of the Escherichia coli lac operon promoter for controlled recombinant protein production[J].Biochemical Society Transactions, 2019, 47(2):755-763.
[11] LOZANO TEROL G, GALLEGO-JARA J, SOLA MART NEZ R A, et al.Impact of the expression system on recombinant protein production in Escherichia coli BL21[J].Frontiers in Microbiology, 2021, 12:682001.
NEZ R A, et al.Impact of the expression system on recombinant protein production in Escherichia coli BL21[J].Frontiers in Microbiology, 2021, 12:682001.
[12] RIBEIRO V T, ASEVEDO E A, DE PAIVA VASCONCELOS L T C, et al.Evaluation of induction conditions for plasmid pQE-30 stability and 503 antigen of Leishmania i. chagasi expression in E.coli M15[J].Applied Microbiology and Biotechnology, 2019, 103(16):6495-6504.
[13] 彭念, 陈婷婷, 王璐, 等.纤维二糖磷酸化酶表达条件的优化及其酶学性质研究[J].食品与发酵工业, 2024, 50(16):125-131.PENG N, CHEN T T, WANG L, et al.Optimizing conditions for expression of cellobiose phosphorylase and determining its enzymatic properties[J].Food and Fermentation Industries, 2024, 50(16):125-131.
[14] 张丽, 应向贤, 毛王伟, 等.裂殖酵母醛酮还原酶基因的克隆与诱导表达条件优化[J].发酵科技通讯, 2018, 47(3):161-165.ZHANG L, YING X X, MAO W W, et al.Cloning, over-expression and optimization of induction conditions of an aldo-keto reductase from Schizosaccharomyces pombe[J].Bulletin of Fermentation Science and Technology, 2018, 47(3):161-165.
[15] SHI Q H, JIA Y T, WANG H B, et al.Identification of four ene reductases and their preliminary exploration in the asymmetric synthesis of (R)-dihydrocarvone and (R)-profen derivatives[J].Enzyme and Microbial Technology, 2021, 150:109880.
[16] DEANE D T, COPE T A, SCHULZ A M, et al.Design, heterologous expression, and application of an immobilized protein kinase[J].Bioconjugate Chemistry, 2023, 34(1):204-211.
[17] REß T N, HUMMEL D W, HANLON D S P, et al.The organic-synthetic potential of recombinant ene reductases:Substrate-scope evaluation and process optimization[J].ChemCatChem, 2015, 7(8):1302-1311.
[18] LEE H J, LEE Y S, CHOI Y L.Cloning, purification, and characterization of an organic solvent-tolerant chitinase, MtCh509, from Microbulbifer thermotolerans DAU221[J].Biotechnology for Biofuels, 2018, 11:303.
[19] TOOGOOD H S, SCRUTTON N S.Discovery, characterisation, engineering, and applications of ene-reductases for industrial biocatalysis[J].ACS Catalysis, 2018, 8(4):3532-3549.
[20] TISCHLER D, G DKE E, EGGERICHS D, et al.Asymmetric reduction of (R)-carvone through a thermostable and organic-solvent-tolerant ene-reductase[J].ChemBioChem, 2020, 21(8):1217-1225.
DKE E, EGGERICHS D, et al.Asymmetric reduction of (R)-carvone through a thermostable and organic-solvent-tolerant ene-reductase[J].ChemBioChem, 2020, 21(8):1217-1225.
[21] WANG H B, PEI X Q, WU Z L.An enoate reductase achr-OYE4 from Achromobacter sp.JA81:Characterization and application in asymmetric bioreduction of C=C bonds[J].Applied Microbiology and Biotechnology, 2014, 98(2):705-715.
[22] M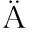 HLER C, KRATZL F, VOGEL M, et al.Loop swapping as a potent approach to increase ene reductase activity with nicotinamide adenine dinucleotide (NADH)[J].Advanced Synthesis &Catalysis, 2019, 361(11):2505-2513.
HLER C, KRATZL F, VOGEL M, et al.Loop swapping as a potent approach to increase ene reductase activity with nicotinamide adenine dinucleotide (NADH)[J].Advanced Synthesis &Catalysis, 2019, 361(11):2505-2513.
Heterologous expression and characterization of Klebsiella sp.ene reductase gene
WANG Wanying1, LI Zhijian1*, LI Zehao1, WANG Zixuan2, ZHANG Jiayue1, WANG Guoguo1, WANG Jingyue2, DUAN Zhirou2, ZHANG Lulu1*
1(College of Food Science and Technology, Henan University of Technology, Zhengzhou 450001, China)2(College of International Education, Henan University of Technology, Zhengzhou 450001, China)
ABSTRACT The ene reductase can reduce the carbon-carbon double bond of α, β-unsaturated alkenes and convert carvone into dihydrocarvone.In this study, an ene reductase gene KlebER2 was cloned from the genome of Klebsiella sp.O852, and was expressed heterologously in Escherichia coli.Besides, the expression conditions of KlebER2 were optimized, and enzymatic properties were explored after purification by nickel column.Finally, the catalytic performance of KlebER2 on dihydrocarvone production was investigated.Results indicated that KlebER2 protein was composed of 369 amino acids, and was a hydrophilic and non-transmembrane protein.The optimal expression condition was induced by 0.8 mmol/L IPTG for 22 h at 20 ℃ and 100 r/min when the concentration of OD600 was 0.5.Enzymatic property studies showed that the optimum reaction temperature and optimal reaction pH value were 30 ℃ and pH 6.0 respectively, and KlebER2 remained good stability below 30 ℃.In addition, KlebER2 was strongly activated by Mn2+, Mg2+, Ba2+, and glycerol, and had a catalytic effect on various unsaturated alkenes.KlebER2 was capable of converting carvone into dihydrocarvone, and the conversion rate was over 50%.These results can provide guidance for the heterologous expression of ene reductase and further industrial applications.
Key words ene reductase; heterologous expression; enzymatic properties; protein purification; dihydrocarvone


 DKE E, EGGERICHS D, et al.Applying a thermostable and organic solvent tolerant ene reductase for the asymmetric reduction of (
DKE E, EGGERICHS D, et al.Applying a thermostable and organic solvent tolerant ene reductase for the asymmetric reduction of ( NEZ R A, et al.Impact of the expression system on recombinant protein production in
NEZ R A, et al.Impact of the expression system on recombinant protein production in