纤维素是自然界分布最广、储量最丰富的可再生资源,其高效炼制对可替代能源的发展至关重要[1]。纤维素降解需要内切-β-葡聚糖酶(endo-1,4-β-D-glucanase,EG, EC 3.2.1.4)、外切-β-葡聚糖酶(exo-1,4-β-D-glucanase, CBH, EC 3.2.1.91)和β-葡萄糖苷酶(β-glucosidase, BGL, EC 3.2.1.21)等多种酶协同发挥作用。EGs在纤维素聚合物内部的非结晶区随机切割,水解β-1,4 糖苷键,主要产物是纤维糊精、纤维二糖和纤维三糖等。CBHs从纤维素链的非还原性末端切割β-1,4-糖苷键,生成可溶性的纤维糊精和纤维二糖。BGLs水解纤维二糖及纤维寡糖生成葡萄糖[2-3]。在纤维素酶系中,BGLs的含量较低,稳定性和活性不足,且常受其产物葡萄糖的反馈抑制,导致纤维二糖的积累;纤维二糖强烈抑制EGs和CBHs的活性,最终影响整个纤维素的糖化效率。因此,通过蛋白质工程技术获得酶学性质更加优良的BGLs成为该领域的一个研究热点[4-5]。
纤维素酶解通常需要在50 ℃、甚至60~70 ℃反应3~5 d,因此,热稳定性是影响BGLs实际催化效率的关键[6]。近二十年来,国内研究者在BGLs的热稳定性分子改造方面取得了一系列进展。BATRA等[7]采用易错PCR对BglI进行改造,三突变G414D/Y722H/ N789D将其在50 ℃和55 ℃的半衰期从0.14 h和0.04 h分别提升到1.52 h和 0.43 h。MATSUZAWA等[8]基于结构比对分析进行设计,获得Tm值提高9 ℃的MeBglD2突变体H8L/N59C/A295G。2018年,本课题组报道了一种基于双层平板的高通量筛选法[9],可快速从大容量随机突变文库中鉴定热稳定性提高的BGLs突变体。采用该方法对β-葡萄糖苷酶Ks5A7[9]、Bgl15[10]和Bgl6-M3[11]进行改造,成功获得热稳定性显著提高的突变体Ks5A7-4R1(T167I/V181F/K186T/A187E/A298G)、Bgl15-5R1(S39T/L42N/V167C/W178L/A251L/E319A/E326P/A396V/L433F)和Bgl6-M7(S30P/P303V/T320R/L440M),其T50值分别比野生型提高25.5、13.1 ℃和21.3 ℃。在上述例子中,最优突变体均是通过有益突变的逐次叠加获得的。除此之外,研究者还使用DNA Shuffling策略,将不同来源的BGLs片段化后进行重组,筛选性能提高的突变体[12]。2023年,RAPP等[13]使用该策略对Bgl3进行改造,获得了4个热稳定性提高的突变体,平均含有69个突变。DNA shuffling的优点是可快速积累多突变叠加效果,缺点是无法确定其中真正发挥作用的单点突变及其分子机制;同时最终突变体中可能积累有害突变,损害有益突变的叠加效果。如果在DNA shuffling的基础上,对其中的有益突变和有害突变进行鉴定,不仅可以利用该策略的优点,而且可避免有害突变的影响,同时增加对其分子机制的了解。
本研究选择β-葡萄糖苷酶突变体Bgl3-6511(含60个单点突变,T50值比野生型提高4.6 ℃)为研究对象,首先对其有益突变和有害突变进行鉴定,然后通过组合突变获得性能更优的突变体,进一步分析其结构基础,并对突变之间的上位性进行了探讨。
1 实验材料
1.1 菌株与基因
菌株:E.coli BL21(DE3),实验室保存;
基因:Bgl3(GenBank:Z29625.1)基因由北京睿博兴科生物技术有限公司合成,并连接于载体pET-28a-tac (+)[9]上。
1.2 仪器与设备
ETC 811型PCR仪,广州东盛生物科技有限公司;Tanon 1600凝胶成像系统,广州誉维生物科技有限公司;安泰SW-CJ-1FD超净工作台,苏州净化设备有限公司;U-2900紫外分光光度计,日本日立公司;SCIENTZ-IID超声波细胞粉碎机,宁波新芝生物科技股份有限公司;Infinite M200酶标仪,瑞士帝肯(Tecan)公司。
1.3 酶和生化试剂
DNA聚合酶Primer Star Max,TaKaRa公司;核酸标准ladder marker,Fermantas公司;硫酸卡那霉素(kanamycin monosulfate,Kana)、4-硝基苯基-β-D-吡喃葡萄糖苷(4-nitrophenyl β-D-glucopyranoside,pNPG)、琼脂粉、琼脂糖、酵母提取物、胰蛋白胨,Sigma-Aldrich贸易有限公司;ABclone,爱博泰克公司;FastPure Gel DNA Extraction Mini Kit,南京诺唯赞生物科技股份有限公司。
2 实验方法
2.1 突变体构建
将文献[13]中获得Bgl3突变体6511的氨基酸序列与野生型Bgl3(GenBank:Z29625.1)的氨基酸序列进行比对,确定突变。根据大肠杆菌的密码子偏好性对PCR引物进行设计,引物具体序列见表1。
表1 本研究所用引物
Table 1 Primers used in this study

突变序列(5′→3′)Bgl3-V2T-FGGAGATATACATATGACCCCGGCGGCGCAGCAGACCGCGACCGCGCCGGBgl3-P3A-FGGAGATATACATATGGTGGCGGCGGCGCAGCAGACCGCGACCGCGCCGGBgl3-A4V-FGGAGATATACATATGGTGCCGGTGGCGCAGCAGACCGCGACCGCGCCGGBgl3-A5R-FGGAGATATACATATGGTGCCGGCGCGTCAGCAGACCGCGACCGCGCCGGBgl3-Q6P-FGGAGATATACATATGGTGCCGGCGGCGCCGCAGACCGCGACCGCGCCGGBgl3-2-6-RCATATGTATATCTCCTTCTTAAAGTTAAACAAAATTATTTCTAGAGGGGAATTGBgl3-Q7E-FGTGCCGGCGGCGCAGGAAACCGCGACCGCGCCGGATGCGGCGCTGBgl3-A9S-FGTGCCGGCGGCGCAGCAGACCTCTACCGCGCCGGATGCGGCGCTGBgl3-T10A-FGTGCCGGCGGCGCAGCAGACCGCGGCGGCGCCGGATGCGGCGCTGBgl3-A11Q-FGTGCCGGCGGCGCAGCAGACCGCGACCCAGCCGGATGCGGCGCTGBgl3-A11Q-RCTGCGCCGCCGGCACCATATGTATATCTCCTTCTTAAAGTTAAACBgl3-P12Q-FCAGACCGCGACCGCGCAGGATGCGGCGCTGACCBgl3-P12Q-RCGCGGTCGCGGTCTGCTGCGCCGCCGGCACCBgl3-D13A-FACCGCGACCGCGCCGGCGGCGGCGCTGACCTTTCBgl3-D13A-RCGGCGCGGTCGCGGTCTGCTGCGCCGCCGGCACBgl3-A14S-FGCGACCGCGCCGGATTCTGCGCTGACCTTTCCGBgl3-A14S-RATCCGGCGCGGTCGCGGTCTGCTGCGCCGCBgl3-A15S-FACCGCGCCGGATGCGTCTCTGACCTTTCCGGAAGBgl3-A15S-RCGCATCCGGCGCGGTCGCGGTCTGCTGCGCCGBgl3-L16T-FGCGCCGGATGCGGCGACCACCTTTCCGGAAGGCBgl3-L16T-RCGCCGCATCCGGCGCGGTCGCGGTCTGCTGBgl3-T17S-FCCGGATGCGGCGCTGTCTTTTCCGGAAGGCTTTCBgl3-T17S-RCAGCGCCGCATCCGGCGCGGTCGCGGTCTGCTGBgl3-E20P-FGCGCTGACCTTTCCGCCGGGCTTTCTGTGGGGCBgl3-E20P-RCGGAAAGGTCAGCGCCGCATCCGGCBgl3-L23V-FTTTCCGGAAGGCTTTGTGTGGGGCAGCGCGACCBgl3-L23V-RAAAGCCTTCCGGAAAGGTCAGCGCCGCBgl3-S26A-FGGCTTTCTGTGGGGCGCGGCGACCGCGAGCTATCBgl3-S26A-RGCCCCACAGAAAGCCTTCCGGAAAGGTCBgl3-S30A-FGGCAGCGCGACCGCGGCGTATCAGATTGAAGGCBgl3-S30A-RCGCGGTCGCGCTGCCCCACAGAAAGCCTTCCGGBgl3-I33V-FACCGCGAGCTATCAGGTGGAAGGCGCGGCCGCGBgl3-I33V-RCTGATAGCTCGCGGTCGCGCTGCCCCACAGAAAGBgl3-Y50F-FAGCATTTGGGATACCTTCGCGCGCACCCCGGGCBgl3-Y50F-RGGTATCCCAAATGCTCGGGGTGCGGCCATCTTCBgl3-A51S-FATTTGGGATACCTATTCTCGCACCCCGGGCCGCBgl3-A51S-RATAGGTATCCCAAATGCTCGGGGTGCGGCCATCBgl3-R52H-FTGGGATACCTATGCGCACACCCCGGGCCGCGTGBgl3-R52H-RCGCATAGGTATCCCAAATGCTCGGGGTGCGGCCBgl3-R56K-FGCGCGCACCCCGGGCAAAGTGCGCAACGGCGBgl3-R56K-RGCCCGGGGTGCGCGCATAGGTATCCCAAATGCTCGGBgl3-V65I-FGGCGATACCGGCGATATCGCGACCGATCATTATCBgl3-V65I-RATCGCCGGTATCGCCGTTGCGCACGCGBgl3-T67A-FACCGGCGATGTGGCGGCGGATCATTATCATCGCBgl3-T67A-RCGCCACATCGCCGGTATCGCCGTTGCGCACBgl3-W73Y-FGATCATTATCATCGCTAGCGCGAAGATGTGGCGBgl3-W73Y-RGCGATGATAATGATCGGTCGCCACATCGCCGGBgl3-E75D-FTATCATCGCTGGCGCGACGATGTGGCGCTGATGBgl3-E75D-RGCGCCAGCGATGATAATGATCGGTCGCCACATCBgl3-A81K-FGATGTGGCGCTGATGAAAGAACTGGGCCTGGGCBgl3-A81K-RCATCAGCGCCACATCTTCGCGCCAGCGATGATAATGBgl3-E82D-FGTGGCGCTGATGGCGGACCTGGGCCTGGGCGCGBgl3-E82D-RCGCCATCAGCGCCACATCTTCGCGCCAGCGATGBgl3-G86K-FGCGGAACTGGGCCTGAAAGCGTATCGCTTTAGCBgl3-G86K-RCAGGCCCAGTTCCGCCATCAGCGCCACATCTTCBgl3-L92V-FGCGTATCGCTTTAGCGTGGCGTGGCCGCGCATTCBgl3-L92V-RGCTAAAGCGATACGCGCCCAGGCCCAGTTCCGCBgl3-A93S-FTATCGCTTTAGCCTGTCTTGGCCGCGCATTCAGBgl3-A93S-RCAGGCTAAAGCGATACGCGCCCAGGCCCAGTTCBgl3-P95S-FTTTAGCCTGGCGTGGTCTCGCATTCAGCCGACC
续表1

突变序列(5′→3′)Bgl3-P95S-RCCACGCCAGGCTAAAGCGATACGCGCCCAGGCCBgl3-I97V-FCTGGCGTGGCCGCGCGTGCAGCCGACCGGCBgl3-I97V-RGCGCGGCCACGCCAGGCTAAAGCGATACGCGCBgl3-L106V-FGGCCGCGGCCCGGCGGTGCAGAAAGGCCTGGBgl3-L106V-RCGCCGGGCCGCGGCCGGTCGGCTGAATGBgl3-Q107E-FCGCGGCCCGGCGCTGGAAAAAGGCCTGGBgl3-Q107E-RCAGCGCCGGGCCGCGGCCGGTCBgl3-K108R-FGGCCCGGCGCTGCAGCGTGGCCTGGATTTTTATCBgl3-K108R-RCTGCAGCGCCGGGCCGCGGCCGGTBgl3-A117T-FTTTTATCGCCGCCTGACCGATGAACTGCTGGCGBgl3-A117T-RCAGGCGGCGATAAAAATCCAGGCCTTTCTGCAGBgl3-A122E-FGCGGATGAACTGCTGGAAAAAGGCATTCAGCCGBgl3-A122E-RCAGCAGTTCATCCGCCAGGCGGCGATAAAAATCCBgl3-K123A-FGATGAACTGCTGGCGGCGGGCATTCAGCCGGTGBgl3-K123A-RCGCCAGCAGTTCATCCGCCAGGCGGCGBgl3-Q126E-FCTGGCGAAAGGCATTGAACCGGTGGCGACCCTGBgl3-Q126E-RAATGCCTTTCGCCAGCAGTTCATCCBgl3-A129V-FGGCATTCAGCCGGTGGTGACCCTGTATCATTGGBgl3-A129V-RCACCGGCTGAATGCCTTTCGCCAGCBgl3-N142D-FCCGCAAGAACTGGAAGACCCGGGCGGCTGGCCGGAACGBgl3-N142D-RTTCCAGTTCTTGCGGCAGATCCCAATGATACAGGGTCGBgl3-P143A-FCAAGAACTGGAAAACGCGGGCGGCTGGCCGBgl3-P143A-RGTTTTCCAGTTCTTGCGGCAGATCCCAATGATACBgl3-P150V-FGGCTGGCCGGAACGCGTGACCGCGGAACGCTTTGCGGAATATGBgl3-P150V-RGCGTTCCGGCCAGCCGCCCGGGTTTTCCAGBgl3-R154H-FCGCCCGACCGCGGAACACTTTGCGGAATATGBgl3-R154H-RTTCCGCGGTCGGGCGTTCCGGCCAGCCBgl3-E157D-FGCGGAACGCTTTGCGGACTATGCGGCGATTGCCBgl3-E157D-RCGCAAAGCGTTCCGCGGTCGGGCGTTCCBgl3-I161L-FGCGGAATATGCGGCGCTGGCCGCCGATGCGCTGBgl3-I161L-RCGCCGCATATTCCGCAAAGCGTTCCGCGGTCGGBgl3-D164R-FGCGGCGATTGCCGCCCGTGCGCTGGGCGATCGCGTGAAAACBgl3-D164R-RGGCGGCAATCGCCGCATATTCCGCAAAGCGTTCCBgl3-L215K-FCATTTAAACCTGGGCCATGGCAAAGCGGTGCAAGCGCTGCGCGBgl3-L215K-FGCCATGGCCCAGGTTTAAATGATGCGCCGCGBgl3-D222A-FGTGCAAGCGCTGCGCGCGCGCCTGCCGGCGGATBgl3-D222A-RGCGCAGCGCTTGCACCGCCAGGCCATGGCCCAGBgl3-R223E-FCAAGCGCTGCGCGATGAACTGCCGGCGGATGCGBgl3-R223E-RATCGCGCAGCGCTTGCACCGCCAGGCCATGBgl3-R223E-FCAAGCGCTGCGCGATGAACTGCCGGCGGATGCGBgl3-A226S-FCGCGATCGCCTGCCGTCTGATGCGCAGTGTAGCBgl3-A226S-RCGGCAGGCGATCGCGCAGCGCTTGCACCGBgl3-D227S-FGATCGCCTGCCGGCGTCTGCGCAGTGTAGCGTGBgl3-D227S-RCGCCGGCAGGCGATCGCGCAGCGCBgl3-Q229R-FCTGCCGGCGGATGCGCGTTGTAGCGTGACCCTGBgl3-Q229R-RCGCATCCGCCGGCAGGCGATCGCGCAGCBgl3-O230A-FCCGGCGGATGCGCAGGCGAGCGTGACCCTGAAC Bgl3-O230A-RCTGCGCATCCGCCGGCAGGCGATCGCGC Bgl3-E246D-RGCTATCGGTCAGCGGGCGCACATGATGAATGTTCAGGGTCBgl3-E246D-FCCGCTGACCGATAGCGACGCGGATGCGGATGCG
以质粒pET-28a-bgl3为模板,使用表1中的引物进行PCR:98 ℃预变性10 min;98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸1 min 30 s,循环30次;72 ℃延伸3 min。胶回收产物使用OKclone试剂盒进行同源重组,连接产物转化BL21(DE3)感受态细胞,涂布于含有Kana(终质量浓度50 μg/mL)的平板,37 ℃过夜。从平板上挑取单菌落,接入含有Kana(终质量浓度50 μg/mL)的LB液体培养基中,37 ℃、200 r/min,培养14 h。提取质粒,选择DNA测序正确的克隆子进行后续实验。
2.2 蛋白表达与纯化
将测序正确的突变体接种于含有Kana(终质量浓度50 μg/mL)的LB培养基,37 ℃,200 r/min培养至OD600值为0.8。随后,加入异丙基-β-D-硫代半乳糖苷(终浓度0.5 mmol/L),25 ℃,200 r/min 培养14 h,诱导蛋白表达。用 8 000 r/min 离心 10 min, 收集菌体。Bgl3及其突变体均在 C 端都带有 6×His 标签, 使用 ProteinIso® Ni-IDAResin 对蛋白进行纯化,使用超滤管将洗脱液置换为 100 mmol/L K2HPO4- KH2PO4缓冲液(pH 6.0),放置于 4 ℃ 保存。使用考马斯亮蓝试剂(Bradford 法[14]) 对纯化蛋白进行定量,以牛血清白蛋白(bovine serum albumin,BSA)溶液制作标准曲线。
2.3 T50值检测
半数失活温度(T50)定义为:在10 min内,50%的酶蛋白失去活性所对应的温度。取 20 μL 适当稀释的酶液于0.2 mL PCR管中,置于不同温度条件(40~70 ℃)温育10 min,然后以 pNPG为底物测定剩余酶活性。以不温育的酶活性为 100%,计算相对活性,使用Origin 9.0 软件中的 S 型函数拟合热失活曲线,计算T50值[15]。
2.4 分子内相互作用分析
野生型Bgl3的晶体结构已经解析(PDB:1GNX),以此为模版,采用SWISS-MODEL(https://swissmodel.expasy.org/)对获得的单点有益突变体和组合突变体M6进行同源建模。使用在线网站 SAVES v6.0(https://saves.mbi.ucla.edu/) 中的 PROCHECK 和Verify3D 功能模块分别对模型质量进行评估。使用在线服务器 PIC(http://pic.mbu.iisc.ernet.in/)[16]和RING(https://ring.biocomputingup.it/)[17]进行蛋白分子内部之间的相互作用分析,采用Pymol (https://www.pymol.org) 进行可视化和图片制作。
2.5 分子动力学模拟
使用软件程序GROMACS 2019.3[18]对Bgl3及其突变体M6进行分子动力学模拟,以Amber ff14SB[19] 为力场。使用H++程序在 pH 6条件下对组成蛋白的氨基酸残基进行质子化处理,将蛋白放置在一个矩形的盒子中,盒子边缘与蛋白表面的距离为 10 Å。基于 TIP3P 水分子模型对盒子进行水分子填充,同时往水盒子里加入 K+和Cl-离子,使最终体系中的阴阳离子浓度达到0.1 mol/L。对模拟体系进行能量最小化处理,即先采用最陡下降法优化 50 000步,之后再通过共轭梯度法优化50 000步。对体系进行限制性平衡模拟,先在 NVT 系综下将模拟体系的温度从0 K逐渐升高至363.15 K,平衡200 ps;然后在 NPT 系综下维持体系环境相对稳定,将体系的压强调整为1 bar,平衡5 ns;采用V-rescale恒温器和Parrinello-Rahman恒压器来平衡温度和压强,耦合常数分别设定为0.1 ps和2 ps。
采用LINCS算法对氢原子进行约束;借助particle-meshEwald(PME) 方法计算长距离的静电相互作用,将截距阈值设定为10 Å以计算范德华相互作用。整个非限制模拟的时长为100 ns,设置步长为2 fs。模拟结束后,每隔100 ps导出1帧产生轨迹,最后使用软件VMD 和PyMOL进行可视化分析。
3 结果与讨论
3.1 氨基酸序列比对
野生型Bgl3与突变体6511的氨基酸序列比对如图1所示,可见6511中含有60个单点突变,主要分布于序列前半部分。从二级结构看,α螺旋中含有28个突变,β折叠中含有6个突变,loop区含有26个突变,分别占比46.7%、10%和43.3%。Bgl3属于糖苷水解酶1家族(glycoside hydrolase family 1,GH1),结构为TIM桶折叠,由8个α螺旋围绕8个平行的β链组成,在(β/α)8拓扑主链上的突变有23个,其中部分突变可能会破坏蛋白的整体结构而损害其稳定性。
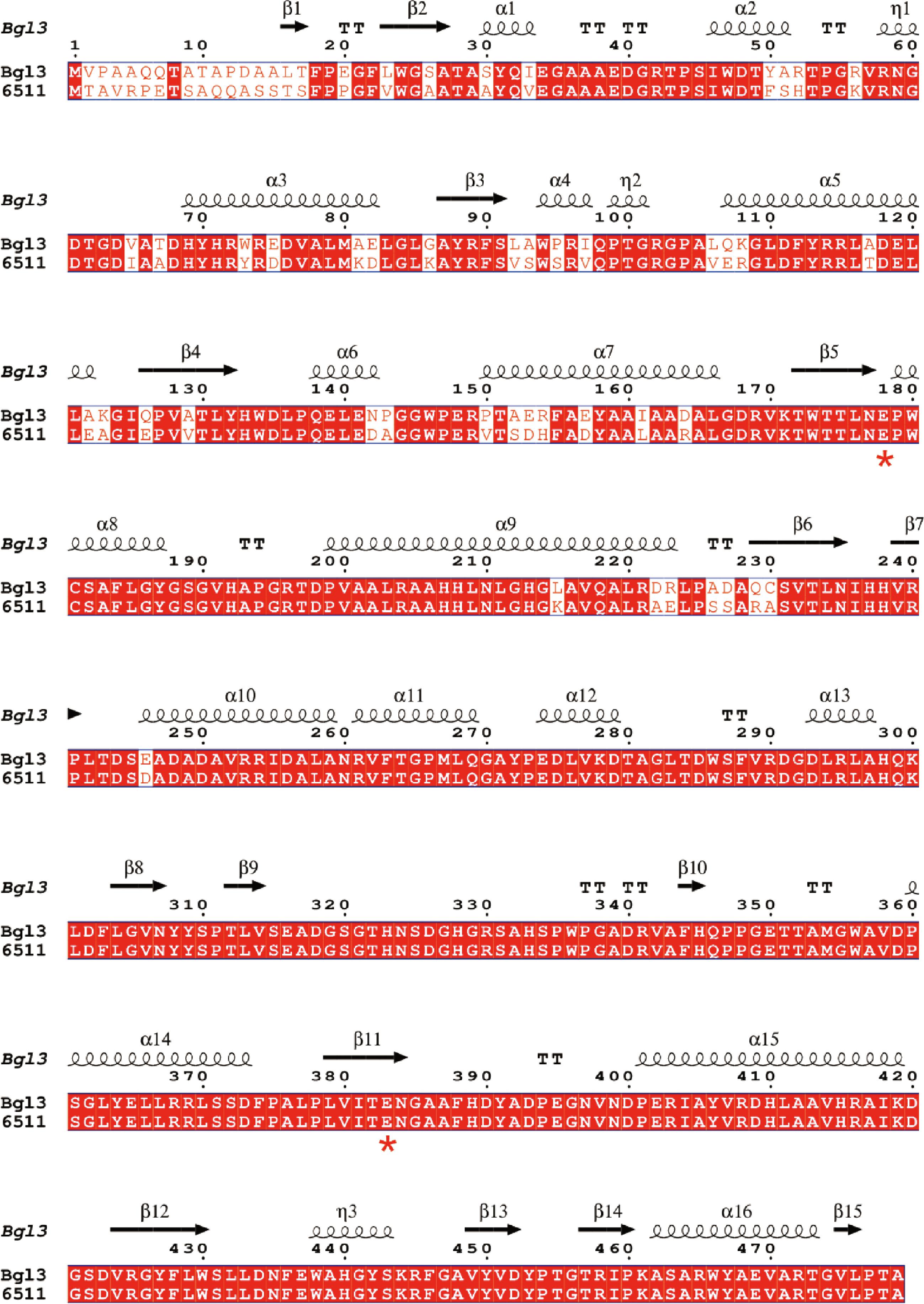
图1 野生型Bgl3与突变体6511的氨基酸序列比对
Fig.1 Amino acid sequence alignment of wild-type Bgl3 and mutant 6511
注:序列比对使用软件MULTALIN[20]进行,Bgl3的二级结构使用ESPRIPT添加[21],催化残基E178和E383用红色*标记。
3.2 有益突变的鉴定
在60个单点突变体中,4个突变体的活性低于Bgl3的1%,无法测定稳定性。其余突变体在不同温度下温浴,通过Origin对温浴后的剩余酶活性进行S型曲线拟合,确定其T50值(图2-a)。Y50F、R52H、R56K、V65I、T67A和P143A的T50值提高幅度大于1 ℃,认为是有益突变(图2-b),它们分别将该酶T50提高3.0、4.2、1.5、2.8、3.2和1.2 ℃。单点突变E75D、G86K、P95S、I97V和L106V 的T50值降低幅度大于1 ℃,认定是有害突变(图2-c),分别将该酶T50降低1.6、1.0、3.4、1.3、1.3 ℃。
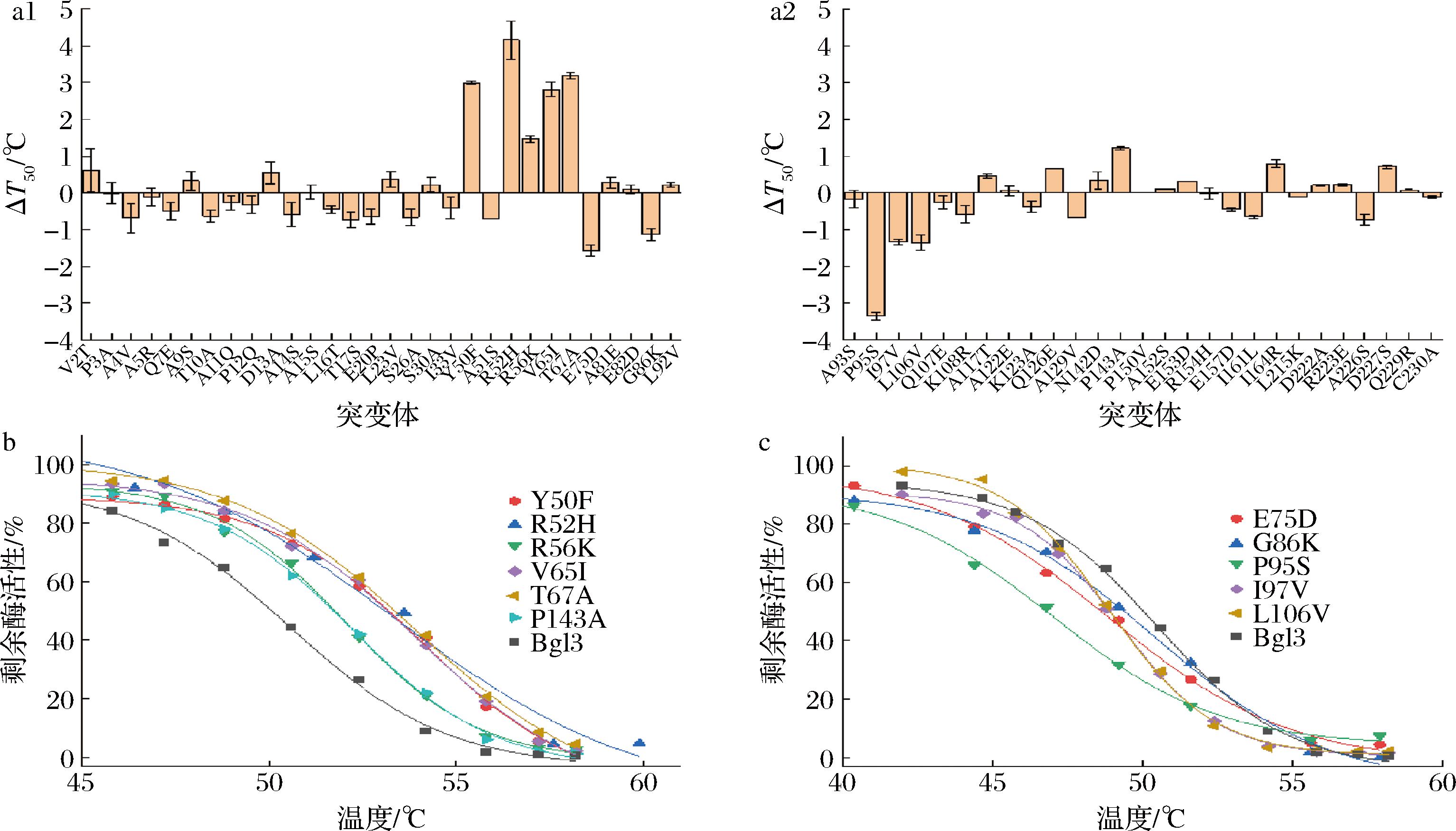
a-60个单点突变对Bgl3 T50值的影响;b-有益单点突变体的T50值拟合曲线;c-有害单点突变体的T50值拟合曲线
图2 单点突变对Bgl3热稳定性的影响
Fig.2 Effects of single point mutations on the thermal stability of Bgl3
注:Bgl3的T50值为50.8 ℃;ΔT50为单点突变体与Bgl3的T50值之差。
在突变体6511包含的突变中,对热稳定性影响不大(此处定义为提高和降低幅度小于1 ℃)的占81.7%,说明通过DNA shuffling获得的突变中大部分是非目标突变。同时,我们鉴定到5个有害突变,其数量和影响均与有益突变相当,说明DNA shuffling虽然可以快速积累有益的突变,但是容易获得有益突变和有害突变叠加的情况,导致有益突变的效应不能发挥或大打折扣。因此,将DNA shuffling和有益单点突变的鉴定结合起来可能是一种更优的酶工程策略。为验证这一猜测,下面将对有益单点突变进行组合,以获得热稳定性优于6511的突变体。
3.3 组合突变
考虑到距离近的突变叠加时更可能相互影响,首先将突变Y50F和R52H、V65I和T67A组合,结果显示,双突变Y50F/R52H(黄色)和V65I/T67A(绿色)分别将酶的稳定性提高5.3、6.8 ℃,均高于单点突变的提高幅度。然后在Y50F/R52H上叠加距离较近的R56K,得到突变体M3(Y50F/R52H/R56K),其稳定性进一步提高(ΔT50值为7.0 ℃)。在三突变基础上继续叠加双突变V65I/T67A,得到突变体M5(Y50F/R52H/R56K/V65I/T67A),将酶稳定性提高12.7 ℃。最后在M5上叠加P143A,获得最优突变体M6,其稳定性比野生型提高13.4 ℃(图3),也远优于突变体6511(ΔT50值为4.6 ℃)。这些结果说明,6511中积累的有害突变的确阻碍了该酶热稳定性的优化进程。
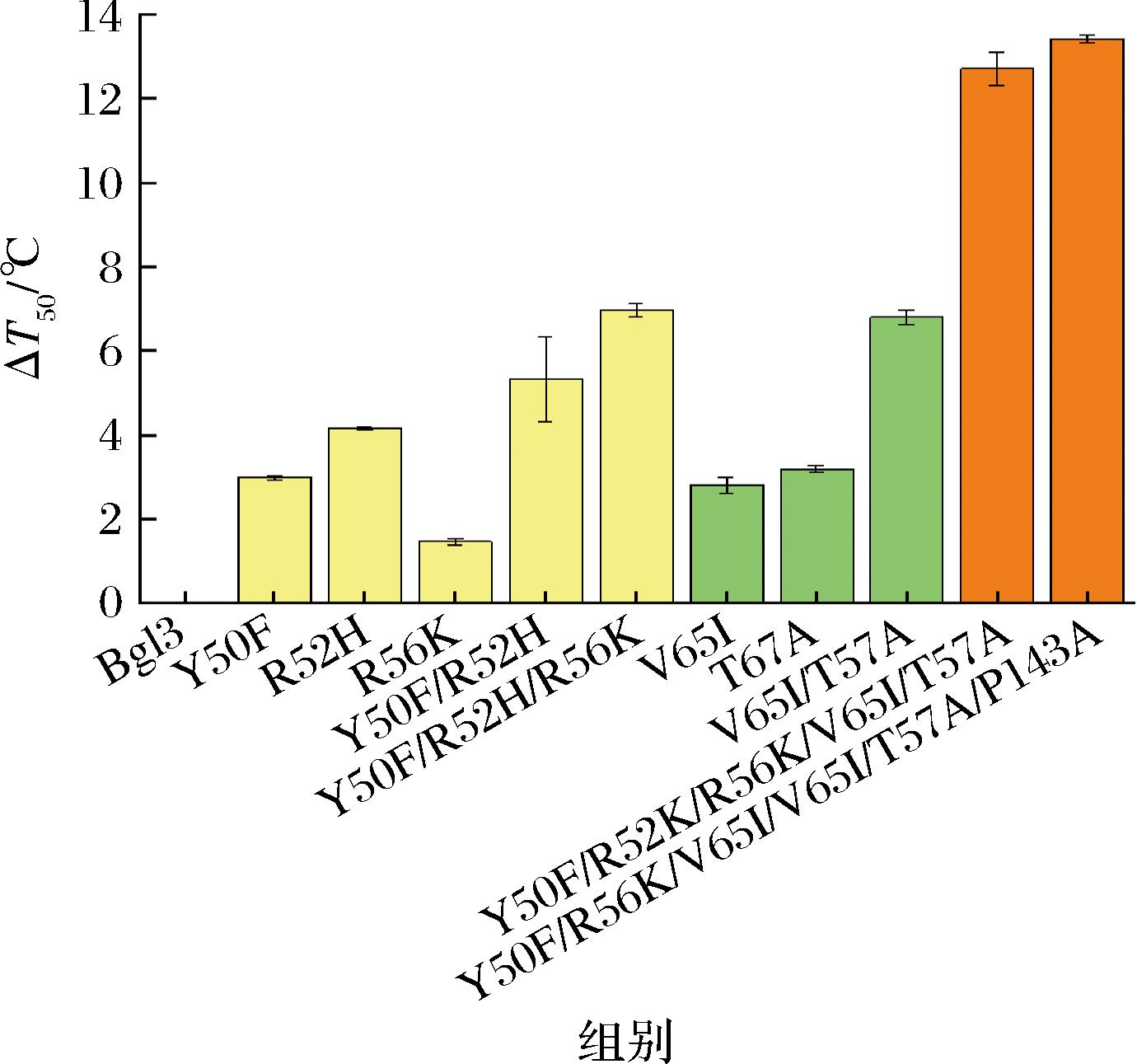
图3 有益单点突变的组合
Fig.3 Combinations of beneficial single point mutations
3.4 热稳定性有益突变的结构基础
为了了解有益突变提高Bgl3热稳定性的分子机制,以Bgl3的晶体结构为模板,对单点有益突变以及组合突变体M6进行同源建模。有益单点突变和有害单点突变在蛋白结构中的位置分布如图4所示,可以看出,6个有益突变中有4个分布于loop区域,仅Y50F位于1个短α螺旋中。而5个有害突变中,有3个(E75D、P95S、I97V)分布于α螺旋中,可能破坏蛋白的二级结构导致稳定性下降。

图4 Bgl3-6511中有益突变(绿色)和有害突变(红色)在蛋白中的分布
Fig.4 Distribution of beneficial mutations (green) and deleterious mutations (red) in Bgl3-6511
注:有益突变和有害突变残基的Cα用小球模型显示,催化残基178E和383E用棍棒模型形式显示。
结构分析显示,Y50F将该位点残基与W43之间的π-π键从5.2 Å缩短为5.0 Å,同时Y50与A193之间的弱氢键(3.6 Å)变成F50与I46位点残基之间的氢键(2.9 Å)(图5-a1和图5-a2);R52H将该残基与D48、D64间的氢键变短,与D64间的盐桥也变短(图5-b1和图5-b2)。所以,上述两个突变可能增强了分子间相互作用,从而提高蛋白质的稳定性。56残基位于蛋白表面(图4),突变前的R和突变后的K均为亲水性带电残基。R56K使其与G190间的氢键从2.7 Å变为2.8 Å,与T53间的氢键从3.3 Å缩短到3.2 Å,但与G195间的氢键消失(图5-c1和图5-c2)。从分子间相互作用来看,R56K可能不利于稳定性,这与实验数据不符(图2-a1)。考虑到此处结构分析仅基于静态模拟结构进行,后续可通过分子动力学从动态角度分析突变对酶分子的影响。V65处于一个疏水口袋中,V65I可能增强了该残基与周围疏水性残基I33、G35、A36、A37、G63和A66之间的相互作用(图5-d1和图5-d2)。T67A缩短了该残基与R458间的氢键,且与I33间形成一个新的氢键(图5-e1和图5-e2);类似地,P143A使该位点残基与E139间形成一个新的氢键,从而提高蛋白的稳定性(图5-f1和图5-f2)。
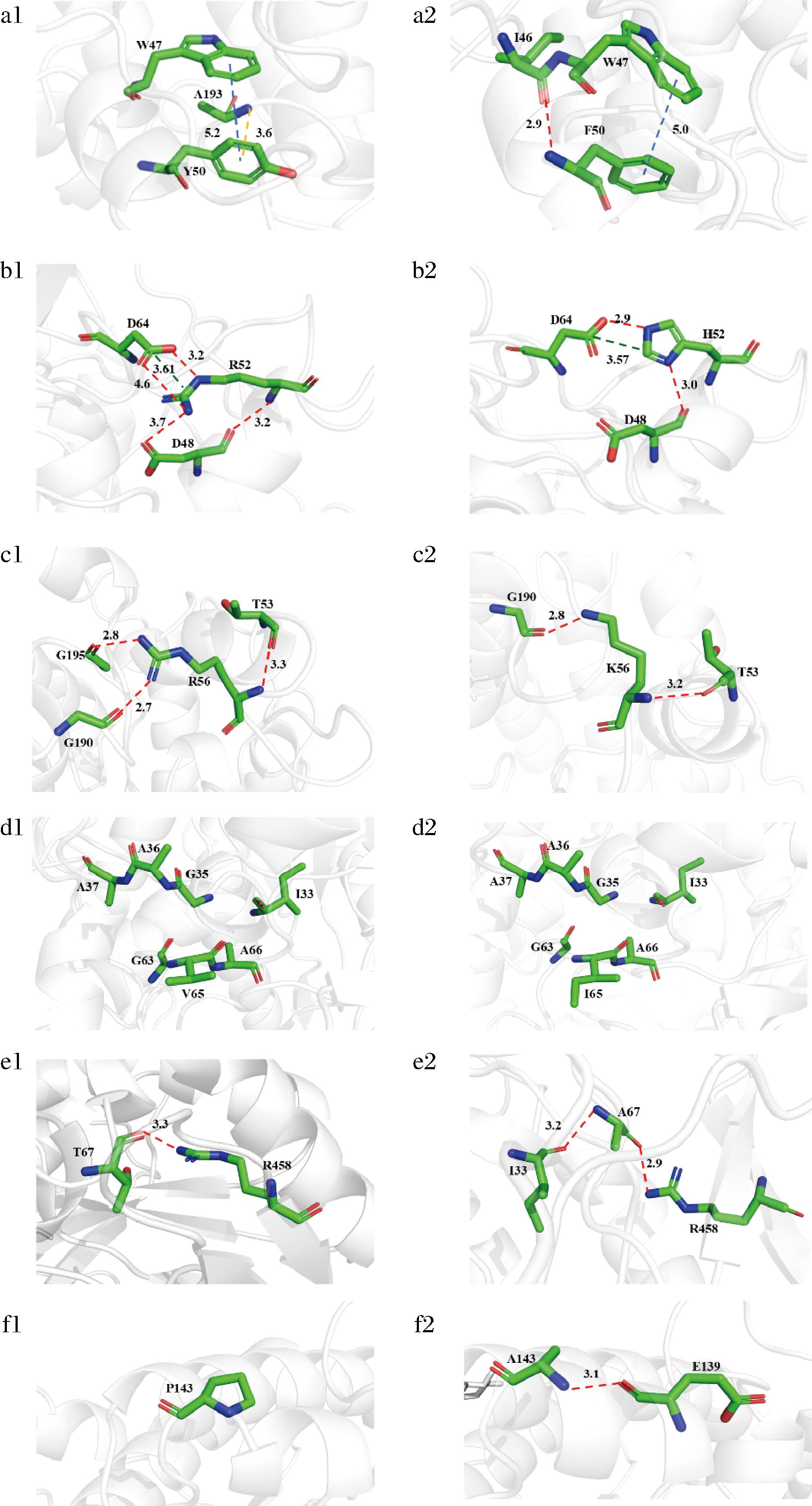
a1~f1-突变前各位点残基与周围残基的相互作用分析;a2~f2-突变后各位点残基与周围残基的相互作用分析
图5 有益突变的结构基础
Fig.5 Structural basis for beneficial mutations
注:红线代表氢键,蓝线代表π-π键,黄线代表π-π键,绿线代表盐桥,其中所有键长的单位均为Å。
为进一步从动态角度分析突变对酶分子的影响,将Bgl3和突变体M6在363.15 K条件下,进行100 ns 的分子动力学模拟。如图6-a所示,在模拟时间内,Bgl3 Cα原子的均方根偏差(root mean square difference,RMSD)在0.25 Å左右,而突变体M6 Cα的RMSD值在0.2 Å左右,说明M6的整体结构比Bgl3更加稳定。均方根波动(root mean square fluctuation,RMSF)分析显示,在突变体M6中,含有5个有益突变的44~68区和P143A的波动均明显小于Bgl3中对应区域(图6-b箭头所示),说明这些有益突变稳定了其所在二级结构。值得注意的是,M6中残基429~447的RMSF值也下降明显(图6-b),该区域位于蛋白的后半部分序列(图1),空间上远离有益突变(大于7 Å),提示有益突变可能通过远程作用影响该区域的刚性[22-23],其具体机制有待后续进一步研究。
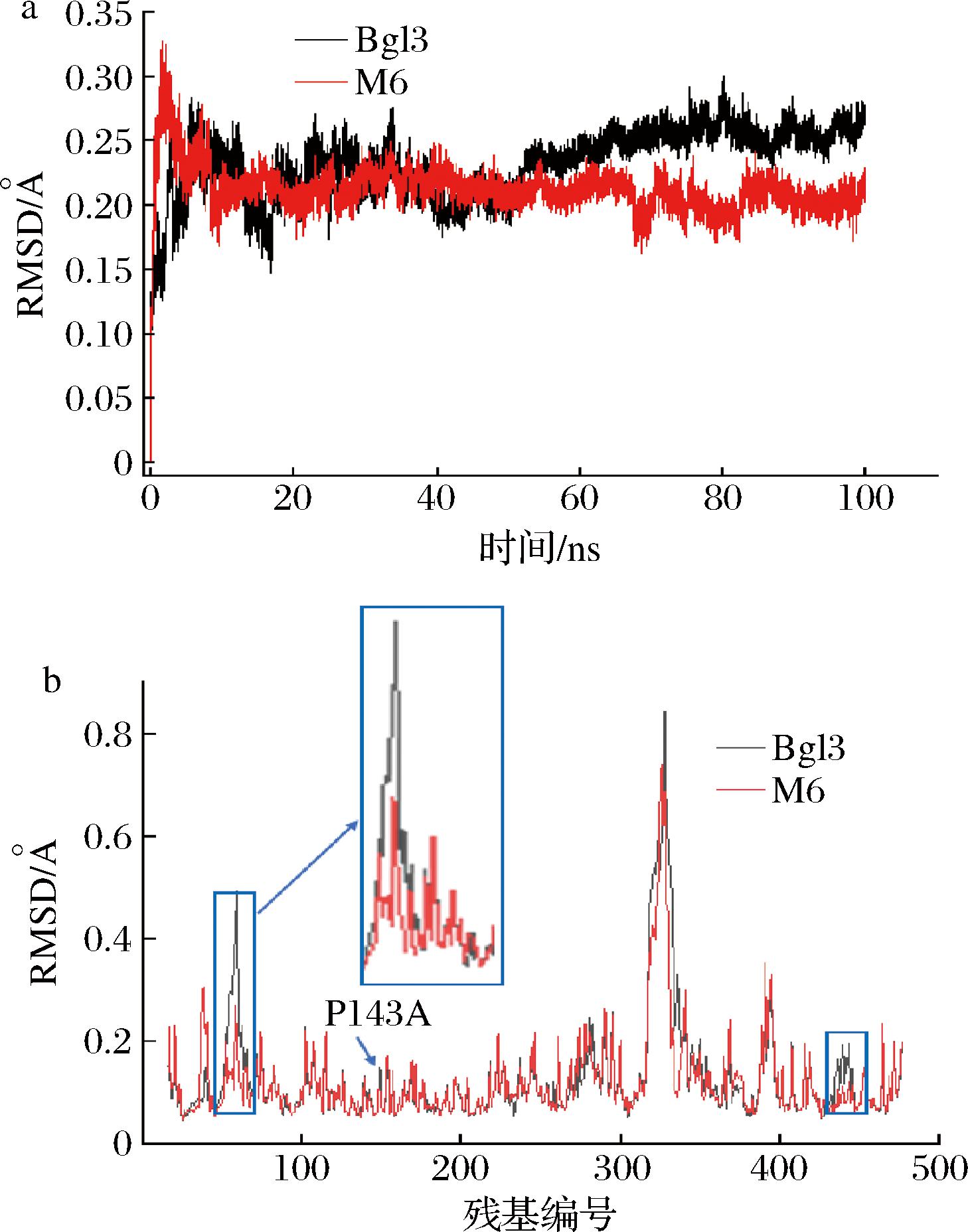
a-RMSD;b-RMSF
图6 Bgl3和突变体M6的分子动力学分析
Fig.6 Molecular dynamics analysis of Bgl3 and mutant M6
将获得的有益突变进行组合是酶工程领域的常用策略,以往认为酶分子内复杂的互作网络常导致上位性[24],使突变叠加的效果难以捉摸[25-27],而且距离较近的位点同时发生突变很可能会相互干扰,更容易产生突变之间的上位性。在本研究中,有益单点突变Y50F、R52H和R56K之间的距离为3~4 Å,V65I和T67A之间距离为3.4 Å,前3个突变之间存在轻微的负上位性(提高幅度之和为8.9 ℃,实际三突变提高7.0 ℃),而后2个突变之间表现为加和效应(提高幅度之和为6.1 ℃,实际三突变提高6.8 ℃)。6个有益单点突变的ΔT50相加为15.8 ℃,而M6实际提高13.4 ℃,说明这些突变之间轻微的上位性并未影响该酶稳定性的持续提高。此外,5个有害突变的ΔT50相加为-10.6 ℃,以加和性粗略估算,有益突变和有害突变的组合将使该酶的热稳定性提高5.3 ℃,而实际上突变体6511的T50值比野生型Bgl3提高了 4.6 ℃,说明有益突变与有害突变之间的上位性也不明显。最近,FAURE等[28]构建了蛋白GRB2-SH3(58个氨基酸残基)的高阶突变文库,对超过1010的序列空间进行实验采样,发现突变对蛋白稳定性的影响可以通过加性模型准确预测。这一结果与本研究的实验数据一致,提示蛋白质的遗传特性可能并不像之前认为的那样复杂。
4 结论
本文以DNA shuffling策略产生的β-葡萄糖苷酶突变体Bgl3-6511为研究对象,鉴定热稳定性有益单点突变,探索其背后的结构基础,并对突变之间的上位性进行讨论。本文获得以下结论:(1)6511中的单点有益突变为Y50F、R52H、R56K、V65I、T67A和P143A,占比10%;有害突变5个,占比8.3%;中性突变49个,占比81.7%。(2)通过组合单点有益突变,获得T50值提高13.4 ℃的突变体M6,其性质提高主要是突变增加了氢键数量、缩短了氢键和π-π键距离以及增强疏水相互作用。(3)有益单点突变之间以及有益突变和有害突变之间存在轻微的负上位性,但并不影响蛋白稳定性的持续优化。
[1] ZHOU Z Y, LIU D H, ZHAO X B.Conversion of lignocellulose to biofuels and chemicals via sugar platform:An updated review on chemistry and mechanisms of acid hydrolysis of lignocellulose[J].Renewable &Sustainable Energy Reviews, 2021, 146:111169.
[2] SINGHANIA R R, PATEL A K, SUKUMARAN R K, et al.Role and significance of beta-glucosidases in the hydrolysis of cellulose for bioethanol production[J].Bioresource Technology, 2013, 127:500-507.
[3] ERKANLI M E, EL-HALABI K, KIM J R.Exploring the diversity of β-glucosidase:Classification, catalytic mechanism, molecular characteristics, kinetic models, and applications[J].Enzyme and Microbial Technology, 2024, 173:110363.
[4] OUYANG B, WANG G P, ZHANG N, et al.Recent advances in β-glucosidase sequence and structure engineering:A brief review[J].Molecules, 2023, 28(13):4990.
[5] 程瑞琛, 刘艳丽, 戴大章.计算机辅助糖苷酶分子设计与改造研究进展[J].分子催化, 2020, 34(5):475-483.
CHENG R C, LIU Y L, DAI D Z.Recent progress in computer-aided design and engineering of glycosidases[J].Journal of Molecular Catalysis (China), 2020, 34(5):475-483.
[6] PATEL A K, SINGHANIA R R, SIM S J, et al.Thermostable cellulases:Current status and perspectives[J].Bioresource Technology, 2019, 279:385-392.
[7] BATRA J, MISHRA S.Organic solvent tolerance and thermostability of a β-glucosidase co-engineered by random mutagenesis[J].Journal of Molecular Catalysis B:Enzymatic, 2013, 96:61-66.
[8] MATSUZAWA T, WATANABE M, YAOI K.Improved thermostability of a metagenomic glucose-tolerant β-glycosidase based on its X-ray crystal structure[J].Applied Microbiology and Biotechnology, 2017, 101(23-24):8353-8363.
[9] CAO L C, LI S F, HUANG X, et al.Enhancing the thermostability of highly active and glucose-tolerant β-glucosidase Ks5A7 by directed evolution for good performance of three properties[J].Journal of Agricultural and Food Chemistry, 2018, 66(50):13228-13235.
[10] CAO L C, CHEN R, HUANG X, et al.Engineering of β-glucosidase Bgl15 with simultaneously enhanced glucose tolerance and thermostability to improve its performance in high-solid cellulose hydrolysis[J].Journal of Agricultural and Food Chemistry, 2020, 68(19):5391-5401.
[11] CAO L C, WANG Z J, REN G H, et al.Engineering a novel glucose-tolerant β-glucosidase as supplementation to enhance the hydrolysis of sugarcane bagasse at high glucose concentration[J].Biotechnology for Biofuels, 2015, 8:202.
[12] YI Z L, ZHANG S B, PEI X Q, et al.Design of mutants for enhanced thermostability of β-glycosidase BglY from Thermus thermophilus[J].Bioresource Technology, 2013, 129:629-633.
[13] RAPP J T, BREMER B J, ROMERO P A.Self-driving laboratories to autonomously navigate the protein fitness landscape[J].Nature Chemical Engineering, 2024, 1(1):97-107.
[14] KRUGER N J.The Bradford method for protein quantitation[J].Methods in Molecular Biology, 1994, 32:9-15.
[15] MUSIL M, KONEGGER H, HON J, et al.Computational design of stable and soluble biocatalysts[J].ACS Catalysis, 2019, 9(2):1033-1054.
[16] TINA K G, BHADRA R, SRINIVASAN N.PIC:Protein interactions calculator[J].Nucleic Acids Research, 2007, 35(Web Server issue):W473-W476.
[17] PIOVESAN D, MINERVINI G, TOSATTO S C E.The RING 2.0 web server for high quality residue interaction networks[J].Nucleic Acids Research, 2016, 44(W1):W367-W374.
[18] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX, 2015, 1:19-25.
[19] MAIER J A, MARTINEZ C, KASAVAJHALA K, et al.ff14SB:Improving the accuracy of protein side chain and backbone parameters from ff99SB[J].Journal of Chemical Theory and Computation, 2015, 11(8):3696-3713.
[20] CORPET F.Multiple sequence alignment with hierarchical clustering[J].Nucleic Acids Research, 1988, 16(22):10881-10890.
[21] ROBERT X, GOUET P.Deciphering key features in protein structures with the new ENDscript server[J].Nucleic Acids Research, 2014, 42(1):W320-W324.
[22] YU H R, DALBY P A.Coupled molecular dynamics mediate long- and short-range epistasis between mutations that affect stability and aggregation kinetics[J].Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(47):E11043-E11052.
[23] YU H R, MA S, LI Y W, et al.Hot spots-making directed evolution easier[J].Biotechnology Advances, 2022, 56:107926.
[24] WEINREICH D M, DELANEY N F, DEPRISTO M A, et al.Darwinian evolution can follow only very few mutational paths to fitter proteins[J].Science, 2006, 312(5770):111-114.
[25] STARR T N, THORNTON J W.Epistasis in protein evolution[J].Protein Science, 2016, 25(7):1204-1218.
[26] MITON C M, BUDA K, TOKURIKI N.Epistasis and intramolecular networks in protein evolution[J].Current Opinion in Structural Biology, 2021, 69:160-168.
[27] PAPKOU A, GARCIA-PASTOR L, ESCUDERO J A, et al.A rugged yet easily navigable fitness landscape[J].Science, 2023, 382(6673):eadh3860.
[28] FAURE A J, MART -ARANDA A, HIDALGO-CARCEDO C, et al.The genetic architecture of protein stability[J].Nature, 2024, 634(8035):995-1003.
-ARANDA A, HIDALGO-CARCEDO C, et al.The genetic architecture of protein stability[J].Nature, 2024, 634(8035):995-1003.