人体肠道的噬菌体库对益生菌和共生微生物构成了显著的环境压力[1]。为了应对诸如噬菌体和质粒等的DNA入侵,细菌演化出了CRISPR(clustered regularly interspaced short palindromic repeats)/Cas(CRISPR-associated genes)系统,这是一种适应性免疫系统,能够通过获得性免疫机制抵御外来DNA的侵袭,且这种抗性可遗传给后代[1]。CRISPR基因座包括CRISPR序列和Cas蛋白编码序列,其中CRISPR序列由重复序列(repeat)和间隔序列(spacer)交替组成[2]。根据Cas蛋白复合物的组成和结构特征,可将CRISPR/Cas分为两大类,其中包括6个类型和33个亚型[2]。在前导区的调控下,CRISPR RNA(crRNA)转录为前体crRNA(pre-crRNA),这些pre-crRNA与反式编码的小RNA(tracrRNA)配对,形成双链RNA(dsRNA)结构,在Cas蛋白和RNase Ⅲ的共同作用下被加工成熟,随后与细菌的Cas核酸酶结合,形成功能性核酸蛋白复合体[3]。Ⅱ型系统(CRISPR/Cas9)包括Ⅱ-A、Ⅱ-B和Ⅱ-C三个亚型,是目前应用最为广泛的基因编辑系统[4]。基于CRISPR/Cas9系统的基因编辑技术已成功应用于卷曲乳杆菌(Lactobacillus crispatus)和植物乳植杆菌(Lactiplantibacillus plantarum)在内的多种乳酸菌[5]。Ⅰ型和Ⅲ型CRISPR/Cas系统也有被开发为基因编辑工具的,但是总体应用较少[6]。
格氏乳杆菌(Lactobacillus gasseri)是一种在人体口腔、胃肠道和阴道常见的微生物,它在肠道菌群早期定植中扮演着关键角色[7]。部分L.gasseri菌株已被证实具有平衡肠道菌群、增强机体免疫力的功能,且其代谢产物具有降低胆固醇水平、预防心血管疾病和维持阴道健康等作用[8-9]。因此,L.gasseri在食品工业、营养补充剂、抗菌剂和降血脂药物中被广泛应用[10-12]。噬菌体作为细菌的天敌,对乳酸菌制品的生产和应用构成了重大威胁[13]。如何实现对L.gasseri的精准基因编辑以获得对噬菌体具有普遍抵抗力的优良菌种,已成为科学界亟待解决的瓶颈问题。SANOZKY-DAWES等[14]对17株L.gasseri的CRISPR系统进行了初步探索,发现6株包含有Ⅱ-A亚型CRISPR/Cas9系统。但随着分子生物学和高通量测序技术的发展,目前已完成了百余株L.gasseri的全基因组测序,这为更加系统且深入地研究其CRISPR/Cas系统的结构及作用机制提供了便利,相关研究结果对后续开发适用于L.gasseri菌株的精准基因编辑技术具有重要意义[15]。
本研究旨在通过生物信息学方法对NCBI GenBank数据库中搜集的142株L.gasseri全基因组序列的CRISPR/Cas系统结构进行分析,以确定其CRISPR/Cas系统的分布和类型,预测tracrRNA的位置和转录机制,分析间隔序列的多样性及来源,最后解析L.gasseri菌株Cas9蛋白识别的前间隔序列邻近基序(protospacer adjacent motif,PAM)序列,以期对现有CRISPR/Cas9基因编辑系统的改良和开发适用于L.gasseri的CRISPR/Cas9基因编辑系统提供参考。
1 材料与方法
1.1 材料
142株L.gasseri全基因组序列从NCBI GenBank数据库(https://www.ncbi.nlm.nih.gov/)获取(NCBI Taxonomy ID:1596)。
1.2 实验方法
1.2.1 CRISPR/Cas系统鉴定
通过CRISPRViz(https://github.com/CRISPRlab/CRISPRviz)鉴定存在的CRISPR序列,并利用CRISPROne(https://omics.informatics.indiana.edu/CRISPRone/)搜索cas基因和位置,确定CRISPR类型。
1.2.2 重复序列二级结构预测
由于CRISPR重复序列部分回文性质,其可能形成稳定的发夹二级结构。使用RNAfold(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi),以最小自由能(minimum free energy,MFE)和默认参数预测RNA二级结构。
1.2.3 TracrRNA位置和转录机制预测
TracrRNA通常在CRISPR/Cas9基因座的3个典型的位置出现:cas9基因上游序列、cas9和cas1基因之间序列、CRISPR阵列的上游。根据CHYLINSKI等[16]提供的方法,通过CRISPROne获得L.gasseri tracrRNA的大概位置。再利用SnapGene工具对cas操纵子进行可视化呈现,同时提取cas9基因上下游500 bp内的基因序列。使用BPROM软件(http://www.softberry.com/berry.phtml?topic=bprom&group=programs&subgroup=gfindb)预测tracrRNA的启动子,ARNold(http://rssf.i2bc.paris-saclay.fr/toolbox/arnold/index.php)预测终止子,最终确定tracrRNA的位置。再利用RNAfold预测tracrRNA的二级结构。
1.2.4 间隔序列多样性和同源性分析
CRISPRViz可以将所有的间隔序列进行相似性比对,并通过不同颜色组合的方格可视化。CRISPRTarget软件(http://crispr.otago.ac.nz/CRISPRTarget/crispr_analysis.html)对142株菌独特的间隔序列进行搜索,查找匹配的原间隔序列(protospacer),最多允许3个碱基不匹配,并对原间隔序列进行溯源。数据库选择GenBank-Phage(default)和Fefseq-Plasmid(default),然后使用R4软件中的Pheatmap软件包对匹配的间隔序列进行分层聚类分析[17]。
1.2.5 PAM序列预测
使用CRISPRTarget软件对所有匹配的间隔序列重新搜索。从已确定的原间隔序列的上下游10个碱基序列。再利用WebLogo网站(https://weblogo.berkeley.edu/logo.cgi)进行可视化,以预测可能的PAM序列。
2 结果与分析
2.1 格氏乳杆菌CRISPR基因座结构
142株L.gasseri菌株中有31株菌(21.83%)包含54个已确定CRISPR位点(表1),其中18株只有1个CRISPR位点,13株含有2~5个不同亚型的CRISPR位点(7株含有2个,3株含有3个,2株含有4个,1株含有5个CRISPR位点)。可见,L.gasseri与其他乳酸菌的CRISPR位点的分布差异较大[17](图1-a)。除菌株SV_Bg7063_mod2和HL75的CRISPR阵列中只有重复序列和间隔序列外,其余29株都含有Cas蛋白编码序列。89.66%(26/29)菌株的CRISPR系统为Ⅱ-A亚型(含有cas1、cas2、cas9和csn2基因),31.03%(9/29)为Ⅰ-E亚型(含有cas1、cas2、cas3、cas5、cas6e、cas7、cas8e和cse2gr11基因),这与前人的研究结果相似[14]。值得注意的是,有6株菌同时含有Ⅱ-A和Ⅰ-E两种CRISPR系统(图1-b)。
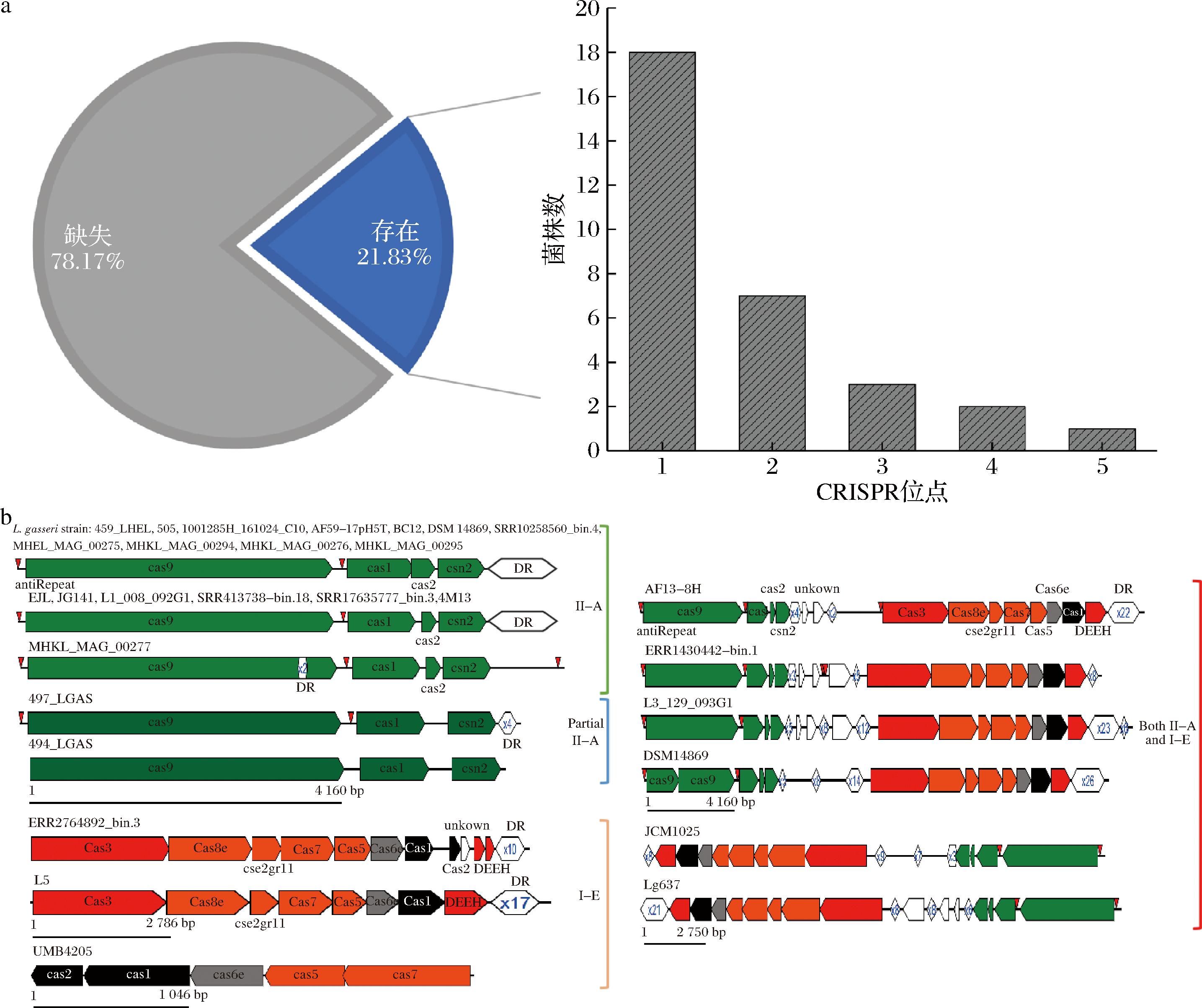
a-CRISPR/Cas系统阳性与阴性菌株的百分比及CRISPR位点分布;b-29株L.gasseri菌株Cas蛋白示意图
图1 L.gasseri中CRISPR/Cas系统的鉴定
Fig.1 Identification of CRISPR/Cas systems in L.gasseri
表1 三十一株L.gasseri菌株中的CRISPR/Cas系统
Table 1 CRISPR/Cas systems in 31 L.gasseri strains

菌株名CRISPR位点数量重复序列数量间隔序列数量cas基因CRISPR/Cas系统类型505187csn2, cas2, cas1, cas9Ⅱ-A亚型1001285H_161024_C1011817csn2, cas2, cas1, cas9Ⅱ-A亚型459_LHEL11716csn2, cas2, cas1, cas9Ⅱ-A亚型494_LGAS25, 44, 3cas9, cas1, csn2部分Ⅱ-A亚型497_LGAS24, 103, 9cas9, cas1, csn2部分Ⅱ-A亚型4M131109cas9, cas1, cas2, csn2Ⅱ-A亚型AF13-8H34, 8, 223, 7, 21cas9, cas1, cas2, csn2,cas3, cas8, cse2gr11, cas7, cas5, cas6e, cas1Ⅱ-A亚型, Ⅰ-E亚型AF59-17pH5T12019csn2, cas2, cas1, cas9Ⅱ-A亚型BC1212322csn2, cas2, cas1, cas9Ⅱ-A亚型DSM 1486945, 8, 14, 264, 7, 13, 25cas9, cas1, cas2, csn2,cas3, cas8e,cse2gr11, cas7, cas5, cas6e, cas1Ⅱ-A亚型, Ⅰ-E亚型EJL198csn2, cas2, cas1, cas9Ⅱ-A亚型ERR1430442-bin.125, 84, 7cas9, cas1, cas2, csn2,cas3, cas8e,cse2gr11, cas7, cas5, cas6e, cas1Ⅱ-A亚型, Ⅰ-E亚型ERR2764892_bin.3_metaWRAP_v1.1_MAG1109cas3, cas8e, cse2gr11, cas7, cas5, cas6e, cas1, cas2Ⅰ-E亚型HL75154--JCM 102538, 9, 77, 8, 6cas1, cas6e, cas5, cas7, cse2gr11, cas8e,cas3, csn2, cas2, cas1, cas9Ⅱ-A亚型, Ⅰ-E亚型JG14111413cas9, cas1, cas2, csn2Ⅱ-A亚型L1_008_092G1_dasL1_008_092G1_metabat.metabat.913332cas9, cas1, cas2, csn2Ⅱ-A亚型L3_129_093G1_dasL3_129_093G1_maxbin2.maxbin.01255, 8, 12, 23, 64, 7, 11, 22, 5cas9, cas1, cas2, csn2,cas3, cas8e,cse2gr11, cas7, cas5, cas6e, cas1Ⅱ-A亚型, Ⅰ-E亚型L5211, 1710, 16cas3, cas8e, cse2gr11, cas7, cas5, cas6e, cas1Ⅰ-E亚型Lactobacillus_gasseri_SV_Bg7063_mod2154- -Lg637421, 8, 8, 620, 7, 7, 5cas9, cas1, cas2, csn2,cas3, cas8e,cse2gr11, cas7, cas5, cas6e, cas1Ⅱ-A亚型, Ⅰ-E亚型MGYG-HGUT-023871109cas9, cas1, cas2, csn2Ⅱ-A亚型MHKL_MAG_002751109csn2, cas2, cas1, cas9Ⅱ-A亚型MHKL_MAG_002761109csn2, cas2, cas1, cas9Ⅱ-A亚型MHKL_MAG_002771109csn2, cas2, cas1, cas9Ⅱ-A亚型MHKL_MAG_0029426, 165, 15csn2, cas2, cas1, cas9Ⅱ-A亚型MHKL_MAG_0029526, 165, 15csn2, cas2, cas1, cas9Ⅱ-A亚型SRR10258560_bin.4_metaWRAP_v1.3_MAG221, 1520, 14csn2, cas2, cas1, cas9Ⅱ-A亚型SRR17635777_bin.3_metaWRAP_v1.3_MAG11211cas9, cas1, cas2, csn2Ⅱ-A亚型SRR413738-bin.1811110cas9, cas1, cas2, csn2Ⅱ-A亚型UMB4205329, 8, 1028, 7, 9cas2, cas1, cas6e, cas5, cas7部分Ⅰ-E亚型
注:-表示无相关信息。
2.2 TracrRNA的位置及转录预测
CRISPR/Cas系统为Ⅱ-A亚型的26株菌中,有24株菌株均识别到2个tracrRNA基因,分别位于cas9基因上游和cas1与cas9基因之间的非编码区(图2),这与CHYLINSKI等[16]的研究结果一致,表明tracrRNA在乳杆菌中的位置可能是相对保守的。但菌株MHKL_MAG_00294仅识别到一个tracrRNA基因即tracrRNA2,MHKL_MAG_00277识别到3个tracrRNA基因即tracrRNA1、tracrRNA2和tracrRNA3(该基因位于CRISPR阵列旁)。而菌株494_LGAS未发现tracrRNA基因。从整体看,tracrRNA的转录方向与cas基因相反,其与重复序列有58.33%的相似度。以菌株505为例,该tracrRNA从5′端起始,为repeat互补的长度约为25 nt的anti-repeat区段,之后是nexus区段,最后是部分折叠的hairpin区段,如图2所示,与CHYOU等[18]的研究结果一致。

图2 L.gasseri菌株505的tracrRNA在CRISPR基因座中的位置及二级结构预测
Fig.2 Location of tracrRNA from L.gasseri strain 505 in the CRISPR motif and prediction of secondary structure
2.3 重复序列的多样性及其二级结构稳定性分析
31株L.gasseri菌株的54个CRISPR位点中包括68个不同的直接重复(direct repeat,DR)序列,CRISPR位点中最少的含有4个DR数,最多的含有33个DR数,大小为28~38 nt,表现出丰富的多样性。同一亚型CRISPR系统的重复序列碱基和序列长度通常保守:图3-a显示Ⅱ-A亚型和Ⅰ-E亚型的DR序列分别聚为一支;图3-b显示Ⅱ-A亚型DR序列平均为36个碱基,而Ⅰ-E亚型为29个,与其他乳酸杆菌相似[17]。随着RNA的转录,连续的单链DR序列能够折叠形成具有2个“环”和1个“茎”的稳定二级结构(图3-c)。68个DR的MFE越低,则其RNA二级结构越稳定。并且茎越长,RNA二级结构越稳定(r=-0.469),且在相同茎长度条件下,GC含量和“G-C”碱基对的数量越多,DR序列的稳定性越高(r=-0.793、-0.833)(表2),与前人研究结果一致[19]。
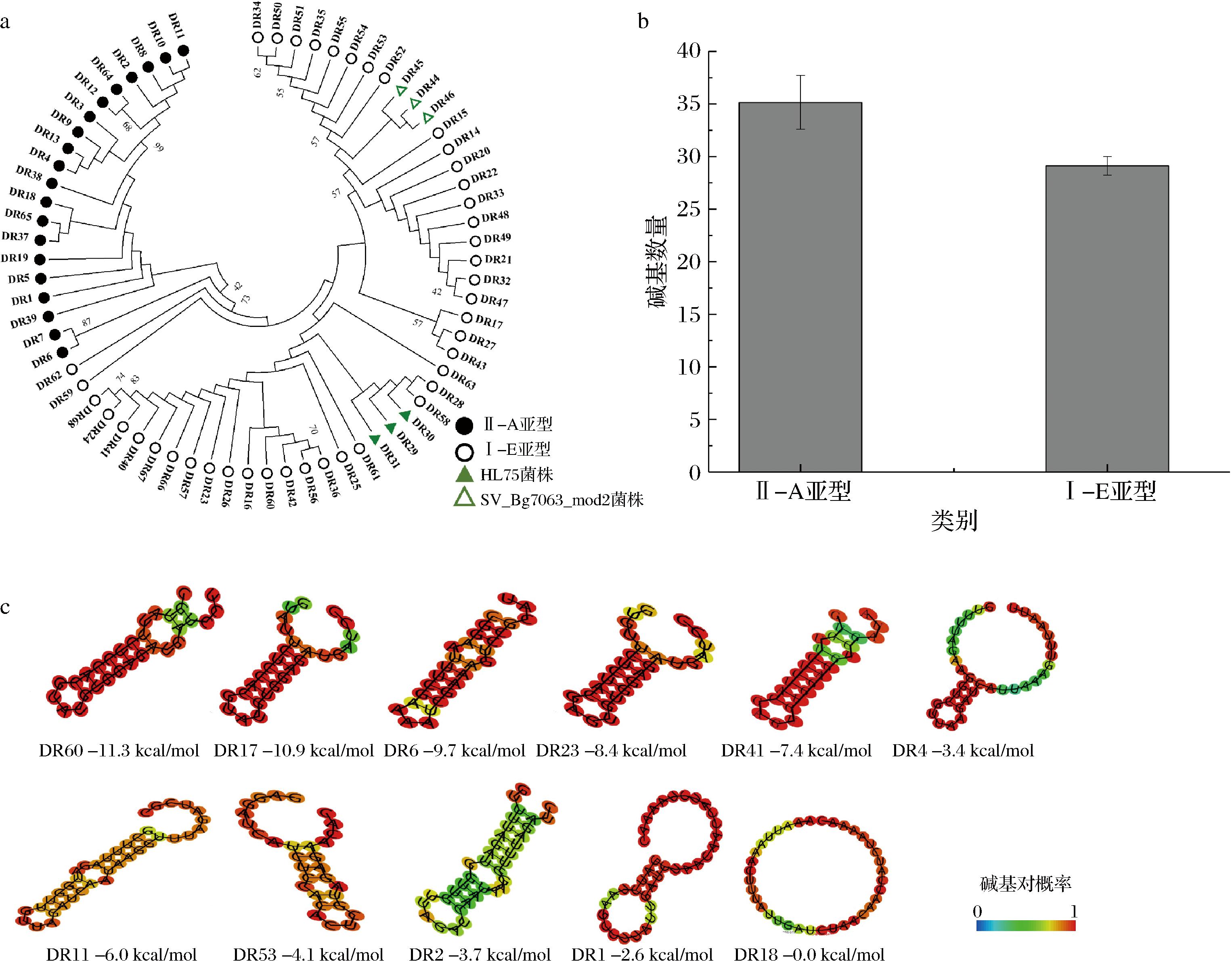
a-L.gasseri CRISPR阵列中68个独特重复序列的系统发育树;b-各亚型中重复序列的核苷酸数;c-部分重复序列(所有结果见补充表3)的二级结构示意图和MFE值
图3 CRISPR重复序列的分析
Fig.3 Analysis of CRISPR repeat sequences
表2 CRISPR重复序列相关结构指标的相关性分析
Table 2 Correlation analysis of CRISPR repeat sequence-related structural indicators
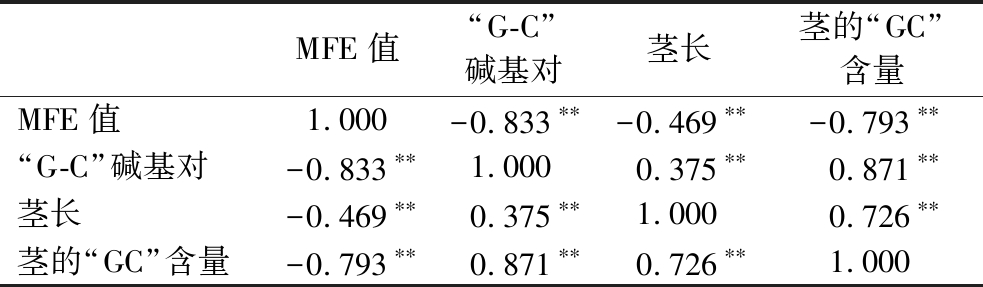
MFE值“G-C”碱基对茎长茎的“GC”含量MFE值1.000-0.833∗∗-0.469∗∗-0.793∗∗“G-C”碱基对-0.833∗∗1.0000.375∗∗0.871∗∗茎长-0.469∗∗0.375∗∗1.0000.726∗∗茎的“GC”含量-0.793∗∗0.871∗∗0.726∗∗1.000
注:**-P<0.01(双端检验),差异具有统计学意义。
2.4 间隔序列多样性与同源性分析
2.4.1 间隔序列多样性分析
L.gasseri CRISPR位点中包含3~32个不等的间隔序列数,分析表明,所有菌株中共有456个独特的间隔序列,其大小为26~38 nt。通过多序列比对发现,Ⅰ-E亚型CRISPR包含22种不同的间隔序列,Ⅱ-A亚型包含14种,其中部分菌株的间隔序列完全或部分相同如电子版增强出版附图1(https://doi.org/10.13995/j.cnki.11-1802/ts.040995)所示,表明它们可能曾经暴露于相似的环境中或相似的移动遗传元件免疫事件中[20]。菌株HL75和SV_Bg7063_mod2与其他菌株间隔序列差异较大,且此2株菌缺乏cas基因。
2.4.2 间隔序列的同源性分析
在456个独特的间隔序列中,有23.90%(109/456)的间隔序列与Streptococcus或Lactobacillus spp.中的噬菌体有同源性,而20.40%(93/456)的间隔序列与Lactobacillus spp.中的质粒有同源性(图4),其中有6.80%(31/456)的间隔序列同时靶向噬菌体和质粒。在31株具有CRISPR位点的L.gasseri菌株中有26株至少含有1个靶向噬菌体的间隔序列(图4-a),28株至少含有一个靶向质粒的间隔序列(图4-b)。其中,菌株DSM 14869的16个间隔序列同源于多达10种不同的噬菌体,而菌株BC12的13个间隔序列同源于11种不同的质粒。进一步分析表明,26株菌中的62个间隔序列可匹配到乳酸杆菌噬菌体JNU_P11的序列;而26株菌中的62个间隔序列与L.gasseri HL70质粒序列同源。但由于噬菌体和质粒序列数据库的限制,仍有62.06%(283/456)的独特的间隔序列未发现其来源。

a-L.gasseri CRISPR/Cas系统间隔区匹配到的噬菌体;b-L.gasseri CRISPR/Cas系统间隔区匹配到的质粒
图4 L.gasseri间隔区匹配的噬菌体和质粒层次聚类分析
Fig.4 Hierarchical clustering analysis of spacer matches in L.gasseri phages and plasmids
2.5 PAM序列的预测
在Ⅱ-A型CRISPR/Cas系统中,PAM序列通常位于原间隔子的3′端[21]。本研究分析了只含有Ⅱ-A亚型的23株菌中98个不同的CRISPR原间隔区3′端侧翼序列,以预测其PAM序列。结果表明,L.gasseri PAM序列的3号位碱基偏好A/G/T,4号位碱基偏好A/T,5、6号位碱基皆偏好A,由此推测L.gasseri CRISPR/Cas系统识别效率最高的PAM序列为5′-AAAA-3′(图5),这与Lactobacillus buchneri的PAM序列一致[22],且与嗜热链球菌中Ⅱ-A亚型CRISPR系统的PAM序列(5′-AGAA-3′)相似[23]。
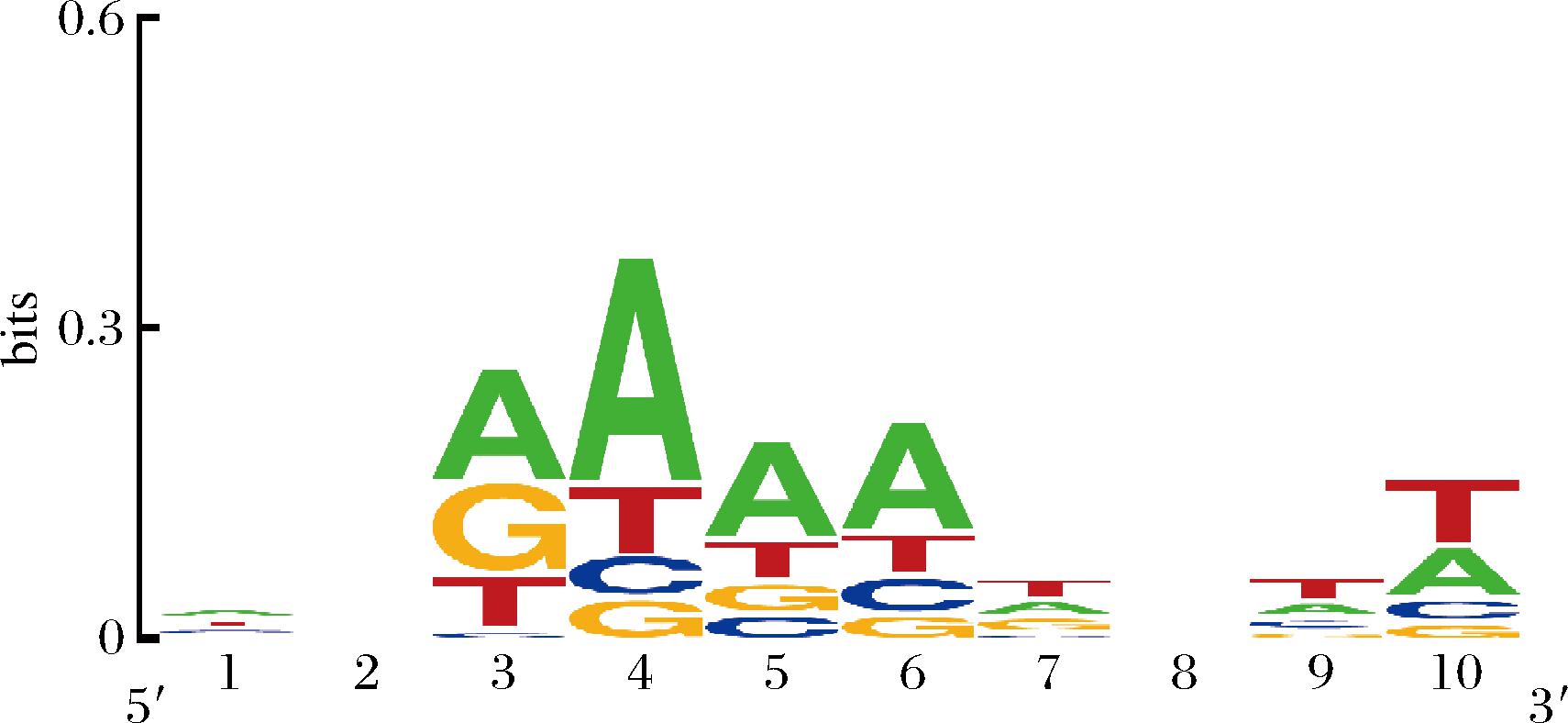
图5 L.gasseri Ⅱ-A 亚型CRISPR/Cas系统的PAM预测
Fig.5 PAM prediction of L.gasseri Ⅱ-A CRISPR/Cas systems
3 结论与讨论
本研究发现142株L.gasseri菌株中有31株(21.83%)共含有54个确定的CRISPR位点,此结果大大低于SANOZKY-DAWES等[14]报道的35%(6/17),而与前人研究得出的20%(4/20)相似[24]。由于本文的样本量远远大于前述研究,因此所得结果更具参考价值,同时本结果也对该菌CRISPR/Cas系统数据库进行了补充。CRISPR/Cas系统在L.gasseri中的流行强度可归因于2个主要因素:(1)生态因素,如氧气需求和温度,这些因素在嗜热细菌和厌氧生活方式相关的细菌中促进了更多系统的发展[25];(2)L.gasseri的CRISPR基因座5′端普遍存在的间隔序列缺失,这影响了CRISPR的保存[15]。本研究结果表明,L.gasseri的CRISPR/Cas系统主要属于Ⅱ-A亚型,其原因可能为:L.gasseri可耐受Cas9蛋白的细胞毒性,且Ⅱ-A亚型系统在其胞内天然活跃,从而高效抵抗入侵的噬菌体和质粒DNA[24]。6株L.gasseri菌中同时含有Ⅱ-A和Ⅰ-E两种CRISPR/Cas系统,这种多亚型CRISPR/Cas系统可能是通过基因水平转移而来,双系统可增加此部分菌株对更多种类的噬菌体或质粒的抵抗力[26]。
近年来,大量细菌的CRISPR/Cas9系统被发现并广泛应用于细菌的基因组编辑,其中以化脓性链球菌(Streptococcus pyogenes)的CRISPR/Cas9(SpCas9)系统最为常用[27]。该菌的CRISPR/Cas9系统属于Ⅱ-A型,由tracrRNA、Cas蛋白(Cas1、Cas2、Cas9和Csn2)和CRISPR阵列组成[28]。CRISPR/Cas系统为Ⅱ-A亚型的26株L.gasseri中,24株都含有完整的CRISPR/Cas9系统,只有2株(494_LGAS和497_LGAS)缺乏Cas2蛋白,而Cas2蛋白是一种将DNA整合到CRISPR阵列中所必需的蛋白质[29]。Ⅱ型CRISPR/Cas系统的激活依赖于反式激活RNA,即tracrRNA,它可用于前体crRNA(pre-crRNA)的成熟,并与pre-crRNA部分互补形成RNA双链体,被宿主RNA特异性核糖核酸酶(RNase Ⅲ)切割,形成crRNA/tracrRNA杂合体,再通过RNA-蛋白相互作用与Cas9蛋白结合并指导切割入侵核酸[30]。被Cas9蛋白识别的crRNA-tracrRNA复合物可分为几个结构域:与重复序列相互作用的抗重复结构域(茎和凸起),以及包含1个nexus区段和2个hairpin区段的尾部[30]。在L.gasseri中,通常只有一个hairpin区段,且研究也表明Cas9蛋白的精氨酸桥螺旋通常与重复-反重复区域的下茎、nexus和第一个hairpin结合成一个活跃的构象[31]。以菌株505为例,其tracrRNA的anti-repeat与repeat互补配对碱基为25 nt,尾部nexus区段有两个茎环结构,最后是一个部分折叠的hairpin区段,与CRAWLEY等[24]观察到的乳酸杆菌的tracrRNA结构相似。此外,每个亚型中tracrRNA的位置可能因系统差异而异,L.gasseri中Ⅱ-A亚型系统的tracrRNA主要存在于cas9基因的上游或下游,与前人研究结果一致[18]。且研究表明在L.gasseri中能够模拟天然crRNA-tracrRNA双链体,比野生型向导更好利用[24]。因此,L.gasseri的CRISPR/Cas9系统具有一定的基因编辑潜力。
前人研究表明,L.gasseri菌株在人体黏膜和肠道等环境中普遍存在,且其具有平衡肠道菌群的功能[32]。本研究结果发现,约1/3的L.gasseri CRISPR间隔序列以链球菌和乳杆菌属生态位的噬菌体或质粒为目标,可见在人类肠道中,各种细菌和病毒之间存在激烈的竞争,而L.gasseri可通过其CRISPR/Cas系统战胜病毒和同生态位细菌的入侵,从而起到调节肠道菌群的功能[1,7]。在本研究中,62.06%的间隔序列未发现其同源性,可能是由于具有同源性的质粒或噬菌体序列仍未被人类发现,也可能是由于本身具有同源性的病毒变异所致[20]。
PAM序列是一种短而保守的序列,在CRISPR/Cas9基因编辑系统的实际应用中,靶位点上错误的PAM序列可导致Cas9无法靶向,进而阻碍基因编辑的成功率[33]。已有研究表明,相比常用的S.pyogenes SpCas9的PAM序列5′-NGG-3′,L.gasseri的PAM碱基更偏好于A[34]。本研究分析了98个不同的CRISPR原间隔区侧翼序列,预测出L.gasseri的PAM序列为5′-AAAA-3′,这正好符合前人的研究结果[22]。但CRAWLEY等[24]预测L.gasseri的PAM序列为5′-cTAAC-3′,而ANDERSON等[35]预测的结果为5′-NTAA-3′,皆与本文结果不同,因此,L.gasseri的Cas9所识别的准确PAM序列仍需通过试验的方法加以验证。
本研究对142株L.gasseri全基因组序列的CRISPR/Cas系统进行了全面分析,发现31株含有CRISPR序列,共包含54个CRISPR位点,其重复序列大小为28~38 nt,间隔序列大小为26~38 nt。29株含有cas基因,包括Ⅱ-A亚型(26株,89.66%)和Ⅰ-E亚型(9株,31.03%)。24株Ⅱ-A亚型菌株包含2个tracrRNA基因,分别位于cas9基因上游和cas1与cas9基因之间的非编码区,tracrRNA转录方向与cas基因相反。比对发现,109个独特的间隔序列靶向噬菌体,93个靶向质粒。预测L.gasseri Cas9蛋白高效识别的PAM序列是5′-AAAA-3′。
[1] STERN A, MICK E, TIROSH I, et al.CRISPR targeting reveals a reservoir of common phages associated with the human gut microbiome[J].Genome Research, 2012, 22(10):1985-1994.
[2] MAKAROVA K S, WOLF Y I, IRANZO J, et al.Evolutionary classification of CRISPR-cas systems:A burst of class 2 and derived variants[J].Nature Reviews.Microbiology, 2020, 18(2):67-83.
[3] DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al.CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J].Nature, 2011, 471(7340):602-607.
[4] JACKSON S A, MCKENZIE R E, FAGERLUND R D, et al.CRISPR-cas:Adapting to change[J].Science, 2017, 356(6333):eaal5056.
[5] 李伟勋, 芦晶, 张书文, 等.CRISPR/Cas基因组编辑技术在乳酸菌中的应用及研究展望[J].微生物学报, 2021, 61(10):2971-2985.
LI W X, LU J, ZHANG S W, et al.Perspectives and applications of CRISPR/Cas-mediated genome editing in lactic acid bacteria[J].Acta Microbiologica Sinica, 2021, 61(10):2971-2985.
[6] LI Y J, PAN S F, ZHANG Y, et al.Harnessing type I and type III CRISPR-Cas systems for genome editing[J].Nucleic Acids Research, 2016, 44(4):e34.
[7] SELLE K, KLAENHAMMER T R.Genomic and phenotypic evidence for probiotic influences of Lactobacillus gasseri on human health[J].FEMS Microbiology Reviews, 2013, 37(6):915-935.
[8] WANG M M, HU T Y, LIN X Q, et al.Probiotic characteristics of Lactobacillus gasseri TF08-1:A cholesterol-lowering bacterium, isolated from human gut[J].Enzyme and Microbial Technology, 2023, 169:110276.
[9] ARTUYANTS A, HONG J, DAUROS-SINGORENKO P, et al.Lactobacillus gasseri and Gardnerella vaginalis produce extracellular vesicles that contribute to the function of the vaginal microbiome and modulate host-Trichomonas vaginalis interactions[J].Molecular Microbiology, 2024, 122(3):357-371.
[10] GUNYAKTI A, ASAN-OZUSAGLAM M.Lactobacillus gasseri from human milk with probiotic potential and some technological properties[J].LWT, 2019, 109:261-269.
[11] DE LIMA M Z T, DE ALMEIDA L R, MERA A M, et al.Crystal structure of a sucrose-6-phosphate hydrolase from Lactobacillus gasseri with potential applications in fructan production and the food industry[J].Journal of Agricultural and Food Chemistry, 2021, 69(35):10223-10234.
[12] OH J K, AMORANTO M B C, OH N S, et al.Synergistic effect of Lactobacillus gasseri and Cudrania tricuspidata on the modulation of body weight and gut microbiota structure in diet-induced obese mice[J].Applied Microbiology and Biotechnology, 2020, 104(14):6273-6285.
[13] ORTIZ CHARNECO G, DE WAAL P P, VAN RIJSWIJCK I M H, et al.Bacteriophages in the dairy industry:A problem solved?[J].Annual Review of Food Science and Technology, 2023, 14:367-385.
[14] SANOZKY-DAWES R, SELLE K, O’FLAHERTY S, et al.Occurrence and activity of a type II CRISPR-Cas system in Lactobacillus gasseri[J].Microbiology, 2015, 161(9):1752-1761.
[15] STOUT E A, SANOZKY-DAWES R, GOH Y J, et al.Deletion-based escape of CRISPR-Cas9 targeting in Lactobacillus gasseri[J].Microbiology, 2018, 164(9):1098-1111.
[16] CHYLINSKI K, LE RHUN A, CHARPENTIER E.The tracrRNA and Cas9 families of type II CRISPR-Cas immunity systems[J].RNA Biology, 2013, 10(5):726-737.
[17] YANG L, LI W X, UJIROGHENE O J, et al.Occurrence and diversity of CRISPR loci in Lactobacillus casei group[J].Frontiers in Microbiology, 2020, 11:624.
[18] CHYOU T Y, BROWN C M.Prediction and diversity of tracrRNAs from type II CRISPR-Cas systems[J].RNA Biology, 2019, 16(4):423-434.
[19] WANG Y, MAO T T, LI Y X, et al.Characterization of 67 confirmed clustered regularly interspaced short palindromic repeats loci in 52 strains of Staphylococci[J].Frontiers in Microbiology, 2021, 12:736565.
[20] LONG J Z, XU Y K, OU L Y, et al.Diversity of CRISPR/cas system in Clostridium perfringens[J].Molecular Genetics and Genomics, 2019, 294(5):1263-1275.
[21] VINK J N A, BAIJENS J H L, BROUNS S J J.PAM-repeat associations and spacer selection preferences in single and co-occurring CRISPR-Cas systems[J].Genome Biology, 2021, 22(1):281.
[22] NETHERY M A, HENRIKSEN E D, DAUGHTRY K V, et al.Comparative genomics of eight Lactobacillus buchneri strains isolated from food spoilage[J].BMC Genomics, 2019, 20(1):902.
[23] PAEZ-ESPINO D, MOROVIC W, SUN C L, et al.Strong bias in the bacterial CRISPR elements that confer immunity to phage[J].Nature Communications, 2013, 4:1430.
[24] CRAWLEY A B, HENRIKSEN E D, STOUT E, et al.Characterizing the activity of abundant, diverse and active CRISPR-Cas systems in lactobacilli[J].Scientific Reports, 2018, 8(1):11544.
[25] WEISSMAN J L, LALJANI R M R, FAGAN W F, et al.Visualization and prediction of CRISPR incidence in microbial trait-space to identify drivers of antiviral immune strategy[J].The ISME Journal, 2019, 13(10):2589-2602.
[26] HAN X, ZHOU X Y, PEI Z M, et al.Characterization of CRISPR-cas systems in Bifidobacterium breve[J].Microbial Genomics, 2022, 8(4):000812.
[27] KNOTT G J, DOUDNA J A.CRISPR-Cas guides the future of genetic engineering[J].Science, 2018, 361(6405):866-869.
[28] HELER R, SAMAI P, MODELL J W, et al.Cas9 specifies functional viral targets during CRISPR-Cas adaptation[J].Nature, 2015, 519(7542):199-202.
[29] NU EZ J K, KRANZUSCH P J, NOESKE J, et al.Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity[J].Nature Structural &Molecular Biology, 2014, 21(6):528-534.
EZ J K, KRANZUSCH P J, NOESKE J, et al.Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity[J].Nature Structural &Molecular Biology, 2014, 21(6):528-534.
[30] LIAO C Y, BEISEL C L.The tracrRNA in CRISPR biology and technologies[J].Annual Review of Genetics, 2021, 55:161-181.
[31] BRINER A E, BARRANGOU R.Guide RNAs:A glimpse at the sequences that drive CRISPR-cas systems[J].Cold Spring Harbor Protocols, 2016 Jul 1;2016(7). DOI: 10.1101/pdb.top090902.
[32] JIA X X, JIA M M, GAO X, et al.Demonstration of safety characteristics and effects on gut microbiota of Lactobacillus gasseri HMV18[J].Food Science and Human Wellness, 2024, 13(2):611-620.
[33] 李婉, 边鑫, 王娜娜, 等.乳酸菌CRISPR-Cas系统研究进展[J].中国乳品工业, 2016, 44(12):22-25; 35.
LI W, BIAN X, WANG N N, et al.Research progress of CRISPR-Cas system in Lactic acid bacteria[J].China Dairy Industry, 2016, 44(12):22-25; 35.
[34] KLEINSTIVER B P, PREW M S, TSAI S Q, et al.Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition[J].Nature Biotechnology, 2015, 33(12):1293-1298.
[35] ANDERSON E M, MCCLELLAND S, MAKSIMOVA E, et al.Lactobacillus gasseri CRISPR-Cas9 characterization in Vitro reveals a flexible mode of protospacer-adjacent motif recognition[J].PLoS One, 2018, 13(2):e0192181.