抗生素耐药性问题已成为全球性的公共卫生危机,造成广泛的发病和死亡,引起国内外的高度关注。联合国粮食及农业组织颁布《2021—2025年抗微生物药物耐药性行动计划》,提出五项关键目标以应对日益严峻的全球耐药性挑战[1]。
细菌广泛应用于食品和饲料及其添加剂的生产,如发酵乳制品的乳酸菌,生产酶制剂、氨基酸等发酵制品的谷氨酸棒杆菌、解淀粉芽胞杆菌等。为了避免这些细菌在使用过程中将耐药性传递至人类,对其进行全面的抗生素耐药性评价尤为重要。早在2002年,联合国粮食及农业组织和世界卫生组织在《食品益生菌评价指南》中就明确指出,应对菌种开展抗生素耐药性等8个方面的安全评价[2]。2013年,我国原国家卫生与计划生育委员会发布《新食品原料申报与受理规定》(国卫食品发23号),其中第十二条规定,“安全性评估报告应当包括微生物耐药性试验报告和产毒能力试验报告”[3]。2019年,农业农村部修订并发布《新饲料添加剂申报材料要求》(农业农村部第226号公告),明确要求微生物饲料添加剂及发酵制品生产菌株应开展菌种鉴定、抗生素耐药性等安全性评价[4]。
耐药基因是细菌抗生素耐药性的核心决定因素,开展基因安全评价能够揭示微生物抵抗药物作用的分子机制,对保障食品安全和维护人类健康具有重要意义。近年来,基因测序技术的飞速发展和不断创新,极大提升了对抗生素耐药性检测与研究的能力。本综述旨在介绍食品和饲料用细菌抗生素耐药性基因安全评价的研究进展,以期为我国食品和饲料用微生物菌种安全评价体系的建立提供科学依据与参考。
1 抗生素
抗生素是细菌(含放线菌)、真菌等微生物在生长繁殖过程中产生的代谢产物,在较低浓度下即可抑制或杀灭其他微生物;现代抗生素亦可由化学合成、半合成,或通过其他非微生物来源获得。20世纪上半叶,抗生素的发现和生产是医学领域最伟大的成就之一,它极大降低了传染病的发病率和死亡率,显著延长了人类寿命,并在动植物疾病预防、促生长等农业实践中具有广泛应用。
根据化学结构与作用机制的不同,可对抗生素进行分类,常见类别包括氨基糖苷类、β-内酰胺类、阳离子多肽类、糖肽类、林可酰胺类、脂肽类、大环内酯类、恶唑烷酮类、酚类、嘧啶类、喹诺酮类和氟喹诺酮类、利福霉素类、链阳霉素类、磺胺类、四环素类等[5](表1)。其中,头孢菌素类(第三、四和五代)、糖肽类、大环内酯类和酮内酯类、多黏菌素类、喹诺酮类被世界卫生组织列入《对人类医学至关重要的抗微生物药物清单》的最高优先级[6]。在评价食品或饲料用细菌耐药性时,应采用国际认可的标准或方法(如美国临床和实验室标准协会、欧盟药敏试验标准委员会的标准)测定特定抗生素对细菌的最低抑菌浓度,重点关注氨苄青霉素、万古霉素、庆大霉素、卡那霉素、链霉素、红霉素、泰乐菌素、克林霉素、四环素、氯霉素、环丙沙星、黏菌素、磷霉素等[7-9],判断菌株是否耐药。
表1 抗生素的主要类别、作用机制、耐药机制及常见耐药基因
Table 1 Major classes of antibiotics, mechanisms of action, mechanisms of resistance, and common resistance genes

抗生素类别(举例)抗生素作用机制耐药机制常见耐药基因氨基糖苷类(庆大霉素、链霉素、卡那霉素)与16S rRNA的30S核糖体亚基相互作用,导致蛋白质翻译或折叠错误,引起细胞死亡;携带正电荷,附着于细胞外膜,引起孔道积聚氨基糖苷修饰酶,如乙酰转移酶、磷酸转移酶和核苷转移酶;16S核糖体甲基化酶;16S RNA基因突变;内流减少和/或外排增加aac(3)-Ia,aadA1,aph(3)-Ia,rmtBβ-内酰胺类(青霉素、头孢菌素、头霉素、碳青霉烯、单环β-内酰胺)通过抑制青霉素结合蛋白使肽聚糖交联,引起细胞壁中的肽链交联,导致细胞裂解产生β-内酰胺酶;修饰青霉素结合蛋白;渗透性降低,外排增加blaCTX-M,blaIMP,blaCMY,blaOXA阳离子多肽类(黏菌素)与脂多糖中的脂质A结合;渗透外膜导致细胞死亡修饰或去除脂质AlpxD, lpsB, mcr-9.1糖肽类(万古霉素)与肽链中的D-丙氨酰-D-丙氨酸结合抑制交联从而抑制肽聚糖的合成对于革兰氏阴性菌,因无法透过外膜为固有耐药;对于革兰氏阳性菌,可通过酶修饰和水解肽聚糖前体;突变可引起膜增厚和通透性降低产生表型上的中介耐药vanB,vanE, vanG林可酰胺类(克林霉素)靶向蛋白质的翻译,尤其是50S核糖体亚基的23S rRNA基因,干扰肽链延长甲基转移酶修饰23S rRNA;表达蛋白使抗生素失活;外排lsaA, lnuA, lmrC脂肽类(达托霉素)插入细胞膜引起去极化,降低产生ATP的能力并导致细胞死亡使细胞壁的正电荷增厚和增加;减少脂肽诱导的膜去极化cls, mprF, pgsA大环内酯类(阿奇霉素、红霉素)通过靶向50S核糖体亚基的23S rRNA,抑制蛋白质的翻译,干扰肽链延长rRNA甲基化酶,可甲基化23S rRNA;核糖体突变;外排;大环内酯磷酸转移酶、酯酶;通过ATP结合盒F(ATP binding cassene F,ABC-F)蛋白保护核糖体ermB, ermX, mefA恶唑烷酮类(利奈唑胺)通过结合50S亚基的23S rRNA限制翻译,并阻止功能性70S亚基的形成修饰23S rRNA,如甲基转移酶;通过ABC-F蛋白保护核糖体cfrB, lmrS, clcD酚类(氯霉素)通过与50S亚基的A位点结合抑制翻译,抑制蛋白质合成50S核糖体亚基的23S rRNA突变;通过乙酰转移酶使抗生素失活;外排catB3,cmrA, cfrB嘧啶类(甲氧苄啶)通过抑制二氢叶酸还原酶影响C1代谢和叶酸合成,阻断四氢叶酸的产生修饰或获取新型二氢叶酸还原酶基因;外排dfrA1, dfrA5, dfrA18喹诺酮类和氟喹诺酮类(环丙沙星)通过与参与DNA超螺旋、链切割和连接的DNA旋转酶和拓扑异构酶IV作用,抑制DNA复制DNA旋转酶或拓扑异构酶IV突变;喹诺酮类药物外排或产生蛋白保护DNA旋转酶和拓扑异构酶qnrA1, norA, pmrA利福霉素类(利福平)与RNA聚合酶结合抑制转录,特别是依赖于DNA的RNA合成药物靶位rpoB突变;酶促核糖基化或利福平失活dnaA, rbpA, efpA链阳霉素类(达福普汀)在肽基转移酶结构域与50S核糖体亚基的23S rRNA结合,干扰肽链延长,影响蛋白质翻译23S rRNA突变;乙酰转移酶修饰链阳霉素;外排lsaA, ermC, vatE磺胺类(磺胺甲二唑)通过抑制二氢叶酸合成酶和抑制细胞生长阻止二氢叶酸的合成二氢叶酸合成酶编码基因和sul1/2基因突变,使编码产物的二氢叶酸合成酶对磺胺敏感度降低sul1, sul2, sul3四环素类(替加环素,四环素)通过结合30S核糖体亚基的16S rRNA抑制翻译,阻止tRNA在A位点与30S结合外排;蛋白质介导的核糖体保护;核糖体突变;酶促药物失活tetW, tetL, tetM
抗生素通过多种机制抑制或杀灭细菌,包括抑制细胞壁合成,改变细胞膜的极性和通透性,干扰DNA解旋、合成或复制过程,阻碍mRNA合成或转录,影响蛋白质翻译或折叠等[5, 10](表1)。
2 抗生素耐药性与耐药基因
细菌抗生素耐药性即细菌对抗生素的抗性。抗生素药物的滥用和过度使用是导致细菌耐药性的主要原因[11]。在治疗感染性疾病过程中,抗生素通常可杀灭敏感菌株,而少量因偶然变异获得耐药性的菌株得以存活并繁殖。随着时间的推移,耐药菌株逐渐在细菌群体中占据优势地位,最终引发整个细菌群体对抗生素产生耐药性。2024年5月,世界卫生组织发布最新《细菌类重点病原体目录》,将耐碳青霉烯类药物鲍曼不动杆菌、耐第三代头孢菌素肠杆菌、耐碳青霉烯类药物肠杆菌和耐利福平结核分枝杆菌列为关键优先级细菌[12],凸显了全球微生物耐药的严峻形势。
细菌对抗生素产生耐药性的机制复杂多样,主要包括降低细胞膜渗透性,外排抗生素,改变、修饰或保护抗生素作用靶位,失活和修饰抗生素以及产生靶标旁路等[5,13](表1)。一些细菌具有固有耐药性,如乳杆菌与明串珠菌的一些物种天然含有对万古霉素不敏感的D-丙氨酰-D-乳酸作为肽聚糖前体而对万古霉素耐药[14]等。另一些细菌通过获得新的遗传物质和耐药机制由原本敏感的菌株变得耐药,即具有获得性耐药。获得性耐药可来自现有染色体基因的点突变,或通过水平基因转移等方式从相同/不同属种的细菌获得新的耐药基因[13,15]。获得性耐药通常由特定遗传基因编码。与点突变相关的耐药基因如23S rRNA基因碱基突变使鼠李糖乳酪杆菌对大环内酯类抗生素耐药[16]。更多的获得性耐药则来源于微生物获取的新耐药基因。表1列举了部分常见抗生素耐药基因。截止2024年10月,根据国际权威的耐药基因数据库CARD (The Comprehensive Antibiotic Resistance Database),已发现并报道与413种细菌相关的5 278种耐药基因。
目前已在食品和饲料用细菌中发现耐药基因。四环素类耐药基因在乳杆菌和双歧杆菌中较常见,包括核糖体保护蛋白基因tetW、tetM、tetS,外排泵基因tetK、tetL等[17]。嗜酸乳杆菌、德氏乳杆菌保加利亚亚种中存在氯霉素耐药基因cat[17],乳杆菌中还发现红霉素耐药基因ermB、ermA、ermC、ermT,大环内酯类抗生素外排泵mefA,林可酰胺转移酶lnuA,链杀阳菌素乙酰转移酶vatE等[18]。在双歧杆菌中,相关研究主要集中在四环素类与大环内酯类抗生素耐药基因,其中tetW在双歧杆菌中以高频次被检出[19]。此外,双歧杆菌可合成非典型的异亮氨酸-tRNA合成酶,不与底物异亮氨酸的竞争物莫匹罗星结合,从而对莫匹罗星高度耐药[20]。在芽胞杆菌中,已报道ermC、tetL、tetM、cfr等耐药基因[17]。
3 全基因组测序(whole genome sequencing,WGS)与耐药基因数据库
以高通量、低成本为特征的下一代测序技术为微生物基因组学研究带来了革命性变革[10]。当前,Illumina、MGI、Ion Torrent、Pacific Biosciences (PacBio)及Oxford Nanopore (Nanopore)是下一代测序技术的主要平台[21],覆盖了短读长和长读长两种主要测序类型。短读长测序(如Illumina和 MGI)以高精度、低成本和高效性著称,其中的DNA被片段化、扩增并测序,所得序列通常较短(约250 bp),借助序列间的重叠区域组装成连续DNA序列,即叠连群(contig)。与之相对,长读长测序(如PacBio和Nanopore)能够产生较长序列(10~100 kb),有助于解决重复序列、变异结构等复杂核酸序列的组装问题,对DNA质量要求高,准确度偏低,且成本更高。对于常见食品或饲料用细菌,结合短读长与长读长测序可获得高质量的完成图基因组序列。
规范的操作规程和严格的数据质量控制对确保WGS分析结果的准确性和可靠性至关重要。2022年,国际标准ISO 23418:2022《食品链微生物学—细菌分型和基因组特性的全基因组测序—一般要求和指南》发布,规定了食品链中细菌全基因组序列测定和分析的最低要求,为WGS技术的标准化应用奠定了基础。欧盟针对食品链微生物的全基因组序列分析,提出包括DNA 提取、文库构建、测序策略、原始数据过滤(如平均 Phred 得分、测序深度、污染检查、基因组完整度)及序列组装(如组装算法、contig数量、基因组大小、完整度等)等在内的质量控制要求[22]。我国对于食品或饲料用微生物菌种,要求应采用二代和三代测序技术获得其完成图基因组序列,并在数据质量控制方面参考国际最新的标准和指南[9, 23]。分析菌种基因组中的耐药基因时,应重点关注完整编码基因,包括其基因名称、定位(染色体或质粒)、编码蛋白的功能、覆盖度、相似性百分比和e值等。
得益于丰富的全基因组序列数据,国际上目前已建立多个耐药基因数据库(表2),其中,CARD、ResFinder、NDARO等综合性数据库涵盖了大量耐药基因序列信息,应用广泛;也有针对β-内酰胺类和万古霉素类等特定抗生素或结核分枝杆菌等特定微生物的耐药基因数据库。此外,ARGs-OAP[24]等基于新算法的耐药基因分析工具目前也在陆续开发中。
表2 微生物抗生素耐药基因数据库
Table 2 Microbial antibiotic resistance gene databases

数据库名称描述耐药基因收录情况序列比对工具发布单位发布时间网站地址当前状态ARDB (Antibiotic Resistance Genes Da-tabase)首个抗生素耐药基因综合数据库,整合了NCBI和Swiss-Prot数据库的耐药基因序列4 554条核心耐药基因序列Blast美国马里兰大学2008年https://ardb.cbcb.umd.edu/不再维护
续表2

数据库名称描述耐药基因收录情况序列比对工具发布单位发布时间网站地址当前状态ResFinder可识别获得性耐药基因和/或染色体突变介导的抗生素耐药与15种抗生素相关的2156个耐药基因,11株菌的点突变ResFinder/PointFinder丹麦技术大学2012年http://genepi.food.dtu.dk/resfinder定期更新CARD (The Compre-hensive Antibiotic Re-sistance Database)基于本体的综合性耐药基因数据库,提供耐药基因、耐药机制、单核苷酸位点多态性(single nucleotide polymor-phism, SNP)、预测模型、可移动遗传元件等信息7 170条本体术语,5 194个参考序列,2 008个SNP,5 242个耐药基因检测模型RGI加拿大麦克马斯特大学2013年https://card.mcmaster.ca/定期更新Resfams基于蛋白家族和隐马尔可夫模型(hidden Markov model, HMM)的数据库,核心数据来自CARD、LacED等,适用于预测未知耐药基因代表主要耐药基因类别的166个HMMsHMMs美国华盛顿大学2014年https://www.dantaslab.org/resfams定期更新NDARO (National Database of Antibiotic Resistant Organisms)整合CARD、Lahey、ResFind-er等多个数据库9 706条耐药基因序列,7 138种抗生素耐药(amti-microbial resistance, AMR)蛋白质AMRFinder-Plus美国国家生物技术信息中心2016年https://www.ncbi.nlm.nih.gov/pathogens/antimi-crobial-resistance/定期更新ARG-ANNOT可检索已和潜在的耐药基因、识别染色体突变 1 689个耐药基因Blast法国艾克斯-马赛大学2014年https://www.mediterranee-infection.com/article.php?laref=282&titer=arg-annot不可访问ARGO (Antibiotic Resistance Genes On-line)最早的耐药基因数据库,仅包含与β-内酰胺类和万古霉素类抗生素相关的耐药基因555个β-内酰胺类耐药基因,115个万古霉素类耐药基因/印度贝拉技术与科学学院2005年http://bioinformatics.org/argo/beta/index.php数据不可访问Lahey list of β-lacta-mases仅包含与β-内酰胺酶相关的耐药基因,根据不同酶的水解和抑制特性将β-内酰胺酶的功能分类更新为3组第一类(Class C):头孢菌素类;第二类(Class A和D):广谱、抗抑菌剂和超广谱β-内酰胺酶和丝氨酸碳青霉烯酶;第三类(Class B):金属β-内酰胺酶/美国雷希诊所2010年https://externalwebapps.lahey.org/studies/不可访问BLDB (Beta-Lacta-mase DataBase)仅包含与β-内酰胺酶相关的耐药基因8 273种酶,1 663种结构,167种突变体,47种水解动力学特征谱Blast法国巴黎萨克雷大学2010年http://bldb.eu/定期更新TBDReaMDB (Tuber-culosis Drug Resist-ance Database)关于结核分枝杆菌耐药突变的综合性数据库,提供核酸与氨基酸水平上由碱基突变导致的密码子变化信息与7类抗生素相关的946种碱基突变信息/美国哈佛大学2009年http://www.tbdreamdb.com不可访问
注:“/”表示信息未提供。
4 固有耐药基因与获得性耐药基因
用于食品或饲料的细菌及其产品应当避免扩充现有耐药基因库或加速耐药基因传播。固有耐药基因是细菌物种内各菌株天生携带且稳定遗传的基因,可赋予细菌在抗生素环境下存活与繁殖的能力[25]。一般认为,固有耐药基因发生水平转移的风险相对较低[25]。与之相对,获得性耐药基因指菌株通过水平基因转移获得的新基因,可使细菌在超过抑制同物种大多数野生型菌株所需抗生素浓度的环境下存活或繁殖[7,25]。获得性耐药基因可整合到染色体中,或位于单独的遗传元件上。鉴于获得性耐药基因存在水平转移风险,国内外食品和饲料相关法规与指南均要求,微生物菌株不应携带对临床上重要抗微生物药物的获得性耐药基因[7-9]。
基于WGS数据的生物信息学分析为判断耐药基因的固有或获得性属性、评估其转移风险提供了全新策略[25]。图1展示了基于全基因组数据的细菌耐药基因分析主要流程。当与数据库中序列比对发现菌种基因组中存在耐药基因时,可从ENA、NCBI等公共数据库或实验室资源中收集目标物种内至少30个不同菌株的全基因组序列,开展严格质量控制,并利用平均核苷酸一致性(average nucleotide identity,ANI)、数字DNA-DNA杂交或系统发育方法,明确其分类学地位。确保所选菌株间为非克隆性关系,即其在流行病学和/或生态学上无直接关联,以避免结果产生偏差。在此基础上,采用Blast等序列比对工具检测目标耐药基因在已收集的全基因组序列集中存在情况及其分布比例。根据比对结果判断耐药基因的固有或获得性属性。若耐药基因广泛存在于同一物种(或亚种)的多数野生型菌株中,且位于染色体上,则判定其为固有耐药基因;反之,判定其为获得性耐药基因[25]。对于获得性耐药基因,若其导致微生物菌种耐药表型,则认为其具有危害性;若未引起耐药表型,则需进一步评估其潜在风险。
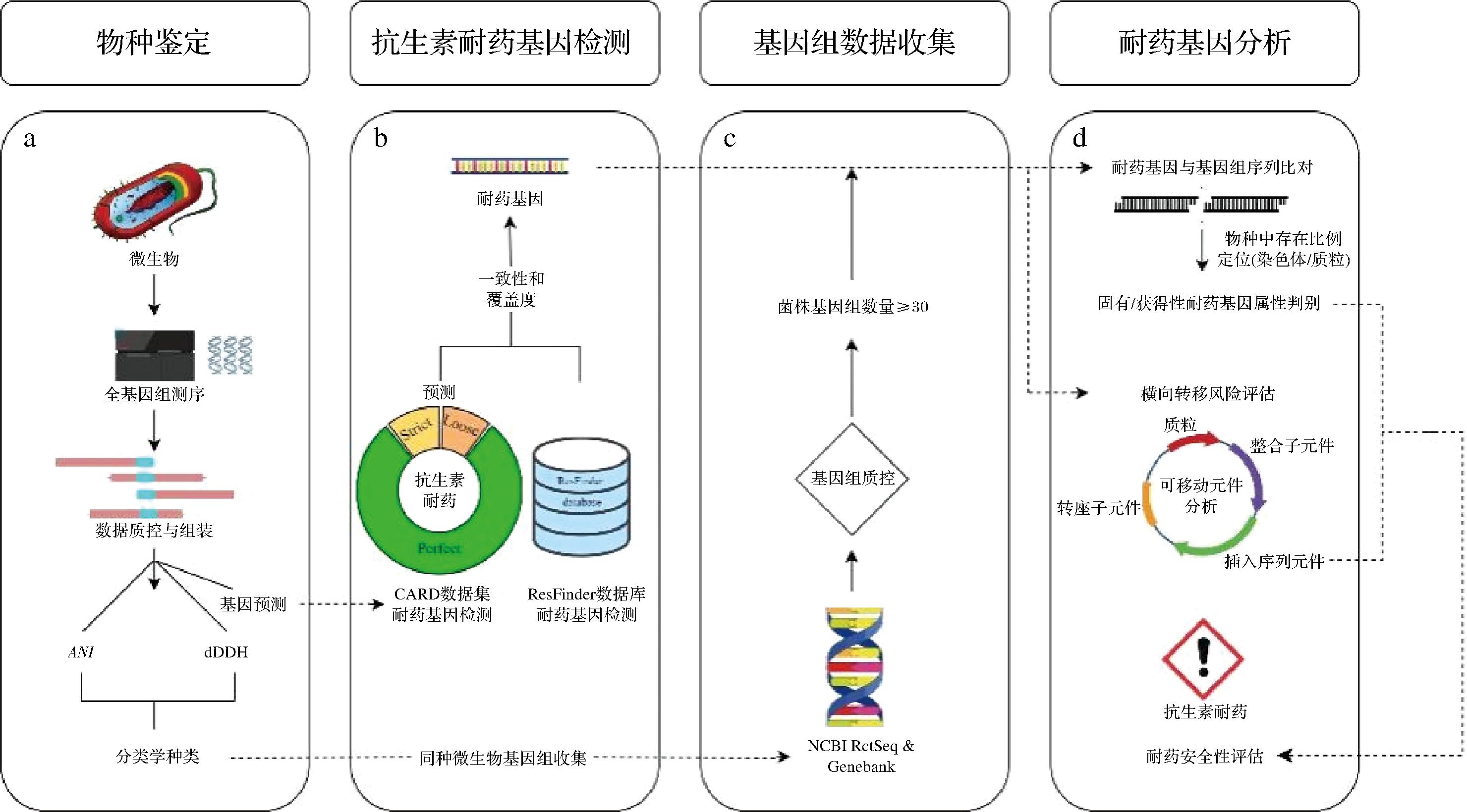
图1 基于全基因组数据的细菌抗生素耐药基因分析流程图
Fig.1 Flow chart of bacterial antibiotic resistance gene analysis based on whole genome data
5 可移动遗传元件(mobile genetic elements, MGE)
获得性耐药基因的获取、积累与传播在很大程度上归咎于MGE的活动。MGEs是指一类能够促进DNA在细胞内(如从染色体到质粒或质粒相互间)或细胞间移动的遗传元件[26]。
质粒、插入序列、转座子和整合子是常见的微生物MGEs[26]。质粒是染色体外的线性或环状 DNA 序列,通常自身包含复制起始序列。当携带耐药基因时,质粒具有将该基因转移至新宿主的风险。质粒的传递性在乳酸菌、乳球菌、肠球菌和链球菌中已有广泛研究[18]。插入序列(insertion sequence, IS)是分散的DNA片段,能够近乎随机地将自身(及相关耐药基因)移动到单个细胞内DNA分子中的新位置。IS携带转座酶基因,其自身不携带其他外源基因,但2个IS可形成复合转座子结构,并将耐药基因作为其中一部分进行移动。已有研究揭示IS6家族成员IS26、IS257和IS1216等在革兰氏阴性和阳性菌的部分耐药基因传播中扮演了关键作用[26]。转座子是一种比IS更大的遗传元件,其末端含有反向重复序列,内部包含转座酶基因和耐药基因等外源基因。转座子具备在DNA上迁移至新位点的能力,这一过程涉及交错切割靶 DNA、将转座子连接到突出单链末端、并填补由此产生的空缺从而完成转座。多种耐药基因与Tn3家族、Tn7样超家族等紧密相关[26]。整合子是一类高效的基因捕获系统,依赖于位点特异性重组机制,能够在既定位点之间迁移耐药基因。整合子的结构包含3个核心元件:酪氨酸重组酶(整合酶)编码基因、整合酶识别的相邻重组位点以及基因盒转录与表达所必需的启动子。上述MGEs参与耐药基因转移的过程示例如图2所示。
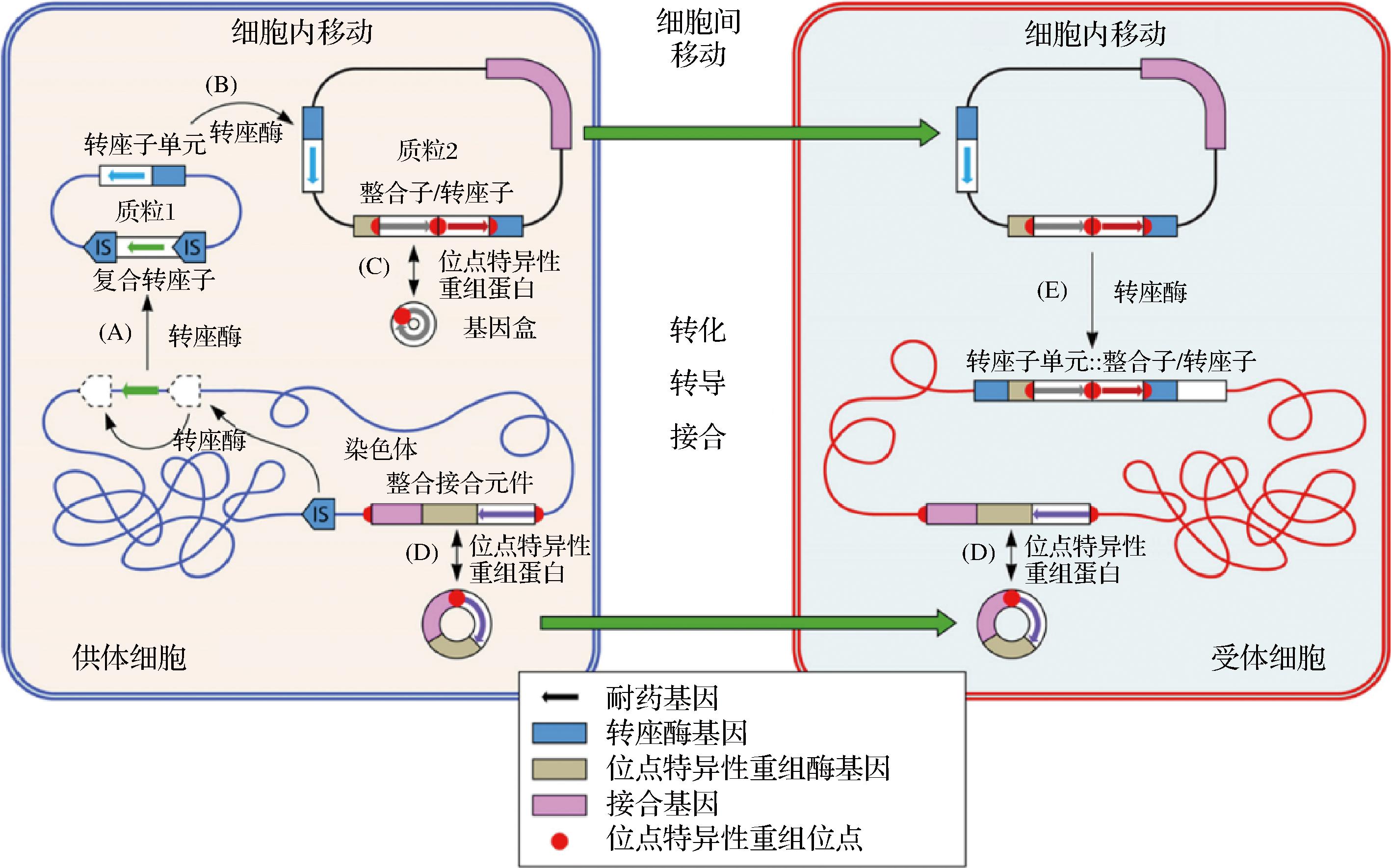
图2 抗生素耐药基因细胞内移动或细胞间转移的相关MGEs及过程示例[26]
Fig.2 Examples of MGEs and processes involved in intracellular mobility or intercellular transfer of antibiotic resistance genes[26]
注:图中包括2个不同菌株或物种的细胞,一个为供体细胞(细胞膜与染色体显示为蓝色,含2个质粒),另一个为受体细胞(细胞膜与染色体显示为红色)。黑色细箭头代表细胞内移动过程,由转座酶与位点特异性重组蛋白介导;绿色粗箭头代表细胞间(水平)转移过程。A-在耐药基因的两侧连续插入相同的 IS 可使其被捕获为复合转座子的一部分移动至另一个 DNA 分子(例如,从染色体到质粒)。B-携带耐药基因的转座子单元可以在质粒之间移动或从质粒移动到染色体,反之亦然。C-基因盒可以通过圆形中间体在整合子之间移动。D-整合接合元件可整合到染色体中,也可作为环状元件被切除,后者可继续由结合进入受体细胞并在特定的重组位点(可逆地)整合至染色体中。质粒可通过接合介导其自身在细胞间的转移,若缺乏接合区域,也可被其他质粒移动(或借助噬菌体转导或转化水平移动)。E-质粒进入受体细胞后,其内部 的转座子和/或整合子及相关耐药基因可移动至受体细胞染色体或其他质粒中。
近年来,研究发现食品和饲料用细菌可通过MGEs传播耐药基因。FATAHI-BAFGHI等[27]对126个益生菌基因组开展分析,发现多数耐药基因由MGEs转移而来。KASZAB等[18]对4 286个乳酸菌全基因组序列分析结果显示,194株菌携带至少一种可转移的耐药基因,且四环素耐药基因tetM和tetW最普遍。T TH等[28]的研究显示,动物双歧杆菌中存在的tetW基因与ISBian1插入序列相关;粪肠球菌中存在的tetM基因与转座子Tn6009相邻;长双歧杆菌、动物双歧杆菌与粪肠球菌分别携带含tetW/N/W和AAC(6′)-Ii的质粒。CHOI等[29]发现屎肠球菌的亚分支II菌株可能通过水平基因转移获得并携带大量的耐药、毒力和细菌素相关基因及MGEs,对人类抗生素产生适应。PlasmidFinder、ISFinder、TransposonPSI、IntegronFinder等专用数据库和软件可用于MGEs分析。然而由于MGEs内常存在重复区、变异区等复杂结构,组装难度较大,且一些低丰度的质粒存在组装遗漏的风险,目前尚未形成标准化的MGEs分析方法,有待进一步深入研究。
TH等[28]的研究显示,动物双歧杆菌中存在的tetW基因与ISBian1插入序列相关;粪肠球菌中存在的tetM基因与转座子Tn6009相邻;长双歧杆菌、动物双歧杆菌与粪肠球菌分别携带含tetW/N/W和AAC(6′)-Ii的质粒。CHOI等[29]发现屎肠球菌的亚分支II菌株可能通过水平基因转移获得并携带大量的耐药、毒力和细菌素相关基因及MGEs,对人类抗生素产生适应。PlasmidFinder、ISFinder、TransposonPSI、IntegronFinder等专用数据库和软件可用于MGEs分析。然而由于MGEs内常存在重复区、变异区等复杂结构,组装难度较大,且一些低丰度的质粒存在组装遗漏的风险,目前尚未形成标准化的MGEs分析方法,有待进一步深入研究。
6 对我国相关法规标准的借鉴意义
抗生素耐药性是菌种安全性评价的重要方面。在我国,食品微生物菌种耐药性管理由国家市场监督管理总局与国家卫生健康委员会共同负责,饲料微生物菌种的耐药性由中华人民共和国农业农村部负责。
近年来,我国已出台了一系列涉及菌种耐药性评价的法规(表3),包括《新食品原料申报与受理规定》(国卫食品发〔2013〕23号)[3]、《新饲料添加剂申报材料要求》(农业农村部第226号)[4]等。在菌种耐药性基因安全评价方面,我国也制定了一系列标准与指南,如《直接饲喂微生物和发酵制品生产菌株鉴定及其安全性评价指南》(农办牧〔2021〕43 号)[9]、GB 31615.2—2025《食品安全国家标准 食品用菌种安全性评价程序》(征求意见稿)等。值得注意的是,《新饲料添加剂申报材料要求》(农业农村部第226号)[4]与新发布的《食品加工用遗传修饰微生物安全性评价申报材料要求(试行)》[30]将基于全基因组序列的耐药基因分析分别纳入新饲料添加剂与食品加工用遗传修饰微生物的申报要求,体现了我国对微生物菌种耐药基因安全评价的重视。然而,在配套法规的标准与技术体系方面尚有待进一步完善。
表3 欧盟和中国与抗生素耐药基因相关法规与标准
Table 3 Regulations and standards about antibiotic resistance genes in European Union and China

国家/地区法规/标准名称发布机构发布时间重点内容欧盟《执行欧盟议会和理事会条例(EC)No 1831/2003的实施细则,关于准备和提供申请及饲料添加剂的评估和批准》(欧盟议会和理事会条例(EC) No 429/2008)欧盟委员会2008年2.2.2.2 微生物所有细菌菌株均应测定其对人类和兽医医学用抗生素的耐药性。若检测到耐药性,则应确定耐药性的遗传基础及其转移至其他肠道群菌的可能性。携带对一种或多种抗微生物药物获得性耐药的微生物菌株不得用作饲料添加剂,除非能够证明该耐药性源自染色体突变且不可转移《用作饲料添加剂或其生产菌株的微生物特征描述指南》欧洲食品安全局2018年2.2.2 应查找全基因组序列中否存在编码或促使对用于人和动物的抗菌剂[至关重要抗微生物药物(critically important antimicrobials, CIA)或高度重要抗微生物药物(highly important antimicrobials, HIA)]产生耐药的基因。应与最新数据库进行比较(例如CARD、ARG-ANNOT、ResFind-er)。分析结果应以表格的形式给出,关注于编码耐药性的完整基因。该表至少应包括基因名称、编码蛋白的功能、相似性百分比和e值《用作生产食品酶的微生物特征描述》欧洲食品安全局2019年1.3 应查找全基因组序列中否存在编码或促使对用于人和动物的抗菌剂(CIA或HIA)产生耐药的基因。应与最新数据库进行比较(例如CARD、ARG-ANNOT、ResFinder)。分析结果应以表格的形式给出,关注于编码耐药性的完整基因。该表至少应包括基因名称、编码蛋白的功能、相似性百分比和e值《关于如何解释“获得性耐药基因”QPS资格的声明》欧洲食品安全局2023年3.3 在欧洲食品安全局风险评估的框架下,与固有耐药相关的比对结果将被视为无关紧要。任何产生耐药表型的获得性耐药基因应被视为具有危害性/关注点。该耐药表型使细菌在高于其他同种菌株所耐受的抗菌药物浓度下存活或繁殖。若获得性耐药基因并未导致表型抗性,则需进一步评估耐药基因是否会表达《关于有意引入食品链的微生物WGS分析要求的声明》欧洲食品安全局2024年2.5.3 当检索抗微生物药物耐药基因时,建议至少与2个维护的数据库进行序列比对,使用覆盖度的最低可用阈值。序列比对时,通常设置序列相似度≥80%、序列覆盖度≥70%中国《新食品原料申报与受理规定》(国卫食品发〔2013〕23号)原国家卫生与计划生育委员会2013年第十二条 (四)微生物耐药性试验报告和产毒能力试验报告《新饲料添加剂申报材料要求》(农业农村部第226号)农业农村部2019年附件1 新饲料添加剂申报材料要求八(四)菌株案例性评价报告。通过微生物表型试验、分子生物学试验和全基因组序列分析,结合相关文献资料,对拟评价菌株的致病性、有毒代谢产物产生能力及抗菌药物耐药性等进行综合评价《益生菌类保健食品申报与审评规定》(征求意见稿)国家市场监督管理总局2019年第四条 (六)菌种(株)的致病性试验、耐药性试验等安全性评价报告《食品加工用遗传修饰微生物安全性评价申报材料要求(试行)》国家食品安全风险评估中心2024年2.6.2 (1)基于全基因组测序进行的毒力基因、耐药基因、毒素产生相关基因等分析;(2)遗传修饰微生物的致病性、耐药性和产毒能力试验报告GB 31615.2—2025《食品安全国家标准 食品用菌种安全性评价程序》国家卫生健康委员会/国家市场监督管理总局2025年4.2 全基因组测序对菌种进行全基因组测序,分别获得其基因组框架图和完成图藻类除外,并对测序数据的毒力基因、耐药基因、毒素产生相关基因等进行综合分析农业行业标准NY/T 1444—2007《微生物饲料添加剂技术通则》农业农村部2007年4.3.2.3 对功能微生物安全性的定性检测至少应包括以下试验:抗生素耐药性的检测国家标准GB/T 23181—2008《微生物饲料添加剂通用要求》国家质量监督检验检疫总局/中国国家标准化管理委员会2008年4.3.2 由具有资质的部门完成微生物饲料添加剂功能菌株的常规耐药性实验团体标准T/CSWSL 016—2019《水产动物饮用乳酸菌筛选标准》北京生物饲料产业技术创新战略联盟2019年5.3.2 将拟评价菌株的基因组序列与已有的数据库(包括但不限于CARD、ARG-ANNOT、ResFinder、VFDB、PAIDB、MvirDB、CGE等)最新版本中存储的序列进行比对,并分析拟评价菌种遗传物质中与抗生素耐药性相关的耐药性基因与致病相关的毒力因子基因的存在情况
续表3
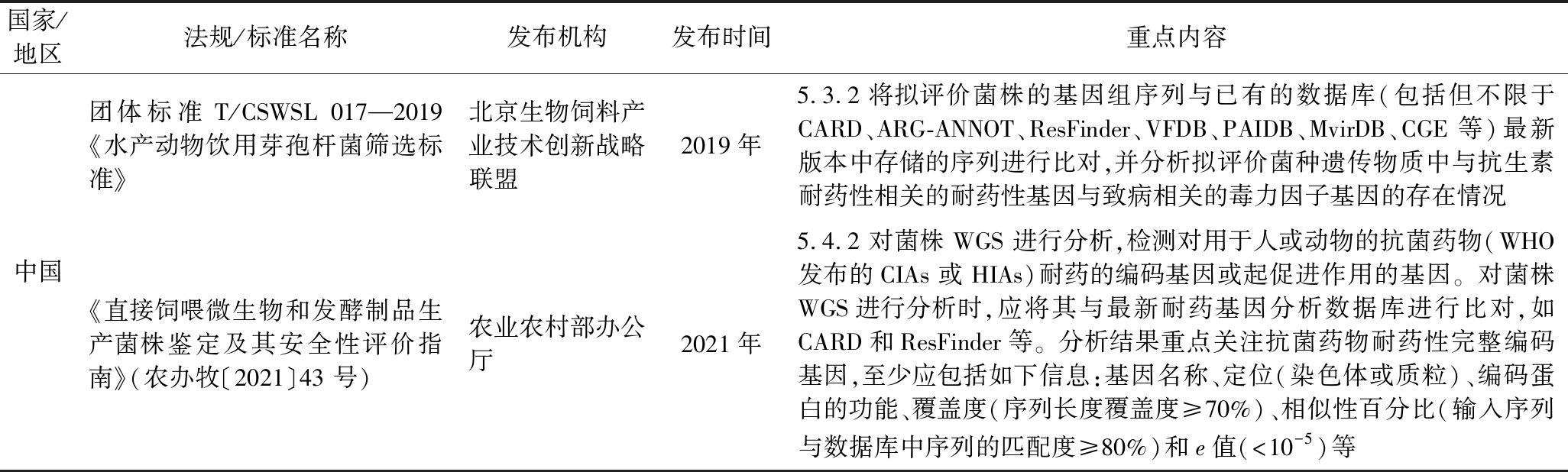
国家/地区法规/标准名称发布机构发布时间重点内容中国团体标准T/CSWSL 017—2019《水产动物饮用芽孢杆菌筛选标准》北京生物饲料产业技术创新战略联盟2019年5.3.2 将拟评价菌株的基因组序列与已有的数据库(包括但不限于CARD、ARG-ANNOT、ResFinder、VFDB、PAIDB、MvirDB、CGE等)最新版本中存储的序列进行比对,并分析拟评价菌种遗传物质中与抗生素耐药性相关的耐药性基因与致病相关的毒力因子基因的存在情况《直接饲喂微生物和发酵制品生产菌株鉴定及其安全性评价指南》(农办牧〔2021〕43 号)农业农村部办公厅2021年5.4.2 对菌株 WGS 进行分析,检测对用于人或动物的抗菌药物(WHO发布的CIAs 或 HIAs)耐药的编码基因或起促进作用的基因。对菌株WGS进行分析时,应将其与最新耐药基因分析数据库进行比对,如CARD和ResFinder等。分析结果重点关注抗菌药物耐药性完整编码基因,至少应包括如下信息:基因名称、定位(染色体或质粒)、编码蛋白的功能、覆盖度(序列长度覆盖度≥70%)、相似性百分比(输入序列与数据库中序列的匹配度≥80%)和e值(<10-5)等
在微生物菌种耐药性评估领域,欧盟、美国等发达国家和地区在监管、法规、标准和技术体系等方面都处于国际领先地位。欧洲食品安全局自2018年起相继发布了《用作饲料添加剂或其生产菌株的微生物特征描述指南》[7]、《用作生产食品酶的微生物特征描述》[8]、《关于有意引入食品链的微生物WGS分析要求的声明》[22]、《关于如何解释“获得性耐药基因”QPS资格的声明》[25]等指南文件,为WGS的数据质控、序列比对阈值设置、耐药基因属性分析等内容提供了有价值的参考(表3)。这些国际经验对我国细菌耐药性评估体系的规范与发展具有重要借鉴意义。
当前,在国际社会共同努力下,标准法规中关于测序数据质量要求、耐药基因与MGEs的标准化分析方法正逐步完善;公共数据库中耐药基因的种类与数量持续增长,为研究人员利用先进的人工智能和深度学习模型预测新型耐药基因奠定了基础[31]。与此同时,可快速、高效和原位筛查耐药基因的新技术不断涌现,如可动态富集和去除特定序列的原位测序(on-site sequencing)[32]、快速即时耐药基因诊断[33]以及新型耐药机制的自动文献挖掘[34]等。这些新技术的应用将进一步促进细菌菌种耐药性评估体系的完善和发展,为保障人类健康、促进畜牧业可持续发展提供有力的技术支撑。
[1] FAO. The FAO Action Plan on Antimicrobial Resistance 2021—2025[M].Rome:FAO, 2021.
[2] FAO/WHO.Joint FAO/WHO working group report on drafting guidelines for the evaluation of probiotics in food[C].Guidelines for the evaluation of probiotics in food.London:FAO/WHO, 2002.
[3] 国家卫生与计划生育委员会. 新食品原料申报与受理规定(国卫食品发23号)[Z].2013.
National Health and Family Planning Commission.New Food Raw Material Registration and Acceptance Regulation (No.23)[Z].2013.
[4] 中华人民共和国农业农村部. 新饲料添加剂申报材料要求(农业农村部第226号公告)[EB/OL].(2019-11-04)[2024-10-16]. https://www.moa.gov.cn/govpublic/xmsyj/201911/t20191107_6331531.htm
Ministry of Agriculture and Rural Affairs of the People's Republic of China. New feed additives declaration material requirements (No. 226)[EB/OL].(2019-11-04)[2024-10-16]. https://www.moa.gov.cn/govpublic/xmsyj/201911/t20191107_6331531.htm
[5] DARBY E M, TRAMPARI E, SIASAT P, et al.Molecular mechanisms of antibiotic resistance revisited[J].Nature Reviews.Microbiology, 2023, 21(5):280-295.
[6] WHO.Critically Important Antimicrobials for Human Medicine[M].6th revision.Geneva:World Health Organization, 2018.
[7] EFSA Panel on Additives and Products or Substances used in Animal Feed (FEEDAP), RYCHEN G, AQUILINA G, et al.Guidance on the characterisation of microorganisms used as feed additives or as production organisms[J].EFSA Journal, 2018, 16(3):e05206.
[8] EFSA PANEL ON FOOD CONTACT MATERIALS E A P A, SILANO V, BAVIERA J M B, et al.Characterisation of microorganisms used for the production of food enzymes[J].EFSA Journal, 2019, 17(6):e05741.
[9] 中华人民共和国农业农村部. 直接饲喂微生物和发酵制品生产菌株鉴定及其安全性评价指南(农办牧〔2021〕43 号)[EB/OL].(2021-11-01)[2024-10-16]. https://www.moa.gov.cn/govpublic/xmsyj/202111/t20211105_6381450.htm
Ministry of Agriculture and Rural Affairs of the People's Republic of China. Guidelines for identification and safety evaluation of Direct-Fed microorganisms and strains for fermentation productions (No. 43 [2021]) [EB/OL].(2021-11-01)[2024-10-16]. https://www.moa.gov.cn/govpublic/xmsyj/202111/t20211105_6381450.htm
[10] BOOLCHANDANI M, D’SOUZA A W, DANTAS G.Sequencing-based methods and resources to study antimicrobial resistance[J].Nature Reviews.Genetics, 2019, 20(6):356-370.
[11] 钱璟, 吴哲元, 郭晓奎, 等.耐药微生物和抗生素耐药基因与全健康[J].微生物学通报, 2022, 49(10):4412-4424.
QIAN J, WU Z Y, GUO X K, et al.Antibiotic-resistant microbes, antibiotic resistance genes and One Health[J].Microbiology China, 2022, 49(10):4412-4424.
[12] WHO.WHO Bacterial Priority Pathogens List 2024:Bacterial Pathogens of Public Health Importance to Guide Research, Development and Strategies to Prevent and Control Antimicrobial Resistance[M].Geneva:World Health Organization, 2024.
[13] VAN HOEK A H A M, MEVIUS D, GUERRA B, et al.Acquired antibiotic resistance genes:An overview[J].Frontiers in Microbiology, 2011, 2:203.
[14] STOGIOS P J, SAVCHENKO A.Molecular mechanisms of vancomycin resistance[J].Protein Science, 2020, 29(3):654-669.
[15] ANDERSSON D I, HUGHES D.Antibiotic resistance and its cost:Is it possible to reverse resistance?[J].Nature Reviews.Microbiology, 2010, 8(4):260-271.
[16] FL REZ A B, LADERO V, PABLO
REZ A B, LADERO V, PABLO  M, et al.Acquired macrolide resistance in the human intestinal strain Lactobacillus rhamnosus E41 associated with a transition mutation in 23S rRNA genes[J].International Journal of Antimicrobial Agents, 2007, 30(4):341-344.
M, et al.Acquired macrolide resistance in the human intestinal strain Lactobacillus rhamnosus E41 associated with a transition mutation in 23S rRNA genes[J].International Journal of Antimicrobial Agents, 2007, 30(4):341-344.
[17] GUEIMONDE M, S NCHEZ B, DE LOS REYES-GAVIL
NCHEZ B, DE LOS REYES-GAVIL N C G, et al.Antibiotic resistance in probiotic bacteria[J].Frontiers in Microbiology, 2013, 4:202.
N C G, et al.Antibiotic resistance in probiotic bacteria[J].Frontiers in Microbiology, 2013, 4:202.
[18] KASZAB E, LACZK L, KARDOS G, et al.Antimicrobial resistance genes and associated mobile genetic elements in Lactobacillales from various sources[J].Frontiers in Microbiology, 2023, 14:1281473.
L, KARDOS G, et al.Antimicrobial resistance genes and associated mobile genetic elements in Lactobacillales from various sources[J].Frontiers in Microbiology, 2023, 14:1281473.
[19] GUEIMONDE M, FL REZ A B, VAN HOEK A H A M, et al.Genetic basis of tetracycline resistance in Bifidobacterium animalis subsp.lactis[J].Applied and Environmental Microbiology, 2010, 76(10):3364-3369.
REZ A B, VAN HOEK A H A M, et al.Genetic basis of tetracycline resistance in Bifidobacterium animalis subsp.lactis[J].Applied and Environmental Microbiology, 2010, 76(10):3364-3369.
[20] SERAFINI F, BOTTACINI F, VIAPPIANI A, et al.Insights into physiological and genetic mupirocin susceptibility in bifidobacteria[J].Applied and Environmental Microbiology, 2011, 77(9):3141-3146.
[21] GOODWIN S, MCPHERSON J D, RICHARD MCCOMBIE W.Coming of age:Ten years of next-generation sequencing technologies[J].Nature Reviews.Genetics, 2016, 17(6):333-351.
[22] EFSA.EFSA statement on the requirements for whole genome sequence analysis of microorganisms intentionally used in the food chain[J].EFSA Journal, 2021, 19(7):e06506.
[23] 国家市场监督管理总局. 保健食品原料用菌种安全性检验与评价技术指导原则(2020年版)[EB/OL].(2020-10-31)[2024-10-16]. https://www.samr.gov.cn/zw/zfxxgk/fdzdgknr/tssps/art/2023/art_d0c45ed1dc6d4ff4a7eb6bb9baac0bb1.htmlState Administration for Market Regulation. Guideline on the safety inspection and evaluation of strains used in health food raw materials[EB/OL].(2020-10-31)[2024-10-16]. https://www.samr.gov.cn/zw/zfxxgk/fdzdgknr/tssps/art/2023/art_d0c45ed1dc6d4 ff4a7eb6bb9baac0bb1.html
[24] YIN X L, ZHENG X W, LI L G, et al.ARGs-OAP v3.0:Antibiotic-resistance gene database curation and analysis pipeline optimization[J].Engineering, 2023, 27:234-241.
[25] EFSA Panel on Biological Hazards (BIOHAZ), KOUTSOUMANIS K, ALLENDE A, et al.Statement on how to interpret the QPS qualification on ‘acquired antimicrobial resistance genes’[J].EFSA Journal, 2023, 21(10):e08323.
[26] PARTRIDGE S R, KWONG S M, FIRTH N, et al.Mobile genetic elements associated with antimicrobial resistance[J].Clinical Microbiology Reviews, 2018, 31(4):e00088-17.
[27] FATAHI-BAFGHI M, NASERI S, ALIZEHI A.Genome analysis of probiotic bacteria for antibiotic resistance genes[J].Antonie Van Leeuwenhoek, 2022, 115(3):375-389.
[28] T TH A G, CSABAI I, JUDGE M F, et al.Mobile antimicrobial resistance genes in probiotics[J].Antibiotics, 2021, 10(11):1287.
TH A G, CSABAI I, JUDGE M F, et al.Mobile antimicrobial resistance genes in probiotics[J].Antibiotics, 2021, 10(11):1287.
[29] CHOI D G, BAEK J H, HAN D M, et al.Comparative pangenome analysis of Enterococcus faecium and Enterococcus lactis provides new insights into the adaptive evolution by horizontal gene acquisitions[J].BMC Genomics, 2024, 25(1):28.
[30] 国家食品安全风险评估中心. 食品加工用遗传修饰微生物安全性评价申报材料要求(试行)[EB/OL].(2024-09-13)[2024-10-16]. https://www.cfsa.net.cn/zxdt/tzgg/2024/14077.shtml
China National Center for Food Safety Risk Assessment. Requirements for application materials for safety evaluation of genetically modified microorganisms for food processing (Trial) [EB/OL].(2024-09-13)[2024-10-16]. https://www.cfsa.net.cn/zxdt/tzgg/2024/14077.shtml
[31] WONG F, ZHENG E J, VALERI J A, et al.Discovery of a structural class of antibiotics with explainable deep learning[J].Nature, 2024, 626(7997):177-185.
[32] KOVAKA S, FAN Y F, NI B H, et al.Targeted nanopore sequencing by real-time mapping of raw electrical signal with UNCALLED[J].Nature Biotechnology, 2021, 39(4):431-441.
[33] VASALA A, HYTÖNEN V P, LAITINEN O H.Modern tools for rapid diagnostics of antimicrobial resistance[J].Frontiers in Cellular and Infection Microbiology, 2020, 10:308.
[34] EDALATMAND A, MCARTHUR A G.CARD*Shark:Automated prioritization of literature curation for the Comprehensive Antibiotic Resistance Database[J].Database, 2023, 2023:baad023.