谷氨酰胺酶(EC 3.5.1.2)是一类酰胺水解酶,能够将1分子的谷氨酰胺通过水解脱酰胺作用生成1分子的谷氨酸和1分子的游离氨。这一反应在食品工业、医药行业等不同领域都有着重要的研究与应用价值[1]。
在食品行业,谷氨酰胺酶的催化作用可以通过水解谷氨酰胺的方式来增强食品的风味,因为该过程产生的谷氨酸是重要的风味氨基酸种类之一[2-4]。尤其是在酱油的酿造过程中,谷氨酰胺酶不仅可以通过这一反应增加酱油的鲜味,同时还一定程度上降低了发酵过程中谷氨酰胺通过非酶性焦化反应生成的无味的焦谷氨酸的水平,从而提升酱油的品质[5-8]。另一方面,由于谷氨酰胺作为癌症细胞的生物能量和生物合成需求的重要碳源[9-10],且在缺氧条件下其消耗量是正常细胞的15倍[11],通过降低血液中谷氨酰胺的水平来控制癌细胞生长的策略是癌症治疗的重要途径之一[12-13],所以在医药行业中,谷氨酰胺酶催化谷氨酰胺水解的作用也具有重要的研究意义。此外,有研究表明谷氨酰胺酶可以通过催化过程提供的氢原子以及释放的酸性物质——谷氨酸来消除自由基[14],从而达到抗氧化的效果,并且谷氨酸也是合成体内强效抗氧化剂谷胱甘肽的重要前体[15]。
在实际的工业应用过程中,往往要求酶具有较好的稳定性能[16],所以耐热酶通常在生产当中更具有优势。耐热酶的最适温度较高,且其分离、纯化、运输等过程均可在常温下进行,活性的保持时间也较长,这使得生产的成本与能耗等方面得到降低。然而天然酶生长条件苛刻且缓慢,产量也欠佳,利用基因工程等方法实现异源表达重组酶是较为有效的解决办法。谷氨酰胺酶在大肠杆菌、芽孢杆菌、乳杆菌、青霉菌、米曲霉菌、酵母以及部分根瘤菌中均有表达[17],有研究通过在大肠杆菌、酿酒酵母、枯草芽孢杆菌、草木樨中华根瘤菌以及原生质体等宿主菌株中进行了异源重组表达[18],其中大肠杆菌被选择为宿主的研究居多,多数研究都提高了谷氨酰胺酶的酶活力,重组酶的最适反应温度基本在30~50 ℃,最适反应pH值为7.0~9.0[7]。本实验室的研究多集中针对一种嗜热古细菌——嗜热玫瑰红球菌(Thermomicrobium roseum DSM 5159),这株嗜热古细菌筛选自美国黄石公园碱性沸泉,所适宜的生长温度为70~75 ℃[19]。本研究在其全基因组数据内预测了一种由279个氨基酸组成的碳氮水解酶家族蛋白(WP_012642565.1),根据美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库对其催化功能给出的预测,了解到该酶可能具有断裂碳氮键,参与有机氮化合物的还原和氮的生成反应的功能。将该基因克隆至pET28a(+)质粒中,转化至大肠杆菌E.coli BL21(DE3)中进行异源可溶性表达,经过Ni-NTA亲和层析纯化得到纯酶,通过基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/ionization time of flight mass spectrometry, MALDI-TOF-MS)鉴定重组表达的产物,进一步发现该重组酶具有谷氨酰胺酶功能,并对其酶学性质进行表征。针对该古生菌来源谷氨酰胺酶的蛋白结构与酶学功能的研究为相关工业生产应用提供了一定的参考价值。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株质粒
嗜热玫瑰红球菌来源的谷氨酰胺酶的氨基酸序列(GenBank:WP_012642565.1)来自NCBI,由天霖生物科技(无锡)有限公司进行全基因合成;质粒pET28a(+)由天霖生物科技(无锡)有限公司提供;大肠杆菌E.coli BL21(DE3)为本实验室保存。
1.1.2 试剂与培养基
L-谷氨酰胺、D-谷氨酰胺、硫酸卡那霉素(kana)、异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactoside,IPTG),上海麦克林生化科技股份有限公司;180 kDa蛋白预染marker、BCA蛋白质定量试剂盒,南京诺唯赞生物科技有限公司;铁锤超级裂菌液,南京艾思易生物科技有限公司;其他试剂均为国产分析纯。
LB培养基(g/L):酵母提取物5.0,胰蛋白胨10.00,NaCl 10.0,琼脂粉(固体培养基)20.0,121 ℃高压灭菌20 min。
TB液体培养基(g/L):酵母提取物24.0,胰蛋白胨12.0,甘油4.0 mL,KH2PO4 2.31,K2HPO4 12.54,121 ℃高压灭菌20 min。
1.2 仪器与设备
高速冷冻离心机,日立(中国)有限公司;酶标仪、Biologic ALX 250分光光度计,赛默飞世尔科技公司;恒温金属浴,杭州博日科技股份有限公司;差示扫描热量仪(nano-differential scanning calorimetry,nano-DSC),美国TA公司。
1.3 实验方法
1.3.1 重组菌株的构建与诱导表达
从NCBI数据库下载嗜热玫瑰红球菌全基因数据预测的碳氮水解酶家族蛋白的氨基酸序列(WP_012642565.1),通过Java Codon Adaptation Tool (JCat)在线软件对氨基酸序列进行密码子优化。送天霖生物科技(无锡)有限公司进行全基因合成,并由公司在质粒pET28a(+)的Nde Ⅰ和Hind Ⅲ两个酶切位点之间插入该段合成基因,构建重组质粒。将重组质粒转化至E.coli BL21(DE3),测序验证正确后,将重组菌株划线于卡那抗性平板培养基,37 ℃倒置培养12 h,挑取单菌落接种于10 mL LB液体培养基(kana,50 μg/mL)中培养12 h(37 ℃,200 r/min),以1%的接种量转接至50 mL LB液体培养基(kana,50 μg/mL)中培养8 h(37 ℃,200 r/min),后以2%的接种量转接至150 mL TB液体培养基(kana,50 μg/mL)中培养2 h(37 ℃,200 r/min)(OD600值达到0.8),然后加入终浓度为0.1 mmol/L的IPTG,诱导培养10 h(28 ℃,200 r/min),收集菌体并贮存于-20 ℃备用。
1.3.2 重组谷氨酰胺酶的分离纯化及SDS-PAGE分析
将菌体重悬于含有150 mmol/L NaCl的20 mmol/L Tris-HCl缓冲液(pH 8.0)中,加入铁锤超级裂菌液(TieChui E.coli Lysis Buffer)并于冰上搅拌10 min破碎细胞,将细胞破碎液在4 ℃条件下进行高速离心。上清液即粗酶液用于可溶性蛋白纯化,利用Ni-NTA亲和纯化法进行纯化,采用SDS-PAGE分析蛋白纯化结果。
1.3.3 MALDI-TOF-MS分析
回收SDS-PAGE凝胶中500 mmol/L咪唑洗脱液组泳道中符合重组酶分子质量大小的单一条带于1.5 mL EP管中,通过脱色、溶化、消化等处理后[20],用MALDI-TOF-MS进行鉴定。
1.3.4 重组谷氨酰胺酶结构及功能预测
通过swiss model在线网站(https://swissmodel.expasy.org/interactive)对碳氮水解酶家族蛋白氨基酸序列(WP_012642565.1)进行三维结构预测。利用Discovery Studio软件,以NCBI数据库预测的催化三联体Glu44-Cys156-Lys115为中心拟定该酶的催化活性中心,分别与多种腈酶超家族的催化底物进行Dock Ligands(LibDock)分子对接,分子对接参数设置如表1所示。
表1 Discovery Studio软件分子对接的参数设置
Table 1 Parameter settings for molecular docking in Discovery Studio software

分子对接参数 具体设置numberofhotspots100dockingtolerance0.25dockingpreferenceshighqualityconformationmethodFASTminimizationalgorithmdonotminimizeparallelprocessingfalse
1.3.5 重组谷氨酰胺酶反应产物液相质谱检测
分别取1 mL重组酶与L-谷氨酰胺在50 ℃,pH值8.0条件下反应1 h后的反应产物、100 mmoL/L谷氨酰胺标准溶液以及100 mmoL/L谷氨酸标准溶液,在国家重点实验室平台采用AccQ·Tag方法通过超高效液相色谱串联四级杆飞行时间质谱联用仪鉴定重组酶反应产物的成分。
1.3.6 重组谷氨酰胺酶活力测定
通过邻苯二甲醛荧光法测定谷氨酰胺酶活力[21]。将360 μL的0.1 mol/L L-谷氨酰胺(pH值8.0)(20 mmol/L Tris-HCl缓冲液)与40 μL酶液的混合物置于50 ℃孵育1 h,加入800 μL 1 mol/L H3PO4溶液终止反应,将不含酶的缓冲液作为空白对照。将以上反应液保存于4 ℃冰箱待测。取40 μL样品与500 μL 0.1 mol/L H3BO3溶液(pH 9.5)混合后,再取混合液30 μL,加入135 μL 50 mmol/L邻苯二甲醛溶液、135 μL 50 mmol/L Na2SO3溶液,55 ℃孵育30 min后立即于黑色酶标板中测定荧光强度(λex:390 nm,λem:538 nm)。以NH4Cl标准溶液测定荧光强度-氨浓度标准曲线。其中50 mmol/L Na2SO3溶液需现配现用,故每次测定均需重新绘制氨标准曲线,且以上所用试剂溶液均由无氨水配制。以上反应均设置3组平行实验。酶活力定义:每分钟催化谷氨酰胺产生1 μmol氨所需要的酶量为1个酶活单位(U)。酶活力计算如公式(1)所示:
![]()
(1)
式中:Δc,将荧光强度增值通过氨标准曲线得到的氨浓度增值,mmol/L; Vt,反应体系体积,mL;VS,酶液样品体积,mL;t,反应时间,min。
1.3.7 重组谷氨酰胺酶的表达优化
采用L9(34)设计正交试验,以每克菌体经纯化得到的重组谷氨酰胺酶对L-谷氨酰胺的酶活力作为参考指标,设置诱导前菌液浓度OD600值、IPTG诱导浓度(mmol/L)、诱导温度(℃)与诱导时间(h)4个因素,如表2所示。经SPSS 26.0分析实验结果,得出最佳诱导培养条件。
表2 L9(34) 正交试验因素与水平表
Table 2 Factors and levels of the L9(34) orthogonal test

因素水平123A诱导前菌液浓度OD600值0.20.40.8BIPTG诱导浓度/(mmol/L)0.050.10.2C诱导温度/℃162837D诱导时间/h61014
1.3.8 重组谷氨酰胺酶的酶学性质测定
1.3.8.1 圆二色谱(circular dichroism,CD)分析与nano-DSC分析
取0.1 mg/mL纯酶液在国家重点实验室平台采用紫外(190~250 nm)CD光谱法测定重组谷氨酰胺酶的各类二级结构的含量[22]。采用nano-DSC测定重组L-谷氨酰胺酶的变性温度(Tm)以及焓变值(ΔH)。取1 mg/mL的纯酶液注入纳米差示扫描量热仪的样品池,以1 ℃/min的速率从40 ℃持续加热至110 ℃。通过Nano Analyze数据分析软件得到实验结果。
1.3.8.2 最适反应温度与pH值测定
在底物缓冲溶液的pH值为8.0的反应条件下,分别在4 ℃或20~100 ℃(间隔10 ℃)中反应1 h后测定酶活力,设定最高酶活力为100%,计算不同温度下重组谷氨酰胺酶的相对酶活力,测定重组酶的最适反应温度。分别在底物缓冲溶液pH值为5.0~10.0(间隔1.0)的反应条件下,在50 ℃恒温金属浴中反应1 h后测定酶活力,设定最高酶活力为100%,计算不同pH值条件下重组酶的相对酶活力,确定其最适反应pH值。
1.3.8.3 温度及pH值稳定性测试
将重组谷氨酰胺酶置于40~80 ℃(间隔10 ℃)温度下孵育2 h后,测定其酶活力,以未做处理的重组酶作为对照组,并定义其酶活力为100%,确定各温度处理下重组酶的残留酶活力。将重组酶置于pH值为4.0~11.0(间隔1.0)的缓冲溶液中,37 ℃孵育2 h后,测定其酶活力,同样以未做处理的重组酶的酶活力设定为100%,确定重组酶的pH稳定性。
1.3.8.4 动力学参数测定
选择不同浓度的L-谷氨酰胺和D-谷氨酰胺(1~30 mg/mL),在50 ℃,pH值为8.0条件下与重组谷氨酰胺酶反应30 min,通过邻苯二甲醛荧光法测定产物氨的生成速率,从而计算其初始反应速率,利用Origin软件中的Michaelis-Menten方程进行非线性曲线拟合,得到其Km和Vmax值。
1.3.8.5 半衰期测定
将重组L-谷氨酰胺酶分别置于40~80 ℃(间隔10 ℃)温度下孵育13 h,其中每隔1 h取1次样进行酶活力测定,以未处理的重组酶作为对照组,并定义其酶活力为100%,确定酶活力在不同温度下衰减至50%所需要的时间,即为相应温度下的半衰期![]()
1.3.8.6 金属离子对酶活力的影响
向酶液中加入不同的工业生产中常见金属离子[23](K+、Ca2+、Mg2+、Mn2+、Fe3+、Fe2+、Zn2+、Cu2+、Co2+、Ni2+、EDTA)至终浓度为1、10、100 mmol/L,置于37 ℃下孵育2 h,后测定重组酶活力,以未处理的重组酶的酶活力为对照组,设定其酶活力为100%,测定在不同金属离子影响下重组酶的酶活力。
2 结果与分析
2.1 重组谷氨酰胺酶的构建、表达与纯化
利用Java Codon Adaptation Tool在线软件对谷氨酰胺酶的氨基酸序列进行了密码子优化,不仅避免了某些限制性内切酶的切割位点以及DNA序列中的非依赖性转录终止子的存在,同时根据宿主菌E.coli BL21(DE3)的密码子偏好性提高重组谷氨酰胺酶的表达效率[24]。如图1-a所示,设计构建了重组质粒,可利用载体质粒pET28a(+)原有的组氨酸标签基因实现对重组蛋白的纯化。SDS-PAGE结果如图1-b所示,分析表明重组蛋白表达量高,且纯化条带单一,分子质量大小约为32 kDa,这与ExPasy网站预测的理论分子质量基本相符。MALDI-TOF-MS结果如图2所示,5条肽段匹配得分有3个可信度单独超过阈值分数,蛋白得分343分,质谱结果显示纯化得到的重组酶与NCBI数据库中预测的目标酶序列基本相同。
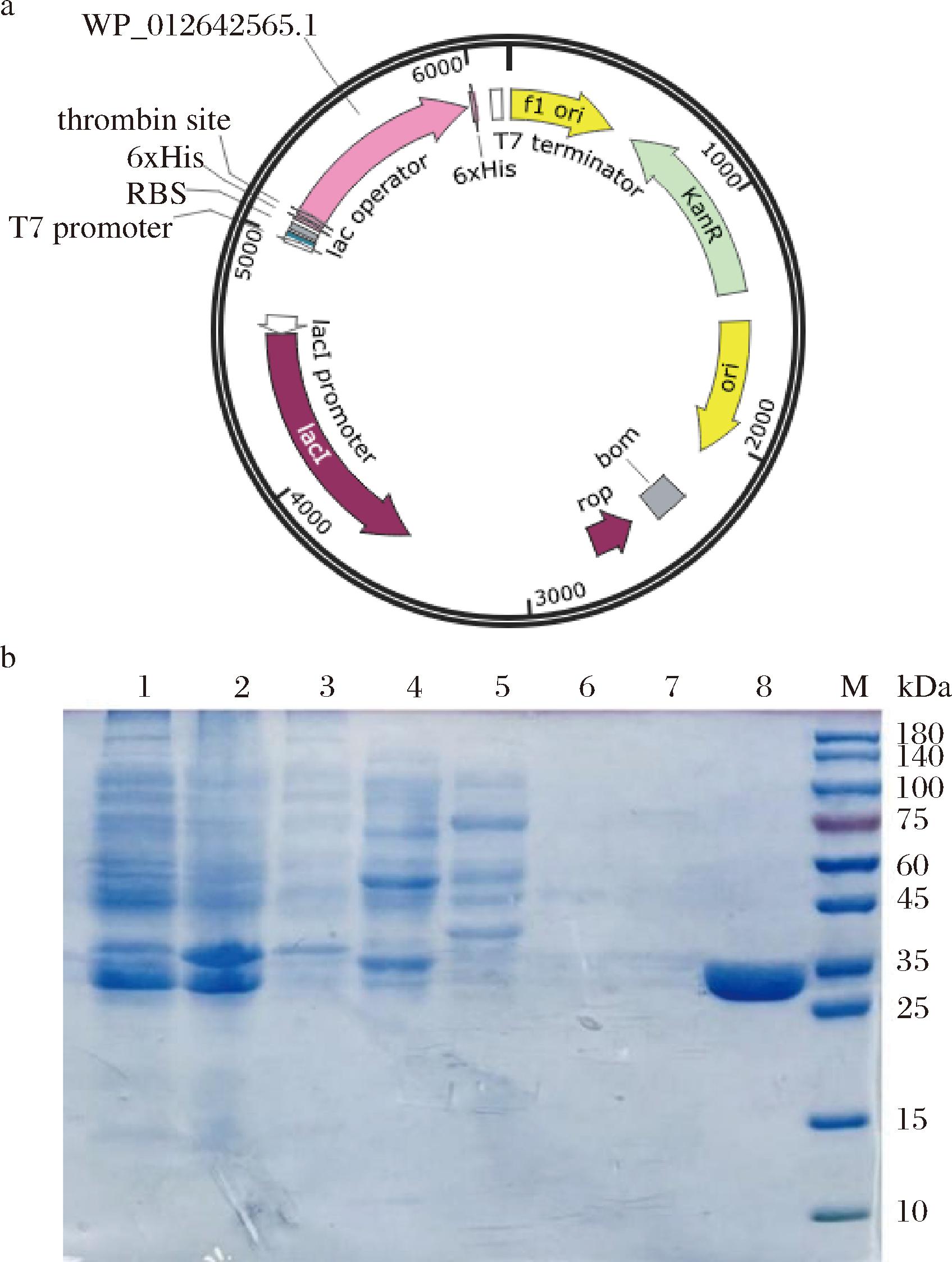
1-粗酶液;2-破碎沉淀;3-流穿液;4~7-20、50、100、200 mmol/L洗脱液;8-纯酶液。
a-重组质粒构建图谱;b-重组谷氨酰胺酶SDS-PAGE分析
图1 重组质粒的构建与表达
Fig.1 Construction and expression of recombinant plasmids
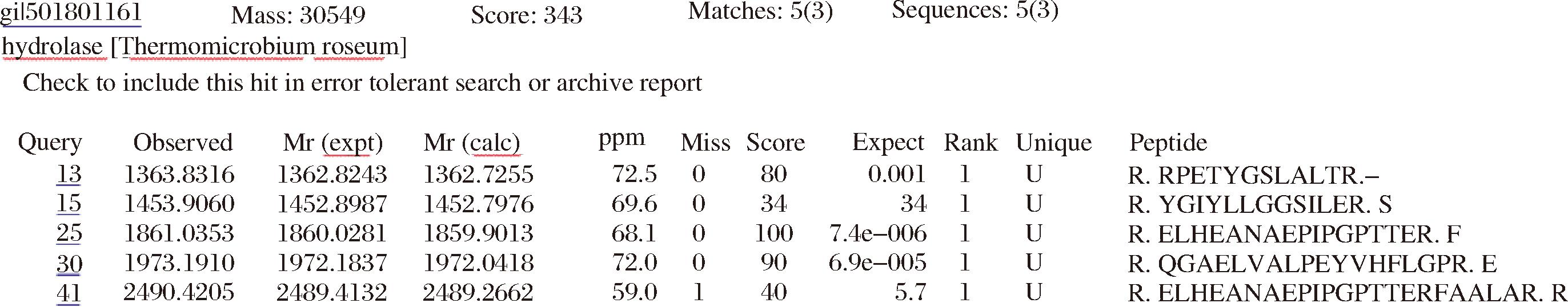
图2 重组谷氨酰胺酶的MALDI-TOF-MS分析结果
Fig.2 MALDI-TOF-MS analysis of recombinant glutaminase
注:MALDI-TOF-MS鉴定后实验样品与NCBI库中该基因编号(gi 501801161)的氨基酸序列相似度最高,表明该实验样品为目标酶。
综上,本实验首次实现了该嗜热古生菌来源的谷氨酰胺酶可溶性表达。
2.2 重组谷氨酰胺酶的功能预测与分析
通过NCBI数据库的Identify Conserved Domains功能对该嗜热玫瑰红球菌来源的谷氨酰胺酶氨基酸序列进行结构域功能预测,结果表明该酶具有以Glu44-Cys156-Lys115所组成的催化三联体,并且属于腈酶超家族,可能具有断裂碳氮键,参与有机氮化合物的还原和氮的生成反应的功能。腈酶超家族(也称为C—N水解酶超家族)包括腈或酰胺水解酶和酰胺缩合酶,基于结构的序列分析细分为13个分支[25-26]。该家族的底物均有C—N键存在,多为腈类或酰胺类物质,通过与腈类底物的氰基碳或酰胺类底物的平面羰基碳相互作用,水解生成相应的酸与游离氨。在swiss model在线网站中查询到了由AlphaFold所预测的该酶的三维蛋白质结构模型。通过Discovery Studio软件与腈酶超家族的几种不同酶类别的常见底物与该三维蛋白质结构模型进行分子对接,对接结果如表3所示,该酶与L-谷氨酰胺的对接得分为77.114 9,可能是其催化作用的反应底物,如图3-a、图3-b)所示,Cys156与L-谷氨酰胺的羧基氧原子通过氢键相互作用,Lys115与羰基氧原子相互作用,Glu44与—NH3基团相互作用,另外Phe119、Tyr157、Arg160、Ala181、Phe182残基也与L-谷氨酰胺分子通过不同的化学键相互作用。将目标酶与其他谷氨酰胺酶比较得出,两者序列与结构上没有显著相似性,多数谷氨酰胺酶属于丝氨酸依赖性β-内酰胺酶和青霉素结合蛋白的超家族,其结构域内的关键催化残基为Ser74、Cys77、Asn126、Cys268和Ser269等,这与腈酶超家族典型的Glu44-Cys156-Lys115催化三联体不同。而嗜热玫瑰红球菌来源的谷氨酰胺酶与同家族内的Nit1/Nit2序列相似度约为35%,其中Nit2可将天然底物α-酮戊二酸单酰胺(α-KGM)[27]水解为α-酮戊二酸(α-KG)与游离氨,谷氨酰胺与α-KGM的差异仅在前者α碳上的氨基与后者α碳的羰基不同。虽然Nit2不具有水解谷氨酰胺的功能,但考虑目标酶序列源自古生菌,在进化过程中处于比较早期的位置,所以该目标酶可能具有水解谷氨酰胺的功能。将该古生菌来源的重组酶与L-谷氨酰胺在50 ℃,pH值为8.0条件下反应1 h,通过液质检测反应产物所得结果如图3-c所示,证明了反应产物中同时含有谷氨酰胺与谷氨酸2种物质,该酶确实具有谷氨酰胺水解酶的功能,能够催化谷氨酰胺断裂碳氮键,产生谷氨酸以及游离氨。
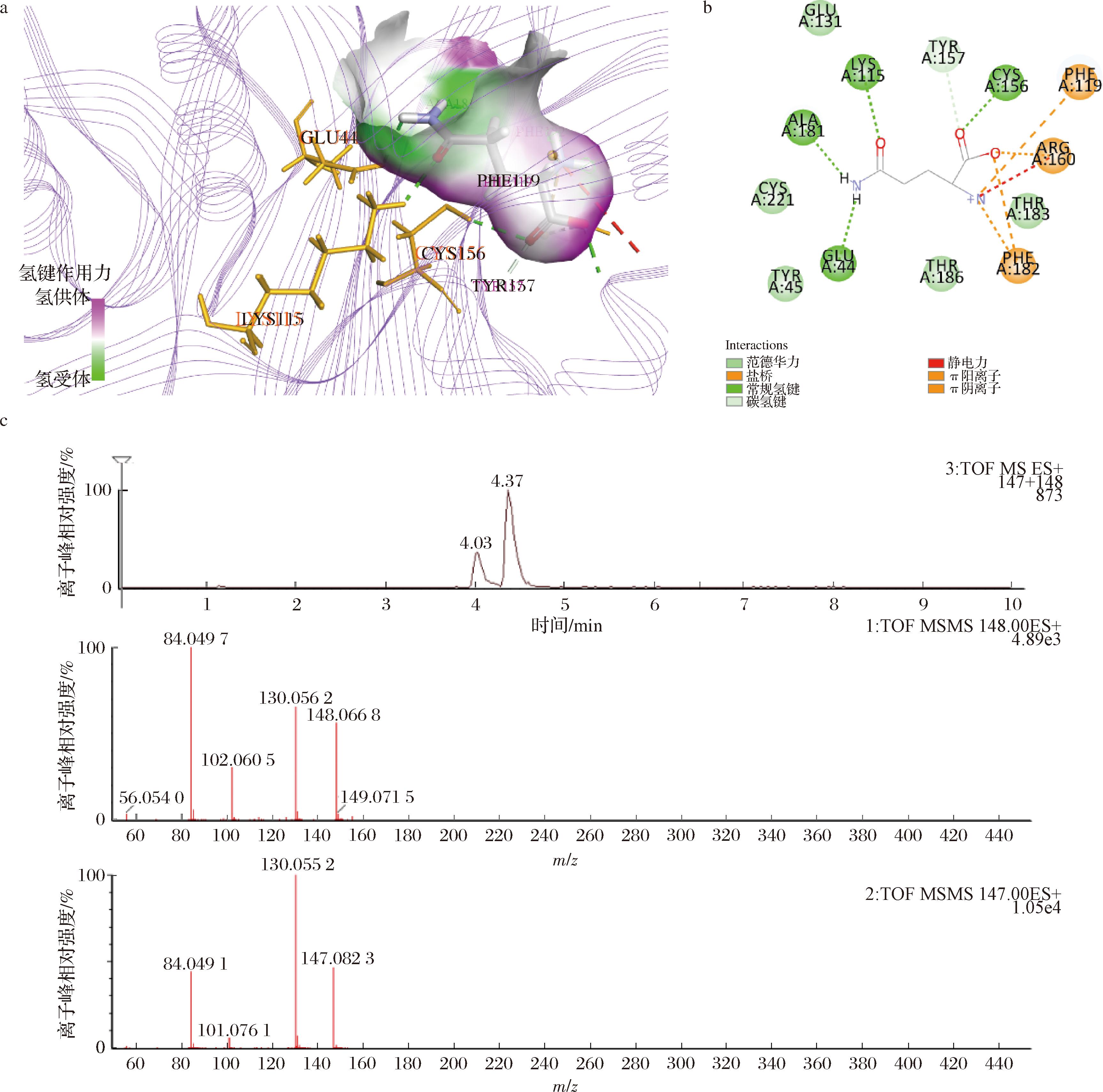
a-重组酶与L-谷氨酰胺分子对接分析(黄色表示Glu44-Cys156-Lys115催化三联体);b-重组酶与L-谷氨酰胺分子对接相互作用二维分析图;c-重组酶与L-谷氨酰胺反应混合物液相质谱分析
图3 重组谷氨酰胺酶功能预测与分析
Fig.3 Functional prediction and analysis of recombinant glutaminase
注:c图中从上至下分别为反应液的液相色谱图、色谱图第一个峰的质谱图、色谱图第二个峰的质谱图。
表3 Discovery Studio软件分子对接结果
Table 3 Molecular docking results from Discovery Studio software
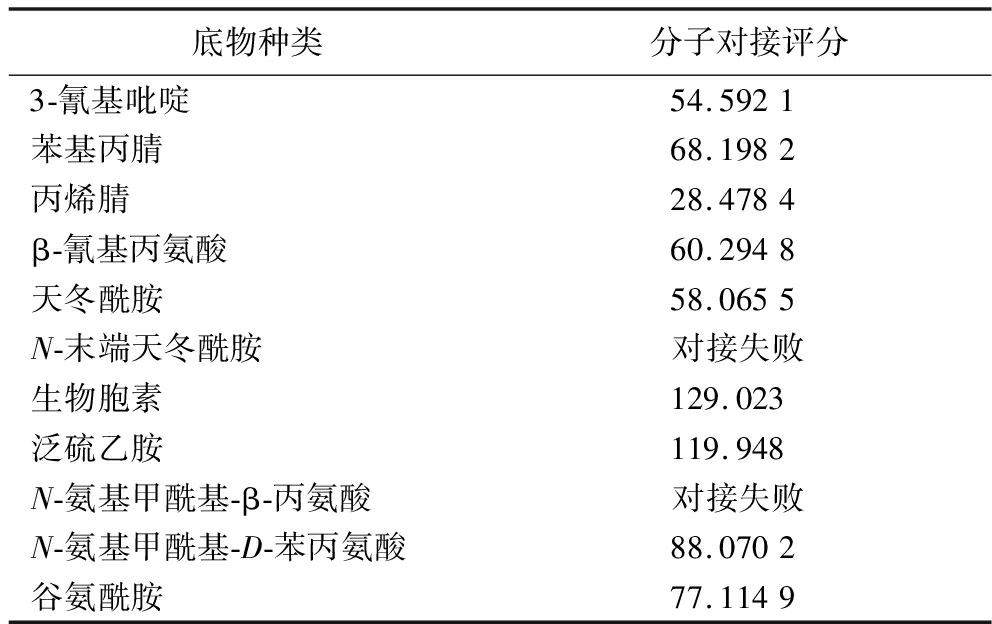
底物种类分子对接评分3-氰基吡啶54.5921苯基丙腈68.1982丙烯腈28.4784β-氰基丙氨酸60.2948天冬酰胺58.0655N-末端天冬酰胺对接失败生物胞素129.023泛硫乙胺119.948N-氨基甲酰基-β-丙氨酸对接失败N-氨基甲酰基-D-苯丙氨酸88.0702谷氨酰胺77.1149
2.3 重组谷氨酰胺酶的表达优化
为优化重组谷氨酰胺酶在重组菌株中的表达,设计4个因素和3个水平的正交试验,每个试验重复3次,SPSS 26.0进行方差分析结果如表4所示。F值的高低表明组间均值的可变性相比组内可变性的大小,即F值越大,因素影响越显著,根据结果得出,影响重组谷氨酰胺酶表达水平的显著程度依次为:IPTG诱导浓度>诱导前菌液浓度OD600值>诱导时间>诱导温度。同时根据SPSS分析得出:诱导前菌液浓度OD600值为0.8,IPTG诱导浓度0.05 mmol/L,诱导温度28 ℃,诱导时间14 h为最佳诱导条件。以此诱导条件进行重组谷氨酰胺酶的表达纯化,得到的重组酶质量由未优化表达条件前(诱导前菌液浓度OD600值为0.8,IPTG诱导浓度0.1 mmol/L,诱导温度28 ℃,诱导时间10 h)的每克菌体0.574 mg提升到了每克菌体1.628 mg,优化后重组酶活力为9.738 U/mL,相较于未优化表达条件前的重组酶活力提高了2.81倍。
表4 主体间效应检验结果表
Table 4 Results of the between-subjects effects test
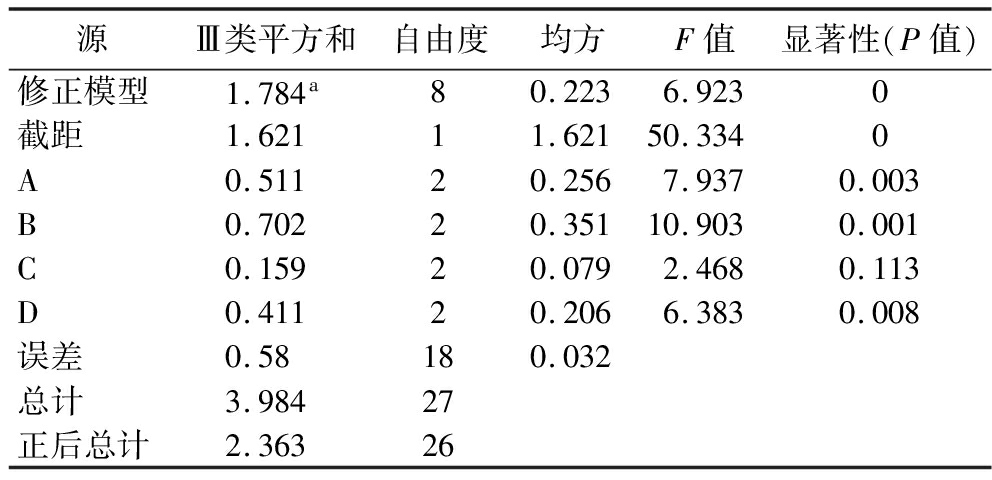
源Ⅲ类平方和自由度均方F值显著性(P值)修正模型1.784a80.2236.9230截距1.62111.62150.3340A0.51120.2567.9370.003B0.70220.35110.9030.001C0.15920.0792.4680.113D0.41120.2066.3830.008误差0.58180.032总计3.98427正后总计2.36326
注:a表示这是一个校正后的模型,其平方和、自由度和均方计算中已排除了未被因子占用的空列(误差列)的影响;F值,该检验的临界值,高F值表明组间均值的可变性比组内可变性较大;P值,反对无效假设的证据,P<0.05意味着有统计学意义。
2.4 重组谷氨酰胺酶的酶学性质
2.4.1 重组谷氨酰胺酶的二级结构和Tm
CD检测结果如图4-a所示,并通过CDNN软件进一步分析,表明重组谷氨酰胺酶由35.78%的α-螺旋,14.07%的β-折叠,16.06%的β-转角以及34.19%无规则卷曲组成。nano-DSC检测结果通过Nano Analyze软件分析,如图4-b所示,纯酶的变性温度(Tm)为94.38 ℃,焓变值(ΔH)为1 672 kJ/mol。重组谷氨酰胺酶的高Tm值表明该酶具有较为优异的热稳定性。
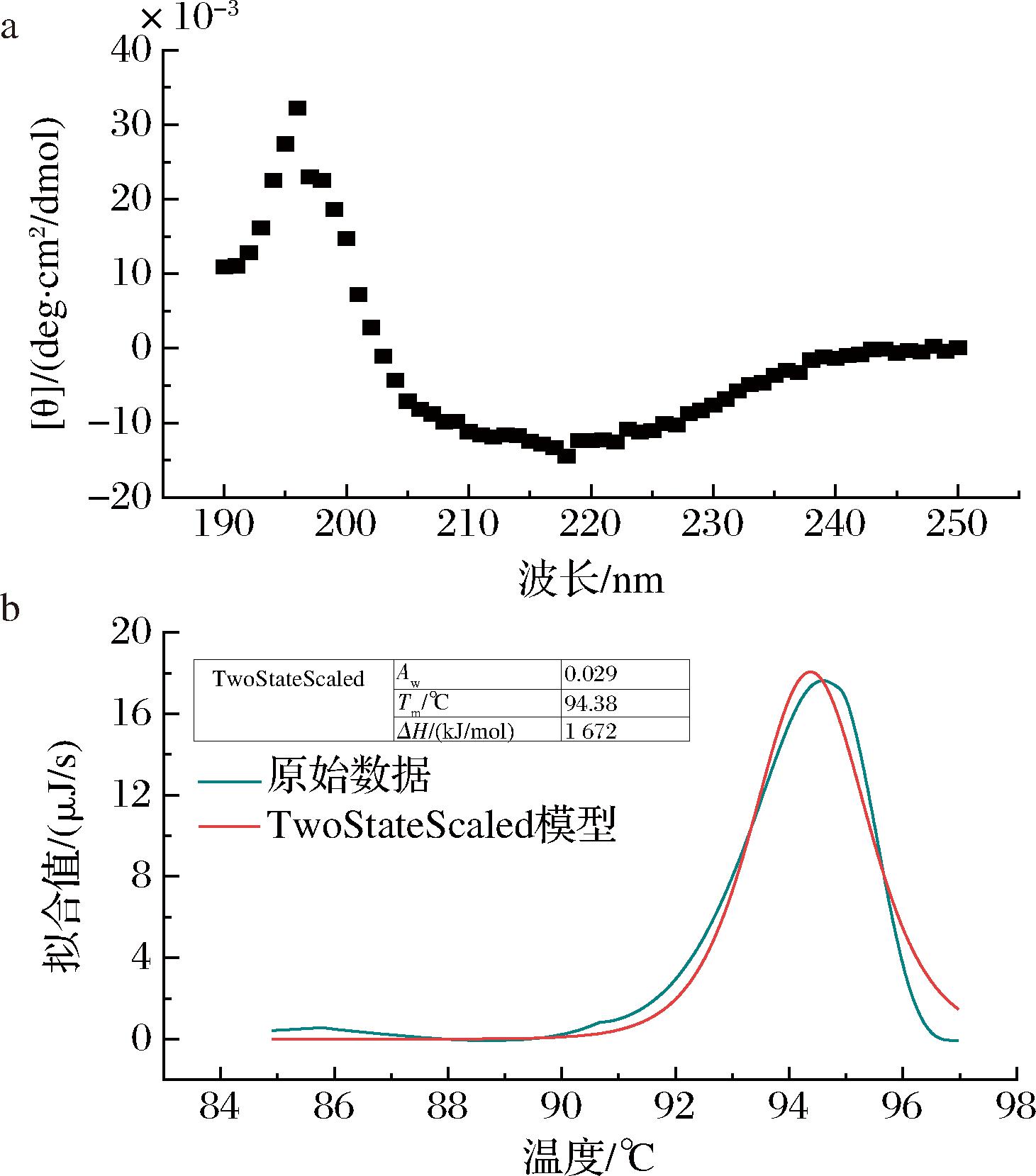
a-重组谷氨酰胺二级结构分析;b- 重组谷氨酰胺酶热变性温度(Tm)分析
图4 重组谷氨酰胺酶的二级结构分析与热变性温度(Tm)分析
Fig.4 Secondary structure analysis and thermal denaturation temperature (Tm) of recombinant glutaminase
2.4.2 重组谷氨酰胺酶的最适反应温度和pH
如图5-a所示,重组酶的最适反应温度为70 ℃,随着反应温度的上升,重组酶与底物L-谷氨酰胺的反应速度加快,4~70 ℃范围内重组酶结构稳定,且较高的温度更有利于嗜热古生菌来源的酶发挥其相应的催化作用。温度上升至70 ℃后,重组酶活力下降,100 ℃反应条件下仍具有部分活性。多数未经改造的属于丝氨酸依赖性β-内酰胺酶和青霉素结合蛋白超家族的谷氨酰胺酶的最适反应温度约为50 ℃或更低,目标重组谷氨酰胺酶的最适温度约为70 ℃,高于前者,更有利于在条件苛刻的工业生产中得到应用。如图5-b所示,重组酶的最适反应pH值为9.0,偏碱性的反应环境可能更有利于反应产物谷氨酸的溶解,从而促进了反应速度的提升,另一方面pH值的改变可能通过改变酶的带电状态来影响酶的活力。
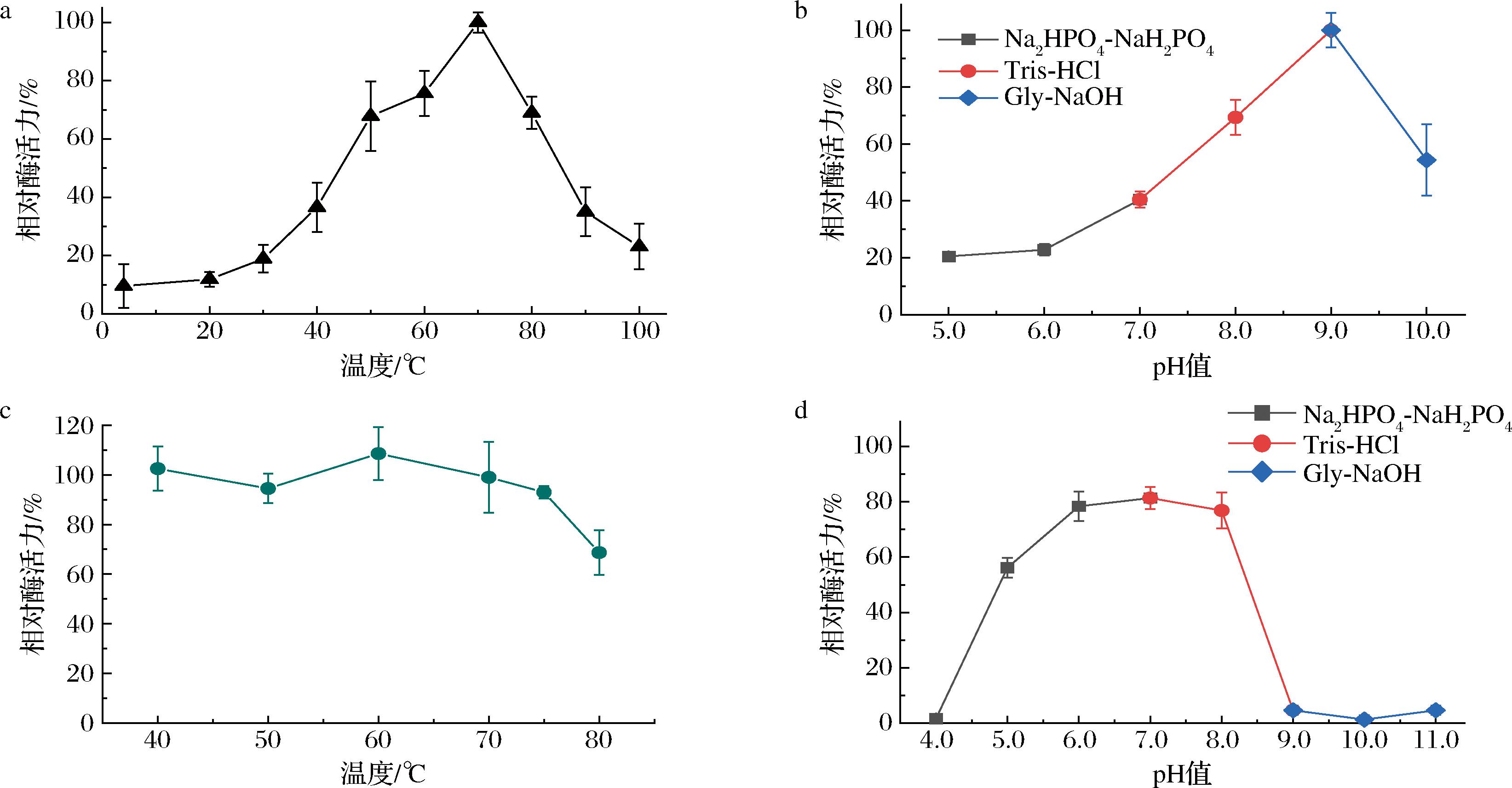
a-最适反应温度;b-最适反应pH值;c-温度稳定性;d-pH稳定性
图5 温度与pH对重组谷氨酰胺酶的影响
Fig.5 Effect of temperature and pH on recombinant glutaminase
2.4.3 重组谷氨酰胺酶的温度和pH稳定性
如图5-c所示,重组酶在4~80 ℃孵育2 h,仍保留60%以上的酶活力,且75 ℃以下酶活力受到温度的影响是较为微小的,这说明重组谷氨酰胺酶的温度稳定性正如预期的良好。如图5-d所示,重组酶在pH值为6.0~8.0的中性缓冲液中孵育2 h酶活力下降至80%,但较为稳定。而在pH值4.0~6.0的酸性缓冲液中孵育2 h后,酶活力受到较大影响,随着pH降低,酶活力逐步下降,在pH值上升至9.0后,酶活力迅速下降,并几乎失去酶活力。这一结果表明该重组谷氨酰胺酶适宜在中性条件下保存,应避免在偏碱性环境条件下保存。
2.4.4 重组谷氨酰胺酶的动力学参数及半衰期
以L-谷氨酰胺和D-谷氨酰胺为底物测定重组酶的反应动力学特性,如图6-a、图6-b所示,当以不同浓度L-谷氨酰胺为底物时,测得其米氏方程为Y=0.198 12X/(26.985 71+X),米氏常数Km为26.986 mmol/L,Vmax为0.198 mmol/(L·min),并计算得kcat/Km为0.397 L/(mmol·min);以不同浓度D-谷氨酰胺为底物时,测得其米氏方程为Y=0.188 27X/(50.837 49+X),米氏常数Km为50.837 mmol/L,Vmax为0.188 mmol/(L·min),kcat/Km为0.200 L/(mmol·min),说明该嗜热古生菌来源的重组谷氨酰胺酶对L-谷氨酰胺的亲和力更好。属于丝氨酸依赖性β-内酰胺酶和青霉素结合蛋白超家族的谷氨酰胺酶往往仅与L-谷氨酰胺发生反应,几乎不与D-谷氨酰胺发生反应,表现出低底物亲和性但高底物选择性[28],而嗜热古生菌来源的重组酶则对2种手性的谷氨酰胺均具有催化活性。在40~70 ℃范围内,该重组谷氨酰胺酶表现出了良好的耐热稳定性,如图6-c所示随着时间的延长,残留酶活力缓慢下降,其半衰期>12 h。75 ℃下,该重组酶活力的半衰期约为6 h,当温度上升至80 ℃,其蛋白结构可能受高温影响,部分发生变性,孵育过程中出现了少量白色沉淀,酶活力随时间延长下降较快,半衰期约为3 h。
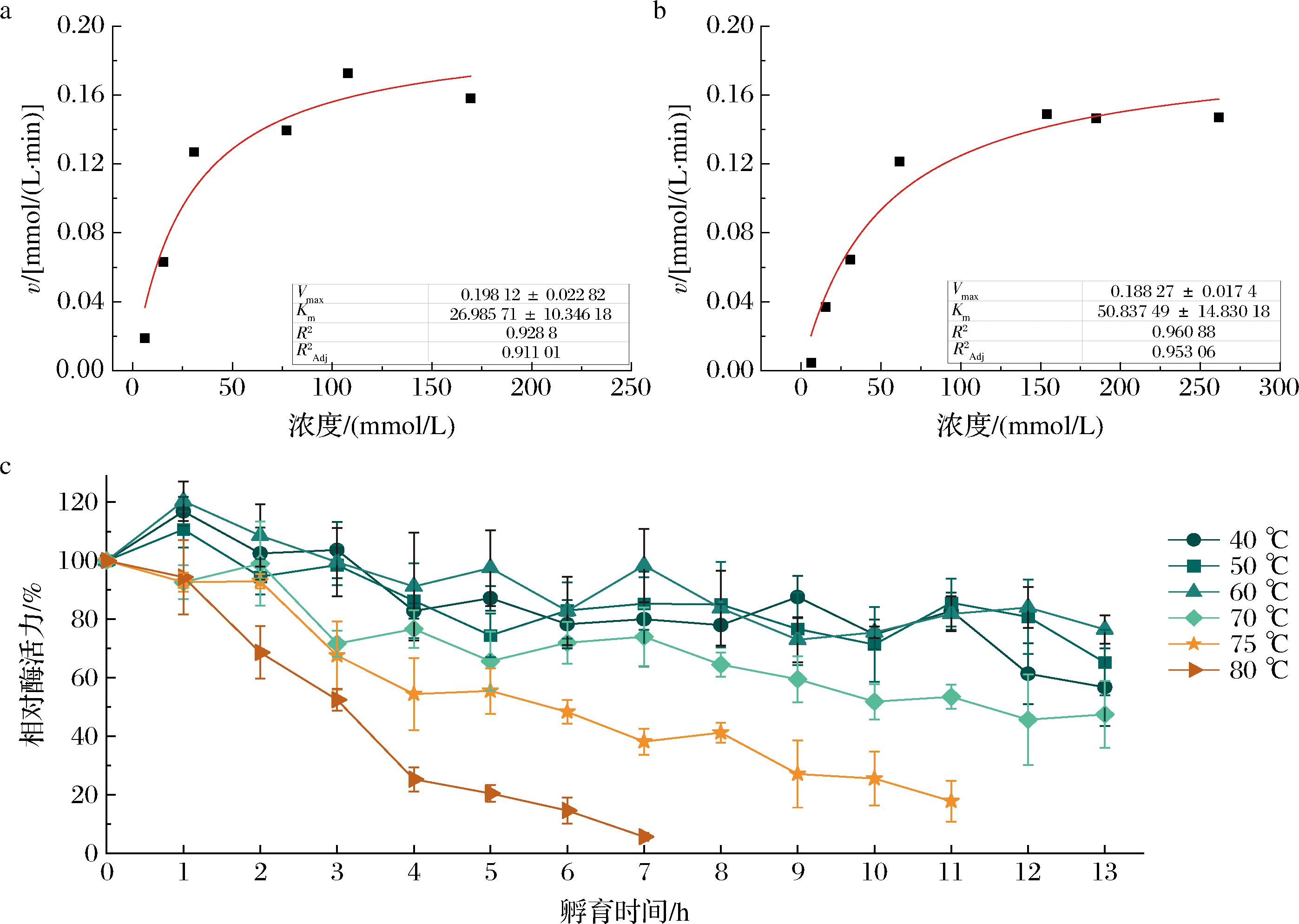
a-重组谷氨酰胺酶以L-谷氨酰胺为底物时的动力学参数分析;b-重组谷氨酰胺酶以D-谷氨酰胺为底物时的动力学参数分析;c-重组谷氨酰胺酶的半衰期分析
图6 重组谷氨酰胺酶的动力学参数与半衰期
Fig.6 Kinetic parameters and half-life of recombinant glutaminase
2.4.5 金属离子对重组谷氨酰胺酶的影响
金属离子多可以通过与底物结合、与酶结合、价态变化以携带电子参与反应以及静电作用等方式影响酶的活力。如图7所示,K+、Ca2+、Mg2+、Fe3+对酶活力影响不大,Mn2+、Fe2+、Zn2+、Cu2+、Co2+、Ni2+对酶活力有不同程度的抑制作用,其中Cu2+的抑制作用最显著,浓度为1 mmol/L的Cu2+即可使重组L-谷氨酰胺酶完全失去酶活力,Zn2+对酶活力的抑制作用次之,浓度为1 mmol/L的Zn2+使得酶活力下降至20%,Mn2+、Fe2+、Co2+、Ni2+ 4种金属离子的抑制影响随浓度的升高而逐步增大。
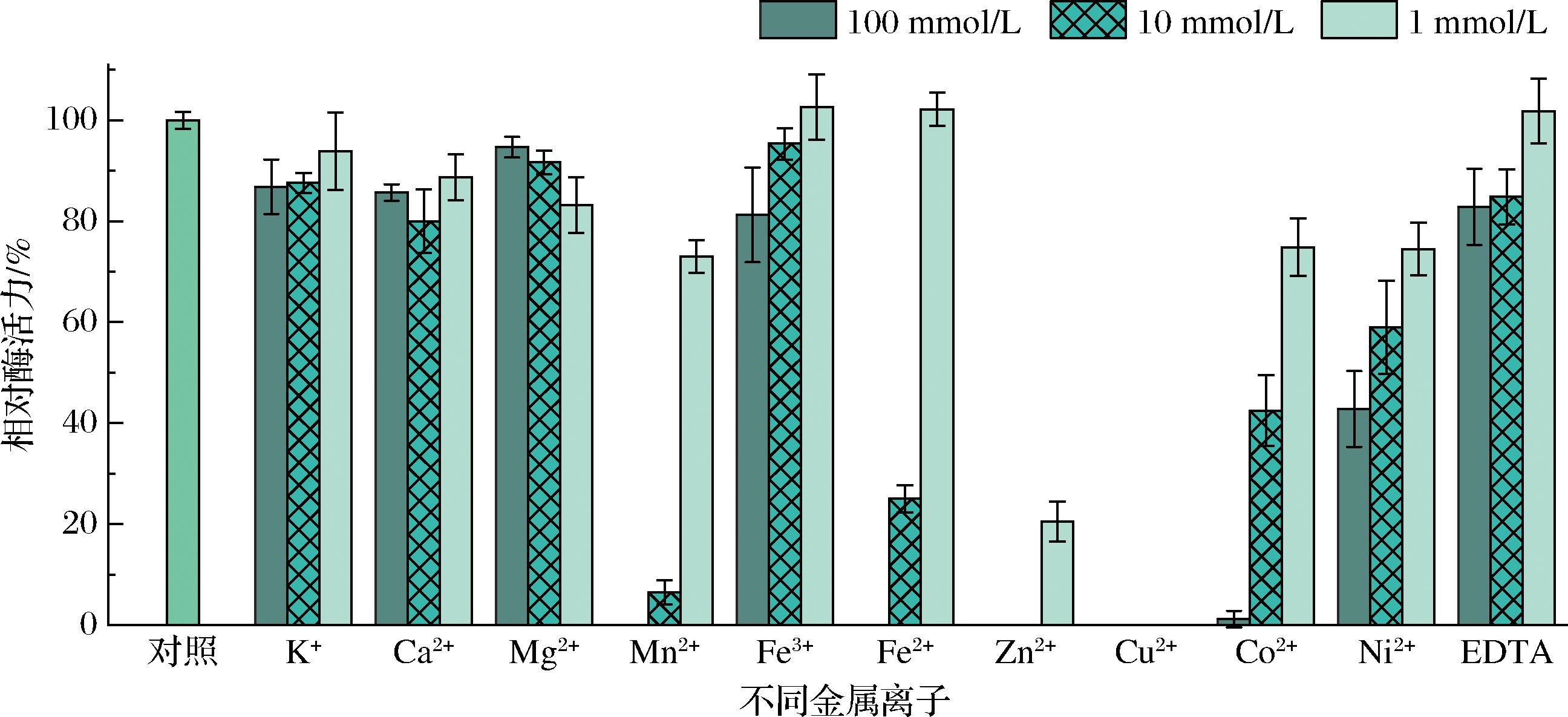
图7 金属离子对重组谷氨酰胺酶的影响
Fig.7 Effect of metal ions on recombinant glutaminase
3 结论与讨论
本研究将筛选自嗜热玫瑰红球菌(T.roseum DSM 5159)的属于腈酶超家族的谷氨酰胺酶基因进行了异源重组表达,发现了该酶具有水解谷氨酰胺产生谷氨酸与游离氨的功能,初步探究了该酶的蛋白质结构,并对纯化后的重组酶进行了酶学性质表征。研究获得的重组谷氨酰胺酶具有良好的热稳定性,最适反应温度为70 ℃,Tm为94.38 ℃,ΔH为1 672 kJ/mol,并且在70 ℃以下,酶活力半衰期>12 h,恒温孵育2 h后酶活力基本保持稳定不变,这说明了该嗜热古生菌来源的重组谷氨酰胺酶在工业应用当中蛋白结构稳定不易变性,酶活力稳定不易失活,在运输贮存方面的成本也较低。重组谷氨酰胺酶在pH值为6.0~8.0的中性环境下,酶活力也较为稳定,随时间延长酶活力下降不明显,但在偏酸性和偏碱性条件下重组酶的耐受性能会大幅下降,在生产应用中,该酶的酸碱稳定性还有待改善。部分金属离子对重组谷氨酰胺酶活力影响较小或几乎没有,但Mn2+、Fe2+、Zn2+、Cu2+、Co2+对酶活力的抑制作用较大,本研究未发现金属离子对重组酶具有激活作用,这与其他腈酶超家族成员酶类相似。来自极端环境的微生物往往是能够适应条件严苛的生产环境的工业酶类的资源摇篮,但相较于已投入商用的酶类,异源表达的重组酶往往具有多方面的劣势,比如低效的酶活力、较差的表达量或可溶性、不良的稳定性等,后续研究将针对各项缺点通过理性设计改造该重组谷氨酰胺酶,以期望获得多方面性能更佳的谷氨酰胺酶,使其能够在多种环境工业中得到广泛应用。另外,通过对该重组谷氨酰胺酶的氨基酸序列及结构初步研究后发现其与多种同家族酶类具有较高的相似性,考虑其来源于古生菌,该酶可能处于进化分支的较前端,通过符合进化趋势的合理改造或许能够挖掘获得该酶的其他酶学功能。
[1] BARZKAR N, SOHAIL M, TAMADONI JAHROMI S, et al.Marine microbial L-glutaminase:From pharmaceutical to food industry[J].Applied Microbiology and Biotechnology, 2021, 105(11):4453-4466.
[2] PARMAR J, TRIPATHI S, KUMAR A.Immunogenicity assessment of antileukemic agent glutaminase from Escherichia coli, Pseudomonas sp., and Bacillus sp[J].Biomedical and Biotechnology Research Journal (BBRJ), 2022, 6(1):138-144.
[3] CRUZAT V, MACEDO ROGERO M, NOEL KEANE K, et al.Glutamine:Metabolism and immune function, supplementation and clinical translation[J].Nutrients, 2018, 10(11):1564.
[4] VO T D, SULAIMAN C, TAFAZOLI S, et al.Safety assessment of glutaminase from Aspergillus niger[J].Food Science &Nutrition, 2020, 8(3):1433-1450.
[5] WAKAYAMA M, YAMAGATA T, KAMEMURA A, et al.Characterization of salt-tolerant glutaminase from Stenotrophomonas maltophilia NYW-81 and its application in Japanese soy sauce fermentation[J].Journal of Industrial Microbiology and Biotechnology, 2005, 32(9):383-390.
[6] YUZUKI M, MATSUSHIMA K, KOYAMA Y.Expression of key hydrolases for soy sauce fermentation in Zygosaccharomyces rouxii[J].Journal of Bioscience and Bioengineering, 2015, 119(1):92-94.
[7] ARYUMAN P, LERTSIRI S, VISESSANGUAN W, et al.Glutaminase-producing Meyerozyma (Pichia) guilliermondii isolated from Thai soy sauce fermentation[J].International Journal of Food Microbiology, 2015, 192:7-12.
[8] MAHAREM T M, EMAM M A, SAID Y A.Purification and characterization of l-glutaminase enzyme from camel liver:Enzymatic anticancer property[J].International Journal of Biological Macromolecules, 2020, 150:1213-1222.
[9] KODAMA M, OSHIKAWA K, SHIMIZU H, et al.A shift in glutamine nitrogen metabolism contributes to the malignant progression of cancer[J].Nature Communications, 2020, 11:1320.
[10] WISE D R, THOMPSON C B.Glutamine addiction:A new therapeutic target in cancer[J].Trends in Biochemical Sciences, 2010, 35(8):427-433.
[11] ANASTASIOU D, CANTLEY L C.Breathless cancer cells get fat on glutamine[J].Cell Research, 2012, 22(3):443-446.
[12] ORABI H, EL-FAKHARANY E, ABDELKHALEK E, et al.Production, optimization, purification, characterization, and anti-cancer application of extracellular L-glutaminase produced from the marine bacterial isolate[J].Preparative Biochemistry &Biotechnology, 2020, 50(4):408-418.
[13] UNISSA R, SUDHAKAR M, REDDY A S K, et al.ChemInform abstract:A review on biochemical and therapeutic aspects of glutaminase[J].ChemInform, 2015, 46(21):201521304.
[14] AMOBONYE A, SINGH S, PILLAI S.Recent advances in microbial glutaminase production and applications-a concise review[J].Critical Reviews in Biotechnology, 2019, 39(7):944-963.
[15] HASANUZZAMAN M, NAHAR K, ANEE T I, et al.Glutathione in plants:Biosynthesis and physiological role in environmental stress tolerance[J].Physiology and Molecular Biology of Plants:An International Journal of Functional Plant Biology, 2017, 23(2):249-268.
[16] SAMOYLOVA Y V, SOROKINA K N, ROMANENKO M V, et al.Cloning, expression and characterization of the esterase estUT1 from Ureibacillus thermosphaericus which belongs to a new lipase family XVIII[J].Extremophiles:Life Under Extreme Conditions, 2018, 22(2):271-285.
[17] 张美丹, 饶志明, 张显, 等.谷氨酰胺酶基因的克隆、表达和酶学性质研究[J].食品与生物技术学报, 2019, 38(10):60-66.ZHANG M D, RAO Z M, ZHANG X, et al.Cloning, expression and characterization of Bacillus subtilis glutaminase[J].Journal of Food Science and Biotechnology, 2019, 38(10):60-66.
[18] BINOD P, SINDHU R, MADHAVAN A, et al.Recent developments in l-glutaminase production and applications- An overview[J].Bioresource Technology, 2017, 245(Pt B):1766-1774.
[19] JACKSON T J, RAMALEY R F, MEINSCHEIN W G.Thermomicrobium, a new genus of extremely thermophilic bacteria[J].International Journal of Systematic Bacteriology, 1973, 23(1):28-36.
[20] XIN Y, HAO M Y, FAN G M, et al.Soluble expression of Thermomicrobium roseum sarcosine oxidase and characterization of N-demethylation activity[J].Molecular Catalysis, 2019, 464:48-56.
[21] LOUIS J R.Enzyme Assays:High-throughput Screening, Genetic Selection and Fingerprinting[M].Weinheim:Wiley‐VCH Verlag GmbH &Co.KGaA, 2005.
[22] PEREZ-IRATXETA C, ANDRADE-NAVARRO M A.K2D2:Estimation of protein secondary structure from circular dichroism spectra[J].BMC Structural Biology, 2008, 8:25.
[23] 仝艳军.肌氨酸氧化酶高效表达、亲和纯化及酶稳定性研究[D].无锡:江南大学, 2015.TONG Y J.Efficient expression, affinity purification and stability of sarcosine oxidase[D].Wuxi:Jiangnan University, 2015.
[24] GROTE A, HILLER K, SCHEER M, et al.JCat:A novel tool to adapt Codon usage of a target gene to its potential expression host[J].Nucleic Acids Research, 2005, 33(Web Server issue):W526-W531.
[25] BRENNER C.Catalysis in the nitrilase superfamily[J].Current Opinion in Structural Biology, 2002, 12(6):775-782.
[26] SILVA TEIXEIRA C S, SOUSA S F, CERQUEIRA N M F S A.An unsual cys-glu-Lys catalytic triad is responsible for the catalytic mechanism of the nitrilase superfamily:A QM/MM study on Nit2[J].ChemPhysChem, 2021, 22(8):796-804.
[27] LIU H J, GAO Y X, ZHANG M Y, et al.Structures of enzyme-intermediate complexes of yeast Nit2:Insights into its catalytic mechanism and different substrate specificity compared with mammalian Nit2[J].Acta Crystallographica.Section D, Biological Crystallography, 2013, 69(Pt 8):1470-1481.
[28] BROWN G, SINGER A, PROUDFOOT M, et al.Functional and structural characterization of four glutaminases from Escherichia coli and Bacillus subtilis[J].Biochemistry, 2008, 47(21):5724-5735.