糖尿病是一种由胰岛素代谢异常或分泌缺陷引起的以高血糖为表征的慢性代谢疾病[1],长期高血糖会导致机体功能障碍以及多个器官和组织衰竭,并伴有代谢性并发症,如神经病变、视网膜疾病、肾脏受损和心血管疾病等[2]。α-葡萄糖苷酶(EC 3.2.1.20)是一种膜结合酶,存在于小肠刷状边缘,可水解末端非还原性α-(1,4)连接的α-葡萄糖残基以释放游离葡萄糖分子,从而导致血糖升高[3]。目前,一些α-葡萄糖苷酶抑制剂(如阿卡波糖)已被用作抗糖尿病药物[4]。然而,临床证据表明,阿卡波糖经常引起胃肠道副作用,如腹胀、腹泻和腹痛[5]。因此,挖掘天然无毒副作用的α-葡萄糖苷酶抑制剂以预防或控制糖尿病非常重要。
生物活性肽是从蛋白质中分离出来的特定氨基酸序列,具有增强免疫能力、降低血脂、降血压、抗氧化、抗菌、抗肿瘤等多种生理功能[6]。生物活性肽的制备方法包括酶解法、微生物发酵法和化学合成法[7]。其中,酶解法被广泛运用,通过不同蛋白酶酶切,可获得不同生理功能的生物活性肽[8]。然而,酶解法仍存在耗时长、成本高、效率低等问题。随着生物信息学的发展以及蛋白数据库、多肽数据库、生物活性预测工具的出现,虚拟筛选技术已成为筛选生物活性肽的有效手段[9]。与传统的酶解、分离纯化方法相比,计算机模拟酶解和分子对接技术极大地缩短了酶解时间,提高了筛选的效率。
核桃(Juglans regia L.)是一种食药同源资源,具有很高的营养价值和生理功能。核桃中含有20%~25%的蛋白质,含有丰富的必需氨基酸,是优质的植物蛋白资源[10]。研究表明,核桃粕蛋白可以释放多种生物活性肽,包括抗氧化肽[11]、降血压肽[12]、降血脂肽[13]等。LUAN等[14]从核桃蛋白酶解物(3~10 kDa)中分离纯化了一种新的五肽LPLLR,且LPLLR对α-葡萄糖苷酶和α-淀粉酶均有抑制作用;李丽琪等[15]的研究表明,核桃多肽、分离纯化后的HT-1、HT-2都具有较好的降糖活性;李丽[16]从核桃多肽中分离纯化出JRP-1和JRP-2,通过酿酒酵母模型和2型糖尿病动物模型实验发现,核桃肽具有一定的降糖功效,并且核桃肽的降糖功效优于核桃蛋白。然而,对于核桃蛋白中的α-葡萄糖苷酶抑制肽的挖掘仍不够全面和深入,因此,对核桃蛋白源资源的开发利用,深入挖掘其中的α-葡萄糖苷酶抑制肽仍具有重要意义。
本研究基于数据库获取的核桃蛋白序列,首先对其进行虚拟酶解,并利用在线工具对肽段的生物活性、毒性和溶解性进行预测,然后采用分子对接技术筛选潜在α-葡萄糖苷酶抑制肽,并通过分子动力学模拟验证肽与α-葡萄糖苷酶结合的稳定性。本研究为快速筛选出潜在的α-葡萄糖苷酶抑制肽提供了新的途径。
1 材料与方法
1.1 在线工具
在线数据库和平台工具信息如表1所示。
表1 在线数据库和平台工具信息
Table 1 Information on online databases and platform tools

名称链接在本研究中的作用ToxinPredhttp://crdd.osdd.net/raghava/toxinpred/毒性预测Uniprothttps://www.uniprot.org/蛋白序列查找BIOPEPhttp://bis.zju.edu.cn/biopepdbr/index.php新颖性检查ProtParamhttps://web.expasy.org/protparam/氨基酸组成PeptideMasshttps://web.expasy.org/peptide_mass/酶切PeptideRankerhttp://distilldeep.ucd.ie/PeptideRanker/活性预测Innovagenhttp://www.innovagen.com/溶解性预测RCSBPDBhttps://www.rcsb.org/蛋白晶体获取SwissADMEhttp://www.swissadme.ch/index.php获得SMILES号A11erTophttps://www.ddg-pharmfac.net/allertop/index.html致敏原预测
1.2 实验方法
1.2.1 核桃蛋白的选择及氨基酸序列获取
通过Uniprot(https://www.uniprot.org/)查找核桃蛋白,选取8条核桃蛋白(表2),将8条核桃蛋白序列复制到BIOPEP中利用“潜在生物活性图谱”数据库[17],查找蛋白质序列中生物活性片段和位置。
表2 核桃蛋白链
Table 2 Walnut protein chains

UniprotID蛋白名称氨基酸数相对分子质量/DaP931982Sseedstoragealbuminprotein13916373A0A2I4E5L6VicilinJugr6.010150257400A0A1L6K37111Sglobulin51058056Q2TPW511SglobulinseedstorageproteinJugr450758144Q9SEW4VicilinJugr2.010159369990Q8ITIForkheadboxproteinG131835384Q9XHP011Sglobulinseedstorageprotein245951830Q26231Periodcircadianprotein10911417
1.2.2 核桃蛋白计算机模拟酶解
采用ProtParam数据库对核桃蛋白的氨基酸组成及占比进行分析对比,根据氨基酸组成初步确定其α-葡萄糖苷酶抑制潜力。利用PeptideMass工具,选择胰蛋白酶、胃蛋白酶、糜蛋白酶(C项至F/Y/W,不在P之前)、蛋白酶K四种酶进行酶解。并计算理论水解度(theoretical degree of hydrolysis, TDH),如公式(1)所示[18]:
(1)
式中:TDH,%;D为蛋白质中肽键总数;d为被裂解的肽键数。
1.2.3 核桃蛋白α-葡萄糖苷酶抑制肽筛选
将计算机模拟酶解所得活性肽通过PeptideRanker在线工具进行活性预测,选择活性评分大于0.5的多肽。将根据评分结果筛选得到的多肽与BIOPEP中报道过的α-葡萄糖苷酶抑制肽进行比较,选取未进行报道过的多肽进行研究。利用Innovagen数据库对筛选的多肽进行溶解性预测,通过ToxinPred软件预测其毒性,采用A11erTop工具进行致敏原预测。
1.3 分子对接
利用ChemDraw 2D软件画出多肽结构,从PDB蛋白质数据库(www.rcsb.org)获取α-葡萄糖苷酶天然晶体结构(PDB代码:3A4A)用ChemDraw 3D构建3D结构,使用AutoDock vina软件进行分子对接,以结合能作为评价指标对α-葡萄糖苷酶抑制肽再次进行筛选。将分子对接的图利用PyMol软件进行可视化处理,找到核桃蛋白α-葡萄糖苷酶抑制肽的作用模式。
1.4 分子动力学模拟
使用Amber24进行分子动力学模拟,力场选择ff14SB分子力场,采用TIP3P水模型,将蛋白与小分子装在正方体水盒子中,模拟中静电相互作用和范德华相互作用的截断均设为1.0 nm,时间步长设为2 fs,静电相互作用长程修正采用PME方法。温度为300 K,压力1 bar。共构建1个体系,蛋白和配体复合物。建立好的体系首先进行能量最小化,而后分别进行NVP平衡动力学200 ps与NPT平衡动力学100 ps,选择V-rescale的方法来热浴耦合系统温度,parrinello-rahman控制压力,分子动力学采样进行100 ns。通过Amber程序自带模块,Cpptraj及Python进行均方根偏差(root mean square deviation, RMSD)、均方根波动(root mean square fluctuation, RMSF)、回旋半径、氢键数目等相关计算。
1.5 数据统计与分析
利用SPSS 16.0进行数据分析,采用GraphPad Prism 8.0.2进行作图。
2 结果与分析
2.1 核桃蛋白序列生物活性
对BIOPEP数据库中的信息进行收集(截至2024年6月19日,在BIOPEP中收录了4 957个具有生物活性的肽段),在核桃蛋白中发现了具有27种已知生物活性的片段(图1),其中血管紧张素转化酶抑制剂、抗氧化剂、二肽基肽酶Ⅳ抑制剂、二肽基肽酶Ⅲ抑制剂的肽段比例较高。由图1可知,核桃蛋白具有多种生物活性片段,因此,挖掘核桃蛋白中的α-葡萄糖苷酶抑制活性肽具有重要意义。
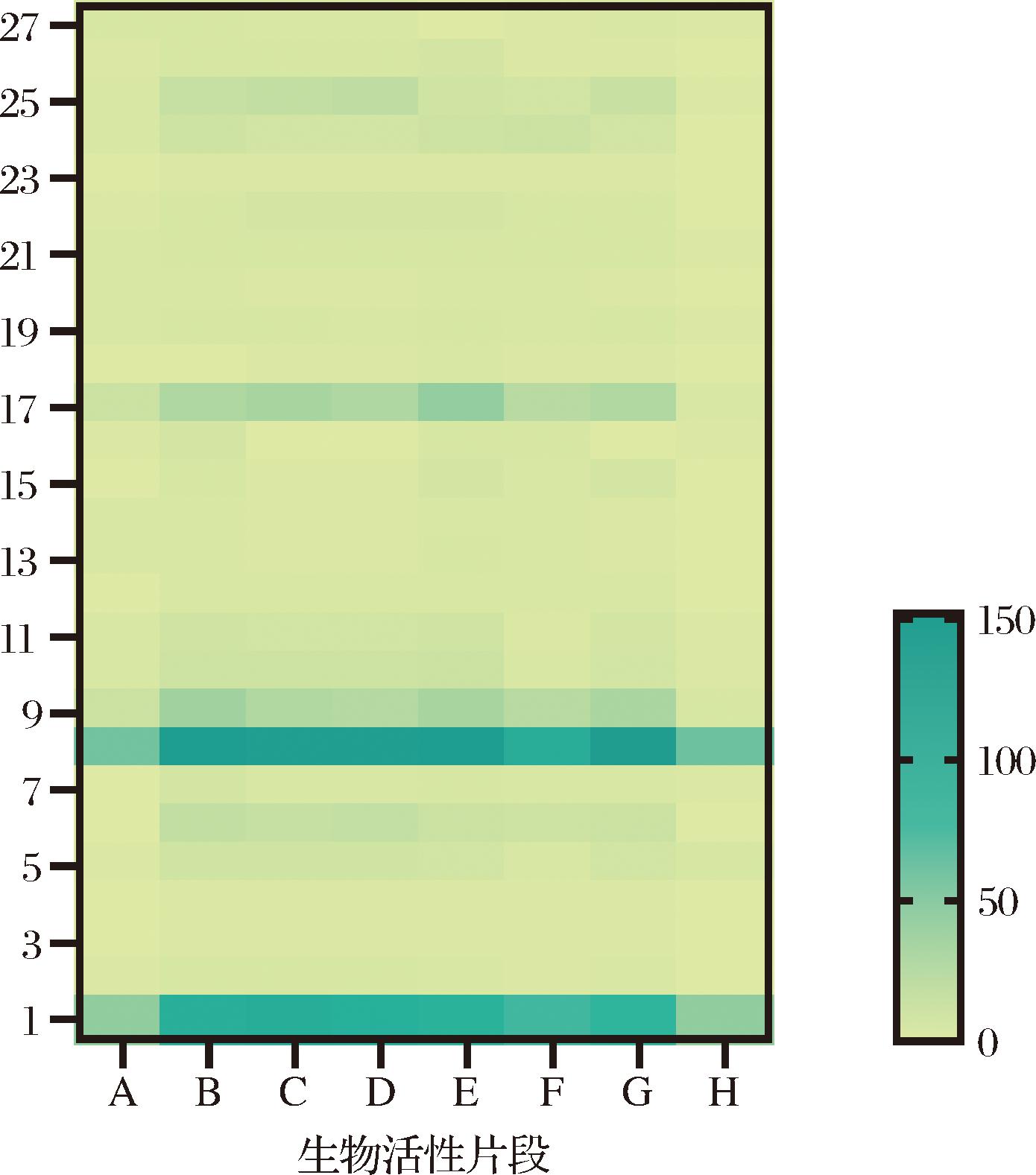
A:P93198、B:AOA214E5L6、C:AOA1L6K371、D:Q2TPW5、E:Q9SEW4、F:Q8IT15、G:Q9XHPO H:Q26231代表8条核桃蛋白的编号;1.血管紧张素转化酶抑制剂;2.CaMPDE 抑制剂;3.PAM 抑制剂;4.xaa-pro抑制剂;5.α-葡萄糖苷酶抑制剂;6.刺激;7.调节剂;8.二肽基肽酶IV抑制剂;9.二肽基肽酶III抑制剂;10.谷氨酸羧肽酶II抑制剂;11.谷氨酸羧肽酶抑制剂;12.激活泛素介导的蛋白水解作用;13.抗病毒;14.抗菌;15.抗血栓;16.抗炎;17.抗氧化剂;18.抗抑郁;19.亮氨酰转移酶抑制剂;20.抗溶血;21.乳糖素抑制剂;22.三肽基肽酶II抑制剂;23.神经溶菌酶抑制剂;24.神经肽;25.肾素抑制剂;26.微管蛋白-酪氨酸连接酶抑制剂;27.细胞溶液丙氨酰氨基肽酶抑制剂。
图1 八条核桃蛋白片段生物活性
Fig.1 Eight biologically active fragments of walnut proteins
2.2 核桃蛋白虚拟酶解
利用Uniprot数据库查找到8条已知核桃蛋白序列,8条核桃序列氨基酸数量分别为139、502、510、507、593、318、459、109个。进一步对核桃蛋白的氨基酸组成进行统计分析,如表3所示,核桃蛋白中含有20种氨基酸,其中,必需氨基酸占比达到30.44%,非极性氨基酸占比为41.14%,极性带正电荷氨基酸(赖氨酸、精氨酸、组氨酸)占比为14.22%,极性带负电荷氨基酸占比为12.15%。
表3 核桃蛋白氨基酸组成
Table 3 Amino acid composition of walnut protein
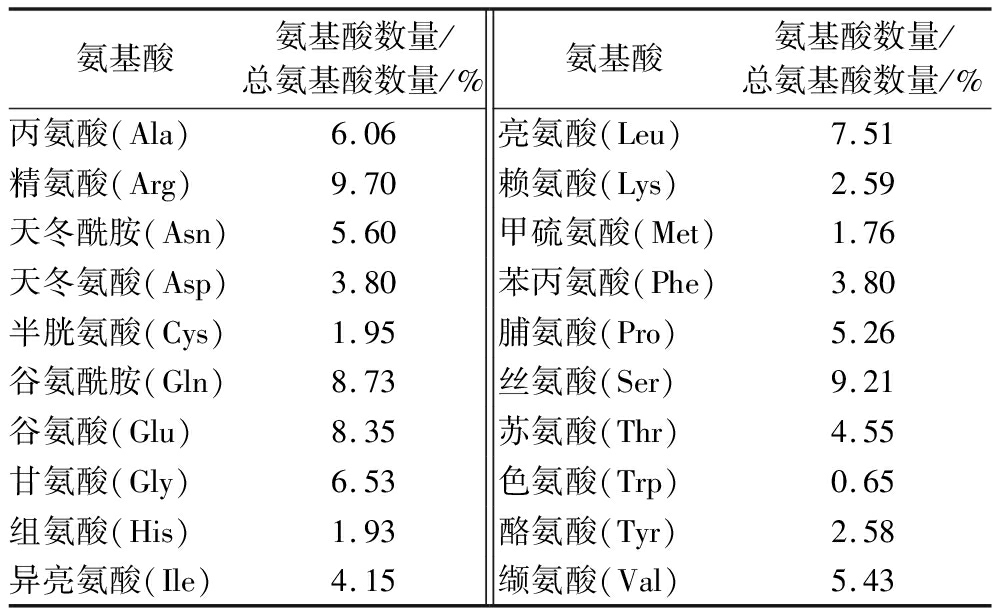
氨基酸氨基酸数量/总氨基酸数量/%氨基酸氨基酸数量/总氨基酸数量/%丙氨酸(Ala)6.06亮氨酸(Leu)7.51精氨酸(Arg)9.70赖氨酸(Lys)2.59天冬酰胺(Asn)5.60甲硫氨酸(Met)1.76天冬氨酸(Asp)3.80苯丙氨酸(Phe)3.80半胱氨酸(Cys)1.95脯氨酸(Pro)5.26谷氨酰胺(Gln)8.73丝氨酸(Ser)9.21谷氨酸(Glu)8.35苏氨酸(Thr)4.55甘氨酸(Gly)6.53色氨酸(Trp)0.65组氨酸(His)1.93酪氨酸(Tyr)2.58异亮氨酸(Ile)4.15缬氨酸(Val)5.43
研究表明肽段中疏水氨基酸残基含量越高,α-葡萄糖苷酶的抑制活性越好。如VILCACUNDO等[19]在体外对藜麦蛋白进行模拟胃肠道消化,分离到3个肽段,发现其中含有3个疏水残基的肽段IQEGGLT对α-葡萄糖苷酶表现出较强的抑制活性;WEI等[20]从芍药“凤丹”种子蛋白中分离出3种新的α-葡萄糖苷酶抑制肽:YFFM、FFFM和YYFM,这些肽中含较高比例的疏水性氨基酸。此外,研究还发现C端精氨酸可以提高肽-酶结合的稳定性,保证肽与酶关键活性氨基酸残基的稳定结合,对α-葡萄糖苷酶抑制活性有积极作用。如LIU等[21]从小麦胚芽中筛选到的肽段LDLQR、AGGFR、LDNFR;HU等[22]从螺旋藻中得到肽段LRSELAAWSR;LUAN等[14]从核桃中得到的衍生肽LPLLR都显示出较好的α-葡萄糖苷酶抑制活性,构效关系分析表明,这些肽段在C-末端都具有精氨酸。而核桃蛋白序列中的疏水性氨基酸占比极高,达到41.14%,此外,精氨酸相对含量达到9.70%,这表明核桃蛋白中含有丰富的α-葡萄糖苷抑制肽,是释放α-葡萄糖苷抑制活性肽的优质来源。
为了进一步挖掘核桃蛋白中的生物活性肽,采用胰蛋白酶、胃蛋白酶(pH>2)、糜蛋白酶、蛋白酶K分别对8条核桃蛋白序列进行虚拟酶解,共得到1 769条多肽(表4)。通过对蛋白酶酶解所得肽段进行统计分析,肽段大多由2~8个氨基酸组成,计算其理论水解度如(图2)所示,其中胃蛋白酶(pH>2)和蛋白酶K对肽段水解能力较强,分别为达到35.67%和30.11%,胰蛋白酶和靡蛋白酶分别为11.63%和10.53%。
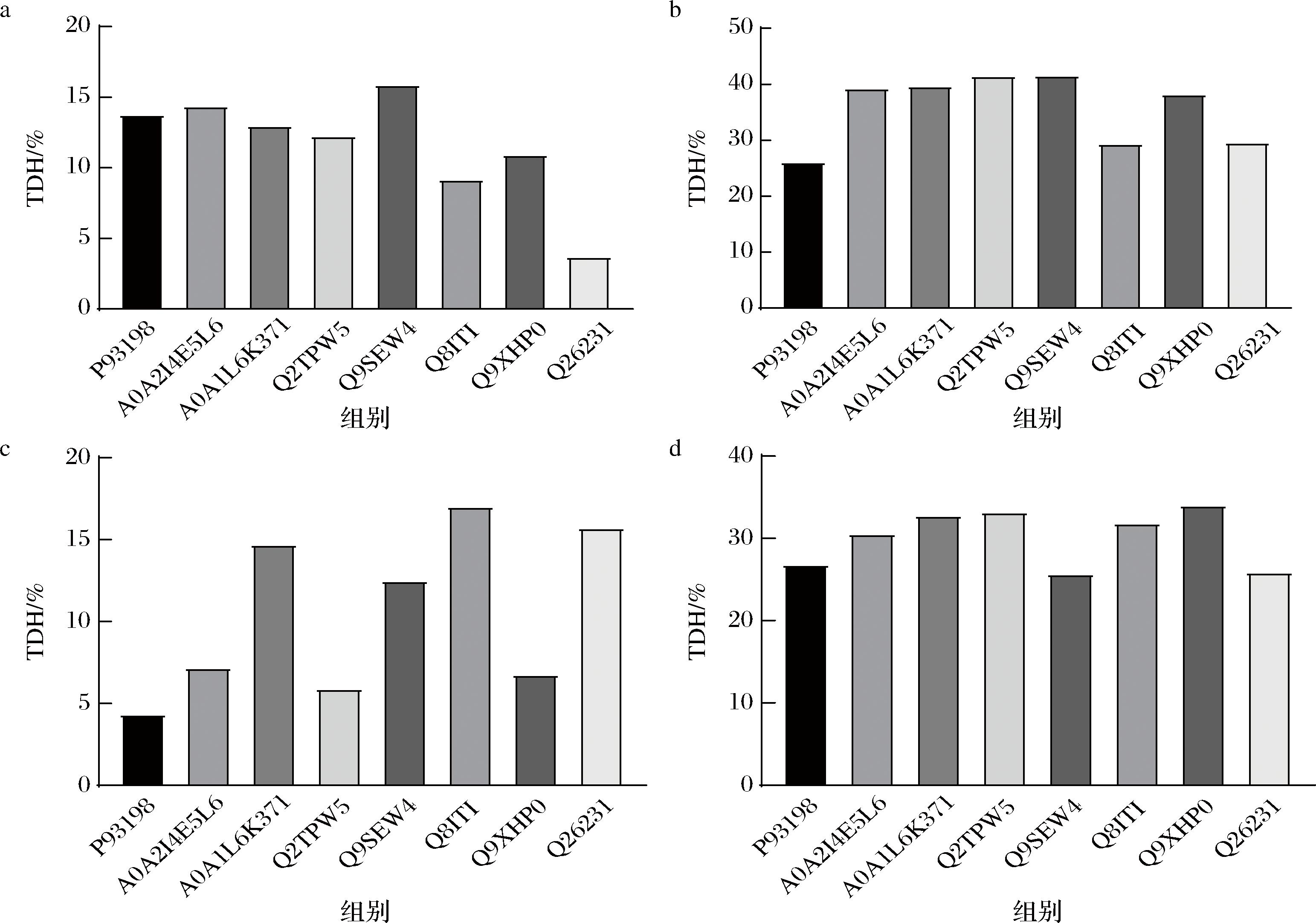
a-胰蛋白酶理论水解度;b-胃蛋白酶理论水解度;c-糜蛋白酶理论水解度;d-蛋白酶K理论水解度
图2 蛋白酶水解核桃蛋白理论水解度
Fig.2 TDH of walnut proteins by proteases
表4 虚拟酶解的多肽数量 单位:条
Table 4 Number of peptides virtually digested

蛋白酶分类二肽三肽四肽五肽其他胰蛋白酶764322胃蛋白酶(pH>2)401991014靡蛋白酶443221蛋白酶K251510817
2.3 核桃蛋白α-葡萄糖苷酶抑制肽筛选
将虚拟酶解所得多肽通过Peptide Ranker数据库进行生物活性预测,活性评分越高,代表肽段生物活性越好;结合BIOPEP-UWM在线工具对已报道过的α-葡萄糖苷酶抑制肽进行对比,得到活性评分大于0.5且未报道过的新颖肽100条(表5)。其中胃蛋白酶和蛋白酶K筛选得到的片段最多,胃蛋白酶酶解所得片段QCF生物活性评分最高为0.968 685。
表5 具有潜在生物活性的肽段
Table 5 Peptides with potential biological activity
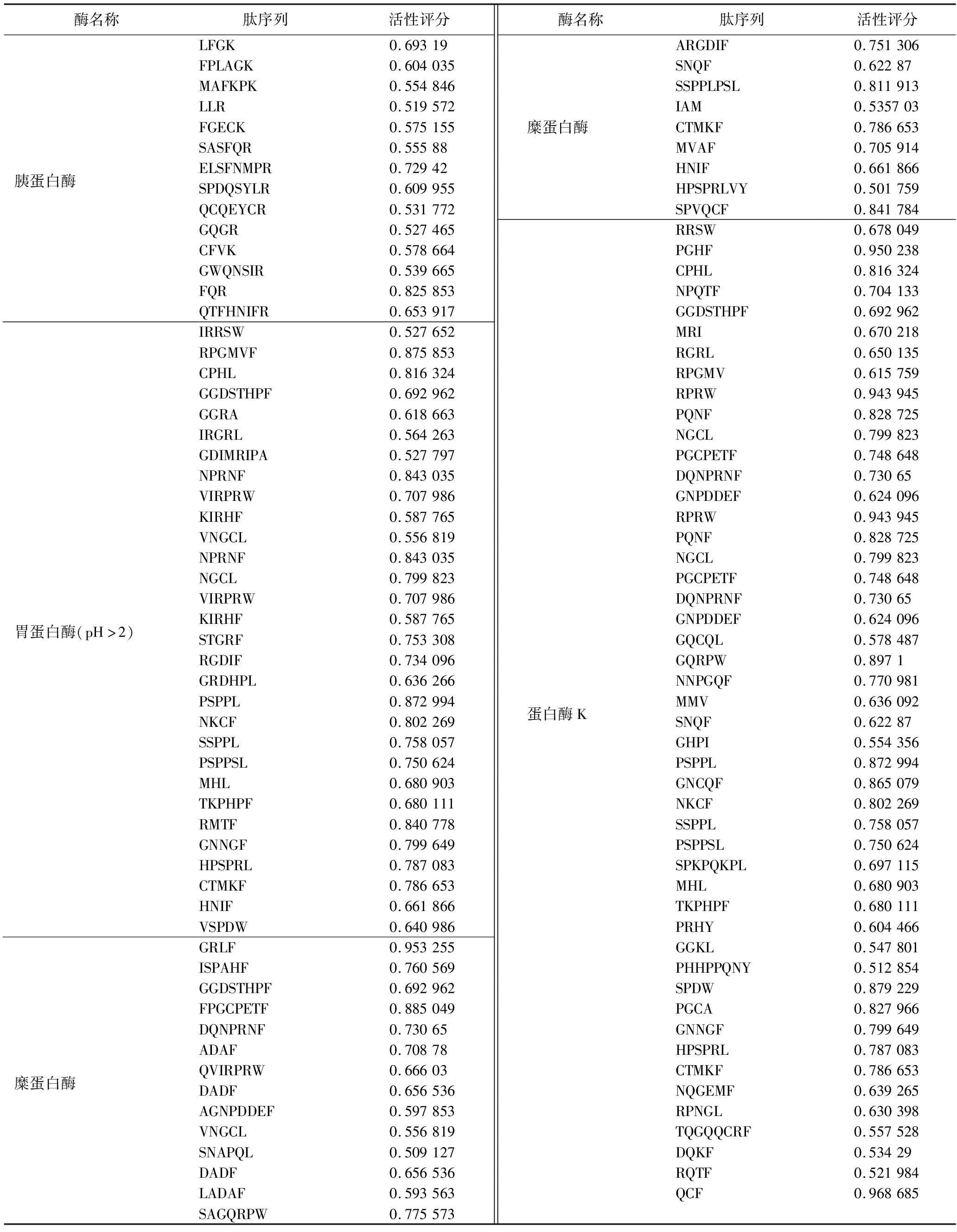
酶名称肽序列活性评分酶名称肽序列活性评分LFGK0.69319ARGDIF0.751306FPLAGK0.604035SNQF0.62287MAFKPK0.554846SSPPLPSL0.811913LLR0.519572IAM0.535703FGECK0.575155糜蛋白酶CTMKF0.786653SASFQR0.55588MVAF0.705914胰蛋白酶ELSFNMPR0.72942HNIF0.661866SPDQSYLR0.609955HPSPRLVY0.501759QCQEYCR0.531772SPVQCF0.841784GQGR0.527465RRSW0.678049CFVK0.578664PGHF0.950238GWQNSIR0.539665CPHL0.816324FQR0.825853NPQTF0.704133QTFHNIFR0.653917GGDSTHPF0.692962IRRSW0.527652MRI0.670218RPGMVF0.875853RGRL0.650135CPHL0.816324RPGMV0.615759GGDSTHPF0.692962RPRW0.943945GGRA0.618663PQNF0.828725IRGRL0.564263NGCL0.799823GDIMRIPA0.527797PGCPETF0.748648NPRNF0.843035DQNPRNF0.73065VIRPRW0.707986GNPDDEF0.624096KIRHF0.587765RPRW0.943945VNGCL0.556819PQNF0.828725NPRNF0.843035NGCL0.799823NGCL0.799823PGCPETF0.748648VIRPRW0.707986DQNPRNF0.73065胃蛋白酶(pH>2)KIRHF0.587765GNPDDEF0.624096STGRF0.753308GQCQL0.578487RGDIF0.734096GQRPW0.8971GRDHPL0.636266NNPGQF0.770981PSPPL0.872994蛋白酶KMMV0.636092NKCF0.802269SNQF0.62287SSPPL0.758057GHPI0.554356PSPPSL0.750624PSPPL0.872994MHL0.680903GNCQF0.865079TKPHPF0.680111NKCF0.802269RMTF0.840778SSPPL0.758057GNNGF0.799649PSPPSL0.750624HPSPRL0.787083SPKPQKPL0.697115CTMKF0.786653MHL0.680903HNIF0.661866TKPHPF0.680111VSPDW0.640986PRHY0.604466GRLF0.953255GGKL0.547801ISPAHF0.760569PHHPPQNY0.512854GGDSTHPF0.692962SPDW0.879229FPGCPETF0.885049PGCA0.827966DQNPRNF0.73065GNNGF0.799649ADAF0.70878HPSPRL0.787083糜蛋白酶QVIRPRW0.66603CTMKF0.786653DADF0.656536NQGEMF0.639265AGNPDDEF0.597853RPNGL0.630398VNGCL0.556819TQGQQCRF0.557528SNAPQL0.509127DQKF0.53429DADF0.656536RQTF0.521984LADAF0.593563QCF0.968685SAGQRPW0.775573
2.4 等电点、静电荷、水溶性、毒性、致敏性预测
采用Innovagen数据库对筛选的核桃蛋白肽进行亲水性(溶解性)预测。利用ToxinPred在线工具对其毒性进行预测,预测结果显示均无毒性。并通过A11erTop在线软件预测其致敏性,发现肽段无致敏原(表6)。最终筛选到得到生物活性肽FQR、NPRNF、NKCF、GRLF、RPRW、GQRPW、SPDW,这也表明从核桃蛋白中释放的生物活性肽安全无毒性,极具开发潜力。
表6 虚拟酶解肽段的理化性质
Table 6 Physicochemical properties of virtual enzymatic peptides
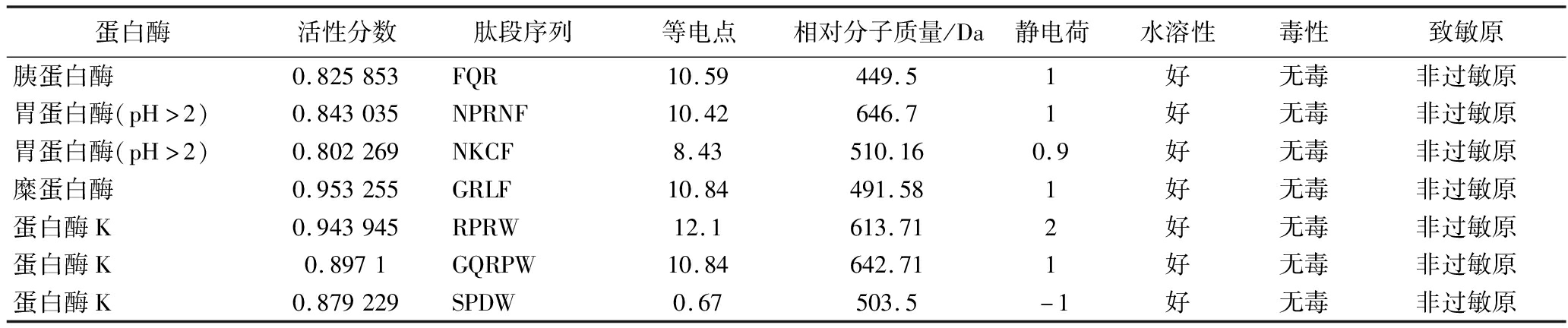
蛋白酶活性分数肽段序列等电点相对分子质量/Da静电荷水溶性毒性致敏原胰蛋白酶0.825853FQR10.59449.51好无毒非过敏原胃蛋白酶(pH>2)0.843035NPRNF10.42646.71好无毒非过敏原胃蛋白酶(pH>2)0.802269NKCF8.43510.160.9好无毒非过敏原糜蛋白酶0.953255GRLF10.84491.581好无毒非过敏原蛋白酶K0.943945RPRW12.1613.712好无毒非过敏原蛋白酶K0.8971GQRPW10.84642.711好无毒非过敏原蛋白酶K0.879229SPDW0.67503.5-1好无毒非过敏原
2.5 分子对接相互作用研究
为了分析多肽与疾病靶点α-葡萄糖苷酶(PDB ID:3A4A)之间的相互作用,筛选最终的α-葡萄糖苷酶抑制肽,利用分子对接技术以结合能作为评价标准。结果表明,7条肽与α-葡萄糖苷酶蛋白晶体具有不同程度的结合,结合能值为-8.3~-9.3 kcal/mol,表明它们与α-葡萄糖苷酶具有良好的结合亲和力,具有稳定的结合能力。
进一步分析肽与靶蛋白之间的结合位点和相互作用,使用利用PyMol软件进行可视化分析。结果如表7和图3所示,在3A4A蛋白对接盒子中,FQR与3A4A分子的氨基酸残基Arg315、Thr310、Ser311、Gln279、Glc601形成传统氢键,与Tyr158形成Pi-Pi键;NPPNF肽段与3A4A分子中Pro312、Arg315、Lys156、Ser241、240形成传统氢键,与Leu313、Phe314形碳氢键,与Asp233、307、242、Tyr158形成静电相互作用。NKCF肽段与3A4A分子中Gln533、Glu277、Arg315、His280、Asp242形成传统氢键,与Glu411形成碳氢键。GRLF肽段与3A4A分子中Arg315、Asn415、Lys156、Ser240、Ser241形成传统氢键,与Leu313形成碳氢键,与Tyr158、Asp242形成静电相互作用;RPRW肽段与3A4A分子中Arg315、Pro312、His80、Lys156形成传统氢键。GQRPW肽段与3A4A分子中Leu13、Lyw156、Tyr158、Arg315、His280、Pro312与Ser304形成碳氢键,与GLU411形成静电相互作用。
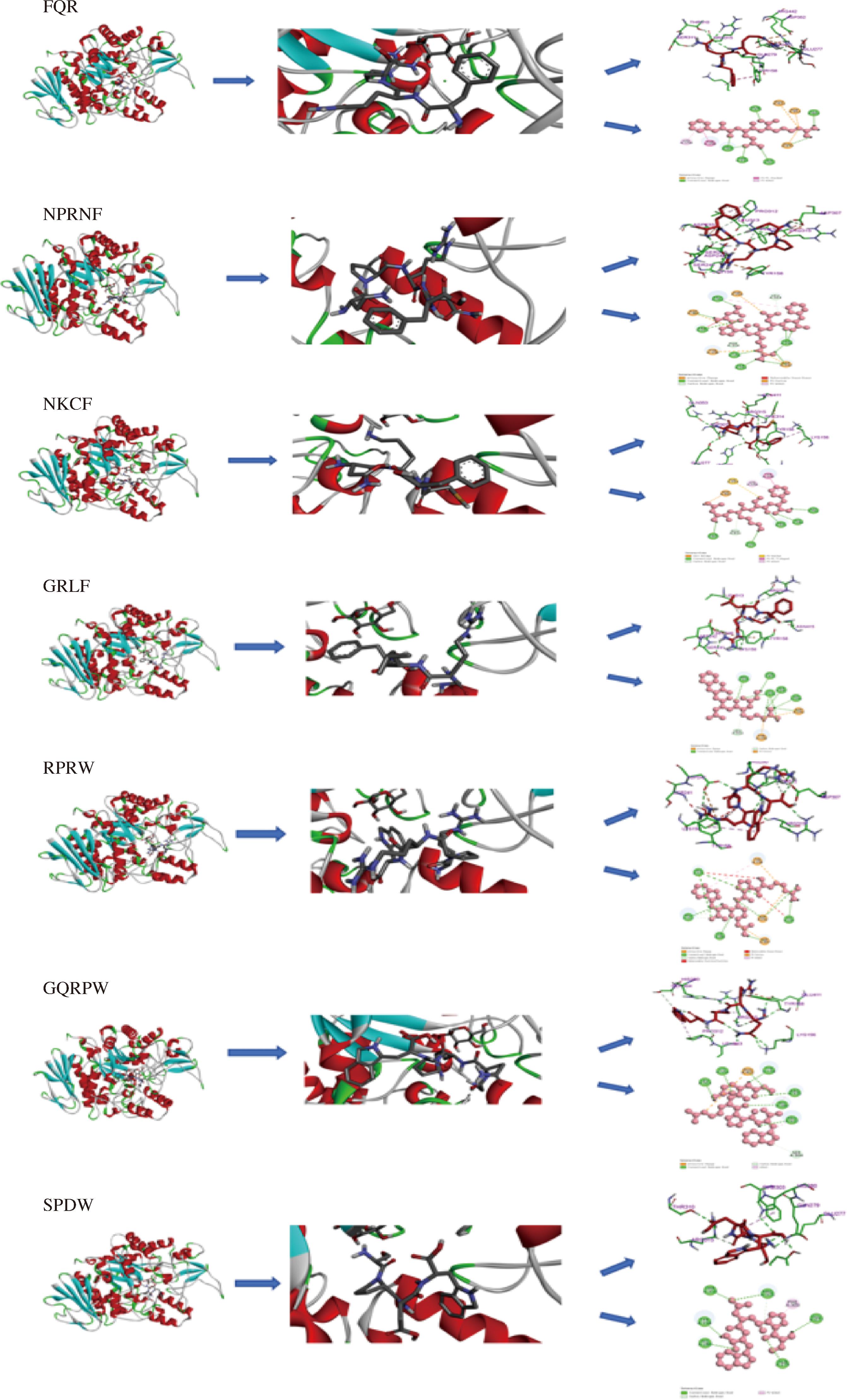
图3 肽段与3A4A蛋白分子对接可视化图
Fig.3 Visualization of peptide docking with 3A4A protein molecule
表7 虚拟筛选确定肽与α-葡萄糖苷酶结合位点
Table 7 Peptide binding sites to α-glucosidase identified by virtual screening
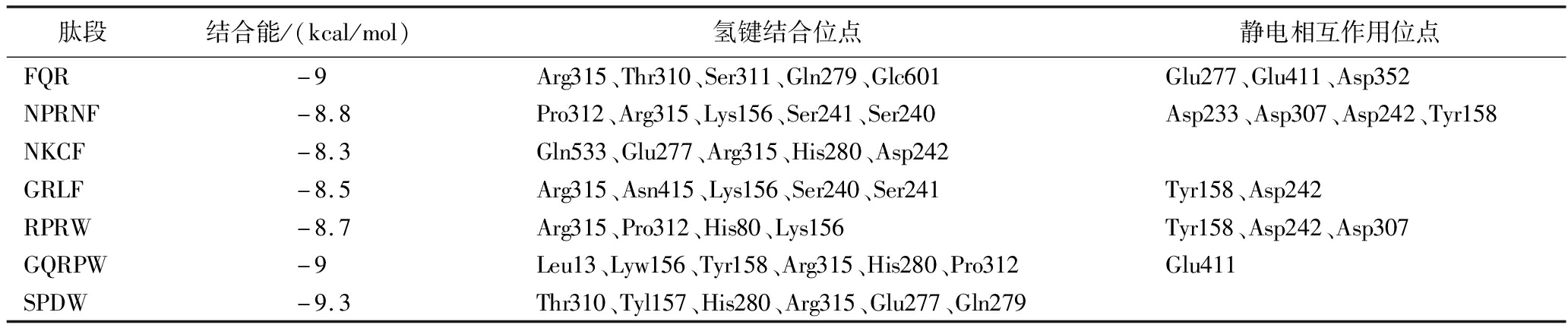
肽段结合能/(kcal/mol)氢键结合位点静电相互作用位点FQR-9Arg315、Thr310、Ser311、Gln279、Glc601Glu277、Glu411、Asp352NPRNF-8.8Pro312、Arg315、Lys156、Ser241、Ser240Asp233、Asp307、Asp242、Tyr158NKCF-8.3Gln533、Glu277、Arg315、His280、Asp242GRLF-8.5Arg315、Asn415、Lys156、Ser240、Ser241Tyr158、Asp242RPRW-8.7Arg315、Pro312、His80、Lys156Tyr158、Asp242、Asp307GQRPW-9Leu13、Lyw156、Tyr158、Arg315、His280、Pro312Glu411SPDW-9.3Thr310、Tyl157、His280、Arg315、Glu277、Gln279
李德龙等[23]通过分子对接技术筛选到药桑中8种成分对α-葡萄糖苷酶具有潜在抑制活性,其中氨基酸残基Asp69、Asp215、Glu277、Asp352与α-葡萄糖苷酶通过氢键相互作用连接,与前人的研究一致,SPDW肽段与3A4A分子中Thr310、Tyl157、His280、Arg315、Glu277、Gln279形成传统氢键,这表明氨基酸残基Glu277是α-葡萄糖苷酶靶蛋白晶体3A4A中的关键结合位点。
近年来,分子对接技术已成为研究人员用于筛选α-葡萄糖苷酶抑制剂的热门的技术手段,并且在这方面做了大量研究。赵晨颖等[24]通过虚拟筛选、分子对接及活性验证得到荭草素和异荭草素对SGLT2和α-葡萄糖苷酶具有一定的抑制活性,分子对接结果表明,5个Arg残基与α-葡萄糖苷酶形成氢键。薛森等[25]以大豆蛋白为研究对象虚拟筛选得到肽段EASY其α-葡萄糖苷酶抑制活性较强,分子对接结果表明,氢键、静电力和盐桥在肽段与α-葡萄糖苷酶间起着重要的作用。本研究结果与前人的研究一致,肽段与α-葡萄糖苷酶分子结合过程中氢键、静电力起着重要的作用。这些研究表明,将虚拟筛选和分子对接方法用于筛选α-葡萄糖苷酶抑制肽具有科学性和可靠性。
2.6 分子动力学模拟
为了进一步验证筛选到的肽段与α-葡萄糖苷酶蛋白复合物间结合的稳定性,选择肽段和蛋白-配体复合物进行分子动力学模拟。根据分子对接结果,肽段SPDW、FQR、GQRPW 3条肽结合亲和力较好,分别为-9.3、-9.0、-9.0 kcal/mol。α-葡萄糖苷酶抑制肽构效关系表明,含有疏水性氨基酸以及C末端含有精氨酸(Arg)的肽是潜在的α-葡萄糖苷酶抑制肽[14],结合构效关系和分子对接结果选择肽段FQR进行分子动力学模拟。
RMSD可用于评估模拟过程中复合物相对于初始结构的整体构象变化,RMSD值越大,表明结构变化越显著。如图4-a所示,在动力学前期(0~50 ns),复合物RMSD变化明显,而在50~100 ns期间趋于平衡,说明在50 ns后复合物构象趋于稳定。整个模拟过程中,RMSD保持相对稳定,表明小分子FQR与受体蛋白的结合始终紧密且稳定。局部氨基酸残基的结构波动由RMSF值反映,RMSF值越大,表明残基越灵活。如图4-b所示,RMSF值在0.2内波动,说明关键残基因受体与配体结合而丧失了灵活性,表明结合稳定。
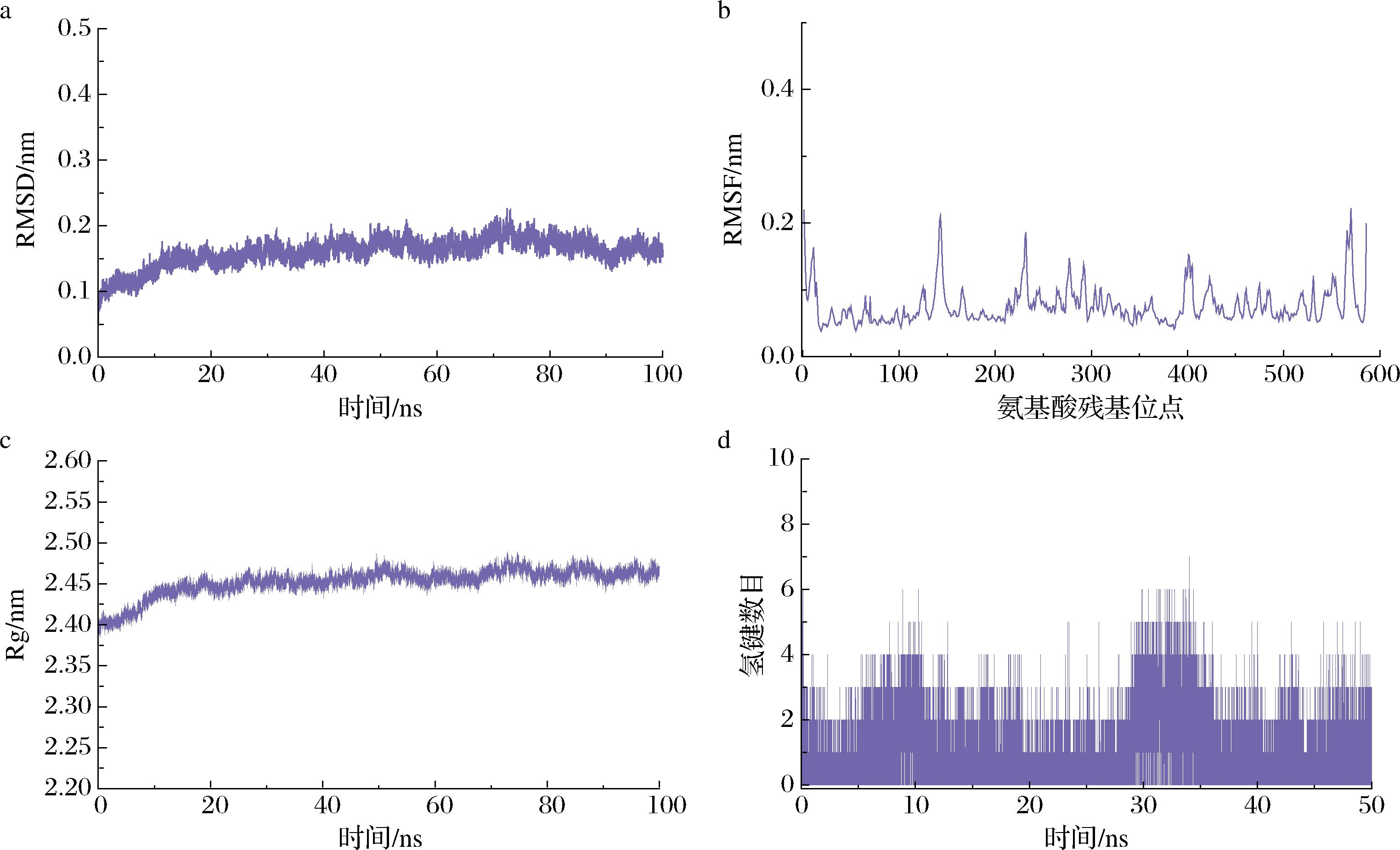
a-RMSD;b-RMSF;c-Rg;d-氢键数目
图4 肽段FQR与蛋白分子动力学模拟分析
Fig.4 Simulation analysis of peptide FQR with protein molecular dynamics
回转半径(radius of gyration, Rg)可用于评价体系的结构紧密程度,值越大表示蛋白质越松散,值越小则表示蛋白质越紧密。从图4-c可以看出,在复合物平衡阶段(50~100 ns),Rg值稳定在2.46 nm。分析氢键信息(图4-d)显示,配体与蛋白质间形成的氢键数量为0~7,表明氢键相互作用对结合有一定的贡献。
3 结论
本文以核桃蛋白为研究对象,利用计算机模拟酶解和在线数据库预测酶解所得肽段的生物活性及理化性质。将筛选所得肽段与α-葡萄糖苷酶靶蛋白进行分子对接,以评价潜在α-葡萄糖苷酶抑制活性。研究结果表明,核桃蛋白共具有20种氨基酸,通过虚拟酶解得到1 769条肽,其中胃蛋白酶(pH>2)理论水解度最高达到35.67%。利用活性预测工具筛选出100条活性评分大于0.5的肽段,对肽段理化性质进一步预测,获得7条具有良好水溶性、无毒且无致敏原的多肽。分子对接结果显示,7条肽对接结合能为-8.3~-9.3 kcal/mol,表现出较强的结合亲和力,其中SPDW结合亲和力最高,其次为GQRPW和FQR,这些肽段主要通过氢键、碳氢键、静电力与α-葡萄糖苷酶晶体蛋白发生稳定结合。通过分子动力学模拟验证,核桃衍生肽FQR与受体蛋白在整个模拟过程中紧密结合,整体构象稳定,表明其为潜在的α-葡萄糖苷酶抑制肽。本文通过计算机模拟酶解,结合多个数据库和在线工具高效筛选出7条潜在的α-葡萄糖苷酶抑制肽,为食源性活性肽的筛选提供了新思路。
[1] SUN H, SAEEDI P, KARURANGA S, et al.IDF Diabetes Atlas:Global, regional and country-level diabetesprevalence estimates for 2021 and projections for 2045[J].Diabetes Research and Clinical Practice, 2022, 183:109119.
[2] LU H, XIE T T, WU Q, et al.Alpha-glucosidase inhibitory peptides:Sources, preparations, identifications, and action mechanisms[J].Nutrients, 2023, 15(19):4267.
[3] 高健健.茶叶中α-葡萄糖苷酶抑制剂高通量筛选及其降糖活性研究[D].北京:中国农业科学院, 2023.GAO J J.High-throughput screening of α-glucosidase inhibitors from tea and their hypoglycemic activity[D].Beijing:Chinese Academy of Agricultural Sciences, 2023.
[4] 苏蓉.藜麦皮中α-葡萄糖苷酶抑制剂的筛选及其作用机制研究[D].西宁:青海大学, 2023.SU R.Screening for α-glucosidase inhibitors and research on effect mechanism of Quinoa Husks[D].Xining:Qinghai University, 2023.
[5] 李炼.临床常用口服降糖药物的潜在代谢性药物相互作用研究进展[J].中国老年学杂志, 2024, 44(3):760-763.LI L.Research process on potential metabolic drug interactions of commonly used oral hypoglycemic drugs in clinic[J].Chinese Journal of Gerontology, 2024, 44(3):760-763.
[6] 王姗姗, 左阮靖, 张玉红.生物活性肽制备方法的研究进展[J].食品科技, 2024, 49(2):255-262.WANG S S, ZUO R J, ZHANG Y H.Recent advances in the production of bioactive peptides[J].Food Science and Technology, 2024, 49(2):255-262.
[7] LI X Y, GUO M L, CHI J T, et al.Bioactive peptides from walnut residue protein[J].Molecules, 2020, 25(6):1285.
[8] 刘雪倩.生物活性肽的分子改造及功能研究[D].福州:福州大学, 2021.LIU X Q.Molecular modification and fuction study of bioactive peptide[D].Fuzhou:Fuzhou University, 2021.
[9] 杜战稳, 陈嘉.模拟酶解海参蛋白制备抗氧化肽及其抗疲劳活性研究[J].食品安全质量检测学报, 2024, 15(8):96-106.DU Z W, CHEN J.Preparation of antioxidant peptides from simulated enzymatic hydrolysis of sea cucumber protein and their anti-fatigue activity[J].Journal of Food Safety and Quality, 2024, 15(8):96-106.
[10] 吴萍, 周际松, 邓乾春, 等.核桃蛋白的结构、营养价值、制备、功能特性及在食品中的应用[J].食品科学, 2024, 45(15):329-337.WU P, ZHOU J S, DENG Q C, et al.Structure, nutritional value, extraction, functional properties of walnut proteins and their application in food:A review[J].Food Science, 2024, 45(15):329-337.
[11] 倪策, 曹天红, 陈敏, 等.核桃粕源抗氧化活性肽的酶解制备及活性分析[J].食品与机械, 2024, 40(5):51-61.NI C, CAO T H, CHEN M, et al.Enzymatic hydrolysis preparation and activity analysis of antioxidant peptides derived from walnut dregs[J].Food and Machinery, 2024, 40(5):51-61.
[12] 刘丹丹.核桃蛋白的抗氧化与降血压活性评价及酶解必要性研究[D].镇江:江苏大学, 2020.LIU D D.Antioxidant and antihypertensive activities evaluation and the enzymatic hydrolysis necessity of walnut protein[D].Zhenjiang:Jiangsu University, 2020.
[13] 李晴.山核桃油降血脂功能活性及其微胶囊化技术研究[D].舟山:浙江海洋大学, 2022.LI Q.Study on hypolipidemic activity and microencapsulation technology of walnut oil[D].Zhoushan:Zhejiang Ocean University, 2022.
[14] LUAN F, PENG L X, LEI Z Q, et al.Traditional uses, phytochemical constituents and pharmacological properties of Averrhoa carambola L.:A review[J].Frontiers in Pharmacology, 2021:12:699899.
[15] 李丽琪, 徐华容, 刘阳, 等.核桃多肽对高糖诱导HepG2胰岛素抵抗模型的影响[J].中国食物与营养, 2018, 24(4):66-70;28.LI L Q, XU H R, LIU Y, et al.Effect of walnut peptide on HepG2 insulin resistance induced by high glucose[J].Food and Nutrition in China, 2018, 24(4):66-70;28.
[16] 李丽.核桃多肽防治糖尿病物质基础及作用机制研究[D].武汉:武汉工程大学, 2016.LI L.Study on the material basis and mechanism of walnut polypeptide in the prevention and treatment of diabetes[D].Wuhan:Wuhan Engineering University, 2016.
[17] 力俊琛, 高昕悦, 江晨怡, 等.模拟酶解优化鸽血红蛋白抗氧化肽酶法制备[J].食品研究与开发, 2023, 44(2):108-115.LI J C, GAO X Y, JIANG C Y, et al.Simulation of enzymolysis to optimize the preparation of pigeon hemoglobin antioxidant peptides[J].Food Research and Development, 2023, 44(2):108-115.
[18] 刘鑫, 罗唐君, 韩丽娟, 等.藏羊血红蛋白抗氧化肽模拟酶解筛选及活性验证[J].食品与发酵工业, 2025,51(7):64-73.LIU X, LUO T J, HAN L J, et al.Simulated enzymatic screening and activity validation of antioxidant peptides from Tibetan sheep hemoglobin[J].Food and Fermentation Industries, 2025,51(7):64-73.
[19] VILCACUNDO R, MART NEZ-VILLALUENGA C, HERN
NEZ-VILLALUENGA C, HERN NDEZ-LEDESMA B.Release of dipeptidyl peptidase IV, α-amylase and α-glucosidase inhibitory peptides from quinoa (Chenopodium quinoa Willd.) during in vitro simulated gastrointestinal digestion[J].Journal of Functional Foods, 2017, 35:531-539.
NDEZ-LEDESMA B.Release of dipeptidyl peptidase IV, α-amylase and α-glucosidase inhibitory peptides from quinoa (Chenopodium quinoa Willd.) during in vitro simulated gastrointestinal digestion[J].Journal of Functional Foods, 2017, 35:531-539.
[20] WEI R T, LIN L K, LI T T, et al.Separation, identification, and design of α-glucosidase inhibitory peptides based on the molecular mechanism from Paeonia ostii ‘Feng Dan’seed protein[J].Journal of Food Science, 2022, 87(11):4892-4904.
[21] LIU W W, LI H Y, WEN Y Y, et al.Molecular mechanism for the α-glucosidase inhibitory effect of wheat germ peptides[J].Journal of Agricultural and Food Chemistry, 2021, 69(50):15231-15239.
[22] HU S F, FAN X D, QI P, et al.Identification of anti-diabetes peptides from Spirulina platensis[J].Journal of Functional Foods, 2019, 56:333-341.
[23] 李德龙, 伊丽则热·艾拜杜拉, 陈冰婷, 等.基于分子对接筛选药桑中抑制α-葡萄糖苷酶活性成分[J].新疆医科大学学报, 2021, 44(11):1275-1281.LI D L, YILZERA Aibaidula,CHEN B T, et al.Screening of α-glucosidase inhibitory components from Morus nigra Linn.based on molecular docking[J].Journal of Xinjiang Medical University, 2021, 44(11):1275-1281.
[24] 赵晨颖, 韩雪, 贺一凡, 等.虚拟筛选荭草素和异荭草素降血糖靶点和体外活性评价[J].现代药物与临床, 2024, 39(2):304-308.ZHAO C Y, HAN X, HE Y F, et al.Hypoglycemic targets of orientin and isoorientin based on virtual screening and in vitro test[J].Drugs and Clinic, 2024, 39(2):304-308.
[25] 薛森, 吴敬凡, 曹淑芳, 等.大豆多肽中α-葡萄糖苷酶和α-淀粉酶抑制剂的虚拟筛选及体外活性验证[J].中国食品学报, 2024, 24(6):44-56.XUE S, WU J F, CAO S F, et al.Virtual screening of α-glucosidase and α-amylase inhibitors from soybean peptides and its activity validation in vitro[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(6):44-56.