甾醇类药物是第二大临床用药[1]。植物甾醇及其衍生物已作为食品添加剂,广泛应用于包括油脂、牛奶、糕点及饮料等在内的各种食品中[2]。近年来,有研究利用各种生物技术,对甾醇分子进行修饰或生物转化,以丰富甾醇母核结构多样性;提高甾醇的脂溶性、水溶性、生物利用度等各种性质;提高其生物转化率等[3-4]。其中,对甾醇分子母核骨架中的羟基进行酯化修饰,是最常见的一种分子修饰。
目前,已报道的用于对甾醇进行酯化修饰的酶蛋白主要是各种脂肪酶(triacylglycerol lipase, EC.3.1.1.3),包括皱褶假丝酵母脂肪酶和南极假丝酵母脂肪酶等[5-6]。但对目前市售商品化脂肪酶催化甾醇酯化(或甾醇酯水解)活性检测分析发现,脂肪酶催化甾醇酯水解(或甾醇酯化)的催化活性普遍偏低,部分脂肪酶甚至完全不具催化活性[7]。虽然甾醇酯酶(cholesterol esterase, EC3.1.1.13)和脂肪酶都是催化羧酸酯键的形成(或断裂),但两者催化底物分子中的酰基受体的分子结构、疏水性等存在显著性的差异。甾醇酯中,酰基受体为甾醇;而甘油三酯分子中,酰基受体为甘油,2种底物对酶蛋白活性中心的结构要求完全不一样[8-9]。
到目前为止,Ophiostoma piceae 胆固醇酯酶(Ophistoma picae steryl esterase,OPE)为仅有的一个微生物源胆固醇酯酶。该酶的基本酶学性质、晶体结构等基本阐明[9],其酶在甾醇酯化、聚酯塑料降解、生物制浆等方面有良好的应用潜力[10-13],然而该酶的异源高效表达一直欠佳[14]。毕赤酵母(Pichia pastoris)表达系统是到目前为止,相对较为成熟的一种真核表达系统,已针对密码子偏好性、转录效率、蛋白质分泌与修饰等技术瓶颈,建立起各种遗传操作体系[15-16]。系列酶蛋白已在P.pastoris中实现了高效表达,包括脂肪酶、溶菌酶、植酸酶和转谷氨酰胺酶等,部分酶种已实现商业化生产[17]。
本研究分别考察了基因拷贝数、重组蛋白分泌信号类型和共表达二硫键异构酶(protein disulfide isomerase, PDI)对ope基因在P.pastoris GS115菌株中表达水平的影响,以期获得高效表达ope基因的重组P.pastoris GS115菌株,为后续实现重组OPE的工业化生产,开发固定化的OPE酶制剂,并进一步推广OPE的应用奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 质粒及菌株
本研究使用的各种质粒、菌株及其基本性质特点如表1所示。所有ope基因序列的5′端携带有编码6个His的密码子序列(CATCACCATCACCATCAC)。
表1 实验使用的质粒及菌株
Table 1 The plasmids and microbial strains used in this research

质粒或菌株名称特点及用途备注P.pastorisGS115表达外源基因的宿主菌株本实验室保存E.coliDH5α克隆菌株、各类质粒的保存及扩繁本实验室保存pPICZαAP.pastoris表达外源基因的穿梭载体。在E.coli和P.pastorisGS115中均为zeocin+抗性本实验室保存pUC57-oafsα-factor信号肽密码子优化序列(简称为OAFS)插入到PUC57质粒HindⅢ、EcoRⅠ上,Amp+,oafs序列由北京擎科生物科技股份有限公司合成[18]pUC57-iafs在α-factor信号肽的甲硫氨酸起始位点和前导体与内源性蛋白酶加工位点之间分别插入AIP和EEAEAEAEPK(简称为IAFS)。Iafs基因序列由北京擎科生物科技股份有限公司合成,并插入到PUC57质粒HindⅢ、EcoRⅠ上,Amp+[19]pEESB-1利用限制性内切酶EcoRⅠ和NotⅠ向pPICZαA载体引入ope基因,zeocin+抗性实验室构建pEESB-2将pEESB-1上信号肽α-factor编码区进行密码子优化(密码子优化后的信号肽简称为OAFS),zeocin+本实验构建pEESB-3将pEESB-1上信号肽α-factor的甲硫氨酸起始位点和前导肽与内源性蛋白酶加工位点之间分别插入AIP序列和EEAEAEAEPK序列的编码序列(该信号肽简称为IAFS),zeocin+本实验构建pEESB-4在pEESB-1基础上,利用定点突变技术,将信号肽α-factor第9位丙氨酸突变为天冬氨酸,第20位丙氨酸突变为苏氨酸(该信号肽简称为MAFS),zeocin+本实验构建pEESB-5在pEESB-1基础上,利用滚环复制技术,将信号肽α-factor的前肽替换成Ost1的对应序列(该信号肽简称为TAFS),zeocin+本实验构建pEESB-6利用BamHⅠ向pEESB-1载体相应位置引入P.pastorisrDNA片段,zeocin+本实验构建pEESB-7利用BamHⅠ向pEESB-2载体相应位置引入P.pastorisrDNA片段,zeocin+本实验构建pEESB-8利用BamHⅠ向pEESB-3载体相应位置引入P.pastorisrDNA片段,zeocin+本实验构建pEESB-9利用BamHⅠ向pEESB-4载体相应位置引入P.pastorisrDNA片段,zeocin+本实验构建pEESB-10利用BamHⅠ向pEESB-5载体相应位置引入P.pastorisrDNA片段,zeocin+本实验构建pPIC9KP.pastoris表达外源基因穿梭载体。在E.coli中为氨苄青霉素抗性,在P.pastoris内为G418抗性实验室保存pPIC9K-pdi利用EcoRⅠ和NotⅠ向pPIC9K载体相应位点插入pdi基因本实验构建P.pastorisGS115/pEESB-1线性化后的pEESB-1经电转导入P.pastoris后筛选获得的重组菌株实验室构建P.pastorisGS115/pEESB-6线性化后的pEESB-6经电转导入P.pastoris后筛选获得的重组菌株本实验构建P.pastorisGS115/pEESB-7线性化后的pEESB-7经电转导入P.pastoris后筛选获得的重组菌株本实验构建P.pastorisGS115/pEESB-8线性化后的pEESB-8经电转导入P.pastoris后筛选获得的重组菌株本实验构建P.pastorisGS115/pEESB-9线性化后的pEESB-9经电转导入P.pastoris后筛选获得的重组菌株本实验构建P.pastorisGS115/pEESB-10线性化后的pEESB-10经电转导入P.pastoris后筛选获得的重组菌株本实验构建P.pastorisGS115/pEESB-11线性化后的pPIC9K-pdi经电转导入P.pastoris/pEESB-10后筛选获得的重组菌株本实验构建
1.1.2 试剂
AxyPrep质粒小量制备试剂盒、AxyPrep DNA凝胶回收试剂盒,爱思进生物技术(杭州)有限公司;各种限制性内切酶等分子生物学试剂,宝生物工程(大连)有限公司;各种引物序列(表2),生工生物工程(上海)股份有限公司合成;基因序列分析,北京擎科生物科技股份有限公司完成;月桂酸对硝基苯酯(4-nitrophenyllaurate, pNPL),SigmaAldrich;其他常规试剂均为市售分析纯。各种微生物培养基具体成分如下:
表2 本研究使用的引物对
Table 2 PCR primers used in this study

引物名称序列(5′-3′)Tm/℃退火温度/℃PCR产物/bp用途αA9D-FwαA9D-RwTTACTGATGTTTTATTCGCAGCATCCTCCAAACATCAGTAAAAATTGAAGGAAATCTCATCG58.655.0555194构建pEESB-4,信号肽α-factor第9位的丙氨酸突变为天冬氨酸αA20T-FwαA20T-RwCATTAGCTACTCCAGTCAACACTACAACAGAAGCTGGAGTAGCTAATGCGGAGGATGCTG59.363.4565197构建pEESB-4,信号肽α-factor第20位的丙氨酸突变为苏氨酸Ost1-1ROst1-1FTCCAAGAGAACCAAACCTGCCTCATCGTTTCGAATAATTAGGTTCTCTTGGAGTCTTCTGCTGCTCCAGTCAACACTACA63.768.1585159构建pEESB-5,利用滚环复制,将α-factor的前导肽置换为Ost1相应序列(第一轮滚环复制)Ost1-2ROst1-2FACATAGGAACAATCCCACAATCCAAGAGAACCAAACCTTTGTTCCTATGTTTTTTCAACGTGTCTTCTGCTGCTCCAGT64.365.4605202α-factor的前导肽置换为Ost1相应序列(第二轮滚环复制)rDNA-FrDNA-RCGCGGATCCTAGTTAGGTTACCGTTTTTCCTAATATTAAAGGATCCCTTCCACCAACAGTCAACCACC64.066.0601199以P.pastoris基因组为模板,PCR扩增rDNA片段pdi-Fpdi-RCCGGAATTCATGCAATTCAACTGGGATATTAAAACTGTGGAAATATGCGGCCGCTTAAAGCTCGTCGTGAGCGTCTGCC62.072.0621568以P.pastoris基因组为模板,PCR扩增pdi5’AOX3’AOXGACTGGTTCCAATTGACAAGCGCAAATGGCATTCTGACATCC5657552034菌落PCR验证重组转化子
注:横线下划线的脱氧核苷酸为:编码突变氨基酸残基的三联体密码子;引入的插入序列;或限制性内切酶识别位点。
最小葡萄糖(minimal dextrose medium, MD)培养基(g/L):酵母氮碱13.4、生物素0.4×10-3、葡萄糖20;
YPD培养基(g/L):酵母提取物10、蛋白胨20、葡萄糖20;
酵母浸出粉胨葡萄糖山梨醇(yeast extract peptone dextrose medium with sorbitol, YPDS)培养基(g/L):酵母提取物10、蛋白胨20、葡萄糖20、山梨醇182.17;
酵母膏胨山梨醇(yeast extract peptone medium with sorbitol, YEPS)液体培养基(g/L):酵母提取物10、蛋白胨20、山梨醇10。
1.2 仪器与设备
GenePulser MXcell转基因仪/电穿孔仪,美国Bio-Rad公司,用于将外源基因导入到毕赤酵母感受态细胞中;UV2600岛津紫外可见分光光度计,日本Shimadzu公司,用于胆固醇酯酶活力分析;AKATA prime plus,瑞典GE Healthcare公司, 用于对重组胆固醇酯酶OPE进行分离纯化。
1.3 实验方法
1.3.1 各类表达载体的构建
本实验中,P.pastoris源rDNA基因片段和伴侣蛋白编码基因pdi克隆使用的引物、PCR扩增使用的退火温度、克隆使用的限制性内切酶如表2所示。
质粒pEESB-1是在pPIC9K-ope(课题组先前构建的质粒)[13]基础上构建的。分别利用限制性内切酶EcoR Ⅰ和Not Ⅰ 对质粒pPICZαA和pPIC9K-ope进行酶切;然后回收ope基因片段和载体pPICZαA的酶切产物;最后用T4DNA连接酶连接,获得的质粒命名为pEESB-1。
对pPICZαA载体上的信号肽α-factor编码区优化产生的4种质粒构建过程分别为:a)信号肽α-factor编码区密码子的优化参照MATHEWS等[18]。脱氧核苷酸序列由北京擎科生物科技股份有限公司完成,优化后的基因序列5′和3′端分别添加限制性内切酶Hind Ⅲ和EcoR Ⅰ识别位点;利用该序列置换质粒pEESB-1质粒对应序列,衍生的质粒命名为pEESB-2;信号肽在后续研究中缩写为OAFS;b)信号肽α-factor氨基酸序列的优化参照XIONG等[19]。脱氧核苷酸序列由北京擎科生物科技股份有限公司完成,优化后的基因序列5′和3′端分别添加限制性内切酶Hind Ⅲ和EcoR Ⅰ识别位点;利用该序列置换质粒pEESB-1质粒对应序列,衍生的质粒命名为pEESB-3;信号肽在后续研究中缩写为IAFS;c)参照BARRERO等[20]的方法,利用定点突变技术,在pEESB-1基础上,将信号肽α-factor第9位的丙氨酸突变为天冬氨酸;第20位的丙氨酸突变为苏氨酸,衍生的质粒命名为pEESB-4;该信号肽在后续研究中缩写为MAFS。定点突变及突变体筛选参照Takara高保真酶PrimeSTAR®HS DNA Polymerase试剂说明书进行;d)参照AZA等[21]的方法,利用滚环复制技术,在pEESB-1基础上,将信号肽α-factor的前导肽置换为Ost1对应序列。衍生的质粒命名为pEESB-5;该信号肽在后续研究中缩写为TAFS。
质粒pEESB-6~pEESB-10分别是质粒pEESB-1~pEESB-5的衍生物。先用限制性内切酶BamH Ⅰ对P.pastoris GS115 rDNA PCR扩增产物、质粒pEESB-1~pEESB-5进行酶切;再用AxyPrep DNA凝胶回收试剂盒分别纯化rDNA酶切产物和 pEESB-1~pEESB-5的酶切产物。最后用T4DNA连接酶连接分别连接rDNA和pEESB-1(或pEESB-2、pEESB-3、pEESB-4和pEESB-5)。连接产物转化E.coli DH5α,抽提转化子的质粒,测序验证,获得重组质粒pEESB-6~pEESB-10。
各类载体构建过程中使用的引物对、退火温度和PCR产物大小等如表2所示。
1.3.2 P.pastoris感受态的制备及其电转化
P.pastoris感受态的制备按照Invitrogen公司的P.pastoris标准操作手册。除pEESB-1和pPIC9K-pdi利用限制性内切酶Sac Ⅰ线性化外,其他各表达质粒均用Sca Ⅰ进行质粒线性化处理。线性化产物在进行电转化前,均需要用AxyPrep DNA凝胶回收试剂盒进行纯化处理。Bio-Rad GenePulser 电转化仪转化P.pastoris参数设置为:1 500 V,25 μF,200 Ω,电击5 ms。
1.3.3 重组P.pastoris转化子的筛选及验证
P.pastoris重组转化子的筛选参照Invitrogen公司的表达载体标准操作手册进行。基于pPIC9K质粒衍生的表达质粒转化P.pastoris获得的重组转化菌株分别利用MD培养基和含有50 μg/mL G418的YPD培养基进行筛选;筛选获得的阳性菌落进一步利用菌落PCR进行验证。基于pPICZαA质粒衍生的各种表达质粒转化P.pastoris获得的重组转化菌株,先用含有100 μg/mL Zeocin的YPDS培养基进行筛选;阳性菌落进一步利用菌落PCR进行验证。
1.3.4 ope基因表达水平差异性筛选
重组P.pastoris转化子的诱导表达及其重组胆固醇酯酶活力的测定参照CEDILLO等[14]并稍作修改。挑取转化子至含500 μL YEPS液体培养基的2 mL离心管中,30 ℃摇床220 r/min培养,每24 h加入体积分数为0.5%的甲醇诱导96 h。取200 μL发酵液检测OD600值,其余发酵液离心收集上清液测定酶活力。
1.3.5 重组胆固醇酯酶活力的测定
胆固醇酯酶活力的测定参照CEDILLO等[14]并稍作修改。反应体系为:180 μL(pH 7.4)20 mmol/L的K2HPO4-KH2PO4缓冲液,10 μL酶液,10 μL 10 mmol/L pNPL。40 ℃下反应5 min,记录410 nm波长下的紫外吸光值的变化。该反应体系下的摩尔消光系数ε=7.489 5×103 L/(μmol·cm)。在40 ℃ pH 7.4的条件下,每分钟催化底物pNPL水解并产生1 μmol pNP所需的酶量,定义为1个酯酶活力单位(U)。
1.3.6 摇瓶复筛
前述每次电转获得的转化子菌株库中,初筛发酵水平较高的10个菌株,将进行摇瓶复筛。具体操作如下:按照1% (体积分数)接种量将每个菌株的种子液接入含50 mL YEPS液体的250 mL三角瓶,30 ℃摇床220 r/min培养,每24 h加入体积分数为0.5%的甲醇诱导96 h。诱导发酵结束后离心收集上清液检测其酶活力,选择发酵液酶活力最高的重组菌株用于后续研究。
1.3.7 YEPS培养基中山梨醇浓度的优化
在YEPS培养基中,分别依次添加体积分数为0%、0.1%、0.2%、0.3%、0.4%和0.5%的山梨醇;然后按照1% (体积分数)的接种量接入P.pastoris GS115/pEESB-11重组菌株,30 ℃摇床220 r/min培养。每24 h添加0.5% (体积分数)甲醇,诱导96 h。分别测定发酵液的OD600值以及发酵上清液的酶活力。
1.3.8 重组OPE的分离纯化
上述摇瓶复筛获得的发酵酶活力最高的重组菌株(P.pastoris GS115/pEESB-11),利用山梨醇浓度优化后的发酵培养基,进一步大量发酵制备重组OPE。发酵液冻干后,溶于适量体积的平衡缓冲液(20 mmol/L pH 7.4 Na2HPO4-NaH2PO4,500 mmol/L NaCl和20 mmol/L 咪唑)中,12 000 r/min离心10 min后,收集上清液,再用0.22 μm滤膜过滤制备粗酶液。粗酶液利用His Trap HP层析柱(1 mL)进行分离纯化,洗脱条件为:20 mmol/L pH 7.4 Na2HPO4-NaH2PO4,500 mmol/L NaCl,300 mmol/L 咪唑,以1 mL/min流速梯度洗脱色谱柱,收集目标蛋白。目标蛋白OPE的纯度用SDS-PAGE检测。
1.4 数据处理
所有的实验均进行3次重复,取平均值;数据的显著性分析用SPSS软件处理;不同毕赤酵母转化子酶活力差异的统计分析用Graphpad Prism分析。
2 结果与分析
2.1 以P.pastoris rDNA为同源重组臂获得的重组P.pastoris转化子表达ope的水平
在前期实验中,以pPIC9K载体和pPICZαA载体作为表达载体,利用限制性内切酶在相应位置引入ope基因后获得的重组转化子经诱导表达后,发酵液中仅检测到极低的酶活力;SDS-PAGE仅在100倍浓缩后的发酵液中检测到微弱条带(未浓缩的发酵液直接电泳观察不到目标条带)[13]。
以核糖体rDNA为重组同源臂(转化pEESB-6质粒)的实验中,一共获得247个P.pastoris重组菌株。如图1所示,大部分重组菌株的胞外酶活力较低,均低于4×10-2 U/OD600;筛选到酶活力最高的转化子(命名为P.pastoris GS115/pEESB-6)为9.05×10-2 U/OD600。摇瓶发酵检测结果显示,P.pastoris GS115/pEESB-6胞外酶活力为0.28 U/mL,较以AOX为重组位点获得的转化子(P.pastoris GS115/pEESB-1)的产胞外酶活力提高了1.65倍。具体见图1。
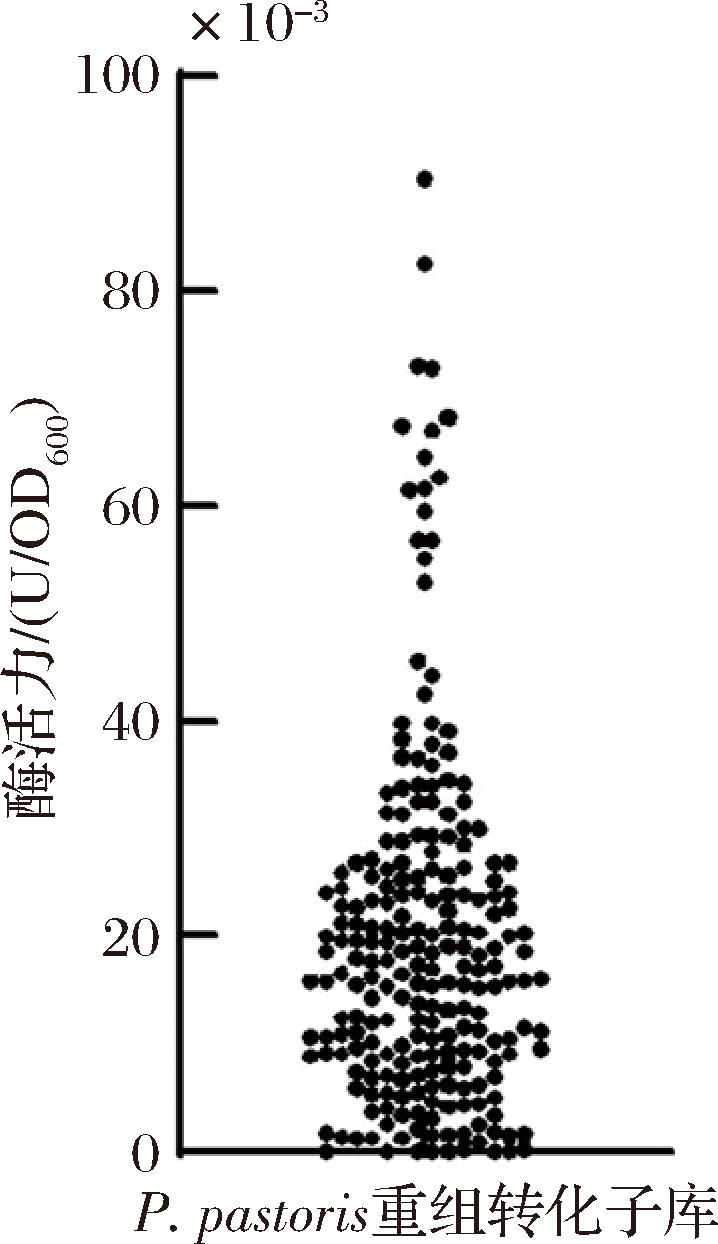
图1 转化pEESB-6质粒后获得的系列重组P.pastoris转化子产胞外胆固醇酯酶水平
Fig.1 OPE yield difference among P.pastoris transformants from plasmid pEESB-6
2.2 信号肽类型对ope基因在P.pastoris 中表达水平的影响
不同类型信号肽对ope基因在P.pastoris 中表达水平的影响如图2所示。4种信号肽中,TAFS和OAFS表现较佳,MAFS表现最差。基于4种信号肽分别获得的系列P.pastoris 重组转化子初筛结果表明:以OAFS、IAFS、MAFS和TAFS作为信号肽获得的重组P.pastoris转化子库中,初筛获得的重组子最高发酵水平分别为1.98×10-1、1.31×10-1、2.17×10-2、2.09×10-1 U/OD600。分别将OAFS和TAFS转化子库中发酵水平最高的10个转化子进行摇瓶复筛,获得发酵水平最好的菌株命名为P.pastoris GS115/pEESB-10(以TAFS作为信号肽),发酵液酶活力为2.55 U/mL,较P.pastoris GS115/pEESB-6菌株相比,胞外胆固醇酯酶OPE提高了9.1倍。
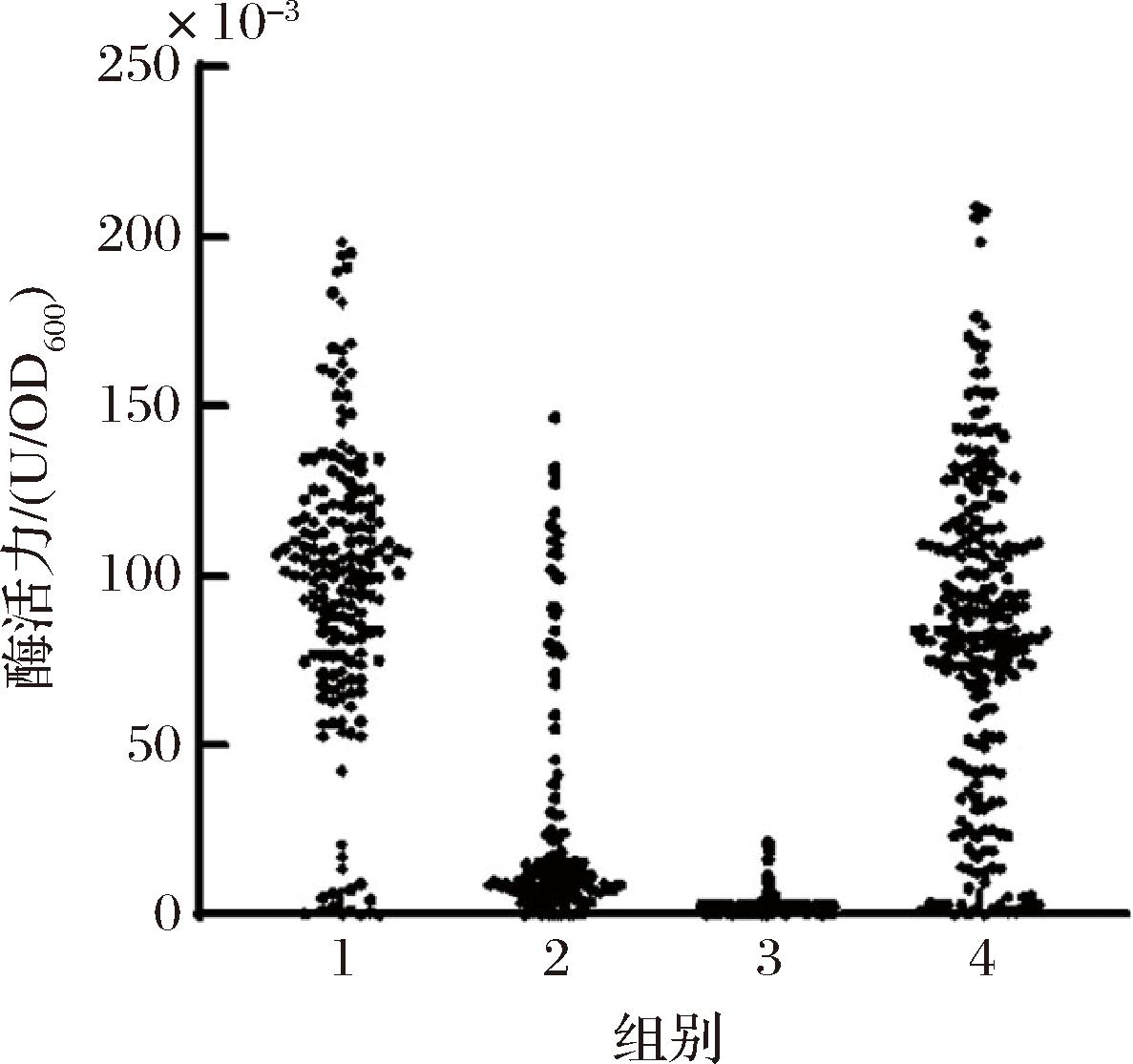
1-质粒pEESB-7经线性化处理后电转化导入P.pastoris菌株后获得的系列重组菌株;2-质粒pEESB-8经线性化处理后电转化导入P.pastoris菌株后获得的系列重组菌株;3-质粒pEESB-9经线性化处理后电转化导入P.pastoris菌株后获得的系列重组菌株;4-质粒pEESB-10经线性化处理后电转化导入P.pastoris菌株后获得的系列重组菌株。
图2 不同信号肽对ope基因在P.pastoris 中表达水平的影响
Fig.2 Effect of signal peptide types on OPE yield from P.pastoris transformants
2.3 共表达二硫键异构酶pdi对ope基因表达水平的影响
pPIC9K-pdi质粒经Sac Ⅰ线性化后,电转化导入P.pastoris/pEESB-10菌株,获得的转化子库中,筛选到P.pastoris/pEESB-11菌株,其胞外胆固醇酯酶活力为(27.4±0.8) U/mL,较P.pastoris/pEESB-10提高了10.8倍;较P.pastoris/pEESB-1菌株提高了161.2倍。不同优化策略提高ope基因表达水平效果如图3所示。在本研究分析的3种因子(基因拷贝数、信号肽类型和共表达伴侣蛋白)中,协助OPE形成二硫键的PDI蛋白的共表达,对OPE高效表达贡献最大;其次是分泌信号。由此说明,基因拷贝数提高到一定程度后,制约重组蛋白表达水平的因素主要是快速翻译合成的蛋白质能否正确折叠和分泌。
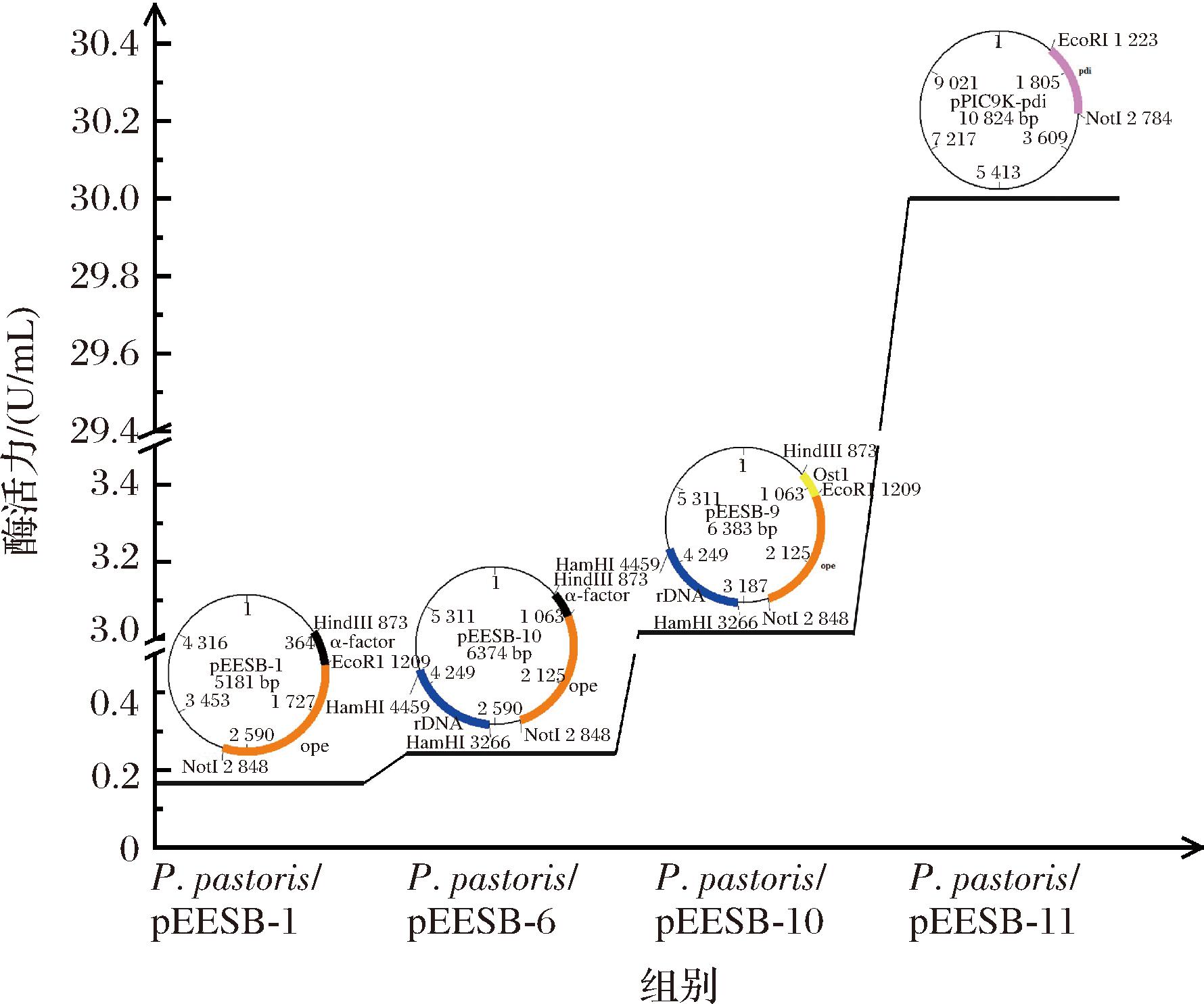
图3 不同P.pastoris重组菌株分泌胞外胆固醇酯酶的水平
Fig.3 Recombinant OPE yield from recombinant P.pastoris strains
利用Ni-NTA色谱柱对P.pastoris/pEESB-11发酵液中的外源蛋白进行分离纯化,获得一条76 kDa的单一条带的蛋白质(图4),与预期的OPE蛋白质分子大小一致。同时在P.pastoris/pEESB-1、P.pastoris/pEESB-6、P.pastoris/pEESB-10和P.pastoris/pEESB-11浓缩后的发酵液中,均存在对应的蛋白质条带(图4)。蛋白质电泳条带也显示,重组OPE可能存在糖基化修饰[11]。
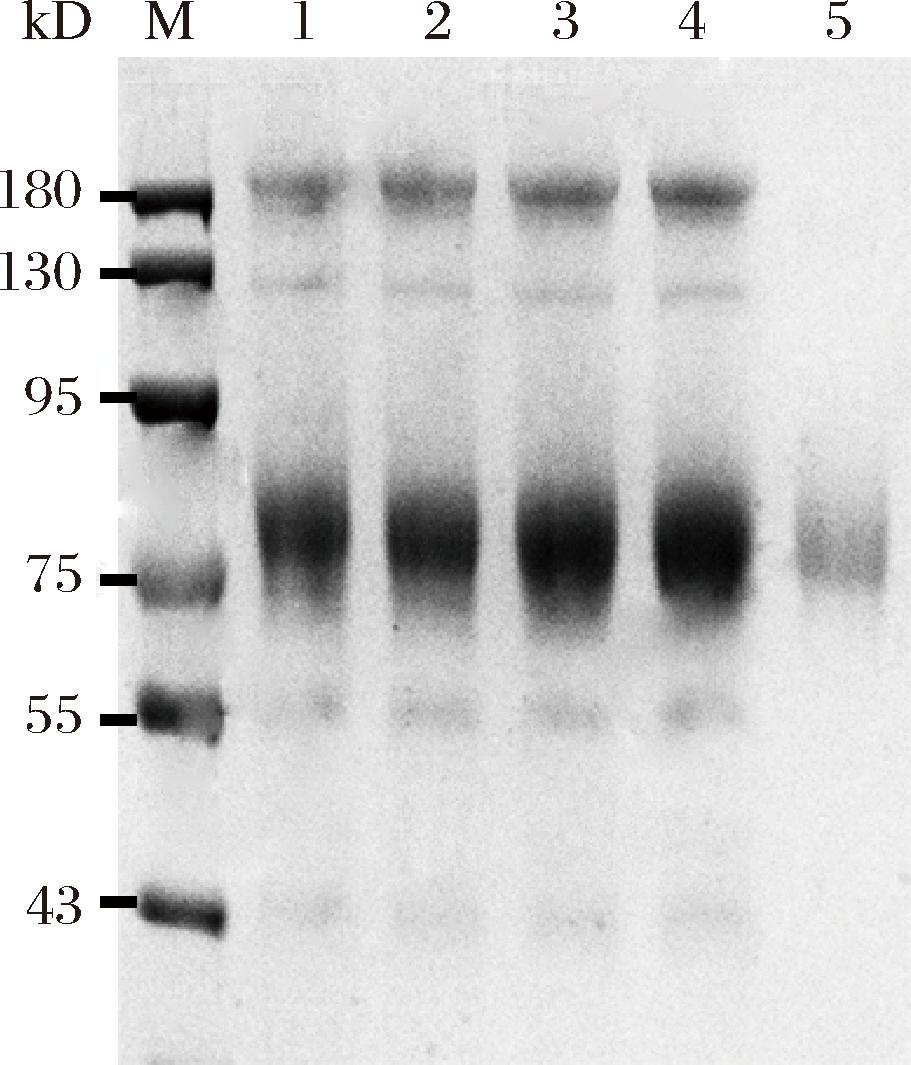
M-Protein Marker;泳道1-P.pastoris/pEESB-1发酵液;泳道2-P.pastoris/pEESB-6发酵液;泳道3-P.pastoris/pEESB-10发酵液;泳道4-P.pastoris/pEESB-11发酵液;泳道5-经Ni-NTA纯化后的重组OPE。
图4 不同菌株诱导表达重组OPE SDS-PAGE电泳
Fig.4 SDS-PAGE analysis of extracellular proteins from recombinant P.pastoris strains
2.4 发酵培养基中山梨醇浓度的优化
YEPS培养基中添加不同浓度山梨醇对P.pastoris/pEESB-11菌株产OPE的影响如图5所示。山梨醇浓度对P.pastoris/pEESB-11菌株产OPE有较大影响。当发酵培养基中添加体积分数为0.1%的山梨醇时,胞外重组OPE产量最高,达29.6 U/mL,随着山梨醇浓度进一步增加,胞外重组OPE产量降低。同时也发现不同山梨醇浓度下,重组P.pastoris/pEESB-11菌株的生物量变化不显著。CEDILLO等[11]也报道YEPS培养基中添加山梨醇作为第二碳源,有利于P.pastoris表达异源蛋白。
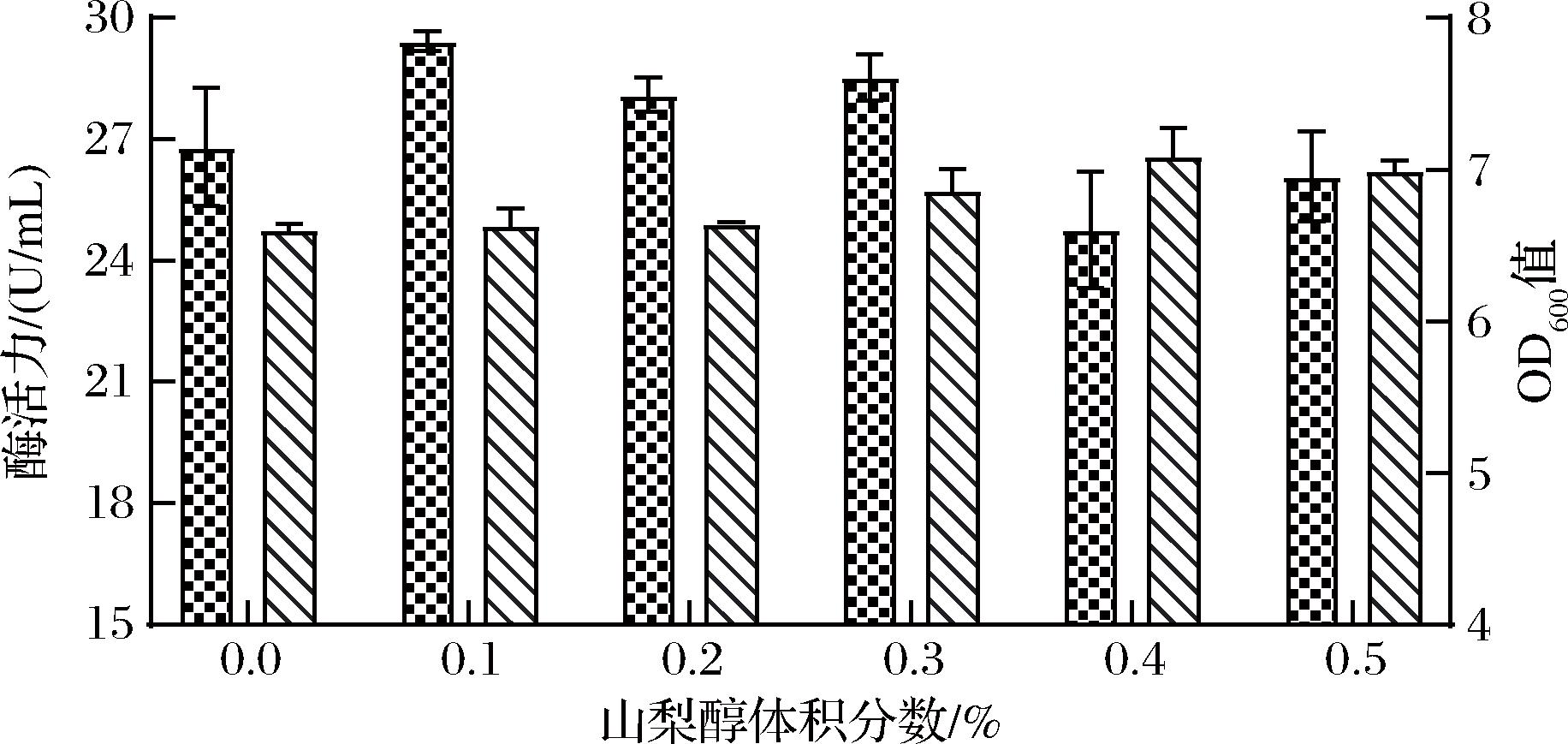
图5 培养基中添加不同浓度山梨醇对重组P.pastoris/pEESB-11菌株产OPE的影响
Fig.5 Effect of sorbitol concentrations in the fermentation medium on OPE yield of P.pastoris/pEESB-11 strain
3 讨论
基因拷贝数、信号肽类型和PDI对外源基因在P.pastoris中高效表达有重要的影响。一定范围内,提高外源基因的拷贝数,可有效提高外源基因的表达量;但对于分泌性蛋白质而言,胞内过表达的外源蛋白,如果不能及时分泌出细胞外,对宿主菌而言,将产生严重的代谢负担[22]。本实验中也发现,外源基因ope的同源重组臂由AOX(质粒pEESB-1)改变为rDNA(质粒pEESB-6)后,发酵液中胆固醇酯酶的酶活力虽有一定程度的提高(图3),但远不及已报道的蛋白酶和脂肪酶等外源基因在P.pastoris中的表达效果[23-24],推测重组P.pastoris/pEESB-11的分泌能力可能制约了重组OPE产量的增加;同时进一步分析发现,OPE蛋白分子内存在二硫键[9],推测ope基因在过表达合成重组OPE蛋白过程中,原始宿主菌的PDI不足以支持新合成的OPE正确折叠,进而影响其产量和活性。在进一步优化分泌信号和共表达PDI后,重组OPE的产量分别提高了9.1、10.8倍。
本研究中P.pastoris GS115转化子诱导外源基因表达使用的发酵培养基为YEPS培养基[14]。在P.pastoris GS115使用手册中,推荐使用的发酵培养基为BMGY(buffered methanol-complex medium)或BMMY(buffered glycerol-complex medium)。CEDILLO等[14]比较了重组P.pastoris GS115 利用YEPS培养基、含甘油的缓冲性完全培养基(BMGY)或含有甲醇的缓冲性基础培养基(BMMY)、含甘油的缓冲性基础培养基(buffered minimal glycerol medium, BMG)或含甲醇的缓冲性基础培养基(buffered minimal methanol medium, BMM)等培养基下,诱导表达重组OPE的发酵水平。实验结果表明,YEPS培养基更有利于重组P.pastoris诱导表达外源基因。从生产角度而言,YEPS培养基更具有价格优势。在使用过程中也观察到在实验室摇瓶发酵水平下,该培养基渗透压过高,不利于发酵液的后续浓缩。YEPS培养基还需要进一步优化,以更有利于重组P.pastoris 生产外源蛋白及其后续的加工。
本研究虽然一定程度上提高了重组OPE的产量,但离工业化生产还有一定的差距。后续研究过程,可进一步对OPE分泌过程中涉及的其他辅助蛋白(如内质网分拣、运输相关蛋白等,Endoplasmic reticulum-associated protein)与ope基因进行共表达[23],以期进一步提高其表达水平。胆固醇酯酶ope在P.pastoris 中的异源高效表达,有助于推动该酶实现工业化生产,并助力健康油脂产业的发展[25]。
[1] ZHENG M M, LIN Z, LIN S J, et al.Chemoenzymatic synthesis of steroidal products:Recent advances[J].European Journal of Organic Chemistry, 2024, 27(5):e202301066.
[2] PAVANI M, SINGHA P, DASH D R, et al.Novel encapsulation approaches for phytosterols and their importance in food products:A review[J].Journal of Food Process Engineering, 2022, 45(8):e14041.
[3] FENG S M, WANG L L, SHAO P, et al.A review on chemical and physical modifications of phytosterols and their influence on bioavailability and safety[J].Critical Reviews in Food Science and Nutrition, 2022, 62(20):5638-5657.
[4] WANG H B, ABE I.Recent developments in the enzymatic modifications of steroid scaffolds[J].Organic &Biomolecular Chemistry, 2024, 22(18):3559-3583.
[5] ZHENG M M, HUANG Q, HUANG F H, et al.Production of novel “functional oil” rich in diglycerides and phytosterol esters with “one-pot” enzymatic transesterification[J].Journal of Agricultural and Food Chemistry, 2014, 62(22):5142-5148.
[6] DA S PEREIRA A, DE SOUZA A H, FRAGA J L, et al.Lipases as effective green biocatalysts for phytosterol esters’ production:A review[J].Catalysts, 2022(1), 12:88.
[7] KONTKANEN H, TENKANEN M, FAGERSTRÖM R, et al.Characterisation of steryl esterase activities in commercial lipase preparations[J].Journal of Biotechnology, 2004, 108(1):51-59.
[8] NAIK S, BASU A, SAIKIA R, et al.Lipases for use in industrial biocatalysis:Specificity of selected structural groups of lipases[J].Journal of Molecular Catalysis B:Enzymatic, 2010, 65(1-4):18-23.
[9] GUTIÉRREZ-FERN NDEZ J, VAQUERO M E, PRIETO A, et al.Crystal structures of Ophiostoma piceae sterol esterase:Structural insights into activation mechanism and product release[J].Journal of Structural Biology, 2014, 187(3):215-222.
NDEZ J, VAQUERO M E, PRIETO A, et al.Crystal structures of Ophiostoma piceae sterol esterase:Structural insights into activation mechanism and product release[J].Journal of Structural Biology, 2014, 187(3):215-222.
[10] CALERO-RUEDA O, GUTIERREZ A, DEL RIO J C, et al.Hydrolysis of sterol esters by an esterase from Ophiostoma piceae:Application to pitch control in pulping of Eucalyptus globulus wood[J].International Journal of Biotechnology, 2004, 6(4):367-375.
[11] CEDILLO V B, PRIETO A, MART NEZ M J.Potential of Ophiostoma piceae sterol esterase for biotechnologically relevant hydrolysis reactions[J].Bioengineered, 2013, 4(4):249-253.
NEZ M J.Potential of Ophiostoma piceae sterol esterase for biotechnologically relevant hydrolysis reactions[J].Bioengineered, 2013, 4(4):249-253.
[12] MOLINA-GUTIÉRREZ M, HAKALIN N L S, RODR GUEZ-SANCHEZ L, et al.Green synthesis of β-sitostanol esters catalyzed by the versatile lipase/sterol esterase from Ophiostoma piceae[J].Food Chemistry, 2017, 221:1458-1465.
GUEZ-SANCHEZ L, et al.Green synthesis of β-sitostanol esters catalyzed by the versatile lipase/sterol esterase from Ophiostoma piceae[J].Food Chemistry, 2017, 221:1458-1465.
[13] LIN H, MU X D, HUANG J Z, et al.Comparative analysis of polyester-hydrolysis activity among three lipolytic enzymes[J].Journal of Chemical Technology &Biotechnology, 2019, 94(8):2522-2528.
[14] CEDILLO V B, PLOU F J, MART NEZ M J.Recombinant sterol esterase from Ophiostoma piceae:An improved biocatalyst expressed in Pichia pastoris[J].Microbial Cell Factories, 2012, 11:73.
NEZ M J.Recombinant sterol esterase from Ophiostoma piceae:An improved biocatalyst expressed in Pichia pastoris[J].Microbial Cell Factories, 2012, 11:73.
[15] THAK E J, YOO S J, MOON H Y, et al.Yeast synthetic biology for designed cell factories producing secretory recombinant proteins[J].FEMS Yeast Research, 2020, 20(2):foaa009.
[16] INGRAM Z, PATKAR A, OH D, et al.Overcoming obstacles in protein expression in the yeast Pichia pastoris:Interviews of leaders in the Pichia field[J].Pacific Journal of Health, 2021, 4(1):2.
[17] YANG X Q, ZHANG Y M.Expression of recombinant transglutaminase gene in Pichia pastoris and its uses in restructured meat products[J].Food Chemistry, 2019, 291:245-252.
[18] MATHEWS D H, DISNEY M D, CHILDS J L, et al.Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure[J].Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(19):7287-7292.
[19] XIONG A S, YAO Q H, PENG R H, et al.High level expression of a recombinant acid phytase gene in Pichia pastoris[J].Journal of Applied Microbiology, 2005, 98(2):418-428.
[20] BARRERO J J, CASLER J C, VALERO F, et al.An improved secretion signal enhances the secretion of model proteins from Pichia pastoris[J].Microbial Cell Factories, 2018, 17(1):161.
[21] AZA P, MOLPECERES G, DE SALAS F, et al.Design of an improved universal signal peptide based on the α-factor mating secretion signal for enzyme production in yeast[J].Cellular and Molecular Life Sciences, 2021, 78(7):3691-3707.
[22] WOLLBORN D, MUNKLER L P, HORSTMANN R, et al.Predicting high recombinant protein producer strains of Pichia pastoris MutS using the oxygen transfer rate as an indicator of metabolic burden[J].Scientific Reports, 2022, 12:11225.
[23] JIAO L C, ZHOU Q H, SU Z X, et al.High-level extracellular production of Rhizopus oryzae lipase in Pichia pastoris via a strategy combining optimization of gene-copy number with co-expression of ERAD-related proteins[J].Protein Expression and Purification, 2018, 147:1-12.
[24] RINNOFNER C, FELBER M, PICHLER H.Strains and molecular tools for recombinant protein production in Pichia pastoris[J].Methods in Molecular Biology, 2022, 2513:79-112.
[25] 李兆丰, 陈福生, 江正强, 等.优质食品酶的催化机制及在高端配料创制方面的应用基础[J].中国食品学报, 2024, 24(5):75-88.LI Z F, CHEN F S, JIANG Z Q, et al.The catalytic mechanism of high-quality food enzymes and their application in the creation of upscale ingredients[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(5):75-88.