沙门氏菌(Salmonella)是一种革兰氏阴性菌,该菌在显微镜下呈现为菌端钝圆的短杆菌,无荚膜,并且多数菌株具有周鞭毛和菌毛。沙门氏菌的最适生长温度为37 ℃,在55 ℃下可部分存活60 min,而在75 ℃水中1 min即可被杀死。此外,它对酸和低温具有一定的耐受性,能够在pH 4.0~6.5的酸性环境及冰箱内长期生存[1]。沙门氏菌主要通过食源性传播,包括感染动物的肉类、奶制品、蔬菜水果等。感染后,患者可能会出现出血性腹泻、腹部绞痛等症状,病程一般为2~9 d,严重时可能影响中枢神经系统或引发急性胃肠炎等疾病,甚至可能导致肠坏死、伤寒、败血症及肠外灶性感染等严重后果[2]。
沙门氏菌作为一种高度致病性的食源性病原菌,其内化机制的研究在食品安全领域中具有举足轻重的地位。沙门氏菌内化机制主要探讨病原菌如何进入并寄生于宿主细胞,从而引发感染的过程。这一过程不仅涉及病原菌与宿主细胞之间的相互作用,还关系到病原菌如何在宿主体内定植、繁殖并传播。主动内化涉及沙门氏菌的鞭毛运动和趋化作用[3]。沙门氏菌能够黏附于植物表面,导致新鲜农产品受到污染;除了黏附外,沙门氏菌还可以通过根系和种子的吸收在土壤中被动内化进入植物组织[4]。尽管在新鲜农产品的加工过程中,消毒剂处理能够有效去除表面黏附的沙门氏菌,但对于已内化的沙门氏菌则难以清除[5]。因此,深入研究沙门氏菌的内化机制,有助于我们更全面地理解该病原菌的致病机理,为制定有效的防控策略提供科学依据,并有助于追踪食品中的污染源。
随着人们生活水平的提高和健康意识的增强,食品安全问题已经成为社会关注的焦点,食品中污染源的追踪与防控策略的紧迫性愈发凸显。食品污染不仅可能来源于原料采购、生产加工、贮存运输等多个环节,还可能受到环境污染、微生物污染等多种因素的影响。一旦食品发生污染事件,迅速准确地定位污染源是防止污染扩散、保障公众健康的关键。通过污染源追踪,可以及时发现并消除潜在的安全隐患,防止类似事件再次发生。同时,污染源追踪也是食品安全监管体系中的重要组成部分,有助于提高食品安全管理的效率和水平。
1 沙门氏菌在果蔬中的内化机制研究
1.1 内化途径
1.1.1 气孔
在众多内化途径中,气孔作为一种关键的入侵通道,通常存在于植物叶片和某些果实的表皮上,是植物与外界进行气体交换和水分散失的主要通道。然而,这些微小的开口也成为了沙门氏菌入侵的潜在途径。沙门氏菌能够利用气孔作为入侵的门户,进入植物组织内部,进而在植物体内定植和繁殖,并可能通过食物链传递给人类和动物,导致一系列健康问题。研究表明,肠道沙门氏菌(Salmonella enterica)聚集在开放的气孔附近并侵入生菜叶片组织[3]。在内化过程中,气孔被用作沙门氏菌内化的营养来源,并为其提供保护性生态位。沙门氏菌通过内化到叶片的气孔及内部隔室中,可以有效地保护自己免受氯或臭氧等消毒剂的侵害[5]。
1.1.2 根部组织
根部组织作为植物与外界环境直接接触的界面,同样构成了沙门氏菌入侵的重要途径。新鲜农产品植物的根部分泌物营养丰富,可将肠道沙门氏菌吸引到植物的根部[6]。沙门氏菌能够利用根部组织中的微小裂缝、伤口或自然开口(如根毛等)作为入侵的切入点;KLERKS等[6]观察到都柏林沙门氏菌(Salmonella enterica serovar Dublin)能够通过侧根周缘穿过皮层和表皮,聚集在根部的营养丰富区域。
1.1.3 损伤部位
在沙门氏菌对植物的入侵过程中,损伤部位常常成为其重要的突破口。这些损伤可能由物理因素(如机械损伤、动物啃食等)、化学因素(如农药使用不当造成的伤害)或生物因素(如其他微生物的侵害)引起。一旦植物表面出现损伤,原本受到保护的内部组织便暴露于外界环境中,从而为病原菌的入侵提供了机会[7]。先前的一项研究表明,与未受损的叶片相比,成年西部花蓟马(Frankliniella occidentalis)侵染的明显受损的番茄叶片上,肠道沙门氏菌的内化量显著更高[8]。
1.2 内化因素
1.2.1 细菌学
1.2.1.1 鞭毛
在沙门氏菌的内化过程中,鞭毛作为一种关键的细菌学因素,扮演着至关重要的角色。鞭毛是细菌细胞表面的一种细长、波状的附属结构,由蛋白质构成,并具备一定的运动能力。在沙门氏菌中,鞭毛不仅赋予细菌在液体环境中的游动能力,还参与了细菌与宿主细胞之间的相互作用,尤其是在内化过程中发挥着重要作用。鞭毛通过其旋转运动,推动细菌向宿主细胞表面移动,并帮助细菌在细胞表面寻找合适的附着位点。KROUPITSKI等[3]报道了肠道沙门氏菌在生菜叶片内化过程中与趋化性和运动性的关联。研究表明,相较于野生株,运动缺陷fliGHI突变体和趋化性缺陷cheY突变体在进入生菜叶片的能力上受到显著抑制。此外,ZARKANI等[9]通过混合感染实验并计算相应的竞争指数(competition indexes,CIs),检验了野生型鼠伤寒沙门氏菌(Salmonella Typhimurium, S.Typhimurium)菌株在番茄植株中的持久性是否优于鼠伤寒沙门氏菌ΔfljBΔfliC双突变体。结果显示,鼠伤寒沙门氏菌ΔfljBΔfliC双突变体与野生型菌株混合感染后,在7 dpi和14 dpi时计算出的CI均与1.0无显著差异,这表明沙门氏菌在植物中的内化和定植能力并不依赖于鞭毛的功能或鞭毛蛋白的表达。
1.2.1.2 菌毛
菌毛是沙门氏菌表面一种重要的附属结构,通常比鞭毛更短、更直,且数量众多,紧密附着于细菌细胞壁上,形成一层致密的覆盖层。菌毛能够特异性地识别宿主细胞表面的受体,并通过与这些受体的结合,使细菌紧密附着于细胞表面。这种紧密的附着不仅为细菌提供了稳定的立足点,还为后续的内化步骤创造了有利条件。ELPERS等[10]利用Tet-on系统揭示了肠道沙门氏菌Saf菌毛及其分泌一型分泌系统的BapA对玉米色拉叶的附着力,从而为其入侵提供了更为有利的条件。此外,肠道沙门氏菌中agfA和agfB突变株对植物组织的附着产生了不同的影响。与野生型相比,仅肠道沙门氏菌菌毛(Curli)成核剂agfB突变株在附着或定居苜蓿芽的能力上有所降低[11]。
1.2.1.3 三型分泌系统
三型分泌系统(type Ⅲ secretion system, T3SS)在沙门氏菌对宿主细胞环境的感知及相关内化基因的表达调控中发挥着重要作用。三型分泌系统由多组分蛋白复合体构成的跨膜通道,能够通过分泌蛋白将毒力蛋白直接注入宿主细胞,从而发挥其致病作用,这一复杂的分子装置广泛存在于许多革兰阴性致病菌中。在沙门氏菌内化菠菜的过程中,仅观察到与T3SS相关基因(如invA、prgH、avrA、ssrB)的上调,而在绿苋菜中则未见此现象。经过5 ℃适应后,菠菜中内化沙门氏菌的prgH转录水平发生了显著变化(FC>60~230),同时有助于pH平衡的赖氨酸转运蛋白cadB在内化细菌中也表现出上调(FC~18)。在20 ℃适应后,菠菜中内化沙门氏菌的spvR转录水平变化最大(FC~1200);而在绿苋菜中,内化沙门氏菌的cadB (FC~300)转录水平也显示出显著变化[12]。在新港沙门氏菌(Salmonella Newport, S.Newport)和鼠伤寒沙门氏菌内化至樱桃番茄的过程中,诱导T3SS的SPI-1基因表达及参与T3SS组装的hilD仅对新港沙门氏菌的初始内化至关重要[13]。
1.2.1.4 其他因素
沙门氏菌能否内化到植物组织中及其存活率还与其代谢状态、适应性以及周围环境相关的外在因素有关[14]。在对数生长期,沙门氏菌通常表现出较强的侵袭能力,这主要归因于其较高的代谢活性和增殖速率。相较之下,处于静止期或衰亡期的沙门氏菌其侵袭能力较弱,因其代谢活动减缓,细胞功能受损[15]。研究表明肠道沙门氏菌在绿叶蔬菜中的内化受到初始接种水平和细菌对轻度胁迫的预适应的影响,在亚致死性干燥、氧化、热休克和酸胁迫下的预暴露,能够促进病原体在菠菜中的内化,与非适应性肠道沙门氏菌相比,预先将肠道沙门氏菌暴露于42 ℃下干燥1.5 h,显著增强了其内化效率[16]。在寒冷(4 ℃,7 d)和饥饿压力(0.85% W/V NaCl,pH 6.6)的预适应条件下,沙门氏菌在芝麻菜、菊苣和菠菜中的内化率相似。此外,在5 ℃和20 ℃下贮存2 h的内化率接近于非适应性病原体。与内化亚群相比,沙门氏菌在绿叶蔬菜上经过5 ℃和20 ℃下贮存2 h和48 h的适应后,诱导了细菌的耐酸性,从而增强了其在pH 2.7下的存活率,存活率为1.5~2.0 lg CFU/g。沙门氏菌在蔬菜提取物中的适应性使其对酸的敏感性增强,而假单胞菌、酵母菌、霉菌、肠杆菌和乳酸菌对生物体的耐酸性影响则相对有限[12]。
1.2.2 植物因素
植物的生理状态是影响病原菌入侵的重要因素。例如,处于生长期或营养充足的植物,其细胞代谢活跃,细胞壁结构相对完整,因此对病原菌的防御能力较强。相反,处于衰老期或营养不足的植物,其细胞代谢减缓,细胞壁结构可能受到损害,从而更容易受到病原菌的侵染。XYLIA等[17]发现,与处于生长期的生菜相比,处于衰老期的生菜更容易受到肠道沙门氏菌内化的影响。此外,植物的组织结构对病原菌的内化过程也具有重要影响。不同植物种类和品种的组织结构存在差异,例如叶片的厚度、气孔的大小和分布等,这些因素都会影响病原菌的入侵路径和内化效率,研究发现生菜叶子比玉米沙拉叶子较易被肠道沙门氏菌内化[4]。最后,植物的防御机制是阻止病原菌入侵的重要防线。植物能够合成多种抗病物质,如酚类化合物和病程相关蛋白等,这些物质能够抑制病原菌的生长和繁殖,甚至直接杀死病原菌[17]。同时,植物还能够通过诱导抗性等方式,提高自身对病原菌的防御能力。KLERKS等[6]通过cDNA-AFLP分析发现,生菜(Lactuca sativa)在被沙门氏菌定植后存在SA定向防御机制,研究表明接种和未接种沙门氏菌血清型的植物之间存在基因差异表达,主要涉及植物胁迫相关基因(如DAD1、PR1、PR4和PR5)、NAM样蛋白基因、OEE3基因、PR1基因、Sec1家族转运蛋白基因、Sec6转运蛋白基因以及bHLH016转录因子基因。
1.2.3 环境因素
环境因素如接种量、温度、土壤类型、雾、光照及pH值等对沙门氏菌的内化过程具有显著影响。这些因素能够影响沙门氏菌的存活、增殖及运动能力,从而间接影响其对宿主细胞的入侵能力。研究表明,沙门氏菌在菠菜、芝麻菜和生菜上的内化量随着接种剂量的增加而显著增加[16]。与在7 ℃贮存的样品相比,在25 ℃下贮存的样品中观察到更高的沙门氏菌浓度[12]。此外,土壤类型也对细菌的内化产生影响,生长在沙质土壤中的植物比生长在肥沃土壤中的植物更易被内化。微生物群分析揭示了不同土壤类型之间细菌组成的显著差异[4]。与未进行喷雾处理的对照组相比,接受喷雾处理组的沙门氏菌内化量增加了10%~30%[18]。KROUPITSKI等[3]发现,光照能够诱导肠道沙门氏菌内化,在光照条件下,GFP标记的肠道沙门氏菌与卷心生菜叶孵育时,细菌聚集在开放气孔附近并侵入叶内组织。与最佳孵化条件相比,肠道沙门氏菌在pH值为5.0的营养水培溶液中生长最慢,这表明与较高pH值的不同营养物质相比,病原体在较低pH值下需要更多时间来适应新的环境条件[17]。
1.3 内化策略
为了克服蔬菜内部组织的物理和化学屏障,沙门氏菌采用了一系列复杂且精细的策略。首先,该病原菌能够利用鞭毛的运动能力,在蔬菜表面进行定向移动以寻找合适的入侵点[3]。这些入侵点可能包括自然开口、伤口或微裂缝等,为病原菌提供了进入蔬菜内部的机会。尽管蔬菜内部存在一系列受体用于识别细菌外部的常见成分并能够激活一系列反应以阻止细菌入侵[19-20],沙门氏菌仍然能够通过T3SS将效应蛋白分泌到宿主细胞中,从而抑制蔬菜的正常先天免疫反应,促进病原菌在宿主内的定植[21]。在拟南芥和红豆的实验中也表明,沙门氏菌的存在会激活先天免疫反应,而消除作为该反应一部分的信号通路则会导致植物体内细菌数量的增加[22-23]。此外,沙门氏菌能够分泌一系列酶类,如细胞壁降解酶,这些酶能够破坏蔬菜细胞壁的主要成分,如纤维素和果胶,从而软化细胞壁并为其入侵开辟道路。通过这些酶的作用,病原菌能够逐步侵蚀蔬菜组织,使其内部暴露并易于感染[24]。沙门氏菌可能利用蔬菜体内的水分和养分流动作为其入侵的辅助途径。在蔬菜生长过程中,水分和养分通过植物的输导组织进行运输。病原菌能够借助这些运输通道,随着水分和养分的流动,进入蔬菜的更深层组织。研究表明,当沙拉叶受损时,释放的微量汁液使沙门氏菌的生长速度比未补充对照组和类宿主组增加了110%[25]。最后,沙门氏菌还可能通过与其他微生物的相互作用来增强其对蔬菜组织的入侵能力。例如,一些微生物可能通过降解细胞壁聚合物或诱导糖的分泌以提供碳源,为病原菌的入侵创造更有利的条件[24,26]。
1.4 内化过程
为了深入探讨沙门氏菌如何克服植物内部组织的障碍并成功内化至蔬菜内部,科学家们利用激光共聚焦显微镜观察沙门氏菌内化过程[3,5]。在实验中,研究人员将感染沙门氏菌的蔬菜样本置于激光共聚焦显微镜下,并利用超高速双转盘系统对样本进行快速扫描。通过调整激光的波长和强度,以及优化成像参数,研究人员能够清晰地观察到病原菌在蔬菜细胞表面的附着、入侵等内化过程。为确定肠道沙门氏菌是否能够在叶表面向气孔移动并发生内化,KROUPITSKI等[3]通过激光共聚焦显微镜以每0.44 s捕获1帧的频率,观察与GFP标记的肠道沙门氏菌共同孵育的生菜中的细菌运动。当实验在光照条件下进行而非在黑暗中时,观察到肠道沙门氏菌细胞向气孔下腔移动并最终消失。
1.5 内化后存活规律与机制
沙门氏菌一旦成功内化至蔬菜细胞内部,其存活机制和规律变得异常复杂且至关重要。内化后的病原菌需应对宿主细胞所带来的多重挑战,包括细胞内的免疫应答、营养物质获取的困难以及代谢环境的适应问题[12,27]。为了在这些不利条件下生存,沙门氏菌利用其独特的代谢途径和适应机制,例如调整代谢速率和合成特定的代谢酶,以适应细胞内的环境[4]。同时,沙门氏菌积极寻找并利用细胞内的糖类、氨基酸、核苷酸等营养物质进行生长和繁殖,通过调节代谢途径来优化营养物质的利用效率[25]。进入细胞后,沙门氏菌还可能通过修改植物细胞的代谢途径和信号传导系统,操纵植物细胞的生理功能,使其更有利于病原菌的生存和繁殖[19-20]。为了逃避或抵抗宿主细胞的免疫应答攻击,沙门氏菌还采取合成抗毒素、抑制免疫细胞活性等策略,从而在细胞内建立稳定的感染灶,持续对宿主细胞造成损害[28]。JECHALKE等[4]通过在鼠伤寒沙门氏菌14 028 s诱导生菜的防御反应中检测到几个与防御机制相关的基因,这些基因编码金属内蛋白酶1、发病机制相关蛋白PR-1、葡聚糖内切-1,3-β-葡萄糖苷酶和乙烯反应性转录因子1B。
2 食品中沙门氏菌污染源的追踪
2.1 污染源追踪的方法与技术
2.1.1 微生物学检测方法
微生物学检测方法通常包括样品的采集与处理、沙门氏菌的分离与培养、鉴定与确认等多个步骤。通过沙门氏菌增菌培养、离心沉淀等,可以提高沙门氏菌的检出率。为了进一步确认病原菌的种类和亚型,还需要进行更深入的血清学试验。血清学试验利用特异性抗体与病原菌表面的抗原结合,实现对病原菌的准确识别。
2.1.2 分子生物学溯源技术
分子生物学溯源技术是一种基于分子标记和遗传信息来追踪物种、物质或生物体起源和历史的科学方法。它通过分析物质中的DNA、RNA、蛋白质或其他生物分子,利用PCR、DNA测序、比较基因组学等先进的分子生物学技术,可以确定它们的起源、进化关系、迁移路径以及变化过程。表1列举了针对食品中沙门氏菌的常见分子生物学检测技术,并在表2中进一步总结了基于抗体和核酸的生物传感方法进行沙门氏菌检测。
表1 针对食品中沙门氏菌的分子生物学检测技术
Table 1 Molecular biology techniques for the detection of Salmonella in foodstuffs

技术优点缺点PCR灵敏度高、特异性强、操作简便易假阳性核酸探针检测技术特异性强复杂繁琐、费用较高基因芯片检测技术强特异性、高灵敏度、高通量检测、简便快速、重复性好仪器价格高昂酶联免疫吸附法(enzymelinkedimmunosorbentassay,ELISA)特异性强、灵敏度高灵敏度低胶体金免疫层析法灵敏度高、特异性强、操作简便、结果准确特异性要求高、样品处理复杂免疫磁珠分离法(immunomagneticseparation,IMS)快速灵敏易受抗体质量影响环介导恒温扩增技术(loop-mediatedisothermalamplification,LAMP)检出限低、特异性好引物要求高、低通量核酸杂交技术特异性好稳定性差基因组测序技术精确度高、信息量丰富繁琐周期长
表2 基于抗体和核酸的生物传感方法检测沙门氏菌
Table 2 Antibody and nucleic acid based biosensing methods for the detection of Salmonella
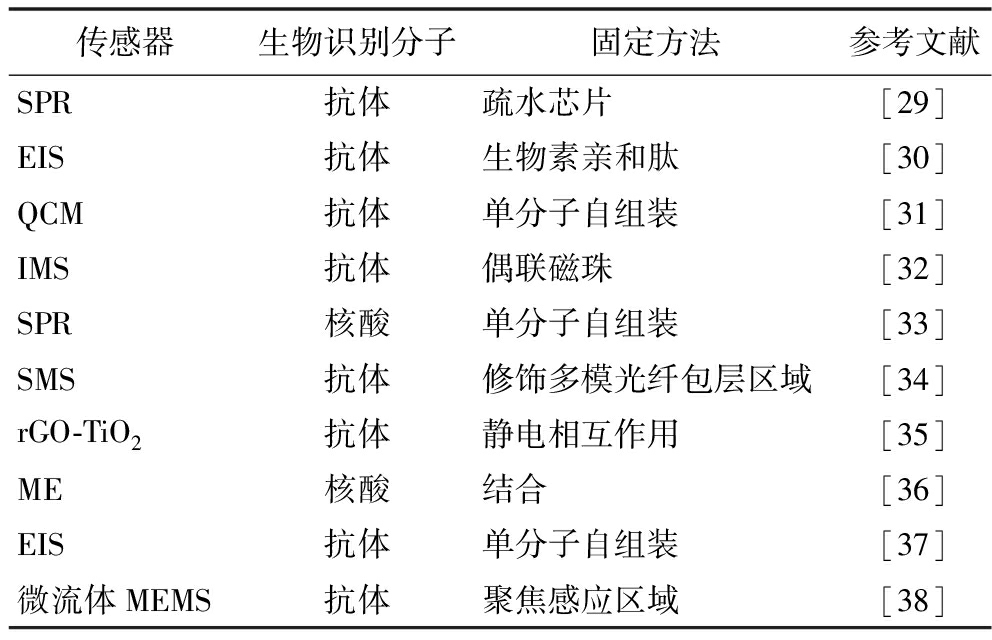
传感器生物识别分子固定方法参考文献SPR抗体疏水芯片[29]EIS抗体生物素亲和肽[30]QCM抗体单分子自组装[31]IMS抗体偶联磁珠[32]SPR核酸单分子自组装[33]SMS抗体修饰多模光纤包层区域[34]rGO-TiO2抗体静电相互作用[35]ME核酸结合[36]EIS抗体单分子自组装[37]微流体MEMS抗体聚焦感应区域[38]
2.2 污染源追踪
2.2.1 实际食品污染事件中的追踪过程
在实际食品污染事件中,追踪沙门氏菌污染源的过程通常涉及多个环节和部门之间的紧密协作。首先,当食品安全事件被报告后,相关部门会立即启动应急预案,组织专业团队进行现场调查和样品采集。这些样品可能包括疑似污染的食品、食品原料、生产环境以及相关人员的生物样本等。随后,通过微生物学检测和分子生物学溯源技术,对采集的样品进行病原菌的分离、鉴定和溯源分析。在溯源过程中,可能会结合流行病学调查、生产流程分析等手段,逐步缩小污染源的范围。最终,通过综合分析各方面的信息和数据,确定污染源的具体位置和原因。
2.2.2 追踪结果的验证与应用
追踪结果的验证是确保溯源准确性的重要环节。在确定了污染源后,相关部门会采取一系列措施对追踪结果进行验证,包括重新采集样品进行复核检测、对溯源过程进行回顾性评估等。通过这些验证步骤,可以确保溯源结果的可靠性和准确性。一旦验证确认,追踪结果将被广泛应用于食品安全事件的处置和后续管理。这包括及时采取措施消除污染源、防止污染扩散、对受污染食品进行召回或销毁等。同时,追踪结果还可以为食品安全监管提供重要依据,帮助相关部门制定更加有效的食品安全防控策略和措施,提高食品安全水平,保障公众健康。
3 沙门氏菌污染的防控策略
3.1 预防措施
为了有效防控沙门氏菌的污染,在生产环节,应确保原料的清洁度和安全性,严格筛选和检验,避免使用受污染的原料。加工过程中,要实施严格的卫生操作规程以及保持生产环境的清洁和卫生。贮存环节则要求控制适宜的温度和湿度,防止食品腐败变质和病原菌滋生。在运输过程中,应确保食品包装完整、温度适宜,并避免交叉污染。
3.2 控制措施
在防控沙门氏菌污染的过程中,通过建立完善的食品安全监测体系,包括定期的抽样检测和风险评估,以便迅速识别出潜在的污染源。一旦发现食品受到沙门氏菌的污染,应立即召回受污染产品、销毁不合格批次。在防控沙门氏菌污染的策略中,对隔离的污染源进行彻底的消毒处理,确保能够杀灭沙门氏菌等病原体。除了使用消毒剂等化学处理方法外,传统的热杀菌技术因其所需的高温可能导致食品的色泽、风味和营养成分发生变化和损失。因此,非热杀菌技术因其节能、高效、安全、经济的特点,以及能够最大限度地保持食品的天然色、香、味,逐渐受到重视。表3列举了针对食品中沙门氏菌的常见非热杀菌技术。
表3 针对食品中沙门氏菌的非热杀菌技术
Table 3 Non-thermal sterilization of Salmonella in foodstuffs
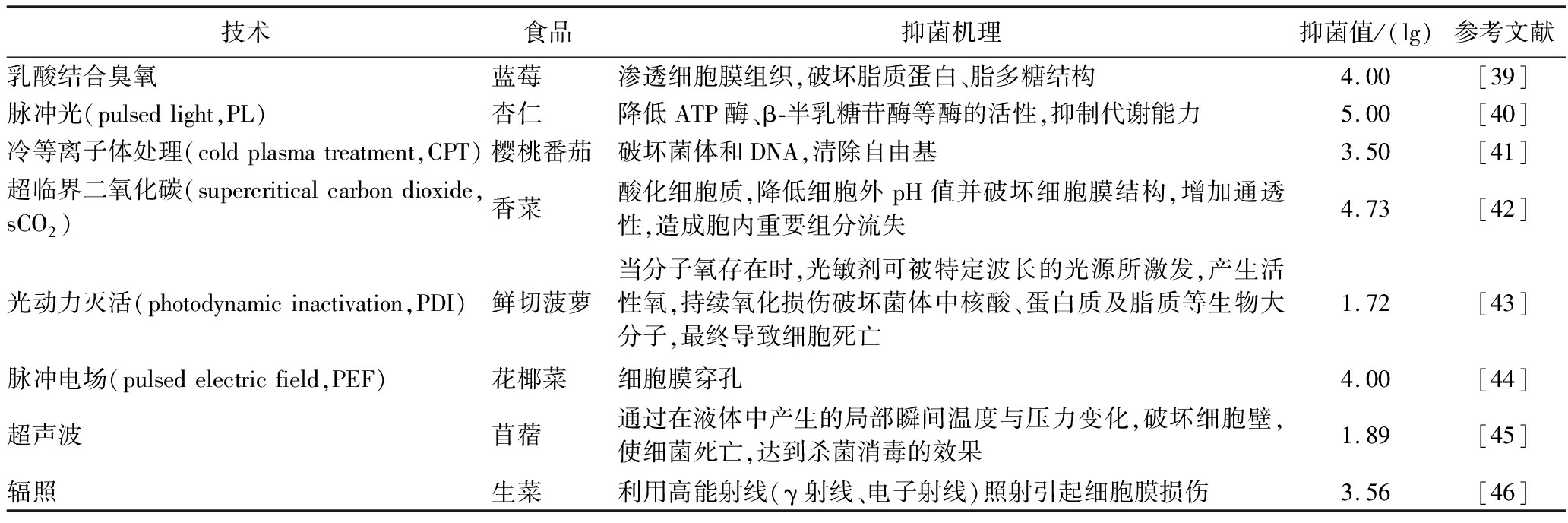
技术食品抑菌机理抑菌值/(lg)参考文献乳酸结合臭氧蓝莓渗透细胞膜组织,破坏脂质蛋白、脂多糖结构4.00[39]脉冲光(pulsedlight,PL)杏仁降低ATP酶、β-半乳糖苷酶等酶的活性,抑制代谢能力5.00[40]冷等离子体处理(coldplasmatreatment,CPT)樱桃番茄破坏菌体和DNA,清除自由基3.50[41]超临界二氧化碳(supercriticalcarbondioxide,sCO2)香菜酸化细胞质,降低细胞外pH值并破坏细胞膜结构,增加通透性,造成胞内重要组分流失4.73[42]光动力灭活(photodynamicinactivation,PDI)鲜切菠萝当分子氧存在时,光敏剂可被特定波长的光源所激发,产生活性氧,持续氧化损伤破坏菌体中核酸、蛋白质及脂质等生物大分子,最终导致细胞死亡1.72[43]脉冲电场(pulsedelectricfield,PEF)花椰菜细胞膜穿孔4.00[44]超声波苜蓿通过在液体中产生的局部瞬间温度与压力变化,破坏细胞壁,使细菌死亡,达到杀菌消毒的效果1.89[45]辐照生菜利用高能射线(γ射线、电子射线)照射引起细胞膜损伤3.56[46]
3.3 应急响应与危机管理
为了有效应对沙门氏菌等食品安全事件,必须建立完善的食品安全应急响应机制,包括明确的应急组织架构、职责分工和操作流程。应急响应机制应涵盖从事件报告、初步调查、风险评估、到应急处置和后续跟踪的全过程,确保每一步骤都有明确的指导和规范。
同时,还需加强应急演练和培训,提高相关人员的应急响应能力和协作水平,确保在食品安全危机面前能够迅速、准确地采取行动,最大限度地减少危害和损失。形成政府、企业和公众三方良性互动、共同维护食品安全的良好局面。
4 结论
沙门氏菌作为一种高度危险的食源性致病菌,其内化机制复杂,能够通过多种途径进入食品链,对公众健康构成严重威胁。本文探讨了沙门氏菌通过气孔、根部组织、损伤部位等途径内化进入果蔬的机制,以及细菌学、植物和环境相关因素对其内化过程的影响。同时,探讨了内化策略、内化过程和内化后的存活规律与机制。尤其对食品中污染源的追踪与防控也至关重要,需要建立完善的监测体系和追溯机制,确保及时发现并隔离污染源。在防控策略上,应注重加强源头控制,提高食品加工和储存环节的卫生标准,同时加强政府部门、企业和公众之间的沟通与协作,形成合力,共同应对食品安全挑战。
[1] 朱奇, 陆斌兴, 覃有泉, 等.沙门氏菌生物学研究进展[J].疾病监测与控制, 2015, 9(7):474-478.ZHU Q, LU B X, QIN Y Q, et al.Research progress on biological Salmonella enteric[J].Journal of Diseases Monitor &Control, 2015, 9(7):474-478.
[2] GAJRAJ R, POORANSINGH S, HAWKER J I, et al.Multiple outbreaks of Salmonella braenderup associated with consumption of iceberg lettuce[J].International Journal of Environmental Health Research, 2012, 22(2):150-155.
[3] KROUPITSKI Y, GOLBERG D, BELAUSOV E, et al.Internalization of Salmonella enterica in leaves is induced by light and involves chemotaxis and penetration through open stomata[J].Applied and Environmental Microbiology, 2009, 75(19):6076-6086.
[4] JECHALKE S, SCHIERSTAEDT J, BECKER M, et al.Salmonella establishment in agricultural soil and colonization of crop plants depend on soil type and plant species[J].Frontiers in Microbiology, 2019, 10:967.
[5] CHAHAR M, KROUPITSKI Y, GOLLOP R, et al.Determination of Salmonella enterica leaf internalization varies substantially according to the method and conditions used to assess bacterial localization[J].Frontiers in Microbiology, 2021, 12:622068.
[6] KLERKS M M, VAN GENT-PELZER M, FRANZ E, et al.Physiological and molecular responses of Lactuca sativa to colonization by Salmonella enterica serovar Dublin[J].Applied and Environmental Microbiology, 2007, 73(15):4905-4914.
[7] SAVATIN D V, GRAMEGNA G, MODESTI V, et al.Wounding in the plant tissue:The defense of a dangerous passage[J].Frontiers in Plant Science, 2014, 5:470.
[8] HARROD V L, GROVES R L, MAURICE M A, et al.Frankliniella occidentalis facilitate Salmonella enterica survival in the phyllosphere[J].PLoS One, 2021, 16(2):e0247325.
[9] ZARKANI A A, L PEZ-PAG
PEZ-PAG N N, GRIMM M, et al.Salmonella heterogeneously expresses flagellin during colonization of plants[J].Microorganisms, 2020, 8(6):815.
N N, GRIMM M, et al.Salmonella heterogeneously expresses flagellin during colonization of plants[J].Microorganisms, 2020, 8(6):815.
[10] ELPERS L, KRETZSCHMAR J, NUCCIO S P, et al.Factors required for adhesion of Salmonella enterica serovar typhimurium to corn salad (Valerianella locusta)[J].Applied and Environmental Microbiology, 2020, 86(8):e02757-19.
[11] BARAK J D, GORSKI L, NARAGHI-ARANI P, et al.Salmonella enterica virulence genes are required for bacterial attachment to plant tissue[J].Applied and Environmental Microbiology, 2005, 71(10):5685-5691.
[12] GRIVOKOSTOPOULOS N C, MAKARITI I P, HILAJ N, et al.Internalization of Salmonella in leafy greens and impact on acid tolerance[J].Applied and Environmental Microbiology, 2022, 88(6):e0224921.
[13] ARISTA-REGALADO A D, BARBA-LE N J, BUSTAMANTE V H, et al.hilD is required for the active internalization of Salmonella newport into cherry tomatoes[J].Journal of Food Protection, 2023, 86(5):100085.
N J, BUSTAMANTE V H, et al.hilD is required for the active internalization of Salmonella newport into cherry tomatoes[J].Journal of Food Protection, 2023, 86(5):100085.
[14] DEERING A J, MAUER L J, PRUITT R E.Internalization of E.coli O157:H7 and Salmonella spp.in plants:A review[J].Food Research International, 2012, 45(2):567-575.
[15] YE B N, HE S K, ZHOU X J, et al.Response to acid adaptation in Salmonella enterica serovar enteritidis[J].Journal of Food Science, 2019, 84(3):599-605.
[16] GRIVOKOSTOPOULOS N C, MAKARITI I P, TSADARIS S, et al.Impact of population density and stress adaptation on the internalization of Salmonella in leafy greens[J].Food Microbiology, 2022, 106:104053.
[17] XYLIA P, CHRYSARGYRIS A, BOTSARIS G, et al.Salmonella Enteritidis survival in different temperatures and nutrient solution pH levels in hydroponically grown lettuce[J].Food Microbiology, 2022, 102:103898.
[18] KIM J, PARK S, LEE J, et al.Internalization of Salmonella in leafy vegetables during postharvest conditions[J].Foods, 2023, 12(16):3106.
[19] NÜRNBERGER T, BRUNNER F, KEMMERLING B, et al.Innate immunity in plants and animals:Striking similarities and obvious differences[J].Immunological Reviews, 2004, 198(1):249-266.
[20] AUSUBEL F M.Are innate immune signaling pathways in plants and animals conserved?[J].Nature Immunology, 2005, 6(10):973-979.
[21] HOTINGER J A, PENDERGRASS H A, MAY A E.Molecular targets and strategies for inhibition of the bacterial type Ⅲ secretion system (T3SS);inhibitors directly binding to T3SS components[J].Biomolecules, 2021, 11(2):316.
[22] INIGUEZ A L, DONG Y M, CARTER H D, et al.Regulation of enteric endophytic bacterial colonization by plant defenses[J].Molecular Plant-Microbe Interactions, 2005, 18(2):169-178.
[23] SCHIKORA A, CARRERI A, CHARPENTIER E, et al.The dark side of the salad:Salmonella typhimurium overcomes the innate immune response of Arabidopsis thaliana and shows an endopathogenic lifestyle[J].PLoS One, 2008, 3(5):e2279.
[24] TAN M S, RAHMAN S, DYKES G A.Relationship between cell concentration and Salmonella attachment to plant cell walls[J].Food Control, 2016, 67:119-126.
[25] KOUKKIDIS G, HAIGH R, ALLCOCK N, et al.Salad leaf juices enhance Salmonella growth, colonization of fresh produce, and virulence[J].Applied and Environmental Microbiology, 2016, 83(1):e02416-16.
[26] BRANDL M T.Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce[J].Applied and Environmental Microbiology, 2008, 74(17):5285-5289.
[27] MENG F H, ALTIER C, MARTIN G B.Salmonella colonization activates the plant immune system and benefits from association with plant pathogenic bacteria[J].Environmental Microbiology, 2013, 15(9):2418-2430.
[28] YUSOF T Y, ONG E B B, TEH A H.RelEB3 toxin-antitoxin system of Salmonella Typhimurium with a ribosome-independent toxin and a mutated non-neutralising antitoxin[J].International Journal of Biological Macromolecules, 2022, 219:1080-1086.
[29] MAZUMDAR S D, BARLEN B, KRAMER T, et al.A rapid serological assay for prediction of Salmonella infection status in slaughter pigs using surface plasmon resonance[J].Journal of Microbiological Methods, 2008, 75(3):545-550.
[30] HU C M, DOU W C, ZHAO G Y.Enzyme immunosensor based on gold nanoparticles electroposition and Streptavidin-biotin system for detection of S.pullorum &S.gallinarum[J].Electrochimica Acta, 2014, 117:239-245.
[31] WANG L J, WANG R H, CHEN F, et al.QCM-based aptamer selection and detection of Salmonella typhimurium[J].Food Chemistry, 2017, 221:776-782.
[32] HERZIG G P D, AYDIN M, DUNIGAN S, et al.Magnetic bead-based immunoassay coupled with tyramide signal amplification for detection of Salmonella in foods[J].Journal of Food Safety, 2016, 36(3):383-391.
[33] DI W T, DU X W, PAN M F, et al.The SPR detection of Salmonella enteritidis in food using aptamers as recongnition elements[J].IOP Conference Series:Materials Science and Engineering, 2017:012114.
[34] KAUSHIK S, PANDEY A, TIWARI U K, et al.A label-free fiber optic biosensor for Salmonella Typhimurium detection[J].Optical Fiber Technology, 2018, 46:95-103.
[35] MUNIANDY S, TEH S J, APPATURI J N, et al.A reduced graphene oxide-titanium dioxide nanocomposite based electrochemical aptasensor for rapid and sensitive detection of Salmonella enterica[J].Bioelectrochemistry, 2019, 127:136-144.
[36] CHEN I H, HORIKAWA S, BRYANT K, et al.Bacterial assessment of phage magnetoelastic sensors for Salmonella enterica Typhimurium detection in chicken meat[J].Food Control, 2017, 71:273-278.
[37] WANG J, LI H H, LI C B, et al.EIS biosensor based on a novel Myoviridae bacteriophage SEP37 for rapid and specific detection of Salmonella in food matrixes[J].Food Research International, 2022, 158:111479.
[38] ABDULLAH A, DASTIDER S G, JASIM I, et al.Microfluidic based impedance biosensor for pathogens detection in food products[J].ELECTROPHORESIS, 2019, 40(4):508-520.
[39] TADEPALLI S, BRIDGES D F, ANDERSON R, et al.Synergistic effect of sequential wash treatment with two different low-dosage antimicrobial washes in combination with frozen storage increases Salmonella Typhimurium and Listeria monocytogenes reduction on wild blueberries[J].Food Control, 2019, 102:87-93.
[40] LIU X J, FAN X T, WANG W L, et al.Wetting raw almonds to enhance pulse light inactivation of Salmonella and preserve quality[J].Food Control, 2021, 125:107946.
[41] KIM J H, MIN S C.Microwave-powered cold plasma treatment for improving microbiological safety of cherry tomato against Salmonella[J].Postharvest Biology and Technology, 2017, 127:21-26.
[42] BOURDOUX S, RAJKOVIC A, DE SUTTER S, et al.Inactivation of Salmonella, Listeria monocytogenes and Escherichia coli O157:H7 inoculated on coriander by freeze-drying and supercritical CO2 drying[J].Innovative Food Science &Emerging Technologies, 2018, 47:180-186.
[43] GHATE V, KUMAR A, KIM M J, et al.Effect of 460 nm light emitting diode illumination on survival of Salmonella spp.on fresh-cut pineapples at different irradiances and temperatures[J].Journal of Food Engineering, 2017, 196:130-138.
[44] SANZ-PUIG M, SANTOS-CARVALHO L, CUNHA L M, et al.Effect of pulsed electric fields (PEF) combined with natural antimicrobial by-products against S.Typhimurium[J].Innovative Food Science &Emerging Technologies, 2016, 37:322-328.
[45] MILLAN-SANGO D, SAMMUT E, VAN IMPE J F, et al.Decontamination of alfalfa and mung bean sprouts by ultrasound and aqueous chlorine dioxide[J].LWT, 2017, 78:90-96.
[46] LIM J S, HA J W.Growth temperature influences the resistance of Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium on lettuce to X-ray irradiation[J].Food Microbiology, 2021, 99:103825.