骆驼乳和牛乳是我国丰富的乳品资源,具有较高的营养价值和功能特性,可以满足各年龄段人群的营养需求。牛乳中含有多种营养素和生物活性成分,包括蛋白质、脂肪、乳糖、灰分、干物质等成分,具有抗氧化、抗癌、促进人类身体发育,提高人体免疫力和预防心血管疾病等作用[1]。骆驼乳与牛乳成分不同,骆驼乳具有较高含量的蛋白质,而且不含β-乳球蛋白和αs1-酪蛋白等易致敏物质,含有特有的免疫球蛋白和高含量的不饱和脂肪酸[2-3]。骆驼乳中这些成分具有多种生物功能,如低致敏、抗炎[4]、抗氧化[5]及减少心血管疾病和糖尿病的风险[6]等功能。骆驼乳与牛乳成分的差异性使其可作为牛乳的优先可替代产品,以满足易致敏人群和乳糖不耐症等特殊人群的需求,但目前对骆驼乳成分和功能特性的研究仍处在初级阶段,并且对骆驼乳和牛乳功能特性的对比研究仍有不足,需要对这2种乳进行深入探究。
外泌体是膜内吞过程形成的双层纳米囊泡,在生理和病理条件下由多种哺乳动物细胞主动释放,大小为40~160 nm不等。这些小囊泡内含脂质、蛋白质、信使mRNA、非编码RNA和DNA,可参与各种生物过程中细胞间的物质运输[7]。乳液是外泌体的丰富来源,乳外泌体具有免疫调节[8]、缓解肠道炎症炎[9]、抗氧化应激[10]等多种生物功能。微小RNA(micro RNA,miRNAs)在哺乳动物中具有高度保守性,作为外泌体信号功能的重要执行者,miRNAs的种类和含量变化是乳外泌体功能特性的重要指标[11]。miRNAs是生物转录后基因表达的重要调节因子,乳外泌体能保护miRNAs免受RNA酶的降解[12],并携带miRNAs靶向与细胞炎症、细胞增殖和细胞凋亡相关途径的基因[13]。除了控制正常的生理过程外,miRNAs还与癌症、免疫性疾病、胃肠道疾病和生殖障碍等病理过程有关[14]。基于高通量测序平台,已了解多种哺乳动物乳汁外泌体中miRNAs的表达谱,包括人乳[15]、驴乳[16]、山羊乳[17]和牛乳[18]等乳液。然而,双峰骆驼乳与荷斯坦牛乳外泌体miRNAs的表达谱研究仍显不足,并且测序技术也制约了对骆驼乳和牛乳外泌体相关功能机制的深入探索,同时也阻碍了外泌体在乳制品开发中的应用。
本研究通过超速离心技术分离乳外泌体,分别构建双峰骆驼乳与荷斯坦牛乳外泌体非编码小RNA(small RNA,sRNA)文库,利用Illumina 6000测序平台开展miRNAs的定量与定性分析,筛选高表达及差异差异表达miRNAs并进行生物信息学分析,旨在阐明2种乳源外泌体miRNAs的生物学功能,为开发基于荷斯坦牛乳与骆驼乳的功能性营养乳制品奠定理论基础。
1 材料与方法
1.1 材料与试剂
研究采集泌乳中期的双峰骆驼乳和荷斯坦牛乳乳液样本。将采集的30份驼乳与牛乳样本随机分为3组,迅速转移至超低温冰箱(-80 ℃)中保存。
MJZol总RNA提取试剂盒、RNA Purification Kit,上海美吉生物医药科技有限公司;miRNAs文库试剂盒,凯杰生物有限公司;CD81抗体,Santa Cruz生物技术公司;TSG-101抗体,艾博抗贸易有限公司;GAPDH抗体,上海优宁维生物科技股份有限公司。
1.2 仪器与设备
TL-48R粉碎研磨仪,上海万柏生物科技有限公司;CR22N/40A高速离心机,日本日立公司;Optime XPN-100超高速冷冻离心机,美国贝克曼库尔特有限公司;NanoDrop 2000超微量分光光度计、Qubit 4.0荧光定量仪,赛默飞世尔科技有限公司;Agilent 5300生物分析仪,安捷伦科技有限公司。
1.3 方法
1.3.1 乳外泌体的提取与鉴定
取5 mL乳样12 000×g、4 ℃离心30 min,重复2次离心除去脂肪和细胞碎片。将上清液以80 000×g、4 ℃离心30 min以分离残留的细胞碎片或酪蛋白。用0.22 μm过滤器清除剩余的细胞碎片,将上清液150 000×g、4 ℃离心4 h,所得沉淀物用PBS重悬后。透射电子显微镜鉴定外泌体形态;纳米颗粒追踪分析技术鉴定外泌体粒径分布;蛋白质印记鉴定外泌体特征蛋白。
1.3.2 小RNA文库构建和Illumina HiSeq测序
从组织样品中提取总RNA,利用超微量分光光度计检测所提RNA的浓度和纯度,琼脂糖凝胶电泳检测RNA完整性,使用生物分析仪测定RNA完整值(RNA integrity number,RIN)值。单次建库要求:RNA总量>1 μg,质量浓度>50 ng/μL,RIN值>7,OD260/280介于1.8~2.2之间。利用试剂盒在3′和5′端连接接头序列,并利用逆转录酶和随机引物转录成cDNA。cDNA经过PCR扩增后富集文库浓度。根据miRNAs的长度分布特点,通过切胶进行文库纯化,并在Illumina NovaSeq 6000测序仪上进行测序。Illumina测序完成后,统计sRNA片段的碱基分布和质量波动,统计碱基含量、碱基质量和碱基错误率分布。使用Fastx-Toolkit过滤原始数据,去除原始数据中的3′接头序列、3′端测序质量较低的碱基(质量值<20)、含未知碱基N、小于18 nt和大于32 nt的序列,得到纯净的sRNA序列。
1.3.3 sRNA的序列注释及miRNAs分析
使用Rfam数据库(http://rfam.xfam.org/)对sRNA进行注释,过滤注释后的非miRNAs序列和重复序列。将比对到参考基因组上的序列先与miRBase 22.0数据库(http://www.mirbase.org/)中该物种的miRNAs成熟体及前体序列进行比对,比对上的即为已知miRNAs,每个miRNAs的表达量通过TPM方法根据每百万转录本的读取数量进行量化。使用软件miRDeep2(https://www.mdc-berlin.de/content/mirdeep2-documentation)将不能比对上Rfam和miRBase的序列比对到参考基因组上,截取其周围序列使用软件进行二级结构预测,根据预测结果利用Dicer酶切位点信息、能量值等特征进行过滤,鉴定出新的miRNAs。
1.3.4 靶基因预测与生物信息学分析
本研究利用动物专用的靶基因预测软件miRanda(http://www.miranda.org/)对所有已知及新预测的miRNAs进行靶基因预测,并对靶基因进行功能注释。本研究对miRNAs靶基因产物进行基因本体论(Gene Ontology,GO)功能分类注释。利用京都基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)数据库,将基因按照参与的通路进行富集分析。使用Fisher法精确检验进行计算通路富集的显著性。采用Bonferroni方法进行检验控制假阳性率。
2 结果与分析
2.1 骆驼乳与牛乳外泌体的鉴定
如图1所示,通过透射电镜观察发现,骆驼乳和牛乳的外泌体均表现为椭圆形囊泡结构,且可以看到典型的双层膜结构。

a-骆驼乳外泌体;b-牛乳外泌体
图1 乳外泌体的形态观察
Fig.1 Morphological observation of milk exosomes
粒径分析结果如图2所示,骆驼乳和牛乳外泌体粒径大多分布于30~160 nm处,牛乳外泌体粒子个数在80 nm处集中分布,骆驼乳外泌体粒子数分布更均匀。所含粒子数分别为1.4×1013/mL和4.5×1013/mL。
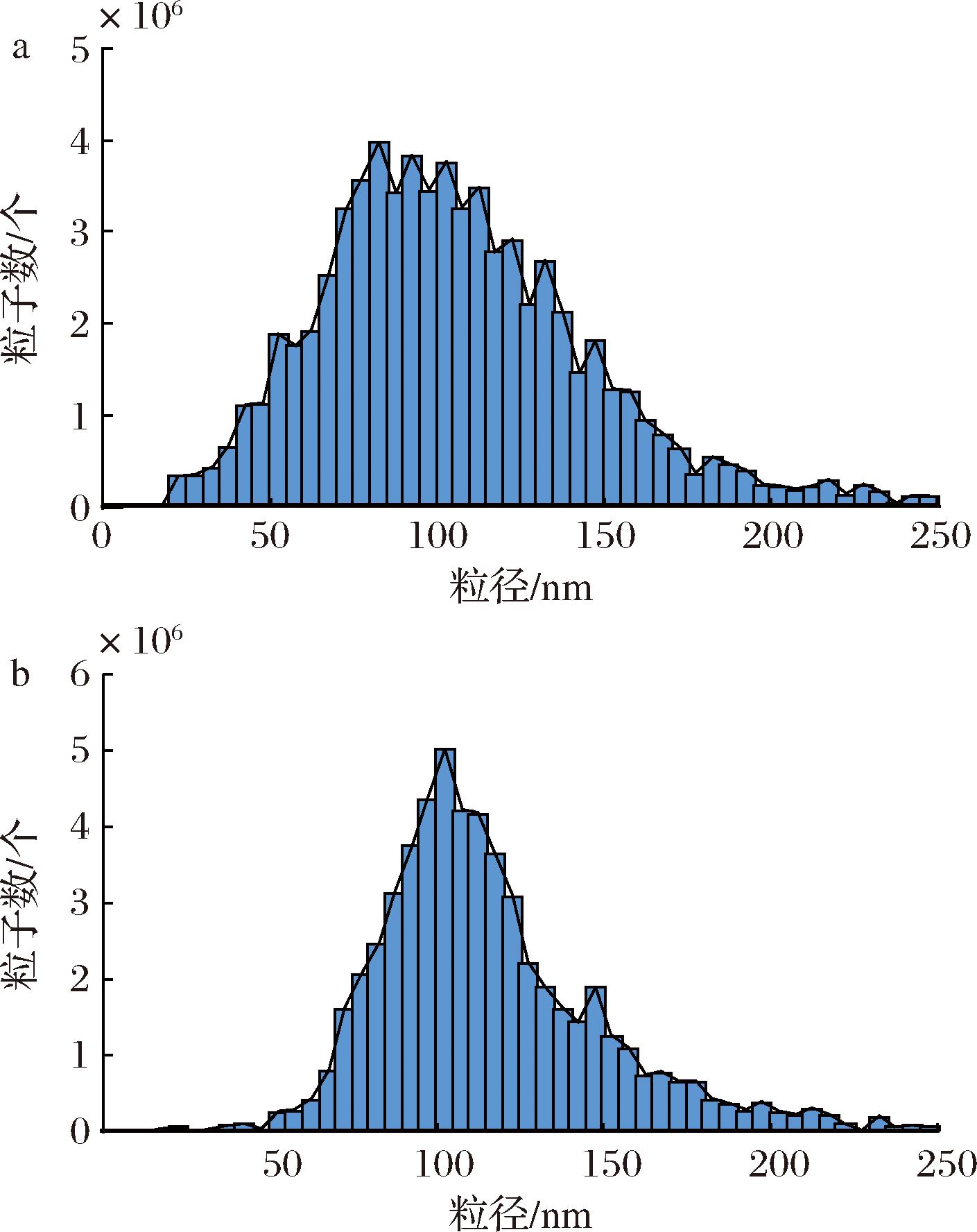
a-骆驼乳外泌体;b-牛乳外泌体
图2 乳外泌体粒径分布
Fig.2 Milk exosomes particle size distribution
蛋白质印记对牛乳外泌体特异性蛋白鉴定如图3所示,在沉淀物中鉴定出TSG101和CD81两种外泌体特异性蛋白,分子质量大小分别为49 kDa和21 kDa,而上清液中几乎不含这2种蛋白。
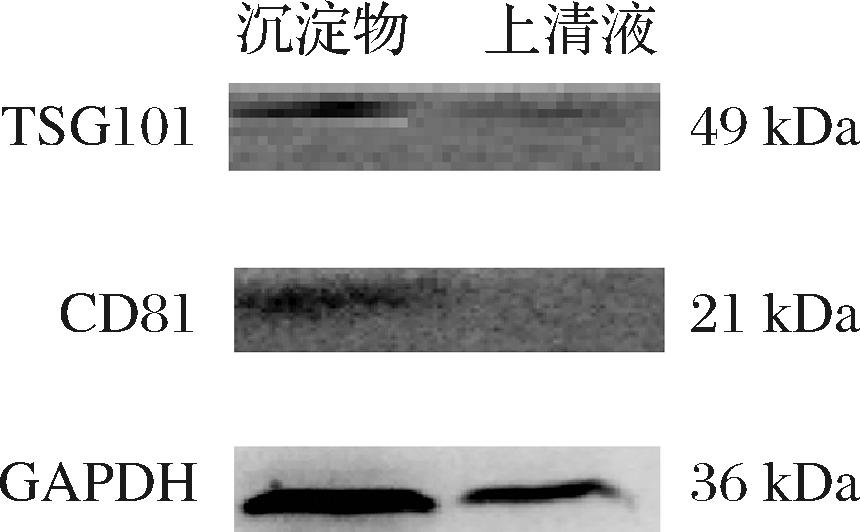
图3 牛乳外泌体的特征蛋白鉴定
Fig.3 Identification of characteristic proteins of bovine milk exosomes
2.2 数据质控和sRNA测序结果
利用Illumina技术对sRNA进行测序,结果如表1所示,骆驼乳和牛乳外泌体测序数据通过质控,除去含有带接头、低质量的序列,分别得到纯净序列8 935 325条(84.83%)和10 288 491条(89.67%)。鉴定到骆驼乳和牛乳外泌体sRNA长度小于18 nt、大于32 nt的序列分别有8 763 534(98.08%)和9 676 474(94.05%)条。将长度筛选后的sRNA定位到参考序列上(骆驼:Camelus_bactrianus_GCF_000767855.1,牛:Bos_taurus_ARS-UCD1.2),统计2个样本所有比对上sRNA的序列后发现基因组比对率分别为24.46%和64.21%。
表1 骆驼乳和牛乳外泌体sRNA测序结果
Table 1 Sequencing results of sRNA in camel and bovine milk exosomes

样品原始序列数/条纯净序列数/条 (比例/%)纯净sRNA总数/条(比例/%)比对上sRNA的序列数/条(比例/%)骆驼乳10 533 2088 935 325(84.83)8 763 534(98.08)2 143 883(24.46)牛乳11 473 77810 288 491(89.67)9 676 474(94.05)6 213 683(64.21)
2.3 sRNA分类及miRNAs首位碱基偏好性
使用Rfam数据库对sRNA分类注释的结果如图4所示,骆驼乳和牛乳外泌体的sRNA均含有外显子、内含子、核糖体RNA、miRNAs、转运RNA、小核和核仁小分子RNA的序列。骆驼乳外泌体中miRNAs仅占sRNA的23.18%,而牛乳外泌体中miRNAs占sRNA的78.41%。
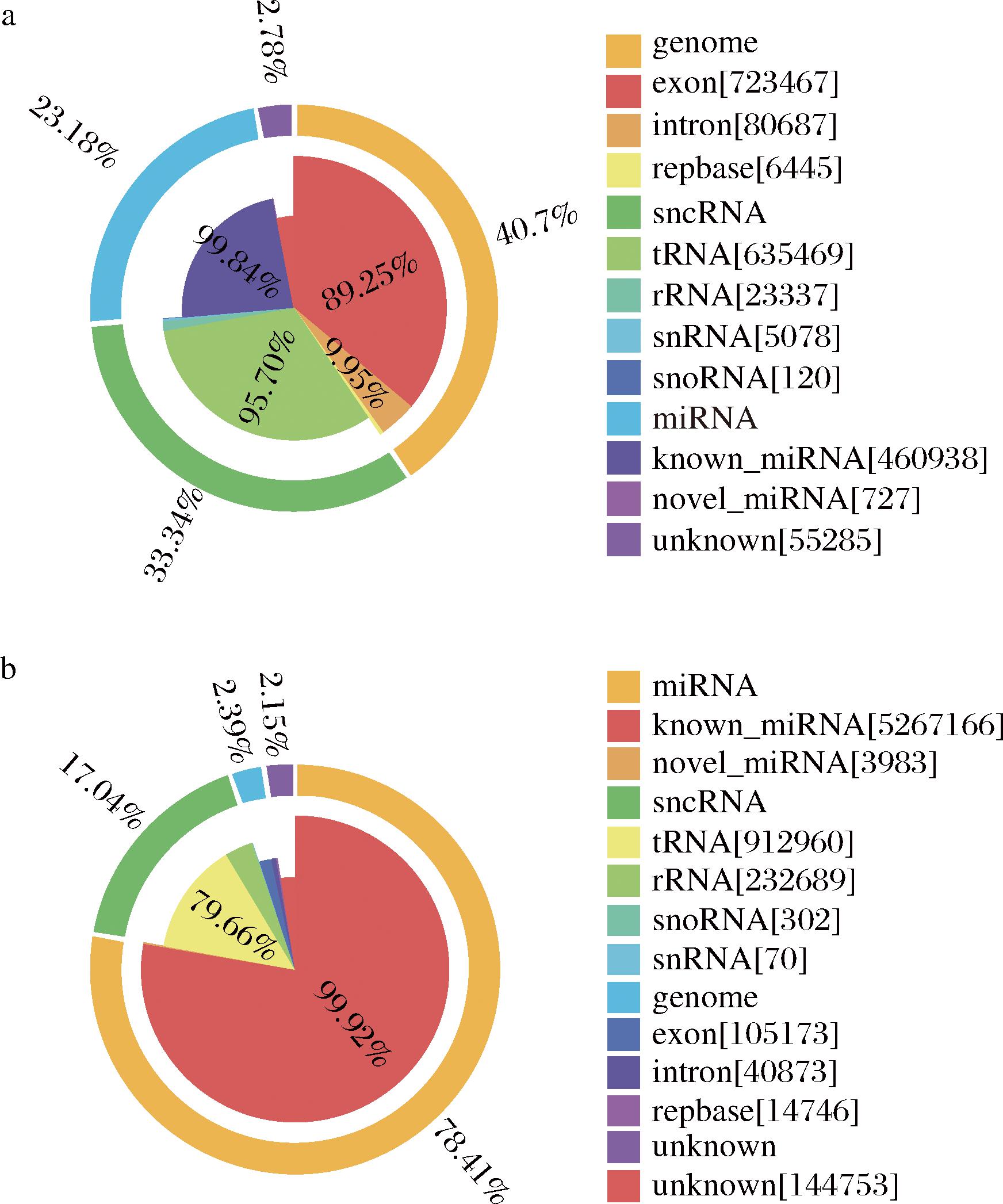
a-骆驼乳外泌体;b-牛乳外泌体
图4 sRNA分类注释
Fig.4 Annotated classification of sRNA
miRNAs的偏好性信息能反映miRNAs的加工和作用机制,因此对18~27 nt的miRNAs序列碱基分布进行统计分析。由图5可知,在20~25 nt长度区间,骆驼乳和牛乳外泌体已知miRNAs均对首位碱基均对U具有显著偏好性,在18 nt、26 nt对碱基A具有显著偏好性。
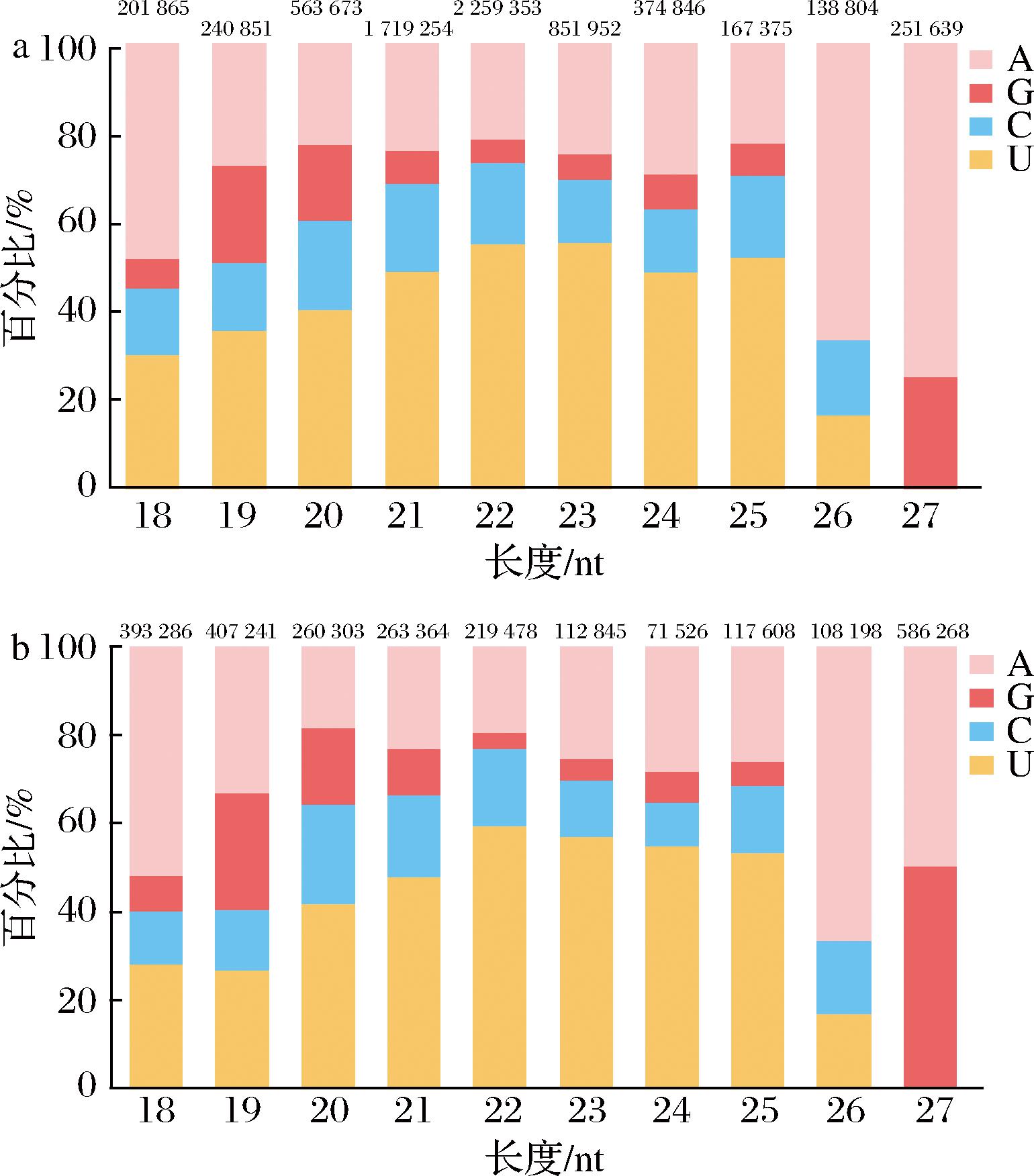
a-骆驼乳外泌体;b-牛乳外泌体
图5 miRNAs首位碱基偏好性分布
Fig.5 Distribution of base preferences in the first place of miRNAs
2.4 识别已知miRNAs及预测新miRNAs
将注释后miRNAs与miRBase数据库比对,鉴定骆驼乳和牛乳外泌体已知miRNAs。如表4可知,在骆驼乳外泌体中共鉴定到10 329条成熟miRNAs序列,种类数为6 985种,在牛乳外泌体中共鉴定到10 329条成熟miRNAs序列,种类数为8 675种。虽然已知的miRNAs种类和总数都很高,但有些miRNAs在miRNAs文库中表达量较高,其中在骆驼乳外泌体中表达最丰富的前35个miRNAs占总数的91.09%,牛乳外泌体中表达最丰富的前35个miRNAs占总数的90.92%。
表2 骆驼乳和牛乳外泌体高表达量miRNAs统计
Table 2 Statistics of highly expressed miRNAs in camel and bovine milk exosomes

骆驼乳外泌体miRNAs表达量/%牛乳外泌体miRNAs表达量/%miR-30a-5p8.13let-7a9.02miR-29b-3p6.92let-7a-5p8.42miR-29a5.73let-7b5.38let-7b4.70let-7b-5p5.38let-7b-5p4.45let-75.20miR-29c-3p4.28miR148a-3p4.52miR-148a-3p3.72let-7-5p3.41let-7a3.65miR-200a-3p3.22miR-29c2.92miR-21-5p3.20miR-1412.48miR-200a3.13miR-30 d-5p2.43miR-200c1.88let-7a-5p2.42miR148a1.81let-72.13miR-211.81miR-141-3p2.09miR-30a1.76miR-29a-3p1.86miR-1071.74miR-30a1.86miR-141-5p1.73let-7f-5p1.84let-7c-5p1.73miR-200a-3p1.84let-7f1.71miR-21-5p1.83miR-261.70miR-200c-3p1.83miR-200a-5p1.70miR-103-3p1.82miR-30a-3p1.70miR-200c1.81miR-26a1.67miR-26b-5p1.80miR-26a-5p1.64miR-26-5p1.80miR-26-5p1.62miR-30 d1.79miR-29a1.59miR-16c-5p1.73miR-29a-5p1.56miR-16c1.69miR-29c-5p1.54miR-16-5p1.68miR-29c1.48miR-148a1.59miR-331.47let-7-5p1.40miR-103-3p1.46miR-161.34miR-30 d1.39miR-29b1.33miR-92a-3p1.31miR-21a1.28miR-1251.18let-7i-5p1.17miR-125b-5p1.01miR-29c-3p0.92miR-125b0.98miR-125a0.83miR-125-5p0.87
在骆驼乳和牛乳外泌体中分别预测了86个和229个新miRNAs。表达量最高miRNAs的是NW_011514130.1_21588和26_35069,分别为16.62%和10.66%,序列为guucaaaucucgguggaac和ggggccgggggcggccgggg,这些新序列能丰富乳外泌体miRNAs的信息。
2.5 靶基因预测及GO功能注释分析
将骆驼乳和牛乳外泌体miRNAs的靶基因进行GO功能富集分析,以了解骆驼和牛全部基因产物的功能分类情况。如图6所示,在细胞组成方面,骆驼乳外泌体和牛乳外泌体miRNAs均参与细胞器、胞器膜、含蛋白质的复合体、细胞外区域和突触这5种条目,且这5种条目所占比例相似。二者不同的是,只有牛乳注释到细胞内部分和膜部分2个条目,骆驼乳外泌体miRNAs的靶基因注释到细胞质的比例最大,为26.93%。
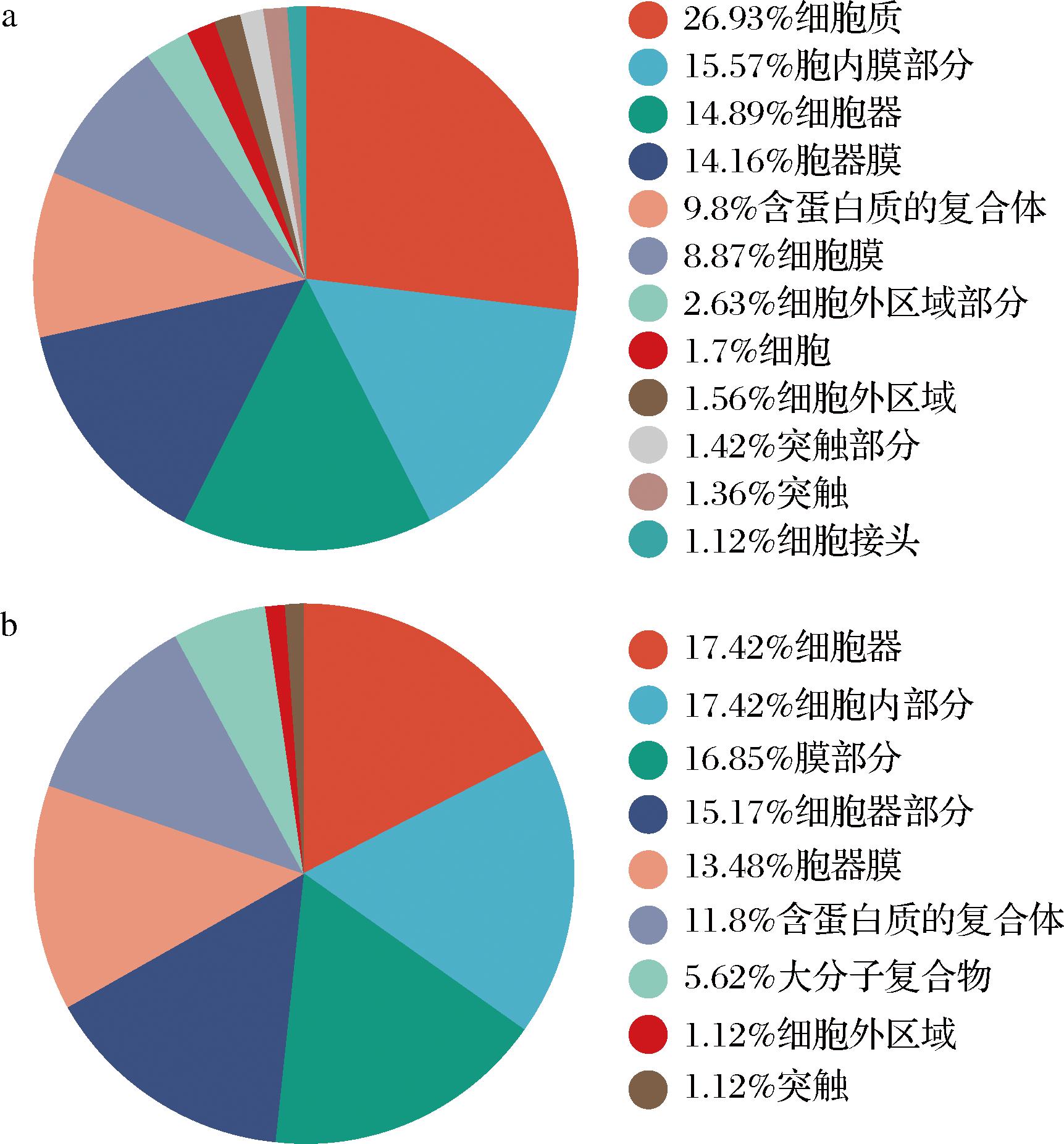
a-骆驼乳外泌体;b-牛乳外泌体
图6 乳外泌体miRNAs靶基因在的细胞成分注释
Fig.6 Annotation of cell composition in the target genes of miRNAs in milk exosomes
在生物学过程方面,骆驼乳与牛乳外泌体miRNAs靶基因在生物调节、细胞成分组织或细胞发生、代谢过程、对刺激的反应和发育过程7种生物过程中显著富集,且比例相似。但骆驼乳与牛乳外泌体miRNAs中靶基因富集在定位、细胞过程和多生物过程3种生物学过程的比例差异较大,分别占总靶基因的7.08%、24.25%和1.65%,而在牛乳中分别占比20.95%、14.86%和10.81%(图7-a)。除和牛乳参与相同的生物学过程外,骆驼乳外泌体miRNAs靶基因还参与多细胞生物体过程(4.65%)、免疫系统过程(2.25%)、生殖过程(1.7%)和生物黏附(1.18%)(图7-b)。分子功能方面上,骆驼乳外泌体miRNAs靶基因有26 729个,牛乳外泌体miRNAs靶基因有24 759个。牛乳外泌体miRNAs的72.73%靶基因在结合功能富集;而骆驼乳外泌体miRNAs的48.44%靶基因在结合功能富集,27.9%靶基因在催化活性富集(图8)。
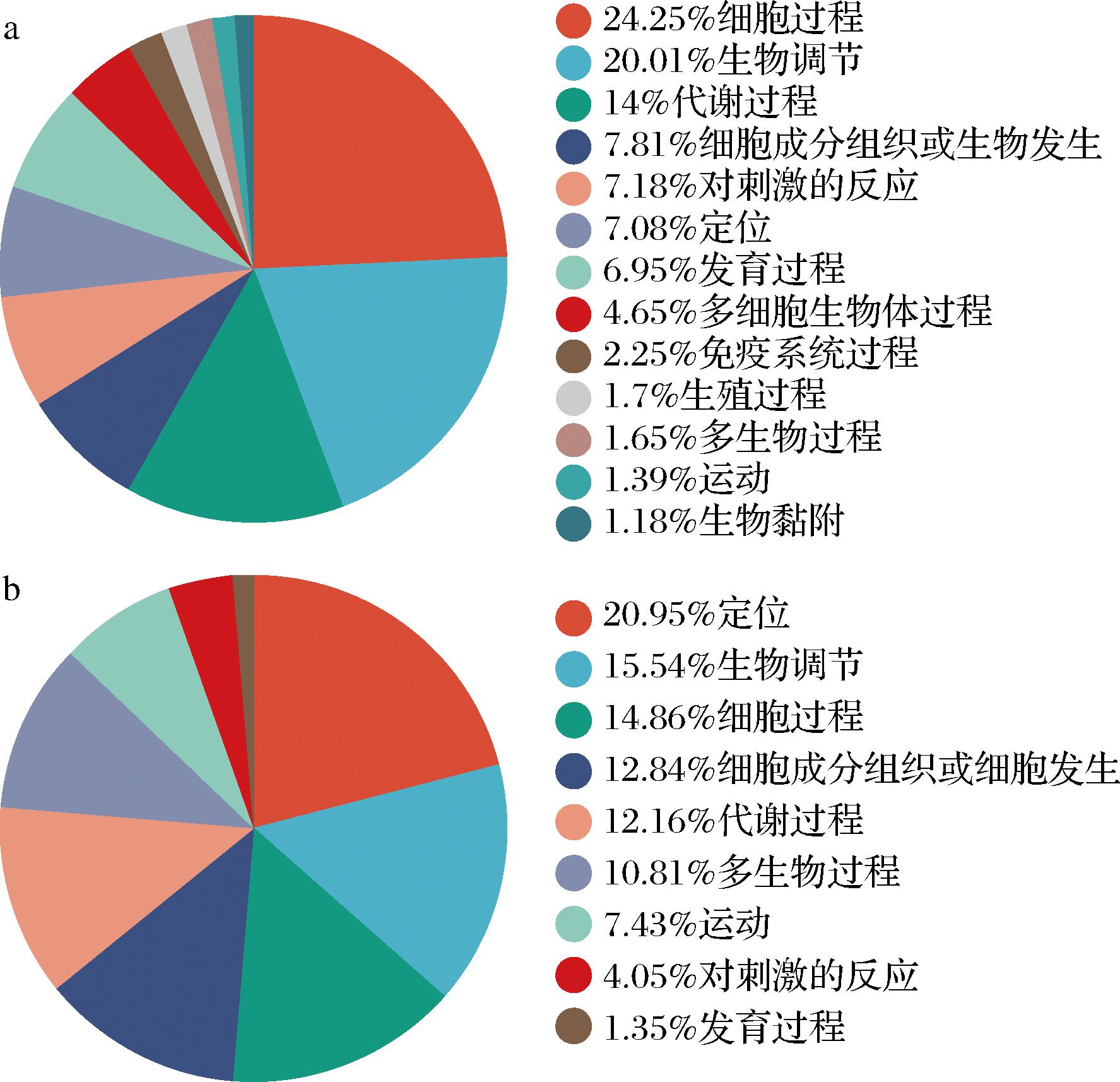
a-骆驼乳外泌体;b-牛乳外泌体
图7 乳外泌体miRNAs靶基因的生物过程注释
Fig.7 Annotation of biological processes in the target genes of miRNAs in milk exosomes
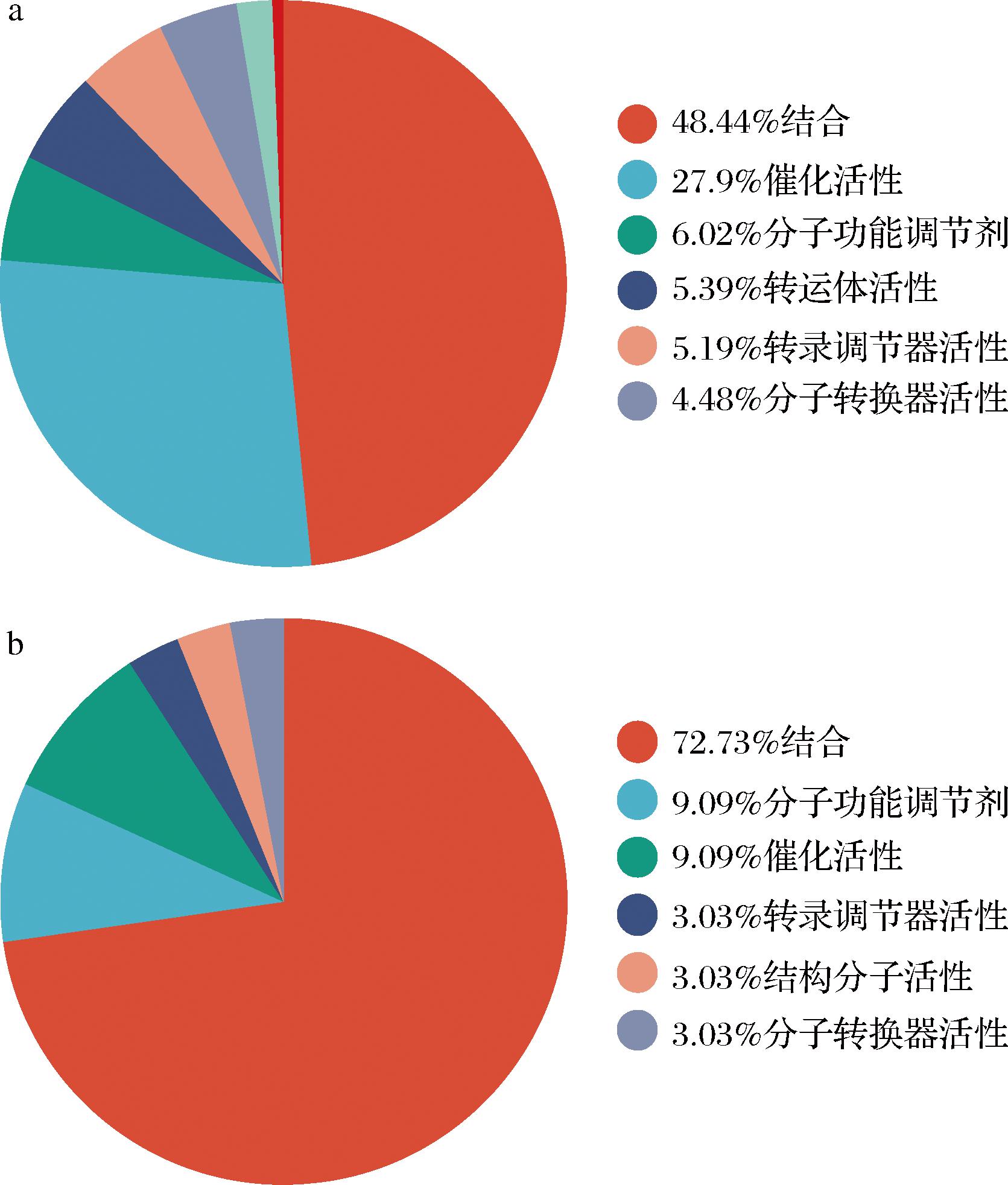
a-骆驼乳外泌体;b-牛乳外泌体
图8 乳外泌体miRNAs靶基因的分子功能注释
Fig.8 Annotation of molecular functions in the target genes of miRNAs in milk exosomes
2.6 靶基因通路富集分析
将骆驼乳和牛乳外泌体miRNAs靶基因富集到KEGG通路的6个条目,分别是环境信息过程、基因信息过程、代谢、细胞过程、生物系统和人类疾病。图9显示了富集显著的前20条通路,大部分通路与人类疾病条目相关。骆驼乳外泌体中miRNAs中共有13 691个靶基因被注释到346条通路中,前20条通路均显著富集(P<0.05),其中癌症中的通路、癌症中的miRNAs、前列腺癌症、胰岛素抵抗、非小细胞肺癌、结直肠癌、胰腺癌、慢性髓系白血病、胶质瘤、黑色素瘤、癌症中的胆碱代谢和糖尿病并发症糖基化终末产物-糖基化终末产物受体(advanced glycation end products-advanced glycation end products,AGE-RAGE)信号通路等12条通路显著富集到人类疾病功能上(图9-a)。
牛乳外泌体中miRNAs中共12 455个靶基因,被富集到356条通路,前20条通路均显著富集(P<0.05),其中癌症中的通路、人类巨细胞病毒感染、糖尿病并发症AGE-RAGE信号通路、胰岛素抵抗、库欣综合征和酒精性肝病等6条通路显著富集到人类疾病功能上。通过分析发现,骆驼乳和牛乳外泌体miRNAs靶基因在癌症中的通路、糖尿病并发症AGE-RAGE信号通路和胰岛素抵抗3条通路上富集(图9-b)。
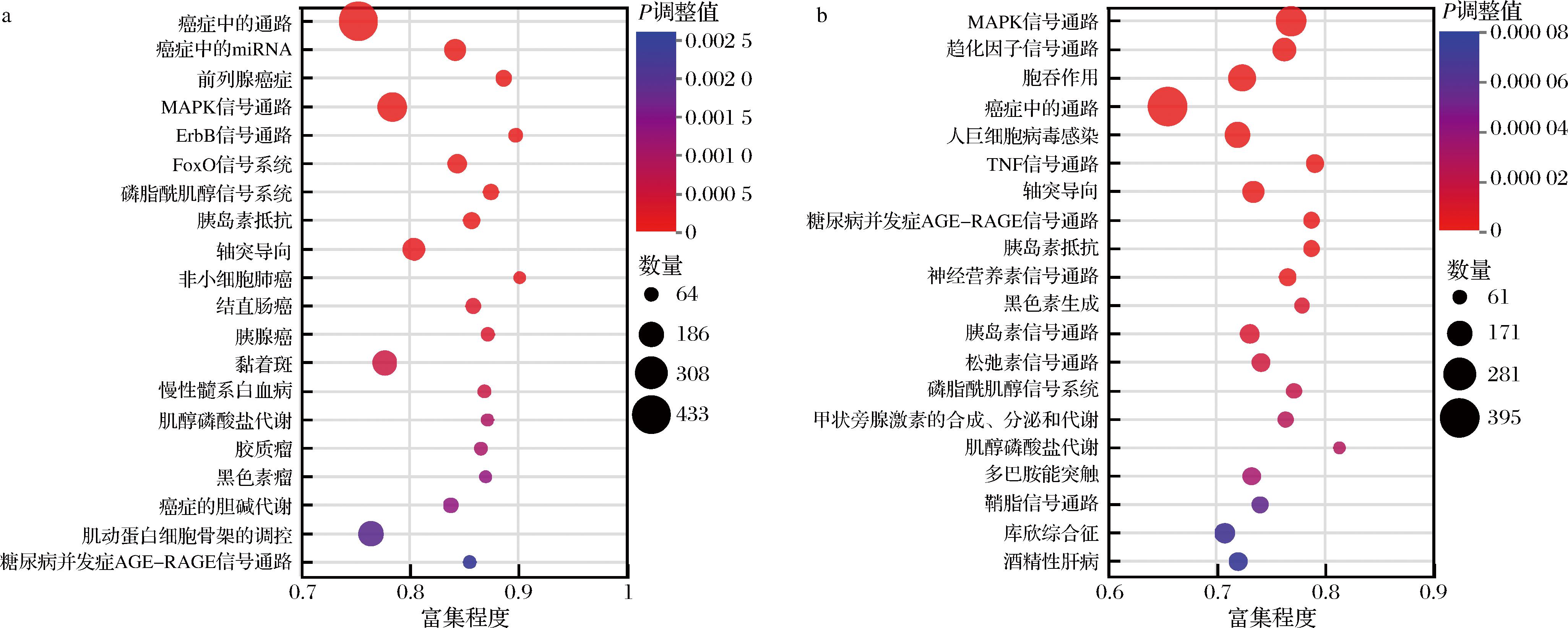
a-骆驼乳外泌体;b-牛乳外泌体
图9 乳外泌体miRNAs靶基因KEGG富集通路
Fig.9 KEGG enrichment pathway of target genes of miRNAs in milk exosome
糖尿病并发症AGE-RAGE信号通路和胰岛素抵抗2条通路均与糖尿病有关,并且牛乳外泌体miRNAs的靶基因显著富集在胰岛素信号系统这一与糖尿病有关生物系统上,因此本研究富集调控这2条通路中的前15个miRNAs。在骆驼乳和牛乳外泌体miRNAs靶基因调控糖尿病并发症AGE-RAGE信号通路中,显著富集的miRNAs分别为miR-185-5p和miR-421-3p(图10-a和图10-b)。而在骆驼乳和牛乳外泌体miRNAs靶基因调控胰岛素代谢通路中,最显著富集的miRNAs分别为miR-10240-5p和miR-92a(图10-a和图10-b)。在骆驼乳外泌体中预测调控胰岛素抵抗通路2种miRNAs的成熟体序列为分别是NW_011514938.1_26311和NW_011514938.1_26312,其成熟体序列分别是NW_011514938.1_26311:gggaggcaggggggagcaga和NW_011514938.1_26312:gggaggcaggggggagcaga。
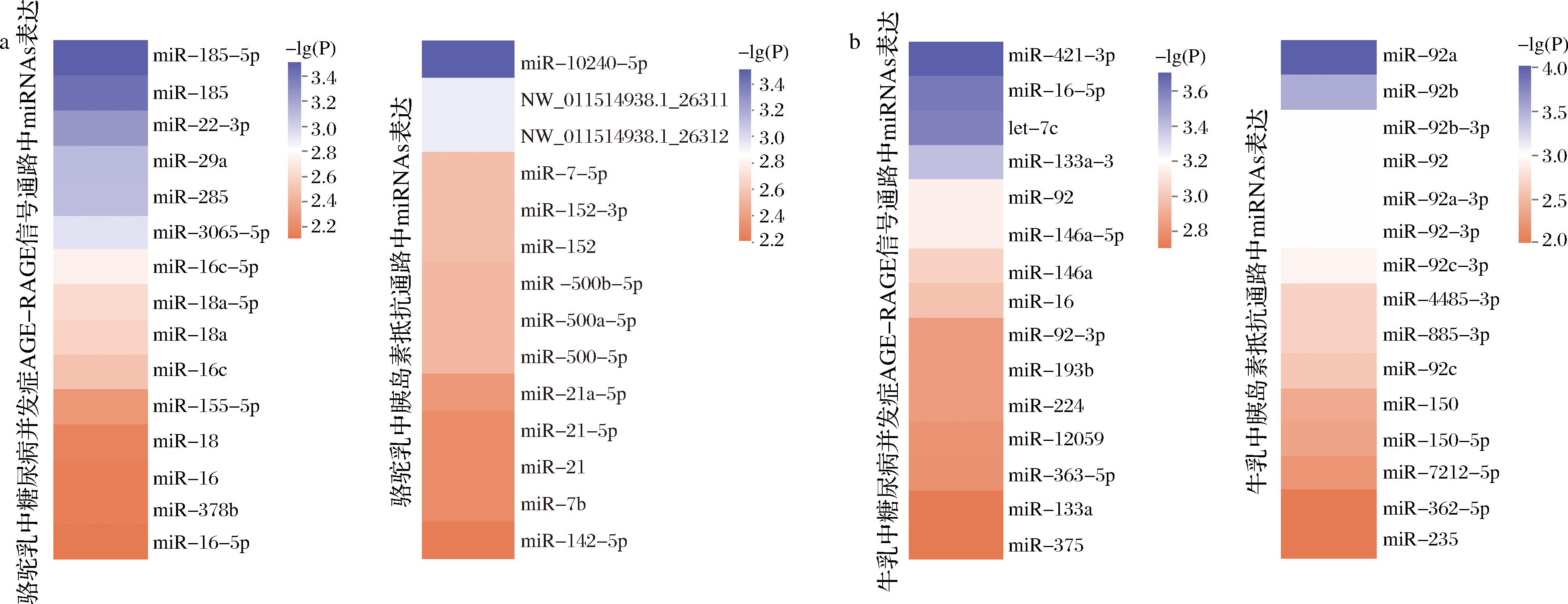
a-骆驼乳外泌体;b-牛乳外泌体
图10 富集乳外泌体糖尿病并发症AGE-RAGE信号通路和胰岛素抵抗通路中miRNAs
Fig.10 Enrichment of miRNAs in AGE-RAGE pathway with diabetes complications and insulin resistance pathways in milk exosomes
3 讨论
通过超高速离心技术,本研究分别从骆驼乳与牛乳样本中分离出外泌体。基于高通量测序技术,鉴定和分析了骆驼乳和牛乳外泌体中miRNAs的表达谱,并对miRNAs的类别及其表达水平进行了系统注释。最后探索了miRNAs的靶基因,从共同富集的KEGG通路中筛选与人类疾病相关的miRNAs。
通过鉴定牛乳外泌体中的sRNA种类和数量发现,miRNAs在sRNA中占比为78.41%,这与miRNAs在牛乳、人乳和驴乳外泌体sRNA占比最多的结果一致[19]。然而,骆驼乳外泌体中miRNAs在sRNA中占比仅为23.18%,这可能与骆驼的泌乳期、产奶量和物种差异等因素有关。通过鉴定,在骆驼乳外泌体中发现6 561种已知miRNAs和86种新型miRNAs;在牛乳外泌体中发现8 675种已知miRNAs和229种新型miRNAs。在已知miRNAs中,let-7b、let-7b-5p、let-7a、let-7a-5p、let-7、miRNA-148a-3p和miR-30a-5p等miRNAs均在骆驼乳和牛乳外泌体中表达量较高。let-7基因是参与正常发育和癌症的关键调节因子,在哺乳动物中高度保守。CENDRON等[20]利用Illumina技术分析1~7胎和2~7个月的牛乳、驴乳、山羊乳和绵羊乳miRNAs表达谱,发现let-7a-5p和let-7b-5p等miRNAs在4种乳中显著表达。let-7家族也在骆驼乳外泌体显著表达,这表明乳外泌体let-7家族miRNAs的高表达和乳相同,都与生长激素、雌激素的表达和催乳素信号通路有关[21]。miRNA-148a-3p是人乳中最丰富的miRNAs之一,这种miRNAs证明对新生儿有积极影响,还与早期婴儿生长和肥胖相关[22]。骆驼乳外泌体中miR-30a-5p表达量最高,它通过靶向TRIF相关接头分子(Trif-related adaptor molecule,TRAM)抑制下游NF-κB炎症通路,是在抵抗肠道上皮炎症方面具有优异效果的分子之一[23]。经过文献检索,这些乳外泌体中高表达的miRNAs在调节细胞生长、增殖、分化和死亡方面具有重要意义。
此外,对miRNAs进行GO富集分析时发现,骆驼乳及牛乳外泌体miRNAs靶基因在细胞组分、生物过程和分子功能等方面富集到多种相同的GO条目。值得注意的是,在分子功能性方面,骆驼乳和牛乳miRNAs靶基因均在结合功能富集,提示了miRNAs对靶基因与其他基因结合可能有调控作用;骆驼乳外泌体miRNAs还在催化活性上显示出巨大潜力,催化活性的提高可通过细胞中多种酶或线粒体来实现,这能加快细胞或生物过程的新陈代谢。对miRNAs进行KEGG富集分析时发现,骆驼乳和牛乳外泌体miRNAs靶基因均显著富集在糖尿病并发症AGE-RAGE信号通路和胰岛素抵抗通路上。而糖基化终末产物(advanced glycation end products,AGE)形成、糖基化终末产物受体(advanced glycation end products,RAGE)上调和胰岛素受体/胰岛素受体底物失调与糖尿病有关[24],这表明骆驼乳和牛乳外泌体miRNAs在糖尿病治疗中具有一定的调节作用。SHANG等[19]的研究表明,人乳、牛乳和驴乳外泌体miRNAs靶基因显著富集在糖尿病并发症AGE-RAGE信号通路上,这表明多种乳外泌体miRNAs均可作为治疗糖尿病的潜在治疗靶点,为糖尿病治疗提供了新的方向。当然,骆驼乳和牛乳外泌体miRNAs的其他功能仍值得被进一步研究。
本研究标志着首次尝试表征新疆双峰驼乳外泌体中的miRNAs,并与荷斯坦牛乳外泌体miRNAs进行比较分析,但也存在某些不足之处:本研究采用蛋白质印记鉴定出牛乳外泌体的特征蛋白TSG101和CD81,由于目前骆驼乳特异性抗体的缺乏,难以使用蛋白质印记法鉴定骆驼乳外泌体;将纯净sRNA序列与参考基因组比对发现,牛乳外泌体sRNA与基因组比对率为64.41%,说明参考基因组选择合适,而且相关实验不存在污染,而骆驼乳外泌体sRNA与基因组比对率较低,仅为23.18%,可能是骆驼RNA数据库尚不完善或者样本杂合度较高导致扩增序列冗余的原因。虽然存在上述不足,但对于骆驼乳和牛乳外泌体miRNAs的表达和对比分析仍然具有重要的研究价值。
骆驼乳作为牛乳的可替代产品,容易被人体消化吸收。近年来随着骆驼乳及其制品不断增加,丰富了以牛乳为主要消费产品的单一的乳品结构。通过比较骆驼乳和牛乳外泌体miRNAs表达谱,为针对糖尿病患者和其他特定人群开发成分和功能相似的功能性乳制品提供理论依据。也为不同物种来源的乳液中生物活性成分的研究提供了参考。
[1] PEREIRA P C.Milk nutritional composition and its role in human health[J].Nutrition, 2014, 30(6):619-627.
[2] 黄新红, 郭文振, 王旭涛, 等. 骆驼乳营养成分变化规律及产业化应用研究[J]. 乳品与人类, 2024, (4): 26-31.HUANG X H, GUO W Z, WANG X T, et al.Nutritional Composition of Camel Milk: Changing Laws and Industrialization Applications, 2024, (4): 26-31.
[3] YADAV A K, KUMAR R, PRIYADARSHINI L, et al.Composition and medicinal properties of camel milk:A review[J].Asian Journal of Dairy and Food Research, 2015, 34(2):83.
[4] ARAB H H, SALAMA S A, EID A H, et al.Camel’s milk ameliorates TNBS-induced colitis in rats via downregulation of inflammatory cytokines and oxidative stress[J].Food and Chemical Toxicology, 2014, 69:294-302.
[5] CHEN Y Z, LI C, GU J, et al.Anti-oxidative and immuno-protective effect of camel milk on radiation-induced intestinal injury in C57BL/6J mice[J].Dose-response, 2021, 19(1):15593258211003798.
[6] KORISH A A.The antidiabetic action of camel milk in experimental type 2 diabetes mellitus:An overview on the changes in incretin hormones, insulin resistance, and inflammatory cytokines[J].Hormon- und Stoffwechselforschung, 2014, 46(6):404-411.
[7] KALLURI R, LEBLEU V S.The biology, function, and biomedical applications of exosomes[J].Science, 2020, 367(6478):EAAU6977.
[8] GOLAN-GERSTL R, REIF S.Extracellular vesicles in human milk[J].Current Opinion in Clinical Nutrition &Metabolic Care, 2022, 25(3):209-215.
[9] REIF S, ELBAUM-SHIFF Y, KOROUKHOV N, et al.Cow and human milk-derived exosomes ameliorate colitis in DSS murine model[J].Nutrients, 2020, 12(9):2589.
[10] IBRAHIM H M, MOHAMMED-GEBA K, TAWFIC A A, et al.Camel milk exosomes modulate cyclophosphamide-induced oxidative stress and immuno-toxicity in rats[J].Food &Function, 2019, 10(11):7523-7532.
[11] ZHANG J, LI S, LI L, et al.Exosome and exosomal microRNA:Trafficking, sorting, and function[J].Genomics, Proteomics &Bioinformatics, 2015, 13(1):17-24.
[12] PENG H, JI W H, ZHAO R C, et al.Exosome:A significant nano-scale drug delivery carrier[J].Journal of Materials Chemistry B, 2020, 8(34):7591-7608.
[13] BARTEL D P.MicroRNAs:genomics, biogenesis, mechanism, and function[J].Cell, 2004, 116(2):281-297.
[14] LI D, YAO X L, YUE J X, et al.Advances in bioactivity of microRNAs of plant-derived exosome-like nanoparticles and milk-derived extracellular vesicles[J].Journal of Agricultural and Food Chemistry, 2022, 70(21):6285-6299.
[15] 罗雨佳, 黄子彧, 林莹莹, 等.不同泌乳期人乳外泌体miRNA表达谱的研究[J].中国食品学报, 2022, 22(11):335-342.LUO Y J, HUANG Z Y, LIN Y Y, et al.Exploration of exosomal miRNA expression profiles in human milk during the different lactation period[J].Journal of Chinese Institute of Food Science and Technology, 2022, 22(11):335-342.
[16] 柴玉霞, 王新宇, 岳喜庆, 等.驴乳外泌体中miRNA的测序与分析[J].食品科学, 2022, 43(14):151-157.CHAI Y X, WANG X Y, YUE X Q, et al.Sequencing and analysis of microRNAs in donkey milk exosomes[J].Food Science, 2022, 43(14):151-157.
[17] 鲁曦, 任珂.山羊乳及绵羊乳外泌体miRNAs表达谱的分析与差异比较[J].食品与发酵工业, 2023, 49(23):134-140.LU X, REN K.Analysis and comparison of miRNAs expression profiles of goat milk and sheep milk exosomes[J].Food and Fermentation Industries, 2023, 49(23):134-140.
[18] 商静雯, 柴玉霞, 曹雪妍, 等.牛乳外泌体中miRNA的测序与分析[J].食品科学, 2023, 44(24):105-111.SHANG J W, CHAI Y X, CAO X Y, et al.Sequencing and analysis of microRNAs in bovine milk exosomes[J].Food Science, 2023, 44(24):105-111.
[19] SHANG J W, NING J T, BAI X, et al.Identification and analysis of miRNAs expression profiles in human, bovine, and donkey milk exosomes[J].International Journal of Biological Macromolecules, 2023, 252:126321.
[20] CENDRON F, ROSANI U, FRANZOI M, et al.Analysis of miRNAs in milk of four livestock species[J].BMC Genomics, 2024, 25(1):859.
[21] ALSAWEED M, LAI C T, HARTMANN P E, et al.Human milk cells contain numerous miRNAs that may change with milk removal and regulate multiple physiological processes[J].International Journal of Molecular Sciences, 2016, 17(6):956.
[22] SHAH K B, FIELDS D A, PEZANT N P, et al.Gestational diabetes mellitus is associated with altered abundance of exosomal microRNAs in human milk[J].Clinical Therapeutics, 2022, 44(2):172-185.e1.
[23] ZHANG C, ZHENG J, HAN X T, et al.Bovine colostrum miR-30a-5p targets the NF-κB signaling pathway to alleviate inflammation in intestinal epithelial cells[J].Journal of Agricultural and Food Chemistry, 2024.
[24] WALKE P B, BANSODE S B, MORE N P, et al.Molecular investigation of glycated insulin-induced insulin resistance via insulin signaling and AGE-RAGE axis[J].Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease, 2021, 1867(2):166029.