葡萄酒生产是一个多种微生物代谢的过程,其中酵母菌不仅参与酒精发酵,在芳香物质的形成中也发挥重要作用[1-2]。本土酿酒酵母菌是“微生物风土”的核心微生物菌群,它能有效促进葡萄酒的生产,并使自然发酵产品具有地域特色[3-4]。酵母菌群的多样性在酿造具有产区特色葡萄酒中起着至关重要的作用。近年来,一些研究调查了同一区域[5]或同一子区域[6]内不同葡萄园酿酒酵母的生物多样性。在较小区域内,本土酿酒酵母的多样性和差异性可能是由地理因素[7]和葡萄品种[8]共同驱动的。
蓬莱产区属温带季风气候,四季分明,雨量集中,依托黄渤海的湿润海风和和煦阳光,赋予了海岸葡萄特有的品质,为产区孕育出丰富多样的本土微生物资源。魏如腾[9]通过对蓬莱产区赤霞珠葡萄酒自然发酵系统中微生物多样性的深入研究,描述了包括有孢汉逊酵母属(Hanseniaspora)和假丝酵母属(Starmerella)在内的众多酵母菌,这些酵母菌在提升葡萄酒品质方面发挥了重要作用;汤晓宏[10]从蓬莱霞多丽葡萄果实和土壤中分离获得接合酵母属(Zygosaccharomyces)、毕赤酵母属(Pichia)和路氏类酵母(ludwigii)等发酵性能优良的酵母菌,为霞多丽葡萄酒的酿造创造了酵母资源条件。结合这2项研究结果,可以推断出蓬莱产区的酵母属、种具有丰富的多样性,并且这种多样性在不同葡萄品种之间存在着差异。然而,关于主栽葡萄品种自然发酵中酵母群落演替规律的研究尚显不足,深入探究此规律对优化酿造工艺、提升葡萄酒品质具有重要意义。
品丽珠(Cabernet Franc)抗寒性强,含糖量高,风味高雅[11],是蓬莱葡萄酒产区主栽酿酒品种之一。蓬莱产区品丽珠葡萄酒果香浓郁,品质优良,但能够展现产区地域特色的酵母菌资源比较匮乏,加强品丽珠葡萄自然发酵过程中微生物多样性的研究对蓬莱品丽珠葡萄酒酵母的开发具有重要理论意义。目前针对葡萄酒发酵中微生物群落的多样性和变化规律的研究多是基于高通量测序技术[12],该技术虽能全面剖析群落构成情况,但物种层面的鉴定结果尚不够详尽,依旧需要与培养组的研究结果进行相互验证[13-14]。
本研究基于可培养的方法,通过连续2年采集蓬莱子产区品丽珠葡萄自然发酵阶段中相关酵母菌落,利用WLN(Wallerstein laboratory nutrient)培养基分离、纯化酵母,对酵母单菌落进行分子生物学鉴定,并利用Interdelta指纹图谱分析技术进一步对酿酒酵母菌株的基因型进行表征,分析酿酒酵母遗传多样性,以期揭示蓬莱葡萄酒产区酵母群落多样性及演替规律,为该地区具有典型酿造特征的酿酒酵母菌株的选育提供理论支撑。
1 材料与方法
1.1 试验材料
葡萄:2年份2个葡萄园(东经120°90′、北纬37°77′)中品丽珠酿酒葡萄品种(表1),编号TF、LF和RF。其中TF和LF为连续2年同一葡萄园,TF和RF两葡萄园相距11 km。
表1 品丽珠葡萄原料信息
Table 1 Raw material information of Cabernet Franc grapes
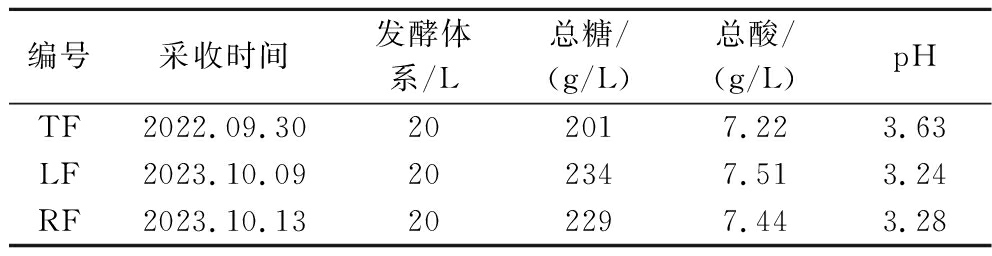
编号采收时间发酵体系/L总糖/(g/L)总酸/(g/L)pHTF2022.09.30202017.223.63LF2023.10.09202347.513.24RF2023.10.13202297.443.28
自然发酵:将新鲜采摘的品丽珠葡萄经人工除梗破碎,装入已灭菌的20 L发酵桶中,于18~22 ℃进行带皮发酵。
取样时期:分别在葡萄醪期(Ⅰ)、发酵启动期(Ⅱ)、发酵旺盛期(Ⅲ,原料糖含量消耗1/4)、发酵后期(Ⅳ,原料糖含量消耗3/4)及发酵末期(Ⅴ,残糖量<4 g/L)这5个发酵阶段进行取样。
1.2 培养基
WLN琼脂培养基:青岛海博生物技术有限公司,灭菌后加入氯霉素100 mg/L。
YPD液体培养基:2%(质量分数,下同)葡萄糖,2%蛋白胨,1%酵母浸粉。
1.3 试剂与仪器
无水乙醇、NaCl、葡萄糖等均为分析纯,天津市科密欧化学试剂有限公司;氯霉素(分析纯),北京博奥拓达科技有限公司;
B-K20型恒温培养箱,上海枫岭生物技术有限公司;C1000型PCR仪,美国BIO-RAD公司;UV755B紫外分光光度计,上海分析仪器总厂;DYYY-10C型电泳仪,北京六一仪器厂;GBOX-EF型凝胶成像系统,英国Syngene公司。
1.4 实验方法
1.4.1 酵母菌株基因组DNA提取及检测
采用石英砂破壁法[15],DNA纯度和浓度使用紫外显微分光光度计检测。
1.4.2 26S rDNA基因D1/D2区测序鉴定
以提取的酵母菌株基因组DNA为模板,NL1(5′-GCATATCAATAAGCGGAGGAAAAAG-3′)和NL4(5′-GGTCCGTGTTTCAAGACGG-3′)为引物,对酵母菌株26S rDNA D1/D2区进行PCR扩增。PCR扩增体系(20 μL体系):模板DNA 2 μL,引物NL1与NL4各0.8 μL,2×Rapid Taq Master Mix 10 μL,以dd H2O补至20 μL。扩增条件:95 ℃预变性3 min,95 ℃变性15 s,55 ℃退火45 s,72 ℃延伸30 s,循环35次;最后72 ℃再延伸5 min。PCR扩增产物交由西安擎科生物科技有限公司进行测序鉴定。将测序结果提交至NCBI的GenBank数据库中与已知序列进行BLAST同源性比对[16]。
1.4.3 Interdelta分型
以delta12(5′-TCAACAATGGAATCCCAAC-3′)和delta21(5′-CATCTTAACACCGTATATGA-3′)为引物,对酿酒酵母菌株进行PCR扩增。PCR扩增体系(20 μL体系):模板DNA 1 μL,引物delta12与delta21各0.8 μL,2×Rapid Taq Master Mix 10 μL,dd H2O补齐至20 μL。扩增条件:95 ℃预变性3 min;95 ℃变性15 s,48 ℃退火45 s,最后72 ℃延伸90 s,共循环35次;72 ℃再延伸5 min。将4 μL的PCR扩增产物置于1%琼脂糖凝胶上,进行电泳(电压130 V,电流90 mA,时间30 min)操作,利用紫外成像仪对电泳结果进行观察和分析。通过比较电泳图,可将酿酒酵母单培养物区分为不同的基因型[17-18]。
1.5 数据处理
所得数据通过Excel 2016、SPSS Statistics 27软件进行分析处理。采用Origin Pro 2024和MEGA 7软件进行绘图。
2 结果与分析
2.1 酵母菌群多样性和动态演替分析
为了检测蓬莱产区品丽珠葡萄自发发酵过程中的酵母种群动态,本试验连续2年在该产区共分离纯化387株野生酵母菌株。结合菌落形态计数和26S rDNA基因测序分析,菌落形态和鉴定结果如电子版增强出版附表1、附表2、附表3所示(https://doi.org/10.13995/j.cnki.11-1802/ts.042102)。
2.1.1 酵母菌群多样性分析
3组发酵体系中的酵母菌群共被鉴定为9属20种,酵母种类丰富,酵母菌各属种所占分离酵母总菌株的比例及交集情况如图1所示。
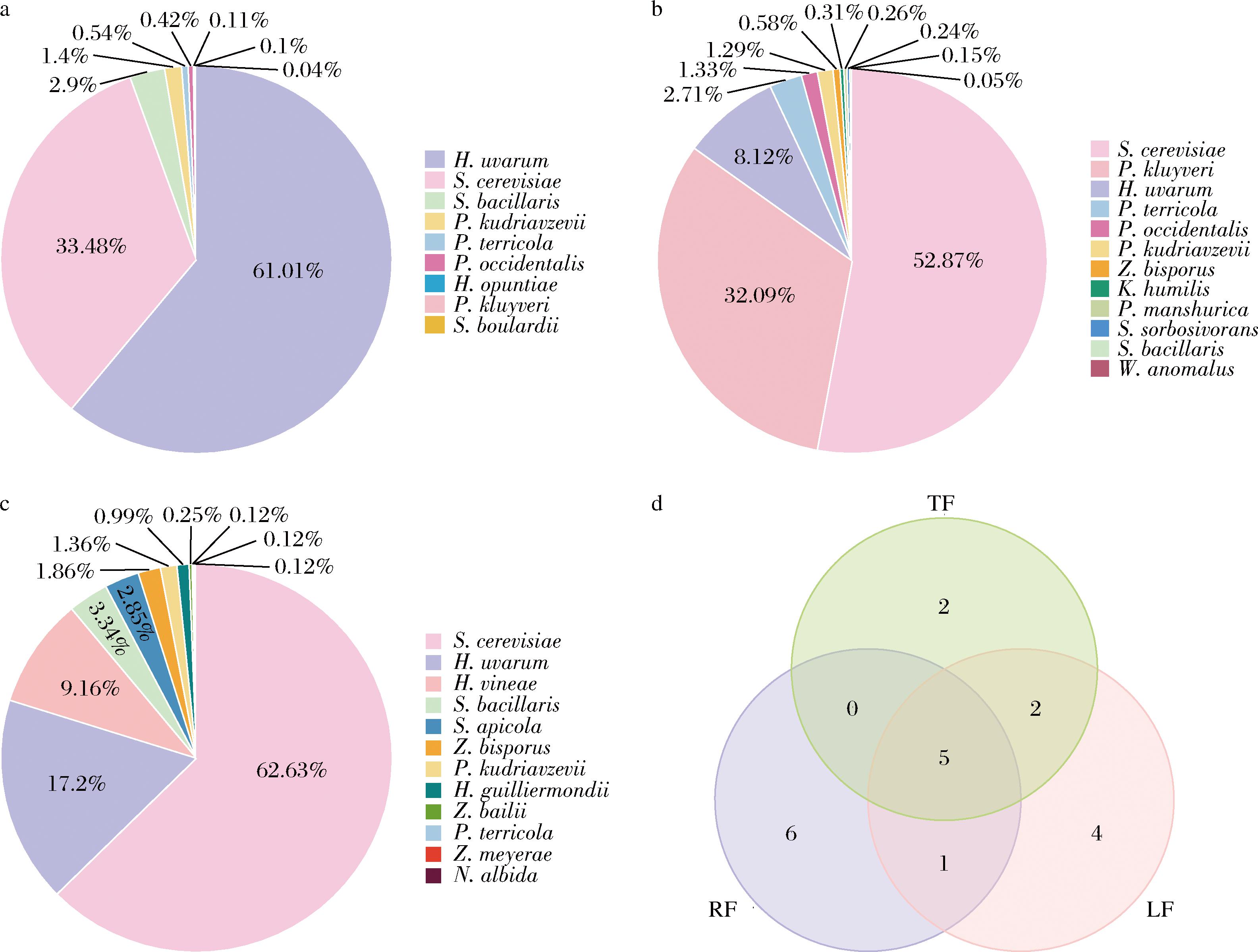
a-TF各酵母菌种所占比例;b-LF各酵母菌种所占比例;c-RF各酵母菌种所占比例;d-酵母属种交互图
图1 三组发酵体系中各酵母属种比例饼图及交互图
Fig.1 Pie charts and interaction plots of the proportion of each yeast genus species in the three fermentation systems
2022年份TF的119株酵母菌鉴定结果分属于4属9种,比例排名前2位的菌种是葡萄汁有孢汉逊酵母(Hanseniaspora uvarum,61.01%)和酿酒酵母(Saccharomyces cerevisiae,33.48%),其次是斯塔梅雷拉假丝酵母(Starmerella bacillaris,2.90%)、库德毕赤酵母(Pichia kudriavzevii,1.40%)、特立科拉毕赤酵母(Pichia terricola,0.54%)、西方毕赤酵母(Pichia occidentalis,0.42%)、仙人掌有孢汉逊酵母(Hanseniaspora opuntiae,0.11%)、克鲁维毕赤酵母(Pichia kluyveri,0.10%)和布拉氏酵母菌(Saccharomyces boulardii,0.04%)。2023年份LF的126株酵母菌鉴定结果分属于7属12种,占分离总菌株比例最高的是S.cerevisiae(52.87%),其次是P.kluyveri(32.09%),还有H.uvarum(8.12%)、P.terricola(2.71%)、P.occidentalis(1.33%)、P.kudriavzevii(1.29%)、双孢合子酵母(Zygosaccharomyces bisporus,0.58%)、矮小假丝酵母(Kazachstania humilis,0.31%)、曼氏毕赤酵母(Pichia manshurica,0.26%)、山梨假丝酵母(Starmerella sorbosivorans,0.24%)、S.bacillaris(0.15%)和异常威克汉姆酵母(Wickerhamomyces anomalus,0.05%)。与2022年(TF)相比,2023年(LF)酵母种类及比例均发生明显变化。2023年份RF的142株酵母菌鉴定结果分属于7属12种,依次为S.cerevisiae(62.62%)、H.uvarum(17.20%)、葡萄酒有孢汉逊酵母(Hanseniaspora vineae,9.16%)、S.bacillaris(3.34%)、蜂房假丝酵母(Starmerella apicola,2.85%)、Z.bisporus(1.86%)、P.kudriavzevii(1.36%)、季也蒙有孢汉逊酵母(Hanseniaspora guilliermondii,0.99%)、拜耳接合酵母(Zygosaccharomyces bailii,0.25%)、梅氏接合酵母(Zygoascus meyerae,0.12%)、P.terricola(0.12%)和浅白隐球酵母菌(Naganishia albida,0.12%)。RF与LF酵母种类总数相同,但菌群结构存在差异。
交互图进一步证实不同年份和不同葡萄园自然发酵过程中酵母菌群群落组成存在差异。3组发酵体系可培养酵母菌种丰富程度由高到低排序为RF=LF>TF。3组发酵体系共有菌种仅5种,分别是S.bacillaris、P.kudriavzevii、P.terricola、H.uvarum、和S.cerevisiae;对于同一葡萄园TF和LF,2个年份共有菌种有7种,差异菌株也有7种;对于同一年份的LF和RF,2个葡萄园共有菌种有6种,差异菌种达12种,菌种差异性更大,据此推测酵母菌群的差异性可能受不同种植区的影响更大。
2.1.2 酵母菌群动态演替分析
3组自然发酵体系中5个发酵时期酵母菌群的相对丰度如图2所示。比较2年份2个葡萄园的品丽珠葡萄酒自然发酵过程可知,发酵过程中各时期酵母的种类与比例均存在差异。在葡萄汁阶段,2022年份TF仅获得2种培养依赖性酵母菌,分别是S.bacillaris(89.89%)和P.occidentalis(10.11%);2023年份LF获得了6种培养依赖性真菌,含量较高的优势菌种为Z.bisporus(52.13%);RF获得了9种培养依赖性真菌,含量较高的优势菌种为H.uvarum(30.96%),其次为H.vineae(23.02%)。进入第二发酵阶段后,3组发酵体系均开始出现酿酒酵母,2022年份TF中酵母菌种增加到7种,H.uvarum(97.43%)成为绝对优势菌种,其他酵母菌种含量较少;而2023年份酵母菌群多样性丰富度开始下降,P.kluyveri(63.23%)成为LF中相对优势菌种,H.uvarum(82.03%)成为RF中含量较高的优势菌种。酿酒酵母出现后,迅速增殖并占据发酵体系的主导地位,体系中物种多样性下降,这与FENG等[18]的研究结论一致。到发酵结束时,LF仅检出S.cerevisiae,而TF和RF中除了S.cerevisiae还有少量P.kudriavzevii。P.occidentalis、P.terricola、P.manshurica、S.sorbosivorans、Z.bisporus、Z.meyerae及W.anomalus在启动发酵前能够存在,但在启动发酵后的任一时期,都难以通过培养获得,这可能是它们的生长受到了乙醇的抑制,或酿酒酵母的拮抗作用[19]。整体而言,2023年份的酵母种类比2022年份多,这可能是受气候因素影响,导致2年份原料糖含量不同,进而影响酵母生长和繁殖,可以推测2023年份的气候条件可能更有利于多种酵母的繁衍发展。
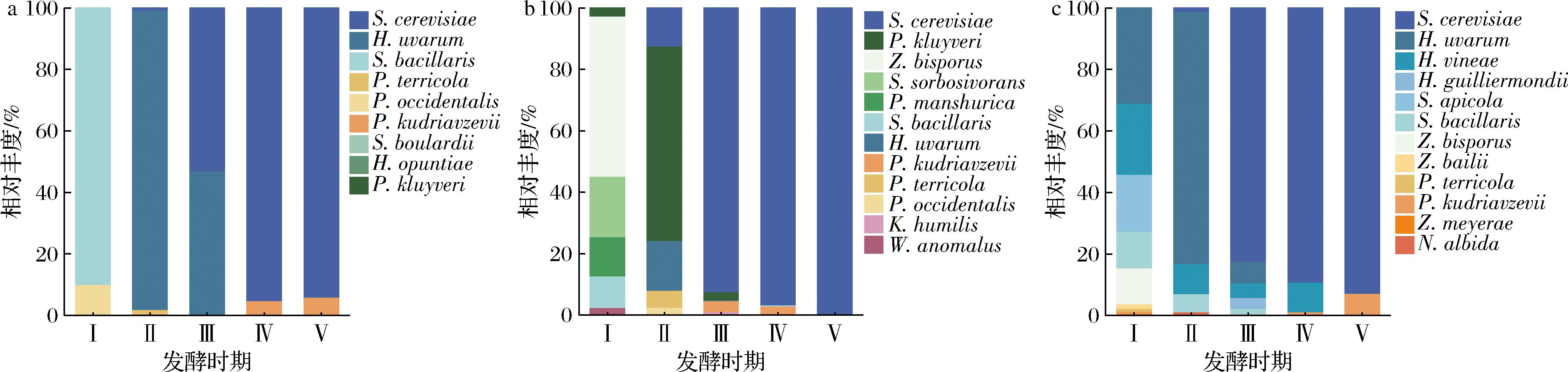
a-TF酵母菌群相对丰度图;b-LF酵母菌群相对丰度图;c-RF酵母菌群相对丰度图
图2 三个发酵体系自然发酵过程中酵母菌群相对丰度图
Fig.2 Plot of relative abundance of yeast flora during natural fermentation in three fermentation systems
综上所述,蓬莱产区品丽珠葡萄酒在自然发酵体系中具有丰富的酵母菌群多样性,菌群分布特征与发酵阶段、年份和种植区有关,说明发酵环境的持续变化对酵母菌群多样性起着重要作用。
2.2 Interdelta指纹图谱分析
上述发酵体系中有248株酵母被鉴定为酿酒酵母。随后利用Interdelta指纹法分析酿酒酵母的遗传多样性,共获得34个基因型,具有较高的遗传多样性,典型Interdelta指纹图谱如图3所示。TF的89株酿酒酵母菌株被区分为20种基因型,编为T;LF的82株酿酒酵母菌株被区分为14种基因型,编为L;RF的77株酿酒酵母菌株被区分为19种基因型,编为R。
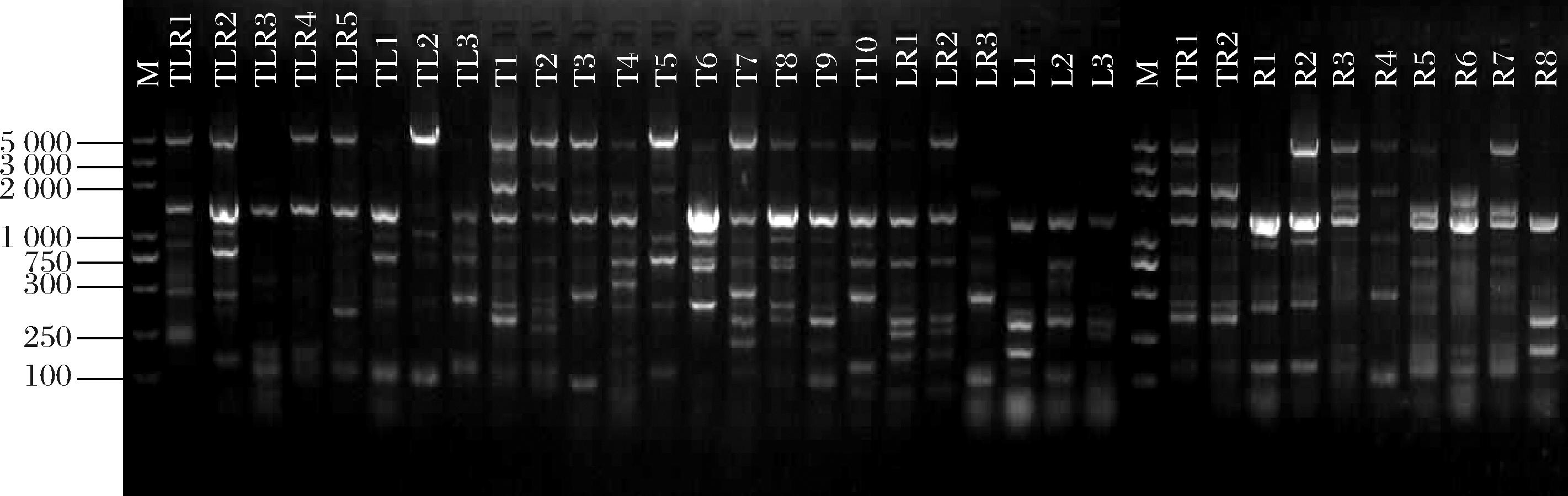
M-5 000 bp marker。
图3 酿酒酵母Interdelta指纹图谱分析图
Fig.3 Interdelta fingerprint analysis of S.cerevisiae strain
3组发酵体系中基因型种类的交集情况如图4所示。3组发酵体系中共有基因型有6种(TLR1~TLR6);仅TF和LF共有基因型有2种(TL1~TL2);仅LF和RF共有基因型有3种(LR1~LR3);仅TF和RF共有基因型有2种(TR1~TR2);TF特有基因型有9种(T1~T9);LF特有基因型有3种(L1~L3);RF特有基因型有8种(R1~R8)。3组发酵体系的酿酒酵母菌株基因型多样性存在差异,这表明酵母菌株的遗传多样性与不同葡萄园及年份密切相关。不同葡萄园和年份的环境条件、原料特性等均可能对酵母菌株的遗传多样性产生影响,进而导致发酵体系中酵母菌株基因型的差异。
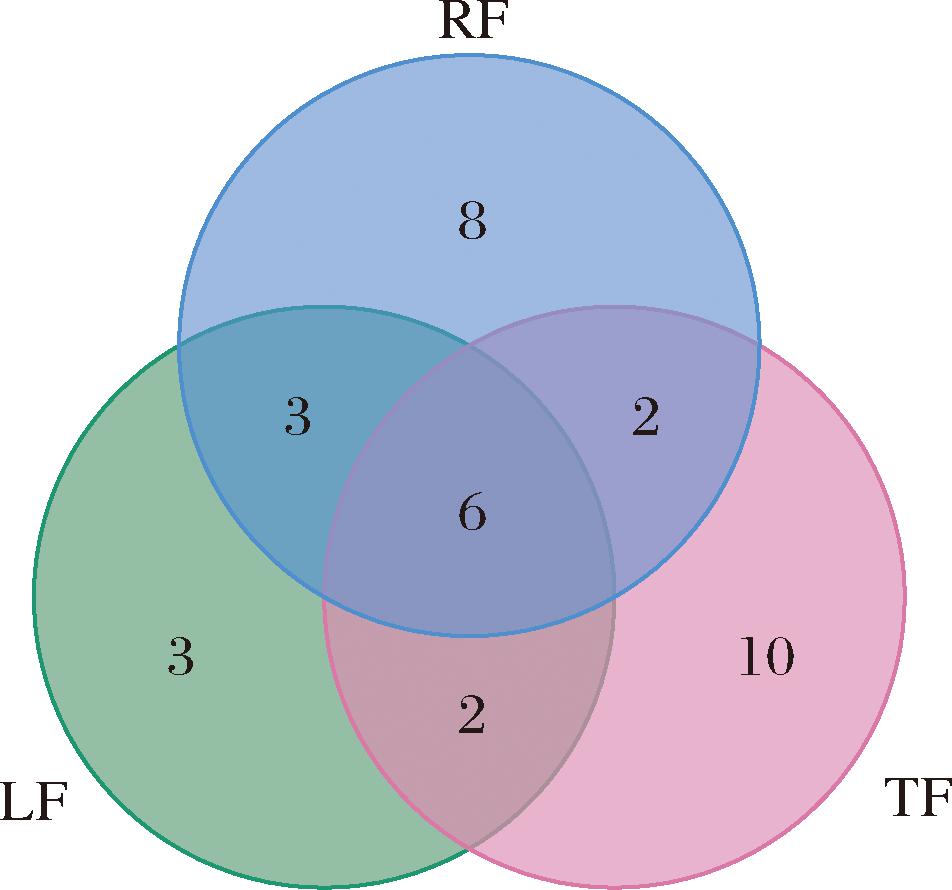
图4 三组发酵体系酿酒酵母菌株基因型种类交互图
Fig.4 Interaction plot of genotypic species of S.cerevisiae strains in three fermentation systems
上述基因型在各发酵时期的分布情况如图5所示,基因型种类的数量和分布呈现出较大差异。在启酵阶段,TF和RF体系中酿酒酵母基因型种类比较单一,分别仅有TR2和TLR5型酿酒酵母菌株,LF体系中有LR1、LR2、TLR1这3种基因型,这可能是由于该阶段发酵体系中酿酒酵母菌株分离量较少。值得注意的是,这些基因型的酿酒酵母菌株在各自的发酵体系中自开始出现至发酵结束的各个阶段均有检测到,具有较强的定殖能力,推测可能是主导发酵的基因型菌株。从发酵旺盛期至发酵结束阶段,3个发酵体系中酿酒酵母基因型均比较丰富,在不同的发酵过程中分离到8~13个基因型,TLR2、TLR5、TR1、TLR6型菌株在TF体系中出现频率或比例较高,LF体系中出现频率或比例较高的为LR1、LR2、TLR1、TLR2型酿酒酵母菌株,TLR3、TLR4、TLR6、R2、R3型菌株在RF体系中出现频率或比例较高,这些遗传菌株对葡萄酒的自然发酵贡献度较大,其他遗传菌株在单个或2个发酵阶段分离,对发酵也有一定的贡献。以上研究结果表明,在整个发酵过程中,3组发酵体系的优势基因型菌株存在差异;优势基因型菌株或源自1~3个发酵体系共有基因型,这些菌株可能具备高效的发酵性能和对环境的良好适应性,可能对葡萄酒发酵起主导作用。
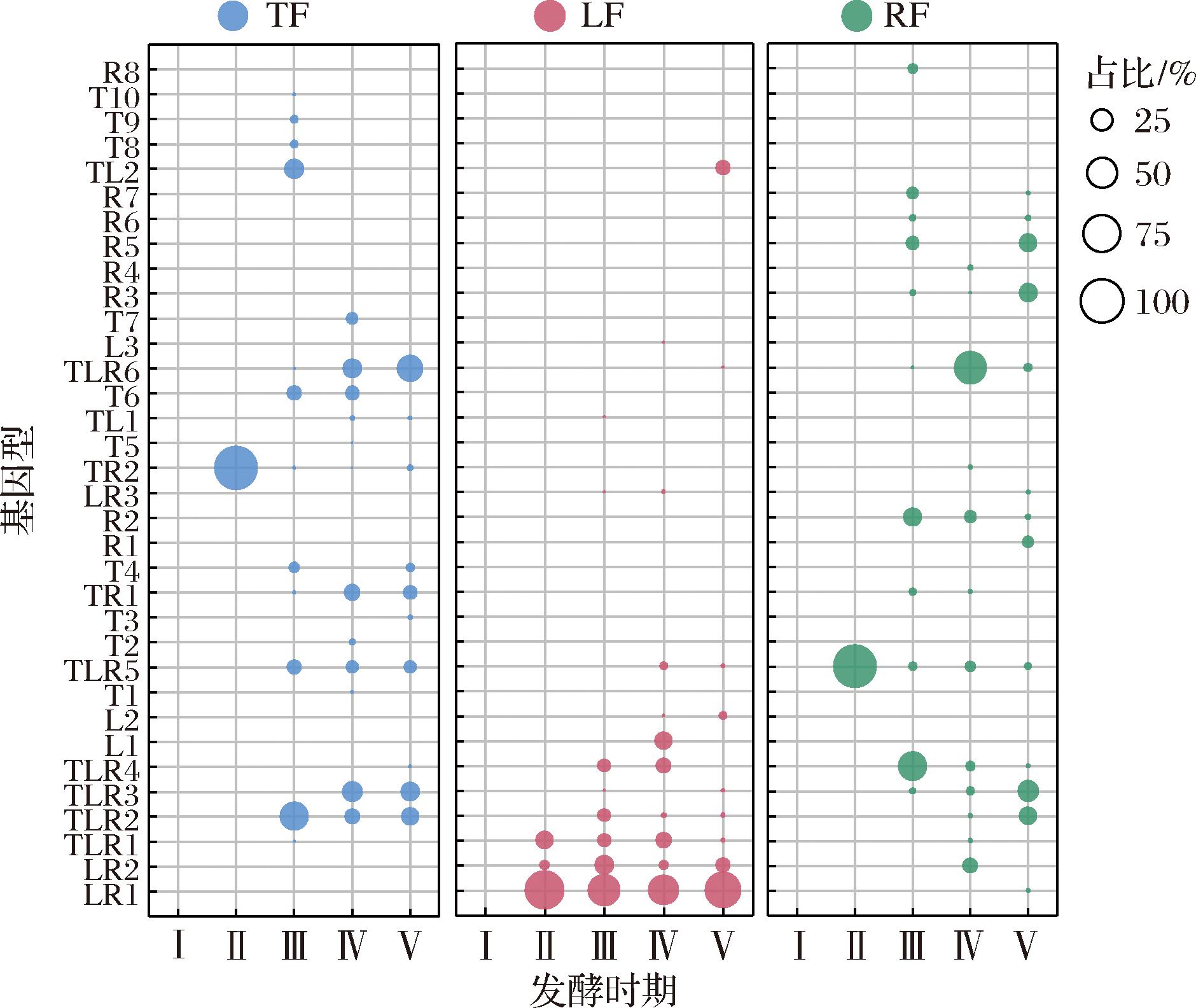
图5 酿酒酵母基因型分布气泡图
Fig.5 Bubble diagram of genotype distribution in S.cerevisiae
为了进一步研究基因型之间的遗传关系,利用Interdelta数据进行UPGMA聚类分析,结果如图6所示。
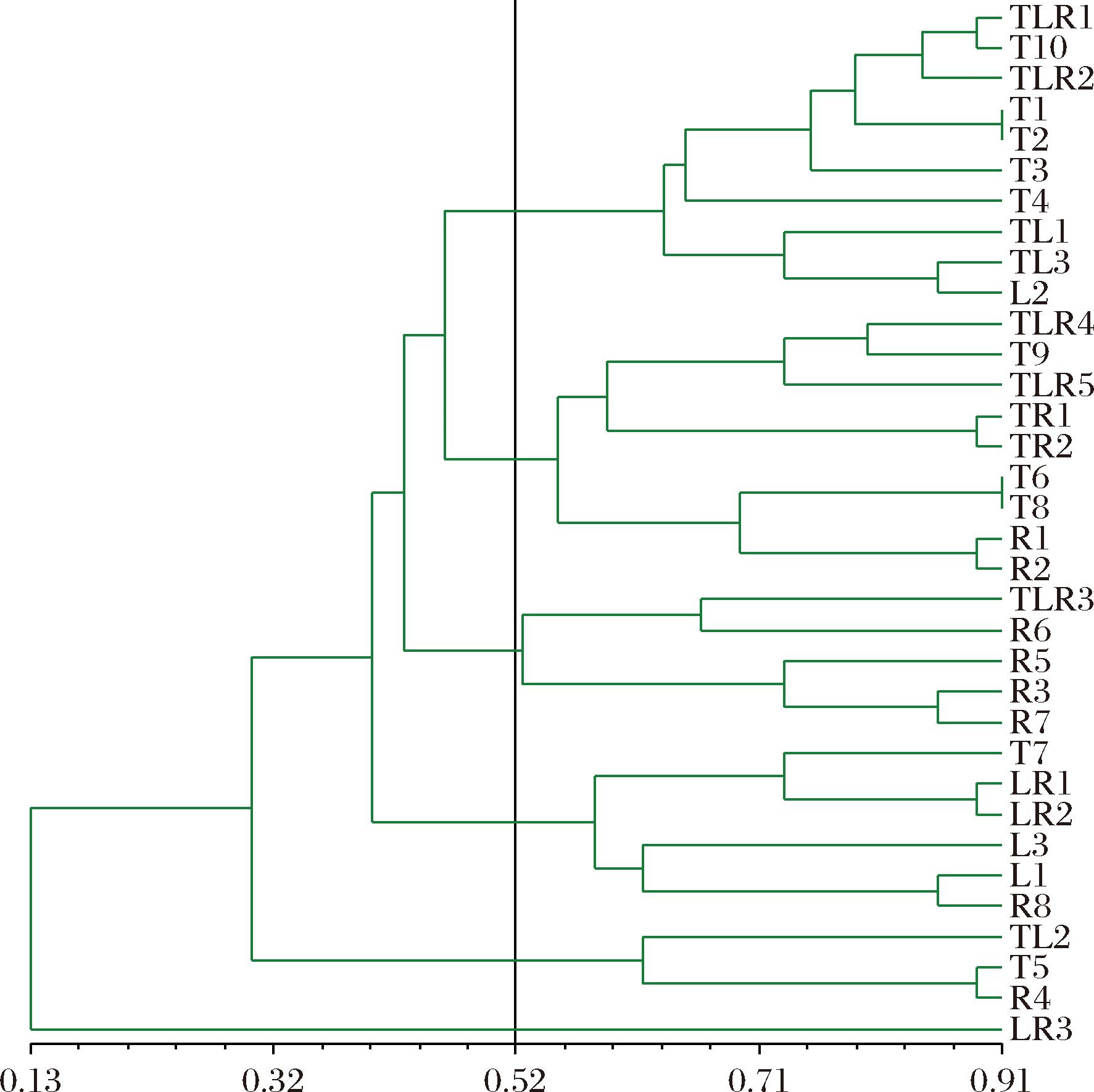
图6 酿酒酵母菌株UPGMA聚类分析图
Fig.6 UPGMA clustering of S.cerevisiae strain
34个基因型在欧氏距离0.52处分为6大类群,其中5个类群由至少2个基因型组成。聚类树状图分析结果显示,第一聚类组收集的10个亲缘关系较近的基因型,源自同一葡萄园的TF与LF发酵体系中特定基因型有8个,另2个为3组发酵体系共有基因型TLR1和TLR2;第三聚类组则收集了4个来自RF发酵体系中特有基因型和1个共有基因型TLR3。此现象表明基因型的聚类受到不同葡萄园的影响,来自同一葡萄园的本土酿酒葡萄菌株可能亲缘关系更近;第2组、第4组、第5组和第6组含有来自2个或3个发酵体系中的基因型,说明同一子区域内本土酿酒葡萄菌株的遗传性具有一定的相似性,这可能意味着它们共享了一些共同的遗传特性或环境适应机制。这些基因型是否可以被认为是该子产区品丽珠葡萄酒的潜在微生物特征还需要进一步的研究。
在本研究中,蓬莱子产区酿酒酵母菌株遗传多样性丰富,且3个发酵体系中酿酒酵母菌株的基因型多样性存在差异,它们在发酵过程中的丰度变化可能取决于2年的气候特征和2个葡萄园的生态条件。
3 结论与讨论
葡萄汁发酵过程中酵母菌群的多样性对葡萄酒的化学成分和感官特性起着非常重要的作用,导致不同产区的葡萄酒具有鲜明的地区差异[20]。本研究以蓬莱海岸带子产区连续2年份2个葡萄园的品丽珠葡萄为原料,研究了3组品丽珠葡萄自发发酵体系中酵母菌群的动态演替,通过菌落形态观察及26S rDNA基因序列分析鉴定获得的387株酵母菌为9个属20个种,表现出蓬莱子产区品丽珠葡萄自然发酵过程中酵母菌群丰富的物种多样性。其中,S.boulardii、P.manshurica、Z.meyerae和K.humilis在葡萄酒领域报道频率相对较低。另外,P.kudriavzevii能够与酿酒酵母共存至发酵结束,具有潜在的发酵性能,CHEN等[21]在对杏酒自然发酵体系的研究中也得到了类似结论;通过进一步对248株酿酒酵母遗传多样性进行表征,获得了34种指纹图谱类型,表现出蓬莱产区酿酒酵母菌株丰富的基因多样性。
3组发酵体系中的酵母菌群组成和结构展现出一定的差异性,这种差异不仅体现在不同年份之间,还体现在不同葡萄园之间。这表明同一子区域内,酵母菌株的多样性和遗传性可能受发酵时期、年份以及种植区域等多种因素的潜在影响,但具体的关联尚需严谨数据验证。在发酵过程中,酵母多样性的变化可能与酵母菌株的酒精耐受性有关,随着酒精浓度的逐渐累积,酒精耐受性较强的酵母菌株逐渐占据主导地位,进一步塑造了酵母菌群的多样性和遗传结构。年份和种植区对酵母菌群多样性和遗传性造成的差异源于气候条件的年际波动及种植区特有的生态环境[22],气候因子、原料特性和栽培管理等因素,它们共同构成独特微环境,对酵母菌群的生长繁殖和遗传特性产生深远影响。因此,来自同一微环境的菌株相似程度更高[23]。
本研究系统认识了蓬莱海岸带子产区品丽珠葡萄自然发酵体系中的酵母菌群多样性及演替规律,为本土特色酵母菌株的选育奠定理论基础,对提升本产区葡萄酒风格独特性具有重要价值。
[1] 张春芝, 江志国.微生物对葡萄酒香气的影响综述[J].中国酿造, 2013, 32(9):28-31.ZHANG C Z, JIANG Z G.Review of microbial effects on wine aroma[J].China Brewing, 2013, 32(9):28-31.
[2] 徐超, 胡文效, 陈明光, 等.葡萄酒发酵香气物质调控研究进展[J].中外葡萄与葡萄酒, 2024(1):81-93.XU C, HU W X, CHEN M G, et al.Advances on regulation of aroma substances in wine fermentation[J].Sino-Overseas Grapevine &Wine, 2024(1):81-93.
[3] GURAKAN G C, AKTUNA I, SEYEDMONIR E.Diversity of wild yeasts during spontaneous fermentation of wines from local grape varieties in Turkey[J].American Journal of Enology and Viticulture, 2022, 73(4):308-320.
[4] BAGHERI B, PHILIPP C, HORACEK M, et al.Microbial diversity in grape musts from Austrian and South African grape varieties and regions[J].BIO Web of Conferences, 2019, 12:02028.
[5] DRUMONDE-NEVES J, FRANCO-DUARTE R, VIEIRA E, et al.Differentiation of Saccharomyces cerevisiae populations from vineyards of the Azores Archipelago:Geography vs Ecology[J].Food Microbiology, 2018, 74:151-162.
[6] CAPECE A, ROMANIELLO R, SIESTO G, et al.Diversity of Saccharomyces cerevisiae yeasts associated to spontaneously fermenting grapes from an Italian “heroic vine-growing area”[J].Food Microbiology, 2012, 31(2):159-166.
[7] SUN Y, QIN Y, PEI Y F, et al.Evaluation of Chinese Saccharomyces cerevisiae wine strains from different geographical origins[J].American Journal of Enology and Viticulture, 2017, 68(1):73-80.
[8] LI R L, LIN M Y, GUO S J, et al.A fundamental landscape of fungal biogeographical patterns across the main Chinese wine-producing regions and the dominating shaping factors[J].Food Research International, 2021, 150:110736.
[9] 魏如腾. 葡萄-葡萄酒自然体系中微生物多样性及其对葡萄酒风味物质的作用[D].杨凌:西北农林科技大学, 2023.WEI R T.Microbial diversity in grape-wine natural systems and its role in wine flavour compounds[D].Yangling:Northwest A&F University, 2023.
[10] 汤晓宏. 蓬莱葡萄酒产区酿酒酵母的选育—中粮君顶酒庄酿酒酵母的筛选[D].济南:齐鲁工业大学, 2014.TANG X H.The breeding of Saccharomyces cerevisiae in Penglai wine region—The screening of Saccharomyces cerevisiae of Chateau junding[D].Jinan:Qilu University of Technology, 2014.
[11] WANG C P, CHEN X Q, REN Y H, et al.Author Correction:Geographical location influence ‘Cabernet Franc’ fruit quality in Shandong province[J].Scientific Reports, 2024, 14:7942.
[12] LIANG L H, MA Y W, JIANG Z Z, et al.Dynamic analysis of microbial communities and flavor properties in Merlot wines produced from inoculation and spontaneous fermentation[J].Food Research International, 2023, 164:112379.
[13] COSTANTINI A, VAUDANO E, PULCINI L, et al.Yeast biodiversity in vineyard during grape ripening:Comparison between culture dependent and NGS analysis[J].Processes, 2022, 10(5):901.
[14] 刘佳禾, 王蕊, 周叶丽, 等.宁夏贺兰山东麓产区维欧尼葡萄酒自然发酵过程中酵母菌群多样性研究[J].中国酿造, 2024, 43(7):73-78.LIU J H, WANG R, ZHOU Y L, et al.Diversity of yeast community during spontaneous fermentation of Viognier wine from the eastern foothills of Helan Mountain in Ningxia[J].China Brewing, 2024, 43(7):73-78.
[15] 周小玲, 沈微, 饶志明, 等.一种快速提取真菌染色DNA的方法[J].微生物学通报, 2004, 31(4):89-92.ZHOU X L, SHEN W, RAO Z M, et al.A rapid method for preparation of fungal chromosome DNA[J].Microbiology, 2004, 31(4):89-92.
[16] KUMAR S, STECHER G, LI M, et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution, 2018, 35(6):1547-1549.
[17] 杨慧敏, 任晓娜, 秦义.四川甘孜州高山葡萄酒产区酵母菌多样性分析[J].微生物学通报, 2022, 49(7):2673-2685.YANG H M, REN X N, QIN Y.Diversity analysis of yeasts in the Ganzi alpine wine region, Sichuan[J].Microbiology China, 2022, 49(7):2673-2685.
[18] FENG L, WANG J M, YE D Q, et al.Yeast population dynamics during spontaneous fermentation of icewine and selection of indigenous Saccharomyces cerevisiae strains for the winemaking in Qilian, China[J].Journal of the Science of Food and Agriculture, 2020, 100(15):5385-5394.
[19] HUANG R, JIANG J, SU Y, et al.Microbial community succession and volatile compounds changes during spontaneous fermentation of Cabernet Sauvignon (Vitis vinifera L.) under rain-shelter cultivation[J].Food Chemistry:X, 2023, 17:100618.
[20]  URANSK
URANSK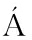 H, VR
H, VR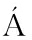 NOV
NOV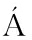 D, OMELKOV
D, OMELKOV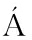 J.Isolation, identification and characterization of regional indigenous Saccharomyces cerevisiae strains[J].Brazilian Journal of Microbiology, 2016, 47(1):181-190.
J.Isolation, identification and characterization of regional indigenous Saccharomyces cerevisiae strains[J].Brazilian Journal of Microbiology, 2016, 47(1):181-190.
[21] CHEN Y, QI J L, YANG H Y, et al.Fungal dynamic during apricot wine spontaneous fermentation and aromatic characteristics of Pichia kudriavzevii for potential as starter[J].Food Chemistry:X, 2023, 19:100862.
[22] VAN LEEUWEN C, SEGUIN G.The concept of terroir in viticulture[J].Journal of Wine Research, 2006, 17(1):1-10.
[23] MART NEZ C, COSGAYA P, V
NEZ C, COSGAYA P, V SQUEZ C, et al.High degree of correlation between molecular polymorphism and geographic origin of wine yeast strains[J].Journal of Applied Microbiology, 2007, 103(6):2185-2195.
SQUEZ C, et al.High degree of correlation between molecular polymorphism and geographic origin of wine yeast strains[J].Journal of Applied Microbiology, 2007, 103(6):2185-2195.