近年来,由于高糖饮食导致患有肥胖、糖尿病和高血压等疾病人数激增,因此低热量的代糖引起广泛关注[1]。D-阿洛酮糖是D-果糖在C3位上的差向异构体[2],其甜度是蔗糖的70%,但热量只有蔗糖的10%[3],同时还具有多种生理功能,例如通过激活葡萄糖激酶积累肝糖原,减少葡萄糖的转运,维持血糖平衡[4];调节脂质代谢,避免脂肪积累[5]。因此,D-阿洛酮糖作为一种低卡路里的甜味剂,备受市场关注。
目前,D-阿洛酮糖工业生产主要通过酶法制备[6],其关键用酶是D-阿洛酮糖3-差向异构酶(D-allulose 3-epimerase,DAEase)[7]。DAEase可以D-果糖为底物,通过差向异构反应生成D-阿洛酮糖[8],但DAEase仍需通过分子改造进一步提升其应用性能以满足日益增长的市场需求。定向进化是提升酶性能的有效分子改造策略,筛选方法的筛选通量是限制其效率的关键因素之一[9]。为实现超高通量筛选,转录因子型生物传感器成为优选工具[10-11]。转录因子型生物传感器是一种利用转录因子与其特定DNA序列之间的相互作用来检测特定信号分子的生物工具[12]。转录因子型生物传感器可以通过改变报告基因的表达水平来反映所响应信号分子的浓度,从而实现定量或半定量的检测[13]。通过将转录因子型生物传感器与荧光激活细胞分选术(fluorescence-activated cell sorting,FACS)相结合,可将筛选通量提高至108个细胞/h,将极大提高文库筛选效率[11]。PANDI等[14]发现了一种来自根瘤农杆菌的转录因子PsiR,可特异性识别D-阿洛酮糖,并调控位于启动子PpsiA下游报告基因的表达。在没有D-阿洛酮糖存在的情况下,PsiR可以特定结合在PpsiA上,阻碍下游报告基因的表达,而D-阿洛酮糖可与PsiR特异性结合并使其从PpsiA上解离下来,从而消除对下游报告基因的阻遏作用。利用这一原理,ARMETTA等[15]开发了一种可特异性响应D-阿洛酮糖的生物传感器并用于DAEase突变体文库的超高通量筛选。
本研究中,为提高Clostridium cellulolyticum H10来源的DAEase(CcDAEase)的比活力,我们首先构建了CcDAEase的易错PCR(error-prone PCR,epPCR)突变体文库,并进一步优化了基于转录因子PsiR的D-阿洛酮糖特异型生物传感器及筛选条件,对CcDAEase突变体文库进行超高通量初筛,并结合微孔板筛选对初筛获得潜在优势突变体进行高通量复筛。最终,经过一轮的文库构建以及筛选获得了比活力提高14.6%的优势突变体H209Q,为D-阿洛酮糖的工业制备提供了理论依据和技术支撑。
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
克隆宿主Escherichia coli JM109,表达宿主E.coli BL21(DE3),重组菌E.coli BL21(DE3)/pET-20b(+)-ccdae以及质粒pSB1C3-psir-PpsiA-mEmeral-T7 term由本实验室保藏。
1.1.2 试验试剂
HEPES缓冲液:20.0 mmol/L HEPES,0.1 mmol/L CoCl2·6H2O,pH 7.5。
纯化缓冲A液:5.8 g/L Na2HPO4,1.5 g/L NaH2PO4,29.3 g/L NaCl,pH 7.5。
纯化缓冲B液:先配制纯化洗脱母液(500.0 mmol/L咪唑,用纯化缓冲A液配制),再与纯化缓冲A液混合即为纯化缓冲B液。
氨苄青霉素(ampicillin,Amp)、溶菌酶、氯霉素(chloramphenicol,Cm),上海生工生物工程股份有限公司;盐酸四环素(tetracycline hydrochloride,Tet)、4-羟乙基哌嗪乙磺酸{2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid,HEPES},上海维塔化学试剂有限公司;常用化学试剂购自国药集团;D-阿洛酮糖液相标品,日本GlyCarbo公司;镍柱填料,美国GE公司;分子操作相关试剂以及酶制剂,宝日医生物技术(北京)有限公司;质粒小提试剂盒与普通琼脂糖凝胶DNA回收试剂盒,天根生化有限公司。
1.1.3 培养基
LB培养基(g/L):酵母粉5.0,NaCl 10.0,胰蛋白胨10.0。
TB培养基(g/L):甘油5.0,胰蛋白胨12.0,酵母粉24.0,KH2PO4 2.3,K2HPO4 12.54。
M9培养基:15.00 g/L磷酸二氢钾,35.50 g/L磷酸氢二钠,2.50 g/L氯化钠,5.00 g/L氯化铵,2.00 mmol/L七水合硫酸镁,0.10 mmol/L无水氯化钙和终质量分数为0.40%的葡萄糖分开灭菌再混合。
1.2 实验方法
1.2.1 筛选质粒的构建
使用引物pBS1C3-psir-PpsiA-mEmerald-ccdae-F/R(CAATTGTACAAAGAGGAGAAACTCGAGGATGAAAC ATGGCATCTATTATGCCTATTGGGAAC/GAGGCCCCA AGGGGTTATGCTAGTCAGTGGTGGTGGTGGTGGTGCT CGA)对ccdae目的基因进行PCR扩增,并使用MEGAWHOP(megaprimer PCR of whole plasmid)[16]将pSB1C3-psir-PpsiA-mEmerald-T7 term载体骨架与目的基因进行连接,得到pSB1C3-psir-PpsiA-mEmerald-ccdae-T7 term质粒,作为筛选质粒。
1.2.2 CcDAEase突变体文库的构建及筛选
以ep-PCR-F/R(GTACAAAGAGGAGAAACTCGA GGATG/TCAGTGGTGGTGGTGGTGGTGCTCGAG)为引物,通过epPCR构建ccdae突变体文库,并转化到E.coli BL21(DE3)中,然后将重组菌置于含20 mmol/L D-果糖以及35 μg/mL Cm的LB中培养10 h,离心收集菌体,用鞘液(PBS缓冲液,pH 7.4)重悬菌体并稀释OD600值至0.3。利用BD FACSArica Ⅲ对文库进行超高通量分选,荧光值超过野生型(wild type,WT)前0.5%的单细胞中分选并涂布到含有35 μg/mL Cm的LB固体培养基上,置于37 ℃恒温培养10 h,然后挑取单菌落到含有100 μL LB培养基(Cm,35 μg/mL)的96微孔板中,并按照BOSSHART等[17]建立的微孔板筛选方法对其进行复筛。
1.2.3 CcDAEase酶学性质测定
1.2.3.1 CcDAEase纯化
将发酵液离心获得菌体,重悬并超声破碎细胞,再经过离心获得破壁上清液,并用0.22 μm水相滤膜过滤获得粗酶液。将粗酶液通过镍离子亲和层析柱,先用含有50 mmol/L咪唑的纯化缓冲B液洗脱杂蛋白,再用含有300 mmol/L咪唑的纯化缓冲B液洗脱目的蛋白。利用截留分子质量为10 kDa的超滤管对含有目的蛋白的洗脱液进行浓缩,并加入HEPES缓冲液替换纯化缓冲B液。最后用Bradford蛋白浓度测定试剂盒测定样品蛋白浓度。
1.2.3.2 CcDAEase比活力测定
酶活力测定:将800 μL含有100 g/L D-果糖的HEPES缓冲液与200 μL酶液充分混合后,置于60 ℃下精准反应10 min,立即将反应液置于沸水中煮10 min。底物溶液需提前在反应温度下预热10 min。
酶活力定义:反应条件为60 ℃与pH 7.5时,每分钟内每生成1 μmol D-阿洛酮糖所用的酶量为1个酶活力单位(U)。
1.2.3.3 CcDAEase最适温度和最适pH值测定
为测定CcDAEase WT与突变体的最适温度,分别在50、55、60、65、70、75 ℃下测定其酶活力,其余反应条件与1.2.3.2节中酶活力测试方法一致,计算对应条件下的相对酶活力,确定WT与突变体的最适温度。
为测定CcDAEase WT与突变体的最适pH值,在60 ℃下,分别添加0.1 mmol/L Co2+的磷酸缓冲液(20 mmol/L,pH 6.0~7.0)、HEPES 缓冲液(pH 7.5~8.0)、Tris-HCI缓冲液(20 mmol/L,pH 8.5)中,在最适反应温度下测定其酶活力,其余反应条件与1.2.3.2节中酶活力测试方法一致,计算对应条件下的相对酶活力,确定WT与突变体的最适pH值。
1.2.3.4 CcDAEase热稳定性测定
为测定WT以及突变体的热稳定性,分别将其置于60 ℃下孵育,定点取样并按1.2.3.2节酶活力测定法测定残余酶活力。
1.2.3.5 CcDAEase最适温度和最适pH值测定
将反应液离心取上清液,用纯水进行适当稀释并通过0.22 μm水系滤膜过滤。使用HPLC测定,具体条件为:Waters e2695(示差检测器),ShodexTM AsahipakTM NH2P-50 4E色谱柱,柱温和检测温度均为35 ℃,流动相为V(乙腈)∶V(水)=75∶25,流速为0.8 mL/min。
2 结果与分析
2.1 D-阿洛酮糖生物传感器的构建
前期研究过程中,首先参照ARMETTA等[15]的前期工作,将D-阿洛酮糖特异型转录因子PsiR、启动子PpsiA、CcDAEase以及报告基因等元件进行组装,获得了重组质粒pSB1C3-psir-PpsiA-mEmerald-ccdae,并转化进E.coli BL21 (DE3),构建了D-阿洛酮糖特异型生物传感器,其原理如图1-a所示。然而,在后期CcDAEase突变体文库筛选过程中,发现该传感器存在一定的缺陷;因此,我们对其进行了相应的优化,例如筛选条件的优化、重组质粒中转录终止子的替换以及表达宿主的筛选等,提升了筛选效果[18]。对优化后的生物传感器进行底物特异性测定,分别将其与D-果糖及D-阿洛酮糖孵育,测定其荧光强度。检测结果如图1-b所示,在无糖或者仅有D-果糖存在情况下,生物传感器不产生荧光信号,但在含有D-阿洛酮糖的情况下,生物传感器的荧光信号会显著提升,表明该传感器对D-阿洛酮糖具有特异性响应,并且具有较高的灵敏度,可用于后续CcDAEase突变体文库的超高通量筛选。
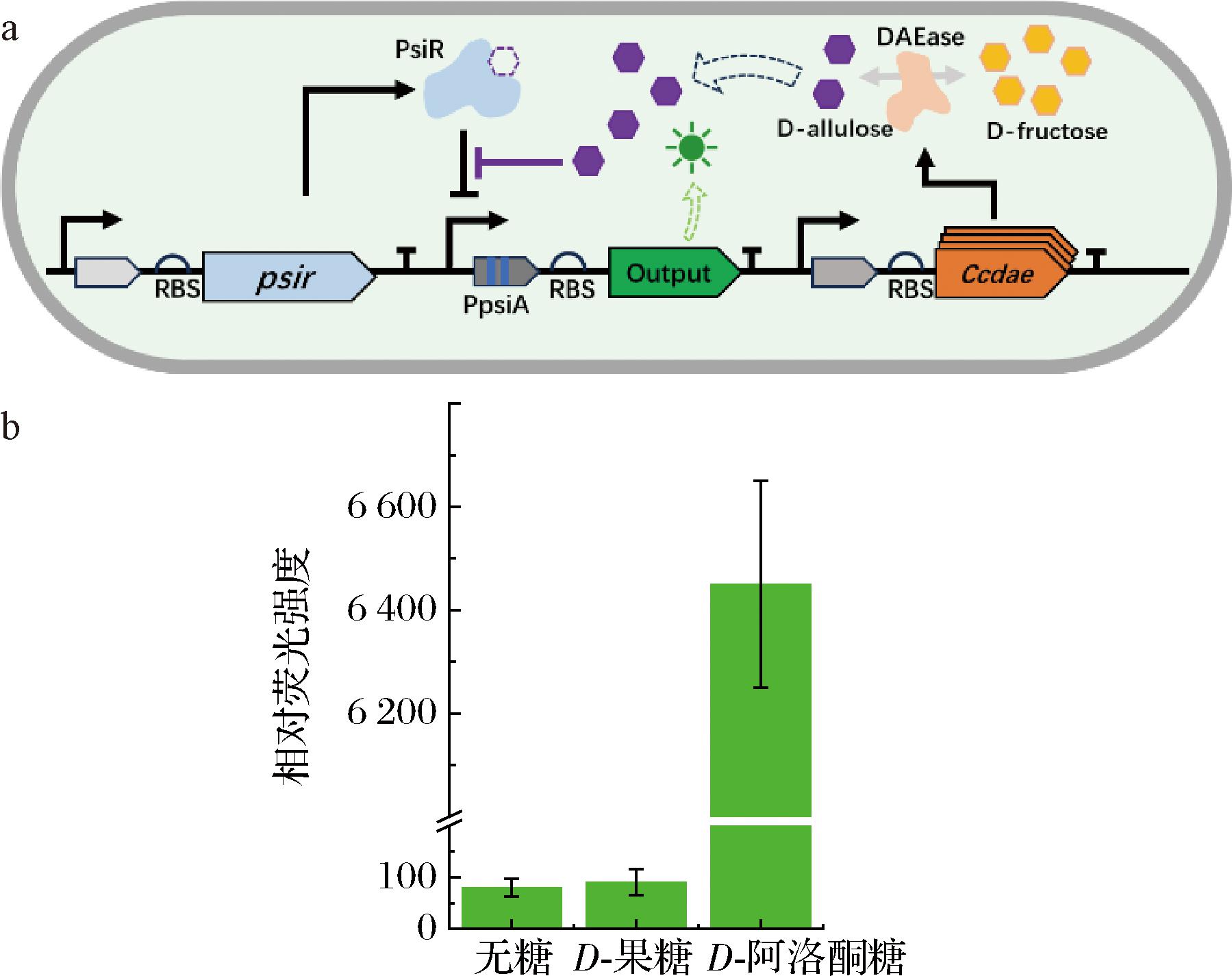
a-PsiR生物传感器示意图;b-对D-果糖以及D-阿洛酮糖的响应情况
图1 PsiR生物传感器示意图及其对D-果糖以及D-阿洛酮糖的响应情况
Fig.1 Schematic diagram of PsiR-based biosensor and its response to D-fructose and D-allulose
注:RBS代表核糖体结合位点。
2.2 CcDAEase优势突变体筛选
首先,构建了CcDAEase epPCR突变体文库,结合生物传感器以及FACS对该文库进行超高通量筛选,以CcDAEase WT的荧光信号为对照,分选了约20 000 个克隆并收集文库中荧光值高于WT前0.5%的单克隆,最终获得100株潜在优势突变体(图2)。然后,通过96孔微孔板法对初筛获得的潜在优势突变体进行高通量复筛,最终,对复筛效果最优的突变体进行测序分析,确定其突变位点是H209Q。为获得足量的CcDAEase蛋白以进行后续的酶学性质表征,将该突变体基因片段与表达载体pET-20b (+)构建重组表达载体pET-20b (+)-ccdae H209Q,并转化至E.coli BL21 (DE3)。
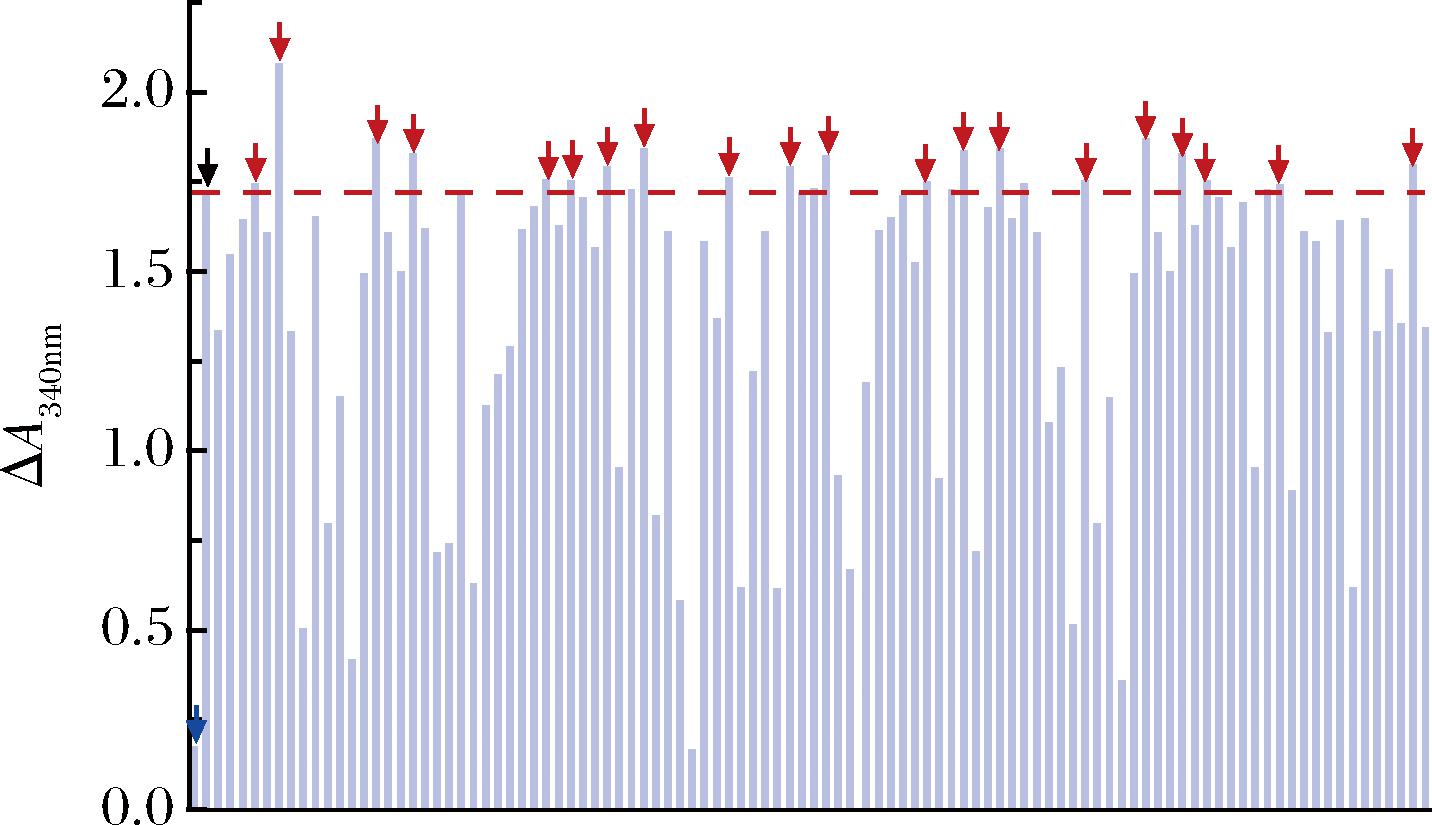
图2 突变体文库复筛结果
Fig.2 Rescreening result of mutant library
注:不加酶的空白对照(蓝色箭头),野生型(黑色箭头),潜在的优势突变体(红色箭头)。
2.3 CcDAEase WT及突变体H209Q的表达纯化及酶学性质测定
分别对CcDAEase WT及突变体H209Q进行重组表达,并通过对组氨酸亲和层析过程中洗脱液咪唑浓度以及洗脱步骤进行优化,并分别对CcDAEase WT及突变体H209Q进行纯化,其纯化结果如图3所示,获得了分子质量符合预期的单一条带,表明获得了纯度较高的目的蛋白。

M-Marker;1-CcDAEase WT纯化蛋白;2-CcDAEase H209Q纯化蛋白
图3 CcDAEase WT及突变体H209Q纯化蛋白SDS-PAGE结果
Fig.3 SDS-PAGE of purified CcDAEase WT and mutant H209Q
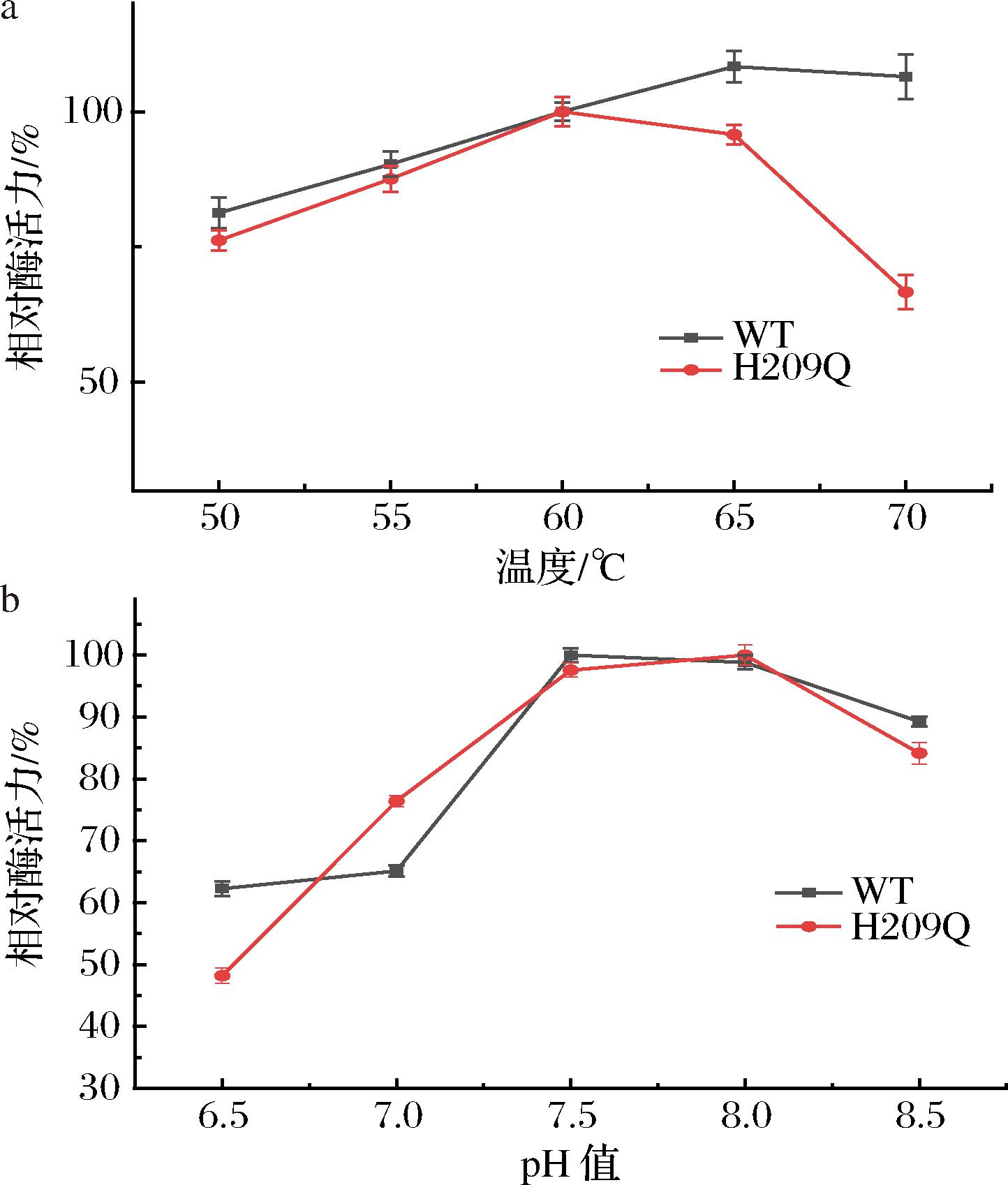
a-最适温度;b-最适pH值
图4 CcDAEase WT与H209Q最适温度与最适pH曲线
Fig.4 Optimum temperature and optimum pH curves of CcDAEase WT and H209Q
对CcDAEase WT与突变体H209Q进行比活力测定,其结果如表1所示,突变体H209Q比活力较WT提高了14.6%,相较于ARMETTA等[15]通过D-阿洛酮糖生物传感器筛选获得的突变体A142 N具有更高的比活力,表明本工作中对该传感器的优化具有一定的应用价值。
表1 CcDAEase WT和H209Q比活力
Table 1 The specific activity of CcDAEase WT and H209Q

酶名称比活力/(U/mg)WT 437.03±8.81H209Q500.81±10.39
为了进一步确定突变体H209Q的最适温度和最适pH值,本文测定了CcDAEase WT与突变体H209Q的酶活力-温度/酶活力-pH值曲线,其结果如图3-a和图3-b所示,WT最适温度为65 ℃,突变体H209Q的最适温度为60 ℃;WT与H209Q的最适pH值分别为7.5和8.0。
为进一步表征热稳定性,分别将CcDAEase WT及H209Q置于60 ℃下孵育,并测定其残余酶活力。结果如图5所示,H209Q的热稳定性略低于WT。这可能是由于H209Q突变位点对该酶的整体稳定性产生了影响,从而降低了酶的热稳定性。
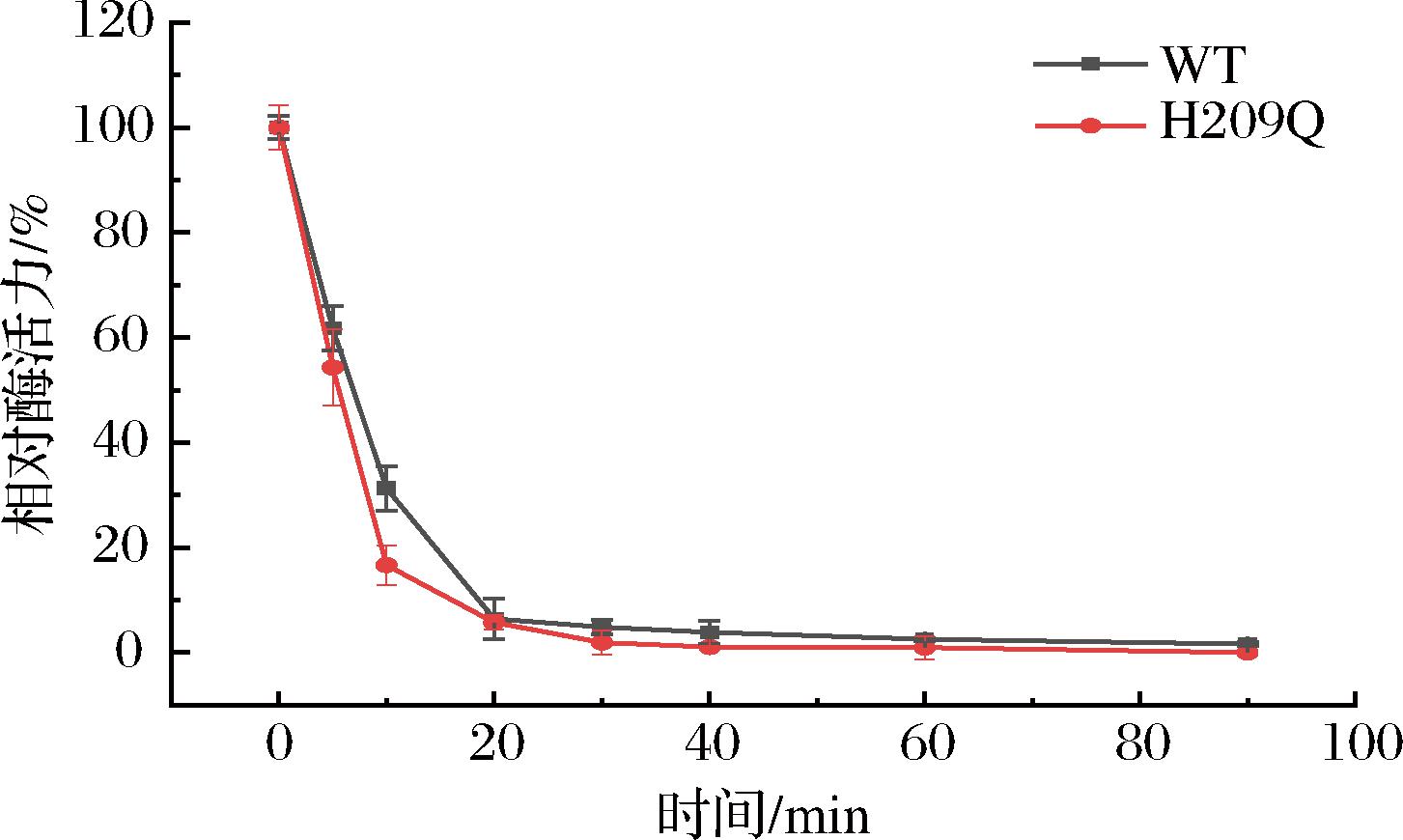
图5 CcDAEase WT及H209Q热稳定性曲线
Fig.5 Thermal stability curves of CcDAEase WT and H2O9Q
2.4 CcDAEase H209Q构效关系分析
通过结构分析发现,H209位点位于酶表面,靠近活性中心的入口处,并且与底物D-果糖以及E34、L182、T184存在氢键相互作用,形成氢键网络维持构象(图6-a),并且可能会影响底物的结合和解离。该位点突变成Q以后,该位点与底物D-果糖的氢键作用消失,导致该区域的氢键网络减弱,因此,可能会增加局部结构的柔性,从而影响了底物的进入和离开速率[19],因此,我们推测这是突变体H209Q比活力提升的可能原因。同时,极有可能是由于突变位点H209Q区域氢键网络的减弱导致了该突变体热稳定性的降低,该结果与前期筛选获得的临近突变位点H207L具有类似效果[20]。后期研究中,可以在突变体H209Q的基础上,继续叠加热稳定性提升的突变位点,以进一步提高其热稳定性及工业应用价值。

a-CcDAEase WT(PDB ID:3VNK);b-突变体H209Q
图6 CcDAEase WT(PDB ID:3VNK)及突变体H209Q结构分析
Fig.6 Structural analysis of CcDAEase WT (DPB ID:3VNK) and mutant H209Q
3 结论
本研究构建并优化了D-阿洛酮糖特异型生物传感器,并将其作为超高通量筛选工具用于CcDAEase定向进化。通过构建CcDAEase epPCR突变体文库,并经过一轮FACS超高通量筛选以及一轮96孔微孔板复筛,成功获得了比活力提升14.6%的优势突变体CcDAEase H209Q。本研究不仅展示了转录因子型生物传感器在酶定向进化中的应用价值,而且为D-阿洛酮糖的工业化生产提供了技术支撑。
[1] ROGERS P J, HOGENKAMP P S, DE GRAAF C, et al.Does low-energy sweetener consumption affect energy intake and body weight? A systematic review, including meta-analyses, of the evidence from human and animal studies[J].International Journal of Obesity (2005), 2016, 40(3):381-394.
[2] KIMURA T, KANASAKI A, HAYASHI N, et al.D-Allulose enhances postprandial fat oxidation in healthy humans[J].Nutrition, 2017, 43:16-20.
[3] CHEN D, CHEN J J, LIU X Y, et al.Biochemical identification of a hyperthermostable L-ribulose 3-epimerase from Labedella endophytica and its application for D-allulose bioconversion[J].International Journal of Biological Macromolecules, 2021, 189:214-222.
[4] HOSSAIN A, YAMAGUCHI F, HIROSE K, et al.Rare sugar D-psicose prevents progression and development of diabetes in T2DM model Otsuka Long-Evans Tokushima Fatty rats[J].Drug Design, Development and Therapy, 2015, 9:525-535.
[5] NATSUME Y, YAMADA T, IIDA T, et al.Investigation of D-allulose effects on high-sucrose diet-induced insulin resistance via hyperinsulinemic-euglycemic clamps in rats[J].Heliyon, 2021, 7(9):e08013.
[6] BEERENS K, DESMET T, SOETAERT W.Enzymes for the biocatalytic production of rare sugars[J].Journal of Industrial Microbiology &Biotechnology, 2012, 39(6):823-834.
[7] ISHIDA Y, KAMIYA T, ITOH H, et al.Cloning and characterization of the D-tagatose 3-epimerase gene from Pseudomonas cichorii ST-24[J].Journal of Fermentation and Bioengineering, 1997, 83(6):529-534.
[8] HU M Y, LI M L, JIANG B, et al.Bioproduction of D-allulose:Properties, applications, purification, and future perspectives[J].Comprehensive Reviews in Food Science and Food Safety, 2021, 20(6):6012-6026.
[9] NIRANTAR S R.Directed evolution methods for enzyme engineering[J].Molecules.2021, 26(18):5599.[10] XIAO D, ZHANG W, GUO X T, et al.A D-2-hydroxyglutarate biosensor based on specific transcriptional regulator DhdR[J].Nature Communications, 2021, 12(1):7108.
[11] LIU Z Z, CHEN S, WU J.Advances in ultrahigh-throughput screening technologies for protein evolution[J].Trends in Biotechnology, 2023, 41(9):1168-1181.
[12] TELLECHEA-LUZARDO J, STIEBRITZ M T, CARBONELL P.Transcription factor-based biosensors for screening and dynamic regulation[J].Frontiers in Bioengineering and Biotechnology, 2023, 11:1118702.
[13] MACHADO L F M, DIXON N.Directed evolution of transcription factor-based biosensors for altered effector specificity[J].Methods in Molecular Biology, 2022, 2461:175-193.
[14] PANDI A, GRIGORAS I, BORKOWSKI O, et al.Optimizing cell-free biosensors to monitor enzymatic production[J].ACS Synthetic Biology, 2019, 8(8):1952-1957.
[15] ARMETTA J, BERTHOME R, CROS A, et al.Biosensor-based enzyme engineering approach applied to psicose biosynthesis[J].Synthetic Biology, 2019, 4(1):ysz028.
[16] MIYAZAKI K.MEGAWHOP cloning:A method of creating random mutagenesis libraries via megaprimer PCR of whole plasmids[J].Methods in Enzymology, 2011, 498:399-406.
[17] BOSSHART A, HEE C S, BECHTOLD M, et al.Directed divergent evolution of a thermostable d-tagatose epimerase towards improved activity for two hexose substrates[J].ChemBioChem, 2015, 16(4):592-601.
[18] LIU Z Z, LIU S H, JIA J Y, et al.Optimization of ultrahigh-throughput screening assay for protein engineering of D-allulose 3-epimerase[J].Biomolecules, 2022, 12(11):1547.
[19] ZHU J X, LI Y, WANG J Z, et al.Adaptive steered molecular dynamics combined with protein structure networks revealing the mechanism of Y68I/G109P mutations that enhance the catalytic activity of D-psicose 3-epimerase from Clostridium bolteae[J].Frontiers in Chemistry, 2018, 6:437.
[20] LIU Z Z, WANG Y F, LIU S H, et al.Boosting the heterologous expression of D-allulose 3-epimerase in Bacillus subtilis through protein engineering and catabolite-responsive element box engineering[J].Journal of Agricultural and Food Chemistry, 2022, 70(38):12128-12134.