生物胺是一类有毒的低分子含氮化合物,通常由相应的氨基酸经微生物脱羧作用形成,广泛存在于泡菜、奶酪和酱油等发酵食品中[1]。常见的生物胺主要有色胺、β-苯乙胺、尸胺、腐胺、组胺、酪胺、亚精胺和精胺[2]。其中,组胺被认为是毒性最强的生物胺,对心血管系统、呼吸系统、消化系统、中枢神经系统、免疫系统和皮肤有不良影响[3]。有报道表明过量摄入组胺会引起包括低血压、头痛、水肿、荨麻疹、休克、血管外平滑肌收缩或松弛、腹部绞痛、腹泻和呕吐等严重的病理反应[4]。2003年,欧盟委员会规定食品中组胺的含量不得超过100 mg/kg[5]。我国传统发酵食品种类繁多,多为自然发酵且部分发酵食品的组胺含量远超其限量标准,易造成食品安全问题[6]。近年来,人们致力于减少发酵食品中的组胺含量,如使用新鲜原料、采取卫生预防措施、辐照、添加精油等,然而,这些措施并不能完全消除食品中的组胺[5]。
微生物降解法被视为控制发酵食品组胺最为有效、绿色的方法,现已运用于豆豉、香肠等多种食品中组胺的控制[7-8]。研究表明,微生物的组胺降解机制在于其自身存在降解组胺的基因[9]。据报道,微生物中存在的组胺降解基因可分为三类:胺氧化酶(amine oxidase,Aos)基因、多铜氧化酶(multicopper oxidase,MCO)基因和胺脱氢酶(amine dehydrogenase,AmDH)基因,由这些基因编码的酶可通过催化组胺进行一系列的脱氨和脱氢反应实现组胺的降解[10]。目前,研究人员已经发现了多种具有组胺降解能力的微生物及其降组胺基因的存在,且不同微生物降解组胺的基因存在差异,从而导致其组胺降解能力的差异[11]。因此,有必要对不同组胺降解菌株降解组胺的关键基因进行探究。
此外,由于食品发酵过程中涉及到相当复杂的微生物群落,且这些微生物在发酵过程中产生多种酶和必需营养素,从而赋予发酵食品独特的风味和营养特性,因此参与发酵的微生物对发酵食品良好品质的形成至关重要[6]。L.plantarum是发酵食品中的常见菌株,广泛存在于各种发酵食品中[12]。研究表明,L.plantarum能够赋予发酵肉制品和发酵果酒以独特的香味[13-14],且L.plantarum的降组胺活性获得广泛认可,现已被运用于多种发酵食品组胺的降解[15]。然而,从自然界筛选的L.plantarum通常具有较低的组胺降解能力[16]。因此,有必要对L.plantarum进行基因改造,通过增强其组胺降解基因的表达增强其降解组胺的能力[17]。mcO基因和gapdH基因被认为是L.plantarum中普遍存在的2种与组胺降解相关的基因[18-19]。褚琪等[17]通过全基因组测序确证了gapdH基因在L.plantarum SGJ-24中的存在;WANG等[20]在大肠杆菌中表达了L.plantarum的gapdH基因,并对GAPDH进行纯化和固定化,提高了GAPDH的组胺降解率;曹玉莹等[21]探究了L.plantarum源mcO基因的异源表达,探究了其酶学性质及其对组胺的降解作用。然而,尚未有研究人员同时研究mcO基因和gapdH基因在L.plantarum降解组胺过程中发挥的作用,导致L.plantarum降解组胺的关键基因尚不清楚,阻碍L.plantarum降解组胺分子机制的阐释,制约高组胺降解能力L.plantarum的分子育种。
因此,本研究从发酵芥菜、发酵酸笋等发酵蔬菜中筛选出一株具有良好组胺降解能力的L.plantarum并对其菌株特性进行分析;通过构建mcO基因和gapdH基因的过表达菌株,评价过表达菌株的降胺能力,明确L.plantarum降解组胺过程中起关键作用的基因,为组胺降解菌株的改良奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株、质粒与引物
L.plantarum CY087为本次实验筛选菌株;质粒pMG36e,淼灵生物科技有限公司;感受态EscherichiA.coli DH5α,擎科生物科技股份有限公司。
本次实验所用到的质粒及引物如表1所示:
表1 实验菌株、质粒和引物
Table 1 Experimental plasmids and primers

注:斜体为保护碱基;下划线代表限制性酶切位点。
名称菌株、质粒特性和引物序列来源质粒pMG36e表达载体,Emr抗性淼灵生物科技pMG36e-mcO携带mcO基因的重组质粒,Emr抗性本研究pMG36e-gapdH携带gapdH基因的重组质粒,Emr抗性本研究菌株Eco-pMG36e携带空质粒pMG36e的E.coli DH5α,Emr抗性本研究Eco-mcO携带重组质粒pMG36e-mcO的E.coli DH5α,Emr抗性本研究Eco-gapdH携带重组质粒pMG36e-gapdH的E.coli DH5α,Emr抗性本研究CY087- pMG36e携带空质粒pMG36e的L.plantarum CY087,Emr抗性本研究CY087-mcO携带重组质粒pMG36e-mcO的L.plantarum CY087,Emr抗性本研究CY087-gapdH携带组质粒pMG36e-gapdH的L.plantarum CY087,Emr抗性本研究引物菌种鉴定引物27F5′-AGTTTGATCMTGGCTCAG-3′[22]1492R5′-GGTTACCTTGTTACGACTT-3′目的基因扩增引物mcO-F5′-GCTCTAGAATGGCAAAAAAAGTCTATACTG-3′本研究mcO-R5′-CCCAAGCTTTTACATTCCTGGCATCCAGACG-3′本研究gapdH-F5′-GCTCTAGAATGTCTGTAAAAATTGGTATTAA-3′本研究gapdH-R5′-CCCAAGCTTTTAGAGAGTGGCGAACTTCAAT-3′本研究验证引物pMG36e-F5′-CCTCCTCATCCTCTTCATCC-3′本研究pMG36e-R5′-TCCAAATATCGTAGCGCC-3′本研究
1.1.2 试剂
市售发酵蔬菜,广东省广州市;蔗糖、甘油、组胺二盐酸盐,天津福晨化学试剂有限公司;EDTA、红霉素,上海源叶生物科技有限公司;1,7-二氨基庚烷,阿拉丁生化科技股份有限公司;限制性内切酶(Xba I、Hind Ⅲ)、2×EasyTaq PCR SuperMix、Goldview 核酸染料,北京全式金生物技术有限公司;T4连接酶,TaKaRa有限公司;细菌基因组提取试剂盒,天根生化科技有限公司;质粒提取试剂盒、胶回收试剂盒,北京金沙生物科技有限公司;革兰氏染色液,比克曼控股有限公司。
甘油管:30 mL丙三醇与70 mL蒸馏水混匀后以800 μL/管分装于2 mL的冻存管中121 ℃灭菌20 min并于-80 ℃进行保存。电击缓冲液:称取34.23 g蔗糖,加入20 mL甘油,用去离子水定容至200 mL,121 ℃灭菌20 min。培养基:MRS肉汤、MRS培养基,源叶生物科技有限公司;LB液体培养基(g/L):蛋白胨10.0,酵母浸粉5.0,氯化钠10.0,pH 7.2~7.4;LB固体培养基:LB液体培养基添加1.5%(质量分数)的琼脂即为LB固体培养基。
1.2 仪器与设备
BLT-470蓝光切胶仪,生工生物工程(上海)股份有限公司;电击杯(间距2 mm)、Power Pac电泳套装、PCR热循环仪(T100TM)、MicroPulserTM型电穿孔仪,美国伯乐公司;MJX-250-Z生化培养箱,上海博讯事业有限公司医疗设备厂;P9PC双光束紫外可见分光光度计,上海美谱达仪器有限公司;ZQZY-88BV振荡培养箱,上海知楚仪器有限公司;LC-20AT高效液相色谱仪,日本岛津公司;D3024R高速离心机,美国赛洛捷克公司。
1.3 实验方法
1.3.1 降解组胺菌株的筛选
按照LUO等[23]的方法对市售发酵蔬菜中的菌株及其组胺降解能力进行筛选和测定。筛选菌株的组胺降解率的计算如公式(1)所示:
组胺降解率/%=
(1)
1.3.2 降解组胺菌株的鉴定
形态学鉴定:将筛选出的组胺降解菌株稀释涂布于固体培养基,37 ℃恒温培养24 h。观察其菌落大小、颜色、表面形态、边缘形状、质地等,并记录观察结果。将菌株进行革兰氏染色后,使用光学显微镜油镜观察其形态。
分子生物学鉴定:以抽提的目标菌株基因组DNA为模板,使用引物对27F/1492R对其16S rDNA序列进行扩增。PCR反应程序为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火1 min,72 ℃延伸2 min,32个循环;最后72 ℃总延伸10 min,扩增产物经0.8%琼脂糖凝胶电泳验证后,送擎科生物科技公司测序,所得序列与NCBI数据库对比进行菌种鉴定。
1.3.3 菌株生长曲线的测定和耐受性分析
生长曲线测定:将OD600值为4.0的菌液按3%接种于5 mL MRS液体培养基中,于37 ℃培养并在第0、2、4、6、8、10、12、14、16、18、20、24 h取样测定菌液的OD600值并绘制其生长曲线。
耐受性分析:NaCl和组胺耐受性:将OD600值为4.0的菌液按3%接种于添加0%、2%、4%、6%、8%、10%(质量分数)NaCl以及0、50、100、200、300、400 mg/mL组胺的5 mL MRS液体培养基中,于37 ℃培养并在第12 h取样测定菌液的OD600值。pH值耐受性:将OD600值为4.0的菌液按3%接种于pH值为3、4、5、6、7的5 mL MRS液体培养基中,于37 ℃培养并在第12 h取样测定菌液的OD600值。
1.3.4 L.plantarum CY087基因组DNA测序与功能注释
收集对数生长期的菌液5 mL低速离心后弃去培养基,用无菌水清洗2次,5 000 r/min离心5 min弃上清液收集沉淀菌体并立即液氮速冻,标记后于-80 ℃保存。处理后的样品经干冰运输送至擎科生物科技有限公司进行后续的测序工作。测序数据经处理后分别与TrEMBL、Swiss-Prot、egg NOG、GO、KEGG和Pfam数据库进行比对以注释其功能。
1.3.5 过表达载体的构建
如图1所示,以提取的L.plantarum CY087基因组DNA为模板,引物对mcO-F/R和gapdH-F/R分别进行PCR扩增得到带有Xba I和Hind Ⅲ酶切位点及相应保护碱基的mcO和gapdH目的基因片段。扩增程序与1.3.2节相同。扩增产物经0.8%琼脂糖凝胶电泳验证后,将目的条带进行切胶回收,得到可用于酶切过程的目的基因片段。
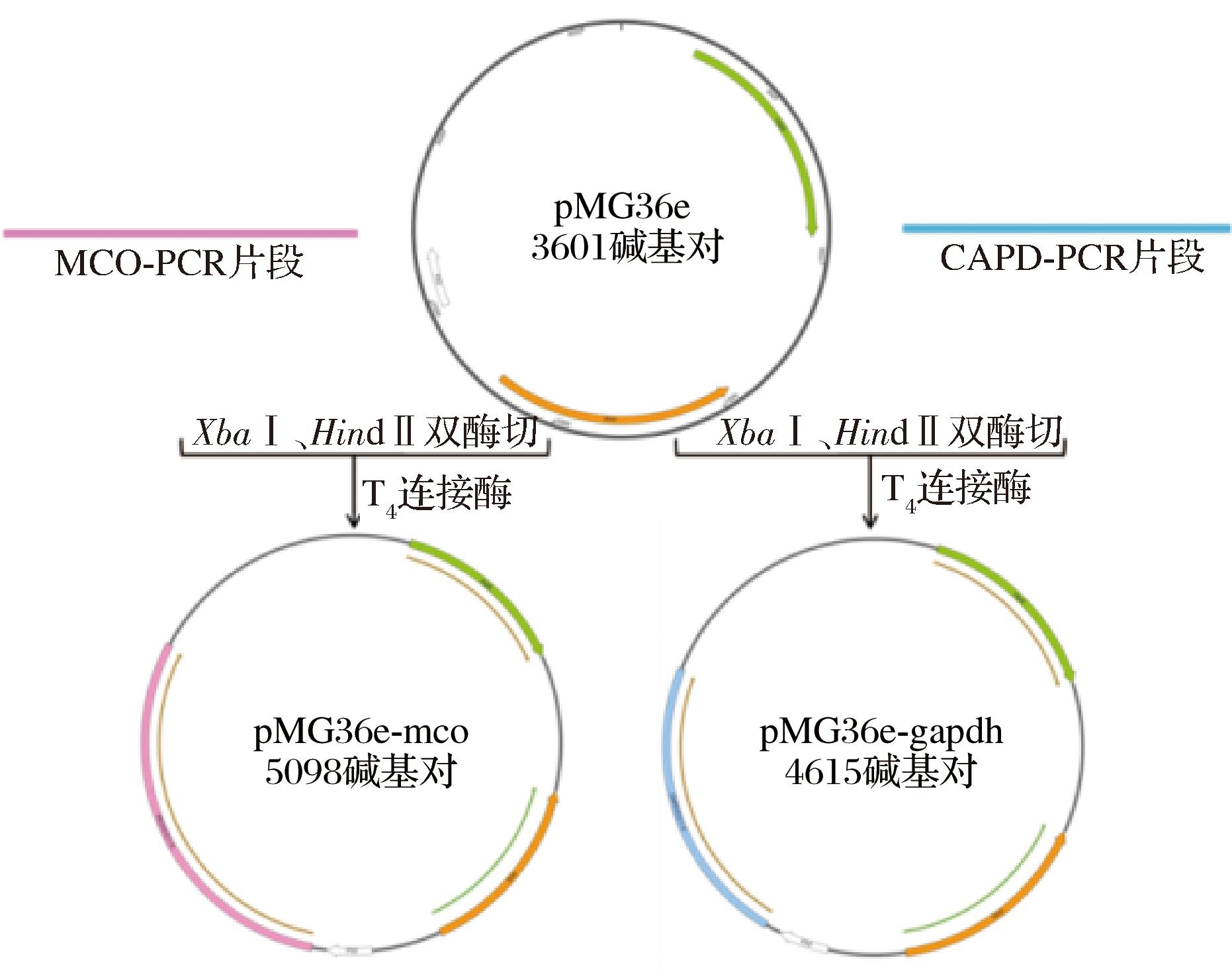
图1 mcO基因和gapdH基因过表达质粒的构建
Fig.1 Construction of mcO gene and gapdH gene overexpression plasmids
将质粒干粉按照说明书转化入E.coli DH5α并抽提得到空质粒,将空质粒和获得的目的基因片段使用内切酶Xba I、Hind Ⅲ按照说明书进行双酶切和酶连。酶连产物参照任大勇等[24]的方法转化进感受态E.coli DH5α,将复壮后的菌液涂布于含500 μg/mL红霉素的LB平板培养基37 ℃倒置培养24 h,将长出的单菌落进行划线扩培,挑取适量菌体,以pMG36e-F/R为引物对进行菌落PCR验证,PCR程序为:95 ℃预变性15 min;95 ℃变性30 s,52 ℃退火1 min,72 ℃延伸2 min,32个循环;最后72 ℃总延伸10 min,扩增产物于4 ℃进行保存。经0.8%琼脂糖凝胶电泳验证成功的转化子接种于含500 μg/mL红霉素的LB液体培养基中过夜培养后置于甘油管中-80 ℃进行保存。
1.3.6 过表达菌株的构建
参照李书鸿[25]的方法进行制备L.plantarum CY087感受态细胞的制备及电转化。电转化电压设置为2.4 kV,脉冲时间控制在4.5~5.0 ms。将复苏后的L.plantarum CY087菌液涂布于含有50 μg/mL红霉素的MRS平板培养基培养过夜,挑取单菌落划线纯化培养后用引物对pMG36e-F/R进行菌液PCR,PCR程序与1.3.4节过表达载体验证相同。经0.8%琼脂糖凝胶电泳验证成功的转化子接种于含50 μg/mL红霉素的MRS肉汤中过夜培养后置于甘油管中-80 ℃进行保存。
1.4 数据处理与分析
运用Graph pad对实验数据进行分析和图表制作;采用ANOVA法分析处理组间的差异显著性;primer 5软件用于引物设计以及碱基序列和氨基酸序列转换;MEGA-X软件用于进化树的构建;DNAMAN用于蛋白质序列的比对。
2 结果与分析
2.1 降解组胺菌株的筛选
从市售发酵芥菜、发酵酸笋等发酵蔬菜样品中筛选了共357株菌,表2为筛选出的46株具有组胺降解能力的菌株。其中,CY087号菌株降解组胺的效果良好且比较稳定。如图2-a所示,以不加菌的降胺模拟体系作为对照组,在摇床培养48 h后,实验组的组胺吸收峰面积明显减小,说明体系中的组胺已被菌株降解。间隔12 h定时取样,绘制了菌株CY087在60 h内的组胺降解曲线(图2-b),菌株CY087在前12 h对组胺的降解速率最快,在48 h时,CY087菌株对组胺的降解速率达到最高(20.16%),之后其降解率趋于平稳。根据王雪郦等[26]对干酪乳杆菌降解生物胺的速率变化的研究,这可能是由于随着培养时间的延长,菌株生长条件的变化使模拟体系内菌体发生自溶,释放出的多肽和游离氨基酸通过一系列水解和脱羧反应生成了组胺,因此使菌株的组胺降解速率逐渐降低。
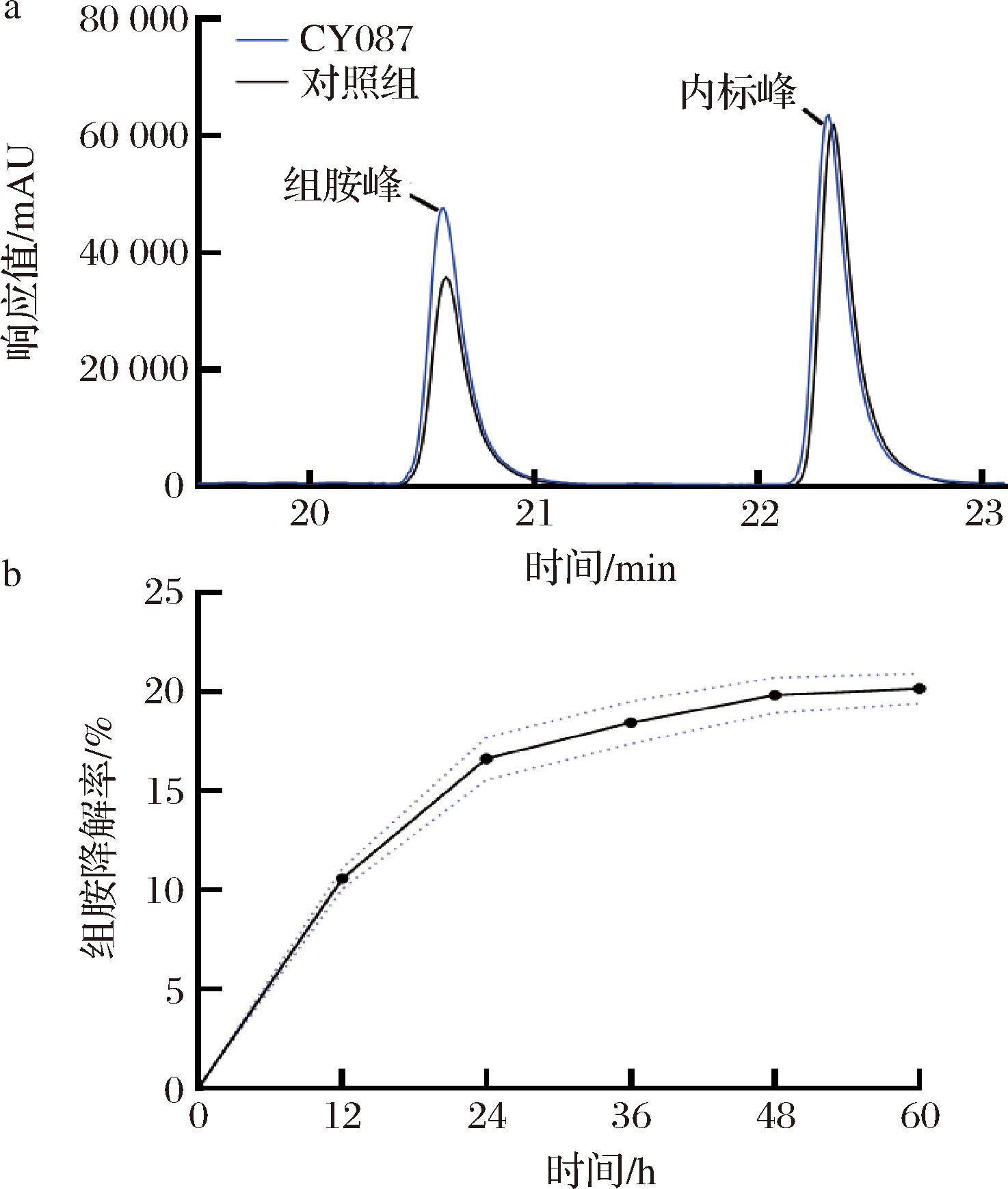
a-模拟体系中组胺含量的测定;b-菌株CY087的组胺降解曲线
图2 模拟体系中组胺含量的测定和菌株CY087的组胺降解曲线
Fig.2 Determination of histamine content in the simulated systemand histamine degradation curve of strain CY087
表2 分离菌株的组胺降解率
Table 2 Histamine degradation rate of isolated strains
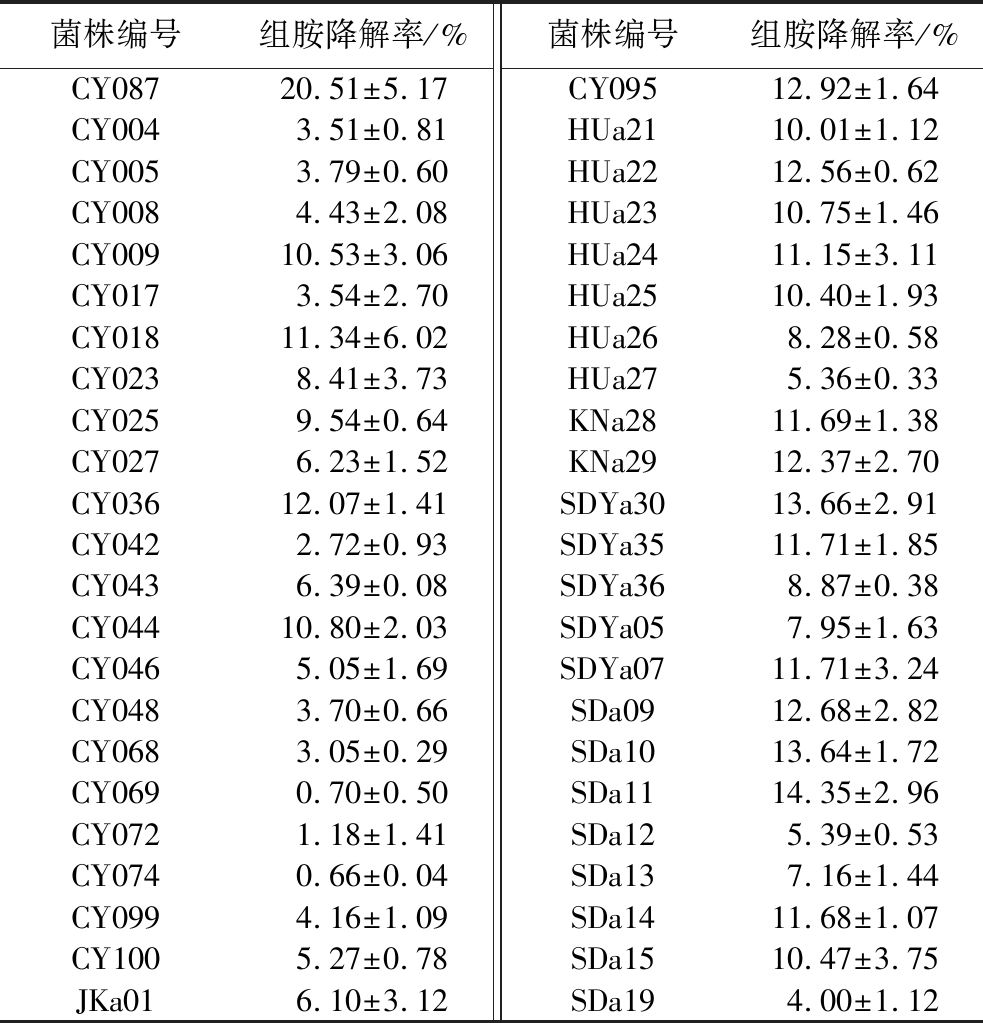
菌株编号组胺降解率/%菌株编号组胺降解率/%CY08720.51±5.17CY09512.92±1.64CY0043.51±0.81HUa2110.01±1.12CY0053.79±0.60HUa2212.56±0.62CY0084.43±2.08HUa2310.75±1.46CY00910.53±3.06HUa2411.15±3.11CY0173.54±2.70HUa2510.40±1.93CY01811.34±6.02HUa268.28±0.58CY0238.41±3.73HUa275.36±0.33CY0259.54±0.64KNa2811.69±1.38CY0276.23±1.52KNa2912.37±2.70CY03612.07±1.41SDYa3013.66±2.91CY0422.72±0.93SDYa3511.71±1.85CY0436.39±0.08SDYa368.87±0.38CY04410.80±2.03SDYa057.95±1.63CY0465.05±1.69SDYa0711.71±3.24CY0483.70±0.66SDa0912.68±2.82CY0683.05±0.29SDa1013.64±1.72CY0690.70±0.50SDa1114.35±2.96CY0721.18±1.41SDa125.39±0.53CY0740.66±0.04SDa137.16±1.44CY0994.16±1.09SDa1411.68±1.07CY1005.27±0.78SDa1510.47±3.75JKa016.10±3.12SDa194.00±1.12
2.2 菌株CY087的鉴定
2.2.1 形态学鉴定
如图3-a所示,菌株CY087在MRS平板培养基上37 ℃培养24 h后,其单菌落形态呈乳白色圆形,表面光滑,菌落规则,边缘整齐,中间凸起,菌落周围与边缘颜色一致,L.plantarum菌落形态特征明显[18]。将菌株CY087进行革兰氏染色,于100×光学显微镜下观察。如图3-b所示,菌株CY087呈革兰氏阳性(紫色),直或弯曲的杆状,无芽孢。

a-菌落形态;b-革兰氏染色光学显微镜形态;c-琼脂糖凝胶电泳图;d-系统进化树
图3 菌株CY087的鉴定
Fig.3 Identification of strain CY087
2.2.2 分子生物学鉴定
对菌株CY087的16S rDNA碱基序列进行检测,将测得的序列与NCBI中的核酸序列数据库进行比对。BLAST结果显示,菌株CY087与NCBI数据库中L.plantarum的CDS序列同源性高达100%。图3-d为L.plantarum CY087的系统进化树,反映了菌株CY087与L.plantarum的高同源性,因此确定菌株CY087属于L.plantarum,将其命名为L.plantarum CY087。
2.3 L.plantarum CY087的生长曲线和耐受性分析
由图可知,0~4 h为L.plantarum CY087的延滞期,由于处于生长初期,培养基内营养物质丰富,因此其生长速率随时间增加逐渐升高。培养4~6 h期间,L.plantarum CY087的生长速率达到最大值,进入生长对数期,之后其生长速率逐渐降低。12 h时,L.plantarum CY087的OD600值达到最大值,此时由于生长环境中营养物质的减少和代谢产物的积累,L.plantarum CY087进入生长的稳定期。
如图4-b所示,与未添加氯化钠的对照组相比,L.plantarum CY087在较高氯化钠浓度下仍具有不同程度的生长繁殖能力可能是由于高盐条件诱导了甜菜碱和四氢吡啶等能够帮助L.plantarum CY087抵御外界渗透压胁迫物质的产生[27]。但不同的氯化钠添加量均使得L.plantarum CY087菌液OD600值显著降低,说明2%~10%质量浓度的氯化钠能够不同程度地抑制L.plantarum CY087的生长繁殖。

a-生长曲线;b-NaCl耐受性测定;c-pH耐受性测定;d-组胺耐受性测定
图4 L.plantarum CY087的生长曲线;L.plantarum CY087耐受性实验
Fig.4 Growth curve of L.plantarum CY087;L.plantarum CY087 tolerance assay
注:ΔOD600值为培养12 h与0 h后测得的OD600值的差值;不同的小写字母表示具有显著性差异(下同)。

a-MCO氨基酸序列比对;b-GAPDH氨基酸序列比对
图5 MCO和GAPDH的氨基酸序列比对
Fig.5 Amino acid sequence comparison of MCO and GAPDH
注:MCO和GAPDH分别代表L.plantarum CY087中的多铜氧化酶和3-磷酸甘油醛脱氢酶;MCO 1~5及GAPDH 1~5分别为NCBI数据库中已有的L.plantarum源MCO和GAPDH氨基酸序列。
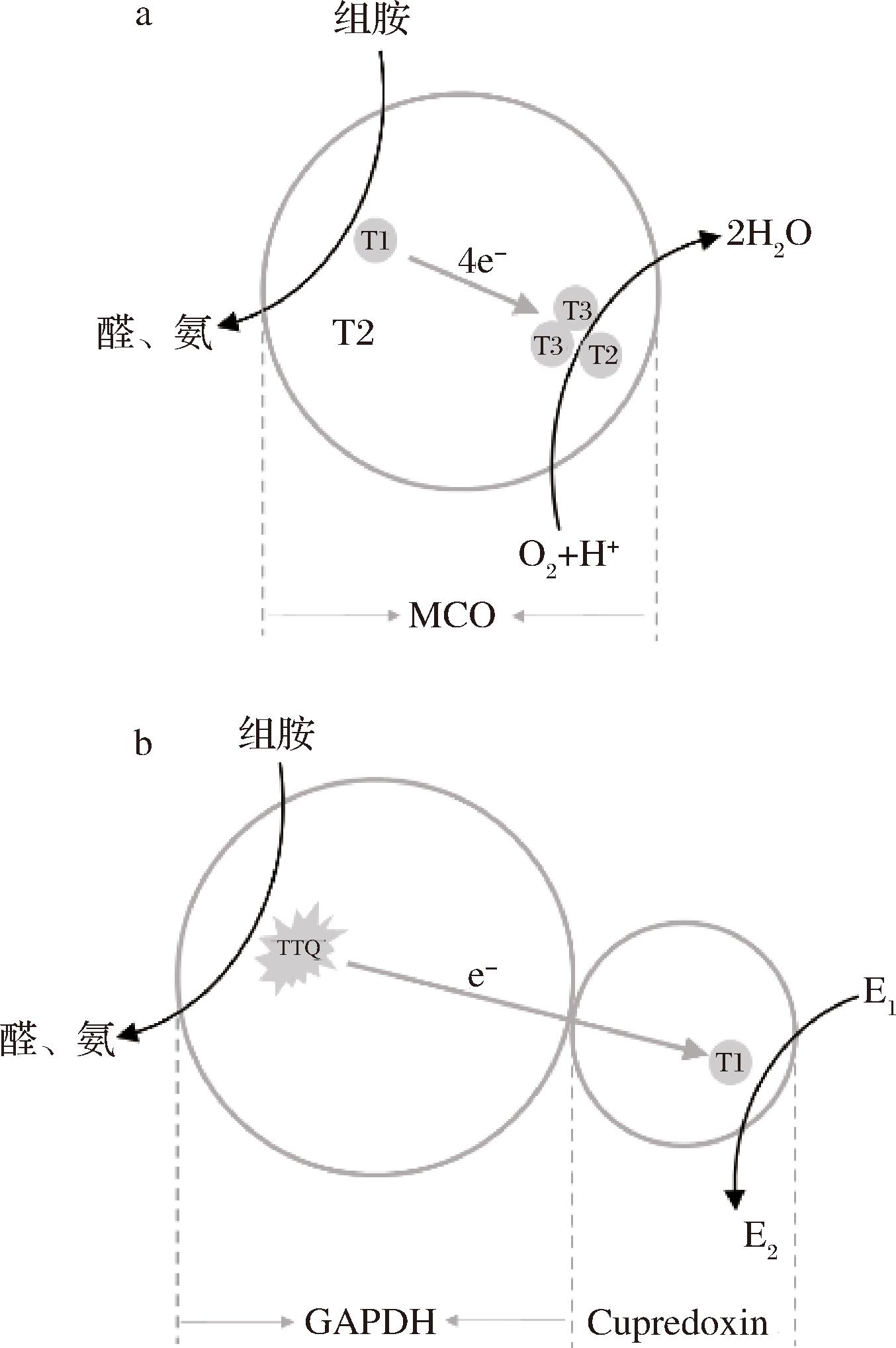
a-MCO组胺降解机制;b-GAPDH组胺降解机制
图6 MCO和GAPDH的组胺降解机制
Fig.6 Amino acid sequence comparison of MCO and GAPDH
注:TTQ-色氨酸-色氨酸醌;T1-Ⅰ型铜;T2、T3-三铜耦合中心;E1-电子受体;E2-还原型电子受体。
研究表明,偏酸或偏碱环境会影响细胞内外电荷平衡,导致细胞膜通透性改变,使营养物质难以进入细胞,从而严重影响菌株的生长代谢[28]。如图4-c所示,L.plantarum CY087在较高的pH(pH 6~7)下生长良好,当pH值降低时L.plantarum CY087菌液的OD600值显著降低,表明了L.plantarum CY087对低pH环境(pH 3~5)的耐受性较差。
与不添加组胺的对照组相比,添加50~400 mg/mL菌液的OD值并未发生显著性降低(图4-d),因此L.plantarum CY087对组胺具有的较高耐受性。这可能与L.plantarum CY087自身的溶质调控系统、糖酵解关键酶调控系统等耐渗透压机制有关[7,29]。
2.4 L.plantarum CY087基因组DNA测序与功能注释
将得到的基因组序列测定数据经处理后分别与TrEMBL、Swiss-Prot、egg NOG、GO、KEGG和Pfam数据库进行比对和功能注释。结果共有3 513个基因得到注释,占基因总数的99.10%。在TrEMBL数据库中得到功能注释的基因较多,为3 506个,占基因总数的98.90%。在Swiss-Prot、egg NOG、GO、KEGG和Pfam数据库中得到注释的基因分别是1 880、2 879、2 732、1 646和2 852个,分别占基因总数的53.03%、81.21%、77.07%、46.43%和80.45%。其中,mcO基因在Swiss-Prot得到注释,gapdH基因在GO、KEGG、nr、Swiss-Prot和TrEMBL数据库中均被注释。将mcO基因和gdpdH基因的碱基序列使用primer 5软件转换成氨基酸序列并与NCBI数据库中的MCO和GAPDH氨基酸序列进行比对(图5)。经软件DNAMAN计算,6种MCO与GAPDH氨基酸序列的一致性分别为97.04%和94.21%,具有多处高度重合的保守氨基酸序列。这说明不同L.plantarum来源的MCO和GAPDH具有高度相似性,即不同L.plantarum的MCO和GAPDH都保持相似的结构特征和功能特性,维持其特定的生物学功能[30]。这可能是L.plantarum普遍具有降解组胺能力的原因。图6为MCO与GAPDH降解组胺的反应机理,两种酶都能将组胺降解为氨和醛类物质[31]。
2.5 过表达载体与过表达菌株的构建
2.5.1 过表达载体的构建
按照1.3.5节所述方法,以L.plantarum CY087基因组DNA为模板,扩增得到mcO(1 506)和gapdH(1 023)两个目的基因的PCR片段。随后,利用内切酶Xba I和Hind Ⅲ对目的基因的PCR片段和空质粒pMG36e同时进行酶切,纯化酶切产物后将mcO和gapdH基因片段连接到载体pMG36e上,得到重组质粒pMG36e-mcO和pMG36e-gapdH。为保证重组质粒携带的基因正确,利用Xba I和Hind Ⅲ双酶切后验证各重组质粒携带的基因大小。结果如图7-a和图7-b所示,重组质粒经双酶切得到2个大小不一的片段,其中以L.plantarum CY087基因组为模板进行PCR得到的mcO(1 523)和gapdH(1 040)目的基因片段与酶切得到的mcO(1 518)和gapdH(1 035)目的基因片段的大小相一致。重组质粒均已构建成功,可用于下一步过表达菌株构建。
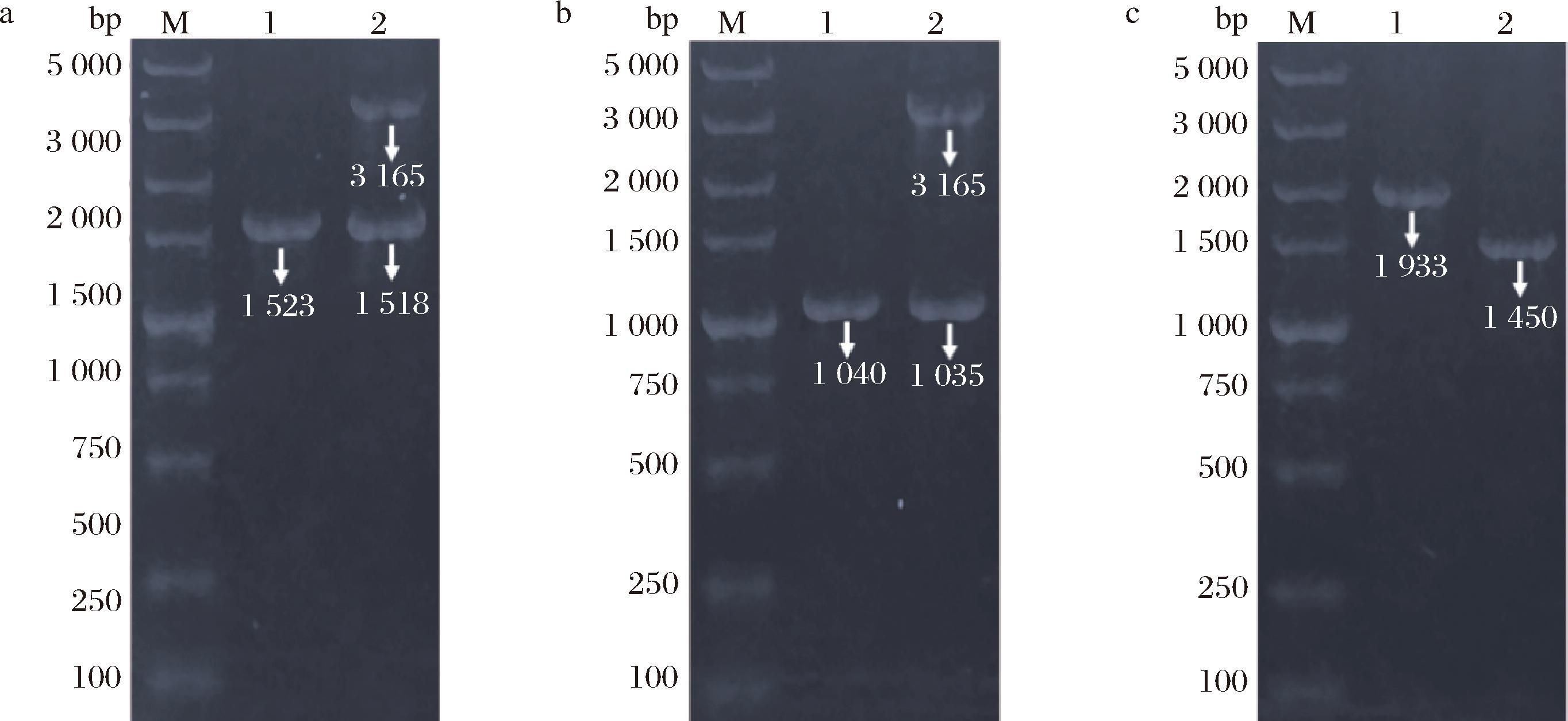
M-LD 5000 DNA Marker
a-pMG36e-mcO的构建及酶切验证(泳道1和泳道2分别为mcO基因的PCR片段和重组质粒pMG36e-mcO的双酶切验证);b-pMG36e-gapdH的构建及酶切验证(泳道1和泳道2分别为gapdH基因的PCR片段和重组质粒pMG36e- gapdH的双酶切验证);c-过表达菌株CY087-mcO和CY087-gapdH的构建和验证(泳道1和泳道2分别为重组质粒pMG36e-mcO和pMG36e- gapdH导入L.plantarum CY087的转化子验证)
图7 重组质粒的构建及酶切验证;过表达菌株的构建和验证
Fig.7 Construction and enzymatic validation of recombinant plasmids; construction and validation of overexpression strains
2.5.2 过表达菌株的构建
将上述构建成功的重组质粒分别转化至E.coli DH5α,随机挑取单菌落进行划线扩培后取适量菌体使用引物对pMG36e-F/R进行菌落PCR验证。成功转化重组质粒pMG36e-mcO和pMG36e-gapdH的E.coli DH5α将分别于1 933 bp和1 450 bp出现目标条带。将验证成功的转化子分别命名为Eco-mcO和Eco-gapdH并置于甘油管中于-80 ℃进行保存。将Eco-mcO和Eco-gapdH分别接入含500 μg/mL红霉素的LB液体培养基扩培并抽提质粒,获得的质粒通过电转化导入L.plantarum CY087中,使用引物对pMG36e-F/R进行验证,如图7-c所示,转化子出现目的条带时,将对应的转化子置于甘油管中于-80 ℃进行保存并以CY087-mcO和CY087-gapdH将其命名。
2.6 mcO基因和gapdH基因过表达菌株组胺降解能力的评价
将成功导入过表达质粒pMG36e-mcO与pMG36e-gapdH的过表达菌株CY087-mcO与CY087-gapdH接入组胺降解模拟体系评价其组胺降解率,以导入pMG36e空质粒的CY087-pMG36e作为对照。各菌株的组胺降解率如图8所示,菌株CY087-pMG36e、CY087-mcO和CY087-gapdH对组胺均具有一定的降解效果,其降解率分别为:20.51%、23.82%和27.06%。CY087-mcO和CY087-gapdH对组胺的降解效率显著高于CY087-36e,表明两个酶基因的强化表达对组胺降解率的提高均发挥了一定的作用。许多文献已经报道了MCO和GAPDH对组胺降解作用[19,30]。此外,CY087-gapdH的组胺降解能力显著高于CY087-mcO,表明与mcO基因相比,gapdH基因的强化表达能够更加有效地增加L.plantarum CY087的组胺降解率。这在相关研究中有类似的结论,JIANG等[32]和CALLEJ N等[33]实现了L.plantarum源gapdH基因和mcO基因在大肠杆菌中的异源表达,且将分离纯化后GAPDH和MCO运用于葡萄酒组胺的降解率,其降解率分别达到85.50%和33.90%。在CALLEJ
N等[33]实现了L.plantarum源gapdH基因和mcO基因在大肠杆菌中的异源表达,且将分离纯化后GAPDH和MCO运用于葡萄酒组胺的降解率,其降解率分别达到85.50%和33.90%。在CALLEJ N等[34]的研究中,L.plantarum的MCO对酪胺具有较高的降解率,但表现出对组胺和腐胺较低的降解率。因此,相比于mcO基因,gapdH基因在L.plantarum CY087降解组胺的过程中发挥更加关键的作用。
N等[34]的研究中,L.plantarum的MCO对酪胺具有较高的降解率,但表现出对组胺和腐胺较低的降解率。因此,相比于mcO基因,gapdH基因在L.plantarum CY087降解组胺的过程中发挥更加关键的作用。
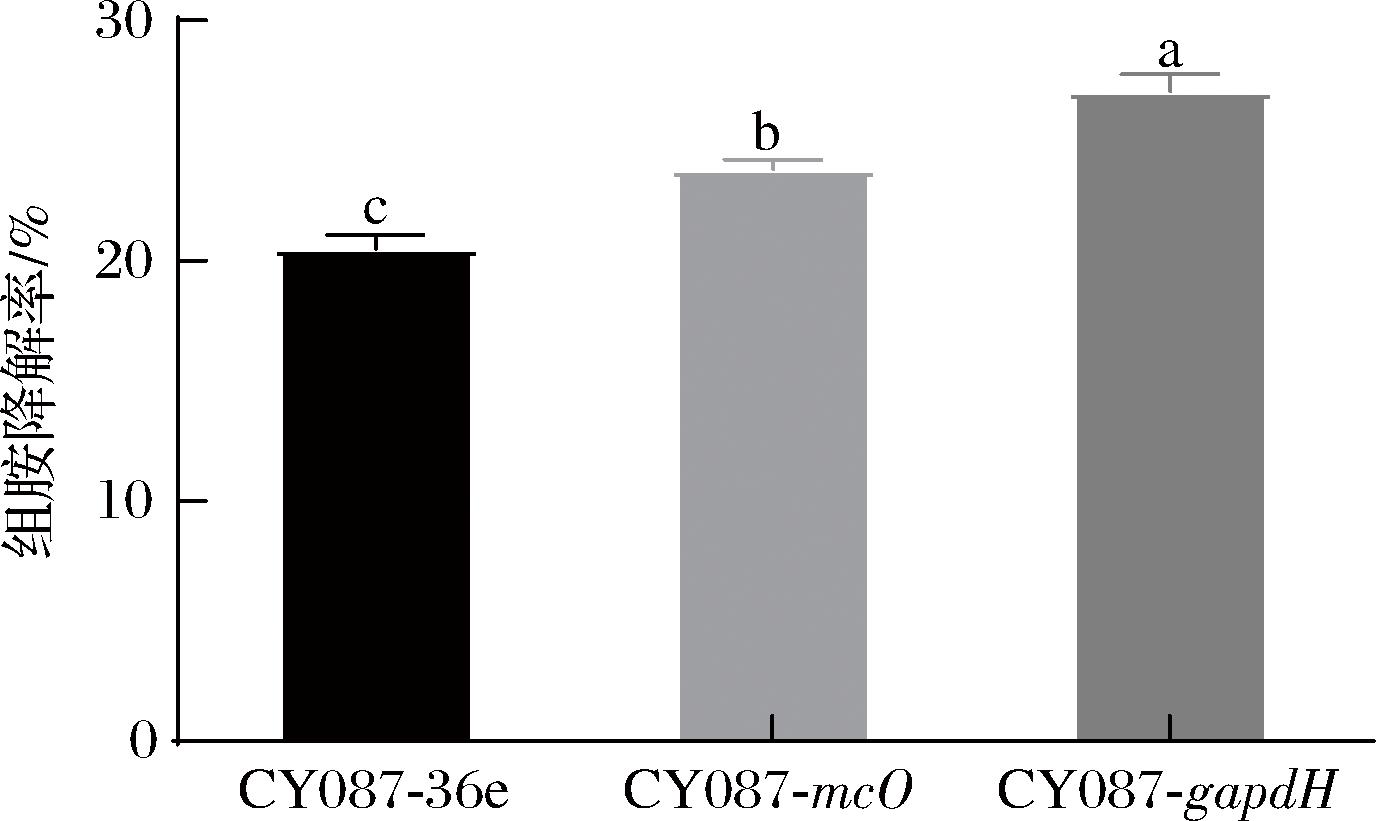
图8 菌株CY087-pMG36e、CY087-mcO和CY087-gapdH的组胺降解率
Fig.8 Histamine degradation rates of CY087-pMG36e, CY087-mcO and CY087-gapdH
3 结论与讨论
本研究从市售发酵蔬菜中筛选出一株具有良好组胺降解能力的L.plantarum CY087,其组胺降解率在48 h达到最大值20.16%。在低pH(pH 3~5)和高NaCl质量浓度(2%~10%)环境下,L.plantarum CY087的生长繁殖受到不同程度的抑制,而其对高质量浓度(400 mg/mL)组胺的良好耐受性。此外,将L.plantarum CY087的MCO和GAPDH氨基酸序列与NCBI数据库中其他L.plantarum MCO和GAPDH的氨基酸序列进行比对,其一致性分别为97.04%和94.21%。将构建成功的过表达菌株CY087-mcO和CY087-gapdH接入降胺模拟体系评价其组胺降解能力,结果显示相比于CY087-mcO,gapdH基因的强化表达菌株CY087-gapdH对组胺具有的更高的降解效率。因此,gapdH基因在L.plantarum CY087降解组胺过程中起更为关键的作用,可作为L.plantarum分子改造的潜在靶点。
[1] MONIENTE M, GARC A-GONZALO D, LLAMAS-ARRIBA M G, et al.Potential of histamine-degrading microorganisms and diamine oxidase (DAO) for the reduction of histamine accumulation along the cheese ripening process[J].Food Research International, 2022, 160:111735.
A-GONZALO D, LLAMAS-ARRIBA M G, et al.Potential of histamine-degrading microorganisms and diamine oxidase (DAO) for the reduction of histamine accumulation along the cheese ripening process[J].Food Research International, 2022, 160:111735.
[2] JIA T T, YU Z.Effect of temperature and fermentation time on fermentation characteristics and biogenic amine formation of oat silage[J].Fermentation, 2022, 8(8):352.
[3] CELIK E E, CANLI M, KOCADAGLI T, et al.Formation of Histamine, phenylethylamine and γ-Aminobutyric acid during sprouting and fermenting of selected wholegrains[J].Food Research International, 2023, 173(Pt2):113447.
[4] MAINTZ L, NOVAK N.Histamine and histamine intolerance[J].The American Journal of Clinical Nutrition, 2007, 85(5):1185-1196.
[5] PI T
T KOVA H,
KOVA H, ![]() L, et al.Detection and relative quantification of amine oxidase gene (yobN) in Bacillus subtilis:Application of real-time quantitative PCR[J].Journal of Food Science and Technology, 2022, 59(3):909-916.
L, et al.Detection and relative quantification of amine oxidase gene (yobN) in Bacillus subtilis:Application of real-time quantitative PCR[J].Journal of Food Science and Technology, 2022, 59(3):909-916.
[6] LI X Y, ZHANG Y, MA X X, et al.Effects of a novel starter culture on quality improvement and putrescine, cadaverine, and histamine inhibition of fermented shrimp paste[J].Foods, 2023, 12(15):2833.
[7] LI L, RUAN L Y, JI A Y, et al.Biogenic amines analysis and microbial contribution in traditional fermented food of Douchi[J].Scientific Reports, 2018, 8(1):12567.
[8] LI L, ZOU D, RUAN L Y, et al.Evaluation of the biogenic amines and microbial contribution in traditional Chinese sausages[J].Frontiers in Microbiology, 2019, 10:872.
[9] GARDINI F, ÖZOGUL Y, SUZZI G, et al.Technological factors affecting biogenic amine content in foods:A review[J].Frontiers in Microbiology, 2016, 7: 1218.[10] LI L, WEN X X, WEN Z Y, et al.Evaluation of the biogenic amines formation and degradation abilities of Lactobacillus curvatus from Chinese bacon[J].Frontiers in microbiology, 2018, 9:1015.
[11] 吴双慧, 牛茵, 何济坤, 等.基于宏基因组技术分析自然发酵羊肉香肠中微生物多样性及生物胺的代谢[J].食品科学, 2023, 44(18):133-140.WU S H, NIU Y, HE J K, et al.Metagenomic analysis of microbial diversity and biogenic amine metabolism in naturally fermented mutton sausage[J].Food Science, 2023, 44(18):133-140.
[12] 陈清爱, 徐道煌, 秦宇昕, 等.植物乳杆菌发酵砀山梨汁香气成分的变化过程分析[J].安徽农业大学学报, 2023, 50(6):1067-1074.CHEN Q A, XU D H, QIN Y X, et al.Analysis of the variation of aroma components in Dangshan pear juice fermented by Lactobacillus plantarum[J].Journal of Anhui Agricultural University, 2023, 50(6):1067-1074.
[13] 易鑫, 谈安群, 欧阳祝, 等.植物乳杆菌混菌发酵对梁平柚果酒理化性质及风味影响[J].食品与发酵工业, 2021, 47(11):180-187.YI X, TAN A Q, OUYANG Z, et al.Effects of Lactobacillus plantarum mixed fermentation on the physicochemical properties and flavor of Liangping pomelo wine[J].Food and Fermentation Industries, 2021, 47(11):180-187.
[14] 张新斌, 胡炜东, 李君冉, 等.植物乳杆菌对发酵肉制品风味影响研究进展[J].肉类研究, 2023, 37(9):70-75.ZHANG X B, HU W D, LI J R, et al.Research progress on the effect of Lactobacillus plantarum on the flavor of fermented meat products[J].Meat Research, 2023, 37(9):70-75.
[15] MAKHAMRUEANG N, SIRILUN S, SIRITHUNYALUG J, et al.Lactobacillus plantarum SK15 as a starter culture for prevention of biogenic amine accumulation in fermented beverage containing Hericium erinaceus mushroom[J].Applied Sciences-Basel, 2021, 11(15):6680.
[16] 史学容, 宋育阳, 秦义, 等.葡萄酒中生物胺的研究进展[J].食品科学, 2023, 44(13):297-308.SHI X R, SONG Y Y, QIN Y, et al.Advances in research on biogenic amines in wine[J].Food Science, 2023, 44(13):297-308.
[17] 褚琪, 张艳芳, 周浩然, 等.L.plantarum SGJ-24全基因组测序及活性相关基因的挖掘和分析[J/OL].食品工业科技, 2023.https://doi.org/10.13386/j.issn1002-0306.2023080245.CHU Q, ZAHNG Y F, ZHOU H R, et al.The whole genome sequencing of L.plantarum SGJ-24 and mining and in-depth analysis of activity related genes[J/OL].Science and Technology of Food Industry, 2023.https://doi.org/10.13386/j.issn1002-0306.2023080245.
[18] 邢鑫, 王鲁良, 张冰艳, 等.GAPDH@Fe3O4固定化酶脱除樱桃酒生物胺的研究及对酒体指标的影响[J].食品与发酵工业, 2023, 49(2):138-145.XING X, WANG L L, ZHANG B Y, et al.Effect of GAPDH@Fe3O4 immobilized enzyme on biogenic amines and basic indicators in cherry wine[J].Food and Fermentation Industries, 2023, 49(2):138-145.
[19] SUN S Y, JIANG D Q, FAN M T, et al.Selection of a versatile Lactobacillus plantarum for wine production and identification and preliminary characterisation of a novel histamine-degrading enzyme[J].International Journal of Food Science and Technology, 2020, 55(6):2608-2618.
[20] WANG L L, FAN M T, XING X, et al.Immobilization of glyceraldehyde-3-phosphate dehydrogenase on Fe3O4 magnetic nanoparticles and its application in histamine removal[J].Colloids and Surfaces B:Biointerfaces, 2021, 205:111917.
[21] 曹玉莹, 王爽, 刘怡宁, 等.植物乳杆菌漆酶的酶学性质及其对生物胺的降解作用[J].食品科学, 2023, 44(12):157-163.CAO Y Y, WANG S, LIU Y N, et al.Enzymatic characteristics of laccase from Lactobacillus plantarum and its effect on degradation of biogenic amines[J].Food Science, 2023, 44(12):157-163.
[22] 陈聪, 汤秀娟, 陈晓松, 等.产酯酶格氏乳球菌的筛选、鉴定与基因组注释[J/OL].食品科学, 2023.http://kns.cnki.net/kcms/detail/11.2206.TS.20231023.0853.002.html.CHEN C, TANG X J, CHEN X S, et al.Screening, identification, and genome annotation of esterase-producing Lactococcus garvieae[J].Food Science, 2023.http://kns.cnki.net/kcms/detail/11.2206.TS.20231023.0853.002.html.
[23] LUO W S, WU W L, DU X Y, et al.Regulation of the nitrite, biogenic amine and flavor quality of Cantonese pickle by selected lactic acid bacteria[J].Food Bioscience, 2023, 53:102554.
[24] 任大勇, 李昌, 秦艳青, 等.植物乳杆菌melA基因的克隆及其作为食品级筛选标记的初步研究[J].食品科学, 2011, 32(9):135-139.REN D Y, LI C, QIN Y Q, et al.Cloning of melA gene from L.plantarum as a food grade selection marker[J].Food Science, 2011, 32(9):135-139.
[25] 李书鸿. 植物乳杆菌YM-4-3菌株中叶酸生物合成途径关键基因folP和folE的功能研究[D].昆明:昆明理工大学, 2018.LI S H.The study of functions on the key genes folP and folE of folate biosynthesis of Lactobacillus plantarum YM-4-3 strain[D].Kunming:Kunming University of Science and Technology, 2018.
[26] 王雪郦, 雷超, 申开卫, 等.干酪乳杆菌(Lactobacillus casei)FV006对发酵食品中生物胺的降解性能[J].食品工业科技, 2023, 44(14):137-144.WANG X L, LEI C, SHEN K W, et al.Degradation performance of biogenic amines in fermented food by Lactobacillus casei FV006[J].Science and Technology of Food Industry, 2023, 44(14):137-144.
[27] IMHOFF J F, RODRIGUEZ-VALERA F.Betaine is the main compatible solute of halophilic eubacteria[J].Journal of Bacteriology, 1984, 160(1):478-479.
[28] MA J G, WANG W, SUN C B, et al.Effects of environmental stresses on the physiological characteristics, adhesion ability and pathogen adhesion inhibition of Lactobacillus plantarum KLDS 1.0328[J].Process Biochemistry, 2020, 92:426-436.
[29] 李东霞, 黎明, 王洪鑫, 等.生物法合成戊二胺研究进展[J].生物工程学报, 2014, 30(2):161-174.LI D X, LI M, WANG H X, et al.Progress in biosythesis of diaminopentane[J].Chinese Journal of Biotechnology, 2014, 30(2):161-174.
[30] AMARI M, ARANGO L F G, GABRIEL V, et al.Characterization of a novel dextransucrase from Weissella confusa isolated from sourdough[J].Applied Microbiology and Biotechnology, 2013, 97(12):5413-5422.
[31] 倪秀梅, 杨涛, 方芳.生物胺降解酶研究进展及其应用[J].微生物学通报, 2021, 48(11):4398-4411.NI X M, YANG T, FANG F.Biogenic amines-degrading enzymes and their applications:A review[J].Microbiology, 2021, 48(11):4398-4411.
[32] JIANG D Q, LI H M, SUN S Y.Verification of a novel glyceraldehyde-3-phosphate dehydrogenase capable of histamine degradation and its preliminary application in wine production[J].Food Science and Biotechnology, 2020, 29(12):1 719-1 726.
[33] CALLEJ N S, SENDRA R, FERRER S, et al.Identification of a novel enzymatic activity from lactic acid bacteria able to degrade biogenic amines in wine[J].Applied Microbiology and Biotechnology, 2014, 98(1):185-198.
N S, SENDRA R, FERRER S, et al.Identification of a novel enzymatic activity from lactic acid bacteria able to degrade biogenic amines in wine[J].Applied Microbiology and Biotechnology, 2014, 98(1):185-198.
[34] CALLEJ N S, SENDRA R, FERRER S, et al.Cloning and characterization of a new laccase from Lactobacillus plantarum J16 CECT 8944 catalyzing biogenic amines degradation[J].Applied Microbiology and Biotechnology, 2016, 100(7):3113-3124.
N S, SENDRA R, FERRER S, et al.Cloning and characterization of a new laccase from Lactobacillus plantarum J16 CECT 8944 catalyzing biogenic amines degradation[J].Applied Microbiology and Biotechnology, 2016, 100(7):3113-3124.