作为公认安全(Generally Recognized as Safe, GRAS)的甲醇营养型微生物,巴斯德毕赤酵母(Komagataella phaffii)已成为外源蛋白表达和高附加值代谢产物的重要生产平台[1]。甲醇氧化的第一步反应产物为甲醛,它在甲基营养菌中具有双重作用:既是关键的代谢中间体,也是潜在的细胞毒素[2]。甲基营养菌需要精确平衡甲醛代谢通量与毒性缓解机制,以此来保护细胞[3-4]。未经改造的K.phaffii菌株通过木酮糖单磷酸(xylulose monophosphate pathway, XuMP)途径同化甲醇,但仅约1/3的甲醇被同化为生物量,其余通过异化途径彻底氧化为CO2,甲醇同化效率较低[5-6]。
研究表明,甲醇代谢瓶颈主要在于醇氧化酶活力不足、木酮糖-5-磷酸(xylulose-5-phosphate, Xu5P)或核酮糖-5-磷酸(ribulose-5-phosphate, Ru5P)再生受限、以及辅因子失衡等[7-8]。由于甲醇同化高度依赖NADPH和碳骨架整合,磷酸戊糖途径(pentose phosphate pathway, PPP)非氧化阶段可能通过多重机制影响甲醇利用效率。该非氧化支路通过核糖-5-磷酸差向异构酶(ribose 5-phosphate epimerase, rpe)、转酮酶(transketolase, tkt)和果糖-1,6-二磷酸酶(fructose 1,6-bisphosphatase, fba)催化的反应,将五碳糖(如核糖-5-磷酸和Xu5P)转化为糖酵解中间产物,如果糖-6-磷酸和三磷酸甘油醛,从而连接甲醇代谢与中心碳代谢网络[9-10]。一方面,rpe调控的Xu5P/R-5-P平衡不仅影响PPP通量,其衍生物赤藓糖-4-磷酸还可作为芳香族氨基酸合成前体,并可能介导甲醛向中心代谢的输入;另一方面,tkt催化的五碳糖与七碳糖互变可补充三磷酸甘油醛,促进甲醛同化并缓解毒性中间体积累[11]。此外,核糖-5-磷酸作为核苷酸合成必需底物,其代谢与甲基转移反应密切相关,如吡咯喹啉醌(pyrroloquinoline quinone, PQQ)辅因子合成[12]。
综上,缓解甲醛毒性、维持XuMP途径高效运行的核心在于底物Xu5P的持续再生。因此,PPP的代谢通量至关重要。有研究表明,过表达PPP中的酶基因或找到对甲醛敏感的启动子有助于解决此问题[13]。表达甲醇芽孢杆菌(Bacillus methanolicus)的非氧化PPP途径,也使甲醇同化效率提升[14];此外,还可以构建关键酶的合成蛋白支架系统,通过空间定位优化提高代谢通量[15]。基于此,在本实验室前期工作阻断甲醇异化途径的基础上,本研究通过增强rpe、tkt和fba基因的表达水平,并结合重组菌株的生长动力学及甲醇消耗特性,系统考察其对K.phaffii甲醇代谢的影响,为进一步解析PPP非氧化阶段在甲醇高效同化中的潜在机制提供指导。加强PPP非氧化支路增加甲醇利用效率的代谢通路如图1所示。
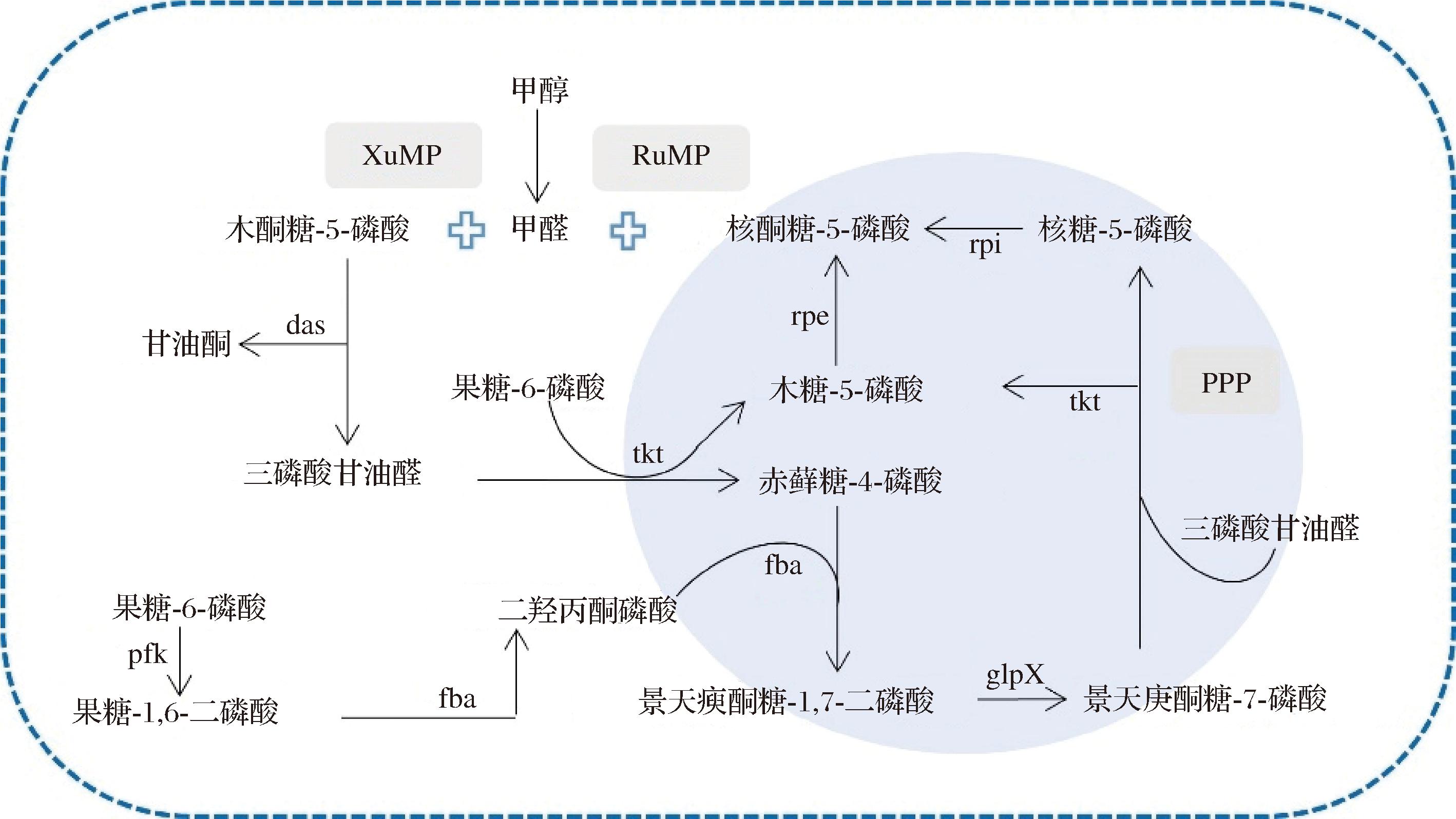
图1 本研究中过表达的靶基因
Fig.1 The target genes overexpressed in this study
注:代谢改造PPP(浅蓝色背景)中的目标基因(红色字体);das (dihydroxyacetone synthase,二羟丙酮合酶);rpi (ribose 5-phosphate isomerase, 核糖5-磷酸异构酶);pfk (fructose-6-phosphate kinase,果糖-6-磷酸激酶);RuMP(ribulose monophosphate pathway, 核酮糖单磷酸途径); glpX(sedoheptulose-1,7-bisphosphatase)。
1 材料与方法
1.1 菌株和质粒
本研究所构建的菌株和质粒见表1。
表1 本研究所用到的菌株与质粒
Table 1 The strains and plasmids used in this study
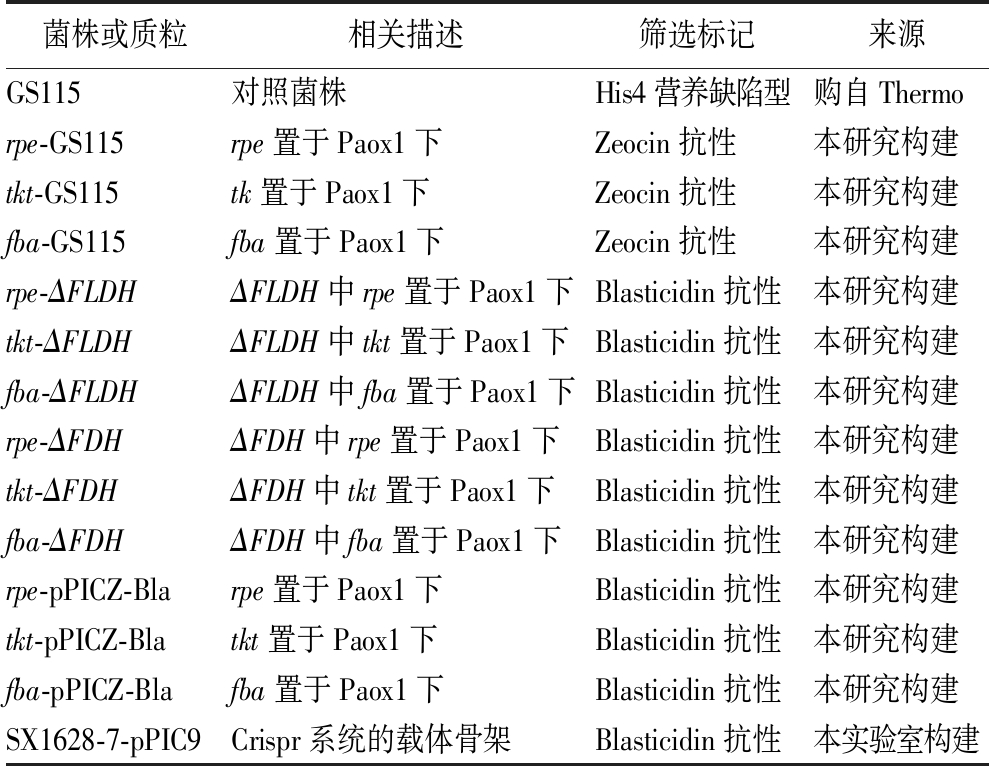
菌株或质粒相关描述筛选标记来源GS115对照菌株His4营养缺陷型购自Thermorpe-GS115rpe置于Paox1下Zeocin抗性本研究构建tkt-GS115tk置于Paox1下Zeocin抗性本研究构建fba-GS115fba置于Paox1下Zeocin抗性本研究构建rpe-ΔFLDHΔFLDH中rpe置于Paox1下Blasticidin抗性本研究构建tkt-ΔFLDHΔFLDH中tkt置于Paox1下Blasticidin抗性本研究构建fba-ΔFLDHΔFLDH中fba置于Paox1下Blasticidin抗性本研究构建rpe-ΔFDHΔFDH中rpe置于Paox1下Blasticidin抗性本研究构建tkt-ΔFDHΔFDH中tkt置于Paox1下Blasticidin抗性本研究构建fba-ΔFDHΔFDH中fba置于Paox1下Blasticidin抗性本研究构建rpe-pPICZ-Blarpe置于Paox1下Blasticidin抗性本研究构建tkt-pPICZ-Blatkt置于Paox1下Blasticidin抗性本研究构建fba-pPICZ-Blafba置于Paox1下Blasticidin抗性本研究构建SX1628-7-pPIC9Crispr系统的载体骨架Blasticidin抗性本实验室构建
1.2 培养基
培养重组大肠杆菌所用的培养基为LB或LLB(根据重组载体抗性选择)。活化酵母所用的培养基为YPD完全培养基,表型验证所用的培养基为YNB(yeast nitrogen base)补加1%(体积分数)甲醇。筛选重组子所用的固体培养基为上述各类培养基加上相应的抗性素。各培养基配方如下:
LB培养基:5 g/L酵母提取物,10 g/L蛋白胨,10 g/L NaCl,pH 7.0,固体培养基需补加20 g/L琼脂,121 ℃高压灭菌15 min。
LLB培养基:与LB培养基成分一样,不同的是NaCl浓度,减半至5 g/L,配制固体培养基时需加入20 g/L琼脂,121 ℃高压灭菌15 min。
YPD培养基:酵母提取物10 g/L,蛋白胨20 g/L,115 ℃高压灭菌30 min。需要指出的是,所用的葡萄糖需单独配制,葡萄糖储存液浓度200 g/L,115 ℃高压灭菌20 min,现用现加至工作液其终质量浓度为20 g/L。同样,固体培养基中额外添加15~20 g/L的琼脂。
YNB培养基:13.4 g/L YNB(含硫酸铵,不含氨基酸),碳源则按需添加。
1.3 重组载体与菌株的构建
1.3.1 重组载体的构建
以F1/R1为引物、pPICZB为模板,扩增产物作为pPICZ-Bla骨架;以以F2/R2为引物、本实验室自主构建的带有Blasticidin的质粒SX1628-7-pPIC9为模板,扩增Blasticidin片段;以上2种扩增产物无缝克隆,至此完成抗性替换,形成新的重组载体pPICZ-Bla。并以此为模板、F3/R3为引物增新的载体骨架;以K.phaffii基因组为模板,F4/R4、F5/R5、F6/R6为引物,分别扩增rpe、tkt、fba基因片段。然后将这3个产物分别与pPICZ-Bla骨架无逢克隆,重组产物即为各自的重组载体。引物序列及各个PCR产物片段大小详见表2。
表2 本研究所用到的引物
Table 2 The primers used in this study

引物序列 (5′ to 3′)PCR产物及其片段长度F1GGTTATGTGTGGGAGGGCTAAGTCCCCCTTTTCCTTTGTCGpPIC骨架, 2 953 bpR1CTTGAGACAAAGGCTTGGCCATGGTTTAGTTCCTCACCTTGTCGF2ATGGCCAAGCCTTTGTCTCAAGBlasticidin, 399 bpR2TTAGCCCTCCCACACATAACCF3TCTAGAACAAAAACTCATCTCAGAAGpPIC-Bla骨架, 3 286 bpR3GGTACCGATCCGAGACGGF4CGGTACCCATGGTTAAAACAATTrpe片段, 732 bpR4GTTCTAGACTATAAATTGGGTTTCGF5CCGTCTCGGATCGGTACCATGTCTGATCTCTTAGCTATCAACACtkt片段, 2 040 bpR5AGATGAGTTTTTGTTCTAGACTACGCATGAACAGACTCAAAAGF6CCGTCTCGGATCGGTACCATGCCAGATCAATTATCATTTTTAAAGAGfba片段,1 124 bpR6AGATGAGTTTTTGTTCTAGATTACAACTGGTTTGCAGTGTGG
通过热激转化的方式将各重组载体其转入大肠杆菌感受态细胞JM109,转化产物恢复培养(37 ℃,100 r/min),低速离心后取100 μL涂布LB+Blasticidin(100 μg/mL)固体平板。次日挑取单克隆,进行菌落PCR验证,结果呈现单一且大小正确的特异性条带的单克隆送测序,以此鉴定重组转化子。
1.3.2 过表达菌株的构建与筛选
重组质粒经Sac Ⅰ线性化后,电转导入K.phaffii GS115、ΔFLDH和ΔFDH感受态细胞(2种敲除菌由本实验室前期构建),复苏培养后,涂布带有Blasticidin抗性的YPDS平板。30 ℃恒温静置培养2~3 d后,挑取单克隆,转入YPD+对应抗性的液体培养基中培养。第2天提取基因组,进行基因组PCR验证并测序,鉴定正确转化子。
1.4 生长曲线的测定
在YNB+1%(体积分数)甲醇培养基中培养各过表达重组菌株,每隔12 h取样,(如果需要)稀释至置信区间内,用分光光度计测OD600值,每个样品至少3个平行。
1.5 实时荧光定量PCR
Takara试剂盒提取毕赤酵母总RNA,Nanodrop超微量分光光度计测定RNA样本的浓度及纯度。经检测质量合格后,去除基因组gDNA后反转成cDNA,作为qPCR模板,反转录试剂为PrimeScript RT Master Mix (Takara)。以肌动蛋白Actin为内参基因,测定相关基因的相对转录水平,所用仪器为ABI Step one plus。
1.6 甲醇残量测定
甲醇含量测定所用方法为HPLC,仪器为高效液相色谱仪(岛津LC-20),色谱柱为Aminex HPX-87H column(300 mm×7.8 mm, Bio-Rad, Hercules, CA)。甲醇标准品体积分数设为0%、0.12%、0.25%、0.50%、1.0%、2.0%、5.0%。分离分析条件:柱温40 ℃,流速0.5 mL/min,流动相为5 mmol/L稀H2SO4,等度洗脱模式,每个样品的洗脱时间25 min。
2 结果与分析
2.1 过表达菌株的表型验证
采用YNB培养基(1%甲醇作为唯一碳源),考察过表达tkt、rpe、fba基因对3种不同遗传背景的K.phaffii菌株(GS115、ΔFLDH和ΔFDH)生长表型的影响,实验结果如图2。
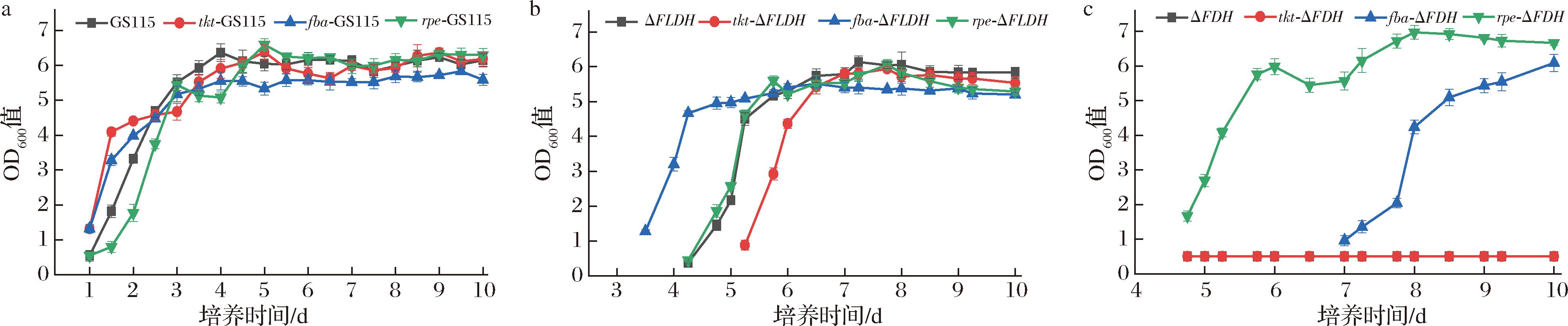
a-GS115为出发菌;b-ΔFLDH为出发菌;c-ΔFDH为出发菌
图2 过表达rpe、tkt和fba重组菌在甲醇基本培养基中的生长曲线
Fig.2 Growth curve of rpe, tkt, and fba overexpressed strains in methanol basic medium
在GS115野生型背景中,tkt、rpe、fba单基因过表达菌株的表型与出发菌株相比未呈现显著差异。在接种初期(前3 d),各菌株的起始生长时间先后顺序为:tkt-GS115>fba-GS115>GS115>rpe-GS115(图2-a)。在此之后,除了fba-GS115生物量一直处在最低,其他3株菌的变化则不稳定,这可能是由于生长后期,培养基中营养物质消耗怠尽,菌体生理状态不佳导致的。具体而言,虽然fba-GS115起始生长排序第二,但最终其最高生物量低于对照菌株(降低约8.4%),tkt-GS115最先开始生长,其最终生物量较亲本菌株约有1.7%的提升,而rpe-GS115生长最晚,最终生物量与对照菌株基本持平。
值得注意的是,在ΔFLDH背景菌株中,rpe-ΔFLDH与出发菌的生长状态最接近,未呈现显著差异。所有重组菌株均表现出明显的生长延迟现象:fba-ΔFLDH、rpe-ΔFLDH和tkt-ΔFLDH的生长延迟分别约为3.5 d、5 d、4 d(图2-b)。然而,这些重组菌株的最终生物量并未超过出发菌株的最大生物量,fba-ΔFLDH、tkt-ΔFLDH分别下降了约4%、8%,只有rpe-ΔFLDH与出发菌基本持平。但fba-ΔFLDH最先进入生长,并很快达到对数期,这已说明该重组菌的甲醇利用能力比其他菌强。
最显著的表型变化出现在ΔFDH背景菌株中。过表达rpe和fba的重组菌株获得了在以甲醇为唯一碳源的基本培养基中的生长能力,而出发菌株则完全不能生长(图2-c)。这说明在PPP非氧化支路与甲醇代谢是存在内部关联的。
2.2 最大比生长速率(μ)分析
最大比生长速率作为微生物发酵动力学的重要参数,表征单位时间内单位质量菌体的增量,是评估微生物生长特性的关键指标。本研究中比生长速率的计算方法为:用2个不同取样时间点样本生物量的增量除以这2个时间点的时间间隔(以小时计)。如图3所示,各重组菌的比生长速率因其各自的出发菌不同而表现出差异。
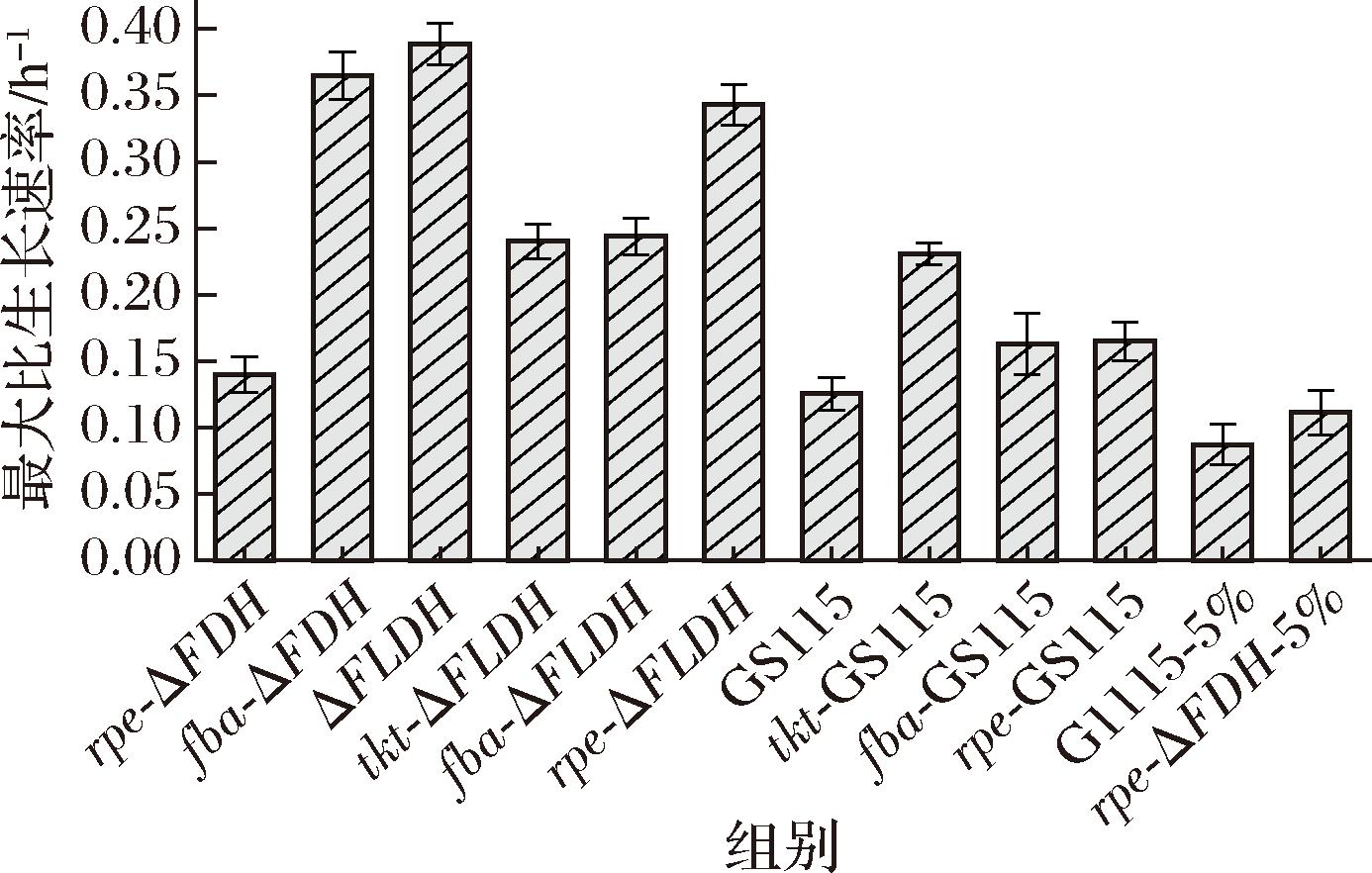
图3 过表达rpe、tkt和fba重组菌株的最大比生长速率
Fig.3 Maximal specific growth rate of recombinant strains overexpressing rpe, tkt, and fba
在以GS115为出发菌的重组菌中,tkt-GS115的最大比生长速率达到0.23 h-1,为本组最高,rpe-GS115(μ=0.163 h-1)次之,fba-GS115(μ=0.164 h-1)最低,但这两者基本无差异。在以ΔFLDH为亲本的重组菌株中,虽然单从最大比生长速率来看,ΔFLDH高达0.39 h-1,但不能忽略的是,fba-ΔFLDH领先生长,最大比生长速率为0.24 h-1。虽然tkt-ΔFLDH起始生长的时间点最晚,但其最大比生长速率与fba-ΔFLDH相当(μ=0.24 h-1)。结合生长曲线,fba-ΔFLDH最先进入生长周期,这充分证明其甲醇代谢的能力要优于其他菌。rpe-ΔFLDH重组菌株的生长最接近出发菌,最大比生长速率为0.34 h-1。关于这种现象的具体原因目前还不确定,但有研究表时fba、tkt和rpe基因产物均含有一个过氧化物酶体靶向信号ePTS1,这暗示这些酶可能被定向转运至过氧化物酶体参与甲醇同化代谢。
值得注意的是,虽然rpe-ΔFDH和fba-ΔFDH的最大比生长速率不是最高(分别为0.14 h-1和0.36 h-1),但考虑到其出发菌株ΔFDH完全不能在甲醇基本培养基中生长,这一结果已经实现了从“不可生长”到“可生长”的突破性转变。如应用到实际生产中,可充分把握这些重组菌生长初期的巨大优势。
目前,国内外关于甲基营养微生物构建的研究进展飞速,其中,HBENNETT等[16]在E.coli通过敲除PPP途径的核糖磷酸异构酶,得到ΔrpiAB突变株能够以甲醇为唯一碳源生长,其最大比生长速率为0.15 h-1。HWU等[17]在E.coli中构建了TAL/FBA突变体,实现了甲酸同化。
2.3 甲醇含量测定方法的可靠性分析
本研究中甲醇含量测定方法为HPLC,按照使用说明书中的相关教程制作标准曲线(图4),相关性系数达到0.999。对应标准曲线对已测样本进行积分处理,得到实际测定的甲醇含量。
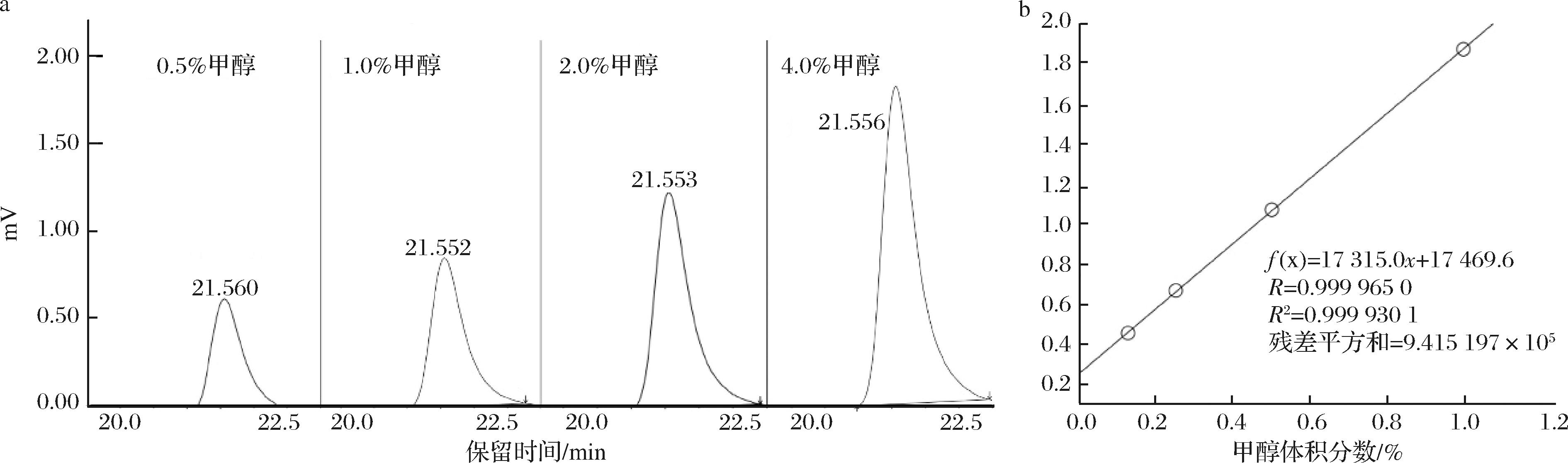
a-各浓度甲醇标准品色谱峰;b-标准曲线
图4 梯度浓度甲醇标准品的色谱峰图及四点校正的标准曲线
Fig.4 Chromatographic peaks of methanol standards across different concentration gradients and standard curve
设置未接种任何受试菌株的空白对照组,考察在同样的条件下不同浓度甲醇的自然挥发量,进而评估挥发量是否对菌株的甲醇利用效率产生实质性的影响。基于此标准曲线,对这些测量值进行积分,结果见图5。
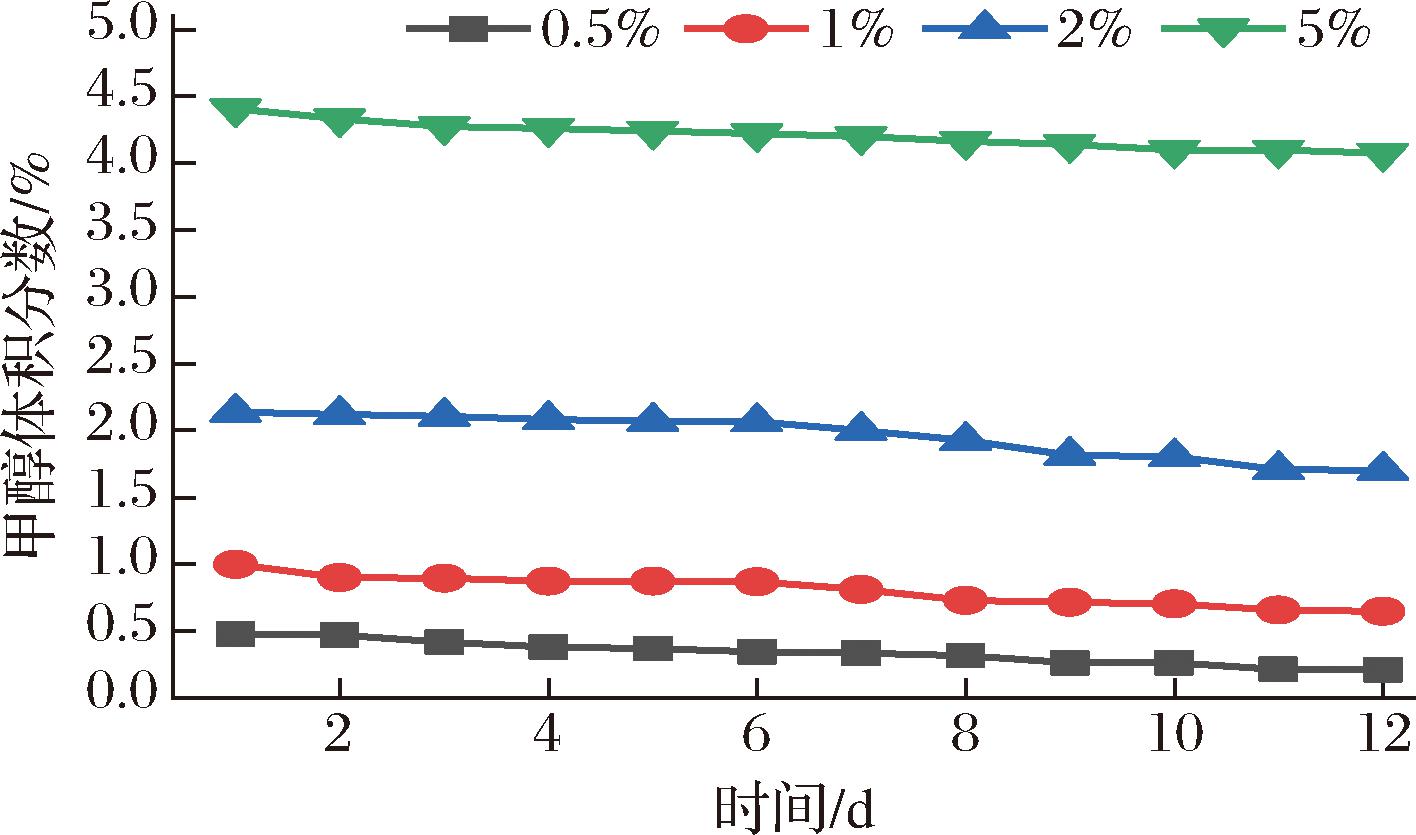
图5 对照培养基中梯度甲醇的自然挥发量
Fig.5 Natural evaporation of methanol with gradient concentrations in control medium
由于正常情况下,6 d即可完成一轮正常的发酵。在此,计算各空白样本的甲醇挥发度,各甲醇体积分数0.5%、1%、2%、5%下的甲醇自然挥发量与初始真实测量值的比值分别为0.28、0.13、0.042、0.034。可见,随着甲醇浓度的降低,其挥发程度逐渐增大。但总体上讲,甲醇挥发对实验结果造成的误差可忽略不计。
2.4 重组菌株的甲醇消耗量分析
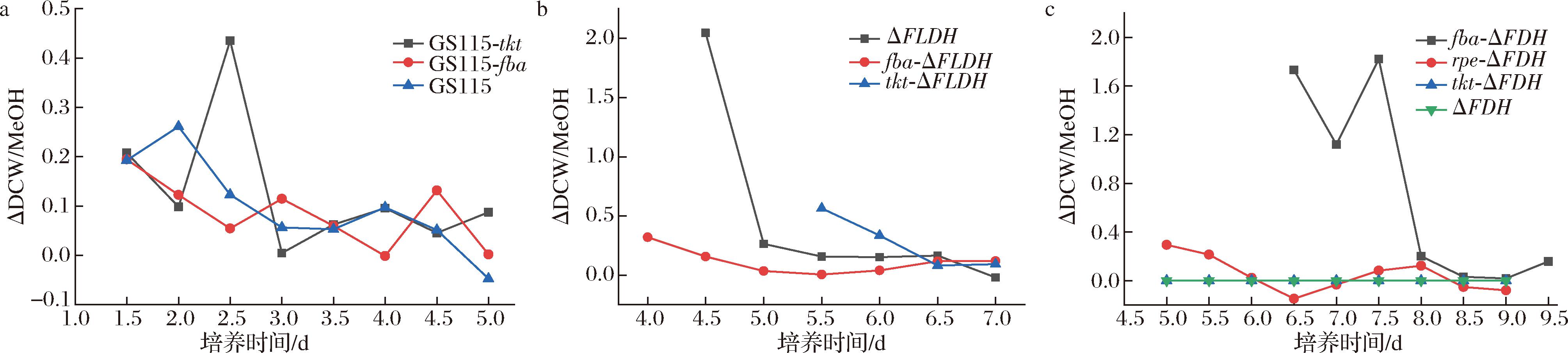
为探究不同重组菌株的甲醇利用能力,在菌株生长进入稳定期后补加甲醇。补料时间点根据各菌株的生长曲线动态来确定。GS115两次补加分别在3、5.5 d,tkt-GS115在2.5、5.5 d,fba-GS115只有1次补加点在2.5 d,rpe-GS115补加点在4 d时;ΔFLDH、rpe-ΔFLDH在6.5 d,fba-ΔFLDH在5.5 d,tkt-ΔFLDH全程并未补加;rpe-ΔFDH在7.5 d补加。
2.4.1 以GS115为出发菌的重组菌株甲醇利用效率分析
本研究选取首次补料时间点(2.5 d)前的实验数据作为甲醇利用效率的评价依据(图6)。具体而言,通过计算该时间段内甲醇消耗量与OD600值增长量的比值(甲醇消耗/ΔOD600值),来表征单位生物量增长所需的甲醇消耗量。该比值越小,表明菌株的甲醇利用效率越高。
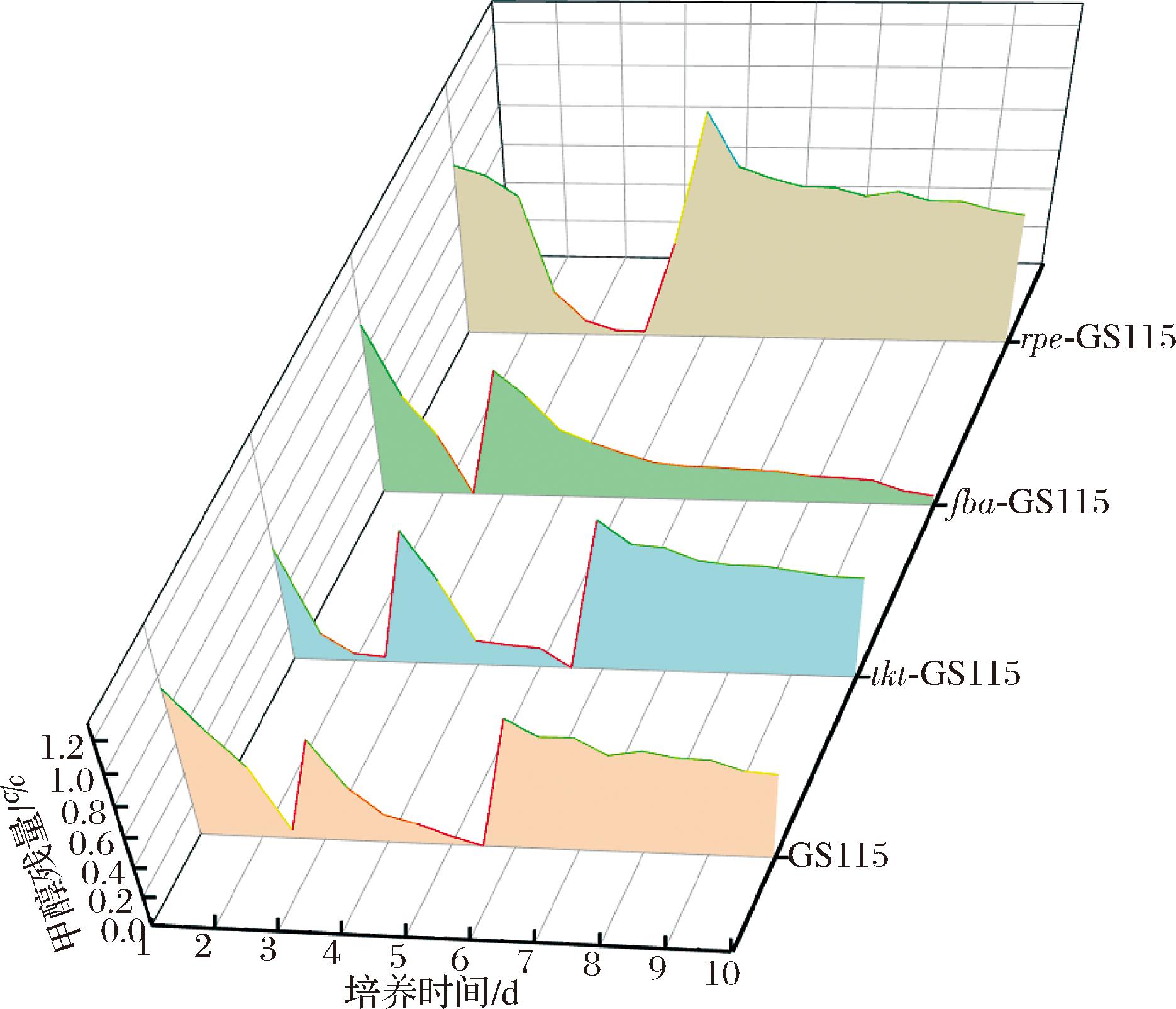
图6 以GS115为出发菌的重组菌的甲醇消耗情况
Fig.6 Methanol consumption profile in recombinant strains using GS115 as host
经精确计算,GS115、tkt-GS115、fba-GS115、rpe-GS115的甲醇利用率分别为0.21、0.20、0.30、0.21。值得注意的是,在这3株重组菌株中,仅fba-GS115的甲醇利用率相对于出发菌株并没有改善。此外,还计算了单位甲醇消耗所产生的生物量的增量(生物量/甲醇),各菌株对应值分别为4.67、5.07、3.36、4.83。该数值越大,说明菌株对甲醇的利用效率越高。
值得注意的是,当对GS115和tkt-GS115二次补加300 μL甲醇后,各菌株OD600值保持平稳或略有下降。这可能是由于此时菌体已处于衰退期,基本停止消耗MeOH。即便重新补加碳源,也未能恢复菌体活力。按照同样的标准,计算得出二次甲醇利用率分别为1.27、0.34(另一种表示方法为0.79、2.96),显然此时的甲醇利用率已明显下降,但tkt-GS115依然保有一定的优势。
2.4.2 以ΔFLDH为出发菌的重组菌株甲醇消耗量分析
针对ΔFLDH系列菌株的详细监测(图7)发现:对照组在培养6 d后甲醇接近耗尽,补加甲醇后OD600值继续增加,在7.25 d达到峰值;fba-ΔFLDH在5 d进入平台期,5.5 d甲醇耗尽后补料仅引起轻微的生长恢复;在7.5 d OD600值达到最高,之后MeOH和OD600值均呈下降趋势。rpe-ΔFLDH则在5.5 d甲醇即将耗尽时仍保持生长,补料后OD600值提升,7.5 d达到最大值。
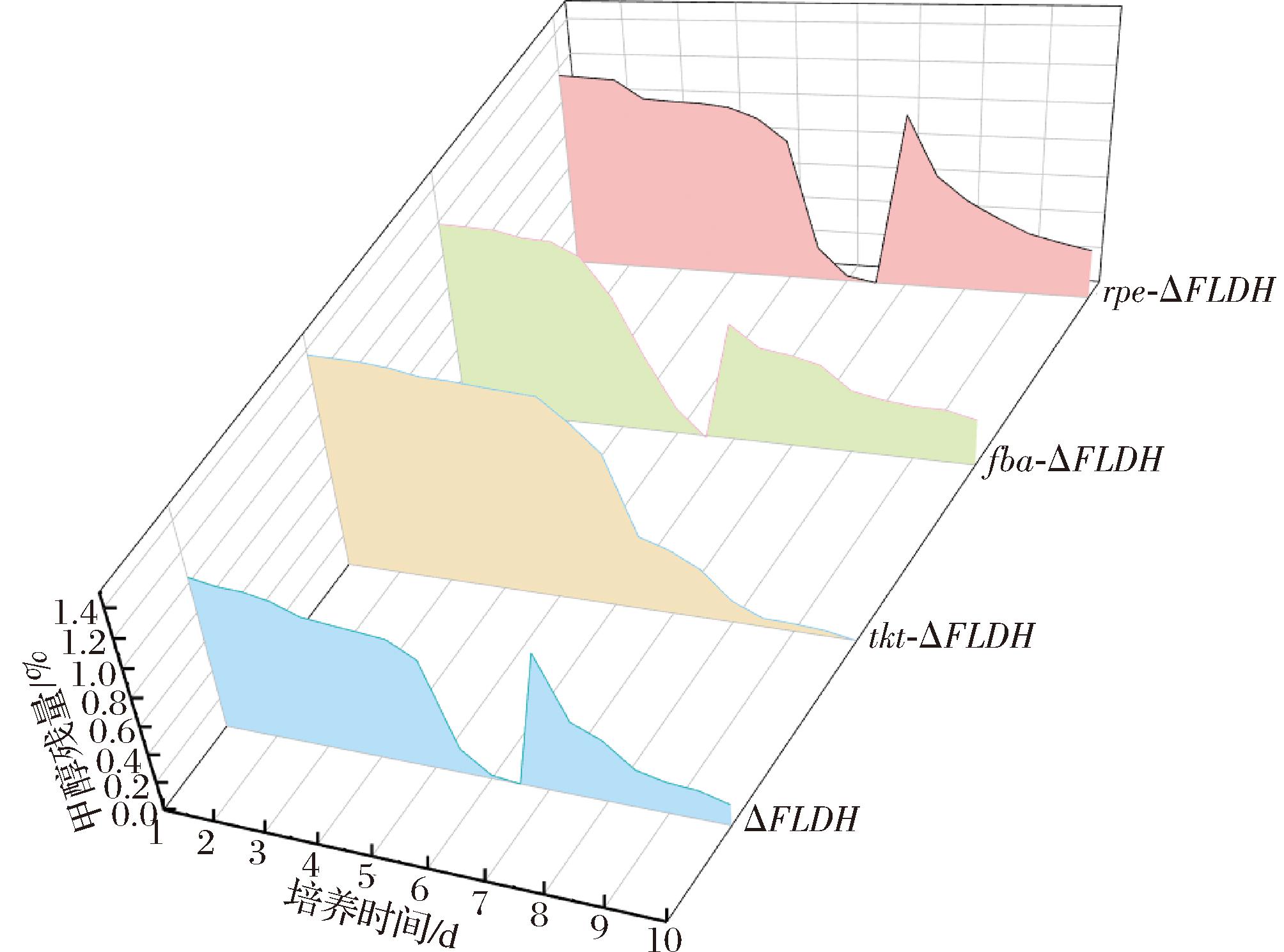
图7 以ΔFLDH为出发菌的重组菌的甲醇消耗情况
Fig.7 Methanol consumption profile in recombinant strains using ΔFLDH as host
按照同样的标准,以甲醇补加时间点前后菌体的OD600值变化与甲醇含量变化,计算得出ΔFLDH、tkt-ΔFLDH、fba-ΔFLDH和rpe-ΔFLDH的甲醇利用率分别为0.42、0.41、0.37、2.74(另一种表示方式下的数值为2.36、2.43、2.70、0.36)。由此可见,最先进入生长状态的fba-ΔFLDH的甲醇利用效率最高,这与生长曲线也是相符合的。虽然tkt-ΔFLDH起始生长的时间最晚,但只有它的甲醇利用率也略有提高。
2.4.3 以ΔFDH为出发菌的重组菌株甲醇消耗量分析
如图8所示,rpe-ΔFDH菌株在第4天开始生长,第6天达到第一个峰值,补加甲醇后OD600值经二次生长并在8 d创下新高。之后略有下降并持平,甲醇也平稳下降。fba-ΔFDH虽然启动生长较晚(第6 d),9.75 d补加甲醇,到第10天仍在持续生长,显示出优异的生长潜力,此时培养基内的MeOH已快耗尽。对照实验表明,ΔFDH和tkt-ΔFDH在实验条件下未能生长,其培养体系中甲醇浓度的轻微降低可归因于自然挥发。
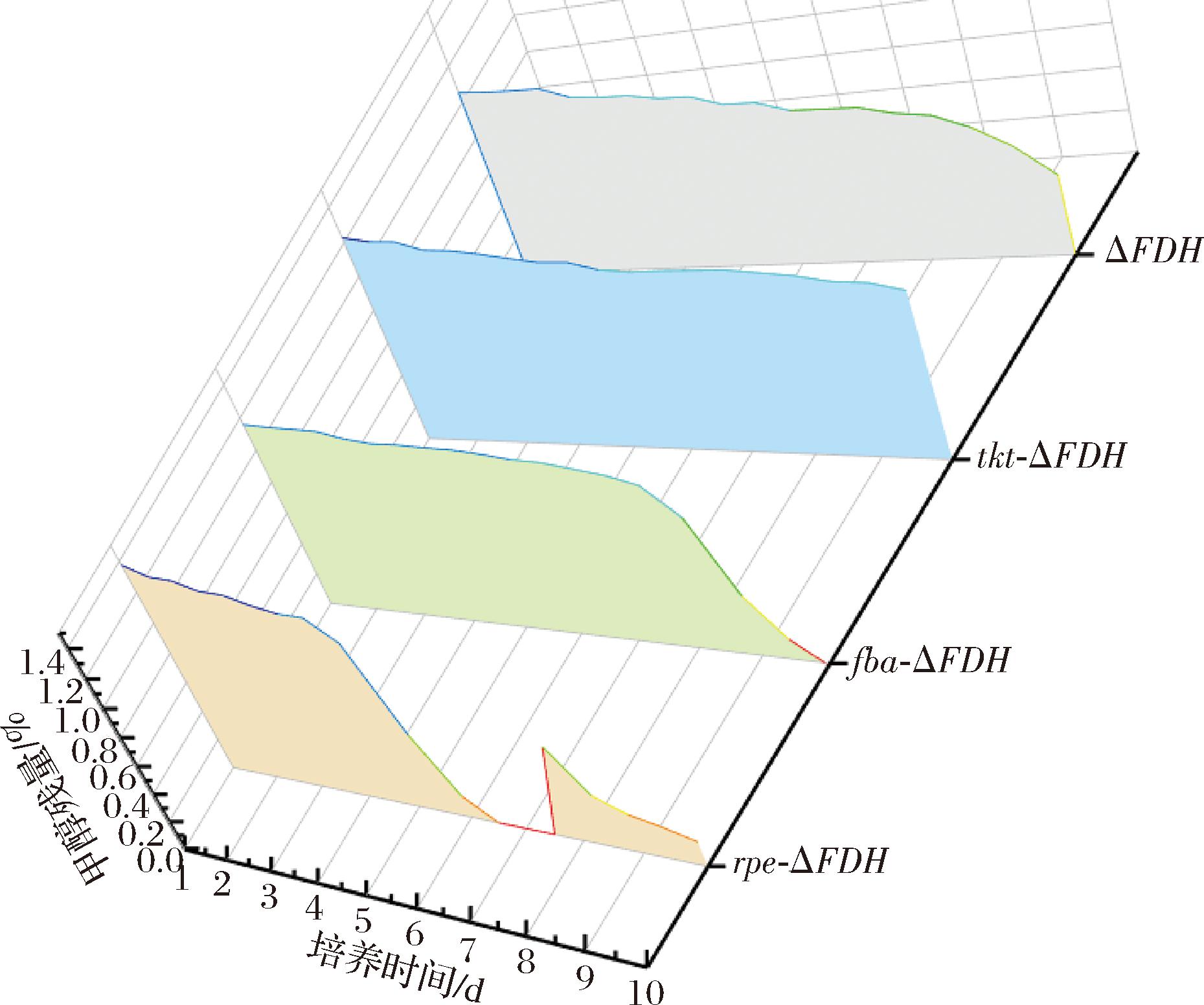
图8 以ΔFDH为出发菌的重组菌的甲醇消耗情况
Fig.8 Methanol consumption profile in recombinant strains using ΔFDH as host
以上结果说明过表达rpe、tkt和fba基因在各自适用的条件下能够提高重组菌的甲醇利用率,但其提升幅度与出发菌株的遗传背景密切相关。进一步说明调控PPP有助于提高K.phaffii的甲醇利用率。
2.5 甲醇同化效率分析
定量分析不同菌株的甲醇同化效率,可作为菌株性能评估的一项重要指标,也为筛选高效工程菌提供数据支撑。甲醇同化效率的高低可以反映菌株利用甲醇的活跃程度,评估甲醇转化为目标产物(如菌体、代谢产物)的效率。通常情况下,同化效率的计算方法需结合具体的代谢途径、碳流向及能量平衡等方面。其中,通过13C标记实验进行的代谢通量分析能够给出各代谢途径节点的甲醇同化量占比(或碳通量分布)。或通过构建稳态模型,用来模拟碳源消耗的动力学特征或关联生长速率等数据[18]。在本研究中,受限于客观条件,仅从单位甲醇消耗产生的生物量这一角度来衡量甲醇的同化效率。具体计算方法如下:
将各菌株的生物量OD600值换算成细胞干重(dry cell weight,DCW),基于本实验室前期工作基础,换算系数为0.3。甲醇质量浓度单位用g/L来表示,经计算,此处换算系数为7.918。分别计算各相邻时间点之间的生物量差值ΔDCW,同步对标同一时间段内的甲醇消耗量ΔMeOH,然后计算两者比值ΔDCW/ΔMeOH,即为甲醇同化效率。基于“2.1节生长曲线”,为了突出差异,对于以GS115为出发菌的重组菌中,只计算比GS115更早生长的fba-GS115和tkt-GS115,对于以ΔFLDH为出发菌的实验组,只计算了fba-ΔFLDH和tkt-ΔFLDH的相关参数,结果见图9。
a- GS115为出发菌;b-ΔFLDH为出发菌;c-ΔFDH为出发菌
图9 各重组菌的甲醇同化效率
Fig.9 Methanol assimilation efficiency of recombinant strains
在以GS115为出发菌的各菌株中(图9-a),最明显的特点是tkt-GS115在2.5 d的甲醇同化效率最高,这与在生长表型分析中的优先生长的事实是一致的。其他时间点并无明确的规律性。而在以ΔFLDH为出发菌的各菌中(图9-b),从起始生长至5.5 d这一时间段,ΔFLDH保持较高的甲醇同化效率,虽然fba-ΔFLDH最先起始生长,经验上认为其利用甲醇的能力更强,但通过它的甲醇同化效率并未超过出发菌ΔFLDH。同样对于最晚生长的tkt-ΔFLDH,它的甲醇同化效率却高于ΔFLDH。直到6.5 d后,各菌株生长已以进入稳定期,甲醇利用效率也趋于平衡。以ΔFDH为出发菌的各菌中(图9-c),rpe-ΔFDH虽然最先生长,但它的甲醇同化效率却低于fba-ΔFDH。这说明分析的角度不同,得出的结论也有差异。起始生长的时间可以从一定程度上表明菌株的甲醇利用能力,但达到稳定期所用的时间及最终生物量也是不可忽略的因素。分阶段分析才能客观反映菌株利用甲醇的特征。
2.6 过表达rpe、tkt和fba后的转录水平分析
为了后续深入探究各基因引起表型或甲醇消耗变化的原因,针对生长有明显变化的重组菌,通过qPCR检测rpe、tkt和fba的对数期转录水平,为进一步解析机制提供基础依据。
由图10可知,rpe-ΔFLDH、rpe-ΔFDH和fba-ΔFDH中的rpe和fba比对照组的表达水平高2~5倍,对于这2株而言,rpe和fba的转录水平提高,对甲醇利用起正向作用。
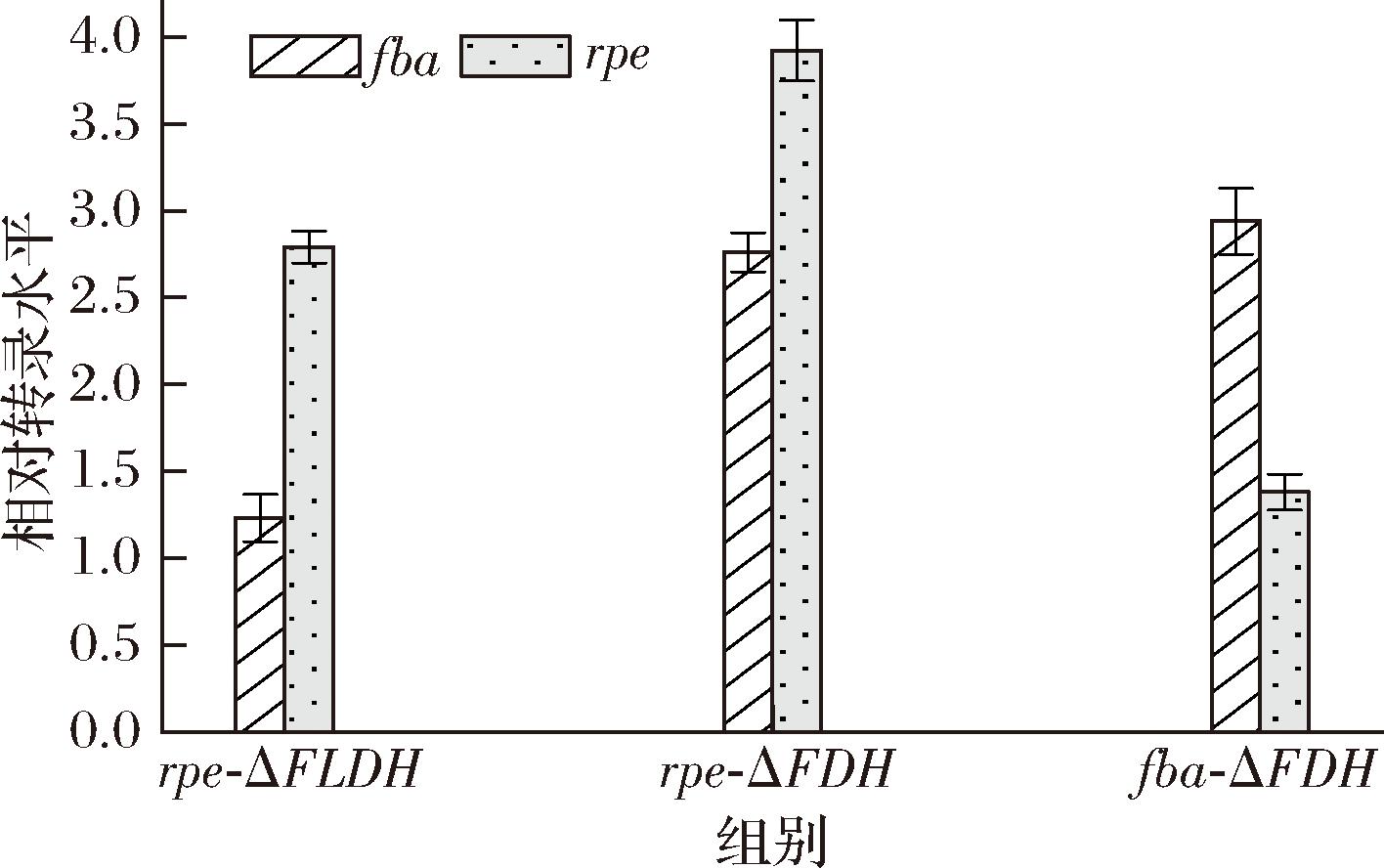
图10 重组菌株中rpe和fba的转录水平
Fig.10 Transcription level of rpe and fba in recombinant strains
3 结论与讨论
在不同出发背景的菌株中分别表达rpe、tkt和fba后,各重组菌的生长特性呈现多样化差异:在以GS115为出发菌的重组菌中,tkt-GS115表现出约1.7%的生物量提升,其最大比生长速率达到0.23 h-1,只有fba-GS115的甲醇利用率没有提高;在以ΔFLDH为亲本的重组菌株中,在培养初期ΔFLDH还未开始生长时,fba-ΔFLDH以最大比生长速率为0.23 h-1的优势领先生长,在本组中,其甲醇利用效率最高;而对于以ΔFDH为出发菌的重组菌中,rpe-ΔFDH和fba-ΔFDH逆转了其亲本菌株ΔFDH不能在甲醇基本培养基中生长的状况,rpe-ΔFDH以最高生物量达到6.97展现出巨大的生长潜力,而fba-ΔFDH的最大比生速率为0.36 h-1。
本研究证明过表达PPP的tkt、fba和rpe能够在不同的出发菌中发挥促进甲醇同化代谢的效果,为后续开发高效甲醇利用的工业菌株奠定了重要的实验基础。
[1] HÇELIK E, ÇAL K P, OLIVER S G. Metabolic flux analysis for recombinant protein production by Pichia pastoris using dual carbon sources: Effects of methanol feeding rate[J]. Biotechnology and Bioengineering, 2010, 105(2):317-329.
K P, OLIVER S G. Metabolic flux analysis for recombinant protein production by Pichia pastoris using dual carbon sources: Effects of methanol feeding rate[J]. Biotechnology and Bioengineering, 2010, 105(2):317-329.
[2] HZHANG M Z, TIAN Y, ZHOU H, et al. Simultaneous elimination mechanism of formaldehyde and acrolein by resveratrol in food and the cytotoxicity of the products[J]. Food Chemistry, 2025, 468:142371.
[3] HBAZURTO J V, BRUGER E L, LEE J A, et al. Formaldehyde-responsive proteins, TtmR and EfgA, reveal a tradeoff between formaldehyde resistance and efficient transition to methylotrophy in Methylorubrum extorquens[J]. Journal of Bacteriology, 2021, 203(9): JB.00589-JB.00520.
[4] HBAZURTO J V, RIAZI S, D’ALTON S, et al. Global transcriptional response of methylorubrum extorquens to formaldehyde stress expands the role of EfgA and is distinct from antibiotic translational inhibition[J]. Microorganisms, 2021, 9(2):347.
[5] HYU Y F, YANG J S, ZHAO F G, et al. Comparative transcriptome and metabolome analyses reveal the methanol dissimilation pathway of Pichia pastoris[J]. BMC Genomics, 2022, 23(1):366.
[6] HWANG S X, FANG J Y, WANG M Y, et al. Rewiring the methanol assimilation pathway in the methylotrophic yeast Pichia pastoris for high-level production of erythritol[J]. Bioresource Technology, 2025, 427:132430.
[7] HKRÜSEMANN J L, RAINALDI V, COTTON C A, et al. The cofactor challenge in synthetic methylotrophy: Bioengineering and industrial applications[J]. Current Opinion in Biotechnology, 2023, 82:102953.
[8] HKELSO P A, CHOW L K M, CARPENTER A C, et al. Toward methanol-based biomanufacturing: Emerging strategies for engineering synthetic methylotrophy in Saccharomyces cerevisiae[J]. ACS Synthetic Biology, 2022, 11(8):2548-2563.
[9] HBERTELS L K, FERN NDEZ MURILLO L, HEINISCH J J. The pentose phosphate pathway in yeasts-more than a poor cousin of glycolysis[J]. Biomolecules, 2021, 11(5):725.
NDEZ MURILLO L, HEINISCH J J. The pentose phosphate pathway in yeasts-more than a poor cousin of glycolysis[J]. Biomolecules, 2021, 11(5):725.
[10] HDMYTRUK O, BULBOTKA N, ZAZULYA A, et al. Fructose-1, 6-bisphosphatase degradation in the methylotrophic yeast Komagataella phaffii occurs in autophagy pathway[J]. Cell Biology International, 2021, 45(3):528-535.
[11] HDE SIMONE A, VICENTE C M, PEIRO C, et al. Mixing and matching methylotrophic enzymes to design a novel methanol utilization pathway in E. coli[J]. Metabolic Engineering, 2020, 61:315-325.
[12] HJOHANSSON B, HAHN-H GERDAL B. The non-oxidative pentose phosphate pathway controls the fermentation rate of xylulose but not of xylose in Saccharomyces cerevisiae TMB3001[J]. FEMS Yeast Research, 2002, 2(3):277-282.
GERDAL B. The non-oxidative pentose phosphate pathway controls the fermentation rate of xylulose but not of xylose in Saccharomyces cerevisiae TMB3001[J]. FEMS Yeast Research, 2002, 2(3):277-282.
[13] HWOOLSTON B M, ROTH T, KOHALE I, et al. Development of a formaldehyde biosensor with application to synthetic methylotrophy[J]. Biotechnology and Bioengineering, 2018, 115(1):206-215.
[14] HROHLHILL J, GERALD HAR J R, ANTONIEWICZ M R, et al. Improving synthetic methylotrophy via dynamic formaldehyde regulation of pentose phosphate pathway genes and redox perturbation[J]. Metabolic Engineering, 2020, 57:247-255.
[15] HCHUANG D S, LIAO J C. Role of cyanobacterial phosphoketolase in energy regulation and glucose secretion under dark anaerobic and osmotic stress conditions[J]. Metabolic Engineering, 2021, 65:255-262.
[16] HBENNETT R K, AGEE A, GERALD HAR J R, et al. Triggering the stringent response enhances synthetic methanol utilization in Escherichia coli[J]. Metabolic Engineering, 2020, 61:1-10.
[17] HWU T, G MEZ-CORONADO P A, KUBIS A, et al. Engineering a synthetic energy-efficient formaldehyde assimilation cycle in Escherichia coli[J]. Nature Communications, 2023, 14:8490.
MEZ-CORONADO P A, KUBIS A, et al. Engineering a synthetic energy-efficient formaldehyde assimilation cycle in Escherichia coli[J]. Nature Communications, 2023, 14:8490.
[18] HPEYRAUD R, SCHNEIDER K, KIEFER P, et al. Genome-scale reconstruction and system level investigation of the metabolic network of Methylobacterium extorquens AM1[J]. BMC Systems Biology, 2011, 5:189.