5-氨基乙酰丙酸(5-aminolevulinic acid,5-ALA)又称δ-氨基乙酰丙酸,是一种存在于自然界的非蛋白原功能性五碳氨基酸[1]。5-ALA是生物合成卟啉、叶绿素、亚铁血红素和维生素B12等四吡咯化合物的共同前体物质[2-5],广泛存在于微生物、植物和动物细胞中,在医药、农业和护肤领域具有重要的应用。例如,在医学领域,5-ALA可用于定位肿瘤[6],实现疾病的早期诊断;5-ALA也可用于肿瘤的光动力治疗[7-9]。在农业领域,5-ALA主要作为除草剂[10]、杀虫剂[11]和植物生长调节剂[12]。另外5-ALA还可用于美容护肤领域,促进新生角质细胞的更替和减少色素过度沉着等[13]。
目前5-ALA的合成方法主要有化学合成法和生物合成法,其中化学合成法成本较高,分离和纯化步骤较多,反应条件严苛,产物收率低[14-15];生物合成法主要有C4和C5两条途径,其中C5途径的酶促反应比较复杂,包括谷氨酰-tRNA合成酶(由gltX编码)、NADPH依赖的谷氨酰-tRNA-还原酶HemA(由hemA编码)和谷氨酸-1-半醛氨基转移酶HemL(由hemL编码)这3种关键酶催化[16-18]。C4途径仅需5-氨基乙酰丙酸合成酶(5-aminolevulinic acid synthase,ALAS)一种酶催化,其以甘氨酸(glycine,Gly)和琥珀酰-辅酶A为底物,在辅酶磷酸吡哆醛(pyridoxal phosphate,PLP)作用下生成5-ALA[19-21]。C4途径在适宜条件下可快速合成产物,反应过程直接、简洁,相比C5途径的多步酶促反应,减少了中间环节的能量损耗和物质消耗,易于进一步的改造和调控。
目前,对C4途径的关键酶——ALAS的研究主要集中在酶的挖掘及表征上,但大多数ALAS都具有酶活力低、热稳定性差、易被血红素抑制等特点[22],这严重限制了其工业化应用。本研究筛选了9种不同来源的ALAS,综合酶可溶性蛋白表达水平以及酶活力,选择了沼泽红假单胞菌(Rhodopseudomonas palustris)来源的RpALAS进行分子改造。目前,CHOI等[23]对RpALAS的研究是将hemA基因克隆到原核表达载体pGEX-KG中,然后在Escherichia coli BL21中表达,并对其进行了酶学性质解析。WANG等[24]通过对RpALAS进行分子改造,获得的组合突变体R97H/T317I的酶活力是野生型(wild type,WT)的1.12倍,并且提高了可溶性蛋白表达水平。本研究对沼泽红假单胞菌来源的RpALAS进行了Alphafold建模和分子对接,利用Fireprot和HotSpot Wizard网站对RpALAS进行理性设计,并通过分子动力学模拟进一步分析了RpALAS热稳定提升的原因,为提高ALAS在工业上的应用潜力奠定了一定的基础。
1 材料与方法
1.1 材料与试剂
1.1.1 主要试剂
Gly,上海麦克林生化科技股份有限公司;琥珀酰-辅酶A、PLP,Sigma(西格玛奥德里奇)公司;葡萄糖、三氯乙酸、冰醋酸、乙酰丙酮,国药集团药业股份有限公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG),上海生工生物工程有限公司。
1 mol/L乙酸钠溶液(pH 4.6):用称量纸准确称取无水乙酸钠8.2 g,溶解于去离子水中,用冰醋酸调至pH值为4.6,去离子水定容至100 mL。
Ehrlich’s试剂:0.2 g对二甲氨基苯甲醛溶于1.6 mL纯度为70%(质量分数)高氯酸中,再用冰乙酸定容至10 mL,现配现用。
1.1.2 培养基
LB培养基(g/L):酵母提取物5.0,胰蛋白胨10.0,氯化钠10.0。固体培养基加入质量分数为2%的琼脂粉。
2×酵母提取物胰蛋白胨(2×yeast extract-tryptone,2YT ) 培养基(g/L):蛋白胨16.0,酵母粉10.0,氯化钠5.0。
1.1.3 质粒与菌株
E. coli BL21(DE3)、E. coli JM109、表达载体pET-28a(+)为实验室保藏;RhoALAS-pET28a、RpALAS-pET28a、BraALAS-pET28a、RsALAS-pET28a、BsALAS-pET28a、EtALAS-pET28a、PbALAS-pET28a、RcALAS-pET28a、TtALAS-pET28a均由本实验室构建。
1.1.4 工具酶
Primer STAR Max DNA聚合酶、Quick Cut Dpn I消化酶,TaKaRa宝日医生物技术有限公司。
1.2 仪器与设备
C1000 Touch PCR仪,美国伯乐(Bio-Rad)公司;Synergy H1酶标仪,美国博腾仪器有限公司;29707636 AKTA pureTM25T蛋白纯化仪,通用电器医疗器械集团;FiveEasy PlusTMFE28pH计,梅特勒-托利多仪器(上海)有限公司;SONICS VCX 750超声波破碎仪,美国SONICS&MATERIALS公司;UV-1800紫外可见分光光度计,上海美谱达仪器有限公司;Mini-PROTEAN®Tetra蛋白电泳仪,美国伯乐(Bio-Rad)生命医学产品有限公司。
1.3 实验方法
1.3.1 突变体构建
以pET-28a-RpALAS基因组为模板,用Primer STAR Max DNA聚合酶进行全质粒PCR。PCR总体系25 μL,其中:上游引物、下游引物各1 μL,质粒模板1 μL,无菌水22 μL。反应条件为:98 ℃预变性1 min,98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸90 s,72 ℃延伸5 min,共30个循环。本研究所用引物如表1所示。PCR产物用DPn I消化质粒模板,消化产物转入JM109感受态,涂布于卡那霉素抗性平板,37 ℃培养箱过夜培养,送至苏州金唯智生物科技有限公司进行测序。测序成功后,化转法将质粒转化至BL21(DE3)感受态细胞中,37 ℃培养箱过夜培养即为重组菌。
表1 本研究验实所用引物
Table 1 Primers used in this study

注:下划线部分是引入突变的位点。
引物名称引物序列(5′→3′)D12Q-FCGGATGCGCTGCAGCGCCTGCATGD12Q-RCTGCAGCGCATCCGCAAAAAATTTGGT73L-FATGGTGGAGACGGCGCTGCGCTTAGGCACT73L-RCCGTCTCCACCATCGCGCV116S-FTACGAGCGGCTATAGCAGCAATCAGACCGGCATV116S-RATAGCCGCTCGTAAACAGCAGCV116D-FTACGAGCGGCTATGATAGCAATCAGACCGGV116D-RATAGCCGCTCGTAAACAGCAGCS123R-FCGCACCCTGGCGAAATTAATTCCGAAS123R-RTTCGCCAGGGTGCGAATGCCGA138E-FTGATTCTGAGCGATGAACTGAACCATAACAGCATGATTGAAA138E-RATCGCTCAGAATCAGGCAGTTCGL139M-FTTCTGAGCGATGCGATGAACCATAACAGL139M-RCGCATCGCTCAGAATCAGGCL139F-FTTCTGAGCGATGCGTTTAACCATAACAGCATGATTGAAL139F-RCGCATCGCTCAGAATCAGGCL139M-FTTCTGAGCGATGCGATGAACCATAACAGE174P-FCGCGCGGTGCCGCCGGGCCGCCE174P-RCACCGCGCGCAGCAGTTE242L-FCGCATTGATATTATTCTGGCGACCTTAGCGAAAGCGTTTGE242L-RAATAATATCAATGCGATGCATCACGCCA243G-FATTGATATTATTGAAGGCACCTTAGCGAAAGCGTTTGGCA243G-RTTCAATAATATCAATGCGATGCATCACGCA246G-FATTGAAGCGACCTTAGGCAAAGCGTTTGGA246G-RTAAGGTCGCTTCAATAATATCAATGCGATGCL252H-FGCGTTTGGCTGCCATGGCGGCTATATTAGL252 N-FGCGTTTGGCTGCAACGGCGGCTATATTAGCGL252D-FGCGTTTGGCTGCGATGGCGGCTATATTAGCGL252C-FGCGTTTGGCTGCTGCGGCGGCTATATTAGCGL252E-FGCGTTTGGCTGCGAAGGCGGCTATATTAGCGL252G-FGCGTTTGGCTGCGGCGGCGGCTATATTAGCGL252K-FGCGTTTGGCTGCAAAGGCGGCTATATTAGCGL252S-FGCGTTTGGCTGCAGCGGCGGCTATATTAGCGL252T-FGCGTTTGGCTGCACCGGCGGCTATATTAGCGL252Y-FGCGTTTGGCTGCTATGGCGGCTATATTAGCGL252I-FGCGTTTGGCTGCATTGGCGGCTATATTAGCL252F-FGCGTTTGGCTGCTTTGGCGGCTATATTAGCL252A-FGCGTTTGGCTGCGCGGGCGGCTATATTAGL252R-FGCGTTTGGCTGCCGCGGCGGCTATATTAGL252 W-FGCGTTTGGCTGCTGGGGCGGCTATATTAGL252Q-FGCGTTTGGCTGCCAGGGCGGCTATATTAL252P-FGCGTTTGGCTGCCCGGGCGGCTATATTAL252V-FGCGTTTGGCTGCGTGGGCGGCTATATTAL252-RGCAGCCAAACGCTTTCGCL252M-FAGCGTTTGGCTGCATGGGCL252M-RGCAGCCAAACGCTTTCGCT288L-FCGCTGGCGGCGATTCGCCATCTGT288L-RGCCGCCAGCGCGGCCGCGCAAATD381P-FCGTATCATGATGACCCGCTGATGGATCGCCTGGCGD381P-RGTCATCATGATACGGGCTCGGGGT396M-FTGGATGTGTGGGAAATGCTGGAACTGCCGCT396M-RTTCCCACACATCCACCAGCG
1.3.2 ALAS的诱导表达、电泳鉴定及纯化
将9种重组菌株的保菌管在LB平板上划线(卡那霉素质量浓度为50 μg/mL),37 ℃培养箱培养过夜。挑取单菌落接种至含有5 mL LB(卡那霉素质量浓度为50 mg/mL)培养基中37 ℃ 200 r/min培养6~8 h,以2%接种量转接至2YT培养基中培养至OD600为0.6~0.8时加入终浓度为0.1 mmoL/L的IPTG诱导,25 ℃,200 r/min培养16~20 h。4 ℃ 8 000 r/min离心10 min收菌。50 mmoL/L Tris-HCl,pH 7.5重悬菌体,超声破碎仪破碎(破3 s停7 s,破碎10 min),将破碎液在离心机4 ℃ 12 000 r/min离心,离心后的上清液即为粗酶液,吸取适量粗酶液进行SDS-PAGE分析重组酶的表达情况。
粗酶液用0.22 μm水系滤膜过滤,蛋白纯化采用1 mL His Trap HP柱子亲和层析,A液(20 mmoL/L咪唑、20 mmol/L NaH2PO4、500 mmol/L NaCl,pH 7.4)、B液(500 mmol/L咪唑、20 mmol/L NaH2PO4、500 mmol/L NaCl,pH 7.4)线性梯度洗脱目的蛋白,收集洗脱液用于后续研究。纯化后的酶液用50 mmoL/L Tris-HCl、100 mmol/L NaCl,pH 7.5、10%(体积分数)甘油于4 ℃层析柜过夜透析。纯酶采用Bradford法测定蛋白浓度、显色法测定比酶活力。
1.3.3 ALAS酶活力测定
酶活力单位(U)的定义:每1 min形成1 μmol ALA所需的酶量定义为一个酶活力单位(1 U)。
酶活力检测体系:总反应体系为200 μL,其中包括:100 mmol/L Gly、0.2 mmol/L琥珀酰-辅酶A、0.1 mmol/L PLP、20 mmol/L MgCl2、50 mmol/L Tris-HCl(pH 7.5),适量的纯酶。把除纯酶以外的反应物在37 ℃金属浴中预热5 min后加入适量纯酶反应5 min,150 μL 10%(质量分数)三氯乙酸终止反应。
显色法测定产物5-ALA:终止反应后4 ℃ 12 000 r/min离心10 min。取200 μL上清液与100 μL 1 mol/L乙酸钠溶液(pH 4.6),50 μL乙酰丙酮溶液100 ℃反应15 min,冷却至室温,加入等量的Ehrlich’s试剂避光反应10 min。然后用酶标仪在554 nm处测定吸光度。
1.3.4 突变体酶学性质测定
最适温度:25、30、35、40、45 ℃下测定酶活力,以测得的最高酶活力定义为100%,计算其他温度下的相对酶活力。
最适pH:分别用50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH 6.0~7.0)和50 mmol/L Tris-HCl缓冲液(pH 7.0~9.0)将pH值调至相应pH后测定酶活力,以测得的最高酶活力定义为100%,计算其他pH下的相对酶活力。
热稳定性测定:将0.2 mg/mL酶液置于45 ℃金属浴中孵育,每隔一段时间取样后冰上冷却5 min,以测得的最高酶活力定义为100%,计算相对酶活力。
半衰期t1/2:把纯酶均稀释到0.2 mg/mL置于45 ℃水浴锅中,孵育不同时间取出后插至冰上冷却5 min后测定其比酶活力,以测得的最高酶活力定义为100%。以在水浴锅中处理酶的时间作为X轴,相对酶活力为Y轴,即可得到纯酶失活曲线,酶活力达到最大酶活力一半的时间即为半衰期t1/2。
1.3.5 结构构建及分子对接
利用Alphafold2.0对沼泽红假单胞菌来源的RpALAS建模,从PubChem数据库(https://pubchem.ncbi.nlm.nih.gov/)下载:PLP、琥珀酰-辅酶A、Gly的结构,使用Schrödinger软件将PLP、琥珀酰-辅酶A、Gly对接到RpALAS的结构模型中。根据该服务器的评分函数和置信度选择最佳的对接结果。蛋白质结构可视化由PyMol展示。
1.3.6 分子动力学模拟
使用GPU版本的GROMACS 2023.2进行分子动力学模拟,并采用Amber 14SB力场,模拟在常压下的300 K和370 K温度条件下进行,总时长为100 ns,蛋白质置于一个TIP3P(transferable intermolecular potential 3-point)水盒中溶解,最小边界距离设为10 Å,添加0.15 moL/L的氯化钠离子以保证体系呈现电中性。利用GROMACS分析套件进行均方根偏差(root mean square deviation,RMSD)分析。
1.4 数据处理
所有数据均表示为3次独立实验的平均值,图中的误差线表示使用Origin 2021计算的标准误差。
2 结果与分析
2.1 ALAS的挖掘
以沼泽红假单胞菌来源的RsALAS为探针,在基因库中筛选并合成了9种ALAS(表2)。
表2 不同来源的ALAS的合成
Table 2 Synthesis of ALAS from different sources
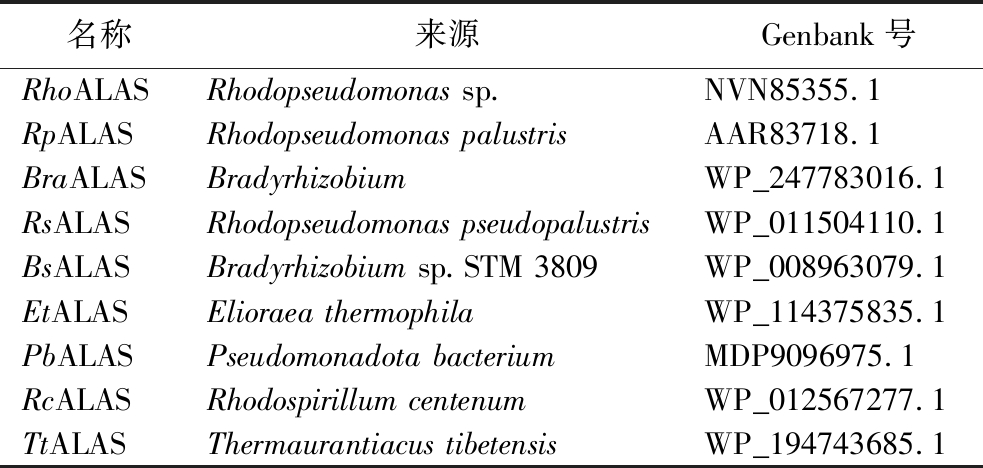
名称来源Genbank号RhoALASRhodopseudomonas sp.NVN85355.1RpALASRhodopseudomonas palustrisAAR83718.1BraALASBradyrhizobiumWP_247783016.1RsALASRhodopseudomonas pseudopalustrisWP_011504110.1BsALASBradyrhizobium sp.STM 3809WP_008963079.1EtALASElioraea thermophilaWP_114375835.1PbALASPseudomonadota bacteriumMDP9096975.1RcALASRhodospirillum centenumWP_012567277.1TtALASThermaurantiacus tibetensisWP_194743685.1
将这9种来源的ALAS在E.coli BL21中进行异源表达,SDS-PAGE检测9种ALAS的蛋白表达水平。结果如图1所示,以未诱导的菌体的上清液和沉淀作为对照,诱导后蛋白质量大小约为46 kDa,其中RhoALAS、RpALAS、EtALAS这3种来源ALAS的表达比较好。
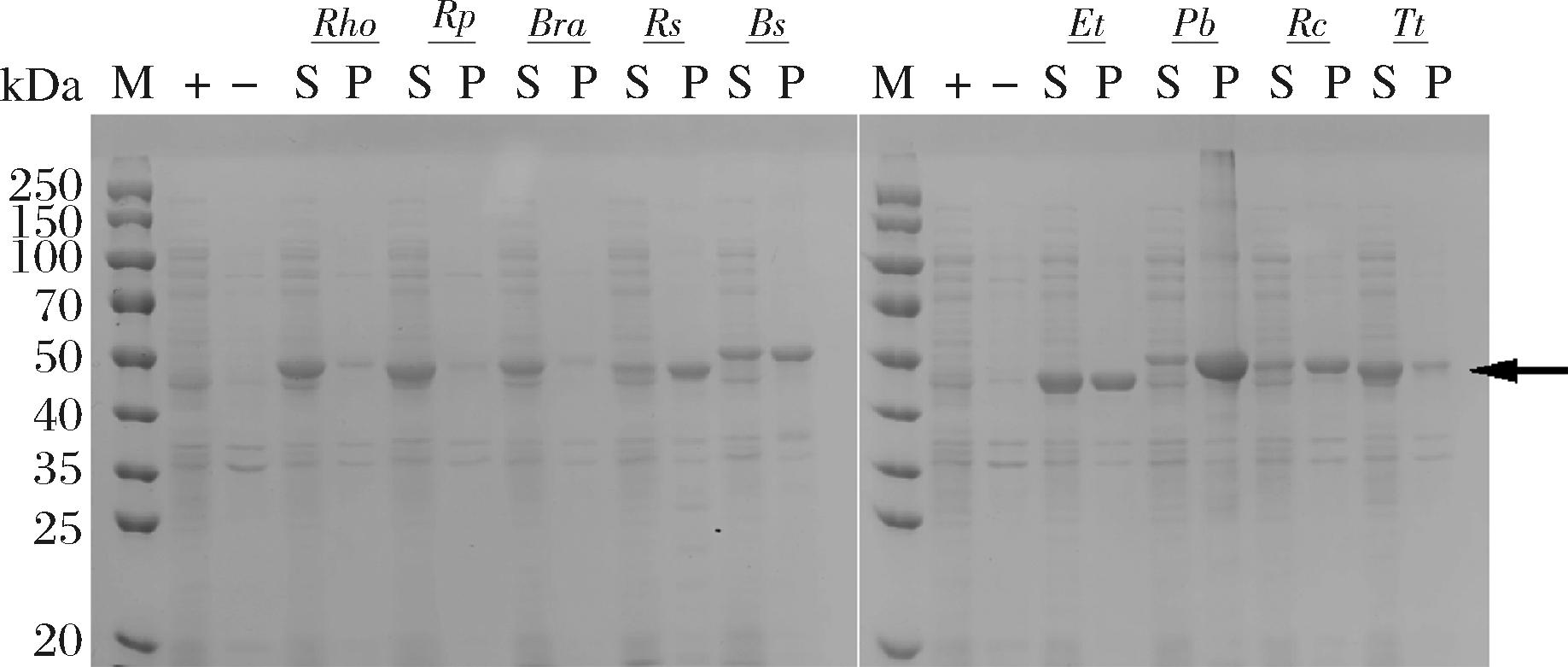
图1 九种来源的ALAS的SDS-PAGE图
Fig.1 SDS-PAGE images of ALAS from nine different sources
注:M:Marker;+:未诱导菌体破碎液上清液;-:未诱导菌体破碎液沉淀;S:诱导后菌体破碎上清液;P:诱导后菌体破碎沉淀。
对诱导后的蛋白进行纯化,透析后的蛋白利用Bradford法测定浓度,分别测定其比酶活力。结果如图2所示,检测到比酶活力最高的是RpALAS,其比酶活力为0.45 U/mg;RhoALAS比酶活力为0.25 U/mg,EtALAS比酶活力为0.09 U/mg。综合考虑蛋白表达水平和酶活力,选取沼泽红假单胞菌来源的RpALAS进行后续分子改造。
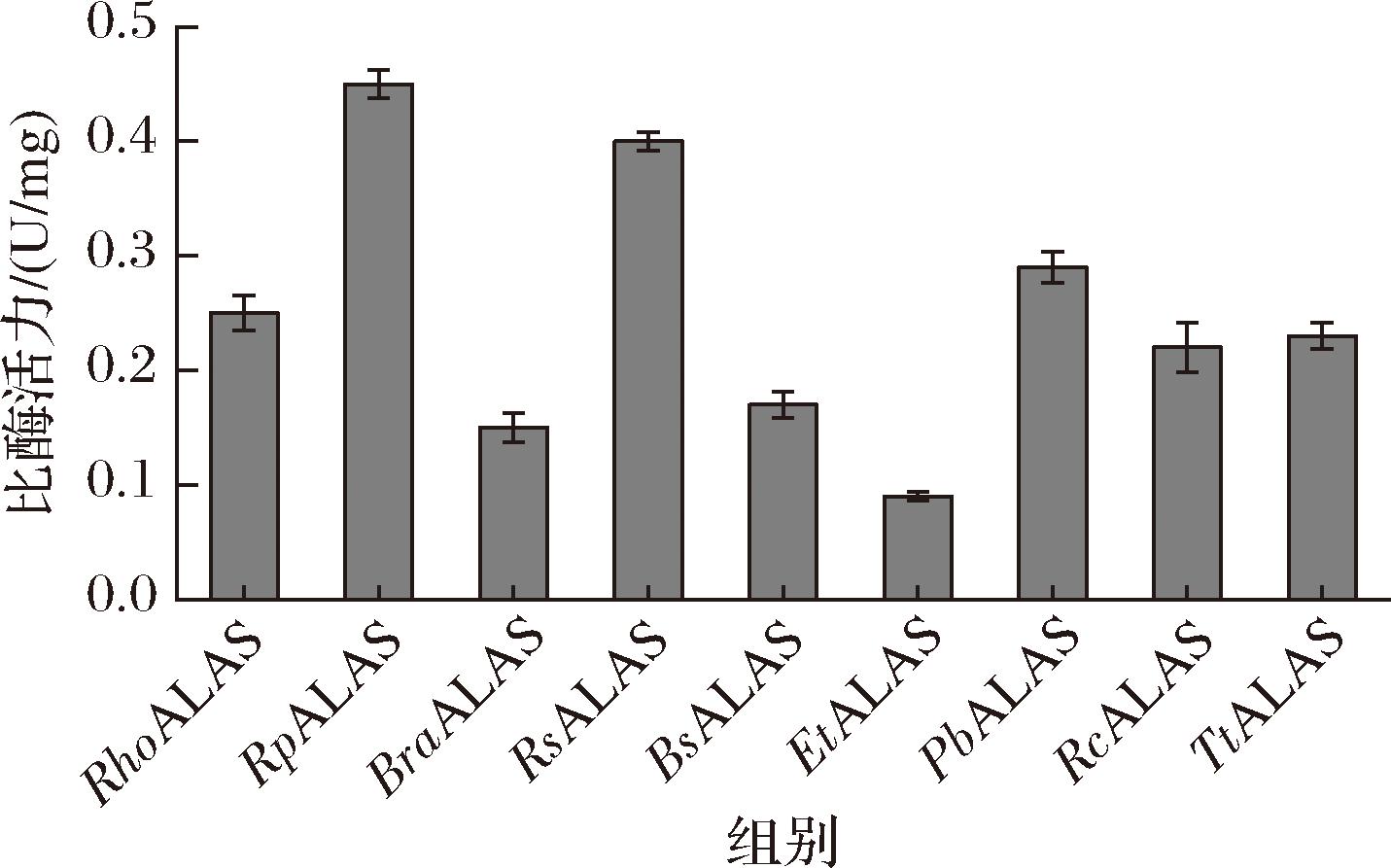
图2 九种ALAS的比酶活力
Fig.2 Nine specific enzyme activities of ALAS
2.2 ALAS分子改造
由于ALAS酶活力低、热稳定性较差,不利于其进行工业化应用,需要对其改造提升其活性和热稳定性。对RpALAS进行结构建模及分子对接,随后将优化过后的结构模型上传至Fireprot网站(https://loschmidt.chemi.muni.cz/fireprot/),根据计算结果中的Rosetta和FoldX值筛选热稳定性提升的潜在位点。设定筛选条件为Rosetta和Fold X的自由能降低绝对值大于1 kcal/moL,在排除绝对保守的氨基酸后,剩余7个单点突变体(表3)。
表3 Fireprot构建的突变体
Table 3 Mutants constructed by Fireprot
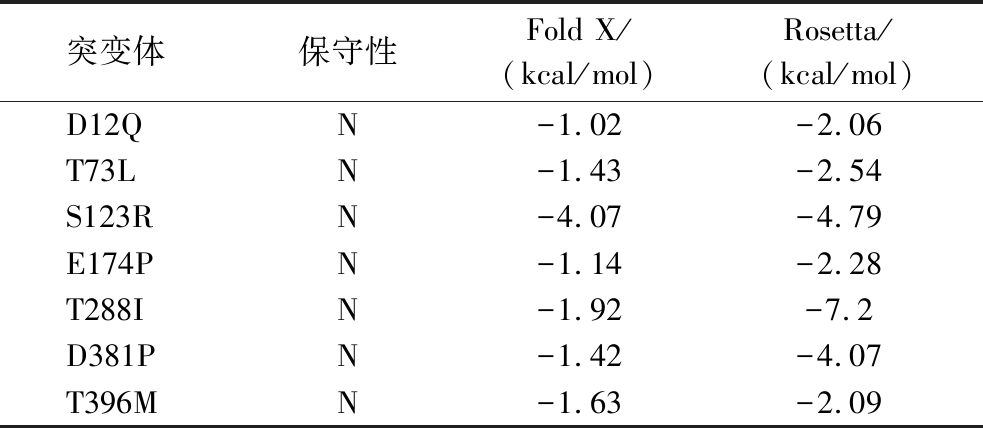
注:N代表non-conserved(非保守的)。
突变体保守性Fold X/(kcal/mol)Rosetta/(kcal/mol)D12QN-1.02-2.06T73LN-1.43-2.54S123RN-4.07-4.79E174PN-1.14-2.28T288IN-1.92-7.2D381PN-1.42-4.07T396MN-1.63-2.09
将筛选获得的7个突变体测定其酶活力和热稳定性,结果如图3所示,7个突变体的酶活力均没有提升,但突变体T73L、T396M在45 ℃孵育30 min后的残余酶活力从WT的38%分别提升至75%和55.3%,残余酶活力分别是WT的1.97和1.46倍。
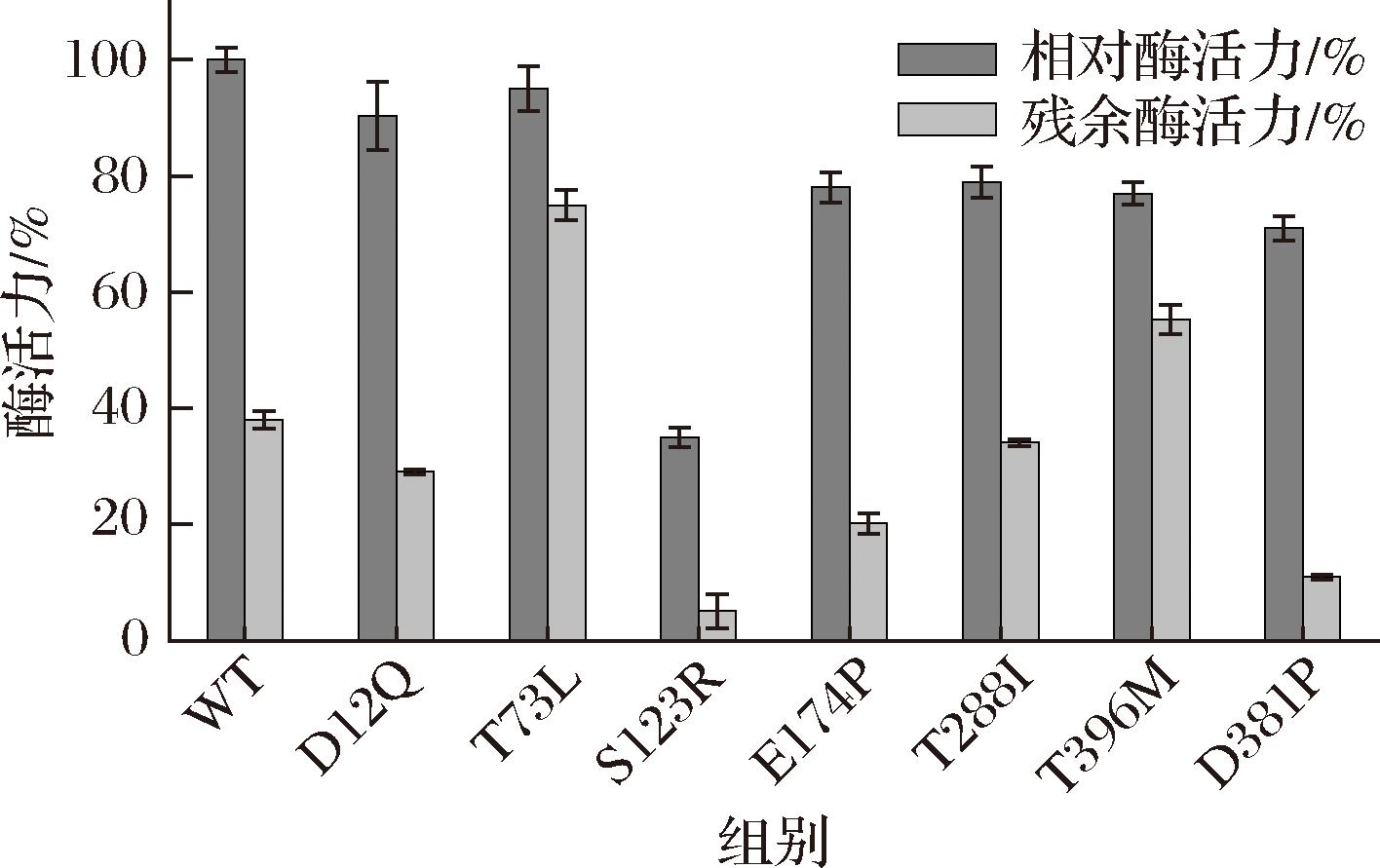
图3 Fireprot设计突变体的酶活力和残余酶活力
Fig.3 Enzyme activity and residual enzyme activity of mutants designed by Fireprot
将RpALAS结构上传至HotSpot Wizard(https://loschmidt.chemi.muni.cz/hotspotwizard/)在线网站进行结构分析,以期望获得酶活力提高的正向突变体。通过序列比对排除绝对保守的氨基酸后,获得了V116S、V116D、A138E、L139F、L139M、A246G、E242L、A243G、L252H共9个突变体。酶活力测定结果如图4-a所示,其中L252H相较WT酶活力提升了10%左右。随后对RpALAS的252位氨基酸进行饱和突变,结果如图4-b所示,发现L252F相对于野生型酶活力大约提升20%。
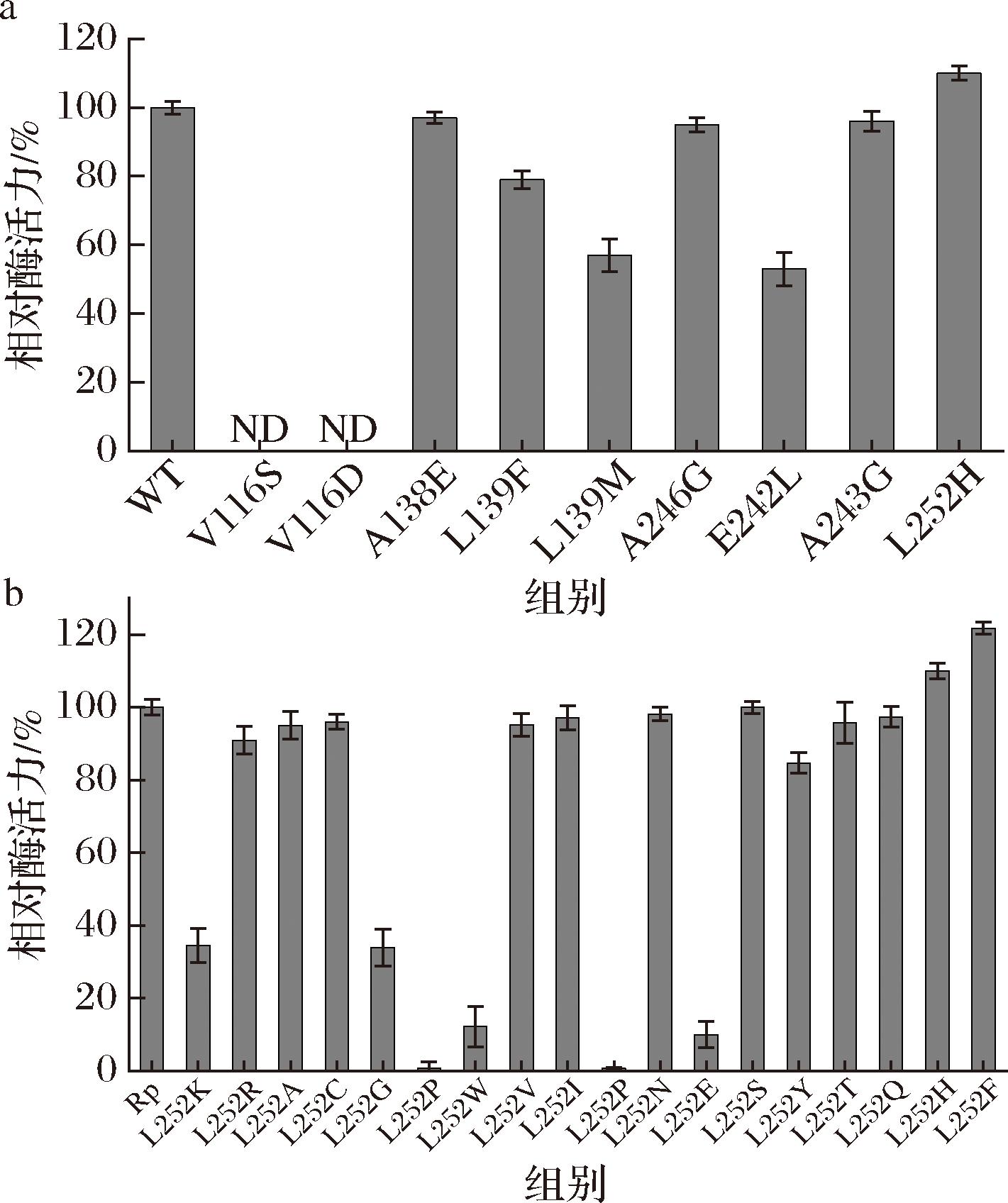
a-HotSpot Wizard在线网站设计的突变体; b-RpALAS中252位氨基酸饱和突变
图4 HotSpot Wizard设计的突变体筛选
Fig.4 Mutant screening by HotSpot Wizard design
注:ND:未检出。
2.3 组合突变
构建突变体T73L/L252F(M1)、T73L/T396M(M2)、L252F/T396M(M3)、T73L/L252F/T396M(M4)。测定每个突变体比酶活力和残余酶活力。结果如图5所示。突变体M1、M2、M3的酶活力相较于WT分别下降3.14%、2.99%、9.47%,M4较WT提升5%,残余酶活力分别是78.25%、37.97%、29.8%、84.92%,突变体M1、M4的残余酶活力分别是WT的2.06、2.23倍,由此确定最佳突变体为M4。
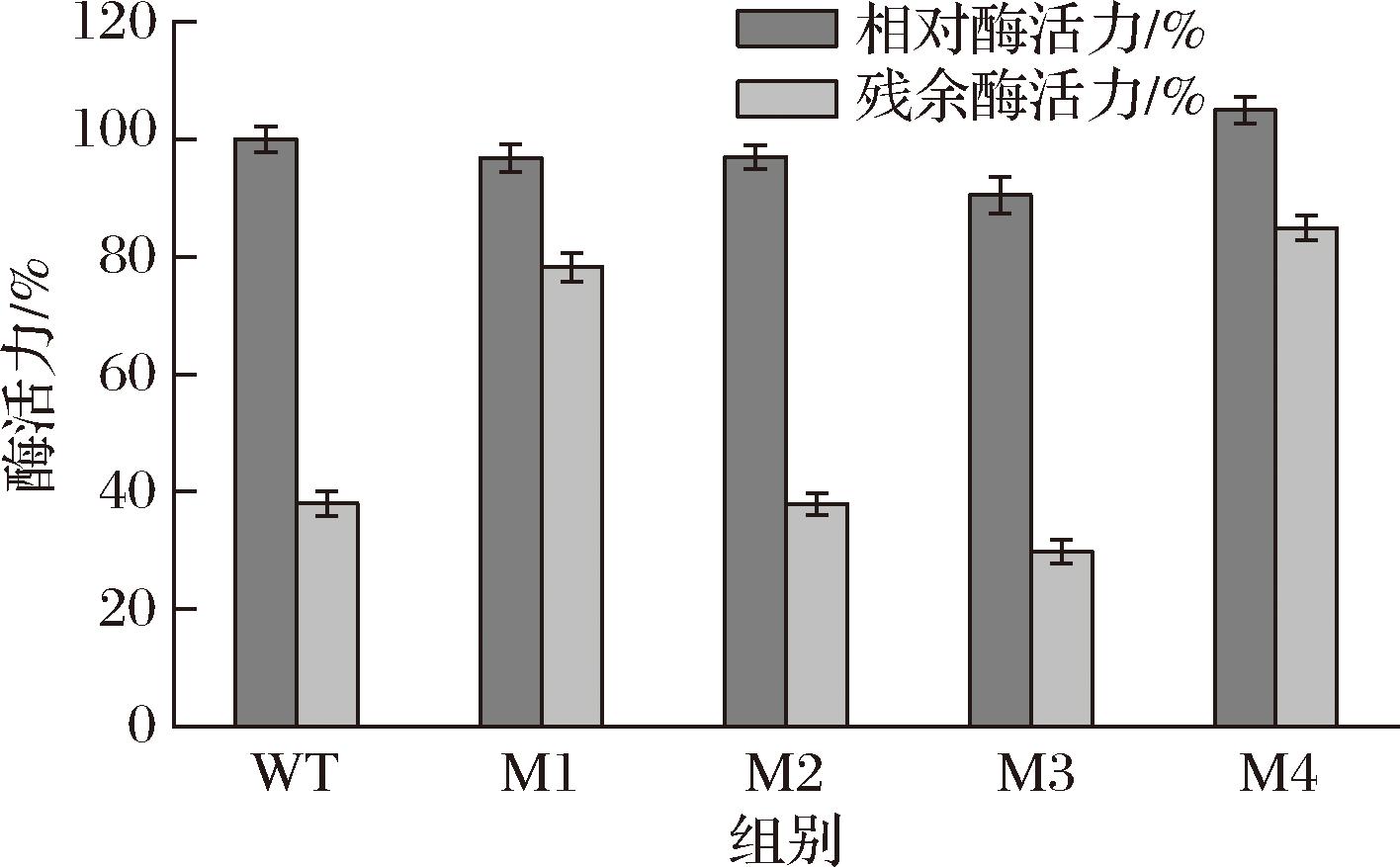
图5 组合突变
Fig.5 Combination mutation
2.4 最佳突变体酶学性质解析
对最优突变体M4进行了酶学性质解析。结果如图6所示,发现WT的最适温度为37 ℃,突变体M4的最适温度为45 ℃,最适温度提高了8 ℃。WT的最适pH值为8.0,突变体M4的最适pH值为7.5,最适pH向中性移动0.5个单位。M4在45 ℃条件下的热稳定性远高于WT。随后计算了WT和突变体M4在45 ℃条件下的t1/2,结果如图6-d所示,WT的t1/2仅有24 min,M4的t1/2达到214.43 min,是WT的8.9倍。
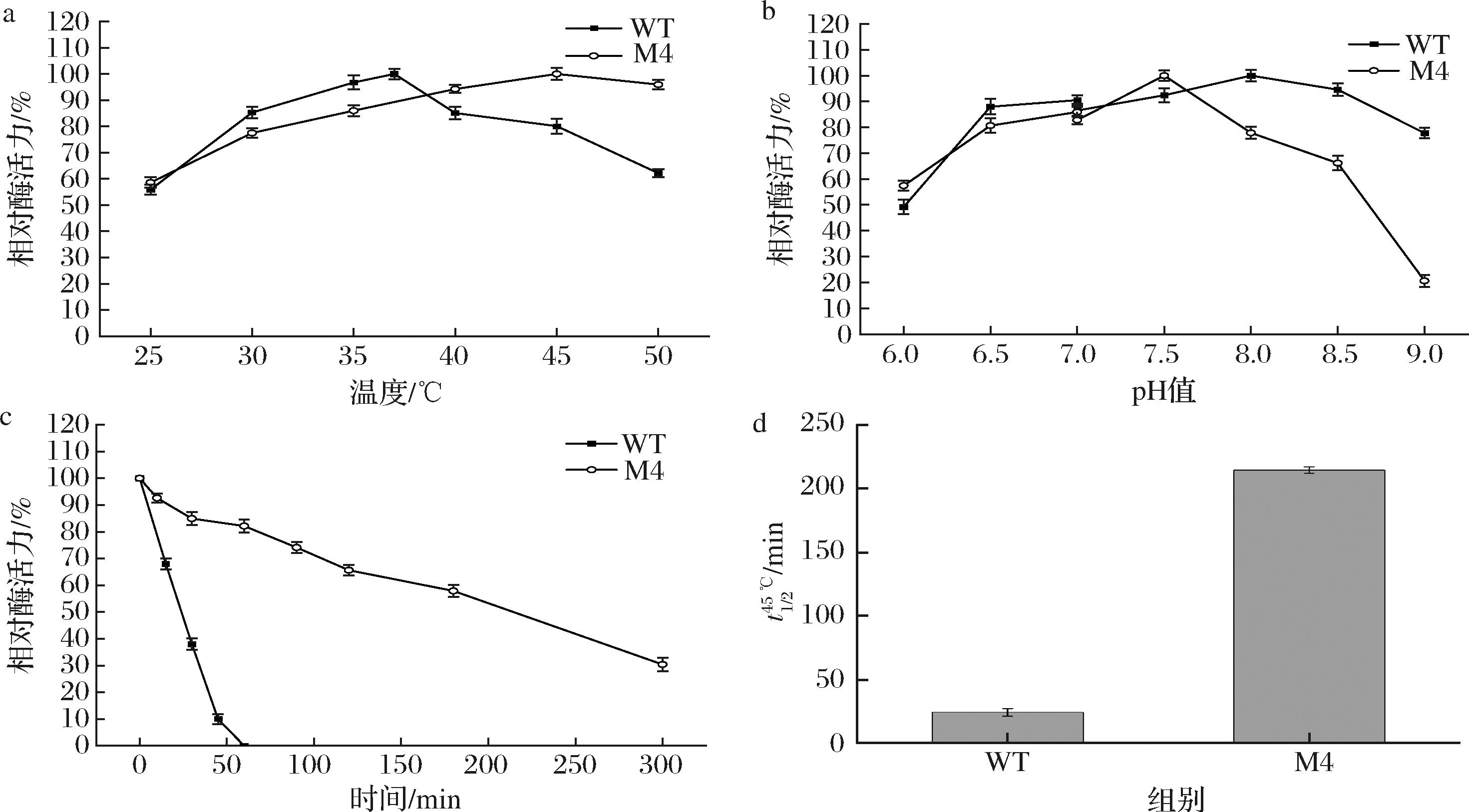
a-最适温度;b-最适pH值;c-45 ℃下的温度稳定性;d-45 ℃下WT和突变体M4的t1/2
图6 酶学性质解析
Fig.6 Enzymatic property analysis
2.5 突变体M4热稳定性增强的分子机制
当第73位的Thr突变成Leu时,虽然其与第69位的Val之间形成的氢键数目减少(图7-a、图7-b),但是蛋白质的热稳定性提升的原因可能是由于新引入的Leu增强了蛋白质内部的疏水相互作用,使得疏水基团聚集在一起,从而避开周围的水分子,形成一个稳定的疏水核心。
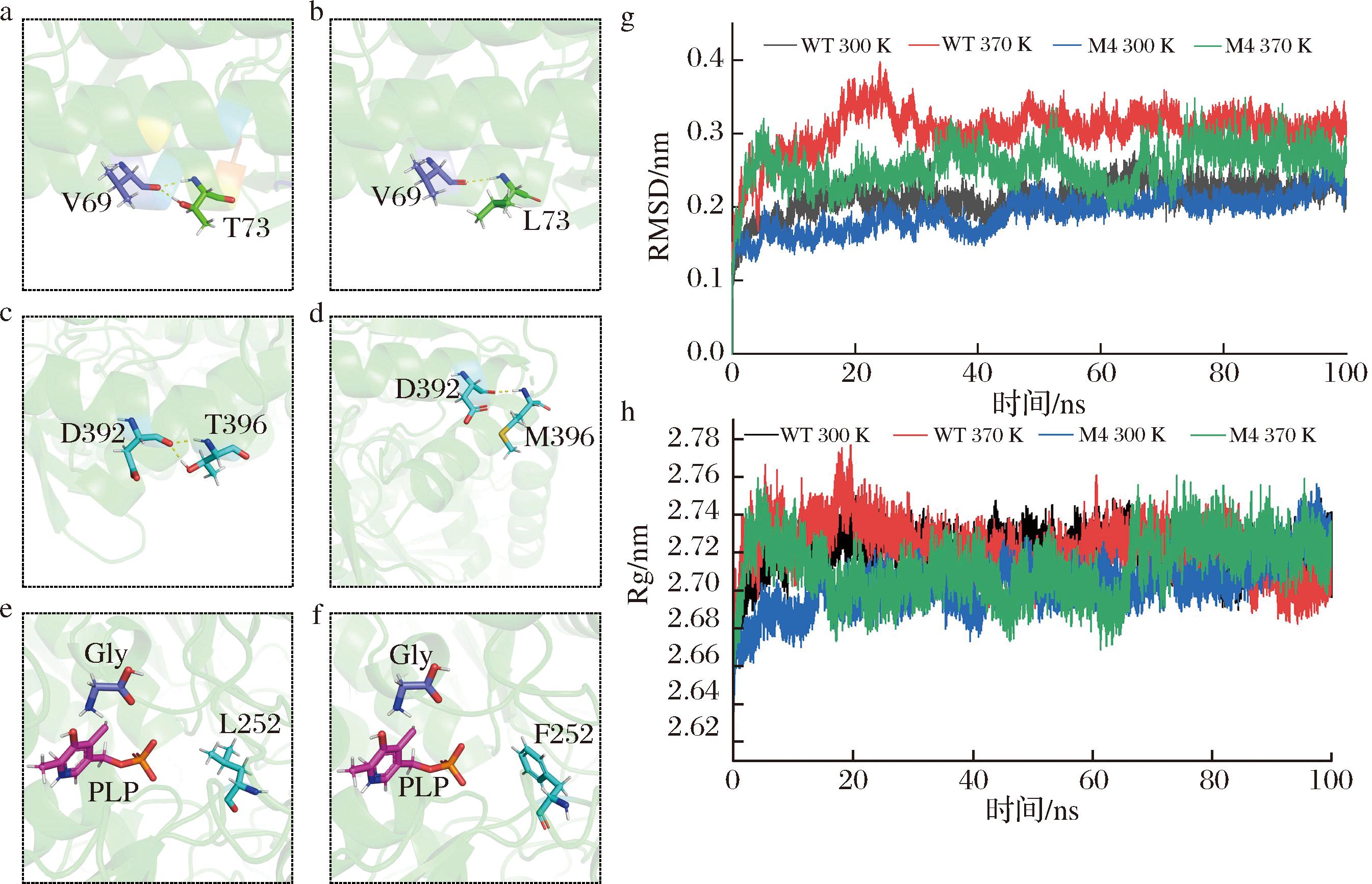
a-WT中T73和V69的相互作用;b-突变体M4中L73和V69的相互作用;c-WT中T396和D392的相互作用; d-突变体M4中M396和D392的相互作用;e-WT中L252残基的相互作用;f-突变体M4中F252位相互作用; g-WT和突变体M4的RMSD值变化;h-WT和突变体M4的Rg值变化
图7 突变体M4热稳定性增强的机制及结构分析
Fig.7 Mechanism and structural analysis of the enhanced thermal stability of mutant M4
如图7-c、图7-d所示,当第396位的Thr突变为Met时,其于D392之间形成的氢键数目减少。但是蛋白稳定性仍然提高的原因可能是第396位的Thr的羟基基团会占据一定空间,可能会引发不合适的空间位阻,致使局部结构无法达成最稳定状态。当其突变为Met时,Met的侧链柔性更佳、空间占位相对更合理,能够优化局部的空间排布,减少不利的空间阻碍,帮助蛋白质达成能量更低、在热环境下更耐受的构象。
如图7-e、图7-f所示,发现L252位于底物PLP和Gly结合口袋附近。当第252位的亮氨酸Leu突变成Phe后,由于Phe含有苯环疏水性更强。在底物结合口袋区域,更强的疏水性能够促进酶与底物紧密结合,构建更稳固的疏水相互作用。这进一步促进酶与底物复合物的形成,从而提升酶活力。
RMSD用于判断在模拟过程中蛋白质是否达到了稳定状态。由图7-g可以看出,在300 K和在370 K模拟条件下,突变体M4的RMSD值均普遍低于WT且更早趋于平衡。这一结果说明,突变体M4较WT的结构更加稳定。
回转半径(radius of gyration,Rg)用于评判在MD模拟过程中蛋白质结构的紧密程度。由图7-h所示,在300 K模拟条件下,野生型的Rg的平均值为2.72 nm,突变体M4的Rg为2.70 nm;在370 K模拟条件下,WT的Rg为2.72 nm,突变体M4的Rg为2.71 nm。发现突变体的Rg值较WT更低,表明M4具有更紧密和热稳定的结构。
本研究获得的组合突变体M4,在45 ℃热处理t1/2达到214.43 min,是WT的8.9倍。先前TAN等[25]对ALAS进行改造后,其突变体H15K、H29R在37 ℃孵育1 h后残余酶活力仅为27%和20%。DU等[26]对荚膜红杆菌(Rhodobacter capsulatus)来源的RcALAS改造获得的组合突变体T6在37 ℃孵育1 h残余活性保持在80.3%。本研究获得的突变体M4的热稳定性较TAN等[25]和DU等[26]获得的突变体具有显著提升,该结果对ALAS的分子改造提供了参考依据。
3 结论
本研究对沼泽红假单胞菌来源的RpALAS进行分子改造,通过Alphafold、Schrödinger软件进行建模和分子对接,Fireprot网站设计获得了2个残余酶活力分别是WT的1.97和1.46倍的突变体T73L和T396M。利用HotSpot Wizard对RpALAS酶活力进行改造,获得酶活力相较于野生型提升20%的突变体L252F。组合突变获得了热稳定显著提升的最优突变体M4,其比酶活力较WT有5%的提升,在45 ℃热处理t1/2达到214.43 min,是WT的8.9倍。这一成果进一步扩充了ALAS突变体酶库同时为后续深入探究ALAS热稳定性和其结构之间的内在关系具有明显优势,为该领域的研究奠定了坚实基础。
[1] 王丽君. 代谢改造重组谷氨酸棒杆菌C4途径高效合成5-氨基乙酰丙酸[D].无锡:江南大学, 2021.WANG L J, Metabolic engineering of C4 pathway in Corynebacterium glutamicum for efficient synthesis of 5-aminolevulinic acid[D].Wuxi:Jiangnan University, 2021.
[2] YI Y C, SHIH I T, YU T H, et al.Challenges and opportunities of bioprocessing 5-aminolevulinic acid using genetic and metabolic engineering:A critical review[J].Bioresources and Bioprocessing, 2021, 8(1):100.
[3] JIANG M R, HONG K Q, MAO Y F, et al.Natural 5-aminolevulinic acid:Sources, biosynthesis, detection and applications[J].Frontiers in Bioengineering and Biotechnology, 2022, 10:841443.
[4] KANG Z, ZHANG J L, ZHOU J W, et al.Recent advances in microbial production of δ-aminolevulinic acid and vitamin B12[J].Biotechnology Advances, 2012, 30(6):1533-1542.
[5] 蒲伟, 陈久洲, 孙村民, 等.琥珀酸脱氢酶或琥珀酰辅酶A合成酶缺失促进大肠杆菌积累5-氨基乙酰丙酸[J].生物工程学报, 2013, 29(10):1494-1503.PU W, CHEN J Z, SUN C M, et al.Deficiency of succinic dehydrogenase or succinyl-CoA synthetase enhances the production of 5-aminolevulinic acid in recombinant Escherichia coli[J].Chinese Journal of Biotechnology, 2013, 29(10):1494-1503.
[6] INOUE K.5-Aminolevulinic acid-mediated photodynamic therapy for bladder cancer[J].International Journal of Urology, 2017, 24(2):97-101.
[7] SWAPNA L A, ALAWAD A O, ABDULLAH ALAMRI L, et al.Efficacy of 5-aminolevulinic acid-mediated photodynamic therapy in patients with nicotine stomatitis[J].Photodiagnosis and Photodynamic Therapy, 2023, 41:103152.
[8] WANG P R, ZHANG G L, ZHANG L L, et al.5-Aminolevulinic acid photodynamic therapy for early-stage lip squamous cell carcinoma[J].Photodiagnosis and Photodynamic Therapy, 2021, 35:102321.
[9] BAO N J, GU T S, ZENG J, et al.Combined therapy of 5-aminolevulinic acid photodynamic therapy and intense pulsed light for Rosacea[J].Lasers in Medical Science, 2022, 38(1):17.
[10] XU L, ZHANG W F, ALI B, et al.Synergism of herbicide toxicity by 5-aminolevulinic acid is related to physiological and ultra-structural disorders in crickweed (Malachium aquaticum L.)[J].Pesticide Biochemistry and Physiology, 2015, 125:53-61.
[11] RHAMAN M S, IMRAN S, KARIM M M, et al.5-aminolevulinic acid-mediated plant adaptive responses to abiotic stress[J].Plant Cell Reports, 2021, 40(8):1451-1469.
[12] LI Y G, ZHANG L Z, YUAN Z Y, et al.MdWRKY71 as a positive regulator involved in 5-aminolevulinic acid-induced salt tolerance in apple[J].Horticultural Plant Journal, 2025,11(4):1397-1413.
[13] LIU J, SHI L, ZHANG L L, et al.Acute acne flare following isotretinoin administration successfully treated by 5-aminolevulinic acid photodynamic therapy[J].Photodiagnosis and Photodynamic Therapy, 2022, 39:102893.
[14] PFALTZ A, ANWAR S.Synthesis of α-aminoketones via selective reduction of acyl cyanides[J].Tetrahedron Letters, 1984, 25(28):2977-2980.
[15] SASAKI K, WATANABE M, TANAKA T, et al.Biosynthesis, biotechnological production and applications of 5-aminolevulinic acid[J].Applied Microbiology and Biotechnology, 2002, 58(1):23-29.
[16] GE F L, LI X K, GE Q R, et al.Modular control of multiple pathways of Corynebacterium glutamicum for 5-aminolevulinic acid production[J].AMB Express, 2021, 11(1):179.
[17] ZHANG J L, WENG H J, ZHOU Z X, et al.Engineering of multiple modular pathways for high-yield production of 5-aminolevulinic acid in Escherichia coli[J].Bioresource Technology, 2019, 274:353-360.
[18] LUO Z S, PAN F, ZHU Y F, et al.Synergistic improvement of 5-aminolevulinic acid production with synthetic scaffolds and system pathway engineering[J].ACS Synthetic Biology, 2022, 11(8):2766-2778.
[19] WANG W Q, XIANG Y L, YIN G B, et al.Construction of 5-aminolevulinic acid microbial cell factories through identification of novel synthases and metabolic pathway screens and transporters[J].Journal of Agricultural and Food Chemistry, 2024, 72(14):8006-8017.
[20] ZHOU H M, ZHANG C Y, LI Z L, et al.Systematic development of a highly efficient cell factory for 5-aminolevulinic acid production[J].Trends in Biotechnology, 2024, 42(11):1479-1502.
[21] PU W, CHEN J Z, ZHOU Y Y, et al.Systems metabolic engineering of Escherichia coli for hyper-production of 5-aminolevulinic acid[J].Biotechnology for Biofuels and Bioproducts, 2023, 16(1):31.
[22] 孟庆龙. 热稳定5-氨基乙酰丙酸合成酶的筛选、表达、酶学性质研究及其应用[D].天津:天津大学, 2016.MENG Q L.Screening, expression, characterization and application of thermostable 5-aminolevulinic acid synthase[D].Tianjin:Tianjin University, 2016.
[23] CHOI H P, HONG J W, RHEE K H, et al.Cloning, expression, and characterization of 5-aminolevulinic acid synthase from Rhodopseudomonas palustris KUGB306[J].FEMS Microbiology Letters, 2004, 236(2):175-181.
[24] WANG Q, JIA M J, LI H J, et al.Design of a genetically encoded biosensor for high-throughput screening and engineering 5-aminolevulinic acid hyper-producing Escherichia coli[J].ACS Sustainable Chemistry &Engineering, 2024, 12(12):4846-4857.
[25] TAN Z J, ZHAO J, CHEN J Z, et al.Enhancing thermostability and removing hemin inhibition of Rhodopseudomonas palustris 5-aminolevulinic acid synthase by computer-aided rational design[J].Biotechnology Letters, 2019, 41(1):181-191.
[26] DU S, ZHENG N, ZHANG Z H, et al.Rational design engineering of 5-aminolevulinate synthase with activity and stability enhancement[J].Journal of Agricultural and Food Chemistry, 2025, 73(3):1892-1901.