海藻糖是由2个吡喃型葡萄糖通过α-1,1糖苷键连接而成的非还原性二糖[1],在动植物和微生物体内作为储能物质和保护生物大分子起重要作用[2-3]。随着生物技术的不断发展,海藻糖被广泛应用于食品、化妆品以及医药行业,因此需要大规模的工业化制备[4-7]。海藻糖的制备方法主要分为生物提取法、微生物发酵法和酶转化法。然而,生物提取法效率低且成本高,微生物发酵法会产生大量难以分离的副产物[8]。相反,酶转化法可以利用酶专一、高效的特性,迅速地将底物转化为海藻糖,具有操作简便、反应迅速和转化率高等优点[9-10]。在酶转化法中,单酶法是通过使用海藻糖合酶催化麦芽糖的糖苷键断裂和重排获得海藻糖,但该过程的可逆性对最终的转化率有不良影响,转化率仅有60%~70%[11-13]。
双酶法制备海藻糖是利用麦芽寡糖基海藻糖合酶(maltooligosyltrehalose synthase, MTSase, 由TreY基因编码)和麦芽寡糖基海藻糖水解酶(maltooligosyltrehalose trehalohydrolase, MTHase, TreZ基因编码)催化麦芽糊精获得,转化率为60%~82%。麦芽寡糖基海藻糖合成酶MTSase首先催化断裂麦芽糊精中还原端的葡萄糖基与相邻葡萄糖基的α-1,4糖苷键,释放最末端的葡萄糖基。该葡萄糖基以α-1,1糖苷键重新连接,生成中间产物麦芽寡糖基海藻糖,随后被麦芽寡糖基海藻糖水解酶MTHase断裂麦芽寡糖基和海藻糖基之间的α-1,4糖苷键,生成产物海藻糖(图1-a)[14]。KATO等[15]应用硫磺矿硫化叶菌(Sulfolobus solfataricus) KM1来源的MTSase和MTHase进行海藻糖双酶法转化,能以平均链长17的淀粉为底物获得83%的转化率,但酶的最适温度达到70 ℃以上,增加了能耗和设备成本。另外,MARUTA等[9]利用来源于节杆菌层(Arthrobacter sp.) Q36的MTSase和MTHase催化直链淀粉,海藻糖转化率达到73%,但是直链淀粉相较糊精价格昂贵,转化周期长,从而使其难以应用于大规模生产。最近的研究表明,4-α葡糖基转移酶(4-α-glucanotransferase, 4αGT, 由MalQ基因编码)通过歧化反应可将裂解的葡聚糖重新转移至短链麦芽寡糖[16],继续被MTSase和MTHase催化(图1-b)。因此,添加4αGT构建三酶催化体系可以进一步提高底物的利用率和海藻糖转化率。此外,这些酶在大肠杆菌中常以包涵体的形式存在[17-18],需要进一步优化以提高可溶性表达水平,从而降低酶制剂生产的成本。
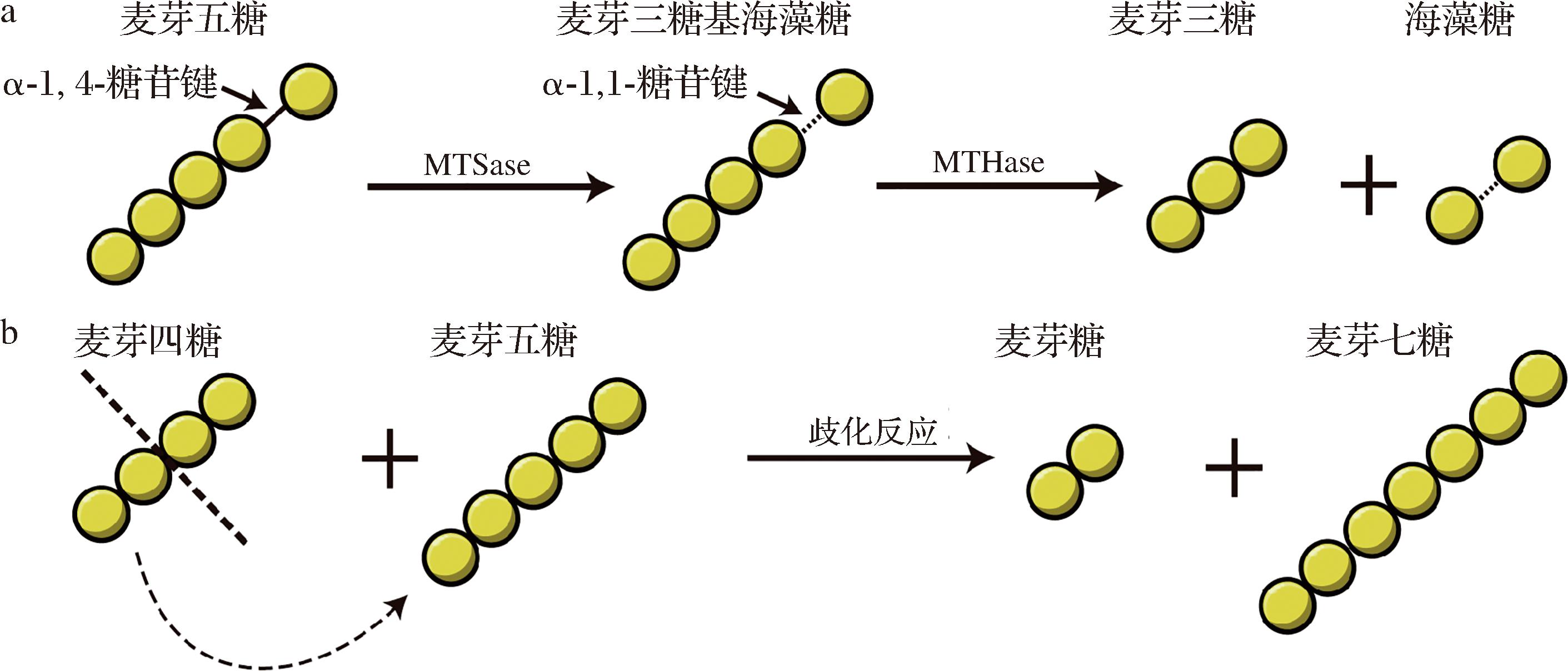
a-MTSase和MTHase催化机制示意图;b-4αGT歧化反应催化机制示意图
图1 MTSase,MTHase和4αGT的反应机制
Fig.1 Reaction mechanism of MTSase, MTHase, and 4αGT
本研究将TreY、TreZ和MalQ分别在大肠杆菌TR07中进行克隆表达;随后根据大肠杆菌密码子偏好性和tRNA适应指数等指标对3个基因进行序列优化,并在酶的N端融合多种蛋白标签以提高它们的可溶性表达水平;在此基础上,通过优化诱导剂浓度、诱导OD600值和诱导温度进一步减少包涵体的生成;最后利用高效表达3种酶的重组菌株进行海藻糖转化率的验证。本研究对降低工业海藻糖制备成本具有重要参考价值。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
菌株大肠杆菌DH5α用于质粒构建,大肠杆菌TR07用于蛋白表达,表达载体pBAD由本实验室保存,本研究中使用的质粒和菌株见表1。
表1 文中所用菌株和质粒
Table 1 Strains and plasmids used in the study

注:*为序列优化后的基因,G:谷胱甘肽巯基转移酶(glutathione S- transferase,GST);S:小泛素样修饰蛋白(small ubiquitin-related modifier,SUMO);M:麦芽糖结合蛋白(maltose binding protein,MBP);N:大肠杆菌转录延长的抗终止因子蛋白(N-utilization substance A,NusA);P:蛋白质二硫键异构酶(protein disulfide isomerase,PDI);T:硫氧还蛋白(thioredoxins,TrxA)。
菌株和质粒 特征来源pBAD大肠杆菌低拷贝质粒,(包含启动子pBAD,卡那霉素抗性)本实验室保藏pBAD-TreYpBAD衍生质粒,大肠杆菌低拷贝质粒,(包含启动子pBAD)本研究pBAD-TreZpBAD衍生质粒,大肠杆菌低拷贝质粒,(包含启动子pBAD)本研究pBAD-MalQpBAD衍生质粒,大肠杆菌低拷贝质粒,(包含启动子pBAD)本研究pBAD-TreY∗pBAD-TreY衍生质粒,(包含启动子pBAD,MTSase经过序列优化)本研究pBAD-TreZ∗pBAD-TreZ衍生质粒,(包含启动子pBAD,MTHase经过序列优化)本研究pBAD-MalQ∗pBAD-MalQ衍生质粒,(包含启动子pBAD,4αGT经过序列优化)本研究pBAD-TreY∗GpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签GST)本研究pBAD-TreY∗SpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签SUMO)本研究pBAD-TreY∗MpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签MBP)本研究pBAD-TreY∗NpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签NusA)本研究pBAD-TreY∗PpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签PDI)本研究质粒pBAD-TreY∗TpBAD-TreY∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签TrxA)本研究pBAD-TreZ∗GpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签GST)本研究pBAD-TreZ∗SpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签SUMO)本研究pBAD-TreZ∗MpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签MBP)本研究pBAD-TreZ∗NpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签NusA)本研究pBAD-TreZ∗PpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签PDI)本研究pBAD-TreZ∗TpBAD-TreZ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签TrxA)本研究pBAD-MalQ∗GpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签GST)本研究pBAD-MalQ∗SpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签SUMO)本研究pBAD-MalQ∗MpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签MBP)本研究pBAD-MalQ∗NpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签NusA)本研究pBAD-MalQ∗PpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签PDI)本研究pBAD-MalQ∗TpBAD-MalQ∗衍生质粒,(包含启动子pBAD,N端融合蛋白标签TrxA)本研究大肠杆菌DH5α大肠杆菌感受态本实验室保藏大肠杆菌TR07大肠杆菌感受态本实验室保藏TR07-1大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD- TreY质粒本研究TR07-2大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD- TreZ质粒本研究TR07-3大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD- MalQ质粒本研究TR07-1∗大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗质粒本研究TR07-2∗大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗质粒本研究TR07-3∗大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗质粒本研究TR07-1∗G大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-G质粒本研究TR07-1∗S大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-S质粒本研究TR07-1∗M大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-M质粒本研究TR07-1∗N大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-N质粒本研究菌株TR07-1∗P大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-P质粒本研究TR07-1∗T大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreY∗-T质粒本研究TR07-2∗G大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-G质粒本研究TR07-2∗S大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-S质粒本研究TR07-2∗M大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-M质粒本研究TR07-2∗N大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-N质粒本研究TR07-2∗P大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-P质粒本研究TR07-2∗T大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-TreZ∗-T质粒本研究TR07-3∗G大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-G质粒本研究TR07-3∗S大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-S质粒本研究TR07-3∗M大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-M质粒本研究TR07-3∗N大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-N质粒本研究TR07-3∗P大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-P质粒本研究TR07-3∗T大肠杆菌TR07衍生菌株,大肠杆菌TR07中转化pBAD-MalQ∗-T质粒本研究
1.1.2 主要试剂
普鲁兰酶、糖化酶、麦芽六糖、麦芽四糖和麦芽三糖样品,上海麦克林生化科技股份有限公司;大肠杆菌感受态制备试剂盒、质粒提取试剂盒、卡那霉素以及其余实验中所有的化学品,生工生物工程(上海)股份有限公司;用于聚合酶链式反应(polymerase chain reaction,PCR)的Max DNA聚合酶和核酸分子质量标准液,宝生物医学科技(北京);无缝克隆试剂盒,上海碧云天生物科技有限公司;蛋白分子质量Marker和蛋白预制胶,翌圣生物科技(上海)股份有限公司;葡萄糖检测试剂盒,北京雷根生物技术有限公司。
1.1.3 培养基及缓冲液
卡那霉素LB液体培养基(g/L):酵母粉5,蛋白胨10,氯化钠10。卡那霉素终质量浓度为50 mg/L。
卡那霉素LB固体培养基(g/L):酵母粉5,蛋白胨10,氯化钠10,琼脂15。卡那霉素终质量浓度为50 mg/L。
自诱导发酵培养基(g/L):酵母粉5,蛋白胨10,葡萄糖25,甘油10,L-阿拉伯糖20,卡那霉素终质量浓度为50 mg/L。
PBS缓冲液(g/L):二水合磷酸二氢钠31.202,无水磷酸氢二钠28.392,氯化钠29.22,pH 6.0。
PB缓冲液(g/L):二水合磷酸二氢钠156,无水磷酸氢二钠142,pH 6.0。
1.2 实验方法
1.2.1 引物设计
根据引物设计的基本原则,使用软件SnapGene 6.0.2构建如表2所示的引物。
表2 文中所用引物
Table 2 Primers used in the study

引物名称序列(5′-3′)作用103 mb-FAACCTAAGAATTCGGTGAGCTCGGTCTG扩增质粒骨架构建质粒pBAD-TreY103 mb-RGCGGGCATGGTACCCATGGTTAATTCCTCCTG103-FTAACCATGGGTACCATGCCCGCTTCAACATATAGGC 扩增TreY基因构建质粒pBAD-TreY103-RGCTCACCGAATTCTTAGGTTTCAACGAGTAGCGCAAC203 mb-FGCTTGGAGTAAGAATTCGGTGAGCTCGGTCTG扩增质粒骨架构建质粒pBAD-TreZ203 mb-RAAACCTTCTATTCATGGTACCCATGGTTAATTCCTCCTG203-FGGTACCATGAATAGAAGGTTTCCAGTATGGGC扩增TreZ基因构建质粒pBAD-TreZ203-RCACCGAATTCTTACTCCAAGCGAACAATCGCAG403 mb-FACCGATTAAGAATTCGGTGAGCTCGGTCT扩增质粒骨架构建质粒pBAD-MalQ403 mb-RTTATCTAACATGGTACCCATGGTTAATTCCTCCTG403-FATGGGTACCATGTTAGATAAAAGGTGTTCAGGAATACTATTGC扩增MalQ基因构建质粒pBAD-MalQ403-RTCACCGAATTCTTAATCGGTGTGGTAGATACGACGAC103op-FTAACCATGGGTACCATGCCCGCTTCAACATATAGGC扩增TreY∗基因构建质粒pBAD-TreY∗103op-RGCTCACCGAATTCTTAGGTTTCAACGAGTAGCGCAAC203op-FGGTACCATGAATAGAAGGTTTCCAGTATGGGC扩增TreZ∗基因构建质粒pBAD-TreZ∗203op-RCACCGAATTCTTACTCCAAGCGAACAATCGCAG403op-FATGGGTACCATGTTAGATAAAAGGTGTTCAGGAATACTATTGC扩增MalQ∗基因构建质粒pBAD-MalQ∗403op-RTCACCGAATTCTTAATCGGTGTGGTAGATACGACGACGST-FCATGGGTACCATGAGCCCGATTCTGGGC扩增GST标签构建质粒pBAD-TreZ∗GGST-RAATCGGGCTCATGGTACCCATGGTTAATTCCTCCTGSUMO-FCATGGGTACCATGAGCGATAGCGAAGTGAACC扩增SUMO标签构建质粒pBAD-TreZ∗SSUMO-RTGTTGAAGCGGGCATAGAACCACCACCACCAGAACMBP-FGGGTACCATGGCCGAAGAAGGTAAACTGG 扩增MBP标签构建质粒pBAD-TreZ∗MMBP-RCCTTCTTCGGCCATGGTACCCATGGTTAATTCCTCCTGNusA-FATGGGTACCATGAACAAAGAAATTTTGGCTGTAGTTGA 扩增NusA标签构建质粒pBAD-TreZ∗NNusA-RTCTTTGTTCATGGTACCCATGGTTAATTCCTCCTGPDI-FATGGGTACCATGCTGCGCCGCGCGC 扩增PDI标签构建质粒pBAD-TreZ∗PPDI-RCGGCGCAGCATGGTACCCATGGTTAATTCCTCCTGTrxA-FCATGGGTACCATGAGCGATAAAATTATTCACCTGACTGA扩增TrxA标签构建质粒pBAD-TreZ∗TTrxA-RTTTATCGCTCATGGTACCCATGGTTAATTCCTCCTG103op-mb-FTGGTGGTTCTATGCCCGCTTCAACATATAGGC扩增质粒骨架构建质粒pBAD-TreY∗G103op-mb-RCATGGTACCCATGGTTAATTCCTCCTG203op-mb-FTGGTTCTATGAATAGAAGGTTTCCAGTATGGGC扩增质粒骨架构建质粒pBAD-TreZ∗G203op-mb-RCATGGTACCCATGGTTAATTCCTCCTG403op-mb-FGTGGTGGTTCTATGTTAGATAAAAGGTGTTCAGGAATACTATTGC扩增质粒骨架构建质粒pBAD-MalQ∗G403op-mb-RCATGGTACCCATGGTTAATTCCTCCTG
1.2.2 序列优化
从NCBI下载TreY、TreZ和MalQ基因序列。综合多种工具分析优化基因序列,先通过Genscript(https://www.genscript.com.cn/gensmart-free-gene-codon-optimization.html)网络在线工具根据宿主密码子偏好性进行序列优化,接着使用Novopro(https://www.novopro.cn/tools/codon-optimization.html)提供的网络在线工具针对5′端序列、mRNA的二级结构和序列的GC含量进行优化,随后利用IDTDNA(https://www.idtdna.com/pages/tools/codon-optimization-tool)工具对tRNA适应性指数和密码子重复率进行优化,将优化后的基因命名为TreY*、TreZ*和MalQ*,最后将TreY、TreZ、MalQ、TreY*、TreZ*和MalQ*基因序列送至南京金斯瑞生物科技有限公司合成。
1.2.3 质粒的构建
以质粒pBAD为模板,以103 mb-F和103 mb-R为引物进行PCR扩增获得线性质粒pBAD;以合成的基因TreY为模板,103-F和103-R为引物进行PCR扩增获得基因片段TreY,PCR体系为模板1 μL、上游引物1 μL、下游引物1 μL、超纯水7 μL、Prime STAR Max DNA聚合酶10 μL。PCR条件:98 ℃预变性5 min,98 ℃变性10 s,60 ℃退火15 s,72 ℃延伸90 s,36个循环,72 ℃延伸5 min。将PCR产物使用无缝克隆试剂盒进行连接,反应液体系为:1 μL TreY片段,1 μL线性质粒载体pBAD,10 μL无缝克隆混合液,8 μL超纯水。反应液于50 ℃水浴15 min后,取5 μL反应液转化入大肠杆菌DH5α感受态中,待菌落生长起来后挑取转化子于液体培养基培养8 h后送测序。将测序正确的转化子命名为pBAD-TreY,按照相同的方法构建表达载体pBAD-TreZ、pBAD-MalQ、pBAD-TreY*,pBAD-TreZ*和pBAD-MalQ*(表1)。
以质粒pBAD-TreY*为模板,103op-mb-F和103op-mb-R为引物进行PCR扩增获得线性质粒pBAD-TreY*;以质粒pBAD-GST为模板,GST-F和GST-R为引物进行PCR扩增获得基因片段GST,PCR体系为:模板1 μL,上游引物1 μL,下游引物1 μL,超纯水7 μL,Prime STAR Max DNA聚合酶10 μL。PCR条件:98 ℃预变性5 min,98 ℃变性10 s,60 ℃退火15 s,72 ℃延伸90 s,36个循环,72 ℃延伸5 min。将PCR产物使用无缝克隆试剂盒进行连接,反应液体系为:1 μL GST片段,1 μL线性质粒载体pBAD-TreY*,10 μL无缝克隆混合液,8 μL ddH2O。反应液于50 ℃水浴15 min后,取5 μL反应液转化入大肠杆菌DH5α感受态中,待菌落生长起来后挑取转化子于液体培养基培养8 h后送测序。将测序正确的转化子分别命名为pBAD-TreY*-G,按照相同的方法构建其他携带促融标签的表达载体(表1)。
1.2.4 重组菌的构建与发酵
提取质粒pBAD-TreY并转化进大肠杆菌TR07中获得重组表达菌株TR07-1,按照相同的方法构建剩余菌株(表1),测序验证后将菌种保藏至-80 ℃冰箱中。将活化的菌株划线接种至卡那霉素LB固体培养基上,在37 ℃培养箱中倒置培养直至菌落生长至合适大小,随后挑取单菌落接种于卡那霉素LB液体培养基中,在30 ℃、220 r/min条件下培养8 h制备发酵种子液,随后将培养完成后的500 μL种子液接种至50 mL自诱导发酵培养基中,继续在相同培养条件下培养24 h。
1.2.5 重组菌细胞破壁和SDS-PAGE电泳分析
培养结束后的菌液使用高速离心机8 000 r/min离心10 min收集细胞,先用50 mL无菌水彻底吹打重悬细胞,之后再次8 000 r/min离心10 min收集细胞,再用50 mL PBS缓冲液彻底吹打重悬细胞。重悬后的细胞利用高压匀浆细胞破碎仪进行细胞破碎,破碎液利用高速离心机10 000 r/min离心30 min获得上清液即粗酶液和沉淀,沉淀中加入50 mL PBS缓冲液并彻底吹打悬浮混匀获得沉淀液。SDS-PAGE分析上清液和沉淀中蛋白表达情况,将30 μL粗酶液和沉淀液与10 μL十二烷基硫酸锂缓冲液混合后于99 ℃热变性10 min作为样品液,使用预制蛋白胶,配合Bio-Rad电泳槽使用,电泳运行条件为:Tris作为电泳缓冲液,电压150 V,时长30~40 min。电泳完成后蛋白胶置于脱色摇床进行考马斯亮蓝染色30 min,后续使用脱色液进行脱色直到蛋白胶背景清晰,最后使用蛋白凝胶成像仪拍照记录,观察蛋白电泳条带是否与理论值相符。
1.2.6 酶活力的测定
MTSase和MTHase的酶活力测定方法参考SUN等[19]的方法并稍作调整。首先,取10 μL稀释至合适浓度的MTSase酶液和90 μL使用PBS缓冲液溶解的10 g/L的麦芽六糖溶液混合于2 mL离心管内,在52 ℃的水浴锅中反应10 min后,100 ℃沸水浴10 min以终止反应。随后向反应体系中加入400 μL无菌水和500 μL 3,5-二硝基水杨酸(3,5-Dinitrosalicylic acid,DNS)溶液沸水反应7 min,再冰水浴降温3 min,使用分光光度计在波长540 nm下测定其吸光度。酶活力单位定义:在52 ℃,其他条件恒定的条件下,每分钟内每消耗1 mmol麦芽六糖所需的酶量为1 U。相似地,MTHase的酶活力测定方法是取10 μL稀释至合适浓度的MTHase酶液和90 μL使用PBS缓冲液溶解的10 g/L的麦芽四糖基海藻糖混合于2 mL的离心管内,在52 ℃的水浴锅反应10 min后,100 ℃沸水浴10 min终止反应。随后向反应体系中加入400 μL无菌水和500 μL的DNS溶液沸水反应7 min,再冰水浴降温3 min,使用分光光度计在波长540 nm下测定其吸光度。酶活力单位定义:在52 ℃,其他条件恒定的条件下,每分钟内每生成1 mmol麦芽四糖所需的酶量为1 U。
4αGT的酶活力测定方法是根据ROTH等[20]的报道并进行调整。首先,取40 μL稀释至合适浓度的4αGT酶液和360 μL使用PBS缓冲液溶解的10 g/L的麦芽三糖溶液混合于2 mL的离心管内,在52 ℃的水浴锅反应10 min,随后100 ℃沸水浴处理10 min终止反应。使用雷根葡萄糖分析试剂盒测定葡萄糖的生成量。酶活力单位定义:在52 ℃,其他条件恒定的条件下,每分钟内每生成1 μmol葡萄糖所需的酶量为1 U。
1.2.7 融合蛋白诱导表达条件的优化
培养条件采用L9(33)正交表进行优化实验,以摇瓶发酵诱导表达的OD600值、温度和诱导剂的浓度为变量(表3)。诱导OD600值分别为0、0.5、1;诱导温度分别为22、25、30 ℃;诱导剂的质量浓度分别为1、2、4 g/L,从而确定对可溶性表达最佳的发酵条件。
表3 酶表达优化的正交试验
Table 3 Orthogonal experiment for enzyme expression optimization
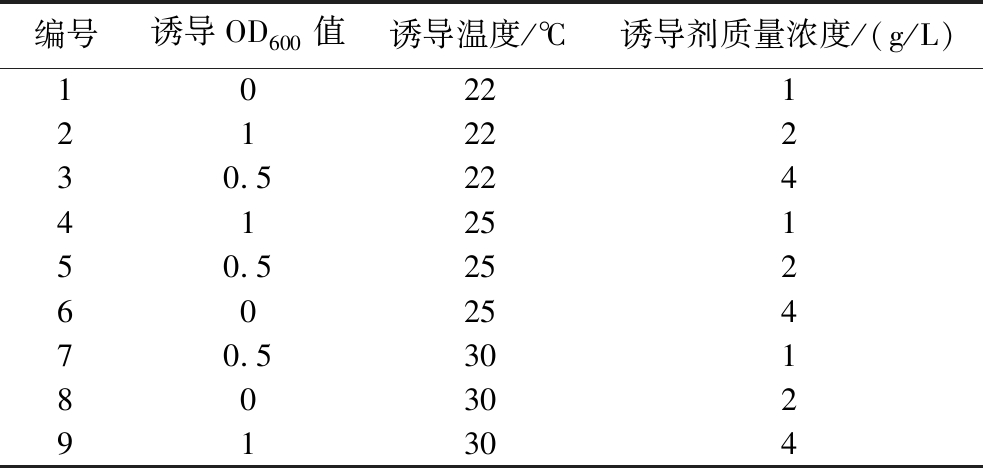
编号诱导OD600值诱导温度/℃诱导剂质量浓度/(g/L)102212122230.52244125150.52526025470.53018030291304
1.2.8 海藻糖的转化和测定
在250 mL摇瓶中加入12 g麦芽糊精和0.5 mL普鲁兰酶,随后分别加入MTSase、MTHase和4αGT粗酶液2 mL,用pH 6.0的PB缓冲液补充反应体系至40 mL,于52 ℃,220 r/min摇床反应24 h,随后向反应体系中加入1 mL糖化酶,继续于52 ℃,220 r/min摇床反应1 h。反应完成后使用0.22 μm滤膜将反应液进行过滤,使用HPLC分析样品。HPLC的具体流程为:Agilent 1260,示差折光检测器,sugar-paktm1色谱柱(Waters,10 μm,6.5 mm×300 mm),流动相:超纯水,流速0.5 mL/min,柱温70 ℃,进样体积为10 μL,每个样品处理13 min。根据海藻糖标品的峰面积计算海藻糖产量,海藻糖转化率的计算如公式(1)所示:
海藻糖转化率![]()
(1)
2 结果与分析
2.1 基因的克隆与表达
首先,构建质粒pBAD-TreY、pBAD-TreZ和pBAD-MalQ(图2-a),以合成的基因TreY、TreZ和MalQ为模板进行PCR获得TreY、TreZ和MalQ的基因片段,经过琼脂糖凝胶电泳验证分别与理论值相符合(图2-b)。随后,与线性质粒pBAD连接以构建质粒,并将它们转化至大肠杆菌TR07感受态中,成功构建重组菌株TR07-1、TR07-2和TR07-3。将重组菌株TR07-1、TR07-2和TR07-3发酵16 h,并对细胞破碎液即粗酶液进行SDS-PAGE分析。结果显示,TR07-1、TR07-2和TR07-3分别在75、60、60 kDa附近有明显的蛋白条带,与三者蛋白理论大小相符合(图2-c)。另外,对TR07-1、TR07-2和TR07-3的酶活力进行测定。其中,TR07-1、TR07-2和TR07-3的酶活力分别为1.46、5.45、1.44 U/mL。这些结果表明,尽管在大肠杆菌中表达的3种酶均有催化活性,但是它们的酶活力均较低,需要进一步提高胞内表达水平。
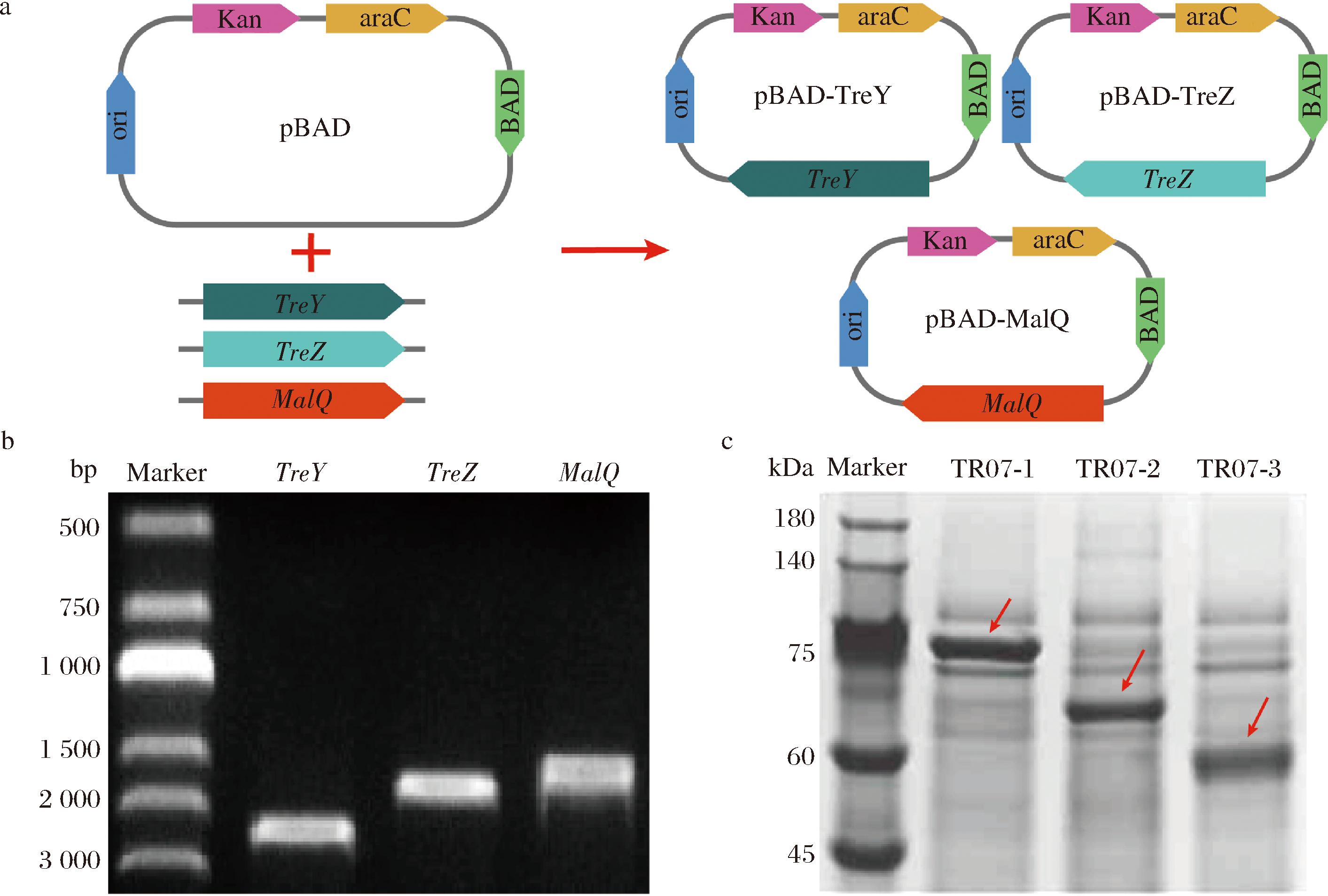
a-质粒构建示意图;b-TreY、TreZ和MalQ的核酸胶图;c-TR07-1、TR07-2和TR07-3的蛋白胶图
图2 TR07-1、TR07-2和TR07-3菌株构建及蛋白表达的SDS-PAGE分析
Fig.2 Construction of TR07-1, TR07-2 and TR07-3 strains and SDS-PAGE analysis of protein expression
2.2 基因序列优化提升表达量
由于TreY、TreZ和MalQ在大肠杆菌中表达时存在密码子偏好性和基因GC含量不合理等问题,因此需要进行序列优化以提升它们的表达水平。根据大肠杆菌密码子偏好性,利用Genscript进行优化并去除可能存在表达限制的密码子;接着使用Novopro针对不合理的mRNA的二级结构和序列GC含量进行优化,并利用IDTDNA优化tRNA适应性指数和密码子重复率(图3-a)。序列优化后,基因TreY*、TreZ*和MalQ*中的GC含量分别由74%、74%和51%优化至61%、61%和54%。其中TreY*和TreZ*基因中的不合理GC含量显著降低。另外,TreY*和TreZ*基因的密码子适应指数提高10%,而MalQ*基因的密码子适应指数提高20%(图3-b)。将3个优化后的基因转入大肠杆菌TR07中获得重组菌株TR07-1*、TR07-2*和TR07-3*,并对它们的发酵上清液和胞内沉淀进行SDS-PAGE分析。如图3-c所示,TR07-1*和TR07-3*蛋白表达量提升明显。另外,酶活力测定结果表明TR07-1*、TR07-2*和TR07-3*发酵上清液中的酶活力分别为2.73、4.93、2.2 U/mL,其中TR07-1*和TR07-3*酶活力相较优化前分别提升了87%和52.8%(图3-c)。这些结果表明基因序列优化一定程度提升了3种酶基因在大肠杆菌中的转录和翻译速率,最终提高了酶的表达量。然而,细胞中存在较多的包涵体(图3-d),这是由于蛋白的错误折叠导致,后续将进一步减少包涵体的生成。

a-序列优化工具的使用流程;b-TreY、TreZ和MalQ序列优化前后的密码子适应指数对比;c-TreY、TreZ和MalQ序列优化前后的酶活力对比; d-TreY、TreZ和MalQ序列优化前后的蛋白胶图
图3 序列优化对酶的表达水平影响
Fig.3 Effect of sequence optimization on enzyme expression levels
注:S为胞内上清液;P为胞内沉淀液。
2.3 蛋白标签融合提高酶的可溶性表达
尽管序列优化提高了蛋白的表达水平,但是重组大肠杆菌的胞内沉淀物中存在一定的包涵体。包涵体形成一般是由于蛋白翻译过程中表达过快或者缺少翻译后修饰导致不正确的折叠,改善包涵体现象的策略通常包括优化诱导表达条件、分子伴侣共表达和促融标签融合表达[21]。在本研究中,首先采用促融标签融合表达的策略。促融蛋白标签不仅能够提高外源蛋白的可溶性水平,还能帮助其正确折叠,能有效降低包涵体的产生。常用的促融标签包括GST、MBP、NusA、PDI、SUMO和TrxA[22]。
6种促融蛋白标签基因分别与TreY*、TreZ*和MalQ*基因融合,构建了18个表达载体(图4-a),随后将这些载体转入大肠杆菌中获得18个重组菌株。发酵结果显示促融标签的融合对菌株的生长有一定影响,其中融合蛋白标签PDI和NusA融合后使菌液的OD600值相较于未融合的分别降低72%和49.6%(图4-c)。分别对发酵上清液和沉淀进行SDS-PAGE分析。重组菌株TR07-1*T、TR07-1*M、TR07-2*N、TR07-3*S和TR07-3*G沉淀中的包涵体现象有一定程度缓解,而TR07-1*S、TR07-1*N、TR07-2*S、TR07-2*T和TR07-3*T的沉淀液中出现更多的包涵体(图4-b、图4-d和图4-f)。另外,发酵上清液中的酶活力测定结果显示,总体而言,连接蛋白标签后的菌株酶活力相较于对照TR07-1*、TR07-2*和TR07-3*的酶活力10、13.01、1.71 U/mL有不同程度的降低。TR07-1*S的酶活力比对照TR07-1*的降低1.1%,而TR07-2*T相较对照TR07-2*减少2.2%(图4-e),这表明蛋白标签的适配性是关键因素。然而,重组菌株TR07-1*S和TR07-2*T中可溶性表达的蛋白和沉淀中包涵体的总量有所提升,因此促进新生肽链的正确折叠有望增加可溶性海藻糖酶的含量。由于诱导温度、诱导时间等会直接影响蛋白折叠,后续将对重组菌株TR07-1*S和TR07-2*T的发酵条件进行优化。同时,融合蛋白标签后TR07-3*的酶活力降低超过40%,表明蛋白标签对4αGT的催化性能产生不良影响,所以选择在未融合促融标签的菌株TR07-3*的基础上进行发酵条件优化。
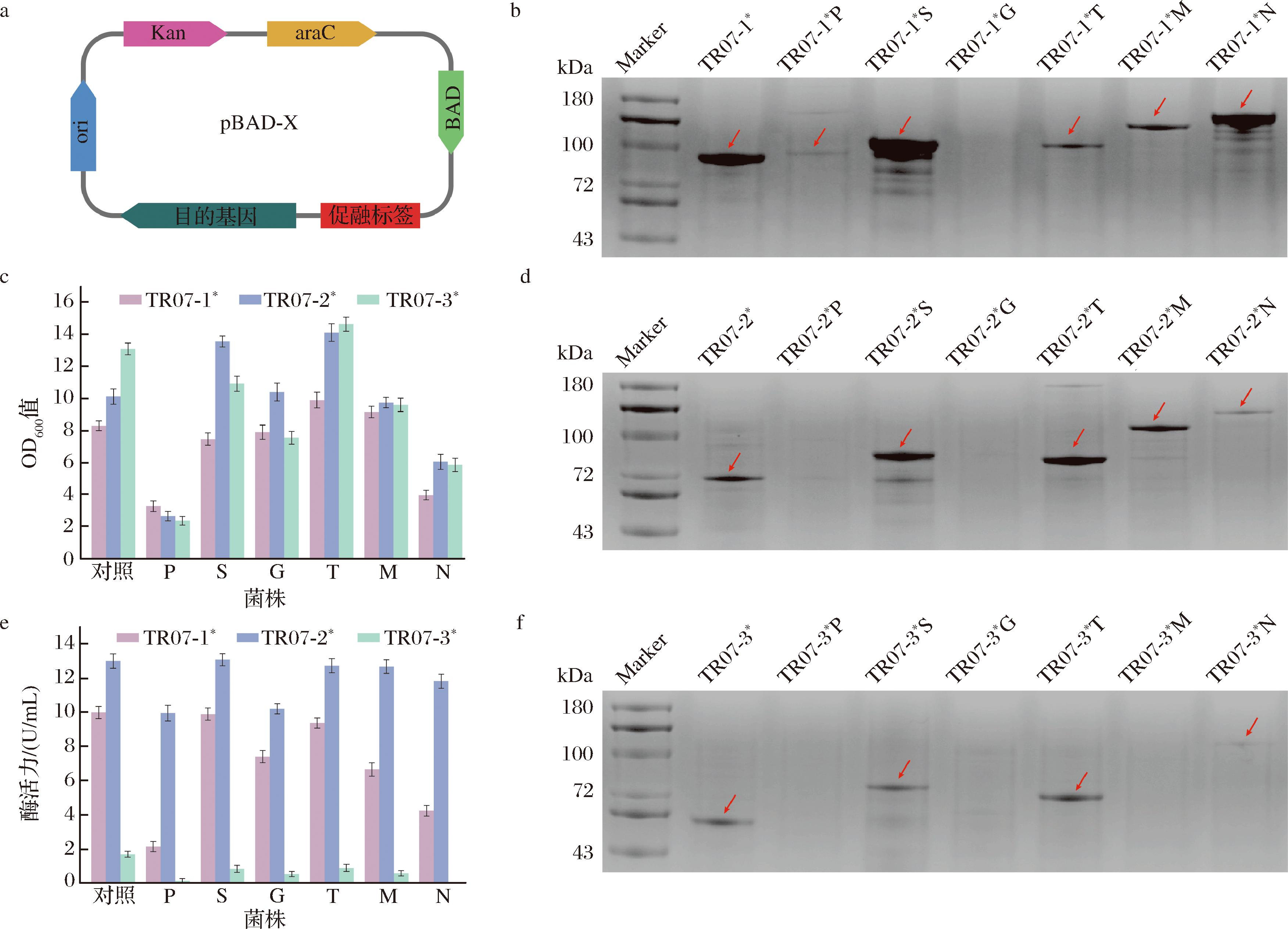
a-融合蛋白质粒示意图;b-促融标签对TR07-1*蛋白表达的影响;c-促融标签对生长的影响;d-促融标签对TR07-2*蛋白表达的影响; e-促融标签对酶活力的影响;f-促融标签对TR07-3*蛋白表达的影响
图4 促融标签对蛋白可溶性表达及菌体生长的影响
Fig.4 Effect of fusion tags on protein solubility and cell growth
注:P:PDI;S:SUMO;G:GST;T:TrxA;M:MBP;N:NusA。
2.4 诱导表达条件优化提升可溶性表达
发酵条件(包括诱导OD600值、诱导剂浓度及诱导温度)显著影响蛋白的可溶性表达水平。合适的诱导OD600值能够在保证菌种正常生长的同时实现较高的蛋白产量;诱导剂浓度对包涵体的形成及蛋白的可溶性表达具有重要影响,同时,不合理的诱导剂浓度会影响菌株的生长。
此外,诱导温度不仅极大影响菌种的生长,造成诱导剂添加的时间形成差异,还会影响多肽之间次级键形成与折叠[23]。本研究针对菌株TR07-1*S、TR07-2*T和TR07-3*在不同的诱导温度、诱导剂浓度和诱导OD600值条件下的酶活力表现进行研究。测定不同条件下各菌株粗酶液的酶活力,如图5所示,TR07-1*S在诱导温度为25 ℃,诱导OD600值为1,诱导剂质量浓度1 g/L的条件下的酶活力最高达到6.49 U/mL,相较原条件提升31.9%。TR07-2*T在诱导温度为30 ℃,诱导OD600值为1,诱导剂质量浓度4 g/L的条件下的酶活力最高达到11.65 U/mL,相较原条件提升15.2%。TR07-3*在诱导温度为30 ℃,诱导OD600值为0,诱导剂质量浓度2 g/L的原条件下的酶活力最高,达到3.4 U/mL。对正交试验的酶活力测定数据进行处理,计算各诱导表达条件下的k值和极差R值,结果如表4所示。TR07-1*S、TR07-2*T和TR07-3*中温度的极差均大于诱导剂浓度和诱导OD600值的极差,表明温度对酶的可溶性表达水平的影响最大。
表4 不同因素下的k值和极差R
Table 4 k values and range (R) under different conditions
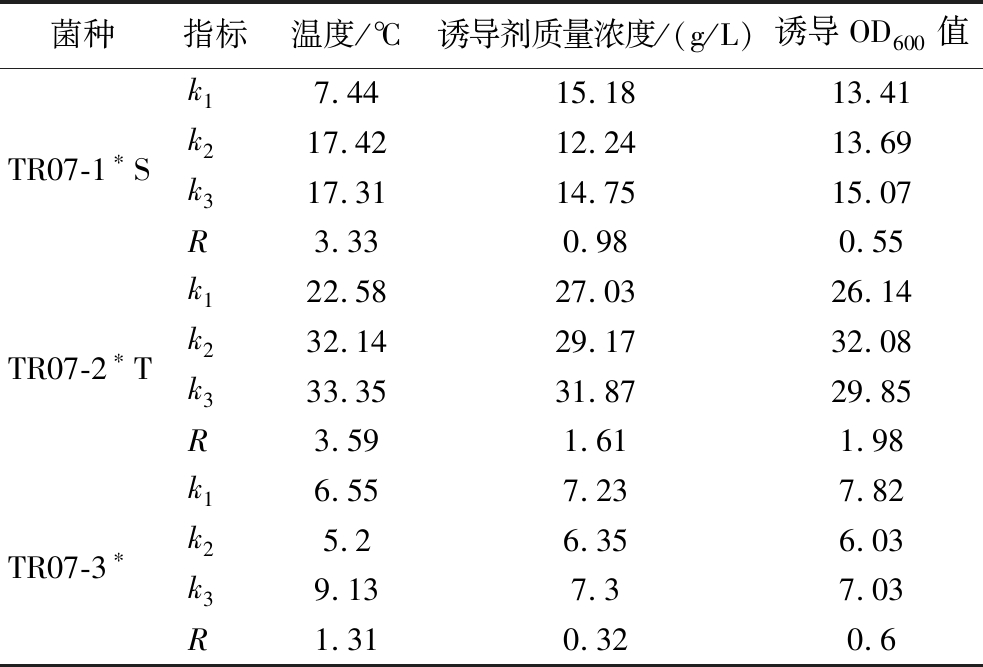
菌种指标温度/℃诱导剂质量浓度/(g/L)诱导OD600值TR07-1∗Sk17.4415.1813.41k217.4212.2413.69k317.3114.7515.07R3.330.980.55TR07-2∗Tk122.5827.0326.14k232.1429.1732.08k333.3531.8729.85R3.591.611.98TR07-3∗k16.557.237.82k25.26.356.03k39.137.37.03R1.310.320.6
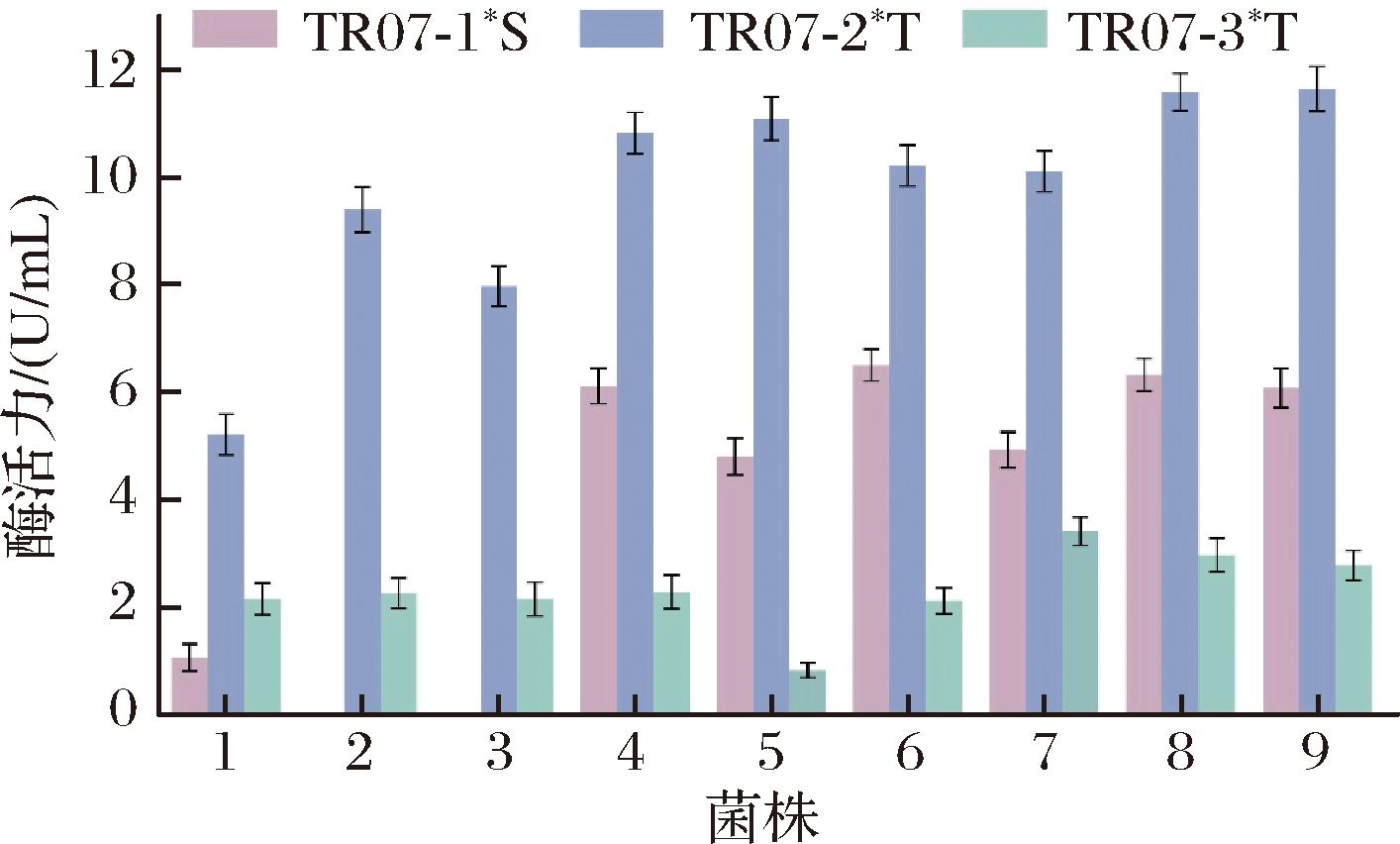
图5 诱导表达条件对酶活力的影响
Fig.5 Effect of induced expression conditions on enzyme activity
注:1:诱导温度22 ℃,诱导OD600值为0,诱导剂质量浓度1 g/L; 2:诱导温度22 ℃,诱导OD600值为0.5,诱导剂质量浓度4 g/L; 3:诱导温度22 ℃,诱导OD600值为1,诱导剂质量浓度2 g/L; 4:诱导温度25 ℃,诱导OD600值为0,诱导剂质量浓度4 g/L; 5:诱导温度25 ℃,诱导OD600值为0.5,诱导剂质量浓度2 g/L; 6:诱导温度25 ℃,诱导OD600值为1,诱导剂质量浓度1 g/L; 7:诱导温度30 ℃,诱导OD600值为0,诱导剂质量浓度2 g/L; 8:诱导温度30 ℃,诱导OD600值为0.5,诱导剂质量浓度1 g/L; 9:诱导温度30 ℃,诱导OD600值为1,诱导剂质量浓度4 g/L。
2.5 融合酶对海藻糖转化率的影响
根据陈春[8]的报道,目前以糊精和淀粉为底物,双酶法制备海藻糖的转化率普遍在60%以上,具有较高的基础水平。其中KIM等[24]利用来源于Brevibacterium helvolum的MTSase和MTHase在37 ℃条件下以1 g/L可溶性淀粉为底物进行海藻糖的转化,最终达到70.4%的海藻糖转化率。为了验证完成优化的酶和诱导表达条件对海藻糖转化率的影响,将对照组(TR07-1和TR07-2的粗酶液),实验组1(TR07-1、TR07-2和TR07-3的粗酶液)以及实验组2(TR07-1*S、TR07-2*T和TR07-3*的粗酶液)均应用于52 ℃条件下麦芽糊精转化海藻糖的实验。通过HPLC分析反应液中海藻糖的产量并计算转化率(图6-a),其中对照组的海藻糖转化率为59.30%,与目前常用的双酶法获得的海藻糖转化率相近(60%)[25-26]。另外,实验组1中海藻糖转化率提升至78.65%(图6-b),表明4αGT的歧化反应对提高海藻糖转化率有显著效果。值得注意的是,实验组2中海藻糖转化率进一步提升至83.33%(图6-b),表明3个酶表达量的提升对海藻糖转化有促进作用。传统双酶法反应结束后体系中残存大量的短链麦芽寡糖,其难以继续与MTSase和MTHase反应生成海藻糖,造成底物浪费,导致转化率和底物利用率较低,阻碍该方法在工业领域大规模使用。而三酶法通过4αGT的歧化反应,催化短链麦芽寡糖合成长链麦芽寡糖,海藻糖的转化得以继续进行,大大提高转化率和底物利用率。此外,提高其表达量还能进一步提升转化率和降低成本。
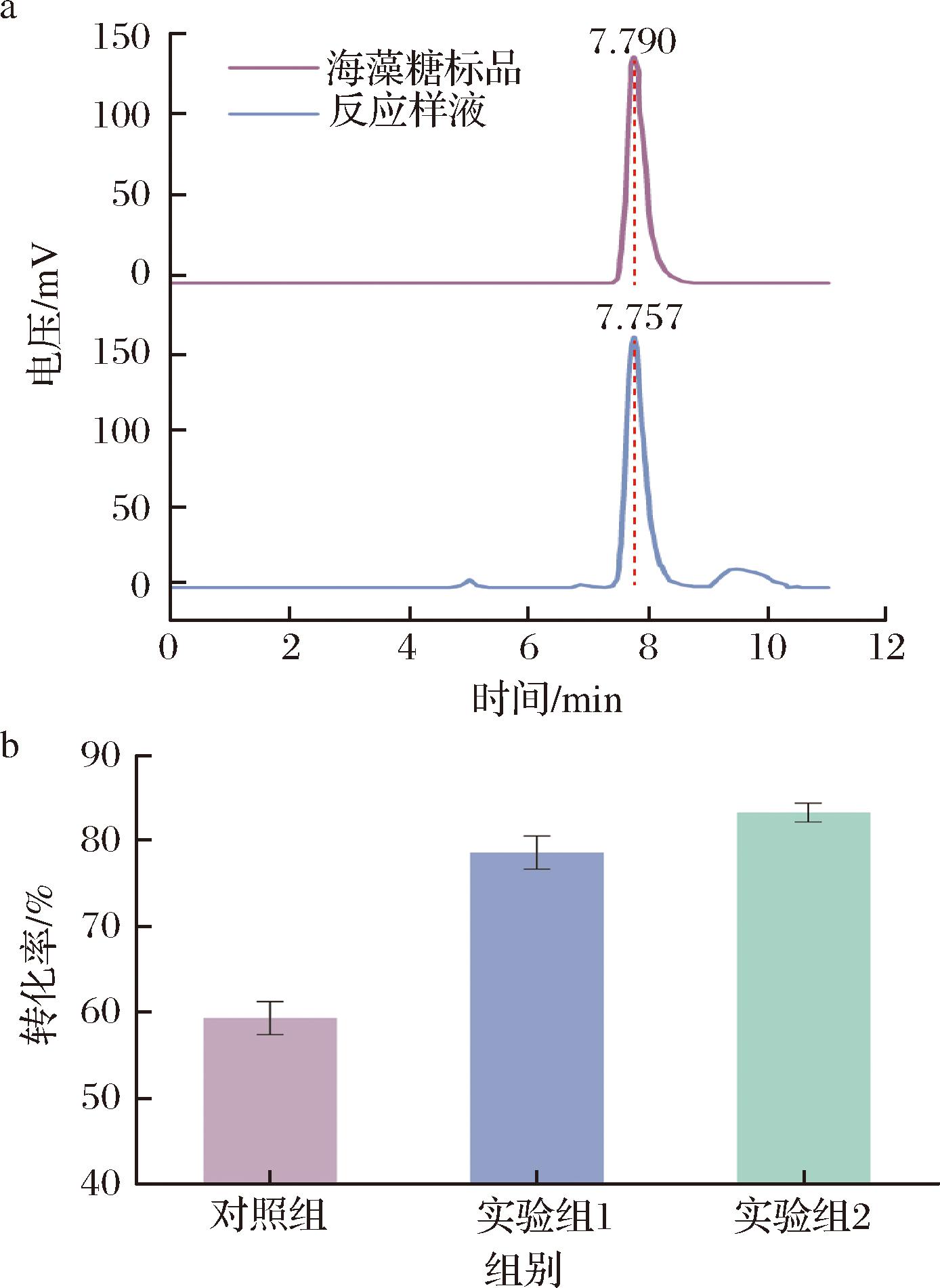
a-海藻糖标品及酶反应液的HPLC结果;b-不同体系对海藻糖转化率的影响
图6 融合酶对海藻糖转化率的影响
Fig.6 Effect of fusion protein on the trehalose conversion rate
3 结论
海藻糖因其稳定性和安全性在多个领域具有广泛的应用潜力,而双酶法在海藻糖的酶法制备中具有重要作用。本研究采用TreY、TreZ和MalQ,将麦芽糊精转化为海藻糖。通过NCBI数据库检索获得上述基因序列,并分析发现,TreY和TreZ基因序列的GC含量偏高(74%),易引发引物设计困难及PCR反应中引物错配率升高,从而降低了后续转化实验的阳性率。通过序列优化,显著降低了GC含量,同时提升了基因在大肠杆菌中的表达量,其中TR07-1*和TR07-3*粗酶液的酶活力分别提高了87%和52.8%。尽管如此,优化后酶的表达仍存在包涵体现象,显著限制了粗酶液的酶活力。为此,本文进一步采用融合促融蛋白标签的策略,在TR07-1*、TR07-2*和TR07-3*的N端通过linker (GGGGS)2连接6种不同的蛋白标签。然而,酶活力测定结果表明,粗酶液的酶活力未得到改善。随后,通过优化酶的诱导表达条件,确定最佳诱导参数,TR07-1*S和TR07-2*T的酶活力分别提高了31.9%和15.2%。最终,以酶活力最高的融合酶TR07-1*S、TR07-2*T和TR07-3*用于海藻糖的转化实验,海藻糖转化率提高至83.33%。本研究使用低成本的麦芽糊精作为原料进行海藻糖的生产,降低生产成本,并且将酶的生产和转化实验的周期压缩至48 h,极大地提高海藻糖的生产效率。此外,本研究中海藻糖转化率是目前报道的更高水平,达到工业化生产需求。
[1] GAO H, GONG J S, SU C, et al.Characterization, heterologous expression and engineering of trehalase for biotechnological applications[J].Systems Microbiology and Biomanufacturing, 2022, 2(3):445-460.
[2] RICHARDS A B, KRAKOWKA S, DEXTER L B, et al.Trehalose:A review of properties, history of use and human tolerance, and results of multiple safety studies[J].Food and Chemical Toxicology, 2002, 40(7):871-898.
[3] ELBEIN A D, PAN Y T, PASTUSZAK I, et al.New insights on trehalose:A multifunctional molecule[J].Glycobiology, 2003, 13(4):17R-27R.
[4] TSENG W C, LIN C R, HUNG X G, et al.Identification of substrate-binding and selectivity-related residues of maltooligosyltrehalose synthase from the thermophilic archaeon Sulfolobus solfataricus ATCC 35092[J].Enzyme and Microbial Technology, 2014, 56:53-59.
[5] AGARWAL N, SINGH S P.A novel trehalose synthase for the production of trehalose and trehalulose[J].Microbiology Spectrum, 2021, 9(3):e01333-21.
[6] KIM H H, JUNG J H, SEO D H, et al.Novel enzymatic production of trehalose from sucrose using amylosucrase and maltooligosyltrehalose synthase-trehalohydrolase[J].World Journal of Microbiology and Biotechnology, 2011, 27(12):2851-2856.
[7] LIU H L, YANG S J, LIU Q, et al.A process for production of trehalose by recombinant trehalose synthase and its purification[J].Enzyme and Microbial Technology, 2018, 113:83-90.
[8] 陈春. Arthrobacter ramosus MTSase/MTHase分子改造及其制备海藻糖研究[D].无锡:江南大学, 2022.CHEN C.Molecular engineering of Arthrobacter ramosus MTSase/MTHase and preparation of trehalose[D].Wuxi:Jiangnan University, 2022.
[9] MARUTA K, NAKADA T, KUBOTA M, et al.Formation of trehalose from maltooligosaccharides by a novel enzymatic system[J].Bioscience, Biotechnology, and Biochemistry, 1995, 59(10):1829-1834.
[10] FANG T Y, HUNG X G, SHIH T Y, et al.Characterization of the trehalosyl dextrin-forming enzyme from the thermophilic archaeon Sulfolobus solfataricus ATCC 35092[J].Extremophiles, 2004, 8(4):335-343.
[11] LIN Y F, SU P C, CHEN P T.Production and characterization of a recombinant thermophilic trehalose synthase from Thermus antranikianii[J].Journal of Bioscience and Bioengineering, 2020, 129(4):418-422.
[12] TRAKARNPAIBOON S, BUNTERNGSOOK B, WANSUKSRIAND R, et al.Screening, cloning, expression and characterization of new alkaline trehalose synthase from Pseudomonas monteilii and its application for trehalose production[J].Journal of Microbiology and Biotechnology, 2021, 31(10):1455-1464.
[13] FANG M, WANG Q, WANG Z, et al.Whole-cell catalytic synthesis of trehalose by Corynebacterium glutamicum displaying trehalose synthase on its cell surface[J].Systems Microbiology and Biomanufacturing, 2023, 3(3):489-497.
[14] CHEN C, SU L Q, XU F, et al.Improved thermostability of maltooligosyltrehalose synthase from Arthrobacter ramosus by directed evolution and site-directed mutagenesis[J].Journal of Agricultural and Food Chemistry, 2019, 67(19):5587-5595.
[15] KATO M, MIURA Y, KETTOKU M, et al.Purification and characterization of new trehalose-producing enzymes isolated from the hyperthermophilic archae, Sulfolobus solfataricus KM1[J].Bioscience, Biotechnology, and Biochemistry, 1996, 60(3):546-550.
[16] PAUL C J, LEEMHUIS H, DOBRUCHOWSKA J M, et al.A GH57 4-α-glucanotransferase of hyperthermophilic origin with potential for alkyl glycoside production[J].Applied Microbiology and Biotechnology, 2015, 99(17):7101-7113.
[17] SU L Q, WU S X, FENG J Y, et al.High-efficiency expression of Sulfolobus acidocaldarius maltooligosyl trehalose trehalohydrolase in Escherichia coli through host strain and induction strategy optimization[J].Bioprocess and Biosystems Engineering, 2019, 42(3):345-354.
[18] HAN C, SU L Q, HONG R Y, et al.A comparative study of maltooligosyltrehalose synthase from Sulfolobus acidocaldarius expressed in Pichia pastoris and Escherichia coli[J].Process Biochemistry, 2017, 60:35-41.
[19] SUN X, YANG J, FU X P, et al.Trehalose production using three extracellular enzymes produced via one-step fermentation of an engineered Bacillus subtilis strain[J].Bioengineering, 2023, 10(8):977.
[20] ROTH C, WEIZENMANN N, BEXTEN N, et al.Amylose recognition and ring-size determination of amylomaltase[J].Science Advances, 2017, 3:e1601386.
[21] DUAN X G, ZHANG X Y, SHEN Z Y, et al.Efficient production of aggregation prone 4-α-glucanotransferase by combined use of molecular chaperones and chemical chaperones in Escherichia coli[J].Journal of Biotechnology, 2019, 292:68-75.
[22] KOSOBOKOVA E N, SKRYPNIK K A, KOSORUKOV V S.Overview of fusion tags for recombinant proteins[J].Biochemistry (Moscow), 2016, 81(3):187-200.
[23] MÜHLMANN M, FORSTEN E, NOACK S, et al.Optimizing recombinant protein expression via automated induction profiling in microtiter plates at different temperatures[J].Microbial Cell Factories, 2017, 16(1):220.
[24] KIM Y H, KWON T K, PARK S, et al.Trehalose synthesis by sequential reactions of recombinant maltooligosyltrehalose synthase and maltooligosyltrehalose trehalohydrolase from Brevibacterium helvolum[J].Applied and Environmental Microbiology, 2000, 66(11):4620-4624.
[25] DE PASCALE D, SASSO M P, DI LERNIA I, et al.Recombinant thermophilic enzymes for trehalose and trehalosyl dextrins production[J].Journal of Molecular Catalysis B:Enzymatic, 2001, 11(4-6):777-786.
[26] HUANG Y, WANG K, WU J, et al.Separate expression and co-expression of MTSase and MTHase from Arthrobacter ramosus S34 in Escherichia coli BL21(DE3)[J].Systems Microbiology and Biomanufacturing, 2024, 4(1):307-317.