胭脂红酸(carminic acid)是一种用途广泛的天然红色着色剂[1],在绘画染料、纺织、食品添加剂、化妆品和制药等领域均有广泛应用。近年来,胭脂红酸的市场需求逐年增加。长期以来,从寄生于仙人掌的雌性胭脂虫的干燥体中提取是目前胭脂红酸的主要生产方式[2]。由于胭脂红酸繁琐低效的提取过程,存在低得率、成本高和质量差异明显等问题[3]。微生物生产胭脂红酸可以避免这些缺点,可能是可持续供应的潜在替代生产方式。生物合成天然聚酮化合物已开展过很多研究,例如红霉素[4]、洛伐他汀[5]、四环素[6]等聚酮化合物已经使用生物法合成并且广泛利用。
胭脂红酸是一种芳香族聚酮化合物,胭脂红酸的合成路径起始于表达芦荟(Aloe arborescens)来源的聚酮合酶OKS(octaketide synthase,OKS)催化7分子的丙二酰辅酶(malonyl-CoA)和1分子的乙酰辅酶A(acetyl CoA )生成线性的非还原的聚酮化合物,再通过链霉菌(Streptomyces sp.R1128)来源的环化酶ZhuI和芳香酶ZhuJ催化线性的非还原的聚酮化合物环化生成蒽酮,然后在氧化酶的催化下生成黄胭脂酮酸。一方面,黄胭脂酮酸可通过糖基化生成DcLL,DcLL在酿酒酵母(Saccharomyces cerevisiae)来源的5-去甲氧磺双醌羟化酶(5-demethoxyubiquinone hydroxylase,CAT5)催化反应下生成胭脂红酸,另一方面,黄胭脂酮酸可通过羟基化生成胭脂酮酸,胭脂酮酸通过糖基化生成胭脂红酸(图1)。目前,胭脂红酸的生物合成的研究较为广泛,在不同的微生物宿主中均有尝试,在构巢曲霉[7]和大肠杆菌[8]中实现了胭脂红酸的从头合成,但是这2种底盘菌株生产胭脂红酸的效率都很低。在酿酒酵母中,基于在构巢曲霉中构建的胭脂红酸合成途径进一步挖掘了关键氧化酶FAD依赖性单加氧酶(FAD-dependent monooxygenase,AptC)和CAT5,构建出了一条新的胭脂红酸合成途径,在5 L发酵罐水平实现了7 983.8 μg/L胭脂红酸的积累[9]。在酿酒酵母中通过挖掘不同的酶和代谢工程改造的方式大大提高了胭脂红酸的生产水平,但是并未达到可进行工业化生产的水平。
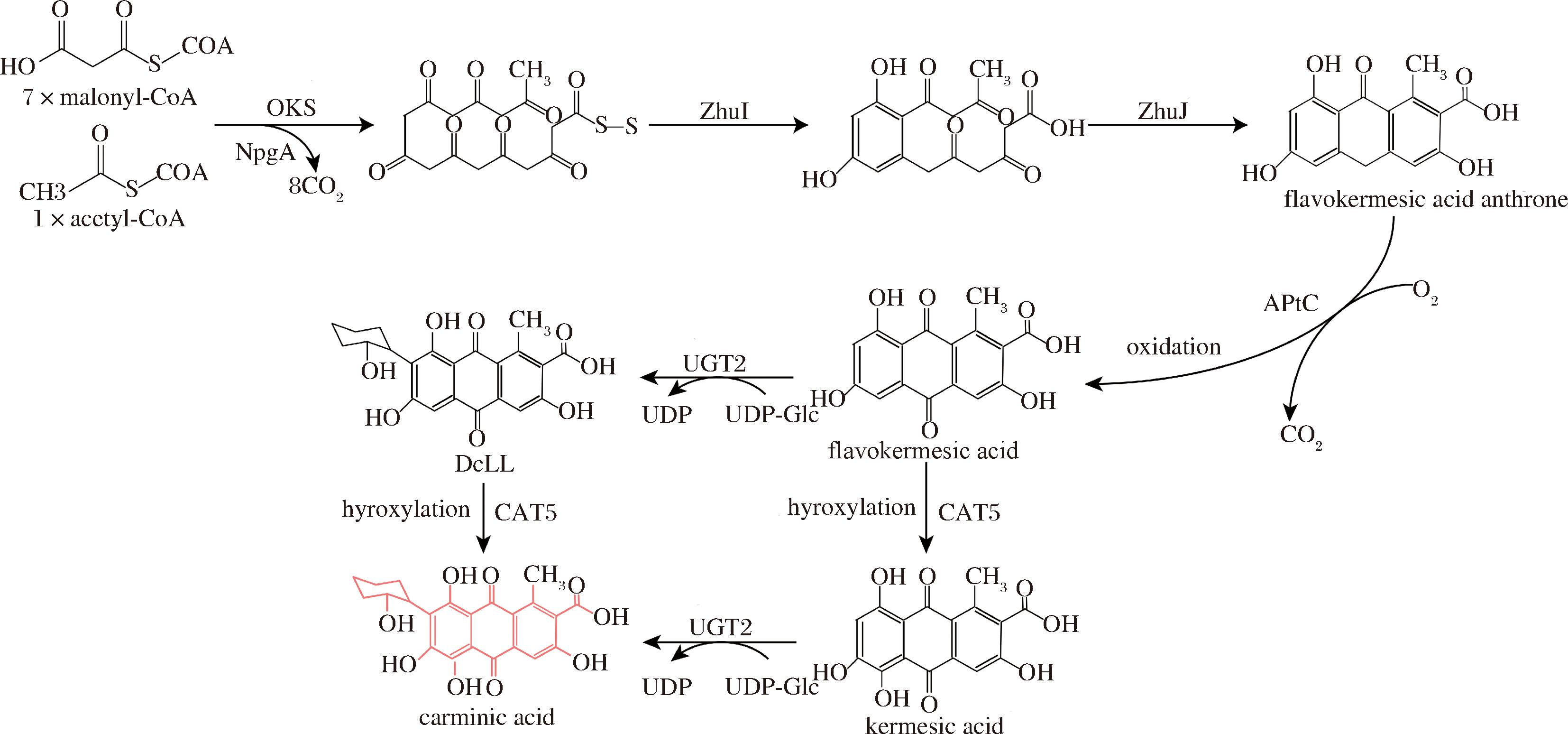
图1 解脂亚洛酵母中胭脂红酸生物合成途径
Fig.1 Biosynthesis pathway of carminic acid in Yarrowia lipolytica
注:oxidation:氧化反应;hyroxylation:羟基化反应;UDP:鸟苷二磷酸;UDP-Glc:鸟苷二磷酸葡萄糖;UDP-葡萄糖醛酸转移酶 (UDP-glucosyltransferase 2,UGT2);构巢曲霉来源的4′磷酸泛硫酰转移酶(4′-phosphopantetheinyl transferase,NpgA)。
解脂亚洛酵母(Yarrowia lipolytica)是一种非传统酵母,以其强大的脂质代谢能力和代谢灵活性著称[10]。相比于传统的模式生物[如大肠杆菌(Escherichia coli)和酿酒酵母],解脂亚洛酵母在利用多种碳源(如甘油、脂肪酸、烷烃等)以及合成脂类衍生天然产物方面具有显著优势[11]。此外,解脂亚洛酵母因富含乙酰辅酶A和丙二酰辅酶A被广泛用于合成许多高价值的化合物[12]。鉴于这些优势,本研究尝试在解脂亚洛酵母中构建胭脂红酸的生物合成路径。由于在酿酒酵母中合成胭脂红酸已经初见成效,本研究选择引入该路径到解脂亚洛酵母中。过氧化物酶体工程在解脂亚洛酵母中也表现出显著效果,例如,通过过氧化物酶体工程实现了角鲨烯的高效生产[13]。另外,解脂亚洛酵母的过氧化物酶体是天然的疏水区室,其中发生的β-氧化是乙酰辅酶A来源的重要代谢过程[14]。因此,过氧化物酶体在胭脂红酸生物合成方面具有很大的发展潜力。
目前尚未有关于胭脂红酸在解脂亚洛酵母中的合成的报道,本研究旨在探究解脂亚洛酵母中构建合成胭脂红酸生物合成路径。通过筛选ACC1突变体[15]、引入非天然乙酰辅酶A合成途径[16]、调节脂质代谢和区室化的策略提高关键前体乙酰辅酶A和丙二酰辅酶A的供应,在解脂亚洛酵母中构建了胭脂红酸的生物合成路径。另外,鉴定发现ylCat5为解脂亚洛酵母中与胭脂红酸生物合成相关的内源性羟基酶。通过调节酶的表达、增加前体供应和修饰β-氧化途径,使胭脂红酸的产量达到1.5 mg/L,最后将胭脂红酸合成途径引入到过氧化物酶体中,使胭脂红酸产量提高到1.9 mg/L。在5 L发酵罐水平优化后,胭脂红酸的积累量达到9.6 mg/L(6.3×10-3 mg/g)。本研究为在解脂亚洛酵母中合成胭脂红酸提供了参考意义。
1 材料与方法
1.1 材料与试剂
1.1.1 实验菌株
大肠杆菌JM109用于质粒的构建及扩增,解脂亚洛酵母PO1f为出发菌株,实验所用菌株见表1。
表1 本研究所用菌株
Table 1 Strains used in this study

菌株宿主菌株描述来源PO1FKU70本研究A1PO1FAXP:: PTEF-OKS-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-SCCAT5-TXPR2本研究A2PO1FAXP::PTEF-PB.1324-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-SCCAT5-TXPR2本研究A3PO1FAXP::PTEF-PB.6324-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-SCCAT5-TXPR2本研究B1PO1FAXP:: PTEF-OKS-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-YLCAT5-TXPR2本研究B2PO1FAXP:: PTEF-OKS-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-OPTSCCAT5-TXPR2本研究B3PO1FAXP:: PTEF-OKS-TXPR2-PTEF-ZhuI-TXPR2-PTEF-ZhuJ-TXPR2,D17::PTEF-APTC-TXPR2-PTEF-UGT2-TXPR2-PTEF-ZhuJ-TXPR2,A3::PTEF-PB.12901-TXPR2本研究C1B2E3::PTEF-ACC1-TXPR2本研究C2B2E3::PTEF-ACC1S667E-TXPR2本研究C3B2E3::PTEF-ACC1S667D-TXPR2本研究C4B2E3::PTEF-ACC1S1178E-TXPR2本研究C5B2E3::PTEF-ACC1S1178D-TXPR2本研究C6B2E3::PTEF-SCACC1-TXPR2本研究C7B2C7::PTEF-MCRC-TXPR2-PGPD-BAUA-TXPR2本研究D1C7E4::PTEF-POX1-TXPR2本研究D2C7E4::PTEF-POX2-TXPR2本研究D3C7E4::PTEF-POX3-TXPR2本研究D4C7E4::PTEF-POX4-TXPR2本研究D5C7E4::PTEF-POX5-TXPR2本研究D6C7E4::PTEF-POX6-TXPR2本研究D7C7E4::PTEF-PEX10-TXPR2本研究D8C7E5::PTEF-ACL1-TXPR2-PTEF-ACL2-TXPR2本研究
1.1.2 主要试剂
2×Taq PCR Master Mix、Phanta®Max Super-Fidelity DNA polymerase,南京诺唯赞生物科技有限公司;快速质粒小提试剂盒、5 min DNA快速纯化试剂盒,北京全式金生物技术有限公司;基因、引物合成及测序,生工生物工程(上海)股份有限公司;胭脂红酸标准品,武汉天植生物技术有限公司。
1.1.3 培养基
用实验室保存的解脂亚洛酵母ku70作为起始菌株,它是在PO1f菌株基础上敲除了基因ku70。采用大肠杆菌JM109菌株构建和扩增质粒。大肠杆菌在37 ℃的条件下,培养于含氨苄青霉素(100 mg/L)的溶源肉汤(LB)或琼脂平板上。解脂亚洛酵母在酵母氮基(yeast nitrogeh base,YNB)培养基(6.74 g/L YNB,含或不含0.47 g/L氨基酸、葡萄糖20 g/L)或酵母膏葡萄糖(YPD)(20 g/L葡萄糖、20 g/L蛋白胨和10 g/L酵母膏)上培养,温度为30 ℃。
1.2 仪器与设备
Mastercycler nexus GX2 PCR基因扩增仪、5425台式高速离心机,德国 Eppendorf 公司;DHP-9082B恒温培养箱,上海跃进医疗器械厂;GelDoc XR+凝胶成像仪,美国伯乐 Bio-Rad 公司;LC-20AT高效液相色谱仪,日本岛津公司;NanoDrop One 超微量分光光度计,美国赛默飞世尔科技公司;MP Fastprep-24 5G 快速样品制备仪,安倍医疗器械贸易(上海)有限公司;5 L生物反应器,T&J生物工程。
1.3 实验方法
1.3.1 目的基因的扩增
来源于芦荟的OKS、链霉菌属R1128的芳香酶ZhuI和环化酶ZhuJ、胭脂虫来源的UDP-葡萄糖醛酸转移酶(UDP-glucosyltransferase 2,UGT2)、构巢曲霉来源的4′磷酸泛硫酰转移酶(4′-phosphopantetheinyl transferase,NpgA)、AptC以及酿酒酵母来源的CAT5由赛索菲合成基因并进行解脂亚洛酵母密码子优化。解脂亚洛酵母的启动子、终止子以及目的基因均由解脂亚洛酵母中基因组为模板扩增。利用Gibson酶构建质粒,通过菌落 PCR 以及测序验证质粒是否构建成功。利用 CRISPR/Cas9 技术整合或者敲除目标基因,在解脂亚洛酵母基因组上设计上下游同源臂,通过酵母菌落 PCR 验证目的基因的敲除或者整合是否成功。研究所用引物见表2。
表2 用于基因扩增的引物
Table 2 Primers used in this study

引物名称序列 (5′-3′)∗OKS-FATGTCTTCTCTGTCTAACGCCTCTCACCOKS-RTTACATCAGGGGCAGAGAGTGCZhuI-FATGCGACATGTCGAGCATACCGTGZhuI-RTTAGGCGGTCACGGTGCCZhuJ-FATGTCTGGCCGAAAGACCTTCCTGZhuJ -RTTAGTCCTCTTCCTCTTGCTCGAACACAptC-FATGACCCTGCCCGTGCTGAptC -RTTAGGCTCGCATCTTCTGCTTCTCNpgA-FATGGTGCAAGACACCTCTTCTGCCNpgA -RTTAAGACAGACAGTTACACACGCCGUGT2-FATGGAGTTCCGACTGCTGATCCTGGUGT2-RTTAGTTCTTCTTCAGCTTCTCAGACTTAGAAGAGSCCAT5-FATGTTATCCCGTGTTTCAGTTTTCAAACCTGSCCAT5-RTTAAATTCTTTCGGCACTCCATATAGCTACTCTGYLCAT5-FATGGCCCGACACGCACTCAGTAAGYLCAT5-RGGGTCGCCGAGCGAGTTTAABAUA-FATGAACCAGCCTTTGAATGTTGCCBAUA-RCGAAGCTCTGAATGGAATCGCTTGAMCRC-FATGGCAGATCTCAGCGCTACCACCMCRC-RGGCGAGCGATCACTGTCTGAACL1-FATGTCTGCCAACGAGAACATCTCCCGACL1-RGTTTCCAAGGCCAAGACTCGATCATAGACL2-FATGTCAGCGAAATCCATTCACGAGGCACL2-RGGCTTCCACTCCTCTCGGAGTTTAAACC1-FATGCGACTGCAATTGAGGACACTAACACGACC1-RGAGCTGCTCAAGGGGTTGTGAPEX10-FATGAGTAGTACTATTCTTTTTTCATCCCGCTTTACCPEX10-RGTCCTTGCCGGACGCGTAAPOX1-FATGGCCAAGGAGCGAGGTAAGACTCPOX1-RCGATGAAATTTGCGATCTCGATGAGTGA
1.3.2 摇瓶发酵
所有小规模发酵均在24深孔板中进行。对于24孔板发酵,菌株在YPD平板上于30 ℃下培养2~3 d。培养结束后,在含有2 mL YPD培养基的24深孔板中随机挑选单菌落,在30 ℃和220 r/min下培养18 h。然后将其转移至新的24孔板中,接种到装有4 mL YPD培养基的24深孔板进行发酵。96 h后测量OD600值,然后处理样品,进行HPLC分析。在补料分批发酵中,将甘油管中的菌株划线在YPD平板上,在30 ℃下培养2~3 d。选择一个接种环,接种到有5 mL YPD培养基中,在30 ℃,220 r/min的条件下摇瓶培养24 h,随后转接到装有100 mL YPD的250 mL摇瓶中,30 ℃,220 r/min的条件培养24 h,随后将其接种到发酵罐。发酵罐接种后的初始OD600值为0.6。在5 L生物反应器中进行发酵。该生物反应器配备了溶解氧(dissolved oxygen,DO)、pH和温度测量探头,并由自动控制的进料系统进料。发酵温度为28 ℃,发酵时间为132 h,初始发酵罐中含有2.5 g/L YPD培养基。每升培养基含20 g蛋白胨、10 g酵母粉和40 g葡萄糖。当初始葡萄糖耗尽时,通过在发酵罐中加入800 g/L葡萄糖来维持葡萄糖质量浓度为1 g/L。对于抗氧化剂抗坏血酸的添加,在发酵时间为24 h时加入,添加量为2 g/L。
1.3.3 检测方法
使用岛津LC-20AT高效液相色谱系统进行分析,该系统配备了可变波长检测器和Agilent ZORBAX Nikpase XDB-C18柱。流动相为乙腈,并添加0.1%(体积分数)无水甲酸,流速1.0 mL/min,进样量10 μL,等度梯度洗脱20 min,检测波长为494 nm。
2 结果与分析
2.1 聚酮合酶PKS(polyketide synthase,PKS)与黄酮酸羟化酶(flavonoid acid hydroxylase)CAT5的筛选与优化
为探索新的聚酮合酶PKS来源,本研究基于前期研究获得的胭脂虫转录组数据,筛选并鉴定出2个具有完整功能结构域的聚酮合酶,分别命名为PB.1324和PB.3976。当将PB.1324和PB.3976基因分别整合至解脂亚洛酵母的E4位点以替代A1菌株中的OKS基因时,构建的菌株A2和A3在发酵液中均未检测到胭脂红酸的生成(图2-A)。尽管PB.1324和PB.3976具备完整的结构域,其催化活性可能受到异源宿主中蛋白质折叠、辅酶需求或其他分子因素的限制,未能有效催化胭脂红酸的合成。

A-聚酮合酶PKS的筛选;B-黄酮酸羟化酶CAT5的筛选;C-氨基酸序列比对结果
图2 聚酮合酶PKS与黄酮酸羟化酶CAT5的筛选
Fig.2 Screening of polyketone synthase PKS with flavonoid hydroxylase CAT5
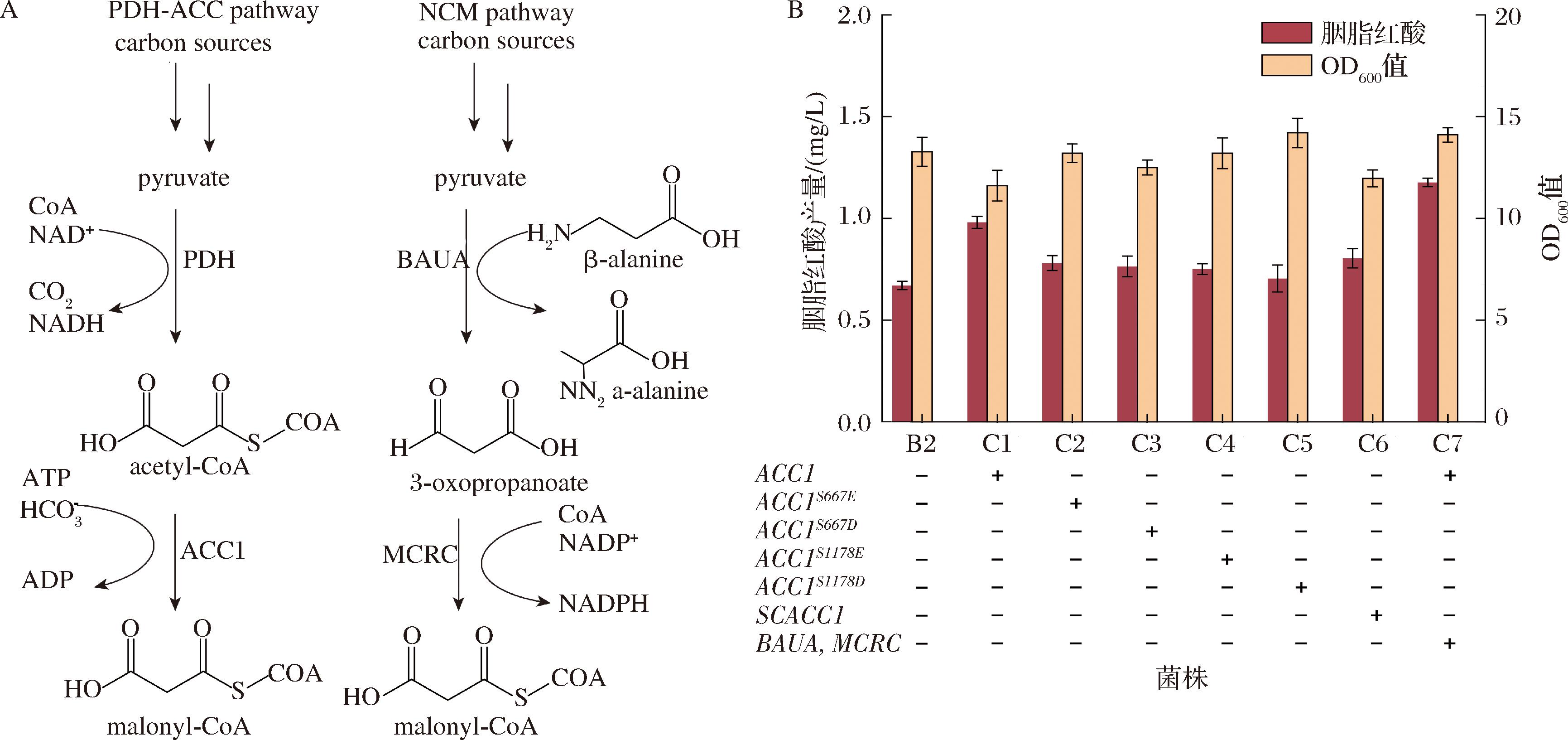
A-天然丙二酰辅酶A合成途经和非天然丙二酰辅酶A合成途径(NCM途径);B-丙二酰辅酶A强化基因的过表达
图3 优化丙二酰辅酶A的供应模块提高胭脂红酸的产量
Fig.3 Optimize the supply module of malonyl-CoA to increase the yield of carminic acid
注:pyruvate:丙酮酸;PDH:pyruvate dehydrogenase,丙酮酸脱氢酶;ACC1:acetyl-CoA carboxylase 1,乙酰辅酶A羧化酶;BAUA:Beta-alanine-pyruvate aminotransferase,β-丙氨酸-丙酮酸转氨酶;β-alanine:β-丙氨酸;3-oxopropanoate:3-氧代丙酸乙酯;MCRC:methyl-coenzyme M reductase operon protein C,丙二酰辅酶A还原酶;NCM pathway:NCM途径。
在胭脂红酸的合成路径中,黄酮酸羟化酶同样对其生产至关重要。研究酿酒酵母中的内源性羟化酶发现,CAT5是关键酶之一。本研究通过与酿酒酵母CAT5氨基酸序列的比对,成功鉴定出解脂亚洛酵母中的一个内源性羟化酶(ylCAT5),其氨基酸序列与酿酒酵母CAT5的相似度超过65%(图2-C)。另外,从胭脂虫转录组数据中挖掘出一个潜在的羟化酶PB.12901。
为比较不同羟化酶对胭脂红酸合成的影响,将酿酒酵母来源的ScCAT5、解脂亚洛酵母来源的ylCAT5以及胭脂虫来源的PB.12901分别整合至解脂亚洛酵母A3位点,构建对应的工程菌株。摇瓶发酵120 h,发现表达ScCAT5的菌株A1获得了最高的胭脂红酸产量(0.5 mg/L)(图2-B),表达YlCAT5的菌株B1产量为0.4 mg/L(图2-B),而表达PB.12901的菌株B3未检测到胭脂红酸生成。为了进一步提高胭脂红酸产量,对ScCAT5基因进行了密码子优化,使其更适应解脂亚洛酵母的表达系统。结果表明优化后的ScCAT5表达菌株B2中胭脂红酸产量达到0.7 mg/L(图2-B),高于未优化的菌株。
2.2 强化丙二酰辅酶A的供给
胭脂红酸的合成需要2个关键前体:1分子乙酰辅酶A和7分子丙二酰辅酶A,其中,乙酰辅酶A羧化是酵母细胞中丙二酰辅酶A形成的内源途径[17]。然而,天然的丙二酰辅酶A生成效率较低[18],限制了胭脂红酸的合成通量。为了提高丙二酰辅酶A的供应,本研究对解脂亚洛酵母编码乙酰辅酶A羧化酶的ACC1进行对比筛选,表达了研究报道的不同ACC1突变体[15],包括ACC1S667E、ACC1S667D、ACC1S1178E、ACC1S1178D,以及酿酒酵母来源的ACC1[19](图3-A),分别构建了菌株C1-C6。结果表明,表达内源ACC1的菌株C1相比对照B3菌株,其胭脂红酸产量提高到1.0 mg/L。然而,表达几种突变体菌株的胭脂红酸产量未显示出明显提升,说明丙二酰辅酶A的供应仍是合成胭脂红酸的瓶颈。为了进一步提高丙二酰辅酶A的供应,又将非天然丙二酰辅酶A合成途径(non-carboxylative malonyl-CoA formation pathway,NCM途径)引入B2菌株中(图3-A),构建了菌株C7,此过程将MCRC和BAUA基因整合到C7位点。结果表明,C7菌株的胭脂红酸产量相较于C1菌株进一步提高到1.2 mg/L(图3-B)。
2.3 调节脂质代谢增加乙酰辅酶A供给
乙酰辅酶A是胭脂红酸合成的重要前体,也是胞内丙二酰辅酶A生成的关键前体[20],鉴于胭脂红酸合成需要更多的丙二酰辅酶A分子,增加乙酰辅酶A的供应对提高其产量至关重要[21]。
本研究拟通过β-氧化途径的改造和相关基因的过表达,提升解脂亚洛酵母中乙酰辅酶A的供给(图4-A),进而提高胭脂红酸的产量。首先,过表达了与β-氧化途径相关的一系列关键基因,包括POX1、POX2、POX3、POX4、POX5、POX6和PEX10。将这些基因分别整合至解脂亚洛酵母E3位点,分别构建了工程菌株D1~D7。摇瓶发酵120 h,结果显示,过表达PEX10的菌株D7,其胭脂红酸的产量略有提高,达到1.4 mg/L(图4-B)。为进一步增加乙酰辅酶A的代谢通量,在D7菌株的基础上又过表达了ATP柠檬酸裂解酶(ATP citrate lyase,ACL)的2个亚基ACL1和ACL2,得到菌株D8,摇瓶发酵结果表明,ACL1和ACL2的过表达显著增强了乙酰辅酶A的生成,进一步将胭脂红酸的产量提高至1.5 mg/L(图4-B)。
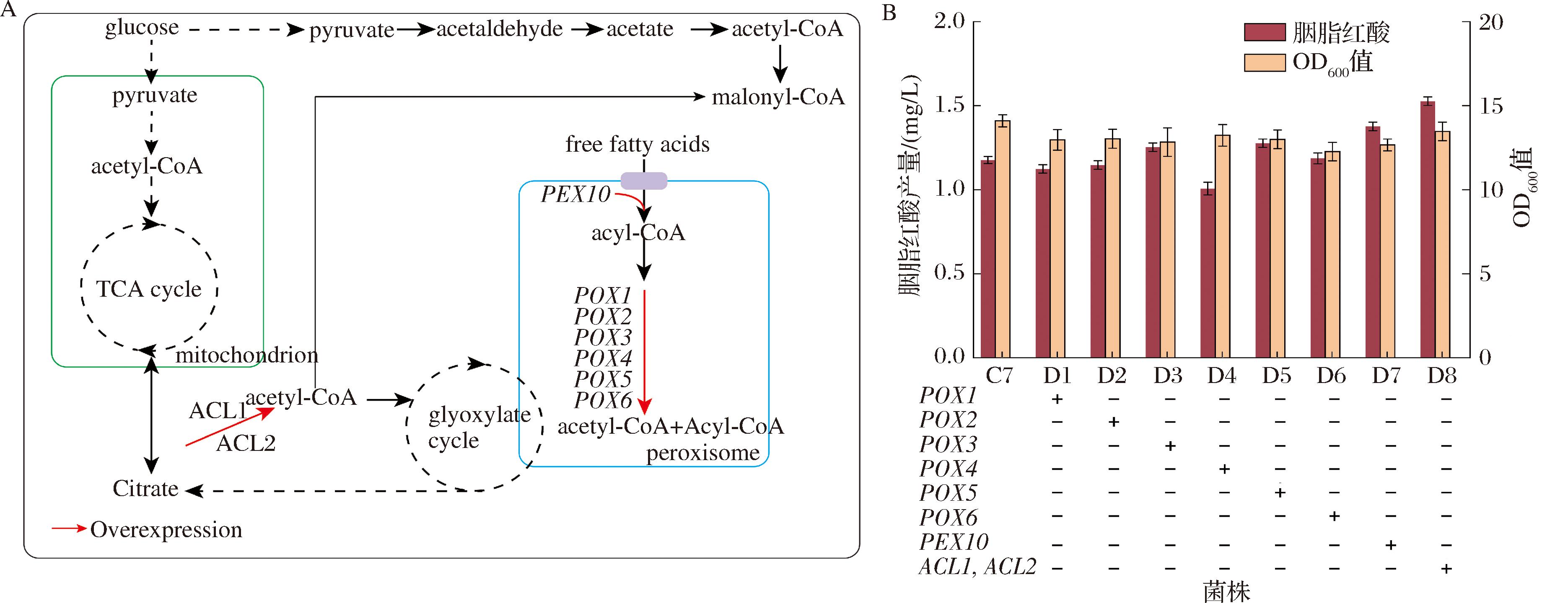
A-调节脂肪酸β-氧化示意图;B-优化乙酰辅酶A的供应模块提高胭脂红酸的产量
图4 优化乙酰辅酶A的供应模块提高胭脂红酸的产量
Fig.4 Optimize the supply module of acetyl-CoA to increase the yield of carminic acid
注:acetaldehyde:乙醛;acetate:乙酸盐;free fatty acids:游离脂肪酸;POX1~6:protoporphyrinogen oxidase1~6,脂肪酰基辅酶A氧化酶;PEX10: peroxisome assembly protein 10,过氧化物酶体生物发生因子10;ACL1,ACL2:ATP-citrate synthase subunit ,ATP 柠檬酸裂解酶;citrate:柠檬酸; glyoxylate cycle:乙醛酸循环;TCA cycle:tricarboxylic acid cycle,三羧酸循环;mitochondrion:线粒体;peroxisome:过氧化物酶体;红色箭头表示过表达。
2.4 构建过氧化物酶体隔室促进胭脂红酸合成
解脂亚洛酵母因其发达的过氧化物酶体结构和脂质代谢能力,在脂肪酸及其衍生物的代谢中发挥了重要作用[22]。过氧化物酶体是解脂亚洛酵母代谢网络的重要组成部分,参与β-氧化、脂质合成和分解等代谢过程[23]。研究表明通过过氧化物酶体的工程改造,可以显著优化解脂亚洛酵母的代谢流,实现高效合成天然产物。尽管通过增强β-氧化途径提高了乙酰辅酶A的通量,但是胭脂红酸的产量提升相对有限。为了进一步提高胭脂红酸的生物合成效率,本研究将参与胭脂红酸合成的7种关键酶分为3组,在信号肽CRMVGKSKL[24]的引导下,将这些酶依次引入至过氧化物酶体(图5-A)。为了验证连接肽的效果,将该信号肽连接到红色荧光蛋白mCherry的C端,与对照相比,携带连接肽的菌株只在过氧化物酶体上检测到荧光信号,表明该连接肽可以成功将酶引入到过氧化物酶体中(图5-C)。发酵结果表明,通过过氧化物酶体区室化策略的引入,有效提升了酶的空间分布和代谢通量,并结合胞质中的胭脂红酸合成途径,从而提高了胭脂红酸的产量。引入全生物合成途径的工程菌株E1,胭脂红酸的产量增加至1.9 mg/L(图5-B)。
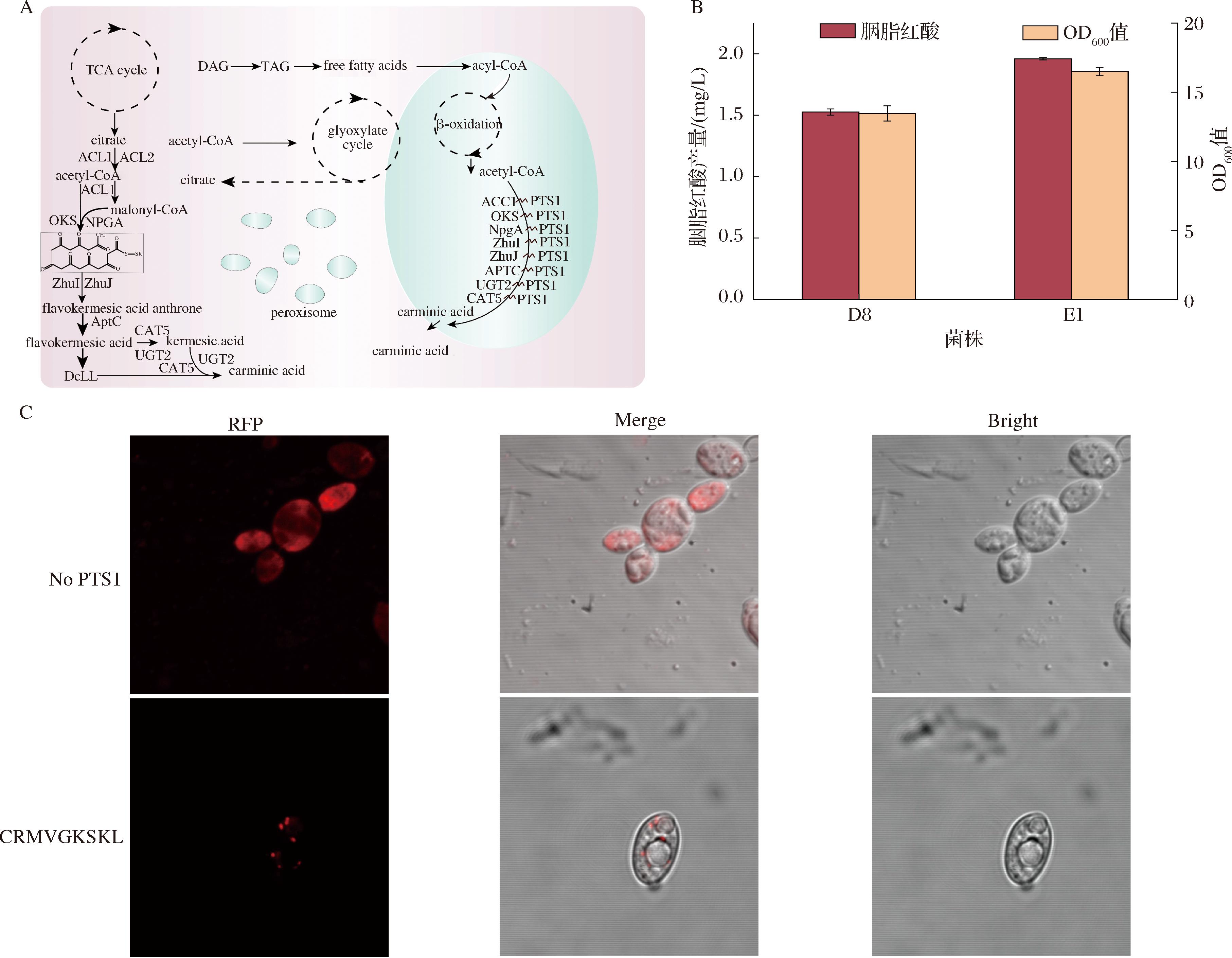
A-增强胭脂红酸合成途径的过氧化物酶体工程;B-过氧化物酶体工程菌株发酵结果;C-红色荧光蛋白定位过氧化物酶体[荧光图像 (左)、合并图像(中)和明场图像(右)]
图5 通过过氧化物酶体工程提高胭脂红酸的产量
Fig.5 Increase the production of carminic acid by peroxisome engineering
注:DGA:酮戊二酸;TAG:甘油三脂;PTS1:1型过氧化物酶体靶向信号肽。
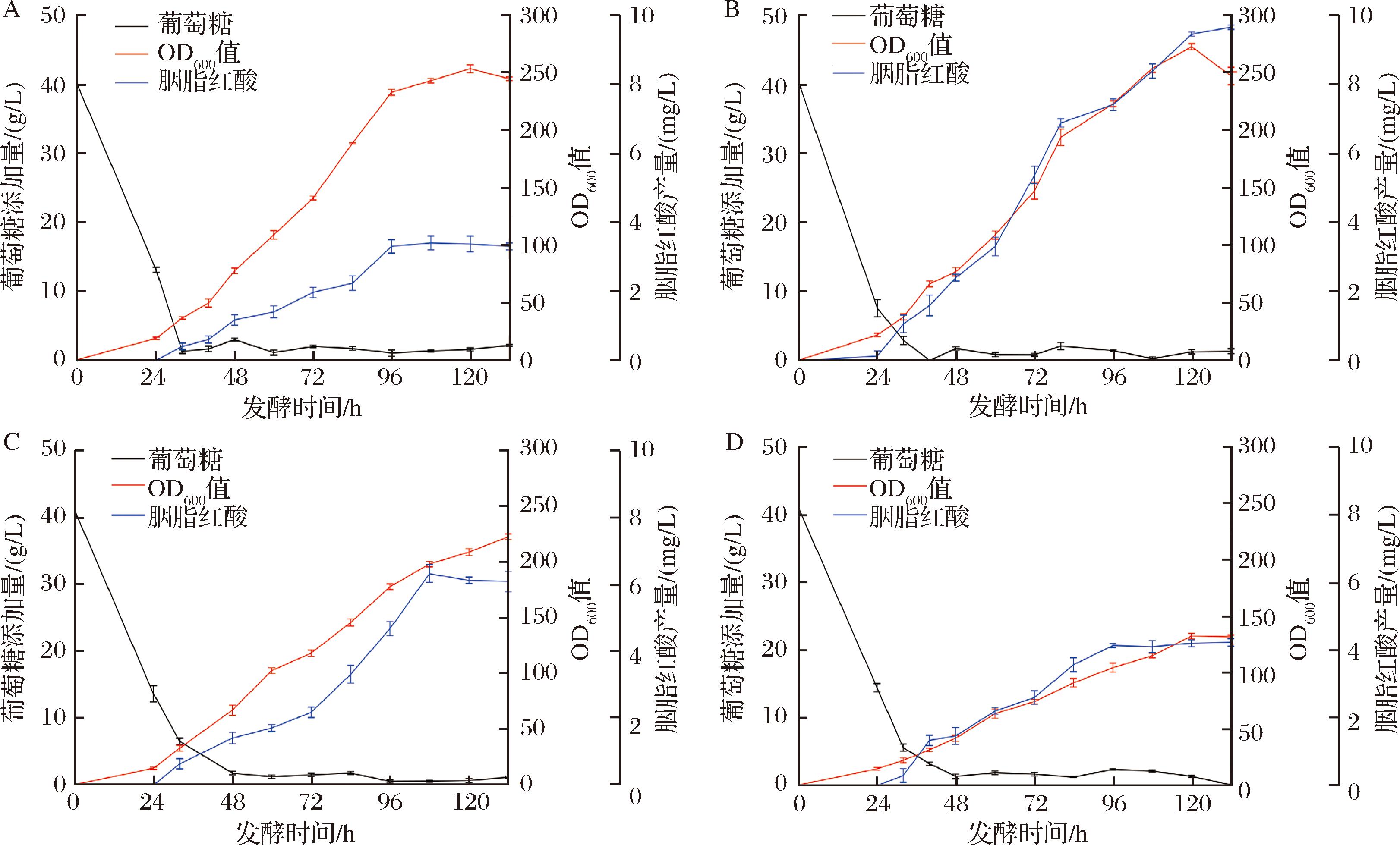
A-不添加抗坏血酸,E2菌株发酵曲线;B-添加抗坏血酸,E2菌株发酵曲线;C-添加0.5% BHT时,E2菌株发酵曲线; D-添加抗坏血酸,无机氮源补料时E2菌株发酵曲线
图6 5 L发酵罐中生产胭脂红酸
Fig.6 Carminic acid is produced in 5 L fermentation tanks
2.5 5 L发酵罐的补料分批发酵
为了进一步提高胭脂红酸的产量,将得到的胭脂红酸产量最高的E1菌株,回补了URA和LEU标签后,在5 L发酵罐扩大培养,研究了添加不同抗氧化剂对胭脂红酸积累的影响。结果表明,不添加抗坏血酸时,发酵液的最高OD600值为250,胭脂红酸产量为3.5 mg/L(2.2×10-3 mg/g)。在24 h时添加0.05%(质量分数)的2,6-二叔丁基对甲酚(butylated hydroxytoluene,BHT)和2 g/L抗坏血酸时,胭脂红酸产量分别为6.4 mg/L(4.1×10-3 mg/g)和9.6 mg/L(6.3×10-3 mg/g)。此外,在添加抗氧化剂的情况下通过补充无机氮源,胭脂红酸产量为4.3 mg/L(5.3×10-3 mg/g)(图6)。本研究所得的胭脂红酸产量已是目前报道的最高水平,为其工业化奠定了基础。
3 结论
随着市场对胭脂红酸需求的不断增长,传统生产方式因其高成本、低效率而面临诸多挑战,微生物合成胭脂红酸成为了备受关注的替代方式。本研究将酿酒酵母中的胭脂红酸合成途径引入至解脂亚洛酵母中,实现了胭脂红酸的生物合成。鉴于胭脂红酸合成途径存在的低酶活问题[7-8],而聚酮合酶作为聚酮化合物合成的起始,本研究尝试在胭脂虫转录组中挖掘新的同工酶,最终鉴定出2个具有完整功能结构域的聚酮合酶PB.1324和PB.3976,但是,将这2个同工酶整合至解脂亚洛酵母基因组后,并未检测到胭脂红酸的生成,表明这些同工酶在该宿主中的功能表现并不理想。此外,针对途径中另一关键酶CAT5,鉴于酿酒酵母和解脂亚洛酵母存在很多同工酶,推测解脂亚洛酵母中也存在相关的羟化酶。在解脂亚洛酵母中鉴定出了同源羟化酶(ylCAT5)。此外在实验室提供的胭脂虫转录组数据中也鉴定出CAT5(PB.12901)。在构建的工程菌株中,内源性羟化酶ylCAT5的整合有效促进了胭脂红酸的合成,而胭脂虫来源的CAT5(PB.12901)则未表现出同样的功能。这一结果表明其在解脂亚洛酵母中的功能并未得到有效表达,进一步表明不同物种间的酶活性和功能性可能存在显著差异,并且胭脂虫来源基因的氨基酸序列与对照OKS序列相似度低,相似度低可能意味着它们的催化活性或底物特异性不同,无法完成目标代谢反应,而且胭脂虫内生产胭脂红酸的途径未被阐明,可能体内胭脂虫合成胭脂红酸的途径与现有的生物合成途径完全不同,也可能胭脂虫来源的酶在解脂亚洛酵母中无法发挥作用。而挖掘出的解脂亚洛酵母来源的CAT5可以实现相应的羟基化作用,不过效果并不如酿酒酵母来源的CAT5。
胭脂红酸合成过程中,乙酰辅酶A和丙二酰辅酶A作为关键底物,而解脂亚洛酵母含有丰富的乙酰辅酶A和丙二酰辅酶A。LIU等[15]将筛选的ACC1突变体引入到工程菌株中成功增强了吉马烯A的生成。因此,本研究对解脂亚洛酵母的乙酰辅酶A和丙二酰辅酶A合成途径进行了系统性优化。通过筛选解脂亚洛酵母内源的ACC1基因及其多个突变体,以及酿酒酵母来源的ACC1基因,并引入了非天然的丙二酰辅酶A合成途径(NCM途径),提升了胭脂红酸的产量。另外,通过引入NCM途径,有效提高了丙二酰辅酶A的通量,为胭脂红酸的生物合成提供了更多前体物质。另一个关键前体乙酰辅酶A是脂肪酸代谢和能量生产的核心中间体,其充足供应对脂质代谢的效率至关重要。因此,在胭脂红酸合成过程提高乙酰辅酶A的供应也是十分必要的。研究表明,通过对β-氧化途径的改造,可以增强油脂的代谢能力,YUE等[25]通过过表达POX1显著提高了(2S)-eriodictyol的产量。本研究通过β-氧化途径增强乙酰辅酶A的生成,并过量表达了内源的ATP依赖性柠檬酸水解酶ACL1和ACL2用于水解脂肪酸β-氧化过程中的柠檬酸,提高了乙酰辅酶A的通量,胭脂红酸产量提高到1.5 mg/L。过氧化物酶体是细胞内执行脂肪酸β-氧化的关键亚细胞器,其特有的酶系统能够高效分解长链脂肪酸,并通过一系列反应生成乙酰辅酶A [21]。PARK等[26]将整个甲羟戊酸途径靶向过氧化物酶体以改善前体供应,显著提高了D-柠檬烯的产生。本研究将整条胭脂红酸合成途径靶向至过氧化物酶体内,胭脂红酸的产量也得到提高,在5 L罐的补料分批发酵过程中,细胞密度是摇瓶水平的十几倍,但胭脂红酸的产量并没有提高到相应的水平,说明代谢通量未必都流向胭脂红酸的合成,大部分营养物质被用于维持细胞的基本代谢和生长,而非产物合成,胭脂红酸积累量还是处在较低的水平,这可能是胭脂红酸合成途径基因的低酶活限制了胭脂红酸的生产。因此,后续的研究工作可以继续挖掘其他更多来源的同工酶,或是通过蛋白质工程和定向进化手段优化限制提升现有酶的活性,以期实现胭脂红酸的高效生物合成。虽然胭脂红酸的产量离工业化生产还有一定差距,但是本研究所呈现的改造策略为胭脂红酸的微生物合成提供了一定参考,也为聚酮类化合物的生物合成提供了研究思路。
[1] GUILLERMIN D, DEBROISE T, TRIGUEIRO P, et al.New pigments based on carminic acid and smectites:A molecular investigation[J].Dyes and Pigments, 2019, 160:971-982.
[2] ZAIMA K, FUKAMACHI A, YAGI R, et al.Kinetic study of the equilibration between carminic acid and its two isomers isolated from cochineal dye[J].Chemical and Pharmaceutical Bulletin, 2017, 65(3):306-310.
[3] DAPSON R.The history, chemistry and modes of action of carmine and related dyes[J].Biotechnic &Histochemistry, 2007, 82(4-5):173-187.
[4] ZHANG Q, WU J, QIAN J, et al.Knocking out of tailoring genes eryK and eryG in an industrial erythromycin-producing strain of Saccharopolyspora erythraea leading to overproduction of erythromycin B, C and D at different conversion ratios: Eeffet of disruption of tailoring genes eryK and eryG[J].Letters in Applied Microbiology, 2011, 52(2):129-137.
[5] KUROKAWA H, ITO H, MATSUI H. Monascus purpureus induced apoptosis on gastric cancer cell by scavenging mitochondrial reactive oxygen species[J].Journal of Clinical Biochemistry and Nutrition, 2017, 61(3):189-195.
[6] ZHANG W J, AMES B D, TSAI S C, et al.Engineered biosynthesis of a novel amidated polyketide, using the malonamyl-specific initiation module from the oxytetracycline polyketide synthase[J].Applied and Environmental Microbiology, 2006, 72(4):2573-2580.
[7] FRANDSEN R J N, KHORSAND-JAMAL P, KONGSTAD K T, et al.Heterologous production of the widely used natural food colorant carminic acid in Aspergillus nidulans[J].Scientific Reports, 2018, 8:12853.
[8] YANG D, JANG W D, LEE S Y.Production of carminic acid by metabolically engineered Escherichia coli [J].Journal of the American Chemical Society, 2021, 143(14):5364-5377.
[9] ZHANG Q, WANG X L, ZENG W Z, et al.De novo biosynthesis of carminic acid in Saccharomyces cerevisiae[J].Metabolic Engineering, 2023, 76:50-62.
[10] ANCHE V C, FAKAS S.Abstract 2069: ATP-citrate lyase regulates lipid biosynthesis in Yarrowia lipolytica[J].Journal of Biological Chemistry, 2023, 299(3S):S437.
[11] SALVADOR L PEZ J M, VIDAL L, ADIUTAMA M P, et al.How do engineered Yarrowia lipolytica strains secrete free fatty acids:Hints from comparative transcriptomics[J].FEMS Yeast Research, 2023, 23:foad027.
PEZ J M, VIDAL L, ADIUTAMA M P, et al.How do engineered Yarrowia lipolytica strains secrete free fatty acids:Hints from comparative transcriptomics[J].FEMS Yeast Research, 2023, 23:foad027.
[12] LIU H H, JI X J, HUANG H.Biotechnological applications of Yarrowia lipolytica:Past, present and future[J].Biotechnology Advances, 2015, 33(8):1522-1546.
[13] NING Y, LIU M S, RU Z Y, et al.Efficient synthesis of squalene by cytoplasmic-peroxisomal engineering and regulating lipid metabolism in Yarrowia lipolytica[J].Bioresource Technology, 2024, 395:130379.
[14] POIRIER Y, ANTONENKOV V D, GLUMOFF T, et al.Peroxisomal β-oxidation:A metabolic pathway with multiple functions[J].Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 2006, 1763(12):1413-1426.
[15] LIU Q, ZHANG G, SU L Q, et al.Reprogramming the metabolism of oleaginous yeast for sustainably biosynthesizing the anticarcinogen precursor germacrene A[J].Green Chemistry, 2023, 25(20):7988-7997.
[16] LI J, MU X, DONG W Y, et al.A non-carboxylative route for the efficient synthesis of central metabolite malonyl-CoA and its derived products[J].Nature Catalysis, 2024, 7(4):361-374.
[17] WAKIL S J.A malonic acid derivative as an intermediate in fatty acid synthesis[J].Journal of the American Chemical Society, 1958, 80(23):6465.
[18] SHI S B, CHEN Y, SIEWERS V, et al.Improving production of malonyl coenzyme A-derived metabolites by abolishing Snf1-dependent regulation of Acc1[J].mBio, 2014, 5(3):e01130-14.
[19] PEREIRA H, AZEVEDO F, DOMINGUES L, et al.Expression of Yarrowia lipolytica acetyl-CoA carboxylase in Saccharomyces cerevisiae and its effect on in-vivo accumulation of Malonyl-CoA[J].Computational and Structural Biotechnology Journal, 2022, 20:779-787.
[20] NIE M Z, WANG J Y, ZHANG K C.Engineering a novel acetyl-CoA pathway for efficient biosynthesis of acetyl-CoA-derived compounds[J].ACS Synthetic Biology, 2024, 13(1):358-369.
[21] SHA Y Y, GE M S, LU M R, et al.Advances in metabolic engineering for enhanced acetyl-CoA availability in yeast[J].Critical Reviews in Biotechnology, 2025,45(4):904-922.
[22] MA Y S, SHANG Y, STEPHANOPOULOS G.Engineering peroxisomal biosynthetic pathways for maximization of triterpene production in Yarrowia lipolytica[J].Proceedings of the National Academy of Sciences of the United States of America,2024, 121(5):e2314798121.
[23] GUO Q, YANG Y X, LI D X, et al.Advances in multi-enzyme co-localization strategies for the construction of microbial cell factory[J].Biotechnology Advances, 2024, 77:108453.
[24] BAKER J J, SHI J, WANG S Y, et al.ML-enhanced peroxisome capacity enables compartmentalization of multienzyme pathway[J].Nature Chemical Biology, 2025,21(5):727-735.
[25] YUE M Y, LIU M S, GAO S, et al.High-level de novo production of (2S)-eriodictyol in Yarrowia lipolytica by metabolic pathway and NADPH regeneration engineering[J].Journal of Agricultural and Food Chemistry, 2024, 72(8):4292-4300.
[26] PARK Y K, SELLÉS VIDAL L, BELL D, et al.Efficient synthesis of limonene production in Yarrowia lipolytica by combinatorial engineering strategies[J].Biotechnology for Biofuels and Bioproducts, 2024, 17(1):94.