为应对外界环境变化,细菌感知环境刺激后通过信号转导途径,将信号传递给效应分子,使其调节体内代谢过程以响应细胞外信号[1]。在信号传递过程中,分子小、易扩散的第二信使分子[2]如环二鸟苷酸(cyclic diguanosine monophosphate,c-di-GMP)和环二腺苷酸(cyclic diadenosine monophosphate,c-di-AMP)等环状二核苷酸(cyclic dinucleotides,CDG)(图1)起到重要作用[3-6]。ROSS等[7]研究木醋酸杆菌纤维素产生机制时首次发现c-di-GMP调控生物膜形成和毒力因子合成,c-di-GMP也被证明是细菌中一种通用且关键的细胞内第二信使[8]。c-di-AMP在研究海栖热袍菌DNA损伤机制过程中首次发现[9],细菌同样利用c-di-AMP进行信号传导[10],缺乏c-di-AMP将无法调控细胞稳态导致细菌死亡[3,11]。CDG在控制细菌细胞壁代谢、钾离子稳态、生物膜形成和DNA修复等生理功能中发挥关键作用[12-13]。
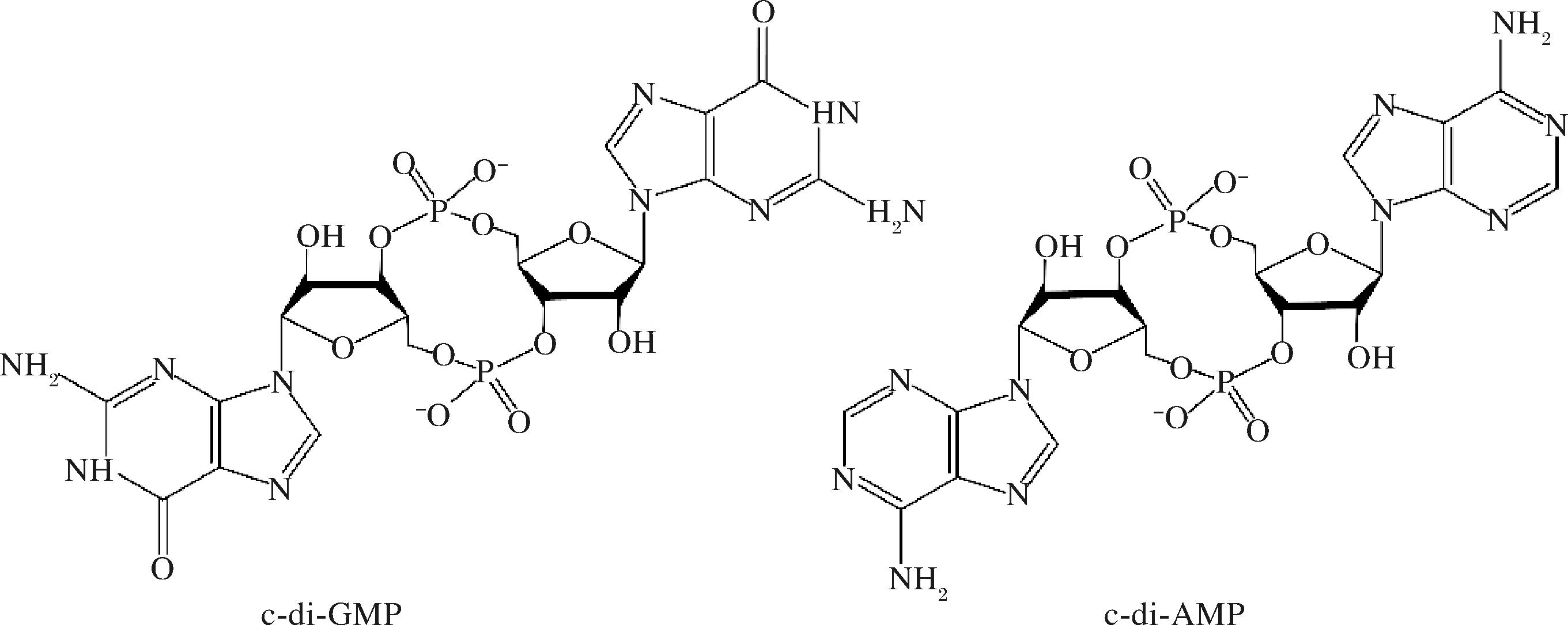
图1 环状二核苷酸
Fig.1 CDG
核糖开关通常位于mRNA 5′非翻译区(5′-untranslational region, 5′-UTR),是一类具有特定结构的非编码型RNA受体[14]。核糖开关折叠形成复杂的三级结构,主要由适配体结构域(aptamer domain)和表达平台结构域 (expression platform) 两部分构成,适配体结构域感知代谢物的浓度变化,引发下游的表达平台结构域的构象变化,进而调节下游基因的表达[15]。这种机制使mRNA可以在“开启”和“关闭”表达的状态之间转换[16]。核糖开关通常通过下游基因的转录终止或阻止核糖体结合mRNA以抑制翻译起始,从而影响微生物表型或生理功能[17]。另外,核糖开关也可以通过直接作用改变RNA的降解速率来控制RNA的代谢过程[18]。
第二信使分子主要通过向下游受体或靶标传导信号而发挥作用[17],除常见的第二信使结合蛋白外,核糖开关(表1)也能作为其信号分子的效应物[18]。核糖开关作为一种潜在调节机制,可进一步完善第二信使分子对mRNA的转录和翻译调控机制,为深入理解细菌全局代谢调控的复杂过程提供新的视角[19]。本文对响应第二信使分子CDG的核糖开关进行综述,系统分析这些核糖开关及其对基因表达调控作用,旨在进一步丰富细菌第二信使分子的信号调控网络。
表1 环状核苷酸核糖开关
Table 1 CDG riboswitch
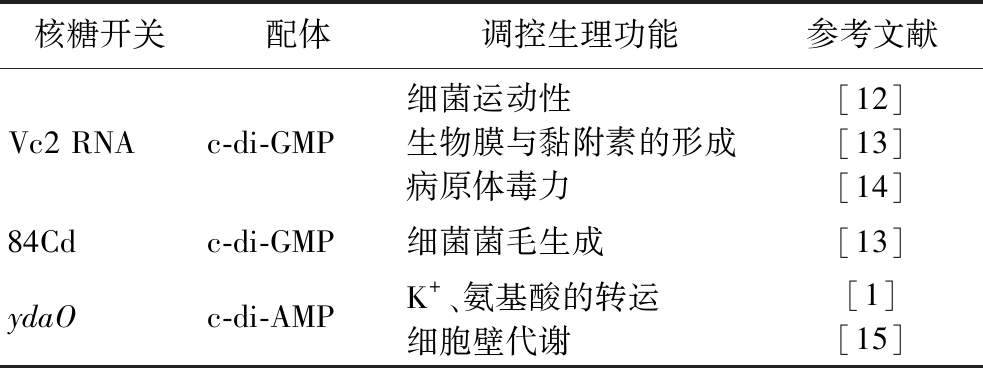
核糖开关配体调控生理功能参考文献Vc2 RNAc-di-GMP细菌运动性生物膜与黏附素的形成病原体毒力[12][13][14]84Cdc-di-GMP细菌菌毛生成[13]ydaOc-di-AMPK+、氨基酸的转运细胞壁代谢[1][15]
1 c-di-GMP核糖开关
c-di-GMP作为细菌中重要的第二信使,参与调节细菌的运动性、毒力基因表达、生物膜和胞外多糖形成及定殖和黏附等生理功能[8,20-21]。细胞内c-di-GMP稳态通过2种功能相反的酶调控(图2):含GGDEF结构域的二鸟苷酸环化酶(diguanylate cyclases, DGC)将2个GTP合成为c-di-GMP;含EAL或HDGYP结构域的磷酸二酯酶(phosphodiesterase, PDE)将c-di-GMP分解为2个GMP [22-23]。
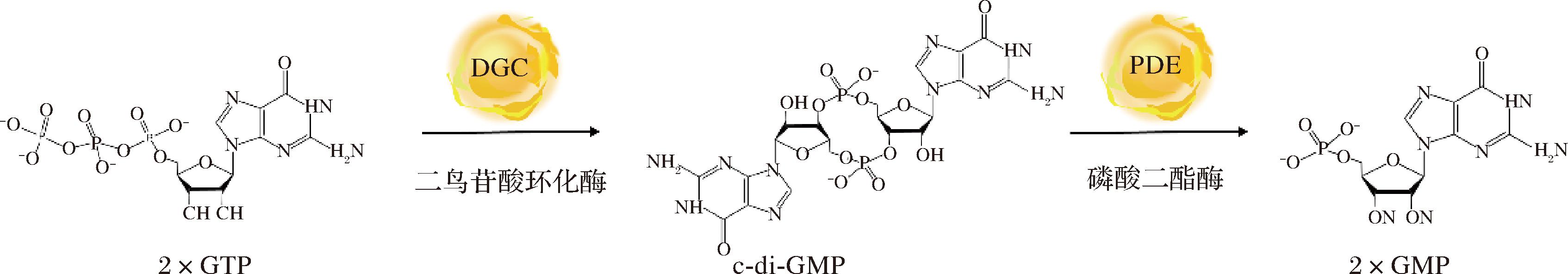
图2 c-di-GMP 稳态
Fig.2 c-di-GMP homeostasis
注:GTP(guanosine triphosphate),三磷酸鸟苷。
2008年SUDARSAN等[24]首次报道霍乱弧菌中c-di-GMP核糖开关,之后在艰难梭菌中发现另一种c-di-GMP核糖开关,并根据核糖开关二级结构及其作用机制的不同,分别命名为Ⅰ类和Ⅱ类c-di-GMP核糖开关[25](图3)。迄今为止,在炭疽杆菌、破伤风梭菌、艰难梭菌和霍乱弧菌等细菌中,通过计算模拟发现约500个Ⅰ类和45个Ⅱ类核糖开关,但目前只有霍乱弧菌Ⅰ类c-di-GMP核糖开关Vc2 RNA和丙酮丁醇梭菌Ⅱ类c-di-GMP核糖开关84 Cd的结构得到解析,也证实它们与c-di-GMP的相互作用机制与调节功能[19]。
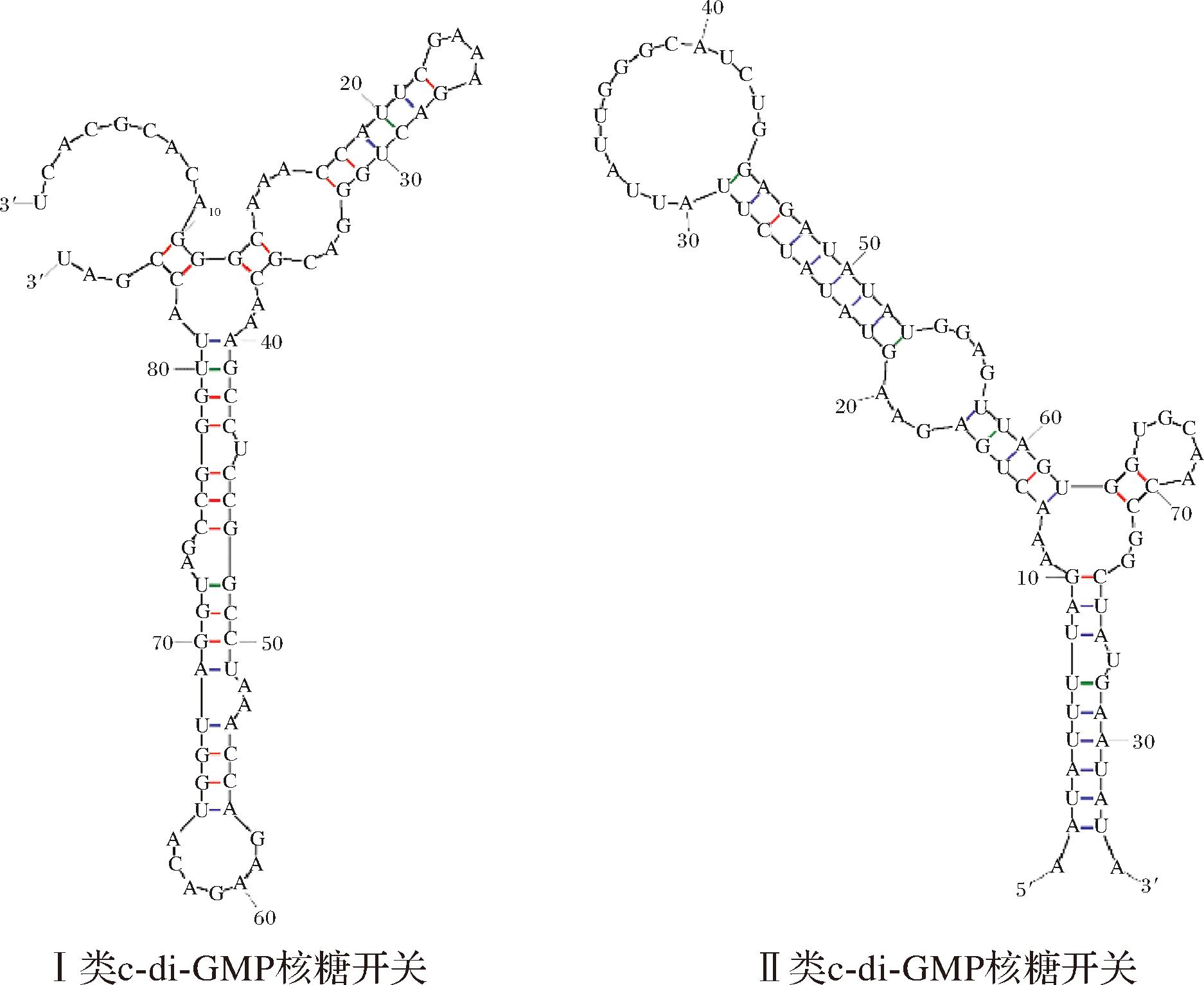
图3 c-di-GMP核糖开关二级结构模型
Fig.3 Secondary structure models of the c-di-GMP riboswitch
注:Ⅰ类序列源自霍乱弧菌B33;Ⅱ类序列源自艰难梭菌630。
1.1 Ⅰ类c-di-GMP核糖开关结构与调节机制
WEINBERG[26]等发现GEMM(the Genes for the Environment, for Membranes, and for Motility)基序能感应细胞代谢物并调节基因表达。SUDARSAN等[24]报道霍乱弧菌tfoX基因上游GEMM序列[27]是一种c-di-GMP核糖开关(并命名为Vc2 RNA),与c-di-GMP的解离常数(KD)约为1 nmol/L,而c-di-GMP的分解产物pGpG和pG(5′ GMP)等c-di-GMP结构类似物不会与Vc2 RNA结合,证实Vc2 RNA能够感知并特异性结合c-di-GMP。SMITH等[17]通过多波长反常色散(multi-wavelength anomalous dispersion,MAD)首次解析Vc2 RNA与c-di-GMP结合的2.7 Å分辨率晶体结构。Vc2 RNA由3个成对螺旋片段P1、P2和P3构成配体识别口袋,P2末端的四元环与P3中四元环受体通过氢键连接保证结构稳定[15,17]。Vc2 RNA以非对称方式识别c-di-GMP,c-di-GMP最上面的鸟苷Gα与P2 5’端第1个未配对的核苷酸G20通过Hoogsteen配对接触,第2个鸟苷Gβ与P3中高度保守的3′核苷酸C92形成标准的沃森-克里克碱基配对,核糖开关的第47位碱基A插在二者之间,辅助P1形成稳定的螺旋结构[17,28]。WOOD等[29]使用单分子荧光共振能量转移(single molecule fluorescence resonance energy transfer,smFRET)技术发现,Vc2 RNA在c-di-GMP存在时才会形成并排结构,四元环与受体以结合的形式存在。另外,有研究者在破伤风梭菌中发现一种新的Ⅰ类c-di-GMP核糖开关Ct-E88 RNA,其结构与Vc2 RNA相似。2.5 mmol/L Mg2+可将Vc2 RNA结构从32 Å压缩至28.5 Å,将c-di-GMP压缩至23.9 Å,而Mg2+对Ct-E88 RNA没有明显作用。小角X射线散射(small angle X-ray scattering,SAXS)和核磁共振(nuclear magnetic resonance,NMR)结果表明,c-di-GMP配体比Mg2+更关键的诱导Ct-E88 RNA折叠和二级结构形成[19]。
Ⅰ类c-di-GMP核糖开关主要在转录层面实现调控作用,其机制涉及终止子或抗终止子的形成[15]。c-di-GMP浓度升高时,Vc2 RNA是促进基因表达的“开启”型开关[27]。c-di-GMP结合Vc2 RNA时,c-di-GMP的一个鸟苷作为螺旋的第一个碱基对嵌入双链体结构元件中,将RNA的3’端锁定为特定构象,触发P1螺旋的形成[30]。P1螺旋是Vc2 RNA表达平台的基本组成部分,c-di-GMP因此能够实现对基因表达的直接控制[17]。但INUZUKA等[31]发现大肠杆菌中Vc2 RNA是“关闭”型开关,在翻译水平上负调控下游基因。
1.2 Ⅱ类c-di-GMP核糖开关结构与调节机制
LEE等[25]在艰难梭菌中首次发现Ⅱ类c-di-GMP核糖开关,根据其含有84个核苷酸命名为84 Cd,与c-di-GMP的KD值约为 200 pmol/L。SMITH等[30]通过单一同晶置换与异常散射(single isomorphous replacement with anomalous scattering,SIRAS)对丙酮丁醇梭菌中Ⅱ类c-di-GMP核糖开关进行2.5 Å分辨率晶体结构解析。核糖开关由4个螺旋状的发卡结构P1、P2、P3和P4组成,折叠成紧密的二级结构,包括螺旋、扭结和假结。假结由P3和P1、P2形成,大多数的保守核苷酸位于假结及其邻近区域[15,30]。假结的独特构造使Ⅱ类c-di-GMP核糖开关能够结合c-di-GMP的鸟苷,并通过独特的凹槽反转结构促进结合,形成稳定的非规范碱基三联体[30]。Ⅱ类c-di-GMP核糖开关与c-di-GMP的特异性识别取决于碱基堆积作用力和碱基非典型配对方式[32]。
Ⅱ类c-di-GMP核糖开关84 Cd也是一种受到c-di-GMP调控的变构核酶。84 Cd位于其相关毒力基因开放阅读框(open reading frame,ORF)的预测起始密码子上游约600个核苷酸处,包含一个Ⅰ组自剪接核酶[15,32]。84 Cd发挥作用需要GTP共同参与mRNA前体的剪接过程,通过自剪接核酶剪接位点的结构变化,控制核糖体结合位点(ribosomebinding site,RBS)的形成,从而在翻译起始水平调控基因表达(图4)[28,32]。在前体RNA中,起始密码子UUG的位置不利于核糖体的结合和翻译的进行。在缺乏c-di-GMP情况下,GTP会攻击起始密码子上游4个核苷酸,导致前体RNA裂解,无法形成完整的RBS,阻止下游基因的翻译[28,32]。84Cd感知并结合第二信使c-di-GMP后发生结构重排,形成抗5’剪接位点(5′ SS)结构,阻止GTP对该位点的攻击。此时,RNA自剪接后的mRNA中起始密码子被暴露出来,其上游也有一个完美距离的RBS序列,使得核糖体可以结合RBS并从起始密码子UUG开启蛋白翻译。
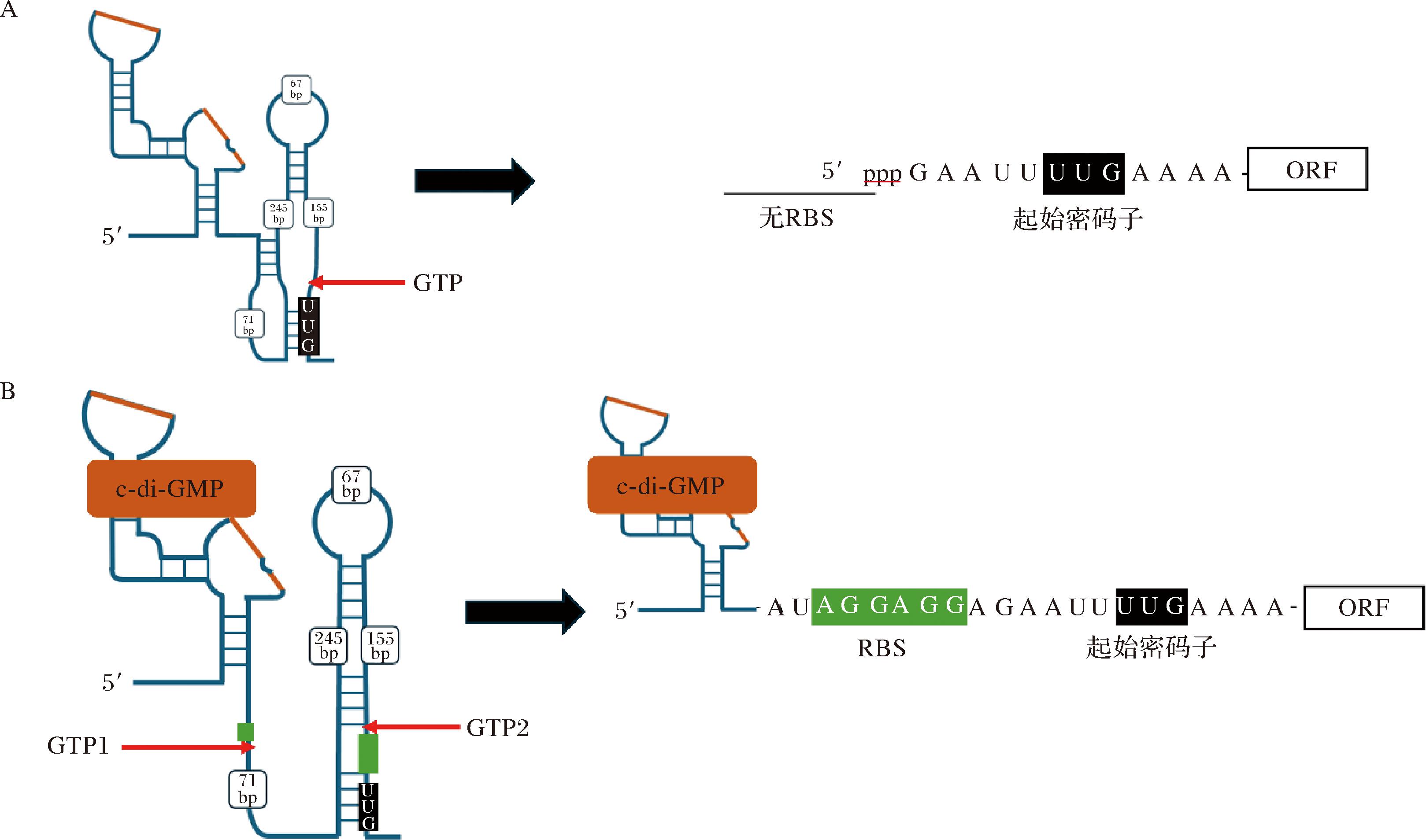
A-无c-di-GMP时GTP攻击前体mRNA;B-c-di-GMP结合84Cd后发生变构,GTP剪切前体mRNA
图4 Ⅱ类c-di-GMP核糖开关的作用机制
Fig.4 Mechanism of action of class Ⅱ c-di-GMP riboswitches
A-Ⅰ类核糖开关;B-Ⅱ类核糖开关
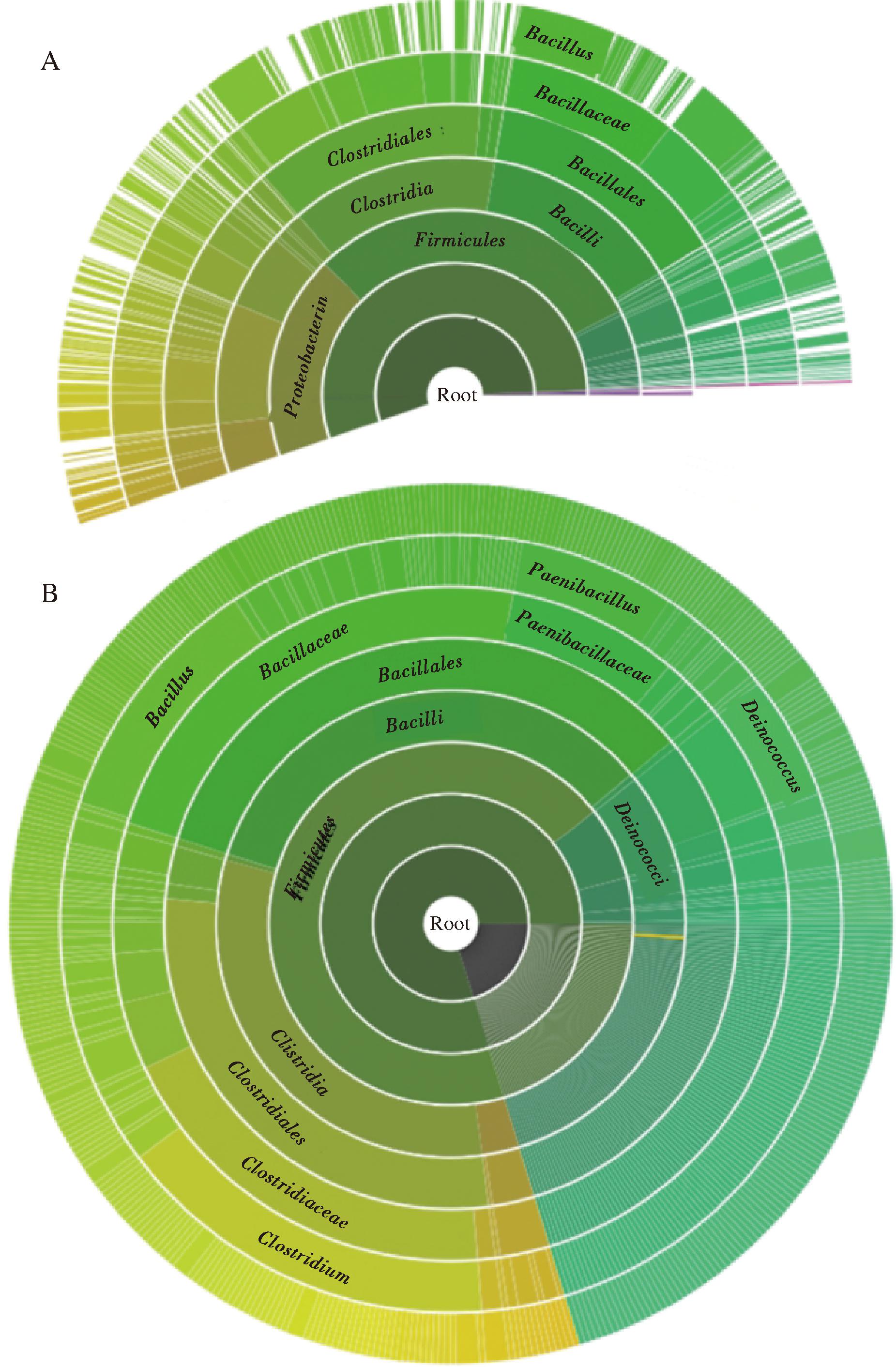
图5 c-di-GMP 核糖开关物种分布
Fig.5 c-di-GMP riboswitch species distribution models
注:数据来源于Rfam数据库。
1.3 c-di-GMP核糖开关对细菌生理功能的调控
c-di-GMP核糖开关在细菌生物膜形成与黏附和细菌运动性等方面发挥关键作用[33-34],如参与新月柄杆菌的细胞形态变化、沙门氏菌对免疫应答的抗性[17,35],以及霍乱弧菌、铜绿假单胞菌和伯氏疏螺旋体等人类病原体毒力基因的调节[36-37](图5)。
1.3.1 调控生物膜的形成和细菌黏附性
c-di-GMP核糖开关在艰难梭菌定植过程起关键作用[38],尤其是调控与表面结构生成相关的基因表达,这些核糖开关在艰难梭菌中大部分高度保守,但也有一些在特定菌株中缺失,可能存在其他替代的调控机制[20,39]。Ⅰ类核糖开关通常响应c-di-GMP起到“关闭”开关的作用,负向调控编码鞭毛、钙结合推定黏附素和锌依赖性金属蛋白酶ZmpⅠ等基因表达[20,40-41]。相反,Ⅱ类核糖开关则作为“开启”开关,正向调控编码细菌表面蛋白及具有胶原结合结构域的黏附素基因表达[42],由ZmpⅠ金属蛋白酶切割,而ZmpⅠ酶受c-di-GMP负向调节。此外,Ⅳ型菌毛(TFP)与黏附上皮细胞和肠道定植有关[20],Ⅱ类核糖开关还控制编码TFP[42]。
1.3.2 调控细菌的运动性
c-di-GMP调控细菌的运动行为以适应复杂多变的生存环境[43-44]。c-di-GMP能抑制霍乱弧菌单极鞭毛驱动的游泳运动[45],同时削弱转录激活因子FlrA的功能、促进弧菌多糖产生,导致细菌运动性下降[46]。PURSLEY等[33]发现转录因子TfoY在低浓度(1.1 μmol/L)c-di-GMP条件下被诱导,促进细菌从密集状态转变为分散状态,而在高浓度(13 μmol/L)c-di-GMP下,Vc2 RNA作为“关闭”开关下调TfoY的表达,抑制分散运动。另外,由于tfoY上游的基因间区有不同的4个转录起始位点,中等(5.2 μmol/L)和高浓度(13 μmol/L)c-di-GMP会使转录产物不包含完整的Vc2 RNA序列,使TfoY不受c-di-GMP负调控。
2 c-di-AMP核糖开关
c-di-AMP是在c-di-GMP之后发现的一种新型第二信使,其生理功能主要包括调节细胞壁合成、维持渗透压平衡、控制细胞内钾离子稳态、促进生物膜形成和参与DNA修复过程[22-23,47-48]。与c-di-GMP相似,c-di-AMP细胞内浓度由二腺苷酸环化酶(diadenylate cyclase,DAC)和PDE控制形成动态平衡(图6),DAC催化ATP或ADP合成c-di-AMP,而PDE将其水解为pApA或AMP[49]。
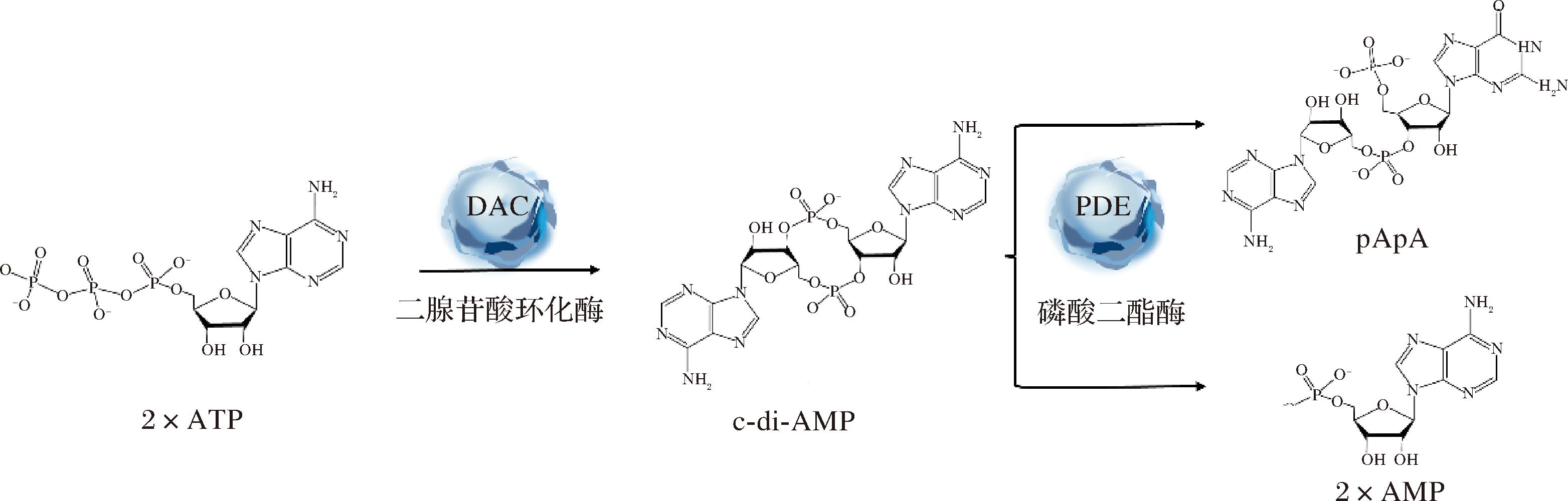
图6 c-di-AMP 稳态
Fig.6 c-di-AMP homeostasis
SMITH等[14]通过诱变研究率先发现存在c-di-AMP核糖开关,双突变体核糖开关G20A/C92U既能识别c-di-GMP也能识别c-di-AMP,且该突变体对c-di-AMP的偏好程度是c-di-GMP的4倍,推测c-di-AMP可以通过核糖开关实现对基因表达的调控作用[3,50]。
BARRICK等[51]在枯草芽孢杆菌中发现具有核糖开关功能的RNA元件ydaO(图7),BLOCK等[52]证实其能够作为一种遗传调节机制调控基因的表达,但其特异性配体并未被鉴定。直到2013年,NELSON等[53]证实ydaO能够特异性响应第二信使c-di-AMP发挥调控作用。
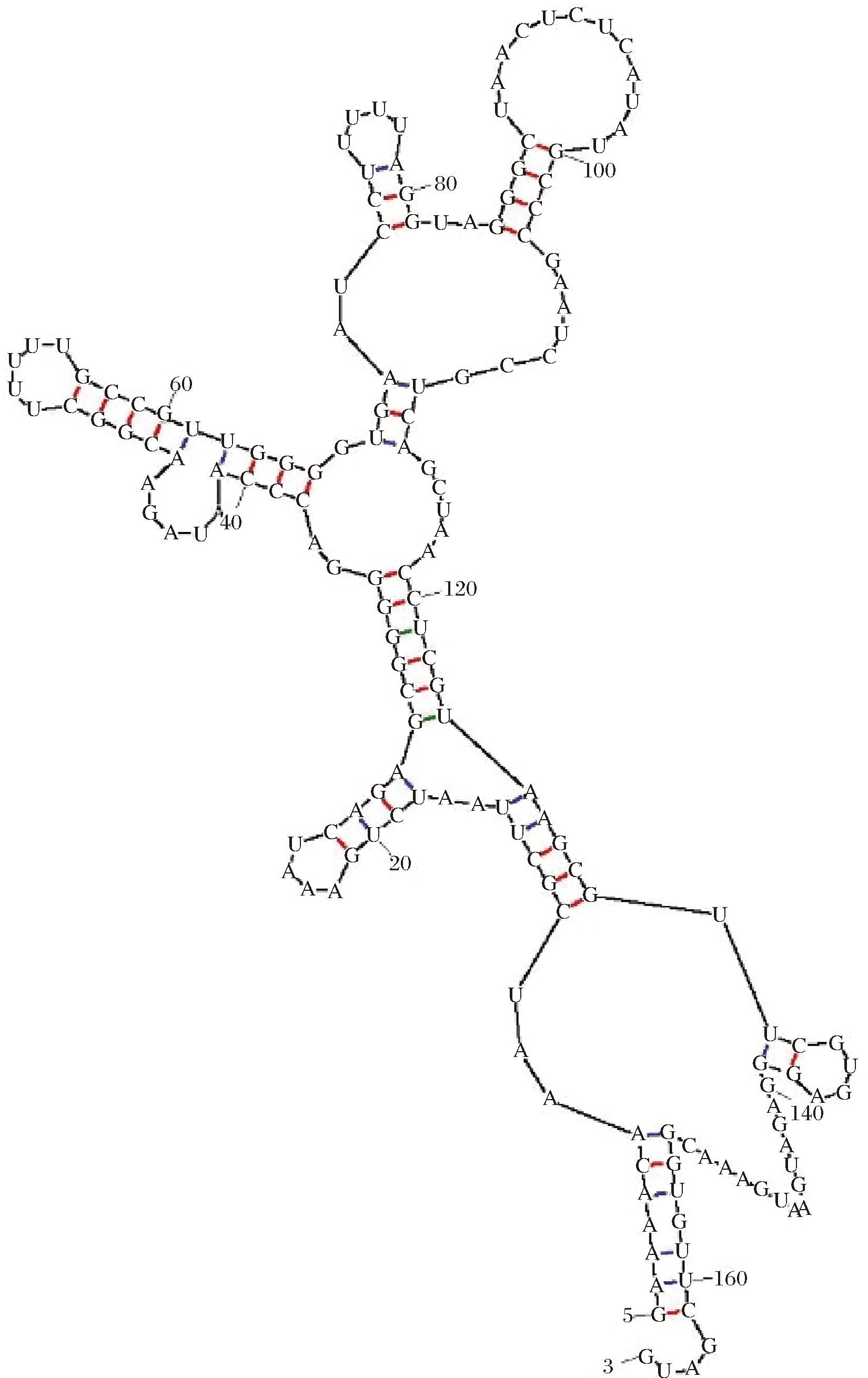
图7 c-di-AMP 核糖开关ydaO二级结构模型
Fig.7 Secondary structure model of the ydaO riboswitch
2.1 c-di-AMP核糖开关结构与调节机制
JONES等[54]测定枯草芽孢杆菌c-di-AMP核糖开关与c-di-AMP结合的共晶体结构,发现此核糖开关是一个能够容纳2个c-di-AMP分子的假对称RNA,结合c-di-AMP引起核糖开关的整体折叠变化,从而调控基因表达。GAO等[55]在3.0 Å分辨率解析拟乙醇热厌氧杆菌和林氏热弧菌c-di-AMP核糖开关晶体结构,发现它们具有高度一致的“正方形”结构,c-di-AMP以延伸构象与RNA相互作用,增强核糖开关的稳定性。另外,配体和RNA之间的碱基堆积作用有助于RNA元件的稳定性,配体与核糖开关螺旋间的氢键对二者结合亲和力和特异性也起作用,共同确保核糖开关对c-di-AMP的特异性识别[55]。ydaO核糖开关具有2个离散的结合口袋,2个c-di-AMP分子分别结合P4-P5和P3(位点1)和P1和P7-P8(位点2)[55]。MEEHAN等[47]发现ydaO核糖开关的2个位点几乎以相同的亲和力识别结合c-di-AMP,4个 腺嘌呤与保守的沃森-克里克G-C对接触,形成Ⅰ型A-次要相互作用(type-Ⅰ A-minor interactions),有助于特异性识别。
2.2 c-di-AMP核糖开关对细菌生理功能的调控
ydaO核糖开关是目前唯一已知的c-di-AMP特异性核糖开关,仅存在于厚壁菌门中能合成c-di-AMP的芽孢杆菌属和梭菌属,主要调控参与离子和氨基酸转运及细胞壁代谢的基因表达[3,56-57] (图8)。目前对ydaO核糖开关的基因调控机制研究较少,枯草芽孢杆菌ydaO核糖开关位于编码钾离子转运蛋白KtrAB和编码高亲和力钾离子转运蛋白的kimA转录本内[58]。ydaO核糖开关结合c-di-AMP导致转录终止,使受其控制的基因表达下调,起到基因“关闭”开关的作用。细胞外高浓度K+会增加细胞内c-di-AMP,进而抑制钾转运系统中核糖开关控制的基因表达[53]。WANG等[59]也发现苏云金芽孢杆菌中c-di-AMP直接促进核糖开关的转录终止,从而抑制操纵子kdpFABC转录。
图8 c-di-AMP 核糖开关物种分布
Fig.8 c-di-AMP riboswitch species distribution models
注:数据来源于Rfam数据库。
3 结语与展望
c-di-GMP和c-di-AMP等CDG核糖开关研究揭示细菌复杂的基因调控机制,为理解细胞信号传导机制提供新的视角。这些第二信使分子通过核糖开关在细菌应对外界环境变化、病原菌致病性等生理功能中发挥重要作用,精确调控细菌的生理过程。尽管在CDG核糖开关的结构及其调节机制等方面研究取得显著进展,但仍然存在较多局限性,特别是核糖开关结构和功能的多样性仍需进一步研究。目前仅少数几个核糖开关的三维结构被解析,不同菌属的同一类核糖开关可能具有不同的折叠模式和识别位点,这些多样性对其功能的发挥是否有影响尚未明确。核糖开关与其他调控网络的相互作用及其在复杂环境中的动态行为也需要进一步探究。高通量测序技术和基于人工智能的大数据分析有助于在全基因组水平快速筛选和识别不同菌属中新的核糖开关。在结构解析方面,冷冻电镜和X射线晶体学等技术的快速发展将有助更深入地理解核糖开关的动态结构基础,高分辨率的核糖开关三级结构研究也将有助于揭示其与CDG结合的分子机制。此外,在开发和改造方面,合成生物学技术可用于设计新型核糖开关,实现对特定基因表达的精确控制或对疾病的精准诊断与治疗。例如,DÜCHS等[60]设计的四环素依赖性核糖开关K19,能够通过响应四环素类药物来激活或抑制基因表达,通过腺病毒载体靶向递送至肝细胞,K19核糖开关精确调控白细胞介素IL-12的表达,有效治疗了小鼠的原位肝肿瘤。改造核糖开关也可用于进一步增强有益菌在肠道的定植能力、提升其抗逆性或增强其特定的益生功能。系统生物学和计算生物学方法可辅助构建核糖开关的调控网络模型,预测核糖开关在不同环境条件下的功能及其对细菌生理过程的影响,为开发新型抗菌策略提供理论基础。总之,深入解析更多种属细菌中未被表征的CDG核糖开关,能够深入理解细菌如何感知并适应环境变化,加深对第二信使分子介导的细菌信号调控网络的认识。
[1] BRIDGES A A, PRENTICE J A, WINGREEN N S, et al.Signal transduction network principles underlying bacterial collective behaviors[J].Annual Review of Microbiology, 2022, 76:235-257.
[2] HENGGE R, PRUTEANU M, STÜLKE J, et al.Recent advances and perspectives in nucleotide second messenger signaling in bacteria[J].microLife, 2023, 4:uqad015.
[3] COMMICHAU F M, DICKMANNS A, GUNDLACH J, et al.A jack of all trades:The multiple roles of the unique essential second messenger cyclic di-AMP[J].Molecular Microbiology, 2015, 97(2):189-204.
[4] DA PURIFICAÇ O A D, DE AZEVEDO N M, DE ARAUJO G G, et al.The world of cyclic dinucleotides in bacterial behavior[J].Molecules, 2020, 25(10):2462.
O A D, DE AZEVEDO N M, DE ARAUJO G G, et al.The world of cyclic dinucleotides in bacterial behavior[J].Molecules, 2020, 25(10):2462.
[5] LU Z Z, FU Y Q, ZHOU X Y, et al.Cyclic dinucleotides mediate bacterial immunity by dinucleotide cyclase in Vibrio[J].Frontiers in Microbiology, 2022, 13:1065945.
[6] LOWEY B, KRANZUSCH P J.CD-NTases and nucleotide second messenger signaling[J].Current Biology, 2020, 30(19):R1106-R1108.
[7] ROSS P, WEINHOUSE H, ALONI Y, et al.Regulation of cellulose synthesis in Acetobacter xylinum by cyclic diguanylic acid[J].Nature, 1987, 325(6101):279-281.
[8] RÖMLING U, GALPERIN M Y, GOMELSKY M.Cyclic di-GMP:The first 25 years of a universal bacterial second messenger[J].Microbiology and Molecular Biology Reviews,2013, 77(1):1-52.
[9] WITTE G, HARTUNG S, BÜTTNER K, et al.Structural biochemistry of a bacterial checkpoint protein reveals diadenylate cyclase activity regulated by DNA recombination intermediates[J].Molecular Cell, 2008, 30(2):167-178.
[10] KALIA D, MEREY G, NAKAYAMA S, et al.Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis[J].Chemical Society Reviews, 2013, 42(1):305-341.
[11] HERZBERG C, MEIßNER J, WARNEKE R, et al.The many roles of cyclic di-AMP to control the physiology of Bacillus subtilis[J].microLife, 2023, 4:uqad043.
[12] YAN H B, CHEN W X.The promise and challenges of cyclic dinucleotides as molecular adjuvants for vaccine development[J].Vaccines, 2021, 9(8):917.
[13] CANCINO-DIAZ M E, GUERRERO-BARAJAS C, BETANZOS-CABRERA G, et al.Nucleotides as bacterial second messengers[J].Molecules, 2023, 28(24):7996.
[14] SMITH K D, STROBEL S A.Interactions of the c-di-GMP riboswitch with its second messenger ligand[J].Biochemical Society Transactions, 2011, 39(2):647-651.
[15] 李新风, 陈芳, 肖金凤, 等.环二鸟苷单磷酸核糖开关的结构与功能[J].生物工程学报, 2017, 33(9):1357-1368.LI X F, CHEN F, XIAO J F, et al.Structure and function of c-di-GMP riboswitches[J].Chinese Journal of Biotechnology, 2017, 33(9):1357-1368
[16] GARST A D, BATEY R T.A switch in time:Detailing the life of a riboswitch[J].Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2009, 1789(9-10):584-591.
[17] SMITH K D, LIPCHOCK S V, AMES T D, et al.Structural basis of ligand binding by a c-di-GMP riboswitch[J].Nature Structural &Molecular Biology, 2009, 16(12):1218-1223.
[18] RICHARDS J, BELASCO J G.Riboswitch control of bacterial RNA stability[J].Molecular Microbiology, 2021, 116(2):361-365.
[19] LUO Y L, CHEN B, ZHOU J, et al.E88, a new cyclic-di-GMP class I riboswitch aptamer from Clostridium tetani, has a similar fold to the prototypical class I riboswitch, Vc2, but differentially binds to c-di-GMP analogs[J].Molecular BioSystems, 2014, 10(3):384-390.
[20] MCKEE R W, HARVEST C K, TAMAYO R.Cyclic diguanylate regulates virulence factor genes via multiple riboswitches in Clostridium difficile[J].mSphere, 2018, 3(5):e00423-18.
[21] HENGGE R, GRÜNDLING A, JENAL U, et al.Bacterial signal transduction by cyclic di-GMP and other nucleotide second messengers[J].Journal of Bacteriology, 2016, 198(1):15-26.
[22] XIONG Z Q, FAN Y Z, SONG X, et al.The second messenger c-di-AMP mediates bacterial exopolysaccharide biosynthesis:A review[J].Molecular Biology Reports, 2020, 47(11):9149-9157.
[23] JENAL U, REINDERS A, LORI C.Cyclic di-GMP:Second messenger extraordinaire[J].Nature Reviews Microbiology, 2017, 15(5):271-284.
[24] SUDARSAN N, LEE E R, WEINBERG Z, et al.Riboswitches in eubacteria sense the second messenger cyclic di-GMP[J].Science, 2008, 321(5887):411-413.
[25] LEE E R, BAKER J L, WEINBERG Z, et al.An allosteric self-splicing ribozyme triggered by a bacterial second messenger[J].Science, 2010, 329(5993):845-848.
[26] WEINBERG Z, NELSON J W, LÜNSE C E, et al.Bioinformatic analysis of riboswitch structures uncovers variant classes with altered ligand specificity[J].Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(11):E2077-E2085
[27] MEIBOM K L, BLOKESCH M, DOLGANOV N A, et al.Chitin induces natural competence in Vibrio cholerae[J].Science, 2005, 310(5755):1824-1827.
[28] KULSHINA N, BAIRD N J, FERRÉ-D’AMARÉ A R.Recognition of the bacterial second messenger cyclic diguanylate by its cognate riboswitch[J].Nature Structural &Molecular Biology, 2009, 16(12):1212-1217.
[29] WOOD S, FERRÉ-D’AMARÉ A R, RUEDA D.Allosteric tertiary interactions preorganize the c-di-GMP riboswitch and accelerate ligand binding[J].ACS Chemical Biology, 2012, 7(5):920-927.
[30] SMITH K D, SHANAHAN C A, MOORE E L, et al.Structural basis of differential ligand recognition by two classes of bis-(3’-5’)-cyclic dimeric guanosine monophosphate-binding riboswitches[J].Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(19):7757-7762.
[31] INUZUKA S, NISHIMURA K I, KAKIZAWA H, et al.Mutational analysis of structural elements in a class-I cyclic di-GMP riboswitch to elucidate its regulatory mechanism[J].Journal of Biochemistry, 2016, 160(3):153-162.
[32] CHEN A G Y, SUDARSAN N, BREAKER R R.Mechanism for gene control by a natural allosteric group Ⅰ ribozyme[J].RNA, 2011, 17(11):1967-1972.
[33] PURSLEY B R, MAIDEN M M, HSIEH M L, et al.Cyclic di-GMP regulates TfoY in Vibrio cholerae to control motility by both transcriptional and posttranscriptional mechanisms[J].Journal of Bacteriology, 2018, 200(7):e00578-17.
[34] LIAO H B, YAN X D, WANG C Y, et al.Cyclic di-GMP as an antitoxin regulates bacterial genome stability and antibiotic persistence in biofilms[J].eLife, 2024, 13:RP99194.
[35] HWANG Y, HARSHEY R M.A second role for the second messenger cyclic-di-GMP in E.coli:Arresting cell growth by altering metabolic flow[J].mBio, 2023, 14(2):e00619-23.
[36] BORDELEAU E, PURCELL E B, LAFONTAINE D A, et al.Cyclic di-GMP riboswitch-regulated type IV pili contribute to aggregation of Clostridium difficile[J].Journal of Bacteriology, 2015, 197(5):819-832.
[37] ANGELONI J, DONG Y Q, WANG Z N, et al.Bacterial second messenger 3′, 5′-cyclic diguanylate attracts Caenorhabditis elegans and suppresses its immunity[J].Communications Biology, 2020, 3:700.
[38] HOMMA M, KOJIMA S.Roles of the second messenger c-di-GMP in bacteria:Focusing on the topics of flagellar regulation and Vibrio spp[J].Genes to Cells, 2022, 27(3):157-172.
[39] PURCELL E B.Second messenger signaling in Clostridioides difficile[J].Current Opinion in Microbiology, 2022, 65:138-144.
[40] CAFARDI V, BIAGINI M, MARTINELLI M, et al.Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins[J].PLoS One, 2013, 8(11):e81306.
[41] BARKETI-KLAI A, HOYS S, LAMBERT-BORDES S, et al.Role of fibronectin-binding protein A in Clostridium difficile intestinal colonization[J].Journal of Medical Microbiology, 2011, 60(8):1155-1161.
[42] HENSBERGEN P J, KLYCHNIKOV O I, BAKKER D, et al.Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831[J].FEBS Letters, 2015, 589(24 PartB):3952-3958.
[43] SOUTOURINA O A, MONOT M, BOUDRY P, et al.Genome-wide identification of regulatory RNAs in the human pathogen Clostridium difficile[J].PLoS Genetics, 2013, 9(5):e1003493.
[44] PURCELL E B, TAMAYO R.Cyclic diguanylate signaling in Gram-positive bacteria[J].FEMS Microbiology Reviews, 2016, 40(5):753-773.
[45] PURCELL E B, MCKEE R W, MCBRIDE S M, et al.Cyclic diguanylate inversely regulates motility and aggregation in Clostridium difficile[J].Journal of Bacteriology, 2012, 194(13):3307-3316.
[46] SRIVASTAVA D, HSIEH M L, KHATAOKAR A, et al.Cyclic di-GMP inhibits Vibrio cholerae motility by repressing induction of transcription and inducing extracellular polysaccharide production[J].Molecular Microbiology, 2013, 90(6):1262-1276.
[47] MEEHAN R E, TORGERSON C D, GAFFNEY B L, et al.Nuclease-resistant c-di-AMP derivatives that differentially recognize RNA and protein receptors[J].Biochemistry, 2016, 55(6):837-849.
[48] CORRIGAN R M, GRÜNDLING A.Cyclic di-AMP:Another second messenger enters the fray[J].Nature Reviews Microbiology, 2013, 11(8):513-524.
[49] MUDGAL S, MANIKANDAN K, MUKHERJEE A, et al.Cyclic di-AMP:Small molecule with big roles in bacteria[J].Microbial Pathogenesis, 2021, 161:105264.
[50] HE J, YIN W, GALPERIN M Y, et al.Cyclic di-AMP, a second messenger of primary importance:Tertiary structures and binding mechanisms[J].Nucleic Acids Research, 2020, 48(6):2807-2829.
[51] BARRICK J E, CORBINO K A, WINKLER W C, et al.New RNA motifs suggest an expanded scope for riboswitches in bacterial genetic control[J].Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(17):6421-6426.
[52] BLOCK K F, HAMMOND M C, BREAKER R R.Evidence for widespread gene control function by the ydaO Riboswitch candidate[J].Journal of Bacteriology, 2010, 192(15):3983-3989.
[53] NELSON J W, SUDARSAN N, FURUKAWA K, et al.Riboswitches in eubacteria sense the second messenger c-di-AMP[J].Nature Chemical Biology, 2013, 9(12):834-839.
[54] JONES C P, FERRÉ-D’AMARÉ A R.Crystal structure of a c-di-AMP riboswitch reveals an internally pseudo-dimeric RNA[J].The EMBO Journal, 2014, 33(22):2692-2703.
[55] GAO A, SERGANOV A.Structural insights into recognition of c-di-AMP by the ydaO riboswitch[J].Nature Chemical Biology, 2014, 10(9):787-792.
[56] ZARRELLA T M, BAI G C.The many roles of the bacterial second messenger cyclic di-AMP in adapting to stress cues[J].Journal of Bacteriology, 2020, 203(1):e00348-20.
[57] FAHMI T, PORT G, CHO K.C-di-AMP:An essential molecule in the signaling pathways that regulate the viability and virulence of gram-positive bacteria[J].Genes, 2017, 8(8):197.
[58] STÜLKE J, KRÜGER L.Cyclic di-AMP signaling in bacteria[J].Annual Review of Microbiology, 2020, 74:159-179.
[59] WANG X, CAI X, MA H D, et al.A c-di-AMP riboswitch controlling kdpFABC operon transcription regulates the potassium transporter system in Bacillus thuringiensis[J].Communications Biology, 2019, 2:151.
[60] DÜCHS M J, KRATZER R F, VIEYRA-GARCIA P, et al.Riboswitch-controlled IL-12 gene therapy reduces hepatocellular cancer in mice[J].Frontiers in Immunology, 2024, 15:1360063.