豆瓣酱和黄豆酱等固态豆类发酵食品作为人们熟知的佐味调料品而广受关注,其以豆类和面粉为原料,经多种微生物协同发酵而成。由于上述发酵食品具有丰富的营养、独特的口感和特殊的风味,深受消费者喜爱[1]。然而,在包装和市场流通阶段,部分豆瓣酱和黄豆酱产品中会出现乳白色小颗粒,即白点物质,严重影响消费者的感官,导致豆瓣酱销售的下降[2-3]。研究者们采用了一系列微生物及化学方法,证明该颗粒的主要成分以游离酪氨酸为主,结合游离苯丙氨酸、富含酪氨酸和苯丙氨酸的短肽及少部分其他有机物,其中酪氨酸在游离氨基酸中约占87%,在水解氨基酸中约占95%[4-6]。上述氨基酸及其短肽均为合成蛋白质的基本氨基酸,对人体无害,但其形成的颗粒口感和外观仍给产品的品质和销售造成一定的困扰[7]。因此,生产企业和研究者近年来采取了一系列方法来控制白点物质的形成,主要包括改良发酵工艺及优化发酵菌株2种方式。闵世豪等[4]通过发酵模拟实验,发现当酪氨酸含量低于0.45 mg/L时豆酱中不会出现酪氨酸结晶;进一步在30 ℃、10%盐度下进行发酵能在保证蛋白质分解水平的同时尽可能减少酪氨酸含量。朱洪康[8]通过对毛霉中酪氨酸羧肽酶酶学性质的研究,证明可以在pH值低于5.66的条件下进行发酵,可以在不影响酶活力的前提下使白点减少50%。陈执[9]筛选出一株酶系缺陷型米曲霉,通过减少大豆蛋白的酶解,降低发酵豆酱中游离酪氨酸含量,进而有效抑制了白点的产生。NIU等[10]筛获得一株能稳定产生少量中性蛋白酶和酸性蛋白酶的米曲霉,在解决白点问题的同时保证了产品品质。
微生物群落是豆瓣酱和黄豆酱等固态豆类发酵食品的发酵及风味产生的关键。不仅有以米曲霉为主要代表的霉菌,还有以埃切假丝酵母为代表的酵母菌,以乳酸菌、杆菌为代表的细菌等[11-13]。这些微生物在发酵过程中发挥不同作用:霉菌与成曲制作和原料利用间关系密切,因其具有丰富的蛋白酶系,可以分解蛋白质、淀粉等大分子原料为后续微生物的生长提供营养基础[14]。邢先雷等[15]通过筛选强化获得一株耐盐的埃切假丝酵母,显著提升了酱醅的风味,证明酵母菌具有发酵产醇类、酯类等香气物质的能力。乳酸菌在豆瓣酱发酵过程中产生有机酸等物质,既能调节pH促进酵母增殖、抑制杂菌生长又能丰富酱醅口感[16]。枯草芽孢杆菌具有与曲霉相似的蛋白质水解能力,被美国食品药监局和中国农业部认定为是非致病性、有良好的蛋白酶分泌能力和发酵基础的菌株[17-19],钟杰[20]通过转录组测序、基因敲除等手段验证了枯草芽孢杆菌对酱香、豉香的风味贡献。因此,通过微生物筛选强化方式,尝试解决白点问题具有可行性。
常压室温等离子体诱变(atmospheric and room temperature plasma, ARTP)以高纯氦气在高频电场中放电产生等离子体为诱变工具,研究表明,氦气作为最小的单原子分子,其放电时所释放出来的等离子体中高能化学活性粒子能量是普通等离子体放电技术的4~5倍,可以对菌株/植物细胞产生多重作用,DNA物质损伤后细胞启动SOS修复机制,SOS修复为一种高容错率修复,在修复过程中会产生种类丰富的错配位点,并最终稳定遗传进而形成突变株。该技术较化学诱变等传统诱变技术具有突变率高、遗传性状稳定、操作简便无毒等优点,配合后期筛选便可得到性状优良的突变株[21-22]。
基于目前白点物质对于豆瓣酱和黄豆酱等豆类发酵食品的困扰,本研究旨在筛选得到具有较强酪氨酸转化能力的微生物菌株,并通过ARTP的方法进一步强化其相关能力,最终在豆瓣酱发酵过程中进行应用。本论文得到的结果能够加深对豆瓣酱发酵过程中酪氨酸代谢的理解,同时获得的较强酪氨酸转化能力的微生物菌株为豆瓣酱白点物质的消除提供了潜在的应用策略。
1 材料与方法
1.1 材料与试剂
豆瓣酱酱醅来自安徽某著名豆瓣酱生产企业;白酒酒醅来自四川某著名白酒生产企业。
酪氨酸筛选培养基 (g/L):葡萄糖1、L-酪氨酸2、氯化钠10、氯化钙0.1,固体加入2%(质量分数)的琼脂粉,115 ℃灭菌15 min。
筛选培养基 (g/L):葡萄糖1、蛋白胨5、L-酪氨酸2、氯化钠10、氯化钙0.1,固体加入2%的琼脂粉,115 ℃灭菌15 min。
含盐筛选培养基 (g/L):葡萄糖1、蛋白胨5、L-酪氨酸2、氯化钠10、氯化钙0.1,按照要求分别添加4%、8%、12%(质量分数)的食盐,固体加入2%的琼脂粉,115 ℃灭菌15 min。
种子培养基 (g/L):氯化钠70、蛋白胨10、酵母粉5,固体加入1.5%~2%的琼脂粉,121 ℃灭菌20 min。
发酵培养基 (g/L):氯化钠70、蛋白胨10、酵母粉5、L-酪氨酸0.5,固体加入1.5%~2%的琼脂粉,121 ℃灭菌20 min。
葡萄糖、氯化钠、氯化钙、琼脂粉、L-酪氨酸、丙三醇、碳酸钠、福林酚标准溶液,国药集团化学试剂有限公司(中国上海);细菌总RNA 抽提试剂盒、酵母粉、蛋白胨,生工生物工程(上海)股份有限公司;TB Green逆转录试剂盒,宝日医生物技术公司。
1.2 仪器与设备
SW-CJ-2FD无菌超净工作台,苏净集团安泰公司;HVE-50全自动高压蒸汽灭菌锅,日本HIRAYAMA株式会社;AL107电子天平,梅特勒-托多仪器有限公司;BSP-250生化培养箱,上海博讯公司;Micro CL 17高速离心机,赛默飞世尔科技(中国)有限公司;UV-2000紫外可见分光光度计,UNICO(上海)仪器有限公司;ARTP-Ⅱ型ARTP诱变育种仪,无锡源清天木生物科技有限公司;GENTIER 96 Real-Time PCR仪,西安天隆科技有限公司。
1.3 实验方法
1.3.1 出发菌株筛选
取豆瓣酱酱醅及白酒酒醅样品各5 g,分别加入装有25 mL无菌生理盐水的锥形瓶中,28 ℃、180 r/min,洗涤1 h。吸取1 mL上清液,用无菌生理盐水梯度稀释至10-5、10-6、10-7浓度,取50 μL均匀涂布于酪氨酸筛选培养基平板中,28 ℃培养24 h后观察平板中的菌落生长情况。挑选菌落形态不同的单菌落,于无盐筛选培养基平板中重新划线,28 ℃培养24 h后观察菌落生长状态。挑取单菌落转接至液体无盐筛选培养基中,28 ℃,180 r/min培养48 h,分光法测培养基中酪氨酸含量变化。将酪氨酸转化率较高的菌株重新划线于筛选培养基斜面,28 ℃培养12 h后,置4 ℃冰箱内保藏。实验过程中每组均设有2个平行。
1.3.2 菌株耐盐性测试
按照1.1节中的方法,配制盐度梯度4、8、12 g/L的含盐筛选培养基,取筛选菌株的保藏斜面,于超净台中,分别挑取筛选得到的出发菌株接种至0 g/L盐度培养基,28 ℃、180 r/min活化24 h。将活化后菌液以1% (体积分数)接种量接种于0~12 g/L盐度梯度培养基,每组均设2个平行。28 ℃、180 r/min培养80 h,期间每隔8 h取样,于600 nm检测菌液吸光值,并以时间为横坐标,OD600值为纵坐标绘制生长曲线。选取耐盐性最好的菌株为出发菌株,菌液保藏于-80 ℃冰箱中。
1.3.3 菌种活化
将保藏于-80 ℃冰箱的菌株甘油管取出,室温融化后,于超净台中以1% (体积分数)接种量接种至种子培养基,37 ℃、180 r/min活化12 h后转接。
1.3.4 菌悬液制备
将活化后的菌株R1以1% (体积分数)接种量接种于装有30 mL种子培养基的锥形瓶中,37 ℃、180 r/min培养12 h至对数期,取1 mL菌液10 000 r/min离心1 min收集菌体,无菌生理盐水重悬,洗涤2次后弃上清液,加1 mL 10%(体积分数)甘油重悬菌体并逐级稀释至106~108 CFU/mL,即制成菌悬液。
1.3.5 诱变致死曲线测定
取菌悬液10 μL均匀涂布于无菌金属载片表面,ARTP仪舱内经紫外照射处理10~15 min后迅速放置载片并关闭舱门。以He为工作气体,入射功率100 W,照射距离2 mm,气流量10 SLM,对菌悬液进行处理。处理时长分别为10、20、30、40 s,处理完成的金属载片置于加有1 mL无菌生理盐水的离心管中振荡洗脱,洗脱液稀释至10-2进行涂布,每处理时长均设2个平行,于37 ℃培养箱中培养12 h后进行菌落计数。根据公式(1)计算致死率并绘制致死曲线,取致死率80%~90%为诱变处理时间进行诱变实验。若出现致死率变化较大的情况,则重新选择时间区间设置处理时长,重复诱变步骤。
致死率![]()
(1)
1.3.6 诱变菌株初筛
诱变后菌液稀释至10-2,取100 μL均匀涂布于筛选培养基平板,每处理时长设置2个平行,置37 ℃培养箱中培养12 h后,观察培养基中菌落生长情况。
1.3.7 诱变菌株复筛
挑选步骤1.2.4节筛选培养平板中生长的单菌落接入发酵培养基,37 ℃、180 r/min培养48 h,计算发酵液中剩余酪氨酸含量,并计算酪氨酸的转化率,以此进行复筛。
酪氨酸含量测定参考GB/T 23527.1—2023 《酶制剂质量要求 第1部分:蛋白酶制剂》的方法:配制质量浓度0、0.125、0.025、0.05、0.1、0.2 g/L的L-酪氨酸标准溶液,取1 mL标准溶液于具塞比色管中,加入5 mL碳酸钠溶液、1 mL福林工作溶液,振荡混匀,于(40±0.2) ℃恒温水浴锅中反应20 min。分光光度计检测波长设为680 nm,以0管为空白进行标零后,测定各标准浓度溶液吸光值,并绘制标准曲线,获得标准曲线[公式(2)],每组均设2个平行。复筛时,取2 mL菌液于离心管中,10 000 r/min离心2 min,取上清液参考上述步骤测得OD680值,根据公式(2)计算发酵液中酪氨酸含量,再根据公式(3)计算酪氨酸转化率,每个菌株均设2个平行。
酪氨酸含量![]()
(2)
酪氨酸转化率/%
(3)
1.3.8 诱变菌株遗传稳定性测试
选择发酵液中酪氨酸含量降低幅度较大的突变株,以1% (体积分数)接种量接种于新鲜发酵培养基内,每48 h进行一次酪氨酸含量检测及传代,连续进行50次传代培养,观察突变菌株的遗传稳定性。
1.3.9 酶基因相对表达量分析
基于实验室前期工作中获得的基因组数据,分析R1中编码酶的基因序列及其对应酶编号,结合KEGG数据库,确认R1中酪氨酸代谢路径酶基因。使用SnapGene软件联合NCBI数据库BLAST分析,获得具有特异性的基因引物。采用实时荧光定量PCR (quantitative real-time PCR, qRT-PCR)技术,以16 s基因为内参,通过测定酶基因相对表达量。将菌株R1在种子培养基中培养至稳定期,提取细菌细胞内总RNA,经逆转录获得cDNA,以cDNA为模板对内参基因及酶基因进行qRT-PCR。使用两步法进行扩增,第一步,预变性:95 ℃,30 s;第二步,扩增:95 ℃,5 s,60 ℃,30 s 设置40个循环,溶解曲线分析步骤采用仪器默认程序。按照2(-ΔΔCT)法计算酶基因相对表达量,具体方法参考文献[23],本实验所用引物信息列于表1。
表1 qRT-PCR引物信息
Table 1 Information of qRT-PCR primers

引物名称序列 (5′-3′)16 s-RACTCCTACGGGAGGCAGCAG16 s-FATTACCGCGGCTGCTGGAST-RGGAAGGTGCTTTTTACTTGTTCCCAST-FCGCTTGATTCTTTCAATGGCTTPPO-FGGTTTCAGCAGACCTTGTCGPPO-RCGATAAAGGACATCATCCGTCC
1.3.10 豆瓣酱发酵实验
将成曲(由某豆瓣酱厂提供)、水以1∶1.5的质量比混合均匀后,加盐至酱醅终盐度为8 g/L,于37 ℃、湿度75%的培养箱中培养3 d,每天搅拌1次。菌株R1与优选菌株于种子培养基中培养至对数期,离心收集菌体,稀释至OD600值为1,于发酵第3天时按5%体积分数分别接种至酱醅,置于自然条件下发酵。发酵第1周每天翻拌1次,之后每周翻拌1次,期间每7 d进行一次取样。
2 结果与分析
2.1 具备酪氨酸转化能力的微生物菌株筛选
2.1.1 微生物菌株筛选
以豆瓣酱酱醅、白酒酒醅等传统发酵食品过程样品为筛选源,通过酪氨酸为唯一氮源的筛选培养基进行涂布培养,共获得4个微生物菌落。为了进一步获得能够在有机氮源存在条件下能够利用酪氨酸的微生物,进一步将上述4个微生物菌株划线于筛选培养基,经过28 ℃培养24 h后,发现上述4株微生物在无盐筛选培养基中均能够生长。通过微生物鉴定,它们分别为盔形毕赤酵母(Pichia manshurica) M1、毛壳菌(Chaetomium globosum) N1、球形赖氨酸芽胞杆菌(Lysinibacillus sphaericus) S1和枯草芽孢杆菌(Bacillus subtilis) R1。在此基础上,测定了4个微生物菌株在培养前后培养基中的酪氨酸含量(表2)。结果发现,盔形毕赤酵母M1对酪氨酸的转化率为39.3%,毛壳菌N1对酪氨酸的转化率为35.9%,球形赖氨酸芽胞杆菌S1对酪氨酸的转化率为46.4%,而枯草芽孢杆菌R1对酪氨酸的转化率为35.1%。由此可见,上述4株微生物菌株均能在有机氮源存在的情况下降低培养基中的酪氨酸含量。
表2 筛选菌株酪氨酸转化率
Table 2 Tyrosine conversion rates of selected strains
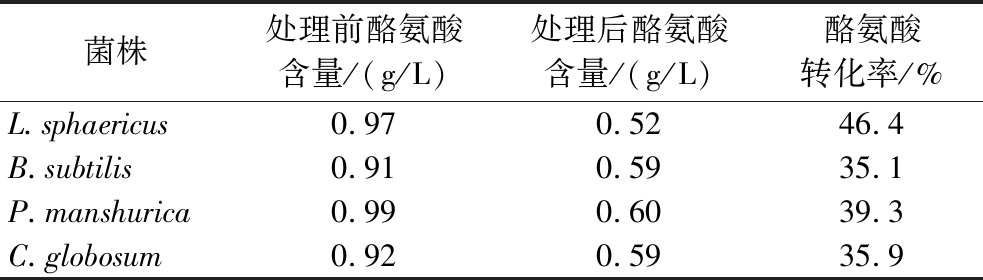
菌株处理前酪氨酸含量/(g/L)处理后酪氨酸含量/(g/L)酪氨酸转化率/%L.sphaericus0.970.5246.4B.subtilis0.910.5935.1P.manshurica0.990.6039.3C.globosum0.920.5935.9
2.1.2 微生物菌株的耐盐性分析
由于传统发酵食品生产过程中通常涉及较高的盐度,因此考察了4个微生物菌株的盐度耐受性。传统发酵豆瓣酱中盐度常在10~16 g/L水平。杨悦等[24]通过实验证明在8.5 g/L的盐度条件下进行发酵,能够较好地平衡豆瓣酱风味与口感,并限制有害微生物生长。因此,本实验考察了菌株在0、4、8、12 g/L盐度下的耐受能力。
如图1所示,4株微生物菌株在0、4 g/L盐度培养基中均具有较好的生长能力。但是当盐度提升至8 g/L或12 g/L时,盔形毕赤酵母M1和毛壳菌N1的生长受到抑制,几乎无法生长。球形赖氨酸芽胞杆菌S1和枯草芽孢杆菌R1的生长在8 g/L和12 g/L盐度培养基中同样受到抑制,但是仍具有一定生长能力。在8 g/L和12 g/L盐度条件下培养80 h后,枯草芽孢杆菌R1溶液的OD600值分别为0.860和0.232,均高于球形赖氨酸芽胞杆菌S1(0.154和0.048)。上述结果说明,枯草芽孢杆菌R1的盐度耐受性优于球形赖氨酸芽胞杆菌S1。因此,选择枯草芽孢杆菌R1进行后续实验。此外,根据其生长曲线可知,R1在不同盐度下培养48 h左右均可进入稳定期,故后续选择培养48 h后进行检测。
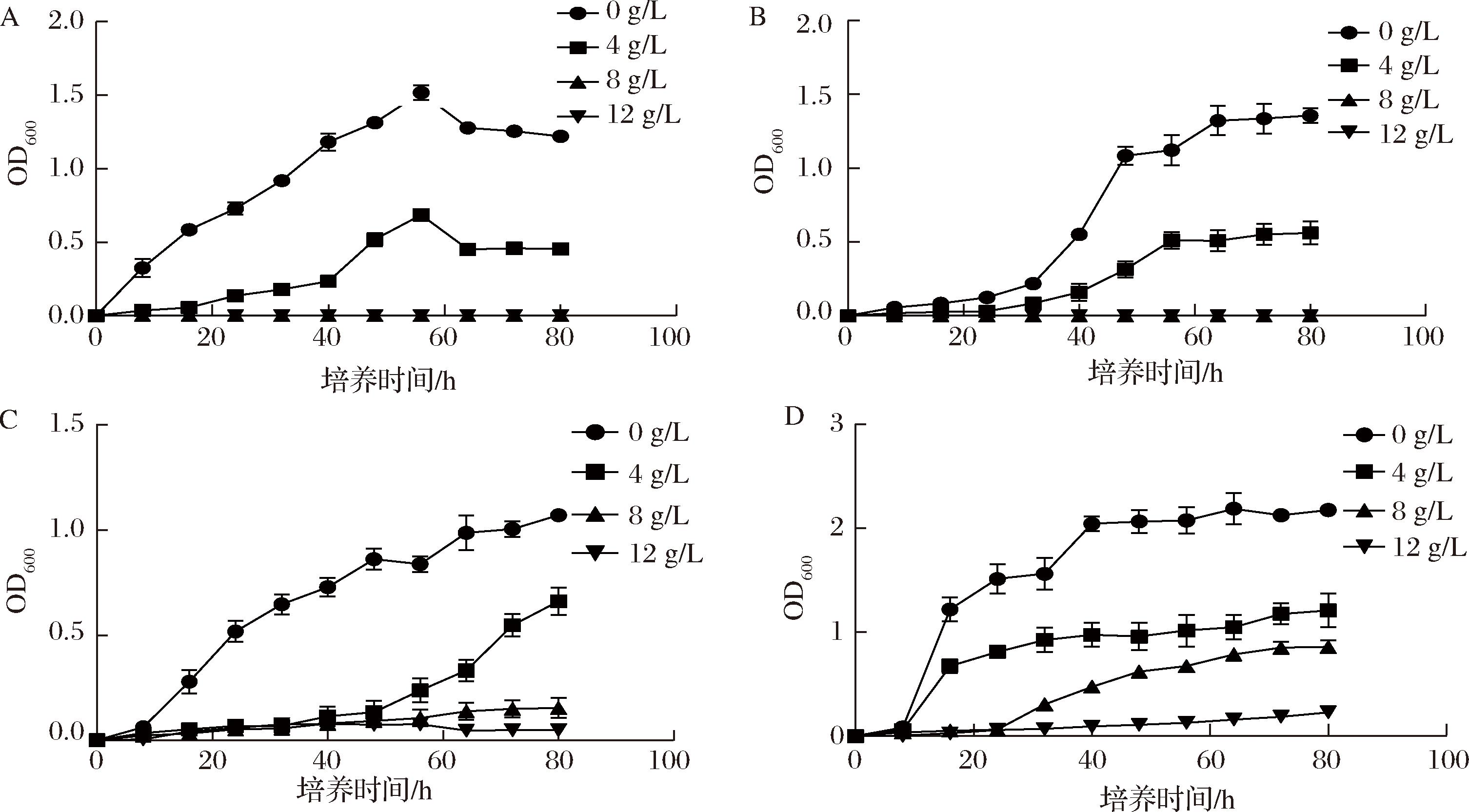
A-盔形毕赤酵母M1;B-毛壳菌N1;C-球形赖氨酸芽胞杆菌S1;D-枯草芽孢杆菌R1
图1 筛选菌株在不同盐度下的生长曲线
Fig.1 Growth curves of screening strains in different salinities
2.1.3 枯草芽孢杆菌R1在不同盐度下的酪氨酸转化率分析
基于1.1节中含盐筛选培养基方案,配制不同盐度培养基(0、4、8、12 g/L),测定了枯草芽孢杆菌R1在不同盐度培养基条件下的酪氨酸转化率。如图2所示,枯草芽孢杆菌R1对于酪氨酸的转化率随着盐度的提高逐渐降低。该菌株在8 g/L盐度下能保持13.01%的转化率,但是当盐度提高至12 g/L时其完全丧失酪氨酸转化活性。尽管目前豆瓣酱盐度的盐度依旧超过12 g/L,但是通过功能微生物强化的方法已经能够实现8%~10%盐度豆瓣酱的成功发酵[24]。因此,本实验将选取8 g/L盐度进行后续实验。
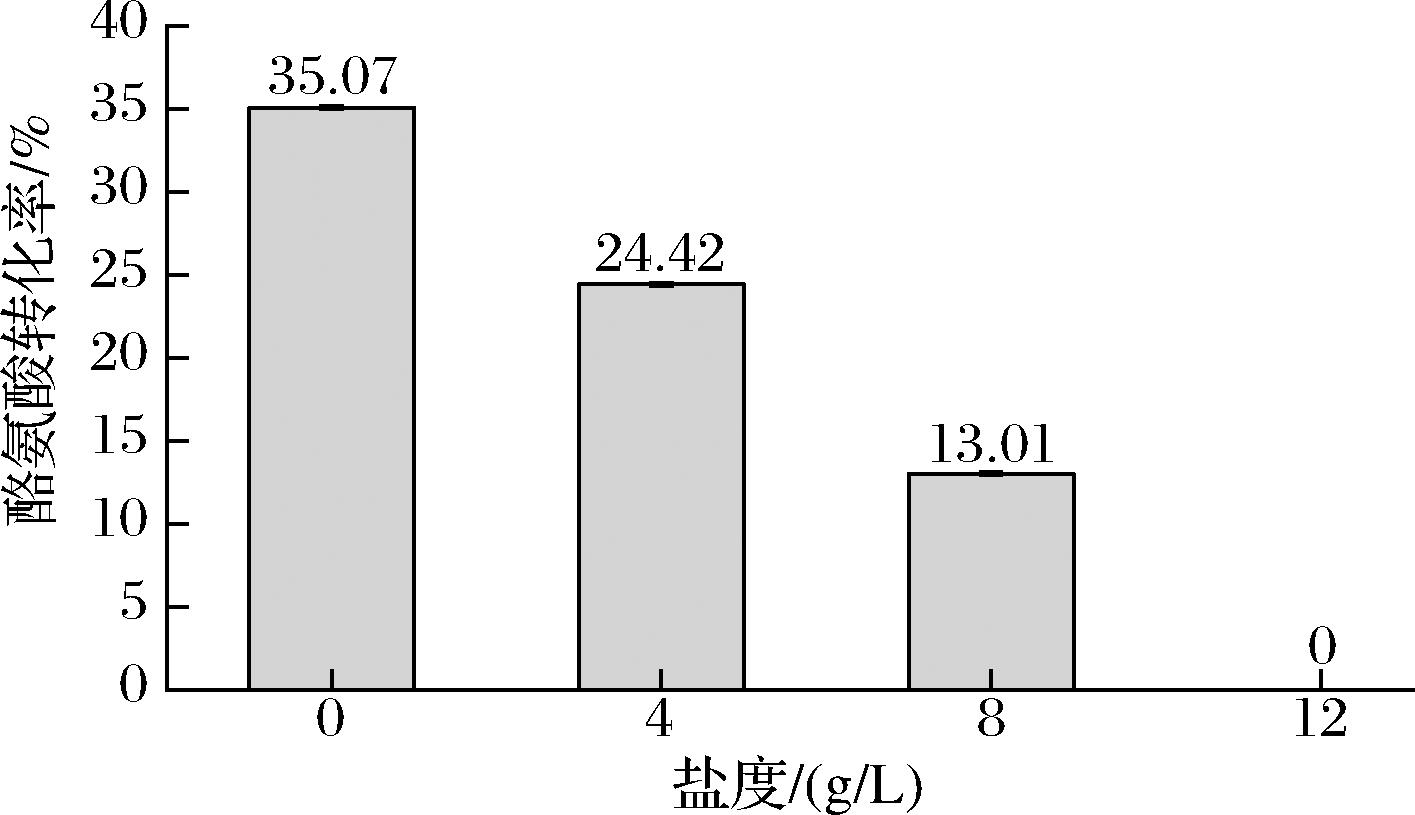
图2 枯草芽孢杆菌R1在不同盐度下的酪氨酸转化率
Fig.2 Tyrosine conversion rates of B.subtilis R1 with different salinities
2.2 枯草芽孢杆菌R1的ARTP诱变
致死率是ARTP诱变条件设定时的关键参考指标。有学者研究发现:致死率处于80%~90%的处理时长是一个较优条件,易获得较理想的突变效果[25]。本研究中,预实验时选择10 s作为处理间隔,于10~20 s出现100%致死点。因此,进一步考察了12~20 s处理后的菌株致死率。如图3所示,处理12、13、14、15 s后枯草芽孢杆菌R1的致死率分别为55%、76%、83%和92%。因此,选择处理14 s为ARTP较优处理时间,并对枯草芽孢杆菌R1进行诱变实验。
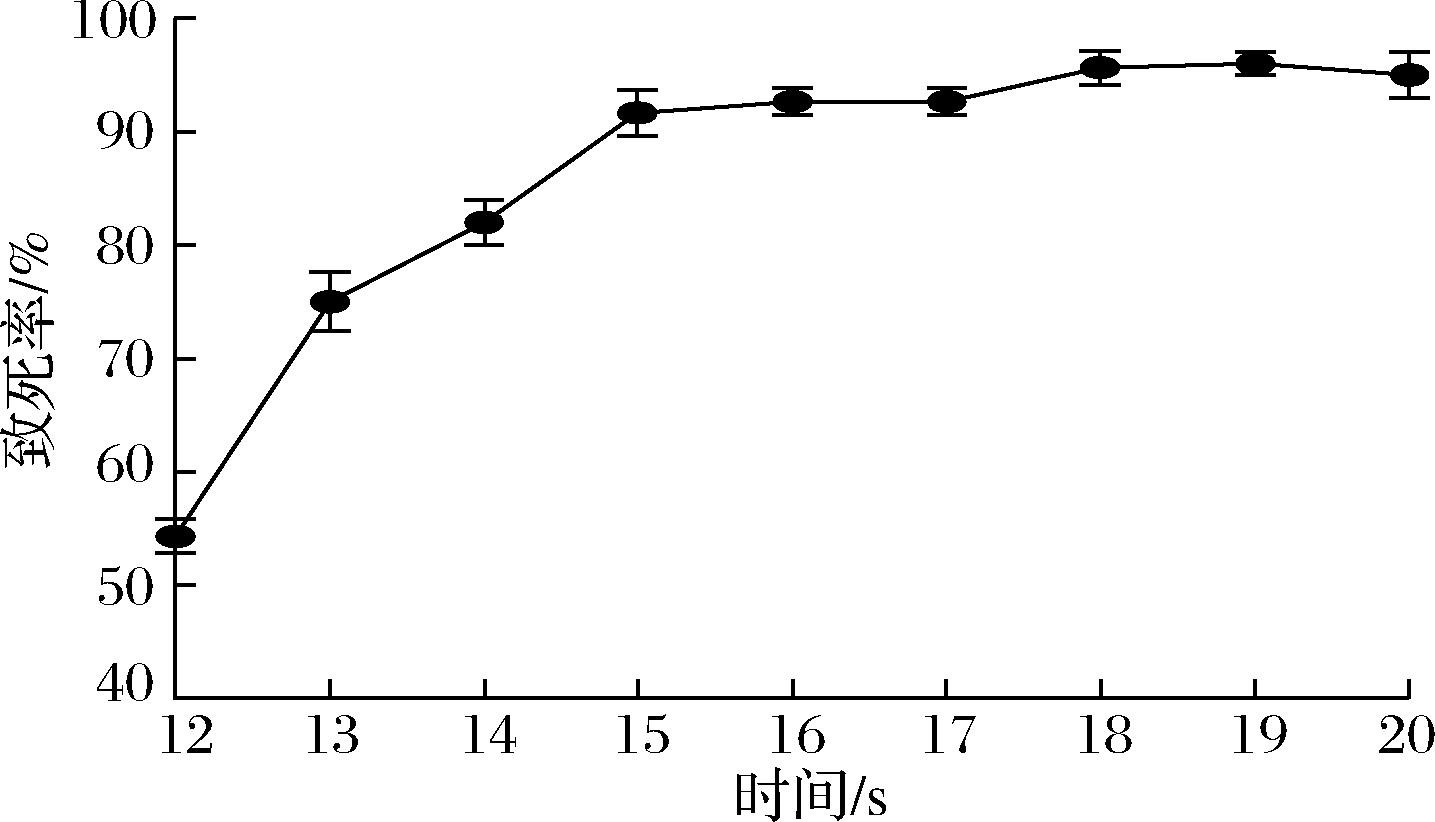
图3 枯草芽孢杆菌R1在不同ARTP处理时间后的致死曲线
Fig.3 Inactivation curves of B.subtilis R1 during ARTP treatment for different time
2.3 诱变菌株的筛选
2.3.1 诱变菌株的初筛
通过含8 g/L盐度的筛选培养基进行诱变菌株的初筛,旨在筛选在有机氮源存在条件下具备酪氨酸利用能力且具有一定耐盐能力的微生物菌株。推测在筛选培养基中长势良好的菌株可能对酪氨酸有较好的利用能力。因此,以出发菌株为对照,挑选筛选培养平板中菌落直径较大的单菌落。ARTP诱变实验共进行了6个批次,共获得233株突变菌株。通过初筛,共有52株诱变菌的菌落大小大于出发菌R1菌株,因此选择该52个菌株进入复筛。初筛突变菌的分布情况如表3所示。
表3 初筛突变菌株分布
Table 3 Distribution of mutant strains in first-round screening
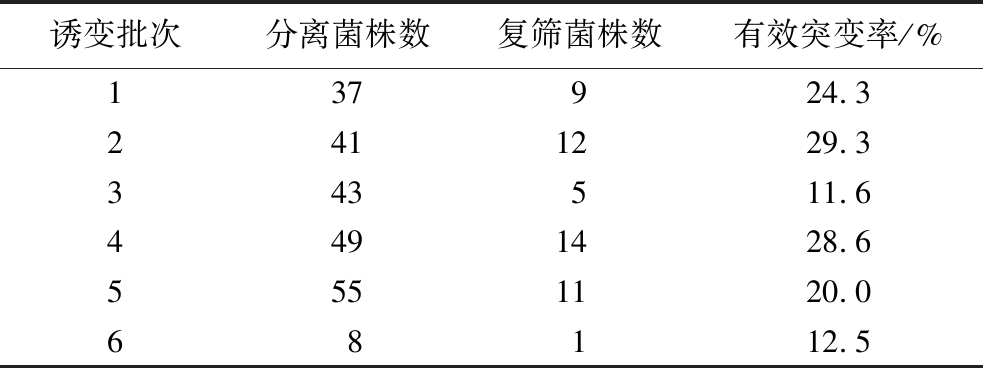
诱变批次分离菌株数复筛菌株数有效突变率/%137924.32411229.3343511.64491428.65551120.068112.5
2.3.2 诱变菌株的复筛
将初筛获得的51个菌株接种进入发酵培养基,测定了发酵前后培养基中的酪氨酸含量,计算并比较了其酪氨酸转化率。如图4所示,在所有诱变菌中,30株诱变菌为正突变,即发酵液中酪氨酸含量减少,其中21株诱变菌的酪氨酸转化率高于R1(14.1%)。在所有正突变中,14号菌株酪氨酸转化率最高,达到33.0%;48号和26号菌株次之,均为31.0%;28号菌株第三,为28.9%;其他22株诱变菌为负突变,即其发酵液中酪氨酸含量增加。因此,选择将具有较高酪氨酸转化率的14、26、28、48号诱变菌株进行平板划线,作为筛选的优选菌株。
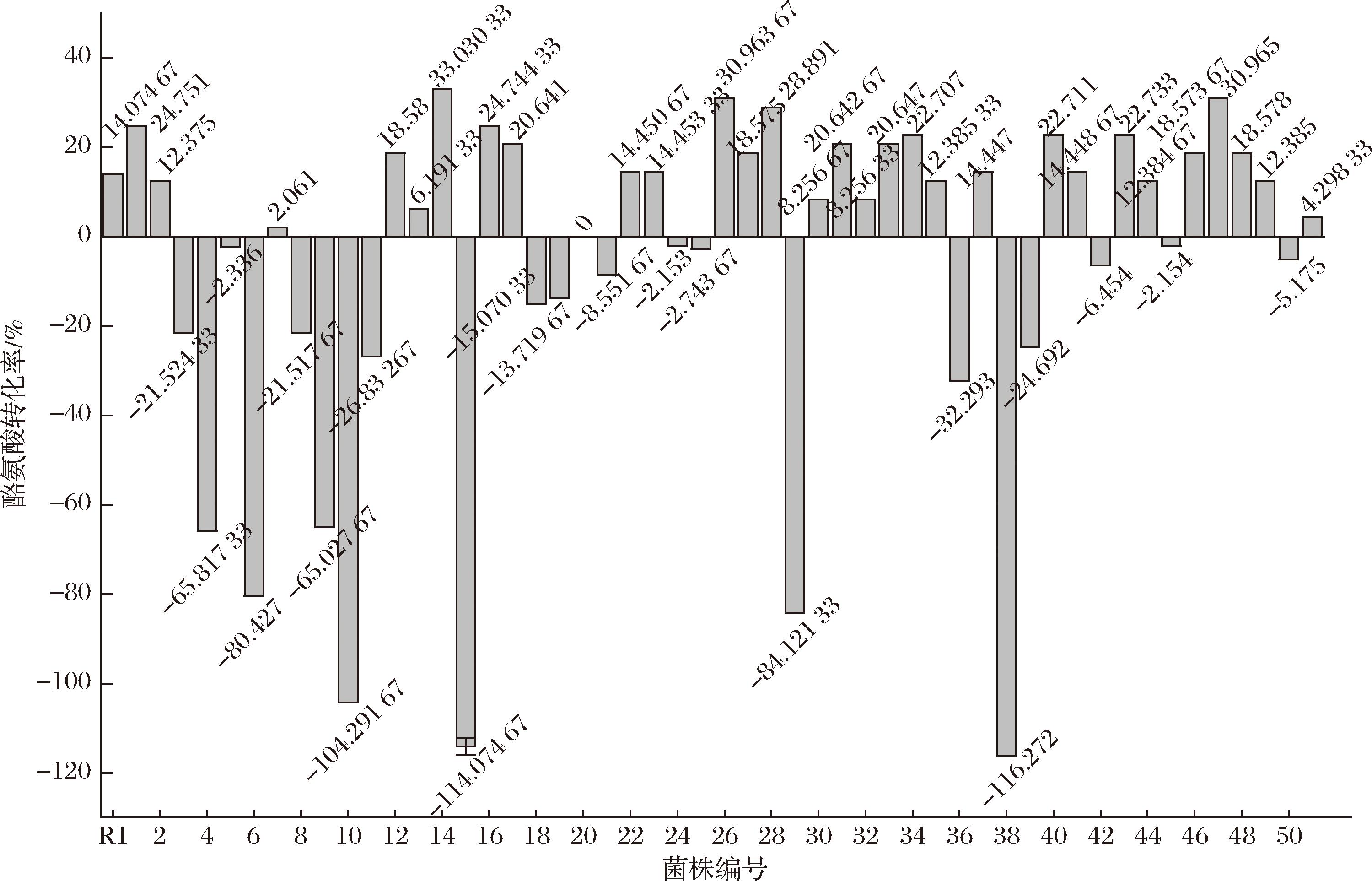
图4 诱变菌株的复筛结果
Fig.4 Results of the second-round screening of mutant strains
2.4 诱变菌株的遗传稳定性分析
将复筛所得的4株酪氨酸转化率提升的突变菌株(14号、26号、28号、48号)接种于发酵培养基,并进行了50代传代实验,期间每次传代时均进行酪氨酸转化率检测并绘制遗传稳定性曲线。如图5所示,该4株突变菌株在传代50次后,其酪氨酸转化率无明显变化,说明其均具有良好的遗传稳定性。根据实验结果,发现与出发菌株R1相比,14号菌株的酪氨酸转化率最高,26号、28号、48号次之,因此将上述菌株重命名为菌株R1-14、R1-26、R1-28和R1-48。
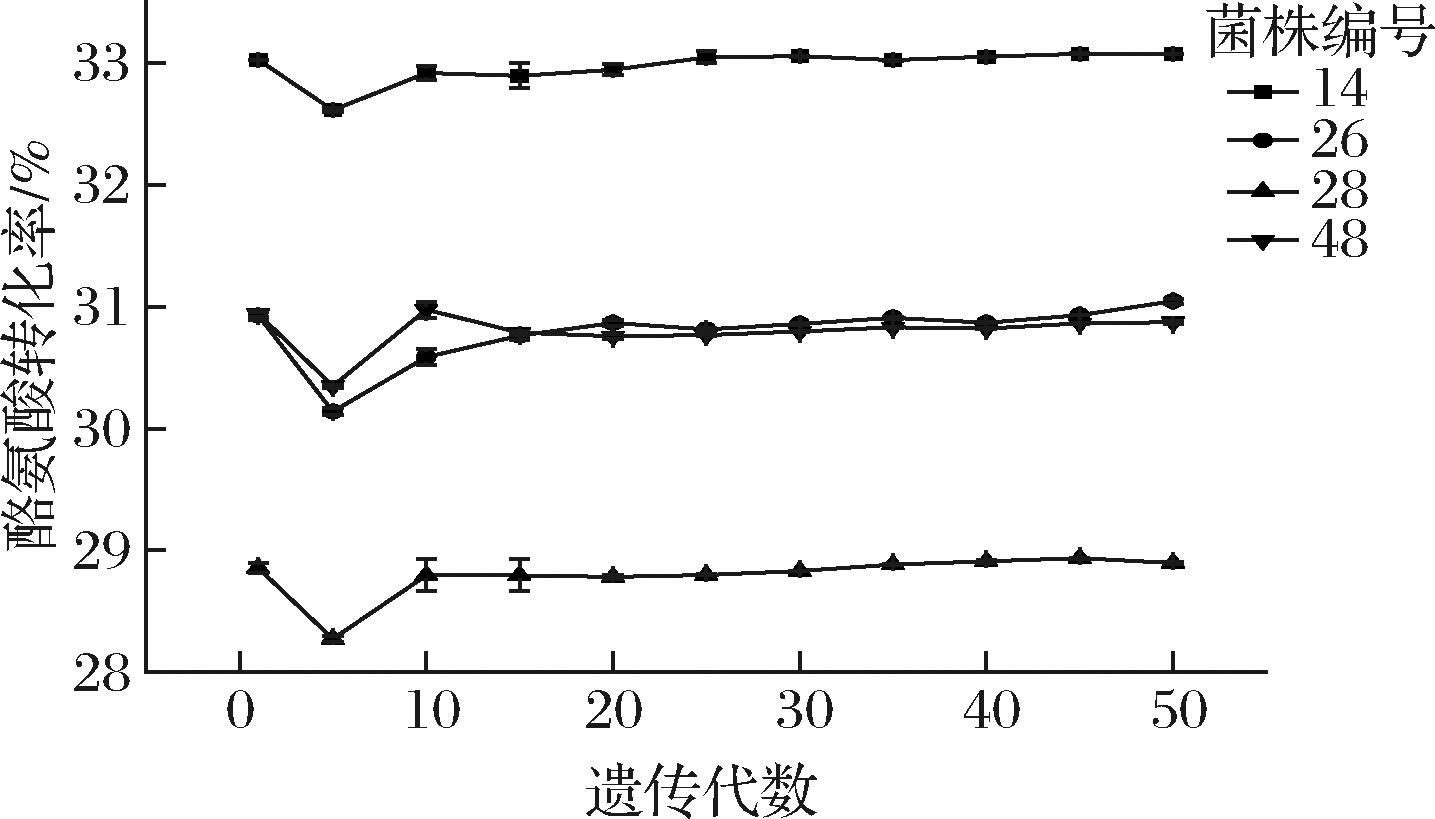
图5 菌株遗传稳定性测试
Fig.5 Genetic stability test of mutant strains
2.5 酪氨酸代谢关键基因分析
2.5.1 酪氨酸代谢路径分析
KEGG是基于大型分子数据集生成的基因组测序和其他高通量实验技术的实用程序数据库资源,其能够通过强大的图形功能来介绍众多的代谢途径以及各途径之间的关系[26]。通过该数据库可以进行L-酪氨酸代谢路径注释,确定L-酪氨酸参与的主要生化代谢途径和信号转导途径。本实验基于实验室前期工作中得到的枯草芽孢杆菌R1基因组数据,分析了编码酶的基因序列及对应的酶编号,联合KEGG数据库中酪氨酸代谢路径所含酶及其编号进行分析,发现枯草芽孢杆菌R1对于酪氨酸的利用主要通过天冬氨酸转氨酶(aspartate aminotransferase,AST,EC 2.6.1.1)和多酚氧化酶(polyphenol oxidase,PPO,EC 1.10.3.1)的催化作用实现(图6)。其中,AST具有以L-天冬氨酸、L-酪氨酸、L-苯丙氨酸、L-色氨酸为底物,生成L-谷氨酸与草酰乙酸的催化能力;PPO则可以催化L-酪氨酸生成L-多巴,最终通过一系列氧化反应产生黑色素,实现酪氨酸的转化。
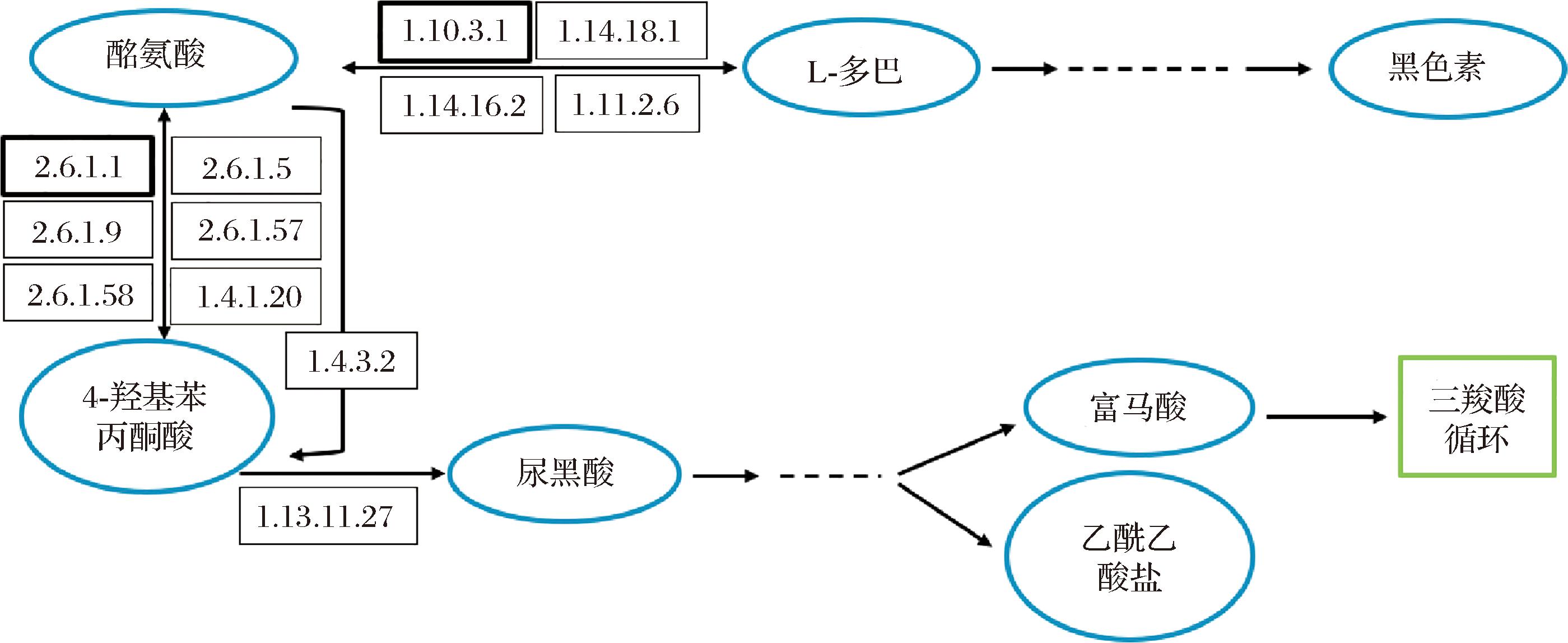
图6 酪氨酸代谢路径
Fig.6 Metabolic pathways of tyrosine in B.subtilis
注:黑色方框内为代谢路径酶编号,黑色加粗方框为枯草芽孢杆菌中含有的酶基因,蓝色椭圆内标有酶催化作用的底物及产物,箭头指明酶催化反应方向,绿色方框内为三羧酸循环代谢路径。
2.5.2 菌株酶基因相对表达量分析
采用qRT-PCR技术,以出发菌R1为对照考察了和4株突变菌在发酵培养基中的AST与PPO编码基因表达量。如图7所示,4株突变菌株的AST的表达量均高于PPO的表达量,其表达量比值约为1.5∶1,说明在突变菌株中AST为主要的酪氨酸代谢途径。与出发菌相比,R1-14菌株的AST基因表达量提高160.3%,而PPO基因表达量提高了48.5%;R1-26菌株的AST基因表达量提高了82.5%,而PPO基因表达量提高了21.4%;R1-28菌株的AST基因表达量提高了82.5%,而PPO基因表达量提高了22.6%;R1-48菌株的PPO基因表达量与出发菌相比没有明显差异,仅提高了4.2%,而其AST基因表达量提高了35.5%。
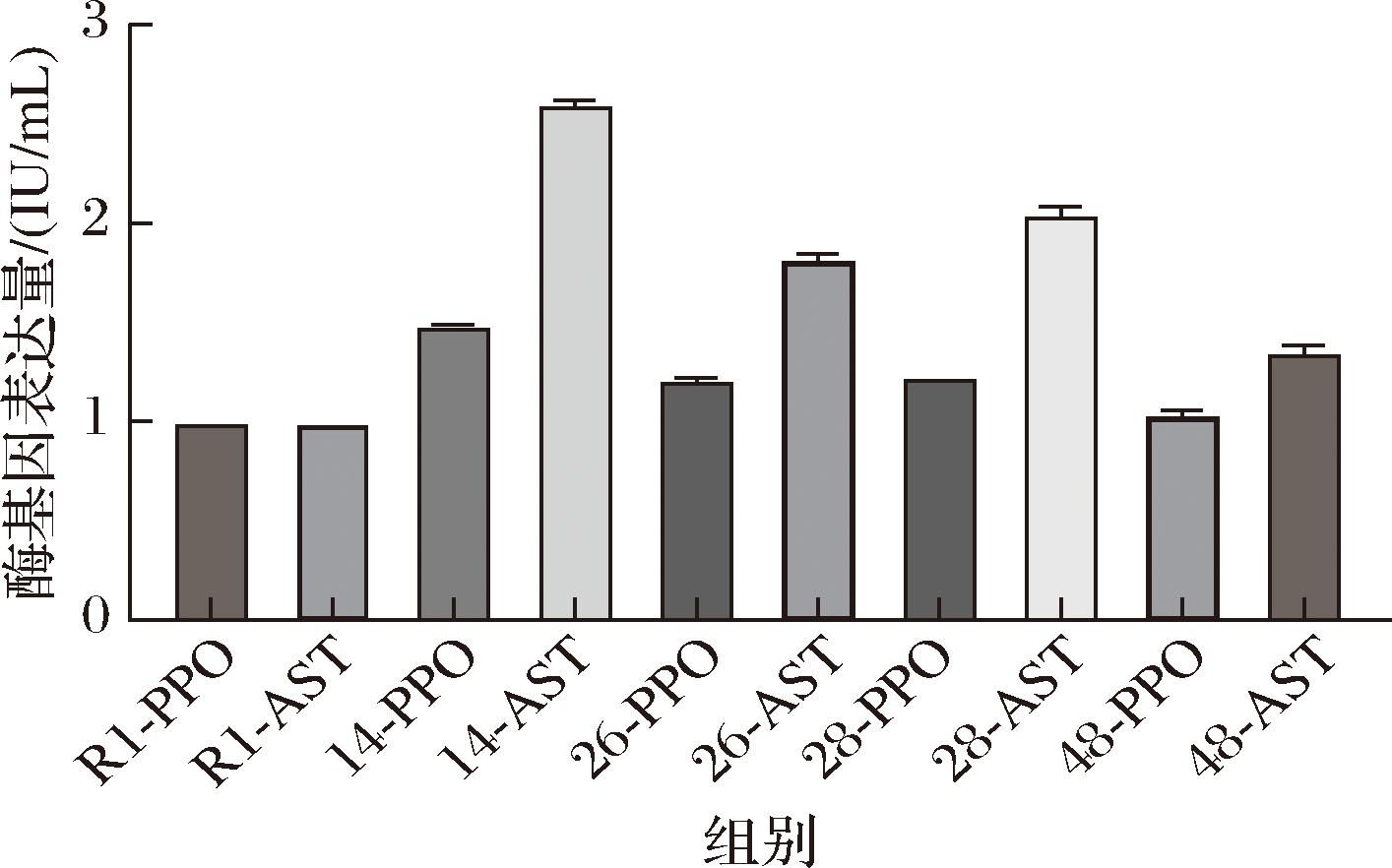
图7 菌株AST与PPO基因表达量
Fig.7 Expression levels of related genes with tyrosine metabolism in original and mutant strains
2.6 诱变菌株在豆瓣酱发酵过程中的应用
将出发菌株R1和诱变菌株R1-14接种于豆瓣酱发酵过程,测定豆瓣酱发酵过程中酱醅中酪氨酸含量的变化趋势,并将豆瓣酱中酪氨酸析出限值以虚线标识,若酱醅中酪氨酸含量超过该限值则易出现白点。如图8所示,发酵起始阶段酱醅的酪氨酸含量均为0.513 g/L。在未接种微生物的空白组样品中,其酪氨酸含量在前期呈下降趋势,而发酵第3周开始缓慢增加,至发酵完成时酪氨酸含量为0.525 g/L;接种出发菌株R1和诱变菌株R1-14的酱醅中的酪氨酸含量变化趋势基本相同。2个实验组的酪氨酸含量均呈现在发酵前15 d快速下降,而在发酵后期基本稳定的特点。接种R1菌株小组发酵结束时酱醅中酪氨酸含量为0.366 g/L,较空白组降低了0.159 g/L,酪氨酸转化率为30.3%。相比之下,接种诱变菌株R1-14的实验组发酵结束后其酱醅中酪氨酸含量为0.285 g/L,较空白组降低了0.240 g/L,酪氨酸转化率为45.7%。由此可见,诱变菌株R1-14的使用能显著降低酱醅中的酪氨酸含量,从而减少豆瓣酱中白点物质出现的可能性。
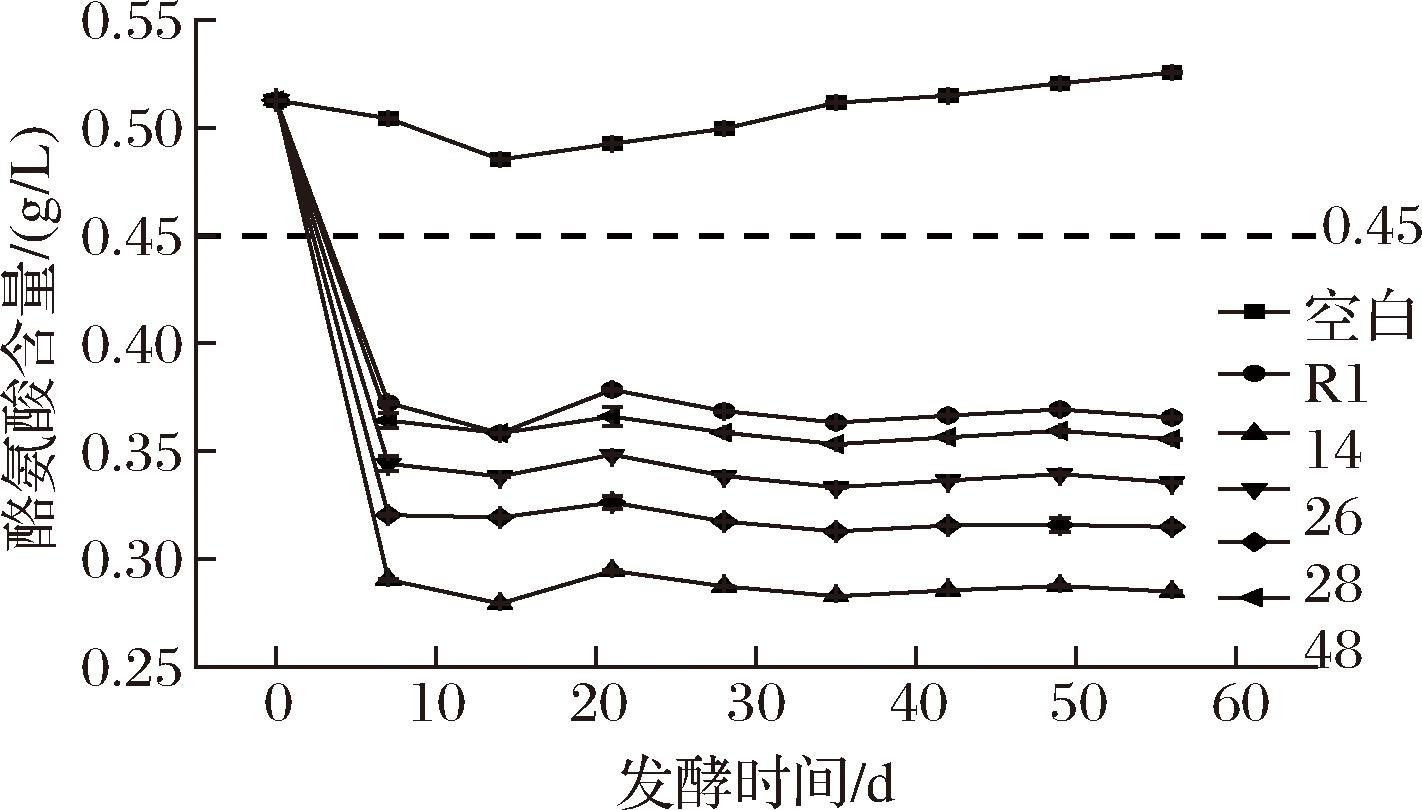
图8 诱变菌株参与豆瓣酱发酵过程中酪氨酸转化能力检测
Fig.8 Concentrations of tyrosine during broad bean paste fermentation with the addition of original and mutant strains
3 结论
从豆瓣酱酱醅和白酒酒醅中筛选得到4株具有酪氨酸利用能力的微生物菌株,并结合菌株耐盐性分析,得到一株具有一定耐盐能力且能在有机氮源存在条件下转化酪氨酸的枯草芽孢杆菌R1。通过ARTP诱变技术,筛选得到4株在8 g/L盐度下酪氨酸转化率显著提高的突变菌株,且均具有良好的传代稳定性。其中,突变菌R1-14菌株的酪氨酸转化率达到33.0%。通过qRT-PCR技术,发现该菌株主要通过天冬氨酸转氨酶途径利用酪氨酸。将该菌株应用于豆瓣酱发酵过程,能显著降低酱醅中的酪氨酸含量,其酪氨酸转化率达到45.7%,远高于出发菌且发酵酱醅中酪氨酸含量低于白点析出限值。因此,利用枯草芽孢杆菌R1-14菌株以减少豆瓣酱或黄豆酱中白点出现的概率具有潜在的可行性。
[1] 张平, 武俊瑞, 乌日娜.大豆发酵食品-豆酱的研究进展[J].中国酿造, 2018, 37(2):6-10.ZHANG P, WU J R, WU R N.Research progress of fermented soybean food-soybean paste[J].China Brewing, 2018, 37(2):6-10
[2] 张锦航, 钟武, 郭小, 等.发酵豆制品中白点防治的研究进展[J].中国酿造, 2021, 40(7):28-32.ZHANG J H, ZHONG W, GUO X, et al.Research progress on prevention and control of white spots in fermented soybean products[J].China Brewing, 2021, 40(7):28-32.
[3] 王立爽, 李文涛.黄豆酱白点性质及形成机理研究进展[J].中外食品工业, 2024(10):34-36.WANG L S, LI W T.Research progress on properties and formation mechanism of white spots in soybean sauce[J].Global Food Industry, 2024(10):34-36.
[4] 闵世豪, 单万祥, 钮成拓, 等.豆酱白点物质组成及模拟发酵条件优化[J].食品与发酵工业, 2020, 46(9):89-93.MIN S H, SHAN W X, NIU C T, et al.Composition of white spots in soybean paste and optimization of simulated fermentation[J].Food and Fermentation Industries, 2020, 46(9):89-93.
[5] 周韬. 黄豆酱白点的形成机理及控制技术研究 [D].天津:天津科技大学, 2021.ZHOU T.Study on the formation mechanism and control technology of white spots in soybean paste [D].Tianjin:Tianjin University of Science and Technology, 2021.
[6] 杨熙, 贾鑫.发酵豆制品的白点问题研究进展[J].现代食品, 2020, 26(17):7-9;12.YANG X, JIA X.Research progress on white spots of fermented soybean products[J].Modern Food, 2020, 26(17):7-9;12.
[7] LIU J B, SONG H L, LIU Y, et al.Discovery of kokumi peptide from yeast extract by LC-Q-TOF-MS/MS and sensomics approach[J].Journal of the Science of Food and Agriculture, 2015, 95(15):3183-3194.
[8] 朱洪康. 绍兴腐乳白点形成与控制的初步研究 [D].无锡:江南大学, 2015.ZHU H K.A preliminary study on the formation and control of white spots in Shaoxing sufu [D].Wuxi:Jiangnan University, 2015.
[9] 陈执. 一种酶系缺陷型酱用曲种的筛选、扩培及应用[J].现代食品, 2023, 29(13):63-66.CHEN Z.Screening, expansion and application of an enzyme deficient type of sauce strain[J].Modern Food, 2023, 29(13):63-66.
[10] NIU C T, MIN S H, JIA Y, et al.Adaptive evolution of Aspergillus oryzae 3.042 strain and process optimization to reduce the formation of tyrosine crystals in broad bean paste[J].Journal of Food Biochemistry, 2019, 43(2):e12716.
[11] 杨代永, 范光先, 汪地强, 等.高温大曲中的微生物研究[J].酿酒科技, 2007, (5):37-38.YANG D Y, FAN G X, WANG D Q, et al.Microbes in high temperature starter[J].Liquor-Making Science &Technology, 2007(5):37-38.
[12] 胡传旺. 酱油发酵过程微生物群落解析及功能研究 [D].无锡:江南大学, 2017.HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains isolated from moromi [D].Wuxi:Jiangnan University, 2017.
[13] 朱海针, 谢卫华, 龙凯, 等.PCR-DGGE技术研究淡豆豉炮制过程中微生物菌群的动态变化[J].中草药, 2017, 48(9):1757-1765.ZHU H Z, XIE W H, LONG K,et al.Study on dynamic changing regularity of microflora in fermentation process of Sojae Semen Praeparatum by PCR-DGGE[J].Chinese Traditional and Herbal Drugs, 2017, 48(9):1757-1765.
[14] 贡汉坤. 传统豆酱自然发酵的动态分析及人工接种多菌种发酵研究 [D].无锡:江南大学, 2004.GUONG H K.Tendency analysis on natural ferment of soy-bean paste and research on fermentation using multi-strains via artificial inoculation [D].Wuxi:Jiangnan University, 2004.
[15] 邢先雷, 左汶骏, 汪溢恒, 等.耐盐产香埃切假丝酵母的筛选、特性分析及其在豆瓣酱的应用[J].食品与发酵工业, 2025, 51(2):68-76.XING X L, ZUO W J, WANG Y H, et al.Screening and characterization of salt-tolerant and aroma-producing Starmerella etchellsii and its application in broad bean paste[J].Food and Fermentation Industries, 2025, 51(2):68-76.
[16] 贾云. 蚕豆酱发酵过程微生物群落结构及其功能分析 [D].无锡:江南大学, 2021.JIA Y.Analysis of microbial community structure and function during broad bean paste fermentation [D].Wuxi:Jiangnan University, 2021.
[17] HONG H A, HUANG J M, KHANEJA R, et al.The safety of Bacillus subtilis and Bacillus indicus as food probiotics[J].Journal of Applied Microbiology, 2008, 105(2):510-520.
[18] 董晨, 曹娟, 张迹, 等.耐高温α-淀粉酶基因在枯草芽孢杆菌中的高效表达[J].应用与环境生物学报, 2008, 14(4):534-538.DONG C, CAO J, ZHANG J, et al.High-level expression of thermostable α-amylase in Bacillus subtilis[J].Chinese Journal of Applied and Environmental Biology, 2008, 14(4):534-538.
[19] 吴拥军, 孟望霓, 李耀中, 等.纯种强化发酵细菌型豆豉研究[J].中国酿造, 2011, 30(9):56-60.WU Y J, MENG W N, LI Y Z, et al.Production of bacterial fermented Douchi by pure fermentation[J].China Brewing, 2011, 30(9):56-60.
[20] 钟杰. 枯草芽孢杆菌酱(豉)香风味基因的筛选及功能研究 [D].贵阳:贵州大学, 2019.ZHONG J.Screening and functional research of the flavor genes about lobster or soya sauce of Bacillus subtilis [D].Guiyang:Guizhou University, 2019.
[21] 康竞艺, 艾奕伶, 刘秀霞, 等.常压室温等离子体结合高通量筛选技术在微生物育种方面的研究进展[J].微生物学通报, 2025, 52(1):78-89.KANG J Y, AI Y L, LIU X X, et al.Research progress in atmospheric and room-temperature plasma combined with high-throughput screening in microbial breeding[J].Microbiology China, 2025, 52(1):78-89.
[22] ZHANG Q, MIAO R Y, FENG R C, et al.Application of atmospheric and room-temperature plasma (ARTP) to microbial breeding[J].Current Issues in Molecular Biology, 2023, 45(8):6466-6484.
[23] 于莹莹. 基于荧光探针技术选育脯氨酸积累的耐高浓啤酒酵母 [D].无锡:江南大学, 2024.YU Y Y.Breedingof beeryeast with proline accumulation in high gravity brewing based on fluorescent probe technology[D].Wuxi:Jiangnan University, 2024.
[24] 杨悦. 豆瓣酱降盐工艺的研究 [D].无锡:江南大学, 2021.YANG Y.Effect of salt reduction on doubanjiang fermentation[D].Wuxi:Jiangnan University, 2021.
[25] 张心青, 张萧萧, 杨传伦, 等.一株对番茄枯萎病有拮抗作用的枯草芽孢杆菌的ARTP诱变与筛选[J].中国农学通报, 2020, 36(26):44-49.ZHANG X Q, ZHANG X X, YANG C L, et al.Bacillus subtilis against tomato Fusarium wilt:ARTP mutagenesis and screening[J].Chinese Agricultural Science Bulletin, 2020, 36(26):44-49.
[26] DAVID C, KONDYLAKIS H.KEGGSum:Summarizing genomic pathways[J].Information, 2024, 15(1):56.