倍半萜内酯是一类具有15个碳原子基本骨架的内酯化合物,主要存在于菊科植物中。这些化合物是传统药用植物中的活性成分,具有多种生物医学活性,包括抗炎、抗氧化、抗真菌和抗癌作用[1-4]。木香烃内酯是自然界中最基本的倍半萜内酯,被认为是合成多种生物活性倍半萜内酯(如愈创木内酯、伪愈创木内酯和xanthanolide)的前体[5-6]。最近,研究人员已经证明木香烃内酯具有显著的抗肿瘤活性,包括抑制肿瘤细胞增殖和诱导凋亡[7]。
木香烃内酯可通过化学合成或从植物根茎中提取获得,但化学合成及植物提取易造成环境污染且效率低下[8]。随着合成生物学的发展以及木香烃内酯生物合成途径的阐明,微生物细胞工厂为获取木香烃内酯提供了一种替代方法。DE KRAKER等[6]最初提出了从法尼基焦磷酸(farnesyl pyrophosphate,FPP)到木香烃内酯(costunolide)的生物合成途径。首先,FPP在吉马烯A合成酶(NSY305N)的作用下生成吉玛烯A(germacrene A),随后在吉马烯A氧化酶(germacrene A oxidase,GAO)的作用下,经过三步氧化反应生成吉马烯A酸(germacrene A acid,GAA)。GAA经木香烃内酯合成酶(costunolide synthase,COS)氧化形成6α-羟基-GAA,最终自发内酯化生成木香烃内酯[9-10](图1)。值得注意的是,GAO和COS均为位于植物内质网膜上的细胞色素P450(cytochrome P450,CYP),这类膜结合的血红素单加氧酶在萜类化合物的生物合成中起着至关重要的作用[11-13]。在催化过程中,CYP需要NADPH(nicotinamide adenine dinucleotide phosphate)、氧气以及细胞色素P450还原酶(cytochrome P450 reductase,CPR)作为电子传递的分子伴侣。近些年来,来自菊科植物(包括向日葵、菊苣和生菜)的几种GAO和COS酶已被成功分离和表征[14]。
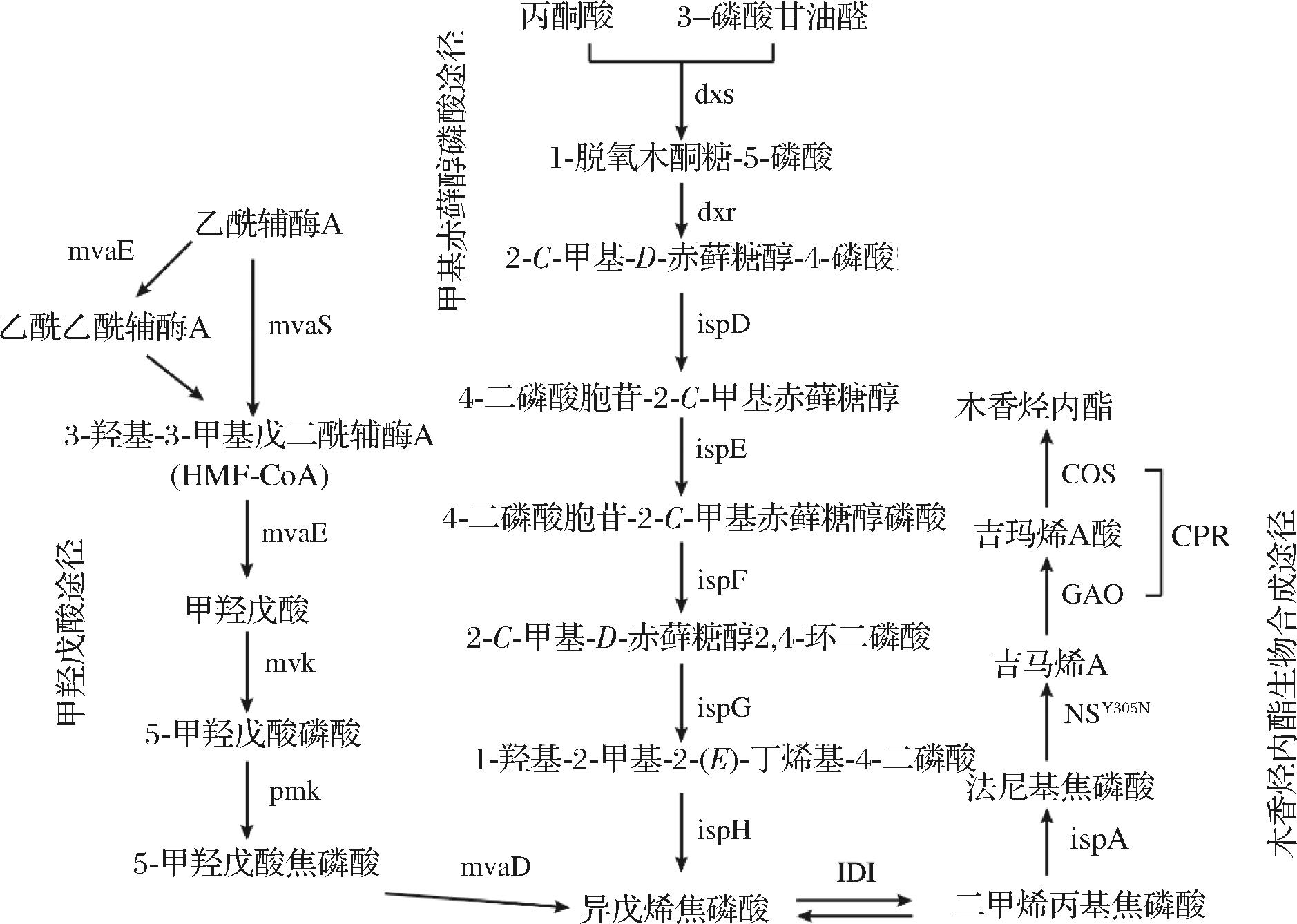
图1 木香烃内酯生物合成途径
Fig.1 Biosynthetic route for costunolide synthesis
注:mvaE:乙酰乙酰辅酶A硫解酶/HMG-CoA还原酶;mvaS:3-羟基-3-甲 基戊二酰辅酶A合成酶;mvk:甲羟戊酸激酶;pmk:磷酸甲羟戊酸激酶; mvaD:磷酸甲羟戊酸脱羧酶;dxs:1-脱氧木酮糖-5-磷酸合成酶;dxr: 1-脱氧木酮糖-5-磷酸还原异构酶;ispD:4-二磷酸胞苷基-2-C-甲基 赤藓糖醇合酶;ispE:4-二磷酸胞苷基-2-C-甲基赤藓糖醇激酶;ispF: 2C-甲基-D-赤藓糖醇2,4-环二磷酸合成酶;ispG:1-羟基-2-甲 基-2-(E)-丁烯基-4-二磷酸合成酶;ispH:4-羟基-3-甲基丁-2-烯基二 磷酸还原酶;IDI:异戊二烯二磷酸异构酶;ispA:法尼基二磷酸合酶; NSY305N:念珠藻合成酶305位突变体;GAO:吉马烯A氧化酶; COS:木香烃内酯合成酶;CPR:细胞色素P450还原酶。
迄今为止,大多数关于木香烃内酯生物合成的研究都是在真核表达系统中进行的,主要是酵母和烟草叶片。LIU等[10]从菊苣中分离出首个具有木香烃内酯合成活性的基因,并将其命名为CiCOS。通过在酵母中共表达TpGAS、CiGAO和CiCOS,成功检测到低水平的木香烃内酯。将这些基因转染到本氏烟草中,产生了60 ng/g的木香烃内酯。IKEZAWA等[9]使用高拷贝质粒pESC-Leu2 d在酿酒酵母中共表达了4个植物基因(GAS、LsGAO、LsCOS和CPR),最终产生9.3 mg/L的木香烃内酯。且进一步发现,尽管来自于向日葵的同源基因CYP71BL1具有GAA的羟基化活性,但在酵母中表达时,它会将GAA氧化为8α-羟基-GAA而非6α-羟基-GAA,因此不能被乳酸化形成木香烃内酯[9]。同样,在转基因酵母的培养过程中,还发现了由于所提出的酸诱导重排而使GAA异常转化为木香酸和冬青酸的情况[15]。大肠杆菌由于其快速的生长周期、易于遗传操作以及不会与内源性CYP产生干扰等原因,成为表达异源CYP的首选细菌细胞工厂[16]。
在本研究中,选择大肠杆菌BL21(DE3)作为合成木香烃内酯的宿主细胞,并引入外源的甲羟戊酸途径以增加FPP的供应。通过对表达体系以及木香烃内酯合成酶的筛选来提高CYP的异源表达水平。随后,引入吉马烯A高产底盘菌株并补加血红素前体以进一步提高木香烃内酯的产量。最终,经发酵条件优化后,在20 mL培养基中合成了6.19 mg/L木香烃内酯,是初始菌株的81倍。研究结果证实了利用大肠杆菌进行木香烃内酯合成的可行性,并为提高植物CYP的原核表达提供了一种参考策略。
1 材料与方法
1.1 材料与试剂
1.1.1 主要试剂
2×Taq PCR Mastermix、2×PrimeStar,Takara;2×Multif Seamless Assembly Mix,Abclonal;引物,苏州金唯智生物科技有限公司。
1.1.2 菌株与质粒
本研究中所使用的质粒与菌株如表1所示,所使用引物如表2所示。
表1 本研究中使用的菌株与质粒
Table 1 Strains and plasmids used in this study

菌株与质粒表型来源菌株大肠杆菌DH5αF-Φ80 lacZΔM15 Δ(lacZYA-argF)U169 recA1 endA1 hsdR17 (rK-, mK+) phoA supE44 thi-1 gyrA96 relA1 λ-实验室贮存大肠肝菌BL21(DE3)F-dcm ompT hsdS(rBmB) + B14:E21 gal λs实验室贮存大肠杆菌Δ7大肠杆菌BL21 (DE3)-ΔpoxB-ΔppsA-ΔptsG-Δcrr-ΔadhE-ΔIdhA-ΔzwfFORDJOUR等[17]CosP大肠杆菌BL21 (DE3) harboring pR-IN∗IGCP本研究构建CosA大肠杆菌BL21 (DE3), pA-ESMPDI+pR-IN∗I本研究构建Cos2大肠杆菌BL21 (DE3), pA-ESMPDI+pR-IN∗I+pT-GCP本研究构建Cos3大肠杆菌BL21 (DE3), pA-ESMPDI+pR-IN∗I + pL-GCP本研究构建Cos4大肠杆菌BL21 (DE3), pA-ESMPDI + pR-IN∗I + pCW-GCP本研究构建Cos5大肠杆菌BL21 (DE3), pA-ESMPDI + pR-IN∗I + pCW-GLsP本研究构建Δ7-CI大肠杆菌Δ7, pA-ESMPDI + pR-IN∗IEKGG + pCW-GCPNAB本研究构建质粒pR-IN∗IGCPRSF ori, KanR, pRSFDuet-T7- IspA-NS-IDI-T7-CiGAOΔ22-CPR-CiCOSΔ22本研究构建pR-IN∗IEKGGRSF ori, KanR, pRSFDuet-T7- IspA-NS-mvaE-mvaS-T7-glK-galP-IDI本研究构建pA-ESMPDIp15a ori, CmR, pACYCDuet-T7-mvaEmvaS-T7-mvk-pmk-mvaD-IDIFORDJOUR等[17]pR-IN∗IRSF ori, KanR, pRSFDuet-T7- IspA-NS-T7-IDIFORDJOUR等[17]pT-GCPColE1(pBR322) ori, AmpR, ptrc99A-CiGAOΔ22-CPR-CiCOSΔ22本研究构建pL-GCPColE1(pBR322) ori, AmpR, pLacUV5-CiGAOΔ22-CPR-CiCOSΔ22本研究构建pCW-GCP ColE1(pBR322) ori, AmpR, pLacUV5/pTac/pTac-CiGAOΔ22-CPR-CiCOSΔ22本研究构建pCW-GLsP ColE1(pBR322) ori, AmpR, pLacUV5/pTac/pTac-CiGAOΔ22-CPR-LsCOSΔ22本研究构建
表2 本研究中使用的引物
Table 2 Primers used in this study

引物序列(5′-3′)IN∗I-V-FCTCGAGTCTGGTAAAGAAACCGIN∗I-V-RTGATATCCAATTGAGATCTGCCACiGAO-FGGCAGATCTCAATTGGATATCAATGCGCCCGAAAAGCAACAACiGAO-RCGCCATGGTATATCTCCTTCTAGATTAAAAGCTCGGCACCAGAACPR-FTCTAGAAGGAGATATACCATGGCGCTGGATAAACTGGATCPR-RCTTATTAAAGTTAAACAAAGGTACCTTACCAGACATCACGCAGGTACiCOS-FTTTGTTTAACTTTAATAAGGAGACTCGAGATGCCGAAAACGAGCAAAAACCiCOS-RCGGTTTCTTTACCAGACTCGAGTTATTTCGCAATGCTTTTCGGAATCptrc99A-FCGGGGATCCTCTAGAGTCGAptrc99A-RCATTATATCTCCTTCTTAAAGTTAAACAAGGTACCGAGCTCGAATTCCATGCP-FCTTGTTTAACTTTAAGAAGGAGATATAATGCGCCCGAAAAGCAACAAAAAACGCP-RTCGACTCTAGAGGATCCCCGTTATTTCGCAATGCTTTTCGGAATCpT-V5-F1TTTACACTTTATGCTTCCGGCTCGTATAATGTGTGGAATTGTGAGCGGATAACpT-V5-R1TTACCAGACATCACGCAGGTApT-V5-F2TACCTGCGTGATGTCTGGTAAGGTACCTTTGTTTAACTTTAATAAGGAGACpT-V5-R2AGCCGGAAGCATAAAGTGTAAACAGCTCATTTCAGAATATTTGCCAGPCW-V-FAATTGTGCCGATTCTGAAAAGCTAAGGATGCGACTATGATGCAACCAGC-RCTCGAGTCTCCTTATTAAAGTTAAACAAAGGLsCOS-FTGTTTAACTTTAATAAGGAGACTCGAGATGCCGAAAACGAGCAAAAACCTGCLsCOS-RTTAGCTTTTCAGAATCGGCACA
1.1.3 培养基与抗生素
LB培养基(g/L):酵母提取物5,蛋白胨5,NaCl 10,主要用于质粒构建菌株及发酵种子液培养。LB固体培养基为上述液体培养基中添加质量分数2%的琼脂粉。
TB培养基(g/L):蛋白胨12,酵母提取物24,甘油4 mL,磷酸二氢钾2.31,磷酸氢二甲12.54,主要用于菌株摇瓶发酵。
抗生素(mg/L):卡那霉素终质量浓度50;氨苄青霉素终质量浓度100;氯霉素终质量浓度35。
1.1.4 标准品
木香烃内酯标准品:CAS编号553-21-9,分子式C15H20O2,分子质量232.318,规格及纯度为≥98%,-20 ℃贮存,源叶生物。
5-氨基乙酰丙酸(5-aminolevulinic acid hydrochloride,5-ALA)盐酸盐标准品:CAS编号5451-09-2,分子式C5H9NO3·HCl,分子质量167.59,规格及纯度为99%,2~8 ℃避光贮存,源叶生物。
乙酸乙酯:CAS编号141-78-6,分子式C4H8O2,分子质量88.11,规格及纯度为≥99.5%,沸点为77 ℃,相对密度0.902,国药沪试。
1.2 仪器与设备
ETC821 PCR基因扩增仪,东胜;759S21023 UV-Vis Spectrophotometer紫外可见分光光度计,上海棱光;LC20AT高效液相色谱,日本Shimadzu;SPD-20A/20AV UV-VIS检测器,日本Shimadzu。
1.3 实验方法
1.3.1 基因克隆和质粒构建
从NCBI数据库(https://www.ncbi.nlm.nih.gov/)获得CiGAO、CiCOS、LsCOS基因的核苷酸序列信息,从Addgene中获得了aaCPRct的核苷酸序列信息,使用TMHMM工具[18]对蛋白质的跨膜结构域进行预测,根据预测结果删除N端疏水跨膜结构域后,由苏州金唯智生物科技有限公司进行密码子优化(参考大肠杆菌BL21)和基因合成。分别用引物CiGAO-F/CiGAO-R;CPR-F/CPR-R;CiCOS-F/CiCOS-R以合成基因CiGAO、CPR、CiCOS为模版,扩增含有CiGAO,CPR,CiCOS的DNA片段;使用引物IN*I-V-F/IN*I-V-R对载体pRSF-Duet进行线性化。分别使用Gibson组装法连接载体和片段,构成质粒pR-IN*IGCP。
使用引物GCP-F/GCP-R以pR-IN*IGCP为模板扩增木香烃内酯生物合成基因簇得到基因片段CiGAO-CPR-CiCOS;使用引物pT-V5-F1/pT-V5-R2以基因片段CiGAO-CPR-CiCOS为模板扩增含有启动子pLacUV5及木香烃内酯生物合成基因簇的线性化基因片段;使用引物ptrc99A-F/ptrc99A-R以pTrc99a为模板扩增线性化载体片段;分别使用Gibson组装法连接载体和片段,构成质粒pT-GCP和pL-GCP。使用引物P-GCP-F/P-GCP-R以质粒pL-GCP为模板扩增基因片段CiGAO-CPR-CiCOS;使用引物PCW-V-F/GC-R以质粒pCWOri为模板线性化载体片段;使用引物LsCOS-F/LsCOS-R以合成基因LsCOS为模版,扩增含有LsCOS的DNA片段;分别使用Gibson组装法连接载体和片段,构成质粒pCW-GCP和pCW-GLsP。
1.3.2 菌株培养
经菌落PCR验证后,从平板上随机挑取3个菌落,接种至装有5 mL LB培养基的50 mL离心管中,在37 ℃和200 r/min下过夜培养。随后将种子液以1.5%的比例接种到装有20 mL TB培养基的150 mL摇瓶中,同时添加10 g/L的葡萄糖作为碳源,在37 ℃和220 r/min下培养5 h,直到OD600值达到0.6~0.8。添加1 mmol/L异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside, IPTG)溶液作为诱导剂,在20 ℃和200 r/min的条件下发酵,48 h后取样检测。
1.3.3 样品处理
发酵完成后,用20 mL乙酸乙酯萃取等体积的发酵培养基,取10 mL乙酸乙酯并将其在旋转真空蒸发器中蒸干。残渣溶于1 mL甲醇中,经0.22 μm滤膜过滤后,用高效液相色谱法进行分析。
1.3.4 木香烃内酯检测
高效液相色谱检测法:色谱柱:C18(4.6 mm×150 mm,5 μm),流动相A:水,流动相B:乙腈;流动相流速为1.0 mL/min,检测波长:213 nm,柱温:30 ℃;检测方法:0~5 min 95% A、5% B,6~40 min 5% B~100% B,41~42 min 5% B,最后维持10 min。使用已知浓度的木香烃内酯标准品绘制标准曲线,将峰面积转换为木香烃内酯浓度进行定量。
2 结果与分析
2.1 木香烃内酯生物合成途径的构建
为了在大肠杆菌中构建木香烃内酯的生物合成途径,本研究选择了实验室先前筛选出的能够从FPP合成吉玛烯A的吉马烯A合成酶突变体(NSY305N),并且引入了来自菊苣的吉马烯A氧化酶(germacrene A oxidase,由基因CiGAO编码)和木香烃内酯合成酶(costunolide synthase,由基因CiCOS编码)以进一步催化木香烃内酯的产生[17]。CHANG等[19]发现,使用从青蒿中分离出的aaCPRct代替来自热带念珠菌的ctCPR,可使大肠杆菌的生产力提高12倍。因此,选择使用aaCPRct作为CYP电子传递的氧化还原伴侣蛋白。在对基因CiGAO和CiCOS进行密码子优化后,使用TMHMM工具[18]预测了蛋白质的跨膜结构域。根据预测结果删除了N端疏水跨膜结构域,随后将其在大肠杆菌BL21(DE3)中表达,以高拷贝质粒pRSF-Duet为载体得到菌株CosP。同时,为了引入外源甲羟戊酸途径以提供高水平的FPP,以质粒pACYC Duet-1为载体在菌株CosP中过表达甲羟戊酸途径的6个基因(mvaE、mvaS、mvk、pmk,mvaD、IDI)以构建菌株CosA。如图2所示,仅依赖大肠杆菌中的甲基赤藓醇磷酸途径的CosP菌株几乎无法产生木香烃内酯。相较于菌株CosP,引入外源甲羟戊酸途径后,菌株CosA产生的木香烃内酯达到0.076 mg/L。
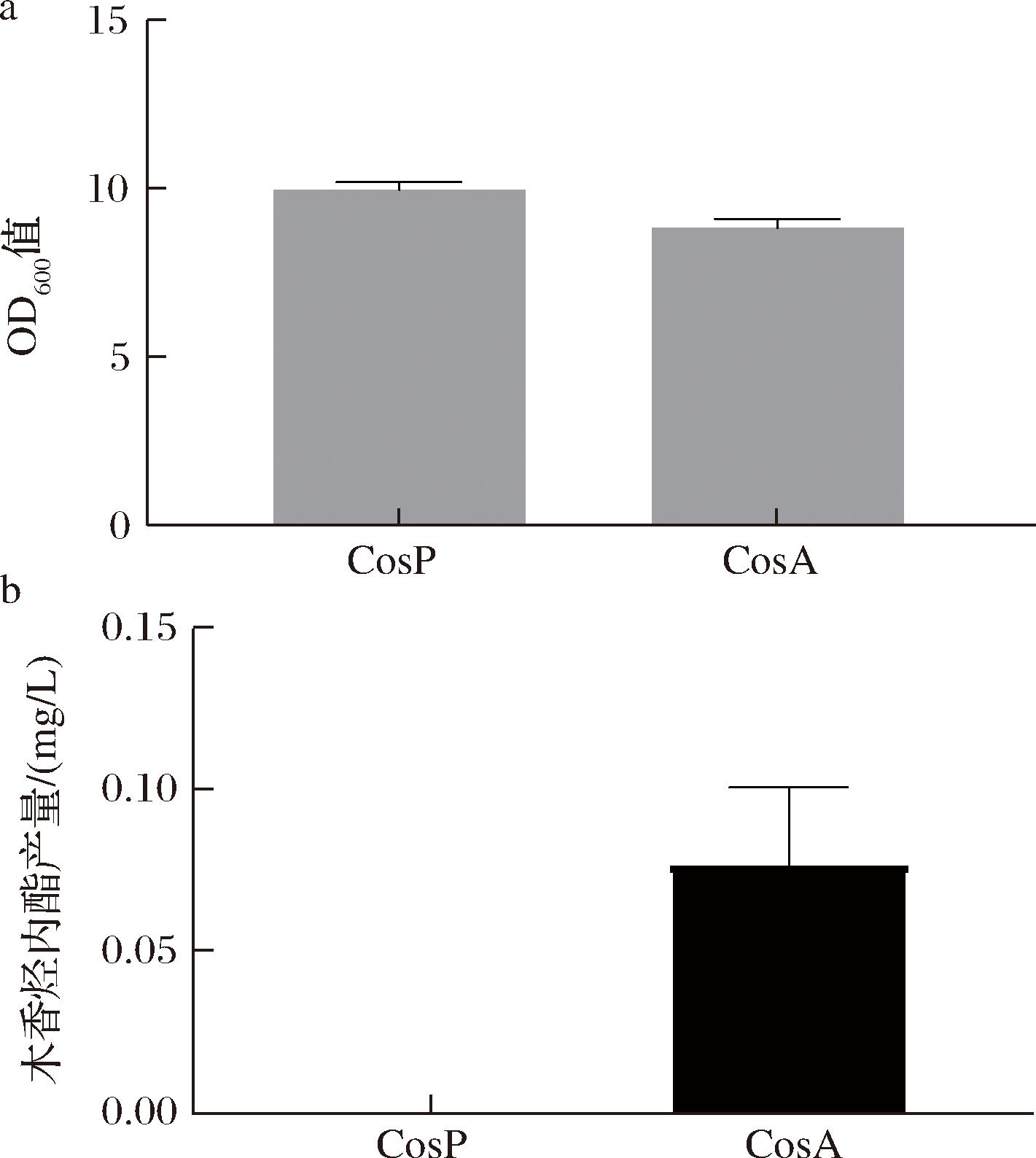
a-菌株CosP及CosA的OD600值;b-木香烃内酯产量
图2 未引入外源甲羟戊酸途径菌株与引入外源甲羟戊酸途径菌株的OD600值和木香烃内酯产量
Fig.2 OD600 and costunolide production between strains with and without exogenous mevalonate pathway introduction
2.2 表达体系及木香烃内酯合成酶的筛选
选择多样化的表达载体对于在大肠杆菌等原核细胞工厂成功表达CYP至关重要[20]。为了确定用于木香烃内酯合成的GAO、COS和CPR的最佳基因表达系统,本研究选择了2个额外的表达载体:pTrc99a(一种大肠杆菌通用表达载体)以及pCWOri(一种特异性针对CYP的表达载体)[21];使用了3种不同的启动子:Ptrc、PLacUV5和Ptac,构建了3种不同的表达系统:pT-GCP、pL-GCP和pCW-GCP(图3-a)。pCWOri质粒包含2个tac启动子和1个LacUV5启动子。这是1个混合启动子,包含trp启动子的-35区和LacUV5操纵子的-10区。由于近乎最佳的共享间距,该启动子在大肠杆菌中被RNA聚合酶强烈识别,从而在表达过程中实现表达水平的精确控制[20]。因此,与其他通用载体相比,pCWOri有助于更精确地调节异源蛋白的表达,从而提高CYP的异源表达水平。通过与吉玛烯A合成质粒(pRSF-IN*I和pA-ESMPDI)共表达,获得了3个菌株:COS-2、COS-3和COS-4(图3-a)。如图3-b所示,与菌株CosA相比,使用Ptrc启动子或PLacUV5启动子表达基因GAO、COS、CPR并没有提高木香烃内酯的产量。使用pCWOri载体的菌株Cos-4的木香烃内酯质量产量为0.117 mg/L,比菌株CosA(0.063 mg/L)高出1.86倍,因而选择菌株Cos-4进行进一步研究。
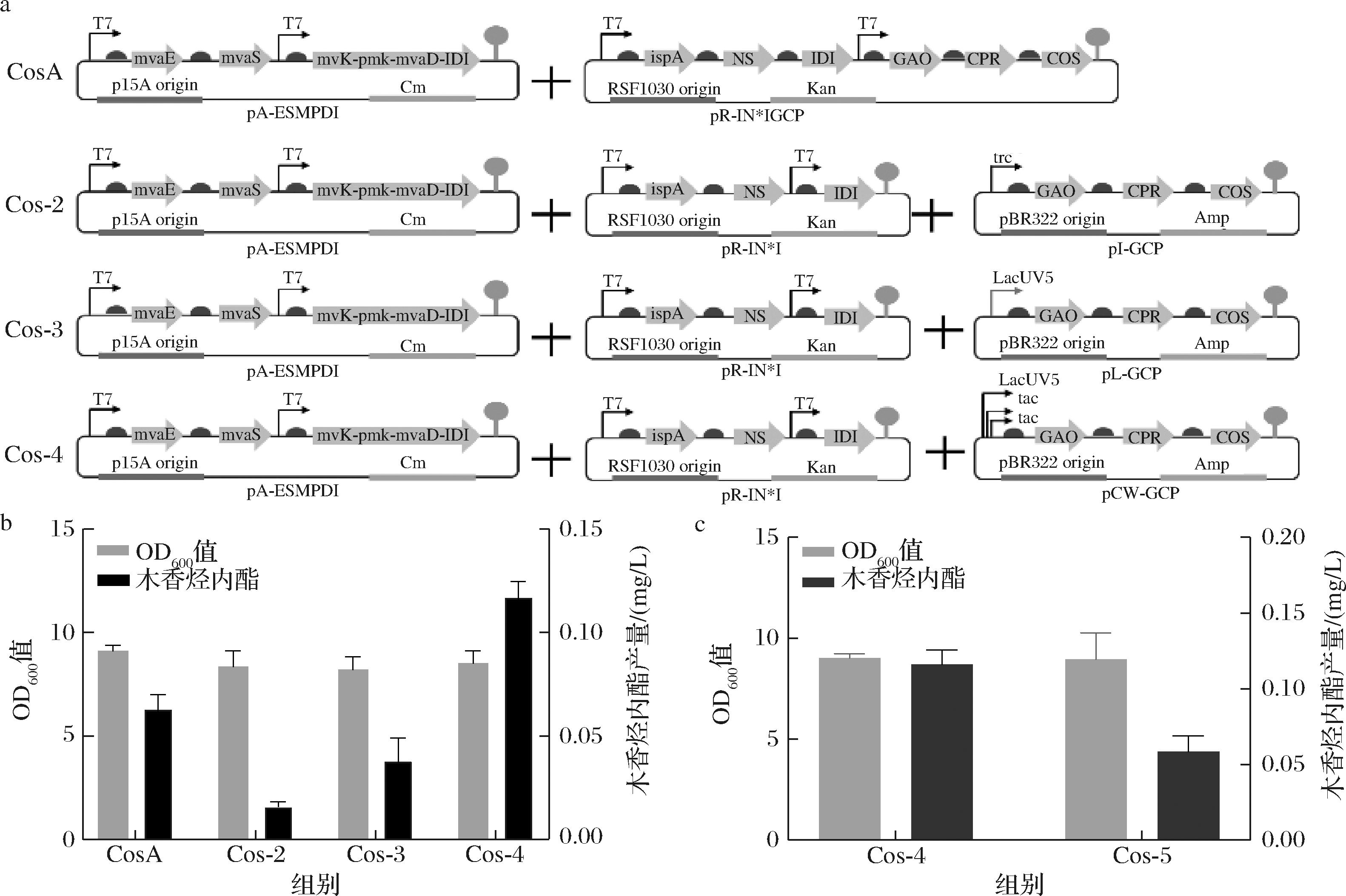
a-表达体系示意图;b-不同表达体系菌株的OD600值和木香烃内酯产量;c-表达不同来源木香烃内酯合成酶菌株的OD600值和木香烃内酯产量
图3 表达体系示意图、不同表达体系菌株及不同来源木香烃内酯合成酶菌株的OD600值和木香烃内酯产量
Fig.3 Schematic diagram of expression system, OD600 and costunolide titer of strains with different expression systems and strains expressing different costunolide synthases
为了获得高效的木香烃内酯合成酶,将来源于生菜的木香烃内酯合成酶(LsCOS)以pCWOri为载体导入大肠杆菌中构建菌株Cos-5。如图3-c所示,表达基因NSY305N、CiGAO和CiCOS的菌株Cos-4产生了0.116 mg/L的木香烃内酯,菌株Cos-5产生了0.058 mg/L的木香烃内酯,仅为前者的50%。这一结果表明,在大肠杆菌中,菊苣来源的木香烃内酯合成酶比生菜来源的更有利于木香烃内酯的合成。
2.3 辅因子增加、代谢工程菌株的应用及发酵条件优化
CYP的催化通常需要血红素作为辅因子。由于大多数微生物宿主中血红素的浓度较低,因此需要补充适量的血红素以确保催化反应的正常进行,而大肠杆菌缺乏血红素摄取系统,因而选择血红素合成前体5-ALA代替血红素进行补充[22-24]。在培养基中加入1 mmol/L 5-ALA以促进CiGAO和CiCOS的完全活性表达。如图4-a所示,补充5-ALA后,木香烃内酯的质量产量提升至0.22 mg/L,是未补充菌株的1.89倍。
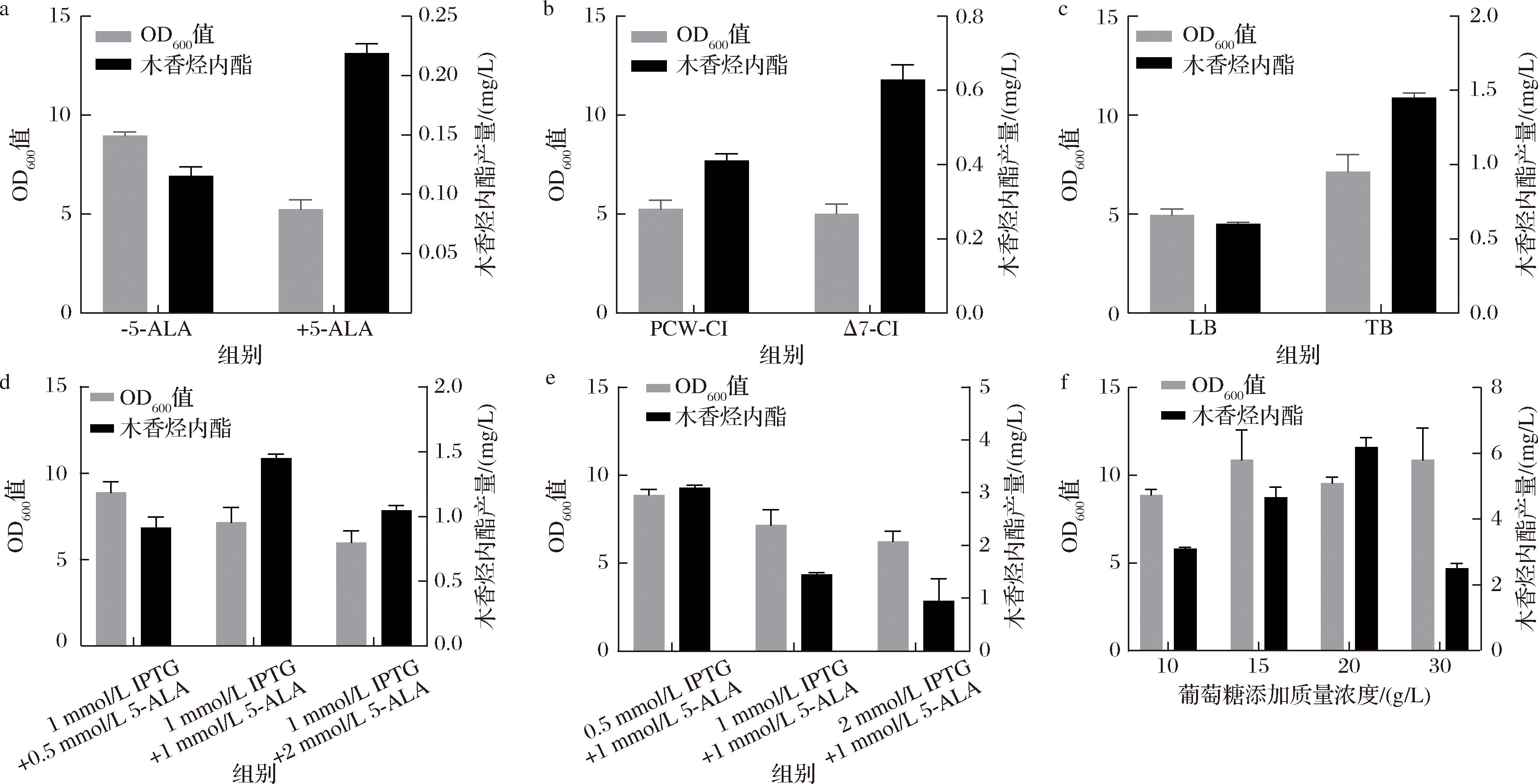
a-未添加5-ALA菌株与添加5-ALA菌株的OD600值和木香烃内酯产量;b-野生型BL21(DE3)与代谢工程底盘菌株的OD600值和木香烃内酯产量;c-使用不同培养基的OD600值和木香烃内酯产量;d-添加不同质量浓度5-ALA后菌株的OD600值和木香烃内酯产量;e-添加不同质量浓度IPTG后菌株的OD600值和木香烃内酯产量;f-添加不同质量浓度葡萄糖后菌株的OD600值和木香烃内酯产量
图4 添加5-ALA、引入代谢工程底盘菌株、发酵条件优化后菌株的OD600值和木香烃内酯产量
Fig.4 OD600 and costunolide titer with addition of 5-ALA, introduction of metabolically engineered chassis strains, and optimization of fermentation conditions
先前,本团队构建了一个高产吉玛烯A的底盘菌株BL21-ΔpoxB-ΔppsA-ΔptsG-Δcrr-ΔadhE-ΔIdhA-ΔZWF[17]。该菌株在野生型BL21(DE3)的基础上敲除poxB、ppsA、ptsG、crr、adhE、IdhA和ZWF等7个基因,从而将代谢通量转向乙酰辅酶A(acetyl-CoA)。此外,通过表达葡萄糖激酶(glk)和半乳糖转运蛋白(galP)以解决因敲除ptsG/crr而导致的葡萄糖缺乏问题[17]。为了进一步提高木香烃内酯的产量,在多基因敲除菌株BL21-ΔpoxB-ΔppsA-ΔptsG-Δcrr-ΔadhE-ΔIdhA-ΔZWF中构建了木香烃内酯合成途径[18]。如图4-b所示,与野生型BL21(DE3)相比,Δ7-CI的木香烃内酯的质量产量提高到了0.63 mg/L。
如图4-a所示,添加一定量的IPTG和5-ALA后,菌体的生长受到严重抑制。为了进一步改善菌体生长情况并提高木香烃内酯的产量,本研究对培养基、诱导剂添加浓度、血红素前体添加浓度以及葡萄糖添加浓度进行优化。以Δ7-CI为发酵菌株,分别选择LB培养基以及TB培养基作为发酵培养基,同时以1 mmol/L IPTG和1 mmol/L 5-ALA为起始浓度,设计正交实验以确定诱导剂和血红素前体的最适添加浓度。相较于LB培养基,使用TB培养基后菌体的生长状况更好(图4-c)。如图4-d和图4-e所示,菌体的生长与IPTG和5-ALA的添加量呈负相关,且木香烃内酯产量并不随IPTG和5-ALA浓度的增加而持续增加。在20 ℃下,添加0.5 mmol/L IPTG和1 mmol/L 5-ALA,木香烃内酯质量产量达到最高,为3.09 mg/L。在此基础上,分别向培养基中添加了10、15、20、30 g/L葡萄糖来进一步确定最适于木香烃内酯合成的葡萄糖浓度。当向培养基中添加20 g/L葡萄糖时,木香烃内酯的质量产量达到了6.19 mg/L(图4-f),达到初始菌株CosA的81倍。
3 结论与讨论
本研究选择大肠杆菌BL21(DE3)作为合成木香烃内酯的宿主细胞,并引入外源的甲羟戊酸途径以增加FPP的供应,在菌株CosA成功构建了木香烃内酯生物合成途径。通过对表达体系以及木香烃内酯合成酶的筛选,木香烃内酯的产量进一步提高,菌株Cos-4的质量产量提高至0.116 mg/L。随后,通过补充血红素前体、引入代谢工程高产底盘菌株以及发酵条件优化,添加0.5 mmol/L IPTG、1 mmol/L 5-ALA以及20 g/L葡萄糖发酵48 h后,在20 mL培养基中合成了6.19 mg/L木香烃内酯,是初始菌株的81倍。
木香烃内酯是一种主要存在于菊科植物中的天然倍半萜烯内酯,具有2个手性中心取代的十元环结构,由(E,E)-1,5-环癸二烯和α-甲基-γ-丁内酯组成。植物中的木香烃内酯含量极低,通过传统提取方法难以实现大规模生产[8]。此外,提取效率往往不令人满意,并且获得高纯度木香烃内酯颇具挑战性。另外,在化学合成方面也存在着如环境污染、原料受限以及安全风险等一系列问题。微生物细胞工厂提供了一种高效、可持续且具有成本效益的方法来生产木香烃内酯,这对推动其在医学领域的应用具有重要意义。
GAO和COS均为植物来源的CYP,这类真核来源的CYP在异源表达时通常会遇到表达困难、表达效率低的问题[20]。在酶的选择上,首先选择了相关报道中异源表达效果最好的菊苣来源的GAO以及COS,并引入细胞色素P450还原酶aaCPRct来提供必要的电子传递。而如实验结果所示,仅靠大肠杆菌内源的甲基赤藓醇磷酸途径无法合成木香烃内酯,因而在此基础上引入外源甲羟戊酸途径以提高所需的FPP通量从而成功合成木香烃内酯。此外,在进一步实验筛选适于在大肠杆菌BL21(DE3)中的CYP异源表达系统和木香烃内酯合成酶时,本研究发现与使用其他表达系统相比,使用pCWOri载体的菌株Cos-4可产生更多的木香烃内酯(图3-b)。且与生菜来源的COS相比,在大肠杆菌中菊苣来源的COS更有利于合成木香烃内酯,这与先前的报道结果一致。
向培养基中添加血红素合成前体能够增强CYP的表达,同时规避大肠杆菌中血红素摄取系统的缺陷。然而,由于前体的细胞毒性以及与诱导剂共同添加所产生的代谢负担会显著抑制细菌生长。因此决定进一步对诱导剂和前体浓度、诱导时机、发酵温度以及培养基进行优化,也在一定程度上减轻了细胞损伤、促进了微生物生长,并提高了目标产物合成的效率。
研究结果证实了利用大肠杆菌进行木香烃内酯合成的可行性,并为其他倍半萜内酯的合成提供了一种参考策略。但真核CYP的异源表达仍然面临着许多的挑战,提高真核CYP异源表达效率,提高改善CYP活性,或许可以成为提高其他倍半萜内酯产量的关键所在。
[1] ZHAO L B, WANG X M, CHANG Q, et al.Neferine, a bisbenzylisoquinline alkaloid attenuates bleomycin-induced pulmonary fibrosis[J].European Journal of Pharmacology, 2010, 627(1-3):304-312.
[2] PENG Z X, WANG Y, FAN J H, et al.Costunolide and dehydrocostuslactone combination treatment inhibit breast cancer by inducing cell cycle arrest and apoptosis through c-Myc/p53 and AKT/14-3-3 pathway[J].Scientific Reports, 2017, 7:41254.
[3] BARRERO A F, OLTRA J E,  LVAREZ M, et al.New sources and antifungal activity of sesquiterpene lactones[J].Fitoterapia, 2000, 71(1):60-64.
LVAREZ M, et al.New sources and antifungal activity of sesquiterpene lactones[J].Fitoterapia, 2000, 71(1):60-64.
[4] WEDGE D E, GALINDO J C G, MAC AS F A.Fungicidal activity of natural and synthetic sesquiterpene lactone analogs[J].Phytochemistry, 2000, 53(7):747-757.
AS F A.Fungicidal activity of natural and synthetic sesquiterpene lactone analogs[J].Phytochemistry, 2000, 53(7):747-757.
[5] SETO M, MIYASE T, UMEHARA K, et al.Sesquiterpene lactones from Cichorium endivia L.and C.intybus L.and cytotoxic activity[J].Chemical &Pharmaceutical Bulletin, 1988, 36(7):2423-2429.
[6] DE KRAKER J W, FRANSSEN M C R, JOERINK M, et al.Biosynthesis of costunolide, dihydrocostunolide, and leucodin.demonstration of cytochrome P450-catalyzed formation of the lactone ring present in sesquiterpene lactones of chicory[J].Plant Physiology, 2002, 129(1):257-268.
[7] KIM D Y, CHOI B Y.Costunolide:A bioactive sesquiterpene lactone with diverse therapeutic potential[J].International Journal of Molecular Sciences, 2019, 20(12):2926.
[8] KASSUYA C A L, CREMONEZE A, BARROS L F L, et al.Antipyretic and anti-inflammatory properties of the ethanolic extract, dichloromethane fraction and costunolide from Magnolia ovata (Magnoliaceae)[J].Journal of Ethnopharmacology, 2009, 124(3):369-376.
[9] IKEZAWA N, GÖPFERT J C, NGUYEN D T, et al.Lettuce costunolide synthase (CYP71BL2) and its homolog (CYP71BL1) from sunflower catalyze distinct regio- and stereoselective hydroxylations in sesquiterpene lactone metabolism[J].Journal of Biological Chemistry, 2011, 286(24):21601-21611.
[10] LIU Q, MAJDI M, CANKAR K, et al.Reconstitution of the costunolide biosynthetic pathway in yeast and Nicotiana benthamiana[J].PLoS One, 2011, 6(8):e23255.
[11] HAMBERGER B, BAK S.Plant P450 s as versatile drivers for evolution of species-specific chemical diversity[J].Philosophical Transactions of the Royal Society B: Biological Sciences, 2013, 368(1612):20120426.
[12] RENAULT H, BASSARD J E, HAMBERGER B, et al.Cytochrome P450-mediated metabolic engineering:Current progress and future challenges[J].Current Opinion in Plant Biology, 2014, 19:27-34.
[13] JIANG B, GAO L, WANG H J, et al.Characterization and heterologous reconstitution of Taxus biosynthetic enzymes leading to baccatin III[J].Science, 2024, 383(6683):622-629.
[14] LI S Y, DU L, BERNHARDT R.Redox partners:Function modulators of bacterial P450 enzymes[J].Trends in Microbiology, 2020, 28(6):445-454.
[15] NGUYEN D T, GÖPFERT J C, IKEZAWA N, et al.Biochemical conservation and evolution of germacrene a oxidase in Asteraceae[J].Journal of Biological Chemistry, 2010, 285(22):16588-16598.
[16] ROSANO G L, MORALES E S, CECCARELLI E A.New tools for recombinant protein production in Escherichia coli:A 5-year update[J].Protein Science, 2019, 28(8):1412-1422.
[17] FORDJOUR E, LIU C L, HAO Y P, et al.Engineering Escherichia coli BL21 (DE3) for high-yield production of germacrene A, a precursor of β-elemene via combinatorial metabolic engineering strategies[J].Biotechnology and Bioengineering, 2023, 120(10):3039-3056.
[18] KROGH A, LARSSON B, VON HEIJNE G, et al. Predicting transmembrane protein topology with a hidden Markov model: Application to complete genomes1[J]. Journal of Molecular Biology, 2001, 305(3):567-580.
[19] CHANG M C Y, EACHUS R A, TRIEU W, et al.Engineering Escherichia coli for production of functionalized terpenoids using plant P450 s[J].Nature Chemical Biology, 2007, 3(5):274-277.
[20] ZELASKO S, PALARIA A, DAS A.Optimizations to achieve high-level expression of cytochrome P450 proteins using Escherichia coli expression systems[J].Protein Expression and Purification, 2013, 92(1):77-87.
[21] BARNES H J, ARLOTTO M P, WATERMAN M R.Expression and enzymatic activity of recombinant cytochrome P450 17 alpha-hydroxylase in Escherichia coli[J].Proceedings of the National Academy of Sciences of the United States of America, 1991, 88(13):5597-5601.
[22] MICHENER J K, NIELSEN J, SMOLKE C D.Identification and treatment of heme depletion attributed to overexpression of a lineage of evolved P450 monooxygenases[J].Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(47):19504-19509.
[23] YUAN X J, RIETZSCHEL N, KWON H, et al.Regulation of intracellular heme trafficking revealed by subcellular reporters[J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(35):E5144-E5152.
[24] QUEHL P, HOLLENDER J, SCHÜÜRMANN J, et al.Co-expression of active human cytochrome P450 1A2 and cytochrome P450 reductase on the cell surface of Escherichia coli[J].Microbial Cell Factories, 2016, 15:26.