L-苯丙氨酸(L-phenylalanine,L-Phe)又称为L-苯基-α-氨基丙酸,属芳香族氨基酸。由于其自身重要的生物学功能,在食品、医药、化工等领域均有广泛的应用,可作为食品添加剂提高食品营养价值,也是多种药物的重要中间体,用于抗癌、抗病毒药物的合成等。随着L-Phe的需求日益增加,对其生产能力提出更高的要求。工业上生产L-Phe的方法主要包括化学合成法、酶法及微生物发酵法等,其中微生物发酵法,即以葡萄糖等廉价底物为原料,通过微生物生长代谢合成产物,具有生产成本低、易于放大、环境污染小等优点[1],是目前国内外生产L-Phe的主要手段。
大肠杆菌因其遗传背景清晰、生长速度快、基因改造工具成熟等优势,成为发酵生产L-Phe的常用菌株而得到广泛研究。如图1所示,在大肠杆菌中,L-Phe生物合成途径主要分为中心代谢途径、莽草酸途径和L-Phe分支途径三部分[2]:底物葡萄糖通过糖酵解途径和磷酸戊糖途径等中心代谢途径生成磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)和4-磷酸赤藓糖(erythrose 4-phosphate,E4P),二者在3-脱氧-D-阿拉伯庚糖酸-7-磷酸合酶(3-deoxy-D-arabino-heptulosonate-7-phosphate synthase,DAHP合酶)的催化下缩合生成3-脱氧-D-阿拉伯庚糖酸-7-磷酸(DAHP);DAHP经莽草酸途径进一步代谢,生成分支酸(chorismic acid,CHA)而进入L-Phe分支途径;经过分支酸变位酶/预苯酸脱水酶(pheA基因编码)和氨基转移酶(ilvE、aspC、tyrB基因编码)的催化合成终产物L-Phe。目前,L-Phe大肠杆菌工程菌的开发主要通过理性代谢工程改造,主要策略包括改造葡萄糖转运系统、解除限速酶反馈抑制、强化莽草酸及L-Phe分支途径、平衡PEP/E4P供给、抑制毒副产物积累及加强L-Phe转运等[3-5]。LIU等[6]对大肠杆菌进行改造,通过灭活磷酸转移酶系统(phosphotransferase system,PTS)和组合调控galP和glk增加PEP供应,同时改造TyrR转录因子、优化aroD启动子等,获得的Xllp21菌株在5 L发酵罐中L-Phe产量达72.9 g/L,是原始菌株的1.62倍;门佳轩等[7]对大肠杆菌进行系统代谢工程改造,通过解除PheA反馈抑制、优化莽草酸途径酶表达强度、引入解除反馈抑制的DAHP合成酶以及敲除丙酮酸激酶PykF构建了一株非缺陷型、无质粒的工程菌株PHE12,L-Phe产量达81.8 g/L;DING等[8]构建E4P生物传感器,经过突变筛选分离得到菌株DP01-H4,在此基础上进一步引入aroG和aroF基因和共表达质粒构建重组菌株DP02,在5 L发酵罐中高效生产L-Phe达到106 g/L,是迄今为止报道的最高产量。
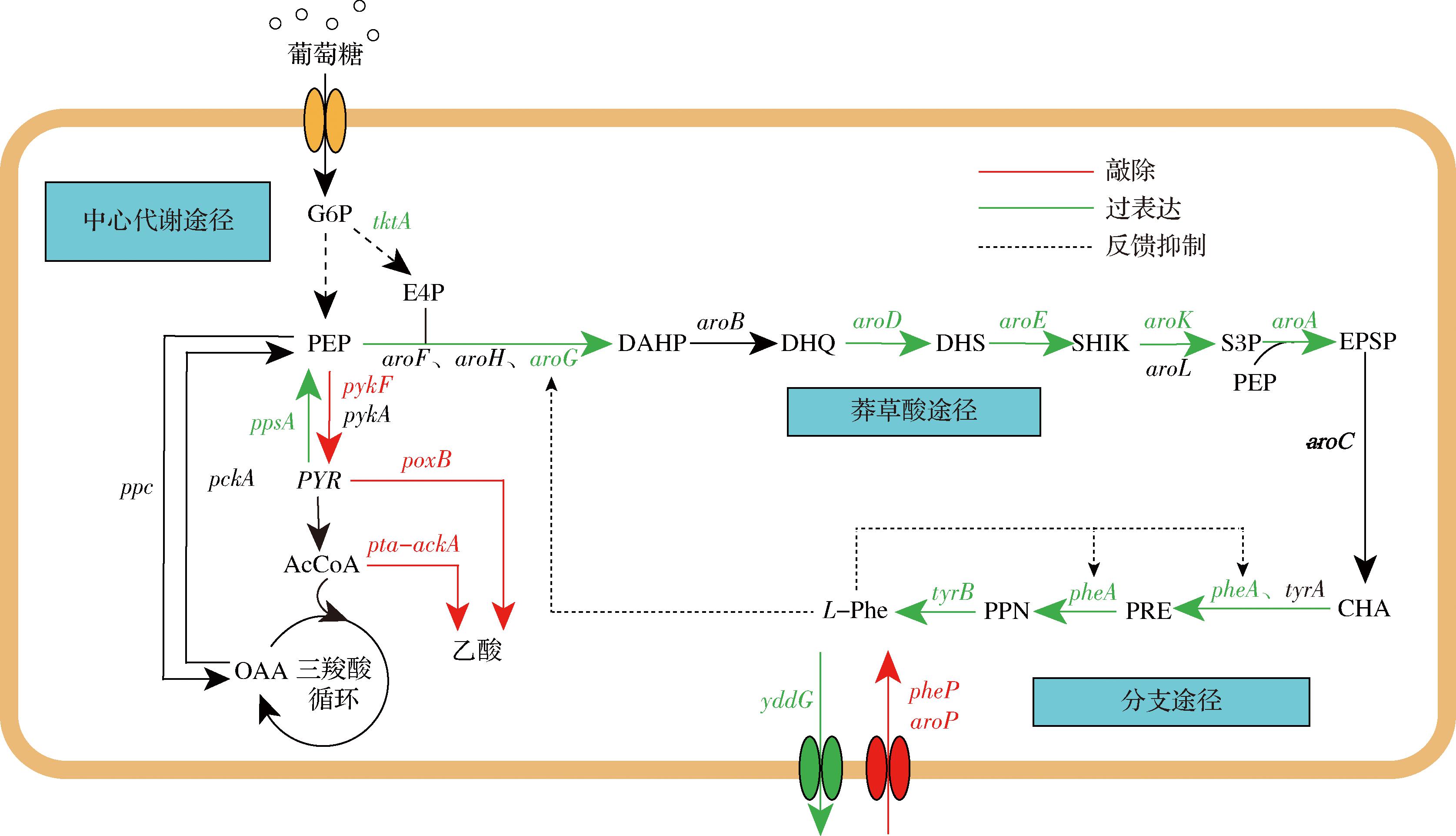
图1 大肠杆菌L-Phe生物合成途径和代谢工程策略
Fig.1 L-Phe biosynthetic pathway and metabolic engineering strategies in E.coli
注:G6P-6-磷酸葡萄糖;PEP-磷酸烯醇式丙酮酸;E4P-4-磷酸赤藓糖;PYR-丙酮酸;AcCoA-乙酰辅酶A;OAA-草酰乙酸;DAHP-3-脱氧-α阿拉伯庚酮糖-7-磷酸;DHQ-3-脱氢奎宁酸;DHS-3-脱氢莽草酸;SHIK-莽草酸;S3P-3-磷酸莽草酸;EPSP-5-烯醇丙酮莽草酸-3-磷酸; CHA-分支酸;PRE-预苯酸;PPN-苯丙酮酸。
尽管传统的遗传修饰策略对代谢途径进行了有效优化,但由于大肠杆菌中L-Phe的代谢网络调控机制较复杂,而常规改造策略对多个基因进行同时修饰的能力较弱,可能导致代谢流阻塞、毒副产物积累、生长生产不平衡等问题,存在一定的局限性。为了规避以上问题,进一步提高工程菌株的生产表现,采用全局转录机器工程(global transcription machinery engineering,gTME)策略,即对细胞转录因子等元件进行突变修饰,改变细胞转录水平,实现复杂代谢网络中多基因同时微调,结合定向进化和筛选技术,筛选出理想突变体,已成为一种开发高效细胞工厂的有效工具[9]。近年来,gTME已成功应用于细胞工厂优化,通过对RpoD、RpoS、RpoE和FecI等σ70因子和CRP、ArcAB、IHF和FNR等转录因子进行突变,有效提高工程菌对乙醇、丁醇和乙酸盐的抗逆耐受性[10-12]、提高目标产物苏氨酸、酪氨酸、透明质酸、番茄红素和3-羟基丙酸[13-16]等产量,具有广阔的应用前景。
本研究旨在构建一株高效合成L-Phe的工程菌株,从基因组层面对大肠杆菌W3110进行改造。首先通过解除反馈抑制、强化莽草酸途径、提高前体物PEP和E4P的供应、截断副产物乙酸合成途径和强化L-Phe向胞外转运等策略,重构L-Phe合成代谢网络;在此基础上采用gTME策略建立突变菌株文库,通过高通量筛选得到理想的高产菌株,为L-Phe生物合成的基础研究和工业化生产提供参考依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
本研究使用的质粒和菌株见电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.042683,下同)、附表2所示,涉及引物见附表3。
表1 莽草酸途径各酶表达水平优化对L-Phe合成的影响
Table 1 Effect of optimizing the expression level of the enzymes of the shikimate pathway on the biosynthesis of L-Phe

菌株基因型OD600L-Phe产量/ g/L PHE-5PHE-4 yciQ Ptrc-tyrB52.33±3.9312.61±0.46PHE-6PHE-5 yjgx PproA-aroA39.90±1.0112.47±1.55PHE-7PHE-5 yjgx PBBa_j23100-aroA44.53±3.5413.40±0.53PHE-8PHE-5 yjgx PproD-aroA42.30±0.6413.15±1.04PHE-9PHE-7 gapC Ptrc-aroK45.36±1.3514.06±1.22PHE-10PHE-7 gapC PBBa_j23105-aroK44.14±1.2713.32±1.35PHE-11PHE-7 yjit Ptrc-aroE L241I 42.05±1.1414.65±0.54PHE-12PHE-7 yjit Ptrc-aroE T61W 41.07±1.9814.77±0.36PHE-13PHE-7 yjit Ptrc-aroE L241I/T61W 43.35±3.3215.12±0.48PHE-14PHE-13 yghx PproD-aroD41.38±2.5515.46±0.43PHE-15PHE-13 yghx PBBa_j23100-aroD41.77±1.2515.96±0.44PHE-16PHE-13 yghx PproA-aroD41.99±2.1315.35±0.35
1.1.2 培养基配方
LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10。
MY1培养基(g/L):酵母粉0.5,NH4Cl 2,(NH4)2SO4 5,KH2PO4 3,K2HPO4 7.3,3-吗啉丙磺酸 8.4,NaCl 0.5,MgSO4 0.24,葡萄糖10。
种子液培养基(g/L):酵母粉10,(NH4)2SO4 5,KH2PO4 3,MgSO4·7H2O 2,柠檬酸钠 2,NaCl 1,CaCl2·2H2O 0.015,FeSO4·7H2O 0.03,另外加入2%(体积分数,下同)的苯酚红溶液,0.1%(体积分数,下同)微混溶液,葡萄糖10。
摇瓶发酵培养基(g/L):酵母粉5,(NH4)2SO4 5,KH2PO4 3,MgSO4·7H2O 2,柠檬酸钠 2,NaCl 1,CaCl2·2H2O 0.015,FeSO4·7H2O 0.03,另外加入2%的苯酚红溶液,0.1%微混溶液,初始葡萄糖10。
发酵罐培养基(g/L):酵母粉3,蛋白胨1,(NH4)2SO4 2,KH2PO4·3H2O 4,MgSO4·7H2O 1.5,柠檬酸1,FeSO4·7H2O 0.02,谷氨酸1,MnSO4 0.01,维生素B1、维生素B3、维生素B5、维生素B12各0.002,0.1%微混溶液,初始葡萄糖20。
微混溶液(g/L):Na2MoO4·2H2O 2.5,Al2(SO4)·18H2O 1.25,NiCl2·6H2O 1.6,CaCl2·2H2O 10,CuSO4·5H2O 0.4,CoCl2·6H2O 1.8,ZnSO4·2H2O 0.5,H3PO3 0.14。
抗生素工作浓度(mg/L):卡那霉素50,氨苄青霉素100,奇霉素100。
1.2 仪器与设备
RePureD(B)基因扩增仪,杭州柏恒科技有限公司:Agilent 1200高效液相色谱仪,安捷伦科技(中国)有限公司;5 L自动控制发酵罐,上海保兴生物设备工程有限公司;V-1200可见分光光度计,翱艺仪器(上海)有限公司;SpectraMax M3多功能酶标仪,美谷分子仪器(上海)有限公司。
1.3 菌株构建方法
基于CRISPR/Cas9技术的多元质粒系统编辑方法对大肠杆菌进行基因改造。该系统包括pGRB和pREDCas9这2个部分。其中pGRB质粒主要由启动子、gRNA-Cas9蛋白结合区域、终止子和氨苄青霉素抗性基因组成。pREDCas9质粒主要由Cas9蛋白、RED重组酶及pGRB质粒消除系统组成,含奇霉素抗性。Cas9蛋白结合pGRB转录出的gRNA形成复合物并识别到基因靶位,实现靶位点基因双链断裂,同时,目的DNA片段与靶位点基因发生同源重组从而被整合到基因组上,实现了目的基因的编辑。
具体操作如下:在靶位点基因上选择合适的20 bp。通过同源重组将20 bp的双链DNA与线性化pGRB结合形成pGRB。使用Primer 5.0以目的基因及靶位点基因为模板设计引物,通过PCR方法扩增目的基因及靶位点基因的上下同源臂,再利用重叠PCR获得目的DNA片段。将pREDCas9质粒导入感受态细胞中,经菌落PCR筛选出阳性转化子后,将阳性菌落制备成新的电转感受态,在制备前加入0.1 nmol/L的异丙基-β-D-硫代半乳糖苷溶液诱导重组酶的表达,将目的DNA片段与pGRB质粒加入制备好的感受态细胞中进行电转化,感受态细胞复苏2 h后,取适量涂布于含氨苄青霉素和奇霉素的LB固体培养基上,32 ℃过夜培养。随机挑选单菌落进行菌落PCR验证,筛选阳性转化子。
筛选得到的阳性菌株接入含有0.2%(体积分数)阿拉伯糖的LB液体培养基中培养12 h,诱导pREDCas9质粒上的pGRB质粒消除系统切割pGRB质粒,最后将带有pREDCas9质粒的菌株接入无抗性的LB液体培养基中于42 ℃培养12 h,诱导温敏质粒pREDCas9消除后得到无质粒的菌株。
1.4 L-Phe发酵生产
1.4.1 摇瓶发酵
活化培养:吸取10~20 μL菌液至装有5 mL LB液体培养基的摇管中,37 ℃,220 r/min振荡培养12 h后,使用接种环在LB平板上划线,37 ℃过夜培养后挑取单菌落,接入液体培养基进行种子培养。
种子培养:活化后的菌株接入装有28 mL种子液培养基的500 mL圆底摇瓶中,加入2 mL 15%(质量分数,下同)的葡萄糖溶液,在37 ℃、220 r/min条件下培养12 h,作为种子液。
摇瓶发酵培养:将3 mL种子液接至装有25 mL发酵培养基的500 mL挡板摇瓶中,加入15%的葡萄糖溶液2 mL,于37 ℃、220 r/min条件下进行培养。发酵过程通过补加氨水维持pH在适宜范围内。当发酵液溶液颜色不再变化时,补加1 mL 60%的葡萄糖溶液。发酵24 h后留样,-20 ℃保存。
1.4.2 发酵罐分批补料发酵
挑选单菌落,接种至含30 mL LB培养基的摇瓶中,于37 ℃、250 r/min下培养过夜;将培养液接种到种子罐中进行发酵培养,设定pH值为7.0,温度37 ℃,溶氧维持在30%左右。当种子罐中OD600值达到15左右,接入装有2 L发酵培养基的5 L发酵罐中,接种量15%。发酵培养条件为pH 7.0、温度37 ℃、溶氧30%。当罐中葡萄糖耗尽时,流加60%葡萄糖溶液,维持葡萄糖质量浓度在0.1~3 g/L。每4 h取样检测发酵液中葡萄糖和L-Phe的质量浓度。
1.5 L-Phe检测基因回路的构建与表征
采用无缝克隆法构建具有L-Phe感应功能的质粒pTE1。利用Prime STAR HS DNA聚合酶,通过PCR扩增线性化载体pTrc99a(引物分别为99a-S和99a-A)、PtyrR-tyrR片段(以W3110基因组为模板,引物分别为tyrR-mtr-S和tyrR-mtr-S)、用引物Pmtr-tyrA-S和gfp-99a-A扩增Pmtr-gfp片段。利用ClonExpress Ⅱ One Step Cloning Kit酶对质粒和目的片段进行同源重组得到质粒pTE1。将质粒pTE1和阳性对照质粒pCON1,分别转化至野生型大肠杆菌W3110,形成菌株TE1和CON1。两菌株分别接种至含有100 mg/L氨苄青霉素的LB培养基中,在37 ℃、250 r/min下培养12 h后,分别接种至含5 mL的MY1培养基的摇管中并保持初始OD600值为0.3。分别向培养基中加入不同浓度的L-Phe(L-Phe最终质量浓度分别为0、0.1、0.5、0.8、1、1.5、2 g/L),培养16 h后,测定细胞密度和荧光强度(激发波长488 nm,发射波长511 nm)。
1.6 rpoD和rpoA基因突变体文库构建与高产菌株筛选
首先利用无缝克隆法构建含有野生型rpoA、rpoD基因的质粒pSL1和pSL2:利用Prime STAR HS DNA聚合酶通过PCR扩增线性化载体pET28a(引物分别为pET28-S、pET28a-rpoA-A和pET28a-rpoD-A)、PropA-rpoA序列(引物PropA-rpoA-S和PropA-rpoA-A)和PropD-rpoD序列(引物PropD-rpoD-S和PropD-rpoD-A),利用ClonExpress Ⅱ One Step Cloning Kit酶进行DNA片段重组分别得到质粒pSL1和pSL2。两质粒分别转化到大肠杆菌XL1-Red中,得到菌株XLS1和XLS2。分别将XLS1和XLS2在含有50 mg/L卡那霉素的LB培养基中培养12 h (37 ℃、250 r/min);随后将培养物转移到含有20 mg/L卡那霉素的新鲜LB培养基中。经过5次传代培养,形成质粒pSL1和pSL2的突变体文库。提取质粒,转化至包含L-Phe检测回路的菌株PHE-51,分别形成菌株PHE-53和PHE-54,得到一系列L-Phe生产的突变菌株。
分别挑选135株PHE-53和135株PHE-54的突变菌单菌落,连同对照菌株PHE-55接种至含有100 mg/L氨苄青霉素和50 mg/L卡那霉素的LB培养基中培养12 h(37 ℃、250 r/min);将种子液接种至含有5 mL MY1培养基的试管中,在37 ℃、250 r/min下培养,18 h后取样检测样品细胞密度和荧光强度(激发波长488 nm,发射波长511 nm)。
挑选与对照菌株相比荧光强度显著提高的3株突变菌,在含有50 mg/L卡那霉素而不含氨苄青霉素的LB培养中传代培养,丢失pTE1质粒。将3株突变菌及对照菌按照1.3.1节中的方法进行摇瓶发酵,培养24 h后检测生物量和L-Phe质量浓度。选取L-Phe质量浓度最高的菌株,提取质粒并进行测序。
1.7 代谢物检测和分析
采用HPLC分析L-Phe和乙酸浓度。L-Phe浓度检测:色谱柱为Agilent C18柱(250 mm×460 mm,5 μm),流动相为10%(体积分数)乙腈溶液。流速1.0 mL/min,柱温40 ℃,紫外检测波长254 nm。乙酸的浓度检测:色谱柱为Bio-Rad Aminex HPX-87H色谱柱(300 mm×7.8 mm,8 μm),流动相为5 mmol/L的硫酸溶液,流速0.6 mL/min,柱温35 ℃,示差检测器。发酵液中葡萄糖质量浓度通过SBA生物传感仪检测。
1.8 数据处理与分析
摇瓶发酵数据为3组平行实验的结果“平均值±标准差”。采用Excel 2016、Origin、Graph Pad prism进行实验数据处理与统计分析,利用t检验双尾分布对发酵数据进行组间单向方差分析。P<0.05时表示差异显著;P<0.01表示差异非常显著。
2 结果与分析
2.1 利用经典代谢工程策略构建L-Phe生产菌株
2.1.1 解除反馈抑制
在L-Phe合成代谢途径中,DAHP合酶及分支酸变位酶/预苯酸脱水酶均受到L-Phe的反馈抑制,是限制L-Phe合成速率的主要因素之一。研究表明,pheA基因编码的分支酸变位酶/预苯酸脱水酶可以通过缺失调节域(R区)来解除L-Phe的反馈抑制[17];而DAHP合酶有3种同工酶(分别由aroG、aroF、aroH基因编码),活性分别受苯丙氨酸、酪氨酸、色氨酸的反馈抑制,其中AroG的第146位氨基酸由天冬氨酸变为天冬酰胺能有效地解除L-Phe的反馈抑制[18]。
本研究构建解除反馈抑制的pheAfbr片段(即删除了pheA基因序列的后260 bp),由启动子Ptrc控制整合到野生型大肠杆菌W3110的tyrR基因位点上,得到菌株PHE-1。在此基础上,整合Ptrc启动子控制的解除反馈抑制的aroGfbr基因(即aroG基因第436位碱基由G变为A),得到菌株PHE-2。为进一步强化代谢通量,在菌株PHE-2基因组上继续整合pheAfbr和aroGfbr基因,分别得到菌株PHE-3和PHE-4。摇瓶发酵结果如图2所示,PHE-4的产量为11.86 g/L,较PHE-1提高了121.27%,较PHE-2提高了51.27%,较PHE-3提高了21.39%,说明解除AroG和PheA的反馈抑制能有效提高L-Phe产量,且适当提高表达量有利于L-Phe的合成。
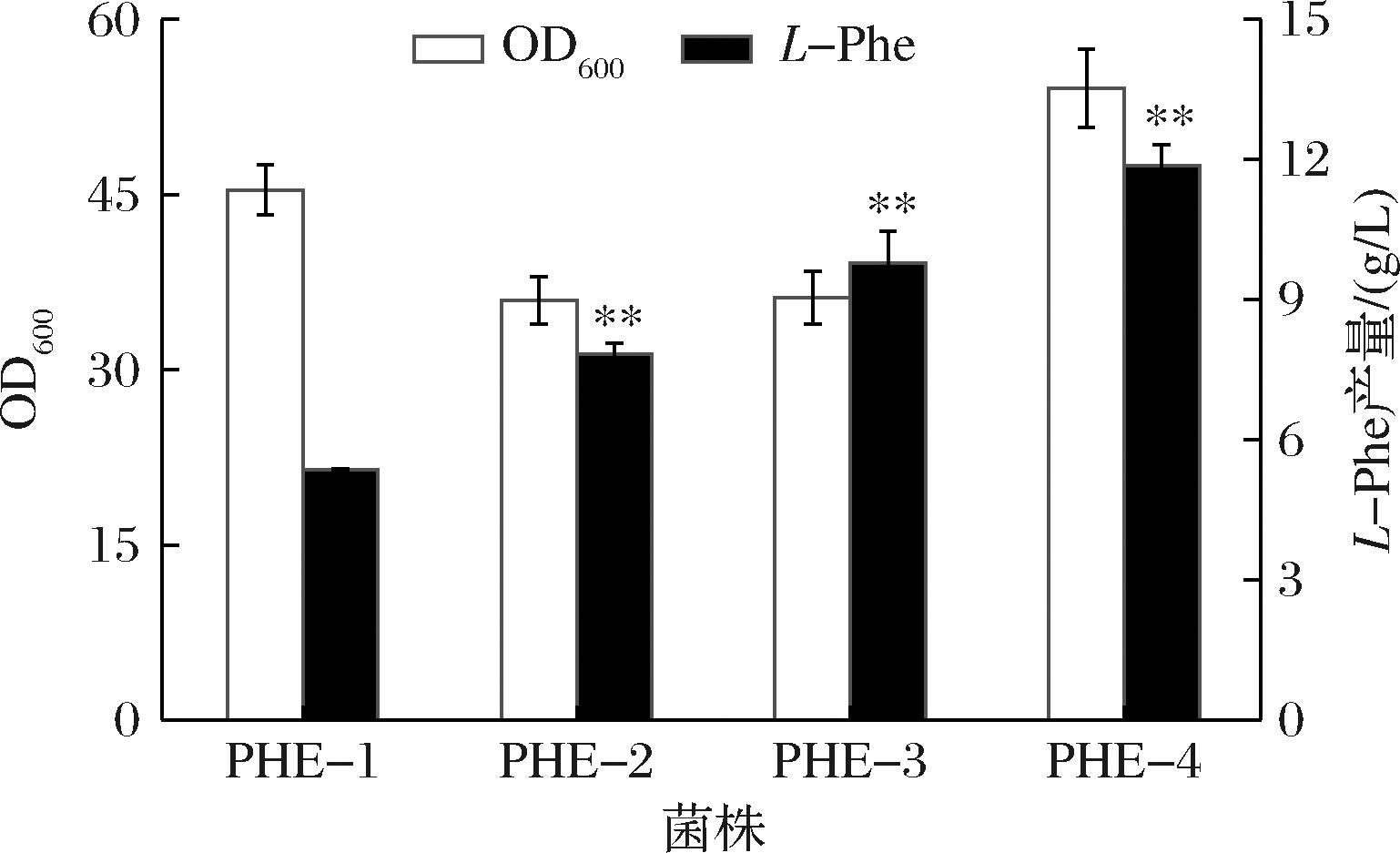
图2 过表达pheAfbr及aroGfbr对L-Phe产量的影响
Fig.2 Effect of overexpression of pheAfbr and aroGfbron L-Phe production
注:*,P<0.05表示差异显著;**,P<0.01表示差异非常显著(下同)。
2.1.2 优化莽草酸途径
莽草酸途径作为连接中心代谢途径和L-Phe分支途径的关键节点,其各酶表达量和活性的精确调控对代谢产物流向具有重要影响。本研究首先通过强化编码转氨酶tyrB基因构建了工程菌株PHE-5,摇瓶发酵证实该菌株的L-Phe产量较原始菌株提高了6.24%。基于PHE-5,进一步进行莽草酸途径的优化。前人研究指出,在莽草酸途径的6种酶中,3-脱氢奎尼酸脱水酶(3-dehydroquinate dehydratase,AroD)、莽草酸激酶(AroL/AroK)和5-烯醇丙酮酰莽草酸-3-磷酸合成酶(AroA)的适度强化可提升L-Phe产量[19],因此本研究对此3种酶的表达水平进行启动子工程优化。此外,aroE基因编码的莽草酸脱氢酶具有双向催化功能,为增强DHS向莽草酸的转化效率,引入aroE基因突变体(aroEL241I、aroET61W、aroET61W/L241I)[20],以期实现代谢途径的进一步优化。
如表1所示,从菌株PHE-5出发,首先在不同强度启动子控制下增强aroA基因的表达(从弱到强分别为PproA、PBBa_j23100和PproD),分别构成菌株PHE-6、PHE-7和PHE-8。摇瓶发酵结果显示,启动子PBBa_j23100调控下过表达aroA可一定程度上提高L-Phe的合成能力,产量达13.40 g/L,较PHE-5提高了6.26%。在PHE-7基础上,对aroK基因的表达进行强化,向基因组中分别整合在较强启动子Ptrc和较弱启动子PBBa_j23105控制下的aroK基因,但二者L-Phe产量与出发菌株相比未显示出明显提升,因此选取PHE-7进行后续改造。为进一步减少莽草酸途径的中间产物积累,在PHE-7中过表达aroE的突变体aroEL241I、aroET61W和aroET61W/L241I,得到的菌株PHE-11、PHE-12和PHE-13与PHE-7相比L-Phe产量均有所提升,表明此3种突变体均表现出较高酶活性,其中PHE-13产量为15.12 g/L,较PHE-7提高了12.84%。在PHE-13的基础上调节基因aroD的表达,结果表明,在中等强度启动子PBBa_j23100调控下过表达aroD可将L-Phe产量提高5.56%。通过对莽草酸途径的优化可知,对AroA、AroE及AroD酶表达水平或酶活性的适度提高可有效疏通莽草酸途径代谢流,提高菌体生产L-Phe的能力,最终菌株PHE-15产量达到15.96 g/L。
2.1.3 平衡前体物的供应
PEP和E4P是莽草酸途径的重要前体,其二者的供应是L-Phe生产的瓶颈之一。其中PEP既参与DAHP的合成,又可以被丙酮酸激酶催化生成丙酮酸而进入三羧酸循环,同时参与PTS代谢等,被多条途径分流,因此其进入莽草酸途径的代谢流受到一定影响。为了提高PEP的供应,本研究尝试弱化或敲除编码丙酮酸激酶的pykF基因,并强化编码磷酸烯醇式丙酮酸合成酶基因ppsA;同时,由于PEP与E4P协同反应缩合形成DAHP,尝试通过适当过表达转酮酶基因tktA增强E4P的合成,平衡E4P与PEP的供应,从而优化碳通量。
如图3所示,本研究对基因tktA、pykF和ppsA的表达水平进行组合优化:其中tktA分别在低、中、高3种强度启动子控制下进行过表达(从弱到强分别为PM1-12、Ptrc和PBBa_j23101);在此基础上,尝试敲除pykF基因或利用中等强度启动子PBBa_j23105控制pykF基因;对于ppsA基因采用中等强度启动子Ptrc和强启动子PBBa_j23100进行过表达,由此共产生27个工程菌株。由摇瓶发酵结果可知,PHE-17、PHE-18和PHE-19的L-Phe产量均低于PHE-15,表明仅过表达tktA基因无法提高L-Phe产量,这可能是由于E4P与PEP的供应不平衡;菌株PHE-20至PHE-28中,pykF基因均被敲除,菌体生长受到严重影响,且L-Phe产量均未提高;PHE-29至PHE-37中,在中等强度启动子PBBa_j23105控制下对pykF进行回补,生长抑制得到缓解。L-Phe产量最高的菌株PHE-34,即tktA(PBBa_j23101)-pykF(PBBa_j23105)-ppsA(Ptrc),其L-Phe产量达17.10 g/L,比PHE-15提高了7.14%,表明对PEP-E4P供应的优化可有效提高L-Phe的生产能力。
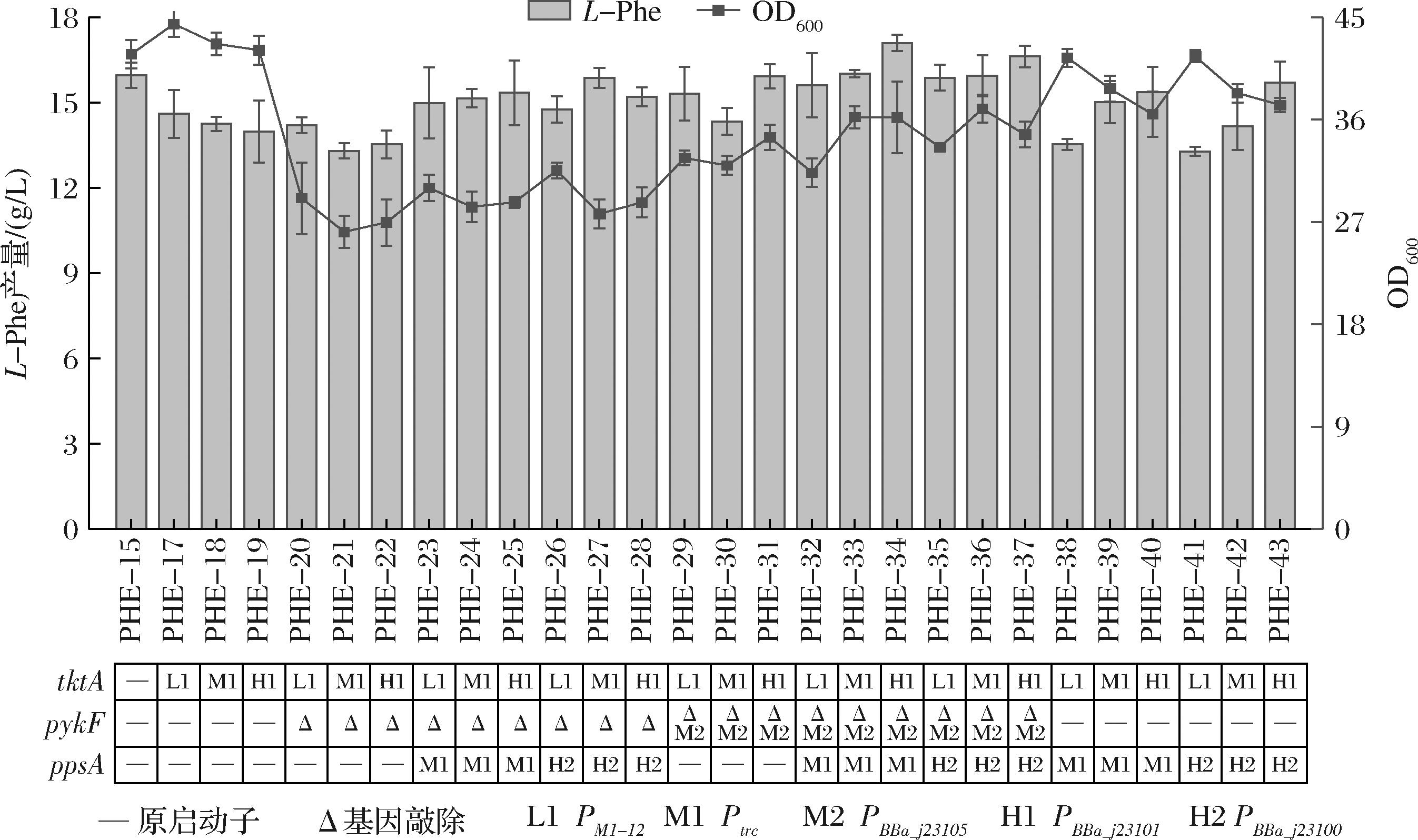
图3 tktA、ppsA和pykF基因的表达水平L-Phe合成的影响
Fig.3 Effect of expression levels of tktA,ppsA,and pykF genes on L-Phe biosynthesis
2.1.4 失活乙酸合成途径
菌株发酵培养过程中,糖酵解途径与产物合成途径不平衡会导致副产物乙酸积累,对菌体生长和生产效率造成不良影响[21],造成碳源浪费而降低L-Phe的得率。乙酸的合成主要通过2种代谢途径,一是由丙酮酸在丙酮酸氧化酶PoxB的催化下获得,二是乙酰辅酶A通过Pta-AckA途径获得。为降低乙酸的生成,本研究在菌株PHE-34的基础上分别失活乙酸激酶编码基因ackA(PHE-44)、丙酮酸氧化酶编码基因poxB(PHE-45)和磷酸乙酰转移酶编码基因pta(PHE-46),探究各靶点的敲除对菌株的细胞生长、乙酸和L-Phe合成的影响。
摇瓶发酵结果如图4所示,敲除ackA、poxB和pta的菌株中乙酸积累量分别降低了63.03%、29.93%及54.93%;在L-Phe产量方面,ackA的敲除使L-Phe的产量提高了5.20%,菌株PHE-44的L-Phe产量达到17.99 g/L,而基因poxB和 pta的敲除对L-Phe的产量没有明显影响;在细胞生长方面,经独立样本t检验进行统计学分析,PHE-44、PHE-45及PHE-46与原菌株的OD600值没有显著差异(P>0.05),因此在本研究中乙酸途径的失活对菌体生长没有显著影响。以上结果表明,基因ackA的敲除有利于L-Phe的合成和减少乙酸的积累,因此选取菌株PHE-44进行后续研究。
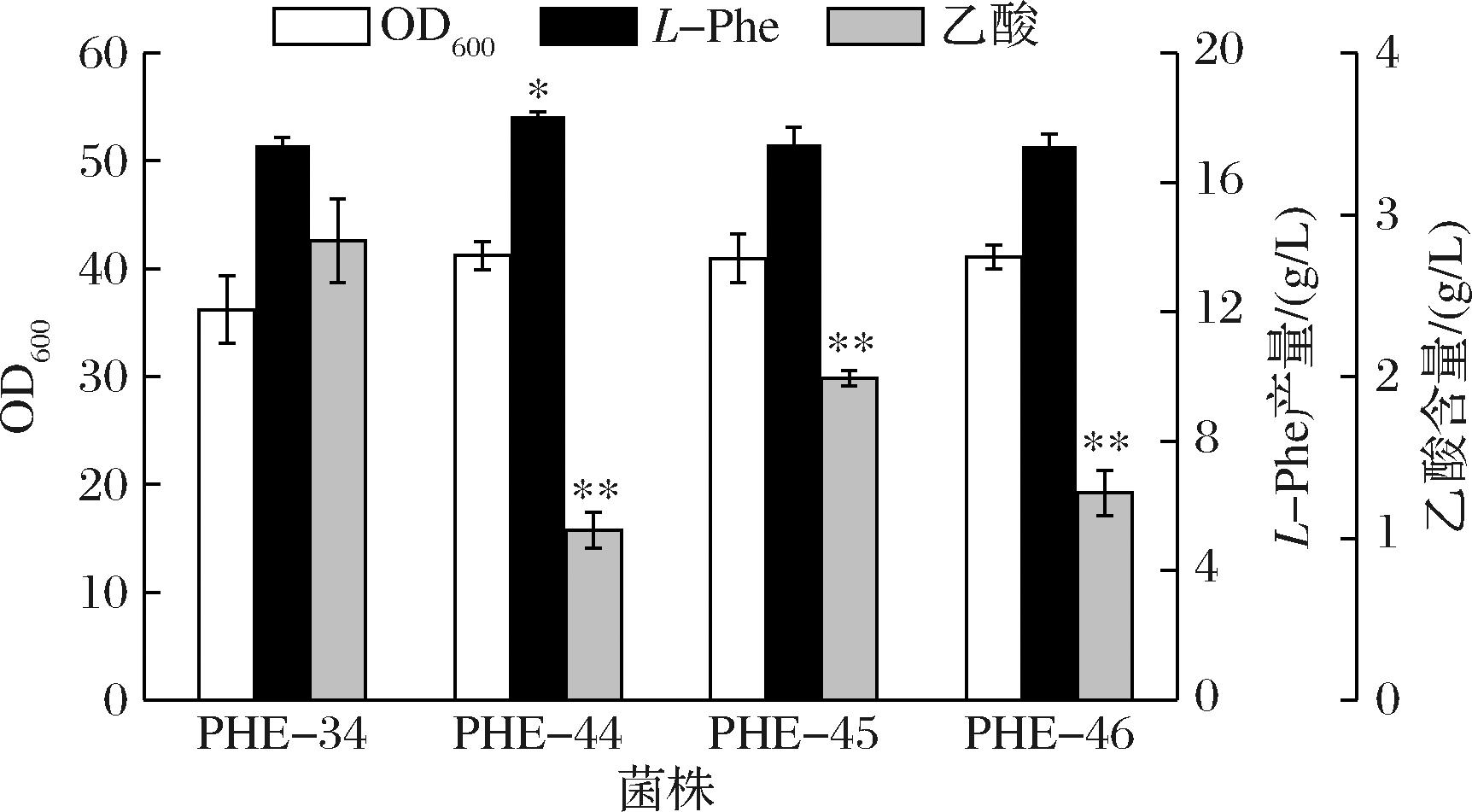
图4 敲除乙酸合成相关基因对L-Phe产量的影响
Fig.4 Effect of deletion of genes related to acetic acid synthesis on the titer of L-Phe
2.1.5 优化L-Phe的转运途径
研究表明,细胞内L-Phe浓度过高会造成反馈阻遏和反馈抑制,同时造成胞内压力和代谢负荷;因此,优化L-Phe的转运途径,促进产物外排可以缓解以上不良影响。改造L-Phe的转运途径主要通过2个方面来进行:一是强化L-Phe向细胞外运输能力,YddG是3种芳香族氨基酸的外向转运蛋白,DOROSHENKO等[22]过表达yddG基因使得胞外L-Phe的积累量提高了3倍;二是弱化外界L-Phe的向内转运,aroP基因编码的芳香族氨基酸转运蛋白及pheP基因编码的L-Phe特异性透过酶是大肠杆菌细胞摄取L-Phe的主要载体。本研究在PHE-44基因组上分别整合了PBBa_j23100和Ptrc启动子控制下的yddG基因,发酵结果显示(图5),胞外L-Phe的积累量分别提高了15.62%和10.69%,表明氨基酸外排蛋白YddG的强化可以有效提高L-Phe的积累能力。在PHE-47的基础上相继敲除了pheP基因和aroP基因,得到菌株PHE-49和PHE-50,L-Phe的产量进一步提高到22.49 g/L和23.76 g/L,证明失活L-Phe向胞内转运蛋白可以提升L-Phe的产量,因此选取PHE-50作为工程菌株进行后续发酵罐测试。
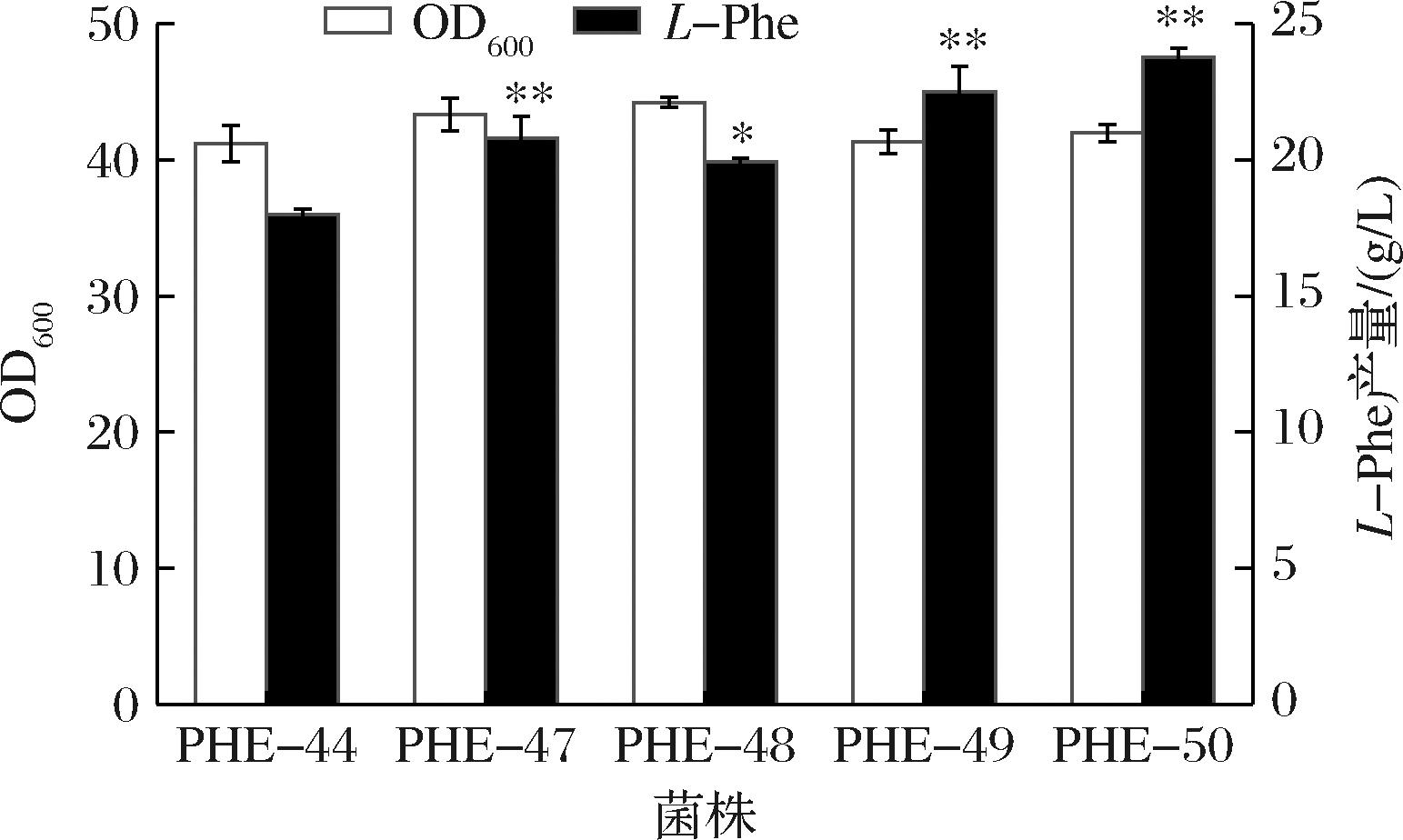
图5 优化L-Phe转运途径对L-Phe产量的影响
Fig.5 Effect of optimization of L-Phe transport pathway on its production
2.2 工程菌株的发酵性能评估
为了对工程菌株的生产性能进行进一步评估,将菌株PHE-50在5 L发酵罐中进行分批补料发酵。发酵过程曲线如图6所示,菌体生物量在发酵初期迅速积累,在24 h后进入稳定期,36 h时OD600值达到最高为123.35,随后OD600值不断下降;经48 h发酵,L-Phe产量达61.49 g/L,糖酸转化率为20%,生产强度为1.28 g/(L·h)。
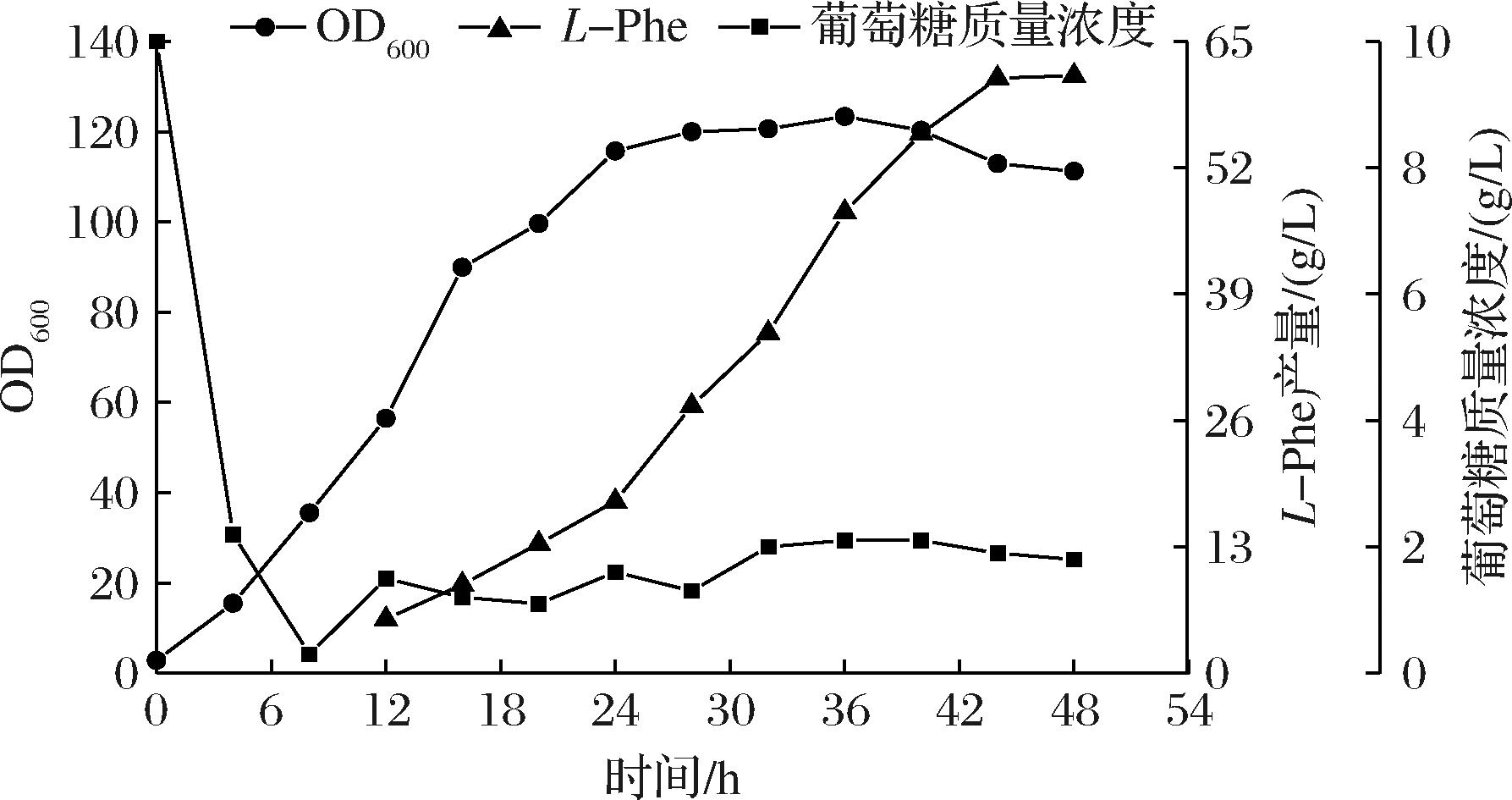
图6 菌株PHE-50在5 L发酵罐分批补料发酵曲线
Fig.6 Fed-batch fermentation curve of strain PHE-50 in a 5 L fermentation tank
2.3 利用gTME筛选高产菌株
以上研究遵循传统代谢工程思路构建了一株L-Phe的合成菌株,尽管该菌株在发酵罐测试中表现出较为稳定的生长和生产能力,但同时体现出发酵后期生物量逐渐下降、生产强度低等局限性。基于L-Phe生产菌株代谢途径的复杂性,为了进一步提高其生物合成能力,本研究采用gTME的方法对菌株进行优化。具体来说,利用大肠杆菌XL1-Red分别对工程菌PHE-50细胞RNA聚合酶中编码σ70因子的rpoD基因和编码α亚基的rpoA基因进行随机突变,建立突变文库;构建基于TyrR转录因子和绿色荧光蛋白的L-Phe检测回路,对突变株进行高通量筛选;对阳性突变株进行基因测序和发酵测试,最终获得生产性能提高的工程菌株。
2.3.1 L-Phe检测基因回路的构建与表征
为了实现工程菌株突变体的高通量筛选,本研究构建了一种响应L-Phe的检测回路。已有报道指出,在大肠杆菌中,编码L-色氨酸特异性转运蛋白的mtr基因表达受TyrR转录因子调控,当存在苯丙氨酸和酪氨酸时,mtr基因表达上调[23-24]。基于此,本研究以pTrc99a为载体,将绿色荧光蛋白基因(gfp)置于Pmtr启动子控制之下,同时引入由原生启动子控制的tyrR基因,构建出L-Phe检测回路质粒pTE1(图7-a)。该设计原理为:在L-Phe质量浓度较高时,gfp基因被激活,展现出较高荧光强度,从而便于筛选高产突变体。
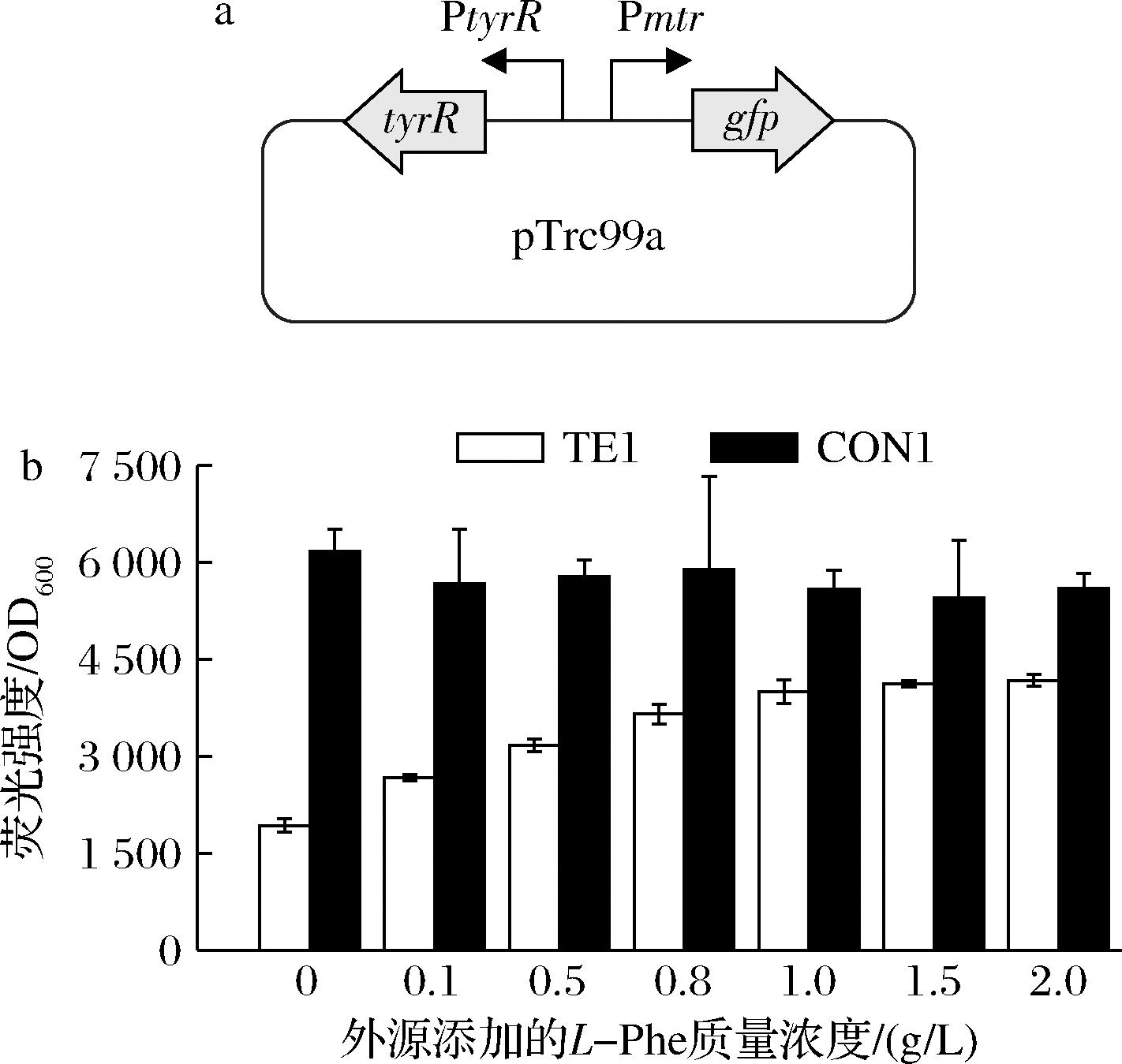
a-L-Phe检测基因线路质粒示意图;b-外源添加L-Phe浓度与荧光强度的关系
图7 L-Phe检测基因回路的构建与表征。
Fig.7 Construction and characterization of L-Phe detection genetic circuit
为表征该检测回路的动态范围及检测能力,将pTE1质粒转化至大肠杆菌W3110中,获得菌株TE1。在菌株培养基中添加不同质量浓度的L-Phe,经摇管培养16 h后测定荧光强度。结果如图7-b所示,在一定范围内,菌株TE1的平均荧光强度与外源添加的L-Phe质量浓度呈正相关关系。当外源添加L-Phe质量浓度为1.5 g/L时,绿色荧光蛋白的表达量相较于基础表达水平提高了2.2倍;而当L-Phe的添加量增至2 g/L时,荧光强度未再增强。此外,将携带组成型启动子Ppdc控制下的gfp基因的质粒pCON1转化至W3110中,得到菌株CON1作为对照组,CON1的荧光强度不受L-Phe质量浓度变化的影响。上述结果充分表明,所构建的基因回路具备预期的L-Phe检测功能,其检测范围确定为0~1.5 g/L。
2.3.2 L-Phe生产菌株的突变与筛选
为了引入L-Phe工程菌株表型多样性,本研究分别建立基因rpoA和rpoD的突变文库。以pET28a为载体,分别构建携带rpoA和rpoD基因的质粒pSL1和pSL2;将两质粒分别转化至大肠杆菌XL1-Red中,通过细胞繁殖进行基因随机突变,产生基因突变文库后将质粒提取转化至携带L-Phe检测回路的工程菌PHE-51中,随机挑选共270个工程菌突变体(其中135个携带rpoA突变基因,135个携带rpoD突变基因)。将270个突变菌株及对照组PHE-55在摇管中培养18 h后取样,在96孔板中分析荧光强度,结果见附图1。270个菌株中,215个突变体比对照菌株荧光强度低,即L-Phe的产量低,55个突变体的L-Phe产量有所提高,其中8个突变体的L-Phe产量提高40%以上,表明gTME策略对工程菌株的优化有效。
选取L-Phe生产能力最强的3株突变菌RpoD15、RpoD129和RpoA133,消除pTE1质粒,在摇瓶中培养以进一步验证其生产性能;同时将pET28a空质粒转化至PHE-50工程菌株作为对照。发酵结果如图8所示,3株突变菌的L-Phe产量均高于对照,其中RpoD129的L-Phe的产量最高,达31.59 g/L,比对照组提高了46.3%。此外,RpoD129和RpoA133的细胞密度(OD600值)分别为36.58和38.74,均高于对照组。以上结果表明,本研究利用gTME策略成功创建了L-Phe生产能力和菌体生长速率均更高的理想表型,选择突变株RpoD129进行后续研究。
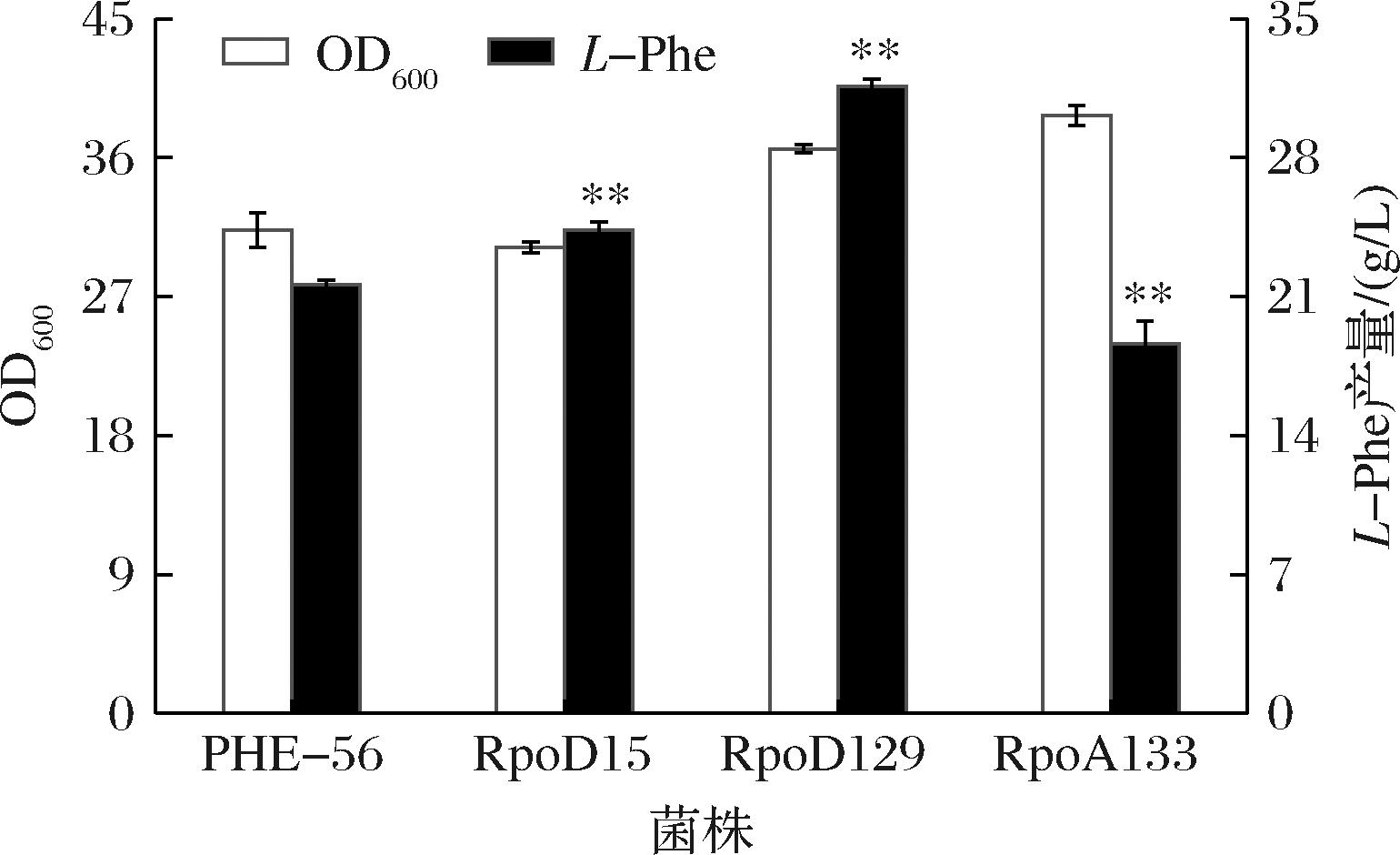
图8 突变菌株与对照菌株PHE-56摇瓶发酵结果
Fig.8 Results of shake flask fermentation of mutant strain and control strain PHE-56
2.3.3 高产突变体的发酵罐测试
为了确定RpoD129的突变体特征,对其携带的pSL2质粒进行测序,结果表明,在启动子和RpoD编码区共有49个单碱基突变位点。据报道,RpoD作为重要σ70因子,协同全局转录因子构成gTME的调控网络,对1 000多个基因的转录水平有调节作用[25];RpoD中的大量点突变对工程菌的代谢网络造成扰动,可能改变菌体生长、L-Phe合成以及菌体抗逆性等相关基因的转录水平,从而实现菌株生产性能的强化。
为了构建一株无质粒的工程菌株,本研究将突变后的PrpoD-rpoD序列整合到PHE-50的基因组中得到PHE-57,并进一步测试其在5 L发酵罐中的生产表现。分批补料发酵结果如图9所示,菌体生物量在24 h后趋于稳定,且在24~48 h期间无明显降低,最高OD600值达到130.45(40 h);L-Phe产量最高达到79.68 g/L,生产强度达1.66 g/(L·h),糖酸转化率为22%。以上结果表明,经gTME策略优化后的菌株PHE-57在生长速率和生产能力方面均显著优于PHE-50。
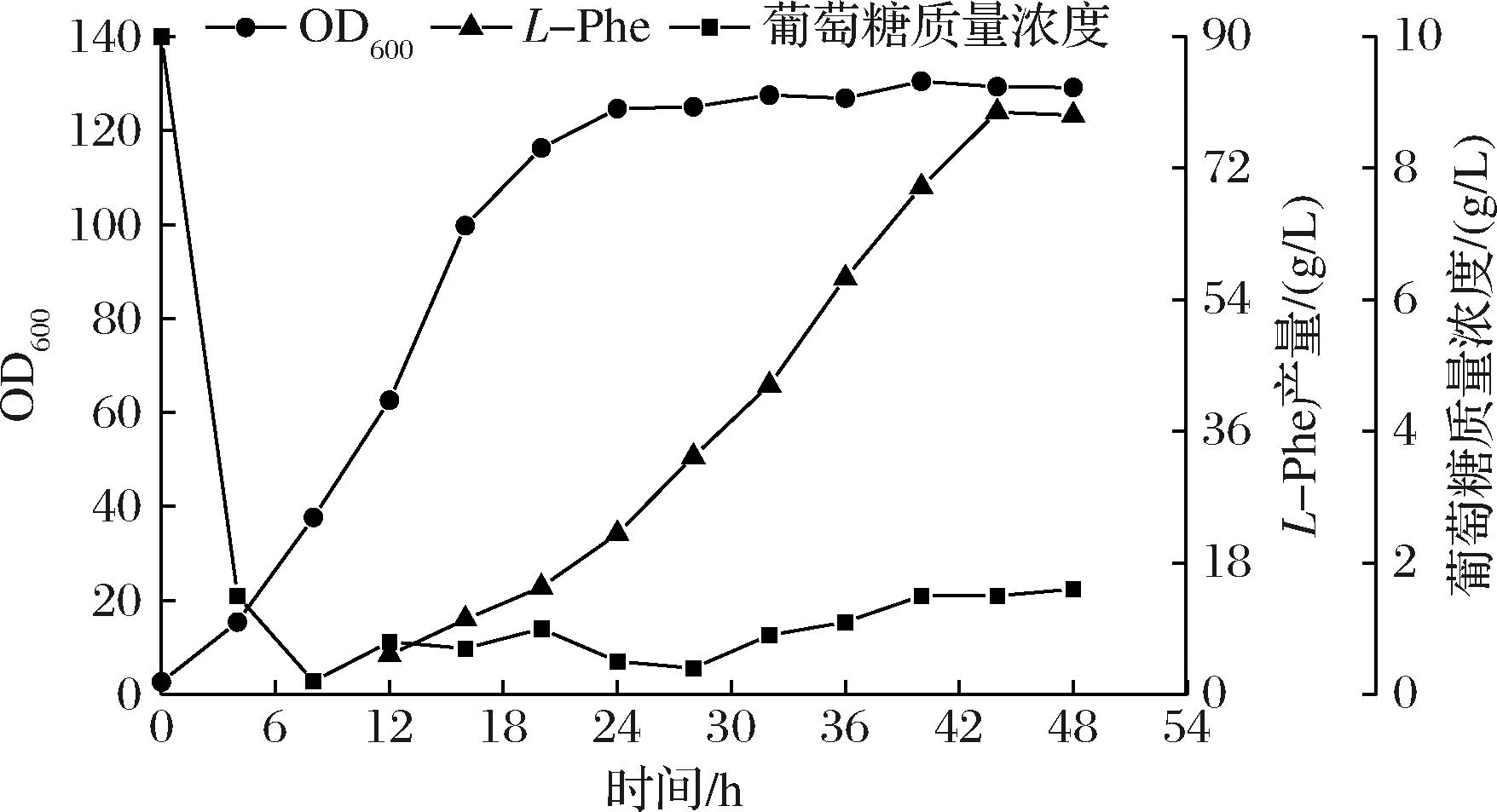
图9 菌株PHE-57在5 L发酵罐分批补料发酵曲线
Fig.9 Fed-batch fermentation curve of strain PHE-57 in a 5 L fermentation tank
3 结论与讨论
本研究首先采用传统代谢工程手段构建了一株生产L-Phe的大肠杆菌工程菌,具体包括:1)在大肠杆菌W3110的基因组上整合双拷贝的pheAfbr和aroGfbr基因,解除L-Phe合成途径的反馈抑制;2)对AroA、AroK、AroE及AroD的表达水平或酶活性进行优化,增强莽草酸途径的代谢通量;3)对基因tktA、pykF和ppsA的表达水平进行组合优化,平衡PEP和E4P的供应;4)敲除乙酸合成相关的ackA基因,减少副产物乙酸合成;5)过表达yddG基因和敲除pheP和aroP基因,强化L-Phe向胞外的转运。以葡萄糖为碳源,工程菌PHE-50在5 L发酵罐分批补料发酵48 h后,L-Phe产量达61.49 g/L,糖酸转化率为20%,生产强度为1.28 g/(L·h)。
在此基础上,通过gTME策略,对L-Phe生产菌株进行优化,成功构建了高产突变体。通过构建基于TyrR转录因子的L-Phe检测回路,结合rpoA和rpoD基因的随机突变文库,筛选理想突变株。构建的无质粒工程菌株PHE-57在5 L发酵罐中表现出良好的生产性能,L-Phe产量达到79.68 g/L,生产强度为1.66 g/(L·h),糖酸转化率为22%。这些结果展示了gTME策略在代谢工程中的高效性,为L-Phe的工业化生产提供了有利的菌株基础。
[1] RODRIGUEZ A,MART NEZ J A,FLORES N,et al.Engineering Escherichia coli to overproduce aromatic amino acids and derived compounds[J].Microbial Cell Factories,2014,13(1):126.
NEZ J A,FLORES N,et al.Engineering Escherichia coli to overproduce aromatic amino acids and derived compounds[J].Microbial Cell Factories,2014,13(1):126.
[2] LIU X Z,NIU H,LI Q,et al.Metabolic engineering for the production of l-phenylalanine in Escherichia coli[J].3 Biotech,2019,9(3):85.
[3] 鄢芳清,韩亚昆,李娟,等.大肠杆菌芳香族氨基酸代谢工程研究进展[J].生物加工过程,2017,15(5):32-39;85.YAN F Q,HAN Y K,LI J,et al.Metabolic engineering of aromatic amino acids in Escherichia coli[J].Chinese Journal of Bioprocess Engineering,2017,15(5):32-39;85.
[4] KOMA D,KISHIDA T,YOSHIDA E,et al.Chromosome engineering to generate plasmid-free phenylalanine-and tyrosine-overproducing Escherichia coli strains that can be applied in the generation of aromatic-compound-producing bacteria[J].Applied and Environmental Microbiology,2020,86(14):e00525-20.
[5] WU J,LIU Y F,ZHAO S,et al.Application of dynamic regulation to increase L-phenylalanine production in Escherichia coli[J].Journal of Microbiology and Biotechnology,2019,29(6):923-932.
[6] LIU Y F,XU Y R,DING D Q,et al.Genetic engineering of Escherichia coli to improve L-phenylalanine production[J].BMC Biotechnology,2018,18(1):5.
[7] 门佳轩,熊博,郝亚男,等.代谢工程优化大肠杆菌高效合成L-苯丙氨酸[J].食品科学,2021,42(2):114-120.MEN J X,XIONG B,HAO Y N,et al.Metabolic engineering of Escherichia coli for efficient synthesis of L-phenylalanine[J].Food Science,2021,42(2):114-120.
[8] DING D Q,LI J L,BAI D Y,et al.Biosensor-based monitoring of the central metabolic pathway metabolites[J].Biosensors and Bioelectronics,2020,167:112456.
[9] 田鑫,戴健欣,田园,等.全局转录机器工程调控微生物代谢的应用进展[J].食品与发酵工业,2022,48(7):298-303.TIAN X,DAI J X,TIAN Y,et al.Progress in the microbial metabolism by global transcription machinery engineering regulation[J].Food and Fermentation Industries,2022,48(7):298-303.
[10] ALPER H,MOXLEY J,NEVOIGT E,et al.Engineering yeast transcription machinery for improved ethanol tolerance and production[J].Science,2006,314(5805):1565-1568.
[11] GAO X,JIANG L,ZHU L Y,et al.Tailoring of global transcription sigma D factor by random mutagenesis to improve Escherichia coli tolerance towards low-pHs[J].Journal of Biotechnology,2016,224:55-63.
[12] ZHANG F,QIAN X H,SI H M,et al.Significantly improved solvent tolerance of Escherichia coli by global transcription machinery engineering[J].Microbial Cell Factories,2015,14:175.
[13] ALPER H,MIYAOKU K,STEPHANOPOULOS G.Construction of lycopene-overproducing E.coli strains by combining systematic and combinatorial gene knockout targets[J].Nature Biotechnology,2005,23(5):612-616.
[14] JIN X,WANG S M,GAO Y P,et al.Combinatorial metabolic engineering of Escherichia coli to efficiently produce L-threonine from untreated cane molasses[J].Bioresource Technology,2025,419:132058.
[15] KLEIN-MARCUSCHAMER D,STEPHANOPOULOS G.Assessing the potential of mutational strategies to elicit new phenotypes in industrial strains[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(7):2319-2324.
[16] WANG K,WANG X,GE X Z,et al.Heterologous expression of aldehyde dehydrogenase from Saccharomyces cerevisiae in Klebsiella pneumoniae for 3-hydroxypropionic acid production from glycerol[J].Indian Journal of Microbiology,2012,52(3):478-483.
[17] XIA T,ZHAO G,JENSEN R A.Loss of allosteric control but retention of the bifunctional catalytic competence of a fusion protein formed by excision of 260 base pairs from the 3′ terminus of pheA from Erwinia herbicola[J].Applied and Environmental Microbiology,1992,58(9):2792-2798.
[18] KIKUCHI Y,TSUJIMOTO K,KURAHASHI O.Mutational analysis of the feedback sites of phenylalanine-sensitive 3-deoxy-D-arabino-heptulosonate-7-phosphate synthase of Escherichia coli[J].Applied and Environmental Microbiology,1997,63(2):761-762.
[19] DING D Q,LIU Y F,XU Y R,et al.Improving the production of L-phenylalanine by identifying key enzymes through multi-enzyme reaction system in vitro[J].Scientific Reports,2016,6:32208.
[20] LI Z D,GAO C,YE C,et al.Systems engineering of Escherichia coli for high-level shikimate production[J].Metabolic Engineering,2023,75:1-11.
[21] BORJA G M,MEZA MORA E,BARR N B,et al.Engineering Escherichia coli to increase plasmid DNA production in high cell-density cultivations in batch mode[J].Microbial Cell Factories,2012,11:132.
N B,et al.Engineering Escherichia coli to increase plasmid DNA production in high cell-density cultivations in batch mode[J].Microbial Cell Factories,2012,11:132.
[22] DOROSHENKO V,AIRICH L,VITUSHKINA M,et al.YddG from Escherichia coli promotes export of aromatic amino acids[J].FEMS Microbiology Letters,2007,275(2):312-318.
[23] BAI D Y,DING D Q,LI J L,et al.Pinpointing the l-phenylalanine binding sites of TyrR using biosensors and computer-aided simulation[J].Biotechnology Letters,2019,41(3):401-408.
[24] MAHR R,VON BOESELAGER R F,WIECHERT J,et al.Screening of an Escherichia coli promoter library for a phenylalanine biosensor[J].Applied Microbiology and Biotechnology,2016,100(15):6739-6753.
[25] FRENDORF P O,LAURITSEN I,SEKOWSKA A,et al.Mutations in the global transcription factor CRP/CAP:Insights from experimental evolution and deep sequencing[J].Computational and Structural Biotechnology Journal,2019,17:730-736.