植物乳植杆菌是一种广泛分布于自然界中的有益菌,其具有优异的益生菌特性和安全性,常用于食品和益生菌生产。它可以从不同的糖中获取能量,并对乳制品、蔬菜、葡萄酒以及人类和动物的胃肠道等多种生态位具有很高的适应性。作为研究最广泛的益生菌乳酸菌之一,植物乳植杆菌已获得欧洲食品安全局(European Food Safety Authority,EFSA) 和美国食品药品监督管理局(Food and Drug Administration,USFDA) 的安全认证,确认其为一般公认安全(Generally Recognized as Safe,GRAS) 微生物[1]。植物乳植杆菌具有抗氧化、调节血脂异常[2]、改善肠道炎症和屏障功能[3-4]、调节肠道内稳态[5]、免疫调节和抗菌等益生菌特性,具有良好的健康促进作用。近年来,植物乳植杆菌的抑菌抗菌功能性研究逐渐受到重视,其产生的多种抗菌化合物,如有机酸、过氧化氢和细菌素[6-7],能够有效对抗多种致病菌[8-9]。特别是细菌素这种由核糖体合成并释放到细胞外空间的生物活性肽或蛋白质,具有高效杀菌或抑菌能力,同时还兼具无毒无害、无抗药性、不易残留和稳定性强的特点,已被用作食品生物防腐剂,鉴于其优异的抗菌活性,使之得以成为抗生素和化学防腐剂的理想替代品[10]。
另一方面,作为宿主的益生菌[11],植物乳植杆菌需要具有一定的低pH值以及高胆盐耐受性[12]、胃肠定殖性[13]以及对肠道黏膜的黏附性[14]等优良特性,证明其在益生菌研究方面的潜力。此外,益生菌可以在胃肠道中存活和增殖,并具有很强的内部转运能力。尽管关于不良事件或安全问题的报道较少,但益生菌的潜在安全问题仍然引起了广泛重视[15]。因此开发新的益生菌菌株必须在使用前进行安全性评估。
本研究通过体外实验对鸭粪源中分离出的植物乳植杆菌WX-1的一些益生菌特性与安全性进行了评估:酸与胆盐耐受性、抑菌性、自聚集和共聚集的能力、抗生素耐药性以及溶血性,从而验证了植物乳植杆菌具有安全性和益生菌特性;此外,利用Illumina MiSeq平台对WX-1进行了全基因组测序,从遗传水平上分析了该菌株的益生菌特性和功能基因组成,采用antiSMASH与BAGEL 4数据库深入挖掘了菌株次级代谢产物和细菌素合成基因簇。本研究从表型特征与基因序列两方面共同对WX-1的益生特性、安全性以及产细菌素潜力进行了评估,为该菌株作为益生菌的开发和应用提供理论依据。
1 材料与方法
1.1 材料与试剂
植物乳植杆菌WX-1由本实验室从养鸭废液中所分离和筛选获得;金黄色葡萄球菌保存于本实验室。
MRS培养基(g/L):酵母浸粉5.0、牛肉浸粉10.0、蛋白胨10.0、葡萄糖20.0、无水乙酸钠5.0、柠檬酸二氨2.0、MgSO4·7H2O 0.5、KH2PO4 2.0、琼脂20.0,液体培养基不加琼脂。
牛肉膏蛋白胨培养基(g/L):牛肉膏3.0、NaCl 5.0、蛋白胨10.0、琼脂20.0,液体培养基不加琼脂。
Ezup柱式细菌基因组DNA抽提试剂盒SK8255,生工生物工程(上海)股份有限公司。
1.2 仪器与设备
BX4研究级正置生物显微镜,日本奥林巴斯光学仪器;101-0AB电热鼓风干燥箱,天津泰斯特仪器有限公司;JA1203电子分析天平,上海舜宇恒平科学仪器有限公司;YXQ-100G立式压力蒸汽灭菌器,上海博讯实业有限公司;ZQLY-180V立式全温振荡培养箱,上海知楚仪器有限公司;SW-CJ-2D型双人净化工作台,苏州净化设备有限公司。
1.3 试验方法
1.3.1 菌株分离纯化与鉴定
将养鸭废液稀释至10-3、10-4和10-5稀释度,涂布于MRS琼脂培养基,于37 ℃培养48 h,挑取单菌落,进行多次划线纯化,直至获得纯化的单菌落。记录菌落形态、大小、颜色以及边缘等特征,经革兰氏染色后在显微镜下观察菌体形态,挑选出具有典型乳杆菌菌体形态的菌株。
采用试剂盒提取其基因组DNA,随后将提取的DNA样本送至生工生物工程 (上海) 股份有限公司进行16S rDNA基因扩增和测序,将得到的菌株序列在NCBI数据库进行BLAST序列比对分析,运用MEGA 11软件构建系统发育树。
1.3.2 酸与胆盐耐受性
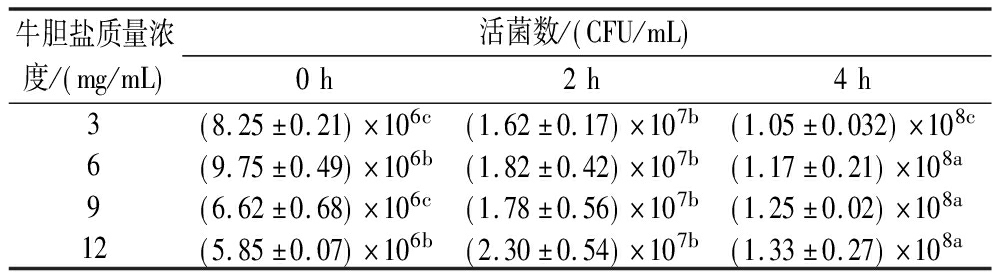
将乳酸菌在pH=3或不同胆汁盐浓度(3、6、9、12 mg/mL)的MRS培养基中分别稀释至1×108 CFU/mL,在37 ℃、180 r/min培养条件下振荡培养4 h以模拟肠道条件,在0、2和4 h取样稀释涂布,通过平板计数法来评估分离的细菌细胞对酸和胆盐的耐受性[15]。以上试验均重复3次。
1.3.3 抑菌能力测定
通过打孔法进行抑菌活性评价试验。WX-1在37 ℃培养12 h,离心收集上清液,将无细胞上清液通过0.22 μm无菌膜过滤以获得不含细胞的无细胞上清液。以金黄色葡萄球菌为指示菌(1×106 CFU/mL),均匀涂抹在固体培养基上,打孔后于孔洞中注入100 μL上清液,37 ℃培养24 h,测定抑菌圈的直径并按公式(1)计算抑菌率[16]。
抑菌率![]()
(1)
1.3.4 自聚集、共聚集能力
自聚集能力:离心以获得培养至稳定期的菌株。沉积物在等体积无菌PBS (pH 7.0)中重悬,并洗涤3次,最终调整至1×109 CFU/mL。将乳酸菌悬浮液涡旋10 s,于37 ℃静置培养8 h。在0、1、5、8 h利用酶标仪测定最上层悬浮液约0.1 mL 的OD600值,自聚集率计算如公式(2)所示[17]:
自聚集率![]()
(2)
式中:At,培养t h后的吸光度;A0,时间为0 h时的吸光度。
共聚集能力:将乳酸菌和金黄色葡萄球菌在各自最适温度下培养16 h,离心后去除上清液,将菌体在无菌PBS (pH 7.0) 中洗涤3次,最终调整OD600值分别为0.25和0.60。将等体积的乳酸菌和金黄色葡萄球菌细菌悬液涡旋10 s混合均匀。在37 ℃培养0、5 h后测量悬液最上层在OD600 nm处的吸光度(Amix),共聚集率计算如公式(3)所示[18]:
共聚集率![]()
(3)
式中:A1和A2分别是两株菌在培养0 h的吸光度;Amix,混合培养5 h后的吸光度。
1.3.5 溶血性试验
采用划线法,将活化后的植物乳植杆菌WX-1接种于哥伦比亚血琼脂平板上,观察目标菌的溶血情况。
1.3.6 抗生素敏感性
采用Kirby-Bauer标准纸片扩散法测定了WX-1的药敏性。将稀释后的WX-1(1×106 CFU/mL) 均匀地擦拭到MRS固体培养基上,用无菌的镊子将药敏纸片均匀放置其上,37 ℃下培养24 h,记录各药敏纸片的抑菌圈直径大小,重复3次试验求平均值。根据美国临床和实验室标准协会 (Clinical and Laboratory Standards Institute,CLSI) 国际标准中对抗菌药物敏感性的最小抑菌圈直径大小来判定菌株的药敏性[19]。
1.3.7 全基因组测序
将2%(体积分数)的WX-1接种至MRS液体培养基中,37 ℃培养24 h后离心取菌体送至上海美吉生物科技有限公司进行全基因组测序。结合二代Illumina HiSeq和三代PacBio Sequel测序平台完成全基因组测序。对测序数据采取质控手段,去除adapter序列,剪切去除5’端含有非A、G、C、T的碱基,修剪测序质量较低的reads末端 (测序质量值 1.3.8 基因组功能注释与分析 利用Glimmer (http://ccb.jhu.edu/software/glimmer/index.shtml),GeneMarkS,Prodigal软件对基因组中的编码序列 (CDS) 进行预测。使用Prokka (version 1.12) 进行蛋白质编码基因的基因预测和计算注释,利用Swiss-Prot、Pfam、NR、COG、KEGG、GO等功能数据库对基因组序列进行功能注释。采用BAGEL4 (http://bagel4.molgenrug.nl) 和antiSMASH软件 (http://antiSMASH.secondarymetabolites.org/),鉴定了次级代谢物生物合成基因簇。 基于16S rDNA测序,选取部分乳酸菌标准菌株的16S rDNA序列,使用MEGA 11构建系统发育树。如图1-a所示,WX-1与植物乳植杆菌(Lactiplantibacillus plantarum) 的同源性最高,进一步确定该菌株为植物乳植杆菌。将WX-1的16S rDNA序列上传至NCBI数据库中,获取登录号PRJNA1202526。植物乳植杆菌WX-1保藏至菌种保藏中心,保藏号为CCTCC NO:M 20242501。植物乳植杆菌WX-1在MRS固态培养基上的菌落较小 (直径约1~2 mm),乳白色,表面光滑微凸起,边缘完整,呈规则圆形 (图1-b);革兰氏染色呈紫色,为革兰氏阳性菌,其细胞为短杆状,单个、成对或短链状 (图1-c)。 a-基于16S rRNA序列建立的系统发育树;b-菌落形态;c-显微形态图 图1 WX-1的鉴定及形态学观察 Fig.1 Identification and morphological observation of WX-1 2.2.1 酸和胆盐的体外耐受性 能否在人类胃肠道恶劣条件下生存是将菌株视为益生菌的一个先决条件。当植物乳植杆菌WX-1在pH为3的环境下进行培养,其活菌数随着培养时间的增加呈现逐渐降低的趋势,在0、2、4 h的活菌数分别为(7.10±0.8)×106、(6.60±0.42)×106、(4.33±0.25)×106 CFU/mL,当培养时间为4 h时菌株存活率仍可达到(61.29±3.48)%。 在不同胆盐浓度下,WX-1的活菌数随着时间的延长呈上升趋势 (表1)。通过酸和胆盐耐受评价,发现菌株WX-1具有较好的酸、胆盐耐受能力。 表1 植物乳植杆菌于不同胆盐浓度下的存活 Table 1 Survival of L.plantarum in different bile salt concentrations 牛胆盐质量浓度/ mg/mL 活菌数/ CFU/mL 0h2h4h3 8.25±0.21 ×106c 1.62±0.17 ×107b 1.05±0.032 ×108c6 9.75±0.49 ×106b 1.82±0.42 ×107b 1.17±0.21 ×108a9 6.62±0.68 ×106c 1.78±0.56 ×107b 1.25±0.02 ×108a12 5.85±0.07 ×106b 2.30±0.54 ×107b 1.33±0.27 ×108a 注:不同小写字母表示差异显著(P<0.05)。 2.2.2 植物乳植杆菌WX-1抑菌能力 对菌株WX-1的无细胞上清液的抑菌能力进行测定。经12 h培养的植物乳植杆菌WX-1上清液对金黄色葡萄球菌有较强的抑制作用,其抑菌圈直径达到(24.93±0.56) mm,抑菌率为(72.3±0.62)%。显著优于刘佳等[20]筛选的Bacillus subtilis 64-3对金黄色葡萄球菌的抑菌效果(最大抑菌圈直径为19.48 mm)以及植物乳植杆菌FCa3L与8PA3对金黄色葡萄球菌的抑菌效果,其最大抑菌圈直径为(7.20±0.45)、(1.75±0.95) mm[21]。 2.2.3 自聚集、共聚集 益生菌需要具备在宿主肠道内有效定植并能阻止病原菌在肠道定殖的能力。乳酸菌的自聚集是体外评估其黏附肠道细胞能力的一个重要指标菌株。根据自聚集率,可将菌株的自聚集能力分为弱(16%~35%)、中等(35%~50%)、强(高于50%)[22],菌株WX-1的自凝能力随时间的延长而增加,在1、5、8 h分别达到(12.40±6.23)%、(27.47±5.16)%、(44.29±4.47)%。显著高于檀茜倩等[23]筛选出的L.plantarum PC715的自聚集能力(27.64%),说明菌株WX-1具有较好的黏附特性。 益生乳酸菌可以通过与病原菌共凝聚来降低胃肠道内病原体的群体水平,从而达到阻止病原体侵染宿主的效果[24]。根据试验结果,菌株WX-1的共凝聚能力为(11.13±4.23)%。 2.3.1 溶血试验 溶血试验结果显示,菌株WX-1未表现出溶血性,具有体外安全性,与赵丽娜[25]等研究结果一致。 2.3.2 抗生素敏感性 虽然已经确定大多数属于乳酸杆菌属的微生物对抗生素敏感,但某些菌株也表现出一定程度的耐药性。研究表明,不同植物乳植杆菌菌株对抗生素的耐药性可能存在差异。结果显示,WX-1对卡那霉素、氨苄西林、头孢噻肟、红霉素、四环素、氯霉素、克林霉素以及青霉素呈现较高敏感性,对氨基糖苷类抗生素:卡那霉素、庆大霉素以及链霉素有耐药性(表2)。由于卡那霉素、庆大霉素以及链霉素能够抑制蛋白质合成,但革兰氏阳性菌细胞壁厚,使得其不能进入细胞壁干扰细胞膜合成,因此具有一定耐药性[26]。 表2 菌株对常用抗生素的敏感性 Table 2 Susceptibility of strains to common antibiotics 抗生素类别抗生素药物质量/μg抑菌圈直径判断标准/mmRIS敏感性β-内酰胺类氨苄西林10≤1314~16≥17S头孢噻肟30≤2223~25≥26S大环内脂类红霉素15≤1314~22≥23S氨基糖苷类卡那霉素30≤1314~17≥18n.r.庆大霉素10≤1213~14≥15n.r.链霉素10≤1112~14≥15n.r.四环素类四环素30≤1112~14≥15S氯霉素类氯霉素30≤1213~17≥18S林可胺类克林霉素2≤1415~20≥20S青霉素类青霉素10≤1314~17≥18S 注:n.r.表示不敏感 将植物乳植杆菌WX-1全基因组序列上传至NCBI获得登录号:PRJNA1202526。使用Circos v.0.64生成菌株WX-1的基因组图谱(图2-a),菌株WX-1基因组长度为3 241 479 bp,GC含量为44.58%,基因组包括3 052个编码序列(coding sequences,CDSs)、16个16rRNA、75个tRNA、38个sRNA,包含一个完整的染色体序列和1个完整的质粒序列。其中染色体序列大小为3 176 250 bp,质粒大小为65 229 bp,GC含量为40.56%。将从数据库(NR、Swiss-Port、Pfam、COG、GO和KEGG)中注释到的基因进行汇总,分别注释到5 204、10 631、2 484、2 357、2 312以及1 476个。并以Lactiplantibacillus plantarum Strain AUSA002、Lactiplantibacillus plantarum strain SCZH-7、Lactiplantibacillus plantarum strain Lap、Lactiplantibacillus plantarum strain BD-LP、Lactiplantibacillus plantarum strain PM8为研究对象,通过BRIG软件进行分析(图2-b)。 a-WX-1染色体圈图;b-以WX-1为参考基因组的Brig分析 图2 植物乳植杆菌WX-1基因圈图 Fig.2 Gene circle map of L.plantarum WX-1 注:a图中圈图最外圈为基因组大小;第2、3圈为正链和负链上的CDS,不同的颜色表示CDS不同的COG的功能分类;第4圈为rRNA和tRNA;第5圈为GC含量,向外的红色部分表示该区域GC含量高于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大,向内的蓝色部分 表示该区域GC含量低于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大;最内圈为GC-Skew值。 为验证菌株之间的亲缘关系,选择5株模式菌进行了平均核苷酸一致性(average nucleotide identity,ANI)的计算。Lactiplantibacillus plantarum WX-1与Lactiplantibacillus plantarum Strain AUSA002、Lactiplantibacillus plantarum strain SCZH-7、Lactiplantibacillus plantarum strain Lap、Lactiplantibacillus plantarum strain BD-LP、Lactiplantibacillus plantarum strain PM8的ANI值分别为99.820 9%、99.820 2%、99.792 3%、99.780 8%以及99.651 5%,均高于99%。 2.5.1 KEGG注释 KEGG数据库将生物学途径分为6个主要类别,每个类别都与注释的基因相关。根据菌株序列与KEGG数据库结果的比较,如图3所示,WX-1被注释为具有1 854个基因,参与了39条KEGG通路。分析表明,WX-1在新陈代谢 (metabolism) 和环境信息处理 (environmental information processing) 两大类别中具有较高的基因注释数量。具体而言,在新陈代谢中共有1 269个基因被注释,占KEGG注释基因总数的68.44%,其中涉及碳水化合物代谢相关的基因最为丰富,氨基酸代谢和次级代谢产物分别包含166和34个基因;在环境信息处理中跨膜转运通路占较大比例,而在跨膜运输通路中基因主要集中在ABC转运蛋白的合成,共注释到93个基因占整个跨膜运输通路的64.13%;翻译通路主要集中在核糖体 (51) 与氨酰基-tRNA生物合成[27],其中占据较大比例的ABC转运蛋白的合成的基因与细菌素合成有较大关联,结合翻译通路占比组成表明了菌株WX-1在氨基酸代谢以及蛋白质合成方面具有较高的潜力。此外,在菌株WX-1中发现与免疫和炎症反应有关的NOD和RIG-I样受体通路,检测到了参与叶酸生物合成的途径,并在次级代谢产物的生物合成及萜类化合物和聚酮类化合物的代谢中分别注释到34和27个基因,其中萜类及聚酮类化合物具有抑菌、调节肠道微生态平衡以及免疫调节的作用是微生物重要的次级代谢产物,具有较好的研究潜力。 图3 KEGG通路分类图 Fig.3 KEGG path classification diagram 2.5.2 COG注释 COG功能注释如图4所示,共注释到2 357个蛋白质编码基因,分布在23个COG条目中。其中,碳水化合物运输和代谢 (carbohydrate transport and metabolism)、转录 (transcription)、氨基酸转运和代谢 (amino acid transport and metabolism)是最大的三大类,分别注释了266个、263个和219个基因,意味着菌株WX-1具有较好的利用碳水化合物和氨基酸的能力。此外,翻译、核糖体结构和生物发生相关基因为212个,在整个注释结果中也占据了较大比例,可以认为菌株WX-1在蛋白质合成方面具有一定的潜力。在菌株WX-1中注释到70个与防御相关的基因,表明其可能具有一定的抵抗外界环境的能力。 图4 COG分类统计柱状图 Fig.4 Statistical histogram of COG classification 2.5.3 GO功能注释 GO数据库共注释了2 312个基因,将其分为生物工程 (biological process)、细胞组分 (cellular component)、分子功能 (molecular function) 3个部分,每部分中均有14类功能得到注释(图5)。通过注释结果表明,该菌株具有合成多种转运蛋白的功能,包括与细菌素合成密切相关的跨膜转运蛋白和ABC转运蛋白等。其中基因组分析显示,注释到与核黄素生物合成相关的基因有9个、叶酸生物合成、相关基因有3个、细菌素免疫相关基因6个、硫胺素生物合成相关基因8个、细菌黏附及6个以及萜类生物合成2个。 图5 GO注释分类统计图 Fig.5 GO annotation classification statistical chart 2.5.4 益生基因注释 通过全基因组测序,在植物乳植杆菌WX-1中发现了一系列可能与植物乳植杆菌常见的益生特性相关的基因,这些基因的存在有助于预测植物乳植杆菌的益生机制。在基因组序列中挖掘到了B族维生素生物合成相关的基因:硫胺素 (thiI、thiD、thiE、hiM)、核黄素 (ribF、ribD、ribE、ribBA、ribH、ycsE、ybjI) 和叶酸 (folC、folP、folB、folK、folE、folA、moaA、moaC、moaB、moaE、ribBA、queD、tilS),使用产维生素的益生菌是一种较好的强化发酵食品或提高益生菌促进健康作用的途径;注释到功能基因clpB、plsC、relA、pyK、guaA、uvrA、tpiA、recA、gadB、Pgi、pgK、relA、atpA等与菌株的耐酸能力有关;sfcA、mtlR、agrA等基因被证明与细菌胆盐耐药性有关[28]。clpL基因已被发现与酸和胆盐抗性相关[29]。此外,已知Fbp基因与细菌黏附能力有关,基因组分析揭示了可能在调节宿主免疫反应中发挥作用的特定基因 (dltB、dltD) 的存在[30]。 利用antiSMASH对全基因数据进行分析,在菌株WX-1中共检测到4个次级代谢产物合成基因簇 (表3)。包括萜烯类 (terpene)、Ⅲ型聚酮类 (T3PKS)、核糖体合成和翻译后修饰肽产物 (RiPP-like)、和环内酯自诱导剂 (cyclic-lactone-autoinducer) (图6)。 表3 植物乳植杆菌次级代谢产物基因簇 Table 3 Gene cluster of secondary metabolites of L.plantarum 区域种类起始/bp终止/bp相似基因簇相似菌株Region1.1核糖体合成和翻译后修饰肽产物361968374118--Region1.2Ⅲ型聚酮类18084301849599--Region1.3萜烯类28529232873804--Region1.4环内酯自诱导剂31108193131530-- 图6 antiSMASH预测细菌素的产生途径 Fig.6 antiSMASH predicts the production pathway of bacteriocin 根据先前的研究表明,T3PKS、萜烯和环内酯自诱导家族与多酮、萜烯和环内酯自身诱导剂的产生有关,这些诱导剂在抗菌活性、抗癌作用、免疫调节和其他生理过程中起着关键作用。 通过BAGEL 4检索WX-1的全基因序列,确定了参与细菌素调控的代谢途径。发现在WX-1菌株中存在一个潜在热点区域 (图7),由63个开放阅读框组成(open reading frame,ORF) 共包含25个细菌素相关基因,含有6种核心肽,分别为Plantaricin K、Plantaricin J、Plantaricin A、Plantaricin N、Plantaricin E、Plantaricin F。其中,PlnJK和PlnEF为ClassⅡb类乳酸菌细菌素,分别由2条寡肽PlnJ、PlnK和PlnE、PlnF组成,是分子质量小且热稳定的一类细菌素,是天然的抗菌物质,对大多数病原菌以及腐败微生物都有抑制作用;PlnA与PlnN为两种细菌素效应物,其中PlnA被认为是一种信号分子,PlnN为非杀菌型细菌素。类似的,在植物乳植杆菌EL2基因组中也存在双肽细菌素PlnEF、PlnJK以及PlnA、PlnN的编码基因[21]。 图7 植物乳植杆菌WX-1基因组BAGEL 4对比结果 Fig.7 Comparative results of genome BAGEL 4 of L.plantarum WX-1 表4列出了可能产生的细菌素基因簇其各个基因的相关信息。orf18、orf20、orf31、orf33、orf36、orf38、orf42、orf56、orf58、orf60等ORFs分别与PlnR、PlnL、PlnP、PlnQ、PlnB、PlnD、PlnI、PlnW、PlnX、PlnY具有同源性,均属于Pln操纵子,参与细菌素的产生。其中,PlnA、PlnB、PlnC与PlnD通过识别信号分子来调节细菌素的产生;存在于细胞膜上的PlnL,PlnR,PlnM和PlnI参与细菌素免疫,赋予产生细菌素的菌株其自身对细菌素的抗性;PlnH、PlnU、PlnV与PlnW通过特异性裂解参与前体肽的鉴定和成熟,与细菌素分泌密切相关,但其确切功能尚不清楚。此外,在WX-1基因组中发现了负责产生AI-2信号分子的LuxS基因,该基因与菌株的双组分系统在调节细菌素的产生中共同起着至关重要的作用;LanT基因编码细菌素ABC-转运蛋白,ATP结合和渗透酶蛋白PlnG,是细菌素合成的核心蛋白[31],通常含有该蛋白的细菌素具有抗菌活性[32],HlyD基因编码的蛋白与PlnH同源,为细菌素ABC转运蛋白辅助因子,细菌素的所有类似物修饰和分泌均依赖于分别编码ABC转运蛋白和复制蛋白的2个基因plnG和plnH,以形成ABC转运系统,从而修饰、成熟和分泌细菌素先导肽。orf31所编码的免疫蛋白PlnP与2家族糖基转移酶和Ⅱ型CAAX氨基肽酶具有显著的同源性。orf58和orf60所编码的PlnX和PlnY是与毒素-康毒素系统具有高度同源的一对蛋白质,通常位于质粒上,但在基因簇中的功能并不明确。 表4 植物乳植杆菌WX-1基因簇注释信息 Table 4 Annotation information of L.plantarum WX-1 gene cluster 蛋白质ID序列ID氨基酸长度功能orf1---orf4WP_015379760.1205半乳糖苷O-乙酰转移酶orf5WP_021356659.1384MFS转运蛋白orf7WP_047672651.1263Cof型HAD-ⅡB家族水解酶orf9WP_024521247.1290AraC家族转录调节因子orf10WP_003641966.1271Cof型HAD-ⅡB家族水解酶orf12EFK30741.1469支链氨基酸转运系统Ⅱ载体蛋白orf15ABD15213.1398逆向转运蛋白orf17---orf18AAS21880.150PlnRorf20ADE08243.1222PlnL173.2 Plantaricin_KWP_003641972.157二肽细菌素植物素JK亚基PlnK172.2 Plantaricin_JWP_003641973.155二肽细菌素植物素JK亚基PlnJorf25---orf26ADN97563.166细菌素免疫蛋白174.2 Plantaricin_NMCG0681161.155细菌素前体肽GlySMDY2S78071.1220糖苷转移酶家族2蛋白orf31ADN97566.1248免疫蛋白PlnPorf33ACO06044.162PlnQ167.2 Plantaricin_AACO06045.164PlnAorf36ADE08252.1442PlnBorf37ADE08253.1247反应调节剂orf38ADE08254.1247PlnDorf42AAS27889.1247膜结合蛋白酶CAAX家族免疫蛋白PlnI171.2 Plantaricin_FAFJ79568.152双肽植物杆菌素EF亚基PlnF170.2 plantaricin_EAGE46854.156双肽植物杆菌素EF亚基PlnELanTACT61243.1716细菌素ABC-转运蛋白 ATP结合和渗透酶蛋白PlnG 续表4 蛋白质ID序列ID氨基酸长度功能HlyDACT61244.1458细菌素ABC转运蛋白 辅助因子PlnHorf52WP_102170862.1229CPBP家族膜内谷氨酸内肽酶orf53WP_021356668.1222CPBP家族膜内谷氨酸内肽酶orf55WP_076636381.1220CPBP家族膜内谷氨酸内肽酶orf56MCG0791852.1231整合膜蛋白PlnWorf58MCG0732421.196PlnXorf60MCG0747765.197推定的车前素生物合成蛋白 PlnYorf63WP_053566442.1769RNA聚合酶回收马达HelD 注:-表示功能未知。 产生细菌素的植物乳植杆菌,其细菌素生产通常是由群体感应(quorum sensing,QS)控制系统[33]调控的。QS通常包括一个由信号分子 (自动诱导剂、AI)、组氨酸激酶(histidine protein kinases,HPK)和反应调节因子 (response regulator,RR)[34] 组成的双组分调控系统 (2 CRS)。这3个组成部分 (AI、HK、RR) 构成了“信号发射、信号接收、响应刺激”的基本信号转导过程。PlnA基因编码II型细菌素肽 (AIP)[35],而LuxS基因编码AI-2作为该系统的信号分子[36]。AI-2经细菌素分泌相关的ABC转运蛋白转运到细胞外,积累到一定浓度后,与具有信号转导能力的膜蛋白PlnB (HK)结合,激活其信号转导活性。随后,位于具有DNA结合结构域和信号接收器结构域的细胞质中的PlnCD (RR)被磷酸化[37]。磷酸化的RR诱导细菌素产生基因集群的表达,刺激启动子的转录,包括编码细菌素前体肽(PlnJK、PlnEF、PlnN),免疫蛋白 (PlnI、RL)和运输蛋白 (PlnGH) 的序列,导致相关蛋白的产生[21]。综上所述,植物乳植杆菌WX-1有相对较完整的调节基因,满足细菌转录调节的基因条件。 植物乳植杆菌是具有EFSA的安全资格认证 (qualified presumption of safety,QPS) 菌株,且为USFDA的GRAS菌株,常被用于食品和益生菌生产[38]。作为一种应用广泛的益生菌[11],植物乳植杆菌具有维持胃肠道菌群平衡[39]、促进消化吸收[40]、增强免疫功能[8]、降低胆固醇[41]等多方面生理功能。随着不同来源的植物乳植杆菌菌株的分离,新型菌株的安全性在一定程度上也会受到关注[15]。基于全基因组测序对植物乳植杆菌的安全性与益生特性进行分析,仅仅停留在理论层面,并且,由于环境条件与基因表达密切相关,联合体外试验与基因层面的分析共同对菌株进行安全评价,对于植物乳植杆菌菌株的安全应用具有重大意义。 本研究通过体外试验对植物乳植杆菌WX-1的益生特性及安全性进行了评估。结果表明菌株WX-1是一种安全的新型菌株,具有较强的环境耐受性 (酸和胆盐)、抗菌活性、自聚集以及共聚集的能力,并且不存在溶血活性。此外对菌株WX-1进行了全基因组测序,经过多个数据库分析比对,确定该菌基因全长为3 241 479 bp,GC含量为44.58%,编码3 052个基因,在基因组数据中注释到与植物乳植杆菌益生特性相关的基因;并且,预测到植物乳植杆菌WX-1存在检测到4个次级代谢产物合成基因簇,以及一条合成Ⅱb类细菌素Plantaricin KJ、Plantaricin N、PlantaricinA和Plantaricin EF基因簇,满足细菌素合成的先决条件。本研究不仅可细菌素的开发与应用提供生物信息基础,也可为植物乳植杆菌WX-1在食品和医药等领域的应用提供理论基础。 [1] SONG X,XIONG Z Q,KONG L H,et al.Relationship between putative eps genes and production of exopolysaccharide in Lactobacillus casei LC2W[J].Frontiers in Microbiology,2018,9:1882. [2] WANG Y,XING X Y,MA Y X,et al.Prevention of high-fat-diet-induced dyslipidemia by Lactobacillus plantarum LP104 through mediating bile acid enterohepatic axis circulation and intestinal flora[J].Journal of Agricultural and Food Chemistry,2023,71(19):7334-7347. [3] LIU Z J,ZHAO J L,SUN R B,et al.Lactobacillus plantarum 23-1 improves intestinal inflammation and barrier function through the TLR4/NF-κB signaling pathway in obese mice[J].Food &Function,2022,13(11):5971-5986. [4] YUE Y,HE Z J,ZHOU Y H,et al.Lactobacillus plantarum relieves diarrhea caused by enterotoxin-producing Escherichia coli through inflammation modulation and gut microbiota regulation[J].Food &Function,2020,11(12):10362-10374. [5] ZHOU X T,ZHANG D D,QI W C,et al.Exopolysaccharides from Lactobacillus plantarum NCU116 facilitate intestinal homeostasis by modulating intestinal epithelial regeneration and microbiota[J].Journal of Agricultural and Food Chemistry,2021,69(28):7863-7873. [6] GUIMAR [7] AHIRE J J,JAKKAMSETTY C,KASHIKAR M S,et al.In vitro evaluation of probiotic properties of Lactobacillus plantarum UBLP40 isolated from traditional indigenous fermented food[J].Probiotics and Antimicrobial Proteins,2021,13(5):1413-1424. [8] CORTÉS-ZAVALETA O,L [9] SILVA D R,DE C [10] 陈志娜,尹琳琳,刘锦,等.1株抑MRSA植物乳植杆菌CHEN1的全基因组测序及产细菌素基因分析[J].食品科学,2024,45(22):43-50.CHEN Z N,YIN L L,LIU J,et al.Whole genome sequencing and bacteriocin gene analysis of Lactiplantibacillus plantarum CHEN1 which inhibits methicillin-resistant Staphylococcus aureus[J].Food Science,2024,45(22):43-50. [11] HAO Y,LI J L,WANG J C,et al.Mechanisms of health improvement by Lactiplantibacillus plantarum based on animal and human trials:A review[J].Fermentation,2024,10(2):73. [12] NATH S,SIKIDAR J,ROY M,et al.In vitro screening of probiotic properties of Lactobacillus plantarum isolated from fermented milk product[J].Food Quality and Safety,2020,4(4):213-223. [13] LIU D M,HUANG Y Y,LIANG M H.Analysis of the probiotic characteristics and adaptability of Lactiplantibacillus plantarum DMDL 9010 to gastrointestinal environment by complete genome sequencing and corresponding phenotypes[J].LWT,2022,158:113129. [14] LIU Y,ZHENG S J,CUI J L,et al.Lactiplantibacillus plantarum Y15 alleviate type 2 diabetes in mice via modulating gut microbiota and regulating NF-κB and insulin signaling pathway[J].Brazilian Journal of Microbiology,2022,53(2):935-945. [15] DENG L Q,LIU L M,FU T Y,et al.Genome sequence and evaluation of safety and probiotic potential of Lactiplantibacillus plantarum LPJZ-658[J].Microorganisms,2023,11(6):1620. [16] ZHENG X,LIANG Q,ZHAO B T,et al.Whole genome sequencing and analysis of probiotic characteristics for Lactiplantibacillus plantarum EL2 isolated from yak yogurt[J].LWT,2024,198:116039. [17] KIM H,KIM J S,KIM Y,et al.Antioxidant and probiotic properties of lactobacilli and bifidobacteria of human origins[J].Biotechnology and Bioprocess Engineering,2020,25(3):421-430. [18] LIN X N,XIA Y J,YANG Y J,et al.Probiotic characteristics of Lactobacillus plantarum AR113 and its molecular mechanism of antioxidant[J].LWT,2020,126:109278. [19] 李禤,贾丹,刘军龙,等.新分离植物乳杆菌的药敏性和抑菌性试验[J].中国兽医科学,2019,49(7):879-886.LI X,JIA D,LIU J L,et al.Antibiotic susceptibility and antimicrobial test of newly isolated Lactobacillus plantarum in vitro[J].Chinese Veterinary Science,2019,49(7):879-886. [20] 刘佳,谢秀丽,牛天贵.一株抑耐甲氧西林金黄色葡萄球菌(MRSA)芽孢杆菌的筛选与鉴定[J].中国农业大学学报,2007,12(6):20-23.LIU J,XIE X L,NIU T G.Screening and identification for Bacillus of having inhibitory effect against MRSA[J].Journal of China Agricultural University,2007,12(6):20-23. [21] 潘果,王云飞,钟忻桐,等.抗鼠伤寒沙门氏菌的乳酸菌细菌素生物学特性及其抑菌机制初步研究[J].饲料研究,2024,47(17):115-120.PAN G,WANG Y F,ZHONG X T,et al.Preliminary study on biological characteristics and inhibitory mechanism of Lactobacillus bacteriocins against Salmonella Typhimurium[J].Feed Research,2024,47(17):115-120. [22] ANGMO K,KUMARI A,BHALLA T C.Probiotic characterization of lactic acid bacteria isolated from fermented foods and beverage of Ladakh[J].LWT-Food Science and Technology,2016,66:428-435. [23] 檀茜倩,麻冰玉,程笑笑,等.一株泡菜中分离高产胞外多糖植物乳杆菌的益生特性分析[J/OL].中国食品学报,2024:1-11.(2024-11-13).https://kns.cnki.net/kcms/detail/11.4528.ts.20241111.1737.009.html.TAN X Q,MA B Y,CHENG X X,et al.Probiotic properties of Lactobacillus plantarum PC715 with high exopolysaccharides(EPS) production isolated from pickled vegetables[J/OL].Journal of Chinese Institute of Food Science and Technology,2024:1-11.(2024-11-13).https://kns.cnki.net/kcms/detail/11.4528.ts.20241111.1737.009.html. [24] HOJJATI M,BEHABAHANI B A,FALAH F.Aggregation,adherence,anti-adhesion and antagonistic activity properties relating to surface charge of probiotic Lactobacillus brevis gp104 against Staphylococcus aureus[J].Microbial Pathogenesis,2020,147:104420. [25] 赵丽娜,李钰炜,张娜,等.新疆赛里木酸奶源降胆固醇植物乳杆菌的筛选鉴定及其安全性研究[J].食品与发酵工业,2023,49(1):79-85.ZHAO L N,LI Y W,ZHANG N,et al.Screening,identification and safety of cholesterol-lowering Lactobacillus plantarum from Sayram Ketteki in Xinjiang[J].Food and Fermentation Industries,2023,49(1):79-85. [26] WON S M,CHEN S Y,PARK K W,et al.Isolation of lactic acid bacteria from kimchi and screening of Lactobacillus sakei ADM14 with anti-adipogenic effect and potential probiotic properties[J].LWT,2020,126:109296. [27] BUSTOS A Y,FONT DE VALDEZ G,FADDA S,et al.New insights into bacterial bile resistance mechanisms:The role of bile salt hydrolase and its impact on human health[J].Food Research International,2018,112:250-262. [28] CHEN C F,YU L L,TIAN F W,et al.Identification of novel bile salt-tolerant genes in Lactobacillus using comparative genomics and its application in the rapid screening of tolerant strains[J].Microorganisms,2022,10(12):2371. [29] TURPIN W,HUMBLOT C,NOORDINE M L,et al.Lactobacillaceae and cell adhesion:Genomic and functional screening[J].PLoS One,2012,7(5):e38034. [30] BHATTACHARYA A,JOISHY T K,KHAN M R.Exploring the probiotic potential,antioxidant capacity,and healthy aging based on whole genome analysis of Lactiplantibacillus plantarum LPJBC5 isolated from fermented milk product[J].BioRxiv,2024:2024.03.14.584937. [31] TIETZ J I,SCHWALEN C J,PATEL P S,et al.A new genome-mining tool redefines the lasso peptide biosynthetic landscape[J].Nature Chemical Biology,2017,13(5):470-478. [32] RIZZELLO C G,FILANNINO P,DI CAGNO R,et al.Quorum-sensing regulation of constitutive plantaricin by Lactobacillus plantarum strains under a model system for vegetables and fruits[J].Applied and Environmental Microbiology,2014,80(2):777-787. [33] PAI A,TANOUCHI Y,COLLINS C H,et al.Engineering multicellular systems by cell-cell communication[J].Current Opinion in Biotechnology,2009,20(4):461-470. [34] KAREB O,AÏDER M.Quorum sensing circuits in the communicating mechanisms of bacteria and its implication in the biosynthesis of bacteriocins by lactic acid bacteria:A review[J].Probiotics and Antimicrobial Proteins,2020,12(1):5-17. [35] MALDONADO-BARRAG [36] CAO Q,MA K,NIE M,et al.Role of luxS in immune evasion and pathogenicity of piscine Streptococcus agalactiae is not dependent on autoinducer-2[J].Fish &Shellfish Immunology,2020,99:274-283. [37] CHO G S,HUCH M,HANAK A,et al.Genetic analysis of the plantaricin EFI locus of Lactobacillus plantarum PCS20 reveals an unusual plantaricin E gene sequence as a result of mutation[J].International Journal of Food Microbiology,2010,141:S117-S124. [38] WIDYASTUTI Y,ROHMATUSSOLIHAT,FEBRISIANTOSA A.The role of lactic acid bacteria in milk fermentation[J].Food and Nutrition Sciences,2014,5(4):435-442. [39] LIU Y,WU J P,XIAO Y,et al.Relief of cadmium-induced intestinal motility disorder in mice by Lactobacillus plantarum CCFM8610[J].Frontiers in Immunology,2020,11:619574. [40] JUNG K,KIM A,LEE J H,et al.Effect of oral intake of Lactiplantibacillus plantarum APsulloc 331261 (GTB1TM) on diarrhea-predominant irritable bowel syndrome:A randomized,double-blind,placebo-controlled study[J].Nutrients,2022,14(10):2015. [41] CHONG H X,YUSOFF N A A,HOR Y Y,et al.Lactobacillus plantarum DR7 improved upper respiratory tract infections via enhancing immune and inflammatory parameters:A randomized,double-blind,placebo-controlled study[J].Journal of Dairy Science,2019,102(6):4783-4797.2 结果与分析
2.1 WX-1鉴定和形态学

2.2 益生特性分析
2.3 安全性评价

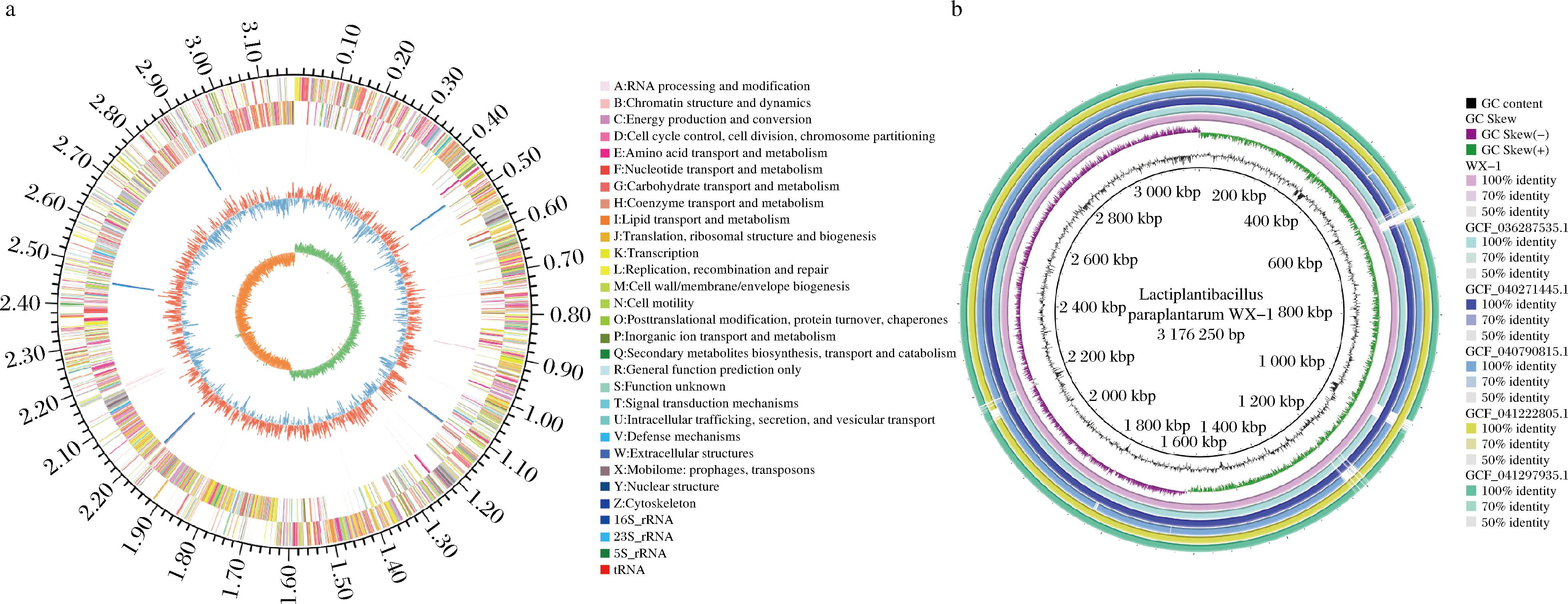
2.4 植物乳植杆菌WX-1全基因组测序与分析
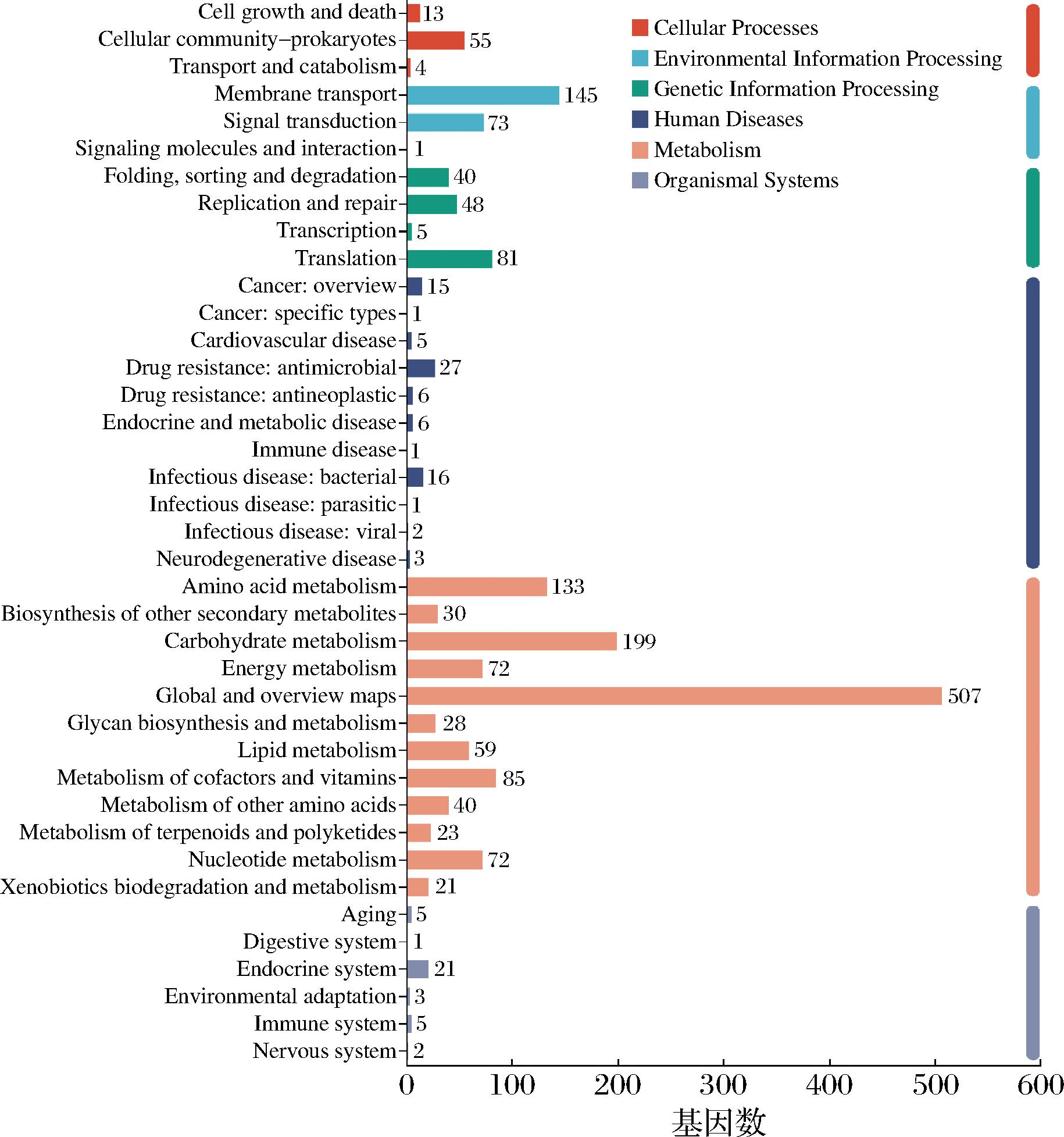
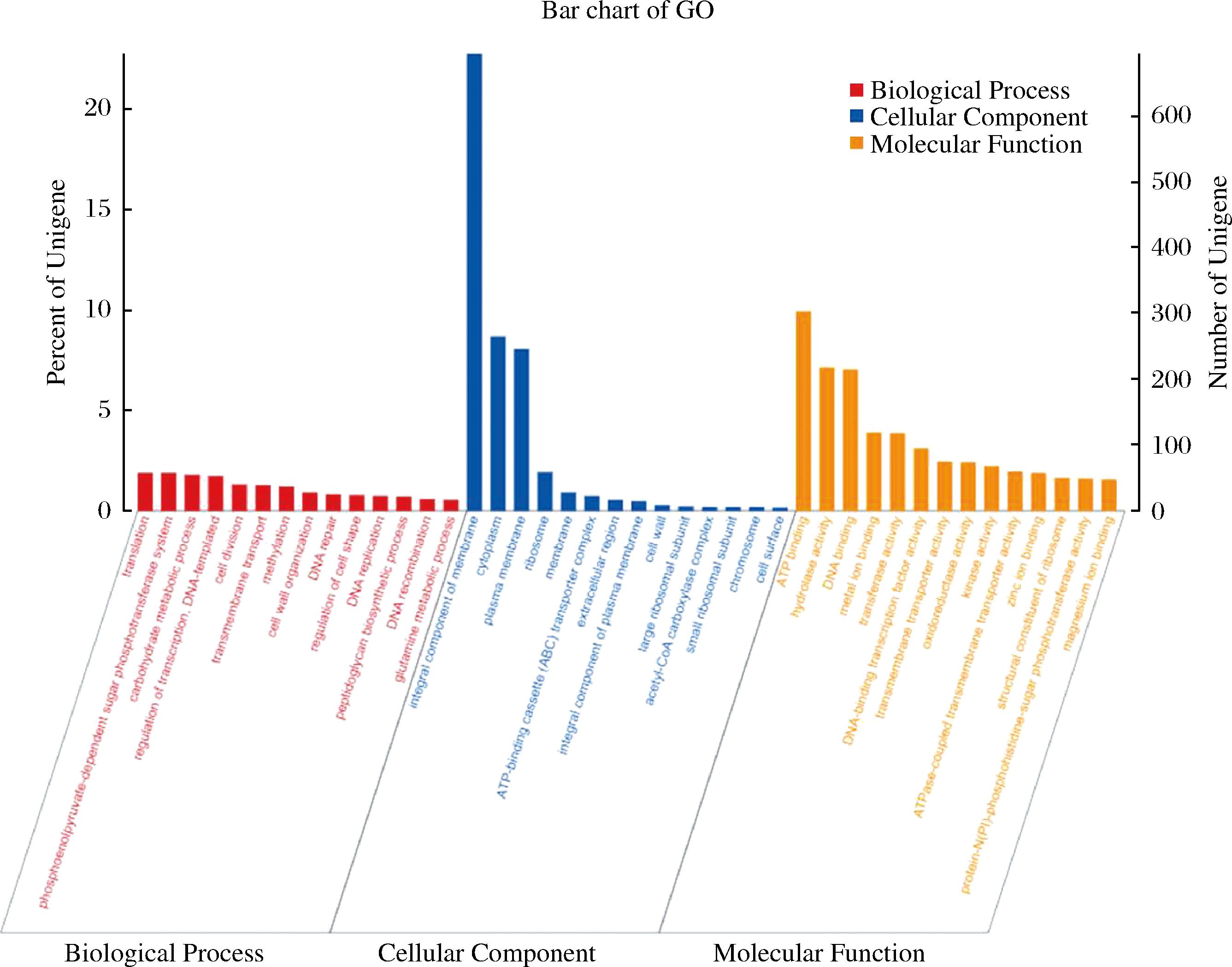
2.5 植物乳植杆菌WX-1基因注释

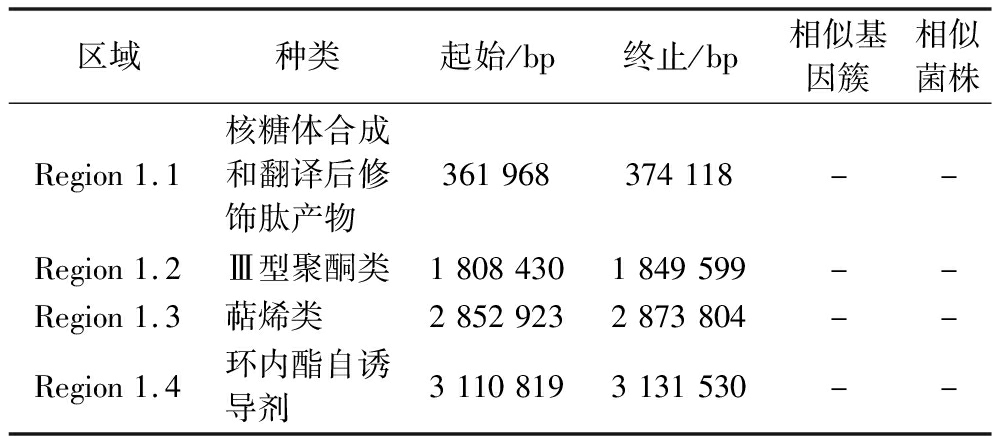
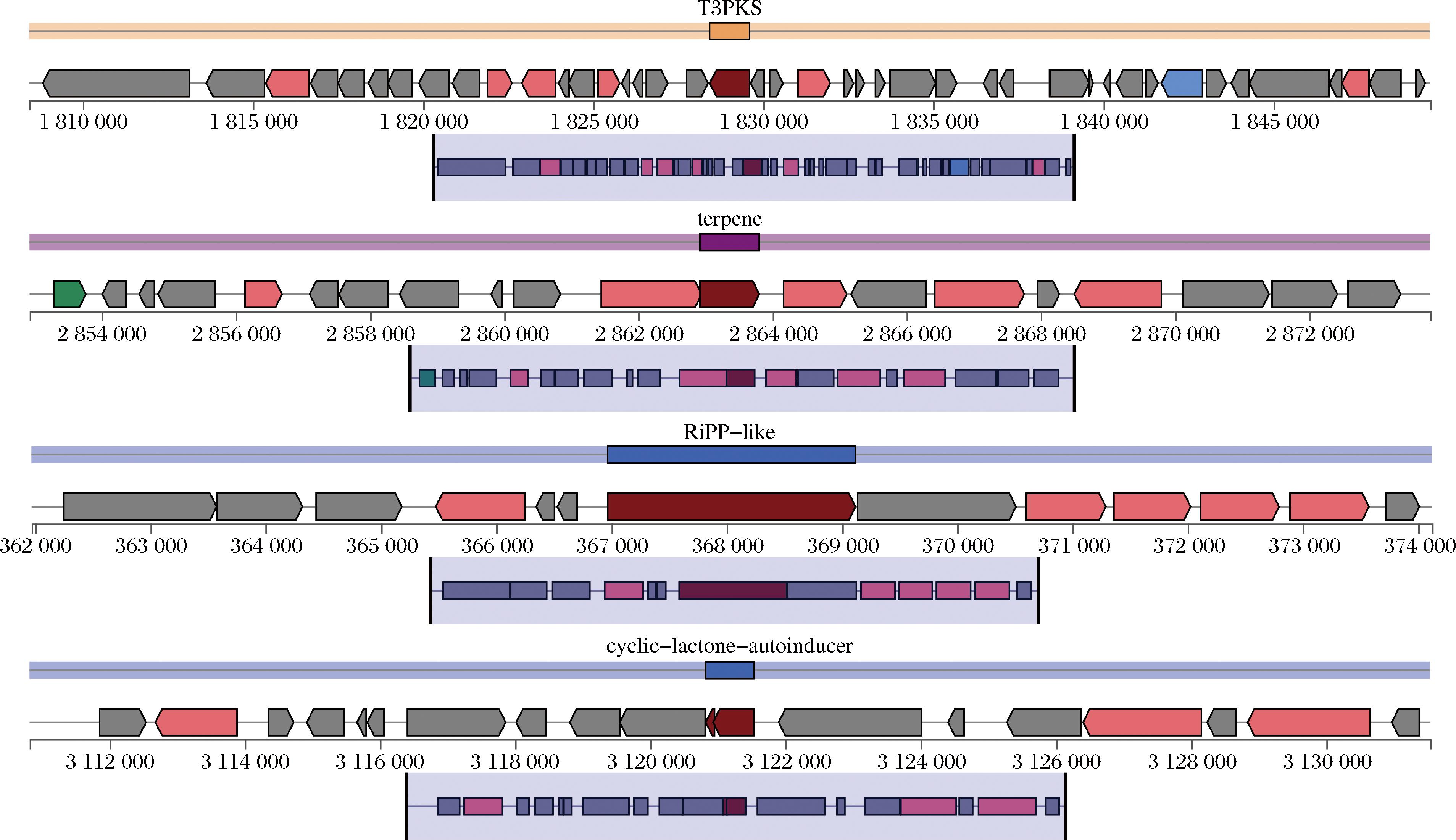
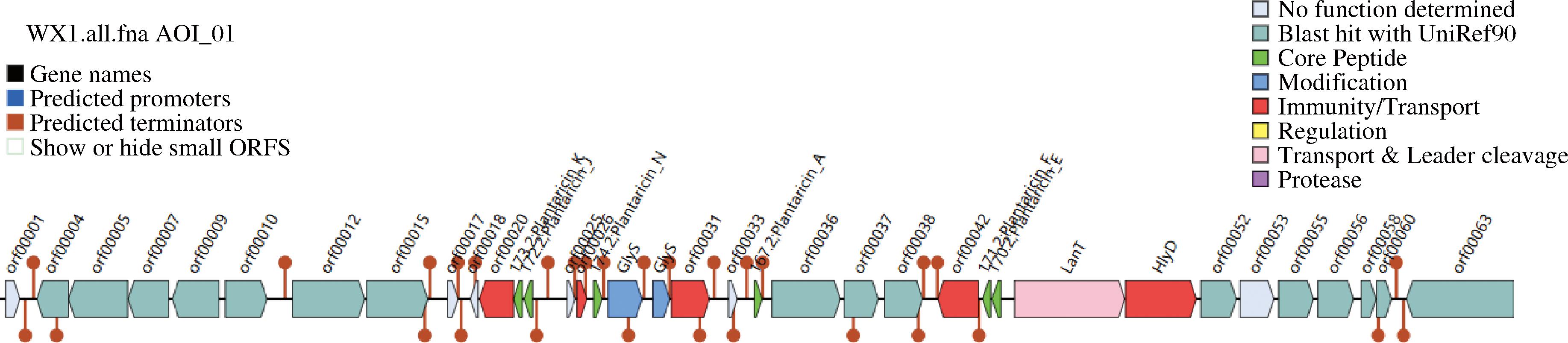
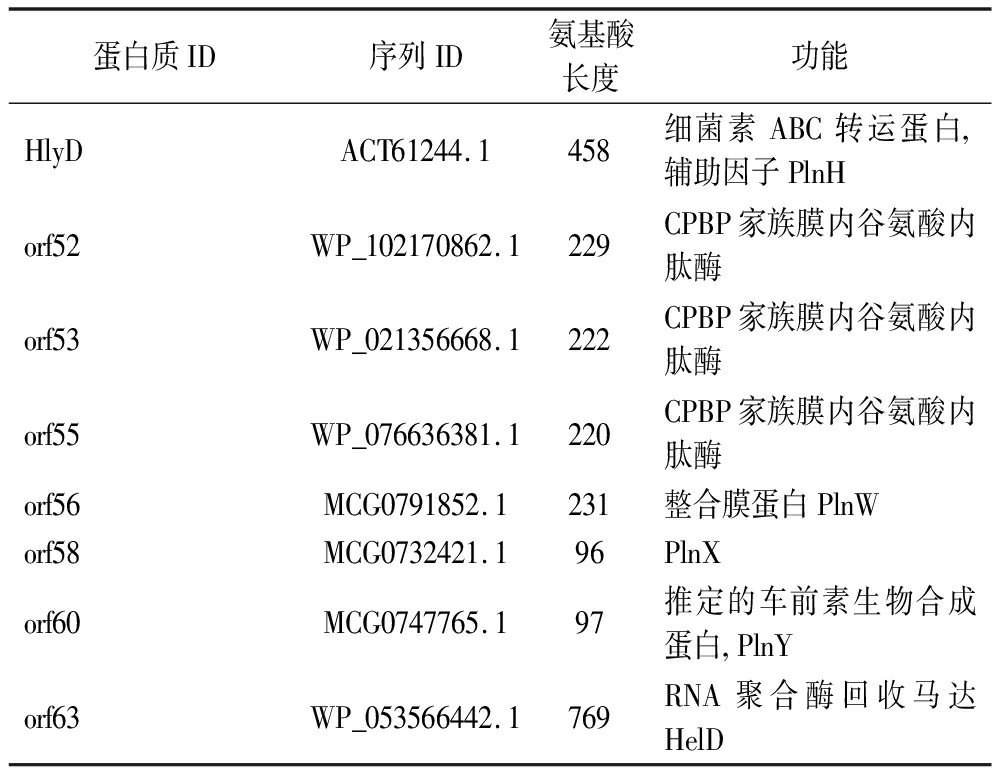
2.6 植物乳植杆菌WX-1次级代谢基因簇预测

3 结论与讨论
 ES A,SANTIAGO A,TEIXEIRA J A,et al.Anti-aflatoxigenic effect of organic acids produced by Lactobacillus plantarum[J].International Journal of Food Microbiology,2018,264:31-38.
ES A,SANTIAGO A,TEIXEIRA J A,et al.Anti-aflatoxigenic effect of organic acids produced by Lactobacillus plantarum[J].International Journal of Food Microbiology,2018,264:31-38. PEZ-MALO A,HERN
PEZ-MALO A,HERN NDEZ-MENDOZA A,et al.Antifungal activity of lactobacilli and its relationship with 3-phenyllactic acid production[J].International Journal of Food Microbiology,2014,173:30-35.
NDEZ-MENDOZA A,et al.Antifungal activity of lactobacilli and its relationship with 3-phenyllactic acid production[J].International Journal of Food Microbiology,2014,173:30-35. SSIA ORLANDI SARDI J,DE SOUZA PITANGUI N,et al.Probiotics as an alternative antimicrobial therapy:Current reality and future directions[J].Journal of Functional Foods,2020,73:104080.
SSIA ORLANDI SARDI J,DE SOUZA PITANGUI N,et al.Probiotics as an alternative antimicrobial therapy:Current reality and future directions[J].Journal of Functional Foods,2020,73:104080. N A,CABALLERO-GUERRERO B,LUCENA-PADR
N A,CABALLERO-GUERRERO B,LUCENA-PADR S H,et al.Induction of bacteriocin production by coculture is widespread among plantaricin-producing Lactobacillus plantarum strains with different regulatory operons[J].Food Microbiology,2013,33(1):40-47.
S H,et al.Induction of bacteriocin production by coculture is widespread among plantaricin-producing Lactobacillus plantarum strains with different regulatory operons[J].Food Microbiology,2013,33(1):40-47.