骆驼是我国荒漠和半荒漠地区的特有畜种,因其独特的役用性能,素有“沙漠之舟”的美誉。驼乳以其独特的营养价值,成为人们追求健康生活的理想选择[1]。过去二十年间,其全球产量从2000年的180万t增至2020年的315万t[2]。驼乳中的蛋白质及其比例与人乳中的蛋白质相似,可作为对牛乳过敏儿童的蛋白质来源之一[3-5]。据报道,驼乳中脂肪含量为(27.2±6.4) g/L,乳糖含量为(43.7±6.1)g/L,蛋白质含量为(25.5±2.7)g/L,灰分含量为(8.7±0.7)g/L,总固形物为(104.2±10.4)g/L[6]。此外,驼乳还含有丰富的烟酸和维生素C,是牛乳的3~5倍[7]。同时,驼乳中富含丰富溶菌酶、乳铁蛋白、乳过氧化物酶和肽聚糖识别蛋白(peptidoglycan recognition proteins,PGRP)等生物活性蛋白[8]。现有的诸多研究证实驼乳对糖尿病[9]、丙型肝炎[10]、肠道菌群[11]、高血压[12]、肺结核[13]等具有辅助治疗作用。
当生乳中菌落总数一旦超标,不仅对消费者的健康构成潜在威胁,还直接影响食品的品质和营养价值[14]。为了防止生乳微生物超标,人员、挤奶环节、乳样的处理、容器等的卫生和清洁极为重要。现有研究证实,驼乳中微生物多样性丰富,包括8 513个操作分类单元,隶属于32门、377属和652种。主要门包括变形菌门、厚壁菌门、热球菌门、拟杆菌门和放线菌门[15]。但是对于不同环境因素对生驼乳微生物组成的影响尚不明确。因此,本研究旨在通过分析不同牧场生乳、挤奶瓶、乳头夹、储奶罐、乳头、饮用水、饲料、粪便和驼舍空气样品之间的微生物多样性,确定造成生乳样品微生物污染关键风险关键点,为有效控制微生物超标,提高乳及乳制品的质量与安全提供依据。
1 材料与方法
1.1 材料与试剂
原料驼乳样品于7月采自内蒙古阿拉善巴丹吉林镇当地牧场样,连续3 d采集3个牧场的驼乳及其他环境样品。
E.Z.N.A.® Soil DNA Kit DNA抽提试剂盒,美国Omega公司;FastPfu PolymeraseDNA抽提试剂盒,美国MP Biomedicals公司;FastPfu Polymerase,北京全式金生物技术有限公司;琼脂糖Agarose 75510019,美国Thermo Scientific公司;DNA凝胶回收纯化试剂盒,中国逾华公司。
1.2 仪器与设备
FastPfu Polymerase,北京全式金生物技术有限公司;T100 Thermal Cycler PCR仪,美国BIO-RAD公司;JY600C电泳仪,北京君意东方电泳设备有限公司。
1.3 实验方法
1.3.1 取样
生乳:从驼场储奶罐中收集生乳,然后将其保存在15 mL的冻存管中;挤奶瓶:用一根无菌拭子擦拭奶瓶的内壁,然后将拭子头放入1.8 mL冻存管中;乳头夹:用一根无菌拭子擦拭乳头夹的内壁,然后将拭子头放入1.8 mL冻存管中;储奶罐:用一根无菌拭子擦拭储奶罐的内壁,然后将拭子头放入1.8 mL冻存管中;乳头:使用无菌棉签擦拭每峰骆驼3个乳头,然后将拭子头放入1.8 mL的冻存管中;饮用水:从每个骆驼养殖场的饮水槽中取等量的水(20 mL),混合后装入50 mL的离心管中;饲料:从骆驼养殖场用无菌采样匙采集相同数量的样本(20 g),混合后装入50 mL的离心管中;粪便:在骆驼排便后,立即使用无菌采样器从每个骆驼群的粪便池中采集1~3 g的样本,将其置于50 mL的离心管内;驼舍空气:采用沉降法收集,经过无菌盐水洗脱后,存放在15 mL的冻存管中。采集完的样品放入液氮罐保藏,所用样品均取3份平行。
1.3.2 微生物多样性测序和数据处理
1.3.2.1 DNA抽提、扩增及测序
根据DNA提取试剂盒(FastDNA® Spin Kit for Soil和E.Z.N.A.® Soil DNA Kit)的使用说明书对DNA进行提取,利用1%琼脂糖凝胶电泳检测。采用338F和806R引物对指定的16S rRNA V3~V4区域PCR扩增,利用2%琼脂糖凝胶电泳检测混合后的产物后纯化,使用DNA凝胶回收试剂盒回收。通过使用QuantiFluorTM-ST蓝色荧光定量系统对PCR产物进行定量,然后按照不同样本所需的要求,调整比例混合。最后使用NEXTFLEX® Rapid DNA-Seq Kit进行建库,利用Illumina公司的Miseq PE300平台进行测序。本测序工作由美吉生物医药科技(上海)有限公司完成。
1.3.2.2 数据处理和分析
所有的数据处理均在美吉生物云平台上进行,优化处理相关数据,获得ASV(amplicon sequence variant)代表序列和丰度信息。基于ASV代表序列及丰度信息,可以进行物种分类学分析、群落多样性分析以及物种差异分析。根据分析结果结合统计等多种分析检验方法,利用SPSS 26.0和Origin 2021软件分析不同样本菌群结构,研究牧场微生物多样性和组成差异。
1.3.3 Source Tracker分析
Source Tracker称目标样本为Sink,微生物污染源或来源的样品为Source;基于贝叶斯算法,探究目标样本(Sink)中微生物污染源或来源(Source)的分析。根据Source样本和Sink样本的群落结构分布,来预测Sink样本中来源于各Source样本的组成比例。
2 结果与分析
2.1 ASV结果分析
2.1.1 稀释曲线分析
稀释曲线不仅可以用来评估样本物种的丰富度,也可以用来判断测序深度是否合理。当稀释曲线趋向平坦时,说明测序数据量足够大,可以覆盖绝大部分ASV。根据图1所示,所测样品都趋向平坦,表明包含所有样本的微生物多样性,测序结果具有可靠性。
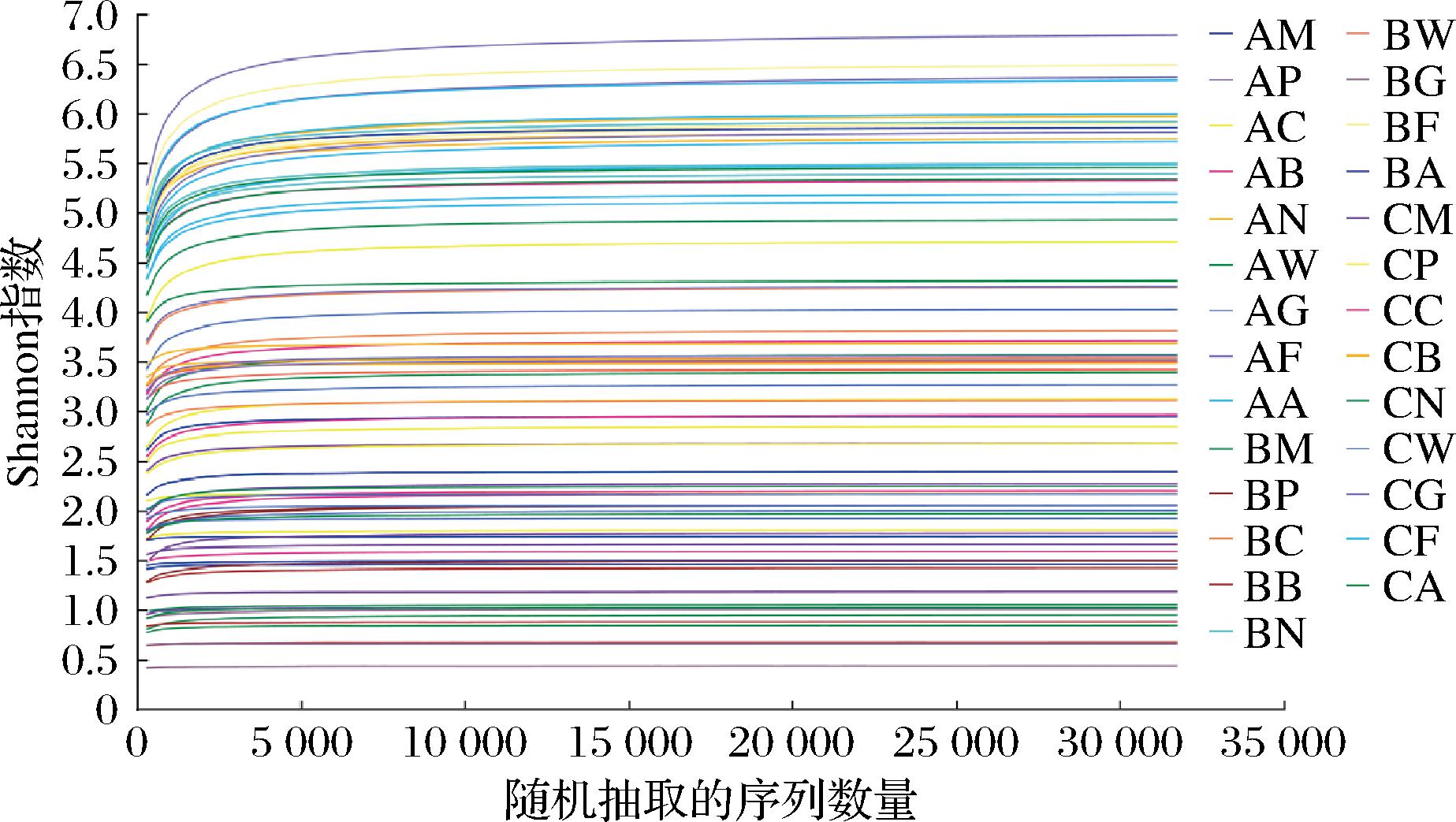
图1 Shannon指数的稀释曲线图
Fig.1 Dilution curve of Shannon index
注:AM-牧场A的生乳样品;AP-牧场A的挤奶瓶样品;AC-牧场A的乳头夹样品;AB-牧场A的储奶罐样品;AN-牧场A的骆驼乳头样品;AW-牧场A的饮用水样品;AG-牧场A的饲料样品;AF-牧场A的粪便样品;AA-牧场A的驼舍空气样品;BM-牧场B的生乳样品;BP-牧场B的挤奶瓶样品;BC-牧场B的乳头夹样品;BB-牧场B的储奶罐样品;BN-牧场B的骆驼乳头样品;BW-牧场B的饮用水样品;BG-牧场B的饲料样品;BF-牧场B的粪便样品;BA-牧场B的驼舍空气样品;CM-牧场C的生乳样品;CP-牧场C的挤奶瓶样品;CC-牧场C的乳头夹样品;CB-牧场C的储奶罐样品;CN-牧场C的骆驼乳头样品;CW-牧场C的饮用水样品;CG-牧场C的饲料样品;CF-牧场C的粪 便样品;CA-牧场C的驼舍空气样品(下同)。
2.1.2 等级-丰度曲线
等级-丰度曲线可以直观地反映样本中物种的丰富度和均匀度。各组样本的总体趋势一致,呈现较为平滑的曲线,表明物种分布相对一致。在水平方向,物种的丰度由曲线的宽度来反映,物种的丰度越高,曲线在横轴上的范围越大;曲线的形状(平滑程度)反映了样品中物种的均度,曲线越平缓,物种分布越均匀。结果如图2所示,BG(饲料)和CB(储奶罐)在水平方向上的范围较大,物种丰度最高。由图3可知,A、B、C三个牧场各鉴别出508、443、447个ASVs,共计1 217个ASVs,其中共同ASVs 38个,各牧场独有ASVs分别为410、329、335个,说明与B、C牧场相比,A牧场的物种更为丰富。
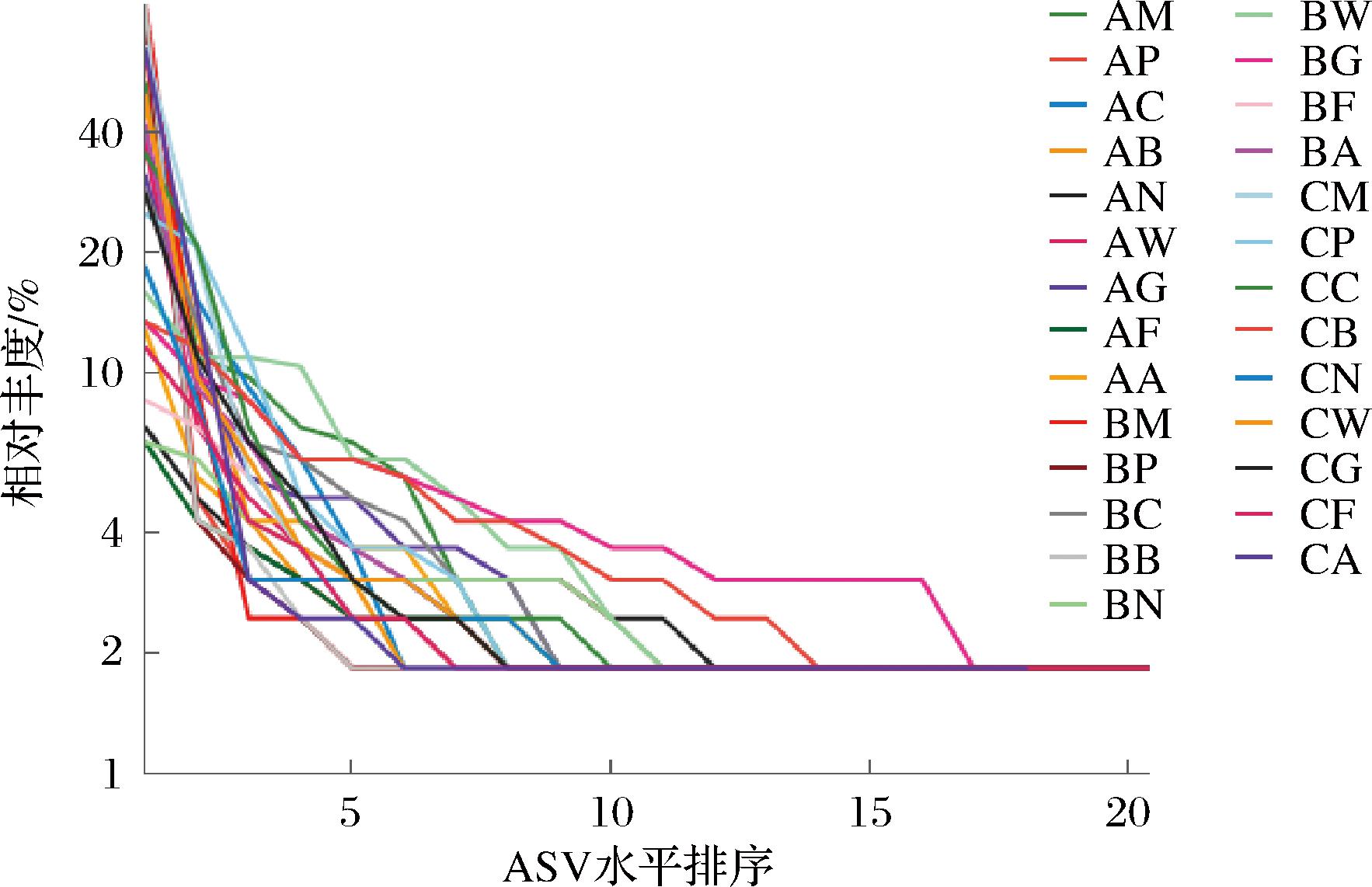
图2 基于ASV数目的等级-丰度曲线
Fig.2 Rank-abundance curve based on the number of ASVs
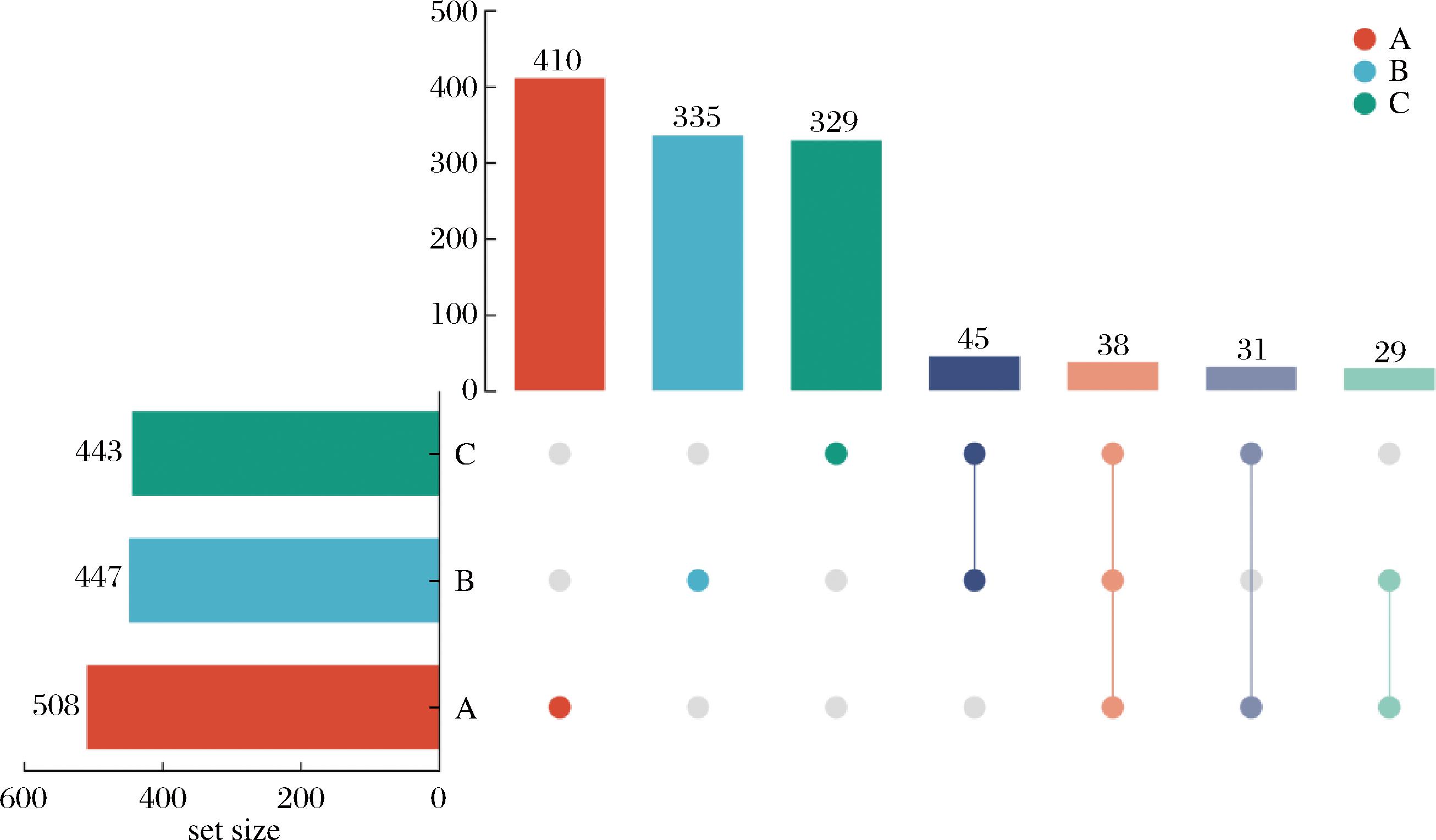
图3 ASV韦恩图
Fig.3 Venn diagram of ASV
2.2 物种组成分析
由图4可知,在属水平上不同样品中微生物种分布存在差异。A牧场乳样相对丰富的属主要为乳球菌属(Lactococcus)占比52.73%、金黄杆菌属(Chryseobacterium)占比15.76%;B牧场乳样中罗思氏菌属(Rothia)的占比最大,高达83.64%;C牧场乳样中优势细菌属以罗思氏菌属(Rothia)和不动杆菌属(Acinetobacter)为主,分别占64.85%、17.58%。先前雷鸣等[16]通过16S rRNA高通量测序发现原料乳中嗜冷菌以黄杆菌属、假单胞菌属、不动杆菌属和嗜冷杆菌属为主,其中假单胞菌属是危害最大的嗜冷菌。
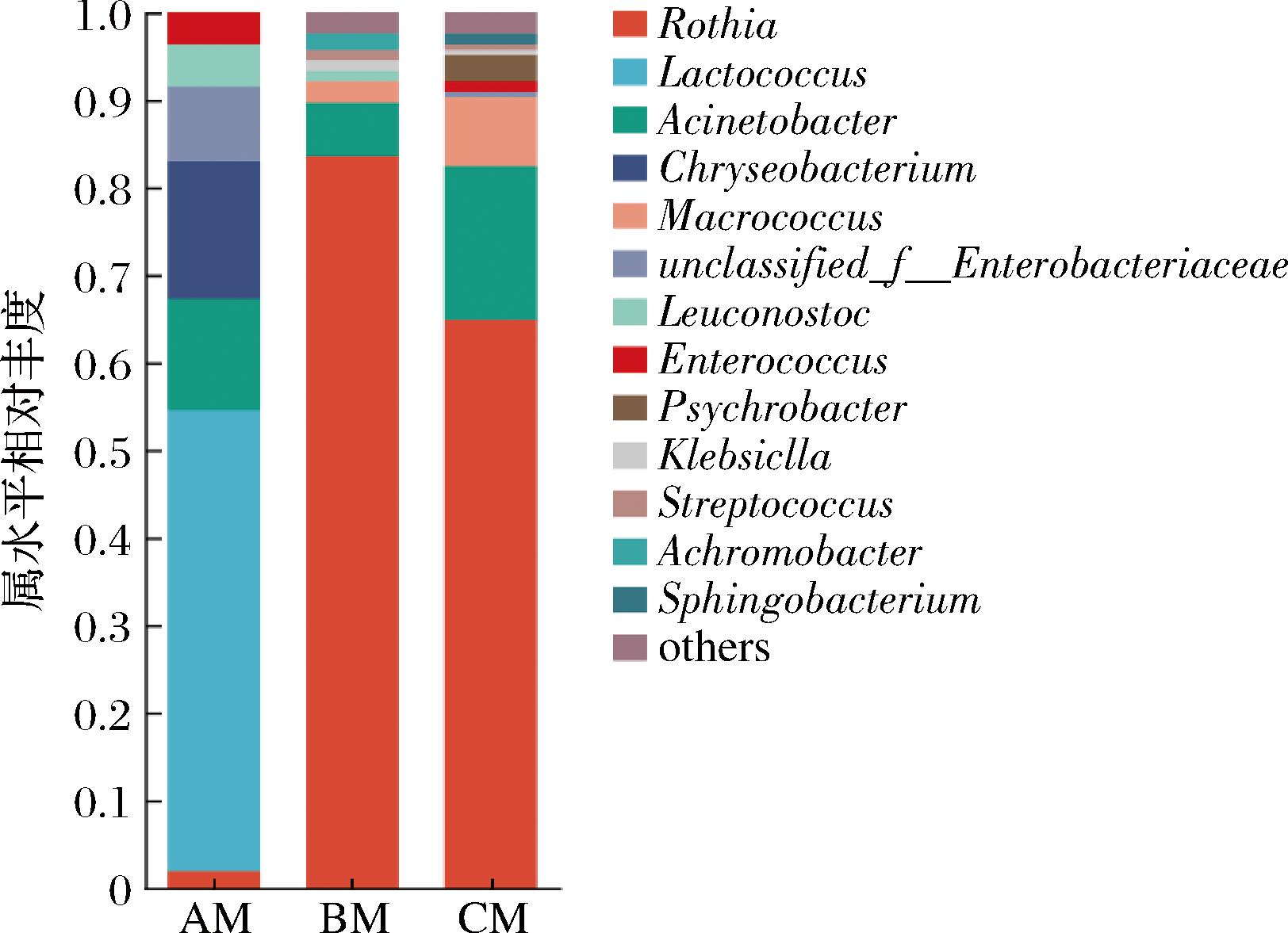
图4 生乳样品中微生物在属水平的相对丰度
Fig.4 The relative abundance of microorganisms at the genus level in raw milk samples
对于挤奶瓶样品(图5-a),在属水平上,A驼场挤奶瓶样品(AP)中优势细菌属为产碱菌属(Alcaligenes)(65.45%)、土地杆菌属(Pedobacter)(4.85%)、假单胞菌属(Pseudomonas)(3.64%);在B、C驼场挤奶瓶样本(BP、CP)中,罗思氏菌属(Rothia)分别占73.94%、24.24%,无色杆菌属(Achromobacter)分别占3.64%、20.00%。张淑媛[17]发现在挤奶的过程中,假单胞菌属、蜡状芽孢杆菌会通过乳房和挤奶用具等途径污染原料乳。因此,导致不同牧场挤奶瓶样品中优势菌群不同主要与挤奶瓶等收集容器的卫生情况与原料乳的质量有关。

a-牧场挤奶瓶样品;b-牧场乳头夹样品;c-牧场储奶罐样品;d-牧场乳头样品;e-牧场饮用水样品;f-牧场饲料样品;g-牧场粪便样品;h-牧场驼舍空气样品
图5 环境样品微生物在属水平的相对丰度
Fig.5 The relative abundance of microorganisms at the genus level in environmental samples
对于乳头夹样品(图5-b),在属水平上,A驼场乳头夹样品(AC)中有3个优势属,分别为不动杆菌属(Acinetobacter)(43.64%)、无色杆菌属(Achromobacter)(16.97%)和黄单胞菌属(Stenotrophomonas)(12.12%);在B驼场乳头夹样品(BC)中有2个优势属,分别为无色杆菌属(Achromobacter)(20.61%)和金黄微菌属(Chryseomicrobium)(13.94%);在C驼场乳头夹样品(CC)中有2个优势属,分别为罗思氏菌属(Rothia)(31.52%)和栖水菌属(Enhydrobacter)(15.76%)。
对于储奶罐样品(图5-c),在属水平上,A驼场储奶罐样品(AB)中相对丰富的属主要为无色杆菌属(Achromobacter),占比46.06%;在B驼场储奶罐样品(BB)中相对丰富的属主要为罗思氏菌属(Rothia),占比82.42%;在C驼场储奶罐样品(CB)中相对丰富的属主要为不动杆菌属(Acinetobacter),占比26.67%和嗜冷杆菌属(Psychrobacter),占比10.30%。
对于骆驼乳头样品(图5-d),在属水平上,在A驼场乳头样品(AN)中的优势属为棒状杆菌属(Corynebacterium),相对丰度为10.91%。B和C驼场乳头样品(BN、CN)中主要微生物分别为棒状杆菌属(Corynebacterium)和无色杆菌属(Achromobacter),分别占比13.33%和12.12%。李强等[18]曾对河南某湖羊养殖场乳房炎病原菌进行分离鉴定,得到葡萄球菌和大肠杆菌2种病原菌。FURSOVA等[19]在俄罗斯中部地区对患有乳房炎的奶牛乳中检出了假单胞菌科、伯克霍尔德菌科以及链球菌科、葡萄球菌科和芽胞杆菌科。本次结果与其所述不一致,说明3个牧场的骆驼健康状况良好。
禽舍作为主要的饲养环境,粪便和饲料等携带着大量致病微生物,如沙门氏菌、黄曲霉菌、大肠杆菌、和金黄色葡萄球菌等[20-21]。TANG等[22]发现在猪舍中的潜在病原体包括不动杆菌、葡萄球菌、假单胞菌、金黄色葡萄球菌等。而且PLEWA等[23]在研究中证实,冬季与夏季禽舍周围环境中的异养细菌和真菌数量差异可达100倍,而葡萄球菌的差异更是高达1 000倍。综上,畜舍中的微生物群落呈现复杂多样性,且不同季节对其影响较大。
对于骆驼饮用水样品(图5-e),在属水平上,A驼场饮用水样品(AW)中的优势属无色杆菌属(Achromobacter)相对丰度达36.36%;在B驼场饮用水样品(BW)中最高的菌属为黄杆菌属(Flavobacteriaceae)相对丰度达18.79%,其次为莱茵海默氏菌属(Rheinheimera)、马赛菌属(Malikia),相对丰度分别为15.76%和12.12%;在C驼场饮用水样品(CW)中丰度较高的菌属有马赛菌属(Malikia)和莱茵海默氏菌属(Rheinheimera),分别为58.79%和10.30%。
对于骆驼饲料样品(图5-f),在属水平上,A驼场骆驼饲料样品(AG)中的优势属为假单胞菌属(Pseudomonas)(43.64%);B驼场饲料样品(BG)中有3个优势属,分别为动性球菌属(Enterobacterales)(8.48%)、无色杆菌属(Achromobacter)(7.88%)、微杆菌属(Microbacterium)(7.27%);C驼场饲料样品(CG)中有3个优势属分别为醋酸杆菌属(Acetobacter)(23.64%)、嗜冷杆菌属(Psychrobacter)(12.12%)、无色杆菌属(Achromobacter)(11.52%)。饲料的质量对养殖动物产品有很大的影响,其中饮食对胃肠道微生物菌群结构起着决定作用,这可能是因为饲料的物理和化学特性决定了不同微生物的结构[24]。
对于骆驼粪便样品(图5-g),在属水平上,在A驼场骆驼粪便样品(AF)中丰度较高的菌属有克里斯滕森菌属(Christensenellae)和毛螺菌属(Lachnospira),分别占8.48%、6.67%;在B驼场骆驼粪便样品(BF)中丰度较高的菌属有克里斯滕森菌属(Christensenellae)和罗姆布茨菌属(Romboutsia),分别为9.09%、7.88%;在C驼场骆驼粪便样品(CF)中,优势菌属为索氏梭菌属(Paeniclostridium),占比10.91%。导致该现象的主要原因可能是粪便中微生物的组成与动物自身因素、饲粮和饲养管理有关[25]。
对于驼舍空气样品(图5-h),在属水平上,在A驼场驼舍空气样品(AA)中最高的菌属为盐水球菌属(Salinicoccus),相对丰度达11.52%,其次为无色杆菌属(Achromobacter)相对丰度为10.91%;在B驼场驼舍空气样品(BA)中优势属无色杆菌属(Achromobacter)相对丰度为41.21%;在C驼场驼舍空气样品(CA)中丰度较高的菌属为无色杆菌属(Achromobacter)占比63.64%。李红梅等[26]采用16S rDNA高通量测序方法揭示了四川省邛崃市金利猪场的空气微生物群落结构,假单胞菌属、放线菌属和链球菌属是优势微生物。POPESCU等[27]研究鉴定了牛舍中的空气微生物,发现主要的微生物有葡萄球菌、肠球菌、肠杆菌和链球菌。蒲施桦等[28]对西南地区不同类型猪舍内空气污染物变化规律研究,发现其中的差异不仅是因为饲养管理需求的不同,最主要的是在于当地的温度和湿度对不同季节、不同时段的通风量的影响,导致舍内污染物浓度和外排空气浓度的变化。
2.3 多样性分析
2.3.1 Alpha多样性分析
Alpha多样性是指一个特定区域或生态系统内的多样性,是反映丰富度和多样性的综合指标。Alpha多样性主要与2个因素有关:一是丰富度(主要包括Chao1指数和ACE指数);二是多样性(Shannon指数和Simpson指数)。
2.3.1.1 生乳Alpha多样性分析
如表1所示,3个牧场的样品的Chaol值、Shannon值和ACE值较接近,说明A牧场驼乳(AM)、B牧场驼乳(BM)、C牧场驼乳(CM)中群落组成相似度高。Shannon值越大,群落的多样性越高,分析的3个牧场样品中,A牧场驼乳的Shannon值比B、C牧场驼乳高,说明A牧场驼乳中的细菌群落最丰富。
表1 A、B、C三个牧场生乳样品的Alpha多样性指数
Table 1 Alpha diversity index statistics of raw milk samples from three pastures A,B,and C
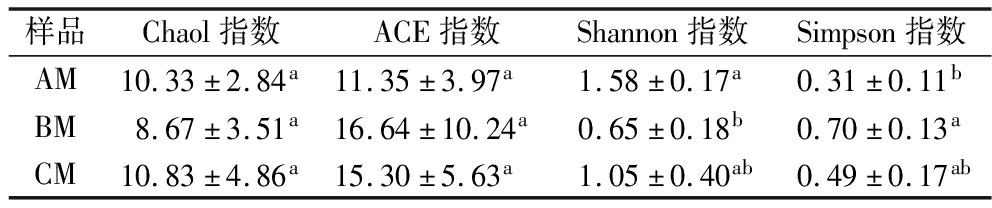
样品Chaol指数ACE指数Shannon指数Simpson指数AM10.33±2.84a11.35±3.97a1.58±0.17a0.31±0.11bBM8.67±3.51a16.64±10.24a0.65±0.18b0.70±0.13aCM10.83±4.86a15.30±5.63a1.05±0.40ab0.49±0.17ab
注:AM、BM、CM分别代表3个牧场的生乳样品;不同字母表示同列数据差异显著(P<0.05)。
2.3.1.2 各牧场样品Beta多样性分析
Beta多样性分析目的是研究样本间的多样性,从整体上分析样本间的相似性或差异性。本研究主要通过非度量多维缩放(nonmetric multidimensional scaling,NMDS)和相似性分析(analysis of similarities,ANOSIM)来呈现Beta多样性分析结果。结果如图6所示,在A牧场中,驼乳(AM)和粪便(AF)样品明显区分其他几组,距离较远说明这2个样品组微生物群落与其他组差异较大;饮用水(AW)、储奶罐(AB)和空气(AA)样品椭圆小,且各样本紧密的挨在一起,说明这3组样品组的微生物种群结构非常相似,且组内微生物种类差异也较少。通过Anosim图发现R值大于0.5,说明组间差异显著大于组内差异,分组有意义。
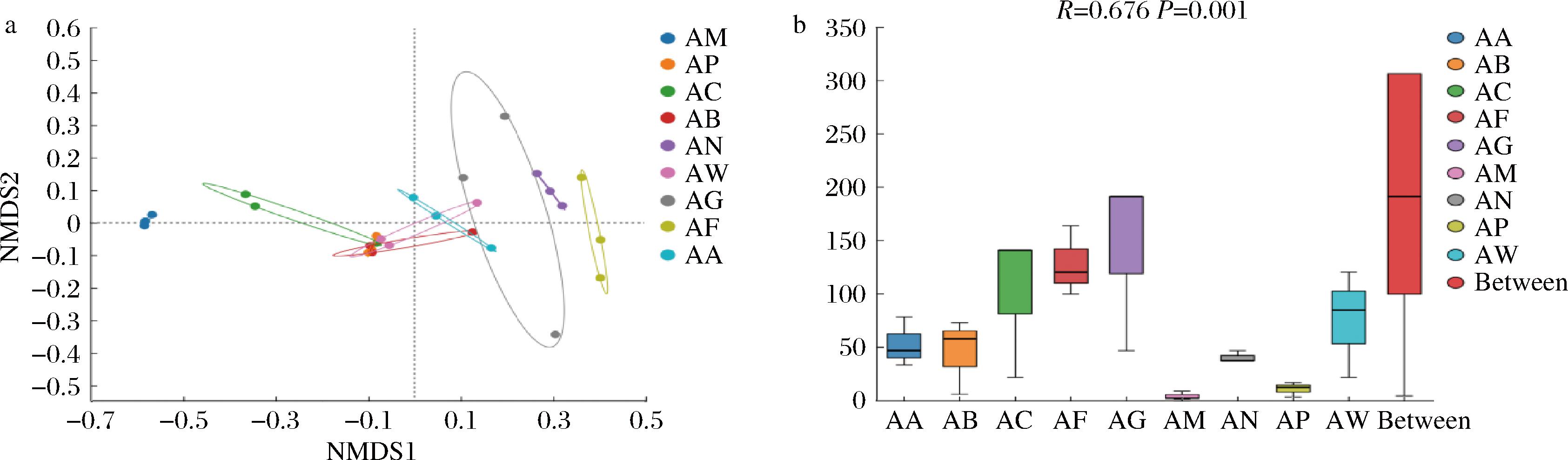
a-A牧场样品基于ASV的NMDS分析;b-A牧场样品Anosim分析
图6 A牧场样品菌群Beta多样性差异分析
Fig.6 Differential analysis of the Beta diversity of the bacterial flora of A pasture samples
结果如图7所示,在B牧场中,BW和BC样品与其他样本没有重合部分,说明其微生物种群结构相差较大。BM与BP、BB都有重叠,说明生乳与其他2个样品间差异不显著。通过Anosim图发现R值为0.8,说明组间差异显著大于组内差异,分组有意义。
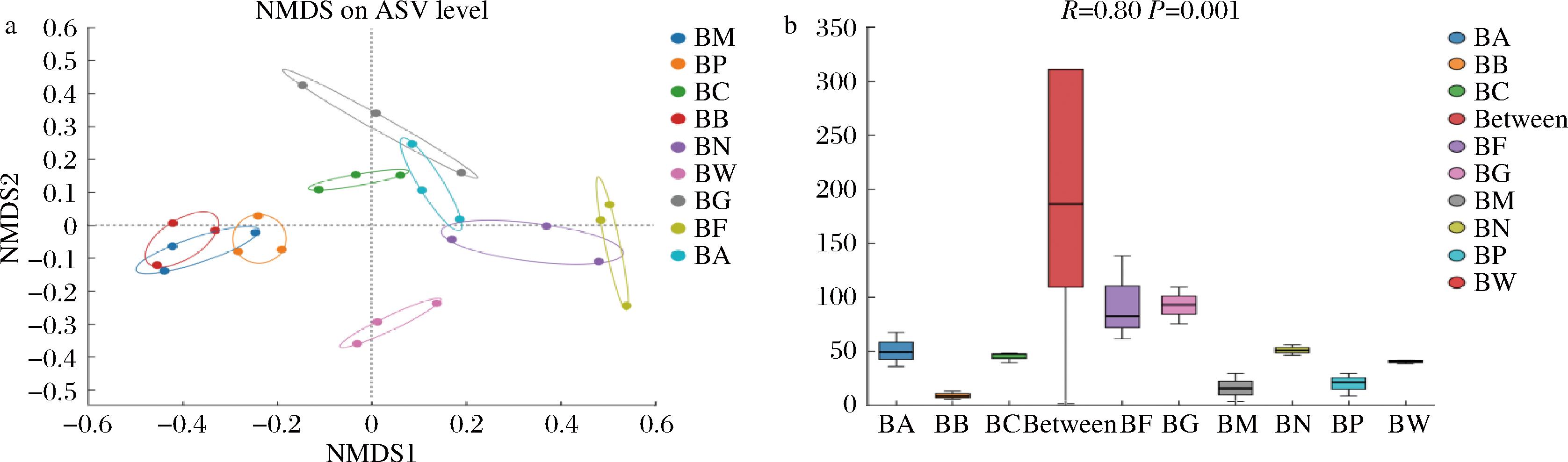
a-B牧场样品基于ASV的NMDS分析;b-B牧场样品Anosim分析
图7 B牧场样品菌群Beta多样性差异分析
Fig.7 Differential analysis of Beta diversity of flora in B ranch samples

a-C牧场样品基于ASV的NMDS分析;b-C牧场样品Anosim分析
图8 C牧场样品菌群Beta多样性差异分析
Fig.8 Differential analysis of beta diversity of bacterial flora in C ranch samples
结果如图8所示,在C牧场中,CW、CP、CM和CA样品组明显区分其他组,说明这几组微生物群落与其他组差异较大。通过观察Anosim图发现所有样品之间都不重叠,该牧场间样品之间相似性较低,其组间差异显著大于组内差异。
2.4 Source Tracker模型
Source Tracker可以追踪微生物来源,分析不同来源的微生物群落的组成,并通过比较来揭示微生物群的相关模式,估算细菌源群落对目标群落的贡献,进而更系统地分析存在的驱动因素。先前杜兵耀[29]已利用Source Tracker模型对生乳中微生物风险关键点分析,确认在郑州牛场和齐齐哈尔牛场最主要风险关键点是奶杯和药浴杯。张喜庆[30]通过Source Track分析证实奶牛舍PM 2.5中细菌主要来源于鼻拭子、饲料、卧床垫料以及粪便,且在各阶段牛舍中所占比例具有一定差异。原料乳中的微生物群落易受到其他因素的影响发生变化,本研究采用Source Tracker模型算法对A、B、C三个牧场的环境微生物进行分析,旨在确定生驼乳微生物风险关键点。从图9能够直观地从科水平上发现3个牧场样品的微生物风险关键点占比。分析发现乳头与A驼场中生乳样品中最主要的微生物具有显著相关性,挤奶瓶与B驼场中生乳样品中最主要的微生物具有显著相关性,C驼场中生乳样品中与最主要的微生物具有显著相关性的是乳头夹。乳头夹是A驼场生乳样品的第二重要的微生物风险关键点,而储奶罐则是B驼场和C驼场生乳样品中的第二重要的微生物风险关键点。
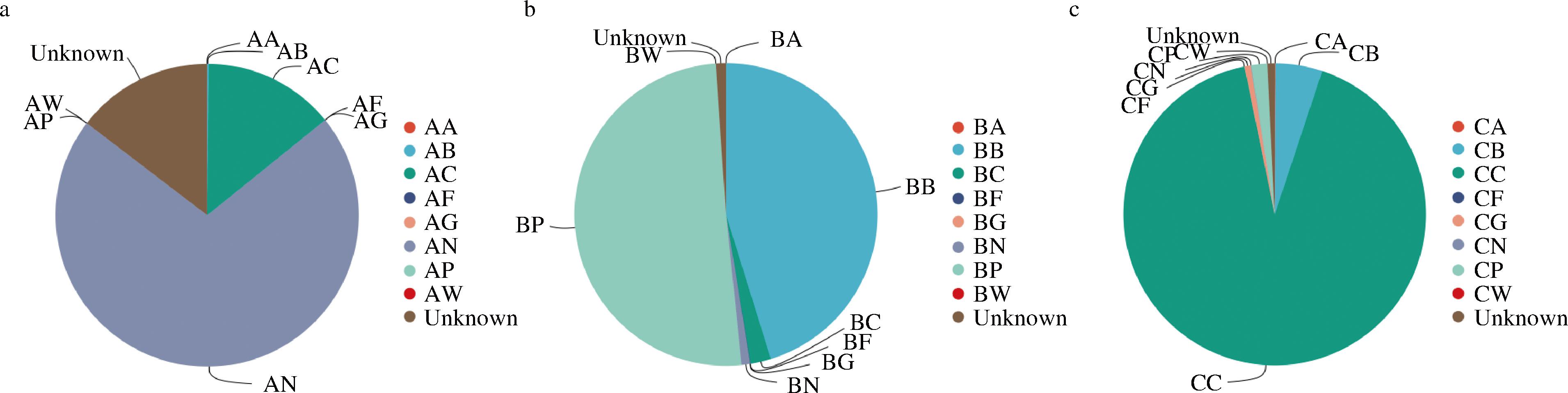
a-A牧场的风险关键点分析;b-B牧场的风险关键点分析;c-C牧场的风险关键点分析
图9 Source Tracker模型推断微生物在科水平的风险关键点
Fig.9 Source Tracker model infers the key risk points of microorganisms at the family level
原料乳是控制乳品质量安全的第一环,需对影响其品质的因素进行分析,进而采取积极有效的控制方法。引起原料乳中出现腐败微生物的原因有奶牛乳腺炎、牧场的卫生条件、季节以及地理位置等多种因素[31]。首先,养殖环节的管理至关重要,这是保证生乳质量安全的基本要求[32]。做好挤奶前后的药浴,可有效减少乳房炎的发生[33]。乳头健康干燥,无污染物或其他异物残留。再者,挤奶设备、器具的卫生情况直接决定了驼奶的细菌数量,挤奶器具消毒不及时会造成菌落总数超标[34]。对挤奶设备进行稳定清洗,定期记录检查。最后,饲料,特别是粗饲料是影响原料奶质量的关键因素之一[35]。采购的饲料需验收合格后才可投喂,科学规划用量,发霉变质的饲料要及时处理。
3 结论
本研究采集3个牧场的生乳样品进行16S rRNA测序结果显示,A驼场生乳中乳球菌属(Lactococcus)的相对丰度占比高达52.73%,而罗思氏菌属(Rothia)是B、C两个驼场主要的微生物,相对丰度分别达到83.64%和64.85%。挤奶瓶、乳头夹、储奶罐、乳头、饮用水、饲料、粪便和驼舍空气样品之间的微生物多样性存在差异。在A、B、C三个牧场中造成生乳样品微生物污染的最主要风险关键点,分别为乳头和乳头夹、挤奶瓶和储奶罐、乳头夹和储奶罐。因此,为减少生驼乳由微生物带来的风险问题,需加强对乳头、乳头夹、挤奶瓶和储奶罐关键点的控制。
[1] KONUSPAYEVA G,FAYE B,LOISEAU G.The composition of camel milk:A meta-analysis of the literature data[J].Journal of Food Composition and Analysis,2009,22(2):95-101.
[2] AIT EL ALIA O,ZINE-EDDINE Y,KZAIBER F,et al.Towards the improvement of camel milk consumption in Morocco[J].Small Ruminant Research,2023,219:106888.
[3] STAHL T,SALLMANN H P,DUEHLMEIER R,et al.Selected vitamins and fatty acid patterns in dromedary milk and colostrum[J].Journal of Camel Practice and Research,2006,13(1):53-57.
[4] AL-JUBOORI A T,MOHAMMED M,RASHID J,et al.Nutritional and medicinal value of camel (Camelus dromedarius) milk[C].WIT Transactions on Ecology and The Environment.WIT Press,2013:221-232.
[5] EL-AGAMY E I,NAWAR M,SHAMSIA S M,et al.Are camel milk proteins convenient to the nutrition of cow milk allergic children?[J].Small Ruminant Research,2009,82(1):1-6.
[6] AL HAJ O A,AL KANHAL H A.Compositional,technological and nutritional aspects of dromedary camel milk[J].International Dairy Journal,2010,20(12):811-821.
[7] EREIFEJ K I,ALU’DATT M H,ALKHALIDY H A,et al.Comparison and characterisation of fat and protein composition for camel milk from eight Jordanian locations[J].Food Chemistry,2011,127(1):282-289.
[8] SINGH R,MAL G,KUMAR D,et al.Camel milk:An important natural adjuvant[J].Agricultural Research,2017,6(4):327-340.
[9] AQIB A I,FAKHAR-E-ALAM KULYAR M,ASHFAQ K,et al.Camel milk insuline:Pathophysiological and molecular repository[J].Trends in Food Science &Technology,2019,88:497-504.
[10] EL-FAKHARANY E M,EL-BAKY N A,LINJAWI M H,et al.Influence of camel milk on the hepatitis C virus burden of infected patients[J].Experimental and Therapeutic Medicine,2017,13(4):1313-1320.
[11] WANG Z X,ZHANG W Y,WANG B N,et al.Influence of Bactrian camel milk on the gut microbiota[J].Journal of Dairy Science,2018,101(7):5758-5769.
[12] YADAV A K,KUMAR R,PRIYADARSHINI L,et al.Composition and medicinal properties of camel milk:A review[J].Asian Journal of Dairy and Food Research,2015,34(2):83.
[13] MAL G,SENA D S,JAIN V K,et al.Therapeutic value of camel milk as a nutritional supplement for Multiple Drug Resistant (MDR) tuberculosis patients[J].Israel Journal of Veterinary Medicine,2006,61:88-91.
[14] 周轩.食品加工过程中微生物污染的控制策略[J].食品安全导刊,2024(8):1-3.ZHOU X.Control strategies for microbial contamination in food processing[J].China Food Safety Magazine,2024(8):1-3.
[15] ZHAO J N,FAN H,KWOK L Y,et al.Analyses of physicochemical properties,bacterial microbiota,and lactic acid bacteria of fresh camel milk collected in Inner Mongolia[J].Journal of Dairy Science,2020,103(1):106-116.
[16] 雷鸣,胡楠,王远一飞,等.低温储存条件对原料乳嗜冷菌及细菌多样性的影响[J].中国乳品工业,2019,47(7):14-18.LEI M,HU N,WANG Y Y F,et al.Effects of low temperature storage conditions on psychrophilic bacteria and bacterial diversity in raw milk[J].China Dairy Industry,2019,47(7):14-18.
[17] 张淑媛.原料牛奶中微生物的污染问题与检测分析[J].饮料工业,2022,25(1):37-39.ZHANG S Y.Pollution problem and detection analysis of microorganisms in raw milk[J].Beverage Industry,2022,25(1):37-39.
[18] 李强,王改玲,赵泽慧,等.河南某湖羊场乳房炎的病原菌分离鉴定及防治[J].动物医学进展,2016,37(4):127-130.LI Q,WANG G L,ZHAO Z H,et al.Isolation and identification of main pathogenic bacteria causing Hu sheep mastitis[J].Progress in Veterinary Medicine,2016,37(4):127-130.
[19] FURSOVA K K,SOKOLOV S L,SHCHANNIKOVA M P,et al.Changes in the microbiome of milk in cows with mastitis[J].Doklady.Biochemistry and Biophysics,2021,497(1):75-80.
[20] 梁雨,邱志刚,李辰宇,等.冬季天津典型集约化畜禽养殖场粪便微生物污染调查[J].环境与健康杂志,2019,36(7):595-598.LIANG Y,QIU Z G,LI C Y,et al.Microbial contamination in feces of typical intensive livestock and poultry farms in Tianjin in winter[J].Journal of Environment and Health,2019,36(7):595-598.
[21] 武侠均.食品与饲料中致病微生物检测技术及展望[J].中国饲料,2022(21):109-117.WU X J.Detection technology and prospect of pathogenic microorganisms in food and feed[J].China Feed,2022(21):109-117.
[22] TANG Q,HUANG K,LIU J Z,et al.Seasonal variations of microbial assemblage in fine particulate matter from a nursery pig house[J].Science of The Total Environment,2020,708:134921.
[23] PLEWA K,LONC E.Analysis of airborne contamination with bacteria and moulds in poultry farming:A case study[J].Polish Journal of Environmental Studies,2011,20(30):725-731.
[24] BELANCHE A,PATRA A K,MORGAVI D P,et al.Editorial:Gut microbiome modulation in ruminants:Enhancing advantages and minimizing drawbacks[J].Frontiers in Microbiology,2021,11:622002.
[25] 刘亭梅.基于微生物组学解析性别对贵州本地羊粪便微生物区系及其发酵产物的影响[D].贵阳:贵州大学,2024.LIU T M.Microbiome-based analysis of the effects of gender on the microbiota of native sheep feces and their fermentation products in Guizhou[D].Guiyang:Guizhou University,2024.
[26] 李红梅,白林,姜冬梅,等.基于16SrDNA高通量测序方法检测猪舍空气微生物多样性[J].中国畜牧杂志,2015,51(3):81-84.LI H M,BAI L,JIANG D M,et al.Microbial diversity of piggery air detected by 16S rDNA high-throughput sequencing[J].Chinese Journal of Animal Science,2015,51(3):81-84.
[27] POPESCU S,BORDA C,DIUGAN E A.Microbiological air quality in tie-stall dairy barns and some factors that influence it[J].African Journal of Agricultural Research,2011,6(32):6726-6734.
[28] 蒲施桦,王浩,龙定彪,等.西南地区不同类型猪舍内空气污染物变化规律[J].猪业科学,2021,38(6):94-98.PU S H,WANG H,LONG D B,et al.Variation of air pollutants in different types of pig houses in southwestern China[J].Swine Industry Science,2021,38(6):94-98.
[29] 杜兵耀.生乳中微生物风险关键点分析及假单胞菌特征研究[D].兰州:兰州大学,2022.DU B Y.Analysis of key points of microbial risk and characterization of Pseudomonas aeruginosa in raw milk[D].Lanzhou:Lanzhou University,2022.
[30] 张喜庆.奶牛舍PM2.5微生物多样性分析及其对大鼠肺脏损伤分子机制研究[D].长春:吉林农业大学,2024.ZHANG X Q.Analysis of microbial diversity of PM2.5 in dairy barn and its molecular mechanism of lung injury in rats[D].Changchun:Jilin Agricultural University,2024.
[31] 邱月,秦雪,马钰,等.原料乳中腐败微生物对液态乳品质的影响及防控[J].食品安全质量检测学报,2023,14(16):8-15.QIU Y,QIN X,MA Y,et al.Effects of spoilage microorganisms in raw milk on the quality of liquid milk and their prevention and control[J].Journal of Food Safety &Quality,2023,14(16):8-15.
[32] 张能飞,王凯,唐都,等.牛场生乳的质量安全管理[J].江西畜牧兽医杂志,2023(1):26-28.ZHANG N F,WANG K,TANG D,et al.Quality and safety management of raw milk in cattle farm[J].Jiangxi Journal of Animal Husbandry &Veterinary Medicine,2023(1):26-28.
[33] 许翊冉,于滨,邱殿锐,等.夏季极端天气条件下奶牛场防范及应对措施[J].粮食与饲料工业,2024(3):41-43.XU Y R,YU B,QIU D R,et al.Prevention and countermeasures of dairy farm under extreme weather conditions in summer[J].Grain and Feed Industry,2024(3):41-43.
[34] 韩海燕,蔡扩军.驼奶质量安全问题的现状及影响因素[J].中国畜牧业,2023(4):28-29.HAN H Y,CAI G J.Current situation and influencing factors of camel milk quality and safety[J].China Animal Husbandry,2023(4):28-29.
[35] 王雷雷,金惠玉.影响原料奶质量安全的因素及控制措施研究[J].中国乳业,2014(8):54-55.WANG L L,JIN H Y.Study on the factors affecting the quality and safety of raw milk and its control measures[J].China Dairy,2014(8):54-55.