己二醛又名1,6-己二醛,是合成橡胶、塑料等高附加值产品的化学原料[1]。具体来说,己二醛可用于合成聚酰胺(如尼龙-6,6)、聚酯和聚氨酯的重要单体,如1,6-己二酸、1,6-己二胺、1,6-己二醇等。目前,己二醛的化学合成法主要有2种,一种是1,6-环己二醇或环己烯的多步氧化反应,另一种是丁二烯的选择性加氢甲酰化,这些方法均存在成本高、不易循环、对环境不友好[2]等问题,导致己二醛售价昂贵,高达600 美元/g。
合成生物学和代谢工程可通过构建独特的代谢途径生产各种化学品[3],多种酶被报道可用于醛类物质的催化合成[4-5]。其中,羧酸还原酶(carboxylate reductase,CARs)(EC 1.2.1.30)借助ATP激活羧酸底物,并利用NADPH可将羧酸底物还原成相应的醛[6-7]。目前,已经鉴定出能够有效合成香草醛、肉桂醛、异丁醛和辛醛的多种CAR[3,8-10]。但是截至目前尚未有二元醛生物合成的系统研究,绝大多数报道都是围绕单醛合成展开,这可能是因为二醛的合成相较单醛更为复杂,猜测是因为经过氧化还原合成一个醛基生成半缩醛后底物结合位点化学能的改变使得第二个醛基的生成更为困难,因此研究生物合成己二醛具有重要意义,可以为脂肪族二醛的生物合成提供一定的借鉴。
然而,大肠杆菌高活性内源醛还原酶(aldehyde reductase,ALR)/醛酮还原酶(aldo-keto reductase,AKR)会自动将醛转化为相应的醇[3],以防止醛基积累[11]。合理敲除大肠杆菌基因组中的ALRs/AKRs基因可减少竞争性醇的形成,RODRIGUEZ等[1]报道了一株能将C2~C12脂肪醛的还原通量显著降低的醛脱氢酶缺陷型大肠杆菌。ALR对己二醛生物合成的影响如图1所示,YahK和YjgB已被报道可以有效地催化己醛[1,8,12];而YbbO对C2-C10醛类底物均具有催化能力[1];同时,YqhD已被鉴定对包含己醛在内的多种碳链长度的醛类物质具有还原酶活性[13-14],而YqhC是YqhD的转录激活因子,且yqhC-yqhD基因簇是醛代谢调控的关键靶点[15],在大肠杆菌K-12 MG1655 RARE菌株中敲除yqhC、yqhD、yahK、yjgB基因可以削弱脂肪醛的过度还原[8]。
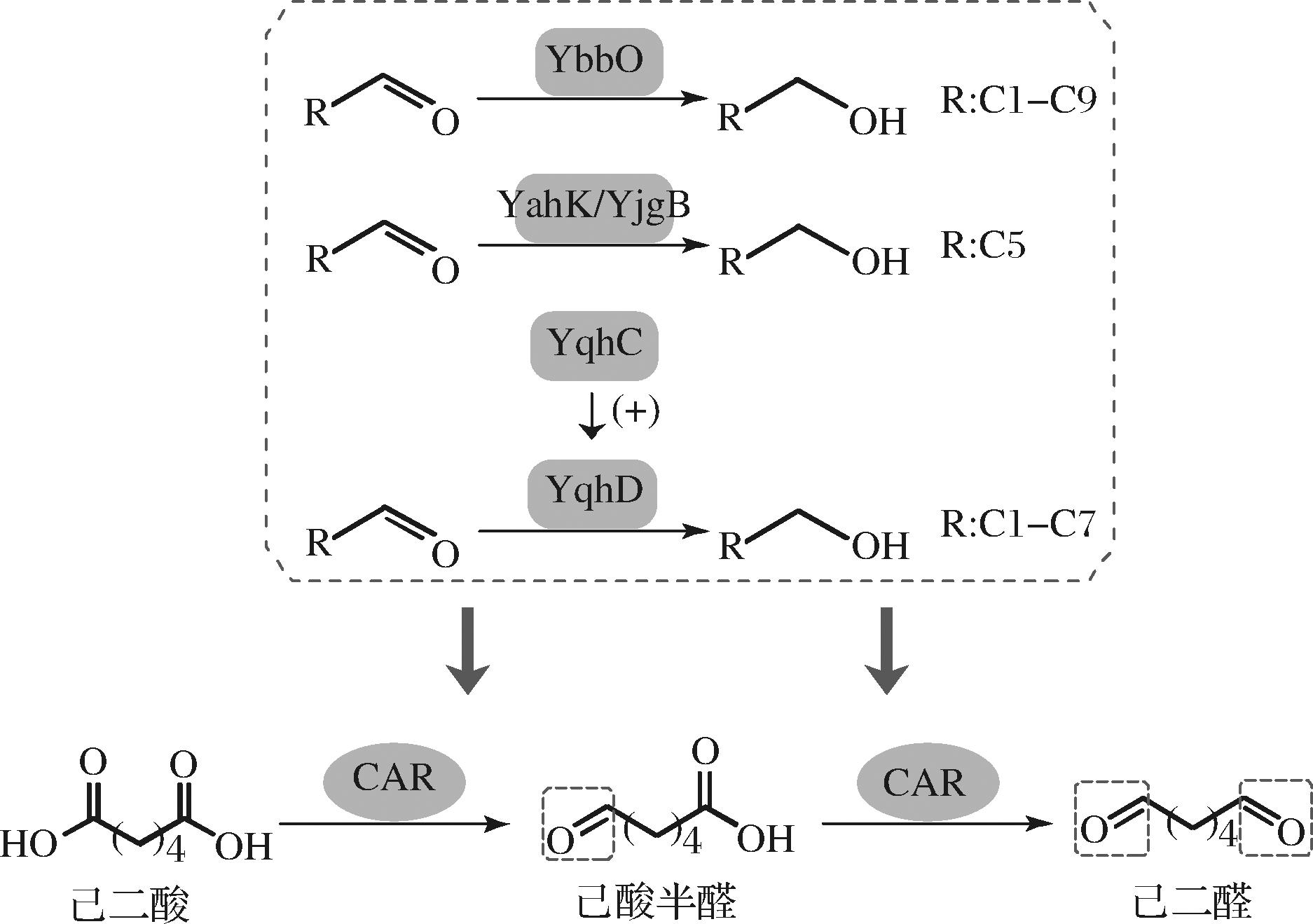
图1 内源醛还原酶对己二醛生物合成的影响
Fig.1 Effect of endogenous aldehyde reductase on the biosynthesis of 1,6-hexanedial
此外,醛对底盘细胞的毒性也是造成胞内产物难以积累的另一原因,具体表现为醛存在时微生物生长受到抑制,其形态也会发生一定变化[16]。究其原因,醛基会与生物靶标上的亲核位点形成共价键,由此形成的加合物会损害包括DNA、RNA、蛋白质和脂质在内的大分子的功能,最终产生细胞毒性[17]。无细胞合成不需维持细胞的生命,而是通过直接添加辅因子、底物而产生目标化学品,能够绕过底物限制[18],避免有毒产物对活体细胞的影响。
在本研究中,首先构建了己二醛合成工程菌株,使用基因编辑技术敲除高活性的ALRs和AKRs,开展CAR同工酶的筛选,并对该菌株的发酵过程进行强化,最后,利用无细胞体外催化以缓解毒性抑制,最终己二醛的产量提高30余倍,达到295.8 mg/L,本研究探讨了己二醛的生物法合成机理,可为其他重要二元醛的生物法合成提供支持与借鉴。
1 材料与方法
1.1 材料和试剂
质粒pRSF-MAB CAR、pRSF-MSM CAR、pRSF-MAV CAR、pRSF-Ni CAR、pRSF-MAB-Ni CAR、pRSF-MAB-MSM CAR、pRSF-MAB-MAV CAR由本实验室保存,E.coli BL21(DE3)△4由本研究改造获得。
1.2 实验方法
1.2.1 菌株培养方法
将储存在-20 ℃下的质粒取出并转化到E.coli BL21(DE3)感受态细胞中。将单个菌落转移到10 mL LB中,过夜培养后以2 mL的接种量接种到50 mL LB培养基中,培养至OD=0.8后,加入1 mmol/L的异丙基β-D-1-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG),并加入5 g/L己二酸底物,转至30 ℃,250 r/min条件下进行发酵实验。
1.2.2 基因敲除
使用JIANG等[19]之前构建的CRISPR-Cas9双质粒系统进行基因组操作。1)首先将质粒pCas转化到待敲除菌株中;2)将质粒pTarget与目的基因上下游500 bp融合片段通过电穿孔共同转化至含有pCas的待敲除菌株中,N20序列由网络工具CRISPOR(http://crispor.tefor.net/)设计;3)验证基因敲除成功后加入100 mmol/L IPTG诱导以删除pTarget,在确认pTarget被删除后,将菌株的培养温度改为42 ℃,以消除质粒pCas。引物合成及Sanger测序在安升达生物科技有限公司和上海生工生物工程公司进行。
1.2.3 发酵优化
底物浓度优化:为探究底物浓度对己二醛产量的影响,控制底物己二酸的质量浓度分别为1、2、3、4、5、6、7、8 g/L,同时控制诱导OD600=0.8,IPTG浓度为1 mmol/L,并通过GC检测己二醛的最终产量。
底物加入时间优化:为探究底物加入时间对己二醛产量的影响,分别在诱导后0、2、4、6、8、10 h后加入1 g/L底物己二酸,同时控制诱导OD600=0.8,IPTG浓度为1 mmol/L,并通过GC检测己二醛的最终产量。
诱导OD600优化:为探究诱导OD600对己二醛产量的影响,分别在OD600为0.4、0.8、1.2、1.6、2、2.4时加入1 mmol/L IPTG,诱导后4 h加入1 g/L己二酸,并通过GC检测己二醛的最终产量。
IPTG浓度优化:为探究IPTG浓度对己二醛产量的影响,在OD600=0.8时分别加入浓度为0.2、0.4、0.6、0.8、1、1.2、1.4 mmol/L的IPTG,诱导后4 h加入1 g/L己二酸,并通过GC检测己二醛的最终产量。
先收集菌体后开展催化优化:在OD600=0.8时加入0.4 mmol/L IPTG后,分别将菌液培养4、8、12、16、20、24 h,收集菌体后控制菌浓相同,分别加入1 g/L己二酸底物进行催化,并通过GC检测己二醛的最终产量。
1.2.4 蛋白表达和纯化
CAR蛋白均是在大肠杆菌BL21(DE3)△4中表达并纯化获得。将含有对应基因表达质粒的大肠杆菌BL21(DE3)△4菌株接种至含10 mL LB培养基的50 mL三角瓶中,并在37 ℃,250 r/min摇床振荡培养过夜。随后,将种子液以2 mL的接种量接种至含100 mL LB培养基的250 mL三角瓶中,再次在37 ℃,250 r/min摇床振荡培养至OD600为0.8时加入1 mmol/L IPTG诱导,转至30 ℃,250 r/min条件下进行菌体培养。孵育12~16 h后使用冷冻离心机5 000 r/min离心7 min,收集菌体。用含20 mmol/L咪唑的pH 7.4的Tris-HCl缓冲液将收集的菌体清洗2遍并重悬,经高压细胞破碎仪破碎。将破碎液使用冷冻离心机以10 000 r/min离心15 min,上清液用于蛋白纯化。蛋白纯化具体步骤可参考Ni-NTA 6FF预装重力纯化柱说明书。将纯化收集的蛋白洗脱液进行SDS-PAGE验证蛋白正确性及纯化的质量。
1.2.5 体外酶催化
CAR体外酶催化体系(5 mL)包括50 mmol/L Tris-HCl(pH 7.5)、1 mmol/L NADPH、10 mmol/L MgCl2、5 mmol/L ATP、5 mmol/L DTT、50 μg/mL纯化CAR、1 g/L己二酸。通过GC对催化产物进行分析。
1.2.6 无细胞催化
在30 ℃下利用不同CAR粗酶液进行无细胞催化反应。催化体系(5 mL)包括50 mmol/L Tris-HCl(pH 7.5)、5 mmol/L DTT、OD=20粗酶液、1 g/L己二酸。通过GC对己二醛的合成进行了分析。
1.2.7 样品处理方法
取500 μL发酵液样品后在8 000 r/min下离心10 min,使菌体与上清液充分分离,取300 μL上清液,用900 μL乙醚萃取3次,取上层有机相,55 ℃水浴蒸发完全后加入300 μL乙腈作为溶剂,并使用0.22 μm滤膜过滤后进行气相分析。
1.2.8 分析方法
通过使用Nexis GC-2030型气相色谱仪进行气相检测。色谱柱为SH-Wax毛细管柱(30 m×0.25 mm×0.25 μm,岛津公司)。色谱条件为初始温度80 ℃,保持1 min,以10 ℃/min的升温速率升至230 ℃,并保持10 min。气相载气为高纯氮气,流速1.2 mL/min。进样器温度230 ℃,进样量1 μL。检测器采用火焰离子化检测器(flame ionization detector,FID),检测器温度250 ℃。
2 结果与分析
2.1 己二醛人工合成途径的构建和验证
大肠杆菌BL21(DE3)生长速度快、转化效率高,并且具有成熟的基因操作系统。本研究将来自Mycobacteroides abscessus的羧酸还原酶MAB CAR(GenBank ID:WP_005082584)在大肠杆菌中进行异源表达,初步构建了己二醛生物合成工程菌株。如图2所示,己二酸首先被还原为己酸半醛,然后第2个羧基也被还原为醛基从而得到己二醛。
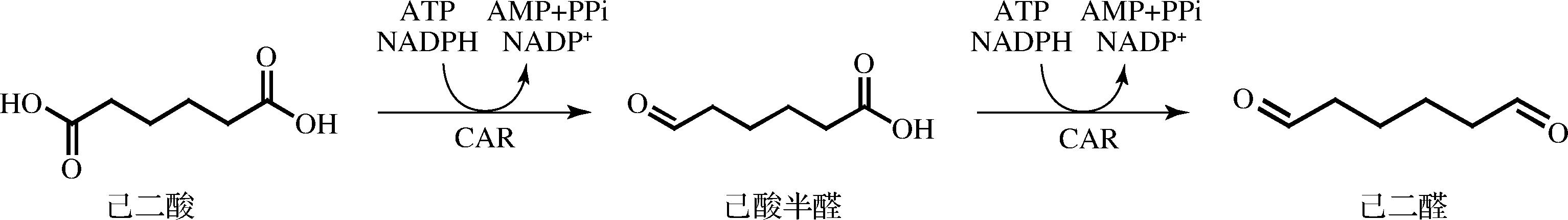
图2 在大肠杆菌中构建己二醛生物合成途径
Fig.2 1,6-Hexanedial biosynthetic pathway constructed in E. coli
为了验证己二醛合成途径的成功构建,利用上述工程菌株进行己二酸催化研究,并进行气质联用分析(图3)。质谱分析(图3-a、图3-b)证实发酵样品与己二醛标准品碎片离子一致。此外,GC分析发现发酵样品和己二醛标准品具有相同的保留时间(图3-c、图3-d)。上述结果证实在大肠杆菌BL21(DE3)中己二醛的成功合成,其产量为8.9 mg/L,为进一步研究己二醛的生物法制备奠定了基础。
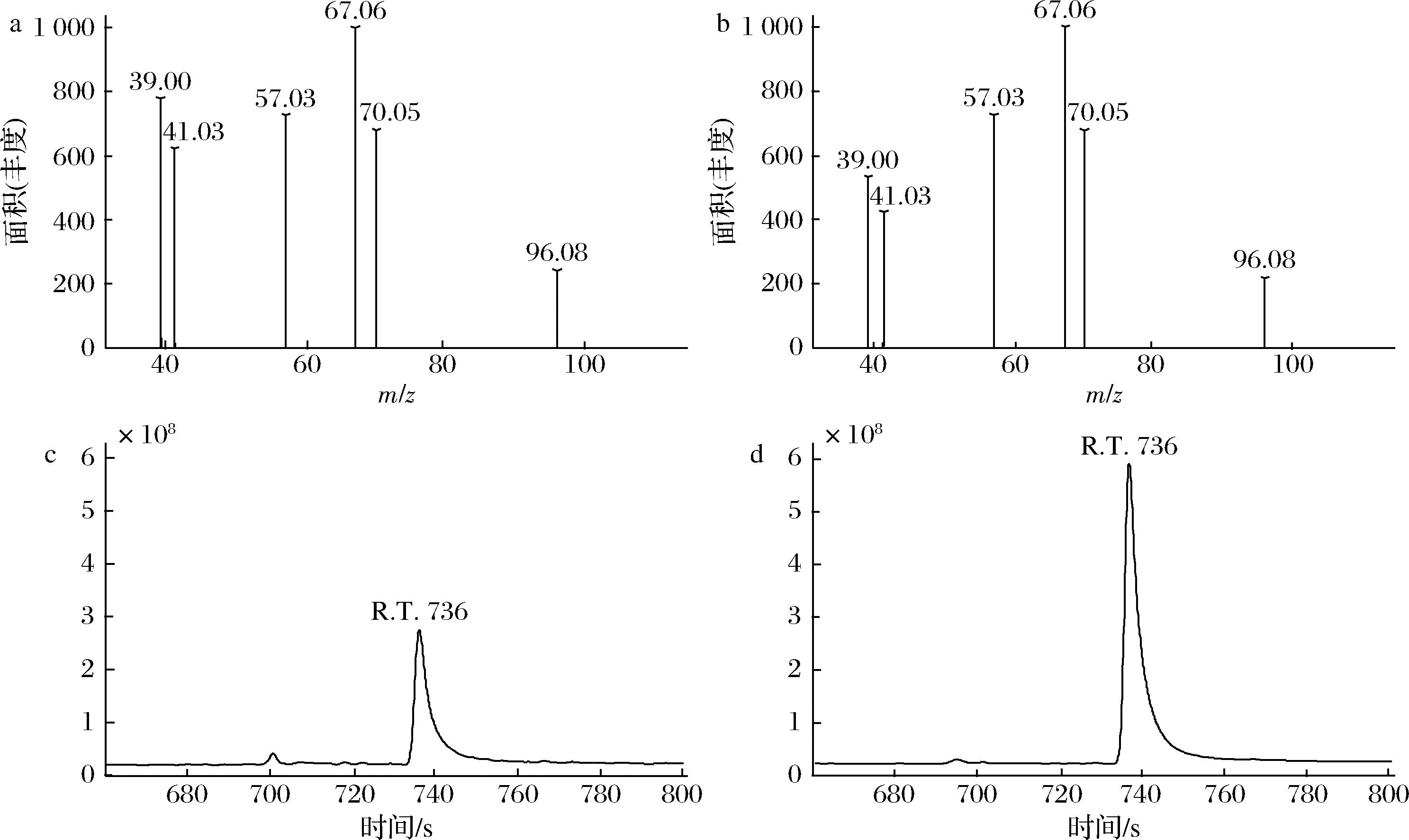
a-发酵样品质谱图;b-己二醛标准品质谱图;c-发酵样品气相图谱;d-己二醛标准品气相图谱
图3 气质联用分析工程菌株发酵产物
Fig.3 GC-MS analysis of fermentation products of the engineered strain
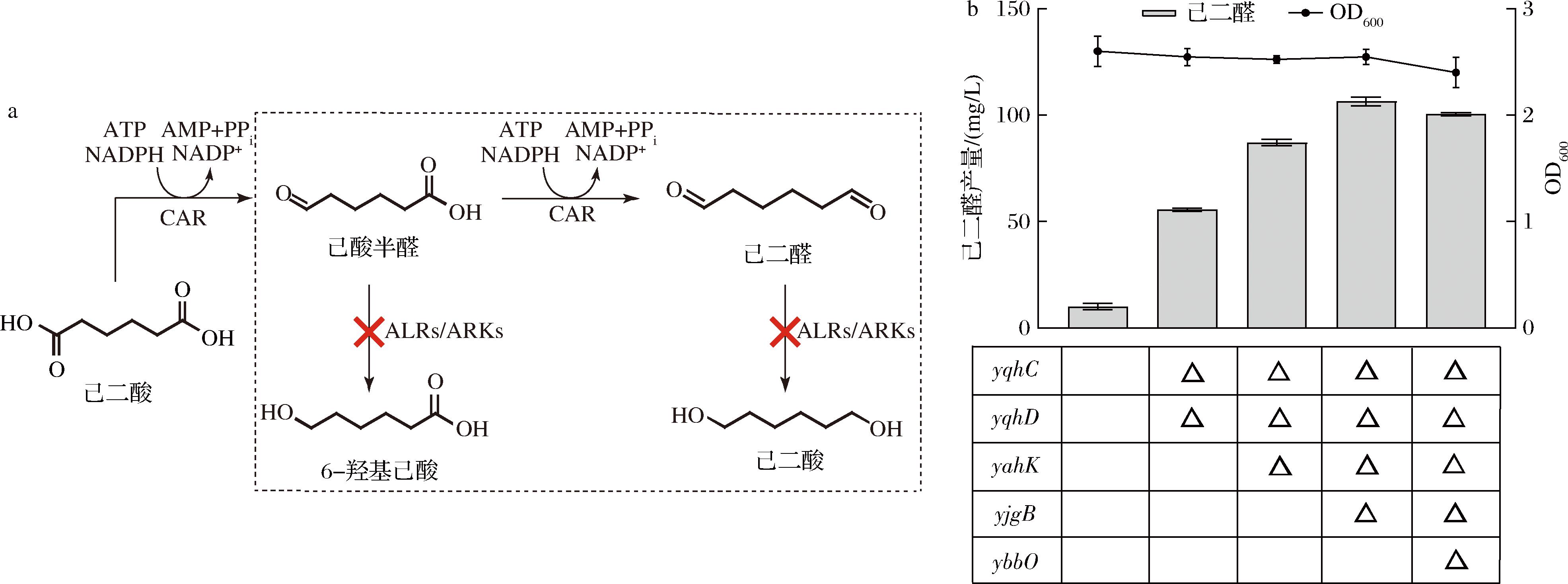
a-己二醛合成副产物;b-基因敲除对己二醛积累和细胞生长的影响
图4 基因敲除对己二醛合成影响
Fig.4 Effect of gene knockout on the synthesis of 1,6-hexanedial
注:“△”表示基因缺失,细胞密度用OD600表示。
2.2 基因敲除强化己二醛合成
大肠杆菌胞内具有高活性的醛还原酶,导致己二醛的胞内合成比单醛更为复杂,其过量还原产物也更具多样性(图4-a)。尽管醛还原酶对单醛的代谢机制已有较多报道,然而其对己二醛的还原潜能及关键作用基因尚未有针对性研究。因此本实验基于醛还原酶对脂肪醛的已知催化活性,敲除对己醛特异性高的yahK、yjgB和ybbO表达基因[1,8,12]及其相关调控靶点yqhC-yqhD[14-15]。
本研究使用己二醛产量表征基因敲除的效果(图4-b),敲除醛代谢调控的关键靶点yqhC和yqhD时发现,双敲除菌株(E.coli BL21ΔyqhCΔyqhD)的己二醛产量较野生型提高5倍至55.5 mg/L,证实己二醛的体内合成受到了内源性醛还原酶的干扰,在此基础上依次敲除yahK和yjgB,己二醛的积累量分别提高到87 mg/L和106.5 mg/L,然而ybbO敲除后己二醛的积累并未进一步增强,这可能是因为YbbO更偏好芳香底物[3]。最终成功获得了适用于己二醛生物合成的底盘细胞,其包含4个基因敲除:yqhC、yqhD、yahK和yjgB,此时己二醛积累量达到106.5 mg/L,比野生菌株提高12倍,且细胞生长情况相比野生型菌株差别不大,该菌株(E.coli BL21 △4)可作为底盘细胞用于后续相关研究。
2.3 CAR同工酶挖掘提高己二醛合成效率
CAR具有十分广泛的底物谱[20],可接受多种脂肪酸作为底物,有研究报道来自海洋分歧杆菌的CAR可催化C4~C18脂肪酸形成相应的醛[21]。此外不同来源的CAR催化效果也大不相同,有研究表明来自Mycobacterium avium subsp.Paratuberculosis的MAV CAR(GenBank ID:WP_003872682)、来自Mycolicibacterium smegmatis MC2155的MSM CAR(GenBank ID:WP_011730755)、来自Mycobacteroides abscessus的MAB CAR(GenBank ID:WP_005082584)和来自Nocardia iowensis的Ni CAR(GenBank ID:OP604544)对己二酸皆具有较高的活性[22]。将上述MAV CAR、MSM CAR、MAB CAR、Ni CAR四种酶在E.coli BL21 △4中分别表达并进行摇瓶发酵(图5-a)。结果发现随着发酵时间的增加,己二醛产量先升高后降低,在24 h达到最高,且4种CAR中MAB CAR效果最好,在5 g/L的己二酸底物下己二醛产量达到110.5 mg/L,其次为Ni CAR,表明在4种CAR中MAB CAR对己二酸具有最好的催化活性。为了研究2种CAR的组合表达效果并与单酶的催化效果进行比较,进一步构建MAB-MSM CAR、MAB-Ni CAR、MAB-MAV CAR双酶表达系统,分别将4种质粒转化至E.coli BL21 △4并在相同的条件下进行摇瓶发酵(图5-b),结果发现双酶的催化效果随时间变化规律与单酶类似,其中最佳的双酶组合是MAB-Ni CAR,己二醛产量较单酶提高至133 mg/L,是原始菌株的14.9倍。
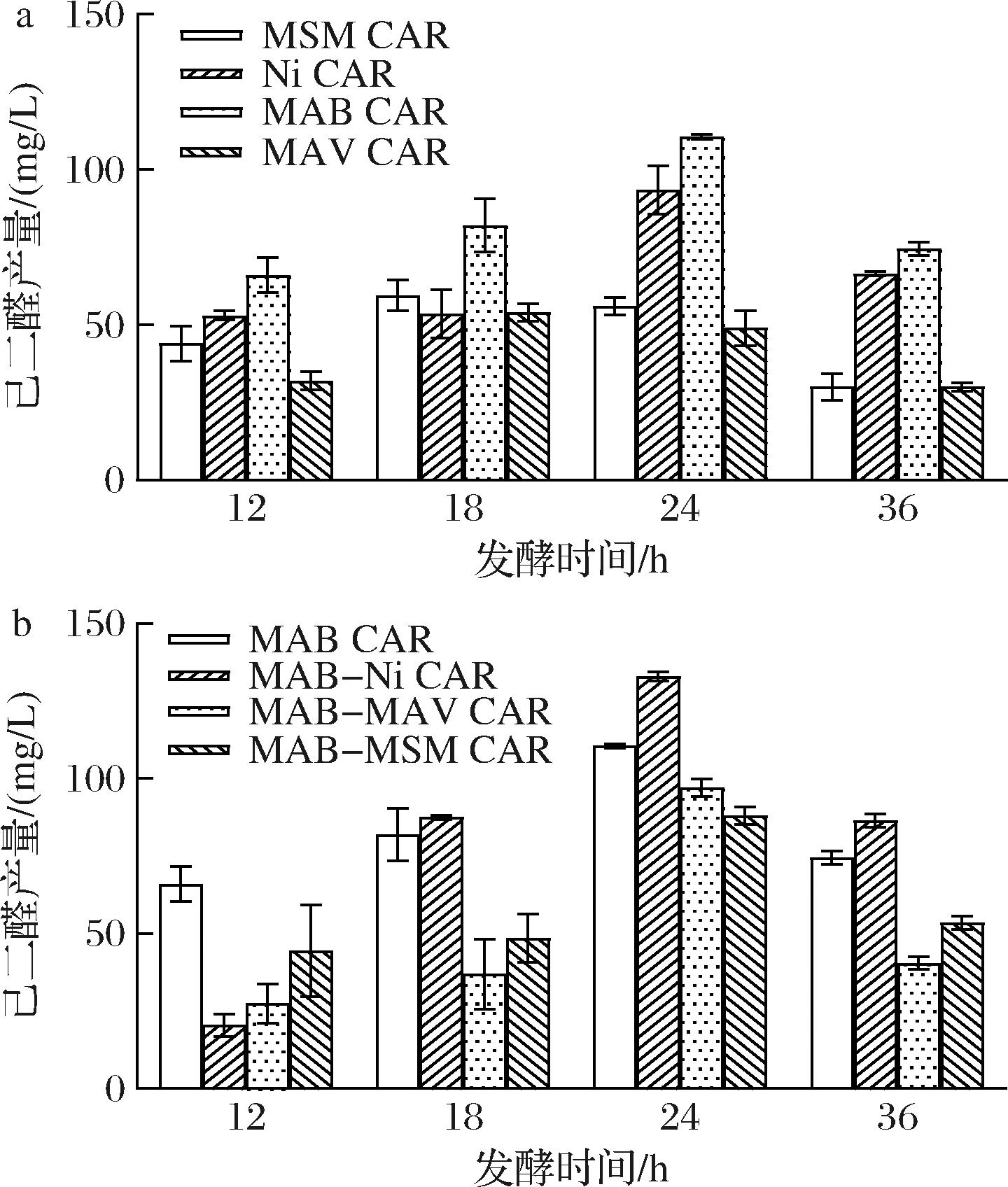
a-带有单个CAR的E.coli BL21 △4菌株;b-带有多种同工酶组合的E.coli BL21 △4菌株
图5 己二醛合成CAR同工酶挖掘
Fig.5 Exploration of CAR isoenzymes for 1,6-hexanedial synthesis
2.4 底物对己二醛合成的影响研究
底物质量浓度是发酵过程中的一个重要影响参数[23]。本研究首先探究了不同的己二酸质量浓度下MAB-Ni CAR的催化效果(图6-a),结果显示较低的底物质量浓度更有利于己二醛的积累,其中1 g/L底物时己二醛产量最高,随着底物质量浓度增加己二醛积累量没有提升,可能是由于底盘细胞的自我保护机制,己二醛的毒性限制了其大量积累。此外,诱导后4 h是加入己二酸的最佳时机(图6-b),己二醛产量最高达到154.2 mg/L,是原始菌株的17.3倍。
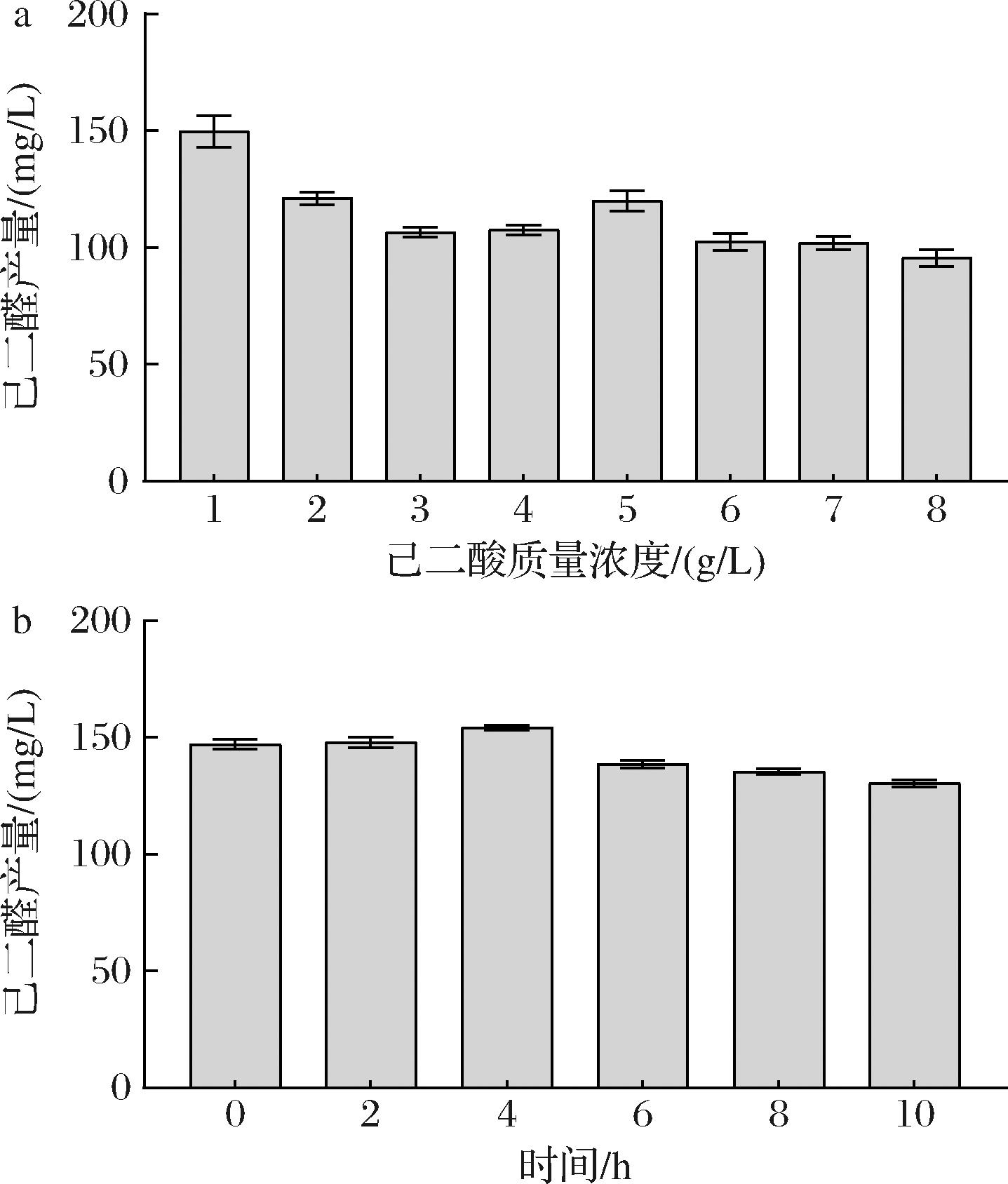
a-不同底物浓度;b-不同底物加入时间
图6 底物对己二醛合成的影响
Fig.6 Influence of substrates on the synthesis of 1,6-hexanedial
2.5 IPTG对己二醛合成的影响研究
IPTG对蛋白表达强度有着重要的调控作用,接下来探究IPTG对MAB-Ni CAR催化的影响(图7),总体来说,加入IPTG时的细胞密度对蛋白表达影响较小(图7-a),OD600为0.8时产生的微弱优势可能是因为大肠杆菌进入对数生长期,菌体生长旺盛,此时开始的蛋白表达和菌体生长速率达到了较好的平衡。同时也比较了不同IPTG浓度对己二醛发酵的影响(图7-b),结果发现IPTG浓度为0.4 mmol/L时效果最好,而更高的IPTG浓度反而对己二醛积累产生了负面作用,可能是高浓度的IPTG使得细胞生长受限[23]进而影响发酵效能。考虑到己二醛对细胞的毒害,为进一步提高己二醛产量,采用先收集菌体后进行催化的方法开展研究,将菌体培养不同时间后收集菌体进行催化(图7-c),结果显示,由于蛋白表达时间过短,己二醛产量在培养时间为4 h时明显较低,并随着培养时间的增加逐步上升,在12 h时达到最高,而后呈现略微下降趋势,表明诱导后的最佳培养时间为12 h,此时获得了178.3 mg/L的己二醛积累,是原始菌株的20倍。
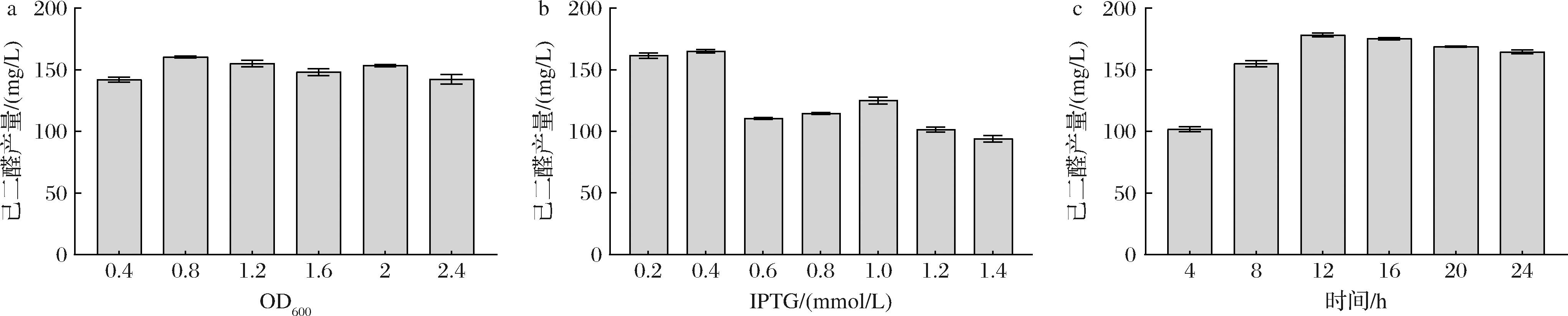
a-不同细胞密度下添加IPTG;b-不同IPTG浓度;c-诱导后不同细胞培养时间
图7 IPTG对己二醛合成影响的研究
Fig.7 Investigation of the effect of IPTG on 1,6-hexanedial synthesis
2.6 己二醛高产菌株的稳定性研究
由于醛类的毒性作用,因此开展了工程菌株的稳定性测试。将pRSF-MAB-Ni导入E.coli BL21 △4,按上述最佳条件在培养过程中直接添加己二酸,结果发现不同转化子之间的己二醛产量存在较大差异(图8-a),其最高产量约为190 mg/L,然而超过80%的转化子产量均低于100 mg/L。另外,观察细胞密度可以发现,携带MAB-Ni CAR的E.coli BL21 △4在不添加己二酸的情况下生长情况更好。将高产菌株作为第二轮筛选的起始菌株,但二次发酵时的己二醛产量均显著降低(图8-b)。此外,添加己二酸的E.coli BL21 △4与对照组表现出相似的生长情况(图8-c),从而证实了己二醛的毒性对工程菌株产生了显著影响[24]。
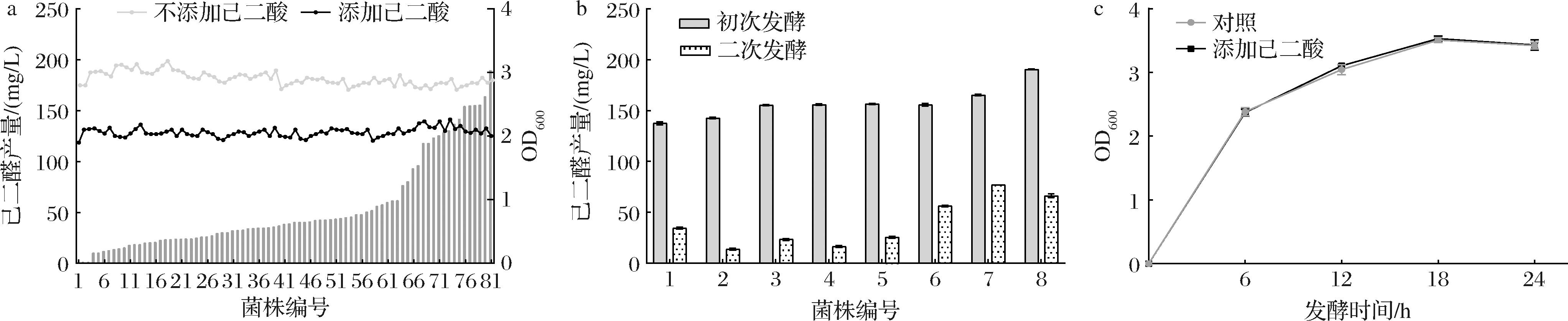
a-81株工程菌株的己二醛催化合成;b-己二醛高产菌株的二次发酵;c-己二酸对菌株生长的影响
图8 己二醛高产菌株的稳定性研究
Fig.8 Investigation of the stability of 1,6-hexanedial-producing strains
2.7 无细胞体系中己二醛的催化合成
在上述研究中,底盘细胞的保护机制可能显著限制了己二醛的积累,因此本研究将不同的CAR纯化后进行体外酶催化,结果表明(图9-a),MAB CAR的催化效果最好,己二醛产量提高至235 mg/L,是全细胞催化己二醛产量的1.32倍,原因可能是体外催化可以在一定程度上减轻己二醛对底盘细胞和酶等生物大分子的毒害。
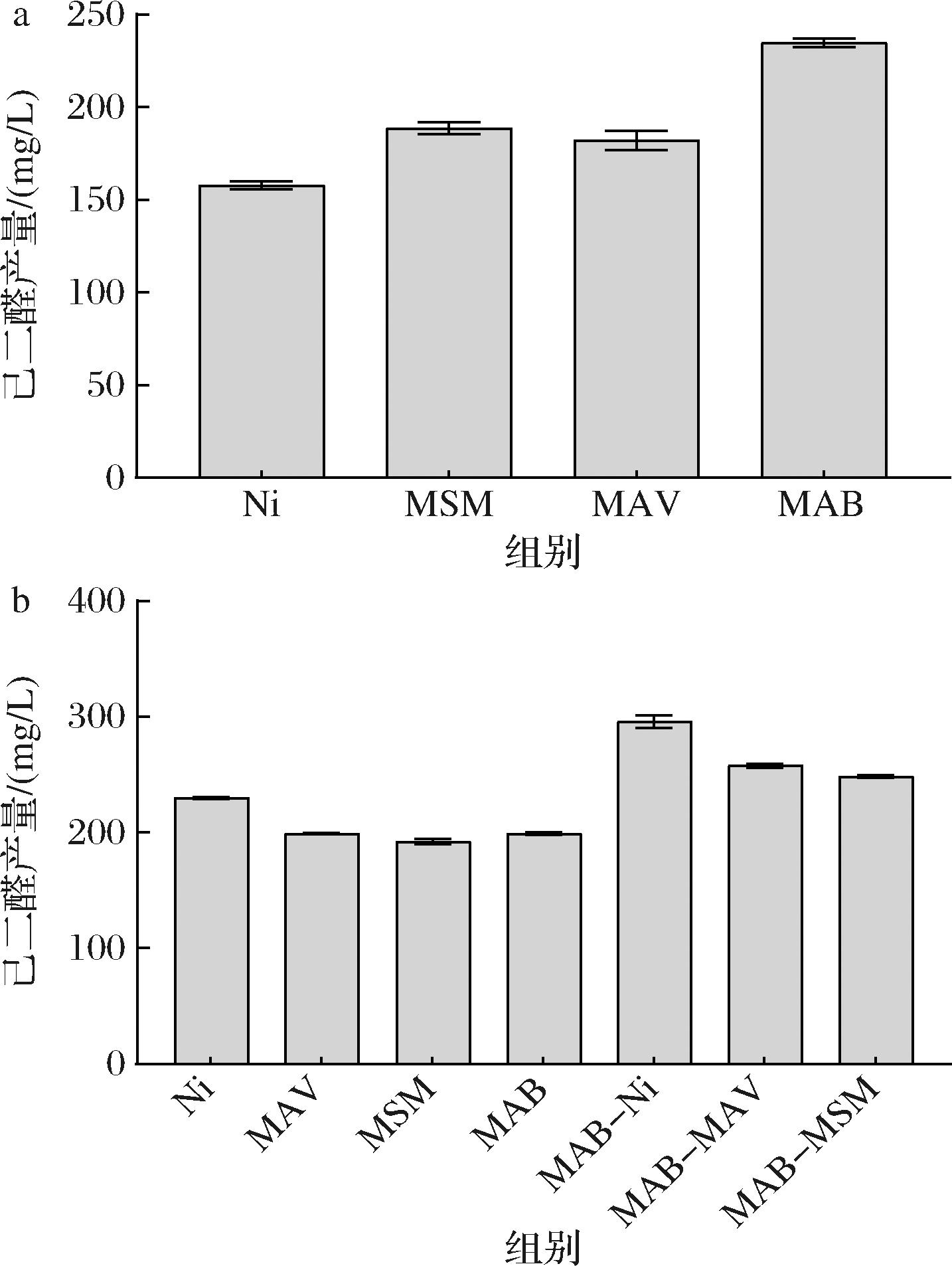
a-体外酶催化;b-粗酶液无细胞系统
图9 无细胞体系中己二醛的催化合成
Fig.9 Catalysis of 1,6-hexanedial in a cell-free system
尽管体外酶催化增强了己二醛的积累,但蛋白纯化这一工艺较为复杂。因此本研究采用无细胞催化体系,利用MAB-Ni CAR在E.coli BL21 △4中表达12 h后的细胞裂解液直接进行催化(图9-b),己二醛的产量提高至295.8 mg/L,是全细胞催化己二醛产量的1.56倍,此时对反应液中残余的己二酸进行检测,发现实际己二酸消耗量仅为0.49 g/L,经计算己二醛的摩尔转化率达到0.77 mol/mol。这一结果表明无细胞催化体系显著提升了己二醛的产量[25],为脂肪族二醛的生物合成研究提供支持和借鉴。
3 结论与讨论
本研究成功在大肠杆菌中构建了己二醛人工合成途径,通过敲除竞争途径副产物积累相关的基因,元件适配组装获得最适羧酸还原酶,结合发酵优化确定了己二醛催化的最适条件,并使用无细胞体系削弱了己二醛的部分毒性作用,最后己二醛产量较初始提高了32.2倍,达到295.8 mg/L,摩尔转化率达到77%。后续可通过开展羧酸还原酶的理性设计来提高催化效率,同时继续敲除冗余基因以进一步减小内源蛋白对己二醛积累的负面影响。本研究填补了己二醛生物合成领域的空白,为后续二醛相关的生物合成研究奠定了基础。
[1] RODRIGUEZ G M,ATSUMI S.Toward aldehyde and alkane production by removing aldehyde reductase activity in Escherichia coli[J].Metabolic Engineering,2014,25:227-237.
[2] TENORIO M J,CHAUDHARI R V,SUBRAMANIAM B.Rh-catalyzed hydroformylation of 1,3-butadiene and pent-4-enal to adipaldehyde in CO2-expanded media[J].Industrial &Engineering Chemistry Research,2019,58(50):22526-22533.
[3] RODRIGUEZ G M,ATSUMI S.Isobutyraldehyde production from Escherichia coli by removing aldehyde reductase activity[J].Microbial Cell Factories,2012,11:90.
[4] KANTER J P,HONOLD P J,LÜKE D,et al.An enzymatic tandem reaction to produce odor-active fatty aldehydes[J].Applied Microbiology and Biotechnology,2022,106(18):6095-6107.
[5] YUE R R,ZHANG Z,SHI Q Q,et al.Identification of the key genes contributing to the LOX-HPL volatile aldehyde biosynthesis pathway in jujube fruit[J].International Journal of Biological Macromolecules,2022,222:285-294.
[6] NAPORA-WIJATA K,STROHMEIER G A,WINKLER M.Biocatalytic reduction of carboxylic acids[J].Biotechnology Journal,2014,9(6):822-843.
[7] DUAN Y T,YAO P Y,CHEN X,et al.Exploring the synthetic applicability of a new carboxylic acid reductase from Segniliparus rotundus DSM 44985[J].Journal of Molecular Catalysis B:Enzymatic,2015,115:1-7.
[8] HORVAT M,WINKLER M.In vivo reduction of medium-to long-chain fatty acids by carboxylic acid reductase (CAR) enzymes:Limitations and solutions[J].ChemCatChem,2020,12(20):5076-5090.
[9] PARK J,LEE H S,OH J,et al.A highly active carboxylic acid reductase from Mycobacterium abscessus for biocatalytic reduction of vanillic acid to vanillin[J].Biochemical Engineering Journal,2020,161:107683.
[10] BANG H B,SON J,KIM S C,et al.Systematic metabolic engineering of Escherichia coli for the enhanced production of cinnamaldehyde[J].Metabolic Engineering,2023,76:63-74.
[11] KUNJAPUR A M,PRATHER K L J.Microbial engineering for aldehyde synthesis[J].Applied and Environmental Microbiology,2015,81(6):1892-1901.
[12] PICK A,RÜHMANN B,SCHMID J,et al.Novel CAD-like enzymes from Escherichia coli K-12 as additional tools in chemical production[J].Applied Microbiology and Biotechnology,2013,97(13):5815-5824.
[13] JARBOE L R.YqhD:A broad-substrate range aldehyde reductase with various applications in production of biorenewable fuels and chemicals[J].Applied Microbiology and Biotechnology,2011,89(2):249-257.
[14] SULZENBACHER G,ALVAREZ K,VAN DEN HEUVEL R H H,et al.Crystal structure of E.coli alcohol dehydrogenase YqhD:Evidence of a covalently modified NADP coenzyme[J].Journal of Molecular Biology,2004,342(2):489-502.
[15] TURNER P C,MILLER E N,JARBOE L R,et al.YqhC regulates transcription of the adjacent Escherichia coli genes yqhD and dkgA that are involved in furfural tolerance[J].Journal of Industrial Microbiology &Biotechnology,2011,38(3):431-439.
[16] ZALDIVAR J,MARTINEZ A,INGRAM L O.Effect of selected aldehydes on the growth and fermentation of ethanologenic Escherichia coli[J].Biotechnology and Bioengineering,1999,65(1):24-33.
[17] GUÉRAUD F,ATALAY M,BRESGEN N,et al.Chemistry and biochemistry of lipid peroxidation products[J].Free Radical Research,2010,44(10):1098-1124.
[18] KELWICK R J R,WEBB A J,FREEMONT P S.Biological materials:The next frontier for cell-free synthetic biology[J].Frontiers in Bioengineering and Biotechnology,2020,8:399.
[19] JIANG Y,CHEN B,DUAN C L,et al.Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J].Applied and Environmental Microbiology,2015,81(7):2506-2514.
[20] GAHLOTH D,ALEKU G A,LEYS D.Carboxylic acid reductase:Structure and mechanism[J].Journal of Biotechnology,2020,307:107-113.
[21] AKHTAR M K,TURNER N J,JONES P R.Carboxylic acid reductase is a versatile enzyme for the conversion of fatty acids into fuels and chemical commodities[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(1):87-92.
[22] VENKITASUBRAMANIAN P,DANIELS L,ROSAZZA J P N.Reduction of carboxylic acids by Nocardia aldehyde oxidoreductase requires a phosphopantetheinylated enzyme[J].Journal of Biological Chemistry,2007,282(1):478-485.
[23] MALAKAR P,VENKATESH K V.Effect of substrate and IPTG concentrations on the burden to growth of Escherichia coli on glycerol due to the expression of Lac proteins[J].Applied Microbiology and Biotechnology,2012,93(6):2543-2549.
[24] LOPACHIN R M,GAVIN T.Molecular mechanisms of aldehyde toxicity:A chemical perspective[J].Chemical Research in Toxicology,2014,27(7):1081-1091.
[25] KUNJAPUR A M,CERVANTES B,PRATHER K L J.Coupling carboxylic acid reductase to inorganic pyrophosphatase enhances cell-free in vitro aldehyde biosynthesis[J].Biochemical Engineering Journal,2016,109:19-27.