磷脂酶D(phospholipase D,PLD;EC 3.1.4.4)是磷脂酶超家族中一类关键功能酶,通过特异性水解磷脂分子中的磷酸二酯键参与两类重要反应:1)水解反应生成磷脂酸和胆碱;2)转磷脂酰反应合成功能性磷脂[如磷脂酰丝氨酸(phosphatidylserine,PS)、磷脂酰乙醇胺及磷脂酰甘油][1]。这些产物在食品添加剂、药物递送系统和生物材料合成领域具有广泛应用[2]。链霉菌来源的PLD因其宽泛的底物谱和高转磷脂酰活性而备受关注[3]。例如源自Streptomyces racemochromogenes 10-3的PLD在单相水体系中可实现82%~97%的PS转化率[4]。然而,野生菌株的低产酶量和长发酵周期限制了PLD的广泛应用。随着分子生物学和基因工程技术的快速发展,构建高产PLD的工程菌株成为突破这一瓶颈的关键。
目前,多种表达系统已被开发用于实现PLD的高水平生产,包括大肠杆菌(Escherichia coli)[5]、枯草芽孢杆菌(Bacillus subtilis)[6]、变铅青链霉菌(Streptomyces lividans)[7]、毕赤酵母(Pichia pastoris)[8]和短小芽孢杆菌(Brevibacillus choshinensis)[9]。尽管如此,高效表达的宿主菌株数量仍然有限。其中,大肠杆菌是最常用的宿主菌株,但内毒素的存在限制了其在食品工业中的应用[10]。相比之下,链霉菌被认为是异源生产重组蛋白的理想宿主,这主要得益于其成熟的蛋白质分泌系统、较低的胞外水解酶活性[11]、无内毒素脂多糖以及高效表达高G+C基因的能力[12]。然而,目前链霉菌表达宿主主要集中在模式菌株S.lividans,但该菌株仍存在表达量低、依赖抗性标记等问题[13]。因此,探索更多适合表达PLD的链霉菌宿主显得尤为重要。
Streptomyces mobaraensis作为谷氨酰胺转氨酶(transglutaminase,TGase)的工业生产菌株,具有良好的生物安全性和强大的蛋白分泌能力[14]。自1989年首次被分离用于TGase工业生产以来,经过长期的诱变选育,其胞外TGase产量已提高12.2倍,蛋白分泌能力显著提高[15]。此外,S.mobaraensis的工业规模发酵工艺已十分成熟,并且可以利用价格低廉的鱼粉等水产和农业副产品作为发酵原料。近年来,基因编辑在S.mobaraensis中取得了重要突破,为其作为重组蛋白生产的宿主菌提供了新的可能性。因此,S.mobaraensis有望成为高产PLD的理想宿主。
本研究基于CRISPR-Cas9技术在S.mobaraensis中表达了来源于S.racemochromogenes 10-3的PLD,并通过启动子优化和双位点整合的策略显著提高了其表达水平。此外,对重组PLD进行了纯化及酶学性质分析,旨在为PLD的工业化生产和应用提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
E.coli DH5α用于质粒构建。S.mobaraensis DSM40587用于PLD的重组表达[15]。pET28a(+)用于基因克隆。质粒pCRISPomyces-2用于链霉菌目的基因的定点整合[16]。pET28a(+)-pld编码S.racemochromogenes 10-3来源的PLD基因(GenBank登录号:AB573232)。pET28a(+)-promoters编码启动子Pgapdh、PrpsL、PermE*和PkasO*[17]。PLD基因和启子基因均由苏州金唯智公司合成并克隆至pET28a(+)。
1.1.2 试剂
同源重组一步克隆试剂盒,北京全式金生物技术有限公司;限制性内切酶、蛋白预制胶、蛋白质Marker、MES蛋白电泳缓冲液等,赛默飞世尔科技有限公司;安普霉素、氯化胆碱、4-氨基安替吡啉等化学试剂、质粒抽提试剂盒、DNA产物纯化试剂盒,上海生工生物工程技术服务有限公司;大豆卵磷脂,上海阿拉丁生化科技股份有限公司;胆碱氧化酶、Bradford蛋白检测试剂盒,上海碧云天生物技术股份有限公司;过氧化物酶,上海源叶生物科技有限公司。其他常规试剂及药品均为国产或进口分装产品。
1.1.3 培养基
LB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10(固体培养基添加20 g/L琼脂粉)。
GYM(glucose yeast malt)琼脂培养基(g/L):葡萄糖10,酵母粉4,麦芽粉3,琼脂粉20。
2×YT(yeast extract-tryptone)(g/L):蛋白胨16,酵母粉10,NaCl 5。
种子培养基(g/L):甘油20,酵母粉5,胰蛋白胨20,K2HPO4 4,MgSO4 2,pH 7.2。
发酵培养基(g/L):甘油20,鱼粉蛋白胨25,酵母粉5,玉米浆粉5.5,(NH4)2SO4 5.5,K2HPO4 2,MgSO4 2,pH 7.2~7.4。
1.2 实验方法
1.2.1 链霉菌整合表达质粒的构建
为在S.mobaraensis的TGase基因位点整合表达PLD,基于CRISPOR(https://crispor.gi.ucsc.edu/)设计相应的sgRNA序列,并合成对应的引物对sgtg-F/sgtg-R。通过单链退火,引物对sgtg-F/sgtg-R形成带有黏性末端的双链sgRNA片段(sgtg)。将该双链sgRNA片段与经限制性内切酶Bbs I切割后的pCRISPomyces-2连接,构建得到含有sgRNA的质粒pCRISPR-sgtg。以S.mobaraensis基因组为模板,使用引物对tg-FF/tg-FR和tg-RF/tg-RR分别扩增获得TGase基因的上游L1000(1 kb)和下游同源臂R1000(1 kb)。同时,以质粒pET28a(+)-pld为模板,使用引物对Sr-F/Sr-R扩增获得C-端融合His标签的PLD基因片段。通过一步克隆试剂盒,将L1000、R1000和PLD基因片段与经Xba I切割后的pCRISPR-sgtg连接,构建得到PLD在TGase基因位点整合表达质粒pCRISPR-sgtg-PgStg-pld(图1-a)。
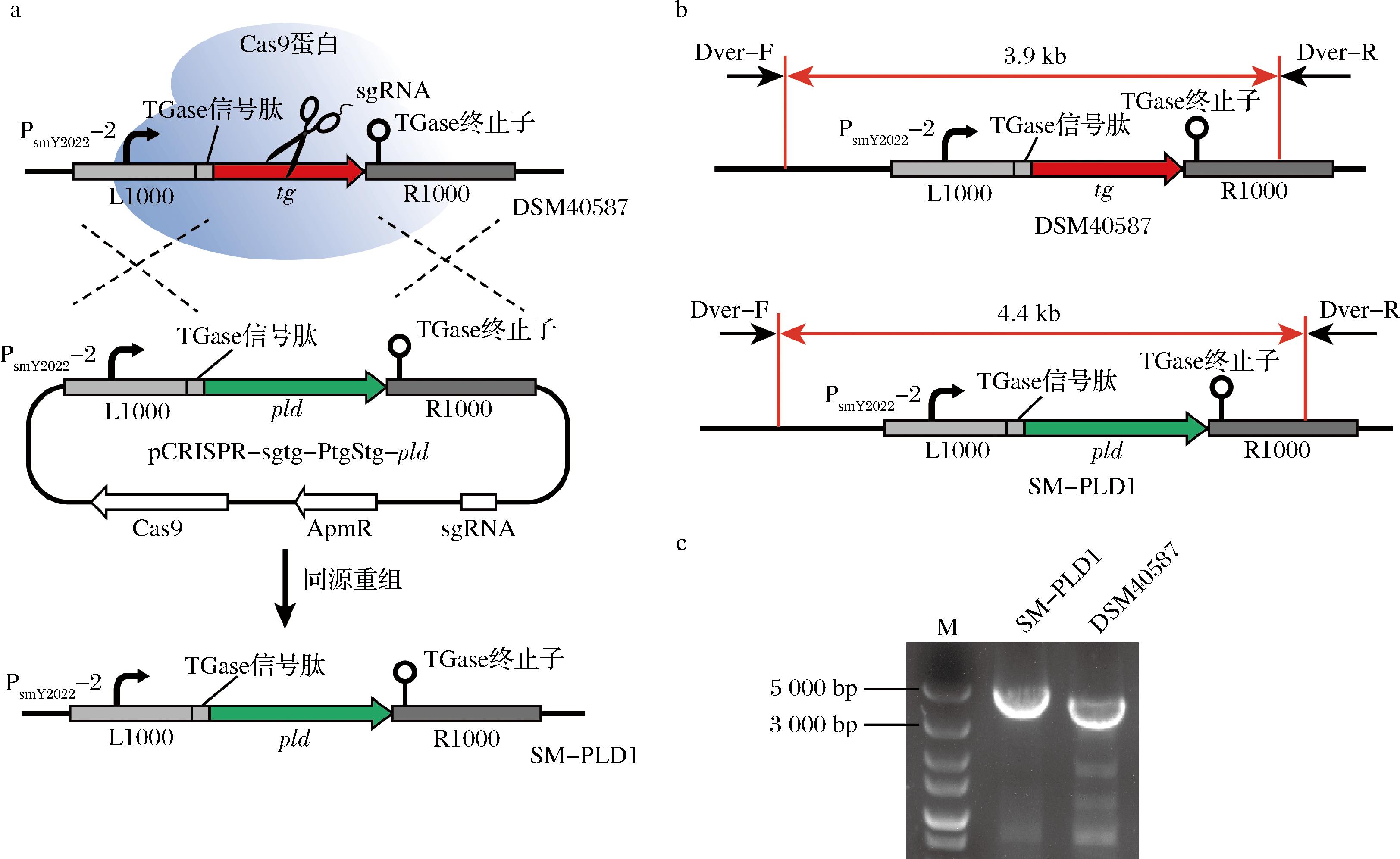
a-基于CRISPR-Cas9的PLD整合原理;b-PCR扩增范围示意图;c-PCR产物的凝胶电泳分析
图1 重组菌株SM-PLD1的构建
Fig.1 Construction of recombinant strain SM-PLD1
为构建受不同启动子驱动的PLD整合表达质粒,以引物对gapdhp-F/gapdhp-R、ermep-F/ermep-R、rpslp-F/rpslp-R和kasop-F/kasop-R分别从pET28a(+)-promoters扩增得到启动子Pgapdh、PermE*、Prpsl和PkasO*的基因片段。以引物对signal-RF/signal-RR从pCRISPR-sgtg-PgStg-pld扩增获得包含PLD及TGase下游同源臂的基因片段WR1000(3 kb)。使用引物对signal-FF/signal-FR从S.mobaraensis基因组扩增获得不包含启动子PsmY2022-2的TGase上游同源臂WL1000(1 kb)。通过一步克隆试剂盒,将Pgapdh、PrpsL、PermE*和PkasO*的基因片段分别与WR1000、WL1000及经Xba I切割的质粒pCRISPR-sgtg连接,分别得到对应的PLD整合表达质粒pCRISPR-sgtg-PgStg-pld、pCRISPR-sgtg-PeStg-pld、pCRISPR-sgtg-PrStg-pld和pCRISPR-sgtg-PkStg-pld。
为在S.mobaraensis壳聚糖酶(chitosanase,Csn)基因位点整合表达PLD,基于CRISPOR设计相应的sgRNA序列,并合成对应的引物对sgcsn-F/sgcsn-R。通过单链退火,sgcsn-F/sgcsn-R形成带有黏性末端的双链sgRNA片段(sgcsn)。将该双链sgRNA片段与经限制性内切酶Bbs I切割后的pCRISPomyces-2连接,获得pCRISPR-sgcsn。利用引物对Whole-F3/Whole-R3从pCRISPR-sgtg-PgStg-pld扩增获得到PLD表达框。以引物对csn-FF/csn-FR和csn-RF/csn-RR从S.mobaraensis基因组扩增Csn基因上游CL1000(1 kb)和下游同源臂CR1000(1 kb)。将PLD表达框、CL1000和CR1000与经Xba I切割后pCRISPR-sgcsn连接,获得在Csn基因位点整合表达PLD基因的质粒pCRISPR-sgcsn-PgStg-pld。上述质粒构建过程中所涉及引物序列如表1所示。
表1 本研究所使用引物序列
Table 1 Primers used in this study

名称序列 5′-3′ sgtg-FACGCAATTTCGACTACGGCTGGTTsgtg-RAAACAACCAGCCGTAGTCGAAATTsgcsn-FACGCCGCCTGGAAACGGGCCAAGTsgcsn-RAAACACTTGGCCCGTTTCCAGGCGSr-FATGGTGATGGTGATGGTGGGCTTGGCACAGGCCGCGGGCGTAGSr-RCCGTCCTTCCGGGCCCCCGCGTCCCCGACCCCCCACCTGGATTCGtg-RFCGGGCGTTTTTTATCTAGCACGTACAGGTACGACGGTGGTGGCtg-RRCACCATCACCATCACCATTGATGTAAGCGGGGAGGGGAGGGGAGtg-FFGGGGGCCCGGAAGGACGGACCGGCGtg-FRACGGTTCCTGGCCTCTAGCCCCGTGGCCCTCGGCAGGCCACGTCsignal-FFACGGTTCCTGGCCTCTAGCCACATCGACCGCGACGTCGACTACsignal-FRGGCTCCAAAACCGGCCACTCCGTCAsignal-RFCGGGCGTTTTTTATCTAGCACGTACAGGTACGACGGTGGTGGCsignal-RRCCCGATCACTCGTCCGGGAGTCGAGgapdhp-FGTGGCCGGTTTTGGAGCCGCTGCTCCTTCGGTCGGACGTGCGTgapdhp-RCCCGGACGAGTGATCGGGGCGTATCCCCTTTCAGATACTCGCACTAAGermep-FCCCGGACGAGTGATCGGGGTGGTGTCCTACCAACCGGCACGATTGTCCermeP-RGTGGCCGGTTTTGGAGCCGGTACCAGCCCGACCCGAGCACrpslp-FCCCGGACGAGTGATCGGGTACGTCTCCGTCGTCTTCTCGAGCTCACGGrpslp-RGTGGCCGGTTTTGGAGCCGCCCTGCAGGCGGAAGTCAGGTAGAkasop-RGTGGCCGGTTTTGGAGCCTGTTCACATTCGAACCGTCTCTGCTkaosp-FCCCGGACGAGTGATCGGGAACTCCCCCAGTCCTGCACGCTGTCDver-FGGTCTCCGGGATTTGTTCGCATGCCDver-RCATCCTCATGTTCGAACCCACGGGCWhole-F3GCTTGGGCTGCAGGTCGACTCTAGAWhole-R3GATTACGAATTCGATATCGCGCGCGGcsn-FFTCGACCTGCAGCCCAAGCACGGAACTCCGTTCCTGATAAGGCGcsn-FRCGGGCGTTTTTTATCTAGCGAGAACCACGCGTTGCGTTCGTCcsn-RFGATATCGAATTCGTAATCGTACCGCATCGACGAGTAGCGCGTCcsn-RRACGGTTCCTGGCCTCTAGCCGGAGCTGACCTGGTCCGACCTGDver-csn-FGGGAAGCAGCAGCACAAATGGCACGADver-csn-RGTCCTCGGCCAGGAACACATGGCCG
1.2.2 S.mobaraensis外源DNA转化
取70 μL的S.mobaraensis原生质体溶液,与7 μL质粒混合后,转移至0.1 cm电转杯(Bio-Rad,美国)。在2 500 V电压和25 Ω电阻的条件下进行1次电击处理。电击完成后,立即加入1 mL预冷的2×YT培养基,将混合液转移至无菌的1.5 mL EP管中,置于30 ℃、220 r/min的摇床中培养3 h。随后,将培养液均匀涂布于含有100 μg/mL安普霉素的GYM平板上培养7~9 d。通过PCR验证和基因测序分析筛选出阳性转化子。其中,引物对Dver-F/Dver-R用于验证在S.mobaraensis tg基因位点整合PLD基因的转化子,引物对Dver-csn-F/Dver-csn-R用于验证在S.mobaraensis csn基因位点整合PLD基因的菌株。引物序列如表1所示。
1.2.3 PLD在S.mobaraensis中的表达及纯化
将S.mobaraensis阳性转化子接种于GYM平板上,于30 ℃培养5~7 d后,刮取孢子和菌丝,接种至含有30 mL种子培养基的250 mL摇瓶中,在30 ℃、220 r/min条件下培养24 h。随后,以8%(体积分数)的接种量将种子液接种至含有30 mL发酵培养基的250 mL摇瓶中,置于30 ℃、220 r/min摇床培养,每隔12 h取样一次,共计培养96 h。取样后,将样品于4 ℃、10 000 r/min离心10 min,收集上清液用于后续分析。
PLD的纯化采用镍柱亲和层析法。将-20 ℃预冷的无水乙醇加入发酵上清液中,调整乙醇终浓度至体积分数70%,于4 ℃保存30 min。4 ℃、4 000 r/min离心10 min,收集沉淀并用纯化缓冲液A液(50 mmol/L磷酸缓冲液,150 mmol/L NaCl,pH 7.4)重悬。将上述溶液经0.2 μm滤膜过滤后,利用镍柱PrePack Ni-NTA Purose 6 FF(嘉兴千纯生物科技有限公司)进行纯化。纯化程序如下:先用体积分数10%纯化缓冲液B液(50 mmol/L磷酸缓冲液,150 mmol/L NaCl,500 mmol/L咪唑,pH 7.4)去除杂蛋白,再用体积分数50%纯化缓冲液B液洗脱目的蛋白。
1.2.4 PLD酶学性质测定
将酶液稀释至0.1~0.2 mg/mL后进行后续酶学性质分析。在不同温度(30~80 ℃)、pH 7.5条件下测定酶的水解活性,以最高酶活力为100%计算相对酶活力,确定酶的最适温度;将酶液置于不同温度(30~60 ℃)、pH 7.5条件下保存,分别在1、2、3、4 h取样测定水解活性,以保温0 h的酶活力为100%计算相对酶活力,以评估酶的温度稳定性;配制不同pH值的底物缓冲液(pH 4~6:乙酸-乙酸钠缓冲液;pH 6~7:MES缓冲液;pH 7~10:Tris-HCl缓冲液;以上缓冲液浓度均为200 mmol/L),在不同pH、50 ℃条件下测定酶的水解活性,以最高酶活力为100%计算相对酶活力,以确定酶的最适pH。
1.2.5 PLD催化PS合成条件及检测
本研究采用双相体系合成PS,具体步骤如下:有机相由4 mL质量浓度为5 mg/mL磷脂酰胆碱(phosphatidylcholine,PC)的2-甲基四氢呋喃溶液组成;水相为1 mL Tris-HCl缓冲液(pH 7.5),其中含有40 mg/mL L-丝氨酸和60 U PLD。反应体系在40 ℃、300 r/min条件下反应12 h。反应结束后,将反应液于4 ℃、7 000 r/min离心10 min,取上层有机相,稀释至适宜浓度后,注入10 μL样品至色谱柱,利用HPLC进行PS定量分析。在绘制PS标准曲线的过程中,先将PS标准品溶于2-甲基四氢呋喃,配制成质量浓度为1 g/L的母液,再梯度稀释为0.1、0.2、0.4、0.6、0.8、1.0 g/L系列标准溶液后进样分析。PS的合成转化率以反应后生成的PS与初始投入PC物质的量比表示。
HPLC检测采用岛津LC-20A型液相色谱仪(配备紫外检测器),色谱柱为安捷伦ZORBAX Rx-SIL(4.6 mm×150 mm,5 μm)。流动相由乙腈、甲醇和85%磷酸溶液按体积比100∶10∶0.8配制,使用前经超声脱气处理。色谱条件:流速1.0 mL/min,柱温30 ℃,检测波长205 nm。
1.2.6 PLD水解活性的测定
PLD的水解活性采用酶联比色法进行测定。具体步骤如下:将150 μL底物溶液与150 μL底物缓冲液(200 mmol/L Tris-HCl,pH 7.5,5 g/L TritonX-100,10 mmol/L CaCl2)混合后,于50 ℃预热5 min;随后加入100 μL稀释至合适浓度的酶液,继续在50 ℃反应10 min;反应结束后,立即加入200 μL反应终止液,并置于100 ℃金属浴中保温15 min以完全终止反应;待反应液冷却至室温后,依次加入200 μL显色液(0.1 mol/L Tris-HCl,pH 8.0,5 mmol/L 4-ATT,8 mmol/L苯酚)、1 U过氧化物酶、1 U胆碱氧化酶,于37 ℃显色60 min后,在500 nm处测量吸光值。在50 ℃、pH 7.5的条件下,酶每分钟催化水解大豆卵磷脂并释放1 μmol胆碱所需的酶量定义为一个酶活力单位。
1.2.7 蛋白质检测
采用Bradford蛋白检测试剂盒测定酶液蛋白浓度。对于SDS-PAGE分析,将30 μL酶液与10 μL 4×loading buffer混合均匀后,于95 ℃加热10 min进行变性处理。依据ThermoFisher公司Bis-Tris预制凝胶说明书进行蛋白分离检测。
2 结果与分析
2.1 产PLD重组S.mobaraensis的构建
来源于S.racemochromogenes 10-3的PLD凭借其高转磷脂酰活性受到研究者的广泛关注[4]。然而,野生型菌株的胞外酶活力仅有0.37 U/mL[4]。尽管S.racemochromogenes PLD基因已经在E.coli、B.subtilis和S.lividans中表达,但最高酶活力仅为30 U/mL(S.lividans)[18]。为实现PLD高效微生物合成,本研究基于CRISPR-Cas9基因编辑系统将S.racemochromogenes PLD基因整合至S.mobaraensis DSM40587的TGase基因位点,构建产PLD重组SM-PLD1(图1-a)。通过PCR扩增和基因测序筛选整合PLD基因的重组菌转化子(图1-b、图1-c)。值得注意的是,在SM-PLD1基因组中,仅有TGase编码基因被PLD基因替换,因此后者的表达受TGase启动子PsmY2022-2及信号肽调控[19](图1-a)。
对SM-PLD1进行摇瓶发酵,并对其胞外上清液进行PLD酶活力检测及SDS-PAGE分析。如图2-a所示,SM-PLD1发酵至36 h,胞外PLD酶活力即达到最大值131.20 U/mL,且发酵至72 h酶活力才出现下降。SDS-PAGE分析显示,发酵12 h即在上清液中出现明显的PLD蛋白条带(理论分子质量:54.9 kDa),并随着发酵时间增加而加粗(图2-b)。在同样的发酵条件下,S.mobaraensis TGase基因缺失菌株(Δtg)发酵上清液中未能检测到PLD酶活力(结果未显示),且未在36 h检测到PLD蛋白条带(图2-b)。上述结果表明,S.racemochromogenes PLD在S.mobaraensis成功实现了PLD的高效表达,且明显高于已报道重组菌的最高水平。
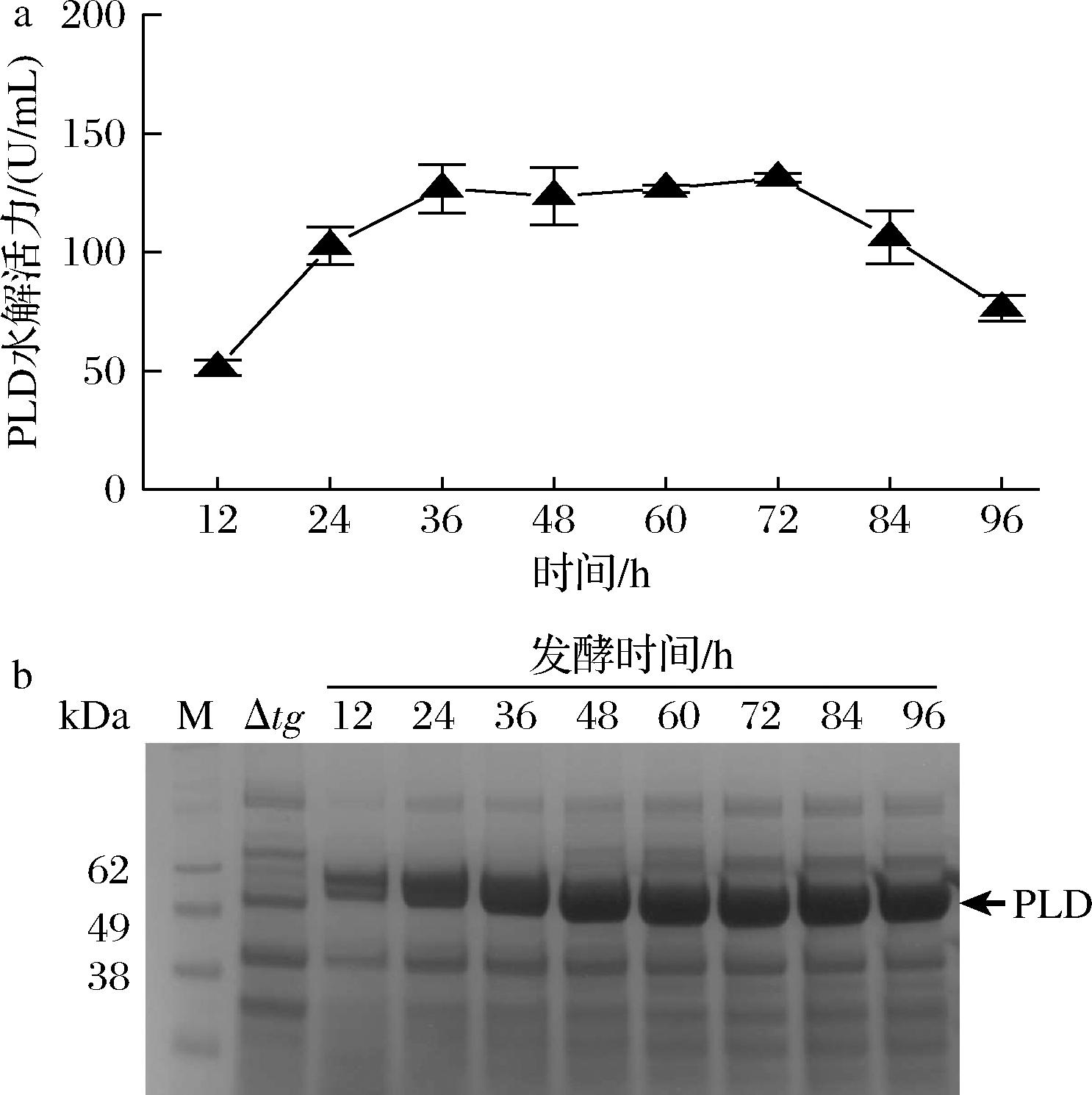
a-发酵上清液水解活性曲线;b-发酵上清液SDS-PAGE分析
图2 重组菌株SM-PLD1发酵过程分析
Fig.2 Fermentation analysis of the recombinant strain SM-PLD1
2.2 启动子优化提高PLD的表达
启动子强度是影响基因表达水平最关键的因素之一[17]。目前,用于链霉菌基因表达的常见强启动子主要包括:Pgapdh(甘油醛-3-磷酸脱氢酶启动子)、PrpsL(30S核糖体蛋白S12启动子)、PermE*(红霉素抗性基因启动子)和PkasO*(SARP型调节因子启动子)等。为进一步提高PLD在S.mobaraensis中的表达水平,基于CRISPR-Cas9基因编辑系统将SM-PLD1中表达PLD的启动子PsmY2022-2分别替换为Pgapdh、PrpsL、PermE*和PkasO*(图3-a)。对重组菌株进行摇瓶发酵,并取发酵至36 h的胞外上清液进行PLD酶活力检测及SDS-PAGE分析。采用Pgapdh的重组菌株(SM-PLD2)的发酵上清液的PLD酶活力最高达到194.29 U/mL,较采用PsmY2022-2的SM-PLD1提高48%(图3-b)。然而,PrpsL对应的重组菌株的胞外PLD活性明显低于PsmY2022-2(图3-b),此外采用PermE*和PkasO*的重组菌株几乎检测不到PLD水解活性(结果未显示)。因此,Pgapdh是上述启动子中最适于PLD在S.mobaraensis表达的启动子。
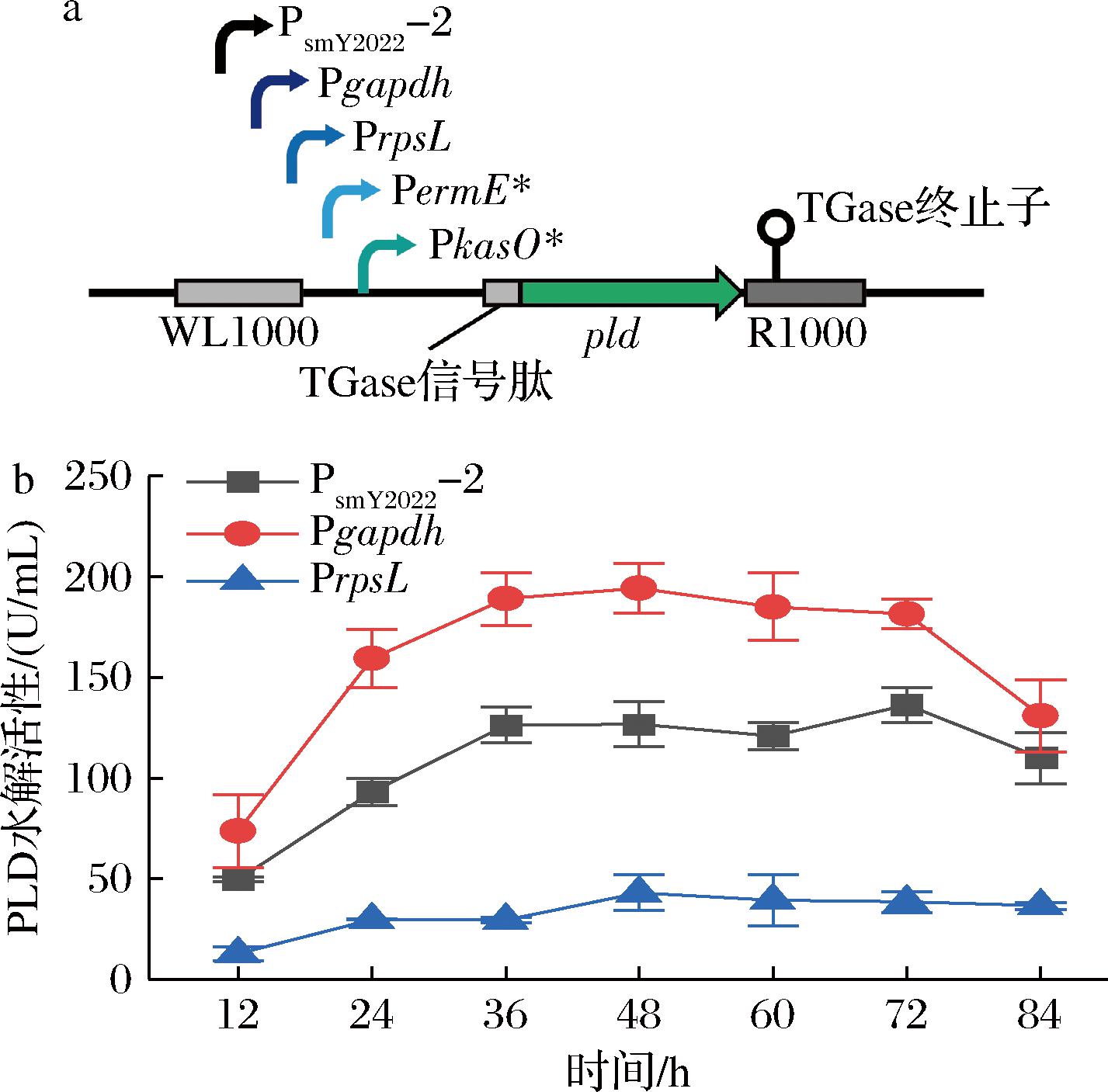
a-PLD基因表达框结构;b-不同启动子表达对PLD合成的影响
图3 启动子对PLD表达的影响
Fig.3 Effect of promoter on the expression of PLD
2.3 增加基因拷贝数提高PLD的表达
一般情况下,增加目标基因拷贝数是提高其在宿主菌中转录水和蛋白表达水平的有效手段[20]。前期研究表明,Csn是S.mobaraensis胞外表达水平较高的酶蛋白之一[19]。为进一步提高PLD在S.mobaraensis中的表达水平,基于CRISPR-Cas9基因编辑系统将以Pgapdh为启动子的PLD表达框整合至SM-PLD2中的Csn基因位点,得到PLD基因双拷贝菌株SM-PLD2d(图4-a)。对SM-PLD2d进行摇瓶发酵,并取胞外上清液进行PLD酶活力检测及SDS-PAGE分析。如图4-b所示,SM-PLD2在36 h的PLD酶活力迅速增加,此后增速明显变缓,到72 h达到最大值;SM-PLD2d胞外PLD水解活性在36 h后的增速依然明显,在60~72 h达到最值244.66 U/mL,较SM-PLD2提高26%(图4-b)。SDS-PAGE分析显示,发酵36 h的SM-PLD2d胞外上清液的PLD蛋白条带较SM-PLD2明显增粗(图4-c)。因此,增加基因拷贝数能显著提高PLD在S.mobaraensis中的表达水平。此前,课题组已在S.mobaraensis筛选得到多个可以高效表达目标基因的位点[19]。后续工作将基于CRISPR-Cas9系统在已知高表达位点整合更多PLD基因,以期提高其在S.mobaraensis中的表达水平。
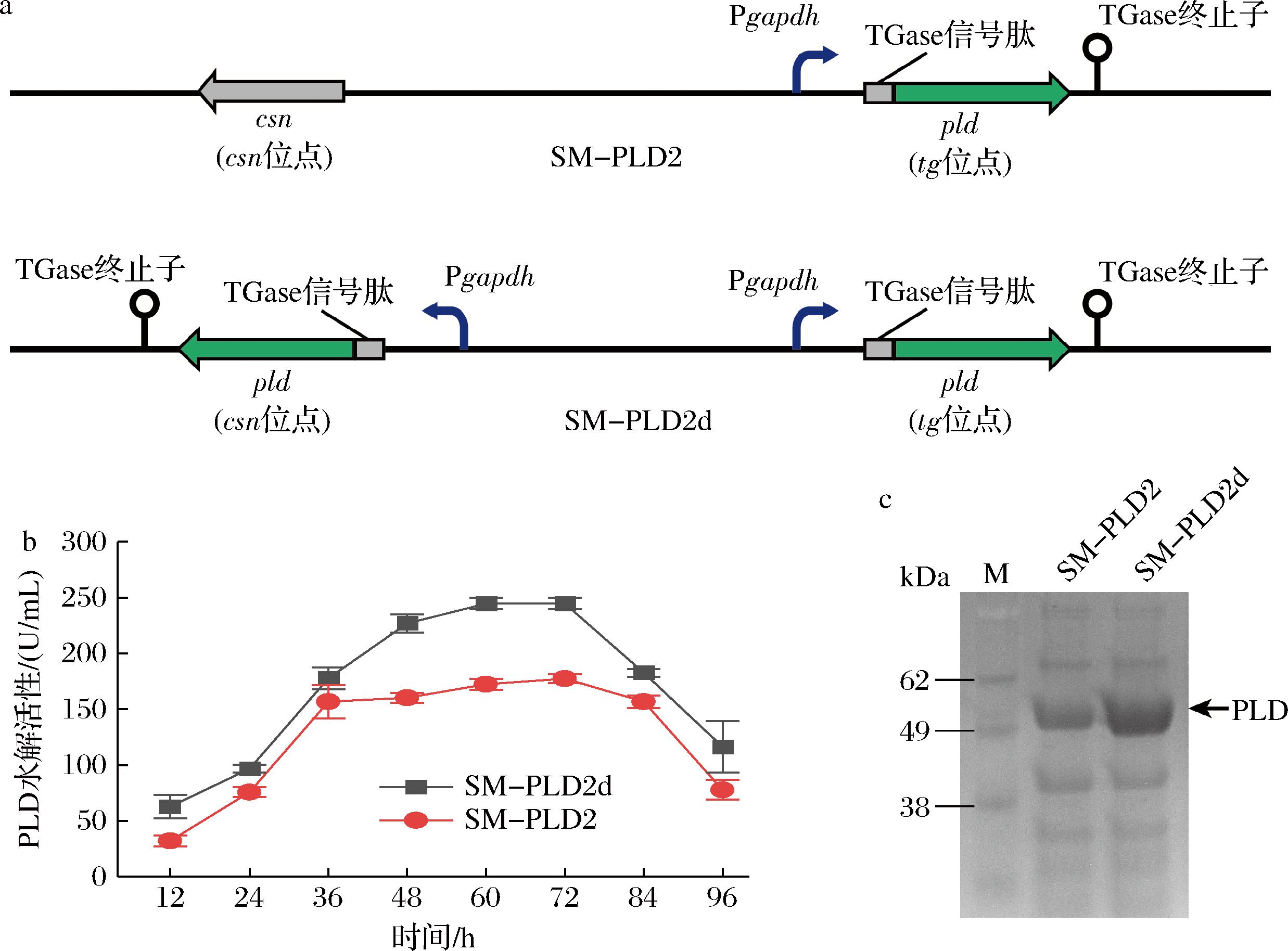
a-PLD基因整合位点;b-发酵上清液PLD酶活力;c-发酵上清液SDS-PAGE分析
图4 基因拷贝数对PLD表达的影响
Fig.4 Effect of gene copy number on PLD expression
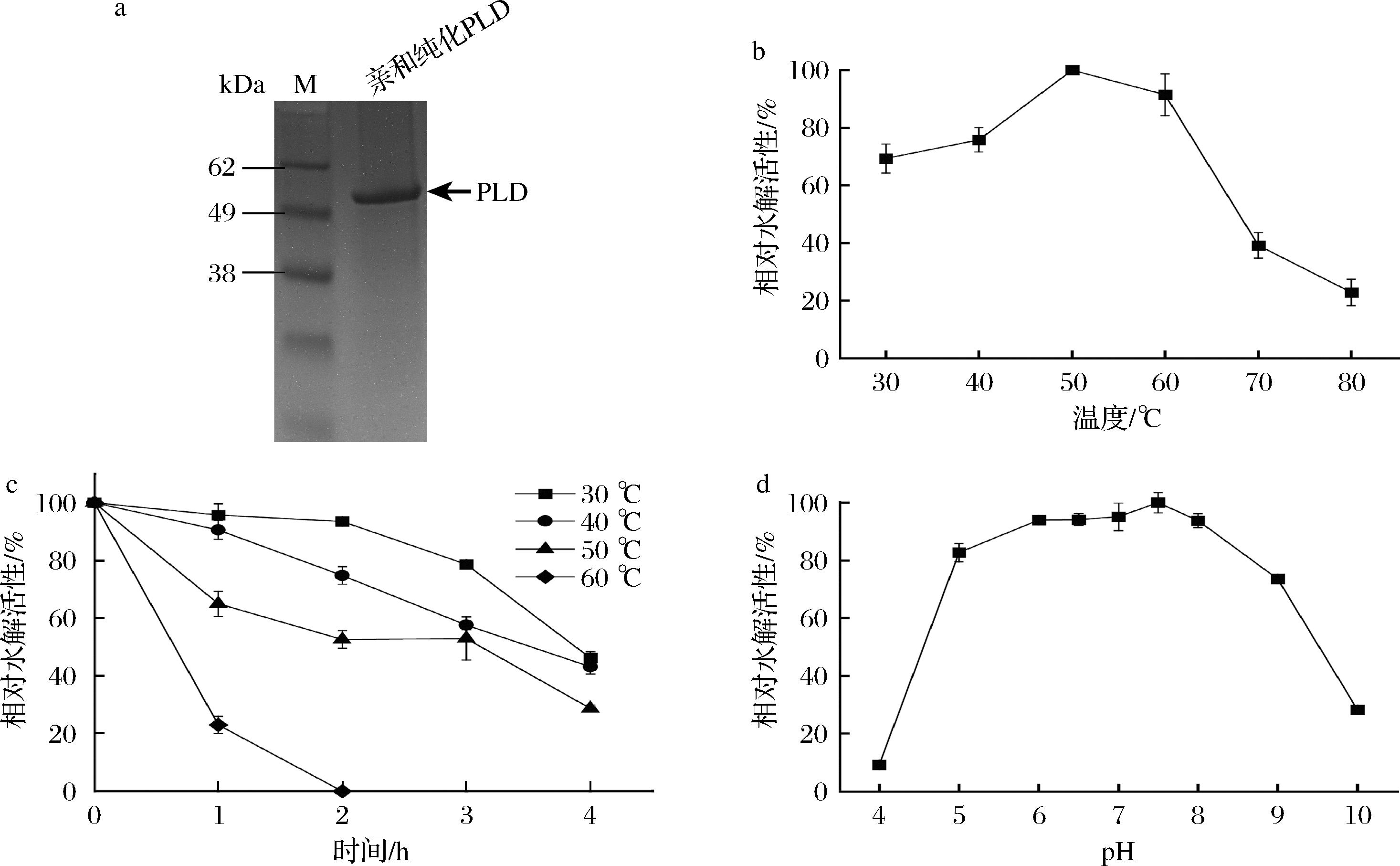
a-纯化后的PLD的SDS-PAGE分析;b-温度对PLD酶活力的影响;c-PLD的温度稳定性;d-pH对PLD酶活力的影响
图5 重组PLD的酶学性质
Fig.5 Enzymatic properties of recombinant PLD
2.4 PLD的纯化及酶学性质研究
为分析重组PLD的酶学性质,采用镍柱亲和层析法从SM-PLD2d的发酵上清液中纯化获得呈现单一电泳条带的重组PLD(图5-a)。进一步对酶的最适反应条件和稳定性进行了分析。如图5-b所示,重组PLD的最适温度为50 ℃,且在30~60 ℃能维持70%以上的催化活性。此外,重组PLD在30~50 ℃下保温2 h后均能保持50%以上水解活性,而在60 ℃保持2 h则完全失活(图5-c)。重组PLD最适反应pH为7.5,并在pH 5~8之间维持80%以上的催化活性(图5-d)。上述结果表明,重组PLD具有良好的热稳定性,以及环境适应性,与直接从S.racemochromogenes 10-3分离得到的PLD的酶学性质基本一致[4]。最适反应条件下(50 ℃和pH 7.5)进行水解反应,重组PLD比酶活力达到726.31 U/mg,优于Thermocrispum sp.[21]、Streptoverticillium cinnamoneum[22]和Streptomyces septatus[23]等来源PLD。基于此,S.mobaraensis表达的重组PLD具有潜在应用优势。
2.5 PLD催化PS合成
本研究利用重组PLD进行PS的酶法合成。为避免传统方法中使用有毒试剂的局限性,尝试采用绿色有机相2-甲基四氢呋喃作为酶催化反应的介质。HPLC分析结果显示,底物PC和产物PS在该体系中实现了良好的分离与检测(图6)。在对照组中仅检测到PC,而在实验组中则清晰检测到PC和PS的存在,表明重组PLD具备合成PS的活性。然而,反应体系中仍有大量底物PC未被完全转化,PS的产量仅为1.22 g/L,对应转化率为23.30%。

a-标准品及样品的HPLC检测;b-PS的标准曲线
图6 高效液相色谱检测分析
Fig.6 Detection and analysis by HPLC
PLD合成PS的效率受多种因素影响,包括有机相的种类,底物PC和L-丝氨酸的含量及比例、双相体系的体积比,以及反应条件(如振荡强度、温度、反应时间等)[24]。此外,通过酶分子改造和固定化[25]同样是提高PLD对功能性磷脂合成效率的重要途径。后续工作将聚焦于反应体系的优化以及PLD的理性改造,以进一步提升其PS合成能力,为实现高效、绿色的PS生产提供理论和技术支持。
3 结论
本研究通过CRISPR-Cas9基因编辑技术,成功将来源于S.racemochromogenes 10-3的PLD基因整合至工业宿主菌株S.mobaraensis DSM40587中,并通过启动子优化和基因拷贝数增加的策略,显著提高了PLD的表达水平。重组菌株SM-PLD2d在摇瓶发酵中胞外PLD酶活力达到244.66 U/mL,表明S.mobaraensis是一个高效的PLD表达宿主。此外,纯化的重组PLD在50 ℃和pH 7.5条件下表现出良好的热稳定性和催化活性,比酶活力达到726.31 U/mg,优于其他来源的PLD,显示出潜在的工业应用优势。
在绿色有机相2-甲基四氢呋喃中,重组PLD能够催化PS的合成,但转化率仅为23.30%,表明反应体系和酶性能仍有待优化。PLD合成PS的效率受多种因素影响,包括有机相种类、底物浓度及比例、双相体系体积比以及反应条件等。未来的研究将聚焦于反应体系的优化、酶分子改造以及固定化技术的应用,以进一步提高PLD的PS合成能力,为实现高效、绿色的PS生产提供理论和技术支持。此外,本研究还为利用S.mobaraensis作为重组蛋白生产的宿主菌提供了新的思路,有望推动其在工业生物技术中的广泛应用。
[1] HUSSAIN M,KHAN I,CHAUDHARY M N,et al.Phosphatidylserine:A comprehensive overview of synthesis,metabolism,and nutrition[J].Chemistry and Physics of Lipids,2024,264:105422.
[2] ZHANG Z X,CHEN M,XU W,et al.Microbial phospholipase D:Identification,modification and application[J].Trends in Food Science &Technology,2020,96:145-156.
[3] ZHANG P,GONG J S,QIN J F,et al.Phospholipids (PLs) know-how:Exploring and exploiting phospholipase D for its industrial dissemination[J].Critical Reviews in Biotechnology,2021,41(8):1257-1278.
[4] NAKAZAWA Y,SAGANE Y,KIKUCHI T,et al.Purification,biochemical characterization,and cloning of phospholipase D from Streptomyces racemochromogenes strain 10-3[J].The Protein Journal,2010,29(8):598-608.
[5] XIONG W D,LUO W Y,ZHANG X L,et al.High expression of toxic Streptomyces phospholipase D in Escherichia coli under salt stress and its mechanism[J].AIChE Journal,2020,66(4):e16856.
[6] ZHANG P,MAO X N,GONG J S,et al.High-level extracellular expression of phospholipase D by combinatorial fine-tuning strategy in Bacillus subtilis for efficient biosynthesis of phosphatidic acid[J].Bioresource Technology,2024,414:131492.
[7] WANG J T,ZHU H H,SHANG H Y,et al.Development of a thiostrepton-free system for stable production of PLD in Streptomyces lividans SBT5[J].Microbial Cell Factories,2022,21(1):263.
[8] TAO X Y,ZHAO M,ZHANG Y,et al.Comparison of the expression of phospholipase D from Streptomyces halstedii in different hosts and its over-expression in Streptomyces lividans[J].FEMS Microbiology Letters,2019,366(5):fnz051.
[9] CHEN S F,XIONG W D,ZHAO X R,et al.Study on the mechanism of efficient extracellular expression of toxic Streptomyces phospholipase D in Brevibacillus choshinensis under Mg2+ stress[J].Microbial Cell Factories,2022,21(1):41.
[10] ZHANG P,GONG J S,XIE Z H,et al.Efficient secretory expression of phospholipase D for the high-yield production of phosphatidylserine and phospholipid derivates from soybean lecithin[J].Synthetic and Systems Biotechnology,2023,8(2):273-280.
[11] HWANG S,LEE Y,KIM J H,et al.Streptomyces as microbial chassis for heterologous protein expression[J].Frontiers in Bioengineering and Biotechnology,2021,9:804295.
[12] SEVILLANO L,VIJGENBOOM E,VAN WEZEL G P,et al.New approaches to achieve high level enzyme production in Streptomyces lividans[J].Microbial Cell Factories,2016,15:28.
[13] OGINO C,KANEMASU M,FUKUMOTO M,et al.Continuous production of phospholipase D using immobilized recombinant Streptomyces lividans[J].Enzyme and Microbial Technology,2007,41(1-2):156-161.
[14] YUAN F,LI G Y,LI Z L,et al.Efficient biosynthesis of transglutaminase in Streptomyces mobaraensis via systematic engineering strategies[J].Current Research in Food Science,2024,8:100756.
[15] YE J C,YANG P H,ZHOU J W,et al.Efficient production of a thermostable mutant of transglutaminase by Streptomyces mobaraensis[J].Journal of Agricultural and Food Chemistry,2024,72(8):4207-4216.
[16] COBB R E,WANG Y J,ZHAO H M.High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J].ACS Synthetic Biology,2015,4(6):723-728.
[17] SHAO Z Y,RAO G D,LI C,et al.Refactoring the silent spectinabilin gene cluster using a plug-and-play scaffold[J].ACS Synthetic Biology,2013,2(11):662-669.
[18] NAKAZAWA Y,SAGANE Y,SAKURAI S I,et al.Large-scale production of phospholipase D from Streptomyces racemochromogenes and its application to soybean lecithin modification[J].Applied Biochemistry and Biotechnology,2011,165(7-8):1494-1506.
[19] 叶佳才.耐热谷氨酰胺转氨酶在茂原链霉菌中的表达研究[D].无锡:江南大学,2024.YE J C.Expression of thermostable transglutaminase in Streptomyces mobaraensis[D].Wuxi:Jiangnan University,2024.
[20] LIU X Q,YANG X Q,XIE F H,et al.Cloning of transglutaminase gene from Streptomyces fradiae and its enhanced expression in the original strain[J].Biotechnology Letters,2006,28(17):1319-1325.
[21] MATSUMOTO Y,KASHIWABARA N,OYAMA T,et al.Molecular cloning,heterologous expression,and enzymatic characterization of lysoplasmalogen-specific phospholipase D from Thermocrispum sp[J].FEBS Open Bio,2016,6(11):1113-1130.
[22] OGINO C,NEGI Y,MATSUMIYA T,et al.Purification,characterization,and sequence determination of phospholipase D secreted by Streptoverticillium cinnamoneum[J].Journal of Biochemistry,1999,125(2):263-269.
[23] HATANAKA T,NEGISHI T,KUBOTA-AKIZAWA M,et al.Purification,characterization,cloning and sequencing of phospholipase D from Streptomyces septatus TH-2[J].Enzyme and Microbial Technology,2002,31(3):233-241.
[24] CHOOJIT S,BORNSCHEUER U T,UPAICHIT A,et al.Efficient phosphatidylserine synthesis by a phospholipase D from Streptomyces sp.SC734 isolated from soil-contaminated palm oil[J].European Journal of Lipid Science and Technology,2016,118(5):803-813.
[25] WU J Q,XU X M,WANG D L,et al.Immobilization of phospholipase D on macroporous SiO2/cationic polymer nano-composited support for the highly efficient synthesis of phosphatidylserine[J].Enzyme and Microbial Technology,2020,142:109696.