肝素作为一种从动物结缔组织肥大细胞中提取的高度硫酸化糖胺聚糖,凭借其独特的抗凝血、降血脂、抗炎及抗肿瘤等多重生物活性,在临床治疗和生物医学领域具有重要应用价值[1-2]。目前商业化肝素原料药主要依赖猪小肠黏膜提取,但存在原料供应不稳定、提取效率低下及产品结构异质性显著等固有缺陷[3]。相较而言,微生物-酶法合成技术因其原料可持续性、工艺环境友好性及产物结构可控性等优势,已成为新型肝素制备策略的研究热点[4-5]。基于大肠杆菌(Escherichia coli)Nissle 1917的微生物合成体系通过发酵获得肝素前体后[6],需经关键性磺基转移反应赋予其抗凝血活性。该过程高度依赖3′-磷酸腺苷-5′-磷酰硫酸(3′-phosphoadenosine-5′-phosphosulfate,PAPS)作为磺酸基供体,然而PAPS的高昂成本与化学不稳定性严重制约了该技术的工业化应用。如图1所示,芳基磺基转移酶 Ⅳ(aryl sulfotransferase Ⅳ,AST Ⅳ)可催化对硝基苯酚磺酸盐(p-nitrophenol sulfate,PNPS)与3′,5′-二磷酸腺苷 (3′-phosphoadenosine-5′-phosphate,PAP)实现磺基供体再生,这为构建经济高效的PAPS再生系统提供了新思路[7-9]。因此,深入解析AST Ⅳ的催化机制、特别是关键氨基酸残基对其酶活性的调控规律,对优化肝素生物合成体系具有重要科学意义。值得注意的是,AST Ⅳ分子内存在二硫键网络且含有5个半胱氨酸残基[10]。作为蛋白质功能结构域的核心元素,半胱氨酸可以通过二硫键维系酶分子的三维构象稳定性、其硫醇基团直接参与催化中心的亲核反应,可逆的氧化还原修饰介导酶活性的动态调节、通过构象重排响应细胞微环境变化,而突变为丙氨酸后,会破坏二硫键的形成,从而改变蛋白质的结构,进而影响其功能[11-14]。为系统探究二硫键形成对AST Ⅳ功能的影响机制,本研究将Cys66、Cys82、Cys232、Cys283、Cys289定点突变成丙氨酸,通过比较不同突变体的酶动力学参数,辅以分子模拟技术揭示关键残基的构效关系。
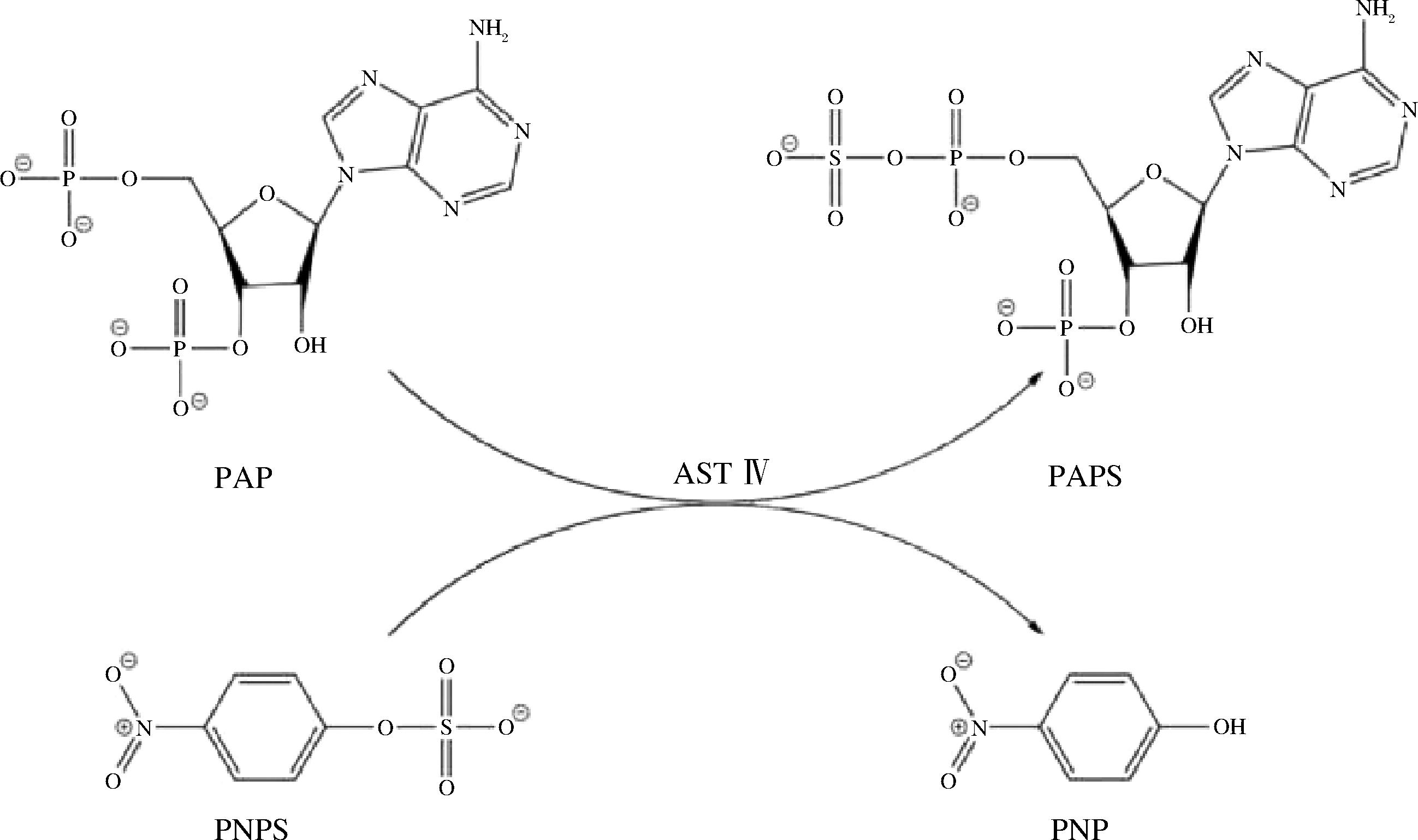
图1 PAPS再生系统
Fig.1 PAPS regeneration system
1 材料与方法
1.1 质粒、菌株和培养基
在本研究中,E.coli BL21 (DE3)、Origami (DE3)以及DH5α被选定作宿主菌株,用于执行基因克隆与表达。所有培养和表达均在LB培养基中进行,该培养基的配方为(g/L):NaCl 5、蛋白胨 5、酵母提取物10。在实验过程中,根据需要添加50 μg/mL的氨苄青霉素以维持选择压力,并添加1 mmol/L的异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)以诱导AST Ⅳ的基因表达。
1.2 构建质粒pET-32b AST Ⅳ
在本研究中,质粒pET-32b AST Ⅳ的构建流程首先通过访问NCBI查询数据库并利用编号为EC_number="2.8.2.1"获取了大鼠AST Ⅳ基因的序列信息。随后,为了提高在宿主细胞中的表达效率,利用VectorBuilder平台(https://www.vectorbuilder.cn/tool/codon-optimization.html)对AST Ⅳ基因进行了密码子优化。密码子优化后的大鼠AST Ⅳ基因(由通用生物合成)被精确地插入到pET-32b质粒的Nco I 和Xho I两个酶切位点之间。通过酶连分子克隆技术准确地插入质粒中。成功构建的质粒被命名为pET-32b AST Ⅳ。为了确认质粒构建的准确性和完整性,进行了DNA测序分析。
1.3 定点突变半胱氨酸
使用Gibson Assembly(无缝克隆)定点突变半胱氨酸,首先对pET-32b AST Ⅳ质粒进行线性化处理,以便于后续的克隆操作,使用高保真PCR Mix(Takara Bio PrimeSTAR® HS)针对目的氨基酸进行PCR扩增。将线性化的载体和目的基因在50 ℃反应20 min(使用Sangon B632219试剂)。这一步骤是Gibson Assembly技术的核心,通过精确的温度和时间控制,实现了载体和插入片段的无缝连接。本研究中使用的寡核苷酸引物列于表1。
表1 使用的寡核苷酸引物
Table 1 Oligonucleotide primers used in this study

引物名称序列 5′→3′ 用途P0741GAAAAAGCGGGCCGCGCCCCGATTTATGCGP0742GCGGCCCGCTTTTTCCAGTTTACCGCCCTG定点突变Cys66P0743TGAAAGGCATTCCGCTGAP0744CCACATCTTTCGCATTGCG检测定点突变P0745AAGTGAGCTATGGCTCATGP0746TCCGGATATAGTTCCTCCTTTC检测定点突变P0749AATTTAAAGCGCCGGGCGTTCCGAGCGGTCTGP0750GCCCGGCGCTTTAAATTCCAGAAACGGCACACG定点突变Cys82P0751AAAATGCGATGACCAATTACACCACCATCCCGP0752ATTGGTCATCGCATTTTCTTTCATTTTTTTGAAGCTGG定点突变Cys232P0753GACCGATGCGGATTTTAAATTTCGCTGCGAACP0754TAAAATCCGCATCGGTCATGGTTTTCGCATAATGCG定点突变Cys283P0755AATTTCGCGCGGAACTCGAGCACCACCACCACP0756CGAGTTCCGCGCGAAATTTAAAATCGCAATCGGTCATG定点突变Cys289
1.4 AST Ⅳ的分子结构建模
通过Expasy-ProtParam以及ProtParam工具预测野生型AST Ⅳ和定点突变半胱氨酸为丙氨酸后的AST Ⅳ理化性质,为了预测AST Ⅳ的三级结构,采用了AlphaFold3进行结构预测。利用其先进的算法预测出蛋白质的三维结构。使用Schrodinger软件的SiteMap工具进行潜在结合位点的预测,该工具能够识别蛋白质表面的结合口袋,为后续的药物设计提供关键信息。此外,为了深入理解PAP分子与预测蛋白之间的相互作用,进一步运用PyMol软件计算了分子与氨基酸残基之间的距离,这一分析有助于揭示分子间相互作用的细节。通过PredictProtein(https://predictprotein.org/)与http://liulab.csrc.ac.cn/ssbondpre[15]对AST Ⅳ中二硫键的存在进行预测。
1.5 AST Ⅳ的纯化
从LB培养基中挑选单个克隆进行过夜培养。随后,将过夜培养物以1%接种量转移到含有250 mL LB培养基的锥形瓶中,在37 ℃、220 r/min的条件下进行摇床培养,直至OD600值达到0.6~0.8。此时,加入IPTG并在22 ℃条件下诱导表达16 h。诱导完成后,将菌液在7 500×g、4 ℃的条件下离心15 min以收集菌体。收集的菌体随后用30 mL缓冲液Ⅰ(0.2 mol/L NaCl,0.02 mol/L Tris-HCl,pH=7.6)重悬,并加入体积分数为1%的苯甲基磺酰氟(benzylsulfonyl fluoride,PMSF)。重悬液在50%功率、5 s运行、5 s停止的条件下超声破碎30 min,然后以7 500 r/min、4 ℃离心30 min,收集上清液,并通过0.22 μm滤膜过滤制成样品。接下来,通过Ni-NTA琼脂糖柱进行亲和色谱法纯化AST Ⅳ。首先,使用80 mL缓冲液Ⅰ平衡Ni-NTA琼脂糖柱,然后上样30 mL样品。接着,使用80 mL缓冲液Ⅱ(0.2 mol/L NaCl,0.02 mol/L Tris-HCl,0.05 mol/L咪唑,pH=7.6)进行杂质洗脱,最后使用80 mL缓冲液Ⅲ(0.2 mol/L NaCl,0.02 mol/L Tris-HCl,0.25 mol/L咪唑,pH=7.6)进行目标蛋白的洗脱,从而获得纯净的AST Ⅳ。为了确定纯化AST Ⅳ的浓度,使用牛血清白蛋白作为标准品进行测量。
1.6 AST Ⅳ 酶活性测定
利用AST Ⅳ催化PAP形成PAPS过程中,形成一种在碱性条件下显色的物质对硝基苯酚(p-nitrophenol,PNP),利用标准曲线计算PNP浓度。酶活性定义为在37 ℃条件下,每分钟合成1 μmol/L PNP所需的酶量,比酶活性定义为U/mg。以100 mmol/L Tris-HCl缓冲液(pH=7.4)、50 mmol/L PNPS、0.1 mmol/L PAP、20 μg的AST Ⅳ蛋白上清液为反应体系,总体积调整至200 μL。将上述混合物置于37 ℃的恒温环境中温育15 min,以确保酶与底物充分反应。温育结束后,使用分光光度计测量反应混合物的OD400值。通过比较反应前后的吸光值变化,可以定量分析AST Ⅳ蛋白的酶活性[16]。
1.7 AST Ⅳ酶动力学常数测定
反应体系为100 mmol/L Tris-HCl (pH=7.4)缓冲液、50 mmol/L PNPS、0.005~0.2 mmol/L PAP、20 μg AST Ⅳ蛋白上清液至200 μL,将上述混合物置于37 ℃的恒温环境中温育5 min,以启动酶促反应,从温育开始后,每隔1 min测量1次反应混合物的OD400值,连续测量15 min。通过记录这一时间序列的吸光值变化,可以实时监测酶促反应的进程。收集的数据通过米氏方程的双倒数作图法进行分析。计算如公式(1)所示,此方法通过绘制1/v对1/S的图,其中v为反应速度(nmol/min),VM为最大反应速度(nmol/min),S为底物浓度(mol/L),Km为米氏常数(mol/L)。通过此图可以求得VM和Km的值,从而了解酶的催化特性和底物亲和力[17]。
(1)
1.8 分子动力学模拟
通过PyMOLWin软件的align功能对AlphaFold3预测的蛋白进行比对,之后通过Discovery Studio 2019 Client软件进行分子动力学模拟,选用的是全原子CHARMM36力场,首先对蛋白样品进行能量最小化消除空间冲突,并且使用TIP3P水模型进行溶剂化,并添加适当的离子以保持系统中性。在NPT系统下进行20 ns的分子动力学模拟,模拟采用周期性边界条件,并用Langevin算法控制温度和压强。
2 结果与分析
2.1 二硫键对AST Ⅳ酶活性的影响
在相同反应条件(50 mmol/L PNPS、0.1 mmol/L PAP、100 mmol/L Tris-HCl溶液)下,分别加入BL21(DE3)和OrigamiB(DE3)2种菌种表达纯化的20 μg AST Ⅳ,如图2~图3所示,BL21(DE3)的AST Ⅳ酶活性为(5.93±1.57) U/mg,显著低于OrigamiB(DE3)的AST Ⅳ酶活性(9.28±0.34) U/mg。鉴于AST Ⅳ蛋白内部含有一对二硫键[10],而大肠杆菌胞内蛋白质的合成和折叠过程通常不利于二硫键的形成。OrigamiB(DE3)菌株具备促进二硫键生成的能力,有助于含二硫键蛋白的正确折叠和活性蛋白的形成,因此在相同条件下,2种菌株表现出不同的酶活性。由此可见,二硫键的折叠程度对AST Ⅳ活性具有显著影响,且折叠程度越高,AST Ⅳ活性越高。
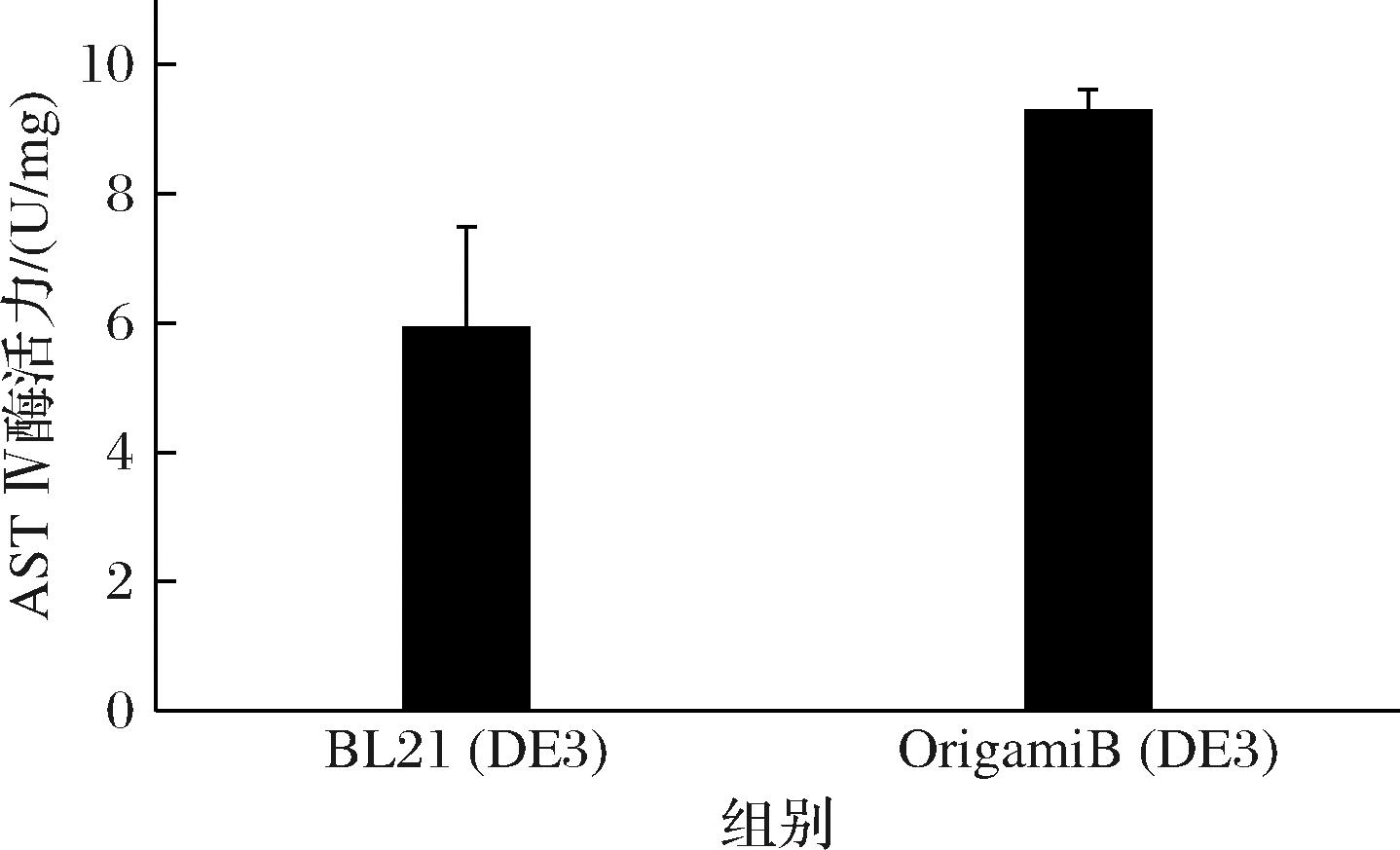
图2 不同菌种的野生型AST Ⅳ酶活性
Fig.2 Enzymatic activity of wild-type AST Ⅳ from different bacterial species
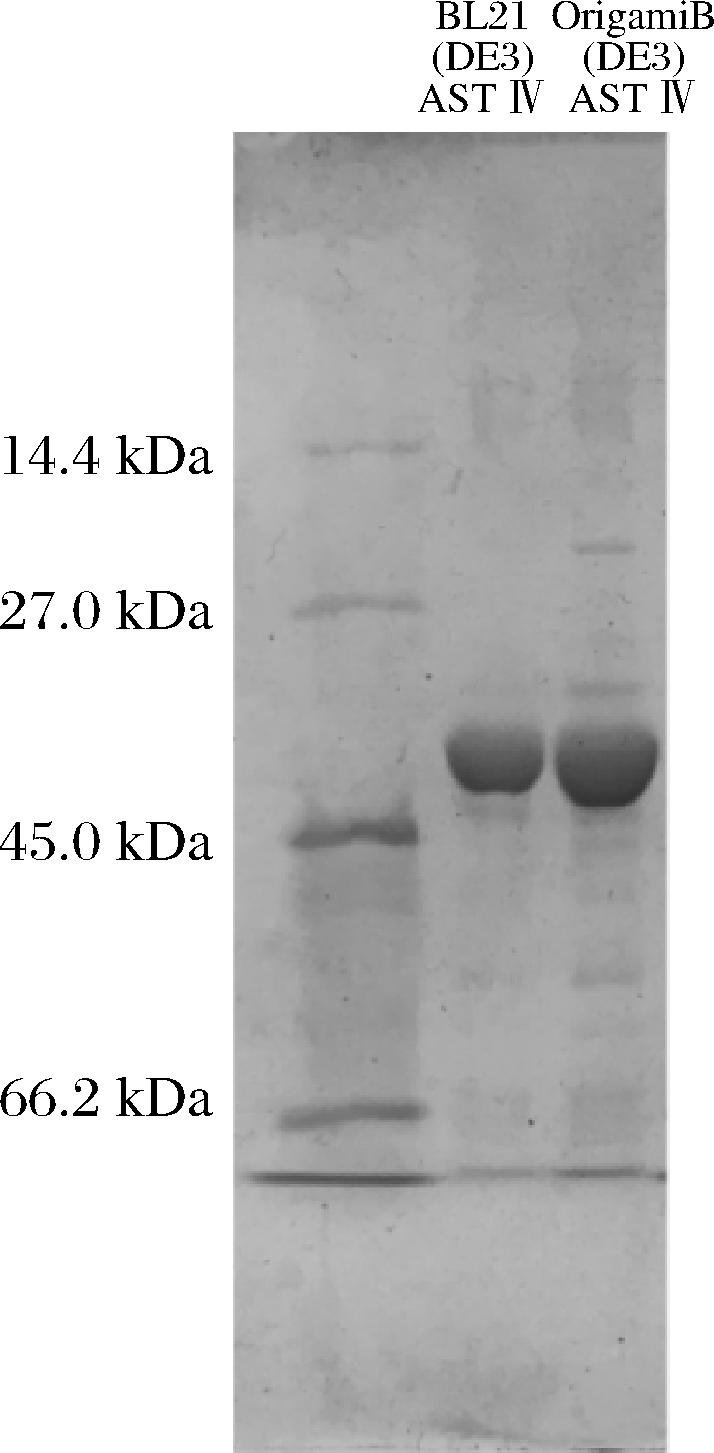
图3 不同菌种表达纯化AST Ⅳ的SDS-PAGE
Fig.3 SDS-PAGE analysis of expressed and purified AST Ⅳ from different bacterial species
2.2 定点突变半胱氨酸AST Ⅳ理化性质
基于AST Ⅳ氨基酸序列,对Cys66、Cys82、Cys232、Cys283、Cys289 分别进行定点突变成丙氨酸。通过Expasy-ProtParam预测蛋白的理化性质,结果如表2所示,突变后的AST Ⅳ分子质量比野生型AST Ⅳ小32.06 Da,而等电点、不稳定系数、疏水性及α-螺旋等并无明显变化。这表明,这些位点的突变对蛋白质的基本理化性质影响较小。
表2 野生型AST Ⅳ和定点突变不同位置半胱氨酸AST Ⅳ的理化性质
Table 2 Physicochemical properties of wild-type AST Ⅳ and site-directed cysteine-to-alanine mutants at various positions
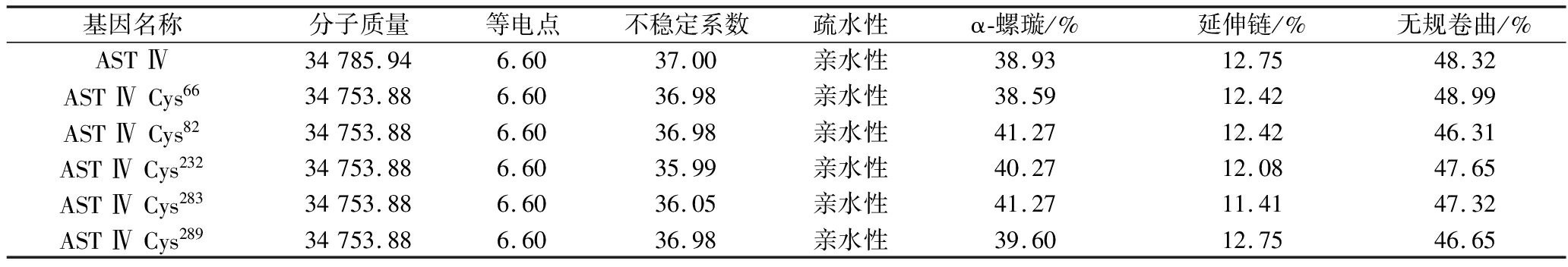
基因名称分子质量等电点不稳定系数疏水性α-螺璇/%延伸链/%无规卷曲/%ASTⅣ34785.946.6037.00亲水性38.9312.7548.32ASTⅣCys6634753.886.6036.98亲水性38.5912.4248.99ASTⅣCys8234753.886.6036.98亲水性41.2712.4246.31ASTⅣCys23234753.886.6035.99亲水性40.2712.0847.65ASTⅣCys28334753.886.6036.05亲水性41.2711.4147.32ASTⅣCys28934753.886.6036.98亲水性39.6012.7546.65
2.3 AST Ⅳ底物结合口袋的结构建模
通过PredictProtein(https://predictprotein.org/)与http://liulab.csrc.ac.cn/ssbondpre网站预测发现Cys66和Cys82形成二硫键,AST Ⅳ通过催化PNPS将磺酸盐基团转移至底物PAP,其催化作用的关键在于PNPS结合区和PAP结合区。通过NCBI上发现人的磺基转移酶4GRA、3U3M和2A3R[18-20],并进行了氨基酸序列比对。图4结果显示,只有Cys66和Cys283是保守的。

图4 AST Ⅳ和人磺基转移酶的序列比对
Fig.4 Sequence alignment of AST Ⅳ and human sulfotransferases
注:序列上方黑色箭头链和黑色螺旋链表示AST Ⅳ的二级结构,加粗字体显示的是AST Ⅳ的底物结合的残基。
为了模拟AST Ⅳ的三维结构,利用AlphaFold3构建了其分子结构模型,并通过Schrodinger软件进行sitemap,分析了PAP分子与预测蛋白之间的相互作用。图5结果表明,参与PAP结合口袋形成的残基Lys45、Gly47、Thr48、Thr49和Lys103在人磺基转移酶中高度保守。同源建模的分子结构模型显示,PAP结合位点在人磺基转移酶中也高度保守[21]。

图5 AST Ⅳ与PAP对接的立体图与不同氨基酸残基图
Fig.5 Stereoview of AST Ⅳ-PAP docking complex and interactions with key amino acid residues
通过Pymol软件对不同半胱氨酸进行可视化,图6结果表明,发现Cys66、Cys82和Cys232位于结合口袋附近,而Cys283和Cys289位于结合口袋的背面。尽管Cys283位于结合口袋的背面,其与底物结合的直接位点有一定距离。这种位置关系可能使得Cys283对酶活性的影响较小,而图4表明Cys283高度保守,可能源于其对三级结构稳定的贡献,其对酶的整体结构、稳定性和功能方面发挥着关键作用,导致其对酶活性的影响更大。
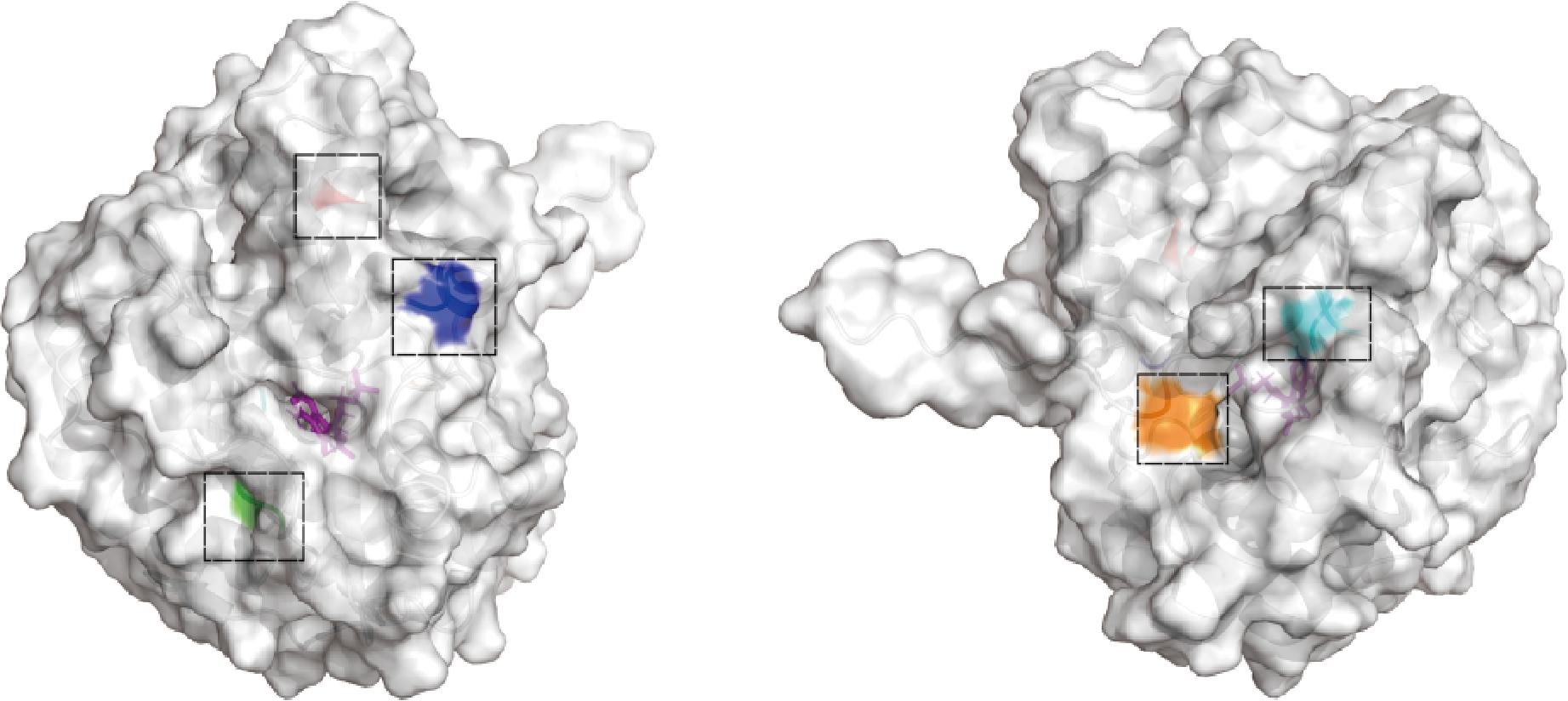
图6 不同半胱氨酸在AST Ⅳ电子云结构中位置
Fig.6 Spatial positions of cysteine residues in AST Ⅳ electron density map
注:紫色代表PNP分子,红色、绿色、蓝色、青色、橙色代表Cys66、Cys82、Cys232、Cys283、Cys289。
2.4 酶学性质参数
为深入探究不同半胱氨酸的定点突变对AST Ⅳ在再生PAPS过程中活性的影响,本研究对野生型及各突变体AST Ⅳ进行了纯化,并对其动力学参数进行了详细分析。结果如表3所示,与野生型相比,Cys66和Cys82突变体对PAP的亲和力显著降低,Km值分别为(0.89±0.04)、(0.95±0.03) mmol/L,同时催化效率也大幅下降,kcat/Km值分别为(80.53±23.44)、(104.85±25.17) L/(s·mol)。结合预测的结果,推测这种现象可能是由于突变破坏了二硫键,进而导致AST Ⅳ活性降低。Cys232突变体表现出更低的PAP亲和力(Km=0.90±0.01) mmol/L,但其催化效率[kcat/Km=(156.34±30.66) L/(s·mol)]却高于野生型,这可能是由于Cys232位于结合口袋附近,该残基的突变可能影响了结合口袋的结构,导致对底物的结合亲和力下降,但该突变可能优化了酶的催化机制,提高了酶的催化效率。Cys283突变体对PAP的亲和力显著降低,Km值为(0.92±0.01) mmol/L,而催化效率kcat/Km值为(116.00±8.30) L/(s·mol),低于野生型的(139.13±25.06) L/(s·mol),这可能是由于Cys283是高度保守的半胱氨酸,而高度保守的半胱氨酸在蛋白质结构和功能中通常具有关键作用,对其进行突变可能导致影响蛋白质的整体稳定性,使其更易发生变性或降解,进而间接影响其活性。Cys289突变体对PAP的亲和力是所有野生型中最低的,并且远低于其余野生型,但是其催化效率kcat/Km为(146.43±35.18) L/(s·mol),与野生型的催化效率相比影响并不明显,这可能是突变使得蛋白整体构象发生变化,Cys289可能在结构中存在更加复杂的角色。值得注意的是,进化保守的Cys66和Cys283其对酶的结构稳定性和功能维持具有关键作用,对其进行突变会导致酶活性降低。
表3 野生型AST Ⅳ和定点突变不同位置半胱氨酸AST Ⅳ的动力学参数的影响
Table 3 Kinetic parameters of wild-type AST Ⅳ and site-directed cysteine mutants at various positions

参数ASTⅣASTⅣCys66ASTⅣCys82ASTⅣCys232ASTⅣCys283ASTⅣCys289Km/ mmol/L 0.80±0.020.89±0.040.95±0.030.90±0.010.92±0.011.02±0.03kcat/s-10.11±0.020.08±0.020.10±0.020.14±0.030.11±0.010.15±0.04kcat/Km/ L/ s mol 139.13±25.0680.53±23.44104.85±25.17156.34±30.66116.00±8.30146.43±35.18
将定点突变后的AST Ⅳ Cys66、AST Ⅳ Cys82、AST Ⅳ Cys232的质粒分别导入到BL21(DE3)和OrigamiB(DE3)菌种中,纯化后测定酶活性。如图7所示,与BL21(DE3)菌种表达的AST Ⅳ酶活性相比,OrigamiB(DE3)菌种表达的AST Ⅳ Cys66和AST Ⅳ Cys82酶活性并无明显变化,分别为BL21(DE3)菌种AST Ⅳ酶活性的98.56%和98.92%,这表明Cys66和Cys82可能形成二硫键,从而导致酶活性并无差异。然而,当定点突变Cys232时,OrigamiB(DE3)菌种表达的AST Ⅳ酶活性达到BL21(DE3)菌种表达的AST Ⅳ酶活性的1.2倍。由此推断,如果在AST Ⅳ中定点突变Cys66和Cys82会破坏二硫键,会对酶活性产生影响。
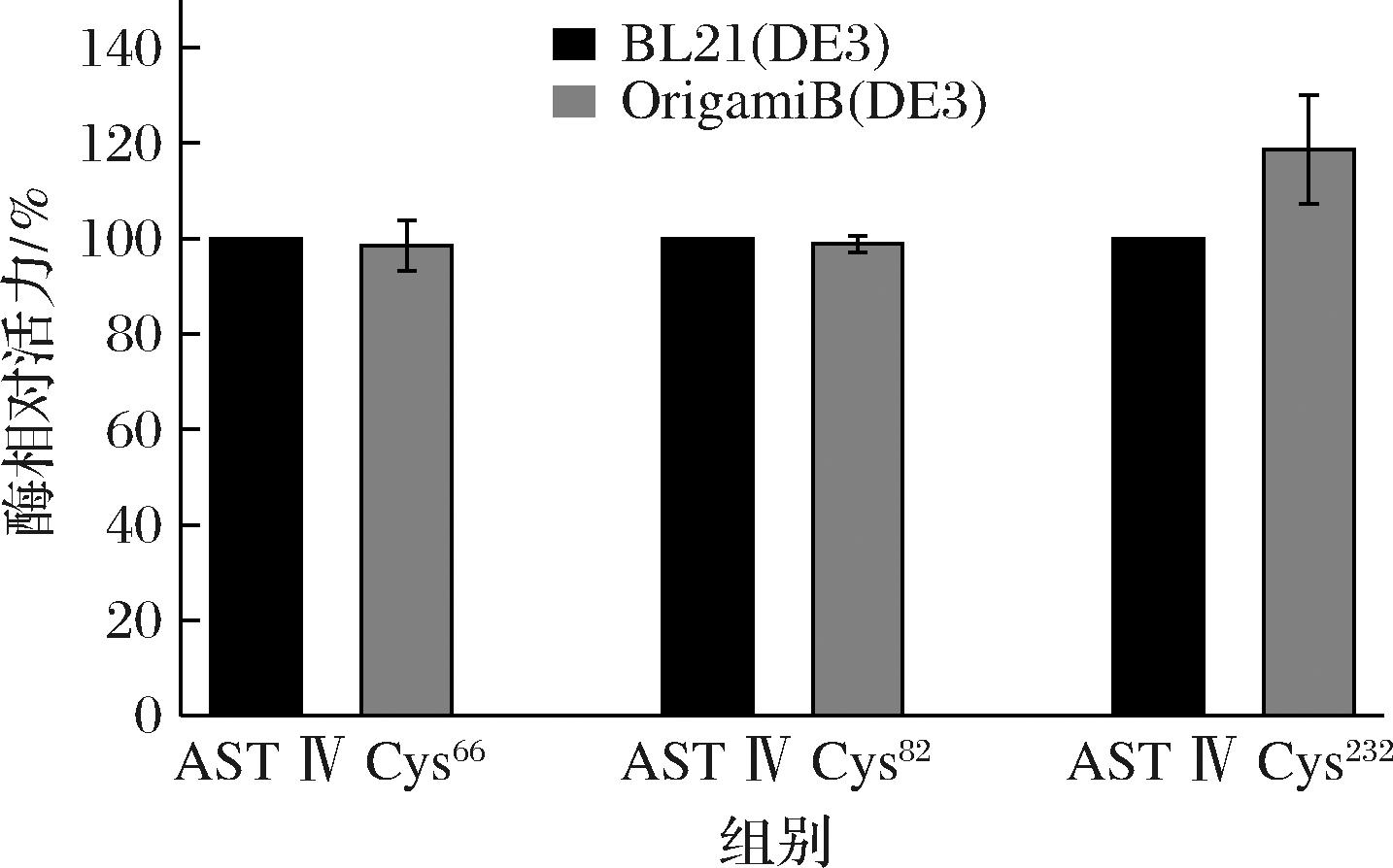
图7 不同菌种的突变型AST Ⅳ酶活性
Fig.7 Enzymatic activity of AST Ⅳ mutants from different bacterial species
2.5 蛋白立体结构对比
在本研究中,借助AlphaFold3预测了AST Ⅳ以及其多个定点突变体AST Ⅳ、AST Ⅳ Cys66、AST Ⅳ Cys82、AST Ⅳ Cys232、AST Ⅳ Cys283、AST Ⅳ Cys289的蛋白立体结构。随后,运用Pymol软件对野生型AST Ⅳ与各定点突变后的AST Ⅳ进行立体结构对比分析。结果如图8所示,所有定点突变后的AST Ⅳ与野生型的均方根偏差(root mean square deviation,RMSD)值均小于0.2 Å,RMSD值是一个衡量蛋白质结构差异的指标,它越小,说明结构越相似。该值小于1 Å时,通常被认为是高度相似的,这表明将特定位置的半胱氨酸定点突变为丙氨酸,对蛋白整体的立体结构并不会产生显著影响,也进一步印证了不同位置半胱氨酸定点突变后的 AST Ⅳ 仍具备活性这一现象。
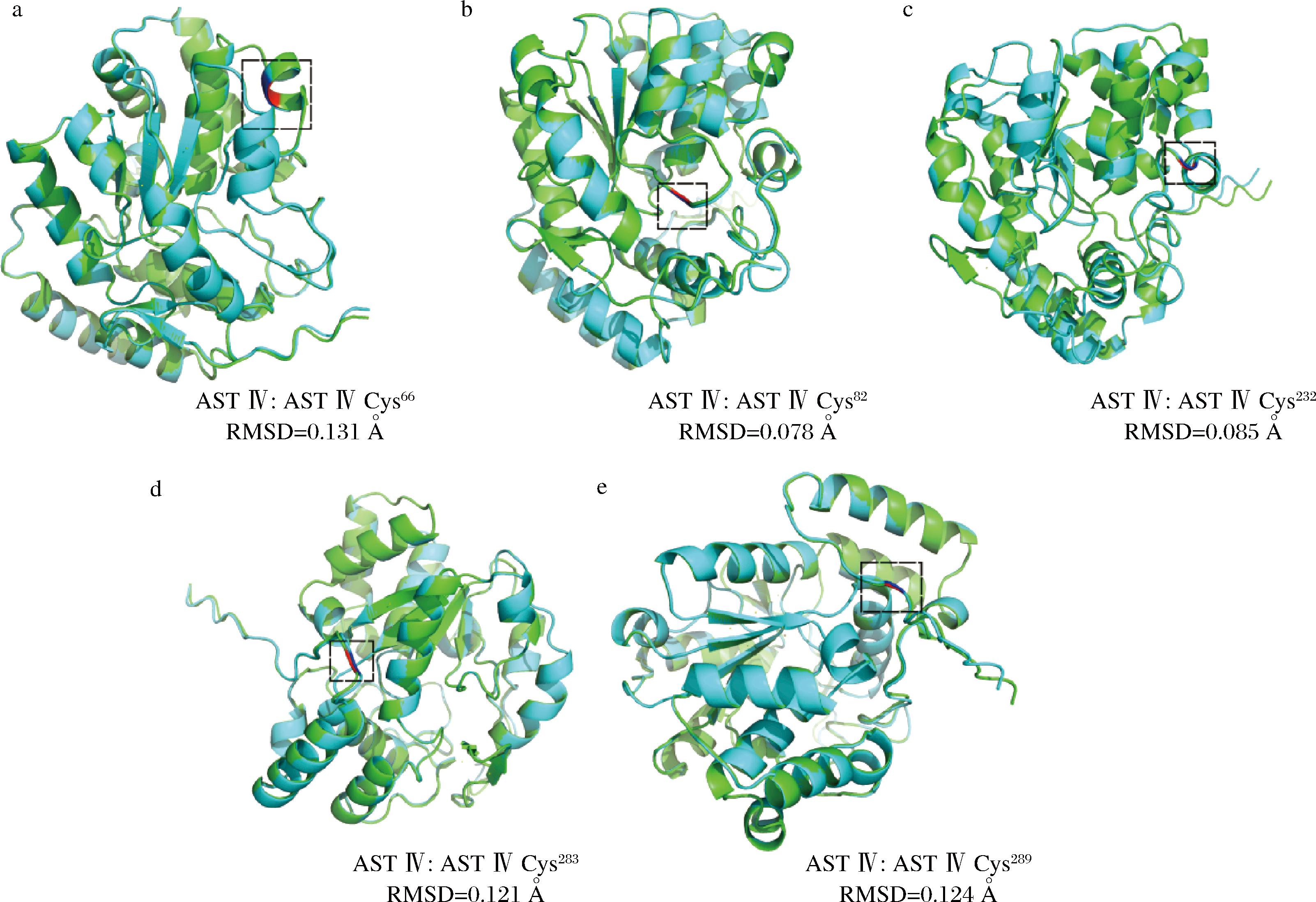
a-AST Ⅳ Cys66;b-AST Ⅳ Cys82;c-AST Ⅳ Cys232;d-AST Ⅳ Cys283;e-AST Ⅳ Cys289
图8 不同AST Ⅳ结构比对图
Fig.8 Structural comparisons of AST Ⅳ variants
注:图中绿色结构为无突变的AST Ⅳ蛋白,青色为定点突变后的AST Ⅳ蛋白,黑色方框为突变区域,其中红色代表半胱氨酸,蓝色代表突变成丙氨酸。
进一步观察发现,对于高度保守的半胱氨酸位点,如Cys66和Cys283,在进行定点突变后,其蛋白立体结构与野生型AST Ⅳ相比,RMSD值较Cys82和Cys232突变型略大,这表明尽管整体结构仍保持稳定,这些高度保守位点的突变可能对蛋白结构的局部细微构象产生相对更为明显的影响。
如图9所示,通过分子动力学模拟,突变型AST Ⅳ Cys66、AST Ⅳ Cys82与野生型相比,在突变位置邻近残基的RMSF值略高于野生型,表明局部结构稳定性下降。这可能是二硫键断开使得其邻近残基的RMSF值增加,进而影响酶的催化效率。
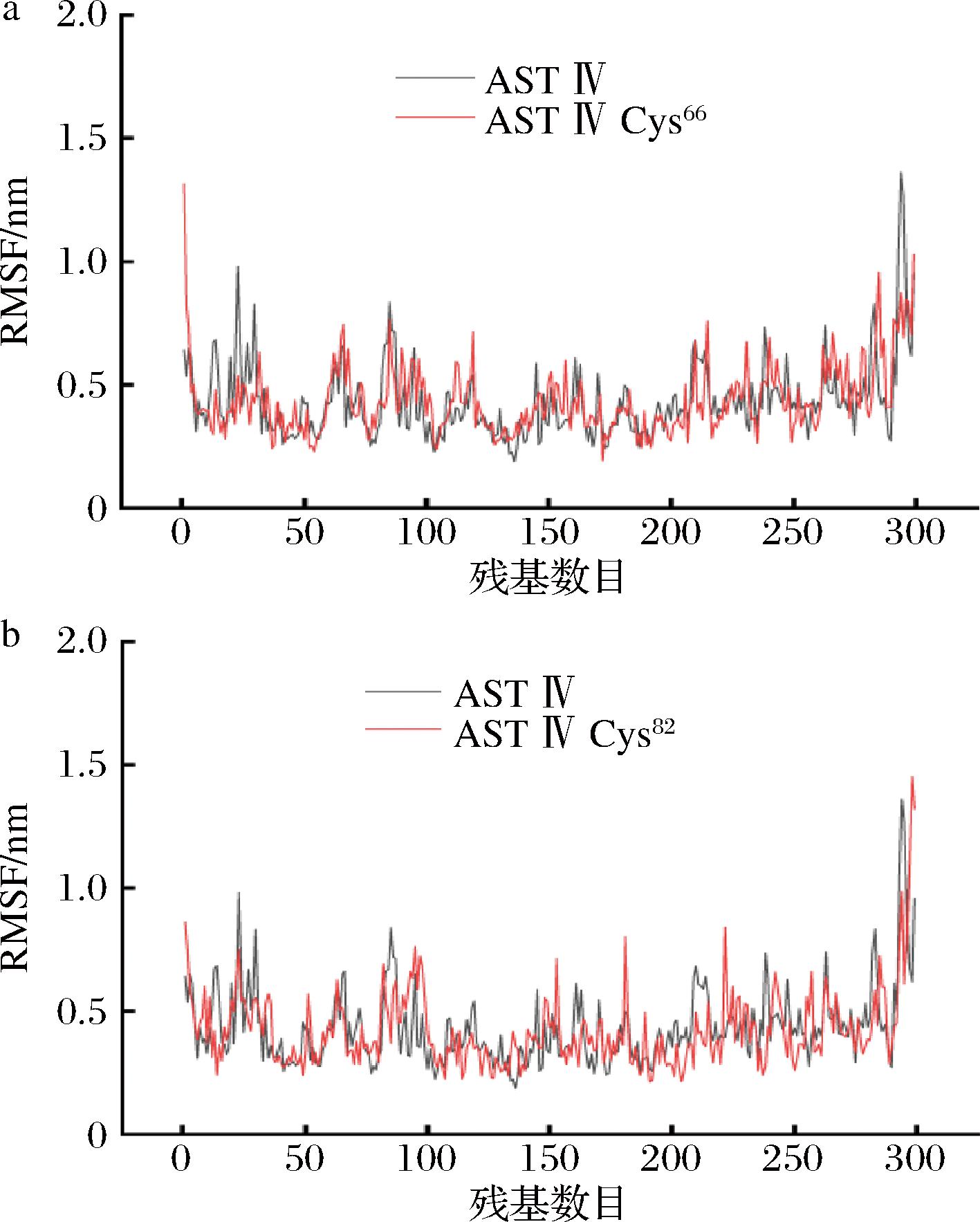
a-AST Ⅳ Cys66;b-AST Ⅳ Cys82
图9 不同蛋白残基的RMSF值
Fig.9 Root mean square fluctuation (RMSF) values of protein residues
3 结论
本研究通过NCBI获取了大鼠AST Ⅳ序列,并进一步优化了表达系统,成功构建了表达载体pET32b-AST Ⅳ。同时,对AST Ⅳ不同位置的半胱氨酸进行了定点突变,包括Cys66、Cys82、Cys232、Cys283、Cys289。并在BL21(DE3)和OrigamiB(DE3)菌种中进行表达,通过添加6×His标签实现了蛋白AST Ⅳ的纯化。
研究结果表明,AST Ⅳ中二硫键的折叠程度对其蛋白活性有影响。具体而言,二硫键的折叠程度越高蛋白的活性越高,AST Ⅳ Cys66、AST Ⅳ Cys82与野生型AST Ⅳ相比,不仅对PAP的亲和力显著降低,Km值分别为(0.89±0.04)、(0.95±0.03) mmol/L低于野生型AST Ⅳ的Km值(0.80±0.02) L/(s·mol),同时催化效率也大幅下降,kcat/Km值分别为(80.53±23.44)、(104.85±25.17) L/(s·mol),低于野生型AST Ⅳ的kcat/Km值(139.13±25.06) L/(s·mol)。通过BL21(DE3)和OrigamiB(DE3)菌种表达定点突变后的AST Ⅳ Cys66、AST Ⅳ Cys82、AST Ⅳ Cys232,AST Ⅳ Cys66和AST Ⅳ Cys82在2种不同菌株中酶活性并无明显变化,而AST Ⅳ Cys232在OrigamiB(DE3)菌种中表达出来的酶活性达到BL21(DE3)菌种表达的AST Ⅳ酶活性的1.2倍。结合分子模拟可以推断在AST Ⅳ中残基Cys66与Cys82之间形成的二硫键对蛋白活性具有重要作用。本研究的发现不仅有助于深入理解AST Ⅳ的结构与功能关系,而且对于高效PAPS再生系统的构建和加速肝素等多糖的生物合成研究具有重要意义。
[1] QIU M,HUANG S J,LUO C H,et al.Pharmacological and clinical application of heparin progress:An essential drug for modern medicine[J].Biomedecine &Pharmacotherapie,2021,139:111561.
[2] HAO C,XU H M,YU L F,et al.Heparin:An essential drug for modern medicine[J].Progress in Molecular Biology and Translational Science,2019,163:1-19.
[3] WARDROP D,KEELING D.The story of the discovery of heparin and warfarin[J].British Journal of Haematology,2008,141(6):757-763.
[4] XU Y M,MASUKO S,TAKIEDDIN M,et al.Chemoenzymatic synthesis of homogeneous ultralow molecular weight heparins[J].Science,2011,334(6055):498-501.
[5] ZHANG X,XU Y M,HSIEH P H,et al.Chemoenzymatic synthesis of unmodified heparin oligosaccharides:Cleavage of p-nitrophenyl glucuronide by alkaline and Smith degradation[J].Organic &Biomolecular Chemistry,2017,15(5):1222-1227.
[6] YU Y Y,GONG B X,WANG H L,et al.Chromosome evolution of Escherichia coli Nissle 1917 for high-level production of heparosan[J].Biotechnology and Bioengineering,2023,120(4):1081-1096.
[7] DATTA P,FU L,HE W Q,et al.Expression of enzymes for 3′-phosphoadenosine-5′-phosphosulfate (PAPS) biosynthesis and their preparation for PAPS synthesis and regeneration[J].Applied Microbiology and Biotechnology,2020,104(16):7067-7078.
[8] AN C Y,ZHAO L,WEI Z J,et al.Chemoenzymatic synthesis of 3′-phosphoadenosine-5′-phosphosulfate coupling with an ATP regeneration system[J].Applied Microbiology and Biotechnology,2017,101(20):7535-7544.
[9] SCHIFF J A.Pathways of assimilatory sulphate reduction in plants and microorganisms[J].Ciba Foundation Symposium,1979(72):49-69.
[10] GRIMSHAW J P A,STIRNIMANN C U,BROZZO M S,et al.DsbL and DsbI form a specific dithiol oxidase system for periplasmic arylsulfate sulfotransferase in uropathogenic Escherichia coli[J].Journal of Molecular Biology,2008,380(4):667-680.
[11] DATTA A K,CHAMMAS R,PAULSON J C.Conserved cysteines in the sialyltransferase sialylmotifs form an essential disulfide bond[J].The Journal of Biological Chemistry,2001,276(18):15200-15207.
[12] HWA J,REEVES P J,KLEIN-SEETHARAMAN J,et al.Structure and function in rhodopsin:Further elucidation of the role of the intradiscal cysteines,cys-110,-185,and-187,in rhodopsin folding and function[J].Proceedings of the National Academy of Sciences of the United States of America,1999,96(5):1932-1935.
[13] LEE J H,BLABER M.Structural basis of conserved cysteine in the fibroblast growth factor family:Evidence for a vestigial half-cystine[J].Journal of Molecular Biology,2009,393(1):128-139.
[14] HORWITZ A H,LEIGH S D,ABRAHAMSON S,et al.Expression and characterization of cysteine-modified variants of an amino-terminal fragment of bactericidal/permeability-increasing protein[J].Protein Expression and Purification,1996,8(1):28-40.
[15] GAO X,DONG X Q,LI X X,et al.Prediction of disulfide bond engineering sites using a machine learning method[J].Scientific Reports,2020,10:10330.
[16] BURKART M D,IZUMI M,WONG C H.Enzymatic regeneration of 3′-phosphoadenosine-5′-phosphosulfate using aryl sulfotransferase for the preparative enzymatic synthesis of sulfated carbohydrates[J].Angewandte Chemie (International Ed),1999,38(18):2747-2750.
[17] XU R R,ZHANG W,XI X T,et al.Engineering sulfonate group donor regeneration systems to boost biosynthesis of sulfated compounds[J].Nature Communications,2023,14:7297.
[18] BERGER I,GUTTMAN C,AMAR D,et al.The molecular basis for the broad substrate specificity of human sulfotransferase 1A1[J].PLoS One,2011,6(11):e26794.
[19] LU J H,LI H T,LIU M C,et al.Crystal structure of human sulfotransferase SULT1A3 in complex with dopamine and 3′-phosphoadenosine 5′-phosphate[J].Biochemical and Biophysical Research Communications,2005,335(2):417-423.
[20] COOK I,WANG T,ALMO S C,et al.The gate that governs sulfotransferase selectivity[J].Biochemistry,2013,52(2):415-424.
[21] ZHOU Z X,LI Q,XU R R,et al.Secretory expression of the rat aryl sulfotransferases IV with improved catalytic efficiency by molecular engineering[J].3 Biotech,2019,9(6):246.