预制菜(预制菜肴)是以一种或多种食用农产品及其制品为原料,添加或不添加调味料等辅料,在不使用防腐剂的前提下,经标准化预加工(如搅拌、腌制、滚揉、成型、炒、炸、烤、煮、蒸等)制成,配以或不配以调味料包,符合产品标签标明的贮存、运输及销售条件,经加热或熟制后方可食用的预包装菜肴[1]。当前,我国预制菜行业正处于快速发展阶段,然而受限于标准体系不完善、监管机制不健全、生产工艺设备落后、创新研发能力不足以及从业人员素质参差不齐等因素,预制菜在食品安全保障方面面临着严峻挑战。
引发预制菜食品安全的因素主要包括微生物污染、添加剂使用不规范、加工过程中产生有毒有害物质等,其中微生物污染是导致食源性疾病最重要的诱因[2-3]。研究表明,在冷链运输过程中,温度波动等因素易导致预制菜受到微生物侵染,进而引发食品腐败变质[4-5]。具体而言,猪肉类制品的优势菌群主要包括假单胞菌属(Pseudomonas)、热杀索丝菌(Brochothrix thermosphacta)和肠杆菌科(Enterobacteriaceae);含鱼类制品在低温环境下主要受腐败希瓦氏菌(Shewanella putrefaciens)和B.thermosphacta等微生物的影响;而甲壳类水产动物在生产贮存过程中易受假单细胞菌等特定腐败菌的污染[6-7],这些微生物污染问题若得不到有效控制,将严重影响预制菜生产企业的产品质量和市场信誉。
目前,预制菜中微生物检测方法主要有生化法、免疫学方法、飞行时间质谱法、高通量测序技术等。自1977年Sanger测序问世以来,微生物检测技术经历了革命性发展[8]。然而,随着研究需求的不断提升,Sanger测序技术的低通量和高成本等局限性日益凸显。近年来,高通量测序(high-throughput sequencing, HTS)技术作为一种快速发展的分子生物学技术,能够在不依赖传统培养方法的情况下,全面解析样品中微生物群落的多样性和组成特征[9-10]。与传统的Sanger测序技术相比,HTS技术具有通量高、成本低、准确性高等显著优势,已成功应用于酸奶、腌渍白菜、玉米等发酵食品和粮食的微生物群落研究[11-13]。基于此,本研究采用16S rDNA高通量测序技术,对市售常见的水产和畜禽类预制菜进行微生物群落分析,旨在探讨不同种类预制菜中微生物菌群结构特征,为预制菜质量控制和食品安全保障提供理论依据和技术支持。
1 材料与方法
1.1 材料与试剂
样品:网购市售预制菜,共计23批,其中鱼肉类预制菜6批、虾肉类预制菜6批次、猪肉类预制菜6批次、鸡肉类预制菜5批次。
蛋白酶K溶液20 mg/mL,Biosharp生物科技公司;无水乙醇,国药集团化学试剂有限公司;氯化钠,广州赛国生物科技有限公司;EDTA(货号1108GR500),广州赛国生物科技有限公司;文库构建试剂盒VAHTS® Universal DNA Library Prep Kit for Illumina V3,VAHTS DNA Adapters set3-set6 for Illumina,南京诺唯赞生物科技股份有限公司。
1.2 仪器与设备
Illumina NovaSeq6000测序仪,美国Illumina公司;Tanon-1600紫外拍胶仪,山西天能科技有限公司;Tissuelyser多样品组织研磨仪,上海净信公司;NanoDrop One spectrophotomete 质检仪器,德国Wilmington公司;Qubit 3.0荧光定量仪,美国Thermo Scientific公司;Gene speed x6微离心机,基因有限公司;H1850台式冷冻离心机,湖南湘仪实验室仪器开发有限公司;VRTEX-5涡旋混匀器,海门市其林贝尔仪器制造有限公司;IA2002电子天平,上海沪析实业有限公司;DYY-6C电泳仪,北京六一生物科技有限公司。
1.3 实验方法
1.3.1 样品处理
预制菜去包装,混合均质,每批样品分别制作3个平行样本,样品信息及分组情况见表1。
表1 样品信息表
Table 1 Sample information

序号样品名称分析样本名称分组名称1分组名称21螺蛳粉酸菜鱼Y1、Y2、Y3ZY12爆香酸菜鱼Y4、Y5、Y6ZY23黑鱼片Y7、Y8、Y9ZY3Y(鱼肉类)4巴沙鱼片Y10、Y11、Y12ZY45黑鱼酸菜鱼Y13、Y14、Y15ZY56三椒水煮鱼Y16、Y17、Y18ZY67麻辣小龙虾X1、X2、X3ZX18新虾麻辣小龙虾X4、X5、X6ZX29麻辣小龙虾X7、X8、X9ZX3X(虾肉类)10龙虾尾X10、X11、X12ZX411十三香味小龙虾X13、X14、X15ZX512麻辣虾尾X16、X17、X18ZX613黄焖鸡J1、J2、J3ZJ114宫保鸡丁J4、J5、J6ZJ215香菇滑鸡J7、J8、J9ZJ3J(鸡肉类)16咖喱鸡肉J10、J11、J12ZJ417歌乐山辣子鸡J13、J14、J15ZJ518邱君扣肉Z1、Z2、Z3ZZ119虎皮梅菜扣肉Z4、Z5、Z6ZZ220梅菜扣肉Z7、Z8、Z9ZZ3Z(猪肉类)21外婆红烧肉Z10、Z11、Z12ZZ422梅菜扣肉Z13、Z14、Z15ZZ523精品梅菜扣肉Z16、Z17、Z17ZZ6
1.3.2 DNA提取
准确称取0.4 g样本于2 mL Bead Tubes中,依据HiPure Soil DNA Mini Kit试剂盒的说明提取样品基因组DNA。使用NanoDrop One spectrophotometer检测DNA纯度,并使用Qubit 3.0荧光计进行DNA定量。
1.3.3 PCR扩增及高通量测序
以稀释后的基因组DNA为模板,使用带Barcode的特异引物进行PCR扩增。采用引物341F(5′-CCTACGGGNGGCWGCAG-3′)和805R(5′-GACTACHVGGGTATCTAATCC-3′)对细菌菌群16S rDNA基因的V3~V4区域序列进行扩增。
PCR扩增条件:98 ℃预变性45 s,98 ℃变性15 s、60 ℃退火30 s,72 ℃延伸50 s,25个循环,最后在72 ℃ 下延伸10 min。测序程序:采用磁珠将扩增产物进行纯化,然后使用Qubit3.0定量,琼脂糖凝胶电泳检测扩增产物条带大小,按照reads数进行pooling混合扩增产物。采用VAHTS® Universal DNA Library Prep Kit 文库构建试剂盒构建文库,文库质检后使用Illumina NovaSeq6000测序仪进行测序。
1.3.4 数据预处理
高通量测序得到的原始图像数据文件经碱基识别(Base Callimg)分析转化为原始数据(Raw reads),应用OIIME软件进行去引物、质量过滤、去噪处理,并检查和剔除嵌合体序列,得到最终有效数据。采用Feature-classifier模块的Fit-classifier-naive-bayes算法对参考数据库进行训练,构建Naive Bayes物种分类器,然后使用Classify-sklearn算法对特征序列进行物种注释。对于细菌、古菌和真核微生物基因,选用Silva数据库(Release138)。
1.4 数据分析
根据操作分类单元(operational taxonomic units,OTU)聚类结果进行丰度处理,在相似性97%的水平上,使用QIIME、Python软件进行不同分类水平上的物种分析,再利用R语言工具,对Alpha、Beta多样性进行评估,探究不同样本或组别间群落结构的差异。
2 结果与分析
2.1 预制菜中微生物多样性分析
2.1.1 OTU统计和特征分析
本研究采用Illumina Nova高通量测序平台对69个预制菜样本进行了二代扩增子测序。在去除低质量序列后经拼接与过滤后得到6 323 428条有效序列,平均碱基长度为221 bp。每个样本至少产生45 351条合格序列。将OTU的代表序列分别与微生物参考数据库Silva和UNITE数据库进行比对,确定各OTU对应的物种分类信息。统计分析结果见表2。
表2 样本各水平OTU物种统计表
Table 2 OTU species statistics at different taxonomic levels in samples

样品种类样品编号门纲目科属种样品种类样品编号门纲目科属种虾肉类X1193696159264127X2193486157262138X3244299167260110X416368411614545X520368011213634X621327910813028X718337611317467X81728608611638X920409914622997X101317457912089X11172967109164101X1217256611016391X13183489145236119X142038101165259136X15203388150235115X161220539113857X171218508712550X181016498412951猪肉类Z136100200289460118Z244108219307459107Z341100202296447118Z41521569012658Z515276410415562Z61728659213643Z71525548712035Z8285312017224770Z921336910619963Z1024408212119852Z1121327711918550Z1221418812816843Z131829618711140Z1420337111719556Z15172762839815Z1624347912320779Z1721317211418363Z181728649011632鱼肉类Y11217447611270Y21216488312069Y31117478412574Y41518468513286Y51420488913289Y61521499614387Y7203471121185110Y819295910115693Y9222980149246143Y10111637628753Y11101540669971Y1291129477146Y1314215010515582Y1419266010415792Y1518245810415269Y16213368121193116Y17182554102180102Y18203167115187111鸡肉类J1914427510671J291234639265J3121841689064J41320508610871J52029599714685J61523508612388J7275112119031394J81423599914342J921357611918151J1021398212722067J113368142227381135J12316012619131778J13192766114201113J14142564128218129J15142363121209132
由表2可知,预制菜样本在不同分类水平(门、纲、目、科、属、种)上所鉴定的OTU数量不同,表明微生物的丰富度在不同分类学等级上存在显著差异。值得注意的是,在种水平识别的OUT数量少于属水平,这可能归因于16S rDNA对亲缘关系较近的物种分辨率不足,导致难以进一步精确分类[14]。
为进一步揭示不同种类预制菜之间共有与特有微生物特征,绘制Venn图(图1)。结果显示,虾肉类、鱼肉类、猪肉类和鸡肉类样本的OTU数目分别为5 335、4 324、11 599、7 600个,其中4种预制菜共有OTU仅为211个,占OTU总数的0.84%(总数25 162)。其中猪肉类特有OTU占总数的40.05%(10 077/25 162),分别是含鱼类、含虾类和含鸡肉类特有OTU的3.56、2.42、1.86倍。这说明猪肉类预制菜的特有微生物明显多于其他类,且四类预制菜均具有各自独特的微生物。
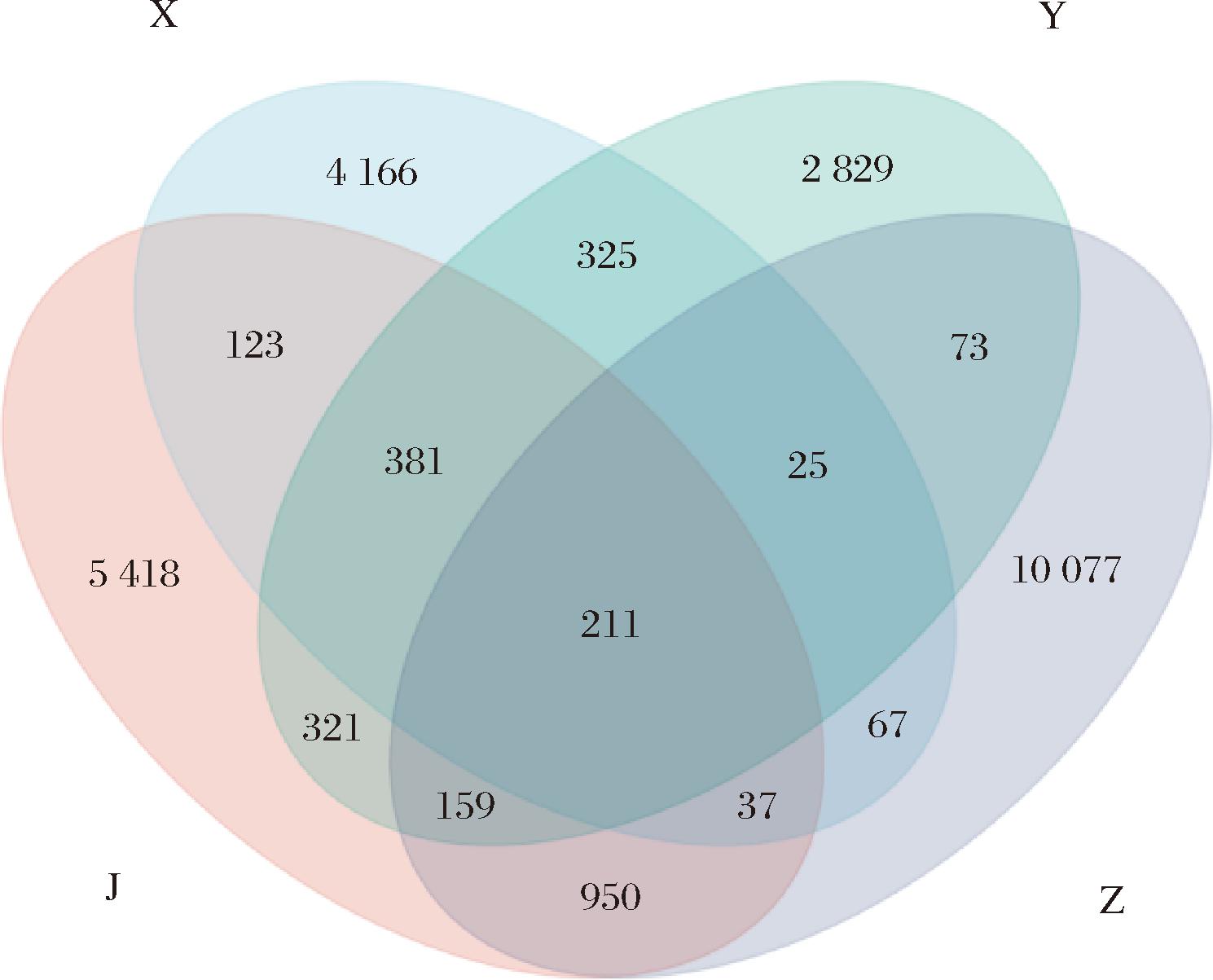
图1 韦恩图
Fig.1 Venn diagram
2.1.2 Alpha多样性分析
Alpha多样性反映的是单个样本的物种丰富度及物种多样性,常用的衡量指标包括ACE指数、Chao1指数、Shannon指数和Simpson指数。ACE和Chao1指数衡量物种丰度即物种数量的多少,指数值越大,样本的物种丰富度越高;Shannon和Simpson指数用于衡量物种多样性,指数值越大,表明样本的物种多样性越高。预制菜微生物群落Alpha多样性分析结果见图2。
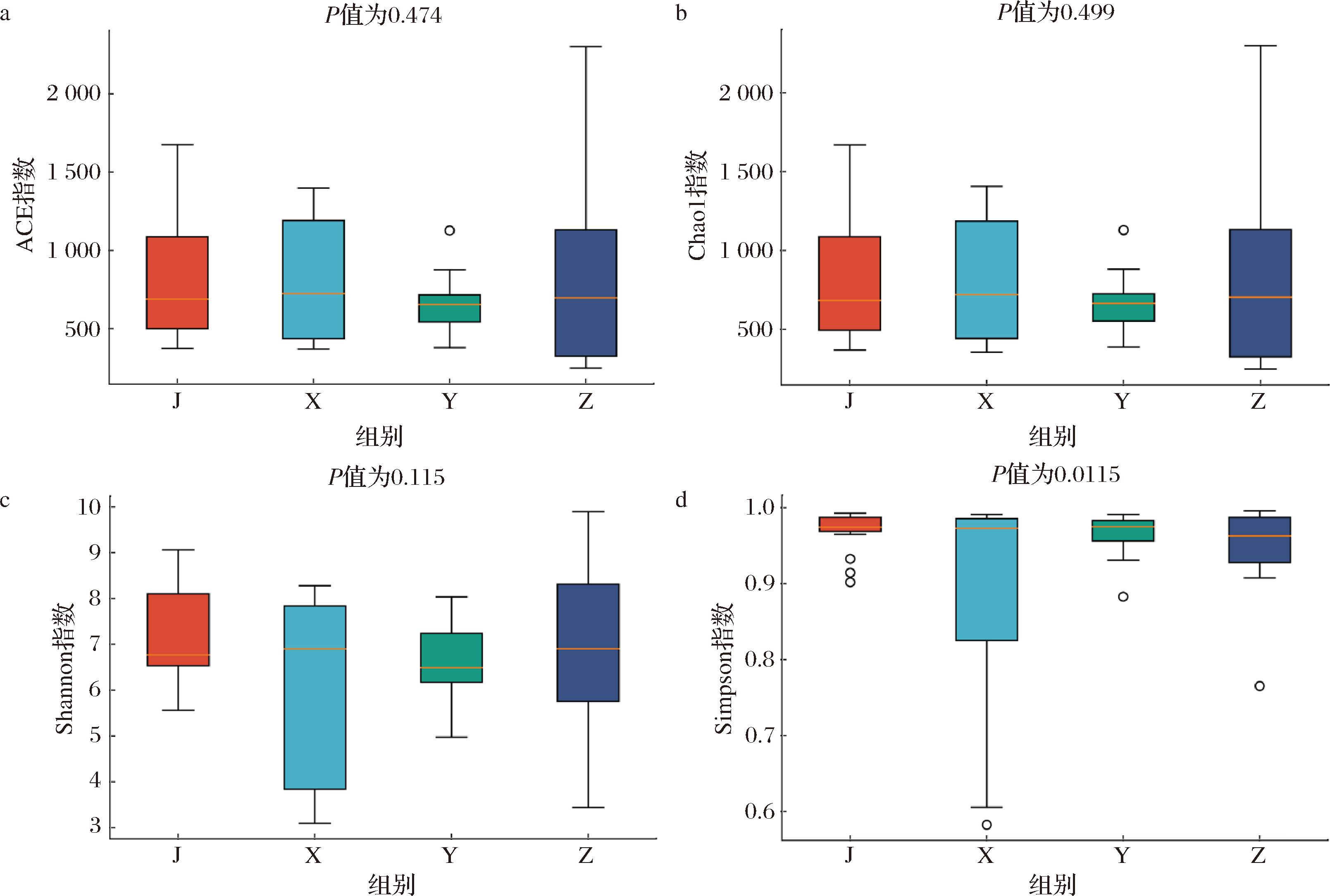
a-ACE指数;b-Chao1指数;c-Shannon指数;d-Simpson指数
图2 四类预制菜的微生物菌群的ACE、Chao1、Shannon、Simpson指数
Fig.2 ACE, Chao1, Shannon and Simpson indices of precooked dishes in four categories
2.1.2.1 ACE和Chao1指数分析
ACE和Chao1指数用于衡量物种丰度即物种数量的多少,ACE、Chao1指数越大,代表物种丰富度越大。由图2可知,四类预制菜的ACE指数排序为:猪肉类(907.00)>鸡肉类(816.43)>虾肉类(804.88)>鱼肉类(662.41);Chao1指数排序为:猪肉类(907.85)>鸡肉类(814.84)>虾肉类(807.43)>鱼肉类(669.18)。由此可看出,猪肉类预制菜的ACE和Chao1指数最大,说明其微生物群落的物种丰富度最高,鸡肉类和虾肉类预制菜的物种丰富度处于中间水平,鱼肉类预制菜的ACE和Chao1指数最低,表明该类型样本的微生物丰富度相对于其他三类预制菜较低。不同预制菜由于原料特性、加工工艺和贮存条件的差异,导致了微生物群落物种丰富度的不同。从原料营养成分分析,猪肉类预制菜含有较高的脂肪含量和水分活性,较其他基质可为多种微生物提供了适宜的生长环境;鸡肉类预制菜的微生物多样性可能受到其相对较低的脂肪含量和加工过程中的热处理影响,限制了某些微生物的生长;虾肉类和鱼肉类预制菜则可能因为其高蛋白和低脂肪的特性,可有效地抑制了微生物的生长和繁殖。此外,这种差异可能还与不同产品的原料质量、加工方式、包装方式和贮存条件等诸多因素有关[15-16]。
2.1.2.2 Shannon指数和Simpson指数分析
Shannon指数和Simpson指数是衡量物种多样性的关键指标。Shannon值越大,说明微生物种类较多;Simpson指数取值范围为0~1,当Simpson指数接近0时,意味着群落物种丰富度和均匀度都很低,整体多样性差。当Simpson指数接近1时,说明群落中存在许多不同的物种,且各个物种的个体数量相对较为均匀,没有明显的优势物种,物种多样性和均匀度都较高,体现出群落具有较高的多样性。由图2可知,四类预制菜的Shannon指数排序为:鸡肉类(7.16)>猪肉类(7.04)>鱼肉类(6.64)>虾肉类(5.99);Simpson指数排序为:鸡肉类(0.968)>鱼肉类(0.966)>猪肉类(0.95)>虾肉类(0.89)。由此可看出,鸡肉类预制菜的微生物多样性最高,表明其微生物群落最丰富且分布最均匀;虾肉类预制菜微生物种类较少且均匀度较低,可能存在优势菌。
2.1.3 Beta多样性分析
本研究使用主成分分析(principal component analysis,PCA)对69个预制菜样本的微生物群落结构间差异进行可视化分析。通过分解方差,将多组数据的差异反映在二维坐标图上。不同样品的距离可以反映样品的差异,2个样品距离越近,则表示组成越相似。由图3可知,PCA1贡献度为58.81%,是区分微生物群落结构的主要维度,PCA2贡献度为12.10%,PCA1和PCA2累计解释了70.91%的微生物群落变异,说明样本的菌群组成存在显著差异,样本分布对分析具有统计学意义。猪肉类和鸡肉类预制菜主要分布与鱼肉类和虾肉类形成明显分离,说明畜禽类预制菜的微生物群落与水产类差异显著;但猪肉类与鸡肉类在PCA2轴也存在距离,表明两者加工工艺或贮存条件的差异导致微生物群落进一步分化。
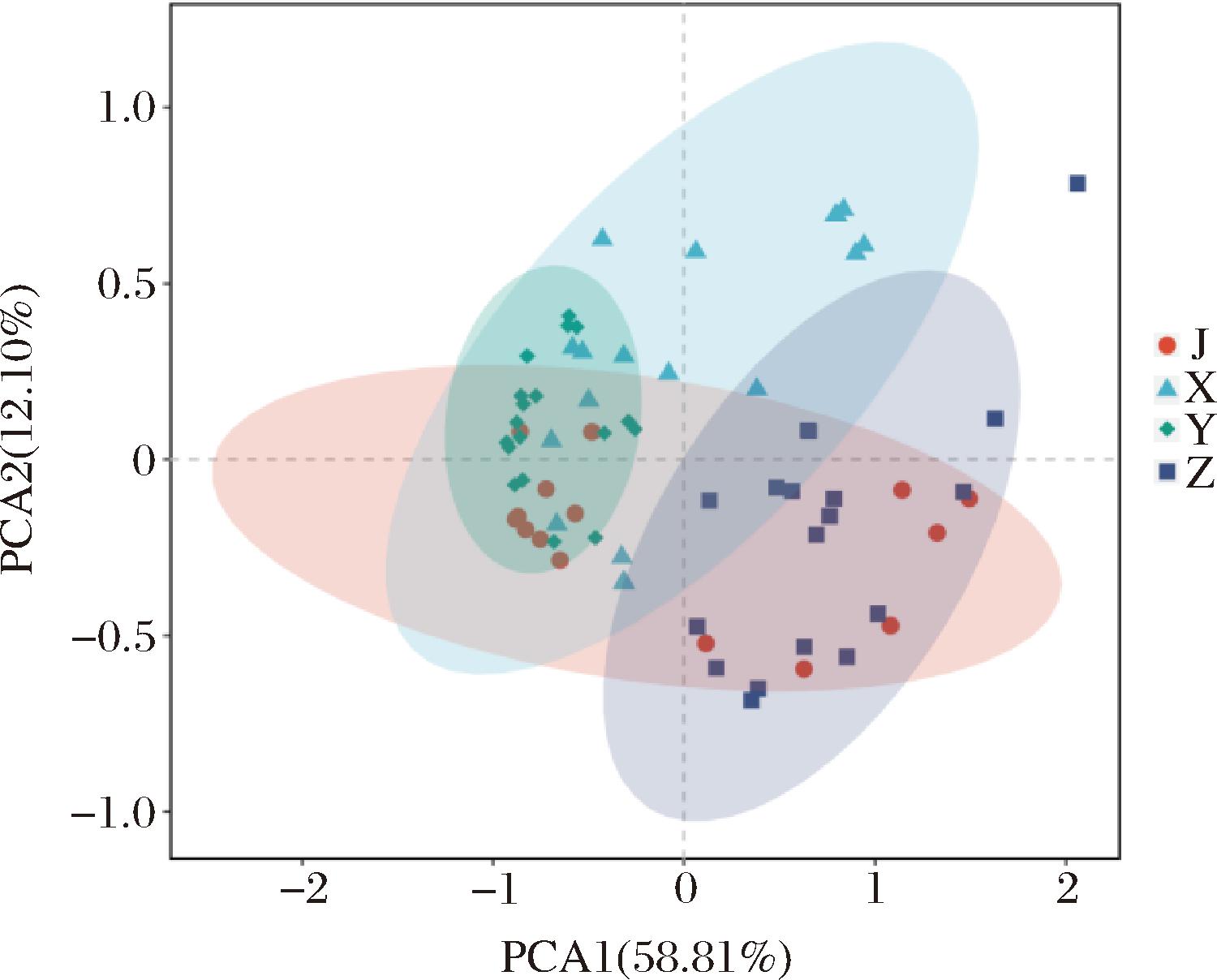
图3 基于weighted_unifrac算法PCA图
Fig.3 PCA diagram based on the weighted_unifrac algorithm
这进一步表明了微生物群落结构可能与其原料种类、加工工艺、贮存条件等因素密切相关。
2.2 微生物菌群结构分析
2.2.1 门水平群落组成
为直观展示不同预制菜样品中微生物群落的组成差异,本研究基于门水平对物种相对丰度进行了柱状图可视化(图4)。为优化视图效果,仅展示丰度排名前10的物种,其余物种统一归类为“others”。
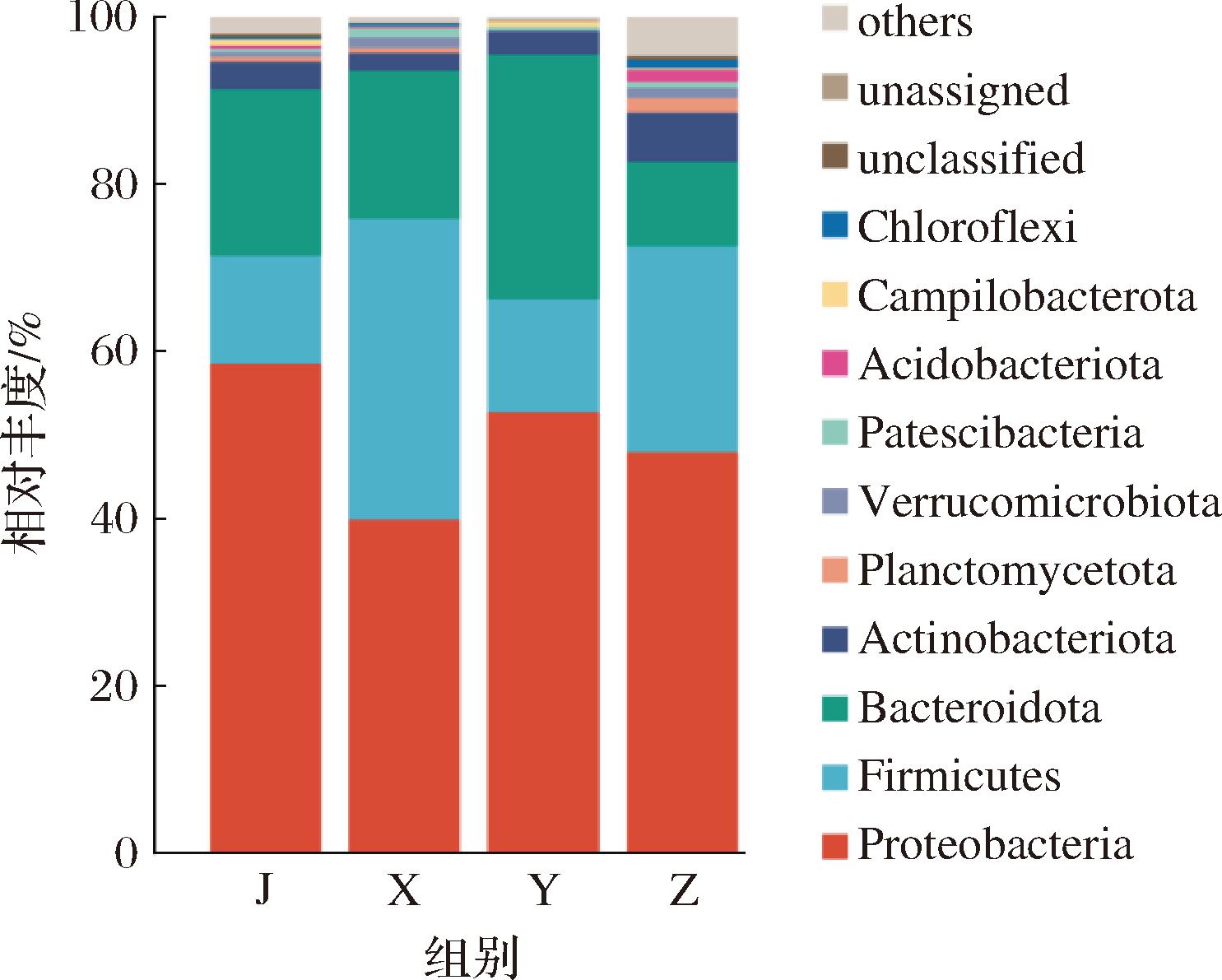
图4 物种相对丰度前10门类柱形图
Fig.4 Histogram of the top 10 phylum in relative abundance of species
从门水平分析结果可以看出(图4),鸡肉类预制菜的菌群以变形菌门(Proteobacteria,58.47%)、拟杆菌门(Bacteroidota,19.84%)和厚壁菌门(Firmicutes,12.98%)为主;虾肉类预制菜的菌群则以变形菌门(39.81%)、厚壁菌门(36.08%)、拟杆菌门(17.67%)为主;鱼肉类预制菜中,变形菌门(52.70%)、拟杆菌门(29.20%)、厚壁菌门(13.56%)占据主导地位;猪肉类预制菜的菌群主要由变形菌门(47.93%)、厚壁菌门(24.65%)、拟杆菌门(10.06%)构成。四类预制菜中变形菌门均为第一优势菌门,但丰度存在差异;虾肉类预制菜中厚壁菌门和鱼肉类预制菜中拟杆菌门丰度突出,显著高于其他组。不同种类预制菜的优势菌群组成表现出明显的多样性,这种差异可能与预制菜的营养成分、加工工艺及原料特性密切相关[17]。据文献报道[18-19],变形菌门是水产动物(如小龙虾肠道化合鱼消化道)中的常见优势菌群。
2.2.2 属水平比较分析
在属水平上对预制菜样品中微生物丰度排名前10的物种进行分析(图5)。结果显示,鸡肉类预制菜中主要优势菌属为不动杆菌属(Acinetobacter,18.90%)、嗜冷杆菌属(Psychrobacter,8.52%)、固氮螺菌属(Azospirillum,5.37%);虾肉类预制菜中主要优势菌属为明串珠菌属(Leuconostoc,10.57%)、不动杆菌属(10.21%)、嗜冷杆菌属(6.67%);鱼肉类预制菜中,主要优势菌属为嗜冷杆菌属(14.88%)、金黄杆菌属(Chryseobacterium,14.65%)、不动杆菌属(9.30%);猪肉类预制菜中,主要优势菌属为不动杆菌属(15.86%)、固氮螺菌属(6.83%)、芽孢杆菌(Bacillus,5.13%)。四类预制菜中优势菌属均含有不动杆菌属,其中鸡肉类预制菜中不动杆菌属丰度最高,虾肉类预制菜含有较高丰度的明串珠菌属,鱼肉类预制菜含有较高丰度的嗜冷杆菌属和金黄杆菌属,猪肉类预制菜含有较高丰度的芽孢杆菌。
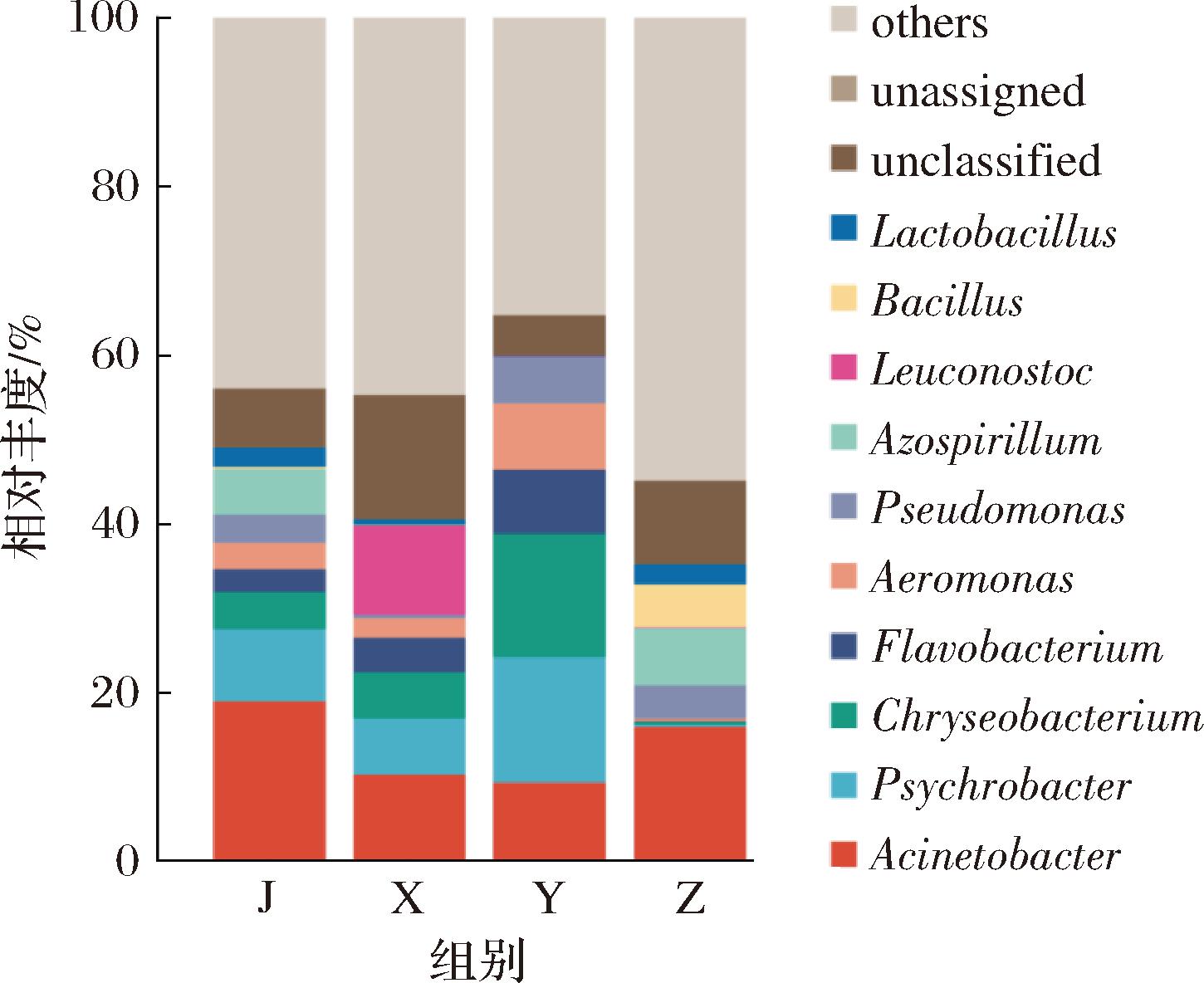
图5 物种相对丰度前10属类柱形图
Fig.5 Histogram of the top 10 genus in relative abundance of species
为进一步分析各类预制菜样品中微生物群落的相似性和差异性,提取了属水平上相对丰度前50的菌群,并绘制了聚类热图(图6)。热图通过颜色变化>和聚类关系直观反映了各样品微生物群落的组成特征。

图6 属水平上物种丰度聚类热图
Fig.6 Clustering heatmap of species abundance on genus
注:横坐标为样本,纵坐标为物种;左侧聚类树代表物种的聚类,上侧聚类树代表样本聚类。不同的颜色代表丰度高低,红色表示物种相对丰度较高,蓝色表示物种相对丰度较低。
从聚类结果来看,猪肉类与鸡肉类预制菜样本的聚类距离较近,它们在微生物群落结构上具有一定的相似性,可能与原料特性、加工工艺较为相似有关,比如它们含有较高丰度的固氮螺菌属,可能在养殖、加工过程中接触到更多的土壤微生物。鱼肉类和虾肉类预制菜样本聚类在一起,但与猪肉类和鸡肉类的样本组距离较远,这可能是因为虾和鱼类属于水产类原料,其蛋白质组成、脂肪类型(富含不饱和脂肪酸)以及水分活性与畜禽肉不同,从而影响微生物群落结构。鱼肉类和虾肉类预制菜优势菌属均有较高丰度嗜冷杆菌属。嗜冷杆菌属隶属于变形菌目,常见于海洋环境、极地地区等低温环境中,也可能存在于冷藏食品中,嗜冷杆菌属中的某些菌种[如肺尖嗜冷杆菌(Psychrobacter pulmonis)和粪嗜冷杆菌(Psychrobacter faecalis)]具有潜在的致病性[20]。
一些低丰度菌属可能在特定条件下对预制菜品质、安全产生重要影响,亦需关注。例如,虾肉类预制菜中检测出的丰度较低的腐败菌包括希瓦氏菌、气单胞菌(Aeromonas)、芽孢杆菌等,这些腐败菌通过分解蛋白质、脂肪和碳水化合物,导致虾肉产生异味、异色和不良质地。检测腐败菌对评估食品安全风险、监控加工卫生和改进保鲜技术具有重要意义,这与毛青秀等[7]的研究结果基本一致;鸡肉类和猪肉类预制菜样品中鉴定出了较低丰度的肠球菌属(Enterococcus),肠球菌能够分解蛋白质,产生氨、硫化氢等异味物质,导致食品腐败变质,食品中检出这种细菌可能是由于生产过程中的卫生条件不佳或交叉污染所致,如屎肠球菌(Enterococcus faecium)可作为冷冻食品的粪便污染指示菌。
3 结论与讨论
3.1 不同种类预制菜微生物多样性
本研究通过高通量测序技术对预制菜样品的微生物多样性进行了分析,共检测到25 162个OTU,表明预制菜中微生物物种丰度较高。然而,不同种类预制菜之间的OTU数量存在显著差异,其中仅有211个OTU为四类预制菜所共有,占总OTU数的0.84%。这一结果表明,不同种类预制菜的微生物组成具有明显的独特性。通过Alpha多样性分析发现,猪肉类预制菜微生物丰富度最高,鱼肉类微生物丰富度最低,鸡肉类微生物多样性最丰富且分布最均匀,虾肉类预制菜微生物种类少且均匀度低。通过Beta多样性分析发现,畜禽类预制菜(猪肉类、鸡肉类)与水产类预制菜(鱼肉类、虾肉类)在PCA图中明显分离,表明原料种类是微生物群落结构差异的核心因素,不同的营养成分为微生物提供了差异化的生长环境,从而导致微生物群落结构和优势菌种的显著差异。例如,虾类和鱼类预制菜富含蛋白质和不饱和脂肪酸[21],而猪肉类预制菜具有饱和脂肪酸和胆固醇含量较高等特征。同时,微生物群落结构还受加工工艺、贮存条件等多因素共同影响。
3.2 不同种类预制菜微生物优势菌群
不同种类预制菜的优势菌群组成也表现出一定程度的相似性和多样性。在门水平上,变形菌门为各类预制菜的第一优势菌门。变形菌门中包含多种与水产品腐败相关的菌属,如嗜冷杆菌属、气单胞菌属和不动杆菌属[22]。这些菌属在冷藏条件下仍具有一定的生长能力,能够分解食品中的蛋白质、脂肪和糖类等营养成分,产生不良气味并改变食品质地,从而导致食品品质下降和货架期缩短。虾肉类预制菜中厚壁菌门和肉类预制菜中拟杆菌门丰度较突出。在属水平上,四类预制菜中优势菌属均含有不动杆菌属,表明其可能广泛存在于肉类预制菜中。其中,猪肉类和鸡肉类预制菜在微生物群落结构上具有一定的相似性,都含有较高丰度的固氮螺菌属,嗜冷杆菌属在鱼肉类和虾肉类预制菜中均表现出较高的丰度,这与冷藏食品中常见的机会致病菌特性一致,而虾肉类预制菜含有较高丰度的明串珠菌属,鱼肉类预制菜含有较高丰度的嗜冷杆菌属和金黄杆菌属,猪肉类预制菜含有较高丰度的芽孢杆菌。值得注意的是,一些低丰度菌属可能在特定条件下对预制菜品质、安全产生重要影响,如虾肉类预制菜中检测出的丰度较低的腐败菌包括希瓦氏菌、气单胞菌、芽孢杆菌,鸡肉类和猪肉类预制菜样品中鉴定出了较低丰度的肠球菌属。
3.3 高通量测序技术在预制菜微生物菌群特征研究中的应用
在微生物种类鉴定领域,传统方法主要依赖于富集、培养、分离纯化,随后通过形态学、生理学和生化实验进行分析。然而,由于培养条件的限制,传统方法难以全面反映预制菜中微生物的真实组成,尤其是未培养微生物的比例往往被低估[23]。相比之下,高通量测序技术能够检测到传统方法无法发现的低丰度微生物种类,从而更全面、准确地揭示预制菜中微生物群落的组成和结构。然而,高通量测序技术也存在一定的局限性,例如16S rDNA高通量测序技术会同时提取预制菜中死菌和活菌的DNA,死菌DNA的存在可能会使测序结果中某些微生物的相对丰度被高估[24],尤其是那些在加工或贮存过程中死亡的微生物。但16S rDNA高通量测序可以提供预制菜中微生物群落的整体组成信息,包括曾经存在过的微生物种类,可以为后续的活菌研究提供重要参考,这对于了解预制菜在加工、贮存和运输过程中微生物群落的历史变化具有重要意义。另一方面,通过分析微生物群落的整体组成,可以识别潜在的腐败菌或致病菌,如有某腐败菌或致病菌的DNA检出,可表明某致病菌其在加工前或加工过程中曾经存在,仍可能提示潜在的食品安全风险,为食品安全控制提供依据。
[1] 惠成章, 刘爱群.我国预制菜产业发展现状及对策[J].辽宁农业科学, 2024(2):69-71.HUI C Z, LIU A Q.The development status and countermeasures of China’s prefabricated vegetable industry[J].Liaoning Agricultural Sciences, 2024(2):69-71.
[2] CANNING M, ABLAN M, CRAWFORD T N, et al.Preparation methods and perceived risk of foodborne illness among consumers of prepackaged frozen vegetables-United States, September 2022.[J].Journal of Food Protection, 2025, 88(2):100440.
[3] 夏琳琳,邱爽,王若彤,等.2011—2020年中国食源性疾病暴发的时空趋势[J].卫生研究,2023,52(2):226-231.XIA L L, QIU S, WANG R T, et al.Foodborne disease outbreaks in China from 2011 to 2020[J].Journal of Hygiene Research, 2023, 52(2):226-231.
[4] 王诗琳,朱慧君,董洪志,等.预制菜产业发展现状与前景分析[J].农产品加工,2023(4):101-104.WANG S L, ZHU H J, DONG H Z, et al.The development status and prospect analysis of pre-made dishes[J].Academic Perodical of Farm Products Processing, 2023(4):101-104.
[5] 王娟,高群玉,娄文勇.我国预制菜行业的发展现状及趋势[J].现代食品科技,2023,39(2):99-103.WANG J, GAO Q Y, LOU W Y.Development status and trends of the pre-prepared food industry in China[J].Modern Food Science &Technology, 2023, 39(2):99-103.
[6] 甄宗圆,胡雪洁,徐留艳,等.肉类微生物多样性分析方法的研究进展[J].生物加工过程,2020,18(3):381-385.ZHEN Z Y, HU X J, XU L Y, et al.Research progress on analytical methods used in meat microbial diversity[J].Chinese Journal of Bioprocess Engineering, 2020, 18(3):381-385.
[7] 毛青秀,彭玲,王建辉,等.甲鱼预制菜加工及贮藏过程中的微生物多样性分析[J].核农学报,2024,38(9):1734-1742.MAO Q X, PENG L, WANG J H, et al.Analysis of microbial diversity of pre-prepared soft-shelled turtle during processing and storage[J].Journal of Nuclear Agricultural Sciences, 2024, 38(9):1734-1742.
[8] 庞煜,马达,王波,等.高通量测序技术在植物内生菌领域研究中的应用[J].生物工程学报,2024,40(10):3395-3406.PANG Y, MA D, WANG B, et al.Application of high-throughput sequencing in research on plant endophytes[J].Chinese Journal of Biotechnology, 2024, 40(10):3395-3406.
[9] AKINTUNDE O, TUCKER T, CARABETTA V J. The Evolution of Next-Generation Sequencing Technologies[M]. High Throughput Gene Screening. New York, NY: Springer US, 2024:3-29.[10] SLATKO B E, GARDNER A F, AUSUBEL F M.Overview of next-generation sequencing technologies[J].Current Protocols in Molecular Biology,2018,122:e59.
[11] BATIONO F, SEYOUM Y, CHOCHOIS V, et al.Bacterial diversity and community structure of some traditional African and European cereal-based fermented foods identified by high-throughput sequencing[J].Food Bioscience,2023,56:103346.
[12] 张咚咚,赵金凤,谢思源,等.基于高通量测序的玉米中微生物多样性分析[J].中国食品学报,2023,23(10):305-314.ZHANG D D, ZHAO J F, XIE S Y, et al.Analysis of microbial diversity in maize based on high throughput sequencing[J].Journal of Chinese Institute of Food Science and Technology, 2023, 23(10):305-314.
[13] 贾晶晶,易洋,韩睿媛,等.高通量测序技术分析腌渍白菜液中原核微生物群落结构[J].中国调味品,2020,45(12):87-90.JIA J J, YI Y, HAN R Y, et al.Analysis of the microbial community structure of prokaryotes in pickled Chinese cabbage liquid by high-throughput sequencing technology[J].China Condiment, 2020, 45(12):87-90.
[14] LUO C, LUO Y, LUO H, et al.High-throughput sequencing identification and multiplex RT-PCR detection of pepper viruses[J].Physiological and Molecular Plant Pathology,2025,136:102555.
[15] 朱迪迪,杨少华,张伟,等.预制菜食品微生物潜在风险点分布及其原因探究[J].山东农业大学学报(自然科学版),2024,55(1):116-122.ZHU D D, YANG S H, ZHANG W, et al.Distribution and causes of potential microbial risk in the prepared dishes industry[J].Journal of Shandong Agricultural University (Natural Science Edition), 2024, 55(1):116-122.
[16] 刘媛,黄宝玲,吴巨贤,等.预制水产品在贮藏过程中的品质劣变机理及调控研究进展[J].热带农业工程,2024,48(2):67-74.LIU Y, HUANG B L, WU J X, et al.Research progress on the mechanisms and regulation of quality deterioration of pre-cooked aquatic products during storage[J].Tropical Agricultural Engineering, 2024, 48(2):67-74.
[17] 邓婕,王秋水,左嘉,等.预制调理食品中的常见微生物风险因子与防控措施[J].分析仪器,2024(3):1-6.DENG J, WANG Q S, ZUO J, et al.Common biosafety issues and control measures in prepared foods[J].Analytical Instrumentation, 2024(3):1-6.
[18] 杨晗月,刘欣,孙正,等.微生物制剂对小龙虾肠道菌群结构的影响[J].饲料研究,2023,46(1):80-84.YANG H Y, LIU X, SUN Z, et al.Effect of probiotics on community structure of crayfish intestinal microbiota[J].Feed Research, 2023, 46(1):80-84.
[19] 徐晟云,陈昆慈,罗青,等.基于16S rRNA高通量测序的鳢肠道微生物群落结构研究[J].大连海洋大学学报,2020,35(5):693-700.XU S Y, CHEN K C, LUO Q, et al.Microbial community in intestine of snakehead based on 16S rRNA high-throughput sequencing[J].Journal of Dalian Fisheries University, 2020, 35(5):693-700.
[20] ISWAREYA LAKSHIMI V, KAVITHA M.Cold-active lipase from Psychrobacter alimentarius ILMKVIT and its application in selective enrichment of ω-3 polyunsaturated fatty acids in flax seed oil[J].Bioprocess and Biosystems Engineering,2025, 48(3):461-481.
[21] GONG H N, WANG B C, JIANG H Y, et al.Quality assessment and microbial community succession of finely chopped common carp (Cyprinus carpio) under low-temperature storage:Implications for nutritional composition and spoilage indicators[J].Food Control,2025,171:111103.
[22] 孟凌云,戴泽川,李娇,等.冷藏小龙虾优势腐败菌的筛选鉴定及细菌群落结构和多样性分析[J].肉类研究,2022,36(6):16-22.MENG L Y, DAI Z C, LI J, et al.Screening and identification of dominant spoilage organisms and analysis of bacterial community structure and diversity in refrigerated crayfish[J].Meat Research, 2022, 36(6):16-22.
[23] 张璟晶,彭庆旺,陈玉勇,等.高通量测序与传统纯培养方法在溱潼鱼丸微生物群落分析中的应用对比研究[J].保鲜与加工,2021,21(6):114-119.ZHANG J J, PENG Q W, CHEN Y Y, et al.Comparative study on application of high throughput sequencing and in microbial traditional pure culture method analysis of Qintong fish balls[J].Storage and Process, 2021, 21(6):114-119.
[24] 曹荣, 张井, 孟辉辉, 等.高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J].食品科学, 2016, 37(24):137-141.CAO R, ZHANG J, MENG H H, et al.Microbial flora analysis of oyster:A comparison between traditional plate culture method and high throughput sequencing[J].Food Science, 2016,37(24):137-141.