乳杆菌(Lactobacillus)作为人体肠道中的重要共生菌的一类,不仅能够调节肠道微生态,还可提高宿主免疫、抗氧化能力、抗癌以及促进营养物质吸收等能力[1-2]。东北酸菜中含有多种优良性能的低温型乳杆菌,可应用到食品和医药等行业中开发出更多益生菌产品。而评价1株乳杆菌是否具有益生功能,首要的标准是其黏附和定植能力,因为这是乳杆菌发挥生理作用的前提条件。在乳杆菌能够抵达胃肠道并开始黏附与定植之前,需耐受胃酸、胆汁等消化液的苛刻环境。在这一过程中,乳杆菌需依次面对口腔中溶菌酶的酶解作用、胃中低pH环境(约2.0~5.0)的酸性胁迫、胆汁的刺激以及胰蛋白酶的蛋白水解作用,同时还需适应肠道的弱碱性条件,这些因素可能会破坏乳杆菌的细胞壁结构,引起细胞内成分的变化,甚至导致乳杆菌死亡[3-5]。因此,乳杆菌必须能够耐受消化道中的不利环境因素,并以足够的活菌数量到达肠道,才能成功黏附并发挥其益生作用。
乳杆菌的黏附和定植能力是评价益生菌的功能特性重要标准之一。黏附过程通常由多种因素相互作用介导,可分为非特异性黏附和特异性黏附[6-7]。非特异性相互作用指氢键、疏水相互作用力和静电相互作用力等物理驱动的作用[8-9]。非特异性黏附通常是微生物与宿主接触的初步阶段,与乳杆菌表面抗原存在联系,这种联系体现在聚集能力、疏水相互作用力以及静电相互作用力[10]。ARGYRI等[11]利用Caco-2细胞模型对发酵橄榄中的菌群进行筛选,成功分离出具有潜在益生特性的乳酸菌株,这为Caco-2细胞模型在体外评估乳杆菌肠道定殖能力的应用提供了实践依据。Caco-2细胞在正常条件下具有明显的极性结构和负电荷,而且乳杆菌黏附筛选也通常采用此种模型[12-13]。因此,深入理解非特异性相互作用与黏附之间的相互作用机制,对于揭示微生物黏附的复杂性、开发新的抗菌策略以及改善宿主的微生物群落平衡具有重要科学意义和临床意义。
虽然已有学者探讨了益生菌黏附能力与其表面特性的关系,但是探讨乳杆菌与宿主细胞产生的不同非特异相互作用力之间相互影响的研究较少。可采用微生物黏附碳氢化合物法(microbial adhesion to hydrocarbons,MATH)模拟乳杆菌与细胞之间产生的疏水相互作用力以及静电相互作用力,以确定的乳杆菌表面特性,包括自聚集、表面疏水和表面电荷与黏附能力之间的相关性。本研究旨在筛选出高黏附乳杆菌,并探讨乳杆菌黏附性与其表面特性之间的关系。
1 材料与方法
1.1 供试菌株、细胞、试剂
菌株:鼠李糖乳杆菌GG(Lacticaseibacillus rhamnosus GG,LGG)来自美国典型培养物保藏中心(American Type Culture Collection, ATCC),保藏编号ATCC 53103。金黄色葡萄球菌(Staphylococcus aureus)来自于中国医学细菌保藏中心(National Center for Medical Culture Collections, CMCC),保藏编号为CMCC(B) 26003。
细胞:Caco-2人结肠腺癌细胞来源于武汉普诺赛生物有限公司。将冷冻保存的细胞置于水浴锅中37 ℃快速解冻,然后将细胞转移到装有5 mL预热的完全培养液[杜氏改良培养基(Dulbecco’s Modified Eagle Medium,DMEM)中分别加入1%(体积分数)双抗和10%(体积分数)血清]的T25瓶中。将细胞接种在T25瓶中,在37 ℃,5%CO2~95%(体积分数)空气中培养至单层。使用1 mL胰蛋白酶消化3~5 min,加入完全培养基使胰酶失活。传代培养(传代比例:1∶3),每2 d更换一次完全培养基。使用胰蛋白酶消化,然后用完全培养基适度稀释,在24孔细胞板中培养至单层。
试剂:MRS肉汤、MRS培养基,北京奥博星生物技术有限公司;青霉素-链霉素的双抗溶液(10 000 units/mL青霉素和10 mg/mL链霉素)、血清、DMEM、胰蛋白酶溶液,上海达特希尔生物科技有限公司;人工唾液、胃液、肠液、猪胃肠道黏蛋白,上海源叶生物科技有限公司;异硫氰酸荧光素酯(fluorescein isothiocyanate, FITC),上海阿拉丁生化科技股份有限公司;赫斯特荧光染料 33342(Hoechst 33342),北京索莱宝科技有限公司。
1.2 仪器与设备
HVA-85高压蒸汽灭菌锅,日本Hirayama公司;BSD-100振荡培养箱,上海博讯实业有限公司;YU-1810紫外可见分光光度计,北京普析通用仪器有限责任公司;激光共聚焦显微镜FV3000,日本奥林巴斯公司。
1.3 实验方法
1.3.1 乳杆菌分离与鉴定
酸菜来自黑龙江省绥化市望奎县敏三乡,取1 mL酸菜汁用无菌PBS(0.01 mol/L, pH 7.4)适度稀释,涂布于MRS-CaCO3固体培养基,在37 ℃无氧条件下恒温培养48 h。采用平板涂布法[14]。在MRS-CaCO3固体培养基上有明显溶钙圈的单菌落进行多次划线分离培养,直到镜检无杂菌,挑选出革兰氏阳性菌株接种斜面,编号并保存。
16S rDNA序列同源性分析;使用细菌基因组试剂盒从乳杆菌中提取总DNA,提取后,使用通用引物(上游引物:5′-AGAGTTTGATCCTGGCTCAC-3′,下游引物:5′-AAGGAGGTGATCCA-3′)进行PCR扩增。将扩增产物发送至吉林库美生物有限公司进行测序,并将单个序列与NCBI网站上的序列进行比较,识别菌株物种。
1.3.2 体外模拟连续消化试验
根据LI等[15]方法略作修改进行体外模拟连续消化实验。将培养18 h的乳杆菌离心(8 000 r/min, 4 ℃, 5 min),无菌PBS(0.01 mol/L, pH 7.4)洗涤2次,之后沉淀重新悬浮于模拟唾液中,37 ℃孵育5 min。然后将沉淀重悬于模拟胃液(pH 3.0)中,37 ℃孵育180 min。最后将菌体沉淀重悬于模拟肠液(添加20 g/L牛胆盐)中,37 ℃孵育120 min。每次孵育后适度稀释采用平板菌落计数法测定乳杆菌的活菌数。
1.3.3 黏附试验
1.3.3.1 猪胃肠黏蛋白模型
参考WU等[5]的方法略作修改进行体外黏附实验。称取一定量的猪胃肠黏蛋白溶于PBS,配置成质量浓度1 mg/mL的黏蛋白溶液。向96孔细胞培养板中加入100 μL黏蛋白溶液,37 ℃孵育60 min后4 ℃ 过夜,然后加入等体积的黏蛋白溶液37 ℃孵育120 min以补足空白位点,在96孔板每孔加入200 μL的109 CFU/mL菌悬液,37 ℃孵育120 min。使用PBS多次洗涤去除未黏附乳杆菌,使用0.1X Triton分离黏附乳杆菌,收集溶液。适当稀释后涂布MRS琼脂板上,培养48 h后计算CFU/mL。按照公式(1)计算黏附率:
黏附率![]()
(1)
式中:X1,黏附前乳杆菌数量;X2,黏附在猪胃肠道黏蛋白上乳杆菌数量。
1.3.3.2 Caco-2细胞模型
采用Caco-2细胞模型对乳杆菌的黏附性能进行评价。
参考ALIZADEH等[16]和JAYASHREE等[17]的方法并略作修改进行体外黏附实验。将培养18 h的乳杆菌使用无抗生素和无血清的DMEM调整菌液浓度至107 CFU/mL,然后与Caco-2细胞单层共孵育120 min。使用PBS洗涤去除未黏附的乳杆菌。使用0.1X Triton溶液,分离黏附的乳杆菌。适当稀释后涂布于MRS琼脂板上,37 ℃培养48 h后计算CFU/mL以确定乳杆菌黏附Caco-2细胞的黏附率。按照公式(2)计算黏附率:
黏附率![]()
(2)
式中:X3,黏附前乳杆菌数量;X4,黏附在Caco-2细胞上的乳杆菌数量。
1.3.4 微生物表面特性评价
1.3.4.1 自聚集能力
根据DEL等[18]的方法并略作修改测定乳杆菌自聚集能力。将培养18 h的乳杆菌离心收集(8 000 r/min, 4 ℃, 5 min)。PBS充分洗涤后,再次重悬于PBS,600 nm波长下调整菌悬液吸光值至0.50±0.02。取4 mL乳杆菌菌液室温放置4 h后测定600 nm下吸光值。按照公式(3)计算乳杆菌自聚集能力:
自聚集能力![]()
(3)
式中:A1,静止前菌悬液吸光值;A2,室温静置4 h后菌悬液吸光值。
1.3.4.2 表面疏水和表面电荷
根据KOS等[9]方法略作修改,采用MATH测定。将培养的乳杆菌离心收集菌体。PBS充分洗涤后再次重悬于0.1 mol/L KNO3溶液(pH 6.2),600 nm波长下调整菌悬液吸光值至0.50±0.02。取3 mL菌悬液分别加入1 mL二甲苯,乙酸乙酯或氯仿,室温下反应10 min,漩涡振荡2 min,再次室温静置30 min。取水相,测定其在600 nm波长下的吸光值。按照公式(4)计算有机试剂对乳杆菌吸附率:
吸附率![]()
(4)
式中:A3,静止前菌悬液吸光值;A4,室温静置30 min后菌悬液吸光值。
1.3.5 相关性分析
使用GraphPad Prism 8.0软件对乳杆菌黏附Caco-2细胞能力与黏附猪胃肠黏蛋白、自聚集能力、表面疏水性和表面电荷等进行相关性分析。
1.3.6 溶血活性
将培养18 h的乳杆菌使用画线法接种在哥伦比亚血琼脂板上[19]。37 ℃培养36 h,观察单菌落周围有无光晕,以LGG为阴性对照,以S.aureus为阳性对照(β溶血)。溶血类型可分为:
(Ⅰ)α溶血:菌落周围产生草绿色半透明溶血环。
(Ⅱ)β溶血:菌落周围出现完全透明的溶血环。
(Ⅲ)γ溶血:菌落周围不产生溶血环。
1.3.7 荧光染色
Caco-2细胞:将Caco-2细胞培养至单层(合流率80%~90%),使用Hoechst(1 μg/mL)染色,在37 ℃,5%CO2~95%空气中孵育30 min。用PBS洗涤去除过量染色剂。
乳杆菌:将乳杆菌悬浮于0.1 mol/L碳酸钠缓冲液中,加入FITC至终浓度2 μg/mL,并在20 ℃下孵育60 min。用PBS洗涤去除过量染色剂,并悬浮于DMEM中。
按照乳杆菌黏附的方法进行实验。在最后孵育后,用PBS洗涤去除未黏附的菌体。使用4%(体积分数)多聚甲醛在室温下固定10 min。然后使用激光共聚焦显微镜采集图像。
1.4 数据分析
该研究中的每个实验均设置3次重复(n=3)。本文数据处理采用SPSS17.0软件,以各数据平均数和标准误差表示。对数据集进行方差分析,以确定数据集之间是否存在显著差异,显著水平为P<0.05。
2 结果与分析
2.1 乳杆菌分离纯化
本实验通过平板涂布法以及16S rDNA同源序列鉴定,从酸菜中分离出47株性状稳定的乳杆菌。其中植物乳植杆菌(Lactiplantibacillus plantarum)30株,分别命名为HS-P1~HS-P30;副干酪乳酪杆菌(Lacticaseibacillus paracasei)12株,分别命名为LP-110~LP-121;鼠李糖乳酪杆菌(Lacticaseibacillus rhamnosus)4株,分别命名为为HM-R2、HM-R3、HM-R7、HM-R9;棒状腐败乳杆菌(Loigolactobacillus coryniformis)1株,命名为HS-LC1。
2.2 乳杆菌消化耐受能力
乳杆菌不仅能为产品带来独特的风味和质地,还可赋予人类和动物健康的功能[20]。乳杆菌主要在肠道中发挥功能,但需先经口腔和胃的消化,保持活力和肠道定植能力对其功能发挥至关重要[21]。本研究通过使从酸菜中分离的47株乳杆菌经过连续的模拟消化液(唾液、胃液和肠液),模拟人体消化过程,探究乳杆菌消化耐受能力。表1为12株耐消化乳杆菌及对照菌株LGG结果,其他乳杆菌结果见电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.042023,下同)。研究结果显示,47株乳杆菌对模拟唾液和胃液均有很好的耐受性。BOVE等[22]认为是由于实验菌株的hsp1基因被模拟唾液刺激而显著上调,提高乳杆菌抗逆性。MANGIA等[23]认为实验乳杆菌在遭受胃部不良环境的胁迫后能够依靠体内稳态机制抵抗,包括调动自身质子移位膜ATP酶和谷氨酸脱羧酶等改善细胞内外酸碱平衡进而维持其活性。而在添加0.2%(质量分数)胆盐的模拟肠液中乳杆菌生存能力显著下降。只有HS-P5、HS-P15、HS-P18、HS-P19、HS-P24、HS-P26、HS-P27、LP-112、LP-116、LP-119、LP-121能够经受模拟肠液的刺激。这可能是由于胆盐可通过插入细菌的细胞膜,破坏其完整性导致。12株乳杆菌表现出耐受性可能是由于乳杆菌表现出胆盐水解酶活性,这种酶能够将胆汁酸解偶联为游离胆汁酸[24]。还有研究显示,在面临渗透压、温度和胆盐等外界刺激时,乳杆菌还会通过增加表面蛋白和多糖等物质以抵御外界刺激[25]。这些因素可能是确保乳杆菌能在连续的模拟消化下存活的原因。
表1 乳杆菌对连续体外消化的耐受能力
Table 1 Lactobacillus tolerance to continuous in vitro digestion
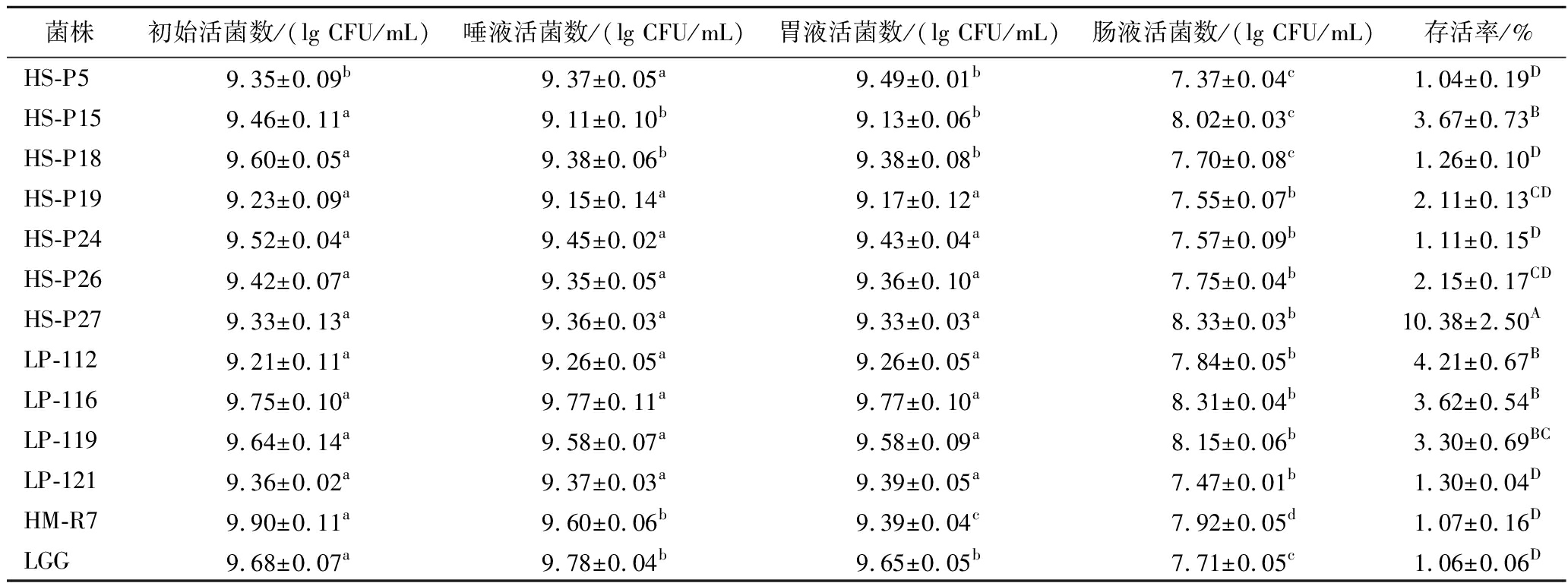
菌株初始活菌数/(lg CFU/mL)唾液活菌数/(lg CFU/mL)胃液活菌数/(lg CFU/mL)肠液活菌数/(lg CFU/mL)存活率/%HS-P59.35±0.09b9.37±0.05a9.49±0.01b7.37±0.04c1.04±0.19DHS-P159.46±0.11a9.11±0.10b9.13±0.06b8.02±0.03c3.67±0.73BHS-P189.60±0.05a9.38±0.06b9.38±0.08b7.70±0.08c1.26±0.10DHS-P199.23±0.09a9.15±0.14a9.17±0.12a7.55±0.07b2.11±0.13CDHS-P249.52±0.04a9.45±0.02a9.43±0.04a7.57±0.09b1.11±0.15DHS-P269.42±0.07a9.35±0.05a9.36±0.10a7.75±0.04b2.15±0.17CDHS-P279.33±0.13a9.36±0.03a9.33±0.03a8.33±0.03b10.38±2.50ALP-1129.21±0.11a9.26±0.05a9.26±0.05a7.84±0.05b4.21±0.67BLP-1169.75±0.10a9.77±0.11a9.77±0.10a8.31±0.04b3.62±0.54BLP-1199.64±0.14a9.58±0.07a9.58±0.09a8.15±0.06b3.30±0.69BCLP-1219.36±0.02a9.37±0.03a9.39±0.05a7.47±0.01b1.30±0.04DHM-R79.90±0.11a9.60±0.06b9.39±0.04c7.92±0.05d1.07±0.16DLGG9.68±0.07a9.78±0.04b9.65±0.05b7.71±0.05c1.06±0.06D
注:同行不同小写字母表示活菌数差异显著(P<0.05),同列不同大写字母表示存活率差异显著(P<0.05)。
2.3 乳杆菌黏附能力
为了研究菌株对肠道黏蛋白和Caco-2细胞的黏附能力,本文采用了平板计数法表征,结果见图1。其中,对猪胃肠黏蛋白的黏附率只有HS-P5菌株显著高于LGG(P<0.05),为(29.50±2.58)%。LP-112、LP-116和LP-121菌株对Caco-2细胞的黏附率显著高于LGG(P<0.05),分别为(19.96±3.39)%、(26.75±6.54)%和(19.48±1.16)%。此外,图2通过荧光染色法更直观地表现出4株高黏附乳杆菌及LGG的黏附效果,细胞周围黏附有大量乳杆菌,且细胞核形态基本完整。其他菌株黏附图片见附图1。考虑到宿主肠道环境的复杂性,肠上皮细胞表面的黏液层会对菌株黏附肠上皮细胞造成空间位阻障碍[25]。对这2种模型进行了深入的相关性分析发现,它们之间存在显著的强正相关(P<0.05),相关系数r=0.633,见图1-C。这表明2个模型在生物学特性或功能上可能具有相似的机制或表现。有研究表明,乳杆菌与其宿主肠道黏蛋白之间的结合主要依赖于菌体表面的3-磷酸甘油醛脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)[26-27]。GAPDH不仅在乳杆菌与肠道黏蛋白的相互作用中发挥关键作用,也是乳杆菌黏附于肠上皮细胞的关键介质[28]。除了有助于乳杆菌黏附肠上皮细胞,还可通过菌株表层蛋白和荚膜多糖等表面物质实现[27]。虽然黏蛋白模型与Caco-2模型在实验结果中显示出显著的相关性,但2种模型之间仍存在一些差异。不同种属的微生物,甚至是同一种属中不同菌株,其表面物质的组成和携带量都可能存在显著差异,这些差异直接影响了它们与宿主细胞或组织黏附的能力[26-27]。这些表面物质与宿主的肠黏蛋白或上皮细胞之间的相互作用可以决定黏附的有效性,并可能影响微生物提供的总体健康益处。
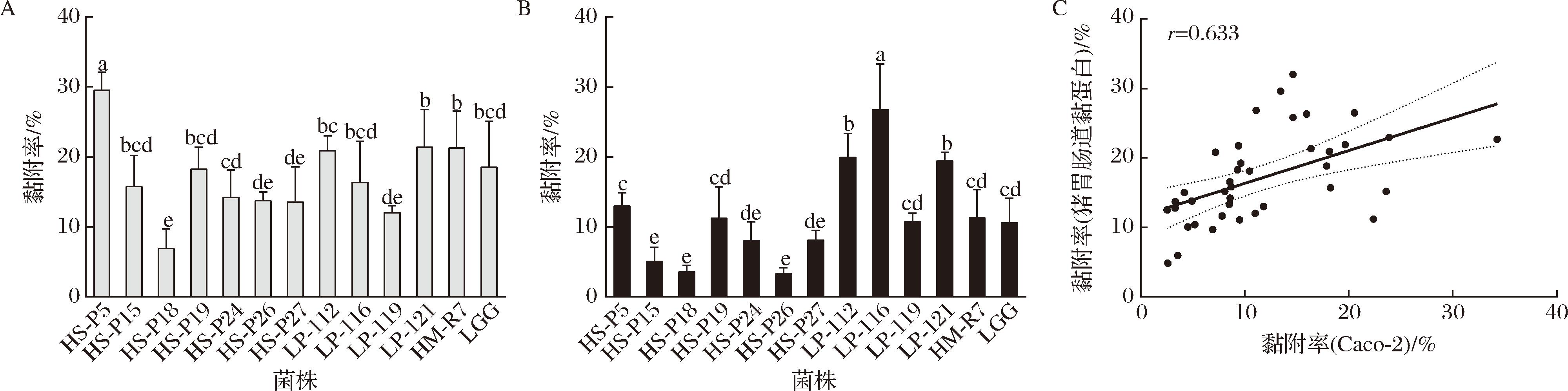
A-乳杆菌黏附猪胃肠道黏蛋白的结果;B-乳杆菌黏附Caco-2细胞的结果;C-乳杆菌对对猪胃肠道黏蛋白与Caco-2细胞黏附能力的相关性分析结果
图1 乳杆菌对黏蛋白和Caco-2细胞黏附能力及相关性分析结果
Fig.1 The adhesion ability of Lactobacillus to mucin and Caco-2 cells and the correlation analysis results
注:不同小写字母表示差异显著(P<0.05)。
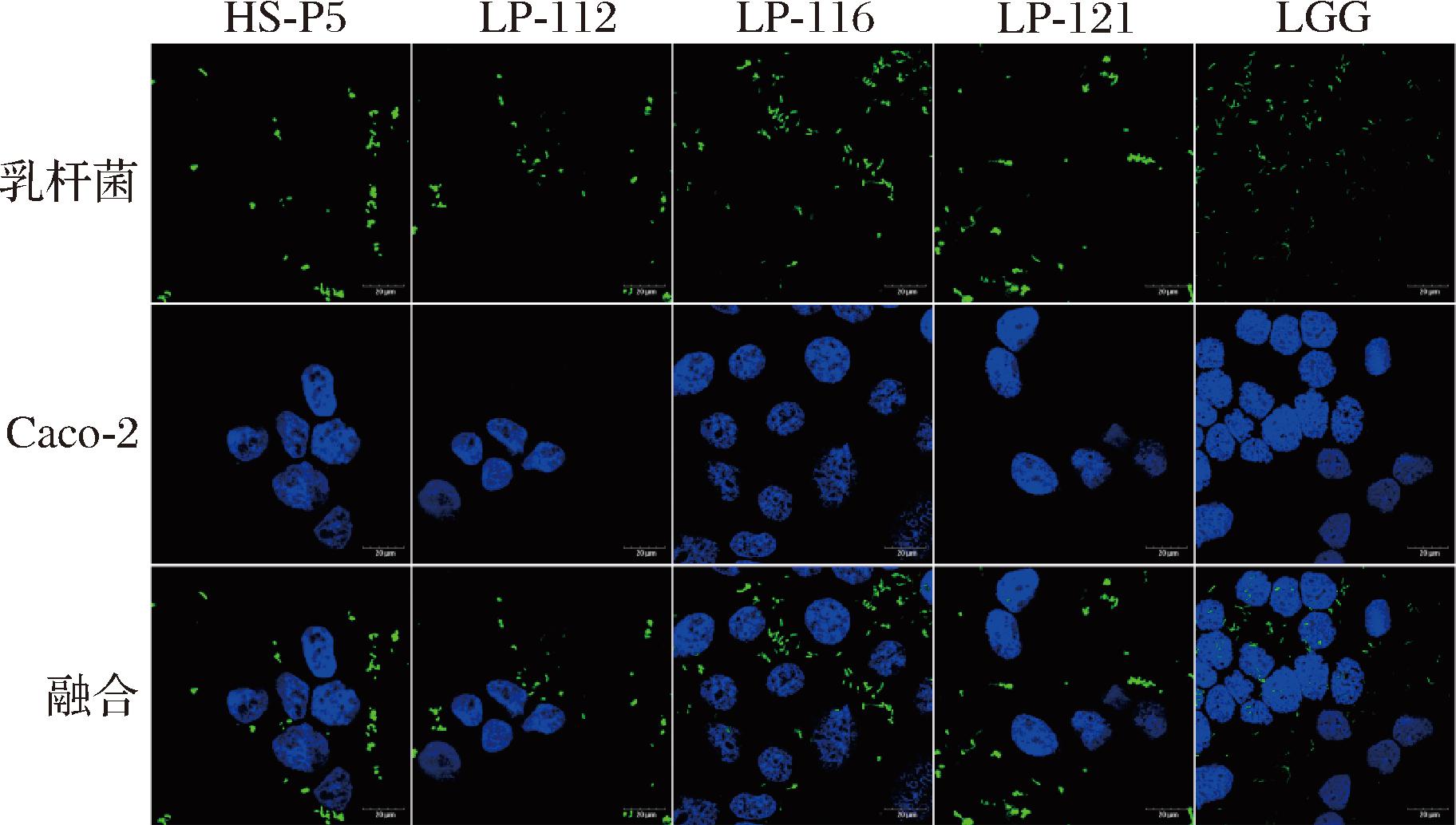
图2 五株乳杆菌与Caco-2细胞孵育2 h
Fig.2 Adherence of 5 Lactobacillus strains incubated with Caco-2 cells for 2 h
注:乳杆菌进行FITC染色(绿色荧光),Caco-2细胞进行Hoechst染色(蓝色荧光)。
2.4 微生物表面特性
为了评价乳杆菌与细胞之间的非特异作用力,采用多种有机溶剂模拟细胞表面性质,即测定乳杆菌表面特性(自聚集能力、表面疏水性和表面电荷),结果如表2所示。不同乳杆菌的自聚集能力呈现显著差异,其中LP-121、LP-112、HS-P26、HS-P19、HS-P5作用效果较好,均显著高于其他菌株(P<0.05)。不同乳杆菌对二甲苯的亲和性(表面疏水)、乙酸乙酯的亲和性(携带负电荷能力)和对氯仿的亲和性(携带正电荷能力)也均呈现显著差异。其中,二甲苯对菌株LP-112、HS-P5、HS-P27、HM-R7、LP-116、LP-119的吸附性显著高于其他菌株(P<0.05);乙酸乙酯对菌株HS-P5的吸附性显著高于其他菌株(P<0.05);氯仿对菌株HS-P27、HS-P5、HM-R7、LP-116、LP-112、HS-P26、LGG、LP-121的吸附性显著高于其他菌株(P<0.05)。
表2 不同乳杆菌的表面特性
Table 2 Surface properties of different Lactobacillus
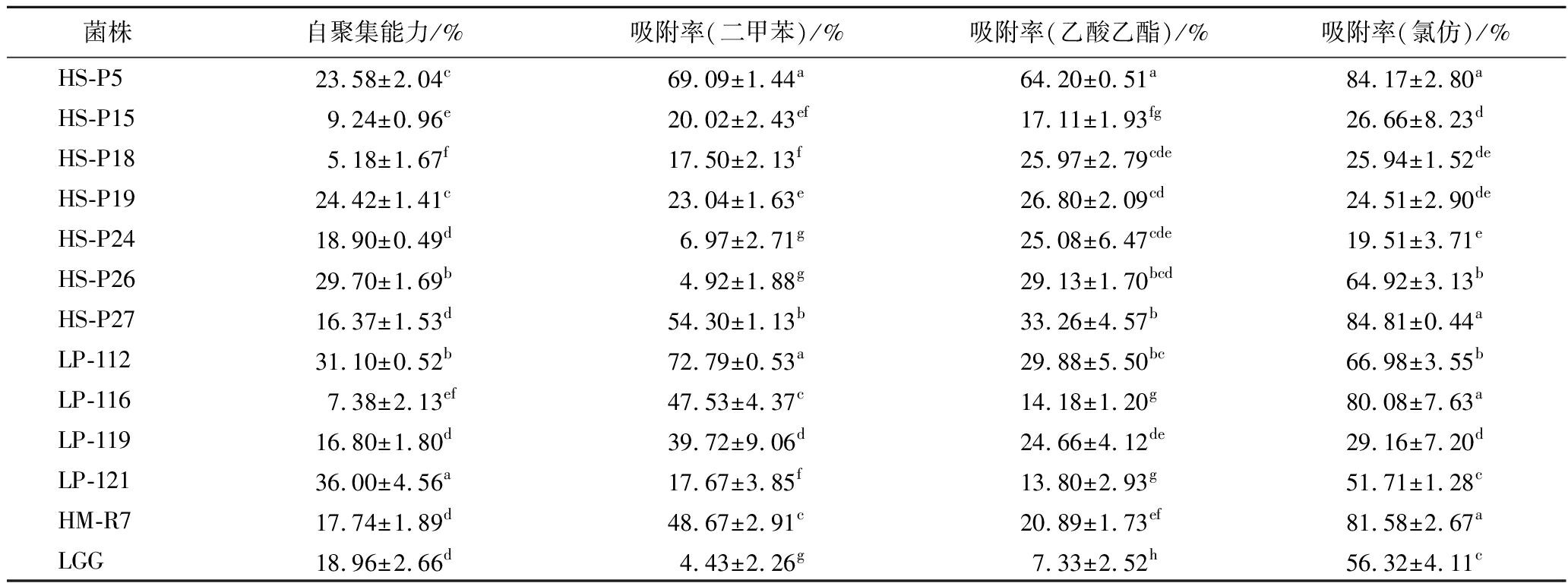
菌株自聚集能力/%吸附率(二甲苯)/%吸附率(乙酸乙酯)/%吸附率(氯仿)/%HS-P523.58±2.04c69.09±1.44a64.20±0.51a84.17±2.80aHS-P159.24±0.96e20.02±2.43ef17.11±1.93fg26.66±8.23dHS-P185.18±1.67f17.50±2.13f25.97±2.79cde25.94±1.52deHS-P1924.42±1.41c23.04±1.63e26.80±2.09cd24.51±2.90deHS-P2418.90±0.49d6.97±2.71g25.08±6.47cde19.51±3.71eHS-P2629.70±1.69b4.92±1.88g29.13±1.70bcd64.92±3.13bHS-P2716.37±1.53d54.30±1.13b33.26±4.57b84.81±0.44aLP-11231.10±0.52b72.79±0.53a29.88±5.50bc66.98±3.55bLP-1167.38±2.13ef47.53±4.37c14.18±1.20g80.08±7.63aLP-11916.80±1.80d39.72±9.06d24.66±4.12de29.16±7.20dLP-12136.00±4.56a17.67±3.85f13.80±2.93g51.71±1.28cHM-R717.74±1.89d48.67±2.91c20.89±1.73ef81.58±2.67aLGG18.96±2.66d4.43±2.26g7.33±2.52h56.32±4.11c
注:同列不同小写字母表示乳杆菌自聚集能力和二甲苯、乙酸乙酯及氯仿对乳杆菌吸附能力等指标有显著差异(P<0.05)。
有研究表明,乳杆菌主要通过2种的黏附机制与宿主细胞建立联系:特异性黏附和非特异性黏附[7]。非特异性黏附通常作为微生物与宿主接触的初步阶段,菌体通过趋化作用接近宿主细胞表面,并通过黏附素与宿主细胞表面发生静电荷相互作用和疏水性相互作用[29]。为了建立乳杆菌黏附性与其非特异性相互作用力之间的联系,将乳杆菌对Caco-2细胞的黏附能力与表面特性进行相关性分析,结果见图3。乳杆菌的黏附能力与其自聚集能力存在中等程度正相关(P<0.05),相关系数r=0.347(图3-A)。具有更强自聚集能力的乳杆菌有助于菌株在肠道上皮细胞表面形成生物膜,从而增强其在肠道中的稳定性和定植能力[30]。黏附能力与疏水性之间存在强相关(P<0.05),r=0.534(图3-B)。黏附能力与乳杆菌携带负电荷能力之间并不存在相关性(r=-0.100)(图3-C)。黏附能力与乳杆菌携带正电荷能力之间存在中等程度相关(P<0.05),r=0.385(图3-D)。具有这些表面特性的微生物可与细胞之间产生趋化作用,即疏水相互作用力和静电相互作用力。乳杆菌和细胞之间更强的疏水相互作用力和静电吸引力,使得趋化作用更为显著,可更有效地识别细胞表面受体。乳杆菌表面具有多种极性或疏水性物质,这些物质赋予了菌体路易斯酸性、路易斯碱性和疏水性等不同的化学性质。正如乳杆菌表面携带的S层蛋白,其亚基通常富含疏水氨基酸,这可能影响着菌体的疏水性[31]。
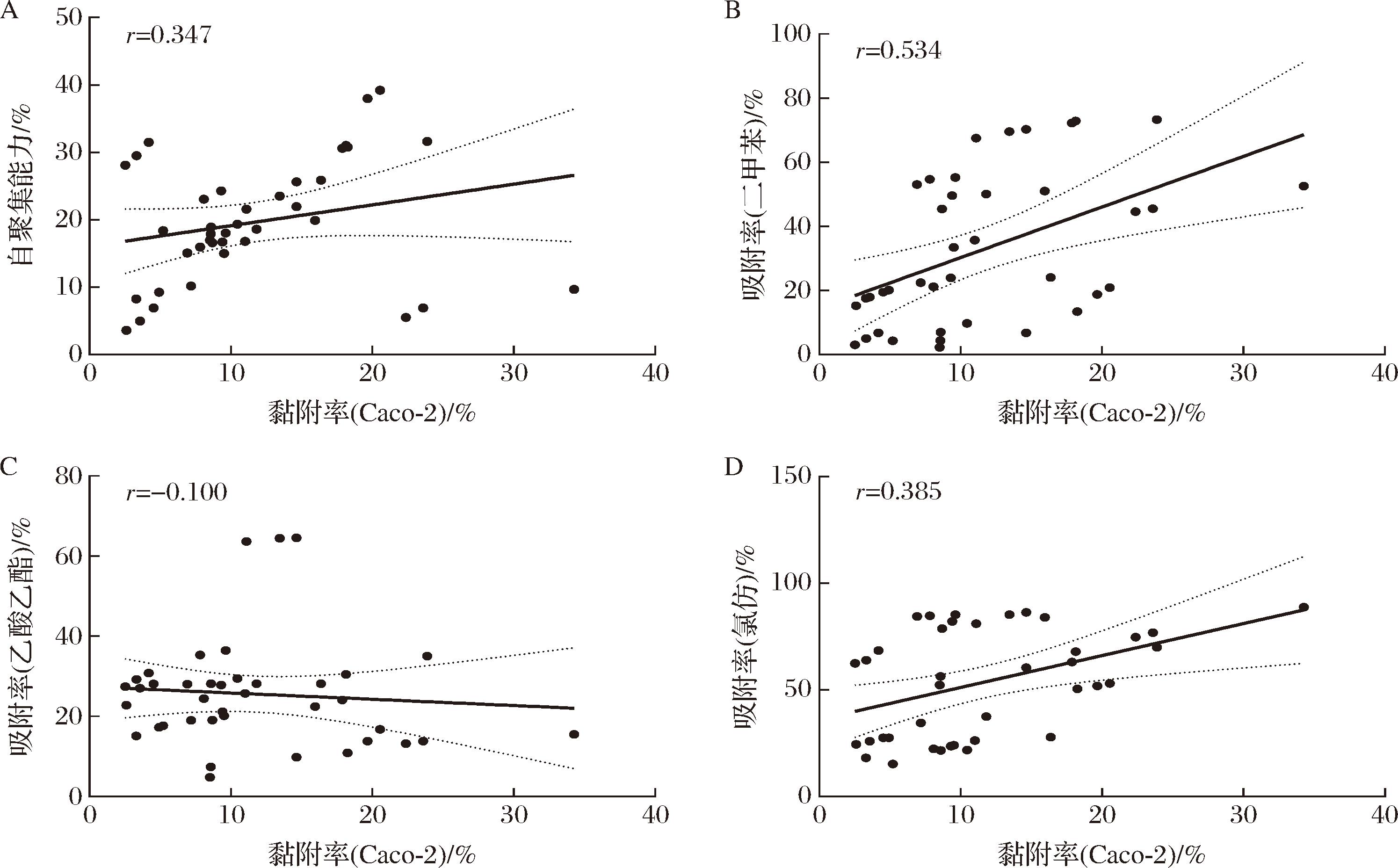
A-乳杆菌黏附能力与自聚集能力的相关性分析;B-乳杆菌黏附能力与对二甲苯吸附的相关性分析;C-乳杆菌黏附能力与对乙酸乙酯吸附的相关性分析;D-乳杆菌黏附能力与对氯仿吸附的相关性分析
图3 乳杆菌对Caco-2细胞的黏附能力与表面特性的相关性分析结果
Fig.3 Results of correlation analysis between adhesion capacity of Lactobacillus to Caco-2 cells and its surface properties
尽管实验结果证实了乳杆菌黏附能力与益生特性之间为正相关,但两者之间仍有差异。HS-P5菌株在自聚集、表面疏水性和静电吸附力(携带正电荷能力)上具有显著优势,然而在细胞黏附能力方面低于LP-112、LP-116和LP-121菌株。这可能是因为HS-P5菌株表面带有丰富的负电荷基团,乳杆菌与细胞间的静电排斥作用削弱了自聚集和疏水作用等产生的趋化作用[32]。以往黏附筛选乳杆菌通常使用细胞模型,但是细胞模型往往成本较高耗时长。先前的研究已经探讨了乳杆菌表面益生特性与黏附能力之间的联系,但这些研究通常只关注了其中的一个方面,并没有进行全面的评估。而本实验通过综合分析发现,虽然益生特性与黏附之间并不存在完全一致性,但是可以作为乳杆菌的初步筛选阶段,大大降低细胞实验的成本。
2.5 乳杆菌溶血活性
乳杆菌的溶血活性被认为是衡量其安全性的重要标准之一[19]。本实验对筛选出的4株高黏附菌株(HS-P5、LP-112、LP-116和LP-121)进行了溶血活性评价,结果见图4。这4株高黏附乳杆菌以及LGG在哥伦比亚血琼脂板上的单菌落周围未出现透明或半透明溶血环,与对照菌株LGG现象一致且未出现阳性对照明显的透明环(β溶血)现象。S.aureus在血琼脂平板上生长速度快于乳杆菌,是因为它可利用血液中的铁离子,并通过溶血素分解血细胞,获得更多营养,从而促进快速生长。表明这4株乳杆菌不具有溶血活性,也意味着菌株在溶血活性方面对于人类是安全的。

图4 四株乳杆菌及对照菌株的溶血活性
Fig.4 Hemolytic activity of four Lactobacillus and control strains
注:LGG的溶血活性为阴性对照,S.aureus的溶血活性为阳性对照。
3 结论
综上,本研究在酸菜中成功分离鉴定出47株乳杆菌,并通过消化模拟反应进一步筛选出12株耐消化的菌株。本实验通过肠道黏蛋白和Caco-2细胞黏附实验,筛选出了HS-P5、LP-112、LP-116和LP-121这4株高黏附性的乳杆菌。12株耐消化菌株的黏附能力与其非特异性相互作用力(包括自聚集能力、表面疏水性和表面电荷)有显著相关性,并且发现4株高黏附菌株拥有出色的表面特性。该研究筛选出4株高黏附力菌株丰富了益生菌资源,为通过微生物表面特性初步筛选高黏附力菌株提供参考。
[1] AMENU DELESA D, JIMMA, BOX E P O. Overview of anticancer activity of lactic acid bacteria[J]. International Journal of Advanced Research in Biological Sciences (IJARBS), 2017, 4(12):166-177.
[2] QAYYUM N, HAN H Y, ISMAEL M, et al. In vitro assessment of antioxidant, antidiabetic, and cholesterol-modulating abilities of lactic acid bacteria: Implications for metabolic health and functional foods[J]. Food Bioscience, 2024, 59:103952.
[3] ERCAN D, DEMIRCI A. Recent advances for the production and recovery methods of lysozyme[J]. Critical Reviews in Biotechnology, 2016, 36(6):1078-1088.
[4] MA J G, WANG W, SUN C B, et al. Effects of environmental stresses on the physiological characteristics, adhesion ability and pathogen adhesion inhibition of Lactobacillus plantarum KLDS 1.0328[J]. Process Biochemistry, 2020, 92:426-436.
[5] WU Z, WANG G, WANG W W, et al. Proteomics analysis of the adhesion activity of Lactobacillus acidophilus ATCC 4356 upon growth in an intestine-like pH environment[J]. PROTEOMICS, 2018, 18(5-6):1700308.
[6] ANTIKAINEN J, ANTON L, SILLANP
 J, et al. Domains in the S-layer protein CbsA of Lactobacillus crispatus involved in adherence to collagens, laminin and lipoteichoic acids and in self-assembly[J]. Molecular Microbiology, 2002, 46(2):381-394.
J, et al. Domains in the S-layer protein CbsA of Lactobacillus crispatus involved in adherence to collagens, laminin and lipoteichoic acids and in self-assembly[J]. Molecular Microbiology, 2002, 46(2):381-394.
[7] ROOS S, JONSSON H. A high-molecular-mass cell-surface protein from Lactobacillus reuteri 1063 adheres to mucus components[J]. Microbiology, 2002, 148(Pt 2):433-442.
[8] ANGELESCU I R, ZAMFIR M, IONETIC E C, et al. The biological role of the S-layer produced by Lactobacillus helveticus 34.9 in cell protection and its probiotic properties[J]. Fermentation, 2024, 10(3):150.
[9] KOS B, ![]() S, et al. Adhesion and aggregation ability of probiotic strain Lactobacillus acidophilus M92[J]. Journal of Applied Microbiology, 2003, 94(6):981-987.
S, et al. Adhesion and aggregation ability of probiotic strain Lactobacillus acidophilus M92[J]. Journal of Applied Microbiology, 2003, 94(6):981-987.
[10] GLEINSER M, GRIMM V, ZHURINA D, et al. Improved adhesive properties of recombinant bifidobacteria expressing the Bifidobacterium bifidum-specific lipoprotein BopA[J]. Microbial Cell Factories, 2012, 11(1):80.
[11] ARGYRI A A, ZOUMPOPOULOU G, KARATZAS K G, et al. Selection of potential probiotic lactic acid bacteria from fermented olives by in vitro tests[J]. Food Microbiology, 2013, 33(2):282-291.
[12] SOPHATHA B, PIWAT S, TEANPAISAN R. Adhesion, anti-adhesion and aggregation properties relating to surface charges of selected Lactobacillus strains: Study in Caco-2 and H357 cells[J]. Archives of Microbiology, 2020, 202(6):1349-1357.
[13] WILLER T, HAN Z F, PIELSTICKER C, et al. In vitro investigations on interference of selected probiotic candidates with Campylobacter jejuni adhesion and invasion of primary chicken derived cecal and Caco-2 cells[J]. Gut Pathogens, 2024, 16(1):30.
[14] YUE X Q, LI X, WU J R, et al. Isolation and identification of Lactobacillus from naturally fermented sauerkraut juices in Xifeng[J]. Advanced Materials Research, 2013, 726-731:147-150.
[15] LI H Y, PENG F, LIN J X, et al. Preparation of probiotic microcapsules using gelatin-xylooligosaccharides conjugates by spray drying: Physicochemical properties, survival, digestion resistance and colonization[J]. Food Bioscience, 2023, 52:102462.
[16] ALIZADEH BEHBAHANI B, NOSHAD M, FALAH F. Inhibition of Escherichia coli adhesion to human intestinal Caco-2 cells by probiotic candidate Lactobacillus plantarum strain L15[J]. Microbial Pathogenesis, 2019, 136:103677.
[17] JAYASHREE S, KARTHIKEYAN R, NITHYALAKSHMI S, et al. Anti-adhesion property of the potential probiotic strain Lactobacillus fermentum 8711 against methicillin-resistant Staphylococcus aureus (MRSA)[J]. Frontiers in Microbiology, 2018, 9:411.
[18] DEL RE B, SGORBATI B, MIGLIOLI M, et al. Adhesion, autoaggregation and hydrophobicity of 13 strains of Bifidobacterium longum[J]. Letters in Applied Microbiology, 2000, 31(6):438-442.
[19] OU D X, LING N, WANG X H, et al. Safety assessment of one lactiplantibacillus plantarum isolated from the traditional Chinese fermented vegetables: Jiangshui[J]. Foods, 2022, 11(15):2177.
[20] FENG J, CEN Q Y, CUI Y R, et al. Lactobacillus rhamnosus: An emerging probiotic with therapeutic potential for depression[J]. Pharmacological Research, 2025, 211:107541.
[21] CABELLO-OLMO M, ARA A M, URTASUN R, et al. Role of postbiotics in diabetes mellitus: Current knowledge and future perspectives[J]. Foods, 2021, 10(7):1590.
A M, URTASUN R, et al. Role of postbiotics in diabetes mellitus: Current knowledge and future perspectives[J]. Foods, 2021, 10(7):1590.
[22] BOVE P, RUSSO P, CAPOZZI V, et al. Lactobacillus plantarum passage through an oro-gastro-intestinal tract simulator: Carrier matrix effect and transcriptional analysis of genes associated to stress and probiosis[J]. Microbiological Research, 2013, 168(6):351-359.
[23] MANGIA N P, SALIBA L, DEIANA P. Functional and safety characterization of autochthonous Lactobacillus paracasei FS103 isolated from sheep cheese and its survival in sheep and cow fermented milks during cold storage[J]. Annals of Microbiology, 2019, 69(2):161-170.
[24] HERN NDEZ-G
NDEZ-G MEZ J G, L
MEZ J G, L PEZ-BONILLA A, TREJO-TAPIA G, et al. In vitro bile salt hydrolase (BSH) activity screening of different probiotic microorganisms[J]. Foods, 2021, 10(3):674.
PEZ-BONILLA A, TREJO-TAPIA G, et al. In vitro bile salt hydrolase (BSH) activity screening of different probiotic microorganisms[J]. Foods, 2021, 10(3):674.
[25] YANG M X, LIANG X H, SONG X Y, et al. Proteomic analysis of milk fat globule membrane protein modulation of differently expressed proteins in Lactobacillus plantarum under bile salt stress[J]. Journal of Agricultural and Food Chemistry, 2024, 72(23):13125-13137.
[26] KINOSHITA H, UCHIDA H, KAWAI Y, et al. Cell surface Lactobacillus plantarum LA 318 glyceraldehyde-3-phosphate dehydrogenase (GAPDH) adheres to human colonic mucin[J]. Journal of Applied Microbiology, 2008, 104(6):1667-1674.
[27] LIANG A J, WANG J L, DING L L, et al. Probiotic properties, whole-genome sequence analysis, and safety assessment of BreviBacillus borstelensis S8[J]. LWT, 2024, 210:116800.
[28] ASHIDA N, YANAGIHARA S, SHINODA T, et al. Characterization of adhesive molecule with affinity to Caco-2 cells in Lactobacillus acidophilus by proteome analysis[J]. Journal of Bioscience and Bioengineering, 2011, 112(4):333-337.
[29] ELLEN R P, LÉPINE G, NGHIEM P M. In vitro models that support adhesion specificity in biofilms of oral bacteria[J]. Advances in Dental Research, 1997, 11(1):33-42.
[30] MILJKOVIC M, BERTANI I, FIRA D, et al. Shortening of the Lactobacillus paracasei subsp. paracasei BGNJ1-64 AggLb protein switches its activity from auto-aggregation to biofilm formation[J]. Frontiers in Microbiology, 2016, 7:1422.
[31] GERBINO E, CARASI P, MOBILI P, et al. Role of S-layer proteins in bacteria[J]. World Journal of Microbiology and Biotechnology, 2015, 31(12):1877-1887.
[32] PIWAT S, SOPHATHA B, TEANPAISAN R. An assessment of adhesion, aggregation and surface charges of Lactobacillus strains derived from the human oral cavity[J]. Letters in Applied Microbiology, 2015, 61(1):98-105.