微生物细胞工厂已被用于生产维生素、甜味剂和氨基酸等食品配料[1]。然而天然的微生物细胞通常倾向于将更多的代谢流用于生长而非产物的合成,严重限制了目标产物的合成效率[2]。因此,如何合理分配细胞生长与产物合成之间的代谢流,是构建高效细胞工厂亟待解决的关键科学问题。近年来,国内外已开发了多种基因表达调控元件用于调节细胞代谢流,如启动子[3]、核糖体结合位点[4]和终止子等。各个表达调控元件又可以彼此耦合,形成基因线路,协同调控胞内多个基因的表达,从而全局调控各代谢途径的通量[5]。在众多基因表达调控系统中,CRISPR(clustered regularly interspaced short palindromic repeats)-Cas系统因具有编程性高、可同时调控多基因表达等优势,已被广泛用于代谢流的精准控制[6]。
CRISPR-Cas系统中的Cas蛋白能够在crRNA的引导下,靶向目标DNA位点,造成DNA双链断裂,因此常被用于基因组编辑。将Cas蛋白突变,使其失去DNA水解酶活性成为dCas(dead Cas)蛋白,所生成的CRISPR-dCas系统则被广泛用于基因表达调控。将dCas蛋白与转录激活结构域(activation domain,AD)融合,所形成的复合体能够在crRNA的引导下靶向目标基因的启动子区域,招募更多的RNA聚合酶,从而强化下游基因的转录。相反的,将dCas蛋白与转录抑制结构域融合,则可降低下游基因的转录水平。相较于传统的调控元件,CRISPR-dCas系统可以通过设计靶向不同目标基因的crRNA阵列,同时调控多个基因的表达,实现代谢网络的全局调控,从而提高目标产物的产量[7-8]。然而,目前在革兰氏阳性菌中构建的CRISPRa调控系统仍存在激活效果较差,难以实现多靶点的原位调控等问题。例如,枯草芽孢杆菌(Bacillus subtilis)作为一种重要的模式工业微生物,具有遗传背景清晰、遗传改造工具完善和蛋白分泌能力强等优点,已被用于生产多种高附加值产物[9],如N-乙酰神经氨酸[5]、多聚谷氨酸[10]等。DONG等[11]和FONTANA等[12]认为在原核生物中构建激活系统还需要更明确的转录激活因子,以及更为明确的激活位点,在枯草芽孢杆菌中报道的具有明确效果的转录激活结构域较少。
在本文中,基于CRISPR-Cas12a(Cpf1)系统,在枯草芽孢杆菌中开发了一种高效的多靶点原位转录调控系统。首先,通过将不同来源的AD与dCpf1的N端融合,筛选得到了2个能够实现多靶点基因激活的dCpf1-RemA和dCpf1-RpoZ复合体,构建了CRISPRa系统。此外,基于dCpf1-RemA和dCpf1-RpoZ复合体,通过改变crRNA位点,构建了抑制基因表达的CRISPRi系统,实现了多基因的双向调控。随后,通过构建蛋白支架、调节dCas蛋白表达量等方法提高了CRISPRa系统的激活效果。最后,使用上述系统原位调控枯草芽孢杆菌中核黄素代谢网络关键基因的表达,通过激活基因zwf和ribA的同时抑制基因ribC的表达,使核黄素产量的提高了3.5倍。该研究为枯草芽孢杆菌的代谢网络调控提供了新工具。
1 材料与方法
1.1 材料与试剂
1.1.1 药品和试剂
本研究中使用的化学物质,生工生物(上海)股份有限公司;T4 DNA连接酶、PrimeSTAR HS DNA聚合酶,宝日医生物技术(北京)有限公司;Taq master mix(Dye Plus)、Phanta Max Master Mix(Dye Plus)、随机突变试剂盒,南京诺唯赞生物科技股份有限公司;无缝克隆试剂盒,上海碧云天生物技术公司;Eco31I酶、质粒提取试剂盒、限制性内切酶、DNA凝胶纯化试剂盒,美国Thermo Scientific;引物合成由苏州GENEWIZ合成并购买。对于常见的细菌培养,使用Luria-Bertani(LB)培养基。大肠杆菌(Escherichia coli)筛选使用氨苄青霉素(100 μg/mL)和卡那霉素(50 μg/mL)。枯草芽孢杆菌筛选使用卡那霉素(50 μg/mL)和氯霉素(5 μg/mL)。
1.1.2 培养基
LB培养基(g/L):胰蛋白胨10、酵母提取物5和NaCl 5。121 ℃ 20 min灭菌。
核黄素发酵培养基(g/L):葡萄糖40,酵母粉7.5,MgSO4·7H2O 1,尿素2,pH 7.2。121 ℃ 15 min灭菌。
1.1.3 质粒和菌株
表1列出了本研究中使用的菌株和质粒。质粒构建均使用大肠杆菌JM109菌株作为宿主,并根据厂家的说明使用T4 DNA连接酶。表2列出了本研究中蛋白支架的氨基酸序列。
表1 本研究中使用的菌株和质粒
Table 1 Strains and plasmids used in this study

菌株/质粒特征来源菌株E.coli JM109recA1, endA1, thi, gyrA96, supE44, hsdR17Δ (lac-proAB)/FΔ[traD36,proAB+, lacΔq, lacZΔ M15]实验室保存B.subtilis 168实验室保存BS-PribT-GFPB.subtilis 168 derivate-PribT-sfGFP本研究BS-PydfK-mCherryB.subtilis 168 derivate,PydfK-mCherry本研究BS-PpdhC-EB.subtilis 168 derivate,PpdhC-erythromycinr本研究BS-PclpQ-ZB.subtilis 168 derivate,PclpQ-Zeocinr本研究BS--PpdhC-E-PclpQ-Z-PribT-GFP-PydfK-mCherryB.subtilis 168 derivate, PpdhC-erythromycinr-PclpQ-Zeocinr-PribT-sfGFP-PydfK-mCherry本研究构建CRISPRa/i系统的质粒pHT01ColE1 Ampr, RepA Cmr, E.coli-B.subtilis shuttle vector[13]pP43 NMKColE1 Ampr, RepU Kanr, E.coli-B.subtilis shuttle vector[13]pHT-XCR6ColE1 Ampr, RepA Cmr, XylR-PxylA-FnCpf1-NgAgo[7]pcrF19 NM2ColE1, pE194ts Kanr, E.coli-B.subtilis shuttle vector, crRNA insertion under Pveg[14]pcrF20 NM2ColE1, Kanr, E.coli-B.subtilis shuttle vector, crRNA insertion under Pveg[14]pHT-dXCR6-AsiAColE1 Ampr, RepA Cmr, LcaI-Pgrac100-dCpf1-Asia本研究pHT-dXCR6-P4ColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-P4本研究pHT-dXCR6-RemAColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-RemA本研究pHT-dXCR6-MalRColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-MalR本研究pHT-dXCR6-RpoZColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-RpoZ本研究pHT-dXCR6-SoxSColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-SoxS本研究构建蛋白支架的质粒pHT-dXCR6-Suntag-RemAColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-Suntag, Pveg-scVF-RemA[15]pHT-dXCR6-Spy-RemAColE1 Ampr, RepA Cmr, LacI-Pgrac100-dCpf1-SpY, Pveg-SpyCatcher-RemA[16]
表2 蛋白支架的氨基酸序列
Table 2 Amino acid sequence of protein scaffold

蛋白支架名称氨基酸序列Sun tagGCN4EELLSKNYHLENEVARLKKscVFMDPIVDMGPDIVMTQSPSSLSASVGDRVTITCRSSTGAVTTSNYASWVQEKPGKLFKGLIGGTNNRAPGVPSRFSGSLIG-DKATLTISSLQPEDFATYFCALWYSNHWVFGQGTKVELKRGGGGSGGGGSGGGGSSGGGSEVKLLESGGGLVQPGGSL-KLSCAVSGFSLTDYGVNWVRQAPGRGLEWIGVIWGDGITDYNSALKDRFIISKDNGKNTVYLQMSKVRSDDTALYYCV-TGLFDYWGQGTLVTVSSYPYDVPDYAGGGGGSGGGGSGGGGSGGGGSLDPGGGGSGSpy TagSpyCatcherAHIVMVDAYKPTKSpy tagMSYYHHHHHHDCDIPTTENLYFQGAMVDSATHIKFSKRDIDGKELAGATMELRDSSGKTSTWISDGQVKDFYLMPGK-YTFVETAAPDGYEVATAITFTVNEQGQVTVNGKATKGDAHAVMVAA
枯草芽孢杆菌168菌株用作起始宿主菌株。所有菌株都是使用WU等[7]之前描述的方法构建的,其中包括创建CRISPR RNA阵列和在CRISPR/Cpf1的帮助下编辑基因组的技术。
1.2 仪器与设备
CLS3603 96孔板,美国Corning公司;Titramax 1000 微孔板振荡器,德国Heidolph公司;SpectraMax M多功能酶标仪,美国Biotek公司。
1.3 实验方法
1.3.1 质粒构建
通过PrimeSTAR酶对DNA片段进行扩增。通过PCR扩增质粒骨架使其线性化。使用T4 DNA连接酶将含有互补序列的DNA片段和质粒骨架连接。重组质粒转化到大肠杆菌中,在固体选择培养基中培养,使用质粒提取试剂盒提取质粒,对重组质粒进行测序验证其正确性。
含有crRNA阵列的pcr20 NM2质粒通过Golden Gate构建。合成包含有全部crRNA阵列的引物对,引物结构如图1-d,通过引物退火使其形成双链DNA。将质粒骨架、crRNA阵列混合物、Eco31I、T4 DNA连接酶混合,在37 ℃、16 ℃转换使crRNA阵列按顺序随机连接在质粒骨架中。
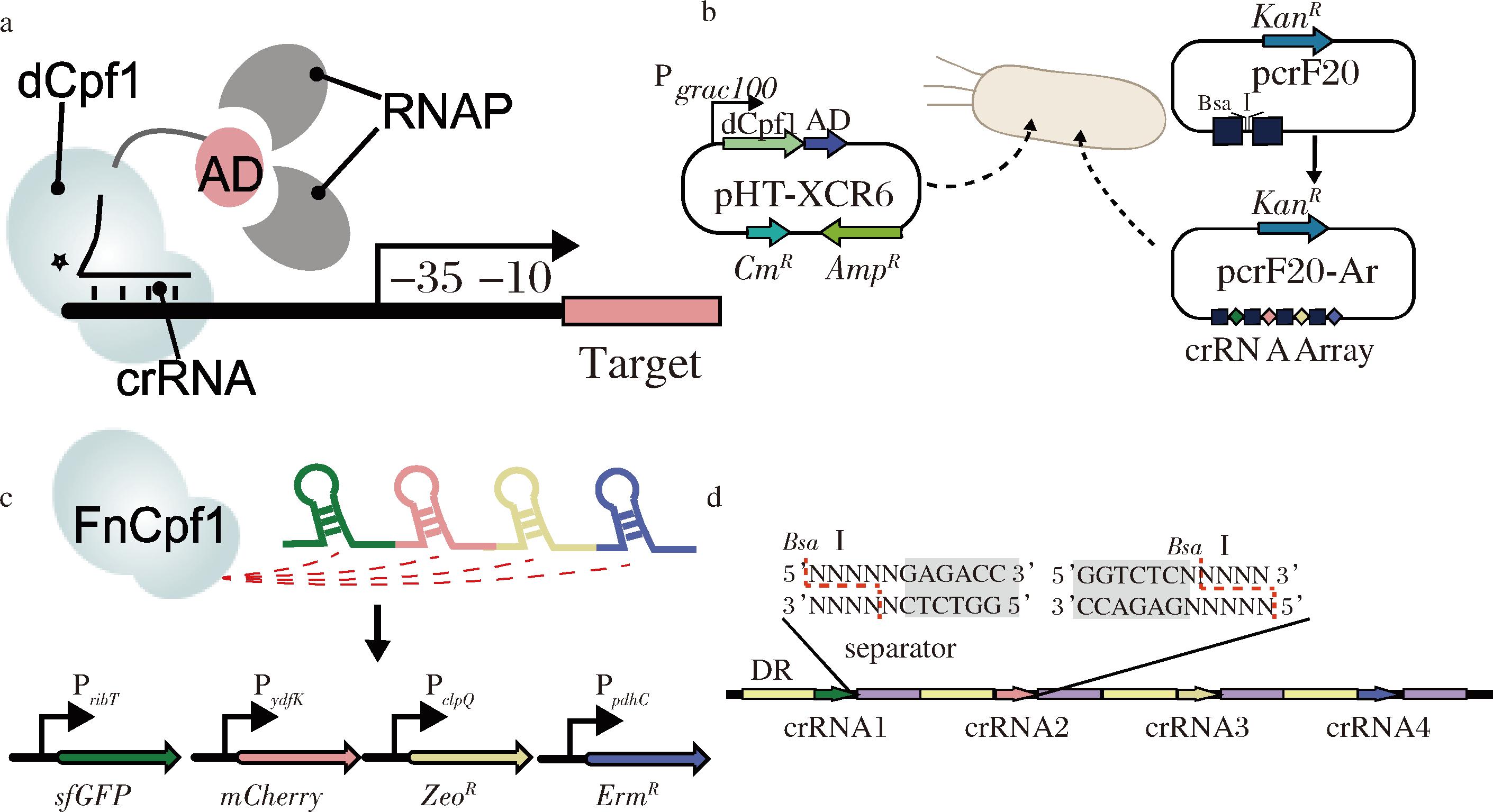
a-CRISPR激活示意图;b-双质粒构建激活系统示意图;c-表征基因示意图;d-Golden Gate组装crRNA阵列示意图
图1 CRISPR激活系统构建示意图
Fig.1 Schematic illustration of CRISPR activation system construction
注:dCpf1:dead Cpf1 失活Cpf1蛋白;Target:靶基因;CmR:氯霉素抗性基因;AmpR:氨苄青霉素抗性基因;Pgrac100:高效启动子;KanR:卡那霉素抗性基因;crRNA array: 向导RNA阵列;ZeoR:博来霉素抗性基因;ErmR:红霉素抗性基因。

a-不同激活结构域对2种荧光基因的激活效果;b-不同激活结构域对红霉素抗性基因的激活效果;c-不同激活结构域对博来霉素抗性基因的激活效果;d-不同激活位点对于激活sfGFP效果的影响;e-不同激活位点对于激活mCherry效果的影响;f-激活结构域在不同位点时对荧光基因的抑制效果
图2 双向调控系统对基因表达的调控效果
Fig.2 The regulatory effect of bidirectional regulatory system on gene expression
注:图中G168表示野生型菌。
1.3.2 CRISPR基因编辑和基因调控
CRISPR基因编辑:在pcrF19 NM2质粒上构建目标位点的crRNA序列和编辑位点的上下游同源臂。同时将pcrF19 NM2和pHT01两个质粒导入到枯草芽孢杆菌内,在含有卡那霉素和氯霉素的固体培养基上进行培养,随后转接到含有3%(体积分数)木糖(500 g/L)的LB液体培养基中过夜培养诱导编辑。
CRISPR基因调控:将含有crRNA序列和调控因子的2个质粒同时导入到枯草芽孢杆菌中,在含有卡那霉素和氯霉素的固体培养基上进行培养,随后转接到含有IPTG的LB液体培养基中过夜培养。
1.3.3 基因表达分析
表达荧光蛋白的菌株,在LB培养基中生长10 h。然后在96孔板中转接20 μL菌液至200 μL的LB培养基中。在多板振荡器(37 ℃,750 r/min)中孵育后,使用微孔板多模式读数器测量600 nm处的吸光度(OD600)、编码绿色荧光蛋白(superfolder green fluorescent protein,sfGFP)荧光(480 nm激发光、523 nm 发射光)和编码红色荧光蛋白mCherry(587 nm激发光、610 nm发射光)。为了排除培养基的背景OD(ODbg)和不含荧光蛋白的菌株的背景荧光(FPbg),使用公式(1)计算相对荧光强度:
(1)
1.3.4 摇瓶发酵
选择新鲜的活化单菌落,并将其转移到15/100 mL LB培养基中。在220 r/min、37 ℃条件下培养10 h,制备种子溶液。将900 μL种子溶液转移到40 mL发酵培养基中,以220 r/min、37 ℃条件下下振荡培养36 h。加入诱导剂IPTG以激活调节系统。
1.3.5 核黄素含量测定
将200 μL发酵培养物加入800 μL 0.05 mol/L NaOH中。在10 000×g离心2 min后,收集上清液,用0.1 mol/L醋酸-醋酸钠缓冲液(pH 4.42)稀释。随后,测量了444 nm处的吸收。核黄素浓度使用公式(2)计算:
(2)
式中:R2=0.999 7;OD444,444 nm处的吸光度值;Y,核黄素质量浓度(mg/L);DF,稀释倍数;OD444通过稀释控制在0.1~0.8。
2 结果与分析
2.1 CRISPRa和CRISPRi系统的设计与构建
首先,通过将不同类型的AD与dCpf1与融合,生成dCpf1-AD复合体,构建枯草芽孢杆菌的CRISPRa系统。分别包括枯草芽孢杆菌内源的3个转录因子RemA、RpoZ、MalR,其中RemA和MalR具有促进RNA聚合酶与DNA结合的功能[17],RpoZ是RNA聚合酶中的ω亚基,具有招募RNA聚合酶的作用(图1-a)。此外,选择了在大肠杆菌中具有激活效果的SoxS作为候选AD[8]。同时,选择了分别来源于大肠杆菌噬菌体和枯草芽孢杆菌噬菌体的AsiA和P4两个蛋白作为候选AD。
为灵活更改crRNA的靶向位点和替换AD,将dCpf1-AD复合体和crRNA阵列分别插入至PHT01和pcrF10 NM2两个质粒中(图1-b)。同时,使用枯草芽孢杆菌中常用的IPTG诱导型启动子Pgrac100表达dCpf1-AD蛋白。此外,为表征dCpf1-AD复合体的多基因转录激活功能,使用了4个不同的枯草芽孢杆菌内源启动子PpdhC、PclpQ、PribT和PydfK分别调控4个报告基因的表达,包括红霉素抗性基因、博来霉素抗性基因、sfGFP、mCherry。随后,将4个基因的表达框分别插入到枯草芽孢杆菌168的基因组中(图1-c),构建菌株BS-PpdhC-E-PclpQ-Z-PribT-GFP-PydfK-mCherry。此外,在构建crRNA阵列时,在4个表征启动子上分别选择了10个crRNA结合位点,所有crRNA结合位点均位于启动子-35区上游以减少对RNA聚合酶的空间位阻[17],且结合位点最远不超过启动子上游400 bp。随后,通过Golden gate技术将靶向4种启动子的crRNA组装(图1-d),创建针对4个启动子不同激活位点的crRNA阵列文库。最后,将上述含有dCpf1-AD复合体和crRNA阵列文库的2个质粒分别转化进入BS-PpdhC-E-PclpQ-Z-PribT-GFP-PydfK-mCherry菌株,构建BS-CRISPRa菌株文库。
首先,利用流式细胞术对BS-CRISPRa菌株文库进行初筛,由于未加入诱导剂时无dCpf1-AD激活因子表达,因此作为筛选的对照菌株。分选sfGFP和mCherry荧光强度均高于对照组的菌株。随后,在含有梯度红霉素和博来霉素的96孔板中筛选流式分选所获得的菌株,获得能够同时激活4个报告基因表达的功能性dCpf1-AD复合体。对筛选得到的菌株进行测序,发现复合体dCpf1-RpoZ和dCpf1-RemA激活效果最为明显,分别能够使sfGFP的表达量提高17.7% 和7.1%、mCherry表达量提高9.3%和21.5%(图2-a)。此外,这2种复合体可以使菌株能够在更高的抗生素浓度下生长,2种复合体分别使菌株红霉素抗性提高50%和61.5%,使菌株博来霉素抗性均提高60%,证实了2种抗性基因同时被激活(图2-b,图2-c)。此外,dCpf1-RpoZ和dCpf1-RemA复合体都具有激活位点偏好性,dCpf1-RpoZ激活的区域位于启动子的-260~-280,而dCpf1-RemA在-170~-180有效,超过-300时激活效果消失(图2-d)。以上结果表明,基于复合体dCpf1-RpoZ和dCpf1-RemA,成功构建了能够激活下游基因转录的CRISPRa系统。
此外,为检测dCpf1-RemA能否用于构建CRISPRi系统,设计靶向启动子-10区的crRNA。结果显示,sfGFP和mCherry的相对荧光强度分别下降75%和55%。为进一步验证dCpf1-RemA是否能够用于同时抑制和激活基因的表达,分别在sfGFP和mCherry 2个基因启动子前不同位点设置crRNA结合位点,结果显示,结合位点在PribT-sfGFP的-175位和PydfK-mCherry的-13位时,使sfGFP表达提高15.2%的同时,mCherry表达降低了61%。结合位点在PribT-sfGFP的-180位和PydfK-mCherry的-27位时,使sfGFP表达降低75.4%的同时,mCherry表达提高了17.3%(图2-e和图2-f)。以上结果表明,dCpf1-RemA可以用于枯草芽孢杆菌内多基因的同时激活和抑制。
2.2 CRISPRa系统的优化
CRISPRa系统的转录激活效果主要取决于RNA聚合酶被招募的数量,因此测试了增加转录激活结构域对激活效果的影响。在酿酒酵母中,在dCas蛋白N端偶连一个蛋白支架的多拷贝招募结构域,同时将被招募的结构域耦连在激活结构域N端,可以招募多拷贝的激活结构域,从而提高CRISPRa系统的激活效果[16](图3-a)。因此,选择了在酿酒酵母中使用的2种蛋白支架Sun Tag和Spy Tag应用于优化枯草芽孢杆菌中CRISPRa系统的激活效果。同时,考虑到蛋白支架拷贝数的不同可能对最终结果产生较大影响,因此测试了不同蛋白支架拷贝数对CRISPRa系统的激活效果。其中Sun Tag使用了6×、10×和24×拷贝,而Spy Tag使用了2×、4×、6×和8×拷贝。将dCpf1蛋白和RemA激活因子的N端分别与蛋白支架的2部分偶连,通过蛋白支架的招募作用将RemA聚集到dCpf1附近,靶向启动子PribT和PydfK-170~-180区域。结果显示,当Sun Tag的拷贝数为10时,CRISPRa系统的激活效果最为显著,相较dCpf1-RemA复合体,sfGFP和mCherry表达量分别提高了14.8%和10.9%(图3-b)。此外,随着蛋白支架拷贝数的增多,CRISPRa系统的激活效果也有所下降。当Sun Tag的拷贝数为24时,其激活效果甚至低于单独偶连RemA的dCpf1-RemA复合体,这可能是由于蛋白支架刚性结构过大,影响了RNA聚合酶正确结合到DNA上。
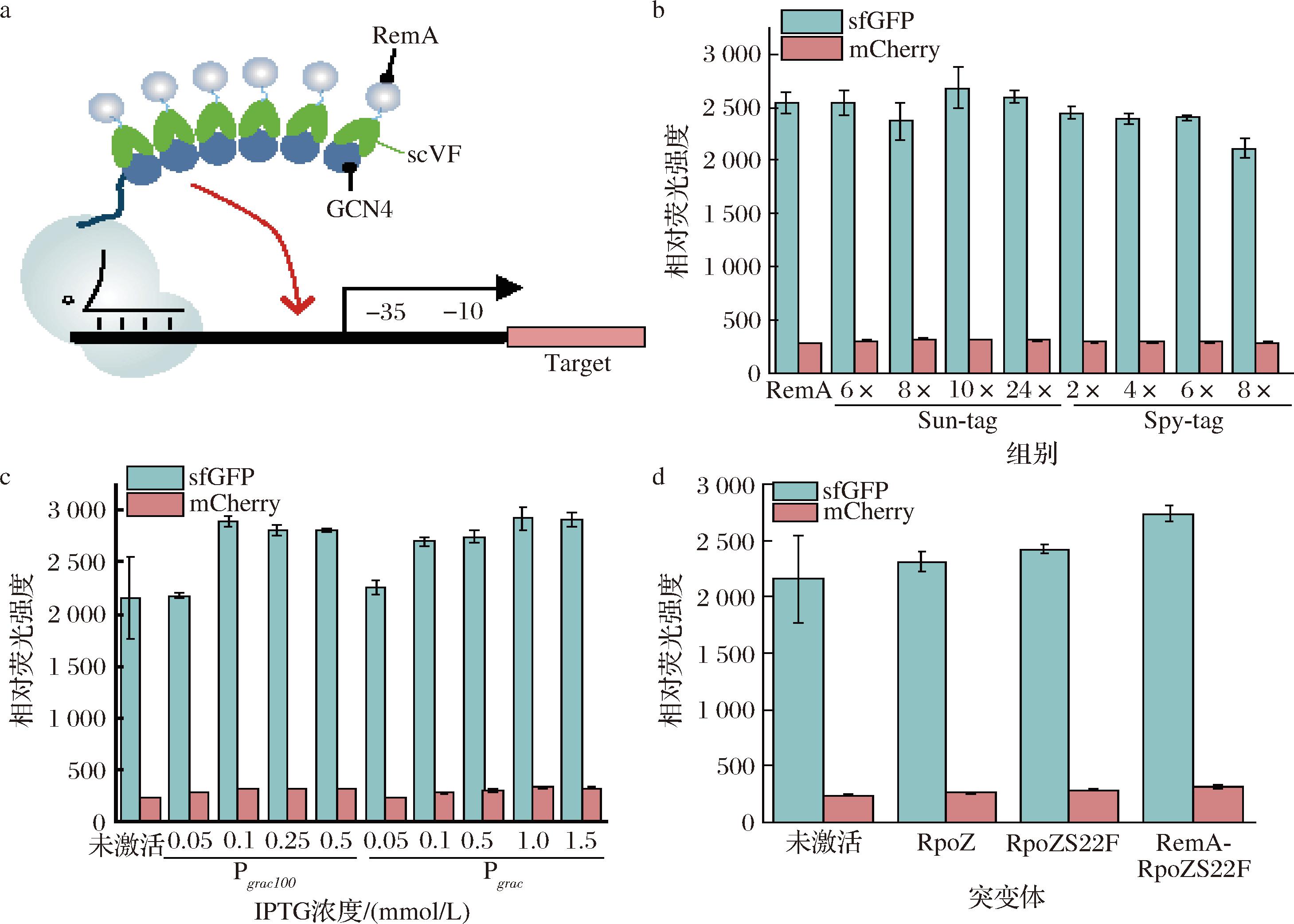
a-蛋白支架示意图;b-2种蛋白支架及不同拷贝数对激活效果的影响;c-不同浓度诱导剂及2种启动子对激活效果的影响;d-突变体对激活效果的影响
图3 双向调控系统的优化效果
Fig.3 Optimization effect of bidirectional regulation system
为进一步提高CRISPRa系统的转录激活效果,测试了不同dCpf1-RemA表达量对激活效果的影响。通过优化IPTG的添加浓度,发现在较低的IPTG诱导浓度下,CRISPRa系统的激活效果反而更高。当IPTG浓度为0.1 mmol/L时,sfGFP和mCherry的荧光强度相比于IPTG浓度为0.5 mmol/L时分别提高了6.1%和2.5%。为进一步验证较低的dCpf1-RemA表达水平是否能够提高激活效果,将原始的Pgrac100启动子替换为了强度更低的启动子Pgrac。在1 mmol/L的IPTG诱导浓度下,sfGFP和mCherry的荧光强度相比于替换启动子分别上升了4.3%和3.8%(图3-c)。
随后,对转录因子RemA和RpoZ进行了随机突变,以期获得具有更强RNA聚合酶招募能力的突变体。使用随机突变试剂盒将随机突变被引入到RemA和RpoZ蛋白中,并将这些突变体与dCpf1融合(图3-d)。为避免突变体的最佳激活位点发生变化,使用crRNA文库筛选激活水平较高的突变体。结果显示,RpoZS22F突变体显示出更好的激活效果,sfGFP和mCherry的荧光强度相比于dCpf1-RpoZ复合物分别提高了4.8%和10.4%(图3-d)。随后,在dCpf1-RpoZS22F复合体上继续耦连转录激活因子RemA,构建了dCpf1-RpoZS22F-RemA双激活结构域复合体,该复合体的激活效果高于dCpf1-RpoZS22F,但低于dCpf1-RemA(图3-d)。
2.3 基于CRISPRa和CRISPRi系统的核黄素代谢网络全局调控
为证明本文构建的CRISPRa和CRISPRi系统能够用于基因表达的原位激活和抑制,将其应用于枯草芽孢杆菌核黄素代谢网络关键基因的激活与抑制。核黄素是一种具有重要工业意义的必需营养素,广泛应用于食品添加剂、药品和动物饲料中[18],而枯草芽孢杆菌是生产核黄素的常用菌株[9](图4-a)。抑制核黄素合成途径关键基因ribC[编码黄素激酶/黄素腺嘌呤二核苷酸(flavin adenine dinucleotide,FAD)合成酶]的表达可以阻碍核黄素转化为FMN[19];激活zwf(编码6-磷酸葡萄糖)的表达可以促使6-磷酸葡萄糖向6-磷酸葡萄糖酸转化,提高核黄素合成的前体浓度[20];同时,ribA(编码GTP环水解酶)能够催化5-磷酸核酮糖合成L-3,4-二羟基-2-丁酮-4-磷酸,提高该酶的表达水平可以促进核黄素合成前体的积累。因此,使用CRISPRa和CRISPRi系统分别调控优化枯草芽孢杆菌G600中ribC、zwf和ribA基因的表达。将带有Sun Tag蛋白支架的转录激活结构域作为转录调节因子插入基因组中,分别在Pzwf和PribA两个启动子的-160~-180 bp分别选择4个crRNA靶向位点用于激活zwf和ribA。同时,构建靶向ribC基因-19 bp的crRNA,降低ribC的转录水平。实验结果显示,当crRNA分别靶向启动子Pzwf的-174 bp处和PribA的-171 bp处时,核黄素产量提升最为明显,达到8.7倍,产量为227.5 mg/L(图4-b和图4-c)。以上研究结果表明本文所构建的CRISPRa和CRISPRi系统可用于枯草芽孢杆菌胞内基因的原位激活和抑制,实现代谢网络的全局调控。
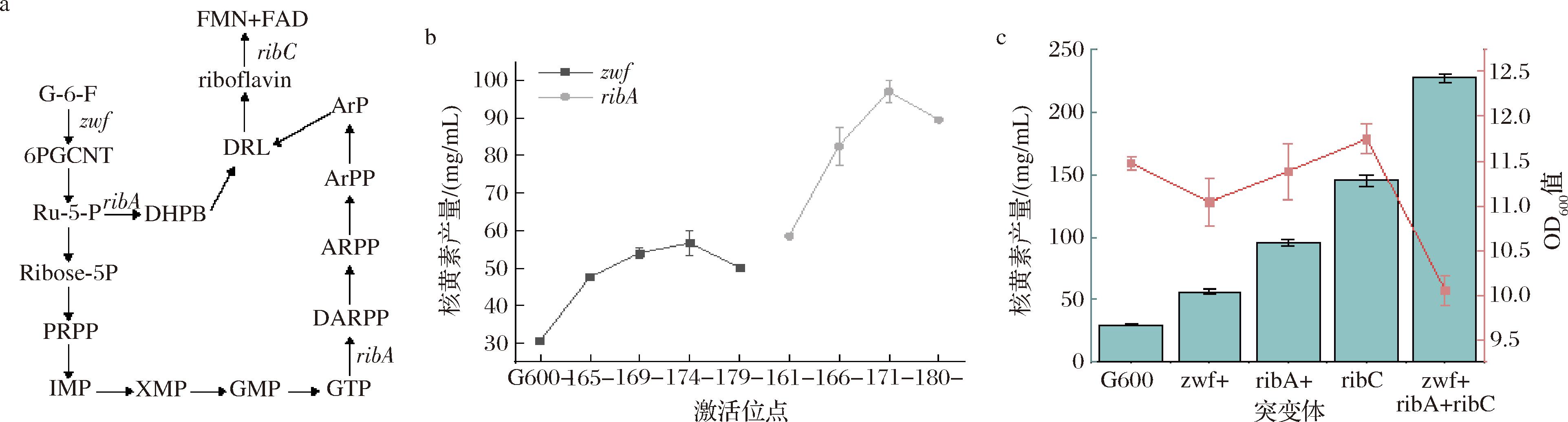
a-核黄素代谢示意图;b-不同激活位点激活分别激活2种基因时对核黄素产量的影响;c-核黄素摇瓶发酵产量
图4 调控系统对核黄素产量的影响
Fig.4 The effect of regulatory system on riboflavin production
注:G-6-F:6-磷酸葡萄糖(glucose-6-phosphate);6PGCNT:6-磷酸葡糖酸内酯(6-phosphogluconolactone);Ribose-5P:5-磷酸核糖(5-phosphate ribose);Ru-5-P:5-磷酸核酮糖(5-phosphate ribulose);DHPB:3,4-二羟基-2-丁酮-4-磷酸(3,4-dihydroxy-2-butanone 4-phosphate);PRPP:5-磷酸核糖-1-焦磷酸(5-phosphoribose-1-phosphate); IMP:肌苷酸(inosine-5′-monophosphate);XMP:黄嘌呤核苷酸(xanthine nucleotide); GMP:鸟苷酸(guanosine monophosphate);GTP:三磷酸腺苷(guanosine triphosphate); DARPP:2,5-二氨基-6-核糖氨基-4(3H)-嘧啶酮-5′-磷酸(2,5-diamino-6ribosamino-4(3H)-pyrimidine ketone-5′-phosphate); ARPP:5-氨基-6-核糖氨基-2,4(1H,3H)-嘧啶二酮-5′-磷酸(5-amino-6-ribosamino-2,4(1H, 3H)-pyrimidinedione-5′-phosphate);ArPP:5-氨基-6-核糖醇氨基-2,4(1H,3H)-嘧啶二酮-5′-磷酸(5-amino-6-ribosylamino-2,4 (1H, 3H)-pyrimidinedione-5′-phosphate);ArP:5-氨基-6-核糖醇氨基-2, 4(1H, 3H)-嘧啶二酮(5-amino-6-ribosyl-amino-2,4 (1H,3H)-pyrimidinedione); DRL:6,7-二甲基-8-核糖基二氧四氢蝶啶(6,7-dimethyl-8-ribosyl dioxotetrahydropteridine);riboflavin:核黄素; FMN:黄素单核苷酸(flavin mononucleotide) 。
3 总结
在本研究中,通过筛选不同来源的转录激活因子,发现转录激活因子RemA与dCpf1融合形成的复合体dCpf1-RemA能够用于构建CRISPRa系统和CRISPRi系统,可同时激活和抑制多个基因的表达。在此基础上,通过结合蛋白支架结构和优化激活因子表达,提高了CRISPRa系统的激活效果。最后,使用构建的CRISPRa系统和CRISPRi系统全局调控枯草芽孢杆菌中核黄素的代谢网络,显著提高了核黄素的产量,证实该系统可应用于枯草芽孢杆菌胞内基因的原位激活和抑制,为枯草芽孢杆菌的代谢网络调控提供了新工具。
[1] ROK C, DAE J, YANG D, et al.Systems metabolic engineering strategies:Integrating systems and synthetic biology with metabolic engineering[J].Trends in Biotechnology, 2019, 37(8):817-837.
[2] DING Q, DIAO W W, GAO C, et al.Microbial cell engineering to improve cellular synthetic capacity[J].Biotechnology Advances, 2020, 45:107649.
[3] ZHOU C Y, YE B, CHENG S, et al.Promoter engineering enables overproduction of foreign proteins from a single copy expression cassette in Bacillus subtilis[J].Microbial Cell Factories, 2019, 18(1):111.
[4] JIANG Z, NIU T F, LYU X Q, et al.Secretory expression fine-tuning and directed evolution of diacetylchitobiose deacetylase by Bacillus subtilis[J].Applied and Environmental Microbiology, 2019, 85(17):e01076-19.
[5] ZHAO L, TIAN R Z, SHEN Q Y, et al.Pathway engineering of Bacillus subtilis for enhanced N-acetylneuraminic acid production via whole-cell biocatalysis[J].Biotechnology Journal, 2019, 14(7):e1800682.
[6] JIAO X, LYU L T, ZHANG Y, et al.Reduction of lipid-accumulation of oleaginous yeast Rhodosporidium toruloides through CRISPR/Cas9-mediated inactivation of lipid droplet structural proteins[J].FEMS Microbiology Letters, 2021, 368(16):fnab111.
[7] WU Y K, LIU Y F, LV X Q, et al.CAMERS-B:CRISPR/Cpf1 assisted multiple-genes editing and regulation system for Bacillus subtilis[J].Biotechnology and Bioengineering, 2020, 117(6):1817-1825.
[8] LIU Y, WAN X Y, WAN B J.Engineered CRISPRa enables programmable eukaryote-like gene activation in bacteria[J].Nature Communications, 2019, 10:3693.
[9] GU Y, XU X H, WU Y K, et al.Advances and prospects of Bacillus subtilis cellular factories:From rational design to industrial applications[J].Metabolic Engineering, 2018, 50:109-121.[10] MASSAIU I, PASOTTI L, SONNENSCHEIN N, et al.Integration of enzymatic data in Bacillus subtilis genome-scale metabolic model improves phenotype predictions and enables in silico design of poly-γ-glutamic acid production strains[J].Microbial Cell Factories, 2019, 18(1):3.
[11] DONG C, FONTANA J, PATEL A, et al.Synthetic CRISPR-Cas gene activators for transcriptional reprogramming in bacteria[J].Nature Communications, 2018, 9:2489.
[12] FONTANA J, SPARKMAN-YAGER D, ZALATAN J G, et al.Challenges and opportunities with CRISPR activation in bacteria for data-driven metabolic engineering[J].Current Opinion in Biotechnology, 2020, 64:190-198.
[13] PHAN T T P, NGUYEN H D, SCHUMANN W.Development of a strong intracellular expression system for Bacillus subtilis by optimizing promoter elements[J].Journal of Biotechnology, 2012, 157(1):167-172.
[14] WU Y K, LI Y, JIN K, et al.CRISPR-dCas12a-mediated genetic circuit cascades for multiplexed pathway optimization[J].Nature Chemical Biology, 2023, 19(3):367-377.
[15] TANENBAUM M E, GILBERT L A, QI L S, et al.A protein-tagging system for signal amplification in gene expression and fluorescence imaging[J].Cell, 2014, 159(3):635-646.
[16] ZHAI H T, CUI L, XIONG Z, et al.CRISPR-mediated protein-tagging signal amplification systems for efficient transcriptional activation and repression in Saccharomyces cerevisiae[J].Nucleic Acids Research, 2022, 50(10):5988-6000.
[17] MURAYAMA S, ISHIKAWA S, CHUMSAKUL O, et al.The role of α-CTD in the genome-wide transcriptional regulation of the Bacillus subtilis cells[J].PLoS One, 2015, 10(7):e0131588.
[18] SCHWECHHEIMER S K, PARK E Y, REVUELTA J L, et al.Biotechnology of riboflavin[J].Applied Microbiology and Biotechnology, 2016, 100(5):2107-2119.
[19] MACK M, VAN LOON A P,HOHMANN H P, et al.Regulation of riboflavin biosynthesis in Bacillus subtilis is affected by the activity of the flavokinase/flavin adenine dinucleotide synthetase encoded by ribC[J].Journal of Bacteriology, 1998, 180(4).
[20] WANG Z W, CHEN T, MA X H, et al.Enhancement of riboflavin production with Bacillus subtilis by expression and site-directed mutagenesis of zwf and gnd gene from Corynebacterium glutamicum[J].Bioresource Technology, 2011, 102(4):3934-3940.