母乳寡糖(human milk oligosaccharides, HMOs)是母乳中含量第三丰富的固体营养物质,主要由D-葡萄糖、D-半乳糖、N-乙酰葡萄糖胺、L-岩藻糖、N-乙酰神经氨酸等5种单体构成,含量约为5~15 g/L[1]。目前,研究人员已在母乳中成功鉴定超过200种不同的寡糖,其中2′-岩藻糖基乳糖(2′-fucosyllactose,2′-FL)在母乳中含量最高,占比达到31%[2]。大量研究证实2′-FL具有提高婴幼儿免疫力[3]、调节肠道菌群[4-5]、促进大脑发育[6-7]等功能,相继通过了国内食品营养强化剂新品种和化妆品新原料的法律备案,市场前景广阔,极具工业生产价值。
2′-FL的生产制备方法主要为化学合成、酶催化和微生物发酵法。化学合成法需要多步反应和复杂的分离提纯过程,从而导致成本高且副反应多;酶法合成具有反应特异性高、副反应少、反应条件温和等优点,但酶的来源有限且稳定性较差,在实际应用中受到一定限制;而微生物发酵法以葡萄糖、甘油、乳糖等廉价材料为原料[8],具有生产效率高、绿色可持续等优势,受到研究人员越来越广泛的关注[9]。目前尚未发现有野生型微生物中具有完整的2′-FL合成途径,而大肠杆菌(Escherichia coli)遗传背景清晰、基因编辑技术成熟[10],具有GDP-L-岩藻糖的合成途径,已成为工业领域用于2′-FL合成的重要模式菌株[11]。在构建2′-FL合成的微生物细胞工厂时,研究人员通常采取多种策略以提高2′-FL的产量。例如,通过敲除与2′-FL合成存在竞争关系的代谢途径,减少底物分流[12-14];强化途径酶以及关键的α1,2-岩藻糖基转移酶的表达[15-16],增强合成通量。综上所述,对2′-FL生物合成代谢通路进行精准调控,并综合运用各类代谢工程策略,将有助于进一步提升2′-FL的产量,有力推动其大规模工业化生产进程。
本研究以E.coli MG1655作为底盘微生物,采用CRISPR/Cas9基因编辑系统将幽门螺旋杆菌(Helicobacter pylori)来源的α1,2-岩藻糖基转移酶(α1,2-fucosyltransferase,FutC)整合至基因组,实现了2′-FL的从头合成。随后,敲除了分支途径基因lacZ,然后利用强组成型启动子PJ23119替换基因组上lacY的原始启动子强化乳糖供给;敲除wcaJ基因阻断了克拉酸途径并利用强启动子Ptac过表达途经酶基因manC、manB、gmd、fcl,强化了前体鸟苷二磷酸(guanosine diphosphate,GDP)-L-岩藻糖的合成,进一步提高2′-FL的生产能力。在此基础上,替换野生型FutC为高活性突变体FutCV93I并增加FutCV93I的拷贝数,获得最终2′-FL生产菌株的摇瓶产量达到6.57 g/L,5 L发酵罐补料分批发酵的2′-FL产量达到31.13 g/L,为后续2′-FL的规模化工业生产提供了重要的数据支持。具体见图1。

图1 重组大肠杆菌合成2′-FL的代谢途径
Fig.1 Metabolic pathway for 2′-FL synthesis in recombinant E.coli strains
注:LON,LON 蛋白酶;lon,编码LON蛋白酶的基因;fkp,编码双功能L-岩藻糖激酶/GDP-L-岩藻糖磷酸化酶的基因;RcsA,荚膜合成调控蛋白A(regulator of capsule synthesis A,RcsA)。
1 材料与方法
1.1 材料与试剂
1.1.1 质粒与菌株
菌株E.coli JM109用于质粒构建,E.coli MG1655用于重组菌株的构建,本研究中使用的质粒和菌株见表1。
表1 本研究中所用菌株和质粒
Table 1 Strains and plasmids used in the study
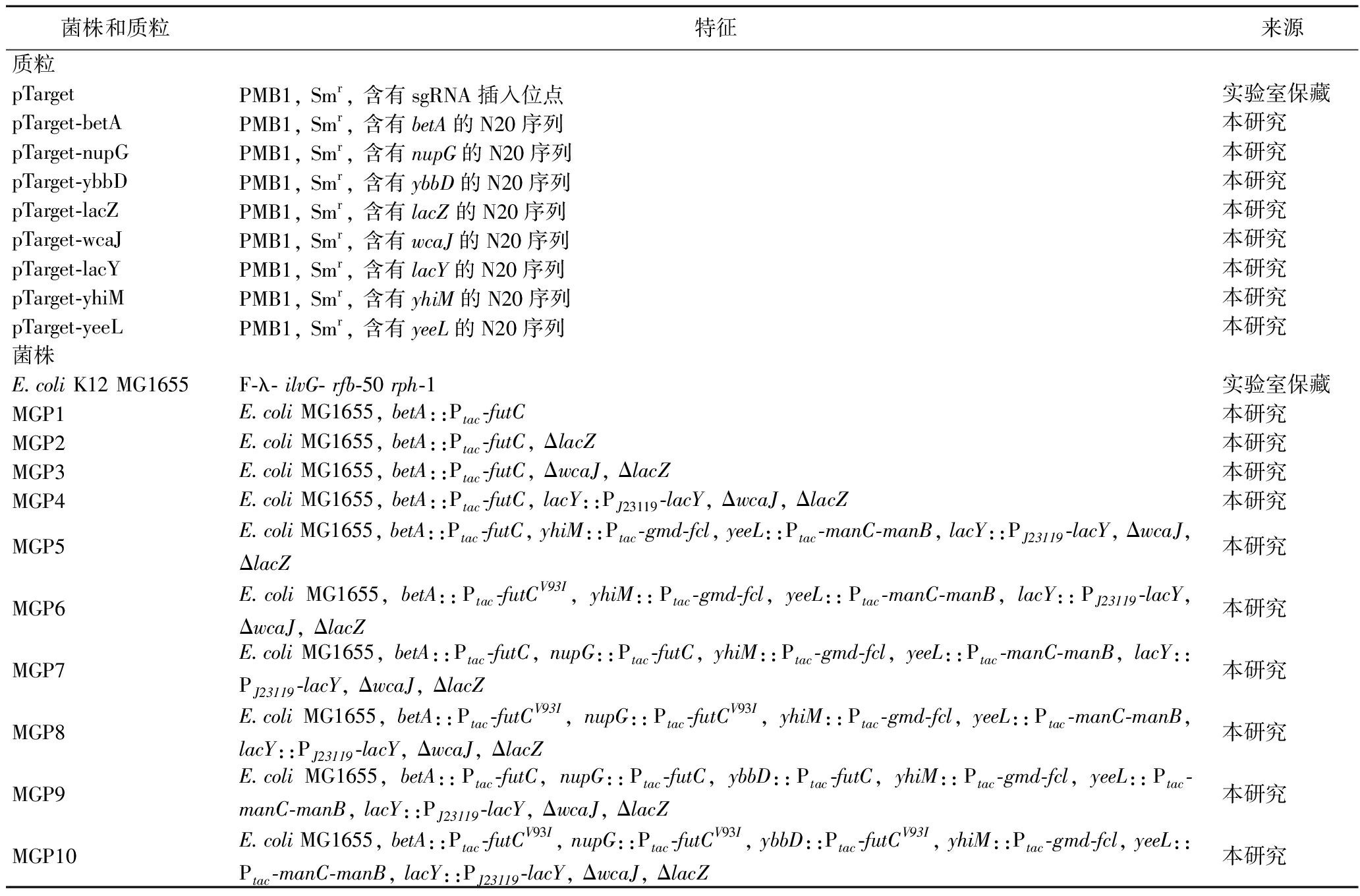
菌株和质粒特征来源质粒pTargetPMB1, Smr, 含有sgRNA插入位点实验室保藏pTarget-betAPMB1, Smr, 含有betA的N20序列本研究pTarget-nupGPMB1, Smr, 含有nupG的N20序列本研究pTarget-ybbDPMB1, Smr, 含有ybbD的N20序列本研究pTarget-lacZPMB1, Smr, 含有lacZ的N20序列本研究pTarget-wcaJPMB1, Smr, 含有wcaJ的N20序列本研究pTarget-lacYPMB1, Smr, 含有lacY的N20序列本研究pTarget-yhiMPMB1, Smr, 含有yhiM的N20序列本研究pTarget-yeeLPMB1, Smr, 含有yeeL的N20序列本研究菌株E.coli K12 MG1655F-λ- ilvG- rfb-50 rph-1实验室保藏MGP1E.coli MG1655, betA::Ptac-futC 本研究MGP2E.coli MG1655, betA::Ptac-futC, ΔlacZ本研究MGP3E.coli MG1655, betA::Ptac-futC, ΔwcaJ, ΔlacZ本研究MGP4E.coli MG1655, betA::Ptac-futC, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP5E.coli MG1655, betA::Ptac-futC, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP6E.coli MG1655, betA::Ptac-futCV93I, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP7E.coli MG1655, betA::Ptac-futC, nupG::Ptac-futC, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP8E.coli MG1655, betA::Ptac-futCV93I, nupG::Ptac-futCV93I, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP9E.coli MG1655, betA::Ptac-futC, nupG::Ptac-futC, ybbD::Ptac-futC, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究MGP10E.coli MG1655, betA::Ptac-futCV93I, nupG::Ptac-futCV93I, ybbD::Ptac-futCV93I, yhiM::Ptac-gmd-fcl, yeeL::Ptac-manC-manB, lacY::PJ23119-lacY, ΔwcaJ, ΔlacZ本研究
1.1.2 培养基及溶液
LB培养基(g/L):称取酵母浸出物5、胰蛋白胨10、氯化钠10,加超纯水溶解后定容至1 L;固体培养基加入20 g/L的琼脂粉。
摇瓶发酵培养基(g/L):称取酵母浸出物24、磷酸二氢钾2.31、甘油20、胰蛋白胨12、磷酸氢二钾12.54、硫酸铵1、氯化钠0.5,加超纯水溶解后定容至1 L,高温灭菌后加入无菌滤膜处理过的七水硫酸镁溶液使其终质量浓度为1.4 g/L。
发酵罐培养基(g/L):称取酵母浸出物24、磷酸二氢钾2.31、甘油20、胰蛋白胨12、磷酸氢二钾12.54、硫酸铵1、氯化钠0.5、柠檬酸2,加超纯水溶解后定容至1 L,高温灭菌后加入无菌滤膜处理过的硫酸镁溶液使其终质量浓度为1.4 g/L和10 mL微量元素溶液。
微量元素溶液(g/L):称取硫酸钠2、七水合硫酸亚铁7.56、五水合硫酸铜0.06、六水合氯化钴0.4、七水合硫酸锌0.64、维生素B1 0.2、生物素0.2,加超纯水溶解后定容至1 L,使用真空抽滤系统和0.22 μm的无菌水系滤膜过滤杂质和细菌。
HPLC流动相:量取浓硫酸550 μL,超纯水定容至2 L,配制终浓度为5 mmol/L的稀硫酸溶液,使用0.22 μm的水系滤膜过滤杂质。
1.2 仪器设备
AIRTECH洁净工作台,苏州安泰空气技术有限公司;RevcoTM ExF立式超低温冰箱,NANO Drop 2000分光光度计,Finnpipette移液器,赛默飞世尔科技公司;C1000 Touch PCR仪,GelDoc Go凝胶成像系统,Gene Pulser Xcell电击转化仪,1 mm电击杯,HPX-87H色谱柱,伯乐生命医学产品上海有限公司;Agilent 1260高效液相色谱仪,安捷伦科技有限公司;MP Fastprep-24 5G快速样品制备仪,安倍医疗器械贸易有限公司;MX-S漩涡混合器,D2012小型离心机,美国赛洛捷克有限公司。
1.3 实验方法
1.3.1 引物设计
根据引物设计的基本原则,使用软件SnapGene构建如表2所示的引物。
表2 文中所用引物
Table 2 Primers used in the study

引物名称序列(5′-3′)lacZ-N20-FAGATTCAGCCGGAAAACCTACCGGATTlacZ-N20-RAATTAATCCGGTAGGTTTTCCGGCTGAwcaJ-N20-FAGATGTGACGCGCGGATCGTTCTGCGTwcaJ-N20-RAATTACGCAGAACGATCCGCGCGTCAClacY-N20-FAAATAGTACATAATGGATTTGTTTTAGAGCTAGAAATAGCAAGlacY-N20-RACAAATCCATTATGTACTATTTACTAGTATTATACCTAGGACTGAGlacZ-U-FGCACTTCAAACTGGCTGGTAATATATTTAAAlacZ-U-RGGAAACAGCTTAATAACCGGGCAGGCCATGTClacZ-D-FTATTAAGCTGTTTCCTGTGTGAAATTGTTATCClacZ-D-RCGGGCAAACAGTCGTTGCTGATTwcaJ-U-FAATGTTTTAATCAACGGTGCCAGGTCCGGGTTAwcaJ-U-RAATAACAGGAACAACGTATGAGCTTACGTGAAAAAACCATCAwcaJ-D-FATACGTTGTTCCTGTTATTAGCCCCTTACCCGCAwcaJ-D-RATCCACGTCTCTCCTGGAACACCGTTGlacY-U-FAGGGCGCATCCGTTTTGGCGAAAAAGAGTAAAACGGCGAGGlacY-U-RTTGACAGCTAGCTCAGTCCTAGGTATAATACTAGTATGTACTATTTAAAAAACACAAACTTTTGGAlacY-D-FACTAGTATTATACCTAGGACTGAGCTAGCTGTCAATTATTTTTGACACCAGACCAACTGGTAAlacY-D-RCGACATTGGCGTAAGTGAAGCGACCCGCATTGACCCTAACGCCTGGGyhiM-N20-FATAAATTTTTATTTATCATGGTTTTAGAGCTAGAAATAGCAAGyhiM-N20-RACCATGATAAATAAAAATTTATACTAGTATTATACCTAGGACTGAGyeeL-N20-FATAAACGGTAGACATTATTAGTTTTAGAGCTAGAAATAGCAAGyeeL-N20-RCTAATAATGTCTACCGTTTATACTAGTATTATACCTAGGACTGAGyhiM-U-FACAGATTTACGACGATATAAAAyhiM-U-RGTAACCTAAGATTAATTCTTATGCTGTTGTTTTTTACyhiM-D-FATTATACGAGCCGATGATTAATTGTCAAGCCTTTTGAACGCTGAAAAGTAGCyhiM-D-RAGCCGAGAAAAATAACCCAATAACACCACAGCCAGTGTTCTTTAyhiM-Ptac-gmd-fcl-FAGAATTAATCTTAGGTTACCCCCGAAAGCGGTCTTyhiM-Ptac-gmd-fcl-RTAATCATCGGCTCGTATAATGTGATCAGACCTTTGTTTAACTTTAAGAAGGAGATATACCATGTCAAAAGTCGCTCyeeL-U-FTATTCTCGCCAAAACACTTTTTAAyeeL-U-RAACCTTACCACCCTCGATTAAACGTTCAGCAGCAATCATAAAyeeL-D-FAAGGTCTGATCACATTATACGAGCCGATGATTAATTGTCAATAACAATCCACTAATGCCCTGGCTTTATCyeeL-D-RGCATCTTTTAATTGTAACGTCGACAATTAATTTAGAAGyeeL-Ptac-manC-manB-FTAATCGAGGGTGGTAAGGTTACTTGTTCAGTAACTCAAGGATAyeeL-Ptac-manC-manB-RGTATAATGTGATCAGACCTTTGTTTAACTTTAAGAAGGAGATATACCATGAGCTCACCTCTTATTCCGbetA-N20-FTGAGAGTTACCGTTCCAGAGGTTTTAGAGCTAGAAATAGCAAGbetA-N20-RCTCTGGAACGGTAACTCTCAACTAGTATTATACCTAGGACTGAGnupG-N20-FAAGAAAAACGGGTCGCCAGAGTTTTAGAGCTAGAAATAGCAAGnupG-N20-RTCTGGCGACCCGTTTTTCTTACTAGTATTATACCTAGGACTGAGybbD-N20-FTATTCATCGTCTGAGCTATAGTTTTAGAGCTAGAAATAGCAAGybbD-N20-RTATAGCTCAGACGATGAATAACTAGTATTATACCTAGGACTGAGbetA-U-FCTGCACGCGAGATATTTCCTCTTCbetA-U-RGGTCTGATCACATTATACGAGCCGATGATTAATTGTCAAGGACCGAATCCTTATATAAACACTGAGGTbetA-D-FTGAAGACGCTGAAAAGCGTCTTTTTTCGTTTTGGTCCCGGTTCCTGCGTTAGTTCACATCbetA-D-RGTGCGGATTAAATCCCGCGACbetA-FutC-FCGGCTCGTATAATGTGATCAGACCTTTGTTTAACTTTAAGAAGGAGATATACCATGGCCTTTAAAGTCGTCCAAATCbetA-FutC-RAGACGCTTTTCAGCGTCTTCAATAATTGGTTTGGTACCGAGTTAGGCGTTGTACTTCTGGCTCnupG-U-FCATCATGTCGATTTCGCAGATCTCTGAAACnupG-U-RGGTCTGATCACATTATACGAGCCGATGATTAATTGTCAATGCGTAATTAGTGGCTAACCGTCnupG-D-FTGAAGACGCTGAAAAGCGTCTTTTTTCGTTTTGGTCCAGGTGACCCGTTTTTTTTATTCTTACnupG-D-RCAATTTGAACAGCATATTGAGGATGACTCGCCGCnupG-FutC-FCGGCTCGTATAATGTGATCAGACCTTTGTTTAACTTTAAGAAGGAGATATACCATGGCCTTTAAAGTCGTCCAAATCnupG-FutC-RAGACGCTTTTCAGCGTCTTCAATAATTGGTTTGGTACCGAGTTAGGCGTTGTACTTCTGGCTCybbD-U-FCATTGTTGTTTTATGCGTAATTACTTATCTybbD-U-RGGTCTGATCACATTATACGAGCCGATGATTAATTGTCAATGGCTTTACACAATAGCCGACATybbD-D-FTGAAGACGCTGAAAAGCGTCTTTTTTCGTTTTGGTCCTCTACTCGTATTCAGTTGTTTATTGAybbD-D-RGGTCAGCACCGGGCCAGCAACGAGCybbD-FutC-FCGGCTCGTATAATGTGATCAGACCTTTGTTTAACTTTAAGAAGGAGATATACCATGGCCTTTAAAGTCGTCCAAATybbD-FutC-RAGACGCTTTTCAGCGTCTTCAATAATTGGTTTGGTACCGAGTTAGGCGTTGTACTTCTGGCTC
1.3.2 重组菌株的构建
(1)重组大肠杆菌基因的敲除和替换lacY原始启动子
为了从E.coli基因组中敲除lacZ和wcaJ基因和替换lacY原始启动子,采用CRISPR/Cas9的基因编辑方法对上述基因进行敲除和替换以及后续质粒pTarget以及质粒pCas9的消除。以质粒pTarget为模板构建sgRNA整合表达质粒,通过CHOPCHOP网站(https://chopchop.cbu.uib.no/)设计整合位点的N20序列。使用引物lacZ-N20-F/lacZ-N20-R、wcaJ-N20-F/wcaJ-N20-R和lacY-N20-F/lacY-N20-R,以pTarget为模板,通过环化PCR,得到引入了N20序列的线性质粒片段,将这些片段分别转入到E.coli JM109中,并将获得的转化子进行测序,获得质粒pTarget-lacZ、pTarget-wcaJ和pTarget-lacY。使用引物lacZ-U-F/R和lacZ-D-F/R;wcaJ-U-F/R和wcaJ-D-F/R;lacY-U-F/R和lacY-D-F/R分别构建无lacZ、无wcaJ和替换lacY启动子的DNA片段。
将实验室保藏的pCas9质粒转化至目的菌株中,正确的转化子制备电转感受态于-80 ℃暂时存放。将上述整合框和对应的pTarget质粒分别转入对应的含有pCas9质粒的重组大肠杆菌电转感受态中,涂布含有卡那霉素和壮观霉素的平板上,于37 ℃倒置培养。将获得的重组大肠杆菌菌株消除质粒pTarget以及质粒pCas9,获得不含质粒的重组菌株。
pTarget质粒消除实验:挑取单菌落接种于1 mL新鲜LB培养基中,添加1 μL质量浓度为50 g/L的卡那霉素和20 μL 质量浓度为10 mmol/L的鼠李糖,37 ℃培养12 h后,将菌液划线于含有卡那霉素的LB平板,继续培养12 h。挑取平板上长出的单菌落分别点接于含有卡那霉素和壮观霉素的双抗LB平板、以及仅含有卡那霉素的单抗LB平板。在单抗平板上生长,而在双抗平板上不生长的菌落即成功消除pTarget质粒。
Cas9质粒消除实验:挑取已消除pTarget质粒的菌落接种于含有5 g/L葡萄糖的新鲜LB液体培养基中,37 ℃培养12 h后,将菌液划线于添加有10 g/L蔗糖和5 g/L葡萄糖的LB平板,继续培养12 h。挑取平板上长出的单菌落分别点接于含有卡那霉素的单抗LB平板、以及不含抗生素的无抗LB平板。在无抗平板上生长,而在双抗平板上不生长的菌落即成功消除Cas9质粒。
(2)重组大肠杆菌基因的过表达
在E.coli MG1655基因组上,选择了常见的稳定的位点yhiM和yeeL[11],并通过CRISPR/Cas9的基因编辑方法将gmd-fcl、manC-manB基因插入到这些位点上。以质粒pTarget为模板构建sgRNA整合表达质粒,通过CHOPCHOP网站设计了整合位点的N20序列。使用引物yhiM-N20-F/yhiM-N20-R和yeeL-N20-F/yeeL-N20-R以pTarget为模板,通过环化PCR,得到引入了N20序列的线性质粒片段,将这些片段分别转入到E.coli JM109中并进行测序,获得质粒pTarget-yhiM和pTarget-yeeL。
使用引物yhiM-U-F/R、yhiM-D-F/R和yhiM-Ptac-gmd-fcl-F/R;yeeL-U-F/R、yeeL-D-F/R和yeeL-Ptac-manC-manB-F/R分别构建yhiM-Ptac-gmd-fcl和yeeL-Ptac-manC-manB整合框,将整合框和对应的pTarget质粒通过电转化法整合至目的菌株中,并消除质粒pTarget以及质粒pCas9,获得不含质粒的重组菌株。
(3)α1,2-岩藻糖基转移酶futC的基因整合
在E.coli MG1655基因组上,选择了常见的稳定的位点betA、nupG和ybbD[11],并通过CRISPR/Cas9的基因编辑方法将α1,2-岩藻糖基转移酶futC基因插入到这些位点上。以质粒pTarget为模板构建sgRNA整合表达质粒,通过CHOPCHOP网站设计整合位点的N20序列。使用引物betA-N20-F/betA-N20-R,nupG-N20-F/nupG-N20-R,ybbD-N20-F/ybbD-N20-R,以pTarget为模板,通过环化PCR得到引入了N20序列的线性质粒片段,将这些片段分别转入到E.coli JM109中,并将获得的转化子进行测序,获得质粒pTarget-betA,pTarget-nupG,pTarget-ybbD。
使用引物betA-U-F/R、betA-D-F/R和betA-FutC-F/R;nupG-U-F/R、nupG-D-F/R和nupG-FutC-F/R;ybbD-U-F/R、ybbD-D-F/R和ybbD-FutC-F/R分别构建betA-Ptac-FutC(V93I)、nupG-Ptac-FutC(V93I)和ybbD-Ptac-FutC(V93I)整合框,将整合框和对应的pTarget质粒通过电转化法整合至对应目的菌株中,并消除质粒pTarget以及质粒pCas9,获得到不含质粒的重组菌株。
1.3.3 摇瓶发酵方法
(1)菌种活化
从-80 ℃冰箱中取出甘油保存菌,放置于冰盒上融化,然后使用接种环蘸取少量菌液划线至LB平板培养基上,得到的单菌落将用于后续的实验中。倒置平板培养基,以防产生的水蒸气将其污染,于37 ℃培养箱中培养12 h。
(2)种子培养
将菌种活化步骤得到的单菌落接种至3 mL的LB培养基中,在37 ℃的振荡培养箱中以220 r/min的转速培养12 h,获得种子培养液。
(3)摇瓶发酵培养
将上述种子液以1%(体积分数)的接种量转接至25 mL的发酵培养基中,按实验需要添加终质量浓度为20 g/L的乳糖,在37 ℃的振荡培养箱中以220 r/min的转速培养72 h,定时取样。
1.3.4 补料分批发酵方法
配制发酵罐发酵培养基置于发酵罐内,750 g/L的甘油母液置于补料瓶中,高温灭菌;配制300 g/L的乳糖母液单独使用滤膜除菌。将甘油菌划线于LB无抗固体培养基,并于37 ℃培养箱中培养12 h。待长出单菌落后,培养一级种子液,挑取单菌落并将其接种至无抗的LB液体培养基中,37 ℃ 220 r/min培养12 h。以1%(体积分数)的接种量,将获得的一级种子液接入到120 mL无抗LB培养基中,继续培养12 h获得二级种子液。将发酵罐中的溶氧和pH校准后,加入乳糖母液使得乳糖的初始质量浓度为25 g/L,并将种子液全部接入发酵罐中。此时发酵罐的参数设置为:温度37 ℃,转速300 r/min,pH通过氨水调节至6.8。发酵过程中,控制甘油的质量浓度在0~3 g/L,乳糖浓度控制在10 g/L,待菌液OD600值超过20后,调节温度至30 ℃。设置溶氧为30%,死区为5%。
1.3.5 HPLC检测方法
取200 μL发酵液,12 000×g离心5 min后加入等体积稀硫酸溶液稀释1倍(补料分批发酵样品需要稀释10倍),取上清过0.22 μm PES滤膜。使用配有示差检测器的HPLC系统和Bio-Rad HPX-87H色谱柱定量检测溶液中的2′-FL和乳糖。工作参数为:柱温55 ℃,流动相为5 mmol/L的稀硫酸,流速为0.5 mL/min,进样量为10 μL。
2 结果与分析
2.1 2′-FL从头合成途径的构建
2′-FL的代谢合成途径分为2种,即以甘油、葡萄糖、蔗糖、甘露糖等为碳源的从头合成途径和以外源添加L-岩藻糖的补救途径[17](图1)。从头合成途径起始于中间代谢物果糖-6-磷酸(fructose-6-phosphate,F-6-P),首先,在甘露糖-6-磷酸异构酶(mannose-6-phosphate isomerase,ManA)和磷酸核糖核酸酶(phosphoribonuclease,ManB)的作用下合成甘露糖-1-磷酸(mannose-1-phosphate,Man-1-P);其次,Man-1-P通过甘露糖-1-磷酸鸟苷基转移酶(mannose-1-phosphate guanylyltransferase,ManC)转化为鸟苷二磷酸-甘露糖(guanosine diphosphate-mannose,GDP-Man)时,需要辅因子鸟苷三磷酸(guanosine triphosphate,GTP)提供磷酸基团;之后,GDP-Man在GDP-甘露糖4,6-脱水酶(GDP-mannose 4,6-dehydratase,Gmd)和GDP-L-岩藻糖合酶(GDP-L-fucose synthase,Fcl)的作用下进一步合成前体GDP-L-岩藻糖;而补救合成途径则以L-岩藻糖作为底物,在双功能L-岩藻糖激酶/GDP-L-岩藻糖磷酸化酶(bifunctional L-fucose kinase/GDP-L-fucose pyrophosphorylase,FKP)以及辅因子ATP和GTP的催化下,经两步反应转化为GDP-L-岩藻糖[18]。最后,GDP-L-岩藻糖在α1,2-岩藻糖基转移酶的作用下脱去GDP基团生成2′-FL。
由于E.coli MG1655具有从头合成前体GDP-L-岩藻糖的调控途径,只需要外源引入α1,2-岩藻糖基转移酶就可以在E.coli中实现从头合成2′-FL。研究表明[19],H.pyloriNCTC11639来源的α1,2-岩藻糖基转移酶FutC催化底物乳糖和前体GDP-L-岩藻糖合成2′-FL的效率较高。为更加稳定的表达FutC蛋白,将futC基因通过CRISPR/Cas9系统整合至E.coli基因组上获得重组菌株MGP1,使用添加了3 g/L乳糖的发酵培养基对上述重组菌株分别进行摇瓶发酵验证,通过HPLC对发酵液进行检测,检测2′-FL产量。结果如图2所示,E.coli MG1655的发酵液中无法检测到2′-FL,但MGP1的2′-FL的产量达到0.1 g/L。
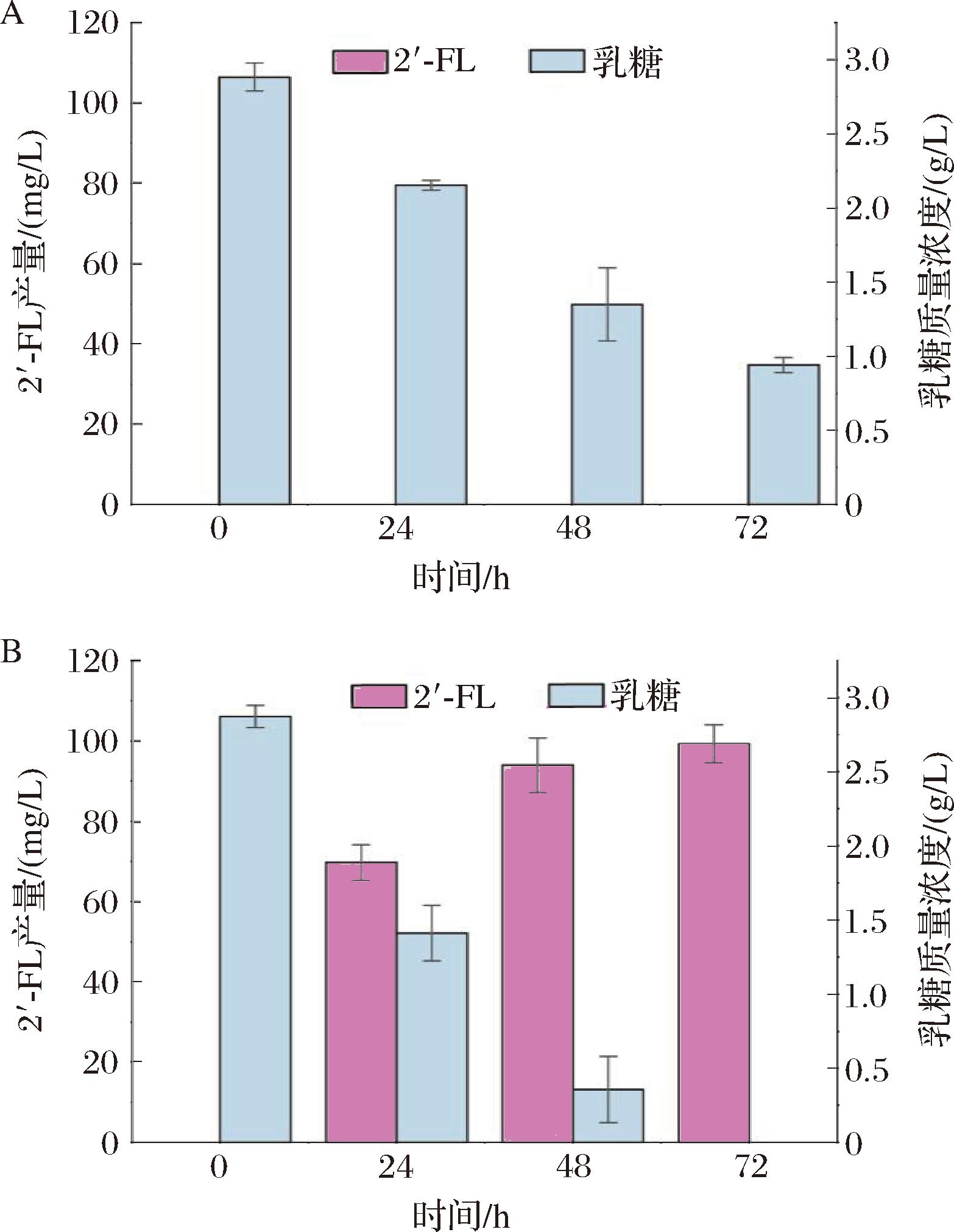
A-菌株E.coli MG1655的发酵验证;B-菌株MGP1的发酵验证
图2 E.coli MG1655和MGP1的发酵验证
Fig.2 Fermentation verification of recombinant strains E.coli MG1655 and MGP1
2.2 分支途径基因的敲除与关键代谢通路的强化
2′-FL的合成中的关键步骤是乳糖的岩藻糖基化,而该过程中两大重要前体为乳糖和GDP-L-岩藻糖。尽管E.coli MG1655中具有合成GDP-L-岩藻糖前体的代谢调控途径,但是GDP-L-岩藻糖同时也是合成荚膜异多糖酸的重要中间代谢产物,可以被克拉酸合成途径消耗利用,导致用于合成2′-FL的GDP-L-岩藻糖前体供应有限[14]。其次,在E.coli乳糖操纵子中,β-半乳糖苷酶对乳糖具有高亲和性,导致转运至胞内的乳糖被快速降解成半乳糖苷和葡萄糖[20]。因此,本研究在MGP1的基础上,敲除克拉酸竞争途径上的基因wcaJ,强化GDP-L-岩藻糖的供应;敲除乳糖操纵子中编码β-半乳糖苷酶基因lacZ,将编码乳糖通透酶基因LacY的启动子替换为组成型启动子PJ23119,提高胞内乳糖的利用效率,将代谢流进一步引向目标代谢产物,从而进一步提高2′-FL产量。使用添加了3 g/L乳糖的发酵培养基对上述重组菌株进行摇瓶发酵72 h后,取样并通过HPLC对发酵液进行检测,检测2′-FL产量和乳糖剩余。结果如图3-A所示,单独敲除lacZ的菌株MGP2的2′-FL产量达到0.45 g/L,继续敲除wcaJ的重组菌株MGP3的2′-FL产量达到0.62 g/L,进一步替换lacY启动子的菌株MGP4的2′-FL产量分别达到0.73 g/L,是初始菌株的7.3倍,说明敲除旁路途径基因并强化乳糖的供应可以有效提升2′-FL的产量。
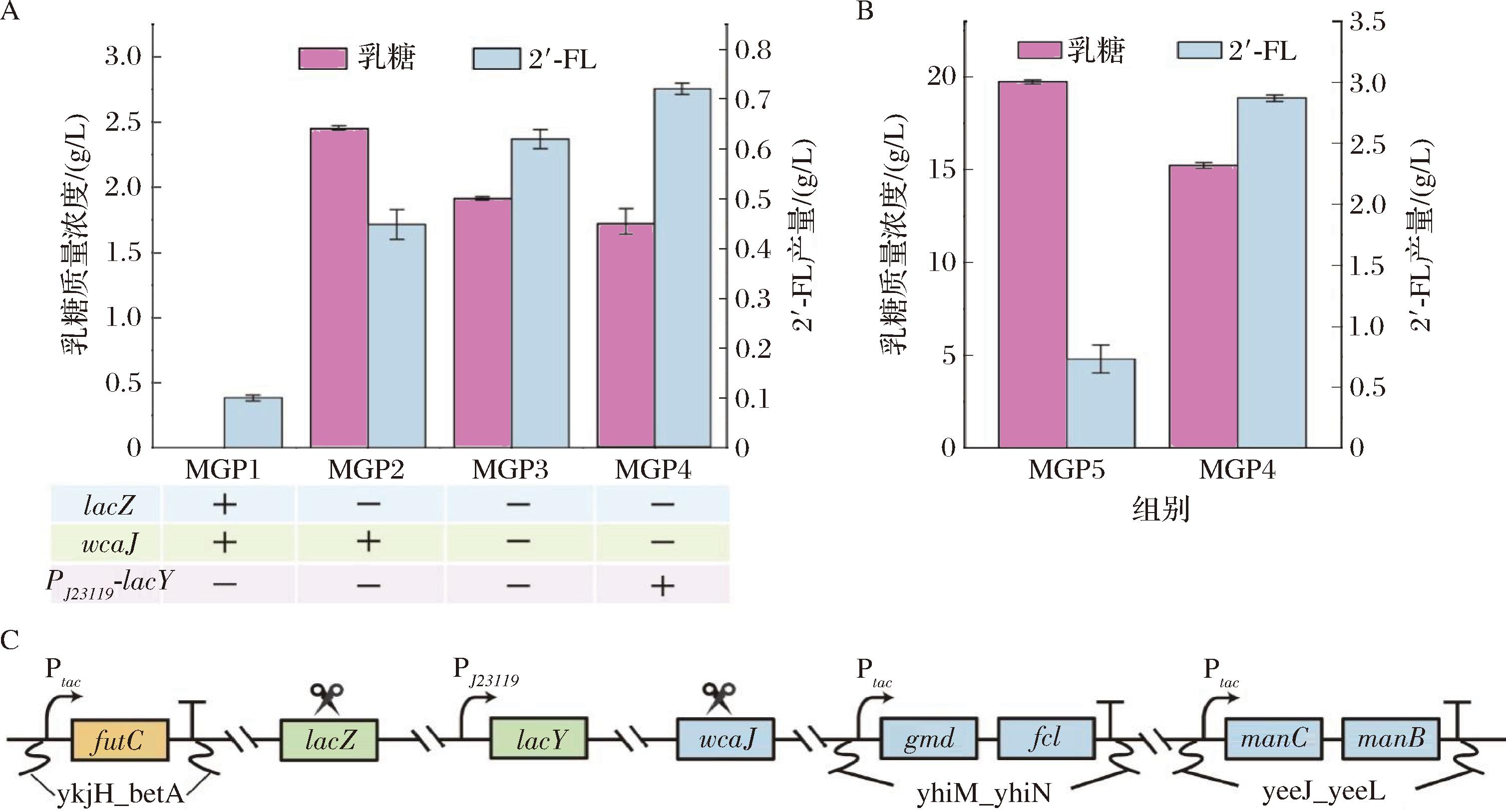
A-分支途径基因的敲除的发酵验证;B-关键代谢通路的强化的发酵验证;C-前体强化菌株的改造示意图
图3 分支途径基因的敲除与关键代谢通路的强化的发酵验证
Fig.3 Fermentation verification of gene knockout in branch pathways and enhancement of key metabolic pathways
在E.coli MG1655中,合成GDP-L-岩藻糖前体的wza基因簇受到Rcs磷酸转移系统中RcsA和RcsB异源二聚体的共同调控,而manB、manC、gmd、fcl是wza基因簇上与2′-FL合成相关的4个途径基因[21]。因此,本研究在MGP4菌株基因组上,分别增加一拷贝组成型强启动子Ptac表达的manB-manC和gmd-fcl来强化从头合成途径(图3-C),获得重组菌株MGP5。使用添加了20 g/L乳糖的发酵培养基对上述重组菌株进行摇瓶发酵72 h后,取样并通过HPLC对发酵液进行检测,检测2′-FL产量和乳糖剩余。结果如图3-B所示,前体GDP-L-岩藻糖合成途径强化的菌株的2′-FL的产量显著提高至2.87 g/L,是MGP4的3.93倍。
2.3 α 1,2-岩藻糖糖基转移酶的突变
目前天然α1,2-岩藻糖基转移酶的普遍具有异源表达差,催化活性低等问题[17, 22],而α1,2-岩藻糖基转移酶是催化供体GDP-L-岩藻糖转移至底物乳糖的半乳糖基以合成2′-FL的一个关键限速酶[10]。本研究利用前期筛选得到的一个活性显著提高的α1,2-岩藻糖基转移酶突变体FutCV93I替换MGP5基因组中的野生型FutC,获得重组菌株MGP6[23]。摇瓶发酵结果显示(图4-A),MGP6的2′-FL产量达到4.07 g/L,较MGP5的2′-FL产量增加了42%。
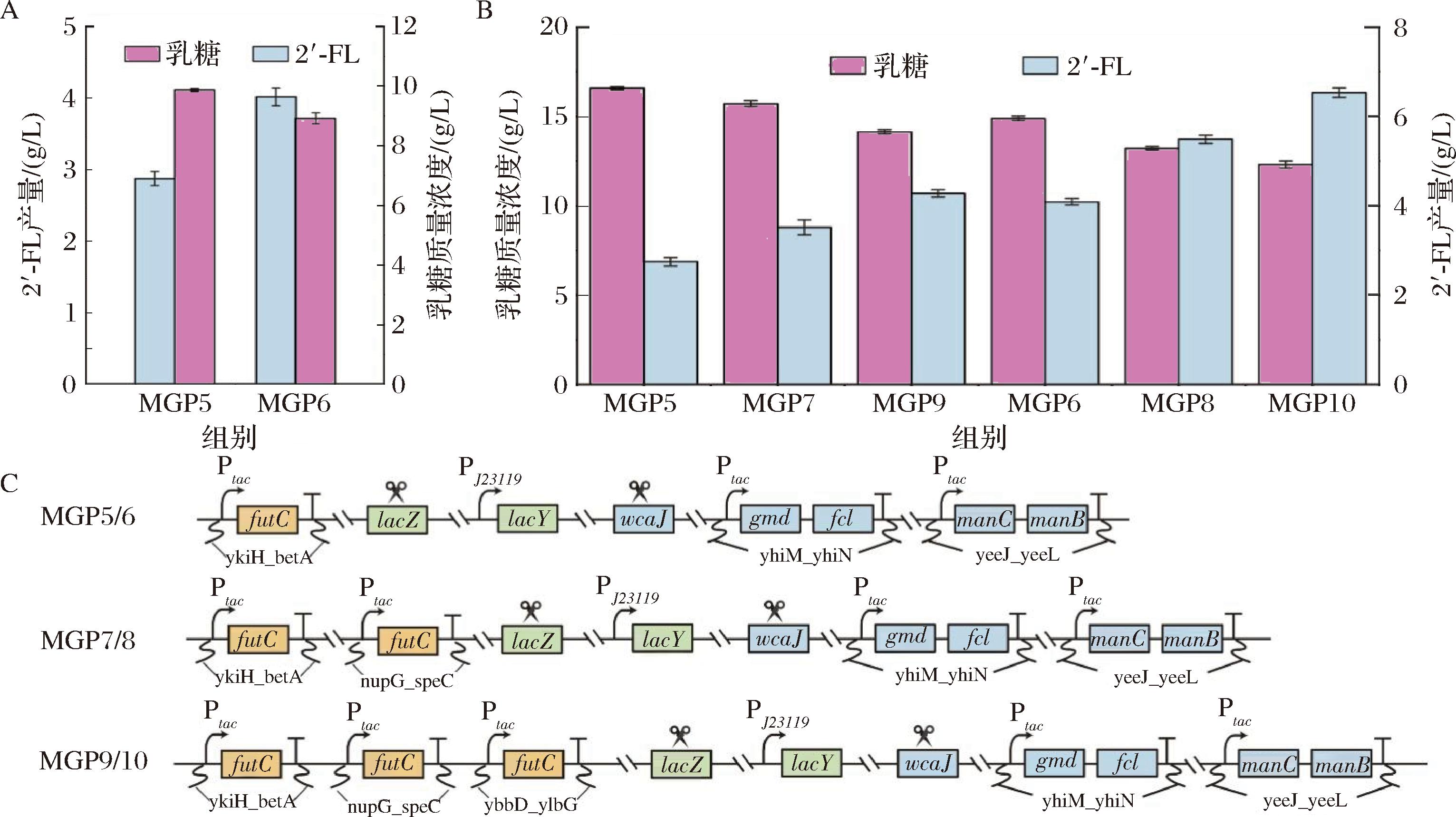
A-突变体FutCV93I对2′-FL合成的影响;B-futC拷贝数增加对2′-FL合成的影响;C-从头合成2′-FL的工程菌株构建示意图
图4 α1,2-岩藻糖基转移酶突变体FutCV93I的发酵验证
Fig.4 Fermentation verification of the α1,2-fucosyltransferase mutant FutCV93I
为了进一步体现突变体FutCV93I在微生物发酵生产2′-FL中的优势,将futC基因通过CRISPR/Cas9系统整合多拷贝至E.coli基因组上从而更加稳定的表达FutC蛋白。由于不同的基因座对外源基因的整合效率和稳定性具有不同的效果,因此,选择大肠杆菌中生长非必需的基因座[11]betA、nupG和ybbD作为futC的整合位点(图4-C),敲入futC或futCV93I构建从头合成2′-FL的工程菌株。如图4-B所示,随着futC基因拷贝数的增加,重组工程菌株的2′-FL产量逐渐增高,含3个原始基因拷贝数的MGP9的2′-FL产量为4.28 g/L,是原始菌株MGP5的1.49倍;而含3个突变基因拷贝数的MGP10的2′-FL产量达到6.57 g/L,是原始菌株MGP5的2.29倍,是单拷贝突变基因菌株MGP6的1.61倍。在基因拷贝数相同的工程菌株对比中,携带突变体的重组菌株展现出更佳的2′-FL生产能力。
2.4 5 L生物反应器扩大培养
进一步的,将MGP10菌株继续在5 L生物反应器中进行扩大培养,发酵过程的甘油质量浓度被控制在0~3 g/L,乳糖的质量浓度则被控制在10 g/L左右,初始培养温度为37 ℃以便菌体的快速生长,当菌体OD600值达到20后将培养温度调整为30 ℃以便2′-FL的生成,发酵结果如图5所示。发酵至30 h时,甘油被完全消耗,此时进行甘油的补料;在整体发酵过程中,2′-FL的合成与菌体量都在稳定增长,在发酵时长到63 h之后,细胞开始迅速衰亡,2′-FL的合成速率开始减缓,直到69 h产物停止合成,最终MGP10的2′-FL发酵产量达到31.13 g/L,是摇瓶发酵产量的4.74倍,最高生产强度可以达到0.45 g/L/h。
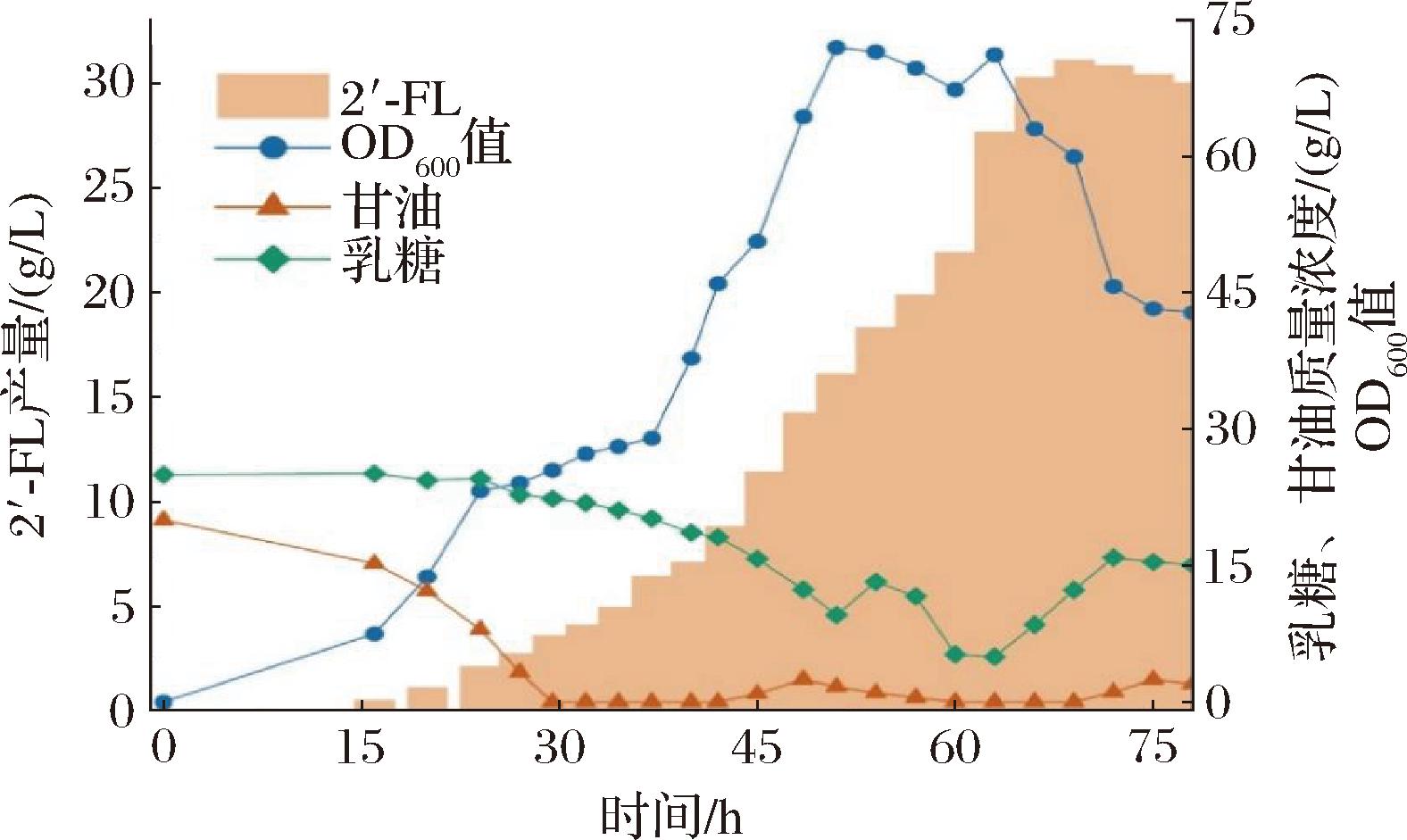
图5 5 L生物反应器上高密度发酵生产2′-FL
Fig.5 High density fermentation of 2′-FL in 5 L bioreactor
3 结论与讨论
本研究以工业模式微生物E.coli MG1655为出发菌株,首先通过整合α1,2-岩藻糖基转移酶基因futC,成功构建大肠杆菌2′-FL从头合成细胞工厂。其次,通过敲除分支途径基因wcaJ和lacZ,并使用强启动子PJ23119替换lacY的原始启动子,利用强启动子Ptac过表达途经酶基因manC、manB、gmd、fcl,强化了GDP-L-岩藻糖的合成和乳糖的供应,从而进一步提高2′-FL的产量。在此基础上,替换原始FutC为突变体FutCV93I并增加FutCV93I的拷贝数,获得2′-FL的生产菌株摇瓶产量达到6.57 g/L,5 L发酵罐补料分批发酵的2′-FL产量达到31.13 g/L。本研究通过基因组整合和组成型表达的策略,实现了以甘油为碳源的2′-FL从头合成,相比于需要添加抗生素及诱导剂的2′-FL的生物合成方法[24],降低了生产成本。此外,高活性突变体FutCV93I的应用,为2′-FL生物合成研究提供了新的思路,对2′-FL的生物合成具有重要参考价值。
[1] YOU R, WANG L, HU M R, et al.Efficient production of 2′-fucosyllactose from fructose through metabolically engineered recombinant Escherichia coli[J].Microbial Cell Factories, 2024, 23(1):38.
[2] ZHU Y Y, WAN L, LI W, et al.Recent advances on 2′-fucosyllactose:Physiological properties, applications, and production approaches[J].Critical Reviews in Food Science and Nutrition, 2022, 62(8):2083-2092.
[3] ENGELS L, ELLING L.WbgL:A novel bacterial α1,2-fucosyltransferase for the synthesis of 2′-fucosyllactose[J].Glycobiology, 2014, 24(2):170-178.
[4] CHEN Q X, YIN Q L, XIE Q G, et al.2′-Fucosyllactose promotes the production of short-chain fatty acids and improves immune function in human-microbiota-associated mice by regulating gut microbiota[J].Journal of Agricultural and Food Chemistry, 2022, 70(42):13615-13625.
[5] ZHU Y Y, CAO H Z, WANG H, et al.Biosynthesis of human milk oligosaccharides via metabolic engineering approaches:Current advances and challenges[J].Current Opinion in Biotechnology, 2022, 78:102841.
[6] ZHANG G F, ZHAO J J, WEN R, et al.2′-Fucosyllactose promotes Bifidobacterium bifidum DNG6 adhesion to Caco-2 cells[J].Journal of Dairy Science, 2020, 103(11):9825-9834.
[7] AYECHU-MURUZABAL V, OVERBEEK S A, KOSTADINOVA A I, et al.Exposure of intestinal epithelial cells to 2′-fucosyllactose and CpG enhances galectin release and instructs dendritic cells to drive Th1 and regulatory-type immune development[J].Biomolecules, 2020, 10(5):784.
[8] ZHU Y Y, ZHANG W L, MU W M.Human milk oligosaccharides:The new gold standard for premium infant formula[J].Journal of Agricultural and Food Chemistry, 2022, 70(7):2061-2063.
[9] YU W W, JIN K, WU Y K, et al.A pathway independent multi-modular ordered control system based on thermosensors and CRISPRi improves bioproduction in Bacillus subtilis[J].Nucleic Acids Research, 2022, 50(11):6587-6600.[10] ZHU Y Y, CHEN R L, WANG H, et al.Elimination of byproduct generation and enhancement of 2′-fucosyllactose synthesis by expressing a novel α1,2-fucosyltransferase in engineered Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2023, 71(12):4915-4923.
[11] LIN L, GONG M Y, LIU Y F, et al.Combinatorial metabolic engineering of Escherichia coli for de novo production of 2′-fucosyllactose[J].Bioresource Technology, 2022, 351:126949.
[12] ZHANG Q W, LIU Z M, XIA H Z, et al.Engineered Bacillus subtilis for the de novo production of 2′-fucosyllactose[J].Microbial Cell Factories, 2022, 21(1):110.
[13] NI Z J, LI Z K, WU J Y, et al.Multi-path optimization for efficient production of 2′-fucosyllactose in an engineered Escherichia coli C41 (DE3) derivative[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:611900.
[14] PARSCHAT K, SCHREIBER S, WARTENBERG D, et al.High-titer de novo biosynthesis of the predominant human milk oligosaccharide 2′-fucosyllactose from sucrose in Escherichia coli[J].ACS Synthetic Biology, 2020, 9(10):2784-2796.
[15] DENG J Y, CHEN C M, GU Y, et al.Creating an in vivo bifunctional gene expression circuit through an aptamer-based regulatory mechanism for dynamic metabolic engineering in Bacillus subtilis[J].Metabolic Engineering, 2019, 55:179-190.
[16] XU M Y, MENG X F, ZHANG W X, et al.Improved production of 2′-fucosyllactose in engineered Saccharomyces cerevisiae expressing a putative α-1,2-fucosyltransferase from Bacillus cereus[J].Microbial Cell Factories, 2021, 20(1):165.
[17] LI M L, ZHANG T, LI C C, et al.Semi-rationally designed site-saturation mutation of Helicobacter pylori α-1,2-fucosyltransferase for improved catalytic activity and thermostability[J].International Journal of Biological Macromolecules, 2024, 259:129316.
[18] 李娜, 徐恺, 李丽, 等.2′-岩藻糖基乳糖的生理功效以及制备方法研究进展[J].食品与发酵工业, 2021, 47(23):265-71.LI N, XU K, LI L, et al.Recent researches in physiological function and manufacturing method of 2′-fucosyllactose[J].Food and Fermentation Industries, 2021, 47(23):265-271.
[19] HUANG D, YANG K X, LIU J, et al.Metabolic engineering of Escherichia coli for the production of 2′-fucosyllactose and 3-fucosyllactose through modular pathway enhancement[J].Metabolic Engineering, 2017, 41:23-38.
[20] ZHANG Y, ZHANG X J, LIU H Y, et al.Efficient production of 2′-fucosyllactose in unconventional yeast Yarrowia lipolytica[J].Synthetic and Systems Biotechnology, 2023, 8(4):716-723.
[21] LIU Y L, ZHU Y Y, WAN L, et al.High-level de novo biosynthesis of 2′-fucosyllactose by metabolically engineered Escherichia coli[J].Journal of Agricultural and Food Chemistry, 2022, 70(29):9017-9025.
[22] LU J G, LV X Q, YU W W, et al.Reshaping phosphatase substrate preference for controlled biosynthesis using a “design-build-test-learn” framework[J].Advanced Science, 2024, 11(22):2309852.
[23] 刘龙, 陈坚, 吕雪芹, 等.α-1,2-岩藻糖基转移酶的高通量筛选方法及筛选得到的突变体:中国, CN119753083A[P].2025-04-04.LIU L, CHEN J, LYU X, et al.High-throughput screening method for α-1,2-fucosyltransferase and the mutants obtained through screening:China, CN119753083A[P].2025-04-04.
[24] 何滋, 梁山泉, 刘丹, 等.工程大肠杆菌利用甘油和乳糖高效合成2′-岩藻糖基乳糖[J].食品科学, 2024, 45(18):99-105.HE Z, LIANG S Q, LIU D, et al.Efficient biosynthesis of 2′-fucosyllactose using glycerol and lactose in engineered Escherichia coli[J].Food Science, 2024, 45(18):99-105.