纳豆激酶是在发酵的大豆中首次发现的,具有良好的溶栓特性,是开发溶栓药物的理想选择[1-2]。随着分子生物技术的发展,利用基因工程和蛋白质工程,已成功实现纳豆激酶编码基因aprN在枯草芽孢杆菌、毕赤酵母、乳酸乳杆菌和大肠杆菌等宿主菌中的克隆表达[3-5]。大肠杆菌中表达的纳豆激酶为无活性的包涵体,其他工程菌的虽能成功表达,但NK产量较低,而枯草芽孢杆菌作为纳豆激酶自然表达菌株,具有生长速率快、分泌能力强、可高效表达外源基因的模式生物[6],可作为实现纳豆激酶可溶性表达的首要选择[7]。
实现目标蛋白的高效表达是目前研究的热点[8-10],而针对目标蛋白序列的优化主要分为两种:基于氨基酸序列的优化及同义突变。前者通过增添、删除或替换氨基酸序列等手段,但无法避免蛋白质性质的改变[11-12];后者在不会改变氨基酸自身的序列同时,还可通过同义密码子改变翻译的起始效率和新生多肽的延伸速率等[13],改变目标蛋白的表达量[14-15]。ZHENG等[16]通过定点诱变和分子动力学模拟发现氢键在纳豆激酶合成中发挥重要作用。柳玫琪等[17]构建3种同义突变体可有效提高植物乳杆菌p-8中乙酰乙酸脱羧酶基因CLA-DC的表达量。
基因的N端编码序列(N-terminal coding sequences, NCSs)在翻译水平上显著影响基因的表达,对细菌基因表达的微调具有重要意义。有研究结果表明,N端编码序列的前10个氨基酸残基对应的密码子形成的mRNA二级结构及每个密码子第3位碱基的G或C频率对其编码的蛋白质表达有显著影响[14,18]。在本研究中,对纳豆激酶的NCSs进行同义突变,通过枯草芽胞杆菌宿主表达,分别构建了4种突变菌株分别为NK1、NK2、NK3、NK4。利用纤维蛋白平板和SDS-PAGE测定比较这4株同义突变体的纳豆激酶活性,为进一步开发纳豆激酶产品和应用提供新的理论依据和方法。
1 材料与方法
1.1 菌株、质粒和仪器
纳豆芽孢杆菌(Bacillus natto)、枯草芽孢杆菌(Bacillus subtilis)168菌株、大肠杆菌JM109菌株和pHY-P43质粒,均为本实验室保存。
PCR仪,伯乐公司;振荡培养箱、恒温培养箱,上海知楚仪器有限公司;酶标仪,赛默飞世尔科技(中国)有限公司;紫外可见分光光度计,上海元析仪器有限公司。
1.2 酶类与试剂
Prime STAR Max Premix(2×)来自 TAKARA Bio Inc,6×Loading Buffer、5 000 marker、质粒提取试剂盒、胶回收试剂盒等,生工生物(上海)股份有限公司;十二烷基硫酸钠(sodium dodecyl sulfate, SDS),化学纯,国药集团化学试剂有限公司;考马斯亮蓝-250,源叶生物有限公司;其余药品和试剂均来自国药集团。
1.3 培养基的配制
Sipizizen感受态培养基:SPI Medium、SPII Medium、50 mmol/L CaCl2、250 mmol/L MgCl2。
平板活化培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,琼脂20,pH值调至7.0,0.1 MPa灭菌20 min。
液体活化培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,pH值调至7.0,0.1 MPa灭菌20 min。
发酵培养基(g/L):大豆蛋白胨50,胰蛋白胨20,酵母提取物20,甘油50,K2HPO4 3.86,KH2PO4 1.62,CaCl2 0.2,MgCl2 0.3,pH值调至7.0,0.1 MPa灭菌20 min。
1.4 NK、NK1、NK2、NK3、NK4五种菌株的构建
引物合成由金斯瑞生物技术有限公司合成(表1)。以aprN基因为模板,对NK前10个残基进行密码子优化并设计出4种突变序列,分别命名为NK1、NK2、NK3、NK4。
表1 目的基因序列
Table 1 Sequences of target genes

序列名称前30个碱基序列(5′→3′)NKGTGAGAAGCAAAAAATTGTGGATCAGCTTNK1ATGCGCTCGAAGAAGCTGTGGATCTCGCTGNK2ATGCGCTCAAAGAAACTGTGGATTTCACTGNK3ATGAGATCTAAAAAATTATGGATTTCATTANK4ATGAGAAGTAAAAAATTATGGATATCACTA
表2 本研究所用引物
Table 2 Primers used in this study

引物名称基因序列(5′→3′)酶切位点NK-FmCGGGATCCGTGAGAAGCAAAAAATTGTGGATBamH ⅠNK-RmCGGAATTCTTATTGTGCAGCTGCTTGTEcoR ⅠNK-FGTGAGAAGCAAAAAATTGTGGATNK-RTTATTGTGCAGCTGCTTGTNK1-F1TGGATCTCGCTGTTGTTTGCGTTAACGTTAATCTNK1-F2CGAAGAAGCTGTGGATCTCGCTGTTGTTTGCNK1-F3CGGGATCCATGCGCTCGAAGAAGCTGTGGATCTCBamH ⅠNK2-F1GATTTCACTGTTGTTTGCGTTAACGTTAATCTTNK2-F2TCAAAGAAACTGTGGATTTCACTGTTGTTTGCGTTAACNK2-F3CGGGATCCATGCGCTCAAAGAAACTGTGGATTTCACTGBamH ⅠNK3-F1GATTTCATTATTGTTTGCGTTAACGTTAATCTTTNK3-F2CTAAAAAATTATGGATTTCATTATTGTTTGCGTTAACGTNK3-F3CGGGATCCATGAGATCTAAAAAATTATGGATTTCATTATTGBamH ⅠNK4-F1GGATATCACTATTGTTTGCGTTAACGTTAATCTTTACNK4-F2GTAAAAAATTATGGATATCACTATTGTTTGCGTTAACNK4-F3CGGGATCCATGAGAAGTAAAAAATTATGGATATCACTATTGBamH Ⅰ
1.4.1 突变序列N1、N2、N3、N4的获取
以提取的纳豆芽孢杆菌全基因组为模板,以NK-F和NK-R引物扩增aprN基因。突变序列扩增采用重叠PCR的方法[19],以aprN为模版使用引物NK1-F1和NK-R进行第一次PCR。验证回收后引物对NK1-F2和NK-R以第一次PCR回收产物为模版进行第二次PCR。验证回收后引物对NK1-F3和NK-Rm以第二场PCR回收产物作为模版进行第三次PCR,经PCR产物纯化后所得即为突变序列N1。后续突变序列N2、N3、N4的构建与N1相似。
1.4.2 重组质粒的构建、蛋白表达
按照20 μL酶切体系使用限制性核酸内切酶BamH Ⅰ和EcoR I在37 ℃分别将pHY-p43质粒和目的基因进行双酶切反应3 h,最后80 ℃水浴20 min灭活。酶切后的质粒和目的基因纯化后,按照连接体系4 ℃过夜连接16 h。连接产物导入大肠杆菌JM109感受态细胞,涂布于Amp抗性平板(100 μg/mL),筛选至阳性转化子后跑胶验证。最后导入枯草芽孢杆菌感受态细胞表达,感受态的制备采用超级感受态法(spizizen)[20]。
1.5 生长曲线的测定
菌体浓度使用酶标仪测定,每组数据做3组平行实验,发酵液每3 h取样,适度稀释使其在OD600值位于0.2~0.8。
1.6 纳豆激酶活力的测定和SDS-PAGE的鉴定
利用纤维蛋白平板法测定酶活力[21]。使用生理盐水将尿激酶标准品配制成一系列浓度为40、60、80、100、130 IU/mL溶液。在纤维蛋白平板上打出直径2 mm小孔,取10 μL尿激酶标准品至孔内,37 ℃培养18 h后测量溶解圈面积,以尿激酶活力(IU/mL)的对数值为横坐标,溶解圈面积(mm2)的对数值为纵坐标,绘制标准曲线(线性回归方程为:y=0.877 99x-3.576 52,R2=0.983 5,x为酶活力的对数值,y为溶解圈面积的对数值)。
取发酵液8 000 r/min,4 ℃离心5 min收集上清液得粗酶液。过0.22 μm膜后置于5 mL离心管中。-20 ℃预冻后,放入冻干机真空干燥除去水分,得纳豆激酶冻干粉。冻干粉溶于生理盐水中,配制10 mg/mL的冻干粉溶液,用于SDS-PAGE。
1.7 NK3菌株纳豆激酶表达发酵条件单因素试验
以初始pH 7、发酵温度37 ℃和发酵时间48 h作为初始发酵条件,分别研究初始pH值(6.0、6.5、7.0、7.5、8.0)、发酵温度(20、25、30、35、40 ℃)和发酵时间(12、24、36、48、56 h)对NK3菌株生产纳豆激酶活力的影响。
1.8 Box-Behnken响应面试验设计与分析
根据单因素试验结果对初始pH(A)、发酵温度(B)和发酵时间(C)3个因素为自变量,纳豆激酶活力(Y)为响应值,设计合理高低水平表(表3)。
表3 响应面因素水平表
Table 3 Factors and levels of response surface
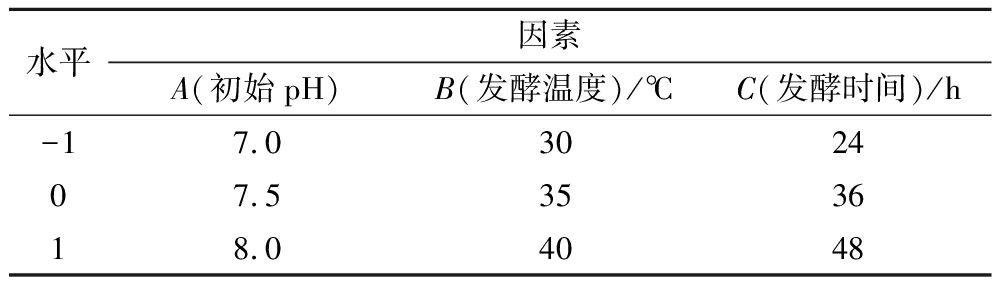
水平因素A(初始pH)B(发酵温度)/℃C(发酵时间)/h-17.0302407.5353618.04048
2 结果与分析
2.1 aprN基因和突变序列的扩增
以提取的纳豆芽孢杆菌全基因组为模板,以NK-F和NK-R为引物对aprN基因进行扩增。经琼脂糖凝胶电泳发现在1 200 bp附近有明显的扩增条带,与预期的aprN基因长度1 146 bp相吻合(图1-a)。
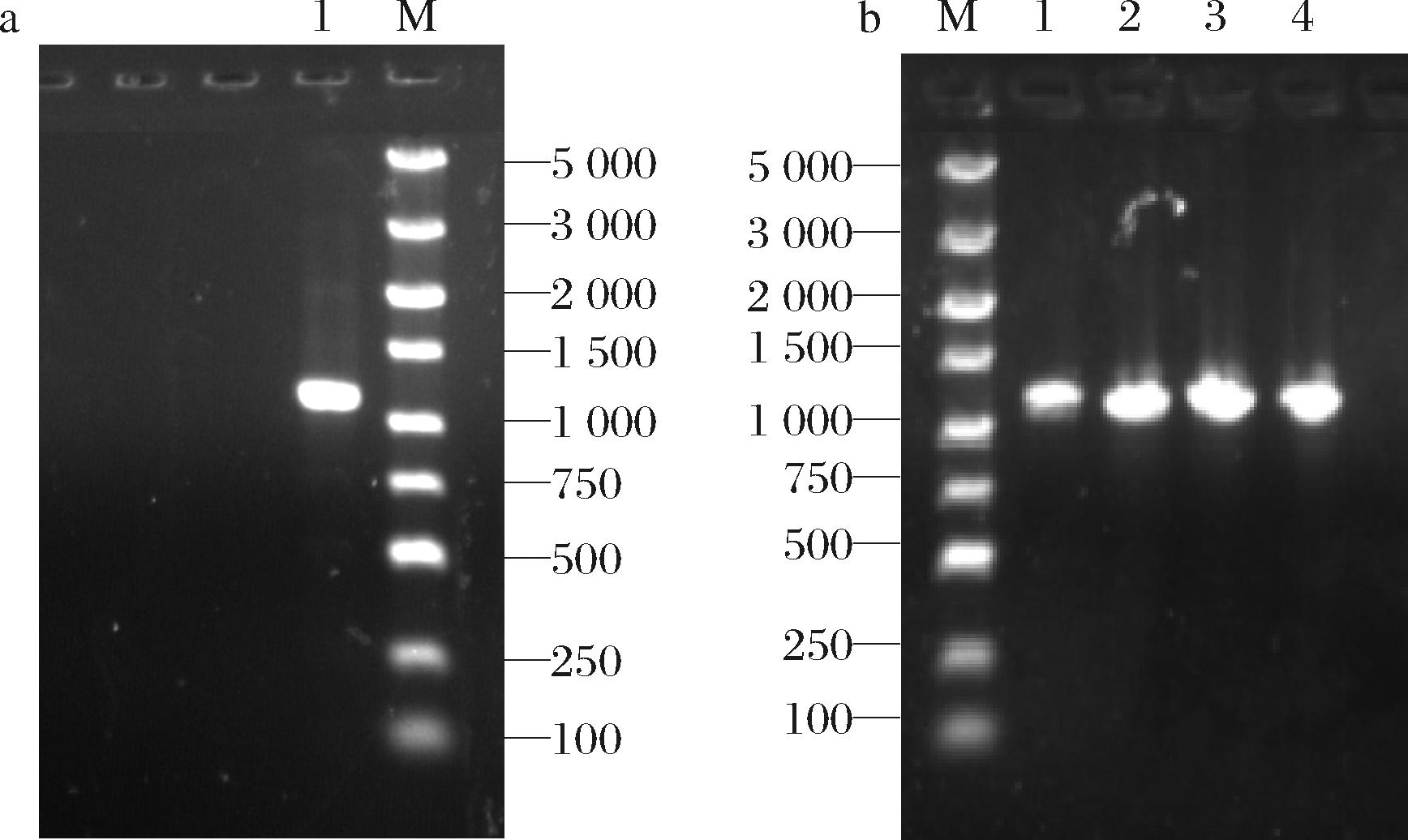
a-aprN克隆产物验证(M-Maker;1-aprN扩增产物);b-突变序列克隆产物验证(1-NK1扩增产物;2-NK2扩增产物;3-NK3扩增产物;4-NK4扩增产物)
图1 aprN和突变aprN序列的扩增
Fig.1 Amplification of aprN and mutant sequences
根据突变序列引物扩增,经琼脂糖凝胶电泳发现在1 200 bp附近有明显的扩增条带,这与预期的突变基因长度1 146 bp相吻合(图1-b)。
2.2 重组质粒双酶切
构建的5种重组质粒进行双酶切(图2),每个泳道均出现2个条带。其中大分子质量条带为双酶切的质粒,大小约为5 500 bp,小分子质量条带为切下的目的基因,大小约为1 200 bp,结果均符合预期,重组质粒构建成功。
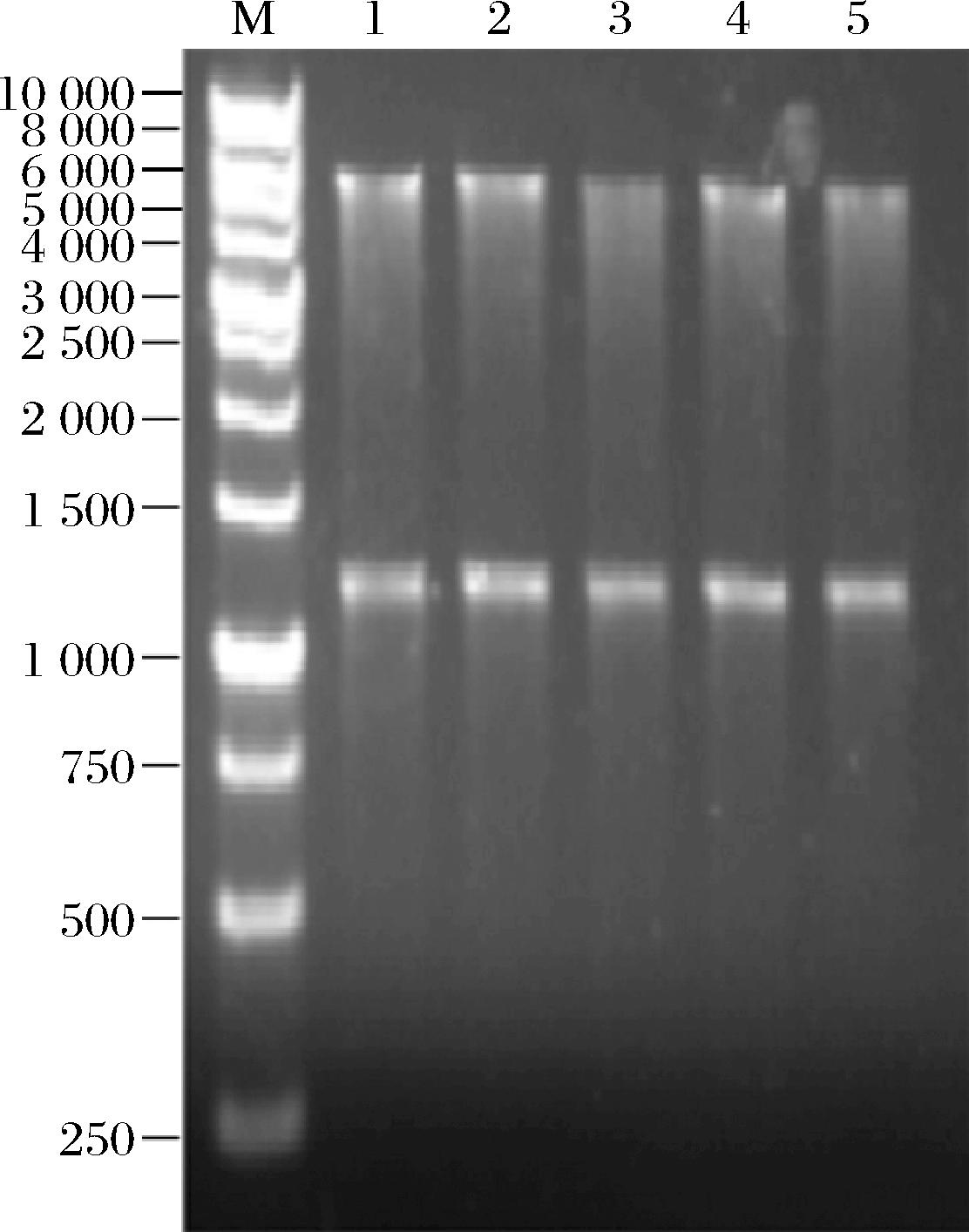
M-Marker;1-双酶切的pHY-P43-NK质粒;2-双酶切的pHY-P43-NK1质粒;3-双酶切的pHY-P43-NK2质粒;4-双酶切的pHY-P43-NK3质粒;5-双酶切的pHY-P43-NK4质粒
图2 重组质粒的双酶切
Fig.2 Double digestion of recombinant plasmids
2.3 重组质粒导入受体细胞转化验证
2.3.1 大肠杆菌转化验证
挑取在氨苄抗性平板上的大肠杆菌单菌落进行菌液PCR验证(图3),由于目的基因大小为1 146 bp,目标条带在1 200 bp附近符合预期。
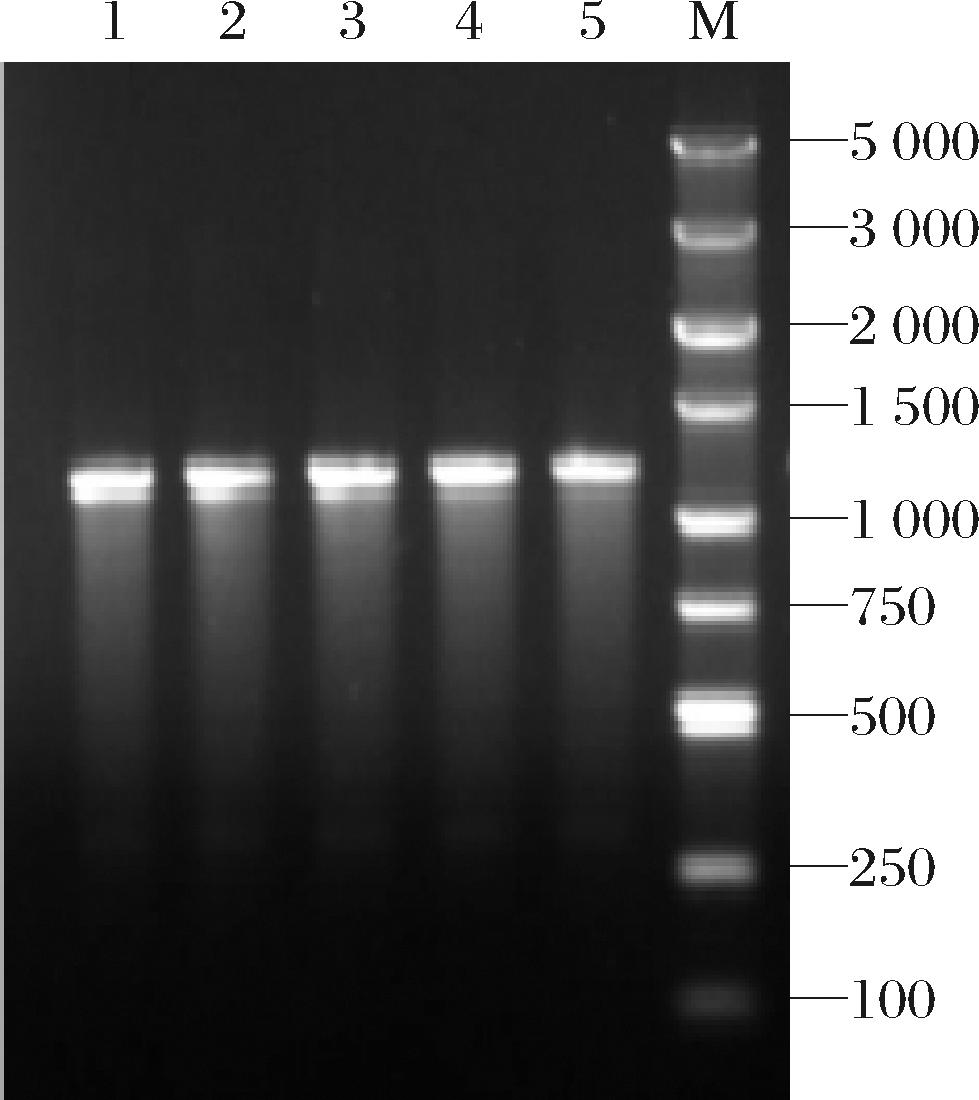
M-Marker;1-NK;2-NK1;3-NK2;4-NK3;5-NK4
图3 大肠杆菌转化验证电泳图
Fig.3 Electrophoretic analysis of successful transformation of mutant sequences in E.coli
2.3.2 枯草芽孢杆菌转化验证
在氨苄抗性平板上挑取枯草芽孢杆菌单菌落培养提取质粒,以质粒为模板利用目的基因上下游引物PCR扩增。目的基因大小为1 146 bp,电泳条带在1 200 bp附近符合预期(图4)。
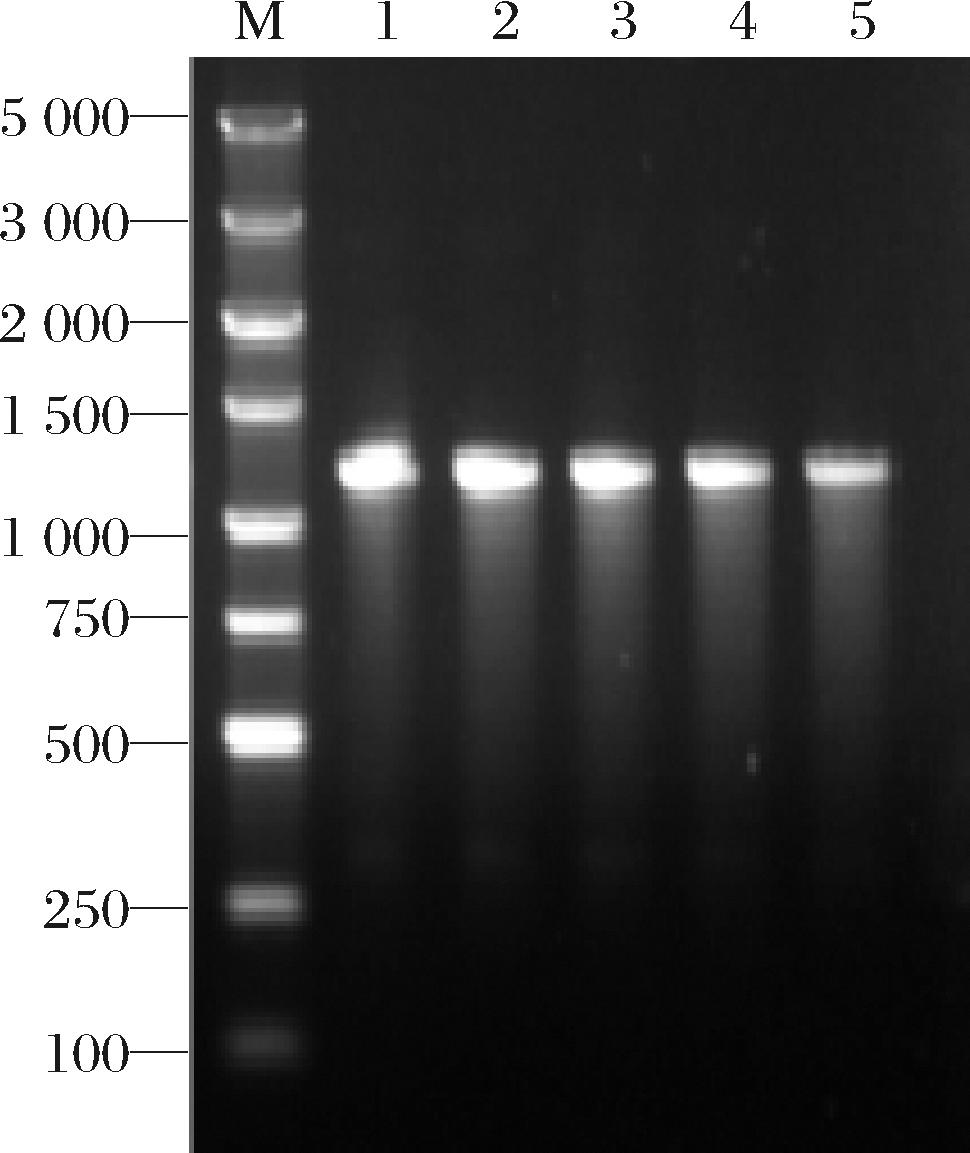
M-Marker;1-NK;2-NK1;3-NK2;4-NK3;5-NK4
图4 枯草芽孢杆菌转化验证电泳图
Fig.4 Electrophoretic analysis of successful transformation of mutant sequences in B.subtilis
2.4 目的基因测序
目的基因序列全长均为1 146 bp,由测序结果(图5)可以看出这5种基因序列同源性高达99%,且序列中前30个碱基的差异均符合预期,说明同义突变序列N1、N2、N3、N4构建成功。

图5 目的基因测序结果
Fig.5 Sequencing results of target genes of mutant strains
2.5 生长曲线的测定
测定构建的5株工程菌的生长曲线,由生长曲线可知(图6),除了在0~12 h的迟缓期,原始菌株NK生物量低于其他菌株外,5株工程菌的生长情况基本一致。在12~24 h时,5株工程菌处于对数生长期。选择同步发酵48 h,以保证纳豆激酶的有效积累。
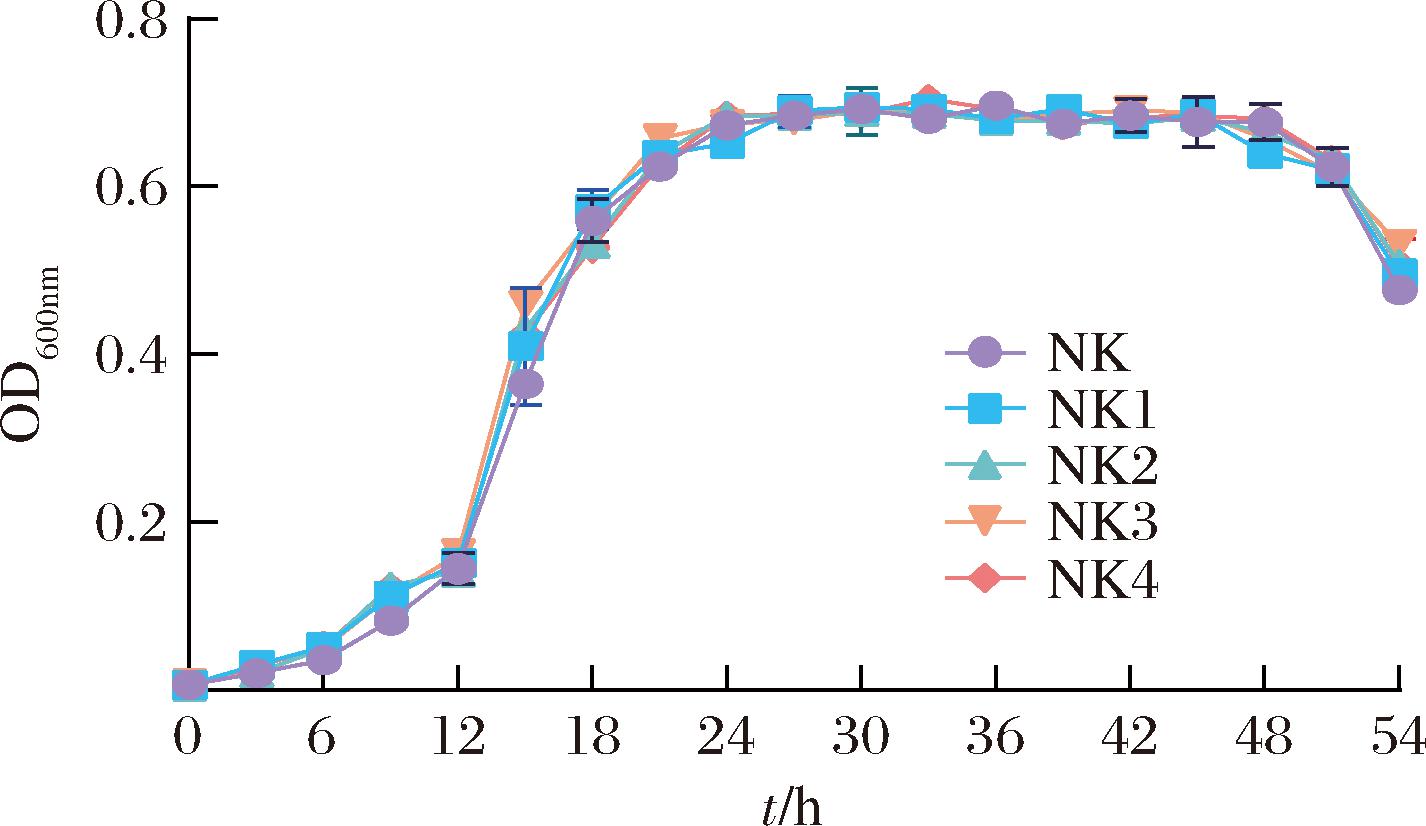
图6 五株工程菌生长曲线
Fig.6 Growth curves of five engineered strains
2.6 突变菌株的酶活力测定
纳豆激酶的分子质量约为28 kDa,5株菌株的冻干粉溶液在29.0 kDa下方均存在明显条带,表明构建的5种工程菌中均成功表达纳豆激酶(图7-a)。
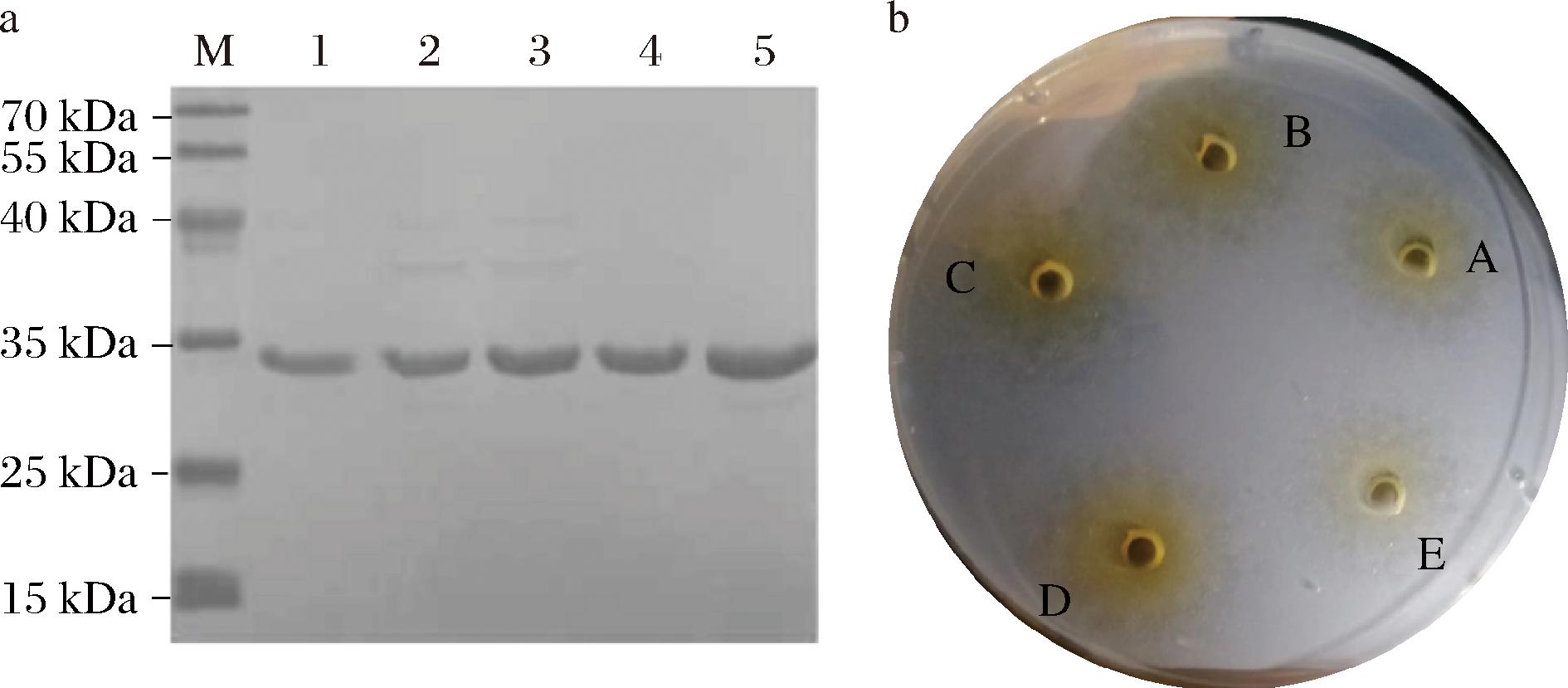
a-五株工程菌的SDS-PAGE分析(M-Maker;1-NK;2-NK1;3-NK2;4-NK3;5-NK4);b-五株工程菌纤维蛋白板的酶活测定(A-NK;B-NK1;C-NK2;D-NK3;E-NK4)
图7 五株工程菌的纳豆激酶活力检测
Fig.7 Nattokinase viability assay of five engineered strains
在相同的培养条件下,对构建的5株工程菌进行发酵,测定纳豆激酶活力(图7-b)。以原始基因aprN构建的NK菌株纳豆激酶活力为83.4 IU/mL;NK1菌株纳豆激酶活力为94.8 IU/mL;NK2菌株纳豆激酶活力为101.9 IU/mL;NK3菌株活性最高,达到124.2 IU/mL,约为原始菌株的1.5倍;NK4菌株活性最低仅为69.4 IU/mL,相比于原始菌株的酶活力下调约17%。
2.7 NK3菌株纳豆激酶表达发酵条件单因素试验
随着初始pH、发酵温度和发酵时间的增加,NK3突变菌株纳豆激酶的酶活力表现为先增加后减少的趋势(图8)。pH值在7.5时,纳豆激酶酶活力达到最大值,这可能是因为pH过酸、过碱对细菌的生长繁殖和酶蛋白的稳定性有影响,而纳豆激酶为碱性蛋白酶在酸性环境中酶活力受到抑制。发酵温度在35 ℃达到最大值,分析原因是温度过高、过低造成菌株生长缓慢,从而影响产物积累。发酵时间为36 h酶活力达到最大值可能是因为发酵时间过短,纳豆激酶未有效积累,而发酵时间过长,后期营养物资缺乏,纳豆激酶被菌株作为营养物质消耗利用。
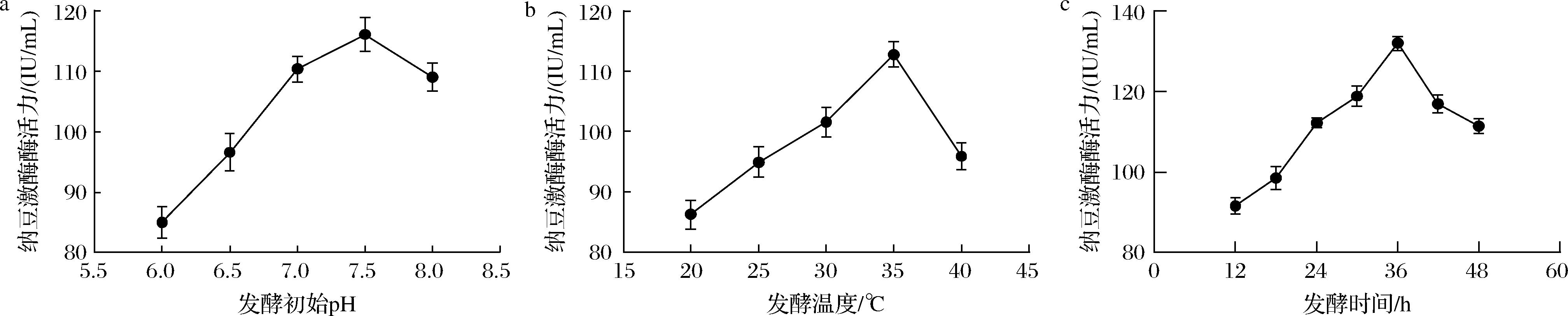
a-初始pH对酶活力的影响;b-发酵温度对酶活力的影响;c-发酵时间对酶活力的影响
图8 各因素对纳豆激酶酶活的影响
Fig.8 Effect of various factors on the enzymatic activity of nattokinase
2.8 响应面实验优化
根据单因素试验结果以初始pH(A)、发酵温度(B)和发酵时间(C)3个因素为自变量,纳豆激酶活力(Y)为响应值,Box-Behnken优化试验及方差分析结果如表4、表5所示。
表4 培养条件优化Box-Behnken试验设计及结果
Table 4 Design and results of Box-Behnken experiments for optimization of culture conditions
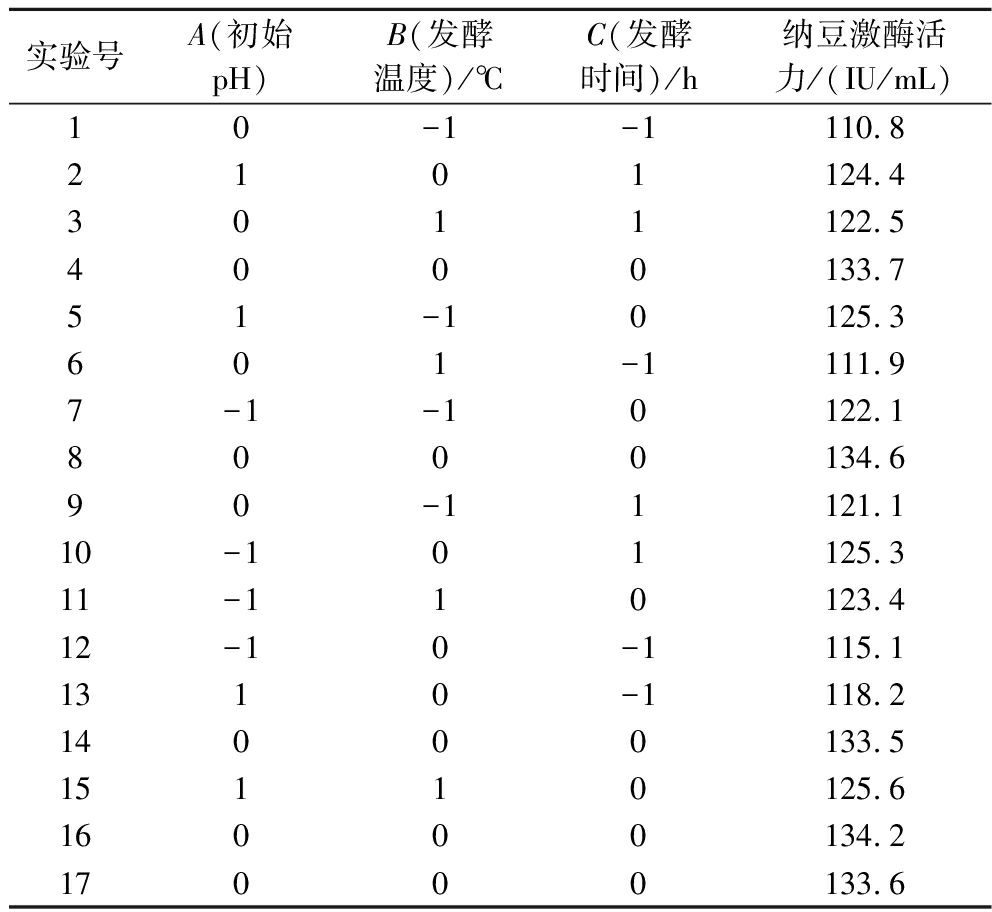
实验号A(初始pH)B(发酵温度)/℃C(发酵时间)/h纳豆激酶活力/(IU/mL)10-1-1110.82101124.43011122.54000133.751-10125.3601-1111.97-1-10122.18000134.690-11121.110-101125.311-110123.412-10-1115.11310-1118.214000133.515110125.616000134.217000133.6
表5 回归模型方差分析
Table 5 Variance analysis of regression model
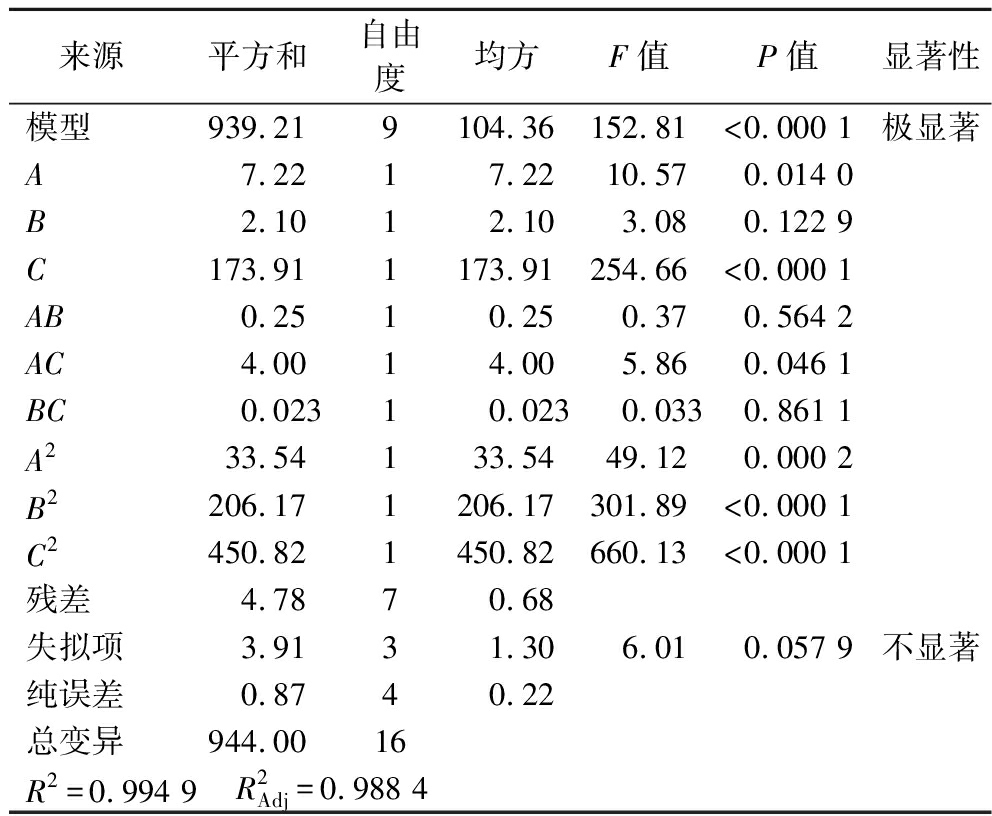
来源平方和自由度均方F值P值显著性模型939.219104.36152.81<0.000 1极显著A7.2217.2210.570.014 0B2.1012.103.080.122 9C173.911173.91254.66<0.000 1AB0.2510.250.370.564 2AC4.0014.005.860.046 1BC0.02310.0230.0330.861 1A233.54133.5449.120.000 2B2206.171206.17301.89<0.000 1C2450.821450.82660.13<0.000 1残差4.7870.68失拟项3.9131.306.010.057 9不显著纯误差0.8740.22总变异944.0016R2=0.994 9R2Adj=0.988 4
对Box-Benhnken试验结果进行响应面分析,得到纳豆激酶活力(Y)对初始pH(A)、发酵温度(B)和发酵时间(C)的多元二次回归方程:Y=0.95A+0.51B+4.66C-0.25AB-1.00AC+0.007 5BC-2.82A2-7.00B2-10.35C2+133.92。
从表4结果,回归模型极显著(P<0.000 1),失拟项(P=0.057 9>0.05)不显著,模型有较好的试验稳定性。模型C达到极显著水平,模型A和模型AC达到显著水平。R2=0.982 0,校正后的决定系数![]() 为0.958 9,两者差值小于0.2,模型有充分的准确性和通用性。
为0.958 9,两者差值小于0.2,模型有充分的准确性和通用性。
由响应面曲面图可知(图9),初始pH和发酵时间的交互作用更显著。利用回归模型,预测的最佳发酵条件为:初始pH 7.56、发酵温度35.18 ℃和发酵时间38.62 h,预测产量134.5 IU/mL。在优化条件下,调整初始pH 7.56、发酵温度35 ℃、发酵时间38 h,摇瓶发酵测得纳豆激酶活力为138.1 IU/ mL,与预测值基本相符。

a-初始pH和发酵温度对纳豆激酶酶活的影响;b-初始pH和发酵时间对纳豆激酶对酶活的影响;c-发酵温度和发酵时间对纳豆激酶酶活的影响
图9 各因素交互作用的响应面曲面图
Fig.9 Response surface plots of factor interactions
3 结论
本实验采用pHY-P43载体,根据枯草芽孢杆菌密码子偏好性,对纳豆激酶N-端前10个氨基酸残基设计4种aprN的同义序列,分别构建了NK、NK1、NK2、NK3和NK4共5株工程菌。测定纤溶活性发现,原始基因序列菌株NK纳豆激酶活力为83.4 IU/mL,NK1菌株的纳豆激酶活力为94.8 IU/mL,NK2菌株纳豆激酶活力为101.9 IU/mL,NK3菌株纳豆激酶活力最高,为124.2 IU/mL,约为原始菌株的1.5倍,NK4菌株纳豆激酶活力最低为69.4 IU/mL,相比于NK菌株酶活力下调约17%。SDS-PAGE获得纳豆激酶的分子质量约28 kDa,表明纳豆激酶检测结果的可信性。响应面优化得到最佳发酵条件:初始pH值为7.56、发酵温度为35 ℃、发酵时间为38 h,纳豆激酶活力为138.1 IU/mL,较未优化前提高11.2%。本研究验证了纳豆激酶N-端前10个氨基酸残基对应的密码子形成的mRNA二级结构及每个密码子第3位碱基的G或C频率对其编码的蛋白质含量有显著影响,为同义突变提高纳豆激酶的异源表达,以及纳豆激酶的产业化应用奠定一定的理论与技术基础。
[1] SHENG Y N, YANG J N, WANG C Y, et al.Microbial nattokinase:From synthesis to potential application[J].Food &Function, 2023, 14(6):2568-2585.
[2] 满丽莉, 向殿军.枯草芽孢杆菌MX-6产纳豆激酶特性分析[J].食品与发酵工业, 2019, 45(6):42-47.MAN L L, XIANG D J.Partial characteristics of nattokinase produced by Bacillus subtilis MX-6[J].Food and Fermentation Industries, 2019, 45(6):42-47.
[3] MODI A, RAVAL I, DOSHI P, et al.Heterologous expression of recombinant nattokinase in Escherichia coli BL21(DE3) and media optimization for overproduction of nattokinase using RSM[J].Protein Expression and Purification, 2023, 203:106198.
[4] CAI D, WEI X, QIU Y, et al.High-level expression of nattokinase in Bacillus licheniformis by manipulating signal peptide and signal peptidase[J].Journal of Applied Microbiology, 2016, 121(3):704-712.
[5] 冯浩, 余凤云, 单凤娟, 等.构建食品级表达纳豆激酶的乳酸乳球菌重组菌株[J].食品科学, 2012, 33(21):208-212.FENG H, YU F Y, SHAN F J, et al.Construction of a strain of recombinant Lactococcus lactis expressing food-grade nattokinase[J].Food Science, 2012, 33(21):208-212.
[6] 李怡欣, 付刚, 马媛媛, 等.碱性蛋白酶SubC在枯草芽孢杆菌中的高效异源表达[J].微生物学通报, 2021, 48(10):3409-3420.LI Y X, FU G, MA Y Y, et al.Efficient heterologous expression of alkaline protease SubC in Bacillus subtilis[J].Microbiology China, 2021, 48(10):3409-3420.
[7] 赵福永, 严寒, 任广旭, 等.重组纳豆激酶的研究进展[J].中国食物与营养, 2019, 25(7):41-45.ZHAO F Y, YAN H, REN G X, et al.Research advancement on recombinant nattokinase[J].Food and Nutrition in China, 2019, 25(7):41-45.
[8] 邓卉, 余丹, 邹成义, 等.大肠杆菌多酚氧化酶的分子克隆及异源高效表达[J].中国饲料, 2024(5):26-31.DENG H, YU D, ZOU C Y, et al.Molecular cloning of the polyphenol oxidase from Escherichia coli and its heterologous high-efficiency expression[J].China Feed, 2024(5):26-31.
[9] 赵越, 王新秀, 吴思, 等.KlacPNP基因密码子优化及在枯草芽胞杆菌中的高效表达[J].食品与发酵工业, 2023, 49(19):53-59.ZHAO Y, WANG X X, WU S, et al.Codon optimization and efficient expression of KlacPNP gene in Bacillus subtilis[J].Food and Fermentation Industries, 2023, 49(19):53-59.
[10] 陈永安, 袁清焱, 李承, 等.快速筛选高效表达重组蛋白毕赤酵母菌株新方法的建立及评价[J].生物工程学报, 2021, 37(3):939-949.CHEN Y A, YUAN Q Y, LI C, et al.Development and evaluation of a novel method for rapid screening of Pichia pastoris strains capable of efficiently expressing recombinant proteins[J].Chinese Journal of Biotechnology, 2021, 37(3):939-949.
[11] DUAN X G, WU J.Enhancing the secretion efficiency and thermostability of a Bacillus deramificans pullulanase mutant (D437H/D503Y) by N-terminal domain truncation[J].Applied and Environmental Microbiology, 2015, 81(6):1926-1931.
[12] WEBER M, BURGOS R, YUS E, et al.Impact of C-terminal amino acid composition on protein expression in bacteria[J].Molecular Systems Biology, 2020, 16(5):e9208.
[13] XU K D, TONG Y, LI Y, et al.Rational design of the N-terminal coding sequence for regulating enzyme expression in Bacillus subtilis[J].ACS Synthetic Biology, 2021, 10(2):265-276.
[14] CHEONG D E, KO K C, HAN Y, et al.Enhancing functional expression of heterologous proteins through random substitution of genetic codes in the 5′ coding region[J].Biotechnology and Bioengineering, 2015, 112(4):822-826.
[15] QUAX T E F, CLAASSENS N J, SÖLL D, et al.Codon bias as a means to fine-tune gene expression[J].Molecular Cell, 2015, 59(2):149-161.
[16] ZHENG Z L, YE M Q, ZUO Z Y, et al.Probing the importance of hydrogen bonds in the active site of the subtilisin nattokinase by site-directed mutagenesis and molecular dynamics simulation[J].Biochemical Journal, 2006, 395(3):509-515.
[17] 柳玫琪, 赵微, 秦雅丽, 等.植物乳杆菌p-8乙酰乙酸脱羧酶的原核表达及活性分析[J].内蒙古农业大学学报(自然科学版), 2022, 43(1):40-47.LIU M Q, ZHAO W, QIN Y L, et al.Prokaryotic expression and activity analysis of Lactobacillus plantarum P-8 acetoacetate decarboxylase[J].Journal of Inner Mongolia Agricultural University (Natural Science Edition), 2022, 43(1):40-47.
[18] GUPTA S K, GHOSH T C.Gene expressivity is the main factor in dictating the Codon usage variation among the genes in Pseudomonas aeruginosa[J].Gene, 2001, 273(1):63-70.
[19] WU J, LI W, ZHAO S G, et al.Site-directed mutagenesis of the quorum-sensing transcriptional regulator SinR affects the biosynthesis of menaquinone in Bacillus subtilis[J].Microbial Cell Factories, 2021, 20(1):113.
[20] ZHOU M J, WU J, HU L X, et al.Enhanced vitamin K2 production by engineered Bacillus subtilis during leakage fermentation[J].World Journal of Microbiology &Biotechnology, 2023, 39(8):224.
[21] 王镭. 纳豆激酶的制备与分离纯化研究进展[J].黑龙江科学, 2019, 10(8):8-10;17.WANG L.Progress in preparation, isolation and purification of nattokinase[J].Heilongjiang Science, 2019, 10(8):8-10;17.