κ-酪蛋白(kappa-casein, κ-CN, 编码基因为CSN3)是牛乳中一种含有少量磷酸基的磷蛋白之一,它与α-、β-酪蛋白共同组成牛乳酪蛋白[1]。κ-酪蛋白除了一般乳蛋白所具有的营养功能外,其水解还可释放出一种抗血栓十二肽,可以抑制ADP诱导的血小板凝集或纤维蛋白原结合过程,即具有抑制血液凝固和抵抗血栓形成的作用[2]。有研究表明,κ-酪蛋白经酶解后产生含糖肽能够刺激新生儿肠道中双歧杆菌生长和抑制大肠杆菌,因而有利于婴幼儿的消化道健康和防止腹泻[3]。有研究者从κ-酪蛋白的胰酶水解物中得到3种颉颃肽,它们在豚鼠回肠中显示出颉颃肽活性,与吗啡颉颃物有同样的效果[4]。因此,将来κ-酪蛋白可能作为食品添加剂,也可用于药物生产,用于治疗腹泻、矿物质吸收障碍和免疫缺乏及保健品的生产,发挥其免疫调节、抗血栓等功效。
目前κ-酪蛋白可以通过分级分离的方式从全脂乳或脱脂乳中沉淀提取,等电点沉淀法是分离牛乳中酪蛋白和非酪蛋白组分最广泛使用的一种方法[5-6]。通过基因工程技术生产牛乳蛋白也是常用的生产手段,研究者通过基因工程技术,选用小鼠[7]、牛、大肠杆菌[8]等生产κ-酪蛋白,但是研究人员对κ-酪蛋白的研究依然不够成熟和完善,例如大肠杆菌中表达量较低,利用哺乳动物细胞生产蛋白则存在操作技术难度大,费用成本高的问题,因此需要探索安全、高效、高产量的表达系统。谷氨酸棒状杆菌(Corynebacterium glutamicum)是一种革兰氏阳性、不产芽孢的兼性厌氧菌,已被广泛应用于化学品、燃料和生物聚合物的工业生产[9-11]。谷氨酸棒状杆菌也被认为是分泌表达重组蛋白的一个有吸引力的宿主,因为它有几个显著的优点:很少将内源性蛋白分泌到胞外,有利于目的蛋白的纯化[12];它可在廉价培养基中生长,已经建立成熟的工业规模发酵[13];无内毒素,是公认安全的菌株,可用于食品级产物的生产[14]。
但谷氨酸棒状杆菌表达系统也存在问题,如外源基因表达困难或表达量较少,重组蛋白表达的溶解性不高,重组蛋白易形成聚集体导致纯化难,为了能够进行后续的结构和功能研究,所获得的重组蛋白必须满足可溶性、稳定性、正确的折叠等要求。SUMO(small ubiquitin-like modifier)标签能够被SUMO蛋白酶特异识别并高效降解,使得后续标签移除过程准确高效[15-17]。SUMO标签在细菌中进行融合表达有着较为广泛地应用[18],不仅能提升重组蛋白的溶解度,也能提高其表达量[19-20]。FH8标签蛋白很小,分子质量只有8 kDa,效果较普通促溶标签蛋白更加明显,所以FH8标签蛋白相较于其他大分子融合标签可以作为一种新的促溶标签使用[21]。
本研究首次实现牛乳蛋白κ-酪蛋白在谷氨酸棒状杆菌中的可溶分泌表达,软件预测发现κ-酪蛋白的固有无序区域,并通过组合筛选促溶标签、信号肽,优化宿主进一步提高谷氨酸棒状杆菌表达系统可溶表达κ-酪蛋白的产量,最终纯化蛋白测定其产量,对谷氨酸棒状杆菌作为宿主表达牛乳蛋白具有重要的指导意义。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒和生长条件
菌株Escherichia coli JM109、C.glutamicum ATCC 13032、表达质粒pGX19及表达优化所用菌株均由本实验室保藏,Cg-ΔclpC为clpC蛋白酶基因敲除菌株,Cg-ΔclpS为clpS蛋白酶基因敲除菌株,MA6为突变菌株,Cg-ΔNCgl1689-ΔNCgl1716为基因NCgl1689敲除菌株,Cg-ΔNCgl1716为基因NCgl1716敲除菌株,Cg-ΔNCgl1689-ΔclpS为NCgl1689和clpS蛋白酶双基因敲除菌株,Cg-ΔNCgl1716-ΔclpS为NCgl1716和clpS蛋白酶双基因敲除菌株。
E.coli和C.glutamicum的氯霉素工作质量浓度分别为30 μg/mL和10 μg/mL。
LBB培养基(g/L):蛋白胨10,酵母提取物5,NaCl 10,脑心浸出液10,用于培养E.coli JM109和C.glutamicum ATCC 13032表达重组蛋白。
LBHIS培养基(g/L):蛋白胨5,酵母提取物2.5,NaCl 5,脑心浸出液18.5,D-山梨醇91,用于C.glutamicum ATCC 13032的活化和恢复培养。
所用质粒见表1。
表1 本研究所用质粒
Table 1 Plasmid in this study

质粒描述来源pGX19表达载体;Cmr本实验室pGX19-CSN3表达κ-酪蛋白;信号肽CspB;Cmr本研究pGX19-S-CspB-CSN3表达κ-酪蛋白;信号肽CspB;SU-MO标签;Cmr本研究pGX19-F-CspB-CSN3表达κ-酪蛋白;信号肽CspB;FH8标签;Cmr本研究pGX19-S-1514-CSN3表达κ-酪蛋白;信号肽1514;SU-MO标签;Cmr本研究pGX19-F-1514-CSN3表达κ-酪蛋白;信号肽1514;FH8标签;Cmr本研究pGX19-S-CAP-CSN3表达κ-酪蛋白;信号肽CAP;SU-MO标签;Cmr本研究pGX19-F-CAP-CSN3表达κ-酪蛋白;信号肽CAP;FH8标签;Cmr本研究pGX19-S-0949-CSN3表达κ-酪蛋白;信号肽0949;SU-MO标签;Cmr本研究pGX19-F-0949-CSN3表达κ-酪蛋白;信号肽0949;FH8标签;Cmr本研究pGX19-S-CspA-CSN3表达κ-酪蛋白;信号肽CspA;SU-MO标签;Cmr本研究pGX19-F-CspA-CSN3表达κ-酪蛋白;信号肽CspA;FH8标签;Cmr本研究pGX19-S-PorB-CSN3表达κ-酪蛋白;信号肽PorB;SU-MO标签;Cmr本研究pGX19-F-PorB-CSN3表达κ-酪蛋白;信号肽PorB;FH8标签;Cmr本研究pGX19-S-Hypothet-ical-CSN3表达κ-酪蛋白;信号肽Hypothet-ical;SUMO标签;Cmr本研究pGX19-F-Hypothet-ical-CSN3表达κ-酪蛋白;信号肽Hypothet-ical;FH8标签;Cmr本研究
1.1.2 主要试剂和仪器
PCR引物,苏州金唯智生物科技有限公司;DL 2 000 DNA maker、DL 10 000 DNA maker,Takara公司;Phanta酶、胶回收试剂盒、质粒提取试剂盒,南京诺唯赞生物技术公司;Hieff Canace酶,翌圣生物公司;同源重组酶,武汉Abclonal公司;氯霉素、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG),上海生工公司;HRP-conjugated 6×His (His),Proteintech公司;ETC821 PCR基因扩增仪,东胜公司;411BR8608 Micro Pulser电转仪,BIO-RAD;759S21023 UV-Vis Spectrophotometer紫外可见分光光度计,上海棱光公司;Megafuge 8高速离心机,Thermo公司。
1.2 实验方法
1.2.1 引物设计与基因合成
牛源κ-酪蛋白(GenBank:NP_776719.1)去除掉存在的信号肽序列和前肽序列后[8],对其进行密码子优化,在C端加上His标签,促溶标签FH8(GenBank:AF213970.1)及SUMO(GenBank:KP405837.1)基因序列由金唯智合成。
所用引物见表2。
表2 本研究所用引物
Table 2 Primers in this study
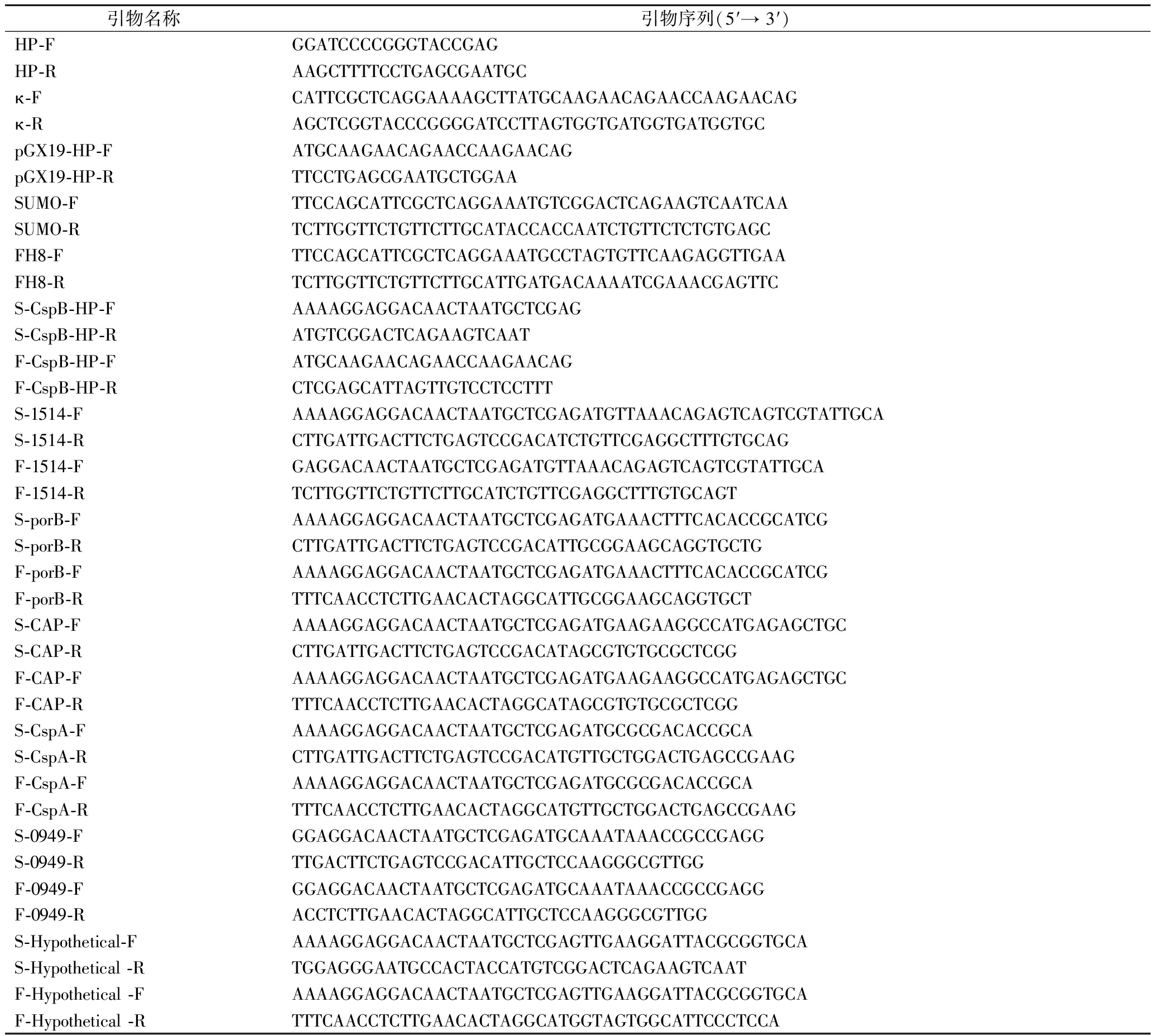
引物名称引物序列(5′→ 3′)HP-FGGATCCCCGGGTACCGAGHP-RAAGCTTTTCCTGAGCGAATGCκ-FCATTCGCTCAGGAAAAGCTTATGCAAGAACAGAACCAAGAACAGκ-RAGCTCGGTACCCGGGGATCCTTAGTGGTGATGGTGATGGTGCpGX19-HP-FATGCAAGAACAGAACCAAGAACAGpGX19-HP-RTTCCTGAGCGAATGCTGGAASUMO-FTTCCAGCATTCGCTCAGGAAATGTCGGACTCAGAAGTCAATCAASUMO-RTCTTGGTTCTGTTCTTGCATACCACCAATCTGTTCTCTGTGAGCFH8-FTTCCAGCATTCGCTCAGGAAATGCCTAGTGTTCAAGAGGTTGAAFH8-RTCTTGGTTCTGTTCTTGCATTGATGACAAAATCGAAACGAGTTCS-CspB-HP-FAAAAGGAGGACAACTAATGCTCGAGS-CspB-HP-RATGTCGGACTCAGAAGTCAATF-CspB-HP-FATGCAAGAACAGAACCAAGAACAGF-CspB-HP-RCTCGAGCATTAGTTGTCCTCCTTTS-1514-FAAAAGGAGGACAACTAATGCTCGAGATGTTAAACAGAGTCAGTCGTATTGCAS-1514-RCTTGATTGACTTCTGAGTCCGACATCTGTTCGAGGCTTTGTGCAGF-1514-FGAGGACAACTAATGCTCGAGATGTTAAACAGAGTCAGTCGTATTGCAF-1514-RTCTTGGTTCTGTTCTTGCATCTGTTCGAGGCTTTGTGCAGTS-porB-FAAAAGGAGGACAACTAATGCTCGAGATGAAACTTTCACACCGCATCGS-porB-RCTTGATTGACTTCTGAGTCCGACATTGCGGAAGCAGGTGCTGF-porB-FAAAAGGAGGACAACTAATGCTCGAGATGAAACTTTCACACCGCATCGF-porB-RTTTCAACCTCTTGAACACTAGGCATTGCGGAAGCAGGTGCTS-CAP-FAAAAGGAGGACAACTAATGCTCGAGATGAAGAAGGCCATGAGAGCTGCS-CAP-RCTTGATTGACTTCTGAGTCCGACATAGCGTGTGCGCTCGGF-CAP-FAAAAGGAGGACAACTAATGCTCGAGATGAAGAAGGCCATGAGAGCTGCF-CAP-RTTTCAACCTCTTGAACACTAGGCATAGCGTGTGCGCTCGGS-CspA-FAAAAGGAGGACAACTAATGCTCGAGATGCGCGACACCGCAS-CspA-RCTTGATTGACTTCTGAGTCCGACATGTTGCTGGACTGAGCCGAAGF-CspA-FAAAAGGAGGACAACTAATGCTCGAGATGCGCGACACCGCAF-CspA-RTTTCAACCTCTTGAACACTAGGCATGTTGCTGGACTGAGCCGAAGS-0949-FGGAGGACAACTAATGCTCGAGATGCAAATAAACCGCCGAGGS-0949-RTTGACTTCTGAGTCCGACATTGCTCCAAGGGCGTTGGF-0949-FGGAGGACAACTAATGCTCGAGATGCAAATAAACCGCCGAGGF-0949-RACCTCTTGAACACTAGGCATTGCTCCAAGGGCGTTGGS-Hypothetical-FAAAAGGAGGACAACTAATGCTCGAGTTGAAGGATTACGCGGTGCAS-Hypothetical -RTGGAGGGAATGCCACTACCATGTCGGACTCAGAAGTCAATF-Hypothetical -FAAAAGGAGGACAACTAATGCTCGAGTTGAAGGATTACGCGGTGCAF-Hypothetical -RTTTCAACCTCTTGAACACTAGGCATGGTAGTGGCATTCCCTCCA
1.2.2 表达载体的构建
κ-酪蛋白表达载体的构建:以HP-F和HP-R环形PCR pGX19质粒,κ-F和κ-R扩增κ-酪蛋白基因,用同源重组酶连接基因和载体,得到表达κ-酪蛋白的载体pGX19-CSN3,该基因在合成时于C端添加了His-tag以便后续纯化和分析。
促溶标签和信号肽组合筛选表达载体构建:以pGX19-HP-F和pGX19-HP-R为引物,pGX19-CSN3为模板扩增质粒,以SUMO-F、SUMO-R和FH8-F、FH8-R引物分别扩增SUMO基因和FH8基因。同源重组酶55 ℃,1 h分别连接pGX19和SUMO、FH8片段得到pGX19-S-CspB-CSN3和pGX19-F-CspB-CSN3。以S-CspB-HP-F、S-CspB-HP-R和F-CspB-HP-F、F-CspB-HP-R为引物分别扩增pGX19-S-CspB-CSN3和pGX19-F-CspB-CSN3载体,S-1514-F、S-1514-R和F-1514-F、F-1514-R引物分别扩增信号肽1514,将纯化后的1514片段分别重组连接到pGX19-S-CspB-CSN3和pGX19-F-CspB-CSN3载体,得到信号肽替换载体pGX19-S-1514-CSN3和pGX19-F-1514-CSN3,其余信号肽筛选质粒均用上述方法构建。
1.2.3 κ-酪蛋白表达菌株及宿主优化菌株的构建
将表1中载体通过电击转化方法转入C.glutamicum ATCC 13032感受态细胞[22],得到表达κ-酪蛋白初始表达菌株,将pGX19-S-CAP-CSN3电击转入Cg-ΔclpC、Cg-ΔclpS、Cg-MA6、Cg-Δ1716、Cg-Δ1689-1716、Cg-Δ1716-clpS、Cg-Δ1689-clpS中。所用菌株信息见表3。
表3 本研究所用菌株
Table 3 Strains used in this study

菌株名称基因型/特征来源E.coli JM109野生型实验室保藏C.glutamicum ATCC 13032野生型实验室保藏C.glutamicum ATCC 13032ΔclpSC.glutamicum ATCC 13032敲除蛋白酶clpS基因实验室保藏C.glutamicum ATCC 13032ΔclpCC.glutamicum ATCC 13032敲除蛋白酶clpC基因实验室保藏MA6C.glutamicum CGMCC1.15647经诱变所得实验室保藏C.glutamicum ATCC 13032Δ1716C.glutamicum ATCC 13032敲除蛋白酶调节亚基Ncgl1716基因实验室保藏C.glutamicum ATCC 13032Δ1716-clpSC.glutamicum ATCC 13032敲除蛋白酶调节亚基Ncgl1716和蛋白酶clpS基因实验室保藏C.glutamicum ATCC 13032Δ1689-clpSC.glutamicum ATCC 13032敲除蛋白酶调节亚基Ncgl1689和蛋白酶clpS基因实验室保藏C.glutamicum ATCC 13032Δ1689-1716C.glutamicum ATCC 13032敲除蛋白酶调节亚基Ncgl1689和蛋白酶1716基因实验室保藏
1.2.4 重组κ-酪蛋白的表达及SDS-PAGE和Western blotting分析
将Cg-PGX19-S-CAP-CSN3、Cg-ΔclpC-PGX19-S-CAP-CSN3、Cg-ΔclpS-PGX19-S-CAP-CSN3、Cg-PF6-PGX19-S-CAP-CSN3、Cg-MA6-PGX19-S-CAP-CSN3、Cg-ΔNcgl1716-PGX19-S-CAP-CSN3、Cg-ΔNcgl1689-PGX19-S-CAP-CSN3、Cg-ΔNcgl1716-ΔclpS-PGX19-S-CAP-CSN3、Cg-ΔNcgl1689-ΔclpS-PGX19-S-CAP-CSN3分别接种于2 mL的氯霉素抗性LBB培养基中,30 ℃、220 r/min摇床培养过夜,转接至5 mL新鲜氯霉素抗性的LBB培养基中,初始OD600值为0.2。当OD600值为1.0时,使用终浓度为1 mmol/L的IPTG诱导24 h后于12 000 r/min离心1 min收集菌体进行蛋白质分析,取离心后菌体,用磷酸盐缓冲液(PBS)清洗菌体3次,并用PBS重悬菌体,超声低温破碎(工作2 s,停2 s,共计9 min),对发酵液及破碎菌体上清液部分进行SDS-PAGE分析及Western blotting检测κ-酪蛋白含量。
Western blotting鉴定方法:将制成的蛋白样品进行SDS-PAGE(分离胶质量浓度为12 g/100 mL,电泳电压为120 V)。在电流作用下,将蛋白质从SDS-PAGE胶转印至固相载体聚偏二氟乙烯膜上,牛奶封闭1 h使抗体只能与特异性蛋白结合,组氨酸(His)抗体孵育1 h,TBST洗涤3次,加入化学发光底物ECL显色液,选用Image Lab软件通过凝胶成像仪进行曝光拍照。
1.2.5 κ-酪蛋白纯化及定量
采用镍预装柱(千纯生物公司)纯化κ-酪蛋白。工程菌发酵结束后收集500 mL发酵液,7 000 r/min离心10 min,弃上清液,将菌体用50 mL 10 mmol/L咪唑重悬,进行高压匀浆破碎(730 kPa破碎30 min)。破碎后菌体8 000 r/min离心40 min,取上清液过柱纯化。
纯化步骤:平衡:用3个体积的去离子水洗柱,再用3个体积10 mmol/L咪唑进行柱平衡;上样:平衡结束后上样,样品需用0.45 μm微孔滤膜过滤;冲洗:用2~3个柱体积的75 mmol/L咪唑冲洗上样后的层析柱;洗脱:用250 mmol/L咪唑进行恒定洗脱;清洗:5个柱体积500 mmol/L咪唑冲洗层析柱,3个体积去离子水冲洗,最后用体积分数20%乙醇冲洗柱子并封柱防止染菌。
牛血清白蛋白(bovine serum albumin,BSA)蛋白定量:将BSA标准品定量稀释至1、0.5、0.25、0.125、0.062 5、0.031 25 mg/mL,利用SDS-PAGE分析蛋白产量和纯度,Image J软件计算SDS-PAGE的灰度值,根据不同浓度BSA标品绘制标准曲线,对κ-酪蛋白进行定量测定。
1.2.6 κ-酪蛋白的二级结构及无序性分析
将κ-酪蛋白氨基酸序列输入AlphaFold的搜索栏,点击“Search”即可出现该蛋白的信息,在“Structure”信息栏中查找其结构图。
进入PONDR网站,网址为http://www.pondr.com/。在“Protein Name”栏中输入“κ-casein”,然后将蛋白氨基酸序列的FASTA格式输入“Sequence”框中,点击“Submit Query”即可获取κ-酪蛋白的有序和无序区域预测图。
进入Iupred2a数据库,网址为https://iupred2a.elte.hu/。在“or paste the amino acid sequence”输入κ-酪蛋白氨基酸序列,随后点击“Sumit”即可获取κ-酪蛋白无序情况的预测结果。
2 结果与分析
2.1 κ-酪蛋白在谷氨酸棒状杆菌中的表达
将经测序验证正确的质粒pGX19-CSN3转入谷氨酸棒状杆菌ATCC 13032中进行摇瓶发酵,IPTG诱导24 h。诱导结束后,取收集发酵上清液作胞外分泌表达样本,将离心沉淀重悬后破碎,收集破碎后液体作胞内全菌表达样品,破碎后上清液作胞内可溶表达样品进行SDS-PAGE和Western bloting分析,如图1所示,与含空载体pGX19的对照菌株相比,引入κ-酪蛋白的菌株发酵上清液、破碎沉淀、破碎上清液中均出现分子质量约为35 kDa的条带,高于目的蛋白的理论分子质量23.8 kDa,这可能是由于κ-酪蛋白属无序蛋白,其中存在大量极性氨基酸,且带较多负电荷,因此存在分子间和分子内的静电排斥,所以蛋白在SDS-PAGE迁移速率会变慢,从而导致目的条带位置变大。SDS-PAGE图中35 kDa处未见明显条带,可见κ-酪蛋白初步表达水平较低,仅Western bloting能检测到。

a-κ-酪蛋白发酵上清液(胞外分泌)、破碎上清液(胞内可溶)和破碎沉淀的SDS-PAGE分析结果;b-κ-酪蛋白发酵上清液、破碎上清液和破碎沉淀的Western bloting分析结果
图1 κ-酪蛋白在谷氨酸棒状杆菌中的初步表达情况
Fig.1 Preliminary expression of κ-casein in C.glutamicum
注:L1、L3、L5分别对应对照菌株Cg-pGX19的发酵上清液、破碎上清液和破碎沉淀;L2、L4、L6分别对应κ-酪蛋白表达菌株Cg-pGX19-CSN3的发酵上清液、破碎上清液和破碎沉淀。
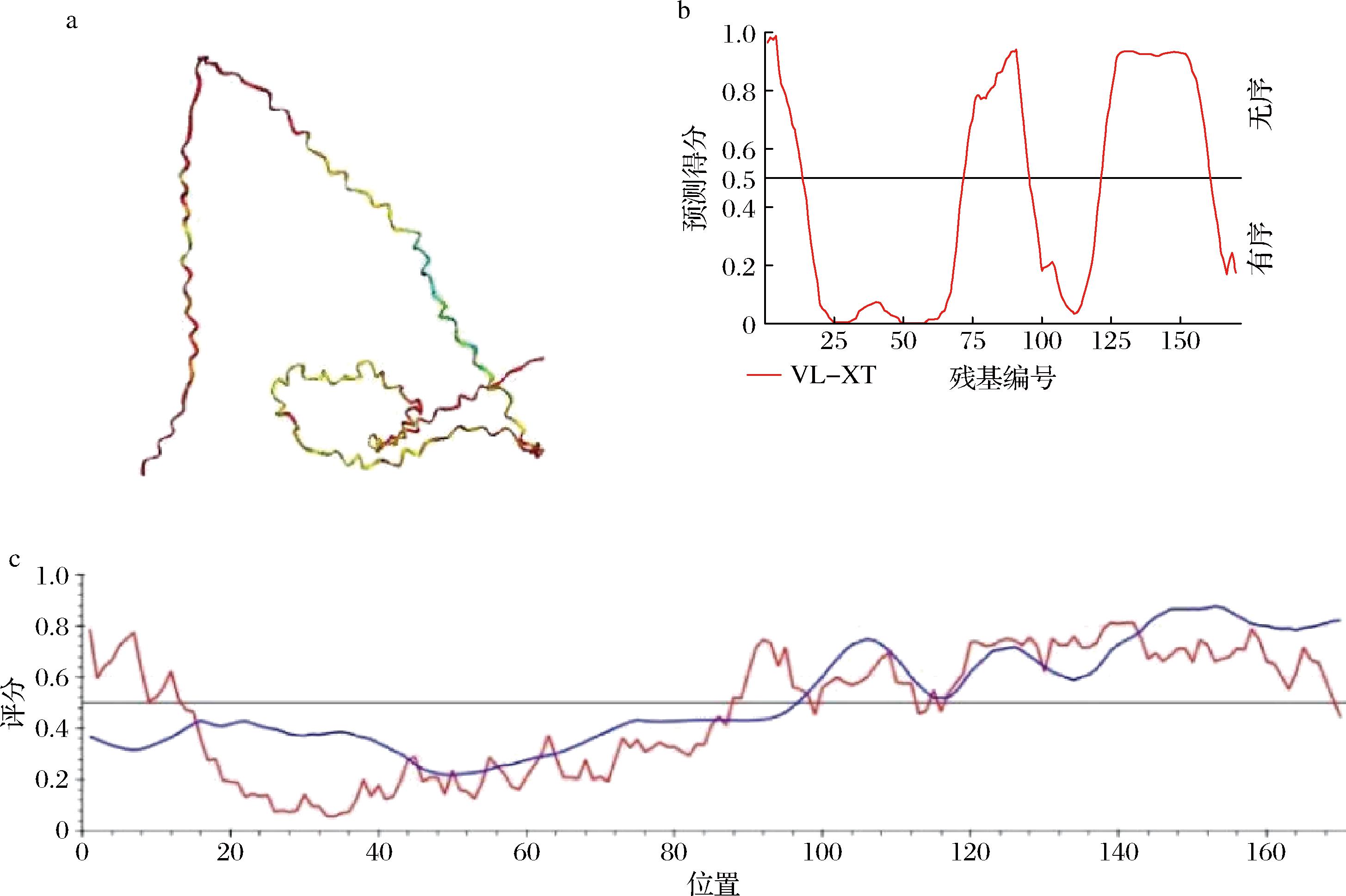
a-AlphaFold网站结构预测结果图;b-PONDR网站无序区域预测(纵坐标为PONDR分数,横坐标为该蛋白氨基酸位置,VSL2 NNP STATISTICS为κ-酪蛋白序列有序和无序区域的氨基酸位置和长度);c-Iupred2a数据库无序预测(红线表示蛋白无序区域预测结果,蓝线为预测的无序结合区)
图2 κ-酪蛋白三级结构及无序性分析
Fig.2 Three-order structure and disorder analysis of κ-casein
2.2 κ-酪蛋白三级结构及无序性分析
在AlphaFold得到κ-酪蛋白的结构图如图2-a所示。根据PONDR网站的无序性预测结果,如图2-b所示,PONDR分数高于0.5的为无序区域,低于0.5分的为有序区域,图中显示κ-酪蛋白共有3个无序部分,分别为第1位氨基酸到第13位氨基酸,第72位氨基酸到第95位氨基酸,第122位氨基酸到第160位氨基酸,其余部分均为有序区域。根据Iupred2a数据库预测,如图2-c所示,观察该图红线部分,分数大于0.5(横线上方)为无序区域,分数小于0.5(横线下方)为有序区域,这和PONDR网站预测结果基本一致。预测结果证实了κ-酪蛋白表观分子质量比实际分子质量大是由于其属于无序蛋白的预测。
2.3 促溶标签和信号肽组合筛选κ-酪蛋白最优表达菌株
为了能够进行后续结构和功能研究,所获得的重组蛋白必须满足可溶性、稳定性等要求,在实际研究中,这些因素通常成为获得一个理想重组蛋白的障碍。一个重组蛋白被宿主表达后,其可溶性就成为首要解决的问题,可添加促溶标签以提高蛋白的可溶性,与此同时,促溶标签的促溶机制并不十分明确,它们各自所适用的范围也不清楚[23],本研究根据文献调研选择2种促溶标签SUMO及FH8进行κ-酪蛋白表达尝试,SUMO标签分子质量大小约为15 kDa,其融合脑钠肽(brain natriuretic peptide,BNP)后脑钠肽的表达量有所提升[24]。FH8为新型促溶标签,分子质量较小约为8 kDa,相比较大的融合标签,小标签更易评估目的蛋白的可溶性[21]。信号肽种类不同,介导外源蛋白分泌的能力也有所差别。为研究不同促溶标签及信号肽对重组牛κ-酪蛋白表达的影响,在促溶标签前端分别引入谷氨酸棒状杆菌常用信号肽CspB、CspA、porB、Cg1514、CgR0949、CAP、Hypothetical(质粒构建如图3-a所示),信号肽参考文献[25],7种信号肽与2种促溶标签组成14种组合(图3-b)探究κ-酪蛋白的表达情况。
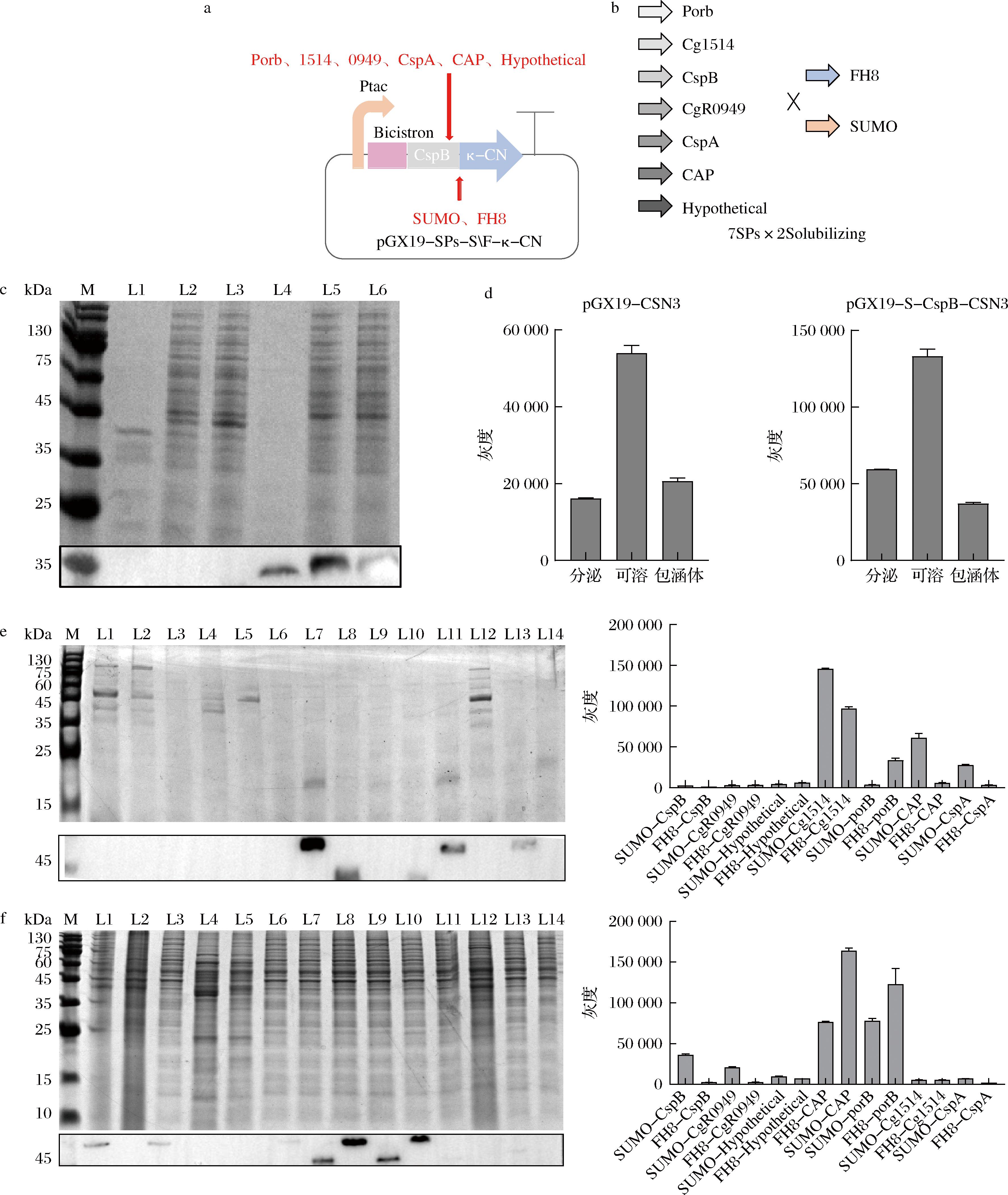
a-信号肽、促溶标签筛选质粒构建示意图(Ptac:诱导型tac启动子;Bicistron:双顺反子;CspB:信号肽;κ-CN:κ-酪蛋白;SUMO、FH8及箭头指向为促溶标签添加位置);b-信号肽与促溶标签组合方式(左侧为7种信号肽,右侧为两种促溶标签);c-Cg-pGX19和Cg-pGX19-S-CspB菌株的SDS-PAGE与Western blotting分析(L1、L2、L3分别对应pGX19的分泌、可溶、包涵体,L4、L5、L6分别对应pGX19-S-CspB-CSN3的分泌、可溶、包涵体);d-Cg-pGX19-CspB-CSN3及Cg-pGX19-S-CspB-CSN3灰度分析;e、f分别为14种信号肽与促溶标签组合的分泌表达、可溶表达SDS-PAGE和Western blotting分析以及灰度分析(L1-Cg-pGX19-S-CspB-CSN3;L2-Cg-pGX19-F-CspB-CSN3;L3-Cg-pGX19-S-CgR0949-CSN3;L4-Cg-pGX19-F-CgR0949-CSN3;L5-Cg-pGX19-S-Hypothetical-CSN3;L6-Cg-pGX19-F- Hypothetical-CSN3;L7-Cg-pGX19-F-CAP-CSN3;L8-Cg-pGX19-S-CAP-CSN3;L9-Cg-pGX19-S-porB-CSN3;L10-Cg-pGX19-F-porB-CSN3;L11-Cg-pGX19-S-Cg1514-CSN3;L12-Cg-pGX19-F-Cg1514-CSN3; L13-Cg-pGX19-S-CspA-CSN3;L14-Cg-pGX19-F-CspA-CSN3 (S为SUMO,F为FH8)
图3 信号肽和促溶标签优化后κ-酪蛋白表达情况分析
Fig.3 Signal peptide and solubilization tag screening

a-改造菌株κ-酪蛋白可溶表达SDS-PAGE及Western blotting分析(L1-Cg-pGX19-S-CAP-CSN3;L2-Cg-ΔclpS-pGX19-S-CAP-CSN3;L3-Cg-ΔclpC-pGX19-S-CAP-CSN3;L4-Cg-Δ-pGX19-S-CAP-CSN3L5L6L7L8);b-对Western blotting进行灰度分析
图4 κ-酪蛋白宿主优化
Fig.4 κ-casein host optimization

a-κ-酪蛋白纯化SDS-PAGE与Western blotting分析(L1~L12-分别为第1~12 mL时的洗脱蛋白样品);b-BSA定量SDS-PAGE分析(L1~L6为不同浓度BSA标准品;L1-1 mg/mL;L2-0.5 mg/mL;L3-0.25 mg/mL;L4-0.125 mg/mL;L5-0.062 5 mg/mL;L6-0.031 25 mg/mL;L7-κ-酪蛋白纯化样品);c-BSA定量标准曲线
图5 κ-酪蛋白纯化及BSA定量
Fig.5 Purification of κ-casein and quantification of BSA
首先在pGX19-CSN3基础上添加SUMO标签进行表达尝试,对其发酵上清液、破碎上清液及破碎沉淀进行SDS-PAGE和Western blotting分析,如图3-c和图3-d结果及灰度分析表明,添加SUMO标签使κ-酪蛋白的胞内可溶表达量有显著提高,约提高2.3倍,分泌水平约提高3.6倍。随后将上述构建的14种表达载体转入C.glutamicum ATCC 13032中进行摇瓶诱导发酵,诱导结束取发酵上清液、破碎上清液及破碎沉淀进行SDS-PAGE和Western blotting分析,同时对发酵上清液及破碎上清液进行灰度分析,图3-e结果显示,由于分泌表达量较低,只能通过Western blotting检测,与SUMO-CspB相比,SUMO-Cg1514组合分泌效果最好。图3-f结果显示,SUMO-CAP组合可溶表达量有明显提升,较SUMO-CspB提高约4倍。
2.4 κ-酪蛋白表达宿主优化
谷氨酸棒状杆菌作为一种常用原核表达宿主,其经济性以及安全性是κ-酪蛋白表达的合适宿主,但不同表达宿主对异源蛋白的表达各有优缺点,对此,为进一步提高κ-酪蛋白的表达量,在前期研究基础上,选择实验室已存8种经ARTP诱变或蛋白酶敲除的改造菌株Cg-ΔclpC、Cg-ΔclpS、Cg-MA6、Cg-ΔNcgl1716、Cg-ΔNcgl1689-ΔNcgl1716、Cg-ΔNcgl1716-ΔclpS、Cg-ΔNcgl1689-ΔclpS进行研究,表达质粒选择前期研究中筛选出的表达元件组合pGX19-S-CAP-CSN3。由于κ-酪蛋白分泌水平表达量较低,本阶段研究只选用胞内可溶表达样品进行分析。与原始菌株C.glutamicum ATCC 13032(图4-b泳道L1)相比,Cg-ΔNcgl1716可溶表达量有显著提升,约3.21倍,MA6提高约2倍,菌株Cg-ΔclpC、Cg-ΔclpS、Cg-ΔNcgl1689-ΔNcgl1716、Cg-ΔNcgl1716-ΔclpS、Cg-ΔNcgl1689-ΔclpS未见有明显提高。
2.5 κ-酪蛋白的发酵制备与蛋白纯化
将目前最优表达系统Cg-Δ1716-pGX19-S-CAP-CSN3置于摇瓶中发酵,在值OD600值为1.0时添加终浓度为1 mmol/L IPTG,诱导发酵结束后,高压匀浆破碎菌体,破碎上清液为κ-酪蛋白胞内可溶蛋白,对上清液使用0.45 μm的纤维滤膜过滤后进行镍柱纯化后并洗脱,如图5-a可见,1~3 mL及6~12 mL洗脱时杂带较少,且45 kDa处可见明显条带。由于蛋白表达量较低,κ-酪蛋白无法应用BCA检测,将BSA蛋白标准品梯度稀释后进行SDS-PAGE分析,经灰度分析拟合标准曲线作为纯化后蛋白产量的测定依据(图5-b、图5-c),纯化后的κ-酪蛋白条带灰度值代入标准曲线处理,得出500 mL发酵液中得到纯化κ-酪蛋白的产量为0.088 mg。
3 结论
随着合成生物学的兴起,人们开始利用一系列模式微生物和植物作为底盘细胞生产异源乳蛋白。目前国内外关于牛源κ-酪蛋白异源表达的研究较少。庞雨[8]以大肠杆菌为底盘细胞异源表达κ-酪蛋白在内的8种牛乳蛋白,而κ-酪蛋白最初未能表达,添加融合标签后才成功表达。相较于产生内毒素的大肠杆菌,本研究以食品级菌株谷氨酸棒状杆菌为底盘菌株生产食用蛋白更为安全。同时在未添加标签时,κ-酪蛋白就能够在谷氨酸棒状杆菌中成功表达,后续通过引入不同促溶标签及信号肽组合介导κ-酪蛋白的分泌及可溶表达,Sec依赖型信号肽CAP与小泛素标签SUMO组合使重组蛋白可溶性表达量更高,经改造菌株优化,κ-酪蛋白的产量提高到原来的3.21倍,最终500 mL发酵液中纯化产量为0.088 mg。经蛋白结构分析及无序性分析,证明κ-酪蛋白中有三部分的无序区域,这为以后生产无膜细胞器提供了有力依据。同时,添加融合标签、表达元件以及宿主优化能够有效提升重组蛋白表达量,为工业化生产水平的进一步方法试验提供了指导,牛乳蛋白在原核中成功表达也为未来乳制品的全合成奠定了坚实基础。
本研究将κ-酪蛋白在谷氨酸棒状杆菌中表达,后续可通过培养基、发酵温度、溶氧等方面进一步优化提高κ-酪蛋白的表达量。同时,目前对于κ-酪蛋白活性的探究报道较少,可进一步探索κ-酪蛋白的简易活性测定方法。本研究中谷氨酸棒状杆菌可溶表达系统的建立,为κ-酪蛋白的重组表达提供了新的宿主选择,也为宿主表达异源重组蛋白可溶性不高的问题提供了新的解决思路和理论依据。
[1] MALIK S, SIDHU N S, KUMAR S, et al.Kappa-casein alleles in Zebu and cross-bred (1/2 Friesian, 1/4 Jersey, 1/4 Hariana) cattle from India using polymerase chain reaction and sequence-specific oligonucleotide probes (PCR-SSOP)[J].Genetic Analysis:Biomolecular Engineering, 1997, 14(2):61-63.
[2] CAPRIOTTI A L, CAVALIERE C, PIOVESANA S, et al.Recent trends in the analysis of bioactive peptides in milk and dairy products[J].Analytical and Bioanalytical Chemistry, 2016, 408(11):2677-2685.
[3] MIGLIORE-SAMOUR D, JOLL S P.Casein, a prohormone with an immunomodulating role for the newborn?[J].Experientia, 1988, 44(3):188-193.
S P.Casein, a prohormone with an immunomodulating role for the newborn?[J].Experientia, 1988, 44(3):188-193.
[4] BERTHOU J, MIGLIORE-SAMOUR D, LIFCHITZ A, et al.Immunostimulating properties and three-dimensional structure of two tripeptides from human and cow caseins[J].FEBS Letters, 1987, 218(1):55-58.
[5] FOX P F.Milk proteins:General and historical aspects[M].Advanced Dairy Chemistry—1 Proteins.Boston, MA:Springer US, 2003:1-48.
[6] O’MAHONY J A, FOX P F.Milk proteins:Introduction and historical aspects[M].Advanced Dairy Chemistry.Boston, MA:Springer US, 2012:43-85.
[7] GUTIÉRREZ-AD N A, MAGA E A, MEADE H, et al.Alterations of the physical characteristics of milk from transgenic mice producing bovine κ-casein[J].Journal of Dairy Science, 1996, 79(5):791-799.
N A, MAGA E A, MEADE H, et al.Alterations of the physical characteristics of milk from transgenic mice producing bovine κ-casein[J].Journal of Dairy Science, 1996, 79(5):791-799.
[8] 庞雨. 牛乳蛋白在大肠杆菌中的异源表达和组合合成[D].北京:中国农业科学院, 2021.PANG Y.Heterologous expression and combinatorial synthesis of bovine milk proteins in Escherichia coli[D].Beijing:Chinese Academy of Agricultural Sciences, 2021.
[9] BECKER J, ROHLES C M, WITTMANN C.Metabolically engineered Corynebacterium glutamicum for bio-based production of chemicals, fuels, materials, and healthcare products[J].Metabolic Engineering, 2018, 50:122-141.
[10] CHENG F Y, YU H M, STEPHANOPOULOS G.Engineering Corynebacterium glutamicum for high-titer biosynthesis of hyaluronic acid[J].Metabolic Engineering, 2019, 55:276-289.
[11] XU G Q, ZHA J, CHENG H, et al.Engineering Corynebacterium glutamicum for the de novo biosynthesis of tailored poly-γ-glutamic acid[J].Metabolic Engineering, 2019, 56:39-49.
[12] YU X Y, LIU X X, GAO X, et al.Development of a novel platform for recombinant protein production in Corynebacterium glutamicum on ethanol[J].Synthetic and Systems Biotechnology, 2022, 7(2):765-774.
[13] PARK S H, KIM H U, KIM T Y, et al.Metabolic engineering of Corynebacterium glutamicum for L-arginine production[J].Nature Communications, 2014, 5:4618.
[14] LIU X X, YANG Y K, ZHANG W, et al.Expression of recombinant protein using Corynebacterium Glutamicum:Progress, challenges and applications[J].Critical Reviews in Biotechnology, 2016, 36(4):652-664.
[15] SATAKARNI M, CURTIS R.Production of recombinant peptides as fusions with SUMO[J].Protein Expression and Purification, 2011, 78(2):113-119.
[16] JOHANSSEN V A, BARNHAM K J, MASTERS C L, et al.Generating recombinant C-terminal prion protein fragments of exact native sequence[J].Neurochemistry International, 2012, 60(3):318-326.
[17] LU W G, CAI X T, GU Z H, et al.Production and characterization of hirudin variant-1 by SUMO fusion technology in E.coli[J].Molecular Biotechnology, 2013, 53(1):41-48.
[18] SINGH A, YADAV D, RAI K M, et al.Enhanced expression of rabies virus surface G-protein in Escherichia coli using SUMO fusion[J].The Protein Journal, 2012, 31(1):68-74.
[19] LI J F, ZHANG J, ZHANG Z, et al.SUMO mediating fusion expression of antimicrobial peptide CM4 from two joined genes in Escherichia coli[J].Current Microbiology, 2011, 62(1):296-300.
[20] WANG Z Y, LI N, WANG Y Y, et al.Ubiquitin-intein and SUMO2-intein fusion systems for enhanced protein production and purification[J].Protein Expression and Purification, 2012, 82(1):174-178.
[21] COSTA S J, COELHO E, FRANCO L, et al.The Fh8 tag:A fusion partner for simple and cost-effective protein purification in Escherichia coli[J].Protein Expression and Purification, 2013, 92(2):163-170.
[22] VAN DER REST M E, LANGE C, MOLENAAR D.A heat shock following electroporation induces highly efficient transformation of Corynebacterium glutamicum with xenogeneic plasmid DNA[J].Applied Microbiology and Biotechnology, 1999, 52(4):541-545.
[23] 李祥魁, 范翠英, 崔亚君, 等.重组蛋白可溶性表达促进标签的研究进展[J].生物技术, 2013, 23(2):93-97.LI X K, FAN C Y, CUI Y J, et al.Recent advances on the solubility-enhancing fusion tags for recombinant protein expression[J].Biotechnology, 2013, 23(2):93-97.
[24] 彭枫, 刘秀霞, 陈静, 等.不同表达模式组合对谷氨酸棒状杆菌中脑钠肽蛋白表达的影响[J].食品与发酵工业, 2018, 44(8):1-7.PENG F, LIU X X, CHEN J, et al.The effect of different expression patterns on B-type natriuretic peptide expression in Corynebacterium glutamicum[J].Food and Fermentation Industries, 2018, 44(8):1-7.
[25] SUN M M, GAO A X, LEDESMA-AMARO R, et al.Hypersecretion of OmlA antigen in Corynebacterium glutamicum through high-throughput based development process[J].Applied Microbiology and Biotechnology, 2022, 106(8):2953-2967.