三乙酸内酯(triacetic acid lactone,TAL)是从植物非洲菊中鉴定和提取出来的一种聚酮化合物[1],其化学名称为4-羟基-6-甲基-2-吡喃酮[2]。在Ⅲ型PKS-2-pyrone合成酶(2-PS)的催化下,一分子乙酰辅酶A分子和两分子丙二酰辅酶A缩合生成一分子TAL[3]。TAL是一种具有广泛工业应用的平台分子:可以作为前体合成具有抗菌、抗真菌和抗病毒活性的间二苯酚和可作为改性剂用于合成木材胶黏剂的间苯三酚[4-6];可以作为前体合成抗生素广藿香酮及其类似物[7];还可以用于生产食品添加剂山梨酸或燃料添加剂乙酰丙酮[8-10]。
与传统的植物提取法或者化学合成法相比,通过微生物工程生产TAL具有原材料价格低、生产成本低、环境友好、副产品少等诸多优势[11-12]。宿主的选择通常取决于异源途径的来源和生产的代谢物类型。与原核微生物相比,真菌宿主由于其工业发酵的天然耐受性而更显优势。酿酒酵母是以生物为基础生产多种化学品和燃料的强大宿主,也是真核生物功能基因组学的重要模式生物,其优势包括遗传背景研究充分,对恶劣环境条件的耐受性强、在遗传上更易于处理和有长期的工业使用历史等[13-14]。前期的工作中,研究者们利用代谢工程的各种手段改造酵母,提高了TAL的发酵产量。CARDENAS等[15]设计改造酿酒酵母的辅助因子和运输途径以增加乙酰辅酶A和NADPH, TAL合成虽然不直接需要NADPH, NADPH的增加仍有益于TAL产量的提高。VICKERY等[16]采用酶工程方法结合体外分析和体内生物合成来提高酿酒酵母TAL的产量,通过2-PS的体外表征构建新的2-PS突变体,缩小活性位点的空腔大小,提高酶的稳定性和活力。SUN等[17]实施木糖-醋酸盐共消耗的代谢工程策略,增加乙酰辅酶A的供应,从而提高TAL的产量。乙酰辅酶A和丙二酰辅酶A分布在酵母的过氧化物酶体等几个亚细胞器中,LIN等[18]利用过氧化物酶体中的乙酰辅酶A更有效地合成TAL。
前述研究主要利用游离质粒系统或者单拷贝整合系统构建酵母微生物细胞工厂合成TAL。然而,由于游离型质粒丢失产生的不稳定性,在培养过程中保持选择压力的需要等原因[19],将途径基因整合到酿酒酵母基因组中是工业化生产的首选方法。但是常使用的整合位点(Trp1、Ura3、Leu2等)在基因组上仅有一个拷贝,因而基因的表达量相比多拷贝的质粒低很多,不利于产量提高。
酿酒酵母基因组存在大量Ty逆转录转座子重复序列。其中最丰富的一类Ty序列,其长末端重复序列被称为Delta序列,基因组含量约300个[20]。有研究者借助Delta整合已经实现异源基因多拷贝整合,SHI等[21]开发了一个基于CRISPR-Cas系统对Delta位点进行切割的平台,实现了酵母基因组大型生化途径的高效、多拷贝、无标记整合,缺点是仅使用一条Delta序列,没有更多的序列以供选择。此外,LIAN等[22]发现可以通过启动子截短和抗生素浓度调节构建拷贝数可调的质粒,进行路径优化以提高异源途径的表达。
因此,本研究考察了多拷贝基因整合对TAL合成的作用。首先采用文献报道中的Delta序列 (本文称为Delta1)[21]表征和优化了多拷贝整合系统。优化后的系统参数应用于新多拷贝序列Delta2,以提供更多位点选择并进一步提高整合拷贝数。酿酒酵母多拷贝整合系统优化完成后,应用该系统构建g2ps-1基因表达载体合成TAL,通过HPLC分析TAL产量,Delta1整合菌株和Delta2整合菌株的最高产量分别是1.50、1.17 mmol/L,比单拷贝菌株产量分别提高了460%和337%。
1 材料与方法
1.1 实验材料
1.1.1 菌株和培养基
采用大肠杆菌JM109构建和扩增质粒,采用LB培养基(5 g/L酵母提取物、10 g/L胰蛋白胨、10 g/L NaCl)在37 ℃、220 r/min的摇床培养。氨苄青霉素、卡那霉素和氯霉素的工作质量浓度分别为100、50、50 μg/mL,用于质粒转化后大肠杆菌的筛选。酿酒酵母菌株采用CEN.PK2-1D(MATα ura3-52 leu2-3,112 trp1-289 his3-Δ1 MAL2-8c SUC2),在YTD培养基或合成完全(SC)培养基于30 ℃、220 r/min条件下培养。YTD培养基:10 g/L酵母提取物、20 g/L胰蛋白胨和20 g/L葡萄糖。SC培养基:6.7 g/L不含氨基酸的酵母氮源、1.27 g/L含除用于营养缺陷型选择的组分外的氨基酸和核苷酸混合物和20 g/L葡萄糖。固体培养基额外添加20 g/L的琼脂。
1.1.2 质粒
表1列出了本研究中使用的所有质粒。表2列出了本研究中使用的所有引物。引物合成及质粒验证测序均由苏州金唯智生物科技有限公司完成。
表1 本研究所用质粒
Table 1 Plasmids used in this study

名称特性来源pYTK001part plasmid entry vector[23]pYTK009pTDH3[23]pYTK055tENO2[23]pYTK056tTDH1[23]pBL_202from pYTK001, carrying pTEF1-sfGFP-tENO1本研究pBL_205from pYTK001, carrying Delta1 sequence left arm本研究pBL_207from pYTK001, carrying Delta1 sequence right arm本研究pBL_213pre-assembly Delta1 integration vector, characterization of sfGFP本研究pBL_223pre-assembled TRP1 integration vector本研究pBL_602pre-assembled HO integration vector本研究pBL_701from pYTK001, carrying pAgTEF-KanR 15-tAgTEF本研究pBL_702from pYTK001, carrying pAgTEF-KanR 20-tAgTEF本研究pBL_703from pYTK001, carrying pAgTEF-KanR 30-tAgTEF本研究pBL_704from pYTK001, carrying pAgTEF-KanR 50-tAgTEF本研究pBL_705from pYTK001, carrying pAgTEF-KanR 100-tAgTEF本研究pBL_706from pYTK001, carrying pAgTEF-KanR Max-tAgTEF本研究pBL_707from pYTK001, carrying pAgTEF-HygR 15-tAgTEF本研究pBL_708from pYTK001, carrying pAgTEF-HygR 20-tAgTEF本研究pBL_709from pYTK001, carrying pAgTEF-HygR 30-tAgTEF本研究pBL_710from pYTK001, carrying pAgTEF-HygR 50-tAgTEF本研究pBL_711from pYTK001, carrying pAgTEF-HygR 100-tAgTEF本研究pBL_712from pYTK001, carrying pAgTEF-HygR Max-tAgTEF本研究
续表1

名称特性来源pBL_713From pBL213, carrying pAgTEF-KanR 15-tAgTEF, Delta1 integration vector本研究pBL_714From pBL213, carrying pAgTEF-KanR 20-tAgTEF, Delta1 integration vector本研究pBL_715From pBL213, carrying pAgTEF-KanR 30-tAgTEF, Delta1 integration vector本研究pBL_716From pBL213, carrying pAgTEF-KanR 50-tAgTEF, Delta1 integration vector本研究pBL_717From pBL213, carrying pAgTEF-KanR 100-tAgTEF, Delta1 integration vector本研究pBL_718From pBL213, carrying pAgTEF-KanR Max-tAgTEF, Delta1 integration vector本研究pBL_719From pBL213, carrying pAgTEF-HygR 15-tAgTEF, Delta1 integration vector本研究pBL_720From pBL213, carrying pAgTEF-HygR 20-tAgTEF, Delta1 integration vector本研究pBL_721From pBL213, carrying pAgTEF-HygR 30-tAgTEF, Delta1 integration vector本研究pBL_722From pBL213, carrying pAgTEF-HygR 50-tAgTEF, Delta1 integration vector本研究pBL_723From pBL213, carrying pAgTEF-HygR 100-tAgTEF, Delta1 integration vector本研究pBL_724From pBL213, carrying pAgTEF-HygR Max-tAgTEF, Delta1 integration vector本研究pBL_725From pYTK001, carrying Delta2 sequence left arm本研究pBL_726From pYTK001, carrying Delta2 sequence right arm本研究pBL_727Pre-assembly Delta2 integration vector, characterization of sfGFP本研究pBL_728From pBL_733, carrying pAgTEF-KanR15-tAgTEF, Delta2 integration vector本研究pBL_729Pre-assembly Delta1 integration vector, characterization of sfGFP and KanR本研究pBL_730Pre-assembly Delta2 integration vector, characterization of sfGFP and KanR本研究pBL_731From pYTK001, carrying pTDH3-g2ps-1-tENO2本研究pBL_732From pBL_742, carrying pTDH3-g2ps-1-tENO2, Delta1 integration vector本研究pBL_733From pBL_743, carrying pTDH3-g2ps-1-tENO2, Delta2 integration vector本研究
表2 本研究所用引物
Table 2 Primers used in this study

名称序列Delta-F1GCATCGTCTCATCGGTCTCACAATTGGAAGCTGAAACGTCTAACGDelta-R1ATGCCGTCTCAGGTCTCAAGGGGAGAACTTCTAGTATATTCTGTATACCTAATATTATAGCCDelta-F2GCATCGTCTCATCGGTCTCAGAGTCTCGAGGATATAGGAATCCTCAAAATDelta-R2ATGCCGTCTCAGGTCTCATCGGTGTTGGAATAGAAATCAACTATCATCTACTAACTDelta1-F1GCATCGTCTCATCGGTCTCACAATGATTTGATAGTTTCGTACCAGTTCGDelta1-R1ATGCCGTCTCAGGTCTCAAGGGTCACTTGCATTAGACAATAACTACTATATTDelta1-F2GCATCGTCTCATCGGTCTCAGAGTCAGGGATGTCATTAATGCATAGTGDelta1-R2ATGCCGTCTCAGGTCTCATCGGCGGTACTCATAAAGCTAGATTTGTTGKAN15-FGCATCGTCTCATCGGTCTCAAACGCGAACATAAACAAAAATGGGTAAGGAAKAN20-FGCATCGTCTCATCGGTCTCAAACGACATCCGAACATAAACAAAAATGGKAN30-FGCATCGTCTCATCGGTCTCAAACGTTCTCACATCACATCCGAACAKAN50-FGCATCGTCTCATCGGTCTCAAACGAAAATCTTGCTAGGATACAGTTCTCKAN100-FGCATCGTCTCATCGGTCTCAAACGTAAAAGGTTAGGATTTGCCACTGKAN MAX-FGCATCGTCTCATCGGTCTCAAACGCTGCAGAGCTTGCCTCGKAN-RATGCCGTCTCAGGTCTCACAGCCAGTATAGCGACCAGCATTCAHYG15-FGCATCGTCTCATCGGTCTCAAACGCGAACATAAACAAAAATGGGTAAAAHYG20-FGCATCGTCTCATCGGTCTCAAACGACATCCGAACATAAACAAAAATGGHYG30-FGCATCGTCTCATCGGTCTCAAACGTTCTCACATCACATCCGAACAHYG50-FGCATCGTCTCATCGGTCTCAAACGAAAATCTTGCTAGGATACAGTTCTCHYG100-FGCATCGTCTCATCGGTCTCAAACGTAAAAGGTTAGGATTTGCCACTGHYG MAX-FGCATCGTCTCATCGGTCTCAAACGCTGCAGAGCTTGCCTCGHYG-RATGCCGTCTCAGGTCTCACAGCCAGTATAGCGACCAGCATTCA2-PS-FGCATCGTCTCATCGGTCTCAAACGCAGTTCGAGTTTATCATTATCAATA2-PS-RATGCCGTCTCAGGTCTCACAGCATTTTTCAAACTGCAAATTCAAGAAAAGFP-FCAGAAAGCGACCACCCAACTGFP-RCGCCTGTTGCTTCCTATGTGALG9-FCGGGAAGCTTGCTCCTGTATALG9-RCTAGCACAGGCAGTGGGAAA
1.1.3 试剂
Taq DNA聚合酶、Pfu DNA聚合酶,康为世纪;PrimeSTAR DNA聚合酶、T4 DNA连接酶、连接酶(ligation mix)、NotⅠ限制性内切酶、酵母培养基-氨基酸缺失混合物,TaKaRa Bio;DNA Marker、酵母氮源基础,翌圣生物;胶回收试剂盒、质粒抽提试剂盒、RT-qPCR酶,诺唯赞;BsmB Ⅰ、Bsa Ⅱ,赛默飞世尔科技公司;胰蛋白胨、酵母提取物,英国Oxoid;葡萄糖、NaCl、乙酸、磷酸二氢钾,国药集团化学试剂有限公司;氨苄青霉素、卡那霉素、氯霉素、G418硫酸盐、潮霉素B(hygromycin B),麦克林;乙酸锂(lithium acetate,LiAc),上海源叶生物科技有限公司;聚乙二醇(polyethylene glycol ,PEG),索莱宝;色谱级乙腈,北京沃凯生物科技有限公司;TAL标准品,泰坦科技探索平台。
1.2 仪器与设备
BD FACSAriaTM Ⅲ高灵敏度流式细胞仪,BD Biosciences;Synergy H1酶标仪,BioTek,;Prominence LC-20A高效液相色谱系统、SPD-20A/20AV紫外-可见光检测器、Shim-pack GIST C18色谱柱,Shimadzu。
1.3 实验方法
1.3.1 质粒构建
本研究主要使用Yeast Toolkit的方法构建质粒,主要借助限制性内切酶BsmB Ⅰ和Bsa Ⅰ通过Golden Gate反应构建质粒[23]。构建的质粒在JM109中转化扩增。采用化转法制备大肠杆菌感受态细胞并转化质粒[24]。
从酿酒酵母基因组中扩增出文献报道的Delta1序列和通过同源序列比对获取的Delta2[21];从载体p19-RiboJ-sfGFP中扩增出绿色荧光蛋白(green fluorescent protein,GFP)基因(sfGFP)(GenBank登陆号:ASL68970)。分别从载体pYTK077和pYTK079中扩增出启动子截短程度依次为100、50、30、20、15 bp的G418基因(KanR)和hygromycin B基因(HygR);编码TAL合成酶的g2ps-1基因(GenBank登陆号:CAA86219)来源于课题组前期的研究,已经进行了密码子优化[18]。采用SDS-LiAc裂解方法提取酵母基因组[25]。
1.3.2 酿酒酵母转化
使用LiAc/鲑鱼精DNA /PEG方法转化线性化的供体DNA,恢复培养1 h后涂布于相应的缺陷型或抗性平板上,37 ℃培养2~3 d[26]。质粒使用Not Ⅰ进行酶切,酶切体系为10 μL(1 μL Not Ⅰ、1 μL缓冲液、300 ng供体DNA和适量ddH2O)。
1.3.3 流式分析
将表征GFP的质粒转化进CEN.PK2-1D,在添加G418或hygromycin B的YTD固体培养基上筛选,将细胞冲洗下来,采用流式细胞仪进行流式分析。以野生型作阴性对照菌,测定GFP阳性细胞的百分比和荧光强度。简单地说,细胞被清洗并重新悬浮在OD600 =0.1的PBS中。将菌体用PBS清洗2次后,用PBS稀释菌液至OD600=0.1。上样体积为1 mL。
1.3.4 荧光强度测量
在酿酒酵母转化筛选平板上挑选绿色荧光菌落,接种于YTD液体培养基。使用酶标仪Synergy H1测量485 nm激发、528 nm发射的GFP和559 nm激发、600 nm发射的mRuby2。
1.3.5 Delta序列的挖掘及序列同源性比对
Delta序列的挖掘:使用SGD酿酒酵母基因数据库(https://www.yeastgenome.org/)下载获取酿酒酵母的基因组文件,在基因组文件中查找选中序列并在网站(https://gggenome.dbcls.jp/sacCer3/25/)上与基因组进行同源比对(错配设置25%)。序列同源性比对:采用SnapGene软件进行,选择双序列比对,输入2个序列之后设定参数为global alignment(Needleman-Wunsch)进行比对。
1.3.6 拷贝数测定
通过实时荧光定量PCR(real-time fluorescence quantitative PCR,RT-qPCR)测定拷贝数。以sfGFP基因和酿酒酵母内源ALG9基因分别作为靶基因和内参基因,以标准曲线法进行拷贝数定量,sfGFP和ALG9的RT-qPCR引物分别为表2中所示GFP-F、GFP-R和ALG9-F、ALG9-R[21]。
1.3.7 HPLC检测TAL
所有的菌株均以OD600=0.3接种于24孔板发酵48 h。发酵液12 000 r/min离心10 min,-20 ℃保存上清液。用0.22 μm滤膜过滤后,采用配备UV-VIS检测器的高效液相色谱系统进行定量。采用Shimadzu Shim-pack GIST C18色谱柱(4.6 mm×150 mm,粒径5 μm)分离代谢物。色谱柱温度为30 ℃。样品用2种溶剂梯度洗脱:1%(体积分数)的乙酸(A)和含1%(体积分数)的乙酸的乙腈(B)。程序设置:溶剂B从2% (体积分数)线性增加到7%(体积分数)(0~5.0 min);然后从7%(体积分数)增加到95% (体积分数)(5.0~7.0 min),95%(体积分数) 保持2 min(7.0~9.0 min);然后从95%(体积分数)降低到2%(体积分数)(9.0~11.0 min);最后2%(体积分数)保持9 min(11.0~20.0 min)。流速1 mL/min,检测波长280 nm。TAL在10.5 min出峰。根据TAL标准品的校准曲线对TAL浓度进行定量,计算时减去酿酒酵母本身在TAL出峰时间出现的小型特征峰。
2 结果与分析
2.1 基于Delta1整合的多拷贝质粒的构建
以Delta1序列为同源臂,GFP作为表征,对抗生素基因(KanR或HygR)启动子进行截短,分别将KanR或HygR的启动子截短至100、50、30、20、15 bp(未截短的启动子作为对照),通过弱化抗生素基因提高选择压力进而提高整合效率,利用Yeast Toolkit的方法构建了如图1所示的用于整合到酿酒酵母Delta1序列上的表达载体。
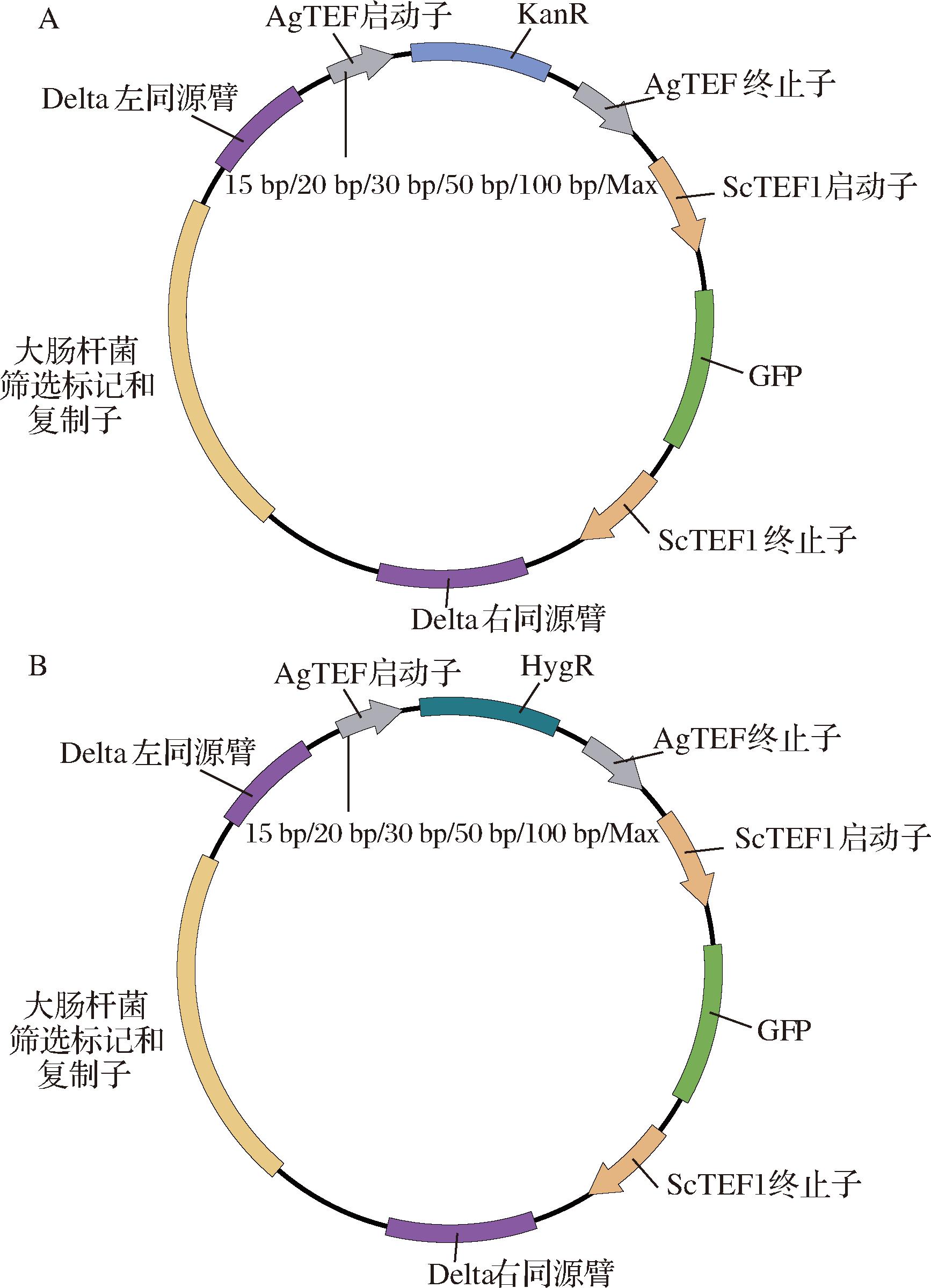
A-G418表达载体;B-hygromycin B表达载体
图1 基于Delta1整合的多拷贝质粒的构建
Fig.1 The physic map of the multi-copy plasmid based on Delta1 integration

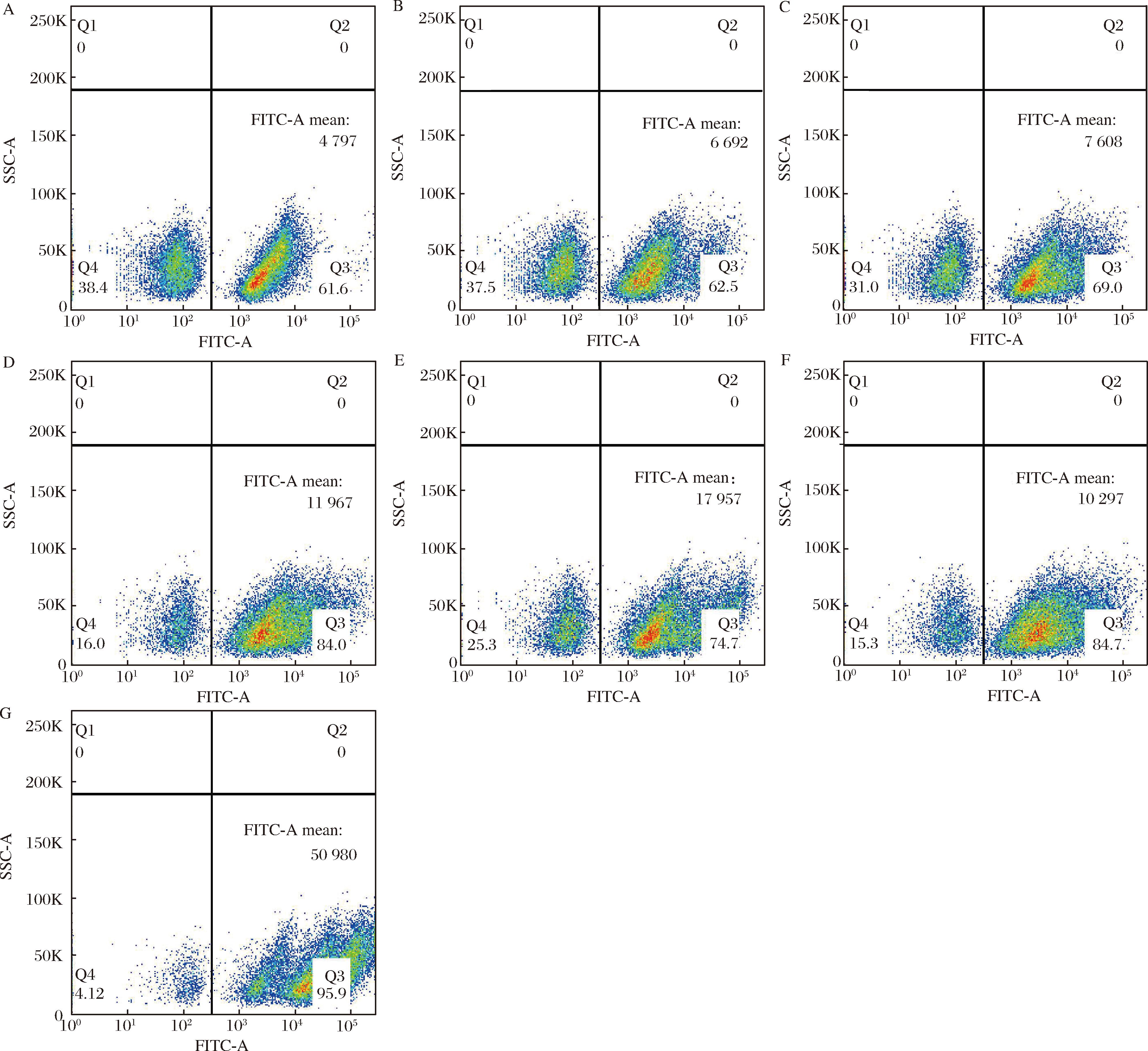
A-Kan Max;B-Kan 100;C-Kan 50;D-Kan30;E-Kan 20;F-Kan 15
图2 G418基因启动子不同截短程度的细胞流式仪检测
Fig.2 Flow cytometry detection of G418 gene promoter with different degrees of truncation
2.2 抗生素基因启动子截短的表征与优化
首先采用文献报道的Delta1在CEN.PK2-1D中进行表征与优化,涂布于添加100 μg/mL的G418或hygromycin B的YTD固体培养基。为了准确比较不同启动子截短程度的影响,将平板菌落冲洗下来后通过流式分析阳性荧光菌群(Q3)占比及其平均荧光强度(fluorescein isothiocyanate-A mean fluorescence intensity,FITC-A mean)(图2、图3),不含GFP的阴性对照菌群(Q4)的平均荧光强度是102左右。

A-Hyg Max;B-Hyg 100;C-Hyg 50;D-Hyg 30;E-Hyg 20;F-Hyg 15
图3 hygromycin B基因启动子不同截短程度的细胞流式仪检测
Fig.3 Flow cytometry detection of hygromycin B gene promoter with different degrees of truncation
如图2所示,对于启动子截短的KanR,Q3的平均荧光强度和启动子截短程度有很好的相关性,启动子截短程度和平均荧光强度大致成正比,尤其是在KanR启动子截短至20、15 bp时,Q3的平均荧光强度分别提高至122 211和91 929;将图2-A与图2-B、图2-C、图2-D、图2-E、图2-F共同比对分析,与启动子未截短的KanR相比,对KanR启动子进行截短后获得的阳性荧光菌群占比均为85%以上。如图3所示,对于启动子截短的HygR,将启动子截短至15 bp时可以获得明显效果,与启动子未截短的HygR相比,此时阳性荧光菌群的平均荧光强度从13 491提高至54 521,阳性荧光菌落占比为78.8%。
这些结果表明通过弱化抗生素基因提高选择压力进而提高高拷贝整合效率这一途径是有效果的。综合分析阳性荧光菌群占比及其平均荧光强度,对于KanR和HygR,均选用15 bp的启动子截短程度进行后续的优化。
2.3 抗生素浓度梯度的优化
为筛选出合适的抗生素浓度,对启动子截短至15 bp的KanR和HygR分别做40、60、80、100、120、140、160 μg/mL一系列抗生素质量浓度的转化,通过流式分析不同抗生素质量浓度下阳性荧光菌群占比及其平均荧光强度。
由图4可以看出,对于启动子截短至15 bp的KanR,选用160 μg/mL的G418质量浓度Q3占比及其平均荧光强度均最高。此外,40 μg/mL G418质量浓度太低未能起到筛选作用而导致糊板,不能在平板上获得有效结果。相似的是,对于启动子截短至15 bp的HygR,hygromycin B质量浓度<120 μg/mL的平板均因浓度太低而糊板,将提升hygromycin B质量浓度至200、300、400 μg/mL则平板上未见菌落生长,因此仅对图5所示hygromycin B浓度进行流式分析。由图5可知,对于启动子截短至15 bp的HygR,选用160 μg/mL的hygromycin B质量浓度Q3占比及其平均荧光强度均最高。实验结果表明多拷贝整合系统最优参数为:启动子截短至15 bp的G418基因与160 μg/mL的G418工作质量浓度。
A-40 μg/mL;B-60 μg/mL;C-80 μg/mL;D-100 μg/mL;E-120 μg/mL;F-140 μg/mL;G-160 μg/mL
图4 不同G418质量浓度的细胞流式仪检测
Fig.4 Flow cytometry detection of cells with different G418 mass concentrations
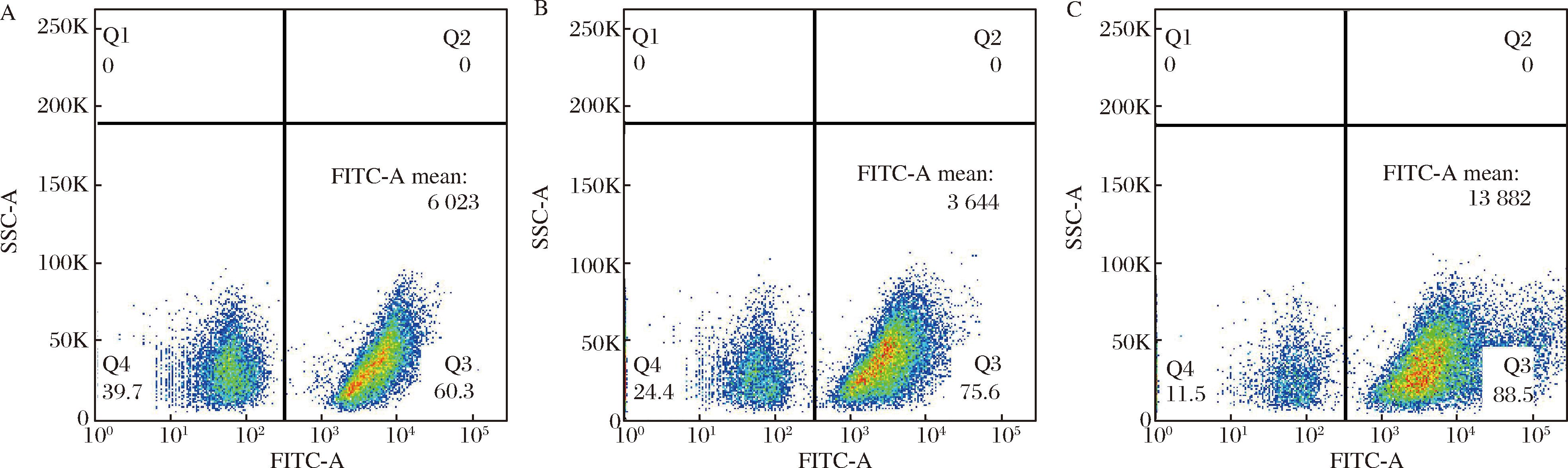
A-120 μg/mL;B-140 μg/mL;C-160 μg/mL
图5 不同hygromycin B质量浓度的细胞流式仪检测
Fig.5 Flow cytometry detection of cells with different hygromycin B mass concentrations
2.4 新多拷贝整合序列Delta2与Delta1序列的比对分析
为扩展上述优化后的多拷贝整合系统的通用性,本文尝试了酵母基因组上的另一重复序列进行多拷贝整合测试。该序列为YERW Delta21,在基因组上有32条高度同源序列,下文将其简称为Delta2序列。
使用SnapGene软件进行Delta1和Delta2两条序列的全局比对,如图6所示,结果显示两条序列一致性为45.86%(205/447),空缺为39.60%(177/447),错配为14.54%(65/447);将Delta1序列的反向互补序列与Delta2进行全局比对显示一致性为43.90%(198/451)。Delta1和Delta2的序列相似性低,存在相互干扰的可能性低。

图6 Delta1和Delta2的序列比对
Fig.6 Sequence alignment for Delta1 and Delta2
2.5 拷贝数测定
采用RT-qPCR测定系统最优参数条件下表征的sfGFP拷贝数并检测对应菌株的荧光强度。结果如图7所示,Delta1位点整合效果优于Delta2,Delta1 位点整合菌株的拷贝数高于Delta2的同时Delta1位点整合菌株的荧光强度也高于Delta2,表明拷贝数与荧光强度有很好的相关性;以Delta1作为整合位点时sfGFP的整合最高可达10个拷贝,以Delta2作为整合位点时sfGFP的整合最高可达7个拷贝。

图7 不同整合位点的拷贝数测定及对应的荧光强度检测
Fig.7 Copy number determination of different integration sites and corresponding fluorescence intensity detection
2.6 TAL的合成与检测
应用优化完成的多拷贝整合系统构建g2ps-1基因表达载体以合成TAL,将g2ps-1基因插入到Delta序列的左同源臂和启动子截短至15 bp的Kan15之间。TAL合成菌株发酵后取上清液进行HPLC检测(图8-B)。与标准品进行对比,结果表明g2ps-1整合菌株成功合成TAL。
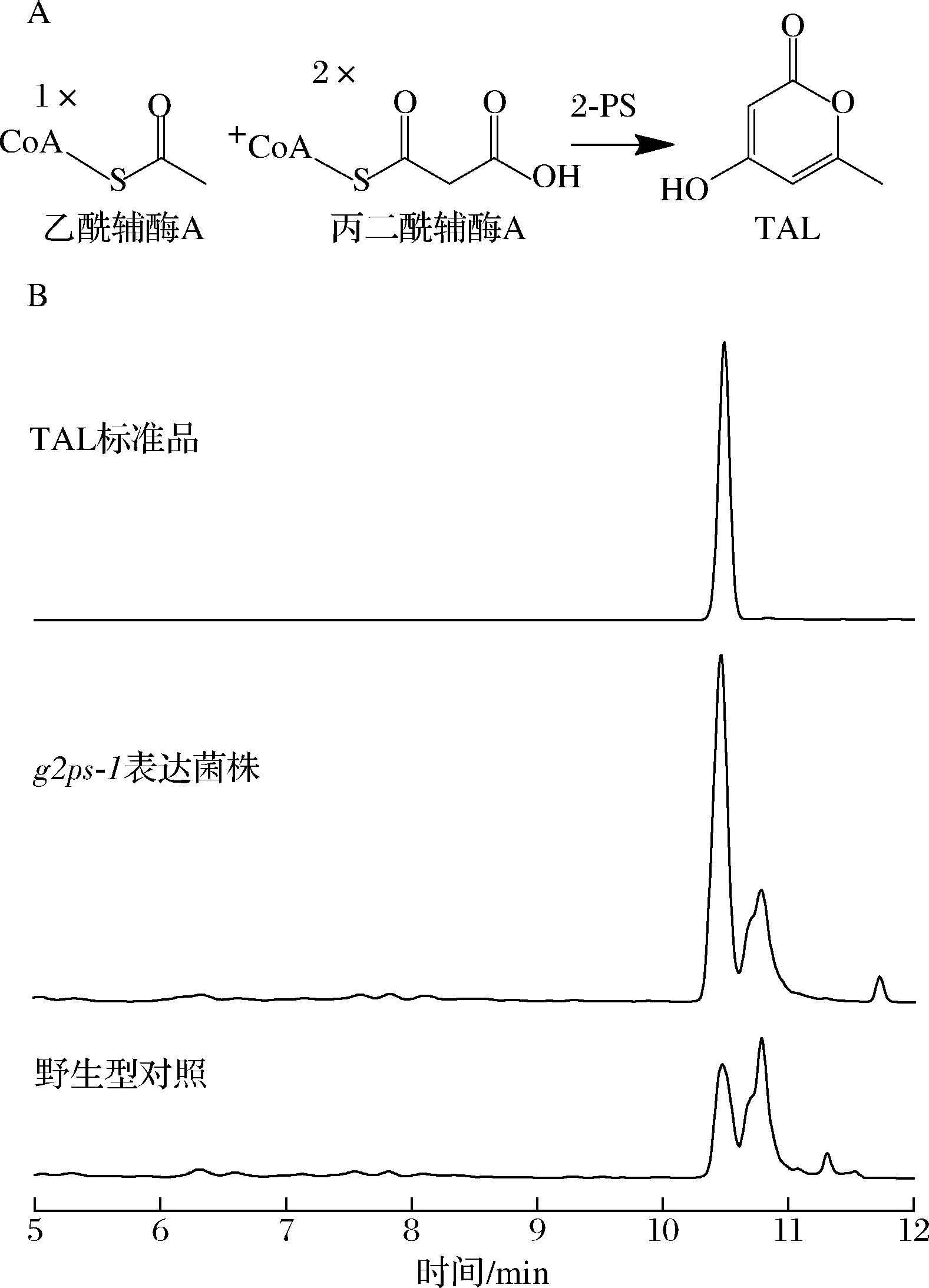
A-TAL的合成途径;B-发酵产物的HPLC检测
图8 发酵产物的 HPLC 检测
Fig.8 Detection of fermentation products by HPLC
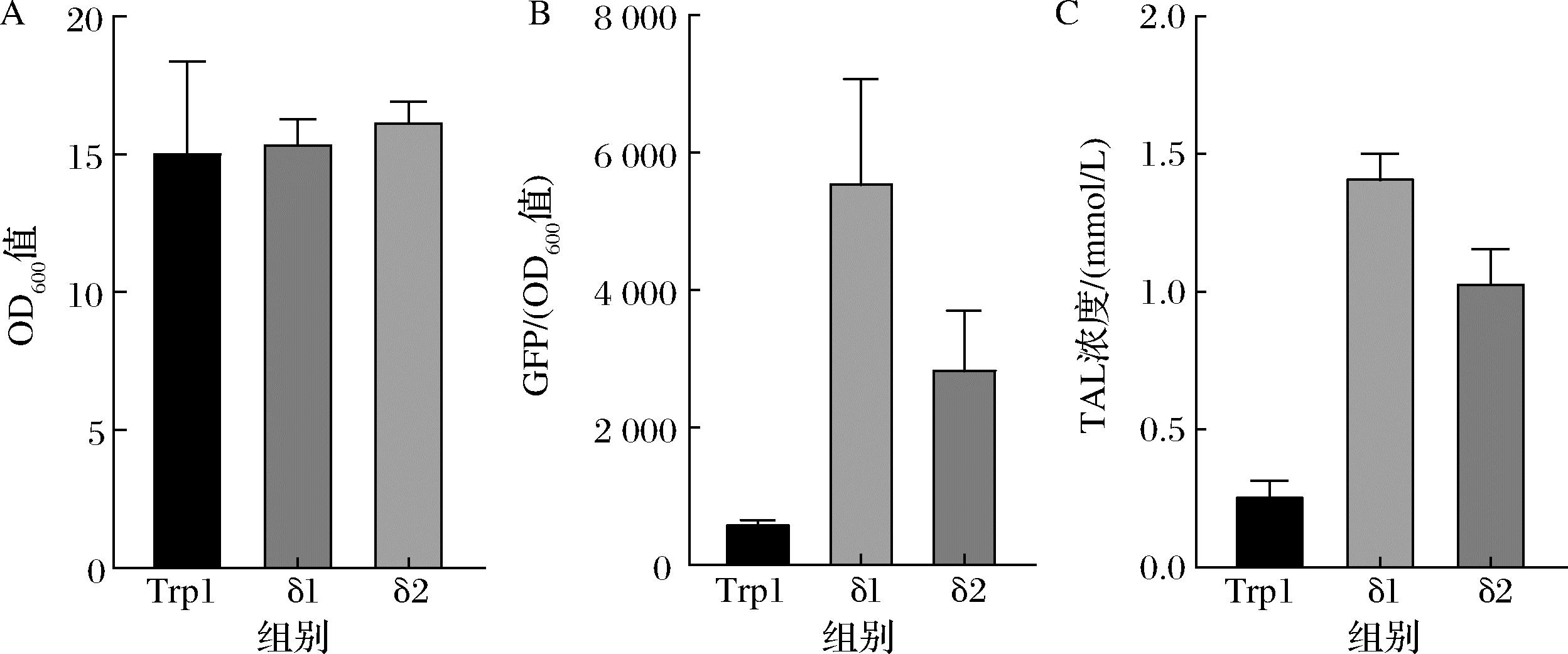
A-菌株生长密度;B-对应菌株绿色荧光强度;C-对应菌株TAL产量
图9 不同位点整合g2ps-1
Fig.9 Integration of the g2ps-1 gene at different loci
首先构建并转化了Delta1或Delta2不同位点整合g2ps-1的表达载体,以Trp1位点单拷贝整合g2ps-1为对照。菌株生长状况如图9-A,各菌株生长未有明显差异;荧光强度分析如图9-B,与Trp1位点的单拷贝整合相比无论是Delta1还是Delta2整合菌株的绿色荧光强度都有很大提高;与绿色荧光强度趋势一致的是菌株生产TAL的浓度,Delta1位点整合菌株的最高产量为1.50 mmol/L,Delta2位点整合菌株最高产量为1.17 mmol/L,与单拷贝对照相比,Delta1位点整合使TAL产量增加了4.6倍,Delta2位点整合使TAL产量增加了3.4倍。
3 结论与讨论
TAL作为最简单的聚酮类化合物,是一种重要的平台分子,作为前体合成各种有机化合物。对于TAL在酿酒酵母中的生产,已有研究采用各式各样的策略提高TAL的产量[15-18]。然而前述的研究均是通过质粒表达系统或者单拷贝整合的方式来实现,仍具有改进空间。鉴于酿酒酵母基因组存在众多重复序列,比如Delta位点,开发利用上述位点,将有利于继续提高酿酒酵母合成TAL的效率。本研究构建了基于Delta整合的酿酒酵母多拷贝整合系统,通过弱化抗生素基因的表达、利用抗生素胁迫提高整合拷贝数和整合效率,应用该系统合成TAL,最终获得产量达1.50 mmol/L的Delta1位点整合菌株和产量达1.17 mmol/L的Delta2位点整合菌株,分别是单拷贝菌株的5.6倍和4.4倍。然而本研究仅使用Delta1或Delta2单一位点整合g2ps-1基因合成TAL,后续研究可考虑将Delta2与Delta1两个整合位点叠加使用,有望进一步提高整合拷贝数和TAL产量。此外本研究只在24孔板中进行小型发酵,还可以通过发酵工艺优化、增加乙酰辅酶A供给等策略进一步提高TAL产量,为构建酿酒酵母聚酮类化合物的高效合成平台提供参考价值。
[1] ECKERMANN S, SCHRÖDER G, SCHMIDT J, et al.New pathway to polyketides in plants[J].Nature, 1998, 396(6709):387-390.
[2] OBYDENNOV D L, EL-TANTAWY A I, SOSNOVSKIKH V Y.Triacetic acid lactone as a bioprivileged molecule in organic synthesis[J].Mendeleev Communications, 2019, 29(1):1-10.
[3] PALMER C M, ALPER H S.Expanding the chemical palette of industrial microbes:Metabolic engineering for type Ⅲ PKS-derived polyketides[J].Biotechnology Journal, 2019, 14(1):e1700463.
[4] HANSEN C A, FROST J W.Deoxygenation of polyhydroxybenzenes:An alternative strategy for the benzene-free synthesis of aromatic chemicals[J].Journal of the American Chemical Society, 2002, 124(21):5 926-5 927.
[5] 刘文杰, 左迎峰, 吴义强, 等.间苯二酚改性木材胶粘剂的研究进展[J].中国胶粘剂, 2017, 26(12):42-46.LIU W J, ZUO Y F, WU Y Q, et al.Research progress of wood adhesives modified by resorcinol[J].China Adhesives, 2017, 26(12):42-46.
[6] KHAN F, TABASSUM N, BAMUNUARACHCHI N I, et al.Phloroglucinol and its derivatives:Antimicrobial properties toward microbial pathogens[J].Journal of Agricultural and Food Chemistry, 2022, 70(16):4817-4838.
[7] YU J, LANDBERG J, SHAVAREBI F, et al.Bioengineering triacetic acid lactone production in Yarrowia lipolytica for pogostone synthesis[J].Biotechnology and Bioengineering, 2018, 115(9):2383-2388.
[8] MEI C A, SCHWARTZ T J, SHANKS B H, et al.Triacetic acid lactone as a potential biorenewable platform chemical[J].Green Chemistry, 2012, 14(7):1850.
[9] ZHOU Y F, DING Y M, GAO W J, et al.Biosynthesis of acetylacetone inspired by its biodegradation[J].Biotechnology for Biofuels, 2020, 13:88.
[10] SCHWARTZ T J, JOHNSON R L, CARDENAS J, et al.Engineering catalyst microenvironments for metal-catalyzed hydrogenation of biologically derived platform chemicals[J].Angewandte Chemie International Edition, 2014, 53(47):12718-12722.
[11] XIE D M, SHAO Z Y, ACHKAR J, et al.Microbial synthesis of triacetic acid lactone[J].Biotechnology and Bioengineering, 2006, 93(4):727-736.
[12] LIU Y M, JIN Y H, XU P, et al.Recent advances and perspectives on the biomass-derived production of the platform chemical triacetic acid lactone by engineered cell factories[J].Biochemical Engineering Journal, 2023, 197:108961.
[13] LUO Y Z, LI B Z, LIU D, et al.Engineered biosynthesis of natural products in heterologous hosts[J].Chemical Society Reviews, 2015, 44(15):5265-5290.
[14] BOND C, TANG Y, LI L.Saccharomyces cerevisiae as a tool for mining, studying and engineering fungal polyketide synthases[J].Fungal Genetics and Biology, 2016, 89:52-61.
[15] CARDENAS J, DA SILVA N A.Engineering cofactor and transport mechanisms in Saccharomyces cerevisiae for enhanced acetyl-CoA and polyketide biosynthesis[J].Metabolic Engineering, 2016, 36:80-89.
[16] VICKERY C R, CARDENAS J, BOWMAN M E, et al.A coupled in vitro/in vivo approach for engineering a heterologous type III PKS to enhance polyketide biosynthesis in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2018, 115(6):1394-1402.
[17] SUN L, LEE J W, YOOK S, et al.Complete and efficient conversion of plant cell wall hemicellulose into high-value bioproducts by engineered yeast[J].Nature Communications, 2021, 12(1):4975.
[18] LIN P X, FU Z H, LIU X X, et al.Direct utilization of peroxisomal acetyl-CoA for the synthesis of polyketide compounds in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2023, 12(6):1599-1607.
[19] GNÜGGE R, RUDOLF F.Saccharomyces cerevisiae Shuttle vectors[J].Yeast, 2017, 34(5):205-221.
[20] GARFINKEL D J.Genome evolution mediated by Ty elements in Saccharomyces[J].Cytogenetic and Genome Research, 2005, 110(1-4):63-69.
[21] SHI S B, LIANG Y Y, ANG E L, et al.Delta integration CRISPR-Cas (Di-CRISPR) in Saccharomyces cerevisiae[J].Methods in Molecular Biology, 2019, 1927:73-91.
[22] LIAN J Z, JIN R, ZHAO H M.Construction of plasmids with tunable copy numbers in Saccharomyces cerevisiae and their applications in pathway optimization and multiplex genome integration[J].Biotechnology and Bioengineering, 2016, 113(11):2462-2473.
[23] LEE M E, DELOACHE W C, CERVANTES B, et al.A highly characterized yeast toolkit for modular, multipart assembly[J].ACS Synthetic Biology, 2015, 4(9):975-986.
[24] CHAN W T, VERMA C S, LANE D P, et al.A comparison and optimization of methods and factors affecting the transformation of Escherichia coli[J].Bioscience Reports, 2013, 33(6):e00086.
[25] L OKE M, KRISTJUHAN K, KRISTJUHAN A.Extraction of genomic DNA from yeasts for PCR-based applications[J].BioTechniques, 2011, 50(5):325-328.
OKE M, KRISTJUHAN K, KRISTJUHAN A.Extraction of genomic DNA from yeasts for PCR-based applications[J].BioTechniques, 2011, 50(5):325-328.
[26] GIETZ R D, SCHIESTL R H.High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J].Nature Protocols, 2007, 2(1):31-34.