非酒精性脂肪肝病(non-alcoholic fatty liver disease,NAFLD)是一种以肝脏脂质过度堆积为特征,排除酒精或其他肝损伤因素导致的慢性代谢类疾病[1]。世界卫生组织估计,全球有超过19亿成年人超重,因此,包括非酒精性脂肪肝病在内的代谢紊乱的发病率有所加剧[2]。NAFLD可能会从单纯的肝脏脂肪积累进展为更严重的非酒精性脂肪肝炎,甚至进一步恶化成肝硬化或肝癌[3],这给医疗保健系统带来了巨大的负担。
目前,NAFLD的发病机制尚不明确,根据“多重平行打击”假说,NAFLD的发生发展与氧化应激、炎症反应以及肠道微生物失调密切相关[4-6]。越来越多证据表明,NAFLD患者的肠道菌群失调会通过肝肠轴从而影响肝脏的糖脂代谢[7]。基于肝肠轴的重要性,最近一些研究试图揭示NAFLD的肠道微生物特征,评估其作为NAFLD诊断生物标志物的适用性,并预测疾病的进展[8-10],这给NAFLD的非侵入性诊断提供了思路。
饮食,作为人类生活中的一个关键组成部分,不仅直接调节宿主的生理健康,还通过影响肠道微生物群及其代谢活动间接发挥作用[11]。饮食、肠道菌群和宿主健康以一种复杂的方式相互作用[12],长时间高热量饮食可促进NAFLD的发生[13]。然而,在大量关于NAFLD肠道生物标志物的研究中[1,14-16],饮食因素并没有被探究,各个研究之间的差异结果是否完全或者部分归因于饮食差异,并且在大量关于非酒精性脂肪肝动物肠道菌群研究中,大多是高脂饮食所致,很少考虑高碳水饮食模式诱导的非酒精性脂肪肝[17]。因此,有必要在研究NAFLD患者的肠道菌群特征的同时增加膳食因素。
在本团队前期研究中发现,NAFLD患者肠道微生物与健康人之间存在显著差异。为了进一步探究不同膳食结构下NAFLD患者的肠道菌群特点,将招募到的NAFLD患者进行亚组分析。通过调研NAFLD患者的膳食情况,基于可以精确到种水平的宏基因组测序手段明确不同膳食结构下的NAFLD肠道菌群特点,可以进一步科学合理地指导NAFLD患者合理膳食,为开发针对性膳食辅助性治疗手段提供帮助。
1 材料与方法
1.1 非酒精性脂肪肝病患者招募
1.1.1 拓募标准
所有招募程序都经过宜兴市人民医院伦理委员会审核(伦理审查意见号:伦审2022科160,审查日期为2022年11月18日),并在中国临床试验注册中心注册(注册号:ChiCTR2200066507)。所有程序均按照《世界医学协会道德准则(赫尔辛基宣言)》执行。所有同意参与本项目的非酒精性脂肪肝病患者均在入组前提供知情同意书。
1.1.2 诊断标准
参照Diagnosis of NAFLD &NASH(NIDDK)和非酒精性脂肪性肝病诊疗指南(中华医学会肝脏病学分会脂肪肝和酒精性肝病学组,2010)对非酒精性脂肪性肝病的诊断,主要结合身体指标、血液指标和B超进行综合分析,在排除酒精性肝损伤、药物性肝损伤和病毒性肝损伤的基础上作出诊断。无饮酒史或折合乙醇摄入量男性<140 g/周,女性<70 g/周;肝脏影像学表现符合弥漫性脂肪肝的影像学诊断标准。
以下3项腹部超声表现中的任意2项者为弥漫性脂肪肝:a)肝脏近场回声弥漫性增强(明亮肝),回声强于肾脏;b)肝内管道结构显示不清;c)肝脏远场回声逐渐衰减。CT诊断脂肪肝的依据为肝脏密度普遍降低,肝/脾CT比值<1.0。
1.1.3 纳入标准
a)符合非酒精性脂肪性肝病的诊断标准;
b)18岁≤年龄≤65岁,男女不限;
c)依从性好,能够按临床试验要求,遵守规定;
d)患者填写书面知情同意书和膳食结构调查表,根据《中国居民膳食营养素参考摄入量速查手册2013版》[18]规定,每日能量摄入中脂肪占能超过30%纳入为高脂肪膳食摄入组,碳水化合物占能超过65%纳入为高碳水膳食摄入组。
1.1.4 排除标准
a)乙醇折合摄入量>40 g/周;
b)在过去6个月内定期摄入非甾体类抗炎药,抗生素和皮质类固醇、抗肿瘤药、抗结核药以及一些中草药及矿物药(大枫子/三氮化二砷);
c)过去6个月内滥用麻醉品,精神药物和卷烟;
d)在过去6个月内摄入引起胃酸缺乏,胺碘酮,丙戊酸钠,泼尼松,他莫昔芬,哌可昔林和甲氨蝶呤,肝脏脂肪诱导药物,激素药物,他汀类药物,抗高血压药和熊去氧胆酸的抗分泌药物;
e)在研究期间或过去3个月内,每周至少2次补充营养素,如益生菌,多种维生素/矿物质,抗氧化剂和ω-3脂肪酸;
f)有影响肝脏病理状况的疾病,胆汁淤积,肝移植,急性全身性疾病,囊性纤维化疾病,肌肉萎缩症,神经系统疾病,胃肠道结构异常,糖尿病,心力衰竭,甲状腺疾病,炎性肠病,肾脏疾病,呼吸衰竭,遗传性血色病,威尔森氏病,α-1抗胰蛋白酶缺乏症,自身免疫性疾病,乳糜泻和任何类型的恶性肿瘤;
g)过去6个月内体重迅速减轻,全胃肠外营养和蛋白质营养不良;
h)孕妇或哺乳期妇女;
i)患者依从性较差,随访可能性差。
1.2 实验方法
1.2.1 实验设计
自2022年11月18日起,于宜兴市人民医院超声科一共招募了150名非酒精性脂肪肝病患者,通过膳食结构调查以及B超检查,筛选出每日能量摄入中脂肪占能超过30%的非酒精性脂肪肝病患者纳入高脂肪膳食摄入组,每日能量摄入中碳水化合物占能超过65%的非酒精性脂肪肝病患者纳入高碳水膳食摄入组。最后筛选出15名高碳水膳食摄入患者与15名高脂肪膳食摄入患者进行亚组分析。
1.2.2 样本的采集
邀请非酒精性脂肪肝病患者在宜兴市人民医院进行检查,由专业医护人员进行B超检查、人体测量评估和血液生化指标检测,空腹采集血清标本,经离心、等体积分装后置于-80 ℃冰箱保存。此外使用装有冰盒的无菌容器收集粪便样品,并立即转移到-80 ℃ 冰箱中后续用于宏基因组分析。
1.2.3 安全性评价
检查项目包括身高、体重、生化指标[谷丙转氨酶(alanine aminotransferase, ALT)、谷草转氨酶(aspartate aminotransferase, AST)、空腹血糖、胰岛素水平、甘油三酯(triglyceride,TG)、总胆固醇(total cholesterol,TC)、高密度脂蛋白胆固醇(high density lipoprotein cholesterol,HDL-C)、低密度值蛋白胆固醇(low-density lipoprotein cholesterol,LDL-C)和C反应蛋白(C-reactive protein,CRP)],B超(肝胆脾胰)检查,项目结果作为安全性评价指标。
1.2.4 宏基因组数据处理和质量控制
宏基因组测序在北京诺禾致源科技股份有限公司Illumina Nova Seq 6000平台上进行,送检的30份样本为采集到的30名非酒精性脂肪肝病患者粪便样本。30个样品的平均测序量为(49.2±4.4)(均数±标准差)百万条reads。原始序列的预处理包括以下步骤:使用Trimmomatic(版本0.39)过滤低质量序列[19],对平均碱基质量分数低于30的序列进行修剪,保留过滤后长度大于60 bp的序列作为质控输出。随后,使用BWA(版本0.7.17)、Samtools(版本1.9)和BEDTools(版本2.30.0)将过滤后的序列比对到人类参考基因组(人类基因组组装GRCh38, hg38),有效地从样品中去除宿主来源基因经过上述质量控制后,平均每个样品保留(37.1±3.8)(均数±标准差)万条reads[20]。
1.2.5 物种注释和功能注释
质控后的高质量序列分别使用MetaPhlAn3和HUMAnN3进行分类和功能注释[21]。HUMAnN3利用合并的双端数据进行注释,随后将获得的计数归一化为相对丰度值。个体细菌功能贡献的分层信息来自代谢途径的标准化丰度数据。使用humann_barplot脚本实现了细菌对单个代谢途径贡献的可视化。物种丰度信息表用于计算α多样性指标。β多样性是使用 Bray-Curtis 指标计算的。线性判别分析效应量(linear discriminant analysis effect size,LEfSE)用于识别MDP-i-KD前后驱动差异的细菌分类群[22]。
1.3 统计分析
采用GraphPad Prism IBM SPSS 22 (IBM, Armonk, NY, USA),和R 4.2.2 (https://www.r-project.org/) 进行统计分析。采用Shapiro-Wilk检验检查数据是否符合正态分布。对于正态分布的数据,进行t检验;对于非参数数据,使用Wilcoxon秩和检验或Fisher精确检验。数据以“平均值±标准差”表示。计数资料之间的比较采用卡方检验。P<0.05被视为具有统计学显著性。
2 结果与分析
2.1 非酒精性脂肪肝病患者基线特征
根据严格的纳入和排除标准,将招募到的非酒精性脂肪肝病患者进行亚组分析,分为15名高碳水膳食摄入组和15名高脂肪膳食摄入组。如表1所示,根据超声检查,2组患者的脂肪肝患病严重程度无显著差异,肝左叶上下径、前后径、右叶肋下斜径和横径都无显著差异;两组患者肝酶指标、甘油三酯、总胆固醇、高低密度脂蛋白、游离脂肪酸、动脉粥样硬化、空腹血糖有和超敏C反应蛋白也无显著差异。
表1 参与者特征
Table 1 Baseline characteristics
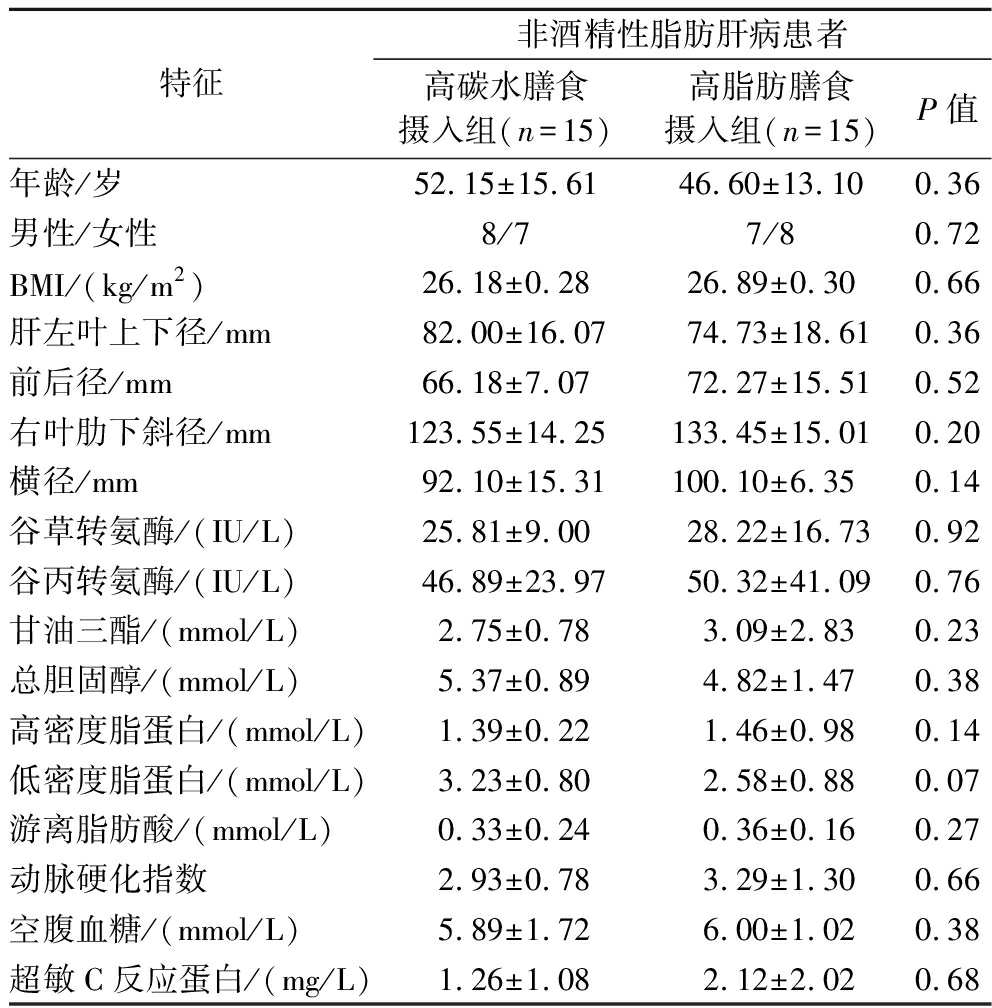
特征非酒精性脂肪肝病患者高碳水膳食摄入组(n=15)高脂肪膳食摄入组(n=15)P值年龄/岁52.15±15.6146.60±13.100.36男性/女性8/77/80.72BMI/(kg/m2)26.18±0.2826.89±0.300.66肝左叶上下径/mm82.00±16.0774.73±18.610.36前后径/mm66.18±7.0772.27±15.510.52右叶肋下斜径/mm123.55±14.25133.45±15.010.20横径/mm92.10±15.31100.10±6.350.14谷草转氨酶/(IU/L)25.81±9.0028.22±16.730.92谷丙转氨酶/(IU/L)46.89±23.9750.32±41.090.76甘油三酯/(mmol/L)2.75±0.783.09±2.830.23总胆固醇/(mmol/L)5.37±0.894.82±1.470.38高密度脂蛋白/(mmol/L)1.39±0.221.46±0.980.14低密度脂蛋白/(mmol/L)3.23±0.802.58±0.880.07游离脂肪酸/(mmol/L)0.33±0.240.36±0.160.27动脉硬化指数2.93±0.783.29±1.300.66空腹血糖/(mmol/L)5.89±1.726.00±1.020.38超敏C反应蛋白/(mg/L)1.26±1.082.12±2.020.68
2.2 不同膳食结构下非酒精性脂肪肝病患者肠道微生物生态总体组成情况
为了探讨不同膳食结构下非酒精性脂肪肝病患者的肠道菌群总体组成情况,用Shannon指数、Chao1指数、ACE指数和Simpson指数来比较高碳水膳食摄入组和高脂肪膳食摄入组肠道菌群的α多样性(图1-A~图1-D)。通过宏基因组测序发现不同饮食结构下2组在物种丰富度和均匀度上没有明显区别。一项关于重金属摄入对肠道菌群的研究同样也表明不同饮食习惯下肠道菌群多样性没有显著改变[23],说明不同饮食结构可能对肠道菌群多样性程度的影响不大。
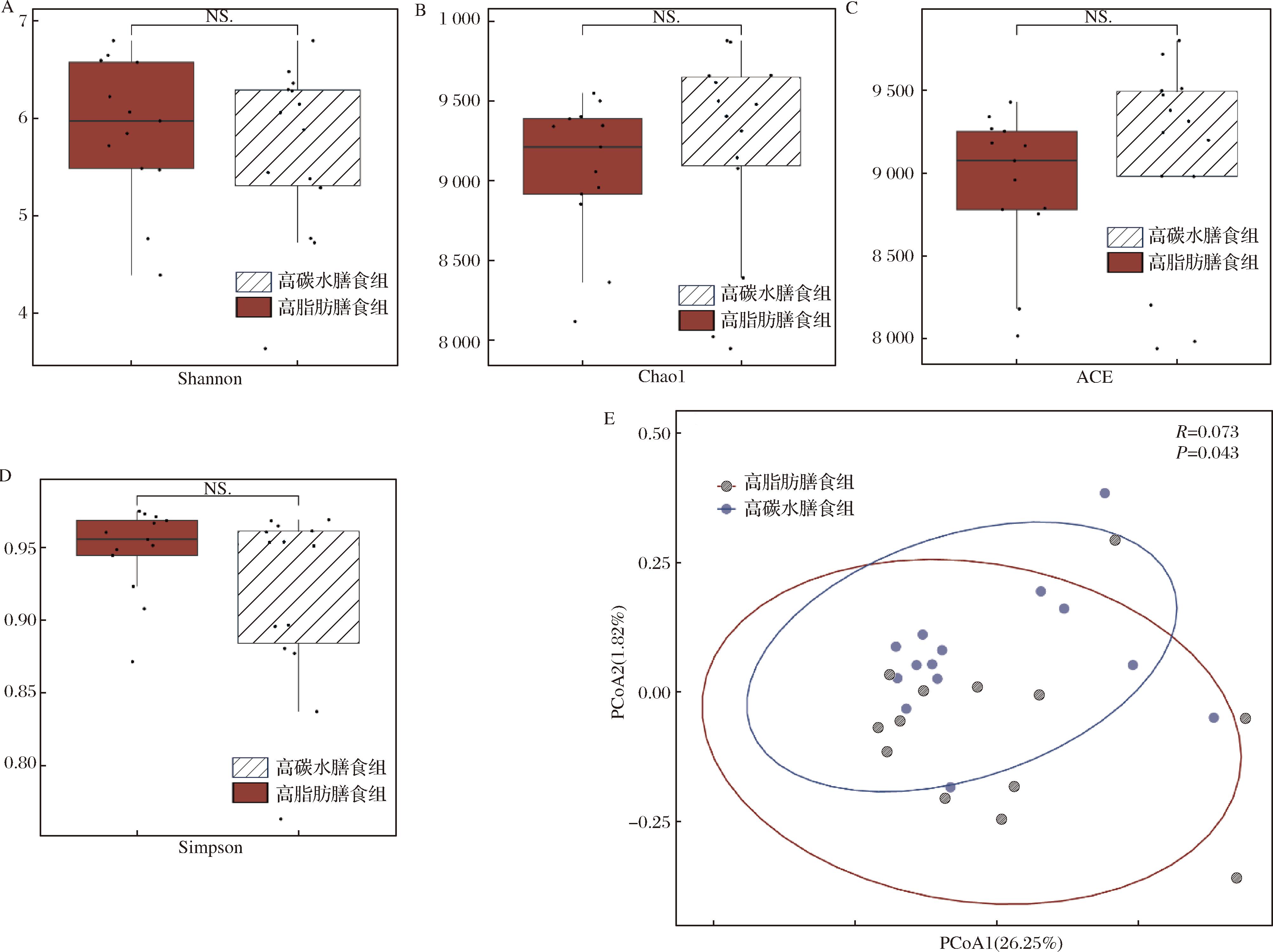
A-α多样性-Shannon指数;B-α多样性-Chao1指数;C-α多样性-ACE指数;D-α多样性-Simpson指数;E-β多样性-布雷-柯蒂斯距离
图1 不同膳食结构下的肠道菌群组成
Fig.1 The composition of gut microbiota under different dietary structures
由图1-E可知,通过主坐标分析表明这2组肠道菌群在种水平上的布雷-柯蒂斯距离存在统计学上的差异(P<0.05),但由于两坐标轴方差解释比例过小,且2组位置基本重合,说明高脂肪膳食摄入和高碳水膳食摄入饮食习惯下患者肠道菌群结构在整体组成上差异不大,因此值得进一步深入研究去揭示这些细微差异的潜在机理。通过图2的堆叠直方图表明,厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和变形菌门(Proteobacteria)都是2组相对丰度较高的4个门。但是不同的是,用Wilcoxon秩和检验确定了4个门存在显著差异。图2-A表明,高脂肪膳食摄入组的厚壁菌门、F/B(厚壁菌门与拟杆菌门的比值)和疣微菌门占比显著高于高碳水膳食摄入组,而梭杆菌门和变形菌门占比显著低于高碳水膳食摄入组。ZHANG等[24]发现小鼠食用高脂饮食30 d后,小肠中菌群的空间分布和菌群组成皆有改变,厚壁菌门和疣微菌门显著增加;DOUGAL等[25]发现高淀粉喂养的老年马肠道菌群中梭杆菌门和变形菌门占比高于高油高纤维喂养。这与本文的研究相符,说明了不同饮食下所诱导的非酒精性脂肪肝病的肠道菌群生态紊乱是不一样的,这对日后的非酒精性脂肪肝动物特异性造模和进一步科学合理地指导非酒精性脂肪肝患者合理膳食有一定意义。

A-门水平的微生物分布;B-厚壁菌门的差异及F/B值(厚壁菌门和拟杆菌门比值);C-梭杆菌门的差异;D-变形菌门的差异;E-疣微菌门的差异
图2 不同膳食结构下的门水平肠道菌群组成
Fig.2 The composition of gut microbiota at the phylum level under different dietary structures
2.3 差异种属筛选
为了鉴定出关键差异属,如图3所示,使用LEfSE分析鉴定出了18个差异显著的属(LDA>2.6,P<0.05)。高脂肪摄入组粪杆菌属(Faecalibacterium)、萨特氏菌属(Sutterellaceae)和罗氏菌属(Rothia)可作为关键菌属。WAN等[26]一项关于217名不同脂肪含量的随机对照试验也表明高脂肪饮食(脂肪40%占能)显著增加了粪杆菌属(Faecalibacterium)。进一步以伪发现率(false discovery rate,FDR)<0.01,fold change>1为标准,筛选差异物种,结果如图4所示,发现普拉梭杆菌(Feacalibacterium prausnitzii)很可能是高脂饮食与高碳水饮食的显著差异菌。一项关于素食者与喜食肉者的肠道菌群的研究表明,素食者的肠道菌群以产气荚膜梭菌(Clostridium perfringens)和多枝梭菌(Clostridium ramosum)为主,长期高水平食肉者的肠道优势菌群则主要是F.prausnitzii [27]。由此推测高脂肪膳食摄入的非酒精性脂肪肝患者长期食用脂肪含量较高的动物肉类所导致肠道优势菌群为F.prausnitzii。同时根据图3和图4表明,高碳水的摄入大大富集了肠道中的梭菌,如C.taeniosporum、C.manihotivorum和C.cellulovorans等。除了梭菌属,还发现高碳水饮食模式下罗姆布茨菌(Romboutsia sp.CE17)显著增加。一项关于90只蛋鸡试验[28]表明,高玉米含量饲料显著增加了Romboutsia丰度。因此,不同饮食结构下的非酒精性脂肪肝病患者所富集的肠道菌群不同,结合不同饮食结构诱导的动物实验所得出的结论,在日后的非酒精性脂肪肝病模型的动物实验中应考虑除了高脂饮食之外的造模模式。

图3 LEfSe线性判别分析筛选差异物种
Fig.3 LEfSe linear discriminant analysis was used to screen differential species
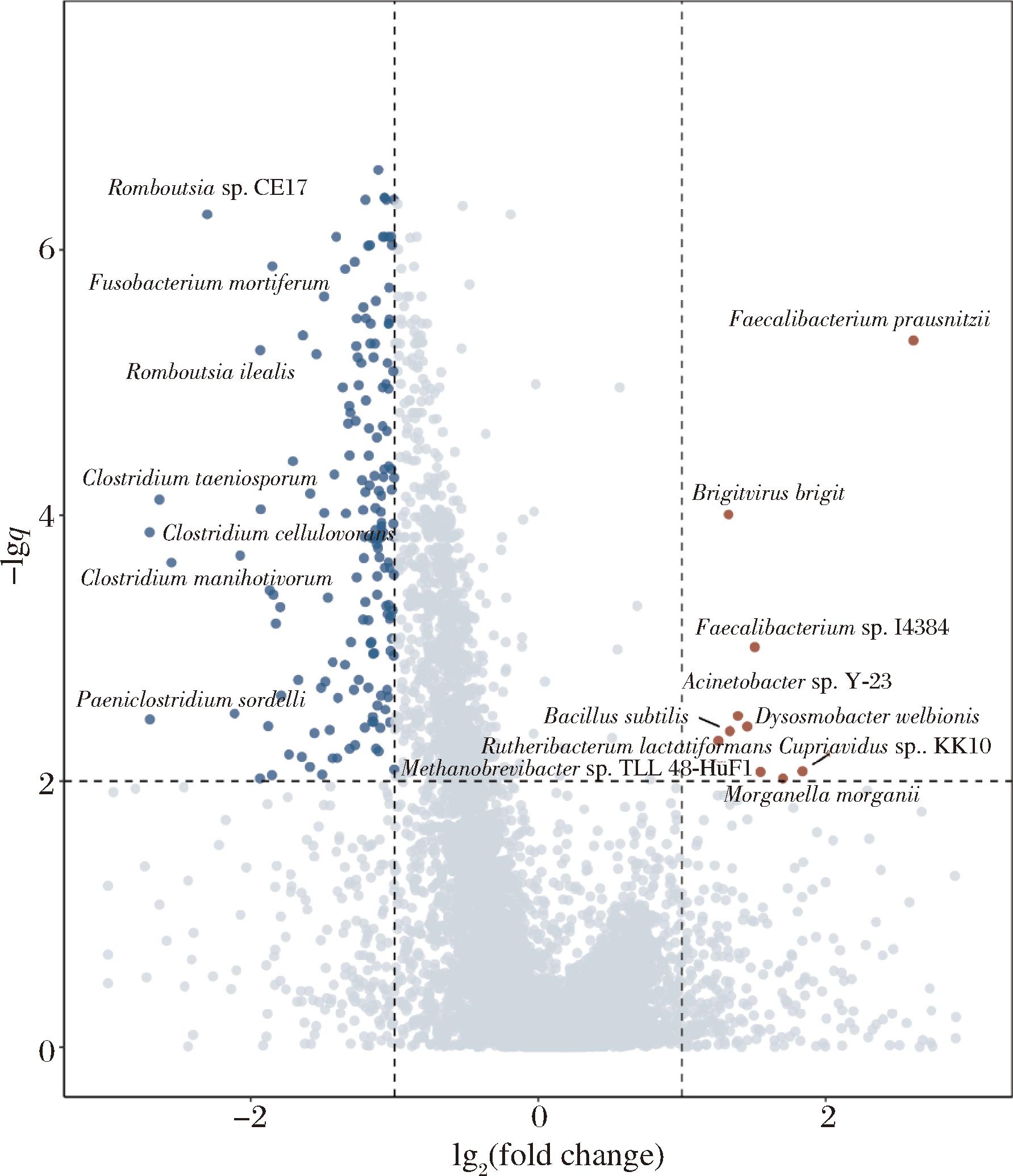
图4 宏基因组物种火山图
Fig.4 Metagenomic species volcano plot
注:蓝色点代表在高碳水膳食组增加物种,红色点代表在高脂肪 膳食组中增加的物种。
2.4 Pathway分析
肠道菌群代谢途径差异分析结果如图5所示,将高脂肪膳食摄入与高碳水膳食摄入做比较,图5右侧为高脂肪膳食摄入显著增加的代谢途径。检测到以下代谢途径:胆汁酸降解途径(bile acid degradation)、碳水化合物生物合成(carbohydrate biosynthesis)、脂肪酸和脂质生物合成(fatty acid and lipid biosynthesis)、短链脂肪酸(fermentation to short-chain fatty acids)、前体代谢物和能力产生(generation of precursor metabolites and energy)和核苷和核苷酸生物合成(nucleoside and nucleotide biosynthesis)等途径。由结果可知,摄入高脂肪的非酒精性脂肪肝患者的胆酸降解途径(PWY-8134:bile acid 7β-dehydroxylation)显著高于高碳水膳食摄入组。胆汁酸合成是胆固醇分解代谢和维持全身胆固醇稳态的最重要途径,与肝脏代谢类疾病密切相关[29]。因此,这一代谢途径与不同的饮食习惯密切相关。同时观察到棕榈酸酯生物合成途径(PWY-5994:palmitate biosynthesis)在高脂肪膳食摄入组显著下降。一项关于淀粉-油酸酯、淀粉-棕榈酸酯、蔗糖-油酸酯和蔗糖-棕榈酸酯4种不同碳水化合物-脂肪组合的配方喂养小鼠的实验表明,棕榈酸酯喂养的小鼠肝损伤程度显著低于其他组[30],由此可以发现即使都是高脂饮食,不同脂质所得到的效果也不一致。

图5 肠道菌群差异代谢通路分析
Fig.5 Analysis of differential metabolic pathways of gut microbiota
根据以上结果,猜测高脂肪膳食摄入的非酒精性脂肪肝患者长期食用脂肪含量较高的动物肉类所导致肠道优势菌群为F.prausnitzii。F.prausnitzii作为主要的丁酸产生菌,产生的丁酸会通过门静脉直接作用于肝脏的脂肪酸受体,如G蛋白偶联受体(G protein-coupled receptors,GPCRs),这些受体参与调控胆汁酸的合成,胆汁酸是激活肝脏和肠道内受体(如FXR和TGR5)的重要信号分子,通过这些受体,胆汁酸参与调控葡萄糖、脂质和能量的代谢[31-32]。因此F.prausnitzii可能通过改变胆汁酸的组成和浓度,间接调节这些信号通路,从而影响宿主的肝脏脂质代谢;而长时间摄入高碳水则富集了大量梭菌,虽然梭菌可能与棕榈酸酯合成通路无直接关系,但大量的梭菌富集可能会影响到宿主肠道中乙酸,乙酸被肝脏细胞用于合成长链脂肪酸,如棕榈酸,此外,乙酸也能通过激活过氧化物酶体增殖物激活受体(peroxisome proliferators-activated receptors,PPARs),调节脂肪酸的合成和贮存,从而间接影响棕榈酸酯合成通路[33-34]。
3 结论
本研究纳入30名不同饮食结构的非酒精性脂肪肝病患者,分为15名高脂肪膳食摄入患者和15名高碳水膳食摄入患者,基于宏基因组学测序方法研究患者的肠道菌群。结果发现,两种膳食模式下的非酒精性脂肪肝病患者肠道菌群存在显著差异,在种水平上,长期高碳水膳食摄入的患者肠道菌群以梭菌为主,长期高脂肪膳食摄入的患者肠道优势菌群则主要是F.prausnitzii,在功能上,高脂肪膳食摄入患者富集在胆酸代谢通路上,而高碳水膳食摄入患者富集在棕榈酸酯合成通路上。这一研究对未来的非酒精性脂肪肝动物特异性造模和进一步科学合理地指导非酒精性脂肪肝患者合理膳食有一定意义。
[1] PEKKALA S.Fecal metagenomics and metabolomics identifying microbial signatures in non-alcoholic fatty liver disease[J].International Journal of Molecular Sciences, 2023, 24(5):4855.
[2] PEREIRA K, SALSAMENDI J, CASILLAS J.The global nonalcoholic fatty liver disease epidemic:What a radiologist needs to know[J].Journal of Clinical Imaging Science, 2015, 5:32.
[3] KLEINER D E, MAKHLOUF H R.Histology of nonalcoholic fatty liver disease and nonalcoholic steatohepatitis in adults and children[J].Clinics in Liver Disease, 2016, 20(2):293-312.
[4] ZHANG X, JI X T, WANG Q, et al.New insight into inter-organ crosstalk contributing to the pathogenesis of non-alcoholic fatty liver disease (NAFLD)[J].Protein &Cell, 2018, 9(2):164-177.
[5] WONG V W S, ADAMS L A, DE LÉDINGHEN V, et al.Noninvasive biomarkers in NAFLD and NASH-current progress and future promise[J].Nature Reviews Gastroenterology &Hepatology, 2018, 15(8):461-478.
[6] MAKRI E, GOULAS A, POLYZOS S A.Epidemiology, pathogenesis, diagnosis and emerging treatment of nonalcoholic fatty liver disease[J].Archives of Medical Research, 2021, 52(1):25-37.
[7] BUZZETTI E, PINZANI M, TSOCHATZIS E A.The multiple-hit pathogenesis of non-alcoholic fatty liver disease (NAFLD)[J].Metabolism-Clinical and Experimental, 2016, 65(8):1038-1048.
[8] PEZZINO S, SOFIA M, FALETRA G, et al.Gut-liver axis and non-alcoholic fatty liver disease:A vicious circle of dysfunctions orchestrated by the gut microbiome[J].Biology, 2022, 11(11):1622.
[9] NASSIR F.NAFLD:Mechanisms, treatments, and biomarkers[J].Biomolecules, 2022, 12(6):824.
[10] MASOODI M, GASTALDELLI A, HYÖTYL INEN T, et al.Metabolomics and lipidomics in NAFLD:Biomarkers and non-invasive diagnostic tests[J].Nature Reviews Gastroenterology &Hepatology, 2021, 18(12):835-856.
INEN T, et al.Metabolomics and lipidomics in NAFLD:Biomarkers and non-invasive diagnostic tests[J].Nature Reviews Gastroenterology &Hepatology, 2021, 18(12):835-856.
[11] AZIZ T, HUSSAIN N, HAMEED Z, et al.Elucidating the role of diet in maintaining gut health to reduce the risk of obesity, cardiovascular and other age-related inflammatory diseases:Recent challenges and future recommendations[J].Gut Microbes, 2024, 16(1):2297864.
[12] DAVID L A, MAURICE C F, CARMODY R N, et al.Diet rapidly and reproducibly alters the human gut microbiome[J].Nature, 2014, 505(7484):559-563.
[13] HYDES T, ALAM U, CUTHBERTSON D J.The impact of macronutrient intake on non-alcoholic fatty liver disease (NAFLD):Too much fat, too much carbohydrate, or just too many calories?[J].Frontiers in Nutrition, 2021, 8:640557.
[14] FEDCHUK L, NASCIMBENI F, PAIS R, et al.Performance and limitations of steatosis biomarkers in patients with nonalcoholic fatty liver disease[J].Alimentary Pharmacology &Therapeutics, 2014, 40(10):1209-1222.
[15] WANG T Y, WANG R F, BU Z Y, et al.Association of metabolic dysfunction-associated fatty liver disease with kidney disease[J].Nature Reviews Nephrology, 2022, 18(4):259-268.
[16] FAZEL Y, KOENIG A B, SAYINER M, et al.Epidemiology and natural history of non-alcoholic fatty liver disease[J].Metabolism-Clinical and Experimental, 2016, 65(8):1017-1025.
[17] FANG Y L, CHEN H, WANG C L, et al.Pathogenesis of non-alcoholic fatty liver disease in children and adolescence:From “two hit theory” to “multiple hit model”[J].World Journal of Gastroenterology, 2018, 24(27):2974-2983.
[18] 潘芳, 朱玲玲, 王宏星.不同饮食结构对围绝经期妇女人体成分和糖代谢的影响[J].广西医学, 2021, 43(6):690-694.PAN F, ZHU L L, WANG H X.Effects of different dietary patterns on body composition and glycemic metabolism in perimenopausal women[J].Guangxi Medical Journal, 2021, 43(6):690-694.
[19] BOLGER A M, LOHSE M, USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics, 2014, 30(15):2114-2120.
[20] QUINLAN A R, HALL I M.BEDTools:A flexible suite of utilities for comparing genomic features[J].Bioinformatics, 2010, 26(6):841-842.
[21] BEGHINI F, MCIVER L J, BLANCO-M GUEZ A, et al.Integrating taxonomic, functional, and strain-level profiling of diverse microbial communities with bioBakery 3[J].Elife, 2021, 10:e65088.
GUEZ A, et al.Integrating taxonomic, functional, and strain-level profiling of diverse microbial communities with bioBakery 3[J].Elife, 2021, 10:e65088.
[22] SEGATA N, WALDRON L, BALLARINI A, et al.Metagenomic microbial community profiling using unique clade-specific marker genes[J].Nature Methods, 2012, 9(8):811-814.
[23] 覃一书, 保欣晨, 汪洁, 等.不同饮食习惯下镉摄入对肠道菌群结构的影响[J].中国环境科学, 2021, 41(8):3896-3905.QIN Y S, BAO X C, WANG J, et al.Effect of cadmium intake on the structure of gut microbiota under different dietary habits[J].China Environmental Science, 2021, 41(8):3896-3905
[24] ZHANG Y L, HU J L, TAN H Z, et al.Akkermansia muciniphila, an important link between dietary fiber and host health[J].Current Opinion in Food Science, 2022, 47.
[25] DOUGAL K, DE LA FUENTE G, HARRIS P A, et al.Characterisation of the faecal bacterial community in adult and elderly horses fed a high fibre, high oil or high starch diet using 454 pyrosequencing[J].PloS One, 2014, 9(2):e87424.
[26] WAN Y, WANG F L, YUAN J H, et al.Effects of dietary fat on gut microbiota and faecal metabolites, and their relationship with cardiometabolic risk factors:A 6-month randomised controlled-feeding trial[J].Gut, 2019, 68(8):1417-1429.
[27] 魏慧, 段丽萍.膳食对肠道菌群结构、代谢和功能影响的研究进展[J].中华消化杂志, 2017, 37(9):642-644.WEI H, DUAN L P.Research progress on the effect of diet on the structure, metabolism and function of intestinal flora[J].Chinese Journal of Digestion, 2017, 37(9):642-644.
[28] WANG C, YANG Y T, CHEN J Y, et al.Berberine protects against high-energy and low-protein diet-induced hepatic steatosis:Modulation of gut microbiota and bile acid metabolism in laying hens[J].International Journal of Molecular Sciences, 2023, 24(24):17304.
[29] CHIANG J Y L, FERRELL J M.Bile acid receptors FXR and TGR5 signaling in fatty liver diseases and therapy[J].American Journal of Physiology-Gastrointestinal and Liver Physiology, 2020, 318(3):G554-G573.
[30] PIERCE A A, PICKENS M K, SIAO K, et al.Differential hepatotoxicity of dietary and DNL-derived palmitate in the methionine-choline-deficient model of steatohepatitis[J].BMC Gastroenterology, 2015, 15:72.
[31] MIQUEL S, MART N R, ROSSI O, et al.Faecalibacterium prausnitzii and human intestinal health[J].Current Opinion in Microbiology, 2013, 16(3):255-261.
N R, ROSSI O, et al.Faecalibacterium prausnitzii and human intestinal health[J].Current Opinion in Microbiology, 2013, 16(3):255-261.
[32] SAYIN S I, WAHLSTRÖM A, FELIN J, et al.Gut microbiota regulates bile acid metabolism by reducing the levels of tauro-beta-muricholic acid, a naturally occurring FXR antagonist[J].Cell Metabolism, 2013, 17(2):225-235.
[33] FROST G, SLEETH M L, SAHURI-ARISOYLU M, et al.The short-chain fatty acid acetate reduces appetite via a central homeostatic mechanism[J].Nature Communications, 2014, 5:3611.
[34] LOUIS P, FLINT H J.Formation of propionate and butyrate by the human colonic microbiota[J].Environmental Microbiology, 2017, 19(1):29-41.