γ-氨基丁酸(γ-aminobutyric acid,GABA),又称4-氨基丁酸,是自然界中普遍存在的一种非蛋白质氨基酸[1-2],广泛分布于动植物和微生物中,具有重要的生理功能[3-7]。此外,GABA还被应用于食品、饲料等众多领域[8-9]。目前生物转化法被广泛用于GABA的合成,生物转化法的主要原理是L-谷氨酸在谷氨酸脱羧酶(glutamic acid decarboxylase,GAD)的作用下,依赖辅酶磷酸吡哆醛(pyridoxal phosphate,PLP),脱去羧基形成一分子CO2生成GABA。该方法具有可选择的微生物种类多、转化率高、污染小、条件温和、分离提纯较为方便等优点[10]。
大肠杆菌具有培养简单、生产周期短等优点,被广泛应用于GABA的生产中[11];但由于大肠杆菌中存在GABA降解的相关途径,因此该方法后期存在GABA消耗的问题[11-13]。在大肠杆菌中,已经确定的GABA降解途径主要是GABA形成琥珀酸后回到三羧酸循环(tricarboxylic acid cycle,TCA循环)[14]。该途径涉及2种酶,即GABA氨基转移酶(GABA transaminase,GABA-T)和琥珀酸半醛脱氢酶(succinic semialdehyde dehydrogenase,SSADH)[15]。GABA-T是PLP依赖型酶,它催化GABA和琥珀酸半醛(SSA)之间的互相转化,这是GABA降解途径的第一步。随后SSADH将SSA转化为琥珀酸(succinic acid,SA),SA进入TCA循环[16]。从理论上说,SSA还可以逆向形成GABA,但是SSADH的活性很高,促进反应朝着形成SA的方向进行[17]。GABA-T包括GABA氨基转移酶(GABAtransaminase,GABT)和β-丙氨酸氨基转移酶(β-alanine aminotransferase,PUUE)2个同工酶[18],它们分别由gabT基因和puuE基因编码[19]。有研究表明,敲除gabT基因可以减少GABA进入下游代谢途径,从而提高GABA的产量[20-21]。IM等[20]将突变后的GAD和谷氨酸/GABA逆向转运蛋白(glutamate/GABAantiporter,GadC)在gabT基因缺失的大肠杆菌中过表达,并利用该菌株以葡萄糖为碳源合成GABA,产量由5.1 mmol/L提高到7.8 mmol/L。PHAM等[21]利用蛋白支架在大肠杆菌中定位了谷氨酸合成酶、谷氨酸脱羧酶和GABA转运蛋白,并敲除基因组上的gabT基因,该基因工程菌以10 g/L的葡萄糖为碳源,产生了1.16 g/L的GABA。但是,YANG等[12]的研究表明,敲除大肠杆菌基因组上的gabT基因会抑制大肠杆菌的生长,对大肠杆菌造成代谢负担。
基因组数据显示大肠杆菌中存在pepD基因,该基因可编码胞质非特异性二肽酶(cytosolic nonspecific dipeptidase,pepD)。该酶是一种锌金属酶,由2个亚基组成,可以水解具有游离氨基和游离羧基组成的二肽,且具有较为广泛的底物谱[22]。在没有金属离子的存在下,水解速率会降低;其最适反应pH值为8.3左右。大肠杆菌来源的二肽酶比酶活力最高可达760 U/mg [23]。JING等[24]研究表明,敲除大肠杆菌基因组上的pepD基因可以有效抑制Ala-Gln二肽降解为L-丙氨酸和谷氨酰胺。
本研究利用CRISPR-Cas9系统构建了pepD基因敲除菌株,敲除后的菌株生长未受影响;并通过质粒回补实验、全细胞催化实验,测定了该酶对GABA的消耗历程。此外,本文利用pepD纯酶对GABA进行催化并借助LC-MS对产物进行了鉴定。本研究对工业化生产GABA具有重要的借鉴意义。
1 材料与方法
1.1 材料
1.1.1 主要试剂
谷氨酸钠和GABA作为反应的底物购自国药集团。苯基异硫酸酯(phenylisothiocyanate,PITC)作为氨基酸衍生试剂购自国药集团。异丙基β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)作为pepD表达的诱导剂购自上海生工。阿拉伯糖作为GadCΔC41表达的诱导剂购自北京伊诺凯。除非另有说明,所有使用的其他试剂都是分析级或色谱级。
1.1.2 质粒与菌株
本研究所使用菌株与质粒见表1。
表1 本研究中使用的主要菌株和质粒
Table 1 Main strains and plasmids used in this study

菌株与质粒性质来源E.coli BL21(DE3)表达宿主实验室保存E.coli JM109克隆宿主实验室保存E.coli BL21(DE3)ΔpepD表达宿主本研究构建pET-28a(+)His标签、Kana抗性、T7启动子实验室保存pET28a-pepDHis标签、Kana抗性、T7启动子本研究构建pKD46-ADI-dCas9-sgRNAP43启动子、Amp抗性、araBAD启动子本研究构建p15A- GadCΔC41araBAD启动子、Cm抗性实验室保存
1.1.3 主要培养基
LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10。
LB固体培养基:LB液体培养基中加入20 g/L的琼脂粉。
2×YT液体培养基(g/L):蛋白胨16,酵母粉10,NaCl 5。
1.2 实验方法
1.2.1 质粒构建
所有片段均由高保真DNA聚合酶扩增。用引物对pET28a-F/pET28a-R构建质粒pET-28a的线性化载体,用引物对 pepD-F/pepD-R,以E.coli基因组为模板,扩增pepD基因片段,利用Gibson组装构建质粒pET28a-pepD。本实验所使用的引物见表2。
表2 本实验所用的引物
Table 2 Primers used in this study
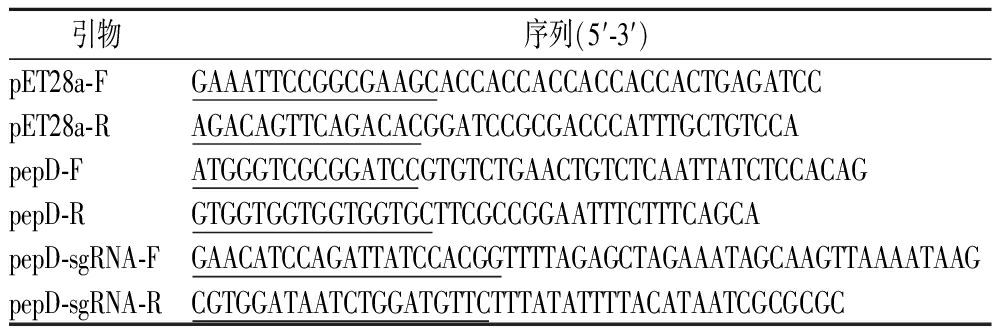
引物序列(5′-3′)pET28a-FGAAATTCCGGCGAAGCACCACCACCACCACCACTGAGATCCpET28a-RAGACAGTTCAGACACGGATCCGCGACCCATTTGCTGTCCApepD-FATGGGTCGCGGATCCGTGTCTGAACTGTCTCAATTATCTCCACAGpepD-RGTGGTGGTGGTGGTGCTTCGCCGGAATTTCTTTCAGCApepD-sgRNA-FGAACATCCAGATTATCCACGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGpepD-sgRNA-RCGTGGATAATCTGGATGTTCTTTATATTTTACATAATCGCGCGC
1.2.2 宿主菌pepD基因的敲除
本文采用CRISPR-Cas9系统进行了pepD基因的敲除,所用的敲基因质粒为pKD46(温度敏感型,含有阿拉伯糖启动子调控的AID-dCas9,P43启动子调控的sgRNA,Ampr)。利用chopchop网站(http://chopchop.cbu.uib.no)设计了sgRNA,用引物对pepD-sgRNA-F/pepD-sgRNA-R进行 PCR 扩增,得到两端为同源臂、中间为 UGI-AID-dCas9-Amp基因片段的线性同源重组片段(大小为9 770 bp)。将同源重组片段化转入E.coli JM109菌株中构建质粒,送至金唯智公司测序,将测序正确的质粒化转入E.coli BL21(DE3)菌株中。在氨苄青霉素抗性平板上挑取转化子接入5 mL试管中,在30 ℃下培养6~8 h后,加入终浓度为10 mmol/L的阿拉伯糖,过夜培养。将过夜培养的菌液稀释106倍后涂布至无抗平板,在42 ℃下培养至形成单菌落。挑取5~10个单克隆用引物pepD-F/pepD-R进行菌落PCR,PCR产物送至金唯智测序,将测序正确的菌落进行保菌。
1.2.3 培养条件及全细胞催化方法
将-80 ℃保存的重组菌株在LB平板(含50 mg/mL 卡那霉素或34 mg/mL氯霉素)上划线活化,挑取单菌落接种至LB液体培养基中,37 ℃振荡培养6~8 h,然后以2%的接种量转接入50 mL的2×YT摇瓶液体培养基中,于37 ℃振荡培养2~3 h至OD600值约为0.6~0.8时,加入0.2 mmol/L IPTG或1.0 mmol/L L-阿拉伯糖诱导培养12~16 h,收集诱导结束的菌体(8 000 r/min,10 min,4 ℃)。
全细胞催化体系:将OD600=15的菌体与1 mol/L的谷氨酸钠或GABA混合,在37 ℃,200 r/min下进行催化,每隔0.5 h或1 h取一次样,反应4 h。然后在100 ℃下灭活10 min。灭活的混合物在12 000 r/min下离心2 min,上清液用HPLC检测MSG和GABA的浓度。
1.2.4 pepD酶的纯化及纯酶反应条件
按1.2.3节所述方法收集诱导结束的菌体,将菌体用HisTrap HP结合缓冲液(20 mmol/L NaH2PO4、500 mmol/L NaCl、10 mmol/L咪唑、pH 7.4)重悬,使用超声破碎仪在冰上破碎15~30 min后,12 000 r/min离心25 min取上清液,用0.22 μm滤膜过滤上清液去除杂质。含pepD的上清溶液使用HisTrap HP纯化柱进行亲和层析,具体操作如下:首先用HisTrap HP结合缓冲液平衡HisTrap HP纯化柱5~10个柱体积,流速为1 mL/min,然后将上清液过HisTrap HP纯化柱,之后使用HisTrap HP结合缓冲液平衡5~10个柱体积,利用洗脱缓冲液(20 mmol/L NaH2PO4、500 mmol/L NaCl、500 mmol/L咪唑、pH 7.4)线性洗脱目的蛋白,收集酶蛋白。在20 mmol/L、pH 7.0 的磷酸缓冲液中过夜透析。用SDS-PAGE检查酶蛋白的纯度,酶浓度用Bradford方法测定。
纯酶反应体系:1 mL体系中,加入0.4 mg/mL酶液、20 mmol/L ATP和1 mol/L GABA,在37 ℃金属浴中分别反应1、2、3、4 h,于100 ℃下高温灭活10 min 终止反应。灭活的反应液在12 000 r/min下离心2 min,上清液用HPLC检测GABA的浓度。
1.2.5 氨基酸检测方法
谷氨酸钠和GABA的浓度均采用HPLC进行检测。样品首先经过PITC衍生处理:取500 μL稀释后的反应液,分别加入250 μL 1 mol/L的三乙胺-乙腈溶液和250 μL 0.1 mol/L的PITC-乙腈溶液,混匀后避光衍生50 min;随后加入750 μL的正己烷,充分振荡以终止反应、静置后待溶液分层。用1 mL的注射器吸取下层溶液并用0.22 μm的有机滤膜进行过滤后所制得的溶液用于氨基酸的检测。然后使用色谱柱La Chrom C18(4.6 mm×250 mm, 5 μm)进行检测,流动相A为80%的乙腈溶液,流动相B为97%的0.1 mol/L乙酸钠和3%的乙腈的混合溶液,流速为0.6 mL/min,检测波长为254 nm,进样量10 μL。梯度洗脱条件为:0~35 min,流动相B由95%降至70%;35~40 min,流动相B由70%升至95%;40~45 min,B流动相浓度不变。
1.2.6 二肽检测方法
将纯酶反应液稀释后,用1 mL注射器吸取样品并用0.22 μm的水系滤膜过滤以除去杂质,所制得的溶液用于二肽的检测。利用LC-MS检测反应液中的二肽,具体检测方法参考文章[25]。
2 结果与分析
2.1 pepD对MSG和GABA的催化
利用基因工程菌生产GABA,存在GABA消耗问题。构建了催化L-谷氨酸粉末合成GABA的基因工程菌,1 mol/L底物的摩尔转化率维持在75%。YANG等[12]通过敲除基因组上gabT基因缓解GABA消耗,但是大肠杆菌的生长受到严重抑制。为了解决该问题,本文从KEGG网站(https://www.kegg.jp/)上寻找GABA的下游代谢途径,发现大肠杆菌中含有一个pepD基因,该基因编码了一种胞质非特异性二肽酶,可能导致GABA参与了成肽反应。基于此,构建了pepD基因敲除菌株(ΔpepD),并通过在其中过表达pepD基因构建了OEpepD菌株,借助2组菌株来确定该酶是否能以GABA为底物进行反应。将OEpepD菌株与ΔpepD菌株分别置于含谷氨酸钠、GABA和二者共存的溶液中,检测2种物质的消耗程度。反应进行3 h后,发现2组菌株的反应液中谷氨酸钠的浓度并没有明显变化,这表明该酶不能以谷氨酸钠为底物催化(图1-a)。然而2组菌株的反应液中GABA的浓度明显下降(图1-b、图1-c)。由于大肠杆菌中GABA进入TCA循环的途径[14]并未被阻断,因此ΔpepD菌株能消耗GABA;但是OEpepD菌株的GABA消耗量比ΔpepD菌株高出17.5%,表明pepD酶催化GABA发生了反应。
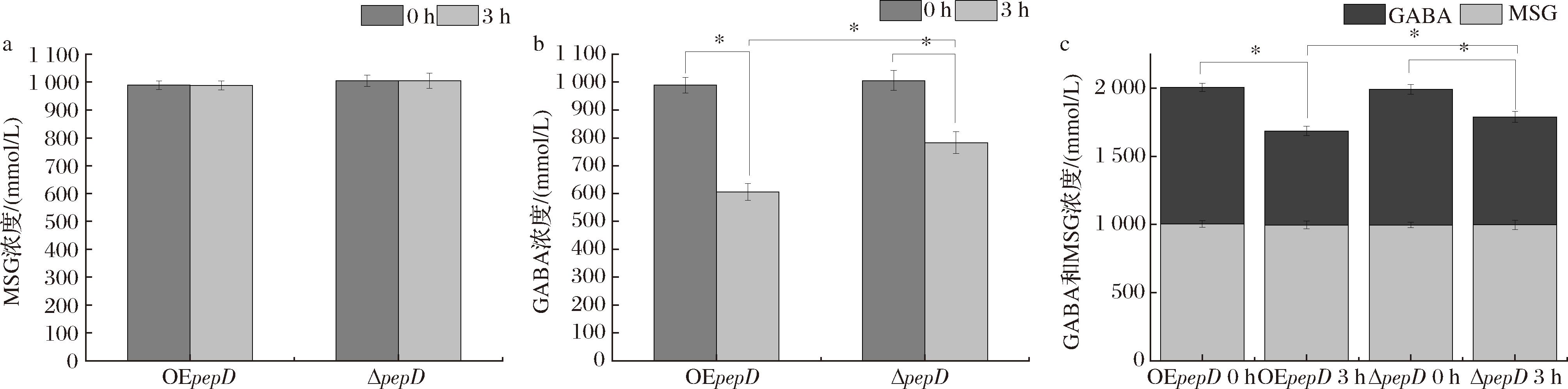
a-MSG为底物;b-GABA为底物;c-MSG和GABA为底物
图1 pepD对MSG和GABA的催化作用
Fig.1 Catalysis of pepD on MSG and GABA
注:*表示在各组间存在显著性差异(P<0.05)。
2.2 全细胞GABA消耗历程
为了探究2组菌株消耗GABA的具体情况,本文对OEpepD菌株与ΔpepD菌株消耗GABA的历程进行了监测,结果如图2所示。2组菌株对于GABA的消耗均集中在前2 h,且OEpepD菌株比ΔpepD菌株高出15.7%,在反应2 h后GABA浓度趋于稳定。这表明大肠杆菌对GABA的消耗主要集中在前2 h。

图2 全细胞GABA消耗历程
Fig.2 Whole-Cell GABA consumption process
2.3 pepD纯酶反应及产物检测
对全细胞催化的反应液进行了LC-MS检测,结果显示有GABA形成的二肽峰,但是吸收峰值较低。为了进一步验证pepD对GABA的催化作用,对其进行表达与纯化,结果如图3所示。以未诱导全细胞作为对照,诱导后的蛋白表达大小在50 kDa左右,与预测性相符合,说明pepD表达成功,目的蛋白可用于进一步的纯酶反应。
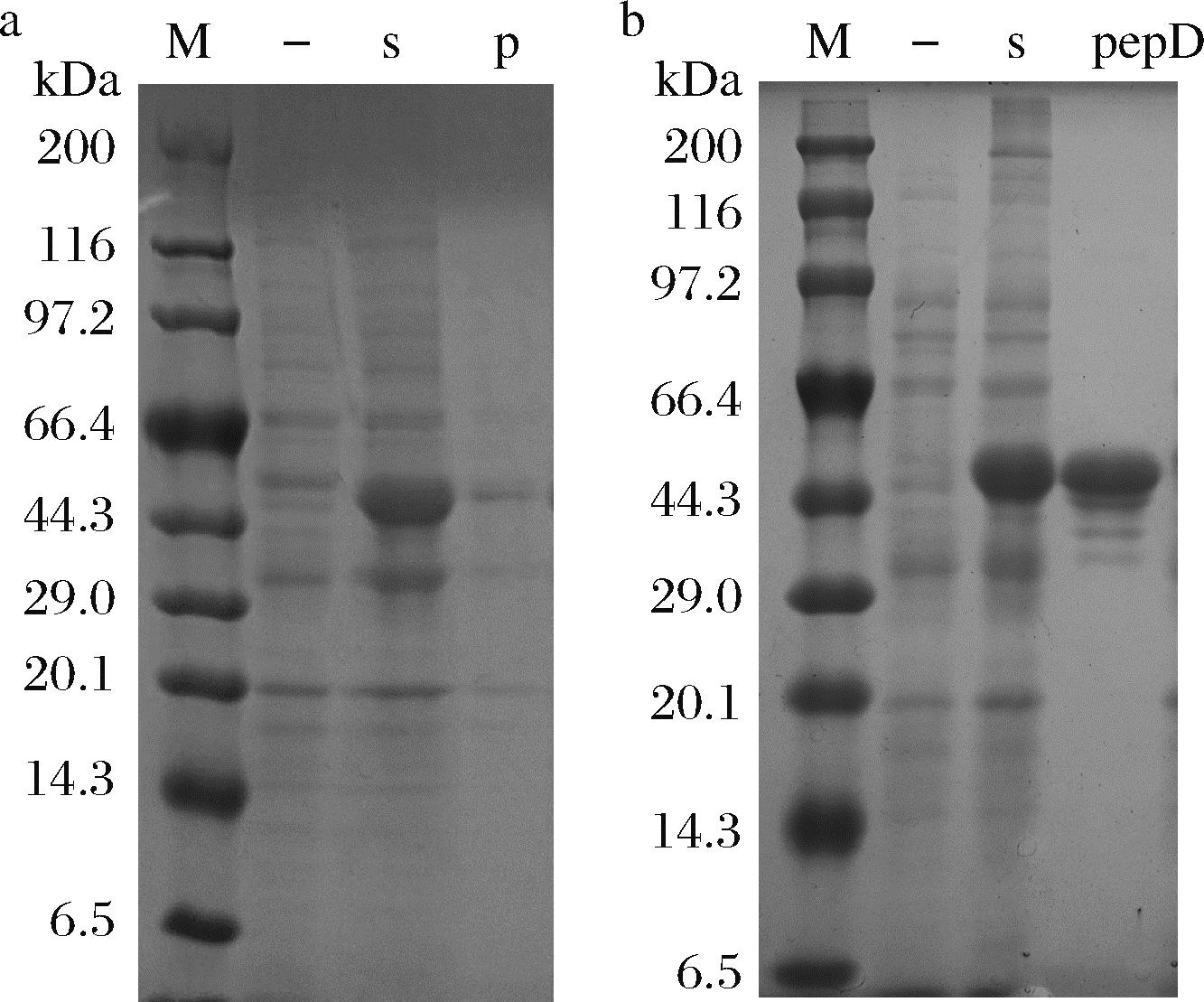
M:Marker;-:未诱导全细胞;s:诱导后破碎上清;p:诱导后破碎沉淀 a-pepD酶过表达;b-pepD酶纯化结果
图3 pepD表达与纯化
Fig.3 pepD expression and purification
本文检测了pepD纯酶催化GABA的反应历程,结果如图4-a所示。在反应前1 h,GABA浓度下降到777.9 mmol/L,1 h后GABA浓度趋于稳定,维持在759.2 mmol/L左右。反应的产物用三重四极杆液质联用仪进行了检测,以反应前的溶液作为对照,反应2 h后的溶液在正离子通道中分子量189.05的位置检测到了明显的吸收峰(图4-b),根据GABA的分子质量推算,该物质是GABA形成的二肽,由于GABA二肽的标准品没有购买到,因此未能对其进行定量分析。

a-纯酶反应历程;b-LC-MS检测结果
图4 pepD纯酶催化
Fig.4 Catalysis of purified pepD
2.4 敲除pepD基因对菌体生长的影响
目前用于提高GABA产量、抑制GABA消耗的主要方法是敲除编码4-氨基丁酸氨基转移酶的基因(gabT),即抑制GABA进入TCA循环[20-21]。但伴随着gabT基因的敲除,细胞生长受到了严重影响,研究表明gabT基因敲除菌株的细胞生物量仅为未敲除菌株的62.9%[12]。
本文对ΔpepD菌株和野生型菌株(WT)进行了细胞生物量的测定,结果如图5所示。ΔpepD菌株和WT菌株在前12 h中OD600值逐渐提升,且二者增幅相似;12 h后二者OD600值均达到7.3左右,后期逐渐趋于平缓。通过对比二者的生长曲线可以发现pepD基因的敲除对于大肠杆菌的生长没有显著影响,该菌株具有工业应用潜力。
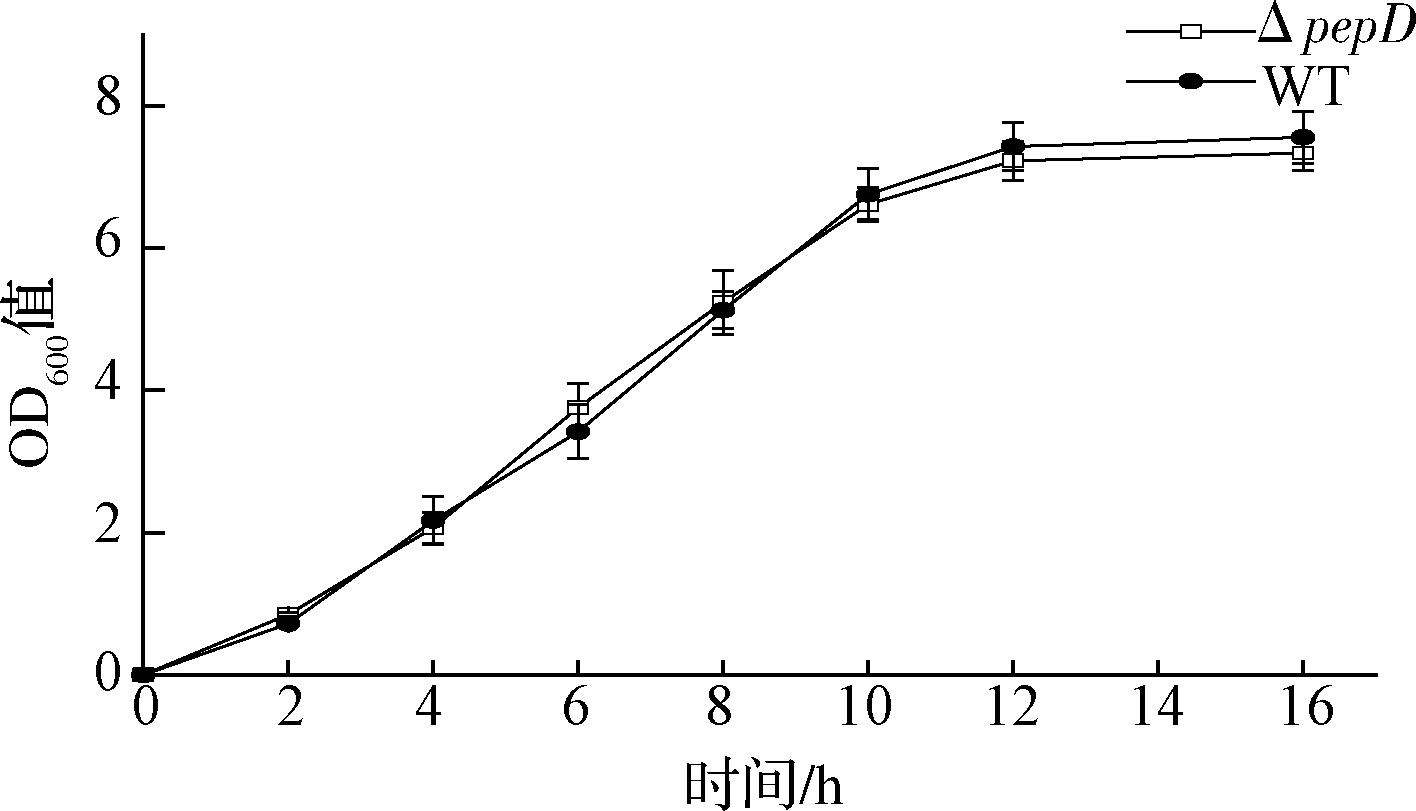
图5 菌体生长曲线
Fig.5 Bacterial growth curve
3 结论与讨论
本研究探索了pepD酶对GABA的催化作用,即pepD酶催化GABA形成二肽。目前关于pepD酶的报道主要集中在其水解二肽的活性,关于pepD酶合成二肽的功能却鲜为人知。有研究表明该酶可以β-丙氨酸和L-组氨酸为底物合成肌肽,需要Mn2+激活[26]。本研究通过纯酶催化GABA,证实GABA可被pepD酶催化形成二肽,该反应是首次被报道。此外,细胞生长检测结果显示pepD基因的敲除不会对大肠杆菌的生长产生不利影响,该发现可有效缓解GABA积累和菌体生长之间的矛盾。GABA的生物合成方法主要是全细胞催化,菌株生物量直接影响生产成本,本研究结果为GABA的工业化生产提供重要的理论基础和实践价值。
[1] 王泳超. γ-氨基丁酸(GABA)调控盐胁迫下玉米种子萌发和幼苗生长的机制[D].哈尔滨:东北农业大学, 2016.WANG Y C.Mechanism of aminobutyric acid (GABA) regulating maize seed germination and seedling growth under salt stress[D].Harbin:Northeast Agricultural University, 2016.
[2] RAMOS-RUIZ R, POIROT E, FLORES-MOSQUERA M.GABA, a non-protein amino acid ubiquitous in food matrices[J].Cogent Food &Agriculture, 2018, 4(1):1534323.
[3] MILOSEVIC L, GRAMER R, KIM T H, et al.Modulation of inhibitory plasticity in basal ganglia output nuclei of patients with Parkinson’s disease[J].Neurobiology of Disease, 2019, 124:46-56.
[4] SHEKARI A, NAGHSHIBAND HASSANI R, SOLEIMANI AGHDAM M.Exogenous application of GABA retards cap browning in Agaricus bisporus and its possible mechanism[J].Postharvest Biology and Technology, 2021, 174:111434.
[5] LYU C J, ZHAO W R, PENG C L, et al.Exploring the contributions of two glutamate decarboxylase isozymes in Lactobacillus brevis to acid resistance and γ-aminobutyric acid production[J].Microbial Cell Factories, 2018, 17(1):180.
[6] JIN L F, CAI Y T, SUN C, et al.Exogenous L-glutamate treatment could induce resistance against penicillium expansum in pear fruit by activating defense-related proteins and amino acids metabolism[J].Postharvest Biology and Technology, 2019, 150:148-157.
[7] 王辉, 项丽丽, 张锋华.γ-氨基丁酸(GABA)的功能性及在食品中的应用[J].食品工业, 2013, 34(6):186-189.WANG H, XIANG L L, ZHANG F H.Function and application in food industry of γ-aminobutyric acid[J].Food Industry, 2013, 34(6):186-189.
[8] 宁亚维, 马梦戈, 杨正, 等.γ-氨基丁酸的制备方法及其功能食品研究进展[J].食品与发酵工业, 2020, 46(23):238-247.NING Y W, MA M G, YANG Z, et al.Research progress in the enrichment process and functional foods of γ-aminobutyric acid[J].Food and Fermentation Industries, 2020, 46(23):238-247.
[9] 吴凡, 谭青松.γ-氨基丁酸在动物营养中的研究进展[J].饲料研究, 2015, (2):61-65.WU F, TAN Q S.Research progress of γ-aminobutyric acid in animal nutrition[J].Feed Research, 2015, (2):61-65.
[10] 侯亚男, 冯昆.中药微生物转化研究进展[J].云南中医中药杂志, 2019, 40(12):66-69.HOU Y N, FENG K.Research progress on microbial transformation of traditional Chinese medicine[J].Yunnan Journal of Traditional Chinese Medicine and Materia Medica, 2019,40(12):66-69.
[11] YU P, REN Q, WANG X X, et al.Enhanced biosynthesis of γ-aminobutyric acid (GABA) in Escherichia coli by pathway engineering[J].Biochemical Engineering Journal, 2019, 141:252-258.
[12] YANG X W, KE C R, ZHU J M, et al.Enhanced productivity of gamma-amino butyric acid by cascade modifications of a whole-cell biocatalyst[J].Applied Microbiology and Biotechnology, 2018, 102(8):3623-3633.
[13] KE C R, YANG X W, RAO H X, et al.Whole-cell conversion of L-glutamic acid into gamma-aminobutyric acid by metabolically engineered Escherichia coli[J].SpringerPlus, 2016, 5:591.
[14] NIEGEMANN E, SCHULZ A, BARTSCH K.Molecular organization of the Escherichia coli gab cluster:Nucleotide sequence of the structural genes gabD and gabP and expression of the GABA permease gene[J].Archives of Microbiology, 1993, 160(6):454-460.
[15] METZER E, LEVITZ R, HALPERN Y S.Isolation and properties of Escherichia coli K-12 mutants impaired in the utilization of gamma-aminobutyrate[J].Journal of Bacteriology, 1979, 137(3):1111-1118.
[16] KUMAR S, PUNEKAR N S, SATYANARAYAN V, et al.Metabolic fate of glutamate and evaluation of flux through the 4-aminobutyrate (GABA) shunt in Aspergillus niger[J].Biotechnology and Bioengineering, 2000, 67(5):575-584.
[17] LE VO T D, KIM T W, HONG S H.Effects of glutamate decarboxylase and gamma-aminobutyric acid (GABA) transporter on the bioconversion of GABA in engineered Escherichia coli[J].Bioprocess and Biosystems Engineering, 2012, 35(4):645-650.
[18] KURIHARA S, KATO K, ASADA K, et al.A Putrescine-inducible pathway comprising PuuE-YneI in which γ-aminobutyrate is degraded into succinate in Escherichia coli K-12[J].Journal of Bacteriology, 2010, 192(18):4582-4591.
[19] PRICE C W, FAWCETT P, CÉRÉMONIE H, et al.Genome-wide analysis of the general stress response in Bacillus subtilis[J].Molecular Microbiology, 2001, 41(4):757-774.
[20] IM D K, HONG J, GU B, et al.13C metabolic flux analysis of Escherichia coli engineered for gamma-aminobutyrate production[J].Biotechnology Journal, 2020, 15(6):e1900346.
[21] PHAM V D, SOMASUNDARAM S, LEE S H, et al.Redirection of metabolic flux into novel gamma-aminobutyric acid production pathway by introduction of synthetic scaffolds strategy in Escherichia coli[J].Applied Biochemistry and Biotechnology, 2016, 178(7):1315-1324.
[22] PATTERSON E K, GATMAITAN J S, HAYMAN S.Substrate specificity and pH dependence of dipeptidases purified from Escherichia coli B and from mouse ascites tumor cells[J].Biochemistry, 1973, 12(19):3701-3709.
[23] HAYMAN S, GATMAITAN J S, PATTERSON E K.The relationship of extrinsic and intrinsic metal ions to the specificity of a dipeptidase from Escherichia coli B[J].Biochemistry, 1974, 13(22):4486-4494.
[24] JING Z Y, XU J, LIU J, et al.Multiplex gene knockout raises Ala-Gln production by Escherichia coli expressing amino acid ester acyltransferase[J].Applied Microbiology and Biotechnology, 2023, 107(11):3523-3533.
[25] J MTGÅRD S, ROBINSON N, MORITZ T, et al.Optimising methods for the recovery and quantification of di- and tripeptides in soil[J].Soil Research, 2018, 56(4):404.
MTGÅRD S, ROBINSON N, MORITZ T, et al.Optimising methods for the recovery and quantification of di- and tripeptides in soil[J].Soil Research, 2018, 56(4):404.
[26] LIU Y R, PAN X W, ZHANG H W, et al.Combinatorial protein engineering and transporter engineering for efficient synthesis of L-carnosine in Escherichia coli[J].Bioresource Technology, 2023, 387:129628.