透明质酸酶是一种糖苷酶,可通过水解作用或β-消除反应降解透明质酸(hyaluronic acid,HA),且对硫酸软骨素(chondroitin Sulfate,CS)、硫酸皮肤素素(dermatan Sulfate,DS)等糖胺聚糖类(glycosaminoglycans, GAGs)物质具有一定程度的降解能力。许多研究表明,GAGs类物质的生物学功能主要取决于链长、分子质量和合成环境[1]。其中,较低分子质量的透明质酸及糖胺寡糖被广泛应用于医药等领域,但通过传统的物理法、化学法制备低分子质量透明质酸的周期长、效率低、成本高,难以满足工业生产的需求。相较而言,透明质酸酶对糖胺类物质的特异性降解,可安全、高效的制备低分子质量透明质酸。
1 透明质酸酶分类
目前,常见的透明质酸酶主要通过物种来源、催化方式以及氨基酸序的同源性来进行分类(图1)。
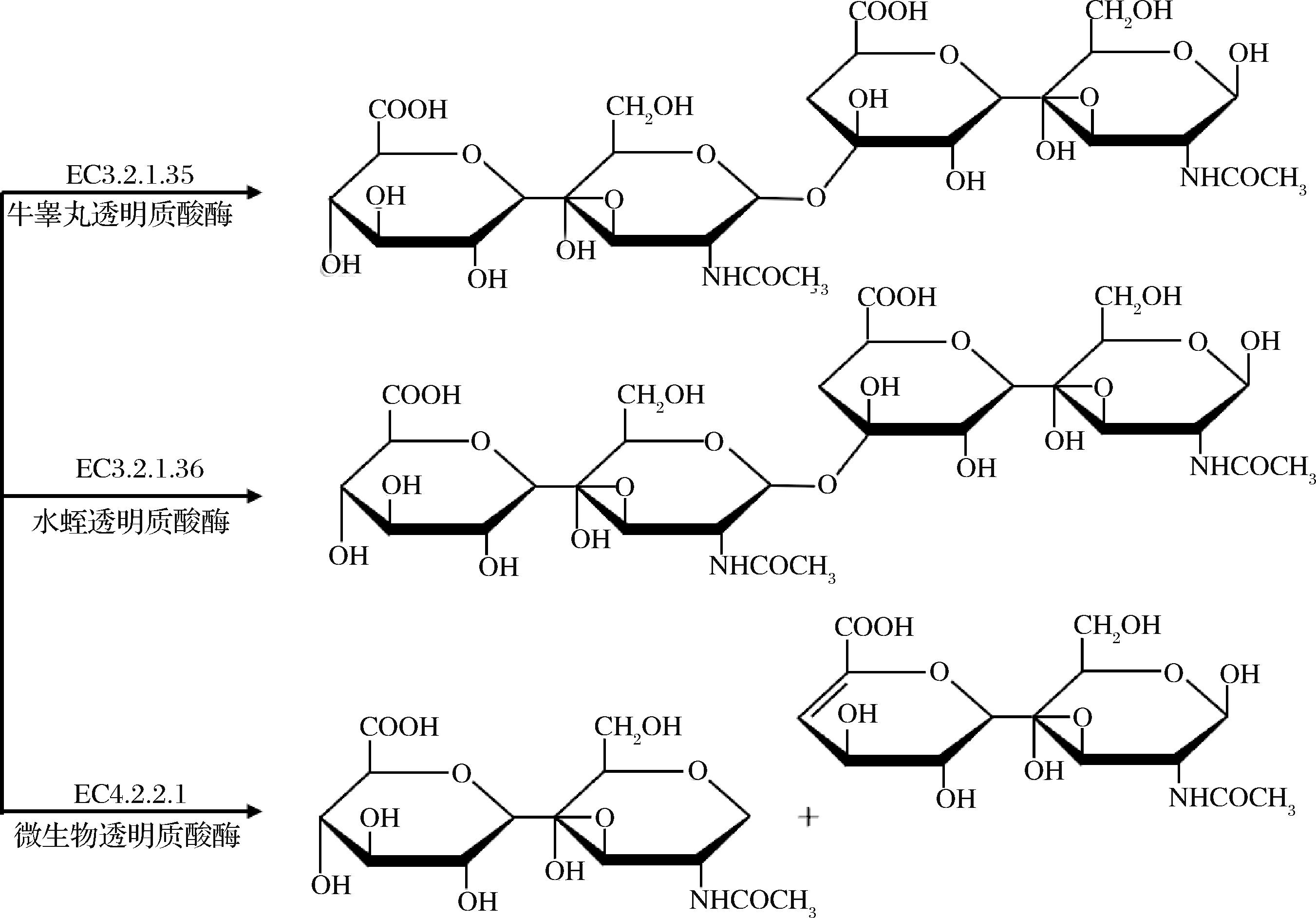
图1 透明质酸酶分类
Fig.1 Classification of hyaluronidases
第一种分类方式根据物种来源将透明质酸酶划分为哺乳动物透明质酸酶、无脊椎动物透明质酸酶、真菌来源透明质酸酶以及细菌、病毒来源的透明质酸酶。
第二种分类方式根据透明质酸酶催化的方式划分为三大家族,该分类方式是MEYER提出的一种最经典的分类方式,如图1所示。第一类是来源于真核动物(脊椎动物和毒液透明质酸酶)的内切透明质酸氨基葡糖苷酶(EC 3.2.1.35),通过水解透明质酸的β-1, 4糖苷键,产生还原性末端为N-乙酰葡萄糖胺的偶数寡糖(4和6)作为主要终产物。该类酶还可作用于硫酸皮肤素、软骨素、硫酸软骨素等物质,并同时具有转糖苷活性。第二类是以水蛭和钩虫唾液腺透明质酸酶为代表的内切透明质酸葡糖醛酸酶(EC 3.2.1.36),通过水解β-1, 3糖苷键,产生还原性末端为葡萄糖醛酸的四糖。该酶专一性较强,仅作用于透明质酸。第三类则是来源于细菌的透明质酸裂解酶(EC 4.2.2.1),通过β-消除反应裂解β-1, 4糖苷键,最终生成不饱和二糖。
第三种分类方式根据氨基酸序列的同源性,将其划分到不同的碳水化合物活性酶组(carbohydrate- active enzymes database,CAZy)中,这种分类方式系统性的反映了透明质酸酶的基因组学、结构、机制、化学组成间的共同关系,能够很好的展现出不同透明质酸酶之间的进化关系以及蛋白质空间结构上的差异。根据这种关系,将常见的透明质酸酶归类到糖苷水解酶家族(glycoside hydrolase,GH)以及糖苷裂解酶家族(polysaccharide lyase,PL)当中。其中,脊椎动物体内的透明质酸酶以及动物毒液来源的透明质酸酶共同属于糖苷水解酶家族56,能够水解β-1, 4糖苷键。水蛭透明质酸酶(leech hyaluronidase, LHyals),能够水解β-1,3糖苷键,被归类到糖苷水解酶家族79当中。细菌和噬菌体来源的透明质酸酶能够通过β-消除反应降解透明质酸,分别被归类到糖苷裂解酶家族8以及糖苷裂解酶家族16当中。
对于一些新发现的具有特殊结构性质的透明质酸酶来说,该分类方式可以很好的解决其归属问题,这点明显优于传统的分类方式。例如,青霉属透明质酸酶作为一种新型的透明质酸酶,其催化活性类似于脊椎动物透明质酸酶,根据推导出的氨基酸序列的同源性分析,该酶无法被归类到MEYER的经典分类方式当中,但根据其结构性质,能够将其归类到多糖水解酶家族16当中[2]。
2 透明质酸酶的性质及催化机理
2.1 透明质酸酶底物特异性及其催化机理
2.1.1 透明质酸酶的底物特异性
除了脊椎动物、水蛭、蜥蜴毒、链霉菌、噬菌体以及石鱼等来源透明质酸酶外,大部分透明质酸酶都具有较为宽泛的底物特异性,可将软骨素等多种糖胺聚糖类物质降解为糖胺寡糖。这可能是由于透明质酸酶是通过软骨素酶的不断进化从而获得了降解透明质酸的能力[1]。此外,每种分类下的透明质酸酶或许具有相同的祖先,因此在进化过程中获得了对底物相类似的作用方式,这在同源性方面有所体现。例如,人源6种透明质酸酶序列和小鼠的6种透明质酸酶相对应序列之间的同源性要大于同一物种内不同透明质酸酶的同源性[3]。
2.1.2 透明质酸酶末端结构对底物特异性的影响
透明质酸酶的生物活性取决于特定的保守结构域(根据CAZy家族划分),通过保留其完整结构域便能够得到具有生物活性的透明质酸酶。例如,SUN等[4]表达了兽疫链球菌透明质酸酶HylB的第128个氨基酸Ala到终止密码子在内的779个氨基酸,得到了具有完整生物活性的透明质酸裂解酶,但其底物特异性尚未明确。此外,决定透明质酸酶底物特异性的结构与具有降解糖胺寡糖活性的结构可能分别取决于透明质酸酶的不同区域。
人源透明质酸酶Hyal-1~Hyal-4以及PH-20的3D模型十分相似,它们主要在C端结构域上有所不同,而催化裂隙和活性位点高度保守。对脊椎动物和类脊椎动物透明质酸酶的空间结构及序列进行分析可知,由连接肽从催化结构域分离的C末端的功能具有最大的可变性和最少的同源性。因此,这些C末端可能为其提供了识别透明质酸以外物质的可能性,通过这种结构上的不同使每一种透明质酸能够实现不同的功能[5]。
细菌来源的透明质酸酶N端结构域具有和真核生物C端结构域类似的组成。据推断,这些结构域能够增加透明质酸酶对透明质酸的亲和力,进而参与到底物的定位当中[5-6]。有研究者仅保留酿脓链球菌噬菌体透明质酸酶的C端结构,发现依旧能够保留其催化活性,但底物特异性发生改变,除了识别透明质酸以外还可以降解硫酸软骨素[7]。
然而单纯通过简单的氨基酸序列同源性的分析比对便将某种酶定义为透明质酸酶的观点应该被摒弃。此前,通过氨基酸序列比对,来自兽疫链球菌的HylZ被认为是一种透明质酸酶,但笔者研究团队通过组建HylZ表达系统证明HylZ是一种β-N-乙酰氨基葡萄糖苷酶,并不具备降解透明质酸的活性[4]。
2.1.3 氨基酸残基对透明质酸酶底物特异性的影响
一些重要的保守残基对透明质酸酶的活性以及特异性有极大影响,例如,Y202、E268和W321在脊椎动物和毒液透明质酸酶的所有序列中都是高度保守的。将E268处的氨基酸残基进行点突变可导致透明质酸酶活性完全丧失[1]。人源透明质酸酶Hyal-4过去被认为是一种透明质酸酶,但目前的研究则认为该酶是一种脊椎动物软骨素酶,对软骨素、硫酸软骨素具有高度特异性[8]。这可能是由于Hyal-4中重要催化残基Y247序列的突变使得其活性改变。因此,通过研究透明质酸酶的保守残基能够更好地揭示其活性位点及其与糖胺聚糖的作用机理。
2.2 常见透明质酸酶催化性质
2.2.1 脊椎动物透明质酸酶
人类基因组中主要以Hyal-1, Hyal-2, Hyal-3, Hyal-4, Hyal-P1(假定蛋白)和PH-20六种透明质酸酶为主。Hyal-2通过糖基磷脂酰肌醇(glycosyl phosphatidyl inositol, GPI)锚定于质膜外侧,可将高分子质量的透明质酸特异性降解成大小约为20 kDa的透明质酸片段[9],随后透明质酸片段进入到质膜内部,经过Hyal-1消化为四糖、六糖。其他物种中也存在该酶,但功能有所差异,例如人Hyal-2与病毒的包膜蛋白结合,并作为功能受体。而小鼠的Hyal-2不与包膜蛋白结合,也不介导任何一种病毒的进入[10]。Hyal-3参与胚胎发生以及干细胞调节且能够在小鼠和人类精子调控中发挥作用,但其催化性能目前尚不明确。
人源透明质酸酶Hyal-P1因为其基因内部存在终止密码子,初步翻译便会流产,因此是一种“伪基因”,但在其他物种可作为具有生理活性的蛋白得到表达。TMEM2是一种具有细胞表面透明质酸活性的跨膜蛋白[11],根据体外研究,该酶在Ca2+存在下将高分子透明质酸裂解成5 kDa的片段[12]。
PH-20是一种具有多种功能的透明质酸酶,其存在于哺乳动物的膜表面以及溶酶体衍生的顶体中,与顶体内膜相连。此外,质膜上该酶仅在中性pH具有活性,顶体中则在酸性和中性pH3.2~9.0下均有活性,这可能是由于不同pH下的酶活性涉及蛋白质中的2个不同结构域。与之类似的是从牛睾丸中提取的牛睾丸透明质酸酶(bovine testis hyaluronidase, BTH)[13],它具有降解多种糖胺聚糖的能力以及一定的转糖苷活性,其发挥转糖苷活性最适pH值为7。常见的脊椎动物透明质酸酶性质如表1所示。
表1 常见脊椎动物透明质酸酶性质
Table 1 Properties of hyaluronidase from common vertebrate

透明质酸酶来源底物特异性产物pH值参考文献Hyal-1血浆、尿液、肝脏、肾脏、脾脏和心脏等HA四糖、六糖3~4[14]Hyal-2肝脏、肾脏、脾、成人大脑、非洲爪蟾、小鼠等HA20 kDa透明质酸4、7.5[14]Hyal-3睾丸、骨髓--3[14]Hyal-4肌肉、胎盘CS、DS--[15]Hyal-P1睾丸和骨髓---[15]PH-20睾丸--4、7.5[16]BTH牛睾丸HA、CS4、CS6、DS透明质酸二糖、透明质酸四糖5、7[13]TMEM2除脑、脾和滑膜外的大多数组织HA5 kDa透明质酸-[12]
注:-代表文献中未提及(下同)。
2.2.2 动物毒液透明质酸酶
动物毒液中普遍存在着透明质酸酶,如蛇、蜘蛛、蝎子、蜜蜂、蜘蛛、蜥蜴、大黄蜂、黄蜂、毛毛虫、石鱼、河豚、狮子鱼、圆锥鱼等,其性质如表2所示。蜂毒透明质酸酶(bee venom hyaluronidase, BVH)主要降解皮肤细胞外基质的透明质酸,也可作用于硫酸软骨素。由于其含量相对于其他毒液透明质酸酶较高,故对该酶的研究较为深入,目前BVH的晶体结构以及酶学特性已被阐明[17],其序列与人睾丸透明质酸酶PH-20类似,因此也被归类为糖苷水解酶家族56。不同动物毒液透明质酸酶对底物的专一性并不相同。例如,蛇毒透明质酸酶可降解透明质酸、软骨素、4-硫酸软骨素、6-硫酸软骨素,生成多种低聚糖,而蜥蜴毒透明质酸酶仅可特异性降解透明质酸。
表2 动物毒液来源透明质酸酶的性质
Table 2 Properties of hyaluronidase derived from animal venom
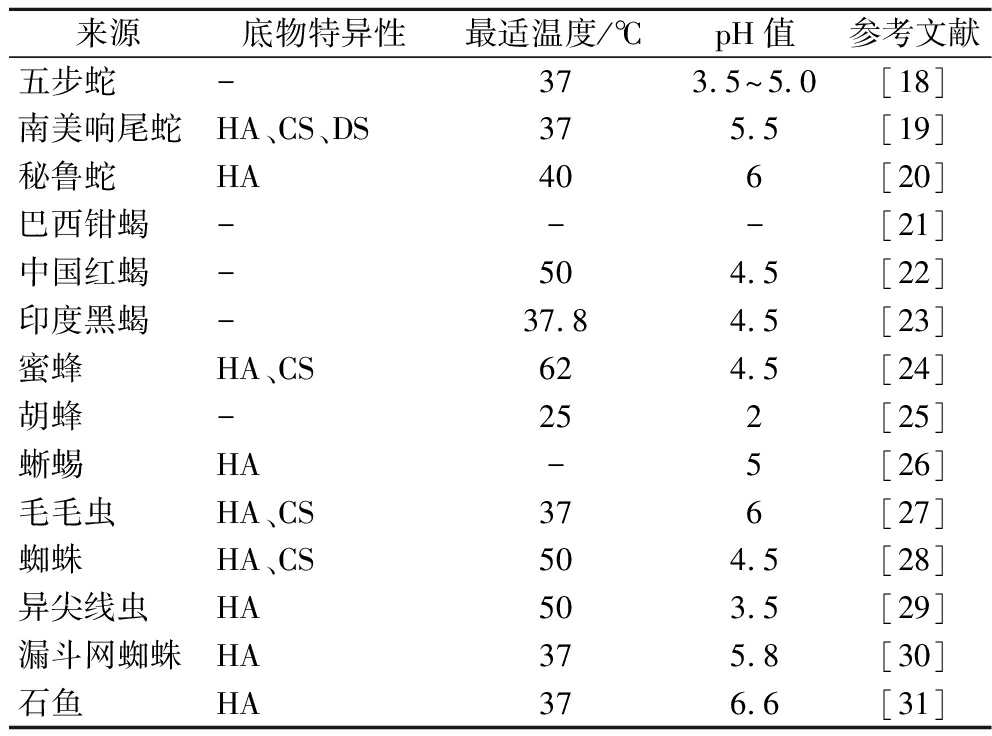
来源底物特异性最适温度/℃pH值参考文献五步蛇-373.5~5.0[18]南美响尾蛇HA、CS、DS375.5[19]秘鲁蛇HA406[20]巴西钳蝎---[21]中国红蝎-504.5[22]印度黑蝎-37.84.5[23]蜜蜂HA、CS624.5[24]胡蜂-252[25]蜥蜴HA-5[26]毛毛虫HA、CS376[27]蜘蛛HA、CS504.5[28]异尖线虫HA503.5[29]漏斗网蜘蛛HA375.8[30]石鱼HA376.6[31]
2.2.3 水蛭透明质酸酶
水蛭来源的透明质酸酶具有高度专一性,仅能作用于透明质酸,对硫酸软骨素、几丁质和肝素具有惰性,且没有转糖苷作用。其最适温度为45 ℃,并观察到对金属离子(Mn2+、Cu2+、Fe3+)的敏感性,最佳pH值为6.5[32]。目前,有研究表明水蛭透明质酸酶具有假定的乙酰肝素酶活性,但尚未发现裂解肝素或硫酸乙酰肝素的能力[33]。由于水蛭透明质酸酶不会带来任何动物交叉感染的风险且酶解得到的产物分子质量范围较窄。因此,重组水蛭透明质酸酶对于临床医学治疗(如外科手术、眼科和内科治疗)以及工业化规模生产分子质量集中的透明质酸寡糖都具有重大意义[34]。
2.2.4 微生物来源透明质酸酶
细菌来源的透明质酸酶在革兰氏阳性细菌中普遍存在,并可作为毒力因子在细菌侵染机制中发挥作用。致病菌利用透明质酸酶降解宿主基质中高分子质量的透明质酸从而突破宿主的防御甚至直接接触细胞组织,并利用产生的寡糖片段诱发炎症反应创造适宜自身生长繁殖的环境[35]。虽然部分革兰氏阴性细菌也能分泌透明质酸裂解酶,但在致病机制中并无作用。微生物来源的透明质酸酶可以通过肽聚糖锚定物锚定到细胞壁上也可以作为胞外酶以游离的形式存在。在通常情况下,它们是周质酶,在周质区域将透明质酸片段降解为不饱和低聚糖。
与透明质酸水解酶通过经典保留机制、底物辅助保留机制、亲核试剂替代保留机制、NAD+依赖保留机制降解糖胺聚糖不同,细菌来源的透明质酸裂解酶通过β-消除反应来生成不饱和糖胺寡糖。其详细催化机制可参考JEDRZEJAS[36]提出的质子接受和捐赠机制。此外在噬菌体、酵母和微霉菌中均检测到透明质酸酶活性(表3)。
表3 微生物来源透明质酸酶性质
Table 3 Properties of hyaluronidase from microorganism
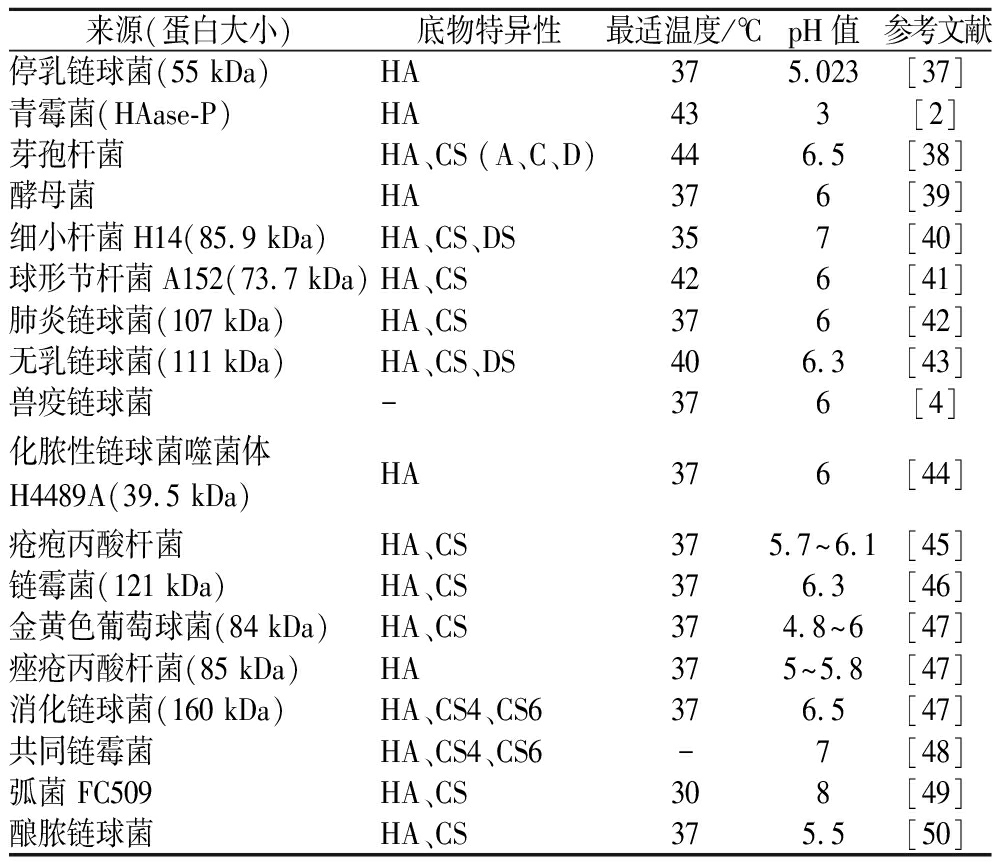
来源(蛋白大小)底物特异性最适温度/℃pH值参考文献停乳链球菌(55 kDa)HA375.023[37]青霉菌(HAase-P)HA433[2]芽孢杆菌HA、CS (A、C、D)446.5[38]酵母菌HA376[39]细小杆菌H14(85.9 kDa)HA、CS、DS357[40]球形节杆菌A152(73.7 kDa)HA、CS426[41]肺炎链球菌(107 kDa)HA、CS376[42]无乳链球菌(111 kDa)HA、CS、DS406.3[43]兽疫链球菌-376[4]化脓性链球菌噬菌体H4489A(39.5 kDa)HA376[44]疮疱丙酸杆菌HA、CS375.7~6.1[45]链霉菌(121 kDa)HA、CS376.3[46]金黄色葡萄球菌(84 kDa)HA、CS374.8~6[47]痤疮丙酸杆菌(85 kDa)HA375~5.8[47]消化链球菌(160 kDa)HA、CS4、CS6376.5[47]共同链霉菌HA、CS4、CS6-7[48]弧菌 FC509HA、CS308[49]酿脓链球菌HA、CS375.5[50]
3 透明质酸酶的生物学应用
3.1 透明质酸酶协助药物扩散吸收
透明质酸酶常用于疾病治疗及检测当中,这与其独特的生物活性有关。如前文所述,在微生物中,透明质酸裂解酶是与病原体引起的发病机制以及疾病发展相关联的毒力因子,可降解基质中的透明质酸等多种糖胺聚糖类物质,使得毒性成分更容易在宿主体内扩散。而毒液透明质酸酶能够分解结缔组织,使得组织坏死程度增加,增加了毒素的扩散和动物组织对毒素的吸收。利用这种机理,研究者在体外、体内和临床研究中利用透明质酸酶微针显著的提高了皮肤的给药效果[51]。换言之,我们可以利用这种性质来提高药物扩散以及吸收速度,减少皮下或肌肉注射药物引起的不适,促进组织中多余液体和渗出血液的吸收,并提高局部麻醉的效果。
3.2 透明质酸酶应用于肿瘤治疗
在肿瘤形成、生长及扩散过程当中,总是伴随着透明质酸酶与组织环境中透明质酸的动态变化,通过研究透明质酸酶的表达与癌细胞的关系能够为癌症治疗提供一种新的思路。透明质酸酶对肿瘤的生命活动具有两面性:一方面,在癌细胞扩散阶段,表达透明质酸酶能够解聚周围组织中的透明质酸,促进肿瘤侵袭;另一方面,在癌症增殖和迁移细胞的细胞基质中发现透明质酸富集,这可能是由于透明质酸的富集有利于形成一种胚胎样环境,促进肿瘤细胞迁移并允许肿瘤细胞通过分泌非特异性抑制或抗炎因子或脱落肿瘤特异性抗原(如淋巴细胞介导的细胞溶解)等机制保护癌细胞免受免疫系统的识别攻击[52-53]。因此,在癌症发展的特定阶段利用透明质酸酶降解癌细胞周围的透明质酸既可以抑制肿瘤的扩散还可以促使淋巴细胞接近肿瘤膜,以增强免疫反应[54]。目前,已通过Hyal-1破坏胶质母细胞瘤中HA的稳定,从而加速癌症治疗[55]。
除了通过对透明质酸的降解间接的对癌细胞起到抑制杀伤作用外,在烟草相关肺癌和上呼吸道癌症中Hyal-1和Hyal-2还可以作为肿瘤抑制蛋白直接发挥作用[56]。此外,在肿瘤治疗中可以将肿瘤细胞的透明质酸酶作为抗癌药物作用的靶位点。有研究显示,卵巢癌伴随着Hyal-1、Hyal-4 mRNA转录水平的提高以及Hyal-2以及Hyal-3 mRNA转录水平的下调[57]。而人睾丸透明质酸酶PH-20也在乳腺癌、前列腺癌和喉癌、黑色素瘤、胶质母细胞瘤和结肠癌的细胞系等恶性肿瘤中被检测到[58]。目前,已有利用类黄酮通过靶向透明质酸酶来调节细胞周期阶段并抑制激素难治性前列腺癌细胞的增殖的相关研究[59]。
4 透明质酸酶应用于制备糖胺寡糖
4.1 糖胺寡糖的生物学功能
相比于高分子质量的糖胺聚糖,糖胺寡糖具有抗氧化、免疫刺激、促进血管生成、癌症以及炎症治疗等多种生理活性,使其在医药领域有着更为重要的作用[60]。在抗氧化衰老方面,郭学平[38]利用芽孢杆菌透明质酸酶制备得到不饱和寡糖,发现其具有降低自由基浓度并进入皮肤表皮层为皮肤提供水分、营养物质从而达到抗衰老和修复紫外线损伤细胞的功效。HA寡聚体(6~20 kDa)是树突状细胞的有效激活剂,可以促进树突细胞成熟,而树突状细胞承担着免疫系统的抗原递呈功能。因此,糖胺寡糖具有良好的免疫刺激功效,可以将其广泛应用于免疫治疗当中。此外,透明质酸寡糖具备诱导新血管的合成的功效。并且相较于小分子质量的透明质酸寡糖(约4 kDa),透明质酸四糖和六糖在促进血管生成效果更加显著。
4.2 透明质酸酶制备糖胺寡糖
相比起化学法和物理法而言,透明质酸酶可更加温和、绿色、高效的降解糖胺聚糖类物质,从而稳定生成特定分子质量大小的糖胺寡糖。因此,通过酶解法制备糖胺寡糖有着广阔的前景。不同物种来源的透明质酸酶所降解产生的糖胺寡糖存在差异,这些差异在主要在于终产物还原性末端的种类和饱和度以及终产物分子质量大小上。我们可以通过控制透明质酸酶的种类以及反应时间从而获取特定的糖胺寡糖类物质,表4为一些常见透明质酸酶所能够降解产生的糖胺寡糖。
表4 糖胺寡糖生物活性研究情况
Table 4 Research on the biological activity of glycosamine oligosaccharides

寡糖类型透明质酸酶来源应用文献透明质酸不饱和二糖芽孢杆菌透明质酸酶抗氧化、抑制皮肤细胞氧化损伤等[61]透明质酸不饱和六糖、八糖链球菌透明质酸裂解酶伤口愈合、细胞修复[62]透明质酸二糖脊椎动物、毒液动物透明质酸酶抑制膀胱癌细胞增殖、促进内皮细胞的分化,诱导内皮细胞对较大HA片段的血管生成作用作出反应[63]透明质酸四糖脊椎动物、毒液动物透明质酸酶诱导热休克蛋的表达,并具有抗凋亡作用,可抑制高温培养中的细胞死亡[64]透明质酸六糖脊椎动物、毒液动物透明质酸酶作为拮抗剂,干扰正常的牛软骨细胞-细胞-基质相互作用、抑制磷酸肌醇生成途径抑制多种肿瘤细胞的锚定及非依赖性生长[65]透明质酸四六糖水蛭、牛睾丸透明质酸酶免疫增殖、促进血管生成、逆转肿瘤细胞耐药性[66]透明质酸十二糖-促进内皮细胞萌发[67]硫酸软骨素四糖牛睾丸透明质酸酶抗炎、抗氧化[68]
当前对透明质酸酶的研究热点主要集中在能够稳定生产透明质酸四糖和六糖的牛睾丸透明质酸酶BTH以及水蛭来源的透明质酸酶LHyals上。WANG等[69]通过向产HA谷氨酸棒杆菌中添加LHyals 至1 500、3 000或6 000 U/mL的终浓度,将使得谷氨酸棒杆所产的HA平均分子质量分别下降到155、91和53 kDa。这两种酶虽然归属于不同的CAZy家族中,终产物的还原性末端不同,但对BTH以及LHyals降解产生的透明质酸四六糖的功效进行对比,发现这两种来源的透明质酸酶所产生的透明质酸四糖和六糖在相同剂量下对免疫增殖、促血管生成作用和逆转肿瘤细胞耐药性均无显著性效果差异,可见分子质量才是对透明质酸性质的决定性因素。
4.3 透明质酸酶的异源表达
虽然一些透明质酸酶具备优秀的降解能力,但由于其在本身宿主细胞中存在表达量较低、稳定性较差或分离纯化困难等因素难以被应用到工业生产当中去。而微生物作为一种优秀的底盘细胞,常作为不同来源的透明质酸酶重组表达的宿主,被广泛应用。例如,毕赤酵母具有中等糖基化的能力,这对于蛋白质形成正确的空间构象极为重要。CHOWPONGPANG等[70]推测人睾丸透明质酸酶PH-20被推测可以在毕赤酵母中得到成功表达,而不至于糖基化程度过高以至于失去部分功能活性。HUANG等[32]首次构建了LHyals 的毕赤酵母表达系统。而LIAO等[71]使用YSD(毕赤酵母表面展示,yeast surface display)系统来促进LHyals的分离和再利用,在蛋白表达和分泌后,通过细胞壁锚定蛋白固定在毕赤酵母的细胞表面,每个循环可将2 000 kDa的透明质酸降解为1.6 kDa。此外,蝎子毒液的第一个重组透明质酸酶也在毕赤酵母中得到表达[21]。
大肠杆菌作为重组表达的常见宿主,多种透明质酸酶在其细胞内得到成功表达。BAKKE等[2]将来自青霉属透明质酸酶在大肠杆菌中成功表达,完成体外制备低分子质量的透明质酸。化脓链球菌H4489A透明质酸裂解酶以及蜂毒透明质酸酶[72-73]均在大肠杆菌中得到成功表达。而来源于兽疫链球菌中的透明质酸酶裂解酶也完成了在大肠杆菌中高活性重组表达,且在优化后的条件下,分别产生46%和31%的透明质酸二糖和透明质酸四糖[74]。陈怡斐等[75]将兽疫链球菌透明质酸酶成功导入大肠杆菌中,利用培养菌株的粗酶液与HA反应75 min,得到分子质量为10 kDa的透明质酸寡糖。
此外,透明质酸酶还在昆虫细胞、枯草芽孢杆菌、中国仓鼠卵巢细胞胞(Chinese hamster ovary,CHO)、谷氨酸棒杆菌等物种中均进行了异源表达。爱思唯尔公司团队克隆了棕色蜘蛛毒液中的透明质酸酶的新亚型,在杆状病毒-昆虫细胞表达系统中表达并完全纯化,从而引入了一种表达蜘蛛重组毒素的新方法[76]。JIN等[77]将水蛭来源透明质酸酶在枯草芽孢杆菌168中实现异源表达,并可控的得到了分子质量在2.2~1 420 kDa之间的透明质酸。任璐[16]将人透明质酸酶PH-20整合到CHO细胞中完成高酶活表达,通过控制纯化后的重组透明质酸酶与透明质酸的反应时间得到了分子质量为26~100 kDa的透明质酸寡糖。
5 展望与结论
透明质酸酶是一种普遍存在于真核生物、原核生物乃至病毒当中的糖苷酶,其被广泛应用于疾病诊断治疗以及低分子质量透明质酸的生产。然而,大量细菌来源的透明质酸酶为透明质酸外切酶,难以高效降解透明质酸,获得的透明质酸范围较广且会产生许多无法回收利用二糖,极大降低了低分子质量透明质酸的产率,经济效益低。而透明质酸内切酶降解获得的低分子质量透明质酸分子质量范围相对集中,但对底盘细胞的要求较高、适应性较差且缺乏用于高效、精细化表达调控的元件。目前,低分子质量透明质酸的制备手段主要通过向高分子质量透明质酸中添加分离纯化得到的透明质酸酶,其高昂的生产成本以及复杂的工艺进一步制约了透明质酸酶在工业领域上的应用。
如前文所述,许多透明质酸酶在结构功能、酶学性质等方面已经得到了充分解析。然而,如何完成透明质酸酶的精细化表达调控以及发酵条件优化等方面还有很多工作要开展,特别是通过一步发酵法直接获得特定分子质量透明质酸的手段亟待建立。因此,未来关于透明质酸酶的研究应包括:a)筛选酶活性更高的透明质酸内切酶,集中获得特定分子质量的透明质酸;b)挖掘更多有潜力的异源表达宿主,实现外源透明质酸酶的高酶活表达;c)构建高效可控的表达元件文库,完成对透明质酸酶可控表达;d)优化发酵工艺,通过一步发酵法直接获得特定分子质量大小的透明质酸。笔者团队已在透明质酸合成菌株中引入不同来源的透明质酸酶并完成透明质酸酶的可控表达,实现了一步发酵法直接合成特定分子质量透明质酸(文章未发表)。
[1] STERN R, JEDRZEJAS M J.Hyaluronidases:Their genomics, structures, and mechanisms of action[J].Chemical Reviews,2006, 106 (3):818-839.
[2] BAKKE M, KAMEI J I, OBATA A.Identification, characterization, and molecular cloning of a novel hyaluronidase, a member of glycosyl hydrolase family 16, from Penicillium spp[J].FEBS Letters, 2011, 585(1):115-120.
[3] JEDRZEJAS M J, STERN R.Structures of vertebrate hyaluronidases and their unique enzymatic mechanism of hydrolysis[J].Proteins, 2005, 61(2):227-238.
[4] SUN X Q, WANG Z, BI Y L, et al.Genetic and functional characterization of the hyaluronate lyase HylB and the beta-N-acetylglucosaminidase HylZ in Streptococcus zooepidemicus[J].Current Microbiology, 2015, 70(1):35-42.
[5] RIGDEN D J, JEDRZEJAS M J.Genome-based identification of a carbohydrate binding module in Streptococcus pneumoniae hyaluronate lyase[J].Proteins, 2003, 52(2):203-211.
[6] RIGDEN D J, GALPERIN M Y, JEDRZEJAS M J.Analysis of structure and function of putative surface-exposed proteins encoded in the Streptococcus pneumoniae genome:A bioinformatics-based approach to vaccine and drug design[J].Critical Reviews in Biochemistry and Molecular Biology, 2003, 38(2):143-168.
[7] MISHRA P, AKHTAR M S, BHAKUNI V.Unusual structural features of the bacteriophage-associated hyaluronate lyase (hylp2)[J].Journal of Biological Chemistry, 2006, 281(11):7143-7150.
[8] RIGDEN D J, JEDRZEJAS M J.Structures of Streptococcus pneumoniae hyaluronate lyase in complex with chondroitin and chondroitin sulfate disaccharides insights into specificity and mechanism of action[J].Journal of Biological Chemistry, 2003, 278(50):50596-50606.
[9] LEPPERDINGER G, STROBL B, KREIL G.HYAL2, a human gene expressed in many cells, encodes a lysosomal hyaluronidase with a novel type of specificity[J].Journal of Biological Chemistry, 1998, 273(35):22466-22470.
[10] REESE K L, ARAVINDAN R G, GRIFFITHS G S, et al.Acidic hyaluronidase activity is present in mouse sperm and is reduced in the absence of SPAM1:Evidence for a role for hyaluronidase 3 in mouse and human sperm[J].Molecular Reproduction and Development, 2010, 77(9):759-772.
[11] NARITA T, TOBISAWA Y, BOBKOV A, et al.TMEM2 is a bona fide hyaluronidase possessing intrinsic catalytic activity[J] Journal of Biological Chemistry, 2023, 299(9):105120.
[12] YAMAMOTO H, TOBISAWA Y, INUBUSHI T, et al.A mammalian homolog of the zebrafish transmembrane protein 2 (TMEM2) is the long-sought-after cell-surface hyaluronidase[J].Journal of Biological Chemistry,2017, 292(18):7304-7313.
[13] NEHMÉ R, NASREDDINE R, ORLIC L, et al.Kinetic theory of hyaluronan cleavage by bovine testicular hyaluronidase in standard and crowded environments[J].Biochimica et Biophysica Acta (BBA-General Subjects), 2021,1865(3):129837.
[14] KRUPKOVA O, GREUTERT H, BOOS N, et al.Expression and activity of hyaluronidases HYAL-1, HYAL-2 and HYAL-3 in the human intervertebral disc[J].European Spine Journal, 2020, 29(3):605-615.
[15] WEBER G C, BUHREN B A, SCHRUMPF H, et al.Clinical applications of hyaluronidase[J].Advances in Experimental Medicine and Biology, 2019, 1148:255-277.
[16] 任璐. 重组人透明质酸酶在 CHO 表达系统中的表达及纯化研究[D].长春:吉林大学, 2019.REN L.Expression and purification of rhPH-20 in CHO expression system[D].Changchun:Jilin University, 2019.
[17] ![]() -HOUSLEY Z, MIGLIERINI G, SOLDATOVA L, et al.Crystal structure of hyaluronidase, a major allergen of bee venom[J].Structure, 2000, 8(10):1025-1035.
-HOUSLEY Z, MIGLIERINI G, SOLDATOVA L, et al.Crystal structure of hyaluronidase, a major allergen of bee venom[J].Structure, 2000, 8(10):1025-1035.
[18] XU X, WANG X S, XI X T, et al.Purification and partial characterization of hyaluronidase from five pace snake (Agkistrodon acutus) venom[J].Toxicon, 1982, 20(6):973-981.
[19] BORDON K C F, PERINO M G, GIGLIO J R, et al.Isolation, enzymatic characterization and antiedematogenic activity of the first reported rattlesnake hyaluronidase from Crotalus durissus terrificus venom[J].Biochimie, 2012, 94(12):2740-2748.
[20] VIVAS-RUIZ D E, GONZALEZ-KOZLOVA E E, DELGADILLO J, et al.Biochemical and molecular characterization of the hyaluronidase from Bothrops atrox Peruvian snake venom[J].Biochimie, 2019, 162:33-45.
[21] AMORIM F G, BOLDRINI-FRANÇA J, DE CASTRO FIGUEIREDO BORDON K, et al.Heterologous expression of rTsHyal-1:The first recombinant hyaluronidase of scorpion venom produced in Pichia pastoris system[J].Applied Microbiology and Biotechnology, 2018, 102(7):3145-3158.
[22] FENG L, GAO R, GOPALAKRISHNAKONE P.Isolation and characterization of a hyaluronidase from the venom of Chinese red scorpion Buthus martensi[J].Comparative Biochemistry and Physiology Part C:Toxicology &Pharmacology, 2008,148(3):250-257.
[23] MOREY S S, KIRAN K M, GADAG J R.Purification and properties of hyaluronidase from Palamneus gravimanus (Indian black scorpion) venom[J].Toxicon, 2006, 47(2):188-195.
[24] REITINGER S, BOROVIAK T, LASCHOBER G T, et al.High-yield recombinant expression of the extremophile enzyme, bee hyaluronidase in Pichia pastoris[J].Protein Expression and Purification, 2008, 57(2):226-233.
[25] RUNGSA P, JANPAN P, SAENGKUN Y, et al.Heterologous expression and mutagenesis of recombinant Vespa affinis hyaluronidase protein (rVesA2)[J].The Journal of Venomous Animals and Toxins Including Tropical Diseases, 2019, 25:e20190030.
[26] TU A T, HENDON R R.Characterization of lizard venom hyaluronidase and evidence for its action as a spreading factor[J].Comparative Biochemistry and Physiology Part B: Comparative Biochemistry, 1983, 76(2):377-383.
[27] DA C B GOUVEIA A I, DA SILVEIRA R B, NADER H B, et al.Identification and partial characterisation of hyaluronidases in Lonomia obliqua venom[J].Toxicon, 2005, 45(4):403-410.
[28] RASH L D, HODGSON W C.Pharmacology and biochemistry of spider venoms[J].Toxicon, 2002, 40(3):225-254.
[29] HOTEZ P, CAPPELLO M, HAWDON J, et al.Hyaluronidases of the gastrointestinal invasive nematodes Ancylostoma caninum and Anisakis simplex:Possible functions in the pathogenesis of human zoonoses[J].The Journal of Infectious Diseases, 1994, 170(4):918-926.
[30] NAGARAJU S, DEVARAJA S, KEMPARAJU K.Purification and properties of hyaluronidase from Hippasa partita (funnel web spider) venom gland extract[J].Toxicon, 2007, 50(3):383-393.
[31] MADOKORO M, UEDA A, KIRIAKE A, et al.Properties and cDNA cloning of a hyaluronidase from the stonefish Synanceia verrucosa venom[J].Toxicon, 2011, 58(4):285-292.
[32] HUANG H, LIANG Q X, WANG Y, et al.High-level constitutive expression of leech hyaluronidase with combined strategies in recombinant Pichia pastoris[J].Applied Microbiology and Biotechnology, 2020, 104(4):1621-1632.
[33] LEVY-ADAM F, MIAO H Q, HEINRIKSON R L, et al.Heterodimer formation is essential for heparanase enzymatic activity[J].Biochemical and Biophysical Research Communications, 2003, 308(4):885-891.
[34] JIN P, KANG Z, ZHANG N, et al.High-yield novel leech hyaluronidase to expedite the preparation of specific hyaluronan oligomers[J].Scientific Reports, 2014, 4:4471.
[35] HYNES W L, WALTON S L.Hyaluronidases of gram-positive bacteria[J].FEMS Microbiology Letters, 2000, 183(2):201-207.
[36] JEDRZEJAS M J.Unveiling molecular mechanisms of pneumococcal surface protein A interactions with antibodies and lactoferrin[J].Clinica Chimica Acta; International Journal of Clinical Chemistry, 2006, 367(1-2):1-10.
[37] STING R, SCHAUFUSS P, BLOBEL H.Isolation and characterization of hyaluronidases from Streptococcus dysgalactiae, S.zooepidemicus and S.equi[J].Zentralblatt Für Bakteriologie, 1990, 272(3):276-282.
[38] 郭学平. 芽孢杆菌 Bacillus sp.A50 透明质酸酶的研究[D].济南:山东大学, 2014.GUO X P.Study on the hyaluronidase from Bacillus sp.A50[D].Jinan:Shandong University, 2014.
[39] SMIRNOU D, ![]() M, KULH
M, KULH NEK J, et al.Characterization of hyaluronan-degrading enzymes from yeasts[J].Applied Biochemistry and Biotechnology, 2015, 177(3):700-712.
NEK J, et al.Characterization of hyaluronan-degrading enzymes from yeasts[J].Applied Biochemistry and Biotechnology, 2015, 177(3):700-712.
[40] SUN J H, HAN X, SONG G R, et al.Cloning, expression, and characterization of a new glycosaminoglycan lyase from Microbacterium sp.H14[J].Marine Drugs, 2019, 17(12):681.
[41] ZHU C L, ZHANG J L, LI L Y, et al.Purification and characterization of hyaluronate lyase from Arthrobacter globiformis A152[J].Applied Biochemistry and Biotechnology, 2017, 182(1):216-228.
[42] JEDRZEJAS M J, MEWBOURNE R B, CHANTALAT L, et al.Expression and purification of Streptococcus pneumoniae hyaluronate lyase from Escherichia coli[J].Protein Expression and Purification, 1998, 13(1):83-89.
[43] JEDRZEJAS M J, CHANTALAT L.Structural studies of Streptococcus agalactiae hyaluronate lyase[J].Acta Crystallographica Section D-Biological crystallography, 2000, 56(Pt 4):460-463.
[44] EL-SAFORY N S, LEE G C, LEE C K.Characterization of hyaluronate lyase from Streptococcus pyogenes bacteriophage H4489A[J].Carbohydrate Polymers, 2011, 84(3):1182-1191.
[45] INGHAM E, HOLLAND K T, GOWLAND G, et al.Purification and partial characterization of hyaluronate lyase (EC 4.2.2.1) from Propionibacterium acnes[J].Journal of General Microbiology, 1979, 115(2):411-418.
[46] TAO L, SONG F, XU N Y, et al.New insights into the action of bacterial chondroitinase AC I and hyaluronidase on hyaluronic acid[J].Carbohydrate Polymers, 2017,158:85-92.
[47] GIRISH K S, KEMPARAJU K.The magic glue hyaluronan and its eraser hyaluronidase:A biological overview[J].Life Sciences, 2007, 80(21):1921-1943.
[48] MESSINA L, GAVIRA J A, PERNAGALLO S, et al.Identification and characterization of a bacterial hyaluronidase and its production in recombinant form[J].FEBS Letters, 2016, 590(14):2180-2189.
[49] HAN W J, WANG W S, ZHAO M, et al.A novel eliminase from a marine bacterium that degrades hyaluronan and chondroitin sulfate[J].Journal of Biological Chemistry, 2014, 289(40):27886-27898.
[50] HYNES W L, DIXON A R, WALTON S L, et al.The extracellular hyaluronidase gene (hylA) of Streptococcus pyogenes[J].FEMS Microbiology Letters, 2000,184(1):109-112.
[51] HU W S, PENG T T, HUANG Y, et al.Hyaluronidase-powered microneedles for significantly enhanced transdermal delivery efficiency[J].Journal of Controlled Release, 2023,353:380-390.
[52] KNUDSON W.Tumor-associated hyaluronan. Providing an extracellular matrix that facilitates invasion[J].American Journal of Pathology, 1996,148(6):1721-1726.
[53] WANG S B, LI Y X, XU C N, et al.An oncolytic vaccinia virus encoding hyaluronidase reshapes the extracellular matrix to enhance cancer chemotherapy and immunotherapy[J].Journal for Immunotherapy of Cancer, 2024,12(3):e008431.
[54] MCBRIDE W H, BARD J B.Hyaluronidase-sensitive halos around adherent cells.Their role in blocking lymphocyte-mediated cytolysis[J].The Journal of Experimental Medicine, 1979,149(2):507-515.
[55] NEVES E R, ANAND A, MUELLER J, et al.Targeting glioblastoma tumor hyaluronan to enhance therapeutic interventions that regulate metabolic cell properties[J].bioRxiv, 2024:2024.01.05.574065.
[56] FROST G I, MOHAPATRA G, WONG T M, et al.HYAL1LUCA-1, a candidate tumor suppressor gene on chromosome 3p21.3, is inactivated in head and neck squamous cell carcinomas by aberrant splicing of pre-mRNA[J].Oncogene, 2000,19(7):870-877.
[57] RIECKS J, PARNIGONI A, GYÖRFFY B, et al.The hyaluronan-related genes HAS2, HYAL1-4, PH20 and HYALP1 are associated with prognosis, cell viability and spheroid formation capacity in ovarian cancer[J].Journal of Cancer Research and Clinical Oncology, 2022,148(12):3399-3419.
[58] STERN R.Hyaluronan metabolism:A major paradox in cancer biology[J].Pathologie Biologie, 2005, 53(7):372-382.
[59] SINGH S, MAURYA A K, MEENA A, et al.Narirutin.A flavonoid found in Citrus fruits modulates cell cycle phases and inhibits the proliferation of hormone-refractory prostate cancer cells by targeting hyaluronidase[J].Food and Chemical Toxicology, 2023,174:113638.
[60] CARTON F, MALATESTA M.Nanotechnological research for regenerative medicine:The role of hyaluronic acid[J].International Journal of Molecular Sciences, 2024, 25(7):3975.
[61] 李强. 不饱和透明质酸二糖的制备和活性研究[D].济南:山东大学, 2013.LI Q.Study on the preparation and biological activities of 4,5-unsaturated hyaluronate disaccharide[D].Jinan:Shandong University, 2013.
[62] TOLG C, TELMER P, TURLEY E.Specific sizes of hyaluronan oligosaccharides stimulate fibroblast migration and excisional wound repair[J].PLoS One, 2014, 9(2):e88479.
[63] JORDAN A R, LOKESHWAR S D, LOPEZ L E, et al.Antitumor activity of sulfated hyaluronic acid fragments in pre-clinical models of bladder cancer[J].Oncotarget, 2017, 8(15):24262-24274.
[64] XU H P, ITO T, TAWADA A, et al.Effect of hyaluronan oligosaccharides on the expression of heat shock protein 72[J].Journal of Biological Chemistry, 2002, 277(19):17308-17314.
[65] OHNO S, IM H J, KNUDSON C B, et al.Hyaluronan oligosaccharide-induced activation of transcription factors in bovine articular chondrocytes[J].Arthritis and Rheumatism, 2005, 52(3):800-809.
[66] 吕梦娴. 透明质酸四糖和六糖的制备及生物活性研究[D].无锡:江南大学, 2016.LYU M X.Study on the preparation and biological activities of hyaluronan tetrasaccharide and hexasaccharide[D].Wuxi:Jiangnan University, 2016.
[67] TAKAHASHI Y, LI L L, KAMIRYO M, et al.Hyaluronan fragments induce endothelial cell differentiation in a CD44- and CXCL1/GRO1-dependent manner[J].Journal of Biological Chemistry,2005,280(25):24195-24204.
[68] 张莲. 硫酸软骨素寡糖的制备及其体外抗氧化活性研究[D].无锡:江南大学, 2012.ZHANG L.Preparation and antioxidant activity in vitro of chondroitin sulfate oligosaccharides[D].Wuxi:Jiangnan University, 2012.
[69] WANG Y, HU L T, HUANG H, et al.Eliminating the capsule-like layer to promote glucose uptake for hyaluronan production by engineered Corynebacterium glutamicum[J].Nature Communications, 2020,11(1):3120.
[70] CHOWPONGPANG S, SHIN H S, KIM E K.Cloning and characterization of the bovine testicular PH-20 hyaluronidase core domain[J].Biotechnology Letters, 2004, 26(15):1247-1252.
[71] LIAO L Z, HUANG H, WANG Y, et al. Yeast surface display of leech hyaluronidase for the industrial production of hyaluronic acid oligosaccharides[J].Engineering Microbiology,2023,3(4):100086.
[72] YANG P F, LEE C K.Purification of recombinant hyaluronan lyase of Streptococcus pyogenes bacteriophage H4489A expressed in Escherichia coli and its application for the specific determination of hyaluronan concentration[J].Carbohydrate Polymers, 2006, 65(2):159-164.
[73] SCHWAIGHOFER A, ABLASSER S, LUX L et al.Production of active recombinant hyaluronidase inclusion bodies from Apis mellifera in E.coli BL21(DE3) and characterization by FT-IR Spectroscopy[J].International Journal of Molecular Sciences, 2020, 21(11):3881.
[74] GUO X P, LIU F, ZHU X Q, et al.Expression of a novel hyaluronidase from Streptococcus zooepidemicus in Escherichia coli and its application for the preparation of HA oligosaccharides[J].Carbohydrate Polymers, 2009, 77(2):254-260.
[75] 陈怡斐, 李茜茜, 荣绍丰, 等.重组兽疫链球菌透明质酸酶在大肠杆菌中的表达及其酶活力[J].中国医药工业杂志, 2015, 46(11):1173-1177.CHEN Y F, LI Q Q, RONG S F, et al.Expression of recombinant hyaluronidase from Streptococcus zooepidemicus in Escherichia coli and its enzyme activity[J].Chinese Journal of Pharmaceuticals, 2015, 46(11):1173-1177.
[76] DE-BONA E, CHAVES-MOREIRA D, BATISTA T B D, et al.Production of a novel recombinant brown spider hyaluronidase in baculovirus-infected insect cells[J].Enzyme and Microbial Technology, 2021, 146:109759.
[77] JIN P, KANG Z, YUAN P H, et al.Production of specific-molecular-weight hyaluronan by metabolically engineered Bacillus subtilis 168[J].Metabolic Engineering, 2016, 35:21-30.