D-阿洛酮糖是一种天然存在的稀有糖,它的甜度约为蔗糖的70%,加工特性等也与蔗糖相近,被评为最具潜力的蔗糖替代品。此外,D-阿洛酮糖对健康有益,例如调节血糖[1]、保护心脏[2]、降低脂质水平[3]和具有抗炎活性等[4]。
目前,D-阿洛酮糖主要通过化学合成和生物合成2种方法来合成。然而,化学合成方法有一些缺点,如催化剂负载高,使用有毒试剂,产生多种副产物,以及需要复杂的保护和去保护步骤[5-7]。而生物合成方法具有反应条件温和、不需要有毒有害试剂、生态友好、选择性好等优点,IZUMORI[8]最初提出利用酮糖3-差向异构酶催化酮糖C3位置实现异构化。然而,酮糖3-差向异构酶存在因热稳定性差而导致转化效率低的问题。
为了提高酮糖3-差向异构酶的热稳定性,陆续报道了多种蛋白质工程改造策略以实现热稳定性提升。例如,通过脯氨酸取代,来自鲍氏梭菌(Clostridium bolteae)的D-阿洛酮糖3-差向异构酶(D-allulose 3-epimerase,DAEase)在55 ℃条件下,外源添加Co2+的半衰期延长至58 min[9]。通过引入二硫键,在70 ℃条件下外源添加Mg2+,刚果盐厌氧菌(Halanaerobium congolense)来源的DAEase的半衰期达到5.2 h,其Tm值从73.5 ℃增加到79.5 ℃10]。利用折叠自由能预测,外源添加Co2+条件下,解纤维素梭菌(Clostridium cellulolyticum)来源的DAEase突变体的Tm值从67.2 ℃提高到79.5 ℃[11]。此外,优化界面相互作用可有效提高菊苣假单胞菌(Pseudomonas cichorii)来源的DAEase的热稳定性[12]。同样,多尔氏菌属(Dorea sp.)的DAEase在50 ℃下,外源添加Co2+的半衰期为20.5 h,Tm值为74.2 ℃[13]。通过优化栖污泥嗜热梭菌(Thermoclostridium caenicola)来源的DAEase的界面区域和重构底物口袋,外源添加Co2+条件下,突变体SaDAE_S191D/M193E/S213C的T5060值从67.0 ℃提高到74.2 ℃[14]。来源于波罗的海红小梨形菌(Rhodopirellula baltica)DAEase通过定向突变,获得突变体L144F在Mn2+存在下,半衰期从52.3 min提高到102.7 min,Tm值从45.7 ℃提高到68.3 ℃[15]。然而,上述工程酶合成D-阿洛酮糖的效率仍不能满足工业生产的要求。
本研究从象牙色克罗彭施泰特氏菌(Kroppenstedtia eburnean)中提取的D-塔格糖3-差向异构酶(D-tagatose 3-epimerase),并命名为KeDt3e,在无外源添加金属离子的情况下仍表现出优异的催化效率。然而,KeDt3e的热稳定性阻碍了D-阿洛酮糖的工业化生产。在此基础上,通过引入二硫键和属性嫁接2种组合策略,提高了KeDt3e的热稳定性,获得了最佳突变体M03。在不需要外源添加金属离子稳定的条件下,最优突变体M03的Tm值从52.7 ℃升高到69.4 ℃;55 ℃时半衰期延长至254.3 min,显著提高54.1倍。通过机制解析发现,在蛋白表面引入二硫键稳定了整体蛋白的稳定性,优化多聚体界面之间的相互作用增强了蛋白的热稳定性。本研究旨在通过理性设计获得一种热稳定性增强的差向异构酶,并对热稳定提高的机制进行解析,为工业化生产D-阿洛酮糖提供实验基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株、质粒与引物
实验所用重组菌株及引物见表1和表2。
表1 本研究所用的菌株和质粒
Table 1 Host strains and plasmids used in this study

质粒和菌株相关特性pET-28apBR322 ori,PT7,KanRpET-28a-1pBR322 ori,PT7,KanR,KeDt3epET-28a-2pBR322 ori,PT7,KanR,KeDt3eK2C/D31CpET-28a-3pBR322 ori,PT7,KanR,KeDt3eH193C/D196CpET-28a-4pBR322 ori,PT7,KanR,KeDt3eA267C/Q271CpET-28a-5pBR322 ori,PT7,KanR,KeDt3eV217C/S279CpET-28a-6pBR322 ori,PT7,KanR,KeDt3eV25C/L30CpET-28a-7pBR322 ori,PT7,KanR,KeDt3eL48C/Q98CpET-28a-8pBR322 ori,PT7,KanR,KeDt3eA195C/A231CpET-28a-9pBR322 ori,PT7,KanR,KeDt3eI198C/A231CpET-28a-10pBR322 ori,PT7,KanR,KeDt3eA37C/I66CpET-28a-11pBR322 ori,PT7,KanR,KeDt3eT107C/W112CpET-28a-12pBR322 ori,PT7,KanR,KeDt3eF155YpET-28a-13pBR322 ori,PT7,KanR,KeDt3eI194FpET-28a-14pBR322 ori,PT7,KanR,KeDt3eA213CpET-28a-15pBR322 ori,PT7,KanR,KeDt3e H193C/D196C/F155YpET-28a-16pBR322 ori,PT7,KanR,KeDt3eH193C/D196C/I194FpET-28a-17pBR322 ori,PT7,KanR,KeDt3eF155Y/I194FpET-28a-18pBR322 ori,PT7,KanR,KeDt3e H193C/D196C/F155Y/I194FE.coli BL21(DE3)for enzyme expression and purificationE.coli 001E.coli BL21(DE3)/(pET-KeDt3e)E.coli 002E.coli BL21(DE3)/(pET-KeDt3eK2C/D31C)E.coli 003E.coli BL21(DE3)/(pET-KeDt3eH193C/D196C)E.coli 004E.coli BL21(DE3)/(pET-KeDt3eA267C/Q271C)E.coli 005E.coli BL21(DE3)/(pET-KeDt3eV217C/S279C)E.coli 006E.coli BL21(DE3)/(pET-KeDt3eV25C/L30C)E.coli 007E.coli BL21(DE3)/(pET-KeDt3eL48C/Q98C)E.coli 008E.coli BL21(DE3)/(pET-KeDt3eA195C/A231C)E.coli 009E.coli BL21(DE3)/(pET-KeDt3eI198C/A231C)E.coli 010E.coli BL21(DE3)/(pET- KeDt3eA37C/I66C)E.coli 011E.coli BL21(DE3)/(pET- KeDt3eT107C/W112C)E.coli 012E.coli BL21(DE3)/(pET- KeDt3eF155Y)E.coli 013E.coli BL21(DE3)/(pET- KeDt3eI194F)E.coli 014E.coli BL21(DE3)/(pET- KeDt3eA213C)E.coli 015E.coli BL21(DE3)/(pET-KeDt3eH193C/D196C/F155Y)E.coli 016E.coli BL21(DE3)/(pET-KeDt3eH193C/D196C/I194F)E.coli 017E.coli BL21(DE3)/(pET-KeDt3eF155Y/I194F)E.coli 018E.coli BL21(DE3)/(pET-KeDt3eH193C/D196C/F155Y/I194F)
表2 本研究所用引物
Table 2 Primers used in this study

突变体引物(5′-3′)H193C/D196C-TGAGGAAGATTGTATTGCATGTGCTATCAGATACACCGGTGATCACH193C/D196C-BTCTGATAGCACATGCAATACAATCTTCCTCAATGTTCATGTGAAAGGTK2C-TGGATCCATGTGTTATGGAGTATATTTTGCTTACTGGGAGGACK2C-BTACTCCATAACACATGGATCCGCGACCCAT D31C-TGGCCTGTGTATCCTGGAAGTCGCCGCAD31C-BCAGGATACACAGGCCCAATTTTTTGACCTTGCGA267C/Q271C-TGACCTGAGCGGTAACTGTACCGAGGAATGTCTGGATCGCGAGCTGA267C/Q271C-BCAGCTCGCGATCCAGACATTCCTCGGTACAGTTACCGCTCAGGTCV217C-TCGTAAATGTCCGGGTAAAGGCAGCATGV217C-BACCCGGACATTTACGATTCGCTTCGCCAATATGS279C-TGCAGAATGTCTGGAATTTGTGAAGGCGGCGS279C-BTTCCAGACATTCTGCCAGCTCGCGATCV25C/L30C-TGAGAAATACGTGCGCAAGTGTAAAAAATTGGGCTGTGACATCCTGGAAGV25C/L30C-BCTTCCAGGATGTCACAGCCCAATTTTTTACACTTGCGCACGTATTTCTCL48C-TCAAAAATGTGAGCGTCTGAAGCAGTTGGCGL48C-BACGCTCACATTTTTGTTCCGGCAGGTTCACQ98C-TATGCACTGTGCGGGTATTCATCGTATCGGCQ98C-BACCCGCACAGTGCATAGCGTCCATAACCTTCTTA195C-TCACATTTGTGATGCTATCAGATACACCGGTGATA195C-BAGCATCACAAATGTGATCTTCCTCAATGTTCATGTGAAAA231C-TGGTCAATGTTTGAAGGACATTCGTTACGACGGTA231C-BATTCGCACAGCCAATATGGAGCTGACCCAGI198C-TGATGCTTGTAGATACACCGGTGATCACCTGI198C-BGTATCTACAAGCATCTGCAATGTGATCTTCCTCA37C-TGTCGCCTGTCTGGGCCTTGTGAACCTGA37C-BGCCCAGACAGGCGACTTCCAGGATGTCCAGI66C-TGCAGGCTGTGGGTTACCGAAAGAGTATGACGTTAGCI66C-BTAACCCACAGCCTGCGGTCAGGATGATT107C/W112C-TCATCGTATCGGCGGTTGTGTTTATAGCTATTGTCCGGTGGACTACTCT107C/W112C-BGAGTAGTCCACCGGACAATAGCTATAAACACAACCGCCGATACGATGF155Y-TCTTAACCGTTATGAGCAGTTCTTGCTGAACGACF155Y-BGAACTGCTCATAACGGTTAAGCGTTTCGATCAACAGI194F-TGAAGATCACTTTGCAGATGCTATCAGATACACCGGTI194F-BAGCATCTGCAAAGTGATCTTCCTCAATGTTCATGTGAAAGGTA213C-TAAAGGCGAATGTAATCGTAAAGTGCCGGGTAAAGGCA213C-BTTTACGATTACATTCGCCAATATGGAGCTGACCH193C/D196C/I194F-TGAGGAAGATTGTTTTGCATGTGCTATCAGATACACCGGTGATCACH193C/D196C/I194F-BTCTGATAGCACATGCAAAACAATCTTCCTCAATGTTCATGTGAAAGGT
1.1.2 实验试剂
商用试剂、标准品和溶剂均无需进一步纯化。D-阿洛酮糖标准品、EDTA-Ca盐,Sigma-Aldrich(上海)贸易有限公司;D-果糖,上海阿拉丁生化科技股份有限公司;蛋白胨、胰蛋白胨、酵母粉,北京百灵威科技有限公司;氯化钴、氯化钠、磷酸二氢钾、磷酸氢二钾、Tris-HCl、咪唑,上海迈瑞尔化学技术有限公司;甘油,TCI(上海)化成工业发展有限公司;异丙基β-D-1-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG)、抗生素,生工生物工程股份公司。所有的化学品和试剂都经过提纯,并在商业上获得。分子生物学试剂如Prime STAR和消化酶试剂,宝生物工程(大连)有限公司;PCR纯化试剂盒,上海碧云天生物技术有限公司;KOD-Plus-Neo,东洋纺(上海)生物科技有限公司。
1.1.3 培养基
LB液体培养基(g/L):酵母粉5,蛋白胨10,NaCl 5,用于质粒构建和种子液活化培养。
LB固体培养基:在LB液体培养基的基础上添加20 g/L琼脂粉,用于单菌落培养。
TB液体培养基(g/L):酵母粉24,胰蛋白胨12,甘油4,磷酸二氢钾2.31,三水磷酸氢二钾16.42,用于重组菌株的培养以实现蛋白表达。
1.2 仪器与设备
HDPN-II-88型恒温培养箱,上海跃进器械厂;GR60DR型灭菌锅,浙江浙科仪器设备有限公司;ZQZY-AF8V型摇床,上海知楚仪器有限公司;Alliance 高效液相色谱系统和Nano-DSC差示扫描量热仪,美国沃特世公司;SW-CJ-1FD型超净工作台,苏州安泰空气技术有限公司;C100型PCR仪,美国BioRad公司;UH-12型高压细胞破碎仪,上海永联生物科技有限公司;5804R型台式高速离心机,德国Eppendorf公司。
1.3 实验方法
1.3.1 蛋白的表达和纯化
在添加卡那霉素(50 μg/mL)的LB培养基中,置于37 ℃,转速为200 r/min的摇床活化含有KeDt3e和突变体的重组大肠杆菌菌株,活化8 h后以2%的接种量转接至含有卡那霉素(50 μg/mL)的200 mL TB培养基中,在37 ℃,200 r/min条件下培养,当菌体浓度培养至OD600值达到0.6~0.8时,加入终浓度0.4 mmol/L IPTG;在16 ℃条件下诱导酶表达18~20 h。
将培养的菌体在4 ℃条件下,以6 000 r/min转速离心10 min收集菌体,重悬于缓冲液A(25 mmol/L Tris-HCl,pH 7.4,150 mmol/L NaCl,20 mmol/L咪唑)。经高压破碎后在10 000 r/min下离心0.5 h收集上清液。随后使用镍柱进行纯化,先使用缓冲液A洗去杂蛋白,再用缓冲液B(25 mmol/L Tris-HCl,pH 7.4,150 mmol/L NaCl,500 mmol/L咪唑)洗脱目的蛋白,最后用洗脱缓冲液C(25 mmol/L Tris-HCl,pH 7.4,150 mmol/L NaCl)将蛋白浓缩置换进行活性测定。
1.3.2 突变文库的构建
采用KOD-Plus-Neo(TOYOBO)全质粒PCR进行突变实验。用消化酶Dpn I对所得PCR产物进行消化处理以消除模板质粒。进一步将10.0 μL的消化产物转化到大肠杆菌BL21(DE3)细胞中,进行表达筛选或DNA测序(GENEWIZ)。
1.3.3 分析检测方法
动力学稳定性-半衰期检测:在1 mL反应体系中,将20 g/L湿菌体置于50 mmol/L Tris-HCI(pH 7.5)缓冲液中,在同一温度下孵育不同时间间隔,添加50 g/L D-果糖进行催化反应,反应时间10 min,反应结束后煮沸10 min终止反应,经10 000 r/min离心10 min后取上清液,测定湿菌体的剩余活性。
热力学稳定性-Tm值测定:称取4~5 g湿菌体,用25 mmol/L Tris-HCI(pH 7.4)缓冲液重悬后,进行高压破碎,经10 000 r/min离心30 min后取上清液,通过镍柱纯化得到纯度较高的目的蛋白,浓缩至质量浓度为4~4.5 mg/mL,使用差示扫描量热仪以1 ℃/min的升温速度,测定酶的熔解温度(Tm)。
初筛检测:将收集到的突变体菌体重悬于pH 7.5的Tris-HCl缓冲液中,置于55 ℃孵育10 min,添加50 g/L D-果糖在55 ℃条件下进行催化反应,反应时间为10 min,反应结束后煮沸10 min终止反应,反应液经3 700 r/min高速离心10 min,取上清液进行D-阿洛酮糖产量检测。
D-果糖和D-阿洛酮糖的检测:D-果糖和D-阿洛酮糖的分析采用Waters e2141 HPLC,采用Sugar-Pak I色谱柱(10 μm,6.5 mm×300 mm),流动相为50 mg/L EDTA-Ca盐,流速0.3 mL/min,柱温60 ℃,检测器温度30 ℃。
1.3.4 分子动力学模拟
模型构建:从本实验室获得X-射线衍射晶体数据,获得KeDt3e的晶体结构(PDB ID:8XIV),基于H++的pKa计算结果和蛋白局部氢键网络,确定带电残基的质子化状态。组氨酸存在3种状态,若组氨酸侧链咪唑环邻位氮质子化,则为HID;若组氨酸侧链咪唑环间位质子化,则为HIE;若组氨酸侧链咪唑环邻位和间位都质子化,则为HIP。根据可视化软件分析His58、His102、His128和His204侧链咪唑环邻位质子化,因此被指定为HID;His97、His186和His209侧链咪唑环间位质子化,因此被指定为HIE。除Glu150被指定为质子化,Asp和其余Glu残基被去质子化,Lys和Arg残基被质子化,最终获得野生型KeDt3e分子动力学的初始结构。以野生型KeDt3e结构为模板通过SWISS-MODEL同源建模获得突变体M03的蛋白结构。
MD模拟:所有MD模拟均使用Amber 16软件包进行[16]。采用Gaussian 16在B3LYP/6-31G(d)水平上对D-果糖的结构进行优化,并采用HF/6-31G(d)计算电荷,由Amber 16软件中的Antechamber模块实现D-果糖部分电荷的约束静电势协议拟合[17-18]。D-果糖的力场参数采用广义力场2.0(gaff2)[19],而标准的Amber14SB力场用于描述蛋白质。每个体系最初用Na+离子中和整个体系的电荷,并在八面体盒子中用显示TIP3P水溶剂化模型[20],每侧缓冲距离为10 Å。接着,进行一系列优化,在此过程中,蛋白质主链重原子的约束逐渐被释放[力常数为10、2、0.1 和0 kcal/(mol·Å2)],并在50 ps内从0 K缓慢加热到338 K,对蛋白质主链重原子施加10 kcal/(mol·Å2)的约束。然后,在恒温常压下进行10 ns MD模拟,以5 kcal/(mol-1 Å2)的力常数抑制蛋白质主链重原子。最后,通过Berendsen恒温法将温度控制在338 K,在恒温常压条件下进行了100 ns的非限制MD模拟。采用粒子网格Ewald方法处理远程静电相互作用[21],对粒子网格Ewald相互作用和范德华相互作用均使用12 Å截断。采用周期性的边界条件通过SHAKE算法[21]计算氢2 fs的时间步长。每2 ps存储一次原子位置,以便进一步分析。检查每个系统的稳定性(结构、能量和温度波动)和收敛性[均方根偏差(root-mean-square deviation, RMSD)]。
2 结果与分析
2.1 KeDt3e特性分析
KeDt3e由289个氨基酸组成,其分子质量为33.0 kDa。KeDt3e的结构是包括8个α-螺旋和8个β-折叠的TIM桶状结构,4个短α-螺旋α1′、α2′、α6′和α8′分别位于β1、β2、β6和β8之后,D-果糖与Co2+结合在TIM桶的中心(图1)。通过序列比对,活性中心的关键残基E150、D183、H209和E244高度保守(图2),具有催化D-果糖生成D-阿洛酮糖的潜力。

图1 KeDt3e结构分析
Fig.1 Structural analysis of KeDt3e

图2 KeDt3e与其他文献报道酮糖3-差向异构酶的序列比对
Fig.2 Sequence comparison between KeDt3e and ketose 3-epimerase reported in other literatures
有文献报道酮糖3-差向异构酶的热稳定性较差,因此从热力学稳定性以及高温下动力学稳定性对KeDt3e进行了评估,KeDt3e的Tm值为52.7 ℃(图3-a),接着通过半衰期表征了KeDt3e在高温下的动力学稳定性,结果表明(图3-b)在55 ℃条件下的半衰期仅为4.7 min,而在60 ℃和65 ℃条件下半衰期太短而无法拟合,因此可以证明KeDt3e的热稳定性较差,需进行蛋白质工程改造以提高酶的热稳定性。
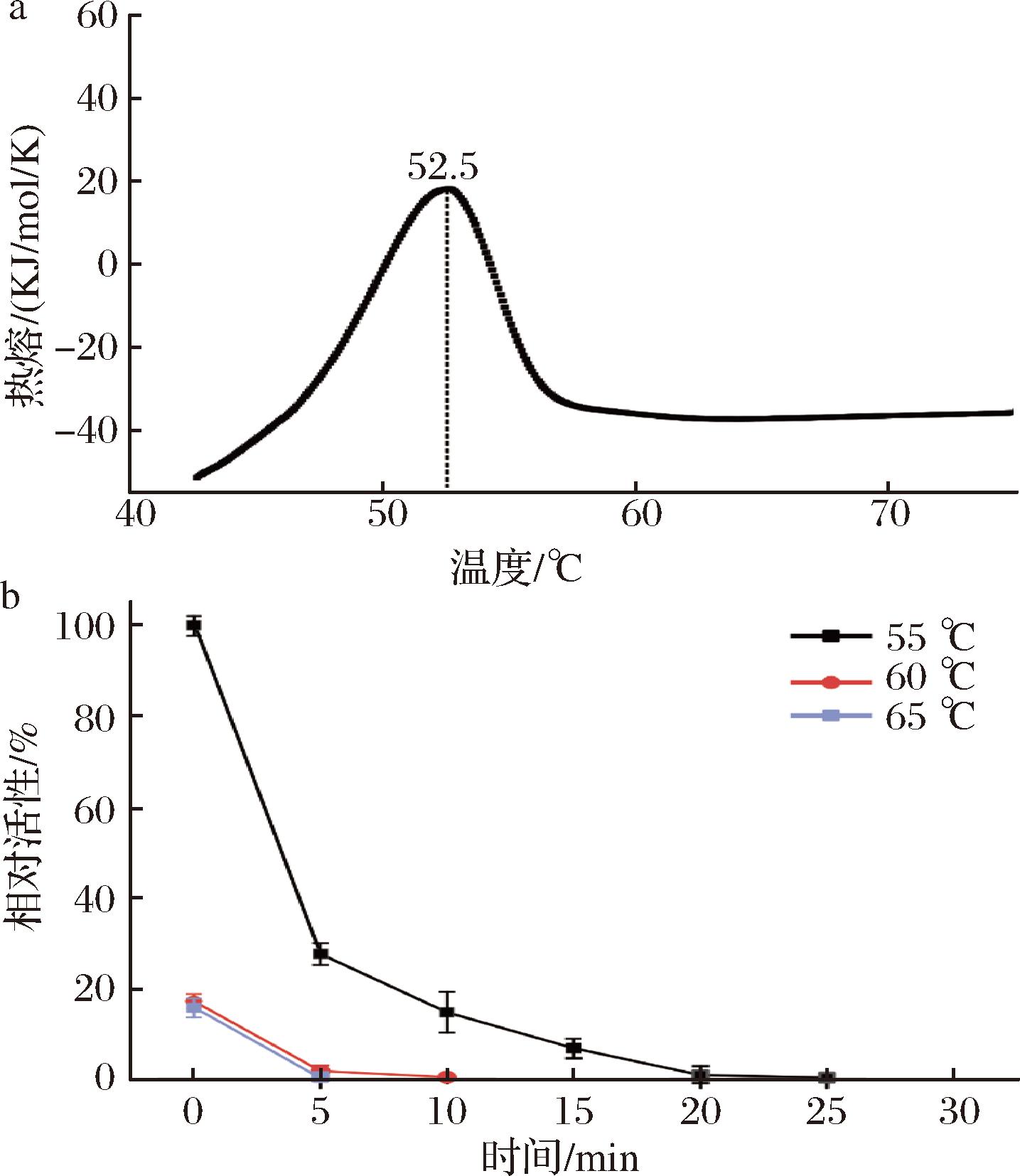
a-KeDt3e的熔解温度;b-KeDt3e在不同温度下的半衰期
图3 KeDt3e的热稳定性表征
Fig.3 Characterization of the thermal stability of KeDt3e
2.2 蛋白质工程改造
引入二硫键是改造热稳定性的通用方法之一。根据MODIP(http://caps.ncbs.res.in/dsdbase/modip.hTml)和cptDomski(http://cptweb.cpt.wayne.edu/)网站预测引入二硫键的位点,即K2C/D31C、H193C/D196C、A267C/Q271C、V217C/S279C、V25C/L30C、L48C/Q98C、A195C/A231C、I198C/A231C、A37C/I66C和T107C/W112C,根据表2引物构建突变文库。第一批构建了双突变体H193C/D196C、A267C/Q271C、V25C/L30C和T107C/W112C以及单突变体K2C、V217C、L48C、A231C和A37C;第二批突变以获得的单突变体为模板进行组合突变,获得双突变体K2C/D31C、V217C/S279C、L48C/Q98C、A195C/A231C、I198C/A231C和A37C/I66C。将上述突变体培养后收集湿菌体,在55 ℃下孵育10 min,进行催化验证,通过HPLC检测反应10 min的D-阿洛酮糖的产量以进行初筛,其中突变体H193C/D196C的产量高于野生型(图4-a);接着准确表征了突变体H193C/D196C的性能,Tm值为52.8 ℃(图4-b);在55 ℃的半衰期为90.4 min,是野生型(4.7 min)的19.2倍(图4-c)。
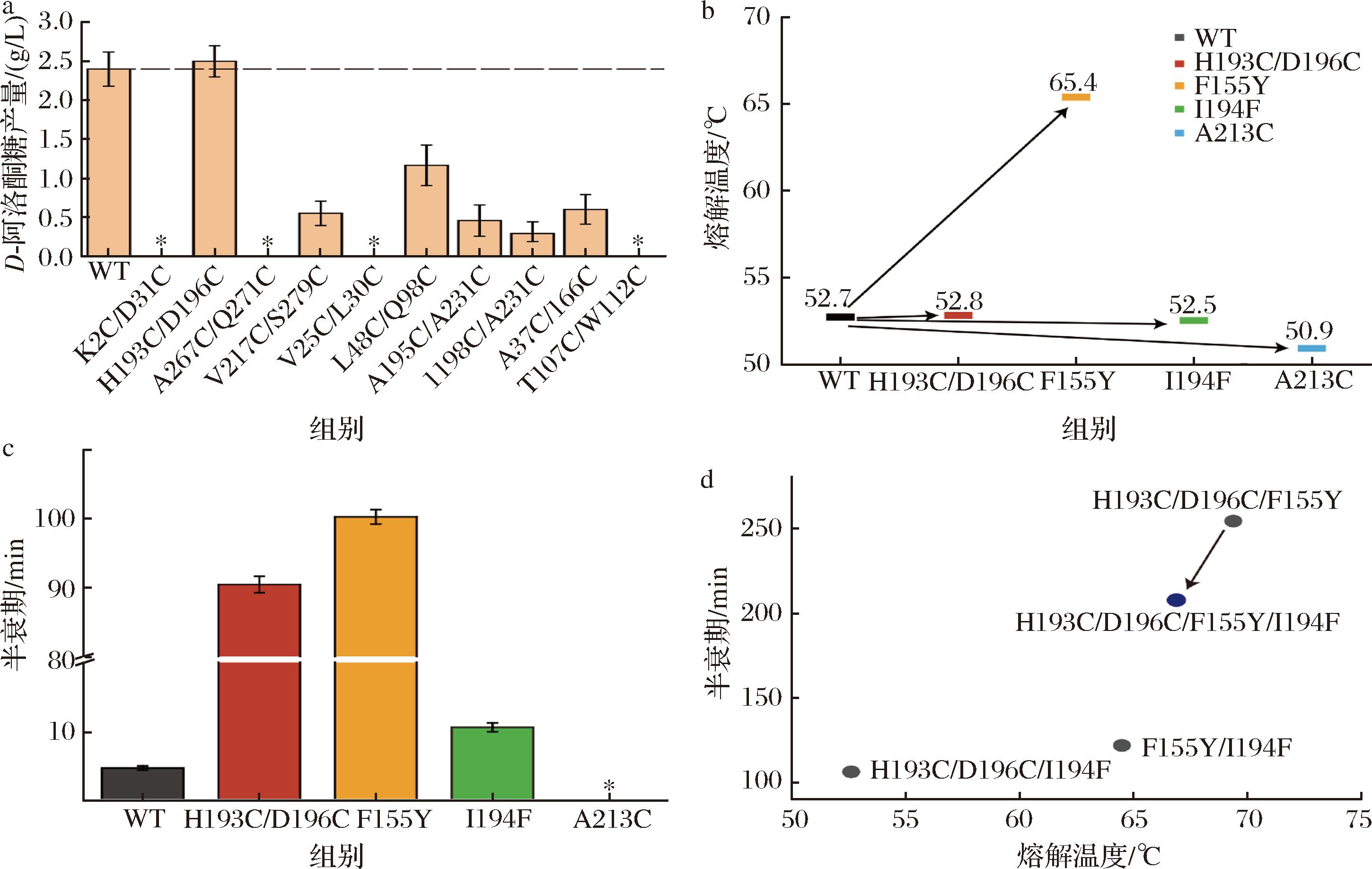
a-引入二硫键突变体初筛检测结果;b-突变体的熔解温度;c-突变体的半衰期;d-组合突变体的性能表征
图4 蛋白质工程改造
Fig.4 Protein engineering modification
注:*代表未检测到。
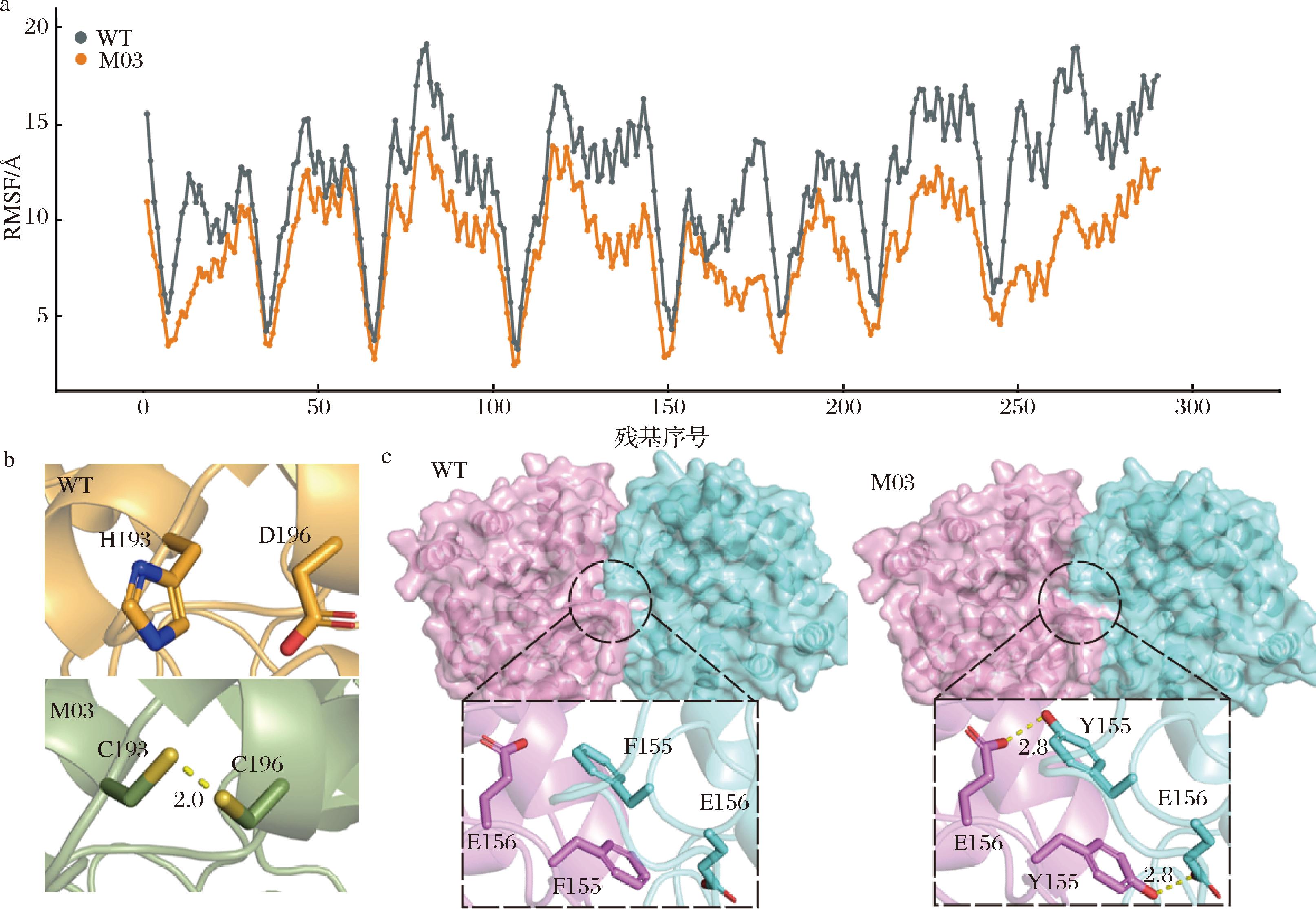
a-KeDt3e与KeDt3eM03的RMSF值;b-KeDt3eM03引入二硫键的相互作用分析;c-KeDt3eM03二聚体界面相互作用分析
图5 突变体KeDt3eM03热稳定性分析
Fig.5 Thermostability analysis of the optimal mutant KeDt3eM03
另外,通过序列比对以及结构比对将文献报道潜在的突变位点进行嫁接验证,构建突变文库F155Y[11]、I194F[15]和A213C[22],并进行性能评估。获得Tm值分别为65.4、52.5、50.9 ℃(图4-b);在55 ℃条件下,F155Y的半衰期为100.2 min,是野生型(4.7 min)的21.3倍,I194F的半衰期为10.6 min,比野生型(4.7 min)高5.9 min(图4-c),A213C的半衰期无法拟合。因此在属性嫁接过程中本研究获得了2个有益突变体F155Y和I194F。
最后,将3个有益突变体进行随机组合进行第一轮组合突变,构建突变文库H193C/D196C/F155Y、H193C/D196C/I194F以及F155Y/I194F。经性能表征获得最优组合M03(H193C/D196C/F155Y),Tm值为69.4 ℃,半衰期为254.3 min(图4-d)。接着以M03为模板与I194F进行迭代组合,获得组合突变体M03/I194F,Tm值(66.9 ℃)低于M03,半衰期(207.6 min)也未明显优于M03,于是放弃I194F的迭代,获得最终突变体M03。
2.3 KeDt3eM03热稳定性提升机制解析
为了阐明最优突变体M03热稳定性提高的原因,以野生型KeDt3e(PDB ID:8XIV)的结构为模板通过同源建模获得了最优突变体M03的结构模型。首先,对构建的M03结构模型进行了MD模拟,研究蛋白质构象的动态变化,比较了野生型KeDt3e与M03的蛋白骨架非氢原子的均方根偏差(root mean square fluctuation, RMSF)。如图5-a所示,突变体M03 loop环区域的RMSF值均显著降低,其整体结构更稳定,原因可能是增强蛋白表面的刚性会影响蛋白折叠,而loop环的稳定是提高蛋白热稳定性的关键因素之一。其中引入二硫键H193C/D196C突变位点位于蛋白表面,位于loop环上的H193C与α-螺旋上的D196C形成稳定的二硫键(图5-b),增强了蛋白表面的刚性,提高了H193所在loop环的稳定性。属性嫁接的F155Y位于界面处,通过SWISS-MODEL同源建模获得了最佳突变体M03和野生型KeDt3e二聚体结构(图5-c)可知,F155Y位于A和B两个单体界面的中心位置,侧链上的羟基与另一条链的E156的侧链形成氢键,通过优化界面的稳定性增强蛋白的稳定性,这与报道的机理一致。
3 结论
本文通过理性设计,采用引入二硫键和属性嫁接策略进行蛋白质工程,获得了热稳定性提高的最佳突变体M03,最佳突变体M03的Tm值从52.7 ℃升高到69.4 ℃;在不需要外源添加金属离子条件下,55 ℃时半衰期延长至254.3 min,显著提高54.1倍。接着分析了性能改善的详细机理,通过改善蛋白表面的稳定性实现提高整个蛋白的刚性,使得蛋白的热稳定性显著提高。本研究获得了一个热稳定性提高的D-塔格糖3-差向异构酶,为实现D-阿洛酮糖的工业化生产提供了实验基础;此外,多种修饰策略的整合同时提高了KeDt3e的热稳定性,在其他酶的蛋白质工程中显示出潜在的应用前景。
[1] FUKUNAGA K, YOSHIMURA T, IMACHI H, et al.A pilot study on the efficacy of a diabetic diet containing the rare sugar D-allulose in patients with type 2 diabetes mellitus:A prospective, randomized, single-blind, crossover study[J].Nutrients, 2023, 15(12):2802.
[2] PONGKAN W, JINAWONG K, PRATCHAYASAKUL W, et al.D-allulose provides cardioprotective effect by attenuating cardiac mitochondrial dysfunction in obesity-induced insulin-resistant rats[J].European Journal of Nutrition, 2021, 60(4):2047-2061.
[3] MATSUO T, TANAKA T, HASHIGUCHI M,et al.Metabolic effects of D-psicose in rats:Studies on faecal and urinary excretion and caecal fermentation[J].Asia Pacific Journal of Clinical Nutrition, 2003, 12(2):225-231.
[4] ZHANG X J, LI A, WANG Y, et al.D-Psicose intake exacerbates dextran sulfate sodium-induced colitis in mice through alteration in the gut microbiota and dysfunction of mucosal barrier[J].Food Science and Human Wellness, 2024, 13(1):173-182.
[5] ANDREANA P R, MCLELLAN J S, CHEN Y C, et al.Synthesis of 2,6-dideoxysugars via ring-closing olefinic metathesis[J].Organic Letters, 2002, 4(22):3875-3878.
[6] NORTHRUP A B, MACMILLAN D W C.Two-step synthesis of carbohydrates by selective aldol reactions[J].Science, 2004, 305(5691):1752-1755.
[7] DONER L W.Isomerization of D-fructose by base:Liquid-chromatographic evaluation and the isolation of D-psicose[J].Carbohydrate Research, 1979, 70 (2):209-216.
[8] IZUMORI K.Izumoring:A strategy for bioproduction of all hexoses[J].Journal of Biotechnology, 2006, 124(4):717-722.
[9] WANG H Y, CHEN J, ZHAO J Y, et al.Improved thermostability of D-allulose 3-epimerase from Clostridium bolteae ATCC BAA-613 by proline residue substitution[J].Protein Expression and Purification, 2022, 199:106145.
[10] ZHU Z L, LI L, ZHANG W, et al.Improving the enzyme property of D-allulose 3-epimerase from a thermophilic organism of Halanaerobium congolense through rational design[J].Enzyme and Microbial Technology, 2021, 149:109850.
[11] CHEN J J, CHEN D, CHEN Q M, et al.Computer-aided targeted mutagenesis of Thermoclostridium caenicola D-allulose 3-epimerase for improved thermostability[J].Journal of Agricultural and Food Chemistry, 2022, 70(6):1943-1951.
[12] BOSSHART A, PANKE S, BECHTOLD M.Systematic optimization of interface interactions increases the thermostability of a multimeric enzyme[J].Angewandte Chemie International Edition, 2013, 52(37):9673-9676.
[13] ZHANG W L, ZHANG Y M, HUANG J W, et al.Thermostability improvement of the D-allulose 3-epimerase from Dorea sp.CAG317 by site-directed mutagenesis at the interface regions[J].Journal of Agricultural and Food Chemistry, 2018, 66(22):5593-5601.
[14] ZHU Z L, GAO D K, LI C, et al.Redesign of a novel D-allulose 3-epimerase from Staphylococcus aureus for thermostability and efficient biocatalytic production of D-allulose[J].Microbial Cell Factories, 2019, 18(1):59.
[15] MAO S H, CHENG X T, ZHU Z L, et al.Engineering a thermostable version of D-allulose 3-epimerase from Rhodopirellula baltica via site-directed mutagenesis based on B-factors analysis[J].Enzyme and Microbial Technology, 2020, 132:109441.
[16] CASE D A, BETZ R M, CERUTTI D S, et al.Amber 16[M].San Francisco:University of California, 2016.
[17] JAKALIAN A, BUSH B L, JACK D B, et al. Fast, efficient generation of high-quality atomic charges. AM1-BCC model: I. Method[J]. Journal of Computational Chemistry, 2000, 21(2): 132-146.
[18] JAKALIAN A, JACK D B, BAYLY C I.Fast, efficient generation of high-quality atomic charges.AM1-BCC model:Ⅱ.Parameterization and validation[J].Journal of Computational Chemistry, 2002, 23(16):1623-1641.
[19] WANG J M, WOLF R M, CALDWELL J W, et al.Development and testing of a general amber force field[J].Journal of Computational Chemistry 2004, 25(9):1157-1174.
[20] JORGENSEN W L, CHANDRASEKHAR J, MADURA J D, et al.Comparison of simple potential functions for simulating liquid water[J].The Journal of Chemical Physics, 1983, 79(2):926-935.
[21] DARDEN T, YORK D, PEDERSEN L.Particle mesh Ewald:An N.log(N) method for Ewald sums in large systems[J].The Journal of Chemical Physics, 1993, 98(12):10089-10092.
[22] TSENG W C, HSU C T, CHANG H C, et al.Fusion of the peptide derived from the acidic tail of alpha-synuclein improves the thermostability and soluble expression of recombinant Agrobacterium sp.D-allulose 3-epimerase[J].Biochemical Engineering Journal, 2021, 165:107828.