母乳寡糖(human milk oligosaccharides, HMOs)是人类母乳中第3大固体成分,仅次于乳糖和脂肪[1],研究表明,HMOs可以作为可溶性受体类似物,在婴儿感染初期阻止病原体与上皮细胞相互作用,从而保护母乳喂养的婴儿免受原生动物、病毒、病原体和真菌的伤害[2-3]。母乳中的HMOs有200多种[4],其中,乳糖-N-二糖(lacto-N-biose,LNB)是一种天然存在于人乳中的HMOs,具有出色的生物活性,作为益生元促进双歧杆菌的增殖[5]。此外,HMOs糖链的核心结构由1-3-连接的乳糖-N-二糖(Galß1-3GlcNAc,LNB,1型链)或1-4-连接的N-乙酰乳糖胺(Galß1-4GlcNAc,LacNAc,2型链)组成,并被岩藻糖残基和唾液酸残基分别通过α1-2/3/4和α2-3/6键修饰,从而形成一系列复杂的HMOs[6]。值得一提的是HMOs中丰富的1型链是HMOs特有的[7],其中1型HMOs:乳糖-N-四糖(lacto-N-tetraose,LNT)和乳糖-N-新四糖(lacto-N-neotetraose,LNnT)是HMOs中含量最丰富、最常见的核心结构[8]。因此,实现LNB的合成对探索HMOs糖链的功能具有重要的意义。
LNB及其衍生物可以通过化学方法合成[9-10],但是基团保护和去保护涉及复杂步骤以及使用有毒试剂使其不符合食品要求。已有研究发现,双歧杆菌具有独特的代谢途径,可以有效地代谢HMOs的糖链单元LNB[11]。LNB通过LNB转运蛋白从细胞外进入细胞质,然后被乳糖-N-二糖磷酸化酶(lacto-N-biose phosphorylase,LnpA)磷酸化,生成半乳糖-1-磷酸(galactose 1-phosphate,Gal-1-P)和各自的N-乙酰己糖胺。最后,Gal-1-P和N-乙酰己糖胺通过“Leloir”途径进入糖酵解和氨基糖代谢循环,该途径由N-乙酰己糖-1-激酶(N-acetylhexosamine 1-kinase,NahK)、UDP-葡萄糖-己糖-1-磷酸尿苷基转移酶(galactose-1-phosphate uridylyltransferase,GalT)和UDP-葡萄糖-4-差向异构酶(UDP-galactose-4′-epimerase,GalE)组成[12-13]。利用该代谢机制,NISHIMOTO等[14]以蔗糖和GlcNAc为底物,通过一锅多酶法合成了LNB,但该方法需要提供昂贵的供体底物二磷酸尿苷葡萄糖(uridine diphosphate glucose,UDP-Glc)以及存在酶提取工艺,会增加生产成本,不适用于大规模经济制备。DU等[8]以半乳糖和GlcNAc为底物,采用ATP再生系统,提高了LNB的转化率,但是半乳糖价格相对较高,并且为了激活ATP再生,需要更多的丙酮酸氧化酶(pyruvate oxidase,PyoD),对于规模化生产,会导致一些额外的成本,大大限制了规模制备及市场化推广。
全细胞法避开了化学法和酶法的缺陷[15],由于不需要酶提取和纯化步骤,已成为一种廉价、高效、可达到食品级的LNB生产方法。LI等[16]通过改造乳酸克鲁维酵母实现了以乳糖为底物的LNB体内合成,乳糖中的葡萄糖单元经过多步催化转化为GlcNAc,半乳糖单元通过消耗ATP转化为Gal-1-P,在LNB磷酸化酶(lacto-N-biose phosphorylase,LnbP)催化下实现了LNB的合成,由于该方法需要提供ATP,而乳酸克鲁维酵母不具备强大的ATP再生系统,产量达到10.41 g/L。因此,实现ATP高效再生是进一步提高LNB产量的关键。
为解决ATP高效再生的问题,本研究提出不依赖ATP的系统来合成LNB,该系统以蔗糖磷酸化酶和LNB磷酸化酶为核心(图1)。但是需要充足的UDP-Glc作为起始“发动催化”物质,这就要求需要提供一定的UTP来实现UDP-Glc的合成。研究发现,酵母有强大的UTP再生系统[17],能用本身存在的酶系统高效地将廉价的UMP转化为UTP[18],由于酵母的来源广泛容易获得、价格低廉,适合工业生产,重要的是酵母自身酶系不代谢LNB。因此,本研究提出了工程菌与酵母利用“LNB磷酸化酶”合成途径耦合发酵,来实现LNB的经济、高效合成策略:(1)酵母利用自身酶系实现UMP到UTP的转化;(2)合成的UTP进入工程菌株E.coli JM109(DE3)(pRSFDuet1-SPase-GalT1和pETDuet1-GalE-BiGalHexNAcP),工程菌将蔗糖转化为UDP-Glc;(3)产生的UDP-Glc进入Gal-1-P循环利用程序,不再需要新的UTP:UDP-Gal通过与Glc-1-P交换实现了Gal-1-P和UDP-Glc的循环再生,整个过程工程菌株不需要直接消耗ATP就可以生成。该思路从设计上避免了以往全细胞合成LNB由于需要能荷高效再生系统而难于大量合成LNB的缺陷。LNB最高产量为25.32 g/L。
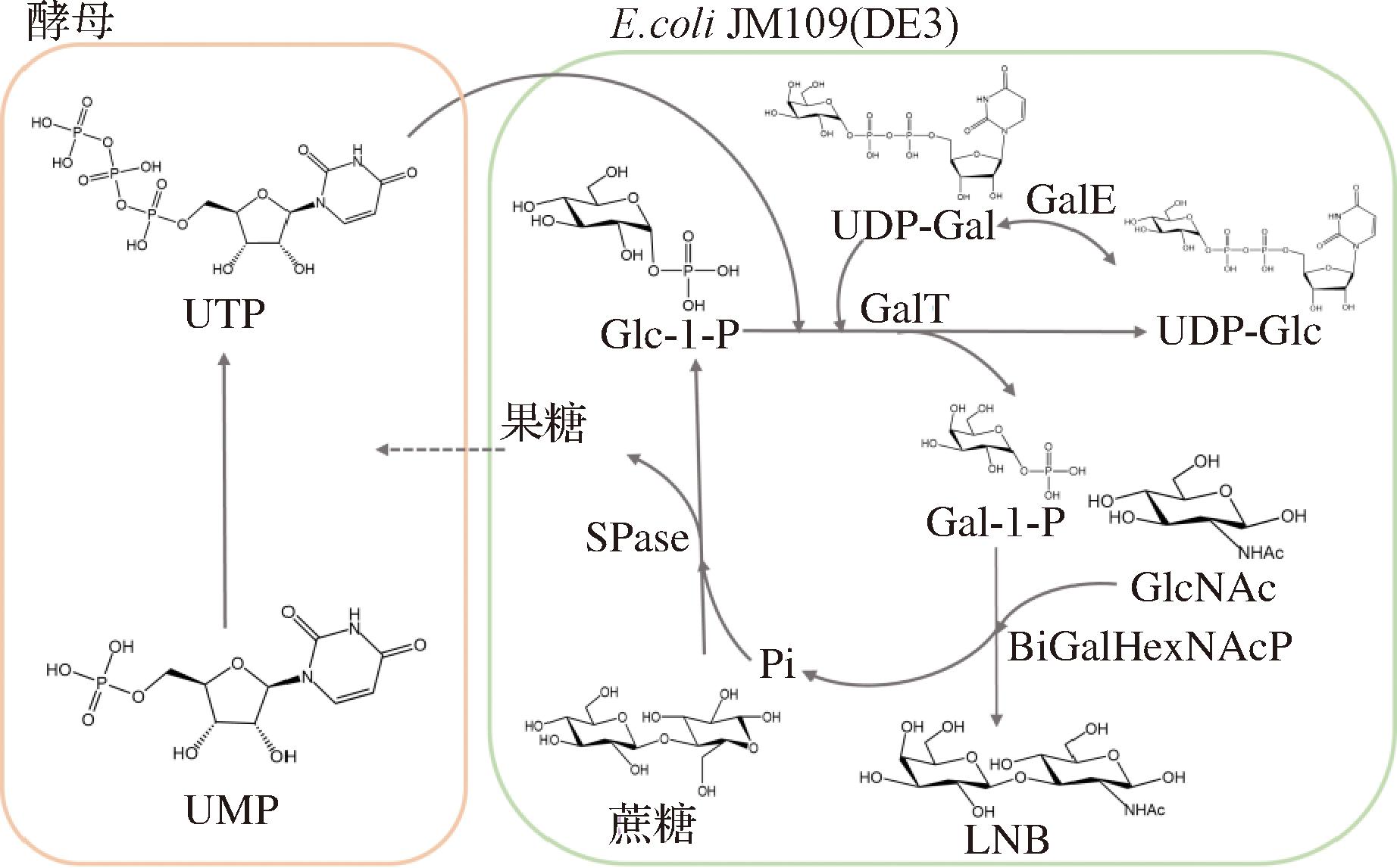
图1 双菌耦合发酵合成乳糖-N-二糖路线
Fig.1 Route for the synthesis of LNB by coupled fermentation of two strains
1 材料与方法
1.1 实验材料
1.1.1 质粒与菌株
本实验所用的质粒和菌株见表1。
表1 本文使用的质粒和菌株
Table 1 Plasmids and strains used in this study
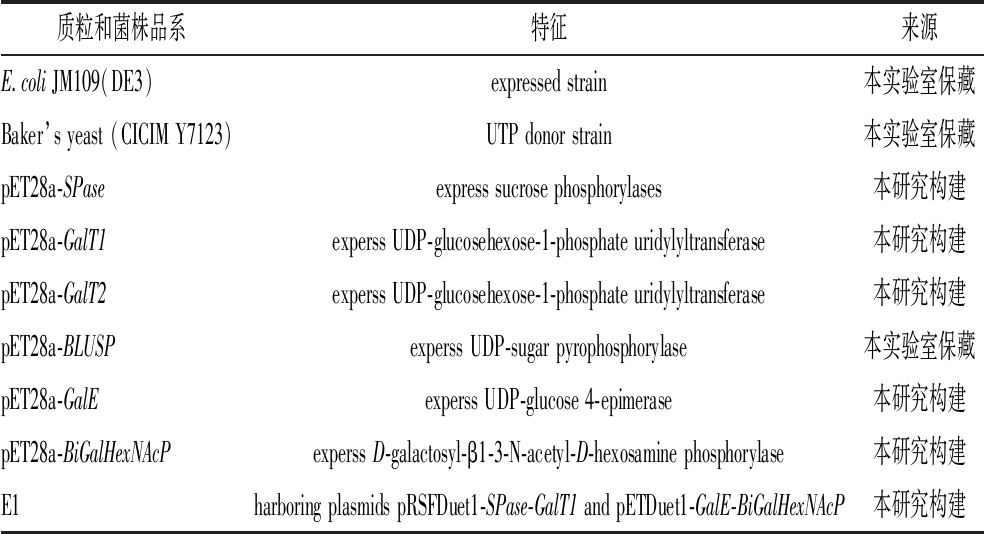
质粒和菌株品系特征来源E.coli JM109(DE3)expressed strain本实验室保藏Baker’s yeast (CICIM Y7123)UTP donor strain本实验室保藏pET28a-SPaseexpress sucrose phosphorylases本研究构建pET28a-GalT1experss UDP-glucosehexose-1-phosphate uridylyltransferase本研究构建pET28a-GalT2experss UDP-glucosehexose-1-phosphate uridylyltransferase本研究构建pET28a-BLUSPexperss UDP-sugar pyrophosphorylase本实验室保藏pET28a-GalEexperss UDP-glucose 4-epimerase本研究构建pET28a-BiGalHexNAcPexperss D-galactosyl-β1-3-N-acetyl-D-hexosamine phosphorylase本研究构建E1harboring plasmids pRSFDuet1-SPase-GalT1 and pETDuet1-GalE-BiGalHexNAcP本研究构建
1.1.2 实验试剂
LNB,荣成市慧海创达生物科技有限公司;蔗糖、GlcNAc,国药集团化学试剂有限公司;UMP,杭州铠朋生物技术有限公司;分子生物学操作试剂,Vazyme公司;其他为国产分析纯试剂。
1.1.3 培养基和培养条件
LB液体培养基(g/L):5酵母提取物、10胰酪蛋白胨和10 NaCl。LB固体培养基:在LB液体培养基中加入20 g/L琼脂粉。灭菌条件均为121 ℃、20 min。
YEPD培养基(g/L):10酵母提取物、20胰酪蛋白胨,20葡萄糖,pH 5.5,灭菌条件115 ℃、20 min。培养条件:将酵母细胞划线平板,挑取平板上的单菌落至100 mL液体培养基,150 r/min、30 ℃摇瓶培养24 h,然后离心(5 min、8 000 r/min),倒掉上清液收集菌体。
1.2 仪器与设备
PCR仪,美国Bio-Rad公司;蛋白电泳仪,上海达燊实业有限公司;高速冷冻离心机,美国Sigma公司;高效液相色谱仪,安捷伦科技公司。
1.3 实验方法
1.3.1 工程菌株的构建
合成肠膜明串珠菌ATCC12291(Leuconostoc mesenteroides ATCC12291)中SPase基因(GenBank ID:D90314.1)序列,长双歧杆菌JCM1217(Bifidobacterium longum subsp.longum JCM1217)中GalT1基因(GenBank ID:WP_007052335.1)序列、产气荚膜梭菌(Clostridium perfringens)中GalT2基因(GenBank ID:WP_011010402.1)序列、长双歧杆菌JCM1217(Bifidobacterium longum subsp.longum JCM1217)中GalE基因(GenBank ID:69578836)序列和婴儿长双歧杆菌(Bifidobacterium longum subsp.Infantis)中BiGalHexNAcP基因(GenBank ID:VUX39206.1)序列,分别以合成序列为模板,用设计的引物进行PCR扩增(表2),扩增后用胶回收试剂盒回收;同样以pET28a质粒为模板,设计有同源臂的引物扩增pET28a质粒,产物回收后通过同源重组的方式连接,将连接的重组质粒转化E.coli JM109(DE3)感受态细胞,然后涂布卡那霉素(Kan)平板,37 ℃过夜培养,挑取多个单菌落进行菌落PCR验证,将条带正确的单菌落送至天霖生物科技无锡有限公司测序,确定构建的重组质粒序列。共表达质粒的构建也采用同源重组的方式构建,从而获得一系列工程菌株。
表2 本文所用引物
Table 2 Primers used in this study

质粒序列(5’-3’)SPase-FGGAGATATACCATGGAAATTCAAAACAAAGCAATGTTGATCACTTATGCSPase-RCTGTTATGAGCAGTGATAATTTGACTCAGAACTAACATATGGCTAGCATGGalT1-FAGATATACCATGGGCCGATCAGCTGACCGAGGTATACGGalT1-RGGCTTCGCTGCGCGTTAAAAGCTTGCGGCCGCAGalT2-FGTGGTGGTGCTCGAGTTAATCGGCGATATCGATAGAGCCCAGalT2-RGGTGTAGTTTGCGAAATCCGCCATATGGCTGCCGCGGalE-FTGGTGGTGCTCGAGTTATTCCGCGTCACGGAAACCGalE-RCACCACCCGTAACCAGAACAGGCGGCCGCAAGCTTGBiGalHexNAcP-FCCGCGCGGCAGCCATATGACCAACACCGGCCGBiGalHexNAcP-RGCCTGGCGTGAAGCCTAACTCGAGCACCACCASPase-1-FactttaataaggagaTATACCATGGAAATTCAAAACAAAGCAATGTTGATCSPase-1-RTGCGGCCGCAAGCTTTTAGTTCTGAGTCAAATTATCACTGCTCATAACAGGalT1-2-FAGTATAAGAAGGAGATATACCATGGGCCGATCAGCGalT1-2-RGAGATCTGCCATATGTTAACGCGCAGCGAAGCCpRSFDuet1-M-FCATCACCATCATCACCACAGCCpRSFDuet1-M-RCTCCTTCTTATACTTAACTAATATACTAAGATGGGGAATTGpRSFDuet1-FCTAGCATAACCCCTTGGGGCpRSFDuet1-RTCTCCTTCTTAAAGTTAAACAAAATTATTTCTAGAGGGGGalE-1-FACTTTAAGAAGGAGATATACCATGGGCAGCAGCCATCGalE-1-RGTGATGATGGTGATGCTCGAGTTATTCCGCGTCACGBiGalHexNAcP-2-FTTAAGTATAAGAAGGAGTATCATATGACCAACACCGGCCGBiGalHexNAcP-2-RCTCGAGTGCGGCCGCTTAGGCTTCACGCCAGGCGATApETDuet1-M-FCATCACCATCATCACCACAGCCpETDuet1-M-RCTCCTTCTTATACTTAACTAATATACTAAGATGGGGAATTGpETDuet1-FCTAGCATAACCCCTTGGGGCpETDuet1-RTCTCCTTCTTAAAGTTAAACAAAATTATTTCTAGAGGGG
1.3.2 重组蛋白的诱导表达和鉴定
单基因工程菌株划线平板,放置37 ℃培养箱过夜培养,挑取单菌落至10 mL LB液体培养基(加入终质量浓度为50 μg/mL的Kan)过夜培养(37 ℃、200 r/min),然后以2%的接种量将种子液转接到100 mL LB(50 μg/mL Kan)培养基,继续在37 ℃、200 r/min下培养2~3 h,当细胞浓度(OD600值)达到0.6~0.8时,加入异丙基-β-D-1-硫代半乳糖苷(IPTG,终浓度为0.3 mmol/L),接着15 ℃、200 r/min条件培养20 h。培养结束后,离心(4 ℃、8 000 r/min、5 min)收集菌体。沸水浴10 min处理少量菌体与蛋白上样缓冲液的混合物,用SDS-PAGE检测蛋白表达。将诱导表达成功的菌体用于发酵耦合。诱导培养共表达菌株的条件与上述一样,除需要加入2种抗生素:Kan和氨苄西林(Amp,终质量浓度为100 μg/mL)。
1.3.3 乳糖-N-二糖的合成
4菌耦合合成LNB的条件:以300 mmol/L蔗糖、20 mmol/L UDP-Glc、50 mmol/L磷酸缓冲液(pH 7.0)、300 mmol/L GlcNAc、20 mL/L酒精、20 mmol/L MgCl2、20 mmol/L二硫苏糖醇(dithiothreitol,DTT)、8 g/L十八胺聚氧乙烯醚、100 g/L E.coli JM109(DE3)/pET28a-SPase、100 g/L E.coli JM109(DE3)/pET28a-GalT1、100 g/L E.coli JM109(DE3)/pET28a-GalE和100 g/L E.coli JM109(DE3)/pET28a-BiGalHexNAcP溶液中,30 ℃、200 r/min反应32 h。
共表达菌株与酵母耦合合成LNB:以300 mmol/L蔗糖、20 mmol/L UMP、50 mmol/L磷酸缓冲液(pH 7.0)、300 mmol/L GlcNAc、20 mmol/L MgCl2、20 mL/L酒精、20 mmol/L DTT、8 g/L十八胺聚氧乙烯醚、100 g/L E1和100 g/L面包酵母(湿重)溶液中,30 ℃、200 r/min反应32 h。
1.3.4 乳糖-N-二糖的分析
薄层层析(thin-layer chromatography, TLC)检测[19]:用正丁醇∶乙酸∶水(体积比)=2∶1∶1展开,茴香醛染色后165 ℃显色5 min。
高效液相色谱检测[8]:色谱柱Aminex HPX-87H Ion Exclusion Column (7.8 mm×300 mm),紫外检测波长210 nm,流动相5 mmol/L H2SO4,柱温60 ℃,流速0.6 mL/min,进样量10 μL。
电喷雾碰撞诱导串联质谱(electrospray ionization collision-induced dissociation tandem mass spectrometry,ESI-CID-MS/MS)分析:将纯化的样品冻干,使用300 μL超纯水将其溶解,采用诱导碰撞解离(collision induced dissociation,CID)解离模式,以氩气为碰撞气,碰撞能量为15~30 eV。
2 结果与分析
2.1 工程菌株蛋白的诱导表达
本研究将经过密码子优化后合成得到的目的基因SPase、GalT1、GalT2、GalE和BiGalHexNAcP克隆到质粒pET28a上,经测序验证后,将重组质粒pET28a-SPase、pET28a-GalT1、pET28a-GalT2、pET28a-GalE、pET28a-BiGalHexNAcP转入E.coli JM109(DE3)中得到工程菌E.coli JM109(DE3)/pET28a-SPase、E.coli JM109(DE3)/pET28a-GalT1、E.coli JM109(DE3)/pET28a-GalT2、E.coli JM109(DE3)/pET28a-GalE和E.coli JM109(DE3)/pET28a-BiGalHexNAcP。用IPTG诱导重组菌株中酶的表达,通过SDS-PAGE进行验证(图2),重组酶均得到良好的表达,与蛋白分子质量预测大小一致(SPase:56.2 kDa,GalT1:45 kDa,GalT2:45 kDa,BLUSP:59.7 kDa,GalE:39 kDa,BiGalHexNAcP:86.5 kDa),说明重组酶在构建的工程菌株中均成功表达。
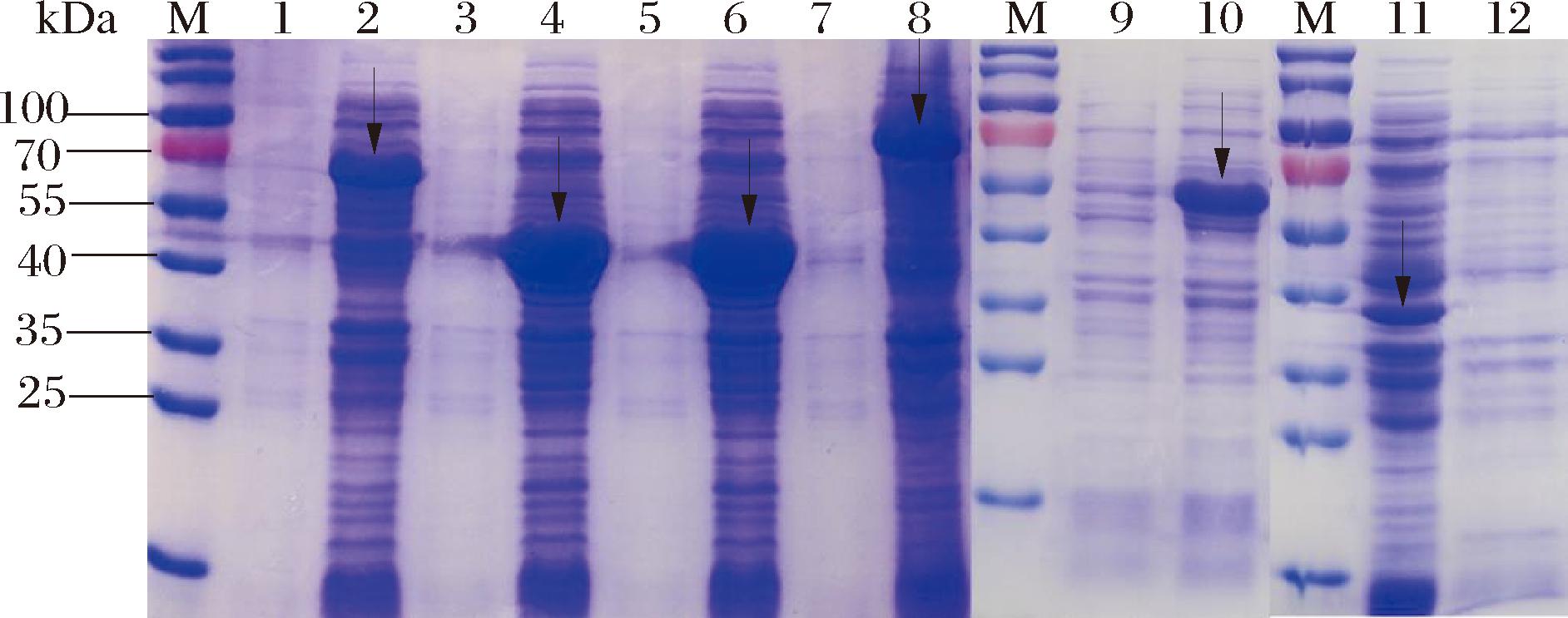
图2 重组酶的SDS-PAGE分析
Fig.2 SDS-PAGE analysis recombinant enzymes
注:M-蛋白质分子质量标准;1-BLUSP诱导前;2-BLUSP诱导后;3-GalT1诱导前;4-GalT1诱导后;5-GalT2诱导前;6-GalT2诱导后;7-BiGalHexNAcP诱导前;8-BiGalHexNAcP诱导后;9-SPase诱导前;10-SPase诱导后;11-GalE诱导后;12-GalE诱导前。
2.2 合成UDP-Glc酶的筛选
由图1可知,UDP-Glc的充足供给是实现UDP-Gal充足获得的关键,是整个合成途径的关键酶,因此对UDP-Glc合成通路的同工酶来自长双歧杆菌(GalT1)、产气荚膜梭菌(GalT2)的UDP-葡萄糖-己糖-1-磷酸尿苷转移酶和来自长双歧杆菌ATCC55813的UDP-糖焦磷酸化酶(UDP-sugar pyrophosphorylase,BLUSP),对三者所表达的酶在全细胞状态下合成UDP-Glc能力进行了筛选。考虑到温度对酶的活力影响最为关键,因此在相同细胞浓度和底物浓度下,比较3株工程菌在不同温度下合成UDP-Glc的能力(图3)。当反应温度为37 ℃时,GalT1合成UDP-Glc产量高于GalT2和BLUSP;在30 ℃条件下3株工程菌产生UDP-Glc的产量高于在37 ℃条件下的产量,而在30 ℃条件下表达GalT1酶的工程菌活性最高,产生UDP-Glc高达10.5 g/L。因此,选取菌株E.coli JM109(DE3)/pET28a-GalT1进行后续的双菌耦合发酵合成UDP-Glc,最佳温度选取30 ℃。
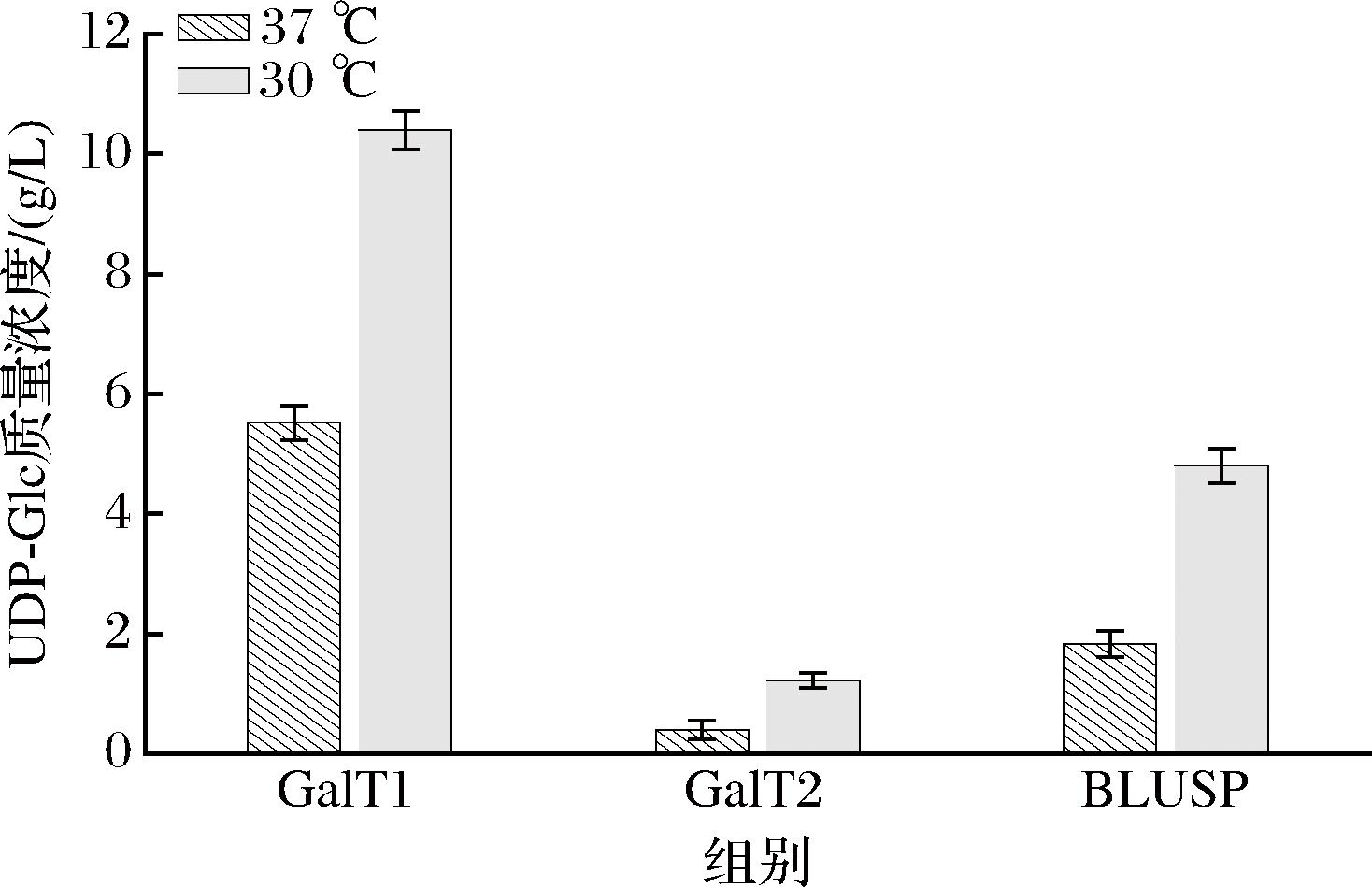
图3 不同工程菌株耦合发酵合成UDP-Glc
Fig.3 The synthesis of UDP-Glc by coupled fermentation of different engineered strains
2.3 多菌株耦合发酵合成LNB体系的建立
为实现LNB的合成,按照设计的路线,以蔗糖、GlcNAc和少量UDP-Glc为底物,通过4菌E.coli JM109(DE3)/pET28a-SPase、E.coli JM109(DE3)/pET28a-GalT1、E.coli JM109(DE3)/pET28a-GalE和E.coli JM109(DE3)/pET28a-BiGalHexNAcP耦合发酵合成目的产物LNB。在耦合发酵的32 h,对不同时间点的发酵液进行取样,通过TLC初步检测(图4-a),发酵液中存在与标品LNB相同比移值(retention factor value,Rf)的斑点,该结果初步表明生成的产物中含有LNB。为了进一步确定产物,采用刮板回收方法,将TLC板上与标品LNB的Rf位置相同的物质回收,并用ESI-CID-MS/MS进一步分析(图4-b),一级质谱图结果显示目的产物在m/z=382.127[M—H]-出现离子峰,与目标物质LNB的荷质比相一致,以上结果表明,以蔗糖、GlcNAc和UDP-Glc为底物,通过四菌耦合发酵成功合成了LNB。为获得发酵产量,用HPLC对发酵产物进行分析(图4-c),发酵液中存在与标品LNB出峰时间相同的物质,通过HPLC定量,当反应时间为20 h时,LNB产量最高,为6.78 g/L(图4-d)。
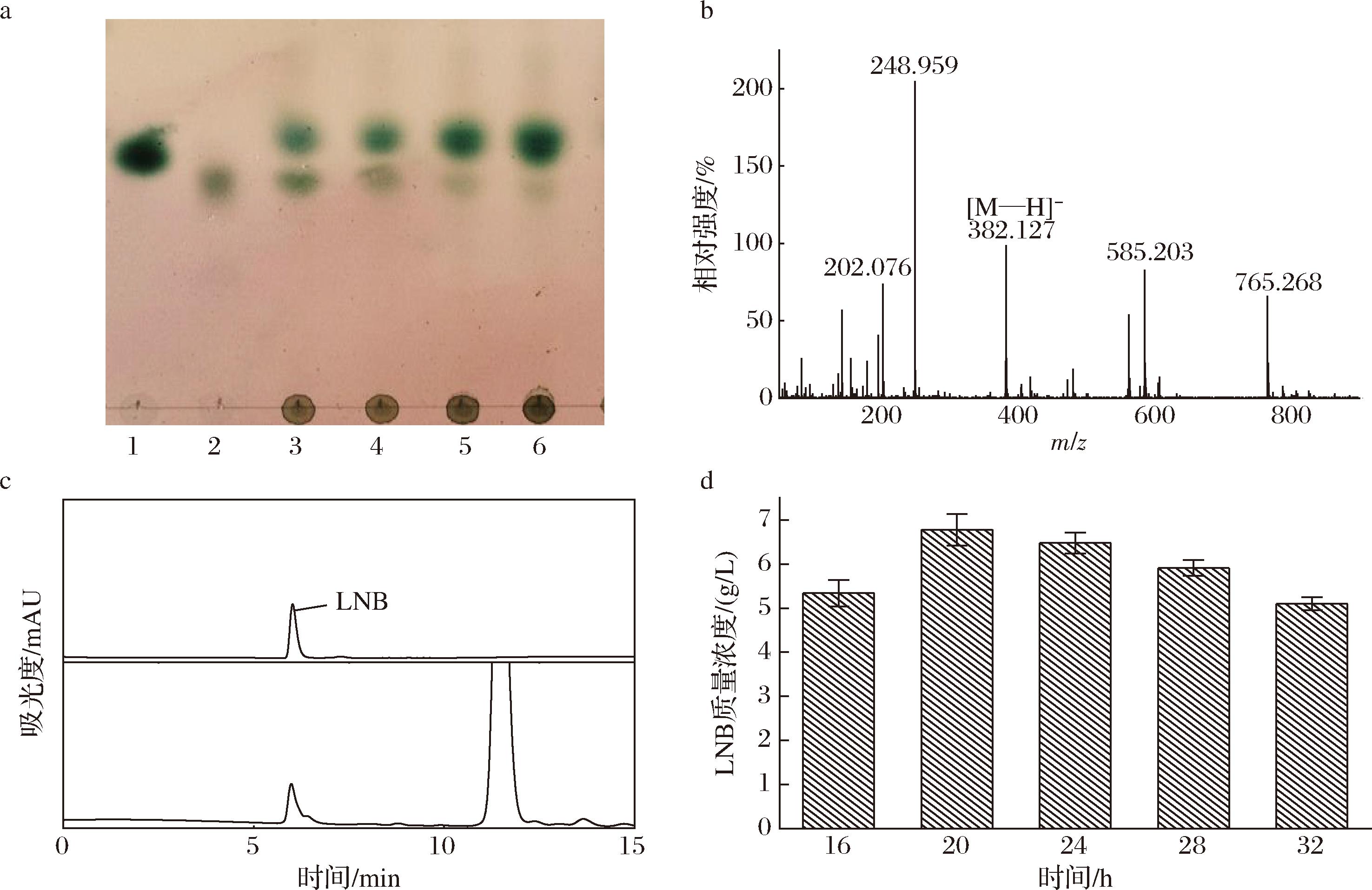
a-TLC图(1-蔗糖标品;2-LNB标品;3~6-20、24、28、32 h四菌耦合发酵产物);b-LNB的ESI-CID-MS/MS一级分析图谱;c-从上至下为LNB标品液相图、4菌耦合发酵产物液相图;d-LNB产量图
图4 四菌耦合发酵合成LNB的检测
Fig.4 Detection of LNB synthesized by coupled fermentation of four strains
2.4 共表达菌株与酵母耦合合成LNB
4菌耦合发酵时LNB产量仅为6.78 g/L,这可能是由于中间产物需要多次进出细胞进行跨膜运输会导致其利用率不断降低,以及4菌耦合发酵时菌浓过高会造成溶氧供给不足,从而导致酵母细胞的UTP再生抑制等问题。因此,采用单菌株全细胞合成LNB的策略来解决以上问题。考虑到酵母UTP再生系统可以将UMP转化为UTP,后者可以直接进入产物合成途径用于UDP-Glc的合成,于是利用酵母与共表达工程菌株E.coli JM109(DE3)(含有质粒pRSFDuet1-SPase-GalT1和pETDuet1-GalE-BiGalHexNAcP)(构建流程见图5-a)耦合发酵来实现LNB的合成。将细胞质量浓度均为100 g/L的共表达菌株和酵母进行双菌耦合发酵,从发酵时间20 h开始,每隔4 h取样,持续到32 h发酵停止。然后将不同时间段取样的发酵液利用HPLC分析,从20 h开始LNB产量逐渐增加,在发酵24 h时产量最高,达到25.32 g/L,随着时间的延长,产量开始降低,到32 h,产量降低到22.56 g/L。这应该是工程菌自身表达的酶酶解合成的LNB用于自身的需求。在最高产量为25.32 g/L时,N-乙酰氨基葡萄糖的摩尔转化率为22.02%(图5-b)。接着,优化耦合体系的菌体浓度(图5-c),菌体质量浓度为120 g/L,LNB产量达到最大,为28.66 g/L,转化率为24.93%。
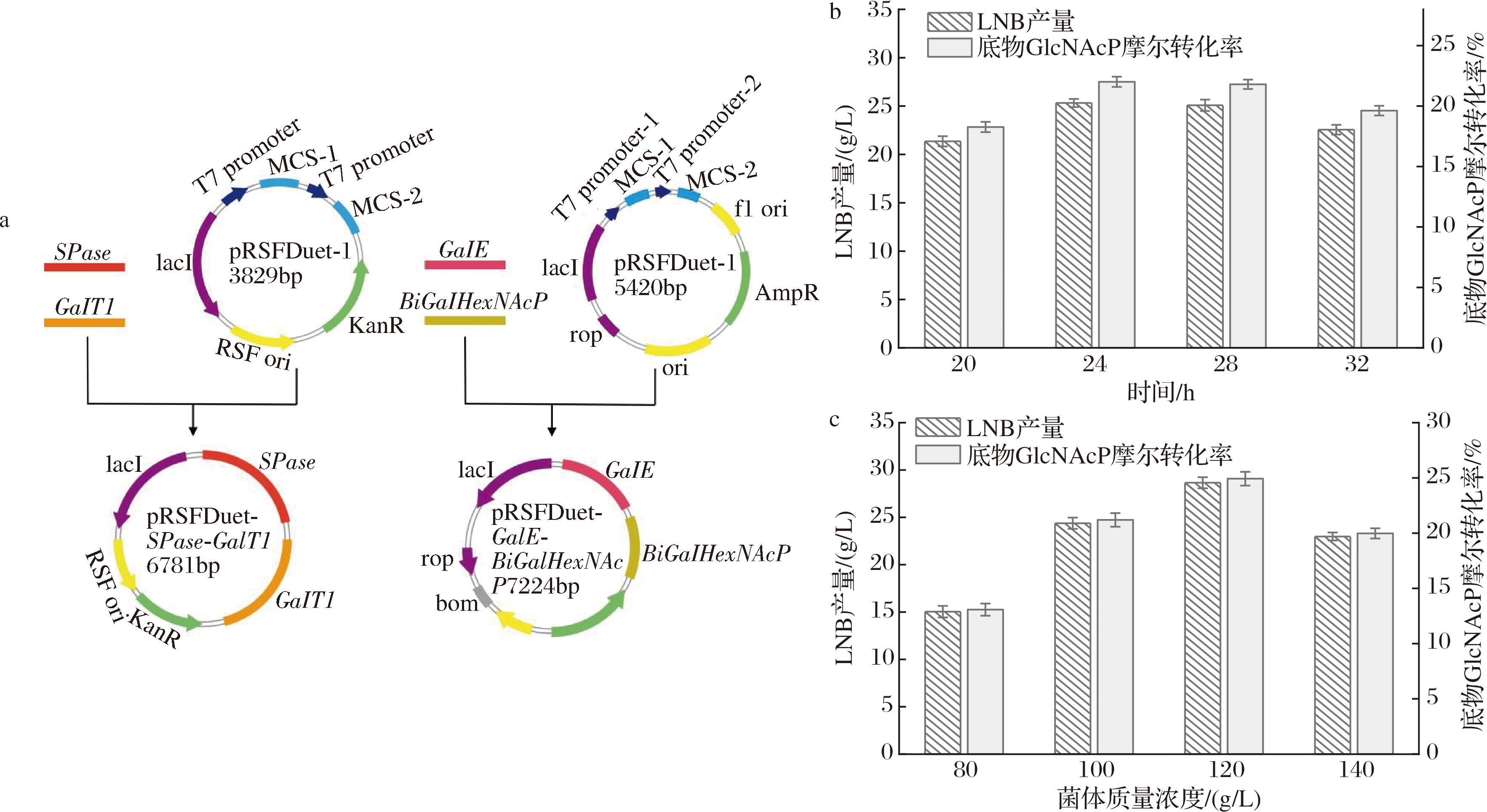
a-共表达质粒的构建流程;b-反应时间对工程菌和酵母耦合合成LNB的影响;c-菌体质量浓度对工程菌和酵母耦合合成LNB的影响
图5 共表达菌株与酵母耦合发酵合成LNB
Fig.5 Synthesis of LNB by coupled fermentation of co-expression strain and yeast
3 结论与讨论
以往合成LNB主要有体外和体内2种方法。报道的体外法合成LNB主要采用包括磷酸化酶在内的多酶耦合催化来实现[8, 14, 20],由于所用酶需要纯化步骤,增加了生产成本。随着合成生物学的发展,利用合成生物学技术实现LNB的发酵生产无疑是降低生产成本的最佳方法。实现细胞工厂内LNB的高效合成需要满足以下条件:a)底物可以进入细胞内;b)整个LNB合成系统非依赖ATP或UTP的再生。LI等[16]通过改造乳酸克鲁维酵母实现了以乳糖为底物的LNB体内合成,产量达到11.39 g/L,但该方法仍然没有解决“非依赖ATP或UTP的再生”问题。
在LNB合成中,最为关键的是Gal-1-P的供给问题,如何解决“非依赖ATP或UTP的再生”实现Gal-1-P的持续合成是关键。本研究提出了实现“非依赖ATP或UTP的再生”细胞工厂持续合成Gal-1-P的方法,该方法不需额外构建ATP再生系统,利用酵母自身的酶系,以少量外源UMP辅因子实现了UTP的供给,在SPase和GalT协同催化下实现了UDP-Glc的合成,然后GalT和GalE协同作用将Glc-1-P和UDP-Gal转化为Gal-1-P和UDP-Glc,实现了Gal-1-P的合成和UDP-Glc的非依赖ATP或UTP的重复再生,最后D-半乳糖基-β1-3-N-乙酰-D-己糖胺磷酸化酶(D-galactosyl-β1-3-N-acetyl-D-hexosamine phosphorylase,BiGalHexNAcP)以廉价GlcNAc为底物,生成了28.66 g/L LNB,这是目前已知的LNB最高产量报道。
但是,目前技术也存在以下2个问题:a)细胞内合成的LNB不能高效地分泌到细胞外;b)果糖没有充分利用,合成途径中每产生一分子LNB就伴随一个分子果糖产生,为实现果糖的充分利用,进一步实现以蔗糖为底物的LNB绿色生产。针对细胞外分泌困难问题,将从通道蛋白的引入着手,筛选合适的通道蛋白,实现LNB的高效外排;针对果糖利用问题,后续考虑引入己糖激酶、GlcN-6-P合成酶(GlcN-6-P synthase,GlmS)和GlcN-6-P乙酰化酶(glucosamine-6-phosphate N-acetyltransferase 1,Gna1),构建GlcNAc合成途径,实现GlcNAc在工程菌中的合成,为进一步降低LNB生产成本奠定基础。
[1] URASHIMA T, ASAKUMA S, LEO F, et al.The predominance of type I oligosaccharides is a feature specific to human breast milk[J].Advances in Nutrition, 2012, 3(3):473S-482S.
[2] ZHU Y Y, WAN L, LI W, et al.Recent advances on 2′-fucosyllactose:Physiological properties, applications, and production approaches[J].Critical Reviews in Food Science and Nutrition, 2022, 62(8):2083-2092.
[3] MOORE R E, XU L L, TOWNSEND S D.Prospecting human milk oligosaccharides as a defense against viral infections[J].ACS Infectious Diseases, 2021, 7(2):254-263.
[4] CHEN X.Chapter four human milk oligosaccharides (HMOS):Structure, function, and enzyme-catalyzed synthesis[J].Advances in Carbohydrate Chemistry and Biochemistry, 2015, 72:113-190.
[5] KIYOHARA M, TACHIZAWA A, NISHIMOTO M, et al.Prebiotic effect of lacto-N-biose I on bifidobacterial growth[J].Bioscience, Biotechnology, and Biochemistry, 2009, 73(5):1175-1179.
[6] BODE L.Human milk oligosaccharides:Every baby needs a sugar mama[J].Glycobiology, 2012, 22(9):1147-1162.
[7] SAKURAMA H, KIYOHARA M, JUN W D, et al.Lacto-N-biosidase encoded by a novel gene of Bifidobacterium longum subspecies longum shows unique substrate specificity and requires a designated chaperone for its active expression[J].Journal of Biological Chemistry, 2013, 288(35):25194-25206.
[8] DU Z Q, LIU Z Y, TAN Y S, et al.Lacto-N-biose synthesis via a modular enzymatic cascade with ATP regeneration[J].iScience, 2021, 24(3):102236.
[9] VETERE A, MILETICH M, BOSCO M, et al.Regiospecific glycosidase-assisted synthesis of lacto-N-biose I (Galbeta1-3GlcNAc) and 3′-sialyl-lacto-N-biose I (NeuAcalpha2-3Galbeta1-3GlcNAc)[J].European Journal of Biochemistry, 2000, 267(4):942-949.
[10] D’ALMEIDA A, IONATA M, TRAN V, et al.An expeditious and efficient synthesis of β-D-galactopyranosyl-(1→3)-D-N-acetylglucosamine (lacto-N-biose) using a glycosynthase from Thermus thermophilus as a catalyst[J].Tetrahedron:Asymmetry, 2009, 20(11):1243-1246.
[11] KITAOKA M.Bifidobacterial enzymes involved in the metabolism of human milk oligosaccharides[J].Advances in Nutrition, 2012, 3(3):422S-429S.
[12] JUN W D, ANDO T, KIYOHARA M, et al.Bifidobacterium bifidum lacto-N-biosidase, a critical enzyme for the degradation of human milk oligosaccharides with a type 1 structure[J].Applied and Environmental Microbiology, 2008, 74(13):3996-4004.
[13] NISHIMOTO M, KITAOKA M.Identification of N-acetylhexosamine 1-kinase in the complete lacto-N-biose I/galacto-N-biose metabolic pathway in Bifidobacterium longum[J].Applied and Environmental Microbiology, 2007, 73(20):6444-6449.
[14] NISHIMOTO M.Large scale production of lacto- N-biose I, a building block of type I human milk oligosaccharides, using sugar phosphorylases[J].Bioscience, Biotechnology, and Biochemistry, 2020, 84(1):17-24.
[15] HAN N S, KIM T J, PARK Y C, et al.Biotechnological production of human milk oligosaccharides[J].Biotechnology Advances, 2012, 30(6):1268-1278.
[16] LI F Y, MA W, LIU L L, et al.Reprogramming the metabolic network in Kluyveromyces lactis with a transcriptional switch for de novo lacto-N-biose synthesis[J].Journal of Agricultural and Food Chemistry, 2023, 71(23):9031-9039.
[17] 周文, 游星, 张洪涛, 等.多细胞耦合转化N-乙酰氨基葡萄糖和乳糖生产唾液酸乳糖[J].生物工程学报, 2023, 39(11):4621-4634.
ZHOU W, YOU X, ZHANG H T, et al.Multicellular coupling fermentation for 3′-sialyllactose conversion using N-acetyl-glucosamine and lactose[J].Chinese Journal of Biotechnology, 2023, 39(11):4621-4634.
[18] 陈德康. 酵母酶系合成三磷酸核苷的研究[D].南京:南京工业大学, 2004.
CHEN D K.Study on synthesis of nucleoside triphosphate by yeast enzyme system[D].Nanjing:Nanjing University of Technology, 2004.
[19] LEE Y C, CHIEN H R, HSU W H.Production of N-acetyl-d-neuraminic acid by recombinant whole cells expressing Anabaena sp.CH1 N-acetyl-D-glucosamine 2-epimerase and Escherichia coli N-acetyl- D-neuraminic acid lyase[J].Journal of Biotechnology, 2007, 129(3):453-460.
[20] CHEN L J, QIN Y M, MA L, et al.Biosynthesis of lacto-N-biose I from starch and N-acetylglucosamine via an in vitro synthetic enzymatic biosystem[J].Synthetic and Systems Biotechnology, 2023, 8(3):555-562.